UNIVERSIDADE FEDERAL DA PARAÍBA
UNIVERSIDADE FEDERAL RURAL DE PERNAMBUCO UNIVERSIDADE FEDERAL DO CEARÁ
PROGRAMA DE DOUTORADO INTEGRADO EM ZOOTECNIA
MASTITE SUBCLINICA CAPRINA: ETIOLOGIA E DIVERSIDADE MICROBIONA DO LEITE POR SEQUENCIAMENTO DO GENE 16S RNAr
ANDREIA BATISTA BEZERRA
AREIA/PB
DEZEMBRO/2016
i
UNIVERSIDADE FEDERAL DA PARAÍBA
UNIVERSIDADE FEDERAL RURAL DE PERNAMBUCO UNIVERSIDADE FEDERAL DO CEARÁ
PROGRAMA DE DOUTORADO INTEGRADO EM ZOOTECNIA
MASTITE SUBCLINICA CAPRINA: ETIOLOGIA E DIVERSIDADE MICROBIONA DO LEITE POR SEQUENCIAMENTO DO GENE 16S RNAr
ANDREIA BATISTA BEZERRA
Zootecnista
AREIA/PB
DEZEMBRO/2016
ii
ANDREIA BATISTA BEZERRA
MASTITE SUBCLINICA CAPRINA: ETIOLOGIA E DIVERSIDADE MICROBIONA DO LEITE POR SEQUENCIAMENTO DO GENE 16S RNAr
Tese apresentada ao Programa de Doutorado Integrado em Zootecnia da Universidade Federal da Paraíba, Universidade Federal Rural de Pernambuco e Universidade Federal do Ceará como requisito parcial para obtenção do título de Doutor em Zootecnia.
Área de Concentração: Produção Animal
Comitê de Orientação:
Prof. Dr. Celso José Bruno de Oliveira – Orientador principal Profa. Dra. Patrícia Emília Naves Givisiez
Prof. Dr. Edgard Cavalcanti Pimenta Filho
AREIA/PB
2016
iii Ficha Catalográfica Elaborada na Seção de Processos Técnicos da
Biblioteca Setorial do CCA, UFPB, campus II, Areia – PB.
B574m Bezerra, Andreia Batista.
Mastite subclínica caprina: etiologia e diversidade microbiona do leite por sequenciamento do gene 16s RNAr / Andreia Batista Bezerra. - Areia: UFPB/CCA, 2016.
xvi, 112 f. ; il.
Tese (Doutorado em Zootecnia) - Centro de Ciências Agrárias. Universidade Federal da Paraíba, Areia, 2017.
Bibliografia.
Orientador: Celso José Bruno de Oliveira.
1. Caprinos – Doenças 2. Mastite caprina – Etiologia 3. Leite de cabras – Microbiologia I. Oliveira, Celso José Bruno de (Orientador) II. Título.
UFPB/CCA CDU: 619:636.39(043.2)
iv
v DADOS CURRICULARES DO AUTOR
ANDREIA BATISTA BEZERRA – Filha de Maria Cleonice Batista Bezerra e Francisco de Assis Bezerra, nasceu no dia 06 de outubro de 1983 na cidade de Esperança, no Estado da Paraíba. Graduou-se em zootecnia pela Universidade Federal da Paraíba em agosto de 2010. No mesmo ano ingressou no Curso de Mestrado em Zootecnia da Universidade Federal da Paraíba, na área de concentração Produção Animal, sob orientação do Prof. Dr. Severino Gonzaga Neto, defendendo a dissertação em outubro de 2012 intitulada por:
―Medidas corporais, características da carcaça e constituintes não carcaça de caprinos Canindé submetidos à restrição alimentar‖. Em março de 2013 ingressou no Programa de Doutorado Integrado em Zootecnia na Universidade Federal da Paraíba, mediante processo seletivo interno, na área de concentração em Produção Animal e linha de pesquisa em segurança alimentar, sob orientação do Prof. Dr. Celso José Bruno de Oliveira, defendendo a Tese em dezembro de 2016.
vi
Não existem pessoas sem conhecimento. Elas não chegam vazias.
Chegam cheias de coisas. Na maioria dos casos, trazem juntas consigo opiniões sobre o mundo, sobre a vida.
Paulo Freire
Transformar o Mundo
Se, na verdade, não estou no mundo para simplesmente a ele me adaptar, mas para transformá-lo; se não é possível mudá-lo sem um certo sonho ou projeto de mundo, devo usar toda possibilidade que tenha para não apenas falar de minha utopia, mas participar de práticas com ela coerentes.
Paulo Freire
vii DEDICO
Aos meus pais, Maria Cleonice Batista Bezerra e Francisco de Assis Bezerra, pelo apoio, compreensão, amor e incentivo a estudar.
A Lavosier Éneas Cavalcante, pelo seu amor e companheirismo.
Aos meus irmãos (Adriana e Leandro) e sobrinhos (Gabriel, Maria Eduarda e Ana Clara), pelo amor que nos uni.
A minha avó, Maria Batista de Araújo (in memoriam), por ensinar a não desistir nos momentos difíceis.
Ao meu primo, Gilvanilton Elias Batista (in memoriam), por me ensinar a ser forte.
A vocês, minha sincera homenagem e eterna gratidão.
viii AGRADECIMENTOS
A Deus, por ter me dado força e coragem em todos os momentos da minha vida.
Aos meus queridos pais, Maria Cleonice Batista Bezerra e Francisco de Assis Bezerra, por todo o amor, educação e incentivo.
As minhas primas, Nathália, Elisângela, Elaine, Aline, Beatriz, Lais e Emily, Luciana e Lizandra pelos momentos de alegrias e pela amizade.
À meu companheiro Lavosier Éneas Cavalcante, pelo seu amor, companheirismo, paciência, apoio nos momentos difíceis e todo amor, amo muito.
Ao meu orientador, Celso José Bruno de Oliveira, pelos valiosos ensinamentos, contribuição e dedicação. Serei sempre grata.
Aos Professores, Dra. Patrícia Emília Naves Givisiez e Dr. Edgard Cavalcanti Pimenta Filho, pelos ensinamentos, dedicação e contribuição.
A banca examinadora, pelas sugestões e contribuição ao trabalho.
Ao Prof. Dr. Walter Esfrain Pereira, pela imprescindível contribuição nos ensinamentos e análises estatísticas, pela dedicação e disponibilidade.
Aos Professores Dra. Suzana Aparecida Costa de Araújo, Dr. Mateus Matiuzzi da Costa, Dr. Lauro Santos Filho, Dr. Paulo Sérgio de Azevedo pela participação no Exame de Qualificação e pelas valiosas contribuições para a melhoria deste trabalho.
Aos funcionários do PPGZ/CCA, Maria das Graças S. C. Medeiros, Carmem e Damião, pela atenção e ajuda que me foram prestadas.
À Daniely Sales e Candice Maria de Leon que me ajudaram bastante com seu apoio, força e alegrias que compartilhamos.
Aos produtores de leite do Cariri Paraibano, que abriram as porteiras das suas propriedades e nos forneceram o leite para concretização deste trabalho.
Ao pessoal que me ajudaram na colheita do leite e processamento das amostras, Magda Fernandes, Jessica Valente, Mauro Saraiva, Silvana Lima, Candice de Leon, Paulo Antônio, Fátima Andrade, Abimael Estevam, Adriano leite, Eudes Fernando, Adelson Santana e Yohana Corrêa.
A todos que direta e ou indiretamente contribuíram para a realização deste trabalho.
Muito obrigada!
ix SUMÁRIO
Página Lista de Tabelas ... X Lista de Figuras ... XIII Resumo Geral ... XV General abstract ... XVI
Considerações Iniciais ... 1
Capítulo I-
Mastite subclínica em cabras: diagnóstico, etiologia e genômica aplicada na determinação do perfil microbiano Introdução... 4Referencial teórico ... 6
Considerações finais... 22
Referências bibliográficas ... 23
Capítulo II-
Investigação longitudinal sobre a etiologia das infecções intramamárias subclínicas em cabras leiteiras usando espectrofotometria de massa MALDI- TOF... Resumo... 36Abstract ... 37
Introdução ... 38
Material e Métodos ... 40
Resultados e Discussão... 44
Conclusões ... 53
Referências Bibliográficas... 54
Capítulo III-
Efeito da infecção intramamária subclínica sobre a produção e qualidade do leite de cabras... Resumo... 61Abstract ... 62
Introdução ... 63
x
Material e Métodos ... 65
Resultados e Discussão... 70
Conclusões ... 76
Referências Bibliográficas ... 77
Capitulo IV- Caracterização da microbiota do leite de cabras com e sem mastite subclínica por sequenciamento do gene 16S RNAr Resumo... 84
Abstract ... 85
Introdução ... 86
Material e Métodos ... 88
Resultados e Discussão... 94
Conclusões ... 106
Referências Bibliográficas ... 107
Considerações finais ... 112
xi LISTA DE TABELAS
Capítulo II-
Investigação longitudinal sobre a etiologia das infecções intramamárias subclínicas em cabras leiteiras usando espectrofotometria de massa MALDI-TOFPágina
Tabela 1.
Ocorrência da positividade do California Mastitis Test (CMT) de glândulas mamárias em três diferentes fases lactacionais em cabras leiteiras: inícial (50 dias de lactação), intermediária (100dias) e final (150
dias)...
42
Tabela 2.
Ocorrência de infecção intramamária em três diferentes fases lactacionais de tetos de cabras leiteiras: inícial (50 dias de lactação), intermediária (100 dias) e final (150 dias) ...44
Tabela 3.
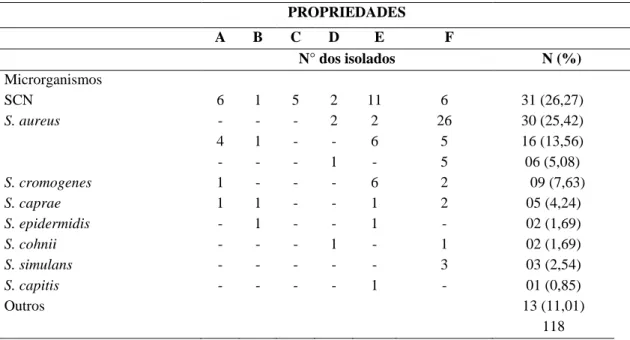
Frequência de microrganismos isolados de amostras de leite caprino identificados pelo MALDI-TOF MS ...46 Tabela 4.
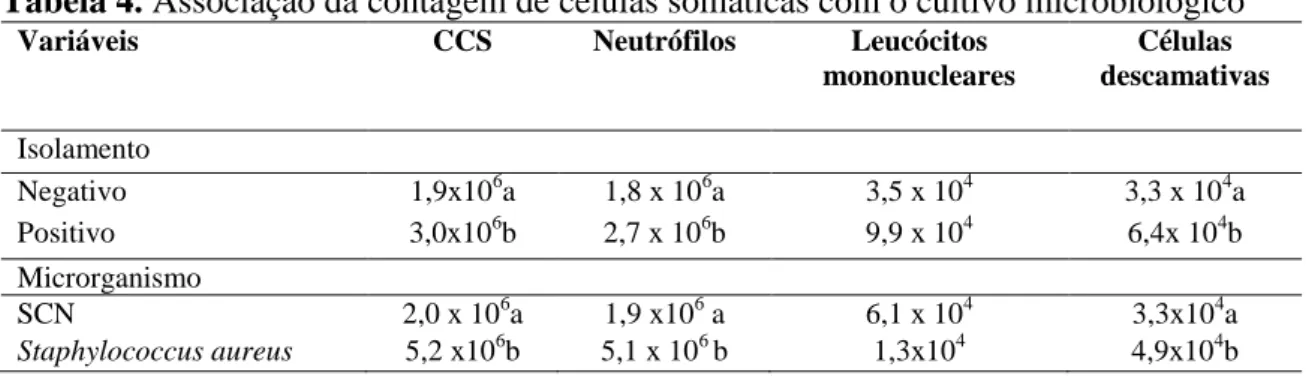
Associação da contagem de células somáticas com o cultivomicrobiológico...
48 Tabela 5.
Contagem de células somáticas e tipos celulares (CCS) deglândulas saudáveis em três diferentes fases lactacionais de cabras leiteiras: inícial (50 dias de lactação), intermediária (100 dias) e
final (150 dias)...
49 Tabela 6.
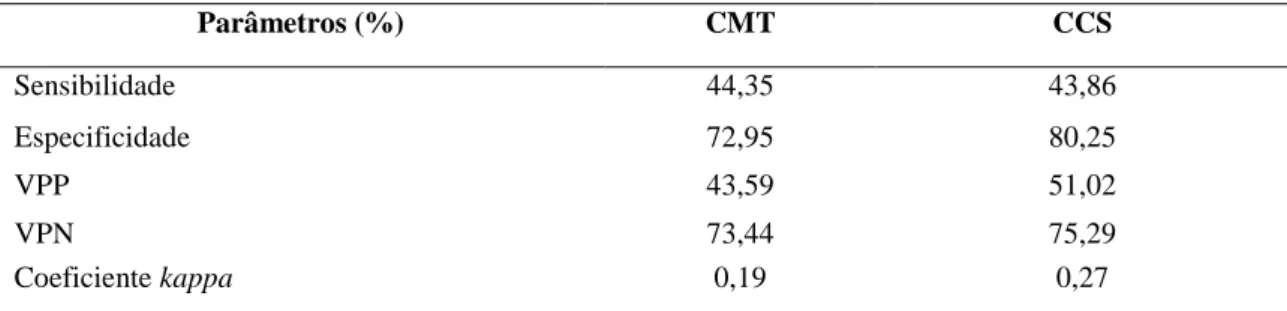
Sensibilidade, Especificidade, Valor Preditivo Positivo, ValorPreditivo Negativo e coeficiente kappa do California Mastitis Test (CMT) e contagem de células somáticas (CCS) com o cultivo
microbiológico...
49 Tabela 7.
Sensibilidade, Especificidade, Valor Preditivo Positivo, ValorPreditivo Negativo e coeficiente kappa do California Mastitis Test (CMT) e Contagem de células somáticas (CCS) com o grupos de
microrganismos...
50
xii LISTA DE TABELAS
Capítulo III Efeito da infecção intramamária subclínica sobre a produção e qualidade do leite de cabras
Página Tabela 1. Ocorrência da infecção intramamária em tetos de cabras das
raças ¾ Parda Alpina e Anglo Nubiana em diferentes períodos
de lactação
...
66 Tabela 2. Estimativas de perdas da produção de leite de cabras ¾ Parda
Alpina e Anglo Nubiana ocasionadas por infecção intramamária por CoNS, infecção por S.aureus e infecção mista por CoNS e S.
aureus através de um modelo linear misto
... 67 Tabela 3. Efeito da infecção intramamária e do agente etiológico sobre a
composição físico-química, CCS e CBT do leite de tetos mamários de cabras através de um modelo linear misto...
68 Tabela 4. Efeito de diferentes classes de CCS sobre a composição físico-
química e contagem de mesófilos aeróbios no leite de tetos mamários de cabras através de um modelo linear misto...
70
xiii LISTA DE TABELAS
Capítulo IV Caracterização da microbiota do leite de cabras com e sem mastite subclínica por sequenciamento do gene 16S RNAr
Página Tabela 1. Identificação das amostras selecionadas e seus respectivos valores de
contagem de células somáticas e cultura microbiológica...
89 Tabela 2. Perfil taxonômico de amostras de leite de tetos com mastite
subclínica e
saudáveis...
94 Tabela 3. Gêneros abundantes entre amostras de leite de tetos com mastite
subclínica e saudável...
96
xiv LISTA DE FIGURAS
Capítulo II-
Investigação longitudinal sobre a etiologia das infecções intramamárias subclínicas em cabras leiteiras usando espectrofotometria de massa MALDI-TOFPágina Figura 1
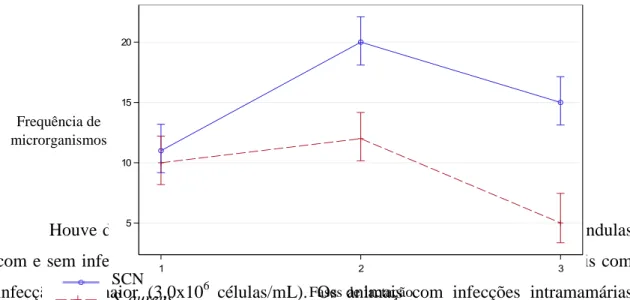
Persistência de infecções intramamárias em cabras leiteiras,ocasionadas por diferentes microrganismos durante uma lactação, por meio da distribuição binomial negativa...
47
xv LISTA DE FIGURAS
Capítulo IV
Caracterização da microbiota do leite de cabras com e sem mastite subclínica por sequenciamento do gene 16S RNArPágina
Figura 1. Construto final para sequenciamento em plataforma Illumina, incluindo os iniciadores específicos para as regi es hipervariáveis do gene 16S RNAr primers 5 e 3 específicos , as sequencias de ligação CS1 e CS2 do sistema Fluidigm e os adaptadores Illumina i5 e i7...
87
Figura 2 Curva de rarefação das amostras quanto à quantidade de
OTUs observadas na amostras...
90
Figura 3 Curvas de rarefação (índice de Shannon) de amostras
positivas e negativas (A) e amostras com isolamento (B)...
91
Figura 4 Analises de coordenadas principais da diversidade β realizado através da distância métrica -UniFrac. (A) Unifrac ponderada, (B) Unifrac não ponderada. Esta Imagem ilustra a diferença na microbiota de amostras com cultura positiva (azul) e amostras de leite com cultura negativa (vermelho).
A análise de UniFrac PCoA foram baseadas nos dados OTU, com somente as três primeiras coordenadas principais.
UniFrac ponderada com PC1= 65,09%, PC2= (10,81%) e PC3= 10,81%, mostra a abundância de organismos observados. UniFrac não ponderada com PC1= 50,36%,
PC2= 13,11% e PC3=
9,55%...
92 Figura 5
Filo de bactérias encontrados em amostras de leite de cabrascom e sem mastite subclínica...
93
xvi MASTITE SUBCLINICA CAPRINA: ETIOLOGIA E DIVERSIDADE MICROBIONA DO LEITE POR SEQUENCIAMENTO DO GENE 16S RNAr
RESUMO GERAL-
Foram desenvolvidos estudos com o objetivo de gerar conhecimentos sobre a etiologia da mastite subclínica, a caracterização do perfil da microbiota de leite de cabras associada à mastite e os efeitos da infecção sobre os parâmetros de qualidade do leite. No primeiro capítulo, apresentamos um referencial teórico abrangendo a etiologia da mastite subclínica e os avanços da genômica relacionados ao estudo da microbiota do leite caprino. No segundo capítulo, objetivou- se identificar as espécies associadas às infecções intramamárias em caprinos em diferentes estágios de lactação. Foram analisadas 390 amostras de leite provenientes de 65 cabras mestiças leiteiras de seis propriedades do Semiárido Paraibano, em três diferentes fases da lactação (inicial, intermediária e final). Oitenta e sete isolados foram identificados através de espectrofotometria de massa – tempo de vôo (MALDI-TOF MS). Os microrganismos mais isolados foram os Staphylococcus coagulase negativos (SCN), com frequências mais elevadas para as espécies Staphylococcus caprae (13,6%), Staphylococcus chromogenes (7,6%), Staphylococcus epidermidis (5,1%), e Staphylococcus warneri (4,2%). No terceiro capítulo, objetivou-se determinar o efeito da infecção intramamária subclínica causada por diferentes agentes sobre a produção e qualidade do leite de cabras. Para isso, foi realizado um estudo longitudinal em 22 cabras leiteiras (12 ¾ cabras alpinas e 10 anglo-nubianas) de uma fazenda no semiárido de Paraíba, Brasil. A infecção intramamária foi associada à redução dos teores de gordura e sólidos totais. No quarto capítulo, objetivou-se caracterizar o perfil da microbiota de animais com e sem mastite por sequenciamento da região V3-V5 do gene 16S RNAr, utilizando-se amplificação. Os resultados da metagenômica através do sequenciamento do gene 16S rRNA reforçaram não apenas a importância do gênero Staphylococcus como o principal causador da mastite em cabras, mas revelaram grupos de microrganismos mais abundantes no leite sem mastite, como os gêneros Nocardioides spp. e Ruminococcus spp.Palavras-chave: MALDI-TOF, metagenômica, microbiologia
xvii SUBCLINICAL MASTITIS GOATS: ETIOLOGY AND MICROBIAL
DIVERSITY OF MILK BY SEQUENCING THE 16S RNAr GENE
GENERAL ABSTRAT – studies were developed with the objective of generating knowledge about the etiology of subclinical mastitis and the characterization of the microbiota profile of goats' milk associated with mastitis and the effects of infection on milk quality parameters. In the first chapter, we present a theoretical reference covering the etiology of subclinical mastitis and the advances in genomics related to the study of the goat milk microbiota. In the second chapter, the objective was identify the species causing intramammary infections in goats at different stages of lactation. For this, a total of 390 milk samples were collected from 65 half-bred dairy goats at six farms in semiarid Paraiba. Eighty-seven isolates were identified by mass spectrometry - time of flight (MALDI-TOF MS). The most common microorganisms associated with mastitis were coagulase negative Staphylococci (CNS), with higher frequencies observed for Staphylococcus caprae (13.6%), Staphylococcus chromogenes (7.6%), Staphylococcus epidermidis (5.1%), and Staphylococcus warneri (4.2%). In the third chapter, the objective was to determine the effect of subclinical intramammary infection caused by different pathogens on the production and quality of goat milk. For this, a longitudinal study was performed in 22 dairy goats (12 ¾ Alpine Goats and 10 Anglo Nubian) from a farm in the semiarid of Paraiba, Brazil. Milk from infected mammary glands showed decreased fat and total solids. In the fourd chapter, the objective was to characterize the microbiota profile of animals with and without mastitis by sequencing the V3-V5 region of the 16S RNAr gene, using amplification. The results of metagenomic through 16S rRNA gene sequencing reinforced not only the importance of the genus Staphylococcus spp. as the main cause of mastitis in goats but also revealed groups of microorganisms more abundant in milk without mastitis, such as the genus Nocardioides spp. and Ruminococcus spp.
Keywords: MALDI-TOF, metagenomics, microbiology
1 CONSIDERAÇÕES INICIAS
A caprinocultura leiteira apresenta-se como uma atividade próspera no panorama atual de desenvolvimento econômico brasileiro e no mundo, cumprindo um papel socioeconômico importante em diversas regiões. No Estado da Paraíba, essa atividade assume um importante papel na diminuição da pobreza, no qual o leite produzido é vendido para programas sociais do Governo, que o distribui para famílias carentes.
Esses programas possibilitaram uma considerável melhoria do setor, fazendo com que houvesse, a cada dia, maiores perspectivas de crescimento. Entretanto, apesar da elevada importância socioeconômica que a caprinocultura leiteira assume, pouca atenção foi dada à qualidade do leite produzido.
O diagnóstico rápido e eficiente da mastite subclínica em cabras contribui para uma melhoria considerável no setor. Contudo, vale ressaltar que, especificamente, na espécie caprina, a aplicação de métodos tradicionais para detecção da mastite caprina, como o método California Mastitis Test (CMT) e contagem de células somáticas (CCS), apresenta limitações, já que foi desenvolvido e patronizado para bovinos, sendo utilizado também para caprinos de forma adaptada. Além das diferenças fisiológicas entre as glândulas mamárias de cabras e vacas, o que explica algumas diferenças no tipo de resposta inflamatória muito pouco se conhece, ainda, sobre a etiologia da mastite em cabras. O conhecimento dos principais agentes causadores da mastite subclínica em cabras no semiárido Paraibano e das alterações no leite causadas pelo processo de infecção intramamária são fundamentais para que tenhamos ferramentas capazes de reduzir os prejuízos causados pela mastite.
O recente progresso de metodologias aplicadas à microbiologia tem aberto novas oportunidades para obtenção de identificação de espécies bacterianas de forma eficiente, trazendo informações relevantes sobre os patógenos que afetam os humanos e animais.
A técnica de espectrometria de massa utilizando o sistema MALDI-TOF MS (Matrix- assisted laser desorption ionization-time of fight mass spectrometry) tem sido utilizada como uma alternativa para identificar mais rapidamente, com menor custo e melhor precisão espécies de bactérias. A metodologia consiste na identificação bacteriana a partir de uma colônia isolada em meios de cultura convencionais ou a utilização do próprio material clínico.
2 Nos últimos anos, a ciência também tem incorporado técnicas baseadas na análise de fragmentos genômicos de microrganismos do ambiente utilizando técnicas independentes de cultivo, especificamente, na determinação e análise da sequência do gene que codifica a subunidade 16S do RNA ribossomal. Essa técnica, independente de cultivo, permite obter todo o acesso da microbioma presente em uma amostra. Assim, o perfil da microbiota do leite de cabras com mastite subclínica poderá contribuir decisivamente no entendimento de fatores críticos associados a enfermidade, proporcionando a exploração de potencias biotecnológicos que poderão beneficiar o setor produtivo e os consumidores.
A presente tese está estruturada em quatro capítulos. No Capítulo I aborda um referencial teórico abrangendo a etiologia da mastite subclínica, os métodos de diagnóstico e suas limitações, e os avanços da genômica relacionados ao estudo da microbiota do leite caprino.
No Capítulo II, utilizou-se o MALDI-TOF para identificar as bactérias associadas às infecções intramamária em caprinos a campo, em diferentes estágios de lactação, a fim de elucidar a relação entre etiologia, estagio de lactação e células somáticas
No Capítulo III, avaliou-se o efeito da infecção intramamária subclínica sobre a produção e os parâmetros de qualidade do leite de cabras.
No Capítulo IV, objetivou-se caracterizar e comparar a diversidade de bactérias presentes no leite de cabras saudáveis e com mastite subclínica por sequenciamento do gene 16S RNAr.
3
CAPÍTULO I
Referencial teórico
Mastite subclínica em cabras: diagnóstico, etiologia e genômica aplicada na determinação do perfil microbiano
4 1. INTRODUÇÃO
A mastite subclínica representa um problema na criação de animais, pois além de causar perdas econômicas, também pode causar riscos à saúde pública (MORONI et al., 2005). A mastite é comumente causada por bactérias, sendo a enfermidade de maior impacto econômico na pecuária leiteira mundial. Além disso, é a principal causa de descarte de animais, e a prevenção e o tratamento são responsáveis pela maior porcentagem do uso de antimicrobianos em rebanhos leiteiros (MAKOVEC; RUEGG, 2003).
Alguns estudos apontam elevadas contagem de células somáticas e crescimento microbiológico em leite de cabra em todo o mundo (OLIVEIRA et al. 2011, KOOP et al., 2012; LEITNER et al., 2012). Portanto, sendo uma ameaça para a saúde pública, pois são frequentes os casos de doenças associadas ao consumo de leite cru ou de derivados produzidos com leite contaminado com microrganismos patogênicos. Deste modo, a precaução e o controle de infecções intramamárias dependem do conhecimento dos padrões de ocorrência da doença, que só se torna possível, através de um estudo epidemiológico (PRESTES et al., 2002).
Com o surgimento da técnica de sequenciamento de nova geração, vários trabalhos foram realizados com o objetivo de ter acesso ao genoma de diferentes microrganismos causadores da infecção intramamária em animais de produção (ALMEIDA et al., 2011; SCHRÖDER et al., 2012). Esses estudos genômicos representaram a evolução do conhecimento dos patógenos causadores dessa enfermidade. Todavia, estima-se que apenas cerca de 0,1 e 1% dos microrganismos presentes em um determinado ambiente são cultiváveis (TORSVIK et al., 2002). A determinação das sequências de apenas um microrganismo isoladamente não é suficiente para identificar toda a microbiota presente em uma amostra. Sendo assim, através da necessidade de caracterizar toda a microbiota de um tecido surgiu a metagenômica, abordagem independente do cultivo microbiológico prévio. A grande vantagem da metagenômica é permitir acesso a genes de uma amostra que não são obtidos por meio do cultivo microbiológico convencional (FALEIRO et al., 2011).
As informações obtidas a partir da análise metagenômica podem ser usadas para determinar a diversidade de uma comunidade e a presença de microrganismos específicos ou dominantes. A abordagem metagenômica permite a visualização dos
5 ecossistemas como unidades biológicas com os seus próprios repertórios genéticos (HANDELSMAN, 2004). Umas das maneiras de se ter acesso a toda microbiota em amostras ambientais é através do sequenciamento do gene 16S ribossomal RNA (rRNA), no qual fornece informações abrangentes sobre os táxons presentes em um ambiente (PATEL et al., 2001).
Diante do exposto, objetivou-se discutir sobre os métodos de diagnóstico da mastite subclínica, etiologia e os avanços da genômica relacionados ao estudo da microbiota do leite de cabras.
6 2. REFERENCIAL TEÓRICO
2.1 Mastite subclínica em caprinos
A caprinocultura vem crescendo muito nos últimos anos, tornando um dos principais setores agropecuário na região Nordeste do Brasil, sendo desenvolvida em sua grande parte em pequenas propriedades com utilização de mão de obra familiar. O efetivo do rebanho mundial de caprinos era de 1.006.785.725 milhões, sendo a maior parte localizadas em regiões em desenvolvimento. No Brasil, a população de caprinos alcançou 8.851.879 cabeças, sendo que 91,6% estão no Nordeste (FAOSTAT, 2015).
Alguns estudos realizados no Brasil apontam elevado índice de mastite na região Nordeste (PEIXOTO et al., 2010; CAVALCANTE et al., 2013), ocasionando um aumento da contagem de células somáticas e aumento das bactérias do leite. Esses resultados são de grande preocupação e pode comprometer o desenvolvimento da indústria de leite caprino.
A mastite do tipo subclínica, é a que mais prevalece nos rebanhos de pequenos ruminantes, cuja frequência estimada se encontra entre 5-30%, podendo ser ainda maior (ISLAM et al. 2012). É uma das enfermidades que ocasiona maiores perdas econômicas em uma atividade que pode chegar até 50%, devido a uma redução na produção e qualidade do leite (FONSECA; SANTOS, 2000). O conhecimento sobre a infecção intramamária subclínica possibilita buscar estratégias para a melhoria da produção e comercialização do leite.
Os fatores que influenciam na susceptibilidade à infecção intramamária incluem:
estágio da lactação; hereditariedade; resistência natural da glândula mamária, idade do animal; ordenha; manejo e patogenicidade do agente etiológico. Fatores como ordenha e manejo merecem atenção especial em função do poder de transmissão da enfermidade (PRESTES et al., 2002). Alguns estudos observaram em cabras que os maiores índices de mastite subclínica ocorre no estágio final da lactação (KOOP et al., 2010; RAZI et al., 2012). Razi et al. (2012) observaram que a associação entre a prevalência de mastite subclínica foi positivamente correlacionada com a idade, ordem de parto e estágio de lactação. A maior prevalência (66,6%) foi detectada a partir do 5° parto, enquanto prevalência mais baixa (4,76%) foi encontrada na segunda paridade.
7 2.2 Métodos de diagnóstico
O diagnóstico da mastite subclínica pode ser realizado por métodos diretos e indiretos. Os métodos diretos baseiam-se na identificação do agente etiológico mediante a demonstração da presença de microrganismos no leite dos indivíduos. Os métodos indiretos baseiam-se em vários critérios de evolução da intensidade da reação inflamatória, que são o California Mastitis Test (CMT) e a contagem de células somáticas (CCS) (MOTA, 2008). Vale ressaltar que esses métodos foram criados e patronizados para a avaliação de qualidade de leite bovino, sendo utilizados também para o leite caprino (MADUREIRA et al., 2010).
O objetivo do CMT é determinar o escore que melhor reflita a quantidade de células somáticas existentes no leite (WINTER et al., 1999). Apresentar um melhor escore de CMT que represente a infecção intramamária em caprinos é muito difícil, isso ocorre porque o estágio de lactação pode influenciar o resultado do CMT. O leite de cabra apresenta altos teores de células somáticas, ocasionadas pela descamação de epitélio alveolar em face de processos fisiológicos, desta forma, podendo interferir nos resultados dos testes para detectar a inflamação intramamária subclínica (FONSECA;
SANTOS, 2000).
Na espécie bovina a interpretação dos resultados do CMT são expressos em cinco escores (zero, traços, 1 +, 2 + ou 3 + sinais positivos) (FONSECA; SANTOS, 2000). Entretanto, devido ao maior número de células somáticas no leite dos animais da espécie caprina, devem-se considerar como negativos os sem reação e fracamente positivos (1+) (CONTRERAS et al., 1996). Mota (2008) estabelece que em caprinos para considerar a infecção instalada deve-se considerar apenas os positivos (2+) e os fortemente positivos (3 +).
Madureira et al. (2010) observaram em caprinos que os escores do CMT negativo, traços, 1+, 2+ e 3+, aumentaram com a elevação da celularidade do leite. Por outro lado, Tonin e Nader filho (2005) observaram que o teste do CMT apresentou melhor conjunto de sensibilidade (57,6%), especificidade (86,0%) e concordância (76,4%) quando o foi considerado qualquer escore de traços com o isolamento microbiológico. Os autores observaram que o CMT seria mais eficiente em detectar
8 glândulas mamárias negativas do que positivas para mastite, devido à alta especificidade do teste em relação ao isolamento bacteriano.
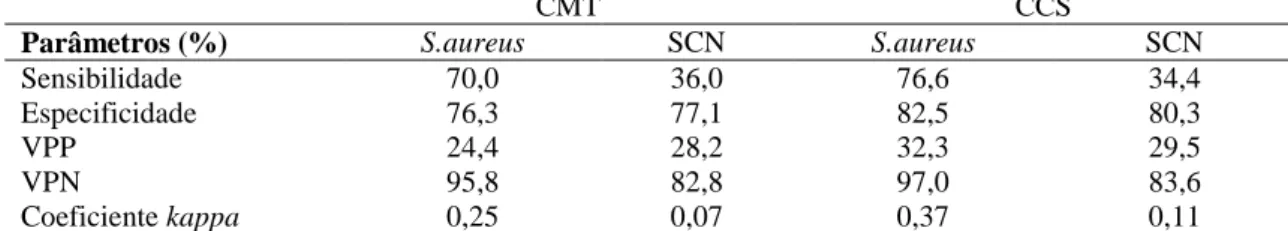
Peixoto et al. (2010) verificaram baixa concordância entre CMT e isolamento microbiológico (Kappa = 0,17) em leite caprino, de rebanhos localizados na região Nordeste do Brasil. Neves et al. (2010) também encontraram baixa concordância (Kappa=0.003) entre o CMT e a infecção intramamária, com baixa sensibilidade (46,7%) e baixa especificidade (60,6%), em cabras no semiárido Paraibano. Porém, McDougall et al. (2010) encontraram em leite caprino na Nova Zelândia 74% de sensibilidade e especificidade para CMT. Perssom et al. (2011) também encontraram uma forte associação entre a CMT com escore 2+ e o isolamento bacteriano, em cabras na Suécia. Recentemente, Roukbi et al., (2015) encontraram uma associação bastante significativa entre o CMT e o isolamento microbiológico, com 90% de sensibilidade, em leite caprino da raça Damascus na Síria.
Santos et al. (2004) ao estudarem a validação do California Mastitis Test como método diagnóstico da infecção intramamária subclínica em cabras. Os autores determinaram que o CMT apresentou 59,2% de sensibilidade, 78,2% de especificidade, 56,9% de Valor Preditivo Positivo (VPP), 79,8% de Valor preditivo negativo (VPN) e coeficiente kappa de 0,37. Neves et al. (2010) observaram que de 261 amostras de leite analisadas, 105 (40,22%) foram positivas no CMT e apenas 30 (11,49%) foram positivas no exame microbiológico, sendo 75 amostras com resultados falso-positivos.
Bezerra et al. (2006) ressaltaram que para evitar resultados falso-positivos, o California mastitis test (CMT) deve ser associado ao exame microbiológico do leite.
A ocorrência do desprendimento das partículas citoplasmáticas em cabras, dependendo do método de contagem, são erroneamente incluídas como células de defesa (PAAPE et al, 2001). O método microscópico direto utilizado para a contagem de células somáticas possibilita a coloração apenas das células nucleadas e permite a exclusão dos corpúsculos citoplasmáticos do leite (ZENG et al., 1997). A contagem eletrônica de células somáticas em equipamentos baseados em citometria de fluxo, são utilizadas para a enumeração das células do leite de vacas (GOMES et al., 2010), o que ocasionam alterações dos resultados quando utilizados em caprinos. Porém, Andrade et al. (2001) encontraram correlação positiva e alta (r=0,76) entre a contagem
9 microscópica e eletrônica por citometria de fluxo em leite de caprinos do tronco Alpino no Brasil.
Vários fatores afetam a contagem de células somáticas, tais como número de lactações, idade do animal e fase da lactação (GOMES, 2003). Boulaaba et al. (2011) verificaram que as proporções de diferentes células de defesa presente no leite de cabras, sem infecção intramamária, foram de 79,2%, 2,8% e 17,8% de leucócitos polimorfonucleares, linfócitos e macrófagos, respectivamente. Leitner et al. (2012) observaram no estágio final da lactação de cabras leiteiras, sem infecção intramamária, uma elevação de leucócitos polimorfonucleares e mononucleados quando comparados a vacas. Outros trabalhos também verificaram aumento da CCS em cabras ,sem infecção intramamária, no estágio final da lactação (GOMES et al., 2006 ; PERSSOM et al., 2014)
Luengo et al. (2004) ao estudarem a variação da CCS de cabras, sem infecção intramamária, verificaram que a idade influenciou a CCS, com maiores contagens em animais mais velhos. Esses autores também observaram que nascimento de animais duplos também aumentava a CCS em cabras leiteiras na Espanha. McDougall e Voermans (2002) observaram que o estro ocasionou o aumento da CCS em cabras Saanen na nova Zelândia. Malheiros Filho et al. (2014) avaliando a qualidade do leite de cabras Alpinas de diferentes genótipos (Parda Alpina, puras, Parda Alpina +SRD), mantidas em regime intensivo de criação no semiárido Brasileiro, observaram que a CCS não apresentou diferença significativa entre os grupos genéticos.
Apesar de vários fatores interferirem na contagem de células somáticas, a mastite é um dos fatores que mais acarreta aumento da CCS no leite (PALES et al., 2005). De acordo com Paape et al. (2001) os neutrófilos representam de 45 a 74% das células somáticas em meios mamários não mastíticos em caprinos, e de 71 a 86% das células em meios mamários mastíticos. Quando os microrganismos invadem a glândula mamária e começam a se multiplicar, o organismo direciona os leucócitos do sangue para a região afetada, com finalidade de combater a agressão tecidual e isso, consequentemente, aumenta a quantidade de células somáticas no leite (PALES et al., 2005). Esse mecanismo de defesa é muito importante para a modulação da resistência da glândula mamária e sua suscetibilidade à infecção, ocorrendo aumento principalmente dos neutrófilos (OVIEDO-BOYSO et al., 2006). Durante a infecção, os
10 neutrófilos circulantes migram para a glândula mamária em resposta a vários mediadores inflamatórios, principalmente às citocinas (TIAN et al., 2005).
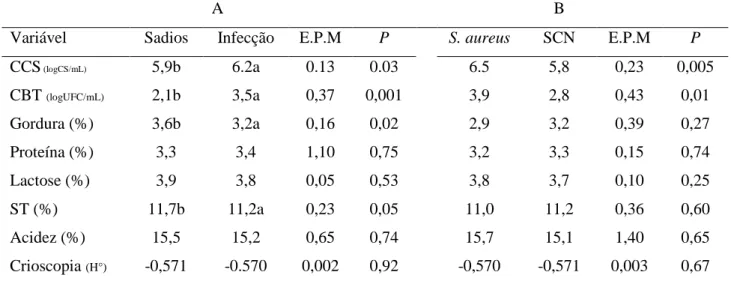
Neves et al. (2010) verificaram que não houve diferenças significativas entre a CCS de amostras positivas e negativas no cultivo bacteriano, com média de 1,39 x 106. Porém, outros trabalhos verificaram que a infecção intramamária aumenta a CCS em caprinos leiteiros (BAGNICKA et al., 2011; GEBREWAHID et al., 2012). Perssom et al. (2011) encontraram para CCS valores mais elevados de sensibilidade (67%) do que especificidade (63%), em cabras leiteiras. Correa et al. (2010) verificaram em cabras que apenas duas metades mamárias apresentaram isolamento bacteriano e CCS elevada.
Em seis fêmeas, os autores observaram contagens de CCS menores que 5x103 cél.mL-1, nos quais consideraram esse valor como limite para a CCS de glândulas mamárias com infecção.
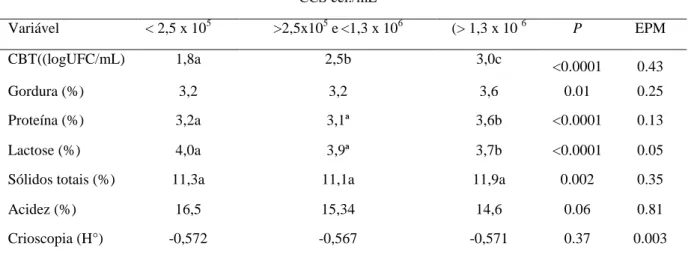
Nos Estados Unidos o limite de CCS para cabras é de 1,0 x 106 cel./mL (PAAPE et al., 2007). No Brasil não há limites máximos oficiais exigidos para a CCS no leite de cabra e medidas regulamentares neste sentido são urgentes. Porém, para que essas medidas sejam adequadas são necessários estudos que verifiquem a acurácia e aplicabilidade desses métodos (MAGALHÃES, 2005).
2.3 Etiologia
A etiologia da infecção intramamária é bastante variável o que torna necessário a identificação dos microrganismos que causam a infecção, sendo de extrema importância para o controle e prevenção da doença e monitoramento dos rebanhos (RIBEIRO et al., 2003).
Dependendo da origem do causador do patógeno as infecções intramamárias podem ser classificadas como ambientais ou contagiosas. Os patógenos contagiosos são os que se disseminam de um teto infectado para outro durante o manejo de ordenha, e estão relacionados com a má higienização dos equipamentos e antissepsia do teto. Os patógenos ambientais são os microrganismo que estão presentes no ambiente, incluindo fezes, solo, e água (BERGONIER et al., 2003).
Os principais gêneros bacterianos encontrados em casos de mastite subclínica em cabras são: Staphylococcus spp., Streptococcus spp., Corynebacterium spp.,
11 Pseudomonas sp., a Mannheimia haemolytica (CONTRERAS et al., 2007). Os Streptococcus mais isolados são os S. agalactiae, S. uberis e S. dysgalactiae. Outras bactérias que também são isoladas em casos de mastite subclínica em cabras são os coliformes (SHEARER, 2003), como a Escherichia coli, Enterobacter spp. e a Klebsiella. spp. Porém, São mais frequentes na etiologia da mastite subclínica em cabras os Staphylococcus coagulase positivos (Staphylococcus aureus) e Staphylococcus coagulase negativos (SCN) (CONTRERAS et al., 2003). Vários estudos encontraram Staphylococcus spp. como um dos principais causadores de infecção intramamária em cabras (LANGONI et al., 2006; PEIXOTO et al., 2010; BAGNICKA et al., 2011; RAZI et al., 2012).
Alguns estudos verificaram os S. aureus como o segundo patógeno mais frequente nos casos de mastite subclínica caprina (MORONI et al., 2005; PEIXOTO et al., 2010). Porém, Cavalcante et al. (2013) verificaram que os S. aureus foram um dos principais patógenos que ocasionaram infecções intramamárias subclínicas em cabras, com 29.5% dos casos obtidas em 9 propriedades no Nordeste Brasileiro. O S.aureus é encontrado na pele dos tetos, camas, mãos do ordenhador, colonizando ou crescendo prontamente na queratina do canal do teto (PHILPOT; NICKERSON, 2000).
A infecção intramamária causadas por Staphylococcus aureus ocorre via teto.
Esse microrganismo invade a extremidade do teto, principalmente nos casos em que há lesão prévia, coloniza a cisterna e se estabelece no tecido secretório. Provavelmente, o S. aureus utiliza proteínas, que estão expostas através de microlesões, como substrato para adesão na colonização e no desenvolvimento das infecções (HERMANS et al., 2010). A patogenicidade de S. aureus é multifatorial, comumente envolve um grande número de enzimas, proteínas extracelulares e outros fatores de virulência. Algumas destas enzimas, incluindo citotoxinas e exoenzimas, proteína A e várias adesinas, fixam- se na parede celular. Acopladas, estas enzimas e proteínas capacitam o microrganismo a escapar das defesas do hospedeiro, aderir às células e moléculas da matriz intercelular, invadir ou destruir as células do hospedeiro, e a se colonizar dentro dos tecidos (VOJTOV et al., 2003).
É amplamente aceito que o aumento da CCS está relacionado com a patogenicidade do agente etiológico envolvido na infecção intramamária (SOUZA et al., 2012). Perssom et al. (2011) encontraram valores mais elevados de CCS (1,0 x106/ml)
12 com isolamento de Staphylococcus aureus em relação a outros microrganismos causadores de mastite subclínica. Bagnicka et al. (2011) observaram que infecções intramamárias causadas por S. aureus aumentam a percentagem de neutrófilos quando comparados com outros microrganismos em cabras leiteiras.
Os Staphylococcus coagulase negativos, apesar de menos patogênicos que o S.
aureus, podem causar infecções intramamárias subclínicas persistentes, como também infecções clínicas. Sua prevalência aumenta frente às deficiências nos sistemas de ordenha, principalmente em relação à má higienização dos tetos (CONTRERAS et al., 2001). Alguns autores encontraram uma maior predominância de Staphylococcus coagulase negativos em caprinos leiteiros com mastite subclínica em relação a outros patógenos (LANGONI et al., 2006, GEBREWAHID et al. 2012, MCDOUGALL et al., 2014).
Os principais Staphylococcus coagulase negativos isolados de caprinos são S.
epidermidis, S. chromogenes, S. simulans, S. caprae e S. agalactiae (CONTRERAS et al. 2003). O isolamento de Staphylococcus coagulase negativos pode causar infecções persistentes, com aumento da Contagem de células somáticas do leite. A inflamação intramamária pode causar injúrias ao tecido mamário, ocasionando queda da produção de leite e também resistência a antimicrobianos (TAPONEN; PYÖRÄLÄ, 2009).
Maroni et al. (2005) observaram que o estágio da lactação influência a ocorrência de infecções causadas por SCN, no qual observaram que isolados desses microrganismos, se repetiam a partir da mesma glândula mamária, em cabras por toda a lactação.
Os microrganismos ambientais mais usualmente encontrados em infecções intramamárias são espécies de bactérias Gram negativas. Os bacilos Gram negativos comumente isolados de infecções mamárias caprinas são Escherichia coli e Pseudomonas aeruginosa (CONTRERAS et al., 2001).
Escherichia coli são habitantes normais do trato gastrointestinal de animais (HOGAN; SMITH, 2003). É uma bactéria Gram negativa anaeróbica facultativa, membro da família Enterobacteriaceae e fermentadora de lactose (CALLAWAY et al., 2003). Outras bactérias que fazem parte do grupo dos coliformes que podem causar mastite subclínica em cabras são os gêneros Enterobacter spp. e a Klebsiella. As infecções por coliformes tendem a ser de curta duração durante a lactação e raramente causam elevada CCS (SMITH; HOGAN, 2008). O meio de entrada das bactérias
13 coliformes para dentro da glândula mamária é através do canal do teto em contato com o ambiente contaminado (HOGAN; SMITH, 2003). Os coliformes não se aderem aos ductos e alvéolos no úbere, em vez disso, eles se multiplicam rapidamente no leite e produzem toxinas que são absorvidas na corrente sanguínea. Como resultado, infecções por coliformes levam à infecção intramamária (QUINN et al., 2002).
Pseudomonas aeruginosa normalmente habita o solo, água, vegetais e é comumente encontrada em equipamentos de ordenha mal higienizados e até mesmo em soluções de desinfecção para tetos, podendo estar presente na pele e fezes (TRABULSI, 2004). Muitas vezes as infecções decorrentes de contaminação por Pseudomonas spp.
estão relacionadas com a incorreta higiene no momento da ordenha e presença de água contaminada (CONTRERAS et al., 2001).
2.4 Genômica
Genômica é a área da ciência que estuda o sequenciamento dos nucleotídeos, mapeamento e análise de genomas, os quais são constituídos por todo o DNA existente nas células de um organismo, inclusive os seus genes estruturais, sequências regulatórias e regiões não codificantes (FURLAN et al., 2007).
A Genômica é subdividida em duas grandes áreas (Genômica Estrutural e Funcional) constituídas de diferentes abordagens com objetivo de gerar informações relevantes sobre a estrutura e o funcionamento de componentes biológicos de um sistema. Tais abordagens são normalmente denominadas de acordo com o princípio central abordado, sucedido pelo sufixo –ômica. Assim, por exemplo, a proteômica é a área da genômica que estuda as proteínas. Os estudos da metagenônima, transcriptômica ou metatranscriptômica, proteômica e metabolômica são conhecidos como ciências ômicas (SOUZA et al., 2014).
A genômica estrutural analisa a estrutura dos genes. Suas abordagens vão desde as ferramentas de mapeamento genético, passando pelo sequenciamento em larga escala de genes e incluindo a análise estrutural e modelagem de proteínas (Proteômica). Seu principal alvo é a geração de dados (sequências gênicas e suas respectivas localizações no cromossomo, estruturas dimensionais de seus produtos de expressão). Genômica funcional é a vertente da genômica cujo objetivo está envolvido na elucidação das funções que cada gene exerce no organismo e, ao mesmo tempo, como esses genes
14 interagem entre si dentro das redes biológicas que controlam as características fenotípicas (MUTCH et al., 2005). As duas abordagens mais utilizadas na genômica funcional têm sido o estudo do perfil global da expressão gênica (transcriptômica) e a análise sistemática das proteínas (proteômica) (FURLAN et al., 2007).
Uma ciência mais recente que vem sendo utilizada para complementar os estudos de genômica funcional é a metabolômica, que estuda a análise sistemática do metaboloma, no qual consiste no conjunto de metabolitos presentes em uma amostra biológica, que são os produtos finais dos processos celulares. O perfil metabólico pode fornecer um panorama geral sobre o estado fisiológico. O metaboloma compreende desde espécies iônicas a carboidratos hidrofílicos, álcoois e cetonas voláteis, aminoácidos e ácidos orgânicos, lipídeos hidrofóbicos e produtos naturais complexos (VILLAS-BÔAS; GOMBERT , 2006).
Por ouro lado, a genômica funcional tem como finalidade o estudo da função dos genes. Sua principal ferramenta é o estudo da expressão gênica diferencial de transcritos de RNA mensageiro (comparação de transcriptomas) entre perfis fisiológicos ou patológicos. A proteômica também estuda a expressão gênica, porém em nível de tradução (perfil de expressão protéica) (SIDER; ZAROS, 2008).
Como os métodos utilizados nas genômica normalmente geram uma quantidade imensurável de informações, a bioinformática tornou-se ferramenta fundamental para analisar e compreender os dados trazidos das diferentes áreas das ômicas, apesar de serem áreas diferentes, a genômica e a bioinformática são, atualmente indissociáveis do ponto de vista prático. Assim, a bioinformática é indispensável para a manipulação dos dados biológicos. Ela pode ser definida como uma modalidade que envolve todos os aspectos de aquisição, processamento, armazenamento, distribuição, análise e interpretação da informação biológica. Pelo meio da combinação de procedimentos e técnicas da matemática, estatística e ciência da computação (KASABOV, 2013;
SCHINDELIN et al., 2012) Essas ferramentas auxiliam a compreender o significado biológico representado nos dados genômicos (SOUZA et al., 2014).
Um dos fatores que mais contribuiu para o rápido desenvolvimento da genômica foi o Projeto Genoma Humano. Esse projeto começou a ser elaborado em 1984 e foi desenvolvido nos anos subsequentes por cientistas de diversos países: Estados Unidos, Japão, Alemanha, Reino Unido, França, e China (LANDER et al., 2001). Esse projeto
15 foi o fator determinante para o desenvolvimento de um setor industrial voltado à criação e aperfeiçoamento de produtos e equipamentos utilizados em pesquisas dessa natureza.
As tecnologias desenvolvidas foram fundamentais para o aumento da eficiência e redução dos custos do processo de sequenciamento (FURLAN et al., 2007).
As bactérias conseguem viver e se desenvolver em alguns ambientes bastante desfavoráveis. Devido à enorme diversidade genética e notáveis adaptações, têm recebido atenção dos pesquisadores da genômica. Em 1994, como uma extensão do projeto genoma humano, o departamento de energia dos Estados Unidos lançou o projeto genoma microbiano, uma ampla força tarefa para sequenciar os genomas microbianos (PALLADINO et al., 2009). Em 1997, a bactéria E. coli foi completamente sequenciada apresentando 4. 403 genes. Atualmente, existem 2.525 projetos de sequenciamento de DNA de bactérias cadastrados no NCBI (National Center for Biotechnology Information: https://www.ncbi.nlm.nih.gov/), sendo 1.017 completos, 961 sendo montado e 547 em andamento (NCBL, 2016).
A caracterização genômica de patógenos de mastite é o principal exemplo da aplicação da genômica estrutural no campo da mastite. Em 2001, foram sequenciadas, pela primeira vez, duas cepas de S.aureus resistentes a meticilina (KURODA et al., 2001). Mais recentemente, Maréchal et al. (2011) sequenciaram o genona de isolados de S.aureus causadoras de mastite subclínica (cepa O46) e mastite gangrenosa (cepa O11) em ovinos. Silva et al. (2011) sequenciaram o genoma completo do Corynebacterium pseudotuberculosis causadores de mastite em bovinos. A mesma equipe também sequenciou o mesmo microrganismo em casos de mastite caprina. A sequência dos nucleotídeos estão depositadas na NCBL (National Center for Biotechnology Information).
2.5 Sequenciamento de DNA
Os estudos em genômica dependem da aplicação conjunta, organizada e sequencial de vários métodos em biologia molecular para atingir objetivos específicos.
No entanto, de todas as técnicas utilizadas, o sequenciamento de nova geração é o componente central da genômica, por gerar as informações até então inacessíveis. O sequenciamento do DNA exibe a estrutura dos genes, permitindo aos pesquisadores
16 deduzir a sequência de aminoácidos dos produtos gênicos. Tal conhecimento permite a manipulação dos genes para compreensão ou alterações de suas funções. A comparação das sequências do DNA revela relações evolutivas que formam um embasamento para a classificação dos microrganismos (BROOKS et al., 2014). Descrevemos aqui alguns aspectos históricos relacionados ao surgimento e evolução do sequenciamento.
O sequenciamento de DNA surgiu em meados do anos 70, quando Frederick Sanger e Alan Coulson publicaram um trabalho descrevendo uma metodologia para determinar a ordem sequencial dos nucleotídeos que compõe a estrutura do DNA, baseada no princípio da terminação controlada da replicação por didesoxinucleotídeos (SANGER; COULSON, 1977a). Sanger et al. (1977b) também publicaram a sequência do genoma completo do fago phi X174 , tendo sido este o primeiro genoma de um organismo a ser completamente desvendado por seres humanos. No mesmo período, surgiu o sequenciamento químico de degradação de bases desenvolvido por Allan Maxam e Walter Gilbert. Os dois métodos são fundamentados na produção de um conjunto de fitas simples de DNA que são separadas pelo princípio de eletroforese (OKUBO, 1992).
No início dos anos 70, o processo era manual, depois de avanços tecnológicos, surgiu o sequenciamento automático com o mesmo princípio do sequenciamento manual. No método manual, a sequência de bases do DNA é lida manualmente, após a exposição das bases identificadas em um filme de raio-X. O sequenciamento automático utiliza sequenciadores com eletroforese vertical em placa ou eletroforese em capilar onde cada ddNTP possui marcação fluorescente, ao contrário do método manual, em que estes apresentam uma mesma marcação radioativa (REGITANO et al., 2007). O sequenciamento do DNA pelo método de Sanger, exige a clonagem do fragmento de DNA e de seus subfragmentos a serem sequenciados em vetores apropriados, como o bacteriófago M13 da E. coli, os quais tem sido vastamente utilizados para esta finalidade (BROOKS et al., 2014).
Devido a necessidade de se trabalhar com quantidades maiores de informações e com um menor tempo, sugiram os sequenciadores de nova geração. Essas novas tecnologias de sequenciamento começaram a ser comercializadas em 2005 e estão em plena evolução. Entre as novas plataformas de sequenciamento, duas já possuem uma vasta utilização em todo o mundo: os equipamentos da Illumina (Illumina-Sollexa, San
17 Diego/Califórnia- EUA) e a plataforma FLX da Roche (Roche GS GLX, Branford, EUA). Outros dois sistemas de sequenciamento que começaram a ser utilizados são a plataforma da Applied Biosystems, denominada SOLiD System, o Heliscope True Single Molecule Sequencing (tSMS), da Helicos. Esses sequenciadores estão divididos em 1ª geração (Degradação química e Interrupção da cadeia-Sanger), 2ª geração (454-Roche, Illumina e SOLID) e 3ª geração (Ion Torrent e PacBio RS) (CARVALHO et al., 2010).
Também foi lançado, recentemente, o de 4ª geração (Nanopore).
2.6 Utilização da metataxigenômica no estudo da mastite
Nas últimas décadas, a determinação do genoma dos microrganismos isolados têm sido úteis na caracterização dos patógenos presentes em amostras de leite, porém, esta técnica necessita de isolamento microbiológico e a maioria dos microrganismos não são cultiváveis. A metagenômica é uma abordagem genômica diferenciada, na medida em que independe do isolamento de um organismo em particular para posterior análise de seu genoma individual. A princípio, a metagenômica poderia acessar 100 % dos recursos genéticos presentes em um ambiente, (FALEIRO et al., 2011).
O termo metagenômica foi descrito pela primeira vez por Jo Handelsman em 1998. O significado do prefixo meta (além, transcendente, em grego) mostra a possibilidade de se isolar e avaliar a diversidade genômica da grande maioria dos microrganismos. As informações obtidas a partir da análise de metagenomas podem ser usadas para determinar a diversidade de uma comunidade e permite a visualização dos ecossistemas (HANDELSMAN, 2004). Assim, tornando possível a caracterização e quantificação de patógenos e bactérias comensais presente em tecidos de animais.
A metagenômica permite a aplicação em diferentes áreas, como, por exemplo, na área de saúde, agricultura e meio ambiente. Na agropecuária, por exemplo, existe potencial no desenvolvimento de metodologias de detecção precoce de patologias vegetais ou animais. Em segurança alimentar, pode‑se monitorar e detectar contaminantes microbianos e detecção de ameaças à produção de alimentos (FALEIRO et al., 2011).
A falta de identificação de microrganismos em cultura, podem ter várias explicações como: falta de simbiose necessária e de nutrientes, compostos inibidores em
18 excesso, incorreta temperatura, acumulo de resíduos a partir do seu próprio metabolismo e taxa de crescimento lenta (SIMU et al., 2004). Em casos dos animais apresentarem mastite clínica e a cultura apresentar-se negativa pode ser devido a incapacidade para cultivar das bactérias responsáveis pela doença, a sua presença abaixo dos limites para detecção ou que a mastite pode ser provocada por microrganismos não bacterianos (KUEHN et al., 2013).
O estudo da metagenômica representa o caminho para a compreensão dos microrganismos causadores da mastite, representando o caminho decisivo para o diagnóstico e tratamento. Alguns estudos com vacas foram realizados determinando a caracterização do metagenoma da microbiota do leite de animais com mastite (OIKONOMOU et al., 2012; KUEHN et al., 2013; FALENTIN et al., 2016), porém, estudos caracterizando a microbiota de leite mastítico com outros animais de produção, como os ovinos e caprinos, ainda não foram publicados.
O conhecimento de toda a microbiota do leite é necessário, pois compreender o perfil dos microrganismos permitirá proporcionar um tratamento adequado. Portanto, a identificação da microbiota do leite deve ser esclarecida para que possamos compreender os microrganismos associados a mastite. A metagenômica aplicada ao conhecimento de um determinado microbiota pode ser realizada através do sequenciamento do gene 16 S RNA ribossomal (PATEL et al., 2001).
2.7 Utilização do gene 16 S RNAr
Woese et al. (1985) comprovaram que a semelhança filogenética entre bactérias poderia ser verificada através da comparação de uma região estável do DNA, como os genes codificadores do rRNA. Esses genes codificadores são pequenas subunidades ribossomais, que são 5S, 16S e 23S. Atualmente, o gene rRNA é bastante utilizado no estudo da caracterização da microbiota de amostras ambientais e de vários tecidos de humanos e animais.
Estudos de metataxigenômica são frequentemente realizados por meio do sequenciamento do gene ribossomal 16S (rRNA 16S), que é de aproximadamente 1500 pb (PATEL et al., 2001). Os genes rRNA 16S tem em sua estrutura uma combinação de regiões variáveis, permitindo a diferenciação de procariotos, o gene possui 9 regiões,
19 V1-V9, que diferem em tamanho e em relação a sua utilidade para a taxonomia microbiana(YARZA et al., 2014). O sequenciamento das primeiras 500 pares de bases, que contém as regiões variáveis V1, V2 e V3, resulta em identificações mais precisas que a região final do gene, que inclui as zonas variáveis V7, V8 e V9 (CAI et al., 2009).
A sequência do gene 16S rRNA fornece identificação em nível de gênero na maioria dos casos (>90%), mas em nível menor para espécies (65 a 83%) (JANDA;
ABBOTT, 2007). A principais razões para se utilizar o rRNA 16S, é que o gene não foi alterado ao longo do tempo ou apresentou baixas taxas de evolução, sendo uma região semiconservativa. Como também o gene rRNA 16S (1500 pb) é suficientemente grande para ser utilizado e facilitar a bioinformática (PATEL, 2001). As desvantagens de utilizar o sequenciamento do rRNA 16s é o custo elevado e o uso de bases de dados públicas, podendo ter sequências de má qualidade com erros, incompletas e com identificação insuficiente (BOUDEWIJNS et al., 2006).
Fundamentalmente, um projeto de metagenômica possui as seguintes etapas:
amostragem dos dados, seguidos pela extração de DNA, sequenciamento, processamento das sequências (reads) e montagem (KUNIN et al., 2008). As ferramentas de bioinformática auxiliam na interpretação dos resultados gerados pelos diferentes sequenciamentos. Permitem identificar um possível papel biológico para os segmentos de DNAs e proteínas, por meio de comparações com sequências e estruturas depositadas em bancos de dados de programas computacionais (FALEIRO et al., 2011).
As ferramentas de bioinformática permitem a análise das sequências, possibilitando a identificação de genes e possíveis rotas metabólicas.
A análise dos dados de metagenômica incide em um processamento com várias etapas, que envolvem: filtragem das leituras por qualidade e tamanho, busca das sequências filtradas contra um banco de dados genéticos com o objetivo de identificar sequências similares conhecidas, atribuição de classificação taxonômica sobre os resultados desta busca, análise estatística das sequências classificadas e não classificadas, resumo e visualização dos resultados (DIAS, 2012). As sequências de nucleotídeos são comparadas com sequências do GenBank (acessadas através do NCBI- National Center for Biotechnology Information) usando o programa BLAST que serve para identificar a qual espécie pertence o DNA sequenciado (MEYER et al., 2009).
Além do NCBI, outros bancos de dados podem ser utilizados o EMBL-EBI EMBL-EBI
20 (European Bioinformatics Institute), DDBJ (DNA Data Bank of Japan), INSDC (união do DDBJ, NCBI e EMBL-EBI).
Ferramentas conhecidas como pipelines metagenômicos, otimizam o tempo de execução da análise de dados de sequenciamento em larga escala, tentando minimizar a intervenção humana neste processo. Essas ferramentas standalone e online foram desenvolvidas e passaram a ser utilizadas para realizar uma análise mais rápida e mais automatizada quanto possível. Alguns exemplos de tais ferramentas são: PANGEA, MOTHUR, RDP classifier, Galaxy e MEGAN (DIAS, 2012).
KUEHN et al. (2013) avaliaram o perfil da microbiota de amostras de leite de vaca com cultura microbiológica negativa e positiva do mesmo animal. Os autores sequenciaram o gene rRNA 16S (Roche 454), as bibliotecas foram feitas com a amplificação da região V1 –V2. Foram identificados seis gêneros comuns em ambas as amostras: Bradyrhizobium, Corynebacterium, Pseudomonas, Ralstonia, Sphingomonas, e Staphylococcus. Os perfis foram significativamente diferente entre as amostras clínicas e saudáveis com maiores quantidades de Brevundimonas, Burkholderia, Sphingomonas, e Stenotrophomonas encontrado em amostras clínicas e Pseudomonas, Psychrobacter, e Ralstonia em amostras saudáveis.
OIKONOMOU et al. (2012) também encontraram diferenças no perfil da microbiota de leite de vacas saudáveis e com mastite. Os autores utilizaram o pirosequenciamento (454) do gene rRNA16S, com amplificação da região V1 e V2. Os patógenos identificados por cultura foram geralmente entre os organismos mais frequentemente detectados por pirosequenciamento (Escherichia coli, Klebsiella spp. e Streptococcus uberis). As amostras que apresentaram cultura negativa, mas que estavam com mastite clínica, foram encontrados patógenos como Escherichia coli, S. aureus, Streptococcus, Arcanobacterium. Com uma maior quantidade de Asticcacaulis nas amostras. Os autores identificaram os genes dos microrganismos através do NCBI, com o BLAST. Outros estudos também utilizaram o gene 16S RNAr para caracterizar o perfil taxonômico do leite de vacas com mastite subclínica (BHANDERI et al., 2014;
FALENTIN et al., 2016), os principais gêneros encontrados foram Staphylococcus ssp.
Bacillus ssp., Bifidobacterium spp., Pseudomonas ssp. Serratia ssp.
Nos estudos de diversidade microbiana é utilizado o conceito de Unidade Taxonômica Operacional. O OTU é apropriado para comparar riqueza relativa quando
21 sequências do gene 16S rRNA são avaliadas de acordo com a mesma região no gene (STACH et al., 2003). Essa unidade taxonômica podem referir-se a qualquer nível taxonômico, como por exemplo, identidade maior ou igual a 97% em um alinhamento de fragmentos do gene 16S rRNA, valor normalmente usado como uma aproximação para espécies. Similaridade maior ou igual a 95%, como uma aproximação para gênero e maior ou igual a 80% como uma aproximação para filo (SCHLOSS:HANDELSMAN, 2005)
Os índices de diversidade utilizados em estudo do perfil da microbiota do leite são diversidade alfa e beta. A diversidade alfa ou local é o número total de espécies em um habitat. Diversidade beta é a de mudança de espécies ao longo de um gradiente ambiental (MAGURRAN, 2004). A primeira medida não paramétrica de diversidade alfa foi proposta por Simpson. O autor sugeriu que a diversidade era inversamente relatada pela probabilidade de que dois indivíduos escolhidos ao acaso pertencessem à mesma espécie (SIMPSON, 1949). A medida de diversidade alfa mais utilizado é o índice de Shannon, pois incorpora tanto a riqueza quanto a equitabilidade. (SHANNON;
WEAVER, 1949).
UniFrac é uma medida de diversidade beta que usa informações filogenéticas para comparar amostras ambientais. UniFrac juntamente com técnicas estatísticas multivariadas padrão, incluindo a análise de coordenadas principais (PCoA), identifica fatores que explicam as diferenças entre as comunidades microbianas (LOZUPONE et al., 2011). A Unifrac ponderada leva em consideração a abundância relativa de cada grupo de sequências, enquanto a uniFrac não ponderada leva em consideração apenas presença-ausência de diferentes UTOs nas amostras (REZENDE, 2016).
22 3. CONSIDERAÇÕES FINAIS
As novas plataformas de sequenciamento proporcionam a vantagem de permitir o sequenciamento altamente representativo de genomas em um único passo e com redução de custo, quando comparados com as metodologias de primeira geração. Seu emprego tem revolucionado a genômica com a geração de dados altamente reprodutíveis. Desta forma, o surgimento dessas novas plataformas permitiram o estudo da metagenômica, no qual representa o caminho decisivo para o controle de infecções.
O estudo da metagenômica da microbiota do leite de cabras é necessário, pois através desta análise poderemos ter conhecimento de quais microrganismos benéficos produzidos estão associados a diminuição da microbiota patogênica no úbere, como também quais os microrganismos podem aumentar os índices de mastite no rebanho.
Sendo assim, conhecimento aprofundado da composição da microbiota causadoras da mastite em animais deverá conduzir a novas intervenções terapêuticas com base na possível modulação desse microbioma.