Universidade Federal do Rio Grande do Norte
Centro de Tecnologia
Programa de Pós-Graduação em Engenharia Elétrica
Dissertação de Mestrado
Um Sistema Computacional para Diagnosticar
Viroses de Plantas Usando a Técnica de PCR com
Construção de “Primers Espécie-Específicos”
AUTOR: Kliger Kissinger Fernandes Rocha
ORIENTADOR: Prof. Dr. Luiz Marcos Garcia Gonçalves CO-ORIENTADOR: Prof. Dr. Paulo Sérgio Marinho Lúcio
Natal/RN – Brasil
Um Sistema Computacional para Diagnosticar Viroses de
Plantas Usando a Técnica de PCR com Construção de
“Primers Espécie-Específicos”
Kliger Kissinger Fernandes Rocha
Aprovada, em 04 de abril de 2005, pela Comissão Examinadora formada pelos seguintes membros:
_____________________________________________________________ Profa. Dra. Eliana Silva de Almeida – TCI-UFAL
_____________________________________________________________ Prof. Dr. José Alfredo Ferreira da Costa – UFRN
______________________________________________________________ Prof. Dr. Paulo Sérgio Marinho Lúcio – UFRN (Co-Orientador)
______________________________________________________________ Prof. Dr. Luiz Marcos Garcia Gonçalves – UFRN (Orientador)
Universidade Federal do Rio Grande do Norte
Centro de Tecnologia
Programa de Pós-Graduação em Engenharia Elétrica
Kliger Kissinger Fernandes Rocha
Um Sistema Computacional para Diagnosticar
Viroses de Plantas Usando a Técnica de PCR com
Construção de “Primers Espécie-Específicos”
ORIENTADOR: Prof. D.Sc. Luiz Marcos Garcia Gonçalves
CO-ORIENTADOR: Prof. D.Sc. Paulo Sérgio Marinho Lúcio
Natal/RN – Brasil Abril de 2005
A Deus onde sempre encontro forças para superar as dificuldades.
Aos meus pais; Consuelo Fernandes Rocha e Manoel Ferreira da Rocha (in memorian), exemplos de vida, mentores da minha evolução profissional e moral.
As minhas irmãs, Kelly Cristina Fernandes
UNIVERSIDADE FEDERAL DO RIO GRANDE DO NORTE
Date: abril de 2005
Author: Kliger Kissinger Fernandes Rocha
Title: Um Sistema Computacional para Diagnosticar Viroses de Plantas Usando a Técnica de PCR com Construção de “Primers Espécie-Específicos”
Department: Programa de Pós-Graduação em Engenharia Elétrica
Degree: M.Sc. Convocation: May Year: 2005
Permission is herewith granted to Federal University of Rio Grande do Norte to circulate and to have copied for non-commercial purposes, at its discretion, the above title upon the request of individuals or institutions.
THE AUTHOR RESERVES OTHER PUBLICATION RIGHTS, AND NEITHER THE THESIS NOR EXTENSIVE EXTRACTS FROM IT MAY BE PRINTED OR OTHERWISE REPRODUCED WITHOUT THE AUTHOR’S WRITTEN PERMISSION.
Agradecimentos
Agradeço principalmente à Deus, inteligência suprema, causa primária de todas as coisas, por
sempre me doar todas as condições necessárias para evoluir como consciência e tantos motivos
para agradecer e ser feliz.
Aos meus amorosos pais, por vários motivos: pela educação fundamentada na ética e no
amor responsável, pelo apoio financeiro, pelos exemplos de seres humanos louváveis na conduta
moral, por terem me concebido com muito amor, pelas renuncias, pela paciência, pelas crenças e
valores Cristãos... Enfim, por dedicarem-se tanto pela felicidade dos filhos.
Às minhas irmãs, pelo convívio amoroso que sempre facilita o meu sucesso e felicidade.
Aos prezados orientadores (Prof. Paulo Marinho e Prof. Luiz Marcos), modelos de
profissionais, por me aceitarem como orientando, pelas valiosas informações para minha
formação profissional, e principalmente por me incentivarem e investirem tanto na minha
qualificação profissional.
À banca examinadora, pelas importantes contribuições para melhorar este trabalho.
À CAPES, pelo apoio financeiro recebido.
Aos amigos: Hani e família, Sheila Mara e Susy; por todos os momentos que passamos
juntos compartilhando sentimentos e idéias que contribuíram para o meu sucesso neste trabalho.
A todos os professores do meu histórico como estudante, pelos ensinamentos que
contribuíram para uma melhor compreensão deste trabalho, e ampliação da minha visão de
mundo. E, nesta fase de Pós-Graduação, agradeço em especial aos seguintes Professores: Dr.
Luiz Affonso Henderson Guedes de Oliveira pela orientação no Estágio Docente; e Dra. Patrícia
Sommer pelo incentivo e apoio.
Aos funcionários da UFRN que colaboraram, principalmente a Santana, por sempre ser
prestativo.
A todos que, de bom grado, contribuíram direta e indiretamente até em anonimato para a
Conteúdo
Resumo
Abstract
1 Introdução 1
1.1 Motivação para o Trabalho. . . 2
1.2 Organização da Dissertação. . . 4
2 Os Primers e a Técnica PCR 5 2.1 A técnica PCR . . . 8
3 O Problema do Projeto e Escolha de Primers 12 3.1 O Método Tradicional . . . 13
3.2 O Problema de Alinhamento. . . 15
3.2.1 O Algoritmo de Força Bruta. . . 15
3.2.1.1 Algoritmo. . . 16
3.3 O Problema da Construção de Primers . . . 18
3.2.1 Temperatura de desnaturação (Tm) e tempo . . . 18
3.3.2 Temperatura de Anelamento (TA) e construção de primer . . . 19
3.3.3 Comprimento do primer. . . 20
3.3.4 Primers degenerados. . . 21
3.3.5 Temperatura de Extensão . . . 22
3.3.6 Número de Ciclos . . . 23
4 Uma Solução Computacional 24 4.1 O Sistema . . . 26
4.1.1 O Módulo de Alinhamento. . . 29
4.1.2 O Módulo de Construção de Primers . . . 30
4.2 Programação, Plataforma e Interface . . . 30
5 Experimentos e Resultados 36 5.1 Experimentos . . . 37
5.1.1 Cenário 1 . . . 38
5.1.2 Cenário 2 . . . 50
Lista de Figuras
1.1 Etapas do ciclo de PCR . . . 1
2.1 Interfaces dos locais na Internet usados para alinhamento de seqüências de genomas 7 2.2 Primer na fita-molde com hidroxila livre e nucleotídeo com fosfato livre . . . 9
2.3 Os primeiros quatro ciclos de uma PCR . . . 9
2.4 Etapas de PCR (amplificação exponencial em 30 ciclos). . . 10
2.5 Termociclador. . . 10
2.6 Ingredientes da reação in vitro da PCR. . . 10
3.1 Exemplos de complementaridade inter- e intra-primers que resulta em problemas. . . 14
3.2 Gel de Eletroforese com vários tamanhos de fragmentos amplificados por PCR . . . . 14
3.3 Funcionamento do Algoritmo de Força Bruta . . . 17
3.4 Seqüências de primers foram derivadas dos alinhamentos múltiplos de seqüência . . 21
4.1 Formato Fasta. . . 27
4.2 Formato do arquivo de primers do programa. . . 32
4.3 Tela inicial do programa. . . 33
4.4 Tela mostra em que sequencia e posição da sequencia se encontra o primer. . . 33
4.5 Tela de resultados que mostra as regiões especificas. . . 34
4.6 Tela de resultados que mostra as regiões universais. . . 34
4.7 Tela mostrando os primers da região específica com seus respectivos parâmetros. . . 13
5.1 Tela de resultados do Entrez no NCBI mostrando 7 genomas de vírus da batata . . . . 39
5.2 Tela de Resultados do Blast-N para Potato Vírus V. . . 41
5.3 Tela de Resultados do Blast-N para Cherry rasp leaf virus RNA2. . . 42
5.5 Tela de Resultados do Blast-N para Potato Vírus Y . . . 45
5.6 Tela de Resultados do Blast-N para Narcissus mosaic virus . . . 47
5.7 Tela de Resultados do Blast-N para Potato Vírus M . . . 48
5.8 Tela de Resultados do Blast-N para Potato Vírus A. . . 49
5.9 Tela de resultados do Entrez no NCBI com 4 genomas de vírus de meloeiro . . . 51
5.10 Tela de Resultados do Blast-N para Cucurbit yellow stunting disorder virus RNA2. . 52
5.11 Tela de Resultados do Blast-N para Cucurbit yellow stunting disorder virus RNA1. . 53
5.12 Tela de Resultados do Blast-N para Melon chlorotic leaf curl virus . . . 54
Lista de Gráficos, Diagramas e Tabelas
3.1 Gráfico de Tempo de Execução de Força Bruta. . . 18
5.1 Gráfico de performance da última versão do algoritmo de Força Bruta . . . 37
4.1 Diagrama de Acessibilidade ao banco de dados. . . 29
4.2 Diagrama de Arquitetura do Sistema. . . 29
4.3 Diagrama do Módulo de Alinhamento. . . 30
4.4 Diagrama do Módulo de Construção de Primers. . . 30
4.1 Soluções do Sistema Dadas aos Diversos Problemas Abordados . . . 45 5.1 Tabela de Primers de Espécies de Viroses de Batata Obtida Pelo Programa Proposto . 39
Resumo
Propõe-se uma solução computacional baseada no desenvolvimento de um software para construir primers espécie-específicos, usados para melhorar o diagnóstico de viroses de planta por PCR. Primers são indispensáveis à reação PCR, além de proporcionar a especificidade do diagnóstico. Um primer é um fragmento de DNA sintético, curto e de fita simples, utilizado como um iniciador na técnica PCR que flanqueia a seqüência que se deseja amplificar. Primers espécie-específicos são primers que só indicam a região bem conhecida de início e término onde a enzima polimerase vai amplificar, de uma determinada espécie, ou seja, é específica para somente uma espécie. Assim, o objetivo principal deste trabalho é automatizar o processo de escolha de primers, otimizando a especificidade dos primers escolhidos pelo método tradicional.
Abstract
It proposes a established computational solution in the development of a software to construct species-specific primers, used to improve the diagnosis of virus of plant for PCR. Primers are indispensable to PCR reaction, besides providing the specificity of the diagnosis. Primer is a synthetic, short, single stranded piece of DNA, used as a starter in PCR technique. It flanks the sequence desired to amplify. Species-specific primers indicate the well known region of beginning and ending where the polymerase enzyme is going to amplify on a certain species, i.e. it is specific for only a species. Thus, the main objective of this work is to automatize the process of choice of primers, optimizing the specificity of chosen primers by the traditional method.
Capítulo 1
Introdução
A Bioinformática visa compreender problemas em que questões biológicas delineiam questões algorítmicas, bem como propor suas soluções. É uma área de pesquisa relativamente nova, com um crescimento substancial de trabalhos.
Na primeira metade da década de 80, foi desenvolvido um método de amplificação de
seqüências de DNA que revolucionou a análise genética nestes últimos anos: a “reação em cadeia da polimerase” (ou PCR, do inglês Polymerase Chain Reaction). Esta técnica possibilita que múltiplas cópias de uma molécula de DNA sejam geradas por meio da amplificação enzimática de uma seqüência de DNA escolhida. A técnica baseia-se na capacidade que a enzima
DNA polimerase tem de replicar seqüências de DNA, em certas condições laboratoriais, a partir de um par de pequenos fragmentos iniciadores da fita molde, denominados de primers.
A Figura 1.1 ilustra a técnica. Através de variações alternadas e cíclicas de temperatura que permitem a desnaturação (ex. 92ºC, abertura da fita dupla de DNA), anelamento (ex. 54ºC, pareamento dos primers ou iniciadores) e extensão (ex. 72ºC, cópia da fita dupla original pela incorporação de nucleotídeos nas fitas complementares). Assim, uma determinada seqüência de DNA é replicada, ciclo após ciclo, em progressão geométrica (figura 2.3), o que torna possível
sua visualização em gel de eletroforese na forma de uma banda (figura 2.7).
O desenvolvimento dessa técnica e suas aplicações concederam ao americano Kary Mullis o Prêmio Nobel em Química em 1993. As aplicações da técnica são inúmeras, conforme pode ser verificado em revisão feita por Mullis, Rerré e Gibbs [23]. A PCR tem sido utilizada, por
exemplo, desde experimentos relacionados ao seqüenciamento de DNA até aplicações comerciais na área de diagnose. Algumas variações da PCR levaram ao desenvolvimento de outras técnicas poderosas na análise de diversidade genética, como: AFLP, RAPD, SAMPL e SSR.
✍ ✎ ✍ ✒ ✔ ✖ ✘ ✚ ✜ ✣ ✥ ✔ ★ ✜ ✪ ✜ ✔ ✮ ✪ ✜ ✱ ✜ ✴ ✵ ✔
O trabalho desenvolvido foi inicialmente motivado pela necessidade de se estabelecer condições técnicas de identificação por PCR de viroses em plantas no Estado do Rio Grande do Norte. Especificamente, procurou-se desenvolver uma ferramenta em bioinformática que aperfeiçoasse a escolha de primers ou oligonucleotídeos para a reação de PCR. Neste trabalho, o aplicativo desenvolvido e otimizado evitaria a fabricação de oligonucleotídeos pouco eficientes na identificação das viroses em plantas caso fossem escolhidos manualmente.
Para o desenvolvimento da ferramenta foi necessário, no entanto, especificar quais os parâmetros que influenciariam na qualidade da amplificação por PCR. Estes parâmetros têm uma influência direta na construção de primers. Por outro lado, há que considerar que a região do genoma viral tem que garantir a especificidade do diagnóstico em nível de espécie do agente causador da virose.
Uma das principais motivações que encontramos é a tentativa de diminuir ou eliminar o prejuízo na fruticultura do Estado do Rio Grande do Norte, onde plantadores têm queimado plantas em áreas agrícolas por causa de suspeita de apenas um ou alguns exemplares da planta com virose. O problema é que muitas vezes não é virose, podendo ser outro agente patogênico ou deficiência ou mesmo o excesso de nutrientes. Mas, para evitar o risco de uma epidemia, infelizmente, quase sempre é preferido o modo drástico de resolver o problema; queimando toda
A motivação biológica de ser um programa de primers para vírus é o fato de que, por serem organismos altamente instáveis, compostos por genes mutantes e recombinantes, os vírus pesquisados apresentam problemas quanto a sua erradicação.
Basicamente, o software contempla dois módulos: um módulo de alinhamento dos genomas de vírus para separar as áreas polimórficas, e o segundo módulo é de construção de primers
específicos para diagnosticarem uma determinada espécie de vírus por PCR. Testes e experimentos foram realizados e os resultados foram satisfatórios para genomas pequenos como os de vírus. O presente trabalho já publicou resultados parciais em pôster no SIBGRAPI 2003 [44], e em artigo completo no 4th IEEE International Symposium on Bioinformatics and
Bioengineering (BIBE 2004) [45].
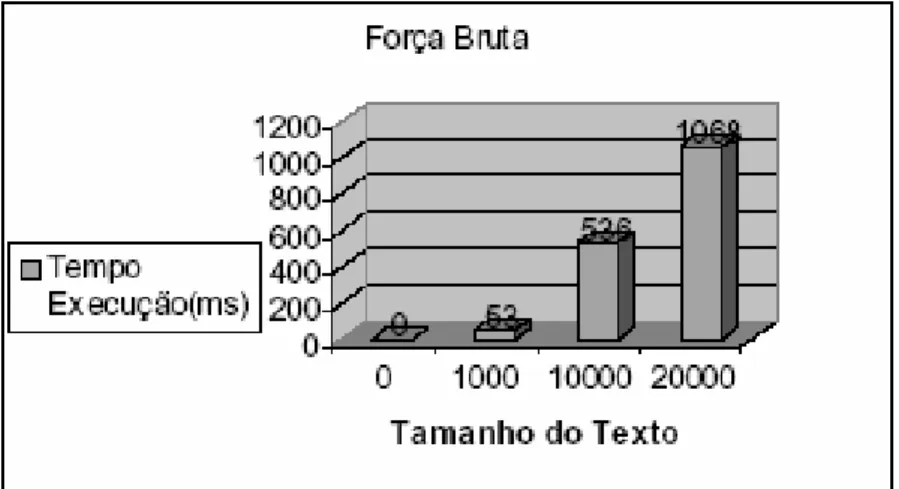
Mesmo usando o Algoritmo da Força Bruta [12], considerado um método simples, uma intensa modificação realizada foi no sentido de evitar comparações desnecessárias e melhorou em 75% a performance de tempo do programa.
A principal questão deste trabalho é propor uma estratégia de escolha de primers eficiente para diagnosticar um patógeno (agente biológico capaz de causar doença) suspeito. A resposta a esta questão avança a metodologia de diagnóstico molecular em nível de espécie, facilitando no processo de tratamento.
Como contribuições deste trabalho podem-se citar a construção de um banco de dados, contendo seqüências de vírus de plantas, e um sistema para alinhar e separar domínios de seqüências. O banco de dados que este trabalho se refere é uma coleção ordenada de arquivos semelhantes, em conformidade com um formato padrão de conteúdo. O banco de dados de arquivos simples pode ser pesquisado devido à indexação. Contudo, à medida que a coleção de arquivos simples fica cada vez maior, torna-se ineficaz trabalhar com ela.
Esta estratégia melhora a mineração de dados no banco de dados, encontrando seqüências e gerando múltiplos alinhamentos. Essas seqüências podem compartilhar similaridades com domínios e diferenciar entre domínios polimórficos. Trabalhou-se com domínios polimórficos
para construir primers com especificidade elevada.
Além disso, o programa desenvolvido possibilita também um estudo de polimorfismo por possuir um módulo de alinhamento indicando regiões com polimorfismo e regiões similares entre duas ou mais espécies de vírus. A construção de um banco de dados contendo arquivos de
O Capítulo 2 trata dos trabalhos relacionados, discutindo a contribuição deste trabalho comparado às publicações estudadas. Um apanhado do estado da arte é apresentado, incluindo informações teóricas básicas, necessárias ao entendimento do problema tratado.
No Capítulo 3, apresentamos o histórico, especificação e detalhamento do problema abordado,
incluindo um estudo dos parâmetros necessários à técnica PCR.
O capítulo 4 aborda a solução teórica encontrada, apontando para possíveis formas de resolver o problema. Neste Capítulo, apresentamos também a solução computacional adotada para solucionar o problema e os módulos do programa implementados.
No capítulo 5 são mostrados os resultados de diversos experimentos e testes realizados para validar o sistema computacional proposto.
Capítulo 2
Os Primers e a Técnica PCR
Muitos trabalhos na área de Biologia Molecular estão relacionados com a construção e escolha de primers para diagnóstico, sendo alguns deles estudados neste trabalho [01 a 11]. A maioria desses usa programas somente para construção de primers sem prever por alinhamento que região do genoma a ser amplificada seria ideal. A proposta deste trabalho é automatizar e aperfeiçoar o processo com a estratégia do alinhamento antes da construção de primers.
Alguns métodos computacionais ou programas estão disponíveis atualmente para a construção de primers [01,02,03], com uma finita probabilidade de produzir erros. Os trabalhos encontrados na literatura descrevem os passos envolvidos no processo e os esforços dispensados para automatizá-lo. A proposta geral é selecionar uma região para construir um primer onde a probabilidade de erro de diagnóstico usando PCR seja baixa [01,03], considerando não somente
as regiões selecionadas visualmente, mas também regiões examinadas estatisticamente.
Convém ressaltar que, em relação aos programas disponíveis na Internet, públicos e privados, o programa proposto neste trabalho tem várias vantagens, sendo a automação do processo de diagnóstico para um usuário sem muitos conhecimentos técnicos de computação uma das principais delas. O programa desenvolvido lista vários primers candidatos com os respectivos atributos para a correta decisão do especialista. No modo como são listados os primers, pode-se distinguir facilmente as vantagens dos candidatos.
metodologia não é aplicada pela maioria dos programas para projetar primers estudados neste trabalho. O programa público Gene Fisher tem a mesma metodologia [40] de busca, mas usa o CLUSTALW ou DCA como programa de alinhamento, dependendo da demora na Internet o processo é moroso e não é considerado totalmente automatizado. Ainda, o programa proposto neste trabalho não tem somente uma funcionalidade básica (construção de primers
espécie-específicos), mas também permite ao pesquisador estudar o polimorfismo de vírus em uma mesma família, e entre taxonomias diferentes.
Existem alguns problemas no caso múltiplo que não existem no caso básico: a pontuação dos alinhamentos; complexidade da abordagem que utiliza programação dinâmica pura (trata-se de
um problema NP - completo); criação de heurísticas que aumentem a velocidade da computação; etc. Existem tanto estudos teóricos que atacam esses problemas quanto algoritmos que implementam essas heurísticas (métodos de alinhamento em estrela, em árvore, e outros), tornando possível, assim, viabilizar o alinhamento de múltiplas seqüências.
O estudo de algoritmos de Bioinformática teria sido incompleto se não fosse abordado um tema cuja importância aumenta a cada dia: a comparação de seqüências genéticas em bancos de dados. A busca de seqüências em bancos de dados permite determinar quais das centenas de milhares de seqüências presentes no banco podem estar relacionadas a uma dada seqüência. Nesse tipo de ambiente, a operação básica consiste em alinhar uma seqüência de consulta com as seqüências do banco de dados.
Os atuais bancos de dados de seqüências já são gigantescos, e continuam a crescer numa taxa exponencial, como por exemplo o “Genbank” em 2004 publicou 44.575.745.176 pares de bases e 40.604.319 seqüências [43]. Isso torna a aplicação de programação dinâmica pura inviável, obrigando o uso de heurísticas, que aumentam bastante a velocidade dos alinhamentos (mas com uma pequena probabilidade de perder alinhamentos verdadeiros).
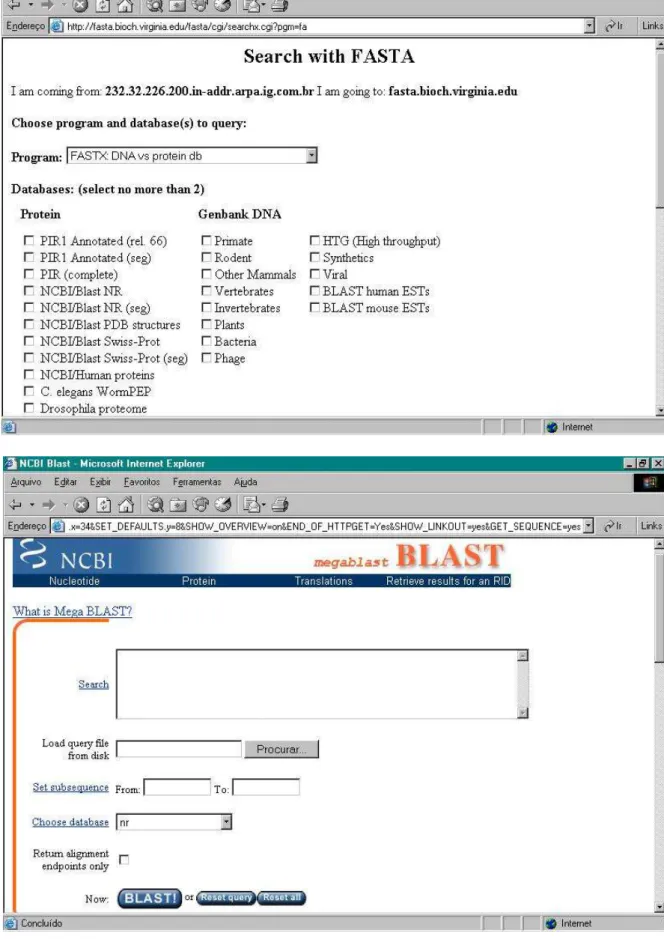
Os dois programas de busca de seqüências mais usados na atualidade são o “FASTA” e o “BLAST” (vide Figura 2.1). O FASTA foi o primeiro de todos os programas do tipo a ser
amplamente utilizado, enquanto o BLAST, posteriormente introduzido, trouxe uma série de refinamentos. Quando de uma busca, ambos aplicam em primeiro lugar métodos heurísticos e, após a obtenção de uma lista inicial de seqüências, métodos baseados em programação dinâmica são usados para, finalmente, gerarem a lista final de hits. Esta palavra hits pode ser identificada
O programa desenvolvido neste trabalho compara as seqüências obtidas de um banco de dados com uma seqüência padrão, identificando e numerando as alterações encontradas. Além disso, é possível no sistema concluir informações importantes cruzando estes resultados de alinhamento com os dados dos respectivos vírus estudados.
O sistema desenvolvido forma uma parte do elo de ligação entre os dados biológicos de vírus,
de um lado, e as informações de seqüências genéticas, de outro, recebendo dados tanto de arquivos Fasta de seqüenciadores quanto dos bancos de dados genéticos de vírus de plantas.
Esta ferramenta computacional é extremamente útil em comparação às outras de domínio público, ao permitir um controle muito mais acurado no projeto de primers específicos, e uma
mineração de dados pelo relacionamento com o banco de dados de vírus.
❉ ❊● ❍ ■ ❏ ❑ ▲ ◆ ❑ ❖ ◗ ❙
A técnica de PCR (do inglês Polymerase Chain Reaction, reação em cadeia da polimerase) consiste numa reação em que uma região pequena e específica do genoma é amplificada por síntese, pela polimerase de DNA. A reação em cadeia da polimerase possibilita a amplificação de uma seqüência rara de DNA a partir de uma mistura complexa, sem a necessidade de clonagem molecular. Esta técnica é amplamente utilizada em pesquisa básica, em medicina forense e no diagnóstico de doenças genéticas e infecciosas.
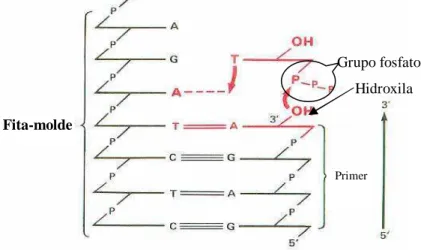
Inicialmente, é necessária a construção por síntese química de dois oligonucleotídeos de DNA ou primers (iniciadores) complementares, as extremidades de cada fita de DNA, flanqueando a região de interesse. Estes oligonucleotídeos servem como iniciadores da síntese de DNA in vitro, que é catalisada pela DNA polimerase, devido ao primer fornecer uma extremidade de hidroxila livre onde a DNA polimerase catalisará a reação deste composto com o grupo fosfato de um nucleotídeo correspondente à base nitrogenada da fita-molde (vide Figura 2.2).
Um ciclo de PCR começa com a desnaturação por calor (95°C), promovendo a separação da fita dupla de DNA. A reação é resfriada na presença de um excesso dos dois oligonucleotídeos, possibilitando a hibridização dos dois iniciadores com a seqüência complementar presente no DNA alvo. Em seguida, a reação é incubada para atividade da DNA polimerase, produzindo
novas fitas de DNAs a partir dos iniciadores e utilizando quatro desoxirribonucleotídeos (dATP, dCTP, dGTP e dTTP) (vide Figura 2.6) [24].
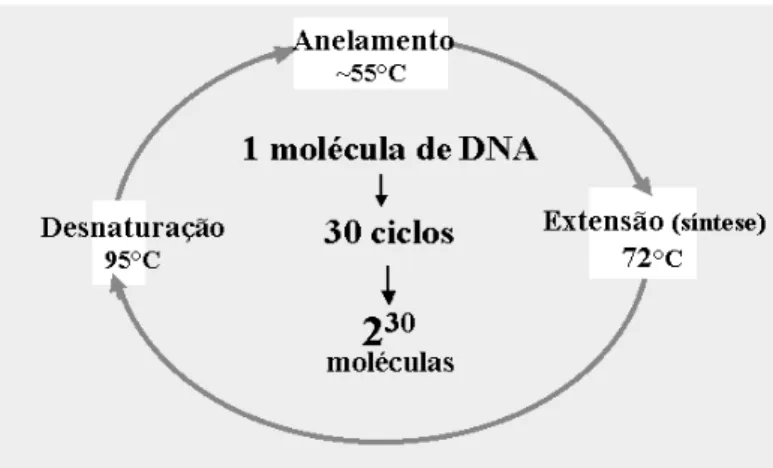
DNA polimerase a partir dos iniciadores, sendo que as fitas de DNA recém sintetizadas servem de molde no ciclo seguinte. Portanto, em cada ciclo é sintetizado o dobro do DNA produzido no ciclo anterior. A Figura 2.3 mostra no terceiro ciclo, duas duplas fitas que apresentam o tamanho correto sendo copiadas (as duas fitas com o mesmo tamanho). No quarto ciclo, 8 duplas fitas que apresentam o mesmo tamanho são copiadas. Usualmente, são realizados entre 20 e 30 ciclos
para amplificação de um segmento de DNA específico dentro de um genoma (Figura 2.4) usando uma máquina termocicladora (máquina que varia a temperatura da PCR em segundos, vide Figura 2.5) e os ingredientes dentro de um tubo onde ocorre a reação mostrada na Figura 2.6.
Figura 2.2: Primer na
fita-molde com hidroxila livre e nucleotídeo com fosfato livre
Grupo fosfato Hidroxila
Fita-molde
Figura 2.4: Etapas de PCR (amplificação exponencial em 30 ciclos)
Figura 2.5: Termociclador
Nas primeiras iniciativas para amplificar fragmentos de DNA, utilizava-se a enzima DNA
polimerase da Escherichia coli, que possui atividade máxima a 37°C. Esta enzima deveria ser adicionada a cada ciclo, pois o passo de desnaturação inativa a enzima. Um importante avanço ocorreu com a descoberta de a enzima Taq DNA polimerase [25] oriunda da bactéria Thermus aquaticus. A Taq DNA polimerase possui atividade ótima a 72°C e permanece razoavelmente
Capítulo 3
O Problema do Projeto e Escolha de Primers
Viroses de plantas constituem um sério problema que afeta a produção de plantas tropicais. No caso de algumas plantas como o mamão, por exemplo, eles são responsáveis pelo abandono da
área contaminada pelo PRSV (Papaya Ring Spot Virus), PMV (Papaya Mosaic Virus) ou PMeV (Papaya Meleira Virus) [27], como tem recentemente ocorrido no Brasil. Outros vírus de planta poderiam ser citados aqui que apresentem as mesmas conseqüências. O controle de vírus de planta empregado em produção de plantas é baseado na identificação dos sintomas da virose que são visíveis quando a contaminação é potencialmente um risco para outras plantas. Estas plantas
são então eliminadas da plantação e normalmente queimadas.
A identificação prévia do vírus de planta pelas técnicas da biologia molecular representa uma maneira eficiente para prevenir contaminações de vírus em grande escala e pode ser empregada em muitas situações como a triagem de plantas importadas pelas instituições de segurança de plantas. Estas técnicas moleculares estão baseadas na PCR que permite a amplificação de seqüências de vírus específicos do DNA da planta afetada.
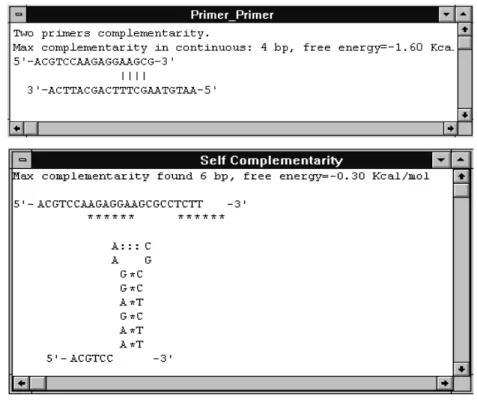
Reações de PCR específicas são mais eficientes usando oligonucleotídeos normalmente com um tamanho entre 16 e 30 bases [05]. Estes primers específicos são escolhidos concordando com seqüências conhecidas presentes no DNA amplificado do genoma, que neste caso são regiões do genoma do vírus. Alguns critérios que permitem uma boa escolha de primers devem ser considerados. É importante, por exemplo, evitar as seguintes falhas: formação de dímeros de primers, auto-complementariedade (vide figura 3.1), temperatura de fusão muito baixa, e/ou estabilidade interna incorreta.
escolha de primers específicos para a identificação de vírus de plantas para serem usados no diagnóstico de viroses deveria considerar, tendo em vista os critérios gerais acima, a complexidade de famílias de vírus de plantas e diferentes espécies. Portanto, a construção do primer é o principal fator a ser considerado e todos os outros parâmetros devem ser considerados na sua construção. Por isso, esta abordagem computacional é eficiente para a identificação de vírus de plantas.
❯ ❱❲
❳ ❨ ❩ ❭ ❪ ❫ ❪ ❵ ❛ ❜ ❫ ❝ ❞ ❝ ❪ ❢ ❜ ❣
A escolha de primers pelo processo tradicional [07] é mais propícia a erros, pois, neste processo, o pesquisador utiliza várias ferramentas diferentes para a escolha do par de primers, conseqüentemente sofre a demora do processo por depender da Internet, e nem sempre os primers escolhidos garantem a eficiência ou especificidade do diagnóstico. Isso acarreta prejuízo com o gasto de primers ineficientes e demora no diagnóstico.
O processo tradicional de escolha de primers tem os seguintes passos:
1. O pesquisador deve procurar as seqüências do genoma do(s) vírus desejados em um banco de dados. O banco mundial de dados genômicos é o Genbank [42]. O processo de copiar a seqüência genômica de interesse do Genebank é demorado por ser bastante requisitado via Internet.
2. Em seguida deve-se fazer um alinhamento múltiplo com as várias seqüências genômicas suspeitas usando algum programa disponível na Internet. Isso é necessário para descobrir se existe uma ou mais regiões espécie-específicas.
3. Tal região deve ter um tamanho de no mínimo 150 bases, pois será amplificada na técnica de PCR pela escolha de dois primers. A visualização pela eletroforese torna-se mais eficiente quanto maior for esta região (vide Figura 3.2). Além do tamanho da região a ser amplificada,
o pesquisador deve também se preocupar em encontrar de 14 a 20 bases iniciadoras nas extremidades desta região, complementares a primers que tenham características similares. 4. Estas características similares devem ser calculadas cuidadosamente seguindo alguns
5. Em seguida deve-se comprar os oligonucleotídeos específicos para o patógeno, de acordo com as características estabelecidas, o que não garante o correto diagnóstico por PCR.
Figura 3.1: Exemplos de complementaridade inter- e intra-primers que resultaria em problemas. Telas de análises feitas usando o programa DNAMAN (Lynnon Biosoft, Quebec, Canadá).
❳ ❥ ❛ ❪ ♠ ❣ ♦ ❜ ❫ ♦ ❣ ❝ ❢ ✇ ❜ ♦ ❢ ❭ ❪
Mesmo usando o Algoritmo da Força Bruta [12], considerado um método simples, uma intensa modificação realizada foi no sentido de evitar comparações desnecessárias e melhorou em 75% a performance de tempo do programa.
O exemplo canônico do algoritmo de Força Bruta é associado com o problema do caixeiro viajante (TSP), um problema clássico NP-completo. Descreve um estilo de programação no qual o programador confia no poder de processamento do computador em vez de usar sua própria
inteligência para simplificar o problema.
Para que a programação do algoritmo de Força Bruta seja considerada boa, dependeria do contexto: se o problema não for terrivelmente grande, o tempo extra do processador gasto em uma solução pelo Algoritmo de Força Bruta pode custar menos do que o tempo que o
programador faria para desenvolver um algoritmo mais inteligente. Adicionalmente, um algoritmo mais inteligente pode implicar uma complexidade de longo prazo e custo de manutenção que se justificaria pela melhoria da velocidade.
Convém ressaltar que foram feitas algumas modificações no algoritmo de Força Bruta. A versão final atende perfeitamente a finalidade do programa, incluindo a especificação do problema e o tempo da solução.
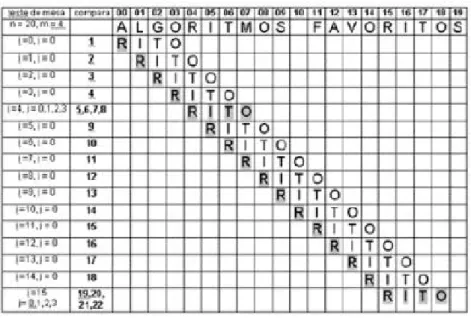
A interface executa um cruzamento de dados para obter a subseqüência, encontrando uma a ser comparada com a seqüência do vírus. A lista de informações seguintes tem que ser armazenada baseando-se em ocorrências de similaridade relativa: a identidade de seqüência comparada, posição inicial e tamanho da subseqüência, e a posição da ocorrência. Veja Diagrama 4.2 para mais detalhes sobre o sistema.
❯ ❱✐ ❱❲
❣ ④ ❪ ❛ ❝ ❭ ❪ ❫ ♦ ⑨ ❪ ❛ ❶ ❜ ❷ ❛ ❸ ❭ ❜
O algoritmo de Forca Bruta é o algoritmo mais simples dentre os algoritmos de alinhamento. Sua simplicidade resulta na sua baixa performance em velocidade, devido ao grande número de comparações, às vezes sem necessidade.
O algoritmo funciona através de indexações na subcadeia B [26] (de tamanho m, indexada por
seqüencialmente cada caracter da cadeia-alvo com o caracter igualmente indexado da subcadeia, enquanto forem iguais, ou até que se chegue ao fim da subcadeia (nesse caso, o casamento de cadeias ocorreu na posição Índice A, e teve início na posição Índice A - m). Caso o fim da subcadeia não tenha sido alcançado, significa que foi encontrado algum caracter diferente na cadeia principal, e então deve se reiniciar todo o processo de comparação, posicionando Índice B no início da subcadeia, e incrementando Índice A de uma posição, como segue no algoritmo apresentado abaixo:
❺ ❻❼ ❻❾ ❻ ❾
❳ ❣ ④ ❪ ❛ ❝ ❭ ❪
➀ ➁➂ ➄ ➅ ➆➇➈ ➄ ➊ ➄ ➅➋ ➍ ➏ ➑ ➅ ➓ ➇➍ ➔➀ → ➣ → ➑ → ➈ ➙ ➛
➜ ➣ ➇ ➅ ➍ ➞ ➍ ➟ ➀ ➔➢ ➇➅ ➆➣ ➂ ➞ ➦ ➇➍ ➈ ➍ ➣ ➧ ➄ ➣ ➙ ➦ ➑ ➔➫ ➇ ➅ ➆➣ ➂ ➞ ➦ ➇➍ ➈ ➍ ➣ ➧ ➄ ➈ ➙ ➫ ➍ ➭➞ ➍ ➟ ➫ ➇➍ ➅➇ ➔ ➲ ➅ ➆➈ ➦ ➆➅ ➍ ➄ ➵ ➄ ➅ ➅➺ ➣ ➵ ➆➍ ➞ ➦ ➑ ➦ ➈ ➀ ➙
➽➣ ➆➵ ➆➄ ➆ ➟➚ ➶ ➛ ➫ ➇ ➍ ➅ ➇ ➟➚ ➘ ➛
➦ ➣ ➷ ➓ ➍ ➣ ➇➄ ➔ ➆➮ ➚ ➣ ➙ ➦ ➔➫ ➇➍ ➅➇ ➚ ➘ ➙ ✃ ➍ ➋ ➍ ❐ ➟➚ ➆➛
❒ ➟➚ ➶ ➛
➦ ➣ ➷ ➓ ➍ ➣ ➇➄ ➔ ➑ ❮❒ ❰ ➚ ➀ ❮❐ ❰ ➙ ➦ ➔❒ ➮ ➚ ➈ ➙ ✃ ➍ ➋ ➍ ❒ ➟➚ ❒ z ➶ ➛
❐ ➟➚ ❐ z ➶ ➛ ➊ ➆➈ ~ ➦ ➣ ➷ ➓ ➍ ➣ ➇➄ ➛ ➫ ➦ ❒ ➚ ➈ z ➶ ➦ ➣ ➇ ➄ ➫ ➇ ➍ ➅ ➇ ➟➚ ❐ ~ ➈ ➛
➽ ➟ ➚ ➆ z ➶ ➛ ➊ ➆➈ ~ ➦ ➣ ➷ ➓ ➍ ➣ ➇➄ ➛ ➊ ➆➈
algoritmo e a segunda traz o número de comparações efetuadas. Portanto, após 22 comparações, o padrão P foi encontrado no texto T, iniciando-se no índice i = 15.
Essas duas comparações seqüenciais e alinhadas são responsáveis pela complexidade quadrática O(m x n) para o pior caso (não encontrar B em A), já que o primeiro laço varre toda a cadeia A (n interações para o pior caso), e para cada caracter de A, é feita uma varredura em B
(m interações para o pior caso).
Esse algoritmo pode ser facilmente mapeado para um algoritmo de busca numa matriz, onde as colunas são os caracteres de A, e as linhas os caracteres de B, e a simples necessidade de varrer toda a estrutura matricial para o pior caso dá-se a complexidade quadrática. Para o melhor
caso, a complexidade é O(1), e no caso médio O(n).
A garantia de que o algoritmo funciona está no fato de que todas as subcadeias possíveis na cadeia-alvo são comparadas até que se encontrem caracteres diferentes nas mesmas.
Por outro lado, o ponto fraco do algoritmo reside nas indexações seqüenciais, e no excesso de comparações desnecessárias realizadas no percurso ao longo da cadeia-alvo. Considerando-se que essa cadeia alvo é um texto de grande porte, como uma revista, ou mesmo um livro, não se poderia dar ao luxo de realizar tamanho número de operações desnecessárias. As modificações do Algoritmo de Força Bruta foram ao sentido de evitar essas operações desnecessárias, e a performance alcançada é satisfatória para o alinhamento de seqüências nucleotídicas de até 40000 bases de tamanho.
Figura 3.3: Funcionamento do Algoritmo de Força Bruta
no caso médio. Ressaltamos que o algoritmo usando Força Bruta pode ser empregado nesta ferramenta, pois dificilmente ela cairia no pior caso que é de O(nm).
Gráfico 3.1: Gráfico de Tempo de Execução de Força Bruta
❯ ❱❯
❳ ❥ ❛ ❪ ♠ ❣ ♦ ❜ ❫ ❜ ❪ ❢ ❭ ❛ ❸ ❶ % ❪ ❫ ♦ ❥ ❛ ❝ ♦ ❛
Existem vários fatores que podem afetar a técnica PCR como: temperatura de desnaturação, número de ciclos, tempo de extensão, uso de primers degenerados, comprimento do primer, temperatura de anelamento. Dependendo destes fatores a PCR pode reagir melhor. Portanto, a construção do primer é o principal fator a ser considerado e todos os outros parâmetros deveriam ser considerados na sua construção.
Para resolver o problema vários fatores que podem afetar a técnica PCR foram tratados, como: temperatura de desnaturação, número de ciclos, tempo de extensão, uso de primers degenerados, comprimento do primer, temperatura de anelamento.
❯ ❱❯ ❱❲
❵ ♦ 3 ♦ ❛ ❜ ❭ ❸ ❛ ❜ ❫ ♦ ❫ ♦ ❢ ❜ ❭ ❸ ❛ ❜ ❶ % ❪ > ❵ A ♦ ❭ ♦ 3 ❪
assegura que as fitas separadas ou desnaturadas não reanelarão ou juntar-se-ão. Adicionalmente, se o ácido nucléico é aquecido num tampão de força iônica menor do que 150mM de cloreto de sódio, a temperatura de fusão é geralmente menor do que 100ºC – isso é o porquê que na PCR se trabalha com temperaturas de desnaturação (Tm) entre 91 à 97ºC.
Sabe-se que a enzima Taq polimerase tem uma meia vida de 30 minutos à 95ºC, por isso, uma enzima não deve fazer mais do que 30 ciclos de amplificação. Entretanto, é possível reduzir a temperatura de desnaturação depois de aproximadamente 10 ciclos de amplificação. Como o principal comprimento do DNA alvo é diminuído, por cópias de 300 pares de bases ou menos, a temperatura de desnaturação pode ser reduzida a 88ºC por cópias com 50% (G+C) [10], ou seja, uma pode desempenhar sua função um pouco mais de 40 ciclos sem muita diminuição da eficiência da enzima.
O tempo em uma determinada temperatura é a razão principal para a desnaturação ou perda da atividade da Taq polimerase. Desta forma, se uma reduz isto, o número de ciclos possíveis pode ser aumentado se a temperatura é reduzida ou não. Normalmente o tempo de desnaturação é 1 minuto à 94ºC. Então é possível, para seqüências de copias curtas, reduzir isto para 30 segundos ou menos. Aumentando a temperatura de desnaturação e diminuindo o tempo pode também funcionar. Innis e Gelfand [5] recomendou 96ºC a 15 segundos.
❯ ❱❯ ❱✐
❵ ♦ ♦ ❛ ❜ ❭ ❸ ❛ ❜ ❫ ♦ ❢ ♦ ❣ ❜ ♦ ❢ ❭ ❪ & ❵ ❜ * ♦ ❞ ❪ ❢ . ❭ ❛ ❸ ❶ 3 ❪ ❫ ♦ ❛ ❝ ♦ ❛
O comprimento do primer e a seqüência são de importância crítica na configuração dos parâmetros de uma amplificação bem sucedida: a temperatura de fusão ou temperatura de anelamento de uma dupla hélice de DNA aumenta com o seu comprimento, e com o aumento do conteúdo (G+C). Uma fórmula simples para calcular o Tm é dada por [9]:
Tm = 4(G + C) + 2(A + T)ºC.
verdadeiro, então as más combinações internas de única-base ou anelamento parcial podem ser tolerados. Isto é bom para amplificar alvos similares ou relacionados. Entretanto, pode conduzir à amplificação "não-específica" e à redução conseqüente no rendimento do produto desejado se a maioria das bases 3' for emparelhada com um alvo.
Uma conseqüência da Ta muito elevada é que uma quantidade muito pequena do produto será produzida, assim como a probabilidade de um anelamento de primer é reduzida. Uma outra consideração importante é que um par de primers com temperaturas muito diferentes de anelamento pode nunca dar rendimentos apreciáveis de um produto único, e pode também resultar em inadvertida amplificação "assimétrica" de fita simples da fita produto mais eficientemente anelada ao primer.
O anelamento não demora muito tempo. A maioria de primers anelarão eficientemente em 30 segundo ou menos, a menos que Ta seja muito próxima da Tm, ou a menos que sejam atipicamente longos.
O comprimento melhor de um primer depende de seu conteúdo (A+T) e do Tm de seu parceiro, se um tem o risco de ter problemas tais como descrito nos parâmetros anteriores. Fora o Tm, uma consideração principal é que os primers devem ser complexos o bastante de modo que a probabilidade do anelamento com as outras seqüências à exceção do alvo escolhido seja
muito baixa. Por exemplo, há uma possibilidade de ¼ (4-1) de encontrar um A, um G, um C ou um T em toda a seqüência dada do DNA; há uma possibilidade 1/16 (16-1) de encontrar qualquer seqüência dinucleotídica (por exemplo: AG); uma possibilidade de 1/256 de encontrar uma dada seqüência de 4-bases. Assim, uma seqüência de dezesseis bases estará
Para a amplificação de seqüências cognatas de organismos diferentes, ou para "PCR evolucionário", um pode aumentar as possibilidades de obter produto construindo primers "degenerados". Estes seriam, de fato, um grupo de primers que têm a possibilidade de anelar em diversas posições na seqüência e amplificar uma variedade de seqüências relacionadas. Para o exemplo, Compton [4] usa grupos de primers de 14 bases com 4 e 5 degenerâncias tanto primers no sentido direto e reverso, respectivamente, para a amplificação da glicoproteína B (gB) de vírus de herpes. A seqüência reversa do primer usada é como se segue:
TCGAATTCNCCYAA
Figura 3.4: Seqüências de primers foram derivadas dos alinhamentos múltiplos de seqüência A Figura 3.5 mostra as posições de má-combinação que foram usadas como degenerâncias de 4-bases para os primers (mostrados como asteriscos; 5 em F e 4 em R).
Apesar da degenerância, os primers podiam ser usados para amplificar uma seqüência de 250 bp de vírus, diferindo na seqüência tanto quanto acima de 50% for a seqüência alvo, e 60% total. Poderiam também ser usados para detectar muito sensivelmente a presença do DNA do vírus da estria do milho contra o DNA genômico do milho, nas diluições abaixo de 1/109 da seiva
infectada.
" $
Esta é normalmente 70 - 72ºC por 0,5 - 3 minutos. A enzima Taq polimerase tem realmente uma atividade específica em 37ºC que é muito perto daquela do fragmento de Klenow da polimerase I do DNA de E coli, que explica o aparente paradoxo que resulta quando um tenta compreender como os primers que anelam em uma temperatura ótima podem então ser alongados em uma temperatura consideravelmente mais alta. A resposta é que a elongação ocorre do momento do anelamento, mesmo se este é passageiro, que resulta em uma estabilidade consideravelmente maior. Em torno de 70ºC a atividade é ótima, e uma extensão do primer ocorre em até 100 bases por segundo. Aproximadamente 1 minuto é suficiente para a amplificação fidedigna das seqüências de 2 kb [5]. Produtos mais longos requerem tempos mais longos: 3 minutos é uma boa medida para 3kb e produtos mais longos. Tempos mais longos podem também ser úteis em ciclos mais atrasados quando a concentração do produto excede a concentração da enzima (> 1nM), e quando dNTP e/ou esgotamento do primer pode se tornar limitante.
Concentrações maiores do que 50mM de cloreto de potássio ou cloreto de sódio inibe a Taq,
as atenuações forem feitas pelos dNTPs, primers e fita molde, todos os quais são quelados e seqüestrados o cátion; destes, os dNTPs são os mais concentrado, assim que [ Mg2+ ] estivesse entre 0,5 - 2.5mM maior do que a concentração de dNTP. Uma titulação deve ser executada com variação da concentração de Mg2+ com todas as combinações novas da fita-molde-primer, como estas podem diferir marcantemente em suas exigências, ainda sob as mesmas condições das concentrações e ciclagem de tempos/temperaturas.
Algumas enzimas não necessitam adicionar proteína, outras são dependentes dela. Algumas enzimas trabalham marcantemente melhor na presença de detergente, provavelmente porque evita a tendência natural da enzima se agregar.
Concentrações de primer não deveriam ir acima de 1uM a menos que houvesse um grau elevado de degenerância; 0.2uM é suficiente para primers homólogos. A concentração do nucleotídeo não necessita estar acima de 50uM cada. Entretanto, os produtos longos podem requerer mais.
✂ ✂ ✆
O número dos ciclos de amplificação necessários para produzir uma banda visível no gel de eletroforese depende em grande parte da concentração inicial do DNA alvo: Innis e Gelfand [5] recomendam de 40 - 45 ciclos para amplificar 50 moléculas alvo, e de 25 - 30 para amplificar as moléculas 3x105 à mesma concentração. Esta desproporcionalidade é devido a um efeito platô
bem conhecido, que é a atenuação na taxa exponencial da acumulação do produto em estágios tardios de uma PCR, quando o produto alcança 0,3 - 1,0 nM (nanomolar). Isto pode ser causado pela degradação dos reagentes (dNTPs, enzima); esgotamento de reagente (primers, dNTPs -formam um problema com os produtos curtos, e tardios para produtos longos); inibição do
Capítulo 4
Uma Solução Computacional
Pelo exposto nos Capítulos anteriores, o projeto de primers é fundamentalmente importante em métodos de detecção baseados em PCR. Os critérios gerais para primers são muito simples. Contudo, é difícil escolher primers bons para uma dada seqüência do molde. Não são somente os cálculos. O mecanismo de alinhamento para escolher a região espécie-específica também é muito sofisticado. Portanto, a ajuda computacional na construção de primer é uma tarefa crítica na Bioinformática.
Há diversos serviços na Internet ou software autônomo fornecidos ao público para construção de primers, tal como PRIDE (35), PRIMER MASTER (36), PRIMO (37), PrimeArray (38), Primer3 (34), Prime (39) e Web Primer (http://genome-www2.stanford.edu/cgi-bin/SGD/web-primer). Os usuários podem definir os parâmetros listados no menu destas ferramentas e então obter diversos pares de primers para a seqüência do molde alvo. Entretanto, a maioria deles somente analisa uma única seqüência de pesquisa. A proposta deste trabalho é definir, por alinhamento de várias seqüências nucleotídicas, uma região ideal para construção de primers. Assim, considerando as limitações descritas no item 3.3 desta dissertação, um grupo de regras pode ser derivado para garantir a especificidade do diagnóstico:
1) Os primers devem ser 18 a 25 bases de comprimento; 2) A composição de base deve ser 50 a 60% (G+C);
3) Temperaturas de desnaturação entre 55 à 80ºC são preferidas;
4) Extremidades 3' de primers não deve ser complementar com outro primer, caso contrário dímeros de primers serão formados preferencialmente a qualquer outro produto. A
auto-complementaridade de primer (habilidade de formar estruturas secundárias tais como frisos) deve ser evitadas (veja Figura 3.1);
5) O produto amplificado por PCR indicado pelo par de primers não deve exceder 1200 pares de bases de comprimento;
Quanto maior for o primer, maior será sua especificidade. Apesar disso, há problemas na PCR com a construção de primers muito extensos. As variações de comprimento de primer de 18 a 25 nucleotídeos são aconselhadas e a configuração padrão do programa é 19. Em primers de tamanho acima de 25 bases há maior probabilidade de formar estruturas secundárias (frisos pela auto-complementaridade) ou dímeros entre o par de primers. Portanto, para diagnóstico, experimentos apontam que os primers não devem exceder de 25 bases.
A composição de bases é importante devido às pontes de hidrogênio que conferem maior estabilidade no anelamento entre citosina e guanina do que em adenina e timina. Por isso um conteúdo de CG estável deve ser a partir de 50% e não pode exceder de 60% devido a elevada reatividade destas bases nitrogenadas em parearem-se formando dímeros entre primers. No
programa proposto a configuração padrão do conteúdo de GC do par de primers varia de 50% a 60%. Primers com Tm menor do que 50ºC são excluídos.
A diferença aceitável de valores de Tm em um par de primers é de 5ºC. Critérios básicos para a seleção de Tm de primer. A temperatura de anelamento (Ta) é determinada pela temperatura de desnaturação (Tm). A temperatura de desnaturação é o parâmetro mais importante porque o termociclador ao elevar e diminuir a temperatura muito rapidamente afeta também os primers que devem ser capazes de anelarem-se a partir de 54 ºC e não deve passar de 80ºC para que possibilite a desnaturação necessária na PCR.
Deve-se ter cautela com a distribuição de nucleotídeos formar um anagrama de bases bem variado, principalmente nas extremidades. Ao usar um par de primers para diagnóstico deve-se preocupar-se se ocorre inter-complementaridade entre os dois primers. Esse cuidado também é nas extremidades porque em um primer também se pode formar estruturas secundárias como um friso ou um círculo devido a complementaridade das extremidades.
O par de primers são os limites da região que vai ser amplificada ou copiada várias vezes. É necessário estabelecer um tamanho mínimo de 100 bases, pois a partir deste tamanho o fragmento pode ser melhor visualizado em um gel de eletroforese. O tamanho máximo deve ser de 1200 bases porque a partir deste tamanho a polimerase terá diminuição de performance. Em um minuto a polimerase é capaz de encadear até 2000 bases.
Há uma preocupação do par de primers possuírem parâmetros como Tm e conteúdo GC similares, devido à variação de temperatura que são submetidos.
O software desenvolvido para diagnóstico de viroses em plantas, ajuda em selecionar primers
O sistema computacional na sua primeira versão consiste de dois módulos: um módulo para alinhamento das seqüências nucleotídicas e separação das regiões polimórficas, outro módulo para construção de primers usando os melhores parâmetros de primers eficientes no diagnóstico por padrão ou configurados pelo pesquisador.
A Tabela 4.1 pode resumir os tratamentos dados aos problemas solucionados neste trabalho.
PROBLEMAS ATUAIS SOLUÇÕES DO SISTEMA Prejuizo no agronegócio do RN e Brasil; Análise prévia do diagnóstico por PCR e
melhor tratamento; Busca de genomas em banco de dados
(demorado);
Busca mais rápida, devido banco de dados localmente instalado;
Alinhamento múltiplo (demorado, falho); Alinhamento mais rápido do que na Internet, correto;
Escolher região a ser amplificada (manual); Escolha automática rápida detalhada (parâmetros);
Parâmetros controlados de modo ineficiente; Controle mais eficiente para diagnóstico;
Problema performance de tempo do algoritmo. Modificações evitando comparações desnecessárias.
Tabela 4.1: Soluções do Sistema Dadas aos Diversos Problemas Abordados
✞
✠ ✡
O objetivo do sistema é automatizar o processo tradicional de escolhas de primers espécie-específicos. O sistema tem dois módulos: um módulo para alinhamento de seqüências e outro para projetar ou construir primers. Este sistema deve ser modelado orientado a objeto usando UML [48]. No processo de desenvolvimento do software orientado a objetos é necessário ter
uma visão mais ampla do que a oferecida pelo modelo estático do sistema, pois este não reflete a mudança dos objetos e seu comportamento através do tempo [47].
Os bancos de dados de arquivos simples são o tipo de banco de dados que os não-especialistas entendem com mais facilidade. Um banco de dados de arquivos simples não é realmente um
A proposta inicial do sistema atendeu as expectativas com um índice unidimensional em um banco de dados de arquivos simples indexado. A coleção de arquivos texto tem o formato Fasta (vide figura 4.1). Muitos usuários de dados de seqüências biológicas armazenam e acessam as seqüências localmente, utilizando o Sequence Retrieval System (SRS), um sistema de indexação de arquivos simples projetado para utilização com dados biológicos.
Figura 4.1: Formato Fasta
O sistema não tem controle de acesso por senha ou preocupações maiores de segurança por não estar acessível pela Internet, e sim localmente. No entanto, faz-se necessário criar um sistema de segurança do banco de dados para desenvolvimento, manutenção, e prevenção de problemas futuros. O sistema sempre estará em fase contínua de desenvolvimento e outros
Analisando o sistema tradicional de escolha de primers espécie específicos constatamos os seguintes problemas:
• A busca em banco de dados públicos, o alinhamento de genomas e a construção de primers são demorados por depender do tráfego via Internet.
• Utiliza-se vários programas diferentes para obter os primers espécie-específicos.
• Cada etapa do processo implica em copiar e colar resultados manualmente, isso pode gerar algum erro.
• A visualização dos dados do processo é menos compreensível, visto que tem interfaces diferentes sendo utilizadas.
• O alinhamento múltiplo usado em um programa público na Internet demora e pode dar dados imprecisos.
O sistema proposto resolve todos esses problemas do método tradicional:
• O processo de construção de primers espécie-específicos é automatizado portanto não é necessário utilizar vários programas, e o tempo de resposta em comparação ao método tradicional é mais rápido.
• Utiliza-se apenas a interface de um programa para obter os resultados.
• Não é necessário copiar e colar manualmente os dados. O fluxo de dados é dirigido para obter os resultados.
• O processo completo para obter os primers espécie-específicos é melhor compreendido, por ter uma única interface, e por ser uma interface compreensível facilitando a visualização dos dados sendo processados ou transformados. E, pode-se voltar etapas do processo para entender o processamento.
• O alinhamento múltiplo de seqüências até 40000 bases de comprimento tem no pior caso um tempo de resposta inferior a três minutos.
As regiões espécie-específicas são processadas em um grupo de regras ou parâmetros otimizados para desenhar primers. No entanto, o usuário tem a liberdade de decidir o tamanho do fragmento que quer amplificar, o tamanho do primers, a média do conteúdo de C+G, e a temperatura de
desnaturação antes do processamento das regiões espécie-específicas para desenhar os primers. Nos resultados finais ou saída do programa, pode-se escolher em uma lista de primers fornecida pelo programa, um par de primers limítrofes de parâmetros similares.
Diagrama 4.1: Acessibilidade do Banco de Dados
Diagrama 4.2: Arquitetura do Sistema
Processando…
Arquivos de vírus de planta
Seqüência de virus query
Detalhando… Arquivo .txt Formato Fasta
BANCO DE DADOS
Infraestrutura de Bioinformática
GERENCIAMENTO DA INFORMAÇÃO
ENTRADA: Seqüência query
Cálculos do pipeline
SAÍDA:
Lista de Primers de vírus / seqüência. Ex.: 3’5’| Tm |C+G| 5’3’ |
AGTC|54 ºC |52%|TCAG
Ambiente de mineração de dados
Região espécie-específica da seqüência
query 3’5’ Ex.: (XXAGTCXX…)
Dados de vírus de planta
VírusPlanta IrParaSeqüênciaVírus
USUÁRIO BANCO DE DADOSControle de Acesso
PROGRAMA
✠ ☛ ☞ ✆ ✎ ✆ ✒
Inicialmente, desenvolveu-se um módulo de alinhamento (vide Diagrama 4.3), que aceita como entrada arquivo texto com seqüências nucleotídicas em formato Fasta de vários comprimentos e fornece como saída: domínios com similaridades e domínios espécie-específicos. Um domínio espécie-específico é uma seqüência do genoma viral que está presente somente em uma das espécies como resultado de uma comparação entre genomas.
O alinhamento é feito usando o algoritmo de Força Bruta modificado, usando uma string ou
tamanho de palavra configurado pelo usuário. A primeira seqüência escolhida no banco de dados é a seqüência query, ou seja, é a seqüência que será fragmentada em várias strings e comparada com as seqüências subseqüentes de modo global. O resultado do alinhamento será mostrado na seqüência query. Depois, pode-se fazer um detalhamento do resultado indicando as
regiões universais e as regiões espécie-específicas.
Diagrama 4.3: Diagrama do Módulo de Alinhamento
✞ ✓
✠ ☛ ☞ ✆ ✕ #
exibida com seus respectivos parâmetros para ajudar o usuário a minimizar primers ineficientes, e estabelecer o tamanho do fragmento de DNA que deve ser amplificado na reação de PCR da
Biologia Molecular. Os parâmetros otimizados já foram listados no conjunto de regras derivados para garantir a especificidade do diagnóstico no início deste capítulo.
A idéia da aplicação é usar regiões espécie-específicas de genomas virais para construir primers de alta especificidade para diagnosticar vírus de plantas usando PCR. Primers devem
estar prontos para anelarem-se ao ácido nucléico alvo em uma localização prevista e ser estendida pela enzima Taq Polimerase.
O resultado é uma lista de primers com seus respectivos parâmetros. Os parâmetros mostrados são: tamanho, porcentagem do conteúdo de C+G, temperatura de desnaturação, e primer complementar. O primer complementar é a versão 5’-3’ do primeiro primer listado. No par de primers deve necessariamente ser um no sentido 3’-5’ e outro no sentido 5’-3’ de primers diferentes e de distância conhecida.
✕ ✘ ✆ ✛ ✢ ✛ ✂
A ferramenta usada no desenvolvimento do sistema foi o Borland Delphi 5.0, para ambiente Windows. A escolha dessa ferramenta decorreu do fato de a mesma ser visual e orientada a objetos, bem como possuir boa integração com aplicações de bancos de dados. Ambos os fatos contribuíram para a diminuição do tempo de desenvolvimento, ao evitar a preocupação com a API do Windows e o acesso às tabelas Paradox.
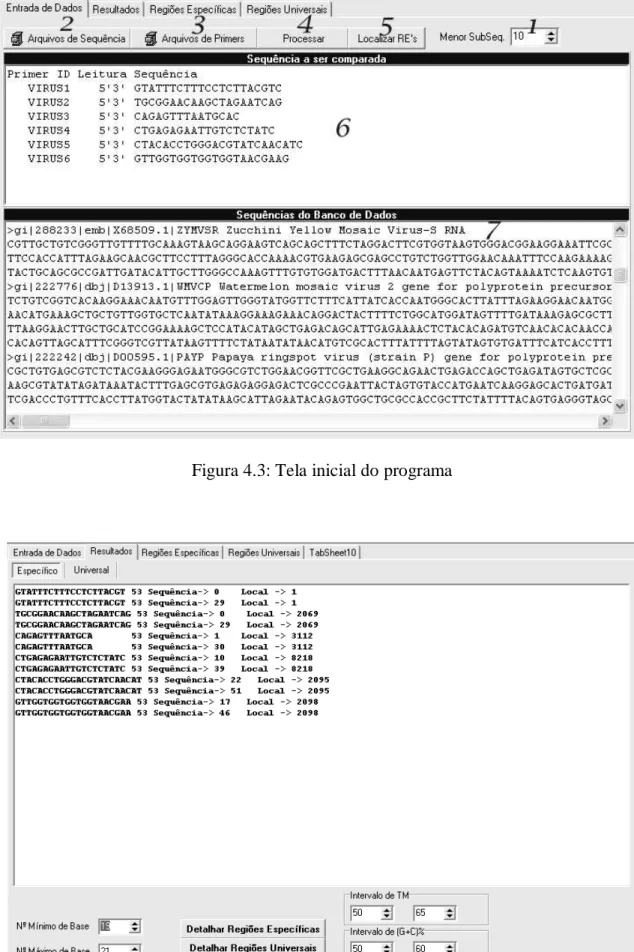
A interface é compreensível, pois é intuitivo o modo de utilizar o programa ao observar as telas. O programa permite que o usuário especifique o tamanho da string de busca ou subseqüência no alinhamento entre seqüências, como mostra o item 1 na tela apresentada na Figura 4.3. O tamanho de string padrão é 10.
Há na tela inicial do programa o botão de arquivos de seqüências (vide figura 4.3, item 2) para acionar a tela de busca do arquivo texto do sistema operacional e assim o usuário pode buscar os arquivos texto com seqüências de vírus em formato Fasta (vide figura 4.1). As seqüências são mostradas no item 7 da figura 4.3 e a primeira seqüência é a seqüência query ou seqüência de busca, onde todas as seqüências subsequentes serão comparadas, e onde os resultados serão mostrados.
Há também o botão de arquivos de primers (vide figura 4.3, item 3). Este botão ao ser acionado mostra a tela de busca do arquivo texto do sistema operacional, e o usuário pode escolher um arquivo texto mostrando primers, cada um já escolhido para um respectivo vírus
(vide figura 4.2). Este arquivo de extensão .txt não tem seqüências no formato Fasta, mas lista primers em texto padronizado para o programa proposto. E mostra os primers específicos de vírus no item 6 da figura 4.3.
O botão Processar mostrado na figura 4.3 no item 4 serve para verificar se os primers são
realmente específicos para as seqüências de vírus adicionadas no programa. Ao acionar este botão uma tela de resultados será exibida mostrando em que ordem de seqüência nucleotídica viral se encontra o primer e sua respectiva posição de base inicial (vide figura 4.4).
(vide figura 4.6). Na tela de resultados (Figura 4.5) são mostradas também as configurações dos primers que serão desenhados. As configurações que podem ser modificadas pelo usuário são: número mínimo de bases, número máximo de bases, intervalo de temperatura de desnaturação (Tm) em graus Celcius, e intervalo de porcentagem de conteúdo G+C. Há tambem na leta de resultados dois botões; um para detalhar as regiões específicas, e outro para detalhar as regiões universais. Ao ser acionado um dos botões ele mostrará uma outra tela (vide figura 4.6) com os primers desenhados de acordo com as configurações estabelecidas na tela de resultados.
A tela de primers específicos mostrada na figura 4.7 mostra as regiões específicas detalhadas
com um número em ordem crescente no sentido 5’-3’, a posição da base inicial e a posição da base final na primeira seqüência viral, e o comprimento da região espécie-específica em número de bases. Ao selecionar um tamanho de região específica, mostrará nesta mesma tela os respectivos primers que podem ser desenhados nesta região de acordo com as configurações estabelecidas. Cada primer de uma região espécie-específica é listado com sua respectiva: seqüência de bases representadas em letras, temperatura de desnaturação em graus Celcius (Tm), porcentagem do conteúdo G+C, tamanho em número de bases, posição na região espécie específica, e o primer complementar. Ao selecionar a seqüência de bases do primer nesta seção, será mostrada na seção Seqüência, as bases pintadas na cor verde e toda a região especie específica pintada na cor vermelha. O usuário pode copiar a seqüência verde. Para saber o tamanho de fragmento de DNA que será copiado várias vezes na técnica e PCR para diagnosticar a espécie de virus, é preciso saber o resultado da subtração da posição inicial do primeiro primer e a posição final do segundo primer da primeira seqüência viral ou seqüência query.
Figura 4.3: Tela inicial do programa
Figura 4.5: Tela de resultados que mostra as regiões especificas
Capítulo 5
Experimentos e Resultados
No sistema desenvolvido, a adaptação do algoritmo de força bruta prolongada. A busca executa uma verificação em todas as seqüências em ordem para obter a subseqüência, usando um tamanho de seqüência de nucleotídeos configurada pelo usuário. Uma lista de subseqüências é armazenada baseada na ocorrência de similaridade relativa: ID da seqüência comparada, posição inicial e tamanho das subseqüências. A figura 4.1 ilustra o sistema final projetado com base em modificações propostas. Este sistema também pode ser usado para análise de polimorfismo.
Realizou-se testes com o Algoritmo de Força Bruta modificado para algumas seqüências de vírus selecionados e os resultados mostram que os primers não são ambíguos para cada seqüência. O tempo gasto para casar uma seqüência única de comprimento 40.000 bases (pior caso de teste) com outra seqüência de 1000 bases, usando uma string de busca de tamanho 10, foi de 2 minutos e 43 segundos, usando uma máquina com processador AMD Athlon 2,4 GHz, 256 MB de memória RAM. Executaram-se vários experimentos com o algoritmo, principalmente testando o tempo de execução com seqüências de tamanhos variados, em relação à versão original. A figura 5.1 mostra um resultado mais extensivo demonstrando a performance do algoritmo. No último caso, o banco de dados de seqüências cresceu, denotando dados mais precisos.
Foi realizado um experimento para algumas seqüências de vírus de planta selecionadas em
banco de dados público e os resultados mostram que os primers também não são ambíguos para cada seqüência. Na versão atual, executou-se uma comparação algorítmica do tempo com tamanhos variáveis da seqüência. O gráfico na figura 5.1 mostra que o programa proposto no pior caso (40.000 pares de bases de comprimento) tem uma performance tempo aproximado de 3
Performance de Tempo do Programa
720 140 37 293 163 157 120 105 94 74 60 43 30 14 0 300 600 900 12000 4 8 12 16 20 24 28 32 36 40
Núm ero de bases da seqüê ncia nucleotídica e m Kb (1000 bases)
Tempo (segundos)
Algoritmo de Força Bruta Algoritmo de Força Bruta Modificado
Gráfico 5.1: Gráfico de performance da última versão do algoritmo de Força Bruta
✞
✥
A simulação é para diagnosticar viroses de batata (Solanum tuberosum). Os genomas completos de viroses de Solanum tuberosum disponíveis no banco de dados de domínio público NCBI são sete, até 16 de setembro de 2004, como mostra na figura 5.3. Em seguida, obteve-se o formato Fasta de cada genoma de vírus da batata. Todos os sete genomas foram processados pelo programa proposto com o cuidado de manter as configurações padronizadas para todos. O alinhamento foi feito com uma string de tamanho 10, e em 25 segundos obteve-se o resultado usando o processador AMD Athlon 2,4 GHz, 256 MB de memória RAM. O programa usa o primeiro genoma da lista como alvo para alinhar os subseqüentes.
Para cada genoma, obteve-se um par de primers com características iguais (tamanho, Tm e conteúdo G+C) configuradas na tabela 5.1. Estas características estão otimizadas segundo Innis e Gelfand [05]. O tamanho do primer foi de 21 bases por ser mais específico e menos provável de formar problemas de complementaridade (vide figura 3.1). Quanto maior o primer mais específico ao alvo, porém também se considera que primers muito longos podem formar frisos com a intra-complementaridade. Tendo em vista isso, as extremidades dos primers foram
escolhidas procurando não formar dímeros entre o par de primers nem frisos intra-primer. A temperatura de desnaturação (Tm em ºC) escolhida foi 54,36 por ser um valor experimentalmente eficiente na reação. A porcentagem de guanina e citosina (% G+C) foi de 52.38% também estável experimentalmente no anelamento da reação de PCR. O tamanho do
fragmento do genoma do vírus amplificado por PCR foi entre 150 à 1200 pares de bases por ser um tamanho de extensão viável para a enzima polimerase expandir na reação. Este tamanho é limitado pelo par de primers escolhido.
Figura 5.1: Tela de resultados do Entrez no NCBI mostrando 7 genomas de vírus da batata.
Os pares de primers específicos obtidos de cada espécie de vírus também foram testados
usando o Blast-n do NCBI e comprova-se que são específicos em nível de espécie, em 16 de dezembro de 2004. E em todos os primers 5`-3` de viroses de batata, constata-se a especificidade com o vírus respectivo, porque o Blast-N alinhou no banco de dados com várias seqüências de genomas parciais e com o genoma completo de cada vírus analisado. Além disso, as outras
✥
O Rio Grande do Norte é o maior exportador de melão do Brasil. Neste cenário simula-se um experimento para diagnosticar viroses do meloeiro (Cucumis melo L). Os genomas completos de
viroses de Cucumis melo L disponíveis no banco de dados de domínio público NCBI são quatro, até 16 de setembro de 2004, como mostra na figura 5.11. Em seguida, obtive-se o formato Fasta de cada genoma de vírus do meloeiro. Todos os quatro genomas foram processados pelo programa proposto com o cuidado de manter as configurações padronizadas para todos. O alinhamento foi feito com uma string de tamanho 10, e em 13 segundos obteve-se o resultado usando um processador AMD Athlon 2,4 GHz, 256 MB de memória RAM. O programa usa o primeiro genoma da lista como alvo para alinhar os subseqüentes.
Para cada genoma obteve-se um par de primers com características iguais (tamanho, Tm e conteúdo G+C) configuradas na tabela 5.2. Estas características estão otimizadas segundo Innis e Gelfand [05]. O tamanho do primer foi de 21 bases por ser mais específico e menos provável de formar problemas de complementaridade (vide figura 3.1). Quanto maior o primer mais específico ao alvo, porém também se considera que primers muito longos podem formar frisos com a intra-complementaridade. Tendo em vista isso, as extremidades dos primers foram escolhidas procurando não formar dímeros entre o par de primers nem friso intra-primer. A temperatura de desnaturação (Tm em ºC) escolhida foi 54,36 por ser um valor experimentalmente eficiente na reação. A porcentagem de guanina e citosina (% G+C) foi de
52.38% também estável experimentalmente no anelamento da reação de PCR. O tamanho do fragmento do genoma do vírus amplificado por PCR foi entre 150 à 1200 pares de bases por ser um tamanho de extensão viável para a enzima polimerase expandir na reação. Este tamanho é limitado pelo par de primers escolhido.
Figura 5.9: Tela de resultados do Entrez no NCBI com 4 genomas de vírus de meloeiro.
Os pares de primers específicos obtidos de cada espécie de vírus também foram testados
Capítulo 6
Conclusão e Perspectivas
Eu obtive um diferencial em relação aos softwares existentes em domínio público e em domínio privado. O diferencial do sistema proposto está na estratégia para realizar um diagnóstico em nível de espécie de modo eficiente. A estratégia é procurar similaridades e não-similaridades nas regiões do genoma do vírus. Regiões onde ocorrem não-não-similaridades são regiões espécie-específicas, ou seja, os primers projetados a partir desta região serão específicos para apenas uma espécie suspeita. O sistema também possibilita ao usuário configurar a estringência do alinhamento, bem como os parâmetros de primers ótimos para o diagnóstico. Estringência é o número de bases da string ou palavra de busca (tamanho da subseqüência). A priori, o sistema tem uma configuração padrão baseada em medidas otimizadas da literatura
pesquisada.
A estratégia de escolher regiões específicas de uma espécie antes da construção de primers é eficiente para diagnosticar uma virose suspeita. A resposta avança então a metodologia de diagnóstico molecular em nível de espécie de vírus e de outros patógenos facilitando no processo
de tratamento. Além de beneficiar o controle de pragas na agricultura de importação e exportação, pode-se fazer um estudo de polimorfismo entre espécie e estudar a filogenia entre espécies de microorganismos.
![Figura 1.1 - Etapas do ciclo de PCR [41]](https://thumb-eu.123doks.com/thumbv2/123dok_br/15550185.94772/14.892.254.687.733.1050/figura-etapas-do-ciclo-pcr.webp)