CENTRO DE BIOCIÊNCIAS
Programa de Pós-Graduação em Biotecnologia - RENORBIO
Mapeamento cromossômico de genes ribossomais em espécies de peixes estuarinos da família Gerreidae (Pisces, Perciformes) – Identificação de uniformidade
cariotípica e sua empregabilidade com fins biotecnológicos - REPROGEN -
LEONARDO LUIZ CALADO
RENORBIO
Programa de Pós-Graduação em Biotecnologia Universidade Federal do Rio Grande do Norte
Centro de Biociências
Laboratório de Genética de Recursos Marinhos - L.G.R.M
Mapeamento cromossômico de genes ribossomais em espécies estuarinas da família Gerreidae – Identificação de uniformidade cariotípica e sua empregabilidade com
fins biotecnológicos - REPROGEN –
LEONARDO LUIZ CALADO
Tese apresentada ao programa de
pós-graduação em Biotecnologia -
RENORBIO/Universidade Federal do Rio Grande do Norte, como parte dos requisitos para obtenção do título de Doutor em Biotecnologia
Orientador: Prof. Dr. Wagner Franco Molina
LEONARDO LUIZ CALADO
Mapeamento cromossômico de genes ribossomais em espécies estuarinas da família Gerreidae – Identificação de uniformidade cariotípica e sua empregabilidade com
fins biotecnológicos - REPROGEN –
Tese apresentada ao programa de
pós-graduação em Biotecnologia -
RENORBIO/Universidade Federal do Rio Grande do Norte, como parte dos requisitos para obtenção do título de Doutor em Biotecnologia
_______________________________________________ Prof. Dr. George Shigueki Yasui
(Membro Externo - USP)
________________________________________________ Profª. Drª. Liliane de Lima Gurgel Souza
(Membro Externo - IFRN)
_______________________________________________ Prof. Dr. José Garcia Junior
(Membro externo – IFRN)
_______________________________________________ Prof. Dr. Marcelo Francisco de Nóbrega
(Membro externo – UFRN)
_______________________________________________ Prof. Dr. Wagner Franco Molina
“....Navegar é preciso, viver não é preciso....”
AGRADECIMENTOS
Primeiramente a Deus, por esta única oportunidade de modificar minha visão de mundo por intermédio de novos conhecimentos e àqueles que de forma direta ou indireta contribuíram para que este feito se concretizasse.
Aos meus queridos pais que me ensinaram tudo o que guardo no meu ser e que sempre, em tudo que fiz foram os responsáveis diretos pelo apoio em todos os âmbitos.
A Anelisa, namorada, esposa, amiga, companheira que aturou ao meu lado mais esta empreitada de vida.
A aquele que foi orientador e mentor, Wagner Franco Molina, que com paciência e sutileza sempre me incentivou e contribuiu para meu engrandecimento enquanto homem e aprendiz de pesquisador.
Aos membros da banca, sempre prontos a contribuir construtivamente para a melhora do trabalho.
A Rede Nordeste de Biotecnologia, RENORBIO, pelas condições dadas para a execução do trabalho e pela oportunidade de ampliação de conhecimento dentro de um multidisciplinar programa.
Ao Conselho Nacional de Pesquisa, CNPq, pelo fomento do projeto por concessão de bolsa, imprescindível para a execução do mesmo.
Aos meus amigos, Uedson, Layse, Pablo, Giroldo, Rodrigo Pantera, Paulo TT, Clóvis, Washington, Valdir pai e filho, Guinga, Amanda, Vanessa, Carla, Eurico Pofessor, Dom Cabeça, Marcela, Mestre Mieli, Rafael Dois de Ouro, Sassá, todos os
“Capoeira”, pela amizade e auxílio nas horas difíceis e ainda bem, nas fáceis
também...
como no mestrado também foram responsáveis por mais esta etapa vencida.
A minha segunda família Lavrense, Rubinho, Rose, Rian, Rubia, Dona Marta, Priscila, Robinho, Dri, Jadir, Patricia e tantos outros que sem eles não estaria cumprindo mais esta etapa de vida.
SUMÁRIO
RESUMO ... i
Abstract ... ii
LISTA DE FIGURAS ... iii
LISTA DE TABELAS ... v
Introdução ... 15
A família Gerreidae – Carapebas e carapicus ... 16
Marcadores citogenéticos ... 18
Objetivos ... 20
Objetivos específicos ... 20
Material e Métodos ... 21
Material ... 20
Técnicas citogenéticas ... 22
Preparações cromossômicas ... 22
Bandamentos cromossômicos ... 23
Identificação de padrões heterocromáticos (bandamento C) ... 24
Detecção de Regiões Organizadoras de Nucléolos (Ag-RONs) ... 24
Fluorocromos GC- e AT-específicos (CMA3 e DAPI) ... 24
Hibridação fluorescente in situ (FISH) – Sondas 18S e 5S ... 25
Microfotografias e captura de imagens ... 26
Referências bibliográficas ... 25
CAPÍTULO I - CYTOGENETIC STUDIES OF ATLANTIC MOJARRAS (PERCIFORMES - GERREIDAE): CHROMOSOMAL MAPPING OF 5S AND 18S RIBOSOMAL GENES USING DOUBLE-FISH Abstract ... 30
Introduction ... 31
Material and Methods ... 32
Specimens and chromosomal preparations ... 332
Chromosomal banding ... 32
Fluorescent in situ hybridization (FISH) ... 33
Results ... 33
Discussion ... 35
Acknowledgements ... 39
References ... 39
CAPÍTULO II - EVOLUTIONARY DYNAMICS OF rDNA GENES IN THE CHROMOSOMES OF THE Eucinostomus FISHES: CYTOTAXONOMIC AND KARYOEVOLUTIVE IMPLICATIONS Abstract ... 46
Introduction ... 47
Material and Methods ... 48
Specimens and chromosome preparations ... 48
Chromosome banding ... 48
Fluorescent in situ hybridization (FISH) procedures ... 49
Results ... 49
Discussion ... 52
Acknowledgments ... 56
CAPÍTULO III - CORRELAÇÃO ENTRE DIVERGÊNCIA CITOGENÉTICA E HIBRIDAÇÃO INTERESPECÍFICA E SEU USO NA PISCICULTURA MODERNA
Hibridação interespecífica e seu uso na aquicultura moderna ... 63
Resumo ... 63
Introdução ... 64
Hibridação na aquicultura ... 64
Citogenética e hibridação ... 66
Hibridação em espécies nativas ... 73
Considerações finais e perspectivas ... 73
RESUMO
Os Perciformes são dominantes no ambiente marinho, contituindo a maior e mais diversificada ordem de peixes dentre os teleósteos. Muitas de suas famílias, como os
Gerreidae, conhecidos popularmente como carapicus, carapebas, ou mojarras, têm um
alto potencial econômico, no que se diz respeito à piscicultura marinha, extrativismo e pesca esportiva. Informações genéticas destas espécies são de fundamental importância para seu manejo e produção. Mesmo assim, das 13.000 espécies de peixes marinhos descritos, apenas 2% foram estudadas sob o ponto de vista citogenético e menos de 1% sobre suas características reprodutivas. A reprodução induzida, a citogenética e a criopreservação de gametas, representam importantes áreas aplicadas de estudo em peixes. No presente trabalho análises citogenéticas foram empregadas na caracterização genética de espécies da família Gerreidae, ocorrentes no litoral do Nordeste do Brasil. Diferentes métodos de identificação de regiões cromossômicas foram empregados por meio de técnicas convencionais (Ag-RONs, bandamento C), coloração com
fluorocromos base-específicos (DAPI-CMA3), e mapeamento cromossômico de genes
ribossomais marcadores DNAr 18S e 5S, através da hibridação in situ com sondas
fluorescentes (FISH). As seis espécies analisadas revelaram marcante conservadorismo cromossômico. Os genes ribossomais 18S e 5S quando analisados em perspectiva filogenética, demonstram dinâmica evolutiva variada, podendo apresentar estase em
alguns grupos e maior dinamismo em outros. As análises por duplo-FISH dos sítios 18S
e 5S se revelaram eficientes marcadores citotaxonômicos nos cariótipos homogêneos deste grupo de espécies. Os padrões cariotípicos identificado, além dos aspectos evolutivos do cariótipo identificados, são sugestivos de baixo potencial de barreiras pós-zigóticas, instigando pesquisas futuras de prospecção de hibridação interespecífica destas espécies de valor comercial.
Abstract
Perciformes are dominant in the marine environment, characterized as the largest and most diverse fish group. Some families, as Gerreidae, popularly known as silver jennies, carapebas, or mojarras have a high economic potential to marine fish farming, natural explotation and game fishing. Genetic information of these species are of fundamental importance for their management and production. Despite exist over 13,000 marine fish species described, only 2% were cytogenetically analyzed and less than 1% have some reproductive characteristics known. Induced breeding, cytogenetic characterization and cryopreservation of gametes, represent important areas in applied fish studies. In this project cytogenetic analyzes were performed to acess genetic aspects of Gerreidae species, distributed in coastal and estuarine regions of Northeast Brazil. Different methods for identifying chromosomal regions were employed using conventional techniques (Ag-NORs, C-banding), staining with base-specific fluorochromes
(DAPI-CMA3), and physical mapping of ribosomal genes 18S and 5S rDNA, through
hybridization in situ with fluorescent probes (FISH). The six species analyzed showed
remarkable chromosome conservatism. The 18S and 5S ribosomal genes when analyzed in phylogenetic perspective demonstrate varied evolutionary dynamics, suggesting ocurrence of stasis process in some groups and greater dynamism in others. Double FISH with 18S and 5S probes showed both how efficient cytotaxonomic markers in the homogeneous karyotypes of this group of species. The karyotypic pattern identified in addition to the evolutionary aspects of karyotype, are suggestive of existence of low potential of post-zygotic barrier, prompting further research to prospect for artificial interspecific hybridization of these species of commercial importance.
Introdução
Figura 1. Mapa da distribuição biogeográfica dos Gerreidae no litoral brasileiro e
suas respectivas áreas de ocorrência. 18
Figura 2. Exemplares da família Gerreidae coletados para análise, Diapterus auratus (a), Diapterus rhombeus (b), Eucinostomus argenteus (c), Eucisnostomus gula (d), Eucinostomus melanopterus (e), Eugerres brasilianus (f)e Ulaema lefroyi
(g). Barra=5cm. 21
Figura 3. Pontos de coleta e espécimes de Gerreidae obtidos ao longo da zona costeira do Rio Grande do Norte. 22
Capítulo I
Figura 1. Karyotypes of Diapterus auratus (a, b), D. rhombeus (c, d) and Eugerres brasilianus (e, f), with the use of conventional staining (a, c, e) and C- banding (b,
d, f). The differentiated interstitial heterochromatic pattern in E. brasilianus can be
observed in (f) Bar=5µm. 34
Figura 2. Nucleolus organizer pairs of D. auratus (a), D. rhombeus (b) (pair 1) and E. brasilianus (c) (pair 6), revealed by silver nitrate, CMA3 and DAPI. Bar=5µm. 35
Figura 3. Chromosomal mapping by double-FISH with 18S rDNA (red) and 5S
rDNA (green) probes in Diapterus auratus (a), D. rhombeus (b) and Eugerres
brasilianus (c). Bar=5µm. 36
Capítulo II
Figura 1. Karyotypes of Eucinostomus argenteus (a, b), E. gula (c, d) and E. melanopterus (e, f) with conventional staining (a, c, e) and C-banding (b, d, f). Bar
= 5μm. 50
Figura 2. Fluorescent pattern of Eucinostomus argenteus chromosomes (a) stained
with CMA3 showing GC-rich pericentromeric heterochromatin on most
chromosome pairs. Detail of the nucleolar organizer pair (pair 22) subjected to silver impregnation and staining with base-specific fluorochrome (b, c, d) and
mapping of telomeric sequences (TTAGGG)n (e, f, g) in E. argenteus (b, e), E. gula
(c, f) and E. melanopterus (d, g), respectively. Bars = 5μm. 51
Figura 3. Location of 18S (red) and 5S (green) ribosomal genes using dual color
Figura 4. Idiogram representing the three stages of chromosomal location of
ribosomal sites in Eucinostomus species. First pattern is represented by both 5S and
18S sites in interstitial position on different chromosome pairs chromosomes; second, represented by both 5S and 18S sites in the terminal position on different chromosomes; and third, both 5S and 18S sites are colocated on a single pair in
interstitial position. 55
Capítulo III
Figura 1. Quantidade de DNA genômico em representantes de três principais Ordens de peixes, evidenciando a grande variação no conteúdo genômico médio em
LISTA DE TABELAS
Capítulo III
Tabela 1. Exemplos de hibridação interespecífica em quatro Ordens de peixes e
Introdução
A captura de espécies aquáticas tradicionais já atingiu a sua capacidade máxima de suporte, cerca de 100 milhões de toneladas por ano. Tendo em vista o progressivo crescimento populacional e subsequente demanda por proteína de origem animal, há a uma possibilidade do esgotamento destes estoques pesqueiros em uma escala global (FAO 2010).
Tentando atenuar essa situação, o cultivo de organismos aquáticos vem se tornando a alternativa mais sustentável ao fornecimento de alimentos de origem marinha para população mundial. O potencial do Brasil essa atividade é enorme, tendo
em vista as suas dimensões continentais de 8.547.404 Km2 e extensa linha costeira.
Além disso, é detentor de algumas das maiores bacias hidrográficas do planeta, representando 12% das reservas mundiais de água doce com 5,5 milhões de ha de águas represadas e condições edafoclimáticas, hidrológicas e topográficas muito favoráveis à
aquicultura (Sidonio et al., 2012).
Praticada em todos os Estados brasileiros, a aquicultura abrange principalmente a piscicultura (cultivo de peixes), a carcinicultura (cultivo de camarões), a malacocultura (cultivo de moluscos: ostras e mexilhões) e a ranicultura (cultivo de rãs). As espécies cultivadas no país, de forma experimental e/ou comercial, somam aproximadamente 64, dentre as quais, algumas descritas de forma geral são apontadas como destaque camarões marinhos (60.128 ton. em 2002), carpas (54.567 ton. em 2002), tilápias (32.460 ton. em 2002) e moluscos (15.533 ton. em 2001) (MMA, 2006).
Na década de 1990 a aquicultura nacional se firmou no contexto econômico brasileiro de fornecimento de alimentos, época na qual a produção chegava à cerca de 25.000 ton/ano. Nos anos 2000 e 2001 estima-se que os diversos setores da mesma (piscicultura, carcinicultura, malacocultura e outros) tenham ultrapassado 150.000 e 210.000 toneladas produzidas respectivamente, e nos 5 anos subseqüentes a aquicultura brasileira cresceu cerca de 25% ultrapassando no ano de 2011 o patamar de 620.000 toneladas e crescimento de 31% em relação ao ano de 2010 (MPA, 2013; Sonoda, 2006).
governamentais para desenvolvimento aquícola para cumprimento de metas de elevação
de até 3 kg/hab/ano. Quando se fala em consumo per capita de pescado no Brasil por
região, aquela que mais consome é a região Norte, com 62,63 kg/hab/ano, seguidos respectivamente pelo Nordeste, 11,54 kg/hab/ano, Sudeste, 3,65 kg/hab/ano, Sul, 3,99 kg/hab/ano e Centro-Oeste 3,20 kg/hab/ano.
O desenvolvimento do setor vem recebendo mais fomento governamental no decorrer dos anos através de criação de linhas de crédito específicas. Assim a publicação de instruções normativas como a abertura da águas da união para o cultivo de organismos aquáticos mediante o Decreto n° 2.869 de 09 de dezembro de 1998 e instrução normativa ministerial n° 9 de 11 de abril de 2001, criação do Ministério da Pesca e Aquicultura (MPA), pela lei nº 11.958 de 29 de Junho de 2009 e a implantação do Plano Safra da pesca e Aquicultura em 25 de outubro de 2012 que visa até 2014 dobrar a produção de pescado fomentando assistência técnica, pesquisas no setor, despescas programadas no período de defeso e em consequência até o ano de 2015 aumentar o consumo per capita por ano de 9 Kg para 13,8 Kg, acima do preconizado pela Organização Mundial da Saúde (OMS).
As particularidades fisiográficas regionais e diversos ecossistemas marinhos no litoral brasileiro permitem caracterizar este território como um ambiente heterogêneo, determinando através das suas características bióticas e abióticas uma rica ictiofauna, tanto em diversidade como em quantidade de espécies, segundo Menezes (2003) desde o Cabo Orange até o Chuí em uma abrangência de 200 milhas náuticas da Zona Econômica Exclusiva, 1297 espécies de peixes marinhos tem ocorrência documentada. Dentre as diversas Ordens de peixes que habitam o litoral, a dos Perciformes se destaca por sua ampla distribuição, não só no Brasil, como em diversos mares e oceanos do mundo. As relações filogenéticas de suas espécies ainda não são bem definidas e são as causas de muitas controvérsias (Lauder & Liem, 1983). Muitas de suas famílias apresentam um alto valor comercial para piscicultura marinha, como também são importantes na pesca comercial e esportiva.
A família Gerreidae – Carapebas e carapicus
acará-peba e acará-picu, que, em linguagem tupi-guarani, significam respectivamente peixe achatado ou largo (Cará-largo) e peixe alongado (Cará-comprido). Abundantes em águas tropicais e subtropicais do Atlântico e do Pacífico, esta família é comumente
encontrada em todo litoral brasileiro (Bordin et al., 2007).
No Brasil, as espécies Eucinostomus melanopterus e Diapterus rhombeus são
seus maiores representantes. São peixes de médio a pequeno porte, comprimidos
lateralmente e caracterizados por suas bocas extremamente protrusivas e reprodução e recrutamento anuais com picos sazonais em meses cuja temperatura são mais altas e o regime de chuvas contribui para um aumento da proporção entre machos e fêmeas e consequentemente à reprodução (Aguirre-León & Díaz-Ruiz, 2006; Olaya-Nieto & Appeldoorn, 2004; Nelson, 1994). São espécies que se alimentam de uma gama de presas de hábitos bentônicos, como anfípodas, crustáceos, bivalves, copépodas,
foraminíferos, ovos de peixes, dentre outros (Kerschner et al., 1985). Geralmente
ocorrem em áreas costeiras rasas e sobre partes inferiores enlameadas e arenosas, nos estuários ou em lagoas hipersalinas.
Devido à sua relativa abundância em ambientes estuarinos, estas espécies têm certa facilidade de captura, o que já constitui um valor comercial agregado (Cervigon, 1993), sobretudo como alternativa de cultivo nas fazendas de camarão espalhadas pelo Nordeste do Brasil. Para os taxonomistas, a importância econômica desigual entre espécies da família Gerreidae, contribuiu de forma controversa para o estabelecimento de caracteres taxonômicos morfológicos com certo grau de subjetividade.
Os membros da família Gerreidae são um dos maiores e representativos grupos de peixes das regiões tropicais e subtropicais, possuindo ampla distribuição na região neotropical (Figura 1), distribuindo-se na maioria dos oceanos, principalmente no Atlântico, Pacífico e Índico. Na costa do Atlântico se distribuem do Estado da Flórida (EUA), Ilhas Bermudas até o estuário do Rio da Prata, na fronteira entre Uruguai e
Argentina, principalmente em áreas estuarinas (Solari et al., 2010). Dos quinze gêneros
já descritos, quatro são constantemente encontrados em águas do Atlântico (Diapterus,
Eucinostomus, Eugerres e Ulaema), totalizando 13 das 44 espécies conhecidas (De La
Cruz Aguero et al., 2011; Andreata, 2006; Gilmore Jr., 2002). São abundantes em áreas
de estuários de rios e mangues, juntamente com representantes das famílias Clupeidae, Engraulidae, Mugilidae e Centropomidae sendo consideradas importantes para a
Figura 1. Mapa da distribuição geográfica dos Gerreidae no litoral brasileiro e algumas de suas principais áreas de ocorrência.
Marcadores citogenéticos
No decorrer das últimas décadas, a genética vem se apresentando como uma ferramenta essencial na elucidação da definição de populações e espécies. Dentre estas linhas de pesquisa, a citogenética tem se constituído uma forte aliada, através da identificação de caracteres citotaxonômicos.
intensivo quanto para elucidação de aspectos da biologia e da estrutura das populações. Neste sentido, as bases da piscicultura moderna vêm demonstrando uma essencial contribuição prestada pela incorporação de metodologias genéticas em espécies sob cultivo, entre elas, a citogenética. Entretanto, apenas 2% das espécies marinhas possuem alguma informação citogenética disponível (Brum, 1996).
Grupo dominante no ambiente marinho, os peixes da ordem Perciformes são caracterizados por uma notável homogeneidade cromossômica, tendo uma porcentagem pequena de seus representantes que carregam os números diploides mais elevados ou abaixo do que seu número básico (Molina & Bacurau, 2006). Estes fatores de homogeneidade podem ser explicados pela ausência de barreiras físicas definitivas, e consequentemente, ausência de isolamento geográfico, associados à grande capacidade de dispersão e distribuição destas espécies (Lacson, 1992). Estes fatores explicam a estabilidade e a ampla distribuição do cariótipo com 48 cromossomos acrocêntricos, indicando um valor modal e a condição conservada desse tipo de cariótipo, proposto
como primitivo para esta ordem (Ohno, 1970; LeGrande & Fitzsimons, 1988; Vitturi et
al., 1992). Este cariótipo basal é encontrado em muitas famílias, como Carangidae
(Caputo et al., 1996), Haemulidae (Brum, 1996), Gerreidae (Molina & Bacurau, 2006;
Ruiz-Carus & Uribe-Alcocer, 2004), dentre outros.
Devido à ampla diversidade dos Perciformes, diversas famílias carecem de informações citogenéticas. Neste sentido, pelo menos um cariótipo de uma espécie está disponível para 50 das 150 famílias de Perciformes, demonstrando com isso, uma lacuna notável sobre o conhecimento citogenético de um grande número de espécies (Brum, 1996). Esta falta de informações pode estar associada à natureza das espécies ou aos parcos esforços dirigidos ao estudo de alguns grupos, sobretudo, espécies estuarinas, como no caso da família Gerreidae (Molina & Bacurau, 2006). Quando existente, a maioria dos dados citogenéticos de espécies marinhas ou estuarinas, é baseado em técnicas convencionais que envolvem a definição da morfologia cromossômica, o bandamento C e a identificação de cístrons ribossomais pela técnica de Ag-RONs (Molina, 2007; Ruiz-Carus & Uribe-Alcocer, 2004).
impactos causados pela sobreexplotação dos recursos pesqueiros, antes que se atinja a capacidade suporte dos mesmos como ocorrem com diversas outras espécies. Em adição a cariotipagem destas espécies tras informações pertinentes para a formação de híbridos que podem auxiliar em pesquisas voltadas para a piscicultura, a poliploidização e a quantificação de conteúdo de DNA.
Objetivos
Este trabalho visou caracterizar detalhadamente os padrões cromossômicos de seis espécies de peixes de importância comercial da família Gerreidae, de ocorrência em áreas estuarinas e ao longo do litoral brasileiro, através de técnicas citogenéticas convencionais e mapeamento físico de genes ribossomais. Espera-se que tais processos subsidiem uma melhor compreensão da taxonomia do grupo, determinação de marcadores citotaxonômicos e nível de divergência cariotípica com vistas ao uso futuro para fins biotecnológicos e de conservação biológica.
Objetivos específicos
Determinação da macroestrutura cariotípica de seis espécies da família
Gerreidae nas áreas estuarinas do litoral brasileiro, identificando suas tendências evolutivas e mecanismos de mudança;
Estabelecimento dos padrões cromossômicos estruturais de seis espécies de
Gerreidae pelo uso de bandamentos cromossômicos para identificação das regiões organizadoras de nucléolos e da heterocromatina constitutiva e pelo
emprego de fluorocromos base-específicos (DAPI - CMA3);
Mapeamento cromossômico dos genes ribossomais 18S e 5S por meio de
hibridação in situ com sondas fluorescentes (FISH);
Correlacionar padrões de divergência cromossômica e a obtenção natural ou
Material e Métodos
Material
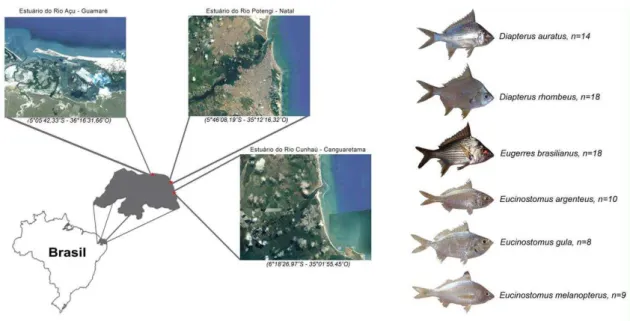
Exemplares das espécies Diapterus auratus Ranzani, 1942 (n=14), D. rhombeus
Cuvier, 1829 (n=18), Eucinostomus argenteus (Baird & Girard, 1855) (n=10), E. gula (Quoiy & Gaimard, 1824) (n=8), E. melanopterus (Bleeker, 1863) (n=9), Eugerres brasilianus (n=18) Cuvier, 1830 (Figura 2) foram coletadas nas áreas dos maiores
Figura 2. Exemplares da família Gerreidae utilizados nas análises citogenéticas.
Diapterus auratus (a), D. rhombeus (b), Eucinostomus argenteus (c), E. gula (d), E. melanopterus (e) e Eugerres brasilianus (f). Barra = 5cm.
Os espécimes coletados foram selecionados mediante suas condições físicas, acondicionados em sacos plásticos com adição de oxigênio puro, onde foram mantidos até a sua chegada e acomodação em tanques, nas dependências do laboratório de Genética de Recursos Marinhos da Universidade Federal do Rio Grande do Norte, para desenvolvimento das práticas citogenéticas.
Figura 3. Pontos de coleta e espécimes de Gerreidae obtidos ao longo da zona costeira do Rio Grande do Norte.
Técnicas citogenéticas
Preparações cromossômicas
A estimulação da divisão celular foi realizada por meio da inoculação intraperitoneal de agentes mitogênicos em cada indivíduo, por um período de 24 a 48
horas (Molina, 2001; Molina et al., 2010). Posteriormente, os animais foram
sacrificados para retirada de fragmentos do rim anterior a partir dos quais suspensões celulares foram empregadas para obtenção de cromossomos mitóticos, segundo a
técnica de Gold et al. (1990).
Bandamentos cromossômicos
As suspensões celulares foram gotejadas em lâminas e coradas com Giemsa 5% em tampão fosfato (pH 6,8) para a caracterização cariotípica dos espécimes. As melhores metáfases foram selecionadas para caracterização do número diploide e definição do cariótipo. Os cromossomos foram classificados em grupos quanto à posição do
centrômero (Levan et al., 1964), e organizados no cariótipo em ordem decrescente de
Identificação de padrões heterocromáticos (bandamento C)
A heterocromatina constitutiva foi evidenciada pelo bandamento C (Sumner, 1972). As lâminas com preparações cromossômicas foram tratadas com uma solução de HCl 0,2 N, à temperatura ambiente, durante 13 minutos e, a seguir, submersas numa solução de hidróxido de bário 5%, à temperatura ambiente, por 1 minuto. Logo após, foram lavadas em solução de HCl 0,2N, incubadas em solução salina 2xSSC aquecida a 60°C por 30 minutos. Os cromossomos foram corados com Giemsa 5%, diluído em tampão fosfato pH 6,8 durante 8 minutos. Após cada um dos passos acima, as lâminas foram lavadas com água destilada e secas ao ar.
Detecção de Regiões Organizadoras de Nucléolos (Ag-RONs)
Para a detecção das regiões organizadoras de nucléolos (RONs) ativas, foi utilizado o procedimento descrito por Howell e Black (1980), empregando nitrato de prata
(AgNO3). As lâminas foram tratadas com uma mistura contendo duas partes de uma
solução aquosa de gelatina 2% (acrescida de ácido fórmico 1% na proporção de 1ml
para cada 100ml de solução) e quatro partes de uma solução de AgNO3 50%. O material
foi recoberto com lamínula e incubado em estufa à 60°C, por aproximadamente 5 minutos. A lamínula foi então retirada com jatos de água destilada e posteriormente os cromossomos corados com Giemsa 5% por cerca de 30 segundos. Após esse período, as lâminas foram lavadas em água corrente e secas à temperatura ambiente.
Fluorocromos GC- e AT-específicos (CMA3 e DAPI)
A coloração dupla por fluorocromos base-específicos (CMA3 e DAPI) seguiu a
metodologia descrita por Christian et al. (1998). Para tanto, as preparações
cromossômicas foram incubadas em formamida 70%/2xSSC à 70ºC por 2 minutos. Posteriormente, as lâminas foram banhadas por duas vezes por 2 minutos em 2xSSC à temperatura ambiente. As lâminas foram então transferidas para uma bateria de álcool 70%, 85% e 100%, e sequencialmente desidratadas por 2 minutos em cada banho. Após
secagem, foram adicionados 80µl de cromomicina A3 em cada lâmina, permanecendo
vezes em tampão PBS 1x, dois minutos em cada banho e imediatamente montadas com 80 µl de solução de DAPI/Antifading (0,2 µg/ml).
Hibridação fluorescente in situ (FISH) – Sondas 18S e 5S
A hibridação in situ fluorescente (FISH), foi realizada de acordo com a
metodologia descrita por Pinkel et al. (1986) com algumas modificações. As sondas
foram marcadas por nick translation (BioNick Labeling System – Invitrogen) de acordo
com as instruções do fabricante. A solução de hibridação consistiu de 200 µl de formamida 50%, 80 µl de sulfato dextrano 50%, 40 µl de 20xSSC, 80 µl de água q.s.p., perfazendo um volume total de 400 µl, sendo adicionado 1,5 µg de sonda (DNA marcado com biotina). Em seguida, a solução de hibridação foi transferida para um banho fervente, durante 10 minutos, para desnaturação do DNA e, imediatamente após, para um recipiente com gelo, impedindo a renaturação por choque térmico. As lâminas, contendo as preparações cromossômicas, foram lavadas com tampão PBS 1x por 5 minutos à temperatura ambiente, sob agitação e, posteriormente desidratadas em série alcoólica (70%, 85% e 100%). Em seguida, foram submetidas a digestão com RNAse (100 µg/ml) por 1 hora e 30 minutos, em câmara úmida a 37°C, e lavadas em soluções salinas. O material foi desidratado em uma série alcoólica, 5 minutos em cada banho e tratado com formamida 70%/2xSSC a 70°C, por 5 minutos, para denaturação dos cromossomos. Uma nova desidratação em série alcoólica foi realizada. Após tal procedimento foi aplicado, sobre a lâmina, 50 µl da solução de hibridação contendo a
sonda denaturada, permanecendo em câmara úmida overnight a 37oC. Transcorrido esse
tempo, as lâminas foram lavadas em solução de formamida 50%/2xSSC a 42oC por 10
minutos, três vezes em 0,1xSSC a 60°C, 5 minutos em cada lavagem e em solução Tween20 (0,05%/2xSSC), sob agitação, por 5 minutos. Em seguida foi realizado um
tratamento com 90 µl de NFDM 5% (non fat dry milk – leite em pó desnatado) em
foram repetidos mais uma vez. Finalizando, a lâmina foi desidratada em série alcoólica (70%, 85% e 100%) durante 5 minutos em cada banho. Os sinais de hibridação foram detectados com avidina-FITC e os cromossomos contra-corados com iodeto de propídio (50 µg/ml) ou DAPI (0,2 µg/ml).
Microfotografias e captura de imagens
As preparações cromossômicas convencionais foram analisadas e fotografadas em microscópio óptico. As preparações de FISH e de fluorocromos base específicos foram analisadas sob aumento de 1000X em fotomicroscópio de epifluorescência Olympus BX50, equipado com sistema digital de captura de imagens DP-70 Olympus.
Referências bibliográficas
Aguirre-León, A. & Díaz-Ruiz, S. 2006. Estructura de tallas madurez gonadica y
alimentacion del pez Diapterus rhombeus (Gerreidae) en el sistema fluvio-deltaico
Pom-Atasta, Campeche, México. Revista de Biologia Tropical 54, 599-611.
Andreata, J.V. 2006. Sobre as espécies de Eucinostomus Baird & Girard in Baird, 1855,
que ocorrem em águas brasileiras (Actinopterygii, Perciformes, Gerreidae). Estudos de Biologia 28, 13-25.
Bordin, D., Costa, M. D. P., Frisanco, D., Duarte, L., Souza-Conceição, J. M. & Spach, H. L. 2007. Estágios iniciais do ciclo de vida de Gerreidae ocorrentes em duas praias estuarinas do litoral de Santa Catarina (Baía da Babitonga, São Francisco do Sul) In:
VIII Congresso de Ecologia do Brasil, Caxambu - MG Anais... Caxambu – MG 2007.
Brum, M. J. I. 1996. Cytogenetic studies of Brazilian marine fish. Brazilian Journal of Genetics 19, 421-427.
Caputo, V., Marchegiani, F. & E Olmo. 1996. Karyotype differentiation between two
species of carangid fishes, genus Trachurus (Perciformes, Carangidae). Marine Biology
Castellanos-Galindo, G.A., Krumme, U., Rubio, E.A. & Saint-Paul, U., 2012. Spatial variability of mangrove fish assemblage composition in the tropical eastern Pacific
Ocean. Reviews in Fish Biology and Fisheries.DOI 10.1007/s11160-012-9276-4
Cervigon, F. M. Los peces marinos de Venezuela, 2nd edition, Vol. 2. Caracas, Fundacion Cientifica Los Roques, 498 p. 1993.
Christian, A., Mcniel, E., Robinson, J., Drabeck, R., Larue, S., Waldren, W. & Bedfort, J. 1998. A versatile image analysis approach for simultaneous chromosome identification and localization of FISH probes. Cytogenetics and Cell Genetics 82, 172-179.
De La Cruz Agüero, J., García Rodríguez, F.J., Cota Gómez, V.M., Chollet Villalpando, J.G. & Vergara Solana, F.J., 2011. Length–weight relations of selected species of the family Gerreidae (Actinopterygii: Perciformes) from the Mexican coast. Acta Ichthyologica Et Piscatoria 41, 67-69.
FAO 2006 Yearbook. Fishery Statistics. Aquaculture production 2004, V. 90, n.2, p. 178. FAO. 2006.
Gilmore Jr. R. G.; Carpenter, K.E. 2002. (ed.) The living marine resources of the Western Central Atlantic. Volume 2: Bony fishes part 1 (Acipenseridae to Grammatidae). FAO Species Identification Guide for Fishery Purposes and American Society of Ichthyologists and Herpetologists Special Publication 5, 601-1374.
Gold Jr., L. C., Shipley, N. S. & Powers, P. K., 1990. Improved methods for working with fish chromosomes with a review of metaphase chromosome banding. Journal of Fish Biology 37, 563-575.
Howell, W. M. & Black, D. A. 1980. Controlled silver staining of nucleolus organizer region with protective colloidal developer: a 1-step method. Experientia 36, 1014-1015.
Lacson, J. M. 1992. Minimal genetic variation among samples of six species of coral reef fishes collected at La Parguera, Puerto Rico, and Discovery Bay, Jamaica. Marine Biology 112, 327-331.
Lauder, C. V. & Liem, K. F. 1983. The evolution and interrelationships of the Actinopterygian fishes. Bulletin of the Museum of Comparative Zoology 150, 95-197.
Legrande, W. H. & Fitzsimons, J. M. 1988. Chromosome numbers of some gulf coast sciaenid fishes. Copeia 2, 491-493.
Levan, A., Fredga, K., Sandberg, A.A. 1964. Nomenclature for centromeric position on chromosomes. Hereditas 52, 201-220.
Ministério do Meio Ambiente/Caderno setorial de recursos hídricos: agropecuária.
2006. Secretaria de Recursos Hídricos. – Brasília: MMA,. 96p.
Ministério da Pesca e Aquicultura. Consumo de pescado no Brasil aumenta 23,7% em
dois anos. Disponível em:
http://www.mpa.gov.br/index.php/imprensa/noticias/2226-consumo-de-pescado-no-brasil-aumenta-237-em-dois-anos. Acesso em: 03 Dez. 2013, 13:18:23
Molina, W.F. 2007. Chromosomal changes and stasis in marine fish groups. in: Pisano E, Ozouf-Costaz C, Foresti F, Kapoor BG (eds): Fish cytogenetics, 69-110.
Molina, W.F. 2001. An alternative method for mitotic stimulation in fish cytogenetics. Chromosome Science 5, 149-152..
Molina, W.F., Bacurau, T.O.F. 2006. Structural and Numerical Chromosome Diversification in Marine Perciformes (Priacanthidae and Gerreidae) The Japan Mendel
Molina W.F., Alves, D.E.O., Araújo, W.C., Martinez, P.A., Silva, M.F.M., Costa G.W.W.F. 2010. Performance of human immunostimulating agents in the improvement of fish cytogenetic preparations. Genetic and Molecular Research 9, 1807-1814.
Nelson, J.S. 1994. Fishes of the world. 3 Ed. New York. John Willey e Sons. p. 600.
Ohno, S. 1970. Evolution by gene duplication. New York. Springer-Verlag, p. 160.
Olaya-Nieto, C.W., Appeldoorn, R.S. 2004. Edad y Crecimiento de la Mojarra Rayada,
Eugerres plumieri (Cuvier), en la Ciénaga Grande de Santa Marta, Colombia. 56th Gulf
and Caribbean Fisheries Institute 55, 337-347.
Pinkel, D., Straume, T., Gray, J.W. 1986. Cytogenetic analysis using quantitative, high-sensivity, fluorescence hybridization. Proceedings of the National Academy of Sciences USA 83, 2934-2938.
Ruiz-Carus, C.R., Uribe-Alcocer B.M. 2004. Karyotype analysis of Eucinostomus
argenteus, E. gula, E. harengulus, and Eugerres plumieri (Teleostei, Gerreidae) from
Florida and Puerto Rico. Environmental Biology of Fishes 67, 269–276.
Sidonio, L., Cavalcanti, I., Capanema L., Morch, R., Magalhães, G., Lima, J., Burns, V., Júnior, A. J. A., Mungioli, R. 2012. Panorama da aquicultura no Brasil: desafios e oportunidades. BNDES Setorial 35: 421 - 463.
Sonoda, D. Y. 2006. Demanda por pescados no Brasil entre 2002 e 2003, 117p, Tese (Doutorado Escola Superior de Agricultura Luiz de Queiroz), Piracicaba, São Paulo 2006
Solari, A., Garcia, M. L., Jaureguizar, J. A. 2010. First record of Eucinostomus
melanopterus, Bleeker 1863 (Perciformes, Gerreidae) from the Río de la Plata estuary.
Journal of Applied Ichthyology 26, 485-487.
Vitturi, R., Catalano, E., Barbieri, R. 1992. Karyological and molecular characterization of Mullus surmuletus and Mullus barbatus (Pisces, Mullidae). Cytologia 57, 65-74.
Wilkins, N. P. 1981. The rationale and relevance of genetics in aquaculture: an overview. Aquaculture 22, 209-228.
CAPÍTULO I
Artigo publicado no jornal Aquaculture Research, 44: 829-835, 2012.
DOI: 10.1111/j.1365-2109.2012.03089.x
Cytogenetic studies of Atlantic mojarras (Perciformes - Gerreidae): chromosomal mapping of 5S and 18S ribosomal genes using double-FISH
Leonardo Luiz Calado1, Luiz Antônio Carlos Bertollo2, Gideão Wagner Werneck Felix
da Costa1 and Wagner Franco Molina1
1 Department of Cellular Biology and Genetics, Biosciences Center, Federal University
of Rio Grande do Norte, Natal, Brazil.
2 Department of Genetics and Evolution, Federal University of São Carlos, São Carlos,
Brazil.
Abstract
Fishes of the family Gerreidae, mainly species of the genera Diapterus and Eugerres,
have high potential for cultivation, because of their saline tolerance. A detailed
cytogenetic analysis of Diapterus auratus, Diapterus rhombeus and Eugerres
brasilianus was conducted using conventional staining, C-banding, Ag-NOR,
AT/GC-specific fluorochrome staining, and mapping of ribosomal sequences with 5S and 18S rDNA probes. All the species exhibited symmetrical karyotype, 2n=48 acrocentric chromosomes. The Ag-NORs and 18S rDNA are present in the interstitial position on
pair 1 (genus Diapterus) and on pair 6 in Eugerres brasilianus. The 5S rDNA sites,
located in the interstitial position (pair 11), are conserved in the three species.
reduced and centromeric bands, differing from E. brasilianus, where, in addition to
these, more prominent interstitial bands were observed. GC-rich regions are located at
ribosomal sites. Karyotypic comparison between Diapterus and Eugerres reveals
similarity in chromosomal macrostructure, differing in C-positive heterochromatin distribution and position of 18S sites, indicating the occurrence of structural microrearrangements. Although complementary analyses are needed, the similarities observed for these and other species suggest the possibility of breaking postzygotic barriers and their potential use, through induced interspecific or intergeneric hybridizations.
Keywords: aquaculture, chromosomal markers, estuarine fish, FISH, karyotypic stasis.
Running title: Chromosomal cytomarkers in Atlantic mojarras
Introduction
Most members of the family Gerreidae inhabit estuaries, hypersaline lakes, as well as freshwater coastal areas. They are usually small to mid-sized, silver colored, with laterally compressed body and have a ventral, protractile mouth as their main characteristic (Cervigon 1993; Nelson 2006). It is represented in the Atlantic by the
genera Diapterus, Eugerres, Eucinostomus, Gerres and Ulaema (Eschmeyer 1998).
Gerreidae are a phylogenetically unstable group, due to complex morphological characters and subjectivity found in some descriptions of their species. A morphological
subdivision has been proposed, individualizing two subgroups, Diapterus and Eugerres,
which exhibit serrated inferior margin of the preoperculum, and Eucinostomus and
Gerres, where it is smooth (Iwatsuki et al. 2001; Ruiz-Carus & Uribe-Alcocer 2003,
2004). The same subdivision has been confirmed by molecular phylogenetic analyses, using mitochondrial 12S and 16S rDNA sequences and Rhodospin and RAG1 nuclear
genes (Chen et al. 2007).
Gerreidae are an important commercial resource on occurrence areas in the
Atlantic (Grijalva-Chon et al. 1996), with diverse species well adapted to the vast
shrimp production areas, indicating their potential use in conjunction with aquaculture,
as suggested for other fish species such as the Asian sea bass (Lates calcarifer), grouper
2005). Indeed, species of the family Gerreidae have been considered for cultivation
(Bensam 2000), with some measure of success in in vitro induction and reproduction, as
evidenced by Eugerres brasilianus, the largest species in the family (Eiras-Stofella &
Fanta 1991; Avila-Poveda & Lamouroux-López 2007).
Chromosomal characteristics of commercially important fish have been increasingly used in the implementation of biotechnological techniques (e.g. Rab 1991;
Flajshans et al. 1992; Jacobina et al. 2011). Although there has been a rise in
cytogenetic studies in marine and estuarine species, information is still very limited or even absent for a number of these groups. In Gerreidae, for example, cytogenetic data are scarce (e.g. Ruiz-Carus & Uribe-Alcocer 2004). With the aim of contributing to future management programs for Gerreidae species, we present the chromosomal
characters of three species from this family: Diapterus auratus (Irish mojarra), D.
rhombeus (rhombic mojarra) and Eugerres brasilianus (Brazilian mojarra), with the use
of conventional staining, detection of C-positive heterochromatin distribution, nucleolus organizer regions (NORs) and GC- and AT-rich chromosomal regions, as well as mapping of ribosomal sequences by double-FISH, with 5S and 18S rDNA probes.
Material and Methods
Specimens and chromosomal preparations
Fifty specimens from the following three species of Gerreidae were analyzed: D.
auratus (n=14), D. rhombeus (n=18) and E. brasilianus (n=18) from the state of Rio
Grande do Norte, Northeast Brazil, collected at the mouth of the Cunhaú River (6⁰18’26.97”S; 35⁰01’55.45”O), in Canguaretama and in the Açu River delta in
Guamaré (5⁰05’42.33”S; 36⁰16’31.66”O). Before chromosomal preparations,
specimens were submitted to in vivo mitotic stimulation for 24 hours, by intramuscular
and intraperitoneal innoculation of fungal and bacterial antigen complexes (Molina
2001; Molina et al. 2010). After specimens were anesthetized with clove oil (eugenol)
and sacrificed, renal tissue was removed for chromosomal preparations. Metaphase chromosomes were obtained from cell suspensions of anterior rim fragments using the
short-term in vitro method (Gold et al. 1990). Cell suspensions were dripped onto slides
covered with a film of distilled water heated at 60oC. Around 30 metaphases were
metaphases were photographed with an Olympus BX50 epifluorescence microscope and Olympus DP70 digital image capture system.
Chromosomal banding
Heterochromatic regions and nucleolus organizer regions were identified by C-
banding (Sumner 1972) and silver nitrate staining – Ag-NORs, respectively (Howell &
Black 1980). CMA3/DAPI fluorochrome staining was used to identify GC- and AT-rich
regions (Barros-e-Silva & Guerra 2009). Slides aged for three days were stained with
CMA3 (0.1 mg.ml-1) for 60 min and restained with DAPI (1 µg.ml-1) for 30 min. Slides
were then mounted in glycerol:McIlvaine buffer pH 7.0 (1:1), aged for three days and analyzed under an epifluorescence microscope equipped with an appropriate filter set.
Fluorescent in situ hybridization (FISH)
Double FISH was performed using the method described by Pinkel et al. (1986),
with modifications. The 18S and 5S rDNA probes were obtained by primer
amplification via polymerase chain reaction (PCR), with DNA from Prochilodus
argenteus (Hatanaka & Galetti 2004) and Leporinus obtusidens (Martins & Galetti Jr
1999), respectively. The 18S rDNA probe was labeled with biotin-dUTP and the 5S rDNA with digoxigenin-dUTP. The 18S probe was detected with avidin-fluorescein isothiocyanate (FITC) conjugate and anti-avidin, whereas the 5S rDNA probe was detected with anti-digoxigenin conjugate and Rodhamin. Images of the best metaphases
were captured under appropriate filters, as described previously for CMA3/DAPI
fluorochromes.
Results
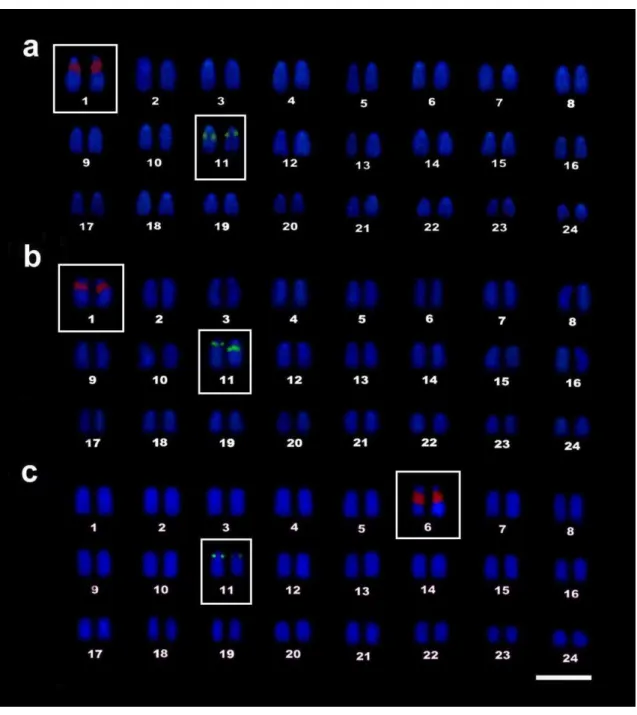
Diapterus auratus, D. rhombeus and E. brasilianus exhibited symmetric
Figure 1. Karyotypes of Diapterus auratus (a, b), D. rhombeus (c, d) and Eugerres brasilianus (e, f), with the use of conventional staining (a, c, e) and C- banding (b, d, f).
The differentiated interstitial heterochromatic pattern in E. brasilianus can be observed
in (f) Bar=5µm.
In species D. auratus and D. rhombeus, NORs are located interstitially on the first
karyotype pair, while in E. brasilianus they are found in the same position on
chromosome pair 6, where they were identified by both silver nitrate impregnation (Fig.
2) and in situ hybridization with 18S rDNA probes (Fig. 3). Chromosome mapping of
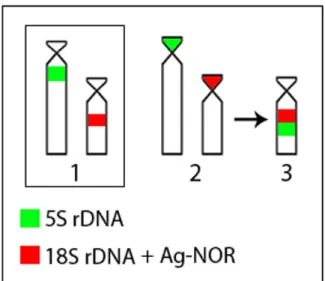
5S rDNA ribosomal subunits showed sites in the interstitial position on a single chromosome pair, apparently homeologous between species and tentatively identified as pair 11 (Fig. 3).
Reduced heterochromatic blocks are present in centromeric regions and
chromosome terminals of three species (Fig. 1b, d, f). In E. brasilianus, in addition to
centromeric regions, more conspicuous heterochromatic interstitial blocks are present
on most chromosomal pairs (Fig. 1f). CMA3/DAPI fluorochrome staining revealed
Figure 2. Nucleolus organizer pairs of
D. auratus (a), D. rhombeus (b) (pair 1)
and E. brasilianus (c) (pair 6), revealed
by silver nitrate, CMA3 and DAPI.
Bar=5µm.
Discussion
Karyotypic analyses of the two Diapterus species and E. brasilianus showed
notable intrageneric similarity, with marked conservatism in most chromosomal characteristics analyzed, as well as a number of common intergeneric characteristics. In general, the data revealed chromosomal patterns typical of several families of
Figure 3. Chromosomal mapping by double-FISH with 18S rDNA (red) and 5S rDNA
(green) probes in Diapterus auratus (a), D. rhombeus (b) and Eugerres brasilianus (c).
Bar=5µm.
Evolutionally, the presence of 2n=48 mono-brachial chromosomes, ribosomal sites situated on a single pair of chromosomes, reduced heterochromatic content dispersed in centromeric and teleomeric regions seem to represent basal conditions for
Perciformes (Galetti et al. 2000; Molina 2007). Indeed, some families in this order have
demonstrated elevated interspecific chromosomal conservatism, despite the use of
different cytogenetic methodologies (Motta-Neto et al. 2011). Some earlier studies on
Eugerres and Eucinostomus (Ruiz-Carus & Uribe-Alcocer 2004; Molina & Bacurau
2006; Cipriano et al. 2008).
The 18S ribosomal sites, as well as the location of Ag-NORs/GC+, showed
equilocal location on chromosome 1 of the karyotype of Diapterus species and on pair 6
of E. brasilianus, indicating that nucleolus organizer regions are characterized by single
sites, a condition considered plesiomorphic in fish (Cross et al. 2006). A similar
situation occurs with mapping of 5S ribosomal sites, in this case located exclusively on pair 11 of the karyotype of the three species. Thus, chromosomes bearing 18S ribosomal
genes in Diapterus species, and 5S, in all the species, are likely homeologous,
reinforcing the similarity and karyotypic symmetry within this family.
The evolution of ribosomal sequences seems to be associated to the level of karyotypic diversification of a group. This, in Characiformes and Salmoniformes, where karyotype structure is more diversified between species, greater intra and interpopulational diversity is observed in terms of location and frequency of 18S and 5S
rDNA sites (e.g. Fujiwara et al. 1998; Ferro et al. 2001; Vicari et al. 2003; Mantovani et
al. 2005; Fernandes & Martins-Santos 2006). On the other hand, in Perciformes, a
markedly conservative group with respect to karyotype macrostructure and heterochromatic content, a more stable condition is usually found at these sites (Molina
& Galetti 2002; Vicari et al. 2006; Motta-Neto et al. 2011). The diversity of situations
observed, for both 18S and 5S rDNA sequences, demonstrates their evolutionary independence, reinforced by frequent non-synthenic arrangements of these genes in fish,
as observed in D. auratus, D. rhombeus and E. brasilianus and suggested as a basal
condition in Teleostei (Martinez et al. 1996; Suzuki et al. 1996; Martins & Galetti 1999,
2000). Although chromosomal mapping of 5S ribosomal sites has been decisive in discriminating several populations and fish species, often phylogenetically close (Aguilar & Galetti 2008; Motta-Neto et al. 2011), this is not the case for Gerreidae,
where intergeneric chromosomal differences are observed only in the positioning of 18S sites, demonstrating recent diversification in this group, or even their slow evolution or evolutionary stasis. This conclusion is corroborated by the short genetic distance
between species of genera Diapterus, Eugerres and Eucinostomus estimated from
allozymic polymorphic systems (Ruiz-Carus & Uribe-Alcocer 2003).
interstitial and terminal regions. In this respect, E. brasilianus differs from Diapterus
species since it exhibits a richer pattern of interstitial blocks. Centromeric distribution of heterochromatin seems to restore the ancestral pattern for the family Gerreidae, given that this same condition is present in a number of other Perciformes, such as Serranidae,
Lutjanidae and Haemulidae (Molina et al. 2002; Aguilar & Galetti 1997; Rocha &
Molina 2008; Motta-Neto et al. 2011).
The sharing of the same pattern of heterochromatic blocks on chromosomes by two Diapterus species, in addition to conservatism of the other characteristics analyzed,
suggests evolutionary maintenance of ample homeologous chromosomal segments between their chromosomes. Thus, differentiated interstitial heterochromatic blocks in
chromosomes of E. brasilianus suggest a more derived condition. Extensive
rearrangements have been suggested as molders of the karyotype of some species of Perciformes (Molina & Galetti 2004). However, a more parsimonious possibility for the occurrence of equilocal heterochromatic regions in several chromosomal pairs, as
observed in E. brasilianus, would be dispersion of repetitive sequences during the
interphase. The physical proximity of these sequences during the interphase, due to the arrangement adopted by chromosomes in the interphase nucleus, might favor their dispersion (Schweizer & Loidl 1987). Indeed, radial metaphase figures oriented with the centromeres, involving non-homologous acrocentric chromosomes, are often found in
chromosomal complements of Perciformes (Aguilar et al. 1998; Molina & Galetti
2002), corroborating this hypothesis.
Phylogenetic proximity, suggested for Diapterus and Eugerres (Chen et al. 2007;
Ruiz-Carus & Uribe-Alcocer 2003), is not ruled out by the chromosomal patterns identified. Karyotypic similarities exhibited by Gerreidae species could establish weak postzygotic barriers, favoring likely interspecific hybridizations, practices routinely used in the commercial production of other fish species.
With the potential use of Gerreidae representatives in fish culture programs, species discrimination using cytogenetic markers could be very useful in the management and individualization of those destined for cultivation, a technique already
used in other fish species (Morán et al. 1994). Chromosomal analyses have been used in
commercially important species to identify chromosomal polymorphisms and their role
in the lethality and productivity of reproducers (Porto-Foresti et al. 1998), determine
ploidy level (Fopp-Bayat & Woznicki 2006) and characterize hybrids (e.g. Galetti 1991;
morphologically and meristically, identifying cytogenetic markers may be particularly appropriate. The present study demonstrated that, despite marked karyotypic similarity,
E. brasilianus can be differentiated from Diapterus species by their C- banding pattern
and nucleolus organizer pairs. Thus, investigating other cytogenetic markers may also allow individualization of additional karyotypic characteristics, with potential use in cultivation and management programs.
Acknowledgements
The authors thank the National Counsel of Technological and Scientific Development (CNPq - Proc. 141234/2009-1) for financial support and the research scholarship awarded to LLC and José Garcia Jr. for taxonomic identification of the specimens.
References
Aguilar C.T., Corrêa M.M.O. & Galetti P.M. (1998) Chromosome associations by
centromeric heterochromatin in marine fishes. Chromosome Science2, 73–76.
Aguilar C.T. & Galetti P.M. (1997) Chromosomal studies in South Atlantic serranids
(Pisces, Perciformes). Cytobios 89, 105–114.
Aguilar C.T. & Galetti P.M. (2008) Chromosome mapping of 5S rRNA genes
differentiates Brazilian populations of Leporellus vittatus (Anostomidae,
Characiformes). Genetic and Molecular Biology 31, 188–194.
Avila-Poveda O.H. & Lamouroux-López S.L. (2006) Saline acclimation of striped
mojarra Eugerres plumieri (Cuvier 1830) and optimal dosage of carp pituitary extract
(CPE) to induce spawning. Reviews in Fish Biology and Fisheries 17, 11-19.
Barros-e-Silva A.E. & Guerra M. (2009) The meaning of DAPI bands observed after
Bensam P. (2000) A review of marine finfish culture experiments in India. In: Marine
Research and Management (ed. by Pillai V.N., Menon N.G.), pp. 802-817. Central
Marine Fisheries Research Institute, Kochi.
Cervigon F. (1993) Los peces marinos de Venezuela, Fundacion Cientıfica Los Roques,
DF, VEN.
Chen W.J., Ruiz-Carus R. & Ortí G. (2007) Relationships among four genera of mojarras (Teleostei: Perciformes: Gerreidae) from the western Atlantic and their
tentative placement among percomorph fishes. Journal of Fish Biology 70, 202-218.
Cipriano R.R., Fenocchio A.S., Artoni R.F., Molina W.F., Noleto R.B., Kantek D.L.Z. & Cestari M.M. (2008) Chromosomal studies of five species of the marine fishes from the Paranaguá bay and the karyotypic diversity in the marine Teleostei of the Brazilian
coast. Brazilian Archives Biology Technology 51, 303-314.
Cross I., Merlo A., Manchado M., Infante C., Cañavate J.P. & Rebordinos L. (2006)
Cytogenetic characterization of the sole Solea senegalensis (Teleostei:
Pleuronectiformes: Soleidae): Ag-NOR, (GATA)n , (TTAGGG)n and ribosomal genes
by one-color and two-color FISH. Genetica 128, 253-259.
Eiras-Stofella D.R. & Fanta E. (1991) Ontogenesis of Eugerres brasilianus (Cuvier,
1830) (Pisces - Gerreidae) obtained by fertilization “in vitro”. Revista de Biología
Marina 26, 21-36.
Eschmeyer W.N. (1998) Catalog of fishes. California Academy of Science, CA, USA.
Fernandes C.A. & Martins-Santos I.C. (2006) Mapping of the 18S and 5S ribosomal
RNA genes in Astyanax altiparanae Garutti & Britski, 2000 (Teleostei, Characidae)
Ferro D.A.M, Néo D., Moreira-Filho O. & Bertollo L.A.C. (2001) Nucleolar organizing
regions, 18S and 5S rDNA in Astyanax scabripinnis (Pisces, Characidae): populations
distribution and functional diversity. Genetica 110, 55-62.
Flajshans M., Rab P. & Dobosz S. (1992) Frequency analyses of active NORs in nuclei
of artificially induced triploid fishes. Theoretical and Applied Genetics 85, 68-72.
Fopp-Bayat D. & Woznicki P. (2006) Verification of ploidy level in sturgeon larvae.
Aquaculture Research 37, 1671-1675.
Fujiwara A., Abe S., Yamaha E., Yamazaki F. & Yoshida M.C. (1998) Chromosomal localization and heterochromatin association of ribossomal RNA gene loci and
silver-stained nucleolar organizer regions in salmonid fishes. Chromosome Research 6,
463-471.
Galetti P.M. (1991) Chromosome complement of induced hybrids of the fish
Prochilodus marggravii and Prochilodus affinis (Prochilodontidae, Characiformes). Revista Brasileira de Genética 14, 203-207.
Galetti P.M., Aguilar C.T. & Molina W.F. (2000) An Overview of marine fish
cytogenetics. Hydrobiologia 420, 55-62.
Gold L.C., Shipley N.S. & Powers P.K. (1990) Improved methods for working with fish
chromosomes with a review of metaphase chromosome banding. Journal of Fish
Biology 37, 563-575.
Grijalva-Chon M., Núñez S., Quevedo R. & Castro L. (1996) Ictiofauna de la laguna
costera La Cruz. Sonora. Ciencias Marinas 22, 129-150.
Howell W.M. & Black D.A. (1980) Controller silver staining of nucleolus organizer
region with protective colloidal developer: a 1 - step method. Experientia 36,
1014-1015.
Iwatsuki Y., Kimura S. & Yoshino T. (2002) A new species: Gerres microphthalmus
(Perciformes: Gerreidae) from Japan with notes on limited distribution, included in the
“G. filamentosuscomplex”. Ichthyological Research 49, 133-139.
Jacobina U.P., Cioffi M.B., Souza L.G.R., Calado L.L., Tavares M., Manzella Jr. J., Bertollo L.A.C. & Molina W.F. (2011) Chromosome mapping of repetitive sequences in Rachycentron canadum (Perciformes: Rachycentridae): implications for karyotypic
evolution and perspectives to biotechnological uses. Journal of Biomedicine and
Biotechnolology 2011, 218-231.
Mantovani M., Abel L.D.S. & Moreira-Filho O. (2005) Conserved 5S and variable 45S
rDNA chromosomal localisation revealed by FISH in Astyanax scabripinnis (Pisces,
Characidae). Genetica 123, 211-216.
Martinez J.L., Morán P., Garcia-Vazquez S. & Pendás A.M. (1996) Chromosomal
localization of the major and 5S rRNA genes in the European eel (Anguilla anguilla).
Cytogenetics and Cell Genetics 73, 149-152.
Martins C. & Galetti P.M. (1999) Chromosomal localization of 5S rDNA genes in
Leporinus fish (Anostomidae, Characiformes). Chromosome Research 7, 363-367.
Molina W.F. (2001) An alternative method for mitotic simulation in fish cytogenetics.
Chromosome Science 5, 149-152.
Molina W.F. (2007) Chromosomal changes and stasis in marine fish groups. In: Fish
Cytogenetics (ed. by Pisano E., Ozouf-Costaz C., Foresti F., Kapoor B.G.), pp. 69-110,
Science Publishers, EN, UK.
improvement of fish cytogenetic preparations. Genetics and Molecular Research 9,
1807-1814.
Molina W.F. & Bacurau T.O.F. (2006) Structural and numerical chromosome
diversification in marine Perciformes (Priacanthidae and Gerreidae). Cytologia 71,
237-242.
Molina W.F. & Galetti P.M. (2002) Robertsonian rearrangements in the reef fish
Chromis (Perciformes, Pomacentridae) involving chromosomes bearing 5S rRNA
genes. Genetics and Molecular Biology 25, 373-377.
Molina W.F. & Galetti P.M. (2004) Karyotypic changes associated to the dispersive
potential on Pomacentridae (Pisces, Perciformes). Journal of Experimental Marine
Biology and Ecology 309, 109-119.
Molina W.F., Maia-LIma F.A. & Affonso P.R.A.M. (2002) Divergence between
karyotypical pattern and speciation events in Serranidae fish (Perciformes). Caryologia
55, 299-305.
Morán P., Pendás A.M., García-Vázquez E. & Izquierdo J. (1994) Genetic variation
among Atlantic salmon in six Spanish rivers. Journal of Fish Biology 45, 831-837.
Motta-Neto C.C., Cioffi M.B., Bertollo L.A.C. & Molina W.F. (2011) Extensive chromosomal homologies and evidence of karyotypic stasis in Atlantic grunts of the
genus Haemulon (Perciformes). Journal of Experimental Marine Biology and Ecology
401, 71-79.
Nelson J.S. (2006) Fishes of the World. John Wiley and Sons New York, NY, USA.
Pendás A.M., Morán P., Martínez J.L. & García-Vázquez E. (1995) Aplications of 5S rDNA in Atlantic salmon, brown trout, and in Atlantic salmon x brown trout hybrid
Pinkel D., Straume T. & Gray J.W. (1986) Cytogenetic analysis using quantitative,
high-sensitivity, fluorescence hybridization. Proceedings of the National Academy
Sciences of the United States of America 83, 2934-2938.
Porto-Foresti F., Oliveira C., Rigolino M.G., Tabata Y.A. & Foresti F. (1998) Possible lethal effect related to an inversion involving the nucleolar organizer region in rainbow
trout, Oncorhynchus mykiss. Cytogenetics and Cell Genetics, 81 131-138.
Ráb P. (1991) Fish cytogenetics and its application in fish reproductive biology.
Bulletin of the Institute of Zoology Academia Sinica 16, 357-373.
Ravisankar T., Sarada C. & Krishnan M. (2005) Diversification of fish culture and exports among major shrimp-producing countries of Asia: A spatial and temporal
analysis. Agricultural and Resource Economics Review 18, 187-195.
Rocha E.C. & Molina W.F. (2008) Cytogenetic analysis in western Atlantic snappers
(Perciformes, Lutjanidae). Genetics and Molecular Biology 31, 461-467.
Ruiz-Carus R. & Uribe-Alcocer M. (2003) Phylogenetic assessment of Eucinostomus
gula, Eugerres plumieri, and Diapterus auratus (Pisces: Gerreidae) based on allozyme
and mtDNA analyses. Caribbean Journal of Science 39, 109-115.
Ruiz-Carus R. & Uribe-Alcocer M. (2004) Karyotype analysis of Eucinostomus
argenteus, E. gula, E. harengulus, and Eugerres plumieri (Teleostei, Gerreidae) from
Florida and Puerto Rico. Environmental Biology of Fishes 67, 269-276.
Schweizer D. & Loidl J. (1987) A model for heterochromatin dispersion and the
evolution of C-band patterns. Chromosomes Today 9, 61-74.
Sumner A.T. (1972) A simple technique for demonstrating centromeric heterocromatin.
Suzuki H., Sakurai S. & Matsuda Y. (1996) Rat rDNA spacer sequences and chromosomal assignement of the genes to the extreme terminal region of chromosome 19. Cytogenetics and Cell Genetics 72, 1-4.
Vicari M.R., Artoni R.F. & Bertollo L.A.C. (2003) Heterochromatin polymorphism
associated with 18S rDNA: a differential pathway among Hoplias malabaricus fish
populations. Cytogenetic and Genome Research 101, 24-28.
Vicari M.R., Moreira F.O., Artoni R. & Bertollo L.A.C. (2006) Basic and molecular cytogenetics in freshwater Cichlidae (Osteichthyes, Perciformes) Karyotypic
CAPÍTULO II
Artigo submetido no Journal of Biotechnology and Biomedicine
Evolutionary dynamics of rDNA genes in the chromosomes of the Eucinostomus fishes: cytotaxonomic and karyoevolutive implications
L. L. Calado, L. A. C. Bertollo§, M. B. Cioffi§, G. W. W. F. da Costa, U. P.
Jacobina, W. F. Molina*
Department of Cell Biology and Genetics, Federal University of Rio Grande do Norte,
Natal, Rio Grande do Norte, Brazil.
§Department of Genetics and Evolution, Federal University of São Carlos, São Carlos,
São Paulo, Brazil.
Abstract
Several chromosomal features of Gerreidae fish have demonstrated to be conservative. In this group, it is unclear whether the high degree of chromosomal stasis is maintained when analyzing more dynamic regions of chromosomes, such as rDNA sites that generally have a higher level of variability. Thus, cytogenetic analyses were performed
on three Atlantic species of the genus Eucinostomus using conventional banding
(C-banding, Ag-NOR), AT and GC-specific fluorochromes and FISH mapping of telomeric sequences and 5S and 18S rDNA sites. The results showed that although the karyotypical macrostructure of these species is similar (2n=48 chromosomes, simple Ag-NORs seemingly located on homeologous chromosomes and centromeric heterochromatin pattern), there are differences in the position of rDNA ribosomal subunits 5S and 18S. Thus, the ribosomal sites have demonstrated to be effective
cytotaxonomic markers in Eucinostomus, presenting a different evolutionary dynamics
in relation to other chromosomal regions and allowing access to important evolutionary changes in this group.