FACULDADE DE MEDICINA VETERINÁRIA E ZOOTECNIA
APLICAÇÃO DE CONJUGADO DE MICROESFERAS DE
POLIESTIRENO PARA PREPARO DE RNA NO
DIAGNÓSTICO DA CINOMOSE CANINA POR RT-qPCR
CLAUDIA DE CAMARGO TOZATO
Botucatu-SP
FACULDADE DE MEDICINA VETERINÁRIA E ZOOTECNIA
APLICAÇÃO DE CONJUGADO DE MICROESFERAS DE
POLIESTIRENO PARA PREPARO DE RNA NO
DIAGNÓSTICO DA CINOMOSE CANINA POR RT-qPCR
CLAUDIA DE CAMARGO TOZATO
Tese apresentada junto ao Programa de Pós-Graduação em Medicina Veterinária para obtenção do título de Doutor.
Orientador: Prof. Adj. João Pessoa Araújo Junior
Botucatu/SP
Claudia de Camargo Tozato. Aplicação de conjugado de microesferas de poliestireno para preparo de RNA no diagnóstico da cinomose canina por RT-qPCR. Defesa: 29/08/2014. Local: FMVZ/UNESP – Campus de Botucatu/SP.
COMISSÃO EXAMINADORA
Prof. Adj. João Pessoa Araújo Junior Presidente e Orientador
Departamento de Microbiologia e Imunologia
Instituto de Biociências – UNESP – Botucatu – São Paulo
Prof. Adj. Marcos Bryan Heinemann Membro Titular
Departamento de Medicina Veterinária Preventiva e Saúde Animal Faculdade de Medicina Veterinária e Zootecnia – USP – São Paulo
Profa. Tit. Clarice Weis Arns Membro Titular
Departamento de Genética, Evolução e Bioagentes
Instituto de Biologia – UNICAMP – Campinas – São Paulo
Profa. Tit. Maria Terezinha Serrão Peraçoli Membro Titular
Departamento de Microbiologia e Imunologia
Instituto de Biociências – UNESP – Botucatu – São Paulo
Prof. Ass. José Paes de Oliveira Filho Membro Titular
Departamento Clínica Veterinária
Faculdade de Medicina Veterinária e Zootecnia – UNESP – Botucatu – São Paulo
Profa. Luciane Alarcão Dias Melicio Membro Suplente
Departamento de Patologia
Faculdade de Medicina de Botucatu – UNESP – Botucatu – São Paulo
Profa. Ass. Márcia Guimarães da Silva Membro Suplente
Departamento de Patologia
Faculdade de Medicina – UNESP – Botucatu – São Paulo
Profa. Adj. Alexandrina Sartori Membro Suplente
Departamento de Microbiologia e Imunologia
DEDICATÓRIA
AGRADECIMENTOS
Registro meus agradecimentos a todos os que compartilharam o trilhar de mais esse caminho percorrido, contribuindo direta e indiretamente, para que eu realizasse esta pesquisa, auxiliando-me e dando-me forças nos momentos em que mais precisei.
Minha gratidão, em primeiro lugar, a Deus, por estar comigo em todos os momentos, sendo meu refúgio e fortaleza nos momentos mais difíceis. A Ele, minha eterna gratidão.
Agradeço especialmente à minha família, pelo apoio para que eu concretizasse
essa pesquisa. Meus pais Elizeu e Cristina: ͆A presença de vocês significou segurança
е a certeza de quе não estou sozinha nessa caminhada͇. Minhas irmãs: Heloisa e
Carolina, pelo amor maior e incondicional.
Ao meu orientador João Pessoa, o qual possibilitou o desenvolvimento deste trabalho, esteve sempre presente durante todo o percurso, me auxiliou, incentivou, orientou e contribuiu para meu crescimento profissional. Levo para a vida um exemplo de educador e de profissional.
A todos os professores, alunos e funcionários do Departamento de Microbiologia e Imunologia do Instituto de Biociências, meus sinceros agradecimentos: Dani, Regis, Peruca, Ana, Larissa, Ivana, Aline, Rafael, Luís, Lulinha, Prof.ª Ângela, Prof.ª Terue, Prof.ª Vera, Prof.ª Sandra, Prof. Ari, Prof. Ramon, Prof.ª Terezinha, Prof.ª Lurdinha e Prof. Maurício. Foram importantes companheiros e auxiliaram na minha vida acadêmica.
Aos companheiros de colaboração prof. Valber e Carolina, pela colaboração e ampliação da aplicação deste trabalho.
Fukuta, Marianna, Thays Mizuki, Jacque, Leila, Sueli, Marlous, Ploc (Elaine), Andreza, Lidiany, Flávio, Curan (Ricardo) e Claudia Filoni. Cada um de vocês tem um lugar especial no meu coração.
Ao Prof. Paes do Departamento de Moléstias Infecciosas que permitiu a coleta de material para realização do trabalho. Ao professor Abelardo e doutorando Otávio Valério da Universidade Federal de Viçosa, por fornecer as células, material indispensável para realização deste trabalho.
À Vivian Zadra, pela amizade que começou com este trabalho e será para toda vida. À Noemi Gardinali, amiga de longa jornada, que mesmo longe sempre esteve presente. À Bruna Bottino, pela sua eterna amizade e companheirismo. À Flávia, companheira de infância, que sempre esteve ao meu lado. À Dani, Mia, Rosani e Lane, minhas queridas amigas de todas as horas.
A todos que contribuíram para minha formação acadêmica, em especial aos alunos Elis, Michele, Marlise, Brígida, Aline, Tonel, Ana, Juliana e professores Alice e Amauri Alfieri, da Universidade Estadual de Londrina, que compartiram o início dos estudos com vírus na minha vida. Ao Prof. Laurenil Gaste (in memorian) que sempre
me incentivou e acreditou em mim, nos deixou o legado da sua bagagem de vida, o ensinamento eterno, sua estrela estará sempre brilhando.
Aos professores Clarice, Marcos, Terezinha, José Filho, membros da banca examinadora, que corrigiram e contribuíram para a edificação dessa tese, muito obrigada pela paciência e dedicação.
LISTA DE TABELAS
TABELA 1. Comparison of absolute viral quantification of urine sample, urine centrifuged, urine ultracentrifuged and urine dilution in the A and B
LISTA DE FIGURAS
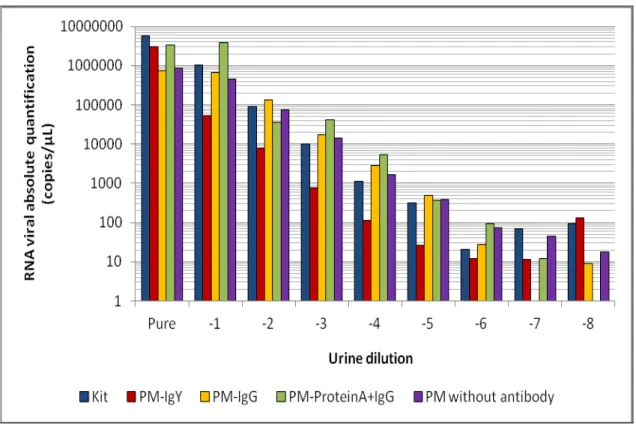
FIGURA 1. VDST under RT-qPCR positive urine inoculation….…..………. 44
FIGURA 2. CDV RNA copies/µL from roller, clarification and concentration steps……….…
45
FIGURA 3. Sucrose concentration in brix scale, protein quantification
(µg/mL) and CDV RNA quantification (copies/µL) of fractions collected from
sucrose gradient……… 46
FIGURA 4. Dot Blot of collected fractions (1-8) in sucrose gradient to CDV purification………
47
FIGURA 5. Analytical sensibility of RT-qPCR using RNA extraction by the conventional method (kit), PM-IgY, PM-IgG, PM-ProteinA+IgG and PM
without antibody……….…….. 49
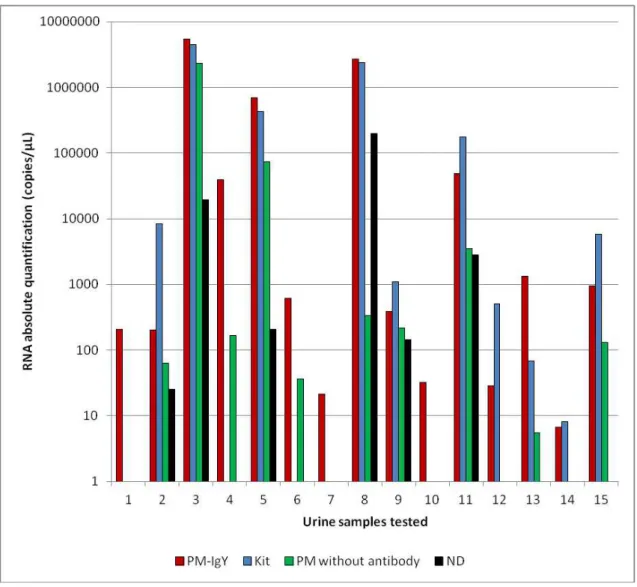
FIGURA 6. Diagnostic sensibility of RT-qPCR using RNA extraction by the conventional method (kit), PM-IgY, PM without antibody and urine directly
in the polypropylene tube (ND)……….…….………....
SUMÁRIO
CAPÍTULO 1………. 01
1. INTRODUÇÃO... 02
2. REVISÃO DE LITERATURA... 04
3. OBJETIVOS... 13
CAPÍTULO 2... 14
4. TRABALHO CIENTÍFICO 1.: Canine Distemper Virus detection by SYBR Green One-Step RT-qPCR assay... 15
CAPÍTULO 3... 31
5. TRABALHO CIENTÍFICO 2.: Application of polystyrene microspheres immunoconjugate in canine distemper diagnosis by RT-pPCR……… 32
CAPÍTULO 4... 57
6. CONCLUSÕES... 58
TOZATO, C.C. Aplicação de conjugado de microesferas de poliestireno para preparo de RNA no diagnóstico da cinomose canina por RT-qPCR. Botucatu, 2014. 63p. Tese (Doutorado) - Faculdade de Medicina Veterinária e Zootecnia, Campus de Botucatu, Universidade Estadual Paulista “Júlio de Mesquita Filho”. Botucatu, São Paulo.
RESUMO
Devido às diversas formas de apresentação clínica e aos sinais clínicos comuns com outras afecções, o diagnóstico laboratorial do CDV (Canine distemper virus) se torna necessário. A RT-PCR seguida da NESTED com amostras de urina é atualmente a técnica mais recomendada para o diagnóstico do CDV. No Capítulo 2, foi desenvolvida a técnica de One-Step-RT-qPCR utilizando o sistema de detecção Syber Green I. Foram testados e comparados três kits comerciais (Sigma®, Promega® e Qiagen®). Foi determinada a sensibilidade analítica de dois sistemas de detecção (Sistema A e Sistema B) utilizando uma curva padrão de RNA sintético. Os três kits comerciais testados detectaram o vírus em 100% das urinas provenientes de animais sintomáticos e o kit da Qiagen® mostrou melhores resultados de reação. O Sistema A detectou aproximadamente 10 cópias de RNA por microlitro e no Sistema B detectou 100 cópias de RNA por microlitro na RT-qPCR e 10 cópias por microlitro quando seguida da NESTED-RT-qPCR. No Capítulo 3 foram desenvolvidos três imunoconjugados de microesferas de poliestireno (MP) utilizando anticorpos policlonais hiperimunes anti-CDV (IgY, IgG e Proteína A+IgG) para o preparo de RNA e aplicação na RT-qPCR. A aplicação dos conjugados foi comparada ao método convencional de extração de RNA por kit comercial. Os conjugados foram padronizados e a sensibilidade analítica foi determinada através de curva padrão de RNA sintético. A sensibilidade diagnóstica foi determinada utilizando-se conjugado com IgY, MP sem anticorpos conjugados e urina adsorvida diretamente nos tubos de qPCR.
TOZATO, C.C. Application of polystyrene microspheres immunoconjugates in canine distemper diagnosis by RT-qPCR. Botucatu, 2014. 63p. Thesis (Doctorade) - Faculdade de Medicina Veterinária e Zootecnia, Campus de Botucatu, Universidade Estadual Paulista “Júlio de Mesquita Filho”. Botucatu, São Paulo.
ABSTRACT
The diagnosis of canine distemper is usually based on clinical suspicion of the disease. However, due to the different clinical forms of the disease and other disorders common to clinical signs, the laboratory diagnosis is necessary. The RT-PCR followed by NESTED is currently the method used to diagnosis of CDV (Canine Distemper Virus). The Chapter 2 aimed to standardized the technique of One-Step RT-qPCR-using Syber Green I system. Three commercial kits (Sigma®, Promega® and Qiagen®) were tested and compared. Analytical
sensitivity of two detection systems (System A and System B) was determined using a standard curve. The three commercial kits tested detected the virus in
100% of samples from symptomatic dogs and the kit of Qiagen® showed better
results. The System A detected approximately 10 RNA copies/µL and System B detected approximately 100 RNA copies/ µL in RT-qPCR and 10 RNA copies/ µL when followed by NESTED-qPCR. In Chapter 3 three immunoconjugates were developed with polystyrene microspheres (PM) using polyclonal hyperimmune anti-CDV antibody (IgY, IgG and protein A+IgG) to prepare RNA to canine distemper molecular diagnosis and comparison with the conventional method of RNA extraction by commercial kit. Conjugates were standardized and the analytical sensitivity was determined by standard curve. The diagnostic sensitivity was determined using IgY-conjugated, PM without conjugated antibodies and urine adsorption directly qPCR in tubes.
1. INTRODUÇÃO
A cinomose é uma doença sistêmica altamente infecciosa que acomete várias espécies de animais e se apresenta de forma enzoótica no mundo. A maior ocorrência e importância econômica desta enfermidade estão relacionadas à infecção em cães jovens não vacinados. A natureza contagiosa e as altas taxas de mortalidade de cães infectados pelo Canine distemper Virus (CDV) torna necessário o desenvolvimento de ferramentas para o diagnóstico acessível da doença.
Atualmente, a RT-PCR e RT-qPCR de amostras de urina são utilizadas para o diagnóstico da doença. Porém essas técnicas apresentam como limitação o custo, o tempo e o risco de contaminação. Microesferas de poliestireno (MP) são amplamente aplicadas no teste de aglutinação em látex (LAT) em uma variedade de testes analíticos para diagnóstico de infecções por ser um método simples, rápido, específico e de baixo custo. Até o presente momento, o LAT não foi desenvolvido para o diagnóstico da cinomose canina, provavelmente pelo baixo limiar de detecção. As microesferas de poliestireno possuem afinidade com proteínas pelas suas características físico-quimicas, são consideradas livres de contaminação e possíveis inibidores de PCR, representando uma prática e eficiente tecnologia para o preparo de ácido nucléico viral, quando previamente conjugadas a um anticorpo específico.
contaminação e inibidores; iii) eliminação da etapa de extração de ácido nucleico que aumenta o custo e o tempo de preparo da amostra.
O CDV foi isolado, propagado e purificado para inoculação em galinha (Galus Galus domesticus) para obtenção de IgY hiperimune. As MP foram conjugadas a IgY hiperimune anti-CDV, IgG de coelho hiperimune anti-CDV e adsorção prévia de Proteína A para posteriormente serem conjugadas com IgG hiperimune de coelho anti-CDV.
Para comparação da sensibilidade analítica, uma urina positiva na RT-qPCR para CDV foi diluída e os três conjugados e MP sem adsorção de anticorpos foram testados.
A sensibilidade diagnóstica foi avaliada utilizando-se o conjugado de MP com IgY, devido aos resultados obtidos de sensibilidade analítica e às vantagens de produção, MP sem adsorção prévia de anticorpos e urina diretamente no strip de qPCR sem MP.
2. REVISÃO DE LITERATURA
2.1. Características gerais da infecção pelo vírus da cinomose canina (CDV - Canine distemper virus)
A cinomose é uma doença infecciosa causada pelo CDV, um membro do gênero Morbillivirus, subfamília Paramyxovirinae, família Paramyxoviridae e
ordem Mononegavirales, que acomete vários animais da ordem Carnívora e é
antigenicamente semelhante ao vírus do sarampo e da peste dos pequenos ruminantes. O vírion pleomórfico com aproximadamente 150 a 250 nm de diâmetro é constituído por envelope lipoproteico, derivado da membrana da célula hospedeira, capsídeo de simetria helicoidal e genoma RNA de fita simples não segmentado de polaridade negativa (GREENE; APPEL, 1998).
O genoma viral contém seis genes que codificam seis proteínas estruturais; uma proteína matriz do envelope (M), duas glicoproteínas de superfície (H e F), duas proteínas associadas à transcriptase (P e L) e uma proteína do nucleocapsídeo (N). O RNA viral é associado à nucleoproteína do capsídeo (N), ao complexo polimerase (polimerase viral (L) e seu cofator fosfoproteína (P)). As proteínas N, P e L e o RNA viral constituem a ribonucleoproteína (RNP), a qual direciona a síntese sequencial do RNA mensageiro. As glicoproteínas do envelope viral do CDV são as proteínas F (fusão) e H (hemaglutinina). A proteína H possui atividade de hemaglutinação, é responsável pela fixação do vírus na célula hospedeira e é alvo de anticorpos neutralizantes. A glicoproteína F é responsável pela penetração do vírus na célula hospedeira por fusão do envelope viral com a membrana celular. Duas proteínas não-estruturais (C e V) são produzidas por duas sequências abertas de leitura alternativa (ORF) do gene P (ZUBRIGGEN et al., 1995; GREENE & APPEL, 1998; LAMB; KOLAKOFSKY, 2001).
molecule) ou CD 150 é uma glicoproteína expressa na membrana celular de células do sistema imune e é o receptor celular para os Morbillivirus. O CDV se liga seletivamente às moléculas SLAM através das suas glicoproteínas de superfície (H e F), permitindo rápida disseminação nos tecidos linfoides. A imunossupressão ocorre não somente pela morte celular induzida pelo vírus, mas também porque o vírus inibe respostas de interferon (IFN) e citocinas de células linfóides via a expressão do gene P e virulência das proteínas V e C (VON MESSLING et al., 2003). Devido à ausência desse receptor apropriado, o CDV não se replica bem e não é facilmente isolado em células epiteliais de linhagem contínua utilizadas em laboratório, tornando o diagnóstico laboratorial dificultoso (GREENE; VALDEVELDE, 2012). Diferentemente dos CDV isolados de amostras de campo, as cepas de vacinas de CDV são adaptadas em cultivo de células VERO (oriundas de rim de macaco verde africano [Cercopithecus aethiops]), apresentam menor patogenicidade e se ligam ao receptor “ heparin-like cell receptor”, presente em células que não pertencem ao sistema imune, como as células epitelias de linhagem contínuas (FUGITA et al., 2007). Após a caracterização da molécula utilizada como receptor para os Morbillivirus,
inicialmente em estudo com o Vírus do Sarampo, foram desenvolvidas células recombinantes para isolamento do CDV, modificadas geneticamente, que
expressam o receptor SLAM de cães (Canis lupus familiaris). As células VERO
dog SLAM tag têm sido amplamente utilizadas para isolamento e diferentes ensaios com CDV (TATSUO et al., 2001; ZIPERLE et al., 2010; PRATAKPIRIYA et al., 2012).
confere imunidade em longo prazo. O gene H codifica uma proteína-chave tanto para o vírus quanto para hospedeiro, pois o vírus utiliza essa proteína para ligação aos receptores da célula na primeira etapa da infecção e a proteína induz resposta imune nos hospedeiros. Entretanto, estudos moleculares demonstraram que a proteína H é pouco conservada, apresentando aproximadamente 10% de variabilidade entre as cepas de CDV (MARTELLA et al., 2006). Com base na diversidade genética do gene H, é possível caracterizar a maioria das cepas de campo do CDV em seis grandes linhagens genéticas, referidas como América 1 e 2, Ásia 1 e 2, européia e Ártica, que são relacionadas de acordo com a distribuição geográfica (MARTELLA et al., 2006; NEGRÃO et al., 2006). Estudos recentes demonstram que substituições de aminoácidos nos sítios 530 e 549 (ambos classificados em domínios de ligação do receptor da proteína H) ocorrem predominantemente em cepas isoladas de CDVs de novas espécies hospedeiras, indicando que a propagação do vírus em espécies não-canina está associada à evolução nesses sítios (PRATELLI, 2011).
A principal forma de transmissão do CDV ocorre através do contato direto dos animais com secreções nasais, orais e urina de animais infectados e em curtas distâncias por aerossóis. Após a infecção, os animais excretam o vírus nos fluidos corporais por períodos prolongados (ARNS et al. 2007). A infecção pelo CDV pode ser subclínica, provocar sinais gastrointestinais e/ou respiratórios, frequentemente associados ao acometimento do sistema nervoso central (SNC) que também podem ocorrer como uma manifestação tardia da infecção sem outros sinais (CARVALHO et al., 2012). Após o contato, o CDV se replica em macrófagos e células linfóides do trato respiratório superior. A disseminação sistêmica é mediada pelas células infectadas, tais como linfócitos, monócitos e plaquetas e/ou através de vírus não associado a células, disseminando a infecção para vários órgãos (ARNS et al., 2007).
(APPEL et al. 1994; HARDER et al., 1997, VAN DE BILDT et al, 2002; VON MESSLING et al., 2003). O crescente número de animais que se infectam naturalmente e surtos em primatas não humanos têm sido alvo de estudos para compreender as mutações envolvidas e o caráter epidemiológico da doença (YOSHIKAWA et al.,1989; QIU et al., 2011; SAKAI et al., 2013).
O controle do CDV é difícil pelo grande número de espécies selvagens que podem ser infectadas. Apesar do CDV acometer uma grande variedade de animais carnívoros, sua maior importância na rotina veterinária está relacionada com manifestações clínicas de cães domésticos. A infecção pelo CDV se apresenta de forma enzoótica no mundo inteiro, ocorrendo com maior frequência em cães jovens não vacinados (LAMB; KOLAKOFSKY, 2001; ARNS et al., 2007).
2.2. Diagnóstico do CDV
O diagnóstico da cinomose é geralmente fundamentado em suspeita clínica e muitas vezes considerado simples. É comum que a doença seja diagnosticada clinicamente pela associação entre idade do animal e sintomatologia clínica sem a utilização do diagnóstico laboratorial. Esta medida pode resultar em diagnóstico equivocado, pois os sinais clínicos são inespecíficos e outras enfermidades podem apresentar sinais clínicos em comum com a cinomose (CORREA; CORREA, 1992; ELIA et al., 2006).
Para o diagnóstico laboratorial, na dependência da apresentação clínica e da evolução da cinomose, o vírus pode estar presente em várias amostras biológicas. Nos diferentes estágios da infecção, amostras biológicas como órgãos, urina, sangue total, leucócitos, fezes, saliva, líquor, secreção respiratória e ocular podem apresentar concentrações variadas do vírus. Nestas amostras são aplicadas técnicas de detecção de antígeno ou de RNA viral, como inoculação em cultivo celular, imunofluorescência direta, ELISA direto com anticorpo de captura, imunoistoquímica e hibridização in situ
podem ser somente realizadas no post-mortem. A urina é a amostra biológica que apresenta maiores vantagens para diagnóstico ante mortem devido à sensibilidade analítica e o método de colheita não invasivo (KIM et al., 2001; AMUDE et al., 2006; ELIA et al., 2006; AMUDE et al. 2007; SAITO et al., 2006).
Desde 1995 a reação em cadeia da polimerase com transcriptase reversa (RT-PCR) tem sido utilizada como ferramenta para o diagnóstico cinomose (SHIN et al., 1995). Trata-se de uma técnica com alta sensibilidade analítica, que varia de acordo com o tipo de amostra, método de extração de RNA e escolha dos primers e até o presente momento, é o método de
diagnóstico recomendado ante mortem da enfermidade (GEBARA et al., 2004;
SAITO et al., 2006).
A amplificação de ácidos nucleicos pela técnica de PCR quantitativa (qPCR) tornou-se padrão para o diagnóstico laboratorial de muitas doenças devido a maior sensibilidade e rápida detecção quando comparada a PCR convencional. A RT-qPCR tem sido empregada com sucesso para detecção e quantificação do CDV (ELIA et al., 2006). No entanto, as técnicas de RT-PCR e RT-qPCR apresentam alto custo devido à tecnologia empregada nas diferentes etapas de isolamento de ácidos nucleicos, amplificação do genoma viral e exigência de elevados níveis de garantia de controle de qualidade laboratoriais. Também foi demonstrada importância de realizar a NESTED-PCR para o CDV quando a PCR convencional é aplicada, uma vez que esta etapa é responsável pelo aumento de 60% da sensibilidade analítica da reação (KIM et al., 2001; RZEŻUTKA; MIZAK, 2002; JÓŹWIK; FRYMUS, 2005).
método de extração de ácido nucleico simples, rápido, barato, que possa ser aplicado na identificação de patógenos diretamente das amostras (UWATOKO et al., 1995).
Outro problema associado à extração de ácidos nucleicos de amostras biológicas por métodos tradicionais está relacionado ao pequeno volume de amostra utilizado, que pode induzir a resultados “falso negativos” em decorrência da inexistência do vírus na amostra selecionada para extração.
2.3. Microesferas de poliestireno
As microesferas de poliestireno, conhecidas como látex, são
amplamente utilizadas no Teste de Aglutinação em Látex (LAT - Látex
Agglutination Test), que é um método diagnóstico onde se utiliza um suporte sólido revestido com determinado anticorpo, mantido em uma solução coloidal, que aglutina na presença do antígeno-alvo formando grumos visíveis.
As partículas de látex foram desenvolvidas em 1947, e o primeiro método de imunoensaio através da aglutinação de látex foi utilizado para a detecção de doença autoimune (SINGER; PLOTZ, 1956). Nos últimos anos, houve um crescente aumento na utilização de métodos de diagnóstico que envolvam procedimentos imunológicos devido a alta especificidade e sensibilidade destes testes. A tecnologia do LAT têm sido aprimorada e aplicada em uma ampla variedade de testes analíticos para diagnóstico de infecções microbianas, doenças autoimunes, dosagem hormonal, detecção de drogas, entre outras. O LAT tem sido amplamente desenvolvido e utilizado como teste direto ou indireto para diagnóstico rápido de doenças virais. Durante a última década, foram publicados mais de 400 trabalhos em revistas de medicina veterinária, onde o LAT foi empregado como análise ou ferramenta de pesquisa. Além disso, mais de 300 diferentes doenças e biomoléculas podem ser detectadas utilizando essa técnica (ORTEGA-VINUESA; BASTOS-GONZÁLEZ, 2001; YOUSIF et al., 2001; MOLINA-BOLÍVAR; GALISTEO-GONZÁLEZ, 2005).
em quantidade suficiente para que para formação do complexo aglutinante visível (ORTEGA-VINUESA; BASTOS-GONZÁLEZ, 2001). Até o presente momento não foi desenvolvido o teste de aglutinação em látex para o diagnóstico da cinomose canina, muito provavelmente pelo baixo limiar de detecção, decorrente da necessidade de um grande número de partículas virais para a aglutinação.
Em relação à adsorção de imunoglobulinas na interface “sólida-líquida”, pequenas alterações nas condições experimentais (pH, força iônica, temperatura), podem resultar em grandes diferenças na adsorção devido às complexas interações envolvidas. Uma das principais características das imunoglobulinas é a propriedade de flexibilidade molecular. Este tipo de flexibilidade é designado para facilitar a formação de complexos antígeno-anticorpo. A flexibilidade das moléculas das classes de IgG de mamíferos difere em suas dimensões assim como em constituição iônica, implicando em taxas de adsorção distintas. Portanto imunoglobulinas provenientes de diferentes animais sensibilizados sob as mesmas condições com o mesmo agente, podem resultar em diferentes taxas de adsorção ao poliestireno, resultando em ensaios com diferentes valores de sensibilidade e especificidade (ORTEGA-VINUESA; BASTOS-GONZALEZ, 2001).
o sistema complemento dos mamíferos. Além disso, em relação à aplicabilidade em imunoensaios, as IgY apresentam ponto isoelétrico na faixa de 5,7-7,6, característica que permite alcançar complexos mais estáveis de látex-anticorpos e a distância filogenética que separa os mamíferos das aves permite que se possa obter IgY contra proteínas altamente conservadas. Devido a essas características, a IgY apresenta grande potencial para ser aplicada em testes imunológicos (ORTEGA-VINUESA; BASTOS-GONZALEZ, 2001).
Outro aspecto relacionado à sensibilidade do LAT, é a conformação posicional que os anticorpos adquirem quando conjugados à partícula de poliestireno, pois as imunoglobulinas têm a capacidade de se ligar aleatoriamente ao látex (MOLINA-BOLÍVAR; GALISTEO-GONZÁLEZ, 2005). A proteína A possui a propriedade de se ligar especificamente à porção Fc da imunoglobulina G de alguns mamíferos. A metodologia de utilização de placas revestidas com a proteína A em poliestireno é aplicada para ensaios de ELISA (Enzyme-Linked Immunosorbent Assay). Os anticorpos conjugados à proteína A expõem exclusivamente o fragmento Fab da imunoglobulina mantendo a orientação “end-on” das imunoglobulinas, permitindo um melhor desempenho do ensaio (NGAI et al., 1993).
2.4. Utilização de partículas ligadas como preparação do RNA para reação de PCR ou RT-qPCR
Existem várias pesquisas visando o desenvolvimento de técnicas para concentração/extração de ácido nucleico viral, com objetivo de aumentar a sensibilidade da reação de PCR (DE PAULA et al., 2001; KITTIGUL et al., 2001; KHONO et al., 2002).
separadas por diferença de campo magnético. A técnica apresenta como vantagens a concentração do agente a partir de um grande volume de fluido e a remoção de inibidores da PCR presente na amostra (OLSVIK, et al., 1994; YAO et al., 2009).
Atualmente, essa tecnologia é utilizada para aumentar a sensibilidade na detecção de patógenos. Inclusive em esferas ligadas a moléculas não específicas, a tecnologia apresenta quantitativamente grande capacidade de concentração e viabiliza a detecção em baixa concentração. Em especial, para vírus RNA, foi demonstrado que a eficiência de concentração foi centenas de vezes superior que o valor teórico, provavelmente devido a remoção de inibidores da polimerase e da transcrição reversa da amostra (SATOH et al., 2003; PARK et al., 2008).
3. OBJETIVOS
3.1. OBJETIVO GERAL
3.1.1. Aprimorar o diagnóstico laboratorial da cinomose canina por RT-qPCR.
3.2. OBJETIVOS ESPECÍFICOS
3.2.1. Padronizar a reação de One-Step-RT-qPCR para detecção e quantificação do vírus da cinomose canina.
3.2.2. Desenvolver, padronizar e aplicar microesferas de poliestireno conjugadas com anticorpos hiperimune anti-CDV de galinha, anticorpos hiperimune anti-CDV de coelho e anticorpos hiperimune anti-CDV de coelho com adorção prévia da Proteína A na reação de RT-qPCR para o diagnóstico da cinomose canina.
3.2.3. Produzir RNA sintético através de clonagem e transcrição in vitro para realização da quantificação absoluta das cargas virais.
3.2.4. Testar e comparar a reação de RT-qPCR e RT-qPCR seguida da NESTED-qPCR para verificação quanto à sensibilidade analítica.
3.2.5. Isolar, propagar e purificar o vírus da cinomose canina.
3.2.6. Inocular o vírus purificado em galinha para produção de IgY hiperimune anti-CDV.
3.2.7. Desenvolver e testar conjugados de imunoglobulinas (IgG, IgG+Proteína A e IgY) com microesferas de poliestireno.
4. Trabalho científico 1.
O artigo está nas normas da revista “Journal of Virological Methods” no formato de Short Communication.
Canine Distemper Virus detection by SYBR Green One-Step RT-qPCR assay
TOZATO, C.C; ARAÚJO JR, J.P*.
Laboratory of Animal and Human Virology, Univ Estadual Paulista, Botucatu,
Department of Microbiology and Immunology, Biosciences Institute,
–18618-970, São Paulo, Brazil.
*Corresponding Author: Laboratory of Animal and Human Virology, Univ
Estadual Paulista – UNESP, Department of Microbiology and Immunology,
Bioscience Institute, Distrito de Rubião Junior, s/n, Botucatu, São Paulo,
80035-050, Brazil. Tel.: +55 (14) 3880-0422, E-mail: jpessoa@ibb.unesp.br.
The authors of the manuscript disclose that there is no conflict of interest and
that there was no commercial relationship with the manufacturers of any
Abstract
Three commercial kits of One-Step RT-qPCR Syber Green based were
evaluated to canine distemper virus (CDV) diagnosis. Two systems of One-Step
Real-time RT-PCR (RT-qPCR) assays were developed to CDV RNA detection:
a RT-qPCR (System A) and a RT-qPCR followed by NESTED-RT-qPCR
(System B). The limits of detection of both systems were determined using
dilution series of 1.1 x 105 to 1.1 x 100 of synthetic RNA. A positive urine sample
was diluted in matrix until 10-5 fold. In addition, the same urine sample (100) was
tested with previously centrifugation and ultracentrifugation. The commercial
kits of one-step RT-qPCR assays detected CDV RNA in 10 (100%) urine
samples from symptomatic animals tested. The limit of detection of RT-qPCR of
System A was 11 copies of RNA/reaction and in the system B was 110 RNA
copies/µL. The System B, showed the limit of detection of 11 copies/µL when
succeed by NESTED-qPCR. The relationship between Ct values and RNA
concentration was linear. The CDV RNA was detected in diluted urine samples
until 10-3 and 10-2 in the System A and System B, respectively. The urine
centrifugation provided increased the analytical sensibility of the test and
showed to be useful for diagnostic routine. The RT-qPCR Syber Green
One-Step based is a sensitivity and specific method that will be available to support
routine canine distemper laboratory and to assist research projects that require
sensitive and quantitative subject.
Canine distemper (CD) is a severe and often lethal infectious disease
that affects dogs and a broad range of terrestrial and aquatic animals. The
disease is caused by Canine distemper virus (CDV), a Morbillivirus of
Paramyxoviridae family. CDV is an enveloped and pleomorphic virion, with
helical capsid that is associated to a single, non-segmented RNA negative
polarity genome. The viral genome contains six consecutive genes that
encodes six structural proteins: nucleocapsid protein (N), two transcriptase
associated proteins (P and L), protein envelope matrix (M), two surface
glycoproteins (F and H) and two nonstructural proteins encoded by two
sequences of alternative open reading frame (ORF) of the P gene (C and V)
(Greene and Valdevelde, 2012). The N gene is the most expressed gene and
well-conserved gene of Morbillivirus that encodes the most abundant structural
viral protein (Diallo, 1990).
The direct contact with nasal, oral secretions and urine of infected
animals is the major route of CDV transmission. Recently, outbreaks of
distemper has been identified in dogs and an increasing number of host
species, which has been the subject many studies to elucidate the potential
mutations and viral mechanisms that may be involved in the host susceptibility
(Bolt et al. 1997; Cottrell et al., 2013; Di Sabatino et al., 2014; Megid et al. 2009;
Megid et al., 2013; Seimon et al., 2013; Zhao et al., 2010). The clinical signs of
canine distemper consist of respiratory and/ or gastrointestinal, associated or
not with neurological symptoms (Greene and Valdevelde et al., 2012).
The diagnosis of canine distemper is often based on clinical suspicion,
similarity of clinical signs with other affections and the high spread of disease,
the laboratory diagnosis is crucial. In the past decade, several gel-based
molecular assays, were developed to CDV detection and the NESTED RT-PCR
has been shown the most sensible method of CDV diagnose (Di Francesco et
al., 2012; Frisk et al. 1999; Headley et al. 2009; Kim et al., 2001; Rosa et al.,
2012; Saito et al., 2006). Afterwards, RT-qPCR assays have been applied to
detect and quantify canine distemper virus in clinical specimens of naturally
infected dogs or to distinguish vaccinal from wild strains, using double step and
real-time TaqMan chemistry (Elia et al., 2006; Fischer et al., 2013; Scagliarini et
al. 2007; Wilkes et al., 2014). Recently, Surface plasmon resonance (SPR) and
electrochemical impedance spectroscopy (EIS) methodologies were also
developed to CDV detection (Basso et al., 2013).
Actually, there is a wide range of commercials reagents and kits of
RT-qPCR that use different chemistry amplification such as probes, DNA
intercalation, reverse transcription in Double-Steps or One-Steps. The
RT-qPCR yields depends on protocols standardization; reagents; gene target,
primers, template preparation and analytical method used. A diagnostic test
must be practical, rapid, sensitive, specific and large scale applicable (Bustin,
2002; Sergerie et al., 2007).
Various samples as blood, conjunctival swab and urine have been used
to CDV diagnosis. The urine has been the sample of choice due to ease
collection, not cause animal damage and mainly showed great sensibility in
different clinical presentations of canine distemper (Gerbara et al., 2004; Elia et
one-step qPCR Syber Green I based, standardized a technique of One Step
RT-qPCR, compared to NESTED-RT-qPCR and determined the analytical
sensibility of the two systems to detect CDV RNA in urine samples from dogs
with clinical suspicion of CDV infection.
Twenty urine samples from dogs were submitted to the Molecular
Diagnostic Laboratory, Institute of Bioscience, Universidade Estadual
Paulista-UNESP, Botucatu, São Paulo, Brazil. Ten urine samples from clinical suspicion
dogs of CD and 10 urines samples from healthy dogs were tested to kit
comparison.
One hundred microliters of urine samples were extracted using Total
RNA Purification Kit (7200, Norgen Biotek Corporation, Canada), according to
the manufacturer and stored at −80°C until further use.
To evaluate the ability of CDV RNA detection, RNAs extracted from 20
urine samples were tested. The RT-qPCR was performed using three
commercial kits of One-Step Syber Green I RT-PCR. The One Step RT-qPCRs
Syber Green I based were performed with QuantFast Sybr Green RT-PCR Kit
(Qiagen®, Crawley, United Kingdom); Sybr Green RT-qPCR (Sigma®, St. Louis,
MO, EUA) and GoTaq One-Step RT-qPCR System (Promega®, Madison, WI
USA) with previously pair of primers described by Elia et al., 2006, according to
each manufacturer instruction. A negative control was performed using
nuclease free water.
The analytical sensitivity of two methods of RT-qPCR were compared:
(System B), both using the QuantFast Sybr Green RT-PCR Kit (Qiagen,
Crawley, United Kingdom) which showed better dissociation curve, time of
reaction and CT average in commercial kits comparison.
The RT-qPCR of System A was performed using the primers pair
previously described by Elia et al. (2006) CDV-For (5’-AGC TAG TTT CAT CTT
AAC TAT CAA ATT-3’) and CDV-Rev (5’-TTA ACT CTC CAG AAA ACT CAT
GC-3’), which produced an amplicon of 83 bp (905–931 and 966–987
nucleotide positions, respectively) with the primers concentrations of systems of
300 nM.
The RT-qPCR of system B was performed using the pair of primers P-1
sense ACAGGATTGCTGAGGACCTAT-3’) and P-2 antisense
(5’-CAAGATAACCATGTACGGTGC-3’) (769–789 and 1055–1035 nucleotide
position of sense and antisense primers, respectively), described by Frisk et al.
(1999) producing an amplicon of 287 bp. The negative reactions were applied to
the NESTED-qPCR reaction carried out with CDV-for and CDV-rev primers pair
using the GoTaq® Green Master Mix (Promega®, Madison, WI, USA)
according to manufacturer's recommendations. The primers concentrations
used were 300 nM and 200 nM in the RT-qPCR and NESTED-qPCR,
respectively. The reactions were performed on a 7300 Real Time PCR System
(Applied Biosystems® Life Technologies Corp., Carlsbad, CA. USA) machine in
a final volume of 20 µL. The negative control was carried out with nuclease free
water.
The synthetic RNA was provided to access the efficiency and analytical
RT-qPCR sample was cloned in pGEM-T easy vector (Promega®, Madison, WI,
USA) and transformed into Escherichia coli DH-5α. The recombinant plasmid
was quantified in NanoDrop®ND-1000 Spectrophotometer. The CDV-plasmid
was treated with restriction enzymes NaeI and NdeI (Fermentas/Thermo
Scientific, Burlington, Canada) and separated by electrophoresis on a 1%
agarose gel. The fragment of 692 bp of CDV containing binding sites of the T7
polymerase was purified using Nucleospin® Gel and PCR Clean Up
(Macherey-NagelTM, Düren, Germany) according to the manufacturer's guidelines. The fragment was in vitro transcribed using MEGAshortscriptTM T7 (InvitrogenTM,
USA) according to the manufacturer's instructions. The product of transcription
was treated with DNAse I (Sigma-Aldrich, St. Louis, MO, EUA) to eliminate
residual DNA and has been tested by RT-qPCR kit without the reverse
transcriptase to accesses RNA purity.
The transcribed RNA was quantified in fluorimetric quatitation using
Qubit® 2.0 Fluorometer (Life Technologies Corporation, Carlsbad, CA), diluted in sample matrix in 10-fold serial dilutions and underwent the A and B systems
in duplicate. The standard curve of both systems was generated using 1.1 x 105
to 1.1 x 100 copies/µl.
One RT-qPCR positive urine sample was diluted until 10-5 in RT-qPCR
negative urine and, after RNA extraction, tested in duplicate for the two
RT-qPCR systems.
Additionally, to verify the RNA amount in the urine sample under
centrifugation, 1 ml of sample was centrifuged for 8,000 x g for 8 minutes and 1
pellet samples were used to RNA extraction as the other samples and
submitted to the two RT-qPCR systems.
All ten RNA extracted urine samples from clinical suspicion dogs (100%)
were positive in the one step RT-qPCR of the three manufacturers. Ten RNA
extracted urines samples from healthy dogs and the negative control did not
showed amplification and no picks in melting curve analyze. The RT-qPCR
duration and average CT values were 2:00h/27.8, 3:20h/24.3 and 2:45h/29.3,
for Qiagen®, Sigma® and Promega®, respectively. The melting temperature
peacks varied in 73,8 °C to 74,5 °C, 74,6 °C to 75,8 °C and 74,2 °C to 75,7 °C
to Qiagen®, Sigma® and Promega®, respectively. The Sigma® kit presented the
lowest average CT of samples. However, in melting curve analysis, the Sigma®
kit shows a broad base, nonspecific peaks and melting temperature variation of
1.2ºC. In Promega® and Qiagen® kit was observed melting temperature
variation of 1,5ºC and 0,7ºC, respectively, but no nonspecific peaks.
The standard curve was generated and showed linearity over the entire
quantification range of both systems. The system A showed slope of −3.5,
coefficient of linear regression (R2) of 0.99 and efficiency of 93.0%. The System
B showed slope of −3.65, linear regression (R2) 0.99 and efficiency of 87.0%.
The system A was able to detect synthetic CDV RNA until 1.1 x10 RNA
copies/µL. The system B amplified the synthetic RNA until 1.1x102 copies/µL in the RT-qPCR reaction and 1.1 x10 RNA copies/µL in NESTED-qPCR reaction.
The System A detected CDV RNA in diluted urine until 10-3 and the
System B until 10-2. The System A showed superior analytical sensibility when
urine at 8,000 x g for 8 minutes showed increase in RNA copies number
detection when compared to the urine about 3.2 and 2.3 times in system A and
B respectively (Table 1.).
Table 1. Comparison of absolute viral quantification of urine dilutions samples, urine centrifuged and urine ultracentrifuged in the A and B systems.
Sample System A (copies/µL) System B (copies/µL)
Urine 100 2.8x105 7.1x104
Urine 10-1 4.0x103 1,1x103
Urine 10-2 622.6 129.394
Urine 10-3 29.3 -
Urine 10-4 - -
Urine 10-5 - -
Urine Centrifuged 9.0x105 1.7x105
Urine Ultra Centrifuged 9.7x105 2.4x105
In this study, we tested three commercial kits of One-step RT-qPCR
Syber Green I based and described two systems that can be used to the CDV
diagnosis. The three commercial kits of One Step RT-qPCR Syber Green I
based were able to detect CDV RNA in urine samples. The efficiency of reverse
(Sergerie et al. 2007). In this study, the Qiagen® kit was faster, with better
melting curve showing better performance.
The One-Step RT-qPCR Syber Green I based of System A detect until
1.1 x 10 RNA copies/µL and was more sensible compared to TaqMan chemistry
using the same primer pair (Elia et al. 2006). The benefits of Syber Green I One
Step RT-qPCR assays over conventional RT-PCR and double-step qPCR
detection methods, particularly to diagnosis propose include their large dynamic
range in ability to be scaled up for high throughput applications and superior
analytical sensibility and specificity due to the curve dissociation (Dupouey et
al. 2014). It is also faster, practical, considering all the required reagents in RNA
transcription reaction, optimizes laboratory time presenting good cost benefit
ratio (Schmittgen et al. 2000). The One-Step RT-qPCR Syber Green I based
can be applied to detect CDV and can be used to target quantification in
academic labor application.
The NESTED reaction usually increases significantly the analytical
sensibility of conventional PCR reactions to CDV detection and must be applied
to diagnosis purpose. However, the qPCR is more sensitive and make it no
longer necessary, optimizing time, money and reducing the risk of amplicons
contamination. The composition of reagents, the gene target and the primers
pair plays a role in the reaction yield and may be selected according to propose.
In this study, the reaction held with primers pair CDV-for and CDV-rev (System
A), showed better efficiency and sensibility when compared to P-1 and P-2
primers pair (System B) reaction that showed the same analytical sensibility
methods that perform the NESTED-PCR there is a high risk of amplicons
laboratory contamination. Whereas qPCR is carried out only from the negative
samples, previously detected in melting curve analyses without opening tubes,
reduces the risk of cross contamination, being a useful option to be applied in
studies that require this tool.
The nucleoprotein gene is considered the most conserved gene of the
Morbillivirus family, is also the most expressed gene due to transcriptional
organization and has been applied to CDV diagnosis. The urine is currently the
best sample for CDV diagnosis ante-mortem and has been usefully applied.
The CDV causes cystitis and inclusion bodies can be found clearly in cytoplasm
or less frequently in nucleus of urinary bladder cells in histology. Most probably,
the RNA virus is detected in the urine due infected urinary epithelial cells flaking
and free virus in the urine. The One-Step RT-qPCR of viral RNA extracted from
samples centrifuged at 8,000 x g for 10 minutes revealed a higher quantity of
RNA virus (about tree fold), most probably due to infected cell sedimentation
containing the intracytoplasmic viral RNA. The ultracentrifugation trying
concentrate viral particles increased sensitivity slightly when compared to
centrifugation. Previously RNA extraction, urine centrifugation at 8,000 x g for 8
minutesis a practical easy to perform, increase diagnostic sensibility and it’s an
important tool particularly in samples with low viral load.
In conclusion, the three kits of one-step RT-qPCR were able to detect
CDV in urine samples and the Qiagen kit was faster, with better melting curve
showing better performance. The paper describes two One-Step RT-qPCR
analytical sensitivity. One-step RT-qPCR Syber Green I based is a sensible,
rapid, and support routine canine distemper laboratory surveillance, facilitates
research projects that require sensitive and quantitative CDV detection. In
addition, the previous centrifugation of urine sample increases the sensibility of
the test.
Acknowledgements
Funding was provided by the State of São Paulo Research Foundation
References
BASSO, C. R.; TOZATO, C.C.; RIBEIRO, M.C.; ARAUJO-JR, J.P.;PEDROSA, V. A. A immunosensor for the diagnosis of canine distemper virus infection using SPR and EIS. Anal. Methods.v.5, p. 5089-5095, 2013.
BOLT, G.; JENSEN, T.D.; GOTTSCHALCK, E.; ARCTANDER, P.; APPEL, M.J.G.; BUCKLAND, R.; BLIXENKRONE-MOLLE, M. Genetic diversity of the attachment (H) protein gene of current field isolates of canine distemper vírus. J. Gen. Virol. v.78, p. 367-372, 1997.
BUSTIN, S.A. Quantification of mRNA using real-time reverse
transcription PCR (RT-PCR): Trends and problems. J. Mol. Endocrinol. v. 29,
p.23-39, 2002.
COTTRELL, W.O.; KELL, M.K.; BROOKS, J.W.; MEAD, D.G.; PHILLIPS, J.E. First report of clinical disease associated with canine distemper vírus infection in a wild black bear (Ursus americana). J. Wildlife Dis. v.49, n.4, 2013.
DIALLO, A. Morbillivirus group: genome organization and proteins. Vet.
Microbiol. v.23, p.155-163, 1990.
DI FRANCESCO, C.E.; DI FRANCESCO, D.; DI MARTINO, B., SPERANZA, R.; SANTORI, D.; BOARI, A.;MARSILIO, F. Detection by hemi-nested reverse transcription polymerase chain reaction and genetic characterization of wild type strains of Canine distemper virus in suspected infected dogs. J. Vet. Diagn. Invest. v. 24, p.107–115, 2012.
DI SABATINO, D.; LORUSSO, A.; DI FRANCESCO, C.E.; GENTILE, L., DI PIRRO, V.; BELLACICCO, A.L.; GIOVANNI, A.; DI FRANCESCO, G.; MARRUCHELLA, G.; MARSILLIO, F.; SAVINI, G. Arctic lineage-canine distemper virus as a cause of death in Apennine wolves (Canis lupus) in Italy.
PLoS One, v.9, n.1, p.1-8, 2014.
rhinoviruses in respiratory samples: a comparison of Taqman probe-SYBR green I and BOXTO based real-time PCR assays. Virol.J., v. 11, n. 31, 2014.
ELIA, G.; DECARO, N.; MARTELLA, V.; CIRONE, F.; LUCENTE, M.S.; LORUSSO, E.; TRANI, L.; BUONAVOGLIA, C. Detection of canine distemper virus in dogs by real-time RT-PCR. J. Virol. Methods. v.36, n.1-2, p.171-6, 2006.
FISCHER, C.D.B.; IKUTA, N.; CANAL, C.W.; MAKJEJCZUK, A.; ALLGAYER, M. da C.; CARDOSO, C.H.; LEHMANN, F.K.; FONSECA, A.S.K.; LUNGE, V.R. Detection and differentiation of field and vaccine strains of canine distemper virus using reverse transcription followed by nested real time PCR (RT-nqPCR) and RFLP analysis. J. Virol. Methods. v.194, n.1-2, p.39-45, 2013.
FRISK, A.L., KÖNIG, M., MORITIZ, A., BAUMGÄRTNER, W. Detection of Canine Distemper Virus Nucleoprotein RNA by Reverse Transcription-PCR Using Serum, Whole Blood, and Cerebrospinal Fluid from Dogs with Distemper.
J. Clin. Microbiol., v.37, n.11, p. 3634-3643, 1999.
GEBARA, C.M.S.; WOSIACKI, S.R.; NEGRA, O.F.J.; OLIVEIRA, D.B.; BELONI, S.N.E.; ALFIERI, A.A.; ALFIERI, A.F. Detecção do gene da nucleoproteína do vírus da cinomose canina por RT-PCR em urina de cães
com sinais clínicos de cinomose. Arq. Bras. Med. Vet. Zootec. v. 56, p. 480–
487, 2004.
GREENE, C.E.; VANDEVELDE, M. Canine Distemper. In: GREEENE, C.E. Infectious Diseases of the dog and cat. Fourth Edition, Elsevier, St. Louis, Missouri, p.25-42, 2012.
KIM, Y.H.; CHO, K.W.; YOUN, H.Y.; YOO, H.S. Detection of canine
distemper virus (CDV) through one step RT-PCR combined with nested PCR. J.
Vet. Sci. v.2, p. 59-63, 2001.
MEGID J.; SOUZA V.A.F.; TEIXEIRA C.R., CORTEZ A.; AMORIM R.L.; HEINEMANN M.B.. Canine distemper virus in a crab-eating fox (Cerdocyon thous) in Brazil: Case report and phylogenetic analyses. J. Wildlife Dis. v. 45, p. 527-530, 2009
MEGID, J.; TEIXEIRA, C. R. ;CORTEZ, A.; HEINEMANN, M. B.; ANTUNES, J.M.A.P. ; FORNAZARI, F.; RASSY, F.B. ; RICHTZENHAIN, L.J . Canine distemper virus infection in a lesser grison (Galictis cuja): first report and virus phylogeny. Pesq. Vet. Bras., v. 33, p. 247-250, 2013.
ROSA, G.N.; DOMINGUES, G.H.; SANTOS, M.M.A.B.; FELIPPE, P.A.N.;SPILKI, F.R.; ARNS, C.W. Detecção molecular e análise ilogenética do gene H de amostras do vírus da cinomose canina em circulação no município de Campinas, São Paulo. Pesq. Vet. Bras. v.32, n.1, p. 72-77, 2012.
SAITO, T.B.; ALFIERI, A.A.; WOSIACKI, S.R., NEGRÃO, F.J.; MORAIS, H.S.A.; ALFIERI, A. A. Detection of canine distemper virus by reverse transcriptase-polymerase chain reaction in the urine of dogs with clinical signs of distemper encephalitis. Res. Vet. Sci. v.80, p.116-119, 2006.
SCAGLIARINI, A.; DAL POZZO, F.; GALLINA, L.; VACCARI, F; MORGANTI, L. TaqMan Based Real Time PCR for the Quantification of Canine Distemper Virus. Vet. Res. Comm., v.31, n.1, p. 261-263, 2007.
SCHMITTGEN, T.D.; ZAKRAJESEK, B.A.; MILLS, A.G.; GORN, V.; SINGER, M.J.; REED, M.W. Quantitative reverse transcription-polymerase chain reaction to study mRNA decay: comparison of endpoint and real-time methods. Anal. Biochem. v. 285, n. 2, p. 194-200, 2000.
SERGERIE, L.; PHELIPE, J.; DUQUETTE, M.; THIBAULT, C.; DELBECCHI, L.; BISSONNETTE, N. Detection limits of several commercial reverse transcriptase enzymes: impaction the low and high abundance
transcript levels assessed by quantitative RT-PCR. BMC Molecular Biology. v.
8, n.93, 2007.
WILKES, R.P.; SANCHEZ, E.; RILEY, M.C.; KENNEDY, M.A. Real-time
reverse transcription polymerase chain reaction method for detection of Canine distemper virus modified live vaccine shedding for differentiation from infection with wild-type strains. J. Vet. Diagn. Invest. v.26, n.1, p.27-34, 2014.
5. Artigo Científico 2
O artigo está de acordo com as normas da revista “Journal of Virological
Methods”.
Application of polystyrene microspheres immunoconjugate in canine distemper diagnosis by RT-qPCR
TOZATO, C.C; ARAÚJO JR, J.P*.
Laboratory of Animal and Human Virology, Univ Estadual Paulista, Botucatu,
Department of Microbiology and Immunology, Biosciences Institute,
–18618-970, São Paulo, Brazil.
*Corresponding Author: Laboratory of Animal and Human Virology, Univ
Estadual Paulista - UNESP, Department of Microbiology and Immunology,
Bioscience Institute, Distrito de Rubião Junior, s/n, Botucatu, São Paulo,
80035-050, Brazil. Tel.: +55 (14) 3880-0422, E-mail: jpessoa@ibb.unesp.br.
The authors of the manuscript disclose that there is no conflict of interest and
there was no commercial relationship with the manufacturers of any products
used in this manuscript.
Abstract
The study shows the use of polystyrene microspheres (PM)
immunoconjudated as an efficient technology to viral RNA preparation and
application in RT-qPCR. The CDV was isolated and purified to antigen
production. Three PM conjugates were developted and standardized using
polyclonal antibodies anti-CDV: IgY, IgG and Protein A+IgG. The analytical
sensitivity was compared through the PMs conjugates, PMs without antibodies
adsorption and the conventional RNA extraction method. The diagnostic
sensitivity was performed using polystyrene microsphere conjugate with IgY
(chicken polyclonal hyperimmune anti-CDV) due to analytical sensitivity results,
and the advantages of production. The application of PM immunoconjugates in
RT-qPCR demonstrated to be a methodology with high analytical and
diagnostic sensitivity. The diagnostic sensitivity of PM-IgY was superior than the
conventional method. The use of conjugate PM-antibody-coated-virus linked
applied in RT-qPCR is an innovative low-cost technology.
1. Introduction
Canine distemper virus (CDV) is an enveloped, single stranded,
non-segmented and negative polarity RNA virus, belonging to the genus
Morbillivirus and Paramyxoviridae family (ICTV, 2013). The canine distemper
(CD) is an important veterinary disease due to lethality, severity of clinical signs
and increasing wide range of susceptible animal species affected (Greene and
Valdevelde, 2012).
The diagnosis of canine distemper (CD) is often based on clinical
suspicion; however, due to the nonspecific symptoms of disease the laboratory
diagnosis is necessary (Elia et al., 2006). The polymerase chain reaction (PCR)
with reverse transcriptase (RT-PCR) has been used as a diagnostic tool and
recently, quantitative PCR (qPCR) has been successfully employed for CDV
detection due to high analytical sensibility and specificity (Di-Francesco et al.,
2012; Frisk et al., 1999; Kim et al.; 2001; Wilkes et al., 2014). Urine has been
showed to be the best sample to CDV molecular diagnosis due to the ability to
detected CDV on different clinical forms of infection andnoninvasive collecting
method (Elia et al., 2006; Gebara et al., 2004; Saito et al., 2006; Shin et al.,
1995).
The viral nucleic acids extraction from biological samples is an important
phase to successful use in PCR and represents much of the reaction cost
(Katayama et al., 2002). The analyte amount, target concentration, sample
contamination and PCR inhibitors eluted are some limitations of conventional
concentrate or extract viral nucleic acid in order to increase PCR sensitivity
(Kittigul et al., 2001; Olsvik, et al., 1994; Yao et al., 2009.).
The polystyrene microspheres (PM), known as latex beads, were
developed in 1947 and have been used to diagnostic agglutination methods.
The specific antibody/antigen is coat to PM held in colloidal solution that
agglutinates in the presence of the antigen/antibody forming visible clumps
(Molina-Bolívar and Galisteo-González, 2005; Ortega-Vinuesa and
Bastos-Gonzalez, 2001; Singer and Plotz, 1956). The variation of immunoglobulin
adsorption in the PM "solid-liquid interface" in experimental conditions is
influenced by pH, ionic strength, and temperature that results in a complex
interactions. Polyclonal antibodies are widely used to polystyrene adsorption
and this interaction is well known. Polyclonal antibodies from eggs yolks (IgY)
can be used in polystyrene adsorption and represent source of large quantities
of desired antibody (Ortega-Vinuesa and Bastos-Gonzalez, 2001).
The positional conformation of immunoglobulin adsorption in PM is
another investigated aspect. Protein A bind specifically to the Fc portion of
some mammals IgG and is used to coat polystyrene plates in ELISA (
Enzyme-Linked Immunosorbent Assay) to increase analytical sensitivity (NGAI et al.,
1993). Due to stability and chemical-physical properties, the PM coated with
specific antibodies presents a new groundbreaking application to capture
virus-target in the sample, lower by centrifugation to direct apply in DNA/RNA
molecular reaction.
The aim of the study was to develop a new method for RNA preparation
Protein A+IgG to capture CDV in urine samples and compare to the
conventional RNA extraction method in molecular diagnosis of canine
distemper.
2. Materials and Methods 2.1. CDV production 2.1.1. Cell line
VERO dog Slam Tag (VDST) cells were cultured using 25 cm2 cell culture
flasks using 10 mL of DMEM high glucose (D7777, Sigma Aldrich, Saint Louis,
USA) added 0.04 M sodium bicarbonate, 0.68 mM L-Glutamine, 1 ml/L MEM
Non-Essential Amino Acid Solution ( M7145, Sigma Aldrich, Saint Louis, MO,
USA), 1 ml/L MEM Vitamin Solution (M6895, Sigma Aldrich, Saint Louis, MO,
USA), 1 ml/L Sodium Pyruvate (S8636, Sigma Aldrich, Saint Louis, MO, USA),
ZeocinTM 1mg/mL (R25001, Life Technologies Corporation, Carlsbad, CA) and
10% fetal calf serum (FCS) (Gibco®, Grand Island, USA) maintained at 37°C.
Subcultures were propagated using 0.2% trypsin in cell dissociation every three
days.
2.1.2. CDV isolation and propagation
One milliliter of dog urine sample, previously positive in RT-qPCR, was
diluted 1:100 in culture medium was filtered through a 0.45 µM pore membrane
directly over the 25 cm2 flasks containing VDST cells at 90% confluence. A
under stirring at 37 °C over 30 min. Then, 5 ml of medium was added and kept
at 37°C. The negative control was carried out with dog RT-qPCR negative urine
inoculum to citopathics effects comparison. To virus propagation, two rollers of
390 cm2 (Corning®) were prepared in the same medium and temperature
conditions of cell line, kept in slow rotation at 37°C. When the cells were
confluent in rollers, the medium VP SFM serum-free medium (Gibco®, Grand
Island, USA) supplemented with 4 mM Glutamax (Gibco ®, Grand Island, USA),
pH 7.4 was used to VDST cultivation to avoid protein contamination derived
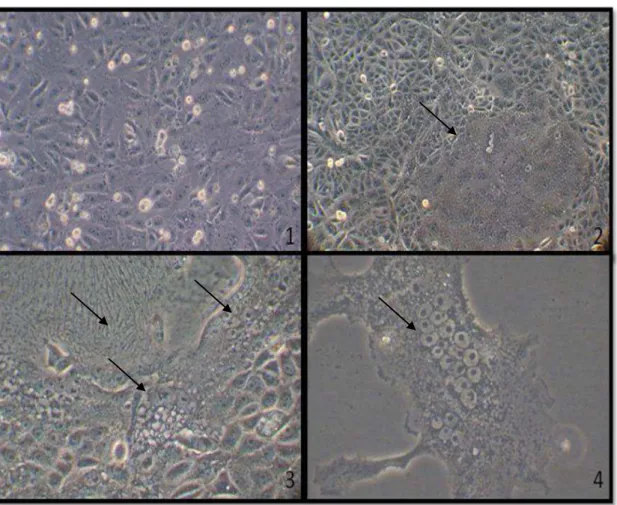
from FCS. The VDST were observed for seven days and no cell morphological
alteration was observed. Then, 1.5 mL of CDV infected cell was filtered in 45
µm membrane directly over the cell and kept in slow rotation at 37°C until total
cells destruction.
2.1.3. CDV purification
A suspension of 80 mL of infected cells was clarified by 1,000 x g for 10 min
at 4°C to CDV purification. The supernatant was ultracentrifuged over a cushion
of 40% sucrose at 50,000 x g for 12 hours at 4°C in SorvallTM 80 Plus
Centrifuge, to virus concentration. The pellet was resuspended using 300 µL of
PBS (0.01 mol/L PO42-, 0.146 mol/L NaCl), pH 7.4 and stored at 4°C after use.
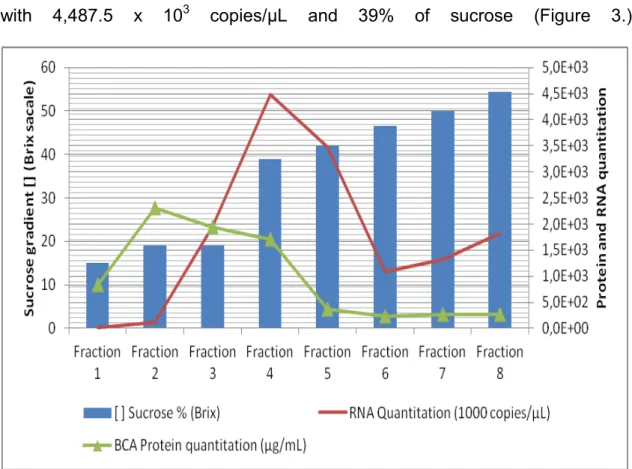
A continuous sucrose gradient of 26 to 60% (w/w) was prepared and 300 µL
of resuspended sample was added under the sucrose gradient and
ultracentrifuged in SorvallTM 80 Plus Centrifugue for 5 h at 54,000 g in angle
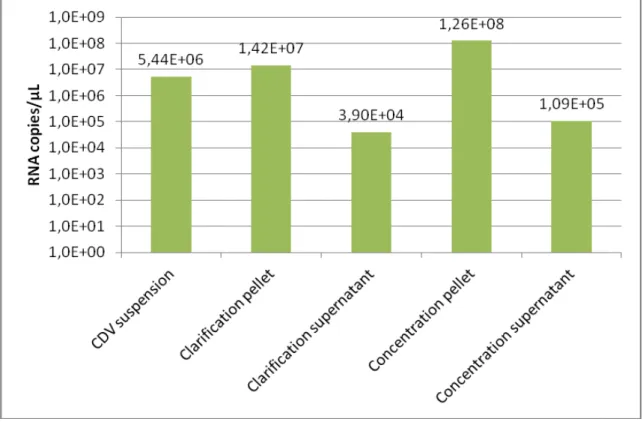
2.1.4. Virus purification fractions analysis
Purification fractions were carefully collected each 500 µL and were
measured by the refractometry index (Brix scale), protein quantification by BCA
method (Smith et al., 1985). The CDV RNA quantification by RT-qPCR was
performed during the virus purification to quantity the amount of viral RNA in the
fractions.
2.1.5. Dot Blot
To access the virus purification fractions specificity, a dot blotting was
performed with Immun-Blot Assay Kit (BioradTM) using 1 µL of collected purified fractions of CDV sucrose in 0.1 nm nitrocellulose membrane, according to the
manufacturer's recommendations with slight modifications. Briefly, the fractions
were tested with rabbit, hyperimmune anti-CDV purified IgG, previously
produced in 1/500 dilution. The immunoassay conjugate bovine IgG anti-rabbit
IgG peroxidase (Santa Cruz BiotechnologyTM, Santa Cruz, USA) was used as
secondary antibody in 1/250 dilution. 2.1.6. Virus dialyze
The virus purification fraction that showed best purification analysis, was
dialyzed using Slide-A-Lyzer Dialysis Cassette G2 10K (Thermo Scientific) in
300 mL PBS (0.01 mol/L PO42-, 0.146 mol/L NaCl), according to the
2.2. Hen inoculation and IgY purification
A hen (Gallus gallus) (Linnaeus, 1758) was immunized intramuscularly with
600 µL of purified virus, previously emulsified with complete Freund's adjuvant
(v/v) at dose of 300 µg/mL in four sites of the pectoral muscle. A second
inoculation was performed after 15 days with the same dose with virus
previously emulsified in incomplete Freund's adjuvant (v/v). Eggs were collected
from 10 days after the second immunization (Johnstone and Thorpe, 1996). The
egg yolk was separated and IgY purification was performed using HiTrap™ IgY
Purification HP Column system (GE Healthcare, Chalfont St. Giles, United
Kingdom), according to the manufacturer's instructions.
2.3. RT-qPCR
The RT-qPCR was performed using the Syber Green RT-PCR Quanti
fast kit (204156, Qiagen, Crawley, UK), with the pair of primers CDV-for
(5’-AGC TAG TTT CAT CTT AAC TAT CAA ATT-3’) and CDV-Rev (5’-TTA ACT
CTC CAG AAA ACT CAT GC-3’) (Elia et al. 2006), using 2 µL of conventional
method extracted RNA (kit) or the PM conjugated urine to a final volume of 20
µL in the 7300 Real Time PCR System machine (Applied Biosystems, Carlsbad,
CA, USA). The data was analyzed in SDS 7300 software as absolute
2.4. Conventional RNA extraction
Total RNA of urine samples and purification fractions were obtained from 100
µL of each sample using Total RNA purification kit (17200; Norgen Biotek
Corporation, Canada), according to the manufacturer's recommendations.
2.5. Polystyrene Microspheres Standardization 2.5.1. PM wash prepare
The PM suspension of 800 µL (LB-8, Sigma Aldrich, Saint Louis, MO, USA) was
diluted in 10 mL of PBS (0.01 mol/L PO42-, 0.146 mol/L NaCl), pH 7.4 (800 µL of
latex in 10 ml PBS). The particles were sonicated with model CV26 sonicator 3
times (30 seconds with an interval of 1 minute, amplitude of 40%) and
centrifuged 3,000 x g for 30 min. The supernatant was removed and the
particles were resuspended in 4 ml PBS (0.01 mol/L PO42-, 0.146 mol/L NaCl),
pH 6.4 to a final concentration of 2% of PM.
2.5.2. Standardization of IgY, IgG and IgG+Protein A conjugate
Three conjugates were produced using IgY, IgG or IgG+Protein A (PM-IgY,
PM-IgG and PM-Protein A+IgG conjugate, respectively). The polyclonal rabbit
IgG anti-CDV was produced previously. The polyclonal anti-CDV IgY and IgG
concentrations were tested to 12.0, 25.0, 50.0, 86.0 and 172.0 µg/mL in 2% of
PM using MES (2-(N-morpholino ethanesulfonic acid) 0.025 M pH 6.0. The
µg/mL concentration and then coupled to IgG concentrations in the same
conditions. The PM was coated with IgY, IgG and Protein A by slow stirring
overnight at room temperature. The conjugates were centrifuged at 3,000 x g
for 30 min and supernatant protein quantification was measured by BCA
method (Smith et al., 1985) to verify the best adsorption parameters.
After determining the best immunoglobulin and protein A adsorption
concentrations, the conjugates coated were centrifuged 3,000 x g for 30 min,
washed with MES (2-(N-morpholino ethanesulfonic acid) 0.025 M pH 6.0 twice
and resuspended in storage buffer (0.1M sodium phosphate, 0.01% glycine and
0.1% NaN3) pH 6.0.
2.5.3. PM conjugates for RNA preparation
Infected cell culture diluted 1:100 in PBS (0.01 mol/L PO42-, 0.146 mol/L
NaCl), pH 7.4 was used as positive control in conjugates standardization.
Twenty microliters of PM-IgY, PM-IgG and PM-Protein A+IgG conjugate were
incubated with 100µL of positive control adsorption in polypropylene strip tube
to qPCR (Corning Axygen® PCR-0208-FCP-C). All steps were performed direct
in qPCR tube. The virus-conjugate adsorption time and temperature were
tested using a chessboard of 15 minutes, 30 minutes, 2 hours and 24 hours
versus 8°C, 37°C, and room temperature.
The particles were centrifuged 3,000 x g for 30 minutes and the supernatant
removed. The particles were washed with 100 µL of PBS, centrifuged and the