RENORBIO
Programa de Pós-graduação em Biotecnologia
Transcriptoma diferencial entre células-tronco
mesenquimais humanas jovens e senescentes
Joana Cristina Medeiros Tavares Natal – RN
ii
Joana Cristina Medeiros Tavares
Transcriptoma diferencial entre células-tronco
mesenquimais humanas jovens e senescentes
Tese de Doutorado apresentada ao Programa de Pós-Graduação em Biotecnologia – RENORBIO, como parte dos requisitos necessários para a obtenção do título de Doutora em Biotecnologia.
Área de concentração: Biotecnologia em Saúde.
Orientadora: Profa. Dra. Silvia Regina Batistuzzo de Medeiros.
iii AGRADECIMENTOS
Aos meus pais por todo amor e ensinamentos dedicados a mim que me fizeram ser quem sou.
Ao meu irmão, André Tavares, por ter feito as escolhas de sua vida sempre pensando em como contribuir para meus estudos.
A toda minha família, por fazer parte do alicerce para a edificação deste sonho, especialmente, à minha prima, Ana Carolina, por sua confiança e apoio na realização dos meus objetivos.
À professora Dra. Silvia Regina Batistuzzo de Medeiros, pela oportunidade dada à minha formação desde a iniciação científica, por ter permitido que eu continuasse desenvolvendo o doutorado após a minha aprovação em concurso para docente na UFRN/FACISA e por confiar na minha capacidade de desenvolver este trabalho.
À UFRN pela estrutura física e pela oportunidade de ter estudado gratuitamente com ensino de alta qualidade durante toda à minha vida acadêmica.
Ao Ministério da Ciência e Tecnologia – MCT, por intermédio do Conselho Nacional de Desenvolvimento Científico e Tecnológico - CNPq e do Ministério da Saúde - MS, por intermédio do Departamento de Ciência e Tecnologia da Secretaria de Ciência, Tecnologia e Insumos Estratégicos - DECIT/SCTIE - Edital CT-Saúde/MS/SCTIE/DECIT/MCT/CNPq Nº 17/2008, pelo apoio financeiro.
iv A Isabella que foi uma grande companheira na realização de todas as etapas experimentais deste trabalho, por sua companhia agradável e inteligente, e por sua amizade verdadeira.
À professora Dra. Vivian Silbiger e ao professor Dr. André Luchessi , pela ajuda nas análises dos dados de microarranjos e de RT-PCR em tempo real, e por suas agradáveis companhias.
À pesquisadora Maria Eugênia do Laboratório de Microarranjos (LMA) – LNBio - Laboratório Nacional de Luz Síncroton –Campinas/SP, por todo seu auxílio técnico nas etapas de hibridização e aquisição dos dados de microarranjos.
Ao Laboratório Nacional de Luz Síncroton – Campinas/SP pela acomodação do alojamento e toda a estrutura física disponível.
À minha grande amiga Adriana Brito, por todo seu apoio para que eu pudesse conciliar o meu trabalho na FACISA com o meu doutorado, compartilhando disciplinas, projetos, reuniões, orientações, e até casa e refeições em Santa Cruz-SC. Obrigada também pelos momentos de descontração na praça Tequinha Farias-SC, por sua ajuda nesta etapa final de organização de tese e, principalmente, por sua sincera amizade.
A Pedro Henrique, pelo seu carinho, companherismo, incentivo e pelos momentos de alegria vividos. Obrigada também à sua família, pela sua receptividade sempre aconchegante e carinhosa, me proporcionando muita paz e descanso.
Aos meus amigos constituintes de minha turma de biologia, especialmente, Aurizângela, Daiane, Denise, Jefferson e Jule, que me proporcionaram muitos momentos de alegria, compartilharam sonhos pessoais e profissionais, e por continuarem dedicando seu carinho e amizade que contribuem para a realização deste trabalho.
v A todos os companheiros e divertidos amigos do LAMA, Jana Dara, Felipe, Nilmara, pelas músicas, danças, risadas e, principalmente, pela amizade e companheirismo verdadeiro.
Ao meu amigo e colega de doutorado, Leonam, pelo seu contagiante otimismo nos experimentos e em sua vida, pelos seus entusiasmantes planos do tipo “cebolinha”, e por sua grande e inestimável amizade.
A toda família Pichorim pelo conforto de sua receptividade, atenção e apoio.
A Mayara e Giselle por todo seu apoio técnico e por sua convivência afável.
Aos alunos de iniciação científica, Guillermo Ortiz Brasil, Isabella Tannus Meira, Luiza Xavier e Thais Pimentel, que contribuíram para realização dos experimentos deste trabalho.
A Guillermo Ortiz, por sua amizade e pelo seu exemplo de perseverança, dedicação e paciência.
A toda família Santos, por todo seu apoio, carinho e incentivo ao longo de toda minha vida acadêmica.
Ao professor Dr. Geraldo Cavalcanti Júnior pela ajuda na realização dos experimentos de citometria de fluxo.
vi LISTA DE FIGURAS
FIGURA 1 Imagens das CTMH jovens e senescentes... 48 FIGURA 2 Imagens das diferenciações osteogênica, adipogência e
condrogênica das CTMH... 49 FIGURA 3 Gráfico mostrando que a comparação entre as CTMH/inv
senescentes e CTMH/inv jovens resultou em maior número de genes diferencialmente expresso do que na comparação entre as CTMH/n senescentes e CTMH/n jovens... 50 FIGURA 4 Classificação funcional dos genes diferencialmente
expressos em CTMH/n senescentes comparadas às CTMH/n jovens... 52 FIGURA 5 Rede de interações formada pelos genes diferencialmente
expressos em CTMH/n senescentes comparadas às CTMH/n jovens... 53 FIGURA 6 Rede de interações formada pelos genes diferencialmente
expressos em CTMH/n senescentes comparadas às CTMH/n jovens no contexto genômico... 54 FIGURA 7 Classificação funcional dos genes diferencialmente
expressos em CTMH/inv senescentes comparadas às CTMH/inv jovens... 55 FIGURA 8 Rede de interações formada pelos genes diferencialmente
expressos em CTMH/inv senescentes comparadas às CTMH/inv jovens... 57 FIGURA 9 Rede de interações formada pelos genes diferencialmente
expressos em CTMH/inv senescentes comparadas às CTMH/inv jovens no contexto genômico... 58 FIGURA 10 Sub-rede da figura 9a, destacando as moléculas que
interage diretamente como gene EGF... 59 FIGURA 11 Diagrama de Venn construído a partir das listas dos genes
vii e CTMH/inv senescentes vs CTMH/inv jovens...
FIGURA 12 Classificação funcional dos 30 genes comuns às duas listas de comparações CTMH/n senescentes vs CTMH/n jovens e CTMH/inv senescentes vs CTMH/inv jovens... 61 FIGURA 13 Diagrama de Venn para identificação dos genes
diferencialmente expressos em ambas as comparações, CTMH/inv jovens vs CTMH/n jovens e CTMH/inv senescentes vs CTMH/n senescentes... 62 FIGURA 14 Cluster hierárquico de todas as probsets diferencialmente
expressas em ambas as comparações CTMH/inv jovens vs CTMH/n jovens e entre as CTMH/inv senescentes vs CTMH/n senescentes... 64 FIGURA 15 Genes mais expressos em CTMH/inv jovens se tornaram
ainda mais expressos em CTMH/inv senescentes... 65 FIGURA 16 Genes menos expressos em CTMH/inv jovens se tornaram
ainda menos expressos em CTMH/inv senescentes... 66 FIGURA 17 Cariótipo parcial das CTMH/inv mostrando a
inv(3)(p13p25~26) e seu
ideograma... 68 FIGURA 18 Representação dos genes diferencialmente expressos em
CTMH/inv jovens comparadas as CTMH/n Jovens nas categorias funcionais de acordo com o IPA Igenuity... 70 FIGURA 19 Redes de interações formadas pela lista dos genes
diferencialmente expressos na comparação CTMH/inv jovens vs CTMH/n jovens... 71 FIGURA 20 Redes de interações formadas pela lista dos genes
diferencialmente expressos na comparação CTMH/inv jovens vs CTMH/n jovens no contexto genômico... 72 FIGURA 21 Representação dos genes diferencialmente expressos em
CTMH/inv jovens comparadas às CTMH/n jovens nas categorias funcionais de acordo com o IPA Igenuity... 74 FIGURA 22 Redes de interações formadas pela lista dos genes
viii CTMH/inv senescentes vs CTMH/n senescentes...
FIGURA 23 Sub-rede de interações da figura 22. Esta rede evidencia os nós que se conectam ao bottleneck EGF... 77 FIGURA 24 Sub-rede de interações da figura 12. Esta rede evidencia
somente os genes que se ligam diretamente a EGF ou aos genes localizados em Inv (3): CNTN3, PDZRN3, LMCD1 e BHLHE40... 78 FIGURA 25 Rede de interações formada pela lista dos genes
diferencialmente expressos apenas na comparação CTMH/inv senescentes vs CTMH/n senescentes inserida no contexto genômico... 79 FIGURA 26 Sub-rede da figura 25, destacando os nós conectados
diretamente ao gene EGF e os gene localizados na região da Inv (3)... 80 FIGURA 27 Gráfico mostrando as categorias funcionais mais
enriquecidas (com valor de p 0,05 e somente aquelas com representação superior à 5%) em ambas as comparações CTMH/inv jovens vs CTMH/n jovens e CTMH/inv senescentes vs CTMH/n senescentes... 82 FIGURA 28 Rede de interações formada pela união entre os genes
ix SUMÁRIO
Lista de Figuras... vi
Resumo... xii
Abstract... Xv 1.0. Introdução... 17
2.0. Revisão Bibliográfica 2.1. Células-tronco mesenquimais humanas... 20
2.2. Cordão umbilical como fonte de CTMH... 23
2.3. Senescência celular... 26
2.4. Estabilidade genômica das CTMH ao longo tempo de cultivo... 29
2.5. Microarranjos e estudos de exprssão gênica em CTMH... 32
3.0. Objetivos... 36
4.0. Material e Métodos... 37
4.1. Isolamento e caracterização das CTMH... 37
4.2. Condições de cultura das CTMH para análise do perfil de expressão gênica... 38
4.3. Caracterização das CTMH senescentes... 39
4.4. Extração de RNA... 39
4.5. Preparação do RNA, hibridização e captura de imagem dos microarranjos... 41
4.6. Análise dos dados de microarranjos... 41
4.7. Avaliação da expressão diferencial por RT-PCR em tempo real... 42
4.8. Classificação funcional dos genes diferencialmente expressos... 44
4.9. Análise de biologia de sistemas... 44
5.0. Resultados 5.1. Caracterização das CTMH jovens e senescentes... 46
5.2. A senescência in vitro afeta o perfil de expressão das CTMH... 50
5.3. Classificação funcional e rede de interações dos genes diferencialmente expressos em CTMH/n senescentes vs CTMH/n jovens... 51
5.4. Classificação funcional dos genes diferencialmente expressos CTMH/inv senescentes vs CTMH/inv jovens... 55
x
5.6. As CTMH/inv tem o transcriptoma distinto das CTMH/n... 62
5.7. Alguns genes localizados na região próxima aos pontos de quebra da inversão em CTMH/inv foram diferencialmente expressos em CTMH/inv... 66
5.8. Classificação funcional e redes de interações dos genes diferencialmente expressos em CTMH/inv jovens comparadas às CTMH/n jovens... 68
5.9. Classificação funcional e redes de interações dos genes diferencialmente expressos em CTMH/inv senescentes comparadas às CTMH/n senescentes... 73
5.10. Análise de interpretação biológica dos genes diferencialmente expressos nas comparações CTMH/inv jovens vs CTMH/n jovens e CTMH/inv senescentes vs CTMH/n senescentes... 80
5.11. Análise da expressão gênica das CTMH por RT-PRC em tempo real... 85
6.0. Discussão... 86
7.0. Conclusão... 116
8.0. Referências Bibliográficas... 118
9.0. Apêndices... 147
A. Tabela da lista de genes diferencialmente expressos em CTMH/n senescentes na comparação CTMH/n senescentes vs CTMH/n Jovens... 147
B. Tabela da lista de genes diferencialmente expressos em CTMH/inv senescentes na comparação CTMH/inv senescentes vs CTMH/inv Jovens... 151
C. Tabela da classificação funcional dos genes diferencialmente expressos em CTMH/n senescentes comparadas às CTMH/n jovens... 162
D. Tabela de Anotação funcional das cinco categorias mais enriquecidas com menor valor de p dos genes diferencialmente expressos em CTMH/n senescentes comparadas às CTMH/n Jovens... 163
E. Tabela da classificação funcional dos genes constituintes da rede de interações formada pela lista dos genes diferencialmente expressos em CTMH/n senescentes comparadas às CTMH/n jovens... 167
F. Tabela da classificação funcional dos genes constituintes da rede de interações no contexto genômico formada pela lista dos genes diferencialmente expressos em CTMH/n senescentes comparadas às mesmas jovens com a adição de 50 genes do genoma humano. 169 G. Tabela da classificação funcional dos genes diferencialmente expressos em CTMH/inv senescentes comparadas às mesmas jovens... 174 H. Tabela da anotação funcional das cinco categorias mais
xi expressos em CTMH/inv senescentes comparadas às CTMH/inv jovens... I. Tabela da classificação funcional dos genes constituintes da rede de interações formada pela lista dos genes diferencialmente expressos em CTMH/inv senescentes comparadas às CTMH/inv jovens... 195 J. Tabela da classificação funcional dos genes constituintes da rede
de interações no contexto formada pela lista dos genes diferencialmente expressos em CTMH/inv senescentes comparadas às CTMH/inv jovens com a adição de 50 genes do genoma humano... 200 K. Tabela da lista de genes da intersecção entre as duas listas das
comparações senescentes vs jovens de CTMH/n e CTMH/inv... 207 L. Tabela da classificação funcional da lista dos 30 genes comuns em
ambas as listas das comparações Senescentes vs jovens de CTMH/n e CTMH/inv... 209 M. Tabela da anotação funcional das cinco categorias mais
enriquecidas com menor p-valor dos 30 genes comuns em ambas as listas das comparações Senescentes vs jovens de CTMH/n e CTMH/inv... 210 N. Tabela de genes diferencialmente expressos em CTMH/inv jovens
na comparação entre as CTMH/inv vs CTMH/n jovens, ranqueados do maior para o menor Fold Change... 214 O. Tabela da lista de genes diferencialmente expressos entre
CTMH/inv senescentes vs CTMH/n senescentes... 218 P. Tabela da lista dos genes diferencialmente expressos da
intersecção entre as comparações CTMH/inv jovens vs CTMH/n jovens e entre as CTMH/inv senescentes vs CTMH/n senescentes. 235 Q. Tabela dos genes localizados próximo à região da inversão que
tiveram sua expressão afetada em CTMH/inv... 237 R. Tabela das categorias funcionais representadas pelos genes
diferencialmente expressos em CTMH/inv jovens da comparação CTMH/inv jovens vs CTMH/n jovens... 238 S. Tabela da anotação funcional das cinco categorias com menores
valor de p representadas pelos genes diferencialmente expressos da comparação CTMH/inv jovens vs CTMH/n jovens... 239 T. Tabela da classificação funcional dos genes constituintes da rede
de interações obtida a partir da comparação CTMH/inv jovens vs CTMH/n jovens por meio do pluggin BINGO do Cytosacape... 249 U. Tabela da classificação funcional dos genes constituintes da rede
de interações obtida a partir da comparação CTMH/inv jovens vs CTMH/n jovens no contexto genômico por meio do pluggin BINGO do Cytosacape... 251 V. Tabela das categorias funcionais representadas pelos genes
diferencialmente expressos em CTMH/inv Senescentes da comparação CTMH/inv vs CTMH/n Senescentes... 253 W. Tabela dos genes classificados nas cinco categorias funcionais
xii de interações no contexto formada pela lista dos genes diferencialmente expressos em CTMH/inv senescentes comparadas às CTMH jovens com a adição de 50 genes do genoma humano... Y. Tabela da classificação funcional dos genes constituintes da rede
de interações no contexto formada pela lista dos genes diferencialmente expressos em CTMH/inv senescentes comparadas às CTMH jovens com a adição de 50 genes do genoma humano... 278 Z. Tabela da comparação dos valores de Fold Change obtidos por
análise de Microarray e de qPCR... 284 10.0. Anexos... 286
xiii RESUMO
xv ABSTRACT
17 1.0. INTRODUÇÃO
As células-tronco mesenquimais humanas (CTMH) são células multipotentes, caracterizadas pela sua capacidade de aderência ao plástico, diferenciação em osteoblastos, adipócitos e condrócitos, apresentam expressão dos marcadores CD105, CD90, CD73 e ausência da expressão de CD45, CD34, CD14, CD11b, CD79α ou CD19 e HLA-DR (Reagan e Kaplan, 2011). As CTMH têm sido consideradas como uma importante alternativa para a terapia celular devido à sua facilidade de obtenção e expansão in vitro, à sua plasticidade funcional, e mais recentemente, por secretar moléculas bioativas com papel na imunomodulação, quimioatração, com efeitos tróficos, neuroproteção, entre outras que auxiliam o reparo tecidual (Eyal Ben-Ami et al., 2011; Honmou et al., 2012; Youwei Wang et al., 2012).
Há evidências de que as células-tronco sofrem senescência celular in vivo que pode ser responsável pelo declínio de suas funções e pela origem das doenças metabólicas, degenerativas, câncer e o envelhecimento dos indivíduos (Revisado por Rodríguez-Rodero, et al. 2011). Esta proposição está em conformidade com a observação de que o envelhecimento afeta a renovação e a capacidade de diferenciação de CTMH residentes no tecido adiposo e na MO de indivíduos mais velhos (Alt et al., 2012; Kretlow et al., 2008; Chen et al., 2009).
18 próprio processo de senescência, ou por outro lado propiciar o desenvolvimento de câncer e de outras doenças relacionadas à idade como aterosclerose e diabetes (Cichowski e Hahn, 2008; revisado por Sikora et al., 2012).
Várias alterações cromossômicas adquiridas durante o cultivo já foram descritas em diferentes tipos de células-tronco, embrionárias (Baker et al., 2007; Mayshar et al., 2010), pluripotentes induzidas, CTMH e neurais (Ben David et al., 2011) que parecem conferir vantagens seletivas e podem aumentar a tumorigenicidade das células, e alterar a sua capacidade de diferenciação (Harrison et al., 2007; Mayshar et al., 2010; Ben-David e Benvenisty, 2011). Por outro lado, há relatos que algumas alterações desaparecem na cultura e não conferem vantagem seletiva (Sensebé et al., 2012; Hussein et al., 2011). A presença de aneuploidia em várias preparações de CTMH da MO depois de cultivo in vitro foi documentada, mas foi associada somente à senescência e não à tumorigenese (Tarte et al., 2010).
Recentemente Duarte e colaboradores (2012) identificaram uma alteração cromossômica constitucional, cariótipo 46,XY,inv(3)(p13p25~26), em CTMH obtidas do endotélio da veia do cordão umbilical de um doador, que são as MSC/inv utilizadas no presente trabalho. As inversões paracêntricas são consideradas como heteromorfismos que não causam problemas à saúde do portador. No entanto, pessoas que a possuem têm maior probabilidade de originar rearranjos não balanceados na sua descendência, e apresentam uma frequência mais elevada de aborto espontâneo em relação à população em geral (Grati et al., 2008). Contudo, nada se sabe sobre o efeito desta alteração cromossômica nas células após sua expansão in vitro, e nem tão pouco sobre o seu comportamento no organismo de um possível receptor. Considerando que a região da inversão (3p25~26) contém vários genes de grande importância biológica, como genes envolvidos com o reparo de DNA e outros responsáveis pelo desenvolvimento de tumores, tais células podem ser mais propensas à transformação espontânea in vitro.
20 2.0. REVISÃO BIBLIOGRÁFICA
2.1. Células-tronco mesenquimais humanas
O termo células-tronco mesenquimais humanas (CTMH) foi introduzido por Caplan, em 1991. As primeiras células-tronco mesenquimais foram isoladas da medula óssea (MO) de roedores e coelhos por Friedenstein e colaboradores (1976), e identificadas como unidades formadoras de colônias semelhantes a fibroblastos, sendo consideradas como células precursoras de fibroblastos. Posteriormente, foi observado que elas tinham a capacidade de se diferenciar em células com características de osteoblastos, condrócitos e adipócitos. Em concomitância a esta descoberta, foi identificada uma população de células aderentes necessárias para o estabelecimento das células estaminais hematopoiéticas não aderentes, em um estudo sobre a hematopoese in vitro desenvolvido por Dexter e colaboradores (1977). A partir deste momento, em referência à sua localização anatômica, um novo termo foi estabelecido, células estromais da MO. Em analogia ao modelo de hematopoese, o termo CTMH foi introduzido para designar uma população de células específicas do estroma da MO - células aderentes que originam os diferentes tipos de tecidos mesenquimais, ósseo, cartilagem e músculos (Revisado por Meirelles e Nardi , 2009). Desde então, células com características semelhantes às CTMH foram isoladas de diferentes tecidos adultos, tais como, tecido adiposo, pele, osso, músculo, cérebro, fígado, rins, pâncreas, placenta, glândulas salivares, polpa dentária, ligamento periodontal, folículo capilar, linfonodos, baço, timo e até menorreia (Revisado por Momin et al., 2010; Ding et al., 2011). Elas também podem ser obtidas a partir de tecidos perinatais, como do cordão umbilical, fluido e membrana amniótica, e córion (Bieback e Brinkmann, 2010; Ding et al., 2011; Hass et al., 2011; Roubelakis et al., 2012).
21 identificação de CTMH: capacidade de adesão ao plástico de cultura; fenótipo positivo para os marcadores de superfície CD105, CD73 e CD90, e negativo para CD45, CD34, CD14 ou CD11b, CD79a ou CD19e HLA-DR; e se diferenciar in vitro em osteoblastos, adipócitos e condrócitos (Dominici et al., 2006).
Em 2008, Crisan e colaboradores documentaram uma subpopulação de células perivasculares humanas que expressam marcadores in situ para ambos os tipos celulares, CTMH e pericitos. Este e outros estudos, que compararam o fenótipo das CTMH obtidas de diferentes fontes com os pericitos, sugerem fortemente que as CTMH sejam pericitos (Revisado por Caplan, 2008), e que sua natureza pleiotrópica seja responsável pelo seu papel na renovação dos tecidos mesenquimais in vivo (Revisado por Singer e Caplan, 2011). Contudo, não há nenhuma confirmação definitiva de que a origem de CTMH do tecido adiposo (ASC) seja de pericitos. Tornando o quadro mais complexo, dados recentes indicam que as CTMH em vários tecidos humanos podem originar-se não só de pericitos (CD146 + CD34-CD31-CD45-), mas também a partir de um subconjunto fenotipicamente distinto de células adventícias (CD34+CD146 -CD31-CD45-) que residem na camada mais externa da parede de vasos maiores. Foi identificado que elas nativamente expressam marcadores de superfície de CTMH (tal como CD90) e comportam-se de maneira semelhante às CTMH após longo tempo de cultura in vitro (Revisado por Strioga et al., 2012).
22 T, B e NK (Natural killer), e da alteração da secreção de citocinas inflamatórias, de imunoglobulinas; secreção de fatores anti-apoptóticos, anti-cicatrizantes, quimioatraentes, neuroprotetores, neurogênicos e angiogênicos; favorecimento do crescimento e da diferenciação local de células-tronco e progenitoras; (Revisado por Singer e Caplan, A.I, 2011; Revisado por Soleymaninejadia et al., 2012; Revisado por Honmou, O. et al., 2012). As CTMH também apresentam baixa expressão de MHC de classe I, MHC de classe II e das moléculas coestimuladoras B7-1/B7-2 (CD80/CD86), conferindo um imunoprivilégio que propicia seu uso em transplantes alogênicos e em doenças auto-imunes (Revisado por Singer e Caplan, 2011; Bem-Ami et al., 2011; Miguel et al., 2012).
Outra característica interessante é que as CTMH podem modular a progressão do tumor e a sensibilidade a fármacos. Muitas evidências demonstram que elas apresentam tropismo para região do tumor, promovem a proliferação das células tumorais e conferem um fenótipo de resistência a fármacos. No entanto, alguns estudos verificaram inibição de crescimento tumoral em leucemias e hepatomas, por exemplo. Ainda não estão elucidados os fatores que definem o papel das CTMH na progressão ou inibição tumoral. Porém, acredita-se que seu tropismo pode favorecer o uso destas células como veículo de fármacos para uma terapia anti-tumoral mais eficiente (Revisado por Houthuijzen et al., 2012).
Um total de 225 estudos clínicos utilizando as CTMH para tratamento de diferentes doenças está registrado no banco de dados do Instituto Nacional de
Saúde dos Estados Unidos
(http://www.clinicaltrials.gov/ct2/results?term=stem+cell+mesenchymal&no_unk =Y, acessado no dia 27 de agosto de 2012). Este número é ainda subestimado considerando que há muitos estudos em andamento que não são registrados em bancos de dados. Ambas as pesquisas clínicas em andamento e a plasticidade funcional das CTMH descrita acima demostram seu potencial de utilização na terapia celular de várias doenças, tais como: cardiopatias, doenças imunológicas, doença de Crohn, angiogênese, diabetes, esclerose múltipla, injúrias cerebrais isquêmicas e terapia contra o câncer (Meirelles e
23 http://www.clinicaltrials.gov/ct2/results?term=stem+cell+mesenchymal&no_unk =Y, acessado no dia 27 de agosto de 2012).
A fonte mais comum para obtenção de CTMH é a medula óssea (MO-CTMH) (Pittenger et al.,1999; Jiang et al.,2002). Apesar da identificação de um novo método (seleção negativa pelo sorteamento magnético seguido pelo FACS - fluorochrome activated cell sorting para a depleção de linhagens hematopoiéticas e endoteliais) de isolar populações mais enriquecidas em células progenitoras mesenquimais/osteoblásticas (Mödder et al., 2012), ainda existe uma necessidade de conhecer mais sobre fontes alternativas de CTMH devido à existência de um número limitado de MO-CTMH disponíveis para utilização autóloga. Há outros problemas em se utilizar MO-CTMH, como o procedimento invasivo de aspiração deste tecido, e a diminuição significativa da frequência e potencial de diferenciação das MO-CTMH com o aumento da idade do doador (Kretlow et al., 2008; Chen et al., 2009).
2.2. Cordão umbilical como fonte de CTMH
O cordão umbilical humano é um anexo fetal originado de um mesoderma extra-embrionário, importante durante a gravidez e que é descartado após o nascimento. Descrita em 1977 por Nanaev e colaboradores, sua arquitetura interna é constituída por vasos sanguíneos, uma veia e duas artérias, envoltos por um tecido conectivo, também chamado de geleia de Wharton, delimitado pelo epitélio umbilical. A geleia é composta por uma matriz esponjosa, rica em fibras de colágenos e proteoglicanas, embebidas por células estromais responsáveis pela síntese dos componentes da matriz (Revisado por Fan et al., 2011). Este tecido conectivo é importante por prevenir a compressão, torção, e o enrolamento dos vasos que realizam fluxo sanguíneo bidirecional entre o feto e a mãe (Can e Karahuseyinoglu, 2007).
24 (Revisado por Troyer, e Weiss, 2008; Fan et al., 2011); derivam de tecidos que são descartados após o nascimento; maior acessibilidade, não há a necessidade de procedimentos invasivos como ocorre na obtenção de MO e tecido adiposo; menor risco de infecção devido à presença da barreira placentária (revisado por Yang et al., 2012); é possível a obtenção de maior número de CMTH que possuem grande potencial proliferativo ao longo tempo do cultivo comparadas às originadas da MO de idosos (Petsa et al., 2009; Revisado por Fan et al., 2011); são hipoimunogênicas, enquanto as CTE podem provocar uma resposta imunológica depois do transplante (Revisado por Bieback e Brinkmann, 2010).
As vantagens descritas acima colocaram as CTMH do cordão como uma importante fonte alternativa de células para uso na terapia celular. Vários estudos já demonstram seu potencial em regeneração cutânea (Revisado Yang et al., 2012), cardiomiogênico (Roura et al., 2010; Bai et al. 2012), em reparo de danos neurológicos (Lu et al., 2012; Dalous et al., 2012, Koh et al.,2008). Em uma consulta ao banco de dados do Instituto Nacional de Saúde dos Estados Unidos (http://www.clinicaltrials.gov), utilizando os termos Umbilical Cord e Mesenchymal Stem Cell, acessado no dia 30 de agosto de 2012, foram encontrados 43 estudos clínicos registrados, incluindo: hepatite auto imune; cirrose biliar primária, reconstituição imunológica de pacientes com HIV; cirrose hepática; doença de Alzheimer; displasia broncopulmonar; esclerose lateral amiotrófica; nefrite lúpica; colite ulcerativa; diabetes tipo 1; cardiopatia dilatada idiopática; ataxia hereditária; esclerose múltipla e neuromielite óptica; distrofia muscular de Duchene; artrite reumatoide e queimadura aguda.
25 sangue do cordão, obtendo sucesso apenas com a MO. Outros estudos também falharam ou tiverem uma eficiência de isolamento muito baixa (Wexler et al., 2003; Romanov et al., 2003). Tais resultados motivaram pesquisas por novas fontes de CTMH, levando à descoberta da presença de CTMH no endotélio da veia do cordão (Romanov et al., 2003; Covas et al., 2003) e da geleia de Wharton (Wang et al., 2004; Fong et al.; 2007). Posteriormente, outros pesquisadores documentaram a presença de CTMH numa região que fica ao redor dos vasos umbilicais (Baksh et al., 2007; Sarugaser et al., 2005).
26 As diferenças citadas acima podem ocorrer devido à aplicação de metodologias distintas de isolamento e condições de cultura, à própria identidade biológica dos doadores, ou por outro lado, sugerir potencialidades distintas para estas populações celulares. Apesar de existirem vários trabalhos sobre CTMH isoladas de tecidos umbilicais, o conhecimento biológico e genético sobre elas ainda é insuficiente para que o seu potencial terapêutico seja explorado com segurança e eficácia, sobretudo no que diz respeito às CTMH isoladas da veia do cordão umbilical humano. Além disso, o uso dessas células na clínica depende de seu isolamento e expansão in vitro. Portanto, informações sobre as propriedades biológicas dessas células após um longo período de cultivo pode auxiliar na elaboração de novas estratégias que assegurem e monitorem a manutenção do fenótipo de célula-tronco, oferecendo maior garantia e segurança para seu uso nos tratamentos clínicos.
2.3. Senescência celular
27 Diabete Mellitus, osteoartrite, aterosclerose, diminuição da cicatrização de feridas e da resposta imune (revisado por Bekaert et al., 2005).
Além do encurtamento dos telômeros, outros fatores estão associados à senescência celular, tais como estresse oxidativo (Toussaint et al., 2000); senescência prematura induzida por estresse (Serrano e Blasco, 2001; Toussaint et al., 2000); ativação de oncogenes (revisados por Prieur e Peeper, 2008); e ativação da DDR (revisado por d‟Adda di Fagagna, 2008). Resumidamente, o mecanismo de senescência celular pode ser explicado da seguinte maneira: danos no DNA (causados por vários tipos de estresse como, disfunção telomérica; quebras de dupla fita; estresse oxidativo; e ativação de oncogenes) resultam na ativação da DDR promovendo a parada no ciclo celular de forma irreversível, mesmo na presença de fatores de crescimento (revisado por Magalhães, 2004).
Alguns estudos tem contribuído para uma contribuição na compreensão de mecanismos moleculares à senescência envolvidos com a parada do ciclo celular. Genes como CCNA2, que promovem a proliferação celular, podem ter sua expressão inibida. Alguns trabalhos demonstram que genes supressores tumorais, como P16/RB e P53/p21, podem levar à formação de SAHF. Estes genes fazem parte da via DDR relacionada à senescência (Revisado por Sikora et al., 2011). Também foi observado que nas células senescentes são formados focos de heterocromatina (SAHF- senescence-associated heterochromatic foci), que podem contribuir para a natureza irreversível da senescência.
28 Wojda et al., 2006) e adquirem um fenótipo secretoma característico, chamado de fenótipo secretório associado à senescência (SASP - senescent-associated secretory phenotype). Ele compreende o aumento no nível de liberação de mais de 40 fatores que atuam na sinalização celular, fatores de crescimento, proteases, entre outros (Revisado por Davalos et al., 2010). Foi proposto que o SASP contribui para a manutenção da senescência no tecido e para o desenvolvimento de doenças relacionadas à idade e dá suporte à carcinogênese (Revisado por Sikora et al., 2011). Contudo, detalhes moleculares das redes de interações de vias relacionadas ao processo de senescência celular ainda precisam ser elucidados para uma melhor compreensão sobre a sua ligação com o envelhecimento in vivo, com o surgimento de doenças e com o câncer.
No indivíduo adulto, a homeostasia do tecido se dá por meio da renovação e diferenciação de células-tronco somáticas, específicas de cada tecido. Durante o envelhecimento, fatores ambientais, o genótipo do indíviduo e fatores estocásticos podem induzir gradualmente alterações genéticas e epigenéticas que causam um declínio das funções de células-tronco que pode ser a origem das doenças metabólicas, degenerativas, câncer e o envelhecimento dos indivíduos (Revisado por Rodríguez-Rodero, et al. 2011). Esta proposição está em conformidade com a observação de que o envelhecimento afeta a renovação e a capacidade de diferenciação de CTMH residentes no tecido adiposo e na MO de indivíduos mais velhos (Alt et al., 2012; Kretlow et al., 2008; Chen et al., 2009), bem como a morfologia das CTMH obtidas da MO em indivíduos idosos (Zaim et al., 2012). As vias envolvidas com as alterações na sequência do DNA no envelhecimento das células-tronco somáticas não são claras, mas alguns estudos sugerem que o comprimento do telômero ou deficiências no reparo possam fazer parte destas rotas (Revisado por Rodríguez-Rodero et al., 2011).
29 maiores; declínio na expressão de marcadores moleculares específicos, bem como, diminuição de seu potencial de diferenciação adipogênico e aumento do osteogênico. Outros autores demonstram que as MO-CTMH perdem gradualmente sua capacidade progenitora (Banfi et al.,2000). Além destas consequências funcionais, o co-cultivo de CTMH com HSC evidenciou que quando foi utilizado CTMH de passagens tardias houve um aumento da proliferação de células progenitoras hematopoiéticas enquanto que com CTMH de passagens iniciais as HSC mantiveram um fenótipo mais primitivo (Walenda et al., 2009). Como já foi descrito que a secreção de fatores de crescimento e citocinas por fibroblasto pode ser influenciada pelo tempo de cultura (Coppe et al. 2008), uma sugestão é que as funções imunomoduladoras de CTMH possam ser alteradas durante a expansão na cultura por alteração no seu perfil secretório.
Os estudos de senescência celular têm utilizado o número de passagens, o tempo para duplicação da população (PD-Population doublings); e a detecção da -galactosidase (SA- -gal), para identificar as células senescentes após sua expansão. O número de passagens representa o número de vezes que a cultura foi expandida. O tempo requerido para isto depende do número de células semeadas inicialmente, o qual afeta o momento em que elas alcançam a confluência, e, consequentemente, a frequência da passagem. Portanto, podem haver diferenças laboratoriais produzindo artefatos no estudo da senescência. O PD é o quociente entre o número de células colhidas dividido pelo número de células que foram inicialmente semeadas. No entanto, este número expressa apenas o número cumulativo de divisões celulares de toda a cultura, independentemente de células individuais; algumas células podem ser perdidas durante a lavagem; e já é documentado que há diferenças entre PD sob mesma condição de cultura entre doadores. O ensaio da enzima SA- -gal é um método bem estabelecido que pode ser aplicado em preparações de CTMH. Embora ele não facilite a quantificação absoluta de células senescentes, a enzima é ativa apenas em senescentes e não em quiescentes, pré-senescentes, ou diferenciadas (Revisado por Wagner et al., 2010). Entretanto, em culturas confluentes pode ocorrer a coloração com a
30 2.4. Estabilidade genômica das CTMH ao longo tempo de cultivo
A proliferação das células em cultivo é usualmente maior do que in vivo, com isso elas estão sujeitas a acumular mais danos no DNA quando expandidas in vitro. Diversos estudos relatam que o tempo de cultivo pode levar à aquisição de alterações cromossômicas em CTMH humanas (Bochkov et al., 2007; Maitra et al., 2005; Sareen et al., 2009; Stephenson et al., 2010; Ueyama et al., 2011;). O mais recente e maior estudo de estabilidade genômica em células-tronco adultas utilizou 400 amostras isoladas de diferentes tecidos (de MO, tecido adiposo, cordão umbilical e tecidos fetais) e nove amostras de CTMH derivadas de CT embrionárias. Estes autores encontraram nove alterações em seis das 144 amostras de CTMH analisadas, uma frequência de 4%, menor do que a encontrada em CT neurais e CT pluripotentes induzidas, ambas apresentaram cerca de 9%. Eles demonstraram que a presença de alterações cromossômicas é uma característica comum de células-tronco propagadas in vitro, e sugeriram que cada tipo de célula-tronco tem uma predisposição a adquirir um conjunto particular de alterações cromossômicas. Em CTMH foi identificada uma tendência ao surgimento de monossomia, uma delas foi do cromossomo 13. Tal alteração é relacionada com tumores mesenquimais, frequente em tumores de tecidos moles e osso (lipomas, condrossarcomas e osteossarcomas) (Ben David et al., 2011). Além disso, em modelos animais, CTMH expandidas in vitro apresentam uma alta susceptibilidade à transformação maligna (Miura et al., 2006; Tolar et al., 2007; Ren et al., 2011).
31 transformação encontrados foram originados de contaminação cruzada (de La Fuente et al., 2010; Torsvik et al., 2010). Contudo, a presença de aneuploidia em várias preparações de MO-CTMH para aplicação clínica foi observada depois de seu cultivo. No entanto, como valores de expressão de genes relacionados à transformação estavam normais, os autores sugeriram que aneuploidia poderia aparecer durante a expansão, mas isso não significaria uma transformação, e sim uma senescência celular (Tarte et al., 2010). Depois disso, as pesquisas clínicas com CTMH na França foram liberadas, mesmo sendo identificadas aneuploidias.
A inexistência de alterações cromossômicas também foi relatada em CTMH do cordão umbilical expandidas in vitro (Spees et al., 2004). Um estudo com infusão de CTMH em modelos animais revelou que em nenhum dos camundongos tratados com CTMH da geleia de Wharton houve o aparecimento de doença e nem reações inflamatórias (Fong et al., 2010; Gauthaman et al., 2012). Estes estudos indicam que as CTMH do cordão umbilical são hipoimunogênicas e não tumorigênicas, com grande potencial para o uso seguro na terapia celular. Nekanti e colaboradores (2010) observaram cariótipos normais de CTMH da geleia de Wharton expandidas até a passagem 20. Duarte e colaboradores (2012) encontraram um grande número de alterações cromossômicas após a criopreservação das UV-CTMH de um dos cordões, entretanto, nenhuma alteração clonal foi observada.
32 2.5. Microarranjos e Estudos de expressão gênica em CTMH
Uma maneira de conhecer a função dos genes é por monitoramento da expressão de milhares de genes em uma amostra sobre uma determinada condição. O conjunto de moléculas de RNA transcritas de um genoma é chamado de transcriptoma. O método mais comum para sua avaliação é o de microarranjos(Auer et al., 2009; Hey e Pepper, 2009). Eles são um conjunto de sequências de DNA (DNA complementar ou oligonucleotídeo) construídas artificialmente, complementares aos transcritos dos genes que se deseja interrogar a expressão, e distribuídas em sítios específicos (spots ou probe cell) em uma superfície sólida. Cada spot contém centenas de cópias das sequências de desoxirribunucleotídeos complementares a um único RNA alvo (Revisado por Bute, 2002).
Na pesquisa médica, o estudo do perfil de expressão por microarranjos surge como uma ferramenta útil para melhor compreensão de doenças, identificação de novos alvos terapêuticos, e subclassificação de doenças para identificar estratégias mais individualizadas de tratamento (Revisado por Auer et al., 2009). Em outras pesquisas básicas e aplicadas, sua análise tem como objetivo acessar o perfil molecular de uma dada amostra; descobrir novos biomarcadores; ou fornecer informações para fazer novas anotações do genoma. As duas análises principais deste método consistem em testar genes diferencialmente expressos entre duas ou mais condições, e identificar as categorias funcionais mais representadas pelos genes diferencialmente expressos em um estudo (Revisado por Kauffmann e Huber, 2010).
33 sequências de sondas com 25 nucleotídeos, conhecidos como um conjunto de sonda (probe set). O agrupamento das intensidades em várias sondas resulta em uma medida única de expressão de um gene. Um dos microarranjos da affymetrix, é o GeneChip® Human Genome U133 plus 2.0 Array (HG U133 Plus 2.0). Neste há 1.300.000 oligonucleotídeos únicos cobrindo mais de
47.000 transcritos e variantes (acessado em:
http://media.affymetrix.com/support/technical/datasheets/hgu133arrays_datash eet.pdf, no dia 08 de setembro de 2012).
Embora ainda haja algumas limitações na técnica de microarranjos, como sua incapacidade de descoberta de novos RNA mensageiros, ela é considerada uma tecnologia consolidada para análise de transcriptoma utilizada a mais de uma década. Mais recentemente, muita atenção tem sido dada ao sequenciamento de RNA (RNA-seq). Esta nova abordagem, uma das técnicas de sequenciamento de última geração (next-generation sequencing – NGS), supera as limitações da primeira. No entanto, acredita-se que sua existência não extinguirá a utilização dos microarranjoss. A escolha deverá se dar pelo custo, disponibilidade, e a necessidade de cada pesquisa (Revisado por Sánchez-Pla et al., 2012).
34 2007; Weng et al., 2011; Kim et al., 2011). CTMH isoladas a partir da veia do cordão são funcionalmente semelhantes às MO-CTMH, mas genes diferencialmente expressos podem refletir as diferenças relacionadas aos seus locais de origem: MO-CTMH seriam mais comprometidas com a osteogênese, enquanto CTMH do cordão seriam mais comprometidas com a angiogênese (Panepucci et al., 2004). Uma comparação, mais recente, de dados de transcriptoma e proteômica entre CTMH da veia do cordão e MO-CTMH, encontrou alguns genes/proteínas diferencialmente expressos, mas que participam de funções celulares semelhantes (Miranda et al., 2012).
Os estudos descritos acima mostram que CTMH residentes em locais distintos guardam muitas semelhanças, sugerindo uma origem comum, e diferenças que possivelmente refletem distintas propriedades biológicas adquiridas pelo seu nicho natural (tais como, potenciais de proliferação e diferenciação), que, talvez, possam ser modificadas com a escolha adequada das condições de cultivo. Por exemplo, a substituição do meio de cultivo de CTMH pelo de MAPC (Multipotent Adult Progenitor Cells), tornou as CTMH capazes de se diferenciar em células semelhantes às endoteliais e aumentou a expressão de cinco transcritos endoteliais (CD34, VWF, FLK1, FLT1, TIE2) (Roobrouck et al., 2011).
35 entanto, esse mesmo estudo detectou diminuição na quantidade da proteína p53 em ASC em P30. O estudo ainda comparou estas linhagens com as mesmas obtidas de macaco Rhesus em passagens iguais, sendo observada uma alteração significativa no perfil de expressão de genes envolvidos com o ciclo celular, resposta ao dano e reparo do DNA, tais como TP53. A diminuição da expressão da p53 pode estar relacionada com a menor taxa de apoptose nas ASC humanas e de células-tronco mesenquimais de MO de Rhesus na passagem 30, bem como é associada com o fato de que estas últimas puderam ser cultivadas além da passagem 30 (Izadpanah et al., 2008).
Uma análise de expressão gênica, por meio de proteômica, de CTMH da geleia de Wharton depois da expansão in vitro até a 12º passagem, revelou que diversas proteínas, incluindo Shootin1, Adenilatoquinase 5 isoenzima e inibidor do ativador do plasminogénio-2 já não são expressas após a passagem 2, sugerindo que a potência proliferativa destas células é reduzida após a sua fase inicial de crescimento in vitro. Este estudo também observou que na última passagem analisada, houve o surgimento de novas proteínas, incluindo, ERO1-alfa, aspartil-tRNA sintetase e prolil-4-hidroxilase, indicando que estas proteínas estejam envolvidas na dificuldade de sobrevivência e diferenciação celular após o tempo de cultivo (Angelucci et al., 2010). O perfil de expressão gênica de CTMH de diferentes origens (líquido amniótico, membrana amniótica, sangue do cordão, e MO) permaneceu estável entre as passagens 3 e 6, durante a cultura in vitro , e depois da criopreservação (Tsai et al., 2007).
36 3.0. OBJETIVOS
O presente trabalho teve como objetivo geral analisar o efeito da senescência no perfil de expressão gênica das CTMH/inv, que têm a inversão cromossômica (inv(3)(p13p25~26), e das CTMH/n, que apresentam cariótipo constitucional normal, sob as mesmas condições de cultivo, bem como comparar, pela primeira vez, o perfil de expressão gênica de CTMH/inv com o perfil de CTMH/n jovens e senescentes.
Objetivos específicos:
Identificar o número de passagens in vitro necessário para obtenção de uma cultura celular senescente;
Verificar se há diferença entre o perfil de expressão de senescentes vs jovens em cada CTMH/inv e CTMH/n;
Verificar se há diferença entre o perfil de expressão de CTMH/inv vs CTMH/n, jovens e senescentes;
Verificar se a expressão diferencial de genes identificados na análise de microarranjos é corroborada por PCR em tempo real; Identificar as categorias funcionais mais representadas pelos
genes diferencialmente expressos para cada comparação;
37 4.0. MATERIAL E MÉTODOS
4.1. Isolamento e Caracterização das CTMH
No presente trabalho, foram utilizadas CTMH obtidas do endotélio da veia do cordão umbilical de três doadores. Os cordões foram coletados na maternidade Januário Cicco da Universidade Federal do Rio Grande do Norte (UFRN). Todas as mães que doaram os cordões assinaram o termo de consentimento livre e esclarecido de acordo com aprovação do Comitê de Ética em Pesquisa da UFRN sob o protocolo n°. FR132464. Os procedimentos realizados para a coleta de todos os cordões, bem como o isolamento, a caracterização imunofenotípica por citometria de fluxo, as diferenciações adipogênica, osteogênica e condrogênica, e a análise citogenética das CTMH do endotélio da veia do cordão seguiram os protocolos realizados por Duarte et al. (2012).
As CTMH isoladas dos três cordões umbilicais foram caracterizadas citogeneticamente por Cornélio (2012), Um dos cordões tem uma alteração cromossômica constitucional: inversão paracêntrica no braço curto do cromossomo 3, cariótipo: 46,XY,inv(3)(p13p25~26) (Duarte et al. 2012), esta linhagem é aqui denominada como CTMH/inv. Muitas células CTMH/inv senescentes apresentaram instabilidade genética, caracterizada pelo surgimento de alterações cromossômicas, especialmente, associações teloméricas (tas) e ganho de material genético do cromossomo 20, que são alterações características de tumor ósseo de células gigantes (TCG). Os outros dois cordões possuem cariótipo constitucional normal, portanto, no presente trabalho as CTMH isoladas de tais cordões serão denominadas CTMH/n.
38 4.2. Condições de cultura das CTMH para análise do seu perfil de expressão gênica
As células recuperadas da criopreservação, conforme protocolo de Duarte et al. (2012), foram plaqueadas em frascos T25 com meio alfa-MEM (Gibco), suplementadas com 10% de SFB (Gibco) e 1% de solução de antibiótico (penicilina e estreptomicina – Sigma ou Gibco) e mantidas à 37 ºC em incubadora umidificada com 5% de CO2. Ao atingir a confluência de 60-70%, as células foram expandidas utilizando 1mL de 0,25% de tripsina/EDTA (Invitrogen). Depois de contadas em hemocitômetro com azul de tripan (Gibco), as células foram plaqueadas novamente em uma concentração de 4000 células/cm2 em diferentes frascos de cultura, este procedimento caracteriza uma passagem (P) celular. As células foram monitoradas em microscópio invertido (CKX 41, OLYMPUS) e a troca de meio foi realizada a cada 72 horas ou quando o meio ficava amarelado, indicativo de mudança de pH.
A expansão teve continuidade até a passagem 18 para obtenção das amostras para análise de expressão gênica das CTMH obtidas de dois cordões (cordão 10 – cariótipo normal - com 9 replicatas experimentais e cordão 04 – inv(3)(p13-25~26) - com 6 replicatas experimentais), e até a 9º passagem para CTMH do outro cordão (cordão 12 – cariótipo normal – com 3 replicatas experimentais), seguindo as mesmas condições de cultivo descritas acima.
Portanto, as passagens de cultura utilizadas para as análises do perfil de expressão gênica foram:
a) Passagem 9 – consideradas como CTMH jovens isoladas do cordão com inversão (grupo CTMH/inv) e de dois cordões com cariótipo constitucional normal (grupo CTMH/n).
b) Passagem 18 – consideradas como CTMH senescentes isoladas do cordão com inversão (grupo CTMH/inv) e de um cordão com cariótipo constitucional normal (grupo CTMH/n).
39 24. Nessa última passagem, as células foram mantidas em cultivo com trocas de meio e monitoradas diariamente pela observação em microscópio invertido.
4.3. Caracterização das CTMH senescentes
As células foram definidas como senescentes ao se visualizar em microscópio invertido (CKX 41, OLYMPUS) as seguintes características: aumento do tamanho celular, alteração na morfologia, aumento na presença de células vacuolizadas, diminuição da capacidade proliferativa e maior proporção de células flutuantes no meio de cultura.
Além disso, foi realizado o ensaio para detecção da atividade da -galactosidase em pH 6,0, utilizando o Kit Senescence - galactosidase da Cell Signaling Technology, conforme as instruções do fabricante (Chemicon, USA) Resumidamente, as células foram fixadas durante 5 min à temperatura ambiente em solução fixadora 1X, em seguida, foram lavadas e incubadas durante a noite à 37 º C com solução de coloração SA- -gal. Posteriormente, as células foram lavadas com PBS e visualizadas utilizando um microscópio de luz. As células que apresentaram coloração verde foram consideradas senescentes. Para evitar a interferência da confluência celular, o ensaio foi realizado em culturas com 60% de confluência em triplicatas experimentais.
4.4. Extração de RNA
O RNA total foi extraído de 1x106 células (~90% de confluência) em dois momentos: na passagem 9, caracterizadas como células jovens das CTMH/inv (obtidas do cordão 04) e CTMH/n (obtidas dos cordões 10 e 12); e na passagem 18, caracterizadas como senescentes das CTMH/inv (obtidas do cordão 04) e CTMH/n (obtidas do cordão 10).
41 4.5. Preparação do RNA, Hibridização e Captura da imagem do Microarranjos.
Do RNA total extraído de cada replicata de todas as CTMH/n e CTMH/inv, jovens e senescentes, 100 ng foi utilizado para a marcação utilizando o GeneChip® 3’ IVT Express Kit (Affymetrix Inc.) de acordo com as instruções do mesmo. Resumidamente, a primeira fita cDNA foi sintetizada pela transcrição reversa utilizando um primer oligo dT etiquetado com um promotor T7 a partir do RNA total à 42 °C por 2 horas. Este cDNA foi então convertido em dupla fita utilizando a complementariedade do promotor T7 da primeira fita do cDNA à 16 °C por 1 hora. Posteriormente, foi realizada uma transcrição in vitro para sintetizar RNA marcado (aRNA) por meio da incorporação de nucleotídeos conjugados com biotina à 40 °C por 16 horas. O aRNA foi então purificado por microesferas magnéticas e 15 μg deste foi fragmentado à 94 °C por 35 minutos. O aRNA purificado e fragmentado foi mantido à -20 ºC até sua hibridização no Laboratório Nacional de Luz Síncroton (LNLS) em Campinas-SP. Depois, 12,5 μg do aRNA fragmentado foi hibridizado no microarranjo Affymetrix Human Genome U133 plus 2.0 arrays, juntamente com os controles e a solução de hibridização conforme indicado pelo Hybridization, Wash, and Stain Kit (Affymetrix Inc). Em seguida, os microarranjos foram mantidos em uma incubadora, Genechip Hybridization Oven-640 (Affymetrix Inc), com rotação de 60rpm à 45ºC por 16 horas. Depois, os microarranjos foram lavados na estação de lavagem, Genechip Fluidics Station-450 (Affymetrix Inc), e corados para amplificação do sinal com o Hybridization, Wash, and Stain Kit (Affymetrix Inc), como indicado pelo manual do fabricante. Os microarranjos foram escaneados com o Affymetrix Gene-Chip Scanner-3000-7G (Affymetrix Inc). A qualidade da hibridização foi avaliada pelo Affymetrix GCOS software seguindo as instruções do fabricante.
4.6. Análises dos dados de microarranjos
42 recomendado pelo fabricante. Subsequentemente, os arquivos CEL foram importados para o programa Partek Genomic Suite (version 6.4; Partek Inc., St. Louis, MO) utilizando o algorítimo RMA para sumarização e normalização com correção de background CG. O sinal de cada transcrito foi calculado utilizando a média das intensidades de cada probset correspondente ao mesmo transcrito. A correspondência entre as replicatas experimentais foi realizada utilizando análise de componente principal e de correlação de Pearson. Posteriormente, para identificação dos genes diferencialmente expressos foi aplicado o teste ANOVA (one-way analysis of variance) com correção Bonferroni. Foi considerado estatisticamente significativo os genes com o valor de p 0,001 com correção Bonferroni e com o fold change 3,0. Os perfis de expressão gênica comparados foram:
a) CTMH/n senescentes (replicatas do cordão 10) vs CTMH/n jovens (replicatas do cordão 10);
b) CTMH/inv senescentes (replicatas do cordão 04) vs CTMH/inv jovens (replicatas do cordão 04);
c) CTMH/inv jovens (replicatas do cordão 04) vs CTMH/n jovens (replicatas do cordão 10 e 12);
d) CTMH/inv senescentes (replicatas do cordão 04) vs CTMH/n senescentes (replicatas do cordão 10).
43 quantificação relativa, utilizando o gene YWHAZ como referência endógena, o qual já foi identificado como melhor endógeno para CTMH de cordão umbilical por Wang et al. (2010). O gene foi eleito como endógeno para o qPCR por meio de análise de variação de seus valores de expressão nos microarranjos. A análise estatística foi feita utilizando o programa geNorm M. O valor de M para YWHAZ foi de 0,142, demonstrando uma expressão estável para este gene.
Para realização do qPCR foram selecionados 11 genes (Tabela 1) a partir da lista do genes diferencialmentes expressos em ambas as comparações CTMH/inv jovens vs CTMH/n jovens e em CTMH/inv senescentes vs CTMH/n senescentes. Foram eleitos conforme seu padrão de expressão, bem como pela sua relevância biológica sugerida pelo Igenuity Analysis Pathway (IPA) (Ingenuity Systems, Redwood City, CA, USA). Todos os ensaios foram sintetizados pelo serviço sondas inventoriadas com marcação FAM (Applied Biosystems).
Os genes ANKRD1 e MMP1 também foram utilizados para comparação entre as CTMH/n (cordão 10) senescentes vs CTMH/n (cordão 10) jovens. Os genes SFRP1, G0S2, ANKRD1 e NDN foram utilizados para a comparação entre as CTMH/inv (cordão 04) senescentes vs CTMH/inv (cordão 04) jovens. Assim, ao total foram 28 comparações de expressão gênica entre os dados de microarranjos e de qPCR.
Gene ID do ensaio
LAMC2 Hs01043711_m1
ANKRD1 Hs00173317_m1
KYNU Hs00187560_m1
MMP1 Hs00899658_m1
MAB21L1 Hs00366575_s1
SFRP1 Hs00610060_m1
NDN Hs00267349_s1
ADORAB2 Hs00386497_m1
CCL7 Hs00171147_m1
G0s2 Hs00274783_s1
ALDH1A1 Hs00946916_m1
YWHAZ Hs03044281_g1
44 4.8. Classificação funcional dos genes diferencialmente expressos Os genes diferencialmente expressos foram submetidos à analise biológica utilizando o programa Igenuity Patway analysis (IPA) – (Ingenuity Systems, Redwood City, CA, USA) – para identificar as categorias funcionais mais representadas pelos genes diferencialmente expressos. Os paramêtros selecionados para esta análise no programa, foram: Reference set – Igenuity Knowlodge Base; Relationship to include – Direta and indirect; Includes Endogenous Chemicall; Filter – consider only molecules and/or relationships where species = human.
4.9. Análises de biologia de sistemas
As redes de interações foram construídas utilizando o software STRING versão 9.0 (Search Tool for the Retrieval of Interacting Genes/Proteins) (Szklarczyk et al., 2012). Para cada lista de genes diferencialmente expressos foram construídas duas redes: uma incluindo somente os genes da lista e outra incluindo genes do genoma humano com o limite de 50 genes. Os parâmetros para construção aplicados foram: organismo, homo sapiens; confidência média; e outros presentes no „default‟ do programa. Todos os métodos de predição foram ativos: Neighborhood, Gene Fusion, Cooccurrence, Co-expression, Experiments, Databases e Textmining. Posteriormente, o arquivo txt sumarizado gerado pelo STRING foi utilizado para exportar para o programa Cytoscape 2.8.2 (Smooth et al., 2010), e subsequentemente, analisado. Para categorização funcional dos genes constituintes da rede foi utilizado pelo pluggin BiNGO 2.44 (Maere et al., 2005). Neste foi utilizado distribuição hipergeométrica, correção de testes múltiplos por meio do algoritmo de False Discovery Rate (FDR) com nível de significância para valor de P < 0.05. O organismo selecionado para a análise foi Homo sapiens.
46 5.0. RESULTADOS
5.1. Caracterização das CTMH jovens e senescentes
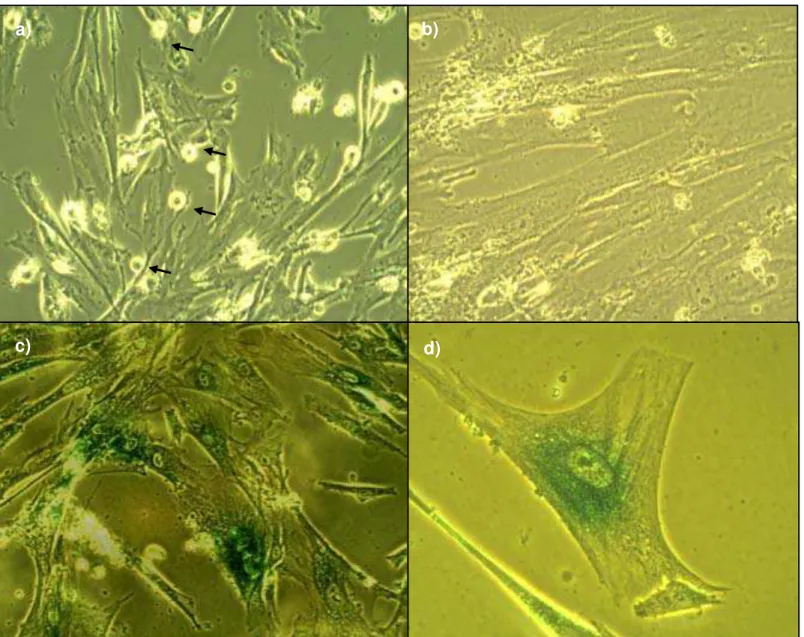
Ambas as CTMH/inv e CTMH/n foram isoladas e expandidas com sucesso conforme descrito por Cornélio (2012). Na 9º passagem, em frascos subconfluentes, não foi detectada nenhuma célula com características de células senescentes. Nesta passagem, foi possível identificar muitas células se dividindo e com morfologia típica de CTMH. Portanto, tal cultura foi considerada jovem (Figura 1a). Já na 18º passagem, também em frasco subconfluente, foi possível detectar pouquíssimas células se dividindo, a maioria delas foi marcada com -galactosidase, apresentando maior tamanho citoplasmático com vacúolos e acúmulo de grânulos. Então, esta cultura foi chamada de senescente (Figura 1b). Além disso, tanto as células jovens quanto as senescentes de ambas CTMH apresentaram os marcadores de superfície celular específicos de CTMH (CD73, CD90 e CD105) e foram capazes de se diferenciar em adipócitos e osteoblastoss (Figura 2a e b). As células jovens de ambas as CTMH foram capazes de se diferenciar em condrócitos (Figura 2c). No entanto, as CTMH/inv sofreram bastante com o processo de senescência in vitro e quando células senescentes apareceram no cultivo, foi difícil expandi-las em número celular suficiente para repetir o experimento de citometria de fluxo com todos os marcadores de linhagem hematopoiética.
Foi identificado que a partir da 16º passagem, em ambas CTMH/inv e CTMH/n, é possível visualizar células senescentes em cultura, no entanto, elas são muito poucas e ainda não comprometem a expansão in vitro. Somente na 18º passagem foram observadas predominância de células senescentes no cultivo, diminuindo a cinética do subcultivo, sendo que após este momento foi mais difícil obter células suficientes para fazer a extração de RNA.
48 Figura 1: a) Cultura jovem. População celular com morfologia fibroblastóide e muitas células se dividindo indicadas
pelas setas (objetiva de 20X); b) Cultura senescente. População celular com tamanho maior com um baixo índice de divisão mitótica (objetiva de 20x). c) Cultura senescente. Células coradas em verde pelo teste por -galactosidase, com formatos poligonais (objetiva de 20x). d) Célula senescente com formato poligonal, vacuolada e citoplasma rico
em grânulos e corada em verde pelo teste -galatosidase (objetiva de 40X).
a) b)
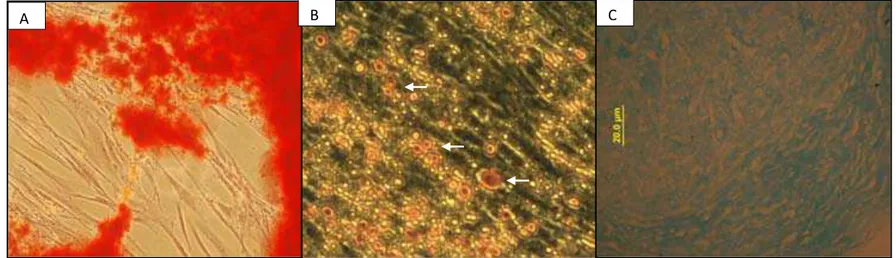
49 Figura 2. A) Diferenciação das CTMH após 21 dias de indução osteogênica, matriz corada com vermelho de alizarina (objetiva de
20x). B) Diferenciação adipogênica após 21 dias de indução, vacuólos adiposos corados por Oil Red O indicados pelas setas
(objetiva 40x). C) Diferenciação condrogênica, matriz extracelular corada em azul celeste pelo corante Alcian Blue (objetiva de
20X).
54 5.2. A senescência in vitro afeta o perfil de expressão das CTMH Para verificar se a senescência in vitro afeta a expressão gênica das CTMH/inv e das CTMH/n, foram feitas comparações entre as células senescentes e jovens de cada grupo: CTMH/n senescentes versus CTMH/n jovens e CTMH/inv senescentes versus CTMH/inv jovens.
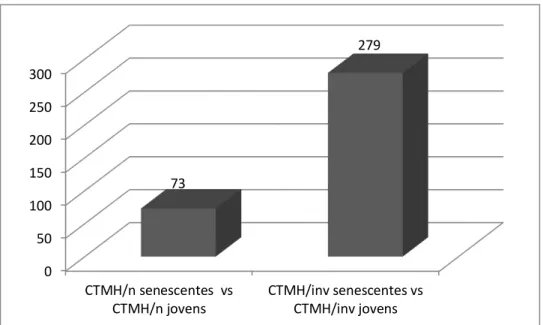
Foram identificados 73 genes diferencialmente expressos, 47 mais e 26 menos expressos em CTMH/n senescentes na comparação CTMH/n senescentes vs CTMH/n jovens (APÊNDICE A).
A comparação entre o perfil de expressão senescentes vs jovens das CTMH/inv resultou em 279 genes diferencialmente expressos identificados, 119 mais e 160 menos expressos em CTMH/inv senescentes (APÊNDICE B). Portanto, a diferença encontrada no perfil de expressão gênica entre as células senescentes e jovens é maior no grupo CTMH/inv do que nas CTMH/n (Figura 3).
Figura 3. Gráfico mostra que a comparação entre as CTMH/inv senescentes e CTMH/inv jovens resultou em maior número de genes diferencialmente expressos (279) do que na comparação entre as CTMH/n senescentes e CTMH/n jovens.
0 50 100 150 200 250 300
CTMH/n senescentes vs CTMH/n jovens
CTMH/inv senescentes vs CTMH/inv jovens 73
51 5.3. Classificação funcional e rede de interações dos genes diferencialmente expressos CTMH/n senescentes vs CTMH/n jovens
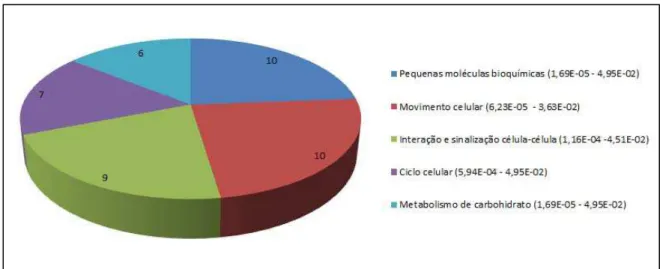
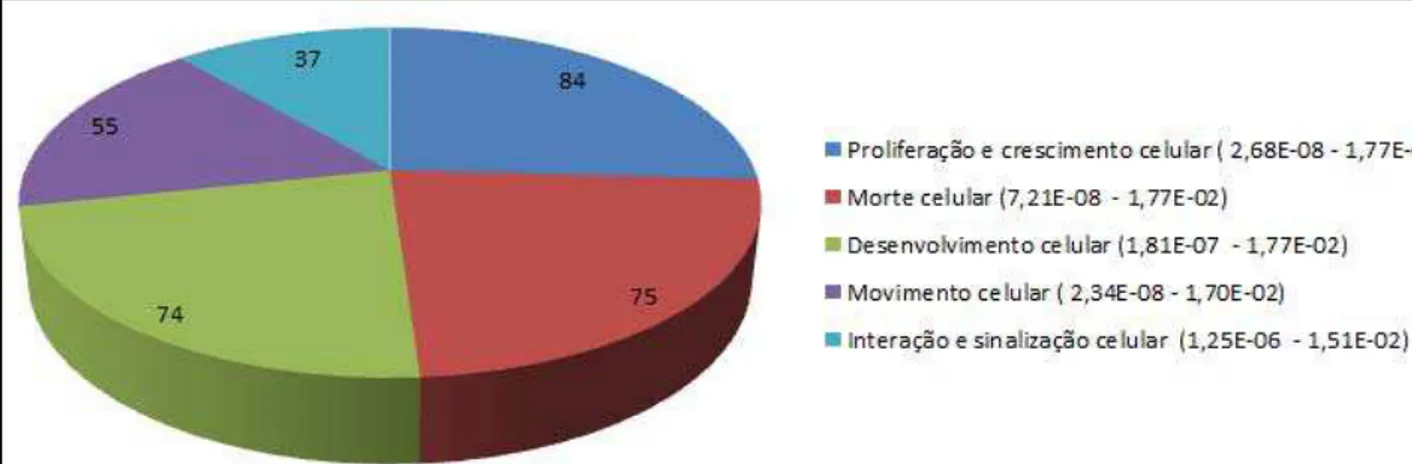
A análise de categoria funcional demonstrou que dentre os 73 genes identificados diferencialmente expressos em CTMH/n senescentes vs as mesmas jovens, 48 foram agrupados em 15 categorias funcionais com valor de p 0.05 e com uma representação biológica ≥ 5% de moléculas (APÊNDICE C). As categorias de maior enriquecimento de acordo com o valor de p foram metabolismo de carboidrato, pequenas moléculas bioquímicas, movimento celular, sinalização e interação célula-célula e ciclo celular (Figura 4 e APÊNDICE D).
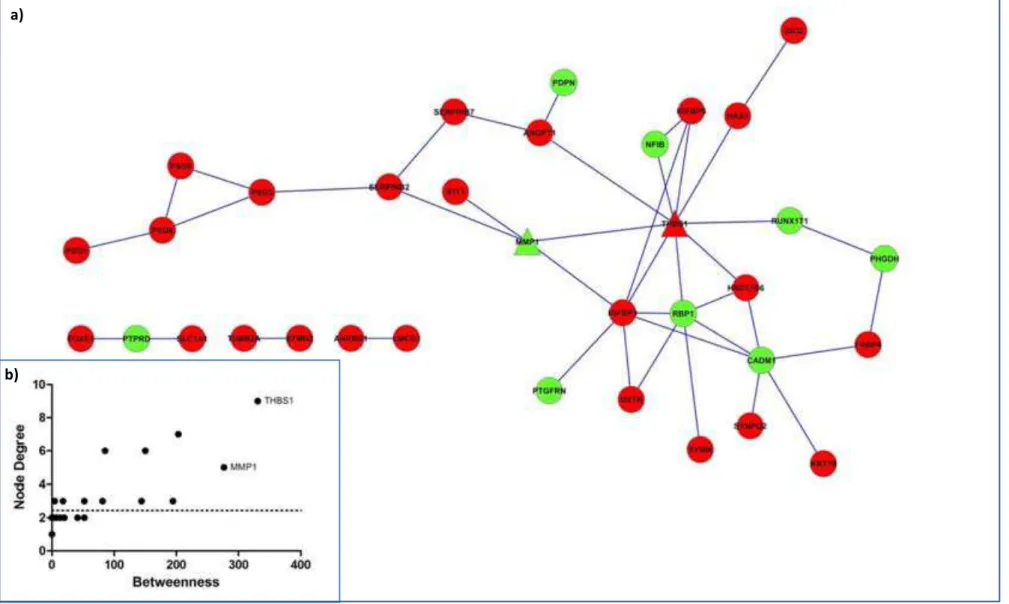
52 Os 73 genes formaram uma rede constituída de 31 nós e 41 interações. Foram identificados MMP1 e THBS1 como gargalos desta rede (Figura 5). A análise de classificação funcional das moléculas presentes nesta rede revelou um maior enriquecimento em processos multi-organismo, regulação de localização e migração celular e, regulação de componentes celulares (APÊNDICE E).
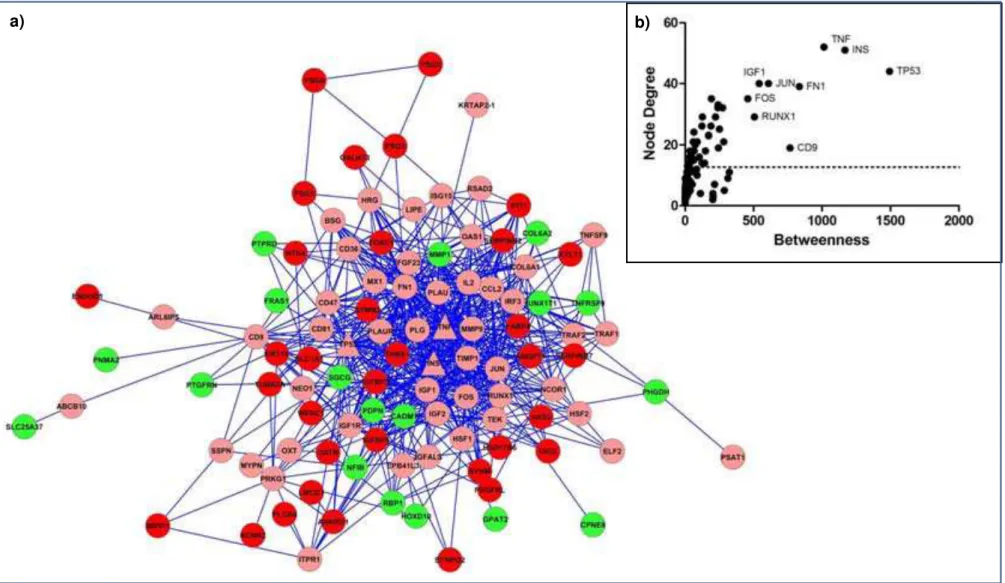
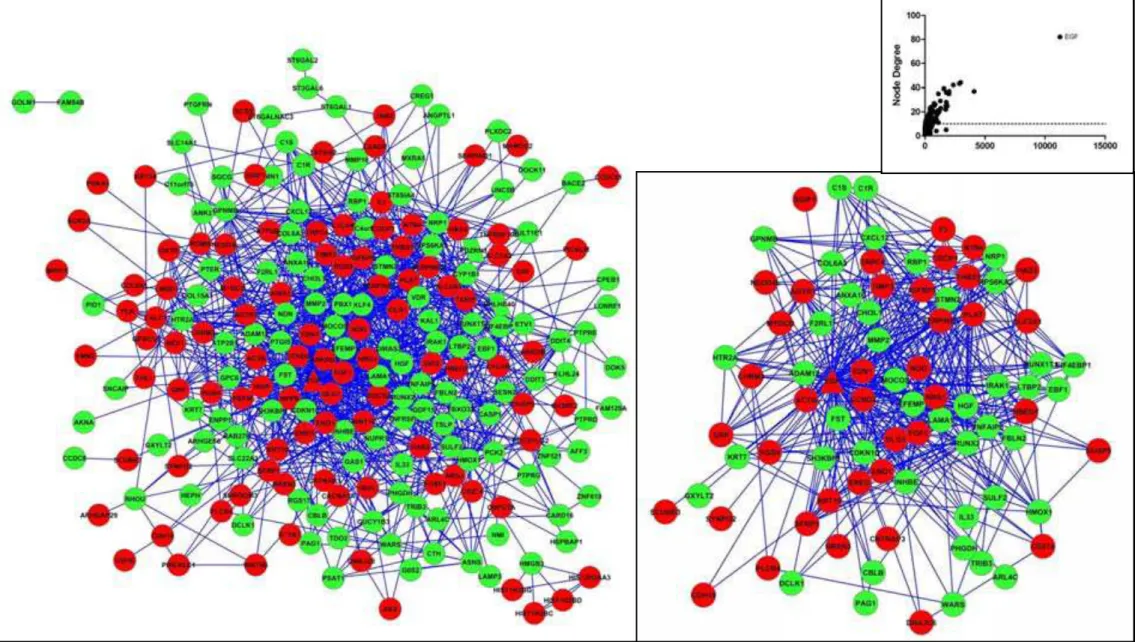
No contexto genômico, uma rede contendo 103 nós e 656 ligações foi formada (Figura 6a), MMP1 e THBS1 não foram identificados como gargalos. Todos os gargalos identificados não estão entre os genes que foram diferencialmente expressos (Figura 6b). Os processos biológicos mais enriquecidos nesta rede foram: regulação de processos biológicos, resposta a estímulos, desenvolvimento e sinalização (APÊNDICE F).
53 a)
b)
54 Figura 6: Rede de interações formada pelos genes diferencialmente expressos em CTMH/n senescentes comparadas às CTMH/n jovens no contexto genômico. As cores vermelhas e verdes representam, respectivamente, genes mais e menos expressos em CTMH/n senescentes. A cor rosa representa os genes do genoma humano que não foram diferencialmente expressos neste estudo. Os nós em forma de triângulo simbolizam genes identificados como gargalos da rede, TP53, TNF e INS. b) Gráfico demonstrando os genes que apresentaram maior node degree e betweeness presente na rede não estão entre os diferencialmente expressos em CTMH/n senescentes.