MAPEAMENTO CROMOSSÔMICO DE DNAs REPETITIVOS
COM FINS BIOTECNOLÓGICOS EM PEIXES MARINHOS DE
INTERESSE COMERCIAL – RACHYCENTRIDAE E LUTJANIDAE
Gideão Wagner Werneck Felix da Costa
Orientador: Prof. Dr. Wagner Franco Molina
Natal/RN
2015
Centro de Biociências
Laboratório de Genética de Recursos Marinhos
Tese apresentada ao programa de Pós‐
Graduação em Biotecnologia ‐ Rede
Nordeste de Biotecnologia (RENORBIO)
como
parte
dos
pré‐requisitos
"Jamais considere seus estudos como uma obrigação,
mas como uma oportunidade invejável para
aprender a conhecer a influência libertadora da
beleza do reino do espírito, para seu próprio prazer
pessoal e para proveito da comunidade à qual seu
futuro trabalho pertencer."
Aos meus pais Francisco Wagner Felix da Costa e Maria Evaneide Pinheiro da Costa, pela
educação, incentivo, carinho, compreensão e por tudo que me proporcionou na vida;
Aos meus irmãos Natanael e Otoniel Wagner, pelo convívio familiar e a Rosa Maria pelo
apoio durante a jornada do doutorado, todo carinho, apoio e amor;
Aos meus demais familiares, que torcem pelo meu sucesso; Aos mestres que um dia fizeram
parte da minha formação;
Aos funcionários da Universidade, que também são parte essencial no processo de ensino;
A Rede Nordeste de Biotecnologia (RENORBIO) pela oportunidade de realizar e alcançar um
objetivo;
A CAPES e ao CNPq pelo apoio fornecido para o desenvolvimento do trabalho;
Ao Professor Dr. Wagner Franco Molina, primeiro por aceitar‐me como orientando desde a
graduação e agora por abraçar mais uma vez esta luta em conjunto comigo, pelo apoio,
sugestões , ensinamentos transmitidos e pelo exemplo de profissionalismo, desempenhando
um papel fundamental na minha formação, muito obrigado!!!!!.
A todos do L.G.R.M, Rodrigo (Pantera), Clovis Motta, Paulo, Amanda, Karlla, Allyson, Inailson,
Aparecida, Juliana, Josy e Jeane, aos amigos que não estão mais presente no laboratório,
parceiros nas coletas e análises, espero não ter esquecido ninguém, pelo incentivo, apoio,
pelas horas de descontração e por tornar o laboratório um lugar harmonioso e descontraído;
Aos professores Dr. Marcelo de Bello Cioffi e Dr. Luiz Antônio Carlos Bertollo, pela parceria
nas elaborações dos artigos;
Ao professor Dr. Paulo Marinho, pela participação e contribuição na qualificação, e por
permitir a utilização do seu laboratório e material;
A professora Dra. Karina Ribeiro, pela participação e contribuição na qualificação;
Aos professores Dr. Marcelo Nóbrega e Dra. Katia Scortecci, pela participação da banca de
defesa e pelas contribuições;
A todos os amigos de fora da UFRN pela parte não científica da história, afinal ela é também
muito importante, diria até essencial, obrigado também;
A todos aqueles que de forma direta ou indiretamente contribuíram para este momento.
SUMÁRIO
1. INTRODUÇÃO GERAL ... 1.1. Considerações sobre Rachycentron canadum
1.2. Considerações sobre a família Lutjanidae 1.3. DNA repetitivo 1.4. Função dos elementos transponíveis na evolução do genoma 1.4.1. Retrotransposons 1.4.2. Transposons 2. OBJETIVOS GERAIS ... 3. OBJETIVOS ESPECÍFICOS ... 4. MATERIAIS E MÉTODOS ... 4.1. Materiais 4.2. Métodos 4.2.1. Preparações de cromossomos e células mitóticas 4.2.2. Bandamento cromossômicos 4.2.2.1. Detecção de heterocromatina constitutiva 4.2.2.2. Fluorocromos GC‐ e AT‐ específicos (MM e DAPI) 4.2.2.3. Obtenção das sondas de DNA para FISH por PCR 4.2.2.4. Obtenção de DNA Cot‐1 4.2.2.5. Oligonucleotídeos enriquecidos com microssatélites 4.2.2.6. Hibridação fluorescente in situ (FISH)
4.2.2.7. Análise das lâminas e idiograma
REFERÊNCIAS ... CAPÍTULO 1: Unusual dispersion of histone repeats on the whole chromosomal complement and their colocalization with ribosomal genes in Rachycentron canadum (Rachycentridae, Perciformes)...
Resumo Introdução Material e Métodos Resultados Discussão Referências
CAPÍTULO 2: Transposable Elements in Fish Chromosomes: A Study in the Marine Cobia Species ... Resumo Introdução Material e Métodos Resultados Discussão Referências CAPÍTULO 3: Structurally complex organization of repetitive DNAs in the genome of Cobia (Rachycentron canadum) ...
Discussão Referências
CAPÍTULO 4: Dinâmica de genes ribossomais e histonas em cinco espécies da família Lutjanidae ...
Resumo Introdução
Material e Métodos Resultados
Discussão Referências
CAPÍTULO 5: Moderada compartimentalização dos elementos Rex1 e Rex3 em Pargos (Lutjanidae, Perciformes) ...
Resumo Introdução
Material e Métodos Resultados
Discussão Referências
CONCLUSÕES ... CONSIDERAÇÕES FINAIS ...
INTRODUÇÃO
Figura 1. Fluxograma do processo produtivo em cativeiro de Rachycentron canadum.
Figura 2. Representação esquemática da constituição dos microssatélites.
Figura 3. Representação de alguns elementos transponíveis (DNA Transposons e Retrotransposons (LTR e não‐LTR). Figura adaptada de Thomas et al. (2010).
MATERIAIS E METÓDOS
Figura 4. Rachycentron canadum (Rachycentridae). Fonte: King SailFish.
Figura 5. Espécies da família Lutjanidae, (a) Lutjanus analis; (b) Lutjanus jocu; (c) Lutjanus alexandrei; (d) Lutjanus synagris e (e) Ocyurus chrysurus.
Capítulo 1
Figura 1. Cariótipos de R. canadum. (a) coloração com Giemsa; em destaque sítios Ag‐
RON, FISH 18S sobre o par organizador nucleolar (par 2) e marcações 5S; (b) bandamento C; (c) genes das histonas H2B‐H2A (verde) e (d) da histona H3 (vermelho). Barra=5µm.
Figura 2. Idiograma representativo dos cromossomos de R. canadum indicando a distribuição do DNAhis H2A‐H2B e H3 e dos sítios ribossomais 18S e 5S.
Capítulo 2
Figura 1. Cariótipos de R. canadum. (a) coloração Giemsa e (b) de bandas C dos
cromossomos de beijupirá. Em destaques nas caixas cromossomos Ag‐NOR‐ rolamento (par de cromossomos 2). (c, d) análises de dual color FISH da distribuição de DNA genômico. TE Tol2 (verde) e DNAr 18S (vermelho); (d) Tol2 (vermelho) e DNAr 5S (verde) (d); (e, f) FISH com sondas Rex1 (e) e (f) Rex3. Os pares organizadores do nucléolos estão destacados nas caixas. Barra = 5 µm.
Capítulo 3
Figura 1. Gel de Agarose de Cot‐1 do Rachycentron canadum. (M) Marcador; (1) DNA
genômico; (2) DNA genômico depois de autoclavado por 5 minutos; (3) DNA Cot‐1 depois de tratamento enzimático.
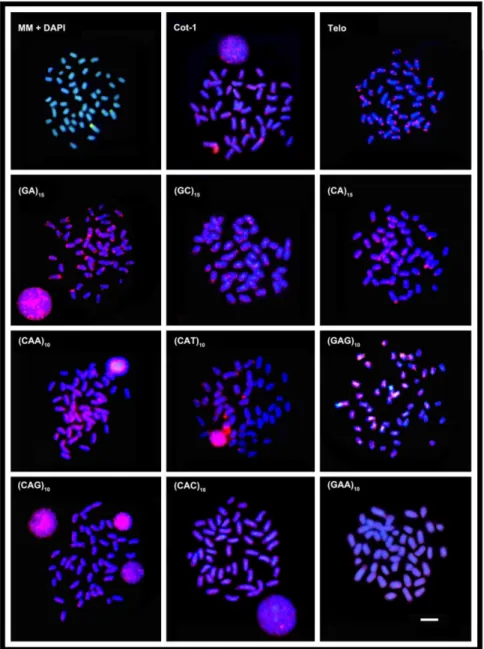
Figura 2. Metáfases de Rachycentron canadum coloração com Mitramicina/DAPI e
hibridização com Cot‐1, (TTAGG)n e nove sondas de microssatélites. Os cromossomos foram contracorados com DAPI (azul). Barra=5µm.
Figura 3. Ideograma dos cromossomos 2 e 3 de Rachycentron canadum. As paletas de
cores destaca a co‐localização de várias sequências repetitivas nos cromossomos. Relatado anteriormente sequências repetitivas, transcricionais e não‐transcricional (Jacobina et al., 2011; Costa et al., 2013; Costa et al., 2014) também foram
adicionados aos dados atuais. Capítulo 4 Figura 1. Cromossomos mitóticos metafásicos de espécies de Lutjanidae hibridizados com sondas DNAr 18S (vermelho), DNAr 5S (verde; coluna da esquerda), e DNAhis H3 (vermelho, coluna à direita). Cromossomos foram contracorados com DAPI (azul). Barra = 5µm.
Figura 2. Distribuição das famílias multigênicas 18S, 5S e DNAhis H3 ou sítios Ag‐RONs em relação aos agrupamentos filogenéticos de espécies de Lutjanidae e relógio molecular da divergências dos clados A, B e C (modificado de Gold et al. (2011), que
incluem as espécies ora analisadas. Os dados para as espécies com asteriscos foram obtidos de Rocha & Molina (2008); Nirchio et al. (2008).
A espécie Rachycentron canadum, conhecida popularmente como beijupirá ou cobia, é o
único representante da família Rachycentridae o qual vem sendo crescentemente utilizado na piscicultura marinha, em cultivos intensivos. Como características vantajosas, é de fácil adaptação, prolífica, possui crescimento precoce em cativeiro e elevado valor comercial. Já as espécies da família Lutjanidae (Lutjanus synagris, Lutjanus jocu, Lutjanus analis, Lutjanus alexandrei e Ocyurus chrysurus) representam um importante recurso pesqueiro em todas as áreas de sua ocorrência. No Brasil a exploração comercial dos Lutjanidae que se iniciou na década de 60 e nos anos 80 já demonstrava um declínio nos volumes de captura. Este fato aponta que os lutjanídeos devem possuir um manejo conservativo. Apesar dos potencias econômicos, pouco se conhece sobre as características genéticas e citogenéticas destas espécies, principalmente no que diz respeito a análise de DNAs repetitivos, os quais que representam a maior parte do genoma dos eucariotos e sendo responsaveis por importantes papeis evolutivos no genoma dos peixes. Dados citogenéticos vem crescentemente sendo empregados em estudos populacionais e com fins biotecnológicos em peixes. As analises citogenéticas foram realizadas utilizando métodos clássicos como coloração com Giemsa, bandamento C e Ag‐RONs, coloração com os fluorocromos base‐específicos (DAPI e MM) e mapeamento cromossômicos de sequências repetitivas dentre as quais, sequências teloméricas, transposons (Tol2), retrotransposons (Rex1 e Rex3), DNA repetitivos (Cot‐1 e microssatélites) e das regiões transcricionalmente ativas dos genes ribossomais 18S e 5S e histonas (H2BA e H3), através da hibridação in situ com sondas fluorescentes (FISH). Os padrões cromossômicos obtidos contribuíram para o conhecimento da organização das sequências repetitivas no genoma das espécies, bem como a diferenciação cariotípica. Padrões incomuns de expansão de sequências histônicas retratam a primeira ocorrência em peixes marinhos. Os dados obtidos fornecem subsídios para o conhecimento genético dos importantes recurso pesqueiros representados pelas espécies aqui analisadas, com vistas a auxiliar o desenvolvimento da piscicultura marinha.
Palavras‐chave: Piscicultura marinha; DNAs repetitivos; processos biotecnológicos; Lutjanidae; Rachycentridae
ABSTRACT
The Rachycentron canadum species, commonly known as beijupirá or cobia is the only
representative of Rachycentridae family which has been increasingly used in marine fish farming, in intensive cultivation. As advantageous features it has easy adaptation, prolific behavior, early growth in captivity and high commercial value. Additionally, specie of Lutjanidae family (Lutjanus synagris, Lutjanus jocu, Lutjanus analis, Lutjanus alexandrei and Ocyurus chrysurus) represents an important fisheries resource in all areas of its occurrence. In Brazil, the commercial exploitation of Lutjanidae which begun in the 60's and 80's, already has showed a decline in catch volumes. This fact suggests that the snappers must have a conservative management. Despite the economic potential, little is known about the genetic and cytogenetic characteristics of these species, especially with respect to repetitive DNA analysis, which represents the major part of the eukaryotes genome, playing important evolutionary roles in the fish genome. Cytogenetic data is increasingly being used in population studies and biotechnological purposes in fishes. The cytogenetical analyzes were performed using classical methods such as Giemsa staining, C‐banding and Ag‐NORs, fluorochromes base‐specific staining (DAPI and MM) and physical mapping of repetitive sequences among which, telomeric sequences, transposons (Tol2), retrotransposons (Rex1 and Rex3), repetitive DNA (microsatellites and Cot‐1) and transcriptionally active regions of the 18S and 5S ribosomal genes and histone (H3 and H2BA) by in situ hybridization with fluorescent probes (FISH). The chromosomal patterns obtained contributed to the organization of repetitive sequences in the genome of the species, as well as karyotypical differentiation. Unusual patterns of histone sequences expansion depict the first occurrence in marine fishes. The obtained data provided subsides to the genetic knowledge of the important fisheries resource represented by the species here analyzed, seeking the marine pisciculture improvement.
Key words: Marine Fish Farming, Repetitive DNAs, biotechnological processes, Lutjanidae, Rachycentridae
1. INTRODUÇÃO GERAL
A pesca é uma atividade milenar que passou por profundas modificações no século
XIX, uma vez que anteriormente a extração de organismos aquáticos era realizada em
pequenas quantidades, sem grandes impactos ambientais (Marrul‐Filho, 2001). As inovações
tecnológicas ocorridas após a Segunda Guerra Mundial provocaram uma rápida expansão da
indústria pesqueira contribuindo para um grande aumento na produção que passou de 4
para 70 milhões de toneladas (Paes, 2002). Entretanto, a partir de 1972, a pesca marítima
mundial começou a sinalizar queda na captura de pescado (Pauly et al., 2002). Desde 1990,
essa atividade estacionou em torno de 80 a 85 milhões de toneladas em que 30 milhões
desta foram, simplesmente, descartadas (Clucas, 1997; Dulvy et al., 2004).
Estatísticas da FAO (2014) têm demonstrado que a captura de espécies aquáticas
economicamente viáveis já atingiu a sua capacidade máxima de suporte, cerca de 100
milhões de toneladas por ano. Isso se deve, isso se deve ao esforço de pesca que vem
aumentando nos últimos anos para suprir a crescente demanda populacional por proteína
de origem animal, indicando uma iminente possibilidade do esgotamento destes estoques
pesqueiros em escala global.
O relatório da FAO (2014) ainda coloca que a produção de pescado global através
de capturas em 2012 foi de cerca de 91,3 milhões de toneladas, das quais aproximadamente
87,3% são de espécies marinhas com vum valor estimado de faturamento primário de 93,9
bilhões de dólares. Quando este numero é somado aos números da produção advinda da
aquicultura, obtém‐se um valor estimado de 158 milhões de toneladas de peixes produzidos
em 2012.
Ainda segundo a FAO (2010 e 2014), a produção mundial da pesca marinha atingiu
um pico de 86,3 milhões de toneladas em 1996 e depois diminuiu ligeiramente para 79,7
milhões de toneladas em 2012, com grandes flutuações interanuais. Em 2008, o Noroeste do
Pacífico teve a maior produção com 20,1 milhões de toneladas (25%) da pesca marinha
mundial, seguido pelo Sudeste do Pacífico, com uma captura total de 11,8 milhões de
toneladas (15%), Pacífico Centro‐Oeste com 11,1 milhões toneladas (14%) e do Atlântico
Nordeste, com 8,5 milhões de toneladas (11%).
A indústria pesqueira mudou a maneira de exploração da pesca em todo o mundo,
sustentável (Morato et al., 2006; Norse et al., 2012). O crescimento populacional e o
aumento no poder de compra fez com que a demanda mundial por pescado crescesse,
aumentando a escassez de peixes na região da plataforma continental e impulsionando a
pesca industrial oceânica pelágica (Watson & Pauly, 2001).
Visando reverter essa situação, o cultivo de organismos aquáticos constitui uma das
alternativas mais viáveis para a continuidade de fornecimento de alimentos para população
mundial. Entre 1987 e 1997 a produção global advinda da atividade aquícola duplicou em
produção e valores, assim como sua contribuição no abastecimento para demanda de peixes
em escala mundial, representando no inicio da década um quarto da quantidade de peixes
consumidos pela população (Cavalli & Hamilton, 2007; Cavalli et al., 2011).
O crescimento da aquicultura pode ser a solução para a preservação de espécies
nas quais seu potencial de exploração pela pesca já não permite um uso sustentável de seus
estoques selvagens (Naylor et al., 2000). Segundo dados da FAO (2010), a aquicultura produz
quase 50% dos peixes consumidos pela população, e esta porcentagem deverá aumentar
para atender à demanda crescente. A aquicultura mundial está concentrada em regiões
tropicais e subtropicais, com atualmente mais da metade do volume produzido proveniente
de águas marinhas e salobras em regiões costeiras e estuarinas, onde destacam‐se a
produção de camarões e peixes de alto valor comercial.
O potencial do Brasil para a piscicultura marinha é enorme, tendo em vista as suas
dimensões continentais, condições climáticas e extensa linha costeira, abrigando portanto
condições edafoclimáticas, hidrológicas e topográficas muito favoráveis. As práticas
aquícolas são bastante diversificadas, com técnicas que vão desde estruturas simples como
criações em nível familiar até iniciativas industriais de larga escala em gaiolas flutuantes no
mar aberto (offshore) (Cavalli et al., 2011).
As atividades aquícolas no Brasil iniciaram‐se na década de 30, intensificando‐se a
partir da década de 70. Já na década de 90 firmou‐se no contexto econômico brasileiro
quanto ao fornecimento de alimentos, época na qual a produção chegava à cerca 25.000
ton./ano, essa atividade vem se expandindo a cada ano no cenário nacional, estimando‐se
que a produção dos diversos setores ligados à atividade aquícola tenha ultrapassado
210.000 toneladas neste século (Sonoda, 2006). Paradoxalmente, a piscicultura marinha
Entretanto, o desenvolvimento da biotecnologia nas ultimas décadas tem auxiliado
a piscicultura moderna através das técnicas de manipulação cromossômicas ou de genes.
Por exemplo, marcadores moleculares vendo sendo utilizados de forma ampla em estudos
de variabilidade genética em espécies com potencial para projetos de piscicultura (Liu &
Cordes, 2004).
Analisando as espécies naturalmente encontradas no litoral brasileiro, Cavalli e
Hamilton (2007), concluíram que o Rachycentron canadum seria uma das espécie que
reuniria as melhores condições para ser cultivada comercialmente, considerando três
critérios de seleção (mercado, potencial de crescimento e disponibilidade de tecnologia de
cultivo).
1.1. Considerações sobre Rachycentron canadum
R. canadum, comumente conhecido como beijupirá, é o único representante da
família Rachycentridae, raramente forma cardumes, entretanto é uma espécie abundante e
adaptada a águas tropicais e subtropicais do Atlântico e do Pacífico, sendo portanto
encontrada naturalmente no litoral brasileiro (Shiau, 2007; van der Velde et al., 2010).
De hábito alimentar predatório, esta espécie inclui em sua dieta peixes e
crustáceos. Entretanto, estudos demonstram que o principal item alimentar encontrado em
espécimes capturadas no litoral pernambucano foram peixes ósseos, com pouquíssimos
crustáceos. Os mesmos estudos também revelam que a ocorrência e a alimentação do
beijupirá devem estar associadas à presença de recifes ao longo do litoral, os quais abrigam
espécies residentes que possuem deslocamento limitados (Meyer & Franks, 1996; Arendt et
al., 2001; Cavalli et al., 2011). É uma espécie de comportamento migratório e, por não
possuir bexiga natatória, apresenta hábito natatório ativo (Shaffer & Nakamura, 1989).
A reprodução do beijupirá ocorre preferencialmente na época da primavera e
verão. Estudos realizados nos Estados Unidos fornecem evidências de que a desova do R.
canadum ocorre de forma parcelada ao longo da temporada reprodutiva, a qual, no
hemisfério norte, se estenderia de abril a setembro (Lotz et al., 1996; Brown‐Peterson et al.,
2001), similar ao que ocorre no litoral de Pernambuco, onde a reprodução ocorre entre os
O potencial aquícola dessa espécie foi primeiramente proposto por Hassler e
Rainville (1975). No entanto, a primeira tentativa de criação do R. canadum provavelmente
ocorreu nos anos 1970, quando pesquisadores da Carolina do Norte, EUA, coletaram ovos na
natureza e conseguiram manter exemplares vivos no laboratório por 131 dias (Hassler &
Rainville, 1975; Cavalli et al., 2011). Em 1990, pesquisadores de Taiwan obtiveram a primeira
desova do beijupirá em cativeiro (Liao et al., 2004; Kaiser & Holt, 2005) e desde 1994,
desovas vêm sendo obtidas rotineiramente a partir de reprodutores produzidos em cativeiro
(Liao et al., 2004).
Examinando o desempenho reprodutivo de indivíduos selvagens de R. canadum,
capturados no litoral de Pernambuco, Peregrino Jr. (2014), analisou um plantel composto
por duas fêmeas (peso de 14 e 16 kg) e quatro machos (cerca de 10 kg), mantidos em
cativeiro em um tanque de 70 t, e observou que entre outubro de 2007 e junho de 2008
produziram 21 desovas espontâneas, sendo que dos 48,7 milhões de ovos produzidos, 49,3%
foram fertilizados. Em cada desova, as fêmeas liberaram de 1,0 a 4,5 milhões de ovos (média
de 2,5 milhões de ovos/desova), confirmando a facilidade de obtenção de desovas
espontâneas em cativeiro, corroborando com o observado em outros países, bem como a
constatação de que as desovas espontâneas em cativeiro se estendem além do observado
na natureza (Chang et al., 1999; Brown‐Peterson et al., 2001; Arnold et al., 2002; Gaumet et
al., 2007).
Os bons resultados obtidos proporcionaram um crescimento rápido na produção
comercial, assim como o desenvolvimento da tecnologia empregada na produção em massa
de jovens, fazendo com que a reprodução em massa fosse alcançada em 1997 (Liao et al.,
2004). Desta maneira, em 2004, a produção de beijupirá em Taiwan alcançou 4.268
toneladas, equivalendo a mais de US$ 20 milhões de dólares (Miao et al., 2009). O sistema
de cultivo, tanques‐rede (ou gaiolas) (Figura 1) adotado em Taiwan, permitiu uma produção
tanto para consumo interno quanto para a exportação, principalmente para o mercado
japonês (Liao & Leaño, 2007).
Figura 1. Fluxograma do processo produtivo em cativeiro de Rachycentron canadum.
Entretanto, no Brasil, apesar da relativa facilidade na obtenção de desovas em
cativeiro, a produção de alevinos em larga escala ainda é limitada. Nos poucos laboratórios
nacionais que trabalham com R. canadum, a criação de larvas é realizada intensivamente
sob condições controladas com o uso de sistemas de manejo de água semiestáticos. Neste
sistema as larvas são normalmente criadas em água marinha (salinidade de 35 ppm),
temperatura entre 26 e 29°C, fotoperíodo natural ou com 13 horas diárias de luz e aeração
constante a uma densidade de até 10 larvas/l (Cavalli & Hamilton, 2009; Cavalli et al., 2011).
Outras características importantes para a aquicultura também são observadas no R.
canadum, como uma relativa tolerância as mudanças de salinidade (Faulk & Holt, 2006),
resposta positiva à tratamento de doenças por vacinação (Lin et al., 2006), adaptabilidade ao
confinamento para o cultivo e aceitação de dietas comerciais extrusadas (Liao et al., 2004;
Liao & Leaño, 2007; Craig et al., 2006).
A produção aquícola mundial do R. canadum tem aumentado rapidamente e a
2010 (Gráfico 1). Enquanto isso, o volume de capturas por meio da pesca manteve‐se
estável, em torno de 10.000 tonelada/ano (FAO, 2014). Em termos de aquicultura, China e
Taiwan são os principais produtores mundiais da espécie segundo estatísticas da FAO (2014).
Outros países como o Brasil, Vietnã, Estados Unidos, Porto Rico, Belize, Bahamas, República
Dominicana, México, Filipinas, Japão, Indonésia, Tailândia, Irã, Martinica, Panamá e
Austrália, veem investindo na produção de R. canadum em cativeiro (Liao & Leaño, 2007;
Benetti et al., 2008; Cavalli & Hamilton, 2009; van der Velde et al., 2010; FAO 2007‐2014).
Gráfico 1. Dados globais na produção em cativeiro de Rachycentron canadum. Fonte: FAO
(2014).
Surpreendentemente, apesar da grande importância econômica apresentada pelo R.
canadum as informações genética sobre a espécie ainda são incipientes. Sendo assim, o
presente estudo teve por objetivo contribuir com o setor produtivo, através do conjunto de
dados citogenéticos, envolvendo métodos modernos e resolutivos. Procurou‐se a obtenção
de subsídios para a caracterização genética da espécie, com a identificação de marcadores
específicos que possam contribuir para programas de manejo e conservação.
1.2. Considerações sobre a família Lutjanidae
To
n
elad
a
(x1000)
Lutjanidae é um grupo composto por 17 gêneros e 120 espécies de peixes marinhos
(Gold et al., 2011), principalmente associados a recifes, um dos principais recursos para a
pesca no Nordeste (Resende et al., 2003). Representantes desta família encontram‐se
distribuídos nos oceanos Atlântico, Indico e Pacíficos, mares tropicais e subtropicais do
mundo, com poucas espécies em ambientes estuarinos (Nelson, 2006).
A família é dividida em quatro subfamílias. Três subfamílias menores são
representadas pelos Paradichthyinae, com dois gêneros monotípicos (Symphorus e
Symphorichthys), Etelinae, com cinco gêneros (Aphareus, Aprion, Etelis, Pristipomoides e
Rhandallichthys) e 19 espécies, e Apsilinae, com quatro gêneros (Apsilus, Lipocheilus,
Paracesio e Parapristipomoides) e 12 espécies. Lutjaninae é a maior subfamília, com três
gêneros monotípicos (Hoplopagrus, Ocyurus e Rhomboplites), os gêneros Macolor e Pinjalo
com duas espécies cada, e o gênero Lutjanus, com 64 espécies (Nelson, 2006).
No Brasil, são relatados quatro gêneros de duas subfamílias (Etelinae e Lutjaninae),
oito espécies do gênero Lutjanus e os gêneros monotípicos Ocyurus, Rhomboplites e Etelis,
difundidos ao longo de todo o litoral brasileiro (Menezes et al., 2003; Floeter et al., 2003;
Moura & Lindeman, 2007). Os Lutjanídeos são espécies de médio a grande porte capazes de
viver em grandes profundidades, e são popularmente conhecidos como pargos ou
vermelhos (Nelson, 2006). São grandes predadores, ocupando um papel importante no
ecossistema marinho (España, 2003). A maioria das espécies desta família possui um
desenvolvimento lento e um extenso período de vida (20 a 30 anos), e são altamente
vulneráveis à sobrepesca (Polovina & Ralston, 1987; Coleman et al., 2000).
As espécies desta família possuem um grande valor comercial, assim como,
importante recurso para a comunidades ribeirinhas que praticam a pesca de subsistência.
Dentre as espécies consideradas comercialmente importante, podemos citar Lutjanus
synagris, Lutjanus purpureus, Lutjanus analis, Lutjanus jocu e Ocyurus chrysurus, entre as
mais valorizadas desta família que são alvo da pesca comercial no litoral brasileiro,
principalmente na região nordeste do país (Resende et al., 2003).
Os dados sobre a pesca de Lutjanidae destacam a preocupação com a sobrepesca
dos estoques comercialmente explorados. A primeira estimativa sobre a captura de
Lutjanidae foi realizada seguindo o modelo de Schaefer (1954) considerando a produção e o
nordeste, no qual o resultados demonstraram uma produção máxima de 4.188 t/ano e
esforço ótimo de 6,0x105 pescadores‐hora (Coelho, 1974).
Outras estimativas de captura máximas e esforço ótimo de pesca foram realizadas
por diferentes estudos, considerando épocas e áreas de pesca distintas. Os maiores valores
de produção máxima e esforço de pesca ótimo foram estimados para a costa
Norte/Nordeste, incluindo dados do período entre 1967 e 1983, utilizando o método de
Schaefer (1954), estimando uma captura máxima de 6.791 t/ano com um esforço de pesca
ótimo de 184,3 x 104 anzóis/dia. No final da década de 80, Ivo e Sousa (1988), estimaram a
captura máxima sustentável de 5.937 t/ano e um esforço de pesca ótimo de 2.074 x 103
anzóis/dia, utilizando o método de Fox (1970), que leva em consideração o nível ótimo de
esforço de captura máxima à ser mantida sem afetar a sustentabilidade do estoque.
Atualmente, a pesca de Lutjanidae evoluiu para a estratificação e diversificação, ao
longo de toda a costa norte e nordeste do Brasil, se estendendo até o sul da Bahia,
contribuindo expressivamente nos desembarques controlados destas regiões. Portanto,
manejar e projetar os rendimentos destas distintas pescarias é o desafio mais relevante na
preservação da diversidade dos estoques e, sobretudo, da atividade pesqueira, que além de
sua relevância cultural, representa o principal sustento de muitas populações costeiras nas
regiões onde os peixes demersais ocorrem (Resende et al., 2003; IBAMA, 2001).
Devido ao seu alto valor econômico e importância alimentar, algumas espécies da
família Lutjanidae estão em sobrepesca, e projetos relacionados a aquicultura marinha
dessas espécies vem sendo desenvolvido o cultivo de juvens ajude a reconstruir as
populações selvagens em programas de melhoria do estoque pesqueiro, o cultivo vem sendo
usado de forma crescente nos Estados Unidos e em outros países como uma ferramenta de
manejo para superar as limitações de capturas (Lorenzen et al., 2010; Saillanti et al., 2013 ).
Apesar da importância ecológica e econômica das 120 espécies conhecidas de
Lutjanidae, estudos citogenéticos ainda são incipientes. Das espécies conhecidas poucas
foram cariotipadas, como por exemplo, Lutjanus argentimaculatus (2n=48a) (Raghunath &
Prasad, 1980), L. russelli (2n=48a, RONs 1 par) (Ueno & Ojima, 1992), Lutjanus analis
(2n=48a, RONs 1 par), L. alexandrei (2n=48a, RONs 1 par), L. synagris (2n=48, RONs 1 par), L.
1‐2 pares), Ocyurus chrysurus (2n=48a, RONs 1 par) (Rocha & Molina, 2008; Nirchio et al.,
2008) e Rhomboplites aurorubens (2n=48 (46a+2st), RONs 1 par) (Nirchio et al., 2009).
Entretanto, a maioria das espécies de Lutjanidae, se conhece apenas o número
cromossômico. Dados sobre a distribuição cromossômica, a composição da heterocromatina
constitutiva, e a distribuição de genes citomarcadores, úteis na investigação das relações
filogenéticas entre espécies dentro de uma família, ainda não foram bem analisados (Sola et
al., 2007).
1.3. DNA repetitivos
Compreendendo cerca de 50% do genoma dos eucariotos, os DNAs repetitivos estão
presentes na maioria dos organismos. O acúmulo dessas sequências repetitivas é
responsável pela variação no tamanho do genoma nos eucariotos (Walsh, 2001; Lopez‐
Flores & Garrido‐Ramos, 2012). Embora não se saiba ao certo a função dos DNAs repetitivos,
estudos sugerem o envolvimento destas sequências no processo de replicação de DNA,
recombinação, expressão gênica e na diferenciação de cromossomos sexuais (Biet et al.,
1999; Liu et al., 2001; Li et al., 2002; Parise‐Maltempi et al., 2007). Segundo Kidwell (2002),
sequências repetitivas também são responsáveis pelas variações cariotípicas observadas em
muito grupos, envolvidas nos rearranjos cromossômicos, tais como deleções, duplicações,
inversões e translocações recíprocas.
Entretanto, as sequências repetidas exercem seu principal papel nos centrômeros e
telômeros dos cromossomos dos eucariotos, desempenhando funções importantes na
manutenção e propagação do material genético, seu estudo proporciona uma melhor
esclarecimento sobre inúmeras questões relacionadas a origem e evolução dos
cromossomos sexuais e dos cromossomos B em peixes (Martins et al., 2004).
Essas regiões, assim como, as posições intersticiais dos cromossomos, são compostas
de heterocromatina, ricas em sequências repetidas que desempenham funções importantes
no comportamento dos cromossomos durante a divisão celular (Csink & Henikoff, 1998).
Entretanto, as sequências repetidas não é exclusividade das regiões heterocromáticas, mas
Estudos genéticos e citogenéticos utilizando a técnica de hibridização in situ com
fluorescência (FISH), vem demonstrando que sequências de DNAs repetidos são uma
poderosa ferramenta genética para estudos evolutivos, de transgenia, da origem dos
cromossomos sexuais e cromossomos supranumerários em peixes (Costa et al., 2013).
Adicionalmente, a aquicultura moderna vem utilizando marcadores de DNAs repetidos, com
o objetivo de melhorar traços importantes, como aumento do crescimento, resistência a
doenças, análise de características quantitativas, cruzamentos seletivos e acesso à
variabilidade genética das populações (Ferreira & Martins, 2008; Oleksiak, 2010; Beardmore
et al., 2001; Jankun et al., 2007).
Classificados de acordo com suas características, os DNAs repetidos compreendem as
famílias multigênicas, as sequências repetidas in tandem e as sequências dispersas no
genoma (Richard et al., 2008).
As famílias multigênicas são compostas por sequencias repetidas de DNAs
codificadores, a exemplo das histonas e dos RNAs ribossomais, formadas por eventos de
duplicação durante a evolução e que as diferenças observadas hoje entre esses genes são
produtos de acúmulos de mutações ocorridas ao longo do tempo. Contudo, são DNAs
repetidos que possuem um número considerável de pseudogenes, mostrando semelhanças
com os genes funcionais da mesma família, entretanto, perderam a capacidade de se
expressar devido as mutações adquiridas (Farah, 2007; Martins, 2007; Richard et al., 2008).
As sequências repetidas in tandem são classificadas em três tipos, levando em
consideração o tamanho da unidade de repetição. Primeiramente, são as sequências
satélites, que consiste em sequências altamente repetidas e organizadas em cadeias,
possuem unidades de repetição com aproximadamente de 100 a 300 pares de base (pb),
podendo variar de 1.000 a mais de 100.000 cópias, estão localizadas principalmente nas
regiões terminais e centroméricas dos cromossomos, constituindo o principal componente
da heterocromatina (Timberlake, 1978). Os DNAs satélites foram encontrados em todos os
genomas das espécies estudadas até o momento, podendo variar de 5% a 30% do tamanho
genômico total (Walsh, 2001).
O segundo tipo de sequências organizadas in tandem, consiste nos minissatélites ou
sequências com número variável de repetições (VNTR – variable number of tandem repeats).
genoma, assim como agrupados nos telômeros. Cada indivíduo apresenta um padrão
particular quanto à quantidade e comprimento das cadeias de minissátelites (Jeffreys et al.,
1985). Com a utilização dos minissatélites, foi possível a realização de estudos de
mapeamento genômico, genética de população, evolução, de seleção e melhoramento e de
ecologia e preservação de espécies, empregando a técnica de DNA fingerprint ou impressão
digital do DNA, com marcadores polimórficos capazes de distinguir o genoma de um
organismo do outro (Harris & Wright, 1995).
O último tipo de elementos repetidos in tandem são os microssatélites, repetições
curtas entre 1 a 5 pb, e pelo menos 30.000 loci diferentes de microssatélites estão presentes
em humanos. Estas sequências são polimórficas devido sua variações em número de
repetições e a alta taxa de mutação, podendo ser mono‐, di‐, tri‐ e tetranucleotídicos (Figura
2). Por apresentarem essas características os microssatélites são utilizados em investigações
criminais e paternidade (Ellegren, 2004; Chistiakov et al., 2006)
Figura 2. Representação esquemática da constituição dos microssatélites.
Os microssatélites estão localizados em sua maior proporção na região
heterocromática do genoma, entretanto, sequências na região eucromática já foram
relatados (Tóth et al., 2000). Assim, funções importantes vem sendo atribuídas aos
microssatélites como a participação na organização da cromatina, na replicação do DNA, na
recombinação e na expressão gênica (Epplen et al., 1996; Biet et al., 1999; Liu et al., 2001; Li
et al., 2002). Adicionalmente, em consequência da sua herança ser co‐dominante,
proporcionam a distinção de homozigotos e heterozigotos, empregando apenas a técnica de
PCR (Polymerase Chain Reaction) com uma limitada quantidade de DNA. Pelas características
que apresentam, estas sequências são excelentes marcadores nos estudos de mapeamento Mononucleotídeos GATCCGATAAAAAAAAAAAAAACGTCGAT
Dinucleotídeos GCTAGCACACACACACACACACATCGATG
Trinucleotídeos CATCGCATCATCATCATCATCATCATGCGT
genômico, genética de populações, epidemiologia molecular, patologia e conservação de
espécies (Chistiakov et al., 2006).
A ultima classe de sequências repetidas de DNA, são as sequências dispersas no
genoma, compreendendo dois tipos de elementos, os transposons e retrotransposons. São
considerados os principais representantes do genoma dos eucariotos, e devido a grande
diversidade de elementos de transposição existente nos diferentes genomas e distintas
sequências nucleotídicas encontradas, foi necessário criar uma classificação que facilitasse
os estudos destas sequências, de acordo com sua organização estrutural e mecanismo de
transposição (Feschotte, 2004; Martins, 2007).
O primeiro tipo são os retrotransposons ou elementos de classe I que transpõem via
transcrição reversa do seu RNA, e o segundo tipo são os transposons ou elementos de classe
II que se movimentam pelo genoma através de cópias de DNA. Diversas famílias de
elementos transponíveis já foram identificados, por exemplo, TC1/mariner em C. elegans e
Drosophila, hobo em Drosophila e Helitrons em Arabdopsis thaliana e C. elegans (Volff et al.,
1999; Volff, 2005; Martins, 2007).
1.4. Funções dos elementos transponíveis na evolução do genoma
Compondo grande parte do genoma de eucariotos e procariotos, os elementos
transponíveis são conhecidos por mudarem de local dentro do genoma, um processo
conhecido como transposição, catalisado por suas próprias enzimas, a transposase. Através
desse movimento de excisão e inserção, a adição de cópias em outros locais do genoma
pode causar mutações e possíveis mudanças fenotípicas, e essas mutações podem contribuir
com o aumento da diversidade existente na natureza, impulsionando a força evolutiva
(Böhne et al., 2008).
Adicionalmente, os elementos transponíveis exercem um papel fundamental na
estrutura e organização dos cromossomos podendo induzir rearranjos cromossômicos ou
atuando na prevenção de perdas teloméricas, perdendo o estereotipo de “parasita
genético” presente no genoma (Charlesworth et al., 2001; Böhne et al., 2008).
Em peixes, alvo de estudo do presente trabalho, uma característica importante do seu
transposons como retrotransposons (Volff et al, 2003), muitos deles também presentes no
genoma de invertebrados, provavelmente fruto de transferência horizontal elementos. No
peixe medaka 7% do seu genoma é composto por elementos transponíveis, Takifugu
rubripes e Tetraodon nigroviridis, contem cerca de 3 a 4% do seu DNA constituído por
transposons (Aparicio et al., 2002; Jaillon et al., 2004; Kasahara et al., 2007).
Embora a diversidade de elementos transponíveis em peixes seja alta o número de
cópias de transposons é aparentemente mais baixo do que em humanos e camundongo.
Muitas classes de retrotransposons foram perdidas na linhagem dos tetrápodes e mantidas
em teleósteos (Böhne et al., 2008). Os retrotransposons encontrados em peixes estão
extremamente compartimentalizados e aglomerados nas regiões heterocromáticas dos
cromossomos, sugerindo uma pressão seletiva contra a inserção destes elementos em
regiões que contenham genes ativos, evitando causar mutações e rearranjos desfavoráveis.
Entretanto, estudos recentes com peixes mostram que esses elementos também se
encontram dispersos nos cromossomos, corroborando com o que também é observado em
mamíferos. (Volff et al., 2003; Ferreira et al 2011; Costa et al., 2013).
Compreender a dinâmica e a evolução dos elementos transponíveis é algo de extrema
importância, uma vez que vem encontrando aplicações em técnicas de biotecnologia como a
transgenia (Davidson et al., 2003), permitindo a inserção de genes no DNA de outros
organismos usando a sua propriedade de inserção e excisão (Miskey et al. 2005; Peng et al.,
2010; Valenzano et al., 2011). Estudos de resistência a doenças ou para obtenção de
biofármacos também vem utilizando elementos transponíveis (Rocha et al., 2003).
1.4.1. Retrotransposons
Encontrados tanto em eucariotos quanto em procariotos, constituem
aproximadamente de 2% do genoma de Drosophila e mais de 40% em plantas, estando
ainda presente nos mais diversos organismos como leveduras, moscas e mamíferos. São os
principais responsáveis pelo aumento ou diminuição do tamanho dos genomas dos
organismos (Feshotte & Prithman, 2007). Eles podem ser classificados em duas categorias,
dependendo da estrutura nucleotídica que flanqueia a sequência:
a. Non‐LTR retrotransposons (non‐Long Terminal Repeat): são os retrotransposons ou retroposons. Não possuem repetições terminais longas (non‐LTR) e consistem em dois tipos,
(1) os SINEs (Short Interspersed Nuclear Elements), que não codificam as proteínas
necessárias para a transcrição reversa e (2) os LINES (Long Interspersed Nuclear Elements)
capazes de codificar as proteínas necessárias para a transcrição reversa (Volff et al., 2003;
Volff, 2005; Richard et al., 2008).
Com sua estrutura e modo de dispersão no genoma, os SINEs formam um classe de
DNA diferenciado de outros DNAs repetitivos, não apresentando sequências para a
produção dos elementos necessários a sua própria retrotransposição, sugerindo a utilização
da maquinaria de transposição de elementos do tipo LINEs (Smit, 1996). São encontrados
em eucariotos distintos como plantas e mamíferos (Ohshima et al., 1993). Correspondendo
cerca de 21% do genoma humanos, os elementos LINEs são encontrados em uma variedade
de organismos incluindo protistas, plantas, insetos, moluscos e vertebrados (Lander et al.,
2001).
b. LTR retrotransposons (Long Terminal Repeats): são elementos estruturalmente semelhantes aos retrovírus, de diferente tamanhos e sequências de nucleotídeos, possuindo
a mesma estrutura básica: uma região central codificante ORF (Open Reading Frames)
flanqueada por longas repetições terminais, sendo em geral limitadas por repetições curtas
invertidas, como encontradas em outros tipos de elementos transponíveis. Esta região
codificante possui um pequeno número de genes, homólogos aos genes gag e pol
encontrado em nos retrovírus, capazes de codificar, respectivamente, uma proteína
estrutural da capsula do vírus e uma transcriptase reversa/integrasse. Esses dois genes tem
papeis importante no processo de transposição (Böhne et al., 2008; Richard et al., 2008). As
três principais famílias de retrotransposons LTR descritas em vertebrados são: Ty1/copia,
Ty3/gypys e família BEL (Eickbush & Malik, 2002).
Figura 3. Representação de alguns elementos transponíveis (DNA Transposons e Retrotransposons (LTR e não‐LTR). Figura adaptada de Thomas et al. (2010).
1.4.2. Transposons
A segunda classe dos elementos repetidos dispersos, são os transposons de DNA, um
componente importante do genoma, contribuindo para a alta taxa de mutação. São
divididos em subclasses ou famílias de acordo com similaridade entre as suas sequências
(Kidwell, 2002; Feshotte & Prithman, 2007; Böhne et al., 2008).
Dependendo da família dos transposons e da espécie analisada, o número de cópias
por genoma pode variar de 10 a milhares (Tafalla et al., 2006), podendo ser encontrados em
diferentes eucariotos, sugerindo que esses elementos divergiram cedo durante a evolução
do genoma, antes mesmo da divergência ocorrida entre as linhagens dos eucariotos
(Feschotte, 2004). A habilidade de dispersão no genoma é relacionada ao mecanismo de
transposição que varia entre os elementos.
O mecanismos mais conhecido de transposição em transposons é o “cut and paste”,
em aumento no número de cópias. Adicionalmente, o elemento excisado nem sempre é
reinserido no genoma, o que pode levar à perda da cópia. Entretanto, a mobilização pode
resultar em um aumento no número de cópias dos transposons (transposição replicativa)
em duas situações distintas. A primeira, durante a replicação do cromossomo, quando a
sequência se mobiliza de uma cromátide já sintetizada para um local ainda não replicado. E
o segundo, quando o gap originado pela mobilização de uma cópia é “reconstruído” pela
maquinaria de reparo de DNA do genoma hospedeiro, seja a partir do cromossomo
homólogo que também possui o transposon, por recombinação de homólogos, ou durante a
fase S do ciclo celular, a partir da cromátide irmã (Kaufman et al., 1992; Miskey et al., 2005;
Feschotte & Pritham, 2007).
Os transposons podem ser classificados em três subclasses: (a) os que se movimentam
pelo mecanismo de “cut and paste”, são os transposons propriamente ditos e são
conhecidas dez famílias para tais elementos: TC1/mariner, haT, elemento P,
MuDR/Fokdback, Cacta, PiggyBac, Pif/Harbinger, Merlin, Transib e Banshee; (b) aqueles que
utilizam provavelmente um mecanismo de replicação por círculo rolante, Helitrons; e (c)
Mavericks, cujo mecanismo de transposição não está ainda bem compreendido. Todos esses
transposons são encontrados nos eucariotos, assim como nos procariotos (Feshotte &
Pritheman, 2007).
Esses elementos podem causar mutações e afetar a expressão ou alterar a estrutura
de genes, durante o seu movimento de transposição, o que pode levar a uma perda de
função dos genes no genoma do organismos (Kappitonov & Jurka, 2007). Entretanto, esses
elementos exercem a funções estruturais e funcionais importantes na evolução genômica
2. OBJETIVO GERAL
Diante do que foi apresentado, o presente estudo teve por objetivo a obtenção de
distintos marcadores cromossômicos em espécies das famílias Rachycentridae e Lutjanidae,
contribuindo assim para o conhecimento da organização genômica dessas espécies, suas
tendências evolutivas, com vistas igualmente a aplicações futuras voltadas ao campo da
3. OBJETIVOS ESPECÍFICOS
Diante das informações apresentadas o presente trabalho teve como objetivos
específicos:
• Investigar a distribuição de DNAr e DNAhis nos cromossomos de R. canadum
utilizando a hibridação in situ fluorescente (FISH) com sondas de DNAr 5S, 18S e
DNAhis H2B‐H2A e H3, com fim de estabelecer a associação entre essas
sequências;
• Mapear sequências de DNAr 5S, 18S e DNAhis H3 em Lutjanus analis, L. jocu, L.
alexandrei, L. synagris e Ocyurus chrysurus a fim de identificar marcadores
citogenéticos e inferências filogenéticas entre as espécies;
• Mapear sequências repetitivas de DNA transponíveis (Tol2) e retrotransponíveis
(Rex1 e Rex3) nos genoma cromossômico de R. canadum, com o intuito de
evidenciar a distribuição e a associação dessas sequências com as regiões
heterocromáticas;
• Averiguar a composição heterocromática nas quatro espécie de Lutjanus e em O.
chrysuru por meio de mapeamento cromossômico de sequências repetidas Rex1
e Rex3;
• Analisar a organização e a associação das porções heterocromáticas com DNA
repetitivos nos cromossomos de R. canadum, por mapeamento cromossômico
a
b
c
e
d
Figura 5. Espécies da família Lutjanidae, (a) Lutjanus analis; (b) Lutjanus jocu; (c) Ocyurus chrysurus; (d) Lutjanus synagris e (e) Lutjanus alexandrei. Barra = 10 cm. Fonte: LGRM/UFRN
4. MATERIAIS E MÉTODOS
4.1. Materiais
Espécimes de Rachycentron canadum (n=40) (Figura 4) foram obtidos a partir de
criadores, em Porto de Galinhas, no estado de Pernambuco. Já espécies da família
Lutjanidae, Lutjanus synagris (n=10), Lutjanus analis (n=8), Lutjanus jocu (n=8), Lutjanus
alexandrei (n=10) e Ocyurus chrysurus (n=5) (Figura 5) foram coletados no litoral do Rio
Grande do Norte, Carnaubinha (5°12'49.53"S; 35°26'2.58"O) e Barra do Rio (5°45'13.21"S;
35°11'48.05"O). Os espécimes foram selecionados, mediantes suas condições físicas,
acondicionadas em sacos plásticos com adição de oxigênio puro, ate a sua chegada e
acomodação em tanques, nas dependências do Laboratório de Genética de Recursos
Marinhos da Universidade Federal do Rio Grande do Norte para posterior desenvolvimento
das práticas citogenéticas.
Figura 4. Rachycentron canadum (Rachycentridae). A imagem não está em
4.2. Métodos
4.2.1. Preparações de cromossomos e células mitóticas
Após estimulação da divisão celular por meio da inoculação de agentes mitogênicos
nos indivíduos por 24‐48 horas (Molina et al., 2010), os animais foram eutanasiados
utilizando óleo de cravo, para retirada do tecido renal adequados às análises citogenéticas.
Fragmentos de tecido renal foram imersos em meio de cultura RPMI 1640 de
acordo com a técnica in vitro preconizada por Gold et al. (1990), onde sucintamente, foram
dissociados em 10 ml de meio de cultura RPMI 1640 através de aspirações com seringas de
vidro de 10 ml até obter um mistura homogênea, onde foi adicionado aproximadamente 125
µl de colchicina 0,025%, deixando agir por 28 minutos em temperatura ambiente.
Posteriormente, o material foi centrifugado por 10 minutos à 900 rpm. Após descarte do
sobrenadante, foi adicionado 10 ml da solução hipotônica de KCl à 0,075M, que após ser
homogeneizada agiu por 30 minutos em temperatura ambiente. A suspensão celular foi pré‐
fixada com 125 µl de fixador de Carnoy [Metanol e Ácido acético (3:1)] recém–preparado e
mantido à ‐20oC. Após homogeneização, a suspensão foi centrifugada por 10 minutos. O
processo de fixação do material foi repetido por três vezes com 6 ml de fixador Carnoy à ‐
20˚C antes das suspensões celulares serem estocadas à ‐20oC em tubos Eppendorf de 2,5 ml.
4.2.2. Bandamento cromossômicos
A partir da suspensão celular obtida pelos procedimentos supracitados, foi realizada
a caracterização cariotípica dos espécimes com uso de coloração convencional. Após
preparação de lâminas coradas com Giemsa 5% em tampão fosfato (pH 6,8), as metáfases
foram selecionadas para definir o número diploide e confecção do cariótipos dos animais
analisados. Os cromossomos foram classificados morfologicamente de acordo com a
literatura e organizados no cariótipo em ordem decrescente de tamanho conforme proposto
4.2.2.1. Detecção de heterocromatina constitutiva
A análise das regiões heterocromáticas foi realizada através do bandamento‐C,
segundo Sumner (1972). Foram utilizadas lâminas com material envelhecido em estufa a
37°C por no mínimo 3 dias, após esse período a lâmina foi imersa em HCl 0,2 N por 14
minutos em temperatura ambiente, sendo lavada com água destilada e seca.
Posteriormente a lâmina foi mergulhada em uma solução a 5% de Ba(OH)2.8H2O à 42°C por
cerca de 1 minuto e 45 segundos sendo rapidamente lavadas em HCl 0,1N e posteriormente
água destilada. Após secar ao ar, a lâmina foi então imersa em solução salina 2xSSC à 60°C
em estufa por 40 minutos. Para análise o material foi corado com Giemsa 5% por 8 minutos.
4.2.2.2. Fluorocromos GC ‐ e AT‐ específicos (MM e DAPI)
Fluorocromos base‐específicos foram utilizados para detectar regiões ricas em
pares de bases, como a mitramicina (MM) para pares de bases GC e DAPI para regiões ricas
em pares de bases AT, de acordo com Schweizer (1976). As lâminas com suspensão celular
foram coradas com 30μl de MM 0,5 mg/ml cobertas com lamínulas e depositadas em
câmara úmida, no escuro, sendo lavadas com água destilada após 30 a 60 minutos, coradas
com 30 μl de DAPI 0,3µg/ml, cobertas por lamínulas e colocada em câmara úmida, no
escuro, por 15 minutos. Posteriormente as lâminas foram lavadas com água destilada e
montadas com tampão glicerol‐Mcllvaine pH 7,0 (1:1) coberto com lamínula e selado com
esmalte incolor. As lâminas foram então guardadas à 4 ˚C em câmara escura e analisadas
após 3 dias com fotomicroscópio de epifluorescência (OlympusTM BX51) com filtros
apropriados, em aumento de 1.000x. As preparações foram capturadas pelo sistema digital
Olympus DP73 com uso do software CellSens Standard 1.7 Full (Olympus Optical Co. Ltd.).
4.2.2.3. Obtenção das sondas de DNA para FISH por PCR
Sondas de DNAr 5S e DNAr 18S foram obtidas por PCR a partir do DNA nuclear de R.
e B 5’‐CAG GCT GGT ATG GCC GTA AGC‐3’ (Pendás et al., 1994), NS1 5’‐GTA GTC ATA TGC
TTG TCT C‐3’ e NS8 5’‐TCC GCA GGT TCA CCT ACG GA‐3’ (White et al., 1990),
respectivamente. A primeira sonda continha uma cópia de DNAr 5S de 200pb e a segunda de
DNAr 18S de 1400pb. A sonda de DNAr 5S foi marcada com biotina‐14‐dATP e a sonda de
DNAr 18S com digoxigenina‐11‐dUTP, ambas por nick translation, seguindo as
recomendações do fabricante (Roche, Mannheim, Alemanha).
As sondas teloméricas foram obtidas utilizando o primer (TTAGGG)n, amplificado
segundo Ijdo et al. (1991) e marcadas usando DIG‐Nick Translation Mix seguindo a
recomendações do fabricante (Roche, Mannheim, Alemanha).
Sondas do DNA transponível (Tol2) foi obtido por PCR a partir do DNA nuclear de R.
canadum, utilizando os primers Tol2 4 F 5’‐ ATA GCT GAA GCT GCT CTG ATC ‐ 3’ e Tol2 4 R 5’‐
CTC AAT ATG CTT CCT TAG G ‐ 3’, Tol2 5 F 5’‐ CTG CTC TGA TCA TGA AAC AG ‐ 3’ e Tol2 5 5’‐
CTT CCT TAG GTT TGA TGG CG ‐3’ (Kawakami & Shima 1999). Ambas as sondas continham
cópia de DNA Tol2 de 200pb. A sonda amplifica com os primes Tol2 4 foi marcada com
biotina‐14‐dATP e a sonda com Tol2 5 com digoxigenina‐11‐dUTP, ambas por nick
translation, seguindo as recomendações do fabricante (Roche, Mannheim, Alemanha).
A amplificação do DNA retrotransponível (Rex1 e Rex3), a partir do DNA genômico
de R. canadum e O. chrysurus, foi realizada utilizando os primers Rex1 F 5’‐ TTC TTC AGT
GCC TTC AAC ACC ‐ 3’ e Rex1 R 5’‐ TCC CTC AGC AGA AAG AGT CTG CTC ‐ 3’, desenhados
para amplificar segmentos Rex1 correspondentes aos domínios de codificação 3‐7 do gene
da transcriptase reversa (RT), e os primers Rex3 F 5’‐ CGG TGA YAA AGG GCA GCC CTG ‐ 3’ e
Rex3 R 5’‐ TGG CAG ACN GGG GTG GTG GT ‐ 3’, para Rex3, utilizados para amplificar os
domínios de codificação 1, 2, 2A, A e B do gene da RT, ambos desenhados a partir dos genes
de Xiphophorus (Volff et al., 1999; Volff et al., 2000) e amplificados segundo Valente et al.,
(2011). As sondas de retrotransposons foram marcadas utilizando nick translation, com
biotina‐14‐dATP para Rex1 e digoxigenina‐11‐dUTP para Rex3, seguindo a recomendações
do fabricante (Roche, Mannheim, Alemanha).
A amplificação dos genes das histonas, a partir do DNA genômico de R. canadum e
O. chrysurus foi realizada utilizando os primers H2BAF 5’‐CCC GAG ATG TGA TGG TAG A‐3’ e
H2BAR 5’‐AGT ACA GCC TGG ATG TTT GGT AA, para H2A‐H2B, e os primers H3F 5’‐ATG GCT