A
IMPORTÂNCIA DA DIAGONAL ÁRIDA(C
AATINGA,
C
ERRADO EC
HACO)
NO ISOLAMENTO DE POPULAÇÕES DE ESPÉCIES DEE
UGLOSSINA(I
NSECTA:
H
YMENOPTERA:
A
PIDAE)
NOS DOMÍNIOS MORFOCLIMÁTICOS DAA
MAZÔNIA E DAM
ATAA
TLÂNTICA–
UM ESTUDO FILOGEOGRÁFICOBelo Horizonte
Carolina Ferreira Cardoso
A
IMPORTÂNCIA DA DIAGONAL ÁRIDA(C
AATINGA,
C
ERRADO EC
HACO)
NO ISOLAMENTO DE POPULAÇÕES DE ESPÉCIES DEE
UGLOSSINA(I
NSECTA:
H
YMENOPTERA:
A
PIDAE)
NOS DOMÍNIOS MORFOCLIMÁTICOS DAA
MAZÔNIA E DAM
ATAA
TLÂNTICA–
UM ESTUDO FILOGEOGRÁFICOTese
apresentada
ao
Colegiado
do
Programa de Pós-Graduação em Ecologia,
Conservação e Manejo de Vida Silvestre do
Instituto
de
Ciências
Biológicas
da
Universidade Federal de Minas Gerais,
como requisito parcial para a obtenção do
grau de Doutora em Ecologia.
Orientador: Prof. Dr. Fernando Amaral da
Silveira
Co-orientador: Prof. Dr. Fabrício Rodrigues
dos Santos
Belo Horizonte
“What makes things baffling is their degree of complexity, not their sheer size… a
star is simpler than an insect.”
“
O que torna as coisas desconcertantes é o seu grau de complexidade e não seu
tamanho... uma estrela é mais simples do que um inseto
.”
—
Martin Rees
Exploring Our Universe and Others
A
GRADECIMENTOSAgradeço ao Fernando por me aceitar como aluna e pela orientação desde a
minha iniciação científica! Ferdinando, obrigada pelas risadas, pelo rock progressivo,
pelo companheirismo, por tantos ensinamentos sobre abelhas e pela sua amizade!
Muito obrigada aos meus pais, Vera e José Celso, por todo amor e carinho e por
me incentivarem a estudar sempre! Pai, o seu interesse pelas plantas (em especial pelas
orquídeas!) e pela vida me contagiaram desde que eu era “pequetita”
! Agradeço
imensamente aos meus sogros queridos, Lola e Fuad (in memorium), por todo carinho
que sempre me deram! Vocês são muito especiais para mim! Obrigada também aos
meus queridos irmãos Renata, Ricardo, Cristiane e Igor, aos meus cunhados e,
especialmente, aos meus sobrinhos Cathatina, João Pedro, Clara, Bernardo, Ana, Lívia e
Tomás por todo carinho e incentivo!
Agradeço ao Canadian Centre for DNA Barcoding (Guelph, Canadá),
especialmente à Dra. Julie K. Stahlhut, pelo sequenciamento de grande parte das
amostras deste projeto.
Muito obrigada ao Prof. Laurence Packer (York University) e toda equipe do
Packer Lab, em especial à Jennifer Albert que foi super prestativa e me ajudou muito
durante o estágio sanduíche. Agradeço ainda aos Profs. Dr. Bridget Stutchbury, Dr. Joel
Shore e Dr. Amro Zayed, da mesma universidade, por disponibilizar material e
equipamentos de seus laboratórios durante o estágio sanduíche. Eu não poderia deixar
de agradecer aos meus amigos Sheila Dumesh, Anne-Isabelle Gravel, Cory S. Sheffield,
Sheila R. Colla, Alana Taylor, Hien T. Ngo, Scott MacIvor, Natalia Veiga, Nick de
Silva pelo apoio e grande amizade durante os seis meses no Canadá!
Muito obrigada a minha amiga Carmenzita (Carmen Yurrita Obliols) pela
confecção dos mapas apresentados nesta tese e pelo apoio e amizade durante o estágio
no Canadá.
Agradeço ao Prof. Dr. André Nemésio (UFU) e aos curadores do Royal Ontario
Museum (Toronto, Canadá) por forneceram valiosos espécimes da Mata Atlântica e da
América Central, respectivamente. Sou muito grata ao Dr. Jerry Rozen e Dr. John
Ascher do American Museum of Natural History de Nova Iorque pela receptividade e
permissão de acesso à grandiosa e importantíssima coleção do AMNH e pelo
fornecimento de amostras incluídas neste estudo. Agradeço também à amiga Margarita
López-Uribe (Cornell University) por fornecer amostras para este estudo e por tantas
dicas sobre PCRs, marcadores nucleares e análises feitas neste estudo. Muito obrigada à
Rose (Roselaini M. C. Silveira) e ao Maverick (Roderic B. Martines) da Biobrasilis por
várias amostras da Amazônia; ao Dr. David Roubik pelas amostras da Guiana Francesa;
ao Prof. Dr. Gabriel Melo (UFPR) pelas amostras de Euglossa bidentata e pela
re-identificação de espécimes do subgênero Euglossa da Mata Atlântica; à Dra. Márcia
Maués e suas orientadas, Mariana Taniguchi e Talyanne Moura, da Embrapa Amazônia
Oriental por amostras do Pará; à Michelle Guimarães (UFCE) pelas amostras de várias
localidades do Ceará e ao José Eustáquio S. Junior pelas amostras de Rondônia.
Agradeço também ao pessoal da APA Rio Vermelho-Humboldt (CEPA
Rudengas) e do CEPA Vila da Glória pelo apoio à nossa expedição de coleta em áreas
de Mata Atlântica de Santa Catarina. Infelizmente, quase nenhuma Euglossina
apareceu... Obrigada ao pessoal do PARNA Ubajara, principalmente a Sra. Nagila M. P.
Gomes e o Sr. Francisco Humberto Bezerra, pelo apoio durante as coletas realizadas
neste parque belíssimo situado nos Brejos de Altitude do Ceará.
sempre disposto a ajudar no que fosse preciso! Agradeço imensamente ao pessoal do
LBEM, especialmente ao José Eustáquio Santos-Junior, à Bárbara R. N. Chaves, à
Gisele P. M. Dantas, à Daniela Lacerda, ao Augusto, ao José, à Larissa e ao Anderson
V. Chaves pelo apoio nos trabalhos de biologia molecular e análises de dados. Obrigada
pela paciência, viu gente!
Obrigada a todo pessoal do Laboratório de Sistemática e Ecologia de Abelhas
pelo apoio e amizade durante todos estes anos! Valeu Vivi’s, Alezita, Rudolf, Rafildo,
Priti, Amandita, Philip’s, Déborah, Igor, Paulita, Maverick, Rose, Rose (do
Zé), Zé,
Ana (que entrou agora e é um doce de pessoa!) e a todos os outros amigos que passaram
por este laboratório e que deixam muita saudade!
Agradeço ao Prof. Dr. João Aguiar Nogueira Batista por permitir a utilização de
equipamentos do Laboratório de Sistemática Vegetal (UFMG), onde fotografias dos
géis de agarose puderam ser feitas.
Muito obrigada ao Prof. Dr. Adalberto José dos Santos do Laboratório de
Aracnologia (UFMG) e ao Prof. Dr. Paulo Christiano de Anchieta Garcia do
Laboratório de Herpetologia (UFMG) por permitir o uso de equipamentos, onde com a
ajuda imprescindível da Priti (Priscila Guimarães Dias), pude tirar fotografias das
abelhas apresentadas nesta tese. Obrigada ao Rudolf (Rodolfo Cesar C. Arantes) pela
doação dos DVDs que foram utilizados para gravar os arquivos das fotos!
Deixo aqui registrado o meu agradecimento à Universidade Federal de Minas
Gerais pela contribuição à minha formação acadêmica, em especial ao Prof. Rogério
Parentoni, que me recebeu de forma tão acolhedora no ex-Laboratório de Ecologia e
Comportamento de Insetos, onde tive a oportunidade de me iniciar no estudo dos
insetos.
Muito obrigada aos Prof. Dr. Eduardo A. B. Almeida (UsP/FFCLRP), Prof. Dr.
Eduardo Leite Borba (UFABC) e Prof. Dr. Gustavo C. S. Kuhn (UFMG) pelas críticas e
sugestões ao projeto desta tese.
Os professores Dr. Almir Pepato (UFMG) e Dr. Gabriel M. Yazbeck (UFSJ)
deram valiosas contribuições para as análises moleculares deste projeto.
Obrigada ao Rafildo (Rafael Ferrari) pela checagem dos caracteres morfológicos
dos espécimes após as análises moleculares.
Obrigada ao pessoal da FUNDEP, principalmente à Carina Souza, pelo apoio
durante a vigência do projeto da FAPEMIG.
Deixo meus agradecimentos ao ICMBio-SISBIO pela licença de coleta e
transporte concedida (N° 26425-1 e 26425-2).
Agradeço imensamente à FAPEMIG (Projeto APQ 2006-10) e ao PRONEX
História Natural (2009) pelo financiamento deste projeto. Agradeço ainda à FAPEMIG
pela bolsa de doutorado e ao Canadian Department of Foreign Affairs and International
Trade (Emerging Leaders in the Americas Program) pela bolsa sanduíche de seis meses
durante o estágio na York University (Toronto, Canadá).
R
ESUMOA subtribo Euglossina (Hymenoptera: Apidae), de ampla distribuição na região Neotropical, possui aproximadamente 220 espécies e é mais diversificada nos ecossistemas florestais. Este estudo avalia a estrutura filogeográfica de espécies/pares-de-espécies irmãs de abelhas Euglossina, que ocorrem tanto na Amazônia (AmF), quanto na Mata Atlântica (AtF). As relações históricas interespecíficas e interpopulacionais são avaliadas aqui por meio da utilização de parte do gene mitocondrial para a subunidade I da citocromo c oxidase (COI) como marcador molecular. É avaliada a hipótese de que a separação da AmF e AtF pela diagonal xérica constituída pela Caatinga, Cerrado e Chaco, entre estes domínios florestais tem atuado no isolamento das populações de espécies de Euglossina, produzindo um conjunto de pares de espécies crípticas. Machos de Euglossa (Glossurella) crassipunctata [n = 98; 579 pares de base (pb)] e de outros quatro pares de espécies com grandes afinidades são incluídos neste estudo: (i) Euglossa (Euglossa) bidentata (n = 10; AmF) e E. (Euglossa) marianae (n = 45; AtF; 532 pb); (ii) Euglossa (Euglossa) mixta (n = 34; AmF) e E. (Euglossa) calycina (n = 17; AtF; 533 pb); (iii) Euglossa (Euglossa) iopyrrha (n = 1; AmF) e E. (Euglossa) botocuda (n = 39; AtF; 503 pb); e (iv) Eulaema (Eulaema) bombiformis (n = 21; AmF e América Central) e E.
(Eulaema) niveofasciata (n = 52; AtF; 558 pb). Através de inferência Bayesiana e redes de
haplótipos é constatado que a diagonal árida da América do Sul tem atuado no isolamento das populações de E. crassipunctata, o que suporta a decisão recente de descrever as populações da AtF como E. clausi, e aquelas da AmF como E. moratoi, restringindo-se o nome E.
crassipunctata apenas às populações da América Central. A sequência de eventos de vicariância
aqui inferida para o par E. calycina/E. mixta é coincidente com aquela previamente obtida para grupos de espécies de Geotrigona e Paratrigona. Pela estimativa prévia da idade mínima de E.
mixta (5 Ma), é possível que a diagonal árida (em uma de suas várias ocorrências, a partir do
final do Mioceno) seja o fator causal da separação entre as populações AtF e AmF neste grupo. Para o grupo crassipunctata, verifica-se o inverso, em concordância com os eventos de vicariância entre grupos de espécies de Partamona e alguns de Geotrigona. A estimativa de idade mínima do grupo crassipunctata (≈ 14,7 Ma) sugere que a separação entre a América Central e AmF seja contemporânea à transgressão do mar Maracaibo (Mioceno médio) e que, a separação entre a AmF e a AtF possivelmente tenha ocorrido no Pleistoceno. A distinção entre as espécies E. bombiformis e E. niveofasciata não é suportada nos resultados aqui apresentados. No entanto, é preciso considerar que diferenças morfológicas, ainda que sutis, congruentes com a disjunção geográfica sugerem a existência de barreiras entre as populações. A baixa estruturação da filogenia do COI encontrada para o par E. niveofasciata/E. bombiformis pode indicar retenção de polimorfismo ancestral que, associada aos baixos valores de divergência interespecífica, suportam a hipótese de separação recente entre estas espécies, coincidente com a formação da diagonal árida da América do Sul no Pleistoceno. É possível que E. niveofasciata e
E. bombiformis sejam entidades reais, não distinguíveis pelo COI, e a análise de outros genes,
juntamente com caracteres morfológicos poderia auxiliar na elucidação deste problema. A maioria das decisões taxonômicas tomadas recentemente foi suportada pelos resultados das análises com o gene COI. Em alguns casos (E. marianae, E. bidentata e E. mixta), este gene sugere a existência de complexos de espécies ainda confundidas sob um mesmo nome. Os dados sugerem que o que é chamado de E. mixta, hoje, possa, na realidade ser um conjunto de espécies, com duas espécies novas a serem descritas, uma da Amazônia e outra da América Central. O nome E. mixta deve ser restrito a uma das populações da América Central. Um estudo cladístico de todo grupo analis (todas as espécies do subgênero Euglossa citadas acima e outras seis não incluídas neste estudo) se faz necessário, assim como o uso de mais marcadores moleculares para que as relações dentro deste grupo possam ser esclarecidas. Ressalta-se a importância da população de E. clausi de Murici, AL, que pode ser considerada uma “Unidade
Evolutiva Significativa”, isolada geograficamente das outras populações.
A
BSTRACTThe subtribe Euglossina (Hymenoptera: Apidae: Apini), broadly distributed in the Neotropical region, is currently represented by approximately 220 species, being most diversified in rainforest ecossystems. This study evaluates the phylogeographic structure of species/sister-species-pairs of Euglossina, with occurrence both in Amazonic (AmF) and the Atlantic (AtF) rainforests. The interspecific and interpopulational historic relationships are evaluated here using as molecular marker part of the mitochondrial gene cytochrome c oxidase subunit I (COI). Here, the hypothesis is tested that the separation between the AmF and the AtF by the arid diagonal composed by the Caatinga, Cerrado and Chaco domains has acted in the isolation of Euglossina populations, producing cryptic-species pairs. Male specimens from Euglossa
(Glossurella) crassipunctata [n = 98; 579 base pairs (bp)] and other four pairs of species with
strong affinity are included in this study: (i) Euglossa (Euglossa) bidentata (n = 10; AmF) and
Euglossa (Euglossa) marianae (n = 45; AtF; 532 bp); (ii) Euglossa (Euglossa) mixta (n = 34;
AmF) and Euglossa (Euglossa) calycina (n = 17; AtF; 533 bp); (iii) Euglossa (Euglossa)
iopyrrha (n = 1; AmF) and Euglossa (Euglossa) botocuda (n = 39; AtF; 503 bp); (iv) Eulaema (Eulaema) bombiformis (n = 21; AmF and Central America) and Eulaema (Eulaema) niveofasciata (n = 52; AtF; 558 pb). Bayesian inference and haplotype networks suggest that the
South American arid diagonal has acted on the isolation of E. crassipunctata, supporting the recent recognition of AtF populations as E. clausi, those from the AmF as E. moratoi, restricting the name E. crassipunctata only to Central American populations. The sequence of vicariant events herein inferred for the E. calycina/E. mixta pair is coincident with that obtained for species groups of Geotrigona and Paratrigona. Based on the previous estimation of the minimum age of E. mixta (5 Ma), it is possible that the arid diagonal (in one of its occurrence since the end of Miocene) is the causal factor of the separation between the populations of AtF and AmF in this group. For the crassipunctata group, the contrary was observed, which agrees with reported vicariant events of species groups of Partamona and some of Geotrigona. Previous estimation of the minimum age of the crassipunctata group (≈ 14.7 Ma) suggests that the separation between Central America and AmF is contemporary with the transgression of the Maracaibo Sea (middle Miocene) and that the break between the AmF and AtF possibly occurred in the Pleistocene. The distinction between E. bombiformis and E. niveofasciata is not supported in the results herein presented. However, it should be considered that morphological differences, albeit subtle, congruent with the geographical disjunction suggest the existence of barriers between these populations. The weak structuring of COI phylogeny found for the E.
niveofasciata/E. bombiformis pair may indicate persistence of ancestral polymorphism which, in
association with low interspecific divergence values, support the hypothesis of recent split between these species lineages, coincident with the formation of the xeric diagonal of South America in the Pleistocene. It is possible that E. niveofasciata and E. bombiformis are real entities, not distinguishable by the COI, and the analysis of other genes, along with morphological characters might help to elucidate this problem. Most of the taxonomic decisions proposed recently have been supported by the results of the analysis with the COI gene. In some cases (E. marianae, E. bidentata and E. mixta), the gene suggests the existence of a species complexes, still assigned under the same name. Data suggest that what is now called E. mixta may actually be a set of species, with two new species yet to be described, one of the AmF and another of Central America. The name E. mixta should be restricted to one of the populations from Central America. A cladistic study of the analis group (all species from the subgenus
Euglossa cited above and six more species not included in this study) is needed, as well as the
use of additional molecular markers so that the relationships inside this group are resolved. The importance of the E. clausi population from Murici (in the state of Alagoas) should be emphasized. It is herein considered as an Evolutionarily Significant Unity, geographically isolated from the other populations.
S
UMÁRIO1. Introdução ... 20
2. Objetivos ... 32
2.1. Objetivos específicos ... 32
3. Material e Métodos... 32
3.1. Amostras ... 32
3.1.1. Espécies estudadas ... 32
3.1.2. Origem das amostras ... 35
3.2. Extração, amplificação e sequenciamento do DNA ... 49
3.2.1. Marcadores moleculares ... 50
3.3. Análise de dados ... 55
3.3.1. Alinhamento ... 55
3.3.2. Seleção do modelo de evolução nucleotídica ... 56
3.3.3. Análises filogenéticas ... 56
3.3.4. Análises populacionais ... 57
4. Resultados ... 58
4.1. “Euglossacrassipunctata” ... 58
4.2. Grupo analis ... 71
4.2.1. Par Euglossa bidentata e Euglossa marianae ... 73
4.2.2. Par Euglossa mixta e Euglossa calycina ... 85
4.3. Par Eulaema niveofasciata e Eulaema bombiformis ... 96
5. Discussão ... 112
5.1. O papel da diagonal árida da América do Sul na diferenciação de populações/especiação de Euglossina dos domínios morfoclimáticos da Amazônia e da Mata Atlântica. ... 112
5.2. O grau de diferenciação entre as espécies é compatível com as hipóteses correntes sobre o tempo de isolamento das Florestas Amazônica e Atlântica? ... 115
5.3. O gene COI dá suporte às decisões taxonômicas recentes em Euglossina baseadas em estudo de caracteres morfológicos? ...... 118
5.4. Implicações para a conservação das espécies de Euglossina nos domínios florestais estudados. ... 118
6. Conclusões ... 120
7. Referências bibliográficas ... 121
L
ISTA DET
ABELASTabela 1. Lista de espécies incluídas neste estudo, número total de machos, data de coleta dos espécimes, país e localidade de origem, amostras com DNA disponível e indisponível e instituição depositária dos espécimes. ... 41 Tabela 2. Sequências dos iniciadores para a amplificação por PCR do gene COI. ... 51 Tabela 3. Reagentes utilizados na PCR para amplificação do gene mitocondrial COI das amostras enviadas para o Biodiversity Institute of Ontario (Canadá) e aquelas amplificadas no LBEM/UFMG. ... 52 Tabela 4. Lista de haplótipos do gene COI de “Euglossa crassipunctata” com os 38 sítios polimórficos. Os pontos (.) indicam igualdade com o haplótipo 1. ... 60 Tabela 5. Número de machos (n) e haplótipos de “Euglossa crassipunctata” das 13 localidades amostradas. Haplótipos destacados em negrito são haplótipos de cópia única. Para cada localidade, Hd = diversidade haplotípica e π = diversidade nucleotídica. (σ = desvio-padrão; n/a = não se aplica)... 61 Tabela 6. Índices de diversidade para os grupos de “Euglossa crassipunctata”: n = número de indivíduos, H = número de haplótipos, Hd = diversidade haplotípica, π = diversidade nucleotídica, S = número de sítios polimórficos. (σ = desvio-padrão; G = grupo) ... 68 Tabela 7. Testes de neutralidade seletiva para os grupos de “Euglossa crassipunctata”: D, teste de Tajima e Fs, teste de Fu. s: número de transições; v: número de transversões; Subst.: número
de substituições. ... 69 Tabela 8. AMOVA usando o modelo de distância Tamura e Nei, considerando G1 (PA), G2 (BA, ES e MG), G3 (AL) e G4 (Costa Rica) definidos na simulação MENCFH/Geneland como grupos e as localidades de coleta como populações de “Euglossa crassipunctata”... 70 Tabela 9. AMOVA usando o modelo de distância Tamura e Nei, considerando G1 (PA), G2 (AL), G3 (norte ES e BA), G4 (Costa Rica) e G5 (MG e parte ES) definidos na simulação MECFH/Geneland como grupos e as localidades de coleta como populações de “Euglossa
crassipunctata”. ... 70 Tabela 10. AMOVA usando o modelo de distância Tamura e Nei, considerando G1 (BA, ES, MG e AL), G2 (PA) e G3 (Costa Rica) definidos pelo NETWORK como grupos e as localidades de coleta como populações de “Euglossa crassipunctata”. ... 70 Tabela 11. AMOVA usando o modelo de distância Tamura e Nei, considerando G1 (Amazônia) e G2 (Mata Atlântica) como grupos e as localidades de coleta como populações de “Euglossa
Tabela 15. Testes de neutralidade seletiva para os grupos do par de espécies Euglossa bidentata e Euglossa marianae: D, teste de Tajima e Fs, teste de Fu. s: número de transições; v: número de
transversões; Subst.: número de substituições. ... 83 Tabela 16. AMOVA usando o modelo de distância Tamura e Nei, considerando G1 (BA + ES) G2 (Peru + AC + RO) e G3 (PA) definidos pelo Geneland (simulação MENCFH) como grupos e as localidades de coleta como populações de Euglossa bidentata e Euglossa marianae. ... 84 Tabela 17. AMOVA usando o modelo de distância Tamura e Nei, considerando G1 (Peru + AC), G2 (RO + parte PA + BA + ES) e G3 (parte PA) definidos pelo Geneland (simulação MECFH) como grupos e as localidades de coleta como populações de Euglossa bidentata e
Euglossa marianae. ... 84
Tabela 18. AMOVA usando o modelo de distância Tamura e Nei, considerando G1 (Peru + AC + RO + parte PA + parte BA + parte ES), G2 (parte Canaã dos Carajás - PA) e G3 (parte BA + parte ES) definidos pelo NETWORK como grupos e as localidades de coleta como populações de Euglossa bidentata e Euglossa marianae. ... 84 Tabela 19. AMOVA usando o modelo de distância Tamura e Nei, considerando G1 (Amazônia), G2 (Mata Atlântica) como grupos e as localidades de coleta como populações de
Euglossa bidentata e Euglossa marianae. ... 84
Tabela 20. Lista de haplótipos do gene COI de Euglossa mixta (H1 a H8) e Euglossa calycina (H9 a H11) com os 33 sítios polimórficos. Os pontos (.) indicam igualdade com o haplótipo 1. A interrogação (?) indica dado faltante. ... 86 Tabela 21. Número de machos (n) e haplótipos de Euglossa mixta e Euglossa calycina das 16 localidades amostradas. Haplótipos destacados em negrito são haplótipos de cópia única. Para cada localidade, Hd = diversidade haplotípica e π = diversidade nucleotídica. (σ = desvio -padrão; n/a = não se aplica) ... 87 Tabela 22. Índices de diversidade para os grupos do par de espécies Euglossa mixta e Euglossa calycina: n = número de indivíduos, H = número de haplótipos, Hd = diversidade haplotípica, π
= diversidade nucleotídica, S = número de sítios polimórficos. (σ = desvio-padrão; G = grupo) ... 94 Tabela 23. Testes de neutralidade seletiva para os grupos do par de espécies Euglossa mixta e Euglossa calycina: D, teste de Tajima e Fs, teste de Fu. s: número de transições; v: número de
transversões; Subst.: número de substituições. ... 95 Tabela 24. AMOVA usando o modelo de distância Tamura e Nei, considerando G1 (BA + ES), G2 (Belize + Honduras) e G3 (RO + PA) definidos pelo Geneland (simulação MENCFH) como grupos e as localidades de coleta como populações de Euglossa mixta e Euglossa calycina. O resultado da AMOVA é idêntico quando se considera os grupos definidos pelo Geneland com a simulação MECFH: G1 (Belize + Honduras), G2 (RO + PA) e G3 (BA + ES). ... 96 Tabela 25. AMOVA usando o modelo de distância Tamura e Nei, considerando G1 (Belize + Gracias a Dios-Honduras), G2 (Honduras, exceto Gracias a Dios), G3 (RO + PA) e G4 (BA + ES) definidos pelo NETWORK como grupos e as localidades de coleta como populações de
Euglossa mixta e Euglossa calycina. ... 96
Tabela 26. AMOVA usando o modelo de distância Tamura e Nei, considerando G1 (Amazônia) e G2 (Mata Atlântica) como grupos e as localidades de coleta como populações de Euglossa
mixta e Euglossa calycina. ... 96
Tabela 27. Lista de haplótipos do gene COI de Eulaema bombiformis (H1 a H19) e Eulaema niveofasciata (H20 a H33) com os 38 sítios polimórficos. Os pontos (.) indicam igualdade com
cópia única. Para cada localidade, Hd = diversidade haplotípica e π = diversidade nucleotídica.
(σ = desvio-padrão; n/a = não se aplica) ... 101 Tabela 29. Índices de diversidade para os grupos do par de espécies Eulaema niveofasciata e Eulaema bombiformis: n = número de indivíduos, H = número de haplótipos, Hd = diversidade haplotípica, π = diversidade nucleotídica, S = número de sítios polimórficos. (σ = desvio -padrão; G = grupo) ... 106 Tabela 30. Testes de neutralidade seletiva para os grupos do par de espécies Eulaema niveofasciata e Eulaema bombiformis: D, teste de Tajima e Fs, teste de Fu. s: número de
transições; v: número de transversões; Subst.: número de substituições. ... 107 Tabela 31. AMOVA usando o modelo de distância Tamura e Nei, considerando G1: Costa Rica + RO + PA (parte) + Ubajara, CE + AL; G2: BA + ES; G3: AM + Tomé-Açu, PA + MA e G4: Guaramiranga, CE definidos pelo Geneland (simulação MENCFH) como grupos e as localidades de coleta como populações de Eulaema niveofasciata e Eulaema bombiformis. .. 108 Tabela 32. AMOVA usando o modelo de distância Tamura e Nei, considerando G1 (RO + parte AM + parte PA), G2 (parte PA + MA), G3 (CE + AL), G4 (BA + ES) e G5 (Costa Rica + parte AM) definidos pelo Geneland (simulação MECFH) como grupos e as localidades de coleta como populações de Eulaema niveofasciata e Eulaema bombiformis. ... 108 Tabela 33. AMOVA usando o modelo de distância Tamura e Nei, considerando G1 (Costa Rica) e G2 (restante das amostras) definidos pelo NETWORK como grupos e as localidades de coleta como populações de Eulaema niveofasciata e Eulaema bombiformis. ... 108 Tabela 34. AMOVA usando o modelo de distância Tamura e Nei, considerando G1 (Mata Atlântica) e G2 (Amazônia) definidos como grupos e as localidades de coleta como populações de Eulaema niveofasciata e Eulaema bombiformis. ... 108 Tabela 35. Divergência genética média entre os grupos de Euglossina calculada pelo modelo de substituição nucleotídica Kimura 2-parâmetros (K2p). Os valores acima da diagonal são os erros padrões e abaixo, as distâncias. Os nomes dos táxons estão de acordo com os clados inferidos através da análise bayesiana, com exceção do par E. bombiformis/E. niveofasciata e do grupo externo. ... 109 Tabela 36. Divergência genética média entre os grupos de Euglossina calculada pelo método de distância p-distance. Os valores acima da diagonal são os erros padrões e abaixo, as distâncias. Os nomes dos táxons estão de acordo com os clados inferidos através da análise bayesiana, com exceção do par E. bombiformis/E. niveofasciata e do grupo externo. ... 110 Tabela 37. Divergência genética média dentro dos grupos (d) de Euglossina calculada pelo modelo de substituição nucleotídica Kimura 2-parâmetros (K2p). Para os grupos que continham apenas um indivíduo, não foi possível calcular esta distância. Os nomes dos táxons estão de acordo com os clados inferidos através da análise bayesiana, com exceção do par E.
bombiformis/E. niveofasciata e do grupo externo. O cálculo através do método p-distance gerou
resultados idênticos ao K2p... 111
L
ISTA DEF
IGURASFigura 1. Mapa de parte da América do Sul com os domínios morfoclimáticos da “diagonal de áreas áridas” (Caatinga, Cerrado e Chaco) separando os de florestas úmidas (Floresta Atlântica e
Amazônia). As setas de sentido duplo representam os caminhos históricos hipotéticos que conectavam a floresta atlântica e a bacia amazônica para as aves Subocines do Novo Mundo segundo Batalha-Filho et al., 2012. A seta cinza escuro corresponde aos contatos antigos (Mioceno), enquanto as setas pretas indicam contatos mais recentes (Plio-Pleistoceno). A seta preta tracejada mostra um contato recente menos comumente observado e a seta preta contínua representa um caminho recente e comumente utilizado. As marcas presentes na caatinga são localidades em que se tem evidência da presença de mata de galeria durante o final do Pleistoceno (> 0,9 mya), com base na paleopalinologia e em espeleotemas (triângulo Oliveira et
al., 1999; círculo Auler e Smart, 2001 e Wang et al., 2004). Adaptado de Batalha-Filho et al.,
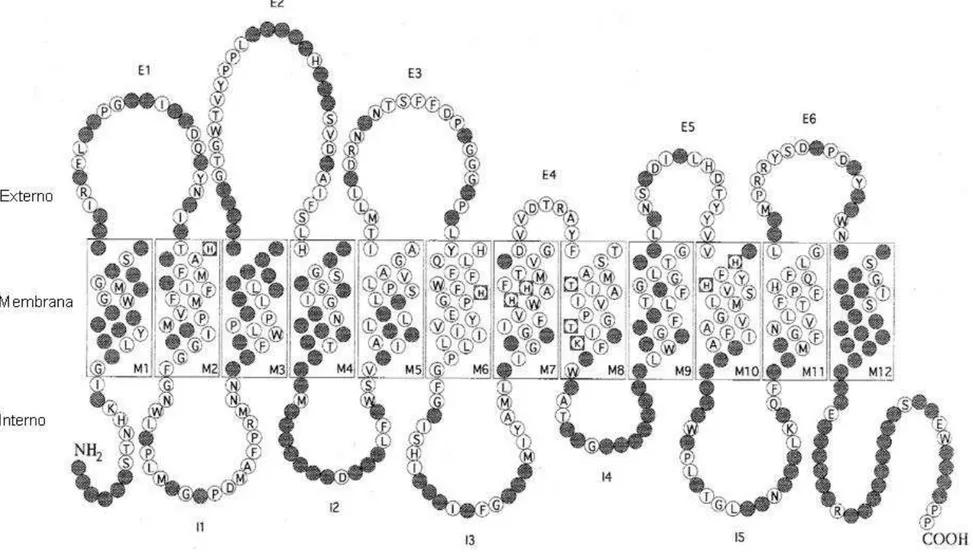
Figura 8. Machos de Eulaema bombiformis (Oriximiná-PA, no espécime: 36061) (a-f) e de Eulaema niveofasciata (Conceição da Barra-ES, no espécime: 49684) (g-l): a, g = habitus, vista lateral (note que em a as faixas dos T1-3 são amarelo-pilosas e em g elas são branco-pilosas); b, h = habitus, vista dorsal (em h é possível notar que o integumento de T2-T3, indicado pela seta, apresenta brilho verde); c, i = cabeça, vista frontal; d, j = cabeça, vista lateral (seta indica a distância clípeo-orbital inferior que é cerca de metade da distância clípeo-orbital superior); e, k = tíbia média com o coxim apontado pela seta branca; f, l = E-5 com cerdas curtas, com detalhe para as cerdas da margem distal ligeiramente maiores indicadas pela seta branca. ... 40 Figura 9. Mapa da América do Sul com destaque para os biomas brasileiros e os pontos de amostragem de Euglossa marianae e Euglossa bidentata. Os círculos e as cruzes pretas referem-se aos pontos de amostragem cujas amostras não estão incluídas nas análises moleculares e os círculos e as cruzes vermelhas referem-se às amostras incluídas nas análises moleculares. ... 45 Figura 10. Mapa da América do Sul com destaque para os biomas brasileiros e os pontos de amostragem de Euglossa calycina e Euglossa mixta. As cruzes pretas referem-se aos pontos de amostragem cujas amostras não estão incluídas nas análises moleculares e os círculos e as cruzes vermelhas referem-se às amostras incluídas nas análises moleculares. ... 46 Figura 11. Mapa da América do Sul com destaque para os biomas brasileiros e os pontos de amostragem de “Euglossa crassipunctata”. As cruzes pretas referem-se aos pontos de amostragem cujas amostras não estão incluídas nas análises moleculares e as cruzes vermelhas referem-se às amostras incluídas nas análises moleculares. ... 47 Figura 12. Mapa da América do Sul com destaque para os biomas brasileiros e os pontos de amostragem de Eulaema niveofasciata e Eulaema bombiformis. Os círculos e as cruzes pretas referem-se aos pontos de amostragem cujas amostras não estão incluídas nas análises moleculares e os círculos e as cruzes vermelhas referem-se às amostras incluídas nas análises moleculares. ... 48 Figura 13. Modelo estrutural bidimensional da cadeia polipeptídica da subunidade I da citocromo c oxidase (COI) de insetos. Há 25 regiões estruturais: 12 hélices intra-membrana
(M1−M12); seis alças (loops) externas (E1−E6); cinco alças internas (I1−I5); extremidades
carboxi (COOH) e amino (NH2) terminais. Os resíduos de aminoácidos inscritos em quadrados tem significância funcional especial e os círculos escuros hachurados representam os resíduos variáveis estre espécies de insetos. Modificado de Lunt et al,.1996. ... 53 Figura 14. Mapa do genoma mitocondrial circular de Apis mellifera. Os genes codificadores das subunidades I, II e III da citocromo c oxidase estão indicados por COI, COII e COIII, respectivamente. A direção da transcrição para cada região codificadora é indicada pelas setas. Adaptado de Crozier e Crozier (1993). ... 54 Figura 15. Filograma da árvore de consenso resultante da inferência Bayesiana para 579 pb do COI de “Euglossa crassipunctata” e duas espécies do grupo externo (Euglossa marianae e
Euglossa mixta). Os valores de probabilidade posterior são apresentados nos nós da árvore. As
correlacionado à frequência dos haplótipos implementado no Geneland para “Euglossa
crassipunctata”. Nas quatro figuras inferiores os eixos horizontais e verticais representam as coordenadas longitudinais e latidudinais, respectivamente, e os pontos pretos representam as localidades amostradas, conforme apontado no mapa superior à esquerda. A cor branca indica maiores valores de probabilidade e, na medida em que a cor se aproxima do vermelho, a probabilidade decresce. O gráfico acima à direita representa o número mais provável de populações (K = 4) após a simulação MCMC. ... 66 Figura 18. Mapas de probabilidade posterior de designação de cinco diferentes populações [G1: PA; G2: AL; G3: ES (Conceição da Barra) + BA; G4: Costa Rica; G5: MG + ES (Linhares e Sooretama)] pelo modelo espacial correlacionado à frequência dos haplótipos implementado no
Geneland para “Euglossa crassipunctata”. Nas três figuras superiores e nas duas à esquerda os eixos horizontais e verticais representam as coordenadas longitudinais e latidudinais, respectivamente, e os pontos pretos representam as localidades amostradas, conforme apontado no mapa inferior central. A cor branca indica maiores valores de probabilidade e, na medida em que a cor se aproxima do vermelho, a probabilidade decresce. O gráfico inferior à direita representa o número mais provável de populações (K = 5) após a simulação MCMC. ... 67 Figura 19. Filograma da árvore de consenso resultante da inferência Bayesiana para 503 pb do COI do grupo analis e duas espécies do grupo externo, “Euglossa crassipunctata” e Euglossa
parvula. Os valores de probabilidade posterior são apresentados nos nós da árvore. As cores
destacam cada clado: E. calycina em laranja, E. marianae (parte 2) em rosa claro, E. bidentata (indivíduo Ebi17) em lilás, E. botocuda em vermelho claro, E. iopyrrha em vermelho escuro, E.
mixta (Amazônia) em amarelo, E. mixta (Honduras) em verde claro, E. mixta (Honduras +
Belize) em verde escuro, E. marianae (parte 1) em rosa escuro, E. bidentata (restante dos indivíduos) em roxo. ... 72 Figura 20. Filograma da árvore de consenso resultante da inferência Bayesiana para 532 pb do COI de Euglossa ... 78 Figura 21. Rede de haplótipos Median-Joining do gene COI de Euglossa bidentata e Euglossa marianae e seus principais agrupamentos. Cada círculo na rede de haplótipos corresponde a um
haplótipo e o seu tamanho é proporcional à frequência do haplótipo. Cada traço na linha que conecta um haplótipo ao outro se refere a um passo mutacional. As cores representam as localidades das amostras e os códigos dos haplótipos estão de acordo com as Tabelas 12 e 13. O grupo destacado em laranja (G1) possui haplótipos da Amazônia e da Mata Atlântica, aquele destacado em amarelo (G2) contem um haplótipo da Amazônia e o vermelho (G3) possui haplótipos da Mata Atlântica... 79 Figura 22. Mapas de probabilidade posterior de designação de três diferentes populações [G1: BA + ES; G2: Peru + AC + RO e G3: PA] pelo modelo espacial não correlacionado à frequência dos haplótipos implementado no Geneland para o par Euglossa bidentata e Euglossa
marianae. Na coluna à esquerda os eixos horizontais e verticais representam as coordenadas
Figura 24. Filograma da árvore de consenso resultante da inferência Bayesiana para 533 pb do COI de Euglossa mixta e Euglossa calycina e duas espécies do grupo externo (“Euglossa
crassipunctata” e Euglossa parvula). Os valores de probabilidade posterior são apresentados nos nós da árvore. As cores destacam cada clado: E. calycina (Mata Atlântica) em laranja, E.
“mixta 1” (Amazônia) em amarelo e E. mixta s. stricto (América Central) em verde, que contem dois clados―E. “mixta 2” [Belize + Honduras (Río Plátano - Gracias a Dios)] em verde escuro e E. “mixta 3” (Honduras) em verde claro. ... 90 Figura 25. Rede de haplótipos Median-Joining do gene COI de Euglossa mixta e Euglossa calycina e seus principais agrupamentos. Cada círculo na rede de haplótipos corresponde a um
haplótipo e o seu tamanho é proporcional à sua frequência. Cada traço na linha que conecta um haplótipo a outro se refere a um passo mutacional. As cores representam as localidades das amostras e os códigos dos haplótipos estão de acordo com as Tabelas 20 e 21. Os grupos destacados em verde (G1 e G2) apresentam haplótipos da América Central, aquele destacado em amarelo (G3) contem haplótipos da Amazônia e o vermelho (G4) da Mata Atlântica. ... 91 Figura 26. Mapas de probabilidade posterior de designação de três diferentes populações [G1: BA + ES; G2: Belize + Honduras e G3: RO + PA] pelo modelo espacial não correlacionado à frequência dos haplótipos implementado no Geneland para o par Euglossa mixta e Euglossa
calycina. Na coluna à esquerda os eixos horizontais e verticais representam as coordenadas
longitudinais e latidudinais, respectivamente, e os pontos pretos representam as localidades amostradas, conforme apontado no mapa superior à direita. A cor branca indica maiores valores de probabilidade e, na medida em que a cor se aproxima do vermelho, a probabilidade decresce. O gráfico abaixo à direita representa o número mais provável de populações (K = 3) após a simulação MCMC. ... 92 Figura 27. Mapas de probabilidade posterior de designação de três diferentes populações [G1: Belize + Honduras; G2: RO + PA e G3: BA + ES] pelo modelo espacial correlacionado à frequência dos haplótipos implementado no Geneland para o par Euglossa mixta e Euglossa
calycina. Na coluna à esquerda os eixos horizontais e verticais representam as coordenadas
longitudinais e latidudinais, respectivamente, e os pontos pretos representam as localidades amostradas, conforme apontado no mapa superior à direita. A cor branca indica maiores valores de probabilidade e, na medida em que a cor se aproxima do vermelho, a probabilidade decresce. O gráfico abaixo à direita representa o número mais provável de populações (K = 3) após a simulação MCMC. ... 93 Figura 28. Filograma da árvore de consenso resultante da inferência Bayesiana para 558 pb do COI de Eulaema bombiformis e Eulaema niveofasciata e duas espécies do grupo externo
(Eufriesea zhangi e Eulaema nigrita). Os valores de probabilidade posterior são apresentados
nos nós da árvore. ... 102 Figura 29. Rede de haplótipos Median-Joining do gene COI de Eulaema bombiformis e Eulaema niveofasciata e seus principais agrupamentos. Cada círculo na rede de haplótipos
corresponde a um haplótipo e o seu tamanho é proporcional à frequência do haplótipo. Cada traço na linha que conecta um haplótipo ao outro se refere a um passo mutacional. As cores representam as localidades das amostras e os códigos dos haplótipos estão de acordo com as Tabelas 27 e 28. O grupo destacado em verde (G1) é formado por um haplótipo da Costa Rica e aquele destacado em laranja (G2) possui haplótipos da Amazônia e da Mata Atlântica. ... 103 Figura 30. Mapas de probabilidade posterior de designação de quatro diferentes populações [G1: Costa Rica + RO + PA (parte) + Ubajara-CE + AL; G2: BA + ES; G3: AM + Tomé-Açu-PA + MA e G4: Guaramiranga-CE] pelo modelo espacial não correlacionado à frequência dos haplótipos implementado no Geneland para o par Eulaema bombiformis e Eulaema
niveofasciata. Nas quatro figuras superiores os eixos horizontais e verticais representam as
probabilidade decresce. O gráfico inferior à direita representa o número mais provável de populações (K = 4) após a simulação MCMC. ... 104 Figura 31. Mapas de probabilidade posterior de designação de cinco diferentes populações [G1: RO + Nhamundá-AM + Oriximiná-PA; G2: PA (Carajás e Tomé-Açu) + MA; G3: CE + AL; G4: BA + ES e G5: Costa Rica + Novo Aripuanã-AM.] pelo modelo espacial correlacionado à frequência dos haplótipos implementado no Geneland para o par Eulaema bombiformis e
Eulaema niveofasciata. Nas três figuras superiores e nas duas à esquerda os eixos horizontais e
verticais representam as coordenadas longitudinais e latidudinais, respectivamente, e os pontos pretos representam as localidades amostradas, conforme apontado no mapa inferior. A cor branca indica maiores valores de probabilidade e, na medida em que a cor se aproxima do vermelho, a probabilidade decresce. O gráfico inferior à direita representa o número mais provável de populações (K = 5) após a simulação MCMC. ... 105
L
ISTA DE ABREVIATURAS,
SIGLAS E UNIDADESABI: Applied Biosystems
AMNH: American Museum of Natural History AMOVA: Analysis of molecular variance
Amplicon: pedaço de DNA que é o produto da reação artificial de amplificação por PCR
apCADfor1: iniciador Forward do gene CAD ap835rev1: iniciador Reverse do gene CAD ≈ : aproximadamente
A, T, C, G: nucleotídeos Adenina, Timina, Citosina e Guanina
Atl: área de endemismo do sudeste Atlântico, que corresponde à região das matas atlânticas, da Bahia até São Paulo, segundo Camargo (1996) e Amorim e Pires (1996).
B10: Fator Bayesiano
Bee-wg-For2: iniciador Forward do gene Wingless Bee-wg-Rev2: iniciador Reverse do gene Wingless BIC: Bayesian Information Criterion
BOLD: Barcode of life database BSA: Bovine Serum Albumin °C: Graus Celsius
C1-N-2191: iniciador Reverse do gene COI
CAD: carbamoyl-phosphate synthetase 2, aspartate transcarbamylase e dihydroorotase CECT-UFMG: Coleção Entomológica das Coleções Taxonômicas da UFMG
Chocó-CA: área de endemismo que corresponde ao noroeste dos Andes e Panamá, América Central e México, segundo Cracraft e Prum (1988).
CNC: Canadian National Collection
COI: subunidade I do gene mitocondrial citocromo c oxidase COII: subunidade II do gene mitocondrial citocromo c oxidase COIII: subunidade III do gene mitocondrial citocromo c oxidase
CPATU: Centro de Pesquisa Agroflorestal da Amazônia Oriental - Embrapa Amazônia Oriental.
CTAB: cetyl trimethylammonium bromide CUIC: Cornell University Insect Collection
D: teste estatístico de neutralidade de Tajima (1989), usado para distinguir entre sequências de DNA que evoluem de forma neutra (aleatória) daquelas que evoluem sob influência de alguma força evolutiva.
ddH2O: água bidestilada e autoclavada
σ: desvio-padrão
DNA: Ácido desoxirribonucléico
dNTPs: Desoxirribonucleotídeo trifosfatado
DZUP-UFPR: Coleção Entomológica Padre Jesus Santiago Moure da Universidade Federal do Paraná
EDTA: ácido etilenodiamino tetra-acético ESS: Effective Sample Size
ESUs: Evolutionary Significant Units (Unidades Evolutivas Significativas) ET kit: kit de sequenciamento para o sequenciador MegaBACE
EUA: Estados Unidos da América
e.g.: do latim exempli gratia, que significa por exemplo
FAPEMIG: Fundação de Amparo à Pesquisa do Estado de Minas Gerais
Fs: teste estatístico de neutralidade de Fu (1997) usado para distinguir entre sequências de DNA que evoluem de forma neutra (aleatória) daquelas que evoluem sob influência de alguma força evolutiva.
FST: Índice de Fixação de Wright
Γ: distribuição gamma pela teoria de probabilidade e estatística G1, G2, G3, G4 e G5: grupos 1, 2, 3, 4 e 5
GeneBank: banco de dados colaborativo de sequências de genes e proteínas
GTR: modelo de substituição nucleotídica General Time Reversible H: número de haplótipos
Hd: disversidade haplotípica
H1, H2, H3, etc.: haplótipo 1, 2, 3, etc.
HKY: modelo de substituição nucleotídica de Hasegawa, Kishino e Yano Km2: quilômetros quadrados
IBAMA: Instituto Brasileiro do Meio Ambiente e dos Recursos Naturais Renováveis IBGE: Instituto Brasileiro de Geografia e Estatística
ICB: Instituto de Ciências Biológicas - UFMG
ICMBio: Instituto Chico Mendes de Conservação da Biodiversidade
K: número máximo de populações HWEL (Hardy-Weinberg equilibrium with linkage equilibrium between loci) estimado após a simulação MCMC no programa Geneland
K2p: modelo de substituição nucleotídica Kimura 2-parâmetros LBEM: Laboratório de Biodiversidade e Evolução Molecular LepF1: iniciador Forward do gene COI
LepR1: iniciador reverse do gene COI Ma: milhões de anos
MCMC: Cadeia de Markov Monte Carlo
MECFH: modelo espacial correlacionado à frequência dos haplótipos MENCFH: modelo espacial não correlacionado à frequência dos haplótipos μL: Microlitros
μM: Micromolar mM: Milimolar
MgCl2: Cloreto de magnésio
MMA: Ministério do Meio Ambiente mtDNA: DNA mitocondrial
NAm: área de endemismo do norte do Amazonas, que corresponde ao Cráton das Guianas-Brasil, segundo Camargo (1996) e Amorim e Pires (1996).
n: número de indivíduos nDNA: DNA nuclear
Nemit: tamanho efetivo populacional do mtDNA
Nenuc:tamanho efetivo populacional do nDNA
ng: nanogramas
NUMT: nuclear mitochondrial DNA ou contaminante do DNA nuclear no DNA mitocondrial pb: pares de bases nucleotídicas
PARNA Ubajara: Parque Nacional de Ubajara, Ceará PCR: reação em cadeia da polimerase
p-distance: algoritmo de distância nucleotídica
PEG: Polietileno Glicol π: diversidade de nucleotídeos PP: probabilidade posterior
PSFR: Potential Scale Reduction Factor P: Valores de significância
ROM: Royal Ontario Museum S: número de sítios polimórficos
s: número de transições
SEAm: área de endemismo que corresponde ao sudeste da Amazônia, abrangendo toda área ao sul dos rios Madeira e Amazonas, segundo Camargo (1996) e Amorim e Pires (1996).
SWAm: área de endemismo que corresponde ao sudoeste da Amazônia, delimitado pelo alinhamento dos rios Uaupés/Negro ao norte, pelos rios Madeira/Mamoré ao sul e pelos Andes a oeste, segundo Amorim e Pires (1996).
T1-3: 1° ao 3° tergos do metassoma dos Hymenoptera UFCE: Universidade Federal do Ceará
UFMG: Universidade Federal de Minas Gerais
UFPR – DZUP: Coleção Entomológica Padre Jesus Santiago Moure do Departamento de Zoologia da Universidade Federal do Paraná
UICN: União Internacional para a Conservação da Natureza v: número de transversões
Wg: wingless
1.
Introdução
Informações geradas sobre relações filogenéticas com o uso de ferramentas da
biologia molecular podem ser associadas ao conhecimento sobre a distribuição
geográfica dos organismos. Isso possibilita a construção de hipóteses sobre biogeografia
histórica, tornando possível a compreensão dos processos de diversificação (Patton et
al., 1994).
A filogeografia é uma área da biogeografia histórica que faz uso de marcadores
moleculares, especialmente de DNA organelar, com o objetivo de compreender
princípios e processos responsáveis pela distribuição geográfica das linhagens
genealógicas que compõem as espécies ou de espécies proximamente aparentadas. Em
estudos com animais, o DNA mitocondrial (mtDNA) possui características desejáveis
para este tipo de pesquisa, particularmente uma taxa evolutiva mais alta que a de genes
nucleares, grande variação intraespecífica, presença de várias cópias, além de possuir
herança exclusivamente materna e não recombinante (Avise et al., 1987; Avise, 2000).
Estudos que utilizam a abordagem filogeográfica podem contribuir para os
esforços de conservação da diversidade biológica por fornecerem evidências para o
reconhecimento de espécies irmãs ou complexos de espécies a partir do que se
considerava uma única espécie com distribuição ampla (Kambhampati et al., 1996;
Buhay e Crandall, 2005; Zayed, 2009 Frankham et al., 2010). A falta de conhecimento
sobre o status taxonômico da espécie alvo de um estudo pode, por exemplo, impedir a
proteção de espécies ameaçadas de extinção ou promover o gasto de recursos e esforços
na conservação de populações de espécies comuns (Frankham et al., 2010). Estudos
com o enfoque filogeográfico podem ainda fornecer subsídios para a proposição de
áreas prioritárias para a conservação, ao apontar as unidades de conservação
intraespecíficas
(“filogrupos” intraespecíficos ou Unidades Evolutivas Significativas –
ESUs da sigla inglesa) definidas por distribuição geográfica e padrões genéticos únicos
(Moritz, 1994; Avise, 2000, 2004).
Os estudos com abordagem filogeográfica podem ser feitos em escala
local/regional, caso o grupo de estudo possua uma capacidade dispersiva limitada, como
por exemplo, peixes e golfinhos de água doce (Bermingham e Martin, 1998; Hollatz et
al., 2011) e salamandras (Garcia-Paris et al., 2000) e, em escala continental, para
aquelas espécies com maior capacidade dispersiva e de distribuição mais ampla
com espécies de ampla distribuição têm revelado, por exemplo, um grande fluxo gênico
ao longo da bacia amazônica (Dick et al., 2003) e a existência de pontos de contato no
Panamá para populações derivadas da América Central e América do Sul (Perdices et
al., 2002; Dick et al., 2003).
Os marcadores de mtDNA, especialmente a subunidade I do gene mitocondrial
citocromo c oxidase (COI), têm-se mostrado eficientes na investigação de padrões
filogeográficos intraespecíficos (e.g. Dick et al., 2004; Norgate et al., 2009;
Batalha-Filho et al., 2010; Brito e Arias, 2010), na caracterização de limites entre espécies
animais, especialmente quando os caracteres morfológicos são de difícil interpretação
(e.g. Brunner et al., 2002; Hebert et al., 2003; Naeole e Haymer, 2003; Schama et al.,
2005; López-Uribe e Del Lama, 2007) e nos casos de detecção de espécies crípticas
(e.g. Kambhampati et al., 1996; Kuhlmann et al., 2007; Gibbs, 2009).
Recentemente, os estudos filogeográficos têm utilizado também marcadores de
DNA nuclear (e.g. Hare e Avise, 1998; Kawakita et al., 2003; Harlin-Cognato et al.,
2007; Rosenblum et al., 2007; Ingvarsson, 2008; Lee e Edwards, 2008; Peters et al.,
2008; Hurt et al., 2009; Werneck et al., 2012), visto que dados de múltiplos loci podem
oferecer informações mais completas sobre a evolução das diversas linhagens e, com
isso, as estimativas dos padrões filogeográficos do grupo de organismos estudado (Hare,
2001). A comparação de árvores de genes de grupos de ligação diferentes possibilita a
detecção de separação incompleta de haplótipos ancestrais, que pode não ser percebida
quando somente uma árvore de gene é analisada (Avise, 2004). Apesar disso, pelo fato
do genoma mitocondrial ser haplóide e transmitido somente pelas fêmeas, estima-se que
o tamanho efetivo populacional do mtDNA (Nemit
) seja um quarto da dos genes nucleares
(N
enuc) e isso os torna menos susceptíveis à separação incompleta de alelos ancestrais, e,
portanto, à incongruência com a árvore de espécies (Moore, 1995).
Outro aspecto que favorece o uso de marcadores nucleares em estudos
filogeográficos é a possibilidade de seleção indireta do mtDNA de artrópodes originária
de um desequilíbrio de ligação devido à herança maternal de micro-organismos
simbiontes (e.g. Wolbachia), o que poderia confundir as interpretações da história
genômica se tomadas somente a partir de dados de seqüência de mtDNA (Hurst e
Jiggins, 2005; Gerth et al., 2011). No entanto, casos de amplificação inadvertida de
sequência de bactéria com primers para COI de artrópodes são raras e quando ocorrem
Neste estudo, examina-se a filogeografia da subtribo de abelhas Euglossina
(Insecta: Hymenoptera: Apidae) sensu Roig-Alsina e Michener (1993). As Euglossina
são abelhas de tamanho médio (8,5 mm) a grande (29 mm), com a maioria apresentando
um colorido metálico e a glossa muito longa, em comparação ao comprimento do corpo
(Michener, 2007). Nesta subtribo são reconhecidos cinco gêneros monofiléticos bem
delimitados (Aglae, Eufriesea, Euglossa, Eulaema e Exaerete) (Michel-Salzat et al.,
2004; Ramírez et al., 2010), sendo dois deles (Aglae e Exaerete) exclusivamente
parasitas de ninhos de Eufriesea e Eulaema (Silveira et al., 2002; Michener, 2007).
Euglossina é um grupo relativamente jovem.
O fóssil mais antigo deste grupo (≈ 20
-22
Ma) possui caracteres sinapomórficos que o colocam no gênero Euglossa atual (Poinar,
1998; Engel, 1999; Roubik e Hanson, 2004) e, pelas análises de datação molecular,
Ramírez et al. (2010) estimam que todas as linhagens existentes de Euglossina
compartilham um ancestral comum mais recente durante os períodos Mioceno-Eoceno
(
≈
27-42 Ma). Há aproximadamente 220 espécies descritas (Nemésio, 2009), das quais a
maioria é solitária, porém algumas espécies de Euglossa e, provavelmente, todas as
espécies de Eulaema formam colônias parassociais com várias fêmeas (Zucchi et al.,
1969; Pereira-Martins e Kerr, 1991; Santos e Garófalo, 1994; Garófalo et al. 1998;
Augusto e Garófalo, 2011). O conhecimento sobre nidificação e estrutura de ninhos é
ainda relativamente escasso para o grupo (Dressler, 1982a).
As Euglossina visitam flores de pelo menos 23 famílias de plantas à procura de
néctar, de pelo menos nove famílias em busca do pólen e de três famílias como fontes
de resinas (Roubik, 1989). Uma característica notável dos machos de Euglossina é a
coleta de substâncias aromáticas geralmente produzidas em flores de orquídeas
(Orchidaceae)
e
de
Araceae,
Bignoniaceae,
Euphorbiaceae,
Gesneriaceae,
Haemodoraceae (Roubik e Hanson, 2004) e Solanaceae (Michener, 2007). Os machos
de Euglossina são os principais, e muitas vezes, os únicos polinizadores das plantas em
que buscam tais fragrâncias (Dressler, 1982a; Roubik, 1989). Dentre as Orchidaceae,
por exemplo, cerca de 650 espécies são sabidamente polinizadas por machos de
Euglossina (Ackerman, 1983). Além disso, estas abelhas já foram vistas coletando
essências de frutos, fungos, excremento de aves, madeira apodrecida e seiva elaborada
de plantas (Roubik, 1989; Cameron, 2004; Michener, 2007).
Os machos de Euglossina, após coletarem essas fragrâncias, que podem ser
monoterpenos (e.g. cineol) ou compostos aromáticos (e.g. eugenol) (Roubik, 1989),
isolaram aproximadamente 60 substâncias diferentes das essências florais coletadas
pelos machos dessa subtribo, sendo que cada espécie possuía de três a dez compostos
aromáticos e as misturas analisadas eram espécie-específicas. Entretanto, ainda é
desconhecida a função biológica que essas substâncias desempenham na biologia dessas
abelhas. Há especulações de que esses compostos sejam importantes na reprodução
desse grupo (Kimsey, 1980; Williams e Whitten, 1983), participando, por exemplo, do
processo de reconhecimento dos machos pelas fêmeas no ato do acasalamento (Eltz et
al., 1999; Eltz et al., 2005b; Zimmermann et al., 2006).
A partir da análise das diferentes substâncias coletadas pelos machos de
Euglossina (Dodson et al., 1969) foi possível sintetizar, em laboratório, diversos
compostos aromáticos que possibilitaram a atração dessas abelhas e, conseqüentemente,
a execução de estudos etológicos (e.g. Kimsey, 1980; Eltz et al., 2005a), ecológicos
(e.g. Janzen, 1981), de composição faunística e sazonalidade (e.g. Roubik e Ackerman,
1987; Rebêlo e Moure, 1995; Neves e Vianna, 1997, 1999; Nemésio e Faria Jr., 2004;
Nemésio e Morato, 2006). Com isso, as descrições das espécies de Euglossina
normalmente são baseadas em holótipos machos e as fêmeas são mais raras nas
coleções entomológicas (Bonilla-Gómez e Nates-Parra, 1992). López-Uribe e Del Lama
(2007) estimam que fêmeas de Euglossa sejam desconhecidas para cerca de 40% das
espécies já descritas.
Euglossina são amplamente distribuídas na região Neotropical, ocorrendo desde
o sul dos Estados Unidos (Minckley e Reyes, 1996; Pemberton e Wheeler, 2006) ao sul
do Brasil (Wittmann et al., 1988) e norte da Argentina (Roubik e Hanson 2004).
Estima-se que esta tribo compreenda cerca de 25% da comunidade de abelhas em
alguns ecossistemas (Roubik e Hanson, 2004), podendo ser encontradas em diferentes
biomas (Silveira et al., 2002), às vezes alcançando altitudes de até 2.700 m (Vergara e
Pinto, apud Roubik, 1989), porém o grupo é mais diversificado nos ecossistemas
florestais (Dressler, 1982a; Morato et al., 1992; Cameron, 2004). Com isso, estudos
sobre as mudanças históricas nos ecossistemas florestais desta região podem auxiliar na
compreensão dos padrões de distribuição e diversificação dessas abelhas.
A América do Sul apresenta essencialmente quatro áreas de florestas úmidas
—
Amazônia, Mata Atlântica, Yungas andinas e Floresta Úmida Chilena (nesta última, as
Euglossina não ocorrem) (Ojeda e Mares, 1989; Hueck, apud de Vivo e Carmignotto,
2004; Hueck e Seibert, apud de Vivo e Carmignotto, 2004; Rizinni, 1997; Eva et al.,
florestas tropicais mais diversificadas do mundo e entre estes dois domínios há uma
faixa de vegetação mais aberta e seca (“diagonal de áreas áridas”), que inclui a Caatinga
(nordeste do Brasil), o Cerrado (região central do Brasil) e o Chaco (partes dos
territórios paraguaio, boliviano, argentino e brasileiro
—
ao sul do Pantanal) (Figura 1 e
Figura 2C) (Ab’Saber, 1977). Esta faixa, conhecida por promover a “maior disjunção da
América do Sul” (Brieger,
apud Costa, 2003), tem sido apontada como uma barreira à
dispersão de espécies entre estas duas regiões de florestas úmidas (e.g. Vanzolini, 1974;
Moojen, apud Costa, 2003; Raven e Axelrod, 1974; Rizzini, apud Costa, 2003; Martini
et al., 2007; Batalha-Filho et al., 2012).
Contudo, esta “faixa xérica” pode não
representar uma região segregante para alguns táxons e sim complementar às florestas
úmidas da Amazônia e da Mata Atlântica, devido a matas de galerias e fragmentos de
florestas decíduas e semi-decíduas que interconectam estes domínios florestais
(Oliveira-Filho e Ratter, 1995; Silva, 1996; Silva e Bates, 2002; Costa, 2003). O estudo
de Faria e Silveira (2011), no entanto, apresenta indícios de que as matas ciliares do
Cerrado aparentemente não atuam como corredores mésicos para a colonização deste
domínio morfoclimático pelas espécies de Euglossina dependentes de ambientes
florestais.
As florestas Amazônica e Atlântica foram provavelmente contínuas no passado
e, com o aumento da aridez no Terciário, houve a formação de uma faixa xeromórfica
entre elas (Bigarella et al., apud Costa, 2003). Há aproximadamente 30.000 anos, no
Pleistoceno (entre 2.588.000
–
11.700 anos atrás), o clima se tornou mais quente e úmido
(Ledru, 1993), com uma provável expansão das florestas úmidas. Ainda no Pleistoceno,
entre 25.000 e 13.000 anos atrás, houve o Último Máximo Glacial, com retração das
florestas úmidas e expansão de um mosaico de florestas abertas e savanas na América
do Sul (Figura 2A). Em seguida, no Holoceno (desde 11.700 anos até o presente) houve
um período mais quente e úmido (entre 9.000 e 5.000 anos atrás) conhecido como o
Ótimo Climático do Holoceno quando possivelmente houve a última junção das
florestas Amazônica e Atlântica (Figura 2B). Este período quente e úmido declinou
gradualmente até aproximadamente 2.000 anos (de Vivo e Carmignotto, 2004).
Estudos botânicos realizados
nos “Brejos de altitude”, que são
encraves de
florestas úmidas da Caatinga, e em florestas costeiras do nordeste brasileiro revelaram
uma disjunção florística com a Amazônia ou com a Mata Atlântica (Rizzini, 1963;
Coimbra-Filho e Câmara, 1996; Bigarella et al., apud Costa, 2003; Wang et al., 2004).
mamíferos, no registro fóssil e em fisiologia ecológica, que uma floresta úmida capaz de
sustentar mamíferos arborícolas típicos de florestas úmidas existiu na área atualmente
ocupada pela Caatinga, e que esta floresta conectava as florestas úmidas da Mata
Atlântica com a porção leste da Amazônia. Coimbra-Filho e Câmara (1996)
argumentam que, ainda no início do século XVI, havia uma continuidade das florestas
na costa atlântica brasileira e que a disjunção vista hoje é resultante de um processo
ininterrupto de degradação ambiental de origem antrópica.
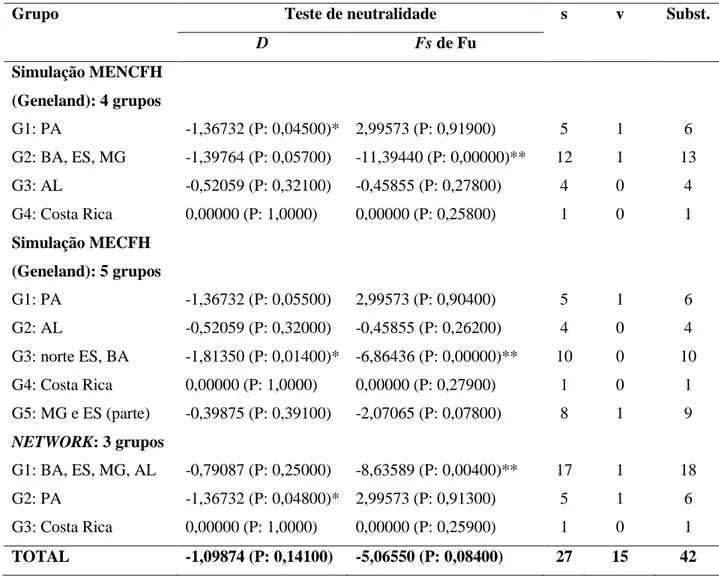
Figura 1. Mapa de parte da América do Sul com os domínios morfoclimáticos da “diagonal de áreas áridas” (Caatinga, Cerrado e Chaco) separando os de florestas úmidas (Floresta Atlântica e Amazônia). As setas de sentido duplo representam os caminhos históricos hipotéticos que conectavam a floresta atlântica e a bacia amazônica para as aves Subocines do Novo Mundo segundo Batalha-Filho et al., 2012. A seta cinza escuro corresponde aos contatos antigos (Mioceno), enquanto as setas pretas indicam contatos mais recentes (Plio-Pleistoceno). A seta preta tracejada mostra um contato recente menos comumente observado e a seta preta contínua representa um caminho recente e comumente utilizado. As marcas presentes na caatinga são localidades em que se tem evidência da presença de mata de galeria durante o final do Pleistoceno (> 0,9 mya), com base na paleopalinologia e em espeleotemas (triângulo Oliveira et al., 1999; círculo Auler e Smart, 2001 e Wang et al., 2004). Adaptado de Batalha-Filho et al., 2012.
Floresta Atlântica
Amazônia
Caatinga
Cerrado
Pantanal

![Figura 17. Mapas de probabilidade posterior de designação de quatro diferentes populações [G1: PA; G2: BA + ES + MG; G3: AL; G4: Costa Rica] pelo modelo espacial não correlacionado à frequência dos haplótipos implementado](https://thumb-eu.123doks.com/thumbv2/123dok_br/15187036.18715/67.892.45.768.105.944/probabilidade-designacao-diferentes-populacoes-correlacionado-frequencia-haplotipos-implementado.webp)
![Figura 18. Mapas de probabilidade posterior de designação de cinco diferentes populações [G1: PA; G2: AL; G3: ES (Conceição da Barra) + BA; G4: Costa Rica; G5: MG + ES (Linhares e Sooretama)] pelo modelo espacial correlacionado à frequência dos haplótipos](https://thumb-eu.123doks.com/thumbv2/123dok_br/15187036.18715/68.1262.92.1126.28.654/probabilidade-designacao-populacoes-conceicao-sooretama-correlacionado-frequencia-haplotipos.webp)