UNIVERSIDADE FEDERAL DO RIO GRANDE DO NORTE CENTRO DE CIˆENCIAS EXATAS E DA TERRA
DEPARTAMENTO DE F´ISICA TE ´ORICA E EXPERIMENTAL PROGRAMA DE P ´OS-GRADUAC¸ ˜AO EM F´ISICA
A INFLU ˆ
ENCIA DO ESQUELETO
AC
¸ ´
UCAR-FOSFATO NO TRANSPORTE
ELETR ˆ
ONICO DA MOL ´
ECULA DE DNA
Ricardo Gondim Sarmento
Orientador: Prof. Dr. EUDENILSON LINS DE ALBUQUERQUE
Tese de mestrado apresentada ao Departamento de F´ısica Te´orica e Experimental da Universidade Federal do Rio Grande do Norte como requisito parcial `a obten¸c˜ao do grau de MESTRE em
F´ISICA.
Dedico a Deus, grande criador presente em todos os momentos da vida, por ter iluminado meus caminhos nos avan¸cos cotidianos na busca da conquista... sei que ela surgir´a triufante hoje ou amanh˜a, pois vale trabalhar e fazer o melhor que posso, aqui e agora, porque a vida se incube de trazer os bons frutos para o futuro.
Agradecimentos
Ao Prof. Dr. Eudenilson Lins de Albuquerque pela oportunidade, ensinamentos, orienta¸c˜ao e pela confian¸ca depositada. Acima de tudo, um profissional dedicado e apai-xonado pela f´ısica, que mant´em seu compromisso em formar novos cientistas, comparti-lhando o seu conhecimento e sua experiˆencia, fornecendo os recursos imprescind´ıveis para forma¸c˜ao de um profissional de excelˆencia.
Aos professores participantes da banca examinadora Prof. Dr. F´abio Ferreira de Medeiros pela eficiente colabora¸c˜ao especialmente nos momentos de d´uvidas, sua paciˆencia e compreens˜ao foram primordiais e ao Prof. Dr. Umberto Fulco pela disponibilidade e contribui¸c˜oes.
A Teresa Cristina Ferreira da Silva, companheira, que esteve comigo nas tristezas e ale-grias, que prudentemente me deu incentivo, al´em de entender meus per´ıodos de ausˆencia, pela sublime paciˆencia e pensamento de dia cada vez melhores, pois com insistˆencia sem-pre, no final, tudo d´a certo!
Ao meu irm˜ao, Renato Gondim Sarmento pela compreens˜ao cotidiana, meus sinceros agradecimentos.
Ao Paulo Dantas Sesion J´unior pela for¸ca e humildade dispensados nas mais diversas situa¸c˜oes, obrigado pela presteza e atendimento imediato, sem modera¸c˜ao quando busquei seu aux´ılio.
de estudo.
Ao professor do Departamento de F´ısica da UESB Dr. Luiz Darcy e sua esposa Cristina, pelos ensinamentos e aten¸c˜ao.
A todos meus colegas que mesmo longe, ou de perto, incentivaram com a coragem em especial a Willian de Souza Santos, Walmir Belinato, Carla Machado e Ubiratan Correia Silva.
Aos meus familiares em especial a Lurdes Gondim Ferreira(tia) e fam´ılia e o carinho basilar de minha av´o Maria.
Aos amigos e colegas da p´os-gradua¸c˜ao Antˆonio de Macedo Filho, Maur´ıcio Lopes de Almeida, Reben Rudson Mendes Gomes, Gabriel Alves Mendes, Carlos Alexandre Amaral Ara´ujo e Leonardo Mafra Bezerril pela agrad´avel convivˆencia durante a trajet´oria desse mestrado.
A secret´aria da P´os-gradua¸c˜ao Celina Pinheiro pela aten¸c˜ao e prontid˜ao.
`
A CAPES pela concess˜ao da bolsa de mestrado.
Resumo
Abstract
´
Indice
Agradecimentos i
Resumo iii
Abstract iv
1
Introdu¸
c˜
ao
1
2
A mol´
ecula de DNA
4
2.1
Introdu¸c˜ao
. . . 42.2
Breve hist´orico dos aspectos bioqu´ımicos e f´ısicos do DNA
. . . 52.3
A estrutura da mol´ecula de DNA
. . . 102.4
A dupla h´elice do DNA
. . . 112.5
O a¸c´
ucar-fosfato
. . . 143
Densidade de estado do DNA de fita dupla
16
3.1Introdu¸c˜ao
. . . 163.2
Modelo te´orico
. . . 203.3
M´etodo da matriz transferˆencia
. . . 253.4
C´alculo da densidade de estado
. . . 323.5
Resultados num´ericos
. . . 434.2
Modelo t´eorico
. . . 52 4.3Resultados num´ericos
. . . 605
Conclus˜
ao
68
CAP´
ITULO 1
Introdu¸
c˜
ao
As c´elulas constituem a unidade b´asica dos seres vivos, elas funcionam cooperativa-mente como parte de um tecido, ´org˜ao ou, independentecooperativa-mente, como um microorganismo unicelular. Os diferentes tipos de c´elulas desempenham fun¸c˜oes espec´ıficas, visando a manuten¸c˜ao da vida no organismo.
As fun¸c˜oes vitais do organismo ocorrem dentro das c´elulas e nelas encontramos as informa¸c˜oes gen´eticas necess´arias que s˜ao transmitidas de uma gera¸c˜ao a outra. Na c´elula existe filamentos espiralados denominados cromossomos. Estes s˜ao constitu´ıdos de ´acido desoxirribonucl´eico, tendo a fun¸c˜ao de coordenar a atividade celular e transmitir as carac-ter´ısticas heredit´arias. H´a dois tipos de ´acidos nucl´eicos, que s˜ao: o Desoxirribo Nucleic Acid (DNA), ou ´acido desoxirribonucl´eico, formado pelo a¸c´ucar chamado desoxirribose. E o Ribo Nucleic Acid (RNA), ou ´acido ribonucl´eico, que ´e formado por outro a¸c´ucar, a ribose.
A mol´ecula do DNA foi uma das maiores descobertas da humanidade, tendo tanta importˆancia que ´e denominada a mol´ecula da vida, pois atrav´es dela conseguimos explicar como as informa¸c˜oes gen´eticas s˜ao copiadas e passadas da c´elula m˜ae para c´elula filha, como tamb´em o processo de muta¸c˜ao, entre outros [1].
regulado-ras ao articularem os aspectos evolutivos, gen´eticos e de desenvolvimento; organizando informa¸c˜ao codificada para s´ıntese de prote´ına. Cada gene ´e formado por uma sequˆencia espec´ıfica, mais ou menos longa, de ´acidos nucl´eicos [2].
A saga para desvendar os mist´erios do mecanismo desses ´acidos, em particular o DNA, proporcionou avan¸cos cient´ıficos, principalmente relacionados a sua estrutura, permitindo entender o funcionamento da vida e da perpetua¸c˜ao das esp´ecies [3]. A heran¸ca gen´etica tem sua base molecular nos processos precisos de replica¸c˜ao e transmiss˜ao do DNA, cons-tituindo desse modo as bases moleculares da heran¸ca gen´etica [4].
Os organismos vivos realizam processos f´ısicos e rea¸c˜oes bioqu´ımicas para seu fun-cionamento. Eles s˜ao regidos por instru¸c˜oes armazenadas em seu genoma - conjunto de genes de um organismo, popula¸c˜ao ou esp´ecie, onde o conte´udo primordial ´e o DNA [5].
O DNA ´e uma macromol´ecula de elevada massa molecular relativa, constitu´ıda de unidades estruturais que se repetem. Por exemplo, o cromossomo humano chamado Ch22 apresenta aproximadamente 6 mm de comprimento e 2×108 nucleot´ıdeos, permanecendo
constante em um volume igual a 500 m3 [6]. Essa macromol´ecula ´e mensurada em
na-noescala, por ter diˆametro de cerca de 2 nm e a curvatura da h´elice possui cerca de 3.4 a 3.6 nm [7]. A quantidade de informa¸c˜oes armazenadas no DNA ´e significante como objeto de pesquisa cient´ıfico-tecnol´ogico em diversas ´areas do conhecimento, pois essa caracter´ıstica da nanoestrutura do DNA permite trabalhos na ´area da Nanociˆencia e Nanotecnologia (N & N).
Desde o final do s´eculo passado, a ascens˜ao dos estudos relacionados ao DNA vem sendo marcada por descobertas intrigantes representadas pelos avan¸cos de pesquisas fascinantes a respeito da sua nanoestrutura, resultando em grandes progressos no estudo f´ısico e bioqu´ımico do DNA no campo da nanotecnologia.
trans-porte de el´etrons na cadeia do DNA ´e um tema intrigante para a comunidade cient´ıfica, j´a que ´e mencionada a possibilidade da constru¸c˜ao de dispositivos nanoeletrˆonicos. H´a estudos e medi¸c˜oes experimentais demonstrando diversos resultados, dentre eles o DNA ´e referido como um fio molecular, levando a uma nova gera¸c˜ao de dispositivos eletrˆonicos e computadores [8]. Muitas s˜ao as controv´ersias sobre a natureza f´ısica envolvendo a condu-tividade do DNA. Al´em disso, este aspecto ´e recente alvo de investiga¸c˜ao, representando um amplo tema para estudo no s´eculo XXI.
As possibilidades de aplica¸c˜ao do DNA na ´area da nanotecnologia tˆem sido sugeridas por ser ideal na fabrica¸c˜ao de dispositivos nanoeletrˆonicos, uma vez que o DNA pos-sui a caracter´ıstica de autoduplica¸c˜ao. Esta particularidade permite a produ¸c˜ao da sua r´eplica sem a necessidade de utilizar equipamentos, desse modo minimiza investimentos na tecnologia de nanofabrica¸c˜ao [9].
Nesta disserta¸c˜ao, analisamos a influˆencia do esqueleto a¸c´ucar-fosfato no transporte eletrˆonico em fitas duplas do DNA, estudando a densidade de estado e a transmitˆancia. Para isto, admitimos que o DNA segue as sequˆencias quasi-peri´odicas de Fibonacci e Rudin-Shapiro e em seguida comparamos com o cromossomo 22 do genoma humano ou Ch22.
CAP´
ITULO 2
A mol´
ecula de DNA
2.1
Introdu¸
c˜
ao
Nos seres vivos encontramos n´umeros distintos de cromossomos, caracterizando cada forma de vida. Essa distin¸c˜ao proporcionou o conhecimento do DNA como a principal fonte b´asica armazenadora da informa¸c˜ao gen´etica. Atualmente, o estudo dessa mol´ecula ´e relevante, trazendo benef´ıcios significativos em ´areas como: a f´ısica, a biom´edica, a engenharia gen´etica, agricultura e entre outras; sobretudo, favorecendo o progresso da Nanotecnologia [10]. O desenvolvimento da ciˆencia envolveu sucessivas etapas, com a rec´ıproca interdependˆencia de cientistas em suas pesquisas para o conhecimento dos seres vivos.
No s´eculo XIX, os estudos da gen´etica tiveram seu pioneirismo com o botˆanico austr´ıaco Gregor Mendel, em 1863, com os trabalhos dos genes das ervilhas. Posterior-mente, foram realizadas investiga¸c˜oes sobre a constitui¸c˜ao qu´ımica, a estrutura e o fun-cionamento dos genes. Assim, constatou-se o interesse em conhecer a heran¸ca gen´etica, atrav´es do estudo envolvendo o controle gen´etico dos caracteres [11].
f´ısico britˆanico Francis Crick revelaram a estrutura helicoidal do DNA. O conhecimento da estrutura ´ıntima do DNA representou um marco na biologia molecular moderna, tendo como consequˆencia, atualmente, algumas aplica¸c˜oes biotecnol´ogicas como os processos de clonagem, os organismos transgˆenicos e o Projeto Genoma Humano [5].
Na contemporaneidade, as controv´ersias cient´ıficas relacionadas ao desvendamento e as v´arias pesquisas do DNA remetem ao estudo tanto bioqu´ımico quanto f´ısico, envol-vendo o futuro dessa mol´ecula, levando-nos ao pr´ospero e atraente ramo nanotecnol´ogico. Portanto, neste cap´ıtulo procuramos apresentar um breve hist´orico descrevendo os con-ceitos bioqu´ımicos e f´ısicos envolvendo o DNA. Al´em disso, ser´a mencionada a influˆencia do esqueleto a¸c´ucar-fosfato nessa mol´ecula.
2.2
Breve hist´
orico dos aspectos bioqu´ımicos e f´ısicos do DNA
As investiga¸c˜oes cient´ıficas envolvendo a descoberta dos genes por Mendel at´e a di-vulga¸c˜ao da estrutura do DNA por Watson e Crick s˜ao importantes para compreendermos as etapas do conhecimento dessa mol´ecula. Brevemente, procuramos recapitular, a busca que levou ao desvendamento do DNA.
Em 1865, Mendel realizou pesquisas com ervilhas, relacionando as caracter´ısticas transmitidas em gera¸c˜oes sucessivas. As distintas caracter´ısticas estariam localizadas e seriam transmitidas em unidades heredit´arias. Os estudos dos genes das ervilhas per-maneceram por longo tempo ignorados pela comunidade cient´ıfica [3]. Por´em, ap´os a concep¸c˜ao da estrutura do DNA, foi necess´ario cerca de meio s´eculo a mais para serem descobertos os processos de decodifica¸c˜ao do DNA, a identifica¸c˜ao e reprodu¸c˜ao de genes isolados e, por fim, o Projeto Genoma Humano. Foi longa a jornada que levou a decifra¸c˜ao dessa mol´ecula polˆemica.
denominado nucle´ına. Prosseguindo os estudos, em 1889, seu disc´ıpulo Richard Altmann mudou o nome para ´acido nucl´eico [12].
No s´eculo XX continuaram as polˆemicas envolvendo o DNA. No ano de 1903, o bioqu´ımico russo-americano Phoebus Aaron Levene constatou que nem todos os ´acidos nucl´eicos continham ribose. Alguns continham um tipo de a¸c´ucar ao qual faltava um ´atomo de oxigˆenio, a desoxirribosa. Depois em 1910, progrediu a verifica¸c˜ao sobre o ´acido nucl´eico, detectando a presen¸ca da ribose estudada por Emil Fischer. Assim, constatou-se que havia, portanto, dois ´acidos nucl´eicos: o ribonucl´eico (RNA) e o desoxirribonucl´eico (DNA).
Nesse mesmo ano, Thomas Hunt Morgan provou que os genes estavam localizados nos cromossomos, o que confirmou a teoria gen´etica da hereditariedade. Outras contribui¸c˜oes foram os estudos sobre a heran¸ca ligada ao sexo, a recombina¸c˜ao dos fatores relacionada `a influˆencia dos cromossomos e o trabalho de pesquisa com a mosca da fruta (Drosophila melanogaster), demonstrando como os genes poderiam sofrer muta¸c˜oes ou serem altera-dos. No ano de 1913, Alfred Sturtevant da equipe de Morgan, iniciou o mapeamento dos cromossomos. E em 1915, Morgan, Sturtevant, Muller e Bridges publicaram The Mechanism of Mendelian Heredity (O Mecanismo da Hereditariedade Mendeliana). A continuidade das pesquisas na d´ecada de 1920 generalizou a id´eia de que o DNA era muito simples, formado de pequenas mol´eculas. Por esse motivo, Levene defendeu que essa simples mol´ecula n˜ao podia carregar o c´odigo gen´etico. Esses resultados, destacando a gen´etica, rendeu a Morgan o Prˆemio Nobel de Medicina, em 1933 [3].
devia-se ao fato das prote´ınas serem macromol´eculas com ampla heterogeneidade e par-ticularidade funcional. No entanto, os conhecimentos eram muito pouco sobre os ´acidos nucl´eicos, pois suas propriedades f´ısico-qu´ımicas eram constantes para codificar a dispari-dade de caracter´ısticas gen´eticas presentes nos seres vivos.
A no¸c˜ao de que o DNA ocorre em quantidade mais constante, seguido das prote´ınas e do RNA, entre as c´elulas do corpo de um indiv´ıduo, assegura que o n´umero de informa¸c˜oes deve ser constante nos indiv´ıduos da mesma esp´ecie. Assim, a dedu¸c˜ao de que o material gen´etico poderia ser o DNA acendeu d´uvida, que persistiu at´e o segundo meados do s´eculo XX.
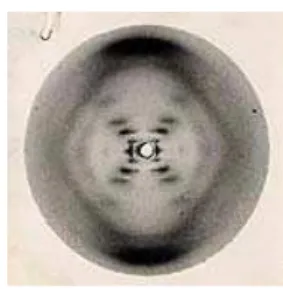
Nos anos 50 aconteceram novas investiga¸c˜oes sobre o DNA a n´ıvel molecular, destacando-se os estudos dos cientistas James Watson e Francis Crick sobre a dupla h´elice do DNA [14]. Contudo, a coopera¸c˜ao cient´ıfica entre outros pesquisadores foi relevante nos estudos do DNA. Os trabalhos paralelos, mas concomitante de cientistas como: Pauling no Institute of Technology na Calif´ornia; Maurice Wilkins e Rosalind Franklin no King’s College em Londres e Watson e Crick na Cambridge University, implicaram no promissor conhecimento do DNA. A contribui¸c˜ao crucial no desvendamento da estrutura do DNA foram `as difra¸c˜oes de raios-X e as medi¸c˜oes desta amostra realizada por Rosalind Franklin [15], ver Fig. 2.1.
Figura 2.1: Imagem do raios-X do DNA.
teoria da evolu¸c˜ao de Darwin e do estudo gen´etico de Mendel [16].
Watson e Crick restringiram-se a correta interpreta¸c˜ao de resultados obtidos por ou-tros pesquisadores na concep¸c˜ao da estrutura ao construir um modelo de DNA de arame e metal, com ˆangulos e dimens˜oes em escala dos espa¸cos interatˆomicos de um segmento de DNA. A imagina¸c˜ao para construir esse modelo foi inspirada no trabalho do renomado qu´ımico americano Linus Pauling ao propor a estruturaα-h´elice como base da estrutura secund´aria das prote´ınas, a qual resultaria do enrolamento dos amino´acidos numa aco-moda¸c˜ao helicoidal. A estrutura apresentava trˆes h´elices, no centro da fibra, unidas por ´ıons de magn´esio, e as bases nitrogenadas, no exterior. A aprecia¸c˜ao que o DNA n˜ao tinha estrutura de h´elice foi rejeitada pelos pesquisadores Wilkins, Franklin e Gosling que desaprovaram o modelo proposto, j´a que n˜ao existiam ´ıons no centro da fibra e os fos-fatos das h´elices n˜ao poderiam ficar juntos porque se repeliriam. Ap´os essa confronta¸c˜ao aconteceram v´arias controv´ersias cient´ıficas sobre constru¸c˜ao do modelo da estrutura do DNA em raz˜ao dos desentendimentos [3]. Pauling principiou analisar o DNA com base na c´opia de imagens mais antigas da difra¸c˜ao dos raios-X, esfor¸cando para findar sua pesquisa sobre o DNA, propˆos uma estrutura helicoidal de trˆes h´elices [17], similar ao primeiro modelo de Watson e Crick.
Os pesquisadores Watson e Crick tinham interesse m´utuo em estudar o DNA, pois constru´ıram modelos em dupla h´elice em conformidade com os dados de cristalografia de raios-X e com a qu´ımica conhecida do DNA. Em uma de suas tentativas sem ˆexito, eles colocaram a cadeia de a¸c´ucar-fosfato no interior da mol´ecula. Em outro ensaio, Watson colocou a cadeia de a¸c´ucar-fosfato no lado exterior da mol´ecula, o que permitiu que as bases nitrogenadas mais hidrof´obicas se abrigassem no interior da mol´ecula, distante do meio aquoso.
Em 1962, o bi´ologo James Watson, o f´ısico Francis Crick e o cristal´ografo de raios-X Maurice Wilkins foram agraciados com o Prˆemio Nobel de Medicina, pela pesquisa que revelou a estrutura do DNA, sendo tamb´em de fundamental importˆancia e reconhecido os estudos de Franklin, que ´e mencionada pelos cientistas em seus livros [3]. ´E not´orio que Watson e Crick trabalharam juntos no in´ıcio de suas carreiras na Universidade de Cambridge, aliando seus recursos interdisciplinares com o franco objetivo de ganhar o Prˆemio Nobel, solucionando o mist´erio do DNA. Eles descobriram a estrutura do DNA, a mol´ecula-chave respons´avel pelo milagre da diversidade de seres vivos, embora origina-dos pelo mesmo processo gen´etico. Estes pesquisadores ainda afirmavam existir muitas permuta¸c˜oes numa mol´ecula longa e, portanto, parece admiss´ıvel que o sequenciamento preciso das bases constitui o c´odigo que cont´em a informa¸c˜ao gen´etica, destacando tamb´em que a estrutura proposta deve ser confirmada pela experimenta¸c˜ao [19].
O descobrimento da estrutura do DNA sinaliza a nova era dos estudos biotecnol´ogicos, pois foi poss´ıvel desvendar o c´odigo gen´etico. Os avan¸cos dos estudos estruturais das prote´ınas por meio da t´ecnica de cristalografia de raios-X proporcionaram o desvenda-mento da estrutura do DNA e o arranjo covalente do pol´ımero de ´acido nucl´eico.
conhecimento desta nanoestrutura. Atualmente, a pesquisa nanotecnol´ogica ´e fundamen-talmente multidisciplinar envolvendo ´areas como: a F´ısica, a Biologia, a Medicina, as Engenharias, a Qu´ımica, a Inform´atica e entre outras.
2.3
A estrutura da mol´
ecula de DNA
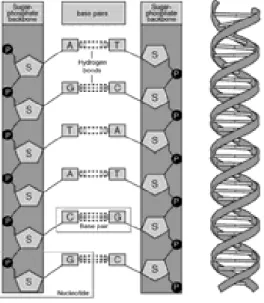
O DNA ´e um pol´ımero formado por seis componentes: uma mol´ecula de a¸c´ucar, um grupamento fosfato e quatro blocos estruturais denominados bases nitrogenadas: a adeni-na, a timiadeni-na, a citosina e a guanina (ver Fig. 2.2). Deste modo, constituem um alfabeto de quatro letras, respectivamente A, T, C e G, codificando a estrutura prim´aria das prote´ınas. As quatro bases do DNA tamb´em podem ser colocadas em uma ordem ´unica para cada ser vivo. O esclarecimento deste c´odigo foi significativo para o desvendamento do DNA. A constitui¸c˜ao da mol´ecula de DNA por uma sequˆencia de nucleot´ıdeos - composto equinolar de uma pentose, um grupo fosfato e uma base (p´urica ou pirim´ıdica) permite a orienta¸c˜ao das liga¸c˜oes entre as trˆes mol´eculas constituintes dos nucleot´ıdeos.
Figura 2.2: Duas sequˆencias complementares de DNA.
atrav´es do grupamento fosfato, s˜ao chamadas liga¸c˜oes fosfodi´ester. Como os nucleot´ıdeos s˜ao unidos por liga¸c˜oes entre seus grupamentos a¸c´ucar e fosfato, frequentemente ´e dito que o DNA possui um esqueleto de a¸c´ucar-fosfato, veja Fig. 2.2.
2.4
A dupla h´
elice do DNA
A coloca¸c˜ao de modelos tridimensionais foi essencial no processo da descoberta da estrutura, esse aspecto formal do modelo pode facilitar a compreens˜ao de v´arios fenˆomenos relacionados ao funcionamento do DNA [19] . A estrutura molecular da dupla h´elice do DNA, proposta por Watson e Crick, fornece que o relacionamento entre seus componentes possibilita o transporte eletrˆonico entre as bases da dupla h´elice [20]. Os pares de bases formados eram desuniformes e a dupla h´elice tinha um diˆametro muito vari´avel [18].
Portanto, torna-se importante o conhecimento estrutural de seus componentes rela-cionados ao funcionamento celular que envolve complementaridade e antiparalelismo da fitas do DNA. Algumas caracter´ısticas do DNA s˜ao facilmente representadas e outras de-mandam esquemas mais elaborados e maior esfor¸co de abstra¸c˜ao para compreens˜ao de sua representa¸c˜ao. Segundo o modelo formulado por Watson e Crick, o DNA ´e constitu´ıdo por duas fitas enroladas para a direita, formando a chamada dupla h´elice. Analogamente, a estrutura do DNA pode ser comparada a uma escada espiralada com dois degraus, sendo que os degraus correspondem `as liga¸c˜oes repetidas de a¸c´ucar-fosfato ao longo de todo o comprimento da mol´ecula.
O grupo fosfato conecta-se com pentose atrav´es de uma liga¸c˜ao fosfodi´ester com a hidroxila ligada ao carbono-5 da pentose. Para a forma¸c˜ao do DNA, ´e necess´ario que ocorra a liga¸c˜ao entre os nucleot´ıdeos, estes formam entre si pontes de fosfato, atrav´es de liga¸c˜oes fosfodi´ester. Essa circunstˆancia determina a forma¸c˜ao da cadeia de DNA. Isto motiva que o crescimento do DNA se fa¸ca na dire¸c˜ao de 5’ para 3’, como demonstra a Fig. 2.3. Assim, s˜ao feitas as liga¸c˜oes entre os nucleot´ıdeos formando a fita de DNA.
Figura 2.3: Representa¸c˜ao da estrutura helicoidal do DNA.
Estudos mostram que existem duas formas de DNA com a h´elice girando para a direita, chamadas A-DNA e B-DNA, e uma outra forma que gira para a esquerda chamada Z-DNA, ver Fig. 2.4.
A - DNA B – DNA Z - DNA
Figura 2.4: Estrutura A-, B- e Z-DNA.
h´elice. Assim, podemos distinguir que: B-DNA tem a dupla h´elice mais longa e mais fina. Para completar uma volta na h´elice s˜ao necess´arios 10 pares de bases. Enquanto, A-DNA possui a forma mais curta e mais grossa. Para completar uma volta na h´elice s˜ao necess´arios 11 pares de bases. Geralmente, o DNA assume a acomoda¸c˜ao B. Quando h´a pouca ´agua dispon´ıvel para interagir com a dupla h´elice, o DNA assume a acomoda¸c˜ao A-DNA. O Z-DNA difere das duas anteriores, j´a que esta configura¸c˜ao ´e mais alongada e mais fina do que o B-DNA. Para completar uma volta na h´elice s˜ao necess´arios 12 pares de bases. O DNA em solu¸c˜ao com altas concentra¸c˜oes de c´ations, assume a conforma¸c˜ao Z-DNA [23].
2.5
O a¸
c´
ucar-fosfato
Como sabemos o a¸c´ucar-fosfato ´e um componente constante nos nucleot´ıdeos com a fun¸c˜ao unicamente estrutural, sendo considerado o esqueleto na mol´ecula de DNA. Para compreender a influˆencia da camada a¸c´ucar-fosfato nas propriedades de transporte no DNA, ´e interessante conceber o a¸c´ucar-fosfato como um suporte, composto alternada-mente de a¸c´ucar e fosfato onde ambos s˜ao polares. O a¸c´ucar-fosfato ´e componente na arma¸c˜ao da nanoestrutura do DNA, definindo tamb´em o sentido da fita. Deste modo, o DNA pode ser dividido estruturalmente em duas por¸c˜oes: o a¸c´ucar-fosfato e as bases nitrogenadas [5].
O conhecimento da sequˆencia de bases em uma fita nos permite deduzir a sequˆencia de bases na fita complementar conforme demonstra na Fig. 2.3. O dobramento em h´elice da mol´ecula de DNA facilita observar a acomoda¸c˜ao ocupada pelo esqueleto a¸c´ucar-fosfato externamente na dupla h´elice, apresentando as bases nitrogenadas no interior.
Esse delineamento dos aspectos f´ısico-qu´ımicos decorrentes da composi¸c˜ao do DNA permite apontar as caracter´ısticas hidrof´ılicas do fosfodi´ester pela presen¸ca das pentoses e o car´ater ´acido conferido pelo grupo fosfato. A solubilidade externa do DNA relacionada com meio hidrof´ılico, ou seja, o esqueleto fosfodi´ester e a neutraliza¸c˜ao das regi˜oes ele-tricamente saturadas atrav´es de pareamentos entre as bases (A-T e C-G) permitiram a existˆencia da regi˜ao hidrof´obica, sem cargas el´etricas livres, localizada no interior do DNA [25].
vez que ele tem a capacidade de automontagem. Isto fornece uma vantagem na produ¸c˜ao de dispositivos nanoeletrˆonicos [8], onde tal possibilidade instiga in´umeros trabalhos de pesquisadores delineando a era da nanoestruturas [9].
CAP´
ITULO 3
Densidade de estado do DNA de fita dupla
3.1
Introdu¸
c˜
ao
A mol´ecula de DNA transporta informa¸c˜ao gen´etica e ´e altamente interessante, pois na classe dos biopol´ımeros ela desempenha um papel importante na eletrˆonica molecular [26]. Isto, deve-se a sua propriedade de auto-replica¸c˜ao e auto-reconhecimento, que s˜ao essenciais para o seu desempenho como transportador do c´odigo gen´etico. Na perspec-tiva cient´ıfica, espera-se que estas propriedades sejam melhor aproveitadas na ´area da nanotecnologia, visando a constru¸c˜ao de circuitos eletrˆonicos [27, 28].
A importˆancia do DNA e as perspectivas que esta mol´ecula pode proporcionar na ´area de nanotecnologia, como a produ¸c˜ao de nanofios [29], ´e devido a sua propriedade de auto-replica¸c˜ao. Por sua vez, h´a grupos de pesquisadores em v´arios pa´ıses interessados em realizar experimentos sobre a condutividade da mol´ecula de DNA. Contudo, os estudos j´a realizados sobre a condutividade da mol´ecula do DNA apresentam alguns resultados contradit´orios, descrevendo o DNA como isolante [30, 31], semicondutor [30], condutor [32, 33] ou supercondutor [34, 35] . A seguir mencionaremos alguns experimentos sobre esses aspectos.
constatado que a mol´ecula se comportava como um condutor de resistˆencia de 2.5 MΩ. Isto corresponde uma resistividade 1mΩ·cm. Neste caso, no qual o DNA comporta-se como um condutor, este resultado tr´as a possibilidade de utiliza¸c˜ao da mol´ecula de DNA em dispositivos nanobioeletrˆonicos.
Em 2000, Porath [30], continuando os estudos sobre a propriedade eletrˆonica da mol´ecula, utilizou o DNA sint´etico poly(dG)poly(dC) como uma sequˆencia de bases peri´odicas. Ao colocar a mol´ecula de DNA de 8 nm entre os eletrodos de platina, mediu-se a condu¸c˜ao el´etrica `a press˜ao atmosfera e no v´acuo (com valor de 10−6
Torr). Foi observado um gap em torno de 2V na curva da corrente-tens˜ao em ambos ambientes, mostrando que a mol´ecula de DNA pode ser isolante [31].
No mesmo ano, um grupo da Universidade de Tecnologia de Delft, na Holanda, di-vulgou um trabalho apontando que o DNA se comporta como isolante [30]. Utilizando um m´etodo denominado de “armadilha eletrost´atica”, aprisionou-se o DNA de fita dupla poly(dG)poly(dC), em torno de 30 pares de bases, entre dois nanoeletrodos met´alicos. Ao submetˆe-lo a uma tens˜ao de 1V, observou-se uma corrente el´etrica no dispositivo. Os resultados indicaram que este DNA sint´etico equivale a um semicondutor degap largo.
Em 2001, A. Yu. Kasumov e seu grupo [34, 35] colocaram o DNA entre eletrodos en-volvidos por uma camada de 2 nm de rˆenio (material supercondutor) e carbono, separados por uma distˆancia de 0,5 mµ, a uma temperatura da ordem de 1K. Ao realizar a medida, foi observado que o DNA apresentava uma resistˆencia inferior a 100 KΩ, o que mostra um efeito de proximidade (EP), ou seja, um comportamento pr´oximo a um supercondutor surge em um condutor normal quando ligamos o DNA aos eletrodos supercondutores. Portanto, a an´alise do EP em uma mol´ecula de DNA fornece que estas mol´eculas s˜ao condutoras e que possuem uma baixa resistˆencia em contato com os eletrodos supercon-dutores.
a interface eletrodo-DNA, a sequˆencia de nucleot´ıdeos da cadeia do DNA, entre outros. Desta forma, podemos designar o DNA como isolante, semicondutor, condutor ou super-condutor. Estes resultados d˜ao uma perspectiva sobre as aplica¸c˜oes bionanotecnol´ogicas na cria¸c˜ao de dispositivos nanoeletrˆonicos de DNA.
Uma aplica¸c˜ao na biotecnologia ´e o chip de DNA, que se trata de um biochip con-tendo milhares de segmentos de DNA ordenados, os quais permitem an´alises simultˆaneas de milhares de marcadores gen´eticos ou sequˆencias DNA (c´opia de DNA). O chip tem duas grandes aplica¸c˜oes: uma ´e a identifica¸c˜ao da sequˆencia (gene/muta¸c˜ao de gene) e a outra ´e a determina¸c˜ao do n´ıvel de express˜ao dos genes. A sua utiliza¸c˜ao se estende nas ´areas da biologia molecular e na medicina [36]. A leitura ´e feita opticamente, mas se alme-jarmos miniaturizar ainda mais esse chip, teria outra forma de leitura a ser desenvolvida, possivelmente, envolvendo a condu¸c˜ao de el´etrons no DNA. Todas as caracter´ısticas e pro-priedades j´a apresentadas fazem dele um dos mais promissores dispositivos na eletrˆonica molecular [37]-[39].
Na literatura, os modelos te´oricos descritos para investigar as propriedades na-noeletrˆonicas do DNA tˆem se tornado mais ousados no intuito de aproximar cada vez mais do caso real. Vamos a seguir comentar alguns trabalhos.
termicamente induzido se torna o mecanismo de transporte dominante [41, 42].
Em 2006, B.P.W. de Oliveira et al.[43] investigou o espectro de densidade de es-tados para estrutura quasi-peri´odica formadas pelas bases nitrogenadas, seguindo duas sequˆencias, a de Fibonacci e a de Rudin-Shapiro. Em seu formalismo matem´atico utilizou o Hamiltoniano tight-binding e a equa¸c˜ao de Dyson, tendo como mecanismo de trans-porte o hopping termicamente induzido. O nosso modelo tamb´em determina o espectro da densidade de estados do DNA, por´em apresenta uma componente a mais, que ´e o grupo a¸c´ucar-fosfato que ser´a apresentado mais adiante. Este trabalho cient´ıfico [43] servir´a de compara¸c˜ao com o nosso modelo e perceber-se-´a que a influˆencia do grupo a¸c´ucar-fosfato apresenta resultados surpreendentes.
Recentemente, alguns pesquisadores tˆem considerado o grupo a¸c´ucar-fosfato, junta-mente com as bases nitrogenadas, em seus modelos, tendo utilizado a t´ecnica de renor-maliza¸c˜ao [44]-[45], geralmente. Essa t´ecnica n˜ao utiliza a liga¸c˜ao entre os grupos a¸c´ ucar-fosfatos, j´a que esta liga¸c˜ao ´e pequena em rela¸c˜ao `a base nitrogenada [46, 47]. Desta maneira, o transporte d´a-se principalmente pelas bases empilhadas ao longo da cadeia.
Em 2006, E. Maci´a [44] investigou analiticamente o espectro de energia e a condutˆancia de Landauer [48] para uma estrutura quasi-peri´odica, seguindo a sequˆencia de Fibonacci, e comparou com a sequˆencia peri´odica do polyGACT-polyCTGA, aplicando a t´ecnica de renormaliza¸c˜ao duas vezes para o DNA de dupla fita (incluindo o grupo a¸c´ucar-fosfato). Neste trabalho, o espectro de energia aparece fragmentado devido ao efeito de ordem da estrutura quasi-peri´odica e uma s´erie de picos de alta condutˆancia surge no espectro de transmiss˜ao da sequˆencia de Fibonacci, indicando a existˆencia de estados estendidos nos sistemas.
corrente-voltagem. Entretanto, ´e relevante destacar que diferentes circunstˆancias podem afetar a liga¸c˜ao entre a estrutura do a¸c´ucar-fosfato e o nucleot´ıdeo em condi¸c˜oes realis-tas. Estes resultados abriram novas perspectivas para trabalhos experimentais, visando controlar a transferˆencia de cargas em nanodispositivos baseado em DNA sint´etico.
Neste cap´ıtulo, iremos estudar as propriedades eletrˆonicas da cadeia de DNA de fita dupla, assumindo a influˆencia do a¸c´ucar-fosfato e fazendo considera¸c˜oes te´oricas sobre sua densidade de estados, considerando um Hamiltoniano do tipo tight-binding dentro do formalismo da equa¸c˜ao de Dyson [50]-[52], que detalharemos a seguir.
3.2
Modelo te´
orico
No modelo que ser´a apresentado, os ´atomos do substrato (S), que comp˜oem o eletrodo no qual a mol´ecula de DNA ´e depositada, podem ser representados pictoricamente como mostra a Fig. 3.1. Esta estrutura foi escolhida pela simplicidade de sua simetria que pos-sibilita determinar os elementos da matriz transferˆencia, que ter´a um papel importante no nosso modelo. Nesta se¸c˜ao e na pr´oxima, vamos estudar uma parte do modelo que corres-ponde ao eletrodo. Para descrever matematicamente esta parte do modelo, utilizaremos o Hamiltoniano tight-binding, ou seja,
H = ω1n|n,1>< n,1|+ω2n|n,2>< n,2|+ω3n|n,3>< n,3|+ω4n|n,4>< n,4| + V12nn[|n,1>< n,2|+|n,2>< n,1|] +V23nn[|n,2>< n,3|+|n,3>< n,2|] + V34nn[|n,3>< n,4|+|n,4>< n,3|] +V41nn[|n,4>< n,1|+|n,4>< n,1|]
+
δ=±1
[V11n,n+δ|n,1>< n+δ,1|+V44n,n+δ|n,4>< n+δ,4|]
+
δ=±1
[V22n,n+δ|n,2>< n+δ,2|+V33n,n+δ|n,3>< n+δ,3|],
onde os ´ındices 1, 2, 3 e 4 correspondem a cada linha da estrutura, V11, V22, V33 e V44
correspondem aos potenciais de hopping entre s´ıtios de mesma linha, V12, V23, V34 e V41
entre s´ıtios de linhas diferentes e ωn
V11
V22
V33 V44 V12
V23
V41
V34
S S S
S
S
S
S
S
S
S
S
S
...
...
...
...
V11
V12
V41
V44
V41 V22
V33
V23 V23
V34 V34
Figura 3.1: O modelo pict´orico do substrato, destacando as liga¸c˜oes c´ıclicas entre os ´atomos que comp˜oem o eletrodo.
Usando o Hamiltoniano na equa¸c˜ao de Dyson
(ω−H)G(ω) = 1. (3.1)
Temos que
< lα|(ω−H)G(ω)|mβ >=< lα|mβ >=δlmδαβ. (3.2)
Agora, escrevendo a rela¸c˜ao de completeza Σl′α′|l
′
α′
>< l′
α′
|= 1. (3.3)
E substituindo a Eq. (3.4) na Eq. (3.3), obtemos
Σl′α′ < lα|(ω−H)|l
′
α′ >< l′α′|G|mβ >=δlmδαβ. (3.4)
Portanto
ω < lα|G|mβ >−Σl′α′ < lα|H|l
′
α′
>< l′
α′
Aplicando o Hamiltoniano, temos
< lα|H|l′
α′
>=< lα|ωn1|n,1>< n,1|l′
α′
>+< lα|ω2n|n,2>< n,2|l′
α′
>
+< lα|ω3n|n,3>< n,3|l′
α′
>+< lα|ωn4|n,4>< n,4|l′
α′
>
+< lα|V12n,n|n,1>< n,2|l′
α′
>+< lα|V12n,n|n,2>< n,1|l′
α′
>
+< lα|V23n,n|n,2>< n,3|l′α′ >+< lα|V23n,n|n,3>< n,2|l′α′ >
+< lα|V34n,n|n,3>< n,4|l
′
α′ >+< lα|V34n,n|n,4>< n,3|l
′
α′ >
+< lα|V41n,n|n,4>< n,1|l
′
α′ >+< lα|V41n,n|n,1>< n,4|l
′
α′ >
+< lα|V11n,n+1|n,1>< n+ 1,1|l′
α′
>+< lα|V11n,n−1|n,1>< n−1,1|l′
α′
>
+< lα|V22n,n+1|n,2>< n+ 1,2|l′
α′
>+< lα|V22n,n−1|n,2>< n−1,2|l′
α′
>
+< lα|V33n,n+1|n,3>< n+ 1,3|l′
α′
>+< lα|V33n,n−1|n,3>< n−1,3|l′
α′
>
+< lα|V44n,n+1|n,4>< n+ 1,4|l′
α′
>+< lα|V44n,n−1|n,4>< n−1,4|l′
α′
> .
(3.6)
Assim, o segundo termo do lado esquerdo da Eq. (3.6) ´e dado por
Σl′α′ < lα|H|l
′
α′
>< l′
α′
|G|mβ >=
+< lm|ωn1|n,1>< n,1|G|mβ >+< lm|ωn2|n,2>< n,2|G|mβ >
+< lm|ωn3|n,3>< n,3|G|mβ >+< lm|ωn4|n,4>< n,4|G|mβ >
+< lm|V12n,n|n,1>< n,2|G|mβ >+< lm|V12n,n|n,2>< n,1|G|mβ >
+< lm|V23n,n|n,2>< n,3|G|mβ >+< lm|V
n,n
23 |n,3>< n,2|G|mβ >
+< lm|V34n,n|n,3>< n,4|G|mβ >+< lm|V
n,n
34 |n,4>< n,3|G|mβ >
+< lm|V41n,n|n,4>< n,1|G|mβ >+< lm|V41n,n|n,1>< n,4|G|mβ >
+< lm|V11n,n+1|n+ 1,1>< n,1|G|mβ >+< lm|V11n,n−1|n,1>< n−1,1|G|mβ >
+< lm|V22n,n+1|n+ 1,2>< n,2|G|mβ >+< lm|V22n,n−1|n,2>< n−1,2|G|mβ >
+< lm|V33n,n+1|n+ 1,3>< n,3|G|mβ >+< lm|V33n,n−1|n,3>< n−1,3|G|mβ >
+< lm|V44n,n+1|n+ 1,4>< n,4|G|mβ >+< lm|V44n,n−1|n,4>< n−1,4|G|mβ > .
Definindo
< lα|G|mβ >=Gαβlm. (3.8)
Com a nova defini¸c˜ao a Eq. (3.8), assume a forma Σl′α′ < lα|H|l
′
α′
>< l′
α′
|G|mβ >= +ωn
1 < lα|n,1> G1n,mβ +ω n
2 < lα|n,2> G2nmβ
+ωn
3 < lα|n,3> G3n,mβ +ω n
4 < lα|n,4> G4nmβ
+V12n,n < lα|n,1> G2β n,m+V
n,n
12 < lα|n,2> G1nmβ
+V23n,n < lα|n,2> G3β n,m+V
n,n
23 < lα|n,3> G2nmβ
+V34n,n < lα|n,3> G4β n,m+V
n,n
34 < lα|n,4> G3nmβ
+V41n,n < lα|n,4> G1n,mβ +V n,n
41 < lα|n,1> G4
β nm
+V11n,n+1 < lα|n,1> G1nβ+1,m+V n,n−1
11 < lα|n,1> G 1β n−1,m +V22n,n+1 < lα|n,2> G2nβ+1,m+V
n,n−1
22 < lα|n,2> G 2β n−1,m +V33n,n+1 < lα|n,3> G3nβ+1,m+V
n,n−1
33 < lα|n,3> G 3β n−1,m +V44n,n+1 < lα|n,4> G4nβ+1,m+V
n,n−1
44 < lα|n,4> G 4β
n−1,m. (3.9)
Reescrevendo a Eq. (3.10), temos a seguinte forma
Σl′α′ < lα|H|l
′
α′
>< l′
α′
|G|mβ >= +ω1nδlmδα1G1n,mβ +ω
n
2δlmδα2G2nmβ
+ω3nδlmδα3G3n,mβ +ω n
4δlmδα4G4nmβ
+V12n,nδlmδα1G2n,mβ +V n,n
12 δlmδα2G1n,mβ
+V23n,nδlmδα2G3n,mβ +V n,n
23 δlmδα3G2n,mβ
+V34n,nδlmδα3G4n,mβ +V n,n
34 δlmδα4G3n,mβ
+V41n,nδlmδα4G1n,mβ +V n,n
41 δlmδα1G4n,mβ
+V11n,n+1δlmδα1G1nβ+1,m+V n,n−1
+V22n,n+1δlmδα2G2nβ+1,m+V n,n−1
22 δlmδα2G2nβ−1,m +V33n,n+1δlmδα3G3nβ+1,m+V
n,n−1
33 δlmδα3G3nβ−1,m +V44n,n+1δlmδα4G4nβ+1,m+V
n,n−1
44 δlmδα4G4nβ−1,m. (3.10)
A equa¸c˜ao acima ´e diferente de zero se l =m, isto ´e Σl′α′ < lα|H|l
′
α′
>< l′
α′
|G|mβ >= +ωn
1δα1G1n,mβ +ωn2δα2G2nmβ
+ω3nδα3G3n,mβ +ω n
4δα4G4nmβ
+V12nnδα1G2n,mβ +V nn
12 δα2G1nmβ
+V23nnδα2G3n,mβ +V nn
23 δα3G2nmβ
+V34nnδα3G4n,mβ +V nn
34 δα4G3nmβ
+V41nnδα4G1n,mβ +V nn
41 δα1G4nmβ
+V11n,n+1δα1G1nβ+1,m+V n,n−1
11 δα1G1nβ−1,m +V22n,n+1δα2G2nβ+1,m+V
n,n−1
22 δα2G2nβ−1,m +V33n,n+1δα3G3nβ+1,m+V
n,n−1
33 δα3G3nβ−1,m +V44n,n+1δα4G4nβ+1,m+V
n,n−1
44 δα4G4nβ−1,m. (3.11)
Substituindo as Eqs. (3.12) e (3.9) na Eq. (3.6), obtemos a seguinte equa¸c˜ao
ωGαβlm−ωn
1δα1G1nmβ −ω n
2δα2G2nmβ −ωn
3δα3G3nmβ −ω4nδα4G4nmβ −Vnn
12 δα1G2n,mβ −V12nnδα2G1nmβ −V23nnδα2G3n,mβ −V
nn
23 δα3G2nmβ −V34nnδα3G4n,mβ −V
nn
34 δα4G3nmβ −V41nnδα4G1n,mβ −V
nn
41 δα1G4nmβ −V11n,n+1δα1G1nβ+1,m−V
n,n−1
11 δα1G1nβ−1,m
−V22n,n+1δα2G2nβ+1,m−V n,n−1
22 δα2G2nβ−1,m
−Vn,n+1δα3G3β −Vn,n
−1
−V44n,n+1δα4G4nβ+1,m−V n,n−1
44 δα4G4nβ−1,m=δlmδαβ. (3.12)
3.3
M´
etodo da matriz transferˆ
encia
Nesta se¸c˜ao, procuramos determinar os elementos da matriz transferˆencia, utilizando a condi¸c˜ao de contorno c´ıclico ou Born-Von Karman para uma cadeia pura (material S), ver Fig. 3.1, onde obtemos o conjunto de equa¸c˜oes:
a) Considerando m= 0 e l = 0, encontramos que i) Para α=β = 1:
ωG1100−ω1SG1100−V12SSG2100−V41SSG4100−V11SSG1110−V11SSG11−10 = 1. (3.13)
ii) Para α= 1 e β = 2:
ωG1200−ω1SG1200−V12SSG2200−V41SSG4200−V11SSG1210−V11SSG12−10 = 0. (3.14)
iii) Para α= 1 e β = 3:
ωG1300−ω1SG1300−V12SSG2300−V41SSG4300−V11SSG1310−V11SSG13−10 = 0. (3.15)
iv) Paraα = 1 e β= 4:
ωG1400−ω1SG1400−V12SSG2400−V41SSG4400−V11SSG1410−V11SSG14−10 = 0. (3.16)
ωG2100−ω2SG2100−V12SSG1100−V23SSG3100−V22SSG2110−V22SSG21−10 = 0. (3.17)
vi) Paraα = 2 e β= 2:
ωG2200−ω2SG2200−V12SSG1200−V23SSG3200−V22SSG2210−V22SSG22−10 = 1. (3.18)
vii) Para α = 2 eβ = 3:
ωG2300−ω2SG2300−V12SSG1300−V23SSG3300−V22SSG2310−V22SSG23−10 = 0. (3.19)
viii) Para α= 2 e β = 4:
ωG2400−ωS
2G2400−V12SSG1400−V23SSG3400−V22SSG2410−V22SSG24−10 = 0. (3.20)
ix) Paraα = 3 e β= 1:
ωG3100−ωS
3G3100−V23SSG2100−V34SSG4100−V33SSG3110−V33SSG31−10 = 0. (3.21)
x) Para α= 3 e β = 2:
ωG3200−ωS
3G3200−V23SSG2200−V34SSG4200−V33SSG3210−V33SSG32−10 = 0. (3.22)
ωG3300−ω3SG3300−V23SSG2300−V34SSG4300−V33SSG3310−V33SSG33−10 = 1. (3.23)
xii) Para α = 3 eβ = 4:
ωG34
00−ω3SG3400−V23SSG2400−V34SSG4400−V33SSG3410−V33SSG34−10 = 0. (3.24)
xiii) Para α= 4 e β = 1:
ωG4100−ω4SG4100−V34SSG3100−V41SSG1100−V44SSG4110−V44SSG41−10 = 0. (3.25)
xiv) Para α= 4 e β = 2:
ωG4200−ω4SG4200−V34SSG3200−V41SSG1200−V44SSG4210−V44SSG42−10 = 0. (3.26)
xv) Para α= 4 e β = 3:
ωG4300−ω4SG4300−V34SSG3300−V41SSG1300−V44SSG4310−V44SSG43−10 = 0. (3.27)
xvi) Para α= 4 e β = 4:
ωG4400−ω4SG4400−V34SSG3400−V41SSG1400−V44SSG4410−V44SSG44−10 = 1. (3.28)
Glm = ⎡ ⎢ ⎢ ⎢ ⎢ ⎢ ⎢ ⎣ G11
lm G12lm G13lm G14lm
G21
lm G22lm G23lm G24lm
G31
lm G32lm G33lm G34lm
G41
lm G42lm G43lm G44lm
⎤ ⎥ ⎥ ⎥ ⎥ ⎥ ⎥ ⎦ , (3.29)
KS =
⎡ ⎢ ⎢ ⎢ ⎢ ⎢ ⎢ ⎣
ω−ωS
1 −V12SS 0 −V14SS
−VSS
12 ω−ωS2 −V23SS 0
0 −VSS
23 ω−ωS3 −V34SS
−VSS
14 0 −V34SS ω−ωS4
⎤ ⎥ ⎥ ⎥ ⎥ ⎥ ⎥ ⎦ , (3.30) e
LSS =
⎡ ⎢ ⎢ ⎢ ⎢ ⎢ ⎢ ⎣
−VSS
11 0 0 0
0 −VSS
22 0 0
0 0 −VSS
33 0
0 0 0 −VSS
44 ⎤ ⎥ ⎥ ⎥ ⎥ ⎥ ⎥ ⎦ , (3.31)
sendo Glm a matriz com elementos da fun¸c˜ao de Green, KS ´e a matriz com os elementos
das energias dos s´ıtios e das energias de intera¸c˜oes entre duas linhas e LSS ´e a matriz
dos potenciais de hopping para s´ıtios localizados em uma mesma linha. Desta maneira, podemos escrever as Eqs. (3.14)-(3.29) em forma de uma equa¸c˜ao matricial:
KSG00+LSSG−10+LSSG10 =I. (3.32)
Agora, fazemos o mesmo procedimento para m = 0 el = 1, temos
KSG10+LSSG00+LSSG20= 0. (3.33)
A matriz de transferˆencia para o sistema ´e dada por:
Substituindo a Eq. (3.35) na Eq. (3.34), temos
KST G00+LSSG00+LSST2G00= 0, (3.35)
que podemos escrever da forma:
KS+LSS(T
−1
+T) = 0. (3.36)
Sendo que a matriz transferˆencia ´e definida como
T = ⎡
⎢ ⎢ ⎢ ⎢ ⎢ ⎢ ⎣
T11 T12 T13 T14
T21 T22 T23 T24
T31 T32 T33 T34
T41 T42 T43 T44
⎤
⎥ ⎥ ⎥ ⎥ ⎥ ⎥ ⎦
. (3.37)
Da Eq. (3.37), retiramos os elementos da equa¸c˜ao matricial:
ω−ωS1 −V11SS[(−T24T33T42+T23T34T42+T24T32T43 (3.38)
−T22T34T43−T23T32T44+T22T33T44)∆
−1
+T11] = 0,
−V12SS−V11SS[(+T14T33T42−T13T34T42−T14T32T43 (3.39)
+T12T34T43+T13T32T44−T12T33T44)∆
−1
+T12] = 0,
−V11SS[(−T14T23T42+T13T24T42+T14T22T43 (3.40)
−T12T24T43−T13T22T44+T12T23T44)∆
−1
+T13] = 0,
−VSS
14 −V11SS[(T14T23T32−T13T24T32−T14T22T33 (3.41)
+T12T24T33+T13T22T34−T12T23T34)∆
−1
+T14] = 0,
−VSS
12 −V22SS[(+T24T33T41−T23T34T41−T24T31T43 (3.42)
+T21T34T43+T23T31T44−T21T33T44)∆
−1
ω−ωS
2 −V22SS[(−T14T33T41+T13T34T41+T14T31T43 (3.43)
−T11T34T43−T13T31T44+T11T33T44)∆
−1
+T22] = 0,
−V23SS−V22SS[(+T14T23T41−T13T24T41−T14T21T43 (3.44)
+T11T24T43+T13T21T44−T11T23T44)∆
−1
+T23] = 0,
−V22SS[(−T14T23T31+T13T24T31+T14T21T33 (3.45)
−T11T24T33−T13T21T34+T11T23T34)∆
−1
+T24] = 0,
−V33SS[(−T24T32T41+T22T34T41+T24T31T42 (3.46)
−T21T34T42−T22T31T44+T21T32T44)∆
−1
+T31] = 0,
−VSS
23 −V33SS[(T14T32T41−T12T34T41−T14T31T42 (3.47)
+T11T34T42+T12T31T44−T11T32T44)∆
−1
+T32] = 0,
ω−ωS
3 −V33SS[(−T14T22T41+T12T24T41+T14T21T42 (3.48)
−T11T24T42−T12T21T44+T11T22T44)∆
−1
+T33] = 0,
−V34SS−V33SS[(T14T22T31−T12T24T31−T14T21T32 (3.49)
+T11T24T32+T12T21T34−T11T22T34)∆
−1
+T34] = 0,
−V14SS−V44SS[(+T23T32T41−T22T33T41−T23T31T42 (3.50)
+T21T33T42+T22T31T43−T21T32T43)∆
−1
+T41] = 0,
−V44SS[(−T13T32T41+T12T33T41+T13T31T42 (3.51)
−T11T33T42−T12T31T43+T11T32T43)∆
−1
+T42] = 0,
−VSS
34 −V44SS[(T13T22T41−T12T23T41−T13T21T41 (3.52)
+T11T23T42+T12T21T43−T11T22T43)∆
−1
ω−ωS
4 −V44SS[(−T13T22T31+T12T23T31+T13T21T32 (3.53)
−T11T23T32−T12T21T33+T11T22T33)∆
−1
+T44] = 0,
sendo ∆ o determinante da matriz transferˆencia.
No modelo de cadeia pura, os s´ıtios de mesma linha e de linhas vizinhas apresentam os mesmos potenciais dehopping V11=V22=V33=V44=V12=V23=V34=V41e as mesmas energias
de s´ıtio ωS
1=ω2S=ω3S=ωS4, de modo que, quando substituindos nas Eqs. (3.39)-(3.54),
obt´em-se as seguintes igualdades:
T12 =T14=T21=T23 =T32 =T34=T41=T43, (3.54)
T11=T22 =T33 =T44, (3.55)
T13=T24 =T31 =T42. (3.56)
Podemos reescrever a Eq. (3.37) como
LSST2+KST +LSS = 0. (3.57)
Substituindo as Eqs. (3.55)-(3.57) na Eq. (3.58), temos um conjunto de equa¸c˜oes:
−V(T112 + 2T122 +T132) + (ω−ωS)T11−2V T12−V, (3.58)
−V(2T11T12+ 2T13T12) + (ω−ωS)T12−V T11−V T13, (3.59)
−V(2T11T13+ 2T122) + (ω−ωS)T13−2V T12. (3.60)
Resolvendo estas equa¸c˜oes, obtemos as seguintes ra´ızes
T12=−
1
onde a=−(ω−ω1S)
VSS
11 .
T13 =−
1 4T12(T12+ 1)
2T12(T12+ 1)b, (3.62)
onde
T11 =−
T13b
4(T12+ 1)3 −
T13c
4(T12+ 1)3 −
T13(+4T124 + 6T123 + 4T122 + 2T12+ 1)
2T12(T12+ 1)
, (3.63)
b = 8T124 + 16T123 + 12T122 + 4T12+ 1
+ 64T6
12+ 192T125 + 224T124 + 128T123 + 40T122 + 8T12+ 1, (3.64)
e
c = 8T124 + 16T123 + 12T122 + 4T12+ 1
− 64T6
12+ 192T125 + 224T124 + 128T123 + 40T122 + 8T12+ 1. (3.65)
3.4
C´
alculo da densidade de estado
O modelo que vamos estudar se trata do DNA de fita dupla aprisionado entre dois eletrodos met´alicos. Neste modelo, inclu´ımos o grupo a¸c´ucar-fosfato (SP), a fim de apro-ximar do caso real, por´em n˜ao levaremos em conta a tor¸c˜ao da h´elice, como podemos ver na Fig. 3.2.
...
...
...
...
SPSPV
11 SPSPV
44 SPG V 12 GCV
23 SPCV
34 SPAV
12 ATV
23 SPTV
34 SPSPV
11 SPSPV
44 SPCV
12 CGV
23 SPGV
34 SPSPV
11 SPSPV
44 SPTV
12 TAV
23 SPAV
34 SPTV
12 TAV
23 SPAV
34 SPSPV
11 SPSPV
44 SSPV
11 SSPV
44 SSPV
11 SSPV
44 SPGV
12 GCV
23 SPCV
34E
L
E
T
R
O
D
O
E
L
E
T
R
O
D
O
SP SP SP SP
SP SP SP SP SP SP
G C A T C G T T A A G C SP SP
Nitro PDF Trial
Figura 3.2: A mol´ecula de DNA ligada aos dois eletrodos met´alicos.Os ´ındices 1, 2, 3 e 4 correspondem a cada linha da estrutura, V11, V22, V33 e V44
correspondem aos potenciais de hopping entre s´ıtios de mesma linha, V12, V23, V34 e
V41 entre s´ıtios de linhas diferentes, ω1n, ω2n, ωn3 e ω4n s˜ao as energias dos s´ıtios (=1) e
os outros ´ındices S, SP, G, C, A e T s˜ao respectivamente, o substrato, a¸c´ucar-fosfato, guanina, citosina, adenina e timina.
O hopping (correla¸c˜ao de longo alcance) no DNA tem sido tema de investiga¸c˜ao pela sua importˆancia no transporte de cargas. As estruturas que apresentam correla¸c˜ao de longo alcance s˜ao as estruturas quasi-peri´odicas, pelo simples fato de conterem auto-similaridade. Portanto, neste presente trabalho, usaremos as sequˆencias quasi-peri´odicas para moldar o nanocircuito de DNA, e dentre as sequˆencias conhecidas, escolhemos as sequˆencias de Fibonacci e a de Rudin-Shapiro (RS) (para uma explica¸c˜ao detalhada destas estruturas quasi-peri´odicas ver [53]).
G
G
C
G
G
G
G
G
G
G
G
G
G
G
G
C
C
C
C
C
C
Figura 3.3: A sequˆencia quasi-peri´odica de Fibonacci para as primeiras gera¸c˜oes, que crescem seguindo a regra de infla¸c˜aoG→GC e C→G.
Nesta primeira etapa, o modelo ser´a constru´ıdo seguindo a sequˆencia de Fibonacci, onde as bases nitrogenadas de uma das fitas crescem seguindo a regra de infla¸c˜aoG→GC
e C →G. De mesmo modo que a fita adjacente deve seguir a regra de infla¸c˜ao C →CG
Para o modelo do DNA, a Eq. (3.1) e, consequentemente, a Eq. (3.13), sofrem algumas altera¸c˜oes devido a ausˆencia dos potˆencias V14, V22 e V33. Os procedimentos
matem´aticos daqui por diante j´a tˆem as devidas altera¸c˜oes, n˜ao h´a necessidade de refazer os mesmos artif´ıcios que foram realizados para cadeia pura, pois o c´alculo anal´ıtico pode ser reproduzido seguindo os mesmos passos. Assim, obtemos
H = ω1n|n,1>< n,1|+ω2n|n,2>< n,2|+ω3n|n,3>< n,3|+ω4n|n,4>< n,4| + V12nn[|n,1>< n,2|+|n,2>< n,1|]
+ V23nn[|n,2>< n,3|+|n,3>< n,2|]
+ Vnn
34 [|n,3>< n,4|+|n,4>< n,3|]
+
δ=±1
[V11n,n+δ|n,1>< n+δ,1|+V44n,n+δ|n,4>< n+δ,4|].
a) Considerando m = 0 e l = 0. i) Para α = β = 1:
ωG1100−ω1SPG1100−V12SP GG2100−V11SP SG1110−V11SP SG11−10 = 1. (3.66)
ii) Para α= 1 e β = 2:
ωG1200−ω1SPG1200−V12SP GG2200−V11SP SG1210−V11SP SG12−10 = 0. (3.67)
iii) Para α= 1 e β = 3:
ωG1300−ωs
1G1300−V12SP GG2300−V11SP SG1310−V11SP SG13−10 = 0. (3.68)
ωG1400−ω1SPG1400−V12SP GG2400−V11SP SG1410−V11SP SG14−10 = 0. (3.69)
v) Para α= 2 e β = 1:
ωG2100−ωG2G2100−V12SP GG1100−V23GCG3100= 0. (3.70)
vi) Paraα = 2 e β= 2:
ωG2200−ωG2G2200−V12SP GG1200−V23GCG3200= 1. (3.71)
vii) Para α = 2 eβ = 3:
ωG2300−ωG
2G2300−V12SP GG0013−V23GCG3300= 0. (3.72)
viii) Para α= 2 e β = 4:
ωG2400−ωG
2G2400−V12SP GG0014−V23GCG3400= 0. (3.73)
ix) Paraα = 3 e β= 1:
ωG3100−ωC
3G3100−V23GCG0021−V34SP CG4100= 0. (3.74)
ωG3200−ωC
3G3200−V23GCG0022−V34SP CG4200= 0. (3.75)
xi) Paraα = 3 e β= 3:
ωG3300−ωC3G3300−V23GCG2300−V34SP CG4300= 1. (3.76)
xii) Para α = 3 eβ = 4:
ωG34
00−ωC3G3400−V23GCG2400−V34SP CG4400= 0. (3.77)
xiii) Para α= 4 e β = 1:
ωG4100−ωSP
4 G4100−V34SP CG3100−V44SP SG4110−V44SP SG41−10 = 0. (3.78)
xiv) Para α= 4 e β = 2:
ωG4200−ω4SPG4200−V34SP CG3200−V44SP SG4210−V44SP SG42−10 = 0. (3.79)
xv) Para α= 4 e β = 3:
ωG43
00−ω4SPG4300−V34SP CG3300−V44SP SG4310−V44SP SG43−10 = 0. (3.80)
xvi) Para α= 4 e β = 4:
Definindo as matrizes
KG=
⎡ ⎢ ⎢ ⎢ ⎢ ⎢ ⎢ ⎣
ω−ωSP
1 −V12SP G 0 0
−VSP G
12 ω−ω2G −V23GC 0
0 −VGC
23 ω−ωC3 −V34SP C
0 0 −VSP C
34 ω−ω4SP
⎤ ⎥ ⎥ ⎥ ⎥ ⎥ ⎥ ⎦ , (3.82) e
LSP S =
⎡ ⎢ ⎢ ⎢ ⎢ ⎢ ⎢ ⎣
−VSP S
11 0 0 0
0 0 0 0
0 0 0 0
0 0 0 −VSP S
44 ⎤ ⎥ ⎥ ⎥ ⎥ ⎥ ⎥ ⎦ . (3.83)
Escrevendo a equa¸c˜ao matricial
KGG00+LSP S(G−10+G10) =I. (3.84)
Fazendo o mesmo procedimento para m = 0 e l = 1, temos
KSG10+LSP SG00+LSS(T G10) = 0. (3.85)
Sabendo que T =G20G
−1
10, obtemos a matriz inversa de Green, que:
G−1
nn =KG+ 2Γ(1), (3.86)
onde
Γ(1) =−LSP S[KS+LSST]
−1
LSP S. (3.87)
(a) Para m = l = 0:
KGG00+LSP SG−10+LSP SPG10=I, (3.88)
onde
LSP SP =
⎡ ⎢ ⎢ ⎢ ⎢ ⎢ ⎢ ⎣
−VSP SP
11 0 0 0
0 0 0 0
0 0 0 0
0 0 0 −VSP SP
44 ⎤ ⎥ ⎥ ⎥ ⎥ ⎥ ⎥ ⎦ . (3.89)
(b) Para m = 0 e l = 1:
KCG10+LSP SPG00+LSP SPG20= 0, (3.90)
onde
KC =
⎡ ⎢ ⎢ ⎢ ⎢ ⎢ ⎢ ⎣
ω−ωSP
1 −V12SP C 0 0
−VSP C
12 ω−ω2C −V23CG 0
0 −VCG
23 ω−ωG3 −V34SP G
0 0 −VSP G
34 ω−ω4SP
⎤ ⎥ ⎥ ⎥ ⎥ ⎥ ⎥ ⎦ . (3.91)
(c) Para m = 0 e l = 2:
KSG20+LSP SG10+LSSG30= 0. (3.92)
Usando as Eqs. (3.89), (3.91) e (3.93) e sabendo que G30 = T G20, o inverso dos
elementos diagonais da matriz de Green ser´a definido por:
G−1
nn =KG+ Γ(1) + Λ(2), (3.93)
onde
Λ(2) =−LSP SP[KC + Λ(1)]
−1
e
Λ(1) =−LSP S[KS+LSST]
−1
LSP S. (3.95)
De maneira an´aloga, para terceira gera¸c˜ao de Fibonacci (trˆes pares de bases), temos que G40=T G30.
(i) Para m = 0 e l= 0:
KGG00+LSP SG−10+LSP SPG10=I. (3.96)
(ii) Para m = 0 e l = 1:
KCG10+LSP SP(G00+G20) = 0. (3.97)
(iii) Para m = 0 e l = 2:
KGG20+LSP SPG10+LSP SG30 = 0. (3.98)
(iv) Para m = 0 e l = 3:
KSG30+LSP SG20+LSSG40= 0. (3.99)
Das equa¸c˜oes acima, chegamos `a seguinte express˜ao para G−1
nn:
G−1
nn =KG+ Γ(1) + Γ(3), (3.100)
Γ(3) =−LSP SP[KC + Γ(2)]
−1
LSP SP, (3.101)
Γ(2) =−LSP SP[KG+ Γ(1)]
−1
G
G
G
G
G
G
G
C
C
C
C
A
A
C
T
Figura 3.4: A sequˆencia quasi-peri´odica de Rudin-Shapiro para as primeiras gera¸c˜oes, que cresce segundo a regra de infla¸c˜ao G→GC,C→GA,A→T C e T →T A.
Depois de sucessivas gera¸c˜oes, encontramos uma rela¸c˜ao para as gera¸c˜oes pares e ´ımpares de G−1
nn da sequˆencia de Fibonacci :
(a) Para gera¸c˜oes pares:
G−1
nn =KG+ Γ(1) + Λ(nF B). (3.103)
(b) Para gera¸c˜oes ´ımpares:
G−nn1 =KG+ Γ(1) + Γ(nF B), (3.104)
onde nF B ´e o n´umero de nucleot´ıdeos conectados e Λ(nF B) e Γ(nF B) s˜ao as rela¸c˜oes de
recorrˆencia, que s˜ao escritas como:
Λ(nF B) =−LSP SP[Ki+ Λ(nF B −1)]
−1
LSP SP (3.105)
e
Γ(nF B) = −LSP SP[Ki+ Γ(nF B −1)]
−1
LSP SP. (3.106)
de Watson e Crick. Essa sequˆencia aproxima mais do modelo do DNA do que a outra, por apresenta as quatros bases nitrogenadas. Reparamos que as duas primeiras gera¸c˜oes de Rudin-Shapiro s˜ao as mesmas para as duas primeiras gera¸c˜oes de Fibonacci, portanto, vamos ter paraG−1
nn, respectivamente:
G−1
nn =KG+ 2Γ(1), (3.107)
G−nn1 =KC + Γ(1) +C(2), (3.108)
onde
C(2) =−LSP SP[KC + Λ(1)]
−1
LSP SP (3.109)
e
C(1) =−LSP S[KS+LSST]
−1
LSP S. (3.110)
Para a terceira gera¸c˜ao de RS, temos (i) Para l=0 e m=0:
KGG00+LSP SG−10+LSP SPG10=I. (3.111) (ii) Para l=1 em=0:
KCG10+LSP SP(G00+G20) = 0. (3.112)
(iii) Para l=2 e m=0:
KGG20+LSP SPG10+LSP SPG30= 0. (3.113)
(iv) Para l=3 e m=0:
KAG30+LSP SPG20+LSP SG40= 0. (3.114)
(v) Paral=4 e m=0:
onde definimos a seguinte matriz:
KA=
⎡ ⎢ ⎢ ⎢ ⎢ ⎢ ⎢ ⎣
ω−ωSP
1 −V12SP A 0 0
−VSP A
12 ω−ω2A −V23AT 0
0 −VAT
23 ω−ωT3 −V34SP T
0 0 −VSP T
34 ω−ω4SP
⎤ ⎥ ⎥ ⎥ ⎥ ⎥ ⎥ ⎦ . (3.116)
Manipulando as Eqs. (3.112) a (3.116) e juntamente com G50=T G40, encontramos o
termo G−1
nn relacionado a esta gera¸c˜ao de RS:
(a) Para gera¸c˜oes pares:
G−nn1 =KG+ Γ(1) +C(N). (3.117)
(b) Para gera¸c˜oes ´ımpares:
G−nn1 =KG+ Γ(1) +A(N), (3.118)
onde
C(N) = −LSP SP[Ki+C(N −1)]
−1
LSP SP, (3.119)
A(N) = −LSP SP[Ki+A(N −1)]
−1
LSP SP, (3.120)
e
A(1) =−LSP S[KS +LSST]
−1
LSP S. (3.121)
Com a matriz:
KT =
⎡ ⎢ ⎢ ⎢ ⎢ ⎢ ⎢ ⎣
ω−ωSP
1 −V12SP T 0 0
−VSP T
12 ω−ω2T −V23T A 0
0 −VT A
23 ω−ω3A −V34SP A
0 0 −VSP A
34 ω−ω4SP
⎤ ⎥ ⎥ ⎥ ⎥ ⎥ ⎥ ⎦ . (3.122)
A densidade de estado (DOS) para os sistemas ´e dada por:
ρ(ω) =−1
πIm[
m
n
Gnn(ω)], (3.123)
3.5
Resultados num´
ericos
Nesta se¸c˜ao, apresentamos os resultados da densidade de estados (DOS) para na-noestrutura da mol´ecula de DNA quasi-peri´odica, que segue as sequˆencias de Fibonacci e Rudin-Shapiro, e tamb´em, para uma parte da sequˆencia do cromossomo humano Ch22. Os c´alculos foram feitos utilizando o Hamiltoniano tipo tight-binding para as bases ni-trogenadas e o grupo a¸c´ucar-fosfato da cadeia bidimensional. Os valores admitidos das energias de ioniza¸c˜ao das bases nitrogenadas s˜ao: ωA = 8.25 eV,ωT = 9.13 eV, ωG = 7.77
eV eωC = 8.87 eV; a energia de s´ıtio da platina (material considerado como eletrodo) vale
ωS = 5.36 eV e a energia de ioniza¸c˜ao do a¸c´ucar-fosfato (ωSP) ´e igual a 12.27 eV [44, 54].
Enquanto, o potencial de hopping entre a base (G, C, A e T) e o a¸c´ucar-fosfato(SP) tem o valor V12 = V34 = 1.5 eV, do eletrodo vale VS = 12 eV [54] e entre os pares de base
G-C e A-T valem VGC = 0.90 eV e VAT = 0.34 eV [44], respectivamente. Agora, o termo
de hopping na interface DNA-eletrodo ´e dado pela diferen¸ca entre a energia de Fermi da platina e os estados HOMO (Highest Occupied Molecular Orbital ou “mais alto orbital molecular ocupado”) do a¸c´ucar-fosfato nos dandoVSP S = 6.91 eV [54]. E por fim, o termo
de hopping entre os grupos a¸c´ucar-fosfatos, que apresenta um valor baixo em rela¸c˜ao as bases, ´e em torno de VSP SP = 0.02 eV [55].
O pico II tem uma correla¸c˜ao forte com a energia de ioniza¸c˜ao da citosina, que ´e em torno de 9 eV. Este resultado ´e interessante porque mostra que embora a quantidade de citosinas ao longo das gera¸c˜oes de Fibonacci seja menor, em compara¸c˜ao a quantidade de guaninas, e sua energia de ioniza¸c˜aoωC seja maior, a densidade de estado mostra um pico
pronunciado (veja os resultados para a d´ecima quinta gera¸c˜ao na Fig. 3.6) compar´avel ao pico III, que ocorre para um valor aproximadamente igual a duas vezes a energia ioniza¸c˜ao da guanina, isto ´e 15.54 eV. Tamb´em podemos observar uma anomalia no espectro do DOS em torno de 10.6 eV, que corresponde a duas vezes o valor ωS, ou seja, 10.72 eV
aproximadamente. Novamente, observamos que o eletrodo deve ser uma caracter´ıstica relevante na escolha do substrato. Al´em disso, podemos ver que a raz˜ao entre as alturas dos picos, para gera¸c˜oes de Fibonacci consecutivas, tende a raz˜ao de ouroτ = (1 +√5)/2, um n´umero intrinsicamente ligado `a sequˆencia de Fibonacci. Os espectros guardam uma rela¸c˜ao auto-similar entre si, atrav´es da raz˜ao de ouro, quando analisamos os perfis dos gr´aficos. A d´ecima gera¸c˜ao de Fibonacci quando multiplicada pelo n´umero da raz˜ao de ouro fornece a d´ecima primeira gera¸c˜ao, com uma boa aproxima¸c˜ao. Da mesma forma, a d´ecima segunda gera¸c˜ao pode ser obtida multiplicando o perfil da d´ecima primeira por τ, e assim por diante.
Por outro lado, o n´umero de estados em cada n´ıvel de energia aumenta com a gera¸c˜ao de Fibonacci para o intervalo de energia de 5.36 eV at´e 15.98 eV, aproximadamente. Observe que este intervalo est´a compreendido quase que totalmente entre a energia de ioniza¸c˜ao do substratoωS e duas vezes a energia de ioniza¸c˜ao da guanina ωG, isto ´e 15.54
eV. No entanto, em torno 12.5 eV, observamos que o DOS ´e quase nulo. Este resultado pode estar relacionado com o potencial dehopping do substrato e/ou a energia de ioniza¸c˜ao do a¸c´ucar-fostato, ambos em torno de 12 eV. Como esta energia ´e alta poucos el´etrons possuem energia suficiente para ocupar estados com este valor de energia ou maior.