RENORBIO
Programa de Pós-graduação em Biotecnologia.
Proteômica diferencial da Chromobacterium violaceum em resposta ao
estresse oxidativo induzido por peróxido de hidrogênio
Fábio Teixeira Duarte
Fábio Teixeira Duarte
Proteômica diferencial da Chromobacterium violaceum em resposta ao
estresse oxidativo induzido por peróxido de hidrogênio
Tese de Doutorado apresentada ao Programa de Pós-Graduação em Biotecnologia – RENORBIO, como parte dos requisitos necessários à obtenção do título de Doutor em Biotecnologia.
Orientadora: Prof.ª Dr.ª Silvia Regina Batistuzzo de Medeiros.
UFRN / Biblioteca Central Zila Mamede. Catalogação da Publicação na Fonte.
Duarte, Fábio Teixeira.
Proteômica diferencial de Chromobacterium violaceum em resposta ao estresse oxidativo induzido por peróxido de hidrogênio / Fábio Teixeira Duarte. – Natal, RN, 2012.
126 f. : il.
Orientadora: Profª. Dra. Sílvia Regina Batistuzzo de Medeiros.
Tese (Doutorado) – Universidade Federal do Rio Grande do Norte. Centro de Tecnologia. Programa de Pós-Graduação em Biotecnologia.
1. Chromobacterium violaceum - Tese. 2. Beta-proteobactéria - Teses. 3. Estresse oxidativo - Tese. 4. Proteônica - Tese. 5. Biotecnologia - Tese. I. Medeiros, Sílvia Regina Batistuzzo de. II. Universidade Federal do Rio Grande do Norte. III. Rede Nordeste de Bioteconologia (Renorbio). IV. Título.
ii
Fábio Teixeira Duarte
Proteômica diferencial da Chromobacterium violaceum em resposta ao
estresse oxidativo induzido por peróxido de hidrogênio
TESE APRESENTADA AO PROGRAMA DE PÓS-GRADUAÇÃO EM BIOTECNOLOGIA (RENORBIO) DA UNIVERSIDADE FEDERAL DO RIO GRANDE DO NORTE, COMO PARTE DOS REQUISITOS NECESSÁRIOS PARA A OBTENÇÃO DO GRAU DE DOUTOR EM BIOTECNOLOGIA.
Examinada por:
Prof.ª Dr.ª Silvia Regina Batistuzzo de Medeiros.
Prof.ª Dr.ª Lucymara Fassarela Agnes de Lima
Prof. Dr. João Paulo Matos Santos Lima
Prof. Dr. Thalles Barbosa Grangeiro
Agradecimentos
A Deus, fonte de todas as minhas conquistas.
À CAPES e ao CNPQ, pelo financiamento do projeto. Aos membros da banca, por aceitarem avaliar o trabalho. Ao RENORBIO, por fornecer esta capacitação.
À professora Dr.ª Silvia Regina Batistuzzo de Medeiros, pela paciência e pela orientação.
Aos componentes do PROTE-CHROMO, Daniel, meu grande amigo, “meu eterno IC”, com quem compartilho este trabalho e sem cuja ajuda não seria possível a
realização. A Vivi, que tanto admiro e inspira a conclusão deste trabalho. E, por fim, a Rai, que acaba de se integrar a esse grupo.
Aos meus amigos e familiares, que tanto cofiam em mim e são fonte de apoio constante.
A todos do LBMG, cujos nomes não citarei, pois tenho certeza de que esquecerei alguém; pela amizade, pela descontração, pela harmonia (às vezes, conturbada), pelas conversas (as vezes meio batidinhas), enfim, por tornar o ambiente de trabalho agradável.
SUMÁRIO
LISTA DE FIGURAS ... ii
LISTA DE ABREVIATURAS ... iii
RESUMO...v
ABSTRACT ... vii
1. INTRODUÇÃO ... 9
2. REVISÃO DA LITERATURA ... 11
2.1. Chromobacterium violaceum ... 11
2.2. Proteômica ... 13
2.3. Estresse oxidativo ... 15
2.3.1. Antioxidantes ... 16
2.3.1.1. Mecanismos moleculares dos sistemas antioxidantes E. coli, B. subtilis e outros procariotos ... 17
2.3.1.1.1. Regulon SoxR ... 18
2.3.1.1.2. Regulon OxyR ... 20
2.3.1.1.3. Regulon PerR ... 22
2.3.1.2. Antioxidantes enzimáticos ... 23
2.3.1.2.1. Superóxido dismutase ... 23
2.3.1.2.2. Catalase ... 24
2.3.1.2.3. Glutationa redutase ... 24
2.4. Relações biotecnológicas em C. violaceum e estresse oxidativo ... 25
3. OBJETIVOS ... 27
3.1. Objetivo geral ... 27
3.1. Objetivos específicos ... 27
4. REFERÊNCIAS BIBLIOGRÁFICAS ... 28
5. ARTIGOS ... 33
6. Considerações finais ... 124
ii
LISTA DE FIGURAS
Figura 1. Colônias de C. violaceum.
Figura2. Principais defesas e endógenas antioxidantes da célula.
Figura 3. Operação do sistema SoxRS em E. coli. Figura 4. Operação do sistema OxyR em E. coli.
iii
LISTA DE ABREVIATURAS
2-D: duas dimensões. ACN: acetonitrile
AHL: N-acil homoserina lactona Apx: peroxidase de ascorbato AsA: ascórbico
BER: “base excision repair”
BLAST: “Basic Local Alignment Search Tool”
BSA: “bovine serum albumin”
Cat: catalase
cDNA: DNA complementar
CRISPR: “clustered, regularly interspaced or short palindromic repeats”
DNA: ácido desoxirribonucleico DTT: Dithiothreitol
DUF: domínio de função desconhecida ERN: Espécies Reativas de Nitrogênio ERO: Espécies Reativas de Oxigênio EtOH: etanol
FDR: “false discovery rate”
GR: glutationa redutase. GSH: glutationa reduzida GST: glutationa S-transferase HOAc: ácido acético
IAM: iodoacetamide
LC: “liquid chromatography”
Meio LB: Meio Luria-Bertani
iv MV: máxima verossimilhança
MV: metilviologênio
NADH: nicotinamida adenina dinucleotídeo
NADPH: nicotinamida adenina dinucleotídeo fosfato NCBI: “National Center for Biotechnology Information”
OD: densidade óptica
ORF: quadro aberto de leitura. pb: “Base pairs”
PCR: reação em cadeia da polimerase pH: potencial hidrogeniônico
Pod: peroxidase
PTM: “post-translational modification”
RNA: ácido ribonucleico
ROS: “reactive oxygen species”
RPM: rotação por minuto
SDS-PAGE: “sodium dodecyl sulfate polyacrylamide gel electrophoresis”
SEPro: “Search Engine Processor”
Sod: superóxido dismutase.
sRNA: pequenos ácidos ribonucleico. TAE: Tris-Acetado e EDTA
v
RESUMO
A -proteobactéria Chromobacterium violaceum é um bacilo Gram-negativo, de vida
livre,saprófito e patógeno oportunista que habita em ecossistemas tropicais e subtropicais dentre eles no solo e na água da Amazônia. Possui grande potencial biotecnológico e, devido a esse potencial, seu genoma foi totalmente sequenciado em 2003. A análise do genoma mostrou que essa bactéria possui vários genes com funções relacionadas à capacidade de sobreviver sob diferentes tipos de estresse ambiental. Objetivando entender a resposta fisiológica de C. violaceum sob condição
de estresse oxidativo, foi aplicada a ferramenta de proteômica de shotgun. Sendo assim, colônias de C. violaceum ATCC 12472 foram cultivadas na presença e na
ausência de 8 mM de H2O2 por duas horas, as proteínas totais da bactéria foram
extraídas, submetidas à SDS-PAGE, coradas e hidrolisadas. Os peptídeos trípticos gerados foram submetidos a uma cromatografia líquida (LC)-linear seguido de espectrômetro de massa (LTQ-XL-Orbitrap) para a obtenção de dados quantitativos e qualitativos. A proteômica de shotgun permite comparar diretamente, em amostras complexas, expressão diferencial de proteínas e revelou que, em C. violaceum, 131
proteínas são expressas exclusivamente na condição controle, 177 proteínas passaram a ser expressas sob estresse oxidativo e 1175 proteínas possuem expressão em ambas condições. A análise dos resultados mostrou que, na condição de estresse oxidativo, essa bactéria muda seu metabolismo, aumentando a expressão de proteínas capazes de combater o estresse oxidativo e diminuindo a expressão de proteínas de processos relacionados com o crescimento bacteriano e catabolismo (transcrição, tradução, metabolismo de carbono e de ácidos graxos). A ferramenta de proteômica com uma abordagem de biologia integrativa forneceu uma visão geral das vias metabólicas envolvidas na resposta de C. violaceum ao
estresse oxidativo, bem como amplificou significativamente a compreensão resposta fisiológica ao estresse ambiental. Ensaios bioquímicos e “in silico” com a ORF
hipotética CV_0868 constataram que esta faz parte de um operon. A análise filogenética de superóxidos dismutase, proteína pertencente tambémb ao operon, mostrou que esse gene está duplicado no genona de C. violaceum e que a segunda
vi
não só o gene sod, mas também todos os genes que compõem este operon foram adquiridos da mesma maneira. Concluiu-se que C. violaceum possui mecanismos
complexos, eficientes e versáteis em resposta estresse oxidativo.
vii
ABSTRACT
The -proteobacterium Chromobacterium violaceum is a Gram-negative, free-living,
saprophytic and opportunistic pathogen that inhabits tropical and subtropical ecosystems among them, in soil and water of the Amazon. It has great biotechnological potential, and because of this potential, its genome was completely sequenced in 2003. Genome analysis showed that this bacterium has several genes with functions related to the ability to survive under different kinds of environmental stresses. In order to understand the physiological response of C. violaceum under
oxidative stress, we applied the tool of shotgun proteomics. Thus, colonies of C. violaceum ATCC 12472 were grown in the presence and absence of 8 mM H2O2 for
two hours, total proteins were extracted from bacteria, subjected to SDS-PAGE, stained and hydrolysed. The tryptic peptides generated were subjected to a linear-liquid chromatography (LC) followed by mass spectrometer (LTQ-XL-Orbitrap) to obtain quantitative and qualitative data. A shotgun proteomics allows to compare directly in complex samples, differential expression of proteins and found that in C. Violaceum, 131 proteins are expressed exclusively in the control condition, 177 proteins began to be expressed under oxidative stress and 1175 proteins have expression in both conditions. The results showed that, under the condition of oxidative stress, this bacterium changes its metabolism by increasing the expression of proteins capable of combating oxidative stress and decreasing the expression of proteins related processes bacterial growth and catabolism (transcription, translation, carbon metabolism and fatty acids). A tool with of proteomics as an approach of integrative biology provided an overview of the metabolic pathways involved in the response of C. violaceum to oxidative stress, as well as significantly amplified
understanding physiological response to environmental stress. Biochemical and "in silico" assays with the hypothetical ORF CV_0868 found that this is part of an operon. Phylogenetic analysis of superoxide dismutase, protein belonging to the operon also showed that the gene is duplicated in genome of C. violaceum and the
viii
manner. It was concluded that C. violaceum has complex, efficient and versatile
mechanisms in oxidative stress response.
9
1. INTRODUÇÃO
A Chromobacterium violaceum, classificada como uma beta-proteobactéria da
família Neisseriacea, se caracteriza como um bacilo Gram-negativo de vida livre, aeróbico, flagelado. Sua característica mais marcante é a coloração violeta, devido à produção de um pigmento, a violaceína, a qual deu nome à espécie. Essa bactéria é encontrada em uma grande variedade de ambientes tropicais e subtropicais, estando muito presente na Amazônia brasileira, principalmente nas águas e nos bancos de areia do Rio Negro.
O pigmento violaceína tem sido alvo de interesse científico. Muitos são os estudos sobre esse metabólito secundário e já foi descrita uma grande variedade de aplicações farmacológicas e biotecnológicas. Dentre essas, podemos citar as ações antibiótica, antiviral, antiprotozoárica, antitumoral, antioxidante, antifúngica, antiúlcera, antidiarreica, citotóxica e ainda a capacidade de induzir apoptose.
Além da violaceína, esse organismo também é capaz de produzir outros bioprodutos com aplicações biotecnológicas. Foi detectado que C. violaceum produz
cianeto, produto que pode ser utilizado para complexar metal pesado e, consequentemente, usado na extração de ouro ou na biorremediação de áreas contaminadas por metal pesado, como garimpos desativados. A produção de um bioplástico biodegradável semelhante ao polipropileno também já foi descrita para esse organismo. A descoberta da produção de uma enzima quitinolítica possibilita o uso desse organismo e/ou produto como bioinseticida. Também foram isolados fármacos com atividade antitumoral.
Devido a esse grande potencial biotecnológico, esse organismo foi o primeiro a ter seu genoma totalmente sequenciado pelo Projeto Genoma Brasileiro, um consórcio brasileiro de sequenciamento genômico. As informações geradas pelo Projeto Genoma foram bastante abrangentes para o entendimento da fisiologia desse micro-organismo. No entanto, parte do seu genoma ainda continua desconhecida, pois é composto de ORFs hipotética e hipotética conservada, revelando que possivelmente podem-se encontrar outras aplicações ainda não descritas.
10
biológicas a ser investigadas, a que se chamou “era pós-genômica”. Esta é
caracterizada, principalmente, pelo uso de ferramentas que buscam compreender melhor a fisiologia dos organismos usados como base nas informações genômicas. Dentre essas ferramentas, estão a trancriptômica, proteômica, metabolômica, interatômica, entre outras. A proteômica foi utilizada como ferramenta para entendimento da resposta fisiológica de C. violaceum ao estresse oxidativo.
A proteômica surge, nessa conjuntura, como uma ferramenta bastante versátil na busca de novas informações, pois tem como principal objetivo a caracterização ou identificação de todas as proteínas expressas por um organismo ou tecido em uma determinada condição. O proteoma difere do genoma, pois este não é tão dinâmico e aquele varia constantemente para cada mudança ambiental sofrida pelos seres vivos, ou seja, para cada condição fisiológica, o organismo possui um proteoma particular.
A proteômica também permite alcançar diferentes objetivos, como o entendimento das vias metabólicas, a caracterização das respostas celulares, a compreensão dos mecanismos de patogenicidade e virulência, bem como a identificação de novos alvos farmacológicos, marcadores biológicos e genes não caracterizados.
11
2. REVISÃO DA LITERATURA
2.1. Chromobacterium violaceum
O gênero Chromobacterium inclui bactérias Gram-negativas de vida livre. Até
2007, o gênero era composto por apenas uma espécie, a Chromobacterium violaceum, mas seis espécies são reconhecidas (Lima-Bittencourt et al., 2011),
dentre elas a C. subtsugae (Martin et al., 2007), C. piscinae, C. pseudoviolaceum
(Kämpfer et al., 2009), C. haemolyticum (Han et al., 2008) e C. aquaticum (Young et al., 2008). Chromobacterium violaceum foi descrita inicialmente por Boisbaudran em
1882 e pertence à família Neisseriacea das -Proteobactérias. É um bacilo Gram-negativo de vida livre, saprófito, aeróbio, flagelado, que vive em uma variedade de ecossistemas em regiões tropicais e subtropicais, incluindo bancos de areia e a água do Rio Negro, na Amazônia brasileira (Durán e Menck, 2001) (Figura 1).
Figura 1. Placa de Petri com colônias de C. violaceum cultivadas em meio de cultura LB
sólido
A principal característica desse micro-organismo é a coloração violeta, devido à produção de um pigmento denominado violaceína, um metabólito secundário. Os genes envolvidos na biossíntese de violaceína apresentam-se em um operon constituído de cinco genes VioABCDE, e a via da biossíntese proposta é
VioA→VioB→VioE→VioD→VioC (Sánchez et al., 2006). As enzimas, produtos
12
de L-triptofano para formar a violaceína (Momen e Hoshino, 2000). No entanto, detalhes e mecanismos regulatórios da biossíntese da violaceína nessa bactéria ainda não são completamente entendidos em nível molecular. A biossíntese da violaceína responde a um mecanismo de sinalização molecular de “quorum-sensing”
(Shinoda et al., 2007). A sinalização molecular é difusível, geralmente designada por
N-acil homoserina lactonas (AHL) (McClean et al., 1997).
A violaceína tem várias atividades biológicas, como antimalárica (Lopes et al.,
2009), antileishimaníaca (Leon et al., 2001), antiviral (Andrighetti-Fröhmer et al.,
2003), antiprotozoárica (Matz et al., 2004), antitumoral (Ueda et al., 1994, Melo et al.,
2000), anti-Mycobacterium tuberculosis (De Souza et al., 1999), bactericida (Durán,
1990), tripanocida (Caldas et aI., 1978; Cavallero et al., 1991), citotóxica em células
de mamíferos (Durán et aI., 1989; Haun et al., 1992), antioxidante (De Azevedo et al., 2000), indutora de apoptose (MeIo et al., 2000), antifúngica (Shirata et aI., 2000),
antidiarreica e protetora de úlceras (Antonisamy et al., 2009), imunomoduladora,
antipirética e analgésica (Antonisamy e Ignacimuthu, 2010). Sua atividade antitumoral é marcada por mecanismos multialvos de citotoxicidade, sendo eficiente contra leucemias, câncer de pulmão, melanoma uveal e linfoma (Durán et al., 2007).
Sabe-se que a violaceína é um potente indutor de apoptose em leucemias (Martins
et al., 2010), em câncer de cólon (de Carvalho, et al., 2006) e coloretal (Kodach, et al., 2006).
Outros potenciais biotecnológicos de C. violaceum incluem a produção de
cianeto (Michaels e Corpe, 1965), a solubilização de ouro (Faramarzi et al., 2004), a
produção de enzimas quitinolíticas (Chernin et al., 1998), a síntese de bioplástico
(Steinbüchel et al., 1993), a biorremediação (Carepo et al., 2004) e a produção de
celulose (Recouvreux et al., 2008). Esse micro-organismo ainda produz o FK228,
que é um depsipeptídio (Shigematsu et al., 1994; Ueda et al., 1994), caracterizado
por Wang et al., (2009) e que se tornou um dos agentes anticancerígenos mais
promissores, intervindo, especificamente, nos mecanismos epigenéticos do câncer (Yoo e Jones, 2006).
Apesar de todo potencial biotecnológico, C. violaceum é considerada um
13
infecções causadas por C. violaceum em humanos foram descritas inicialmente em
1927 na Malásia (Sneath et al., 1953). Essas infecções são raras, no entanto,
possuem altos níveis de mortalidade, mais de 57% (Macher et al., 1982).
Devido à importância farmacológica, médica e biológica e ao grande potencial biotecnológico, o genoma Chromobacterium violaceum da linhagem ATCC 12472 foi
totalmente sequenciado (Brazilian National Genome Project Consortium, 2003;
www.brgene.lncc.br; www.ncbi.nlm.nih.gov: access number NC_005085). A análise
do genoma mostrou que esse organismo possui vários genes relacionados à tolerância ao estresse e à aclimatação ambiental, apontando para uma versatilidade fisiológica quanto às variações ambientais (Hungria et al., 2004).
2.2. Proteômica
A análise proteômica consiste na caracterização em larga escala do conjunto de proteínas expressas a partir de um genoma em uma célula ou tecido (Yarmush e Jayaraman, 2002). O termo proteínas vem do grego, proteios, que significa “mais importante”. As proteínas são formadas de polímeros de aminoácidos e são as operadoras-chave e força motriz de todas as atividades celulares. Elas atuam como enzimas, anticorpos, hormônios, componentes estruturais, receptores celulares, substâncias de reserva, entre outras. O aumento no uso da ferramenta de proteômica se dá pelo desenvolvimento, pela integração e pela automatização de uma variedade de técnicas e equipamentos que permitem separar, identificar, quantificar, caracterizar e observar mudanças pós-traducionais, processamento, danos e degradação de proteínas (Hecker et al., 2010), bem como relacionar essa
informação com a obtida por outras abordagens através da Bioinformática. A evolução da espectrometria de massas associada à bioinformática permitiu, nos últimos anos, um grande avanço da proteômica.
14
Os projetos proteomas têm gerado vários dados, permitindo alcançar diferentes objetivos, segundo Hecker et al. (2010), como: a) esclarecer as proteínas
envolvidas em rotas metabólicas relacionadas aos diferentes processos celulares; b) identificar novos alvos farmacológicos e marcadores biológicos relacionados ao processo de estabelecimento de progressão de doenças; c) identificar moléculas bioativas a partir de extratos biológicos naturais, levando ao desenvolvimento de novos fármacos; d) caracterizar as respostas celulares a determinadas drogas, doenças e mudanças ambientais; e) compreender melhor os mecanismos de patogenicidade e a virulência de micro-organismos; f) identificar novos genes, entre outros.
O primeiro estudo de proteomas bacterianos em reposta a danos oxidativos refere-se à análise de gel 2-D de Salmonella após a indução de estresse oxidativo
por H2O2. Esse trabalho demonstrou uma maior expressão de aproximadamente
trinta proteínas, as quais fazem parte dos regulons SoxR e OxyR, que estão relacionados à função antioxidante (Christian et al., 1985). Atualmente, outros
trabalhos também avaliaram a resposta de micro-organismos ao estresse oxidativo. Dentre eles, Kim et al. (2010) analisaram Salmonella enterica em resposta ao
peróxido de hidrogênio e identificaram que 76 proteínas são expressas diferencialmente em resposta ao peróxido. Também observaram que, possivelmente, a proteína SipC está relacionada à sobrevivência desse patógeno durante a infecção. Wolf et al. (2008) investigaram a resposta celular de Staphylococcus aureus ao estresse oxidativo por meio de proteômica diferencial.
Nessa análise, a adição de peróxido de hidrogênio, diamida e metilviologênio (MV) - um agente gerador de superóxido em células em crescimento exponencial - revelou complexas mudanças no padrão de expressão proteica. As proteínas especialmente envolvidas na desintoxicação, nos sistemas de reparo de DNA e no metabolismo intermediário foram encontradas por serem superexpressas.
Com C. violaceum, os estudos de proteômica são bastante reduzidos e com
resultados insipientes. Há somente uma recente publicação, Baraúna et al. (2011),
15
é bastante afetado devido à repressão das enzimas componentes do ciclo do ácido cítrico.
2.3. Estresse oxidativo
A Terra é o único planeta do sistema solar que possui oxigênio em sua atmosfera. No início da sua formação, não existia esse elemento na forma molecular no ambiente atmosférico, caracterizando-o como redutor (Scandalios, 2005). No entanto, com o advento dos seres autotróficos fotossintetizantes, ele passou a fazer parte dos elementos, sendo encontrado em 53,8% da crosta terrestre e constituindo 21% da composição do ar. Segundo Cabsicol et al., (2000), esse evento é
considerado uma das primeiras ocorrências de poluição no planeta.
Para os seres vivos, o oxigênio é de grande relevância à vida, pois, utilizando-o cutilizando-omutilizando-o aceptutilizando-or final de elétrutilizando-ons na cadeia respiratória, é putilizando-ossível a futilizando-ormaçãutilizando-o de uma quantidade bem maior de energia quando comparada à quantidade de energia formada na fermentação anaeróbica. No entanto, quando os seres são expostos ao oxigênio em altas concentrações, é possível haver a morte celular (Dunning et al.,
2009) devido à formação de grande quantidade de EROs.
Radicais livres são definidos como qualquer espécie química que contenha um ou mais elétrons desemparelhados, sendo assim altamente reativos e com meia vida curta. Eles atuam tentando doar o seu elétron desemparelhado a qualquer molécula vizinha ou mesmo tentando coletar um elétron das moléculas no seu entorno para se tornarem estáveis. Essa atividade pode danificar qualquer biomolécula (Muller et al., 2007).
O oxigênio na forma molecular (O2) é pouco reativo, mas tem a possibilidade
de ser convertido a estados excitados chamados de EROs - espécies reativas de oxigênio, as quais incluem radical superóxido (O2-•), peróxido de hidrogênio (H2O2),
radical hidroxila (OH•) e oxigênio “singlet” (O21) (Quan et al., 2008). Também podem
ocorrer radicais que contêm nitrogênio designado ERNs (espécies reativas de nitrogênio). As mais importantes ERNs são o óxido nítrico (NO•) e o peróxido nítrico
(OONO•−) (Li e Moore, 2007). Essas espécies reativas podem ser formadas por
16
envolvem variações ambientais como estresse hídrico, estresse salino, metais pesados, extremo de temperatura, radiação, poluição, herbicida, defensivos agrícolas, drogas, estresse físico e também respostas a estresse biótico, como ataque de patógenos (Lushchak, 2011).
Mais de 90% das EROs produzidos pelos seres vivos são provenientes da cadeia transportadora de elétrons, do retículo endoplasmático, das membranas plasmática e nuclear e dos sistemas fotossintéticos (Starkov, 2008). Uma menor parcela é gerada por algumas enzimas conhecidas como oxidase, por meio da auto-oxidação de diferentes moléculas. As oxidases responsáveis por produzir EROs são NADPH oxidase e xantina oxidase (Puddu et al., 2008).
As EROs são necessárias em baixas concentrações para vários processos fisiológicos de sinalização e regulação (Quan et al., 2008). No entanto, quando
presentes em altas concentrações e com reatividade química elevada, conduzem danos a biomoléculas como DNA, RNA, proteínas e lipídios. Esses danos podem ser: oxidação de proteínas, lesões no DNA, distúrbios de homeostase intracelular de íons cálcio livres e peroxidação lipídica (Miller, 2007).
2.3.1. Antioxidantes
Para evitar os danos causados pelas EROs, os seres vivos desenvolveram vários mecanismos de defesa, denominados genericamente como mecanismos antioxidantes. Em procariotos, esses mecanismos podem ser classificados como enzimáticos e não enzimáticos, conforme a estrutura química. Os mecanismos de proteção não enzimáticos destacam compostos com propriedades intrínsecas
antioxidantes, como o -caroteno, α-caroteno, α-tocoferol (Vitamina E), ácido lipóico, flavonoides, glutationa reduzida (GSH), ácido ascórbico (AsA - Vitamina C), entre outros (Valko et al., 2007). O NADPH e o NADH são moléculas que ajudam a manter
o ambiente redutor (Mailloux et al., 2011). As enzimas descritas são superóxido
dismutase (Sod), catalase (Cat), peroxidase (Pod), peroxidase de ascorbato (Apx), glutationa redutase (GR) e glutationa S-transferase (GST) (Lee e Lee, 2000). Os
antioxidantes podem ter ação “scavenger”, quando agem transformando o radical
17
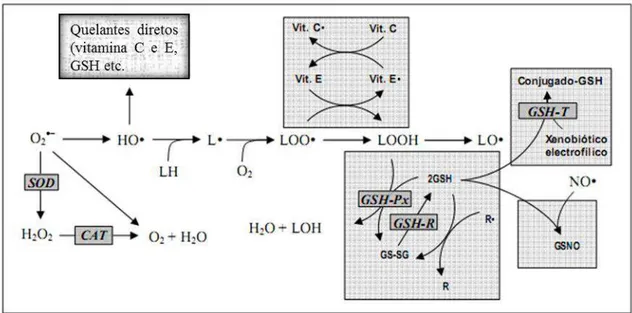
Figura 2. Principais defesas endógenas celulares antioxidantes. As defesas antioxidantes estão representadas em caixas de texto cinza; e as enzimas antioxidantes, com escritas em itálico. As EROs e outros componentes relacionados ao estresse oxidativo são: ânion superóxido (O2−), peróxido de hidrogênio (H2O2) , radical hidróxido (HO•), lipídeos de membrana(LH), radical lipídico
(L•), radical peroxila (LOO•), lipídeo hidroperóxido (LOOH), óxido nítrico (NO•) e álcoois (LOH).
Defesas antioxidantes: superóxido dismutase (SOD), catalase (CAT), glutationa peroxidase (GPH-Px), glutationa redutase (GPH-R), glutationa (GSH), glutationa dissulfito (GS-SG), α- tocoferol ou
vitamina E (vit. E), radical de vitamina E (vit. E), vitamina C (vit. C), radical de vitamina C (vit. C•), não
radical (R), radical (R•), S-nitrosoglutationa (GSNO). Adaptado de Ferreira et al. (2007).
2.3.1.1. Mecanismos moleculares dos sistemas antioxidantes de E. coli, B. subtilis e outros procariotos
A resposta dos sistemas antioxidantes bacterianos para estresse oxidativo é bem caracterizada em Escherichia coli e Bacillus subtilis. Os modelos mais
estudados abrangem os aspectos moleculares e funcionais. A resposta ao estresse oxidativo em E. coli é coordenada principalmente por dois sistemas bem
caracterizados agrupados em dois regulons: o SoxRS e o OxyR. O SoxRS responde
a estresse induzido pelo ânion superóxido, enquanto o OxyR é induzido por peróxido
de hidrogênio. Em ambos os casos, a detecção envolve a oxidação das moléculas sensoras (Lushchak, 2011). Em C. violaceum, ambos os mecanismos foram
18
proteção específica contra estresse oxidativo (Vaconcelos et al., 2003). B. subtilis
apresenta um mecanismo análogo a OxyR, o PerR (regulador de resistência a peróxido), que é o principal regulador da resposta ao estresse provocado por peróxido de hidrogênio nessa bactéria (Duarte e Latour, 2010). Também em B. subtilis, foi descrito outro mecanismo bioquímico sensível a estresse por peróxido.
Ele é regulado por OhrR, um membro de uma família conservada de fatores transcricionais sensíveis a peróxido orgânico. OhrR reprime a transcrição do gene de resistência ohrA por ligação cooperativa (Sukchawalit et al., 2001). No genoma
de C. violaceum, está presente um operon composto por ohrA (CV0209) e ohrR
(CV0210) (Hungria et al., 2004). Em análise genômica, Hungria et al. (2004) também
observam outras ORFs em C. violaceum que podem ter funções relacionadas à
resistência ao estresse oxidativo, como ohrB (CV2493) e ao hidroperóxido e
relacionadas às da família de proteínas MutT/nudix (CV0032, CV1112, CV1586, CV1767, CV3401), que removem precusores de nucleotídeo oxidado. Também estão presentes, no genoma de C. violaceum, o gene dsbA (CV3998), codificante
para principal oxidase presente no periplasma bacteriano, e outras ORFs induzidas por peróxidos de hidrogênio, como dps (CV4253) e fur (CV1797).
2.3.1.1.1. Regulon SoxRS
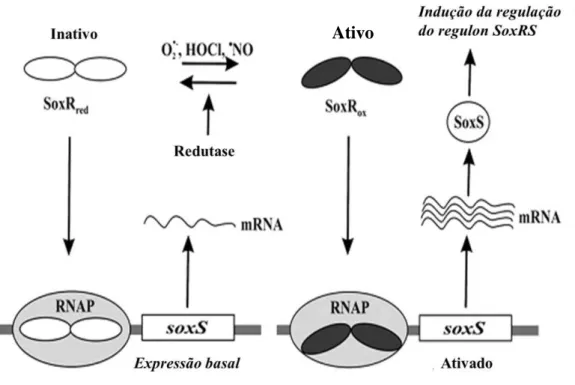
Em E. coli, a ativação do regulon SoxRS ocorre em duas etapas. Inicia-se
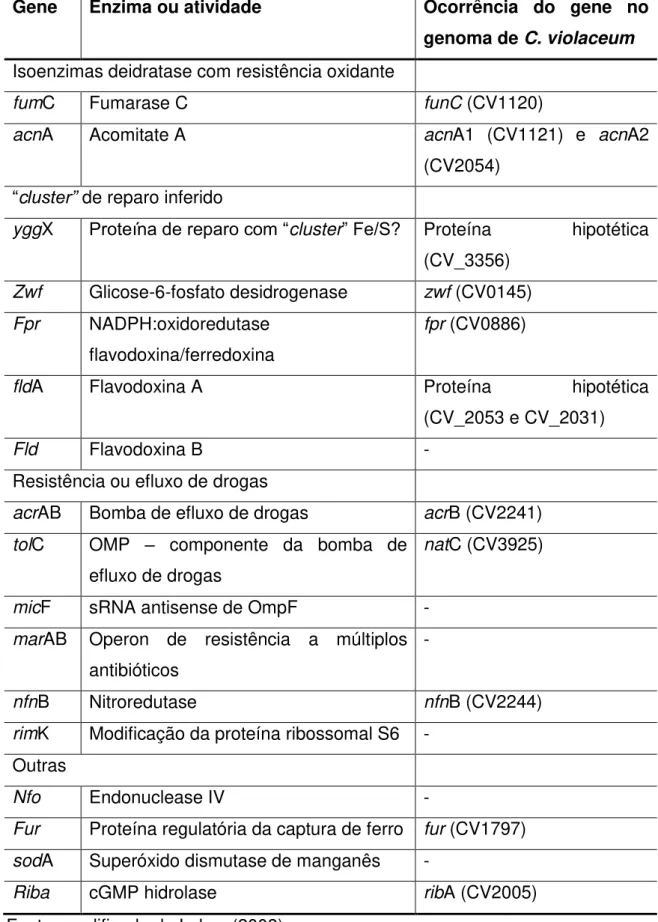
com a ativação da proteína SoxR pela oxidação por compostos geradores de superóxido, tais como paraquate e peróxido de hidrogênio. SoxR oxidada induz à expressão de um segundo fator transcricional, o SoxS (Figura 3), o qual atua regulando positivamente a transcrição de 9 a 40 genes que codificam para proteínas antioxidantes e reparadoras de danos oxidativos (Tabela 1). O regulon SoxRS funciona como parte de um sistema induzível de proteção contra agentes xenobióticos e também pode agir na resistência a macrófagos, pois é induzível por óxido nítrico e ácido hipocloroso (Lushchak, 2011). Em C. violaceum, soxR
(CV2793) está presente, no entanto, os genes soxS, micF, inaA e sodA não foram
19
Tabela 1. Genes induzidos pelo sistema SoxRS em E. coli e a ocorrência no
genoma de C. violaceum.
Gene Enzima ou atividade Ocorrência do gene no
genoma de C. violaceum Isoenzimas deidratase com resistência oxidante
fumC Fumarase C funC (CV1120)
acnA Acomitate A acnA1 (CV1121) e acnA2
(CV2054)
“cluster” de reparo inferido
yggX Proteína de reparo com “cluster” Fe/S? Proteína hipotética
(CV_3356)
Zwf Glicose-6-fosfato desidrogenase zwf (CV0145) Fpr NADPH:oxidoredutase
flavodoxina/ferredoxina
fpr (CV0886)
fldA Flavodoxina A Proteína hipotética
(CV_2053 e CV_2031)
Fld Flavodoxina B -
Resistência ou efluxo de drogas
acrAB Bomba de efluxo de drogas acrB (CV2241) tolC OMP – componente da bomba de
efluxo de drogas
natC (CV3925)
micF sRNA antisense de OmpF - marAB Operon de resistência a múltiplos
antibióticos
-
nfnB Nitroredutase nfnB (CV2244)
rimK Modificação da proteína ribossomal S6 -
Outras
Nfo Endonuclease IV -
Fur Proteína regulatória da captura de ferro fur (CV1797) sodA Superóxido dismutase de manganês -
Riba cGMP hidrolase ribA (CV2005)
20
Figura 3. Operação do sistema SoxS de E. coli. A proteína SoxR reduzida pode ligar-se ao promotor
do gene soxS. No entanto, só sua forma oxidada tem atividade de autoativação e ativação da
expressão de genes alvos. Alguns de seus produtos têm funções claramente de proteção. O mecanismo de inativação da proteína SoxR ainda não é conhecido. Adaptado de Lushchak (2011).
2.3.1.1.2. Regulon OxyR
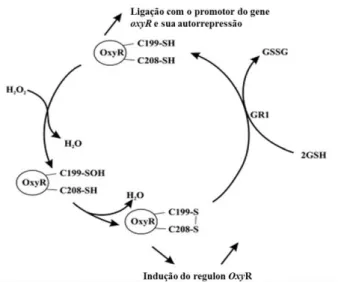
A proteína OxyR é um sensor que responde ao aumento de peróxido de hidrogênio no ambiente intracelular em E. coli. Essa proteína possui duas formas,
uma reduzida e outra oxidada, das quais somente esta tem atividade de ativador transcricional, sendo responsável pela ativação do regulon OxyR (Netto et al., 2007)
(Figura 4). Esse regulon, uma vez ativado, induz à transcrição de uma série de genes cujos produtos têm funções relacionadas à proteção contra estresse oxidativo provocado pelo peróxido de hidrogênio (Tabela 2)(Imlay, 2008).
21
Figura 4. Operação do sistema OxyR em E. coli. A oxidação da proteína OxyR a torna ativada e
possibilita sua ligação aos promotores dos genes alvos, estimulando a expressão gênica. A forma ativada da proteína OxyR é inativada pela ação da tiorredoxina. A autorrepressão do gene oxyR é
proporcionada pelo estado reduzido da proteína OxyR. Adaptado de Lushchak (2011).
Tabela 2. Membros do regulon OxyR de E. coli e a ocorrência no genoma de C. violaceum.
Função Resposta de OxyR em E.
coli
Ocorrência em
C. violaceum
“Scavenging” de H2O2
AhpCF -
Catalase Kate
“Scavenging” de ferro Dps dps (CV4253)
Controle de entrada de ferro
Fur fur (CV1797)
Redução de disulfeto
Tiorredoxina C trxC (CV1106)
Glutarredoxina A -
Glutationa redutase -
DsbG (redutase periplasmica) dsbG (CV2637)
Importador de cátions divalentes
MntH mntH (CV0576)
Síntese de grupo heme Ferroquetalase - Reunião de cluster FeS SufABCDE -
22
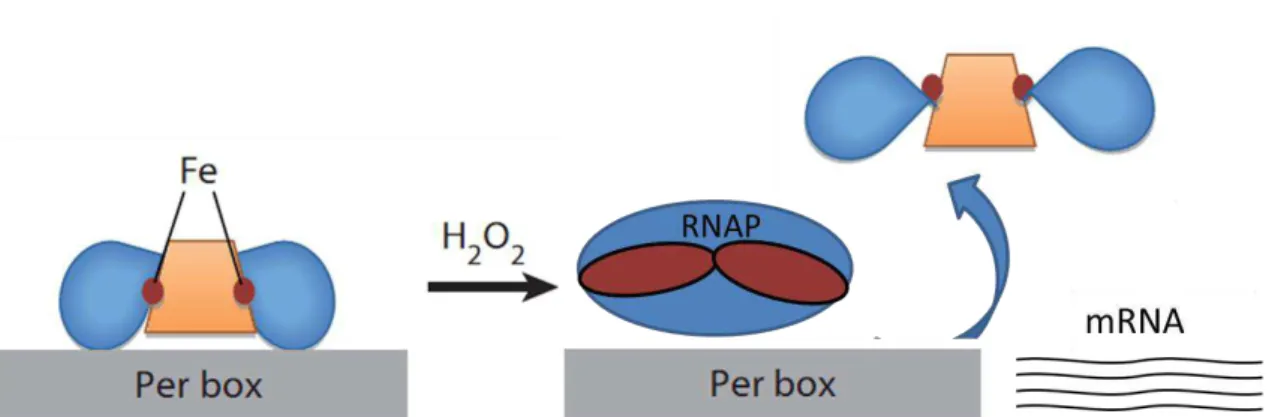
2.3.1.1.3. Regulon PerR
Em B. subtilis, a proteína PerR foi identificada como Fur (regulador de captura
de ferro), um membro da família de proteínas metalo-reguladoras. A proteína PerR está ativa como um repressor da transcrição quando se liga a um único átomo de ferro. Após a exposição ao peróxido de hidrogênio, o átomo de ferro é oxidado em uma reação direta de Fenton, a qual gera um radical ferril e/ou radical hidroxila. A oxidação do íon ferro provavelmente facilita a dissociação do metal da proteína. A metaloproteína sem a ligação ao ferro perde a capacidade de ligação ao DNA, permitindo a indução da expressão dos genes cuja transcrição é normalmente
bloqueada (Figura 5). Os genes induzidos codificam para proteínas “scavengers” de
peróxido de hidrogênio e outras enzimas de defesa em bactérias (Tabela 3). Vários genes regulados por PerR também são homólogos ou análogos àqueles pertencentes a regulon OxyR (Imlay, 2008).
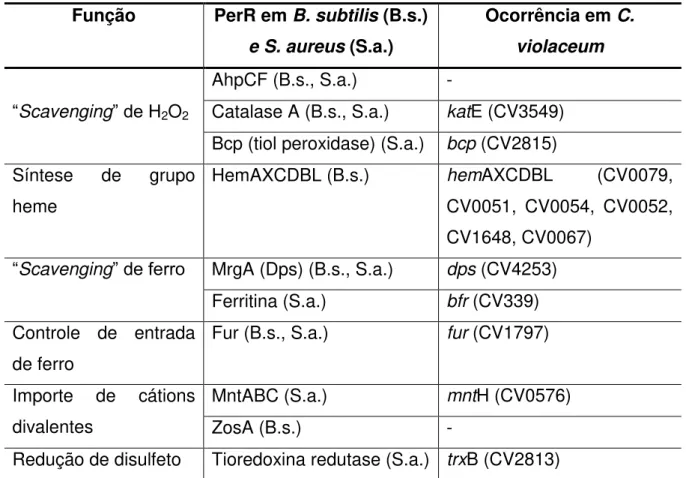
Tabela 3. Membros do regulon PerR de B. subtilis e S. aureus. A ocorrência no
genoma de C. violaceum.
Função PerR em B. subtilis (B.s.) e S. aureus (S.a.)
Ocorrência em C. violaceum
“Scavenging” de H2O2
AhpCF (B.s., S.a.) -
Catalase A (B.s., S.a.) katE (CV3549)
Bcp (tiol peroxidase) (S.a.) bcp (CV2815)
Síntese de grupo heme
HemAXCDBL (B.s.) hemAXCDBL (CV0079,
CV0051, CV0054, CV0052, CV1648, CV0067)
“Scavenging” de ferro MrgA (Dps) (B.s., S.a.) dps (CV4253)
Ferritina (S.a.) bfr (CV339)
Controle de entrada de ferro
Fur (B.s., S.a.) fur (CV1797)
Importe de cátions divalentes
MntABC (S.a.) mntH (CV0576)
ZosA (B.s.) -
Redução de disulfeto Tioredoxina redutase (S.a.) trxB (CV2813)
23
Figura 5. Operação do sistema PerR de B. subilis. A figura mostra as alterações estruturais sofridas
pela proteína PerR quando oxidada pelo H2O2. PerR box é uma região de dupla simetria, o que
possibilita a ligação de PerR. A Proteína PerR na forma ativada de regulador transcricional com ligação a átomo de ferro. H2O2 reagindo com o átomo de ferro da proteína PerR, tornando-a inativa
como repressor da transcrição e liberando a expressão dos genes desse regulon. Adaptado de Zuber, (2009).
2.3.1.2. Antioxidantes enzimáticos
2.3.1.2.1. Superóxido dismutase
A Sod (E.C. 1.15.1.1) está presente em organismos aeróbios e anaeróbios facultativos. Essa enzima caracteriza o grupo da metaloenzima que catalisa a formação de peróxidos de hidrogênio (H2O2) a partir de radicais superóxidos (O2-•),
consumindo-os e, portanto, livrando a célula de riscos de oxidação por esses radicais. A Sod é a primeira enzima de defesa contra danos provocados por EROs nas células (Winterbourn, French e Claridge, 1978).
As Sods são as únicas enzimas cujas atividades interferem nas concentrações de O2-• e H2O2, os dois substratos da reação de Haber-Weiss que
originam os radicais OH-•. Provavelmente por isso, elas representam o mecanismo central de defesa oxidante (Valko et al., 2007).
24
em bactérias e mitocôndria; Fe-SODs, geralmente em procariotos, algas, cloroplastos de algumas plantas. No genoma de C. violaceum, não foi encontrado o
gene da Mn-Sod (Hungria et al., 2004), no entanto foi detectada a duplicação do
gene Fe-Sod (CV0867 e CV2504).
2.3.1.2.2. Catalase
A Catalase (E.C. 1.11.1.6) é uma enzima tetramérica que contém um grupo heme e é encontrada em praticamente todos os organismos vivos. É um componente de defesa antioxidante primário e exerce funções importantes: a decomposição do peróxido de hidrogênio (H2O2) em água e oxigênio (H2O + O2) e a oxidação de
compostos hidrogenados como metanol, etanol, ácido fórmico e fenóis. Devido à sua ampla distribuição e à capacidade de degradar rapidamente o peróxido de hidrogênio (H2O2), concluiu-se que a Cat desempenha um papel fundamental nos
sistemas que capacitam os organismos a viverem em ambientes aeróbicos (Scandalios, 2005). No genoma de C. violaceum, está presente o gene katE
(CV3549), que codifica para uma catalase.
2.3.1.2.3. Glutationas S-transferase
A glutationa (GSH), γ-L-glutamil-L-cisteinil-glicina, é o grupo tiol não proteico mais abundante encontrado na célula de muitos organismos. Em E. coli, a GSH não
é essencial, no entanto, desempenha um papel crítico na proteção contra estresse ambiental, que inclui: choque osmótico, proteção contra toxinas e estresse oxidativo induzido por peróxido de hidrogênio e alquil hidroperóxido (Masip, Veeravalli e Georgiou, 2006). As espécies reativas são removidas via oxidação da glutationa reduzida, já a remoção dos hidroperóxidos se dá pela ação da glutationa peroxidase via catálise enzimática (Martin e Teismann, 2009). Os sistemas tiorredoxinas e/ou GSH/GSSR são os principais agentes redutores nos citoplasmas bacterianos. Trx tem atividade redutora de elétrons de NADPH através de Trx redutase (TrxR). Em E. coli, os genes que codificam para proteínas do sistema Trx têm sua expressão
25
vioalceum, é observada a presença do gene trxB de uma Trx redutase (CV2813) e
três ORFs (CV1107, CV3555, CV3787) que codificam para glutationa pexodase (Hungria et al., 2004)
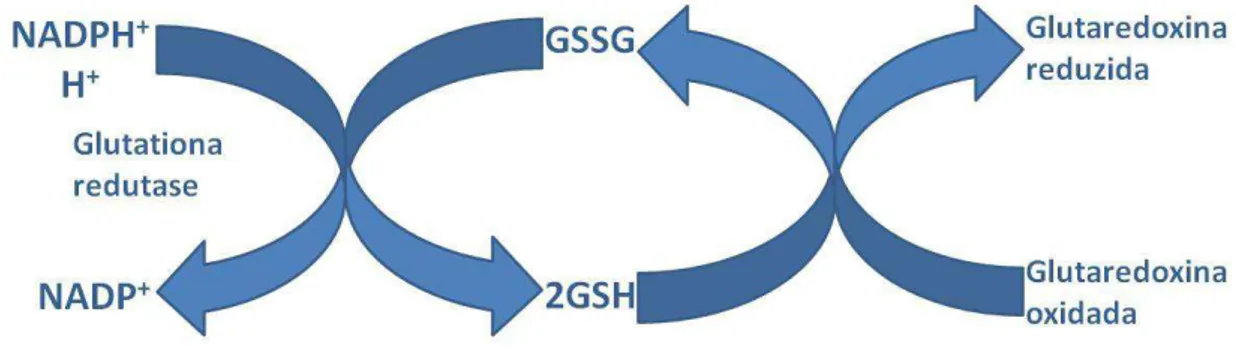
A glutationa redutase, GR (E.C.1.6.4.2), está presente em todos os domínios da vida, ocorrendo de procariotos até eucariotos mais complexos, como plantas e animais (Noctor et al., 2002). Essa enzima atua na regeneração da glutationa
reduzida (GSH) a partir da sua forma oxidada (GSSG) (Gratão et al., 2005) e é
regulada positivamente durante a indução do regulon OxyR (Green e Paget, 2004). O sequenciamento do genoma de C. violaceum revelou a presença dos dois genes
relacionados à biossíntese de GSH, gshA (CV4276) e gshB (CV4276). Também
revelou algumas glutationa-S-transferases (CV0194, CV0289, CV0905, CV0972, CV1086, CV1164, CV1775, CV2424, CV2745, CV3024, CV3306, CV4373) que estão relacionadas com os mecanismos de protecção e podem desempenhar um papel crucial na adaptabilidade (Hungria et al., 2004). No entanto, o gene da
glutationa redutase (GR) não foi identificado.
Figura 6. Ciclo de oxidação-redução das glutationas. Adaptado de Pallardó et al. (2009).
2.4. Relações biotecnológicas de C. violaceum e o estresse oxidativo
26
C. violaceum, organismo conhecido pelo potencial biotecnológico, revelou,
pelo sequenciamento total do seu genoma, aproximadamente 1700 ORFs caracterizadas como hipotéticas ou hipotéticas conservadas, mostrando que grande parte do seu genoma ainda possui potencial a ser explorado (Vasconcelos et al.,
2003). Duarte et al. (2004), realizando análise in sílico das vias de reparo de DNA,
não observaram aspectos muito diferentes desses mecanismos quando comparados com E. coli. Dessa maneira, por se tratar de um organismo de vida livre,
27
3. OBJETIVO
3.1. Objetivo geral
O objetivo geral deste trabalho foi estudar o proteoma diferencial por
shotgun em Chromobacterium violaceum em resposta ao estresse
oxidativo provocado pelo peróxido de hidrogênio.
3.2. Objetivos específicos
Obter o perfil de crescimento de colônias de C. violaceum sob condição de
estresse oxidativo.
Dosar atividade de catalase em extratos proteicos totais de C. violaceum.
Identificar as proteínas expressas pela bactéria quando em presença de peróxido do hidrogênio.
28 4. REFERÊNCIAS BIBLIOGRÁFICAS
ANTONISAMY, P.; KANNAN, P.; IGNACIMUTHU, S. Anti-diarrhoeal and ulcer-protective effects of violacein isolated from Chromobacterium violaceum in Wistar rats. Fundamental & clinical pharmacology, v. 23, n. 4, p. 483-90, ago 2009.
ARAÚJO, G. C. B., TATTO, E. OLIVEIRA, W. K. et al, Investigação de óbitos humanos de causa desconhecida ocorridos no Rio Grande do Norte e de epizootia em sagüis (Callithrix jaccus) no
Parque das Dunas, Rio Grande do Norte, N° 07 - Boletim eletrônico Epidemiológico – SVS. 2004. BARAÚNA, R. A.; CIPRANDI, A.; SANTOS, A. V. et al. Proteomics Analysis of the Effects of Cyanate
on Chromobacterium violaceum Metabolism. Genes, v. 2, n. 4, p. 736-747, 19 out 2011.
BATTISTONI. A. Role of prokaryotic Cu, Zn superoxide dismutase in pathogenesis. Biochem Soc Trans. n. 31 p.1326-92003, 2003.
BERGONIZI, C. Sopra un nuovo bacterio colorato. Annuar Soc. Nat. Modena, Series 2, v. 14, p. 149-158, 1881.
BRITO, C. F. A. D.; CARVALHO, C. B.; SANTOS, F. et al. Chromobacterium violaceum genome:
molecular mechanisms associated with pathogenicity. Genetics and molecular research : GMR, v. 3, n. 1, p. 148-61, jan 2004.
CABISCOL, E.; ROS, J. Oxidative stress in bacteria and protein damage by reactive oxygen species.
Science, p. 3-8, 2000.
CALDAS, L.R., LEITÃO A.A.C., SANTOS, S.M., E TYRRELL, R.M. Preliminary experiments on the photobiological properties of violacein, Intern. Symp. Curr. Topics Radiol. Photobiol. Academia
Brasileira de Ciência. Rio de Janeiro, 121–132, 1978.
CAREPO, M. S. P.; AZEVEDO, J. S. N. D.; PORTO, J. I. R. et al. Identification of Chromobacterium violaceum genes with potential biotechnological application in environmental detoxification. Genetics
and molecular research : GMR, v. 3, n. 1, p. 181-94, jan 2004.
CAVALLERO, D., DURÁN, N. e HAUN, M. Trypanocide. State of Art. Memórias do Instituto Oswaldo Cruz, v. 86, p. 29, 1991.
CHEN, C.H., LIN, L.C., LIU, C.E., YOUNG, T.G. Chromobacterium violaceum bacteremia: a case
report. J Microbiol Immunol Infect.36:141-4, 2003.
CHERNIN, L. S.; WINSON, M. K.; JACQUELYN, M. et al. Chitinolytic Activity in Chromobacterium
violaceum : Substrate Analysis and Regulation by Quorum Sensing. JOURNAL OF BACTERIOLOGY,
n. 17 p. 4435–44, set 1998.
CHRISTMAN, M. F.; MORGAN, R. W.; JACOBSON, F. S.; AMES, B. N. Oxidative Stress and Some Heat-Shock Proteins in Salmonella typhimurium. Physiological Reviews, v. 41, p. 753-762, July
1985.
AZEVEDO, M. B. M. D. E.; ALDERETE, J.; JAIME, A.; FALJONI-ALARIO, A.; HAUN, M. Biological
Activities of Violacein , a New Antitumoral Indole Derivative , in an Inclusion Complex with -Cyclodextrin. Journal Of Inclusion Phenomena And Macrocyclic Chemistry, p. 93-101, 2000.
29
DE SIQUEIRA, I. C., DIAS, J., RUF, H., RAMOS, E. A., MACIEL, E. A., ROLIM, A., LABUR, L., VASCONCELOS, L. S., C. Chromobacterium violaceum in siblings, Brazil. Emerging Infectious
Diseases. n. 11 p.1443-5, set 2005.
DE SOUZA, A. O., AILY, D. C. G., SATO, D. N., DURAN, N. In vitro activity of violacein against Mycobacterium tuberculosis H sub(37)Ra. Revista do Instituto Adolfo Lutz [Rev. Inst. Adolfo Lutz]. n. 58, p. 59-62. 1999.
DIAS, J. P.; SILVANY, C.; SARAIVA, M. M.; RUF, H. R.; GUZMÁN, J. D. Cromobacteriose em Ilhéus,
Bahia : investigação epidemiológica clínica e laboratorial. Revista da Sociedade Brasileira de Medicina Tropical, v. 38, n. 6, p. 503-506, 2005.
DUARTE, F. T.; CARVALHO, F. M. D.; BEZERRA E SILVA, U. et al. DNA repair in Chromobacterium violaceum. Genetics and molecular research : GMR, v. 3, n. 1, p. 167-80, jan 2004.
DUARTE, V.; LATOUR, J.-M. PerR vs OhrR: selective peroxide sensing in Bacillus subtilis. Molecular bioSystems, v. 6, n. 2, p. 316-23, fev 2010.
DUNNING, S.; HANNIVOORT, R. A; BOER, J. F. DE; et al. Superoxide anions and hydrogen peroxide
inhibit proliferation of activated rat stellate cells and induce different modes of cell death. Liver
international : official journal of the International Association for the Study of the Liver, v. 29, n. 6, p. 922-32, jul 2009.
DURÁN, N., CAMPOS, V., RIVEROS, R., JOYAS, A., PEREIRA, M. F., HAUN, M. Bacterial chemistry-III: preliminary studies on trypanosomal activities of Chromobacterium violaceum products. An Acad
Bras Cienc. 1989.
DURÁN, N., CAMPOS, V., RIVEROS, R., JOYAS, A., PEREIRA, M. F., HAUN, M., Bacterial chemistry-III: preliminary studies on trypanosomal activities of Chromobacterium violaceum products. An Academia Brasileira Ciência. n. 61 p. 31-6, mar 1989.
DURÁN, N. e MENCK, C. F. Chromobacterium violaceum: a review of pharmacological and industiral
perspectives. Critical reviews in microbiology, v. 27, n. 3, p. 201-22, jan 2001.
FARAMARZI, M. A; STAGARS, M.; PENSINI, E.; KREBS, W.; BRANDL, H. Metal solubilization from metal-containing solid materials by cyanogenic Chromobacterium violaceum. Journal of biotechnology, v. 113, n. 1-3, p. 321-6, 30 set 2004.
FERREIRA, I. C. F. R., ABREU, R. M. V. Stress oxidativo, antioxidante e fitoquímicos. Sociedade Portuguesa de Bioanalistas da Saúde. p. 32-39, 2007.
GRATÃO, P. L., POLLE, A., LEA, P. J., AZEVEDO, R. A. Making the life of heavy metal-stressed plants a little easier.Functional Plant Biology. n. 32 p. 481-494, 2005.
GREEN, J. e PAGET, M. S. Bacterial redox sensors. Nature reviews. Microbiology, v. 2, n. 12, p. 954-66, dez 2004.
HAN, X. Y., HAN, F. S., SEGAL, J. Chromobacterium haemolyticum sp. nov., a strongly haemolytic
species. International journal of systematic and evolutionary microbiology, v. 58, n. Pt 6, p. 1398-403, jun 2008.
HAUN, M., PEREIRA, M.F., HOFFMANN, M.E., et al. Bacterial chemistry. VI. Biological activities and cytotoxicity of 1,3-dihydro-2H-indol-2-one derivatives. Biological Reseacher. n. 25 p. 21-5, 1992.
KÄMPFER, P.; BUSSE, H.-J.; SCHOLZ, H. C. Chromobacterium piscinae sp. nov. and Chromobacterium pseudoviolaceum sp. nov., from environmental samples. International journal of
30
KODACH, L. L.; BOS, C. L.; DURÁN, N. et al. Violacein synergistically increases 5-fluorouracil
cytotoxicity, induces apoptosis and inhibits Akt-mediated signal transduction in human colorectal cancer cells. Carcinogenesis, v. 27, n. 3, p. 508-16, mar 2006.
LEE, D.; LEE, C. Chilling stress-induced changes of antioxidant enzymes in the leaves of cucumber: in gel enzyme activity assays. Plant science : an international journal of experimental plant
biology, v. 159, n. 1, p. 75-85, 16 out 2000.
LEON L.,L., MIRANDA C.,C., De SOUZA A.,O., DURAN N. Antileishmanial activity of the violacein extracted from Chromobacterium violaceum. Journal of Antimicrobial Chemotherapy. n. 48 p. 449–
450, set 2001.
LOPES, S. C. P.; BLANCO, Y. C.; JUSTO, G. Z. et al. Violacein extracted from Chromobacterium violaceum inhibits Plasmodium growth in vitro and in vivo. Antimicrobial agents and chemotherapy,
v. 53, n. 5, p. 2149-52, maio 2009.
LIMA-BITTENCOURT, C. I.; ASTOLFI-FILHO, S.; CHARTONE-SOUZA, E.; SANTOS, F. R.; NASCIMENTO, A. M. A. Analysis of Chromobacterium sp. natural isolates from different Brazilian
ecosystems. BMC microbiology, v. 7, p. 58, jan 2007.
LUSHCHAK, V. I. Adaptive response to oxidative stress: Bacteria, fungi, plants and animals.
Comparative biochemistry and physiology. Toxicology & pharmacology : CBP, v. 153, n. 2, p. 175-90, mar 2011.
MACHER, A. M., CASALE, T. B., FAUCI, A.S. Chronic granulomatous disease of childhood and
Chromobacterium violaceum infections in the southeastern United States. Ann Intern Med. n. 97 p.
51-5, Jul 1982.
MAILLOUX, R. J.; LEMIRE, J.; APPANNA, V. D. Metabolic networks to combat oxidative stress in Pseudomonas fluorescens. Antonie van Leeuwenhoek, v. 99, n. 3, p. 433-42, mar 2011.
MARTIN, H. L.; TEISMANN, P. Glutathione--a review on its role and significance in Parkinson’s
disease. FASEB journal : official publication of the Federation of American Societies for
Experimental Biology, v. 23, n. 10, p. 3263-72, out 2009.
MARTIN, P. A W.; GUNDERSEN-RINDAL, D.; BLACKBURN, M.; BUYER, J. Chromobacterium subtsugae sp. nov., a betaproteobacterium toxic to Colorado potato beetle and other insect pests.
International journal of systematic and evolutionary microbiology, v. 57, n. Pt 5, p. 993-9, maio 2007.
MATZ, C., DEINES, P., BOENIGK, J. et al. Impact of Violacein-Producing Bacteria on Survival and
Feeding of Bacterivorous Nanoflagellates Impact of Violacein-Producing Bacteria on Survival and Feeding of Bacterivorous Nanoflagellates. Society, 2004.
MCCLEAN, K. H.; WINSON, M. K.; FISH, L. et al. Quorum sensing and Chrornobacteriurn violaceurn :
exploitation of violacein production and inhibition for the detection of N-acyl homoserine lactones.
Molecular Microbiology, n. 143 p. 3703-11, 1997.
MELO, P. S., MARIA, S. S., VIDAL, B. C., HAUN, M., DURÁN, N. Violacein cytotoxicity and induction of apoptosis in V79 cells. In Vitro Cell Dev Biol Anim. n. 36 p. 539-43, set 2000.
Mergaert, J., Schirmer, A., Hauben,L., Mau, M., Hoste, B., Kersters, K., Jendrossek, D., Swings, J. Isolation and identification of poly(3-hydroxyvalerate)-degrading strains of Pseudomonas lemoignei.
Intern. J. Syst. Bacteriol. 46: 769-773 (1996).
MERGAERT, J., SCHIRMER, A., HAUBEN, L., et al. Isolation and identification of
31
MICHAELS, R.; CORPE, W. A. Cyanide Formation By Chromobacterium Violaceum. Journal of
bacteriology, v. 89, n. 1, p. 106-12, jan 1965.
MILLER, G. W. Paraquat: the red herring of Parkinson’s disease research. Toxicological sciences :
an official journal of the Society of Toxicology, v. 100, n. 1, p. 1-2, nov 2007.
MOMEN, A. Z. e HOSHINO, T. Biosynthesis of violacein: intact incorporation of the tryptophan molecule on the oxindole side, with intramolecular rearrangement of the indole ring on the 5-hydroxyindole side. Biosci Biotechnol Biochem. n. 64 p. 539-49, mar 2000.
MULLER, F. L.; LUSTGARTEN, M. S.; JANG, Y.; RICHARDSON, A.; REMMEN, H. VAN. Trends in oxidative aging theories. Free radical biology & medicine, v. 43, n. 4, p. 477-503, 15 ago 2007.
NETTO, L. E. S.; OLIVEIRA, M. A. DE; MONTEIRO, G. et al. Reactive cysteine in proteins: protein
folding, antioxidant defense, redox signaling and more. Comparative biochemistry and physiology.
Toxicology & pharmacology : CBP, v. 146, n. 1-2, p. 180-93, 2007.
NOCTOR, G.; FOYER, C. H. ASCORBATE AND GLUTATHIONE: Keeping Active Oxygen Under Control. Annual review of plant physiology and plant molecular biology, v. 49, p. 249-279, jun 1998.
NOCTOR, G.; FOYER, C. H. ASCORBATE AND GLUTATHIONE: Keeping Active Oxygen Under Control. Annual review of plant physiology and plant molecular biology, v. 49, p. 249-279, jun 1998.
PALLARDÓ, F. V.; MARKOVIC, J.; GARCÍA, J. L.; VIÑA, J. Role of nuclear glutathione as a key regulator of cell proliferation. Molecular aspects of medicine, v. 30, n. 1-2, p. 77-85, 2009.
PUDDU, P., PUDDU, G. M., CRAVERO, E., et al A. The molecular sources of reactive oxygen species
in hypertension. Blood Press. n. 17 p. 70-7, 2008.
QUAN, L.-J.; ZHANG, B.; SHI, W.-W.; LI, H.-Y. Hydrogen peroxide in plants: a versatile molecule of the reactive oxygen species network. Journal of integrative plant biology, v. 50, n. 1, p. 2-18, jan 2008.
RECOUVREUX, D. O. S.; CARMINATTI, C. A; PITLOVANCIV, A. K. et al. Cellulose biosynthesis by
the beta-proteobacterium, Chromobacterium violaceum. Current microbiology, v. 57, n. 5, p. 469-76, nov 2008.
SÁNCHEZ, C., BRAÑA, A. F., MÉNDEZ, C., SALAS, J.A. Reevaluation of the violacein biosynthetic pathway and its relationship to indolocarbazole biosynthesis. Chembiochem. n. 7 p.1231-40, Aug 2006.
SCANDALIOS, J. G. Oxidative stress: molecular perception and transduction of signals triggering antioxidant gene defenses. Brazilian journal of medical and biological research = Revista brasileira de pesquisas médicas e biológicas / Sociedade Brasileira de Biofísica, v. 38, n. 7, p. 995-1014, jul 2005.
SHIGEMATSU N, UEDA H, TAKASE S, TANAKA H, YAMAMOTO K, TADA T. FR901228, a novel antitumor bicyclic depsipeptide produced by Chromobacterium violaceum No. 968. II. Structure determination. J Antibiot (Tokyo). n. 47 p. 311-4, mar 1994.
SHINODA, K.; HASEGAWA, T.; SATO, H. et al. Biosynthesis of violacein: a genuine intermediate,
protoviolaceinic acid, produced by VioABDE, and insight into VioC function. Chemical communications (Cambridge, England), n. 40, p. 4140-2, 28 out 2007.
SHIRATA, A., TSUKAMOTO, T., YASUI, et al. Isolation of bacteria producing bluishpurple pigment
32
SNEATH, P.H, WHELAN J.P, BHAGWAN SINGH, R, EDWARDS, D. Fatal infection by
Chromobacterium violaceum. Lancet, n. 265, p. 276-7, 8 Aug 1953
UEDA, H., MANDA, T., MATSUMOTO, S., et al. K. FR901228, a novel antitumor bicyclic depsipeptide
produced by Chromobacterium violaceum No. 968. III. Antitumor activities on experimental tumors in
mice. J Antibiot (Tokyo). n. 47 p. 315-23, mar 1994.
VALKO, M.; LEIBFRITZ, D.; MONCOL, J. et al. Free radicals and antioxidants in normal physiological
functions and human disease. The international journal of biochemistry & cell biology, v. 39, n. 1, p. 44-84, jan 2007.
VASCONCELOS, A. T. R., ALMEIDA, D. F., HUNGRIA, M., GUIMARÃES, C. T. et al. The complete
genome sequence of Chromobacterium violaceum reveals remarkable and exploitable bacterial
adaptability. Proceedings of the National Academy of Sciences of the United States of America, v. 100, n. 20, p. 11660-5, 30 set 2003.
WANG, C.; WESENER, S. R., ZHANG, H., CHENG, Y.-Q. An FAD-dependent pyridine nucleotide-disulfide oxidoreductase is involved in nucleotide-disulfide bond formation in FK228 anticancer depsipeptide.
Chemistry & biology, v. 16, n. 6, p. 585-93, 26 jun 2009.
WINTERBOURN, C. C. , FRENCH, J. K, CLARIDGE, R. F Superoxide dismutase as an inhibitor of reactions of semiquinone radicals. FEBS Lett, n. 94 p.269–272, 1978.
YARMUSH, M. L.; JAYARAMAN, A. Advances in proteomic technologies. Annual review of biomedical engineering, v. 4, p. 349-73, jan 2002.
YOO, C. B.; JONES, P. A. Epigenetic therapy of cancer: past, present and future. Nature reviews. Drug discovery, v. 5, n. 1, p. 37-50, jan 2006.
YOUNG, C.-C.; ARUN, A B.; LAI, W.-A. et al. Chromobacterium aquaticum sp. nov., isolated from
spring water samples. International journal of systematic and evolutionary microbiology, v. 58, n. Pt 4, p. 877-80, abr 2008.
33
5. Artigos
34
SHOTGUN PROTEOMICS OF CHROMOBACTERIUM
VIOLACEUM: Comparison of proteome changes elicited by
hydrogen peroxide
Fábio Teixeira Duarte1,Daniel Chaves de Lima1, Viviane Katielly Silva Medeiros2, Gabriel Duarte Tavares Araújo3; Gilberto Barbosa Domont3*; Silvia Regina Batistuzzo
de Medeiros1*
1Laboratório de Biologia Molecular e Genômica (Universidade Federal do Rio
Grande do Norte), 2Colegiado de Medicina (Universidade Federal do Vale do São Francisco), 3Unidade Proteômica (Universidade Federal do Rio de Janeiro).
*Corresponding authors:
Mailing address:
Silvia Regina Batistuzzo de Medeiros, Departamento de Biologia Celular e Genética, Centro de Biociências, Universidade Federal do Rio Grande do Norte, Campus Universitário s/n, Lagoa Nova, Natal-RN, CP 1575, ZC 59072-970, Brazil.
Tel: # 55.84.3211-9209 Fax # 55.84.3215-3346 E-mail address: sbatistu@cb.ufrn.br
Gilberto Barbosa Domont, Unidade Proteômica, Laboratório de Química de Proteínas, Bloco A - sala 543, Centro de Tecnologia, Universidade Federal do Rio de Janeiro, Av. Athos da Silveira Ramos, 149, Cidade Universitária, Rio de Janeiro, RJ, 21941 - 909- Brazil.
35
ABSTRACT
The -proteobacterium Chromobacterium violaceum is a free-living bacillus,
Gram-negative, saprophyte and an opportunistic pathogen that inhabits tropical and subtropical ecosystems, including soil and water of the Amazon region. Its genome was completely sequenced in 2003 because of a biotechnological potential. The genome analysis showed that this bacterium has several genes with functions related to the ability to survive under different environmental stress. Aiming to understand the physiological response of C. violaceum to oxidative stress, a differential protein
expression study was conducted. To this end, total proteins extracted from control and treated cultures of C. violaceum for two hours with 8mM hydrogen peroxide were
analyzed using shotgun proteomics. Analysis revealed 131 proteins exclusively expressed in control condition, 177 proteins that began to be expressed under
oxidative stress and 1175 proteins expressed in both conditions. Results showed that under oxidative stress the bacterium increases the abundance of proteins able to reverse the harmful effects and decreases the abundance of proteins responsible for bacterial growth and catabolism (transcription, translation, carbon and fatty acids metabolism). The proteomic approach provides an overview of the metabolic pathways involved in the response of C. violaceum to oxidative stress, as well as
amplifies the understanding of the physiological response to environmental stress. In conclusion, C. violaceum elicits a complex, efficient and versatile mechanism in
response to oxidative stress.
36
Introduction
Chromobacterium violaceum is a free-living gram-negative saprophytic
bacillus, an opportunistic pathogen that inhabits the soil and waters of the Rio Negro in the Brazilian Amazon [1]. The organism is known for its abilities to produce
cyanide [2], solubilize gold [3], produce chitinolytic enzymes [4] and synthesize
bioplastics [5]; it can be used for bioremediation [6], production of cellulose [7] and as an anticancer agent [8]. Its primary characteristic is the production of a violet
pigment, violacein, with several pharmacological and biotechnological applications [9]. Due to a significant potential, the full genome was sequenced in 2003, identifying a circular chromosome of 4,751,080 bp containing 4,431 ORFs [10].
Genome analysis showed that this organism contains several genes associated to environmental acclimation and stress tolerance, indicating
physiological versatility in regard to environmental changes [11]. Analyzed the DNA repair genes [12] of this organism and recorded an absence of PolII and LexA, key proteins in pathways related to genome stability maintenance. The LexA protein acts as a repressor of the SOS regulon, a physiological response provoked by the
presence of DNA damage [13]. As yet there are no studies investigating the C. violaceum response to environmental stress.
In bacteria, several environmental factors induce a change in the redox state of the cellular environment and lead to oxidative stress. These include exposure to acids, high salt concentrations and treatment with antimicrobial components, as well as other biotic and abiotic factors [14].
37
induces oxidative stress and is considered one of the more stable ROS. It can
accumulate to significant levels within the cell and is a widely used chemical model in experimental studies of oxidative stress. It is produced endogenously within
organisms as a byproduct of oxidative phosphorylation and other reactions. In mammals, plants and even bacteria, it is produced in elevated levels as a form of defense against pathogens [16].
A number of studies into the modulation of gene expression, such as
transcriptome and proteome, use hydrogen peroxide as a model to analyze oxidative stress [17–23]. These investigations detected changes in the expression pattern of several genes, including those involved in general stress response. They also found that exposure to hydrogen peroxide induces the gene expression and mechanisms associated with oxidative stress, such as genes that encode catalase, thioredoxin reductase and peroxidase, mechanisms of iron and manganese homeostasis and SOS response.
High peroxide levels in the cellular environment can damage proteins with prosthetic groups of iron, releasing them. Iron, in turn, reacts with hydrogen peroxide through the Fenton reaction, forming highly reactive species such as hydroxyl radical (•OH) [24], which oxidize biomolecules including lipids, DNA and proteins [25].
The intense selective pressure imposed by oxidative stress has resulted in several detoxification and defense pathways [26]. Chromobacterium violaceum is
subject to these pressures due to its lifestyle, where it is constantly exposed to
38
investigate the protein differential expression under hydrogen peroxide-induced oxidative stress, using proteomic tools.
Material and Methods
Reagents and chemicals
Reagents used in electrophoresis were obtained from GE Health Care (Uppsala, Sweden) and those employed to make the culture medium were from Fisher BioRagents (Fisher Scientific). For protein measurement we used the Bradford reagent (Bio-Rad) and for hydrolysis sequencing grade-modified trypsin (Promega, Madison, WI). MilliQ water was used for all the experiments (Millipor-e, MA).
Bacterial strain, maintenance and growth conditions
The bacterial strain used in this study was Chromobacterium violaceum ATCC
12472. It was inoculated into Petri dishes with LB (Luria-Bertani) agar and incubated at 28 °C for 24 h. Individual colonies were aerobically pre-inoculated into 50 mL Falcon tubes containing 5 mL aliquots of liquid LB and submitted to agitation at 200 rpm and 28 °C for 16 hours. Pre-inoculated samples were transferred to 250 mL Erlenmeyer flasks in a fresh medium at a ratio of 10:1. Bacteria were cultivated under the same conditions until reaching the exponential growth phase in an optical density (OD) at 600 nm of 0.4 – 0.5. Control samples remained under growth conditions and for treatment these were added to the 8 mM H2O2 cultures, continuing cultivation for