Clones de Streptococcus pyogenes associados a elevada frequência de colonização da orofaringe
Texto
(2) UNIVERSIDADE DE LISBOA FACULDADE DE CIÊNCIAS DEPARTAMENTO DE BIOLOGIA VEGETAL. Clones de Streptococcus pyogenes associados a elevada frequência de colonização da orofaringe. Paula Maria Monteiro Fernandes. Orientadora: Prof. Doutora Ilda Sanches (FCT-UNL/CREM) Orientadora Interna: Prof. Doutora Lélia Chambel (FCUL/BioFIG). Mestrado em Microbiologia Aplicada 2011.
(3) Clones de Streptococcus pyogenes associados a elevada frequência de colonização da orofaringe. Paula Maria Monteiro Fernandes. Tese de Mestrado 2011. Esta tese foi realizada no Centro de Recursos Microbiológicos (CREM/FCTUNL) sob a orientação externa da Prof. Doutora Ilda Sanches e no Centro de Biodiversidade, Genómica Integrativa e Funcional (BioFIG/FCUL) sob a orientação interna da Prof. Doutora Lélia Chambel no âmbito do Mestrado em Microbiologia Aplicada da Faculdade de Ciências da Universidade de Lisboa..
(4) “A maior parte das gaivotas não se querem incomodar a aprender mais que os rudimentos do voo, como ir da costa à comida e voltar. Para a maior parte das gaivotas, o que importa não é saber voar, mas comer. Para esta gaivota, no entanto, o importante não era comer, mas voar”.. Richard Bach.
(5) AGRADECIMENTOS. Pela contribuição para a realização do presente trabalho, gostaria de manifestar os meus agradecimentos: À Professora Doutora Ilda Sanches, orientadora externa desta tese de Mestrado, pela disponibilidade, ensinamentos, críticas, sugestões e incentivo demonstrados ao longo do decorrer da minha Tese. À Professora Doutora Lélia Chambel, orientadora interna desta tese de Mestrado, pela sua disponibilidade, saber, experiência, sugestões e incentivo demonstrados ao longo do decorrer da minha Tese. Ao Professor Doutor Rogério Tenreiro, Coordenador do Mestrado de Microbiologia Aplicada, pelo incentivo e por ter disponibilizado as condições necessárias à realização da parte inicial do trabalho desta Tese, no Instituto de Ciência Aplicada e Tecnologia - ICAT. À Professora Doutora Teresa Semedo, pela integração e apoio concedido no início do trabalho de presente Tese, no ICAT. À Doutora Sandra Chaves, pelos esclarecimentos pontuais na fase inicial laboratorial do meu trabalho, no ICAT. Às companheiras de trabalho e de almoço no ICAT: Joana e Lara. Ao Renato Pires, pela prontidão e esclarecimentos acerca do meu trabalho laboratorial desenvolvido na FCT/UNL e por me ter facultado os seus resultados ainda não publicados, no âmbito da sua Tese de Doutoramento, na qual está incluída esta Tese de Mestrado. Ao Doutor João Carriço, Ana Friães e Catarina Silva Costa da Unidade de Microbiologia Molecular e Infecção do Instituto de Medicina Molecular (IMM) (responsável: Prof. Doutor Mário Ramirez), todo o apoio na utilização do “software” BioNumerics. À Márcia Rato, pelos esclarecimentos prestados enquanto desenvolvia o meu trabalho prático na FCT/UNL. Ao Renato Pires, à Márcia Rato e à Joana Ministro, pelo companheirismo demonstrado no trabalho de Laboratório da FCT/UNL e nas pausas de almoço e lanche. À Drª Maria da Luz Antunes, Documentalista da Escola Superior de Tecnologia de Lisboa, pela prontidão na facilitação de artigos científicos para a redacção da presente Tese. Às colegas do IPS, pelas trocas de turnos de serviço, de forma a conciliar o serviço e o trabalho laboratorial desta Tese. Aos amores da minha vida, José e Magda, por todo o amor e carinho que me dedicaram durante esta etapa da minha vida. Aos meus pais, José e Noémia, pela compreensão, apoio e toda ajuda prestada..
(6) Resumo. Os estudos de prevalência ou transversais epidemiológicos de Streptococcus do Grupo A são muito frequentes, quando os isolados são associados a diversas infecções no entanto, estudos deste tipo são raros quando os isolados são associados a colonização. Pretendeu-se identificar os clones de S. pyogenes por métodos genotípicos (“Pulsed Field Gel Electrophoresis”- PFGE, emm “typing”, “Multilocus Sequence Type”- MSLT) em 420 isolados de colonização da cavidade orofaringea de crianças, funcionários e familiares de dez Creches/Jardins de Infância, associados a uma elevada taxa de colonização (14%). O PFGE, sendo um método de elevado poder discriminatório, revelou uma heterogeneidade de padrões, a maior parte (397 isolados) com relação clonal. Os tipos emm mais frequentes nos isolados de colonização foram o emm 1, 3 e 12 que, comummente, se encontram associados a isolados invasivos (emm 1 e 3) e não invasivos (emm 12). O PFGE, o emm “typing” e o “Multilocus Sequence Type” definiram as linhagens de S. pyogenes. Algumas delas encontram-se amplamente disseminadas pelo mundo (ST 36, ST 39 e ST 46), outras apresentam uma grande longevidade (ST 15, ST 28, ST 36, ST 55) e todas associadas a infecção, sugerindo que os portadores assintomáticos podem ser reservatório de estirpes virulentas e que podem disseminá-las na comunidade.. Palavras chave: Streptococcus pyogenes; colonização; portadores assintomáticos saudáveis; clones; linhagens genéticas..
(7) Abstract. The epidemiological prevalence studies of the Lancefield group A streptococci (GAS) are frequently related with infections isolates but are rare among colonization isolates. The aim of our study was to identify the S. pyogenes clones by genotypic methods (Pulsed Field Gel Electrophoresis- PFGE, emm typing and Multilocus Sequence Type- MLST) among the 420 colonization isolates collected from the oropharyngeal cavity of healthy child and contact groups that attend to Day Care Centers, associated with high colonization rates (14%). The heterogeneity patterns revealed by PFGE are an omen of its high discriminatory power and allowed to verify that, from the 420 isolates, 397 were clonally related. The emm types emm 1, emm 3 and emm 12 were the most prevalent in colonization isolates and, usually, emm 1 and 3 are associated an invasive infection while emm 12 is associated with noninvasive infection. The genetic lineages of S. pyogenes were defined by PFGE, emm typing and MLST. Some of them are world widely disseminated (Sequence Type- ST 36, ST 39 and ST 46) and others persist for long decades (ST 15, ST 28, ST 36, ST 55). All ST were associated with infection, suggesting that the asymptomatic carriers may be considered virulent strains reservoirs and can spread them in the community. Keywords: Streptococcus pyogenes; colonization; healthy asymptomatic carriers; clones; genetic lineages..
(8) Índice 1- Introdução. 1. 1.1- Características do género Streptococcus. 1. 1.2- Caracterização da espécie Streptococcus pyogenes. 1. 1.3- Patologias associadas a Streptococcus do Grupo A. 2. 1.3.1- Infecções supurativas. 2. 1.3.2- Infecções não supurativas ou sequelas. 3. 1. 4- Constituintes celulares e factores de virulência de S. pyogenes 1.4.1- Constituintes celulares intrínsecos:. 3 3. 1.4.1.1- Proteína M. 3. 1.4.1.2- Proteína T. 4. 1.4.1.3- Ácido hialurónico. 5. 1.4.2- Proteínas extracelulares. 5. 1.4.3- As exotoxinas pirogénicas. 5. 1. 5- Transmissão da infecção. 6. 1.6- Objectivos deste estudo. 6. 2- Material e Métodos. 7. 2.1- Participantes/Amostra. 7. 2.2- Conservação das estirpes bacterianas. 7. 2.3- Estirpes bacterianas e condições de crescimento. 8. 2.4- Identificação presuntiva de Streptococcus pyogenes. 8. 2.4.1- Testes Serológicos. 8. 2.4.1.1- Hidrólise do L-pirrolidonil-β-naftil-amido (PYR). 8. 2.4.1.2- Determinação do grupo A de Lancefield. 8. 2.4.1.3- T-“typing”. 8. 2.4.2- Teste de susceptabilidade à bacitracina. 8. 2.5- Métodos genotípicos. 9. 2.5.1- Electroforese em Gel de Agarose por Campo Pulsado- “Pulsed Field Gel Electrophoresis, PFGE”. 9. 2.5.1.1- Propagação das culturas. 9. 2.5.1.2- Lavagem celular e ajuste da concentração celular. 9. 2.5.1.3- Preparação dos discos de agarose. 10. 2.5.1.4- Lise celular. 10. 2.5.1.5- Desproteinização. 10. 2.5.1.6- Lavagem para remoção da proteinase K. 11. 2.5.1.7- Restrição do DNA com a enzima SmaI. 11. 2.5.1.8- Preparação do gel com os discos de DNA. 11. 2.5.1.9- Coloração do gel e digitalização da imagem. 11. 2.5.1.10- Análise dos perfis de PFGE. 11.
(9) 2.5.2- emm “typing”. 12. 2.5.3- MLST- “Multilocus Sequence Typing”. 12. 2.5.3.1- Análise das sequências no “software DNAStar- SeqMen”. 12. 2.5.3.2- Análise dos dados de MLST- “eBURST”. 13. 2.5.4- Inferência Estatística. 13. 3- Resultados. 14. 3.1- Amostra. 14. 3.2- Taxa de colonização. 14. 3.3- Teste de sensibilidade à bacitracina. 15. 3.4- T “typing”. 15. 3.5- Critérios para avaliação dos métodos de tipificação. 16. 3.5.1- Tipabilidade. 16. 3.5.2- Reprodutibilidade. 16. 3.5.3- Poder discriminatório/ Índice de Simpson/ Intervalos de Confiança. 16. 3.6- PFGE- “Pulsed Field Gel Electrophoresis” 3.6.1- Tipabilidade. 17 17. 3.6.2- Prevalência dos padrões de PFGE nos DCCs e sua localização Geográfica. 17. 3.6.3- Reprodutibilidade de PFGE. 19. 3.6.4- Dendrograma. 20. 3.7- emm-“typing”. 22. 3.7.1- Tipabilidade. 22. 3.7.2- Diversidade dos tipos emm. 22. 3.7.3- Distribuição dos tipos emm, de 2000 a 2007. 23. 3.7.4- Análise da distribuição dos tipos emm na Primavera, no Outono e no Inverno. 24. 3.8- Determinação do Coeficiente de Wallace. 25. 3.9- MLST- “Multilocus Sequense type”. 26. 3.9.1. Tipabilidade. 26. 3.9.2- “eBURST”. 26. 3.9.3- Comparação de isolados de colonização de S. pyogenes com os da base de dados do MLST 3.10- Linhagens Genéticas. 27 30. 3.11- Poder Discriminatório e Intervalos de Confiança de PFGE, emm “typing” e MLST 31 3.12- Inferência Estatística 3.12.1-Teste de independência do Qui-Quadrado- simulação de Monte-Carlo. 31 31. 4- Discussão/Conclusão. 34. 5- Referências. 37.
(10) 6- Anexos. 40. Anexo I. 40. Anexo II. 43. Anexo III. 44. Anexo IV. 46. Anexo V. 46. Anexo VI. 46. Anexo VII. 47. Anexo VIII. 49. Anexo IX. 50. Anexo X. 51.
(11) 1- Introdução 1.1- Características do género Streptococcus. Actualmente existem 17 géneros diferentes de cocos Gram positivos, sendo um deles o género Streptococcus (1), o qual vai ser objecto do nosso estudo. Os cocos agrupam-se em cadeias, e apresentam outras características, tais como: catalase negativa (não hidrolisam o peróxido de hidrogénio devido à ausência de catalase), anaeróbios facultativos, imóveis e não formam esporos. Têm como seu habitat preferido o Homem e os Animais, podendo causar-lhes infecções. A classificação das espécies do género Streptococcus, clinicamente significativas, foi difícil devido à falta de características fenotípicas decisivas (3). Em 1994, Stackebrandt, E. & Goebel, B.M. (2) descobriram que as relações filogenéticas das espécies procariotas podem ser estabelecidas com base na sequência do 16S rRNA para níveis de homologia inferiores a 97% e o seu posicionamento taxonómico pode ser confirmado por hibridização DNA-DNA, quando a homologia é superior a 97% (3). Assim, em 2002, Facklam, R.R. (1) apresenta a árvore filogenética de 55 espécies incluídas no género Streptococcus, baseada na análise da sequência do 16S rRNA. 1.2- Caracterização da espécie Streptococcus pyogenes. Segundo Facklam, R.R. (1) a hemólise das colónias em agar de sangue ajuda na classificação do género Streptococcus ao nível da espécie. As colónias de S. pyogenes têm diâmetro superior a 0,5 mm, variam entre o transparente e o translúcido, convexas, circulares, brilhantes, rodeadas por uma ampla β-hemólise em agar suplementado com de sangue de carneiro como se pode observar na fig.1. A hemólise parcial, muitas das vezes apresenta a coloração verde em torno das colónias em meio com sangue e designa-se por α-hemólise, ao passo que γ-hemólise corresponde à ausência de hemólise em torno das colónias em agar de sangue. Esta característica fenotípica é muito usada em Microbiologia Médica (4).. Figura 1- β-Hemólise de S.pyogenes. (http://spyogenes.mlst.net/). Outra característica fenotípica da espécie é a presença, na parede celular de S. pyogenes, de um polissacárido antigénico constituído por N-acetil-β-D-glucosamina ligada a um polímero de ramnose, cuja. 1.
(12) reactividade serológica permite classificar esta espécie como pertencente a Streptococcus do Grupo A (GAS) (5). Até à data conhecem-se doze genomas de S.pyogenes: dois M1, um M2, dois M3, um M4, um M5, um M6, dois M12, um M18 e um M28 (6). Também se sabe que o teor de G+C das espécies piogénicas do GAS varia entre os 38,5% e os 38,6% e que o seu genoma varia entre os 1,85-1,9 Mb (7). A espécie mais patogénica deste género é S. pyogenes, descrevendo-se de seguida as doenças mais características desta espécie. 1.3- Patologias associadas a Streptococcus do Grupo A Streptococcus do Grupo A (GAS) coloniza1 a garganta e a pele, podendo vir a ser responsável por inúmeras infecções supurativas e não supurativas (sequelas). Madigan, M.T., et al., (8) refere que a microbiota de um indivíduo depende bastante das condições a que se encontra exposto e que é muito diversificada, podendo diferir bastante entre indivíduos e até mesmo numa dada população, como se pode observar na tabela 1. Tabela 1– Géneros representativos da microbiota humana.. Madigan, M.T., et al., (8).. 1.3.1- Infecções supurativas Nas infecções supurativas existem as infecções piogénicas (faringite e tonsilite), a celulite, o impétigo, a erisipela, a escarlatina, a fasceíte necrosante (NF- “Necrotizing Fasciitis”), a síndrome do choque tóxico estreptocócico (STSS- “Streptococcal Toxic Shock Syndrome”) e a miosite estreptocócica. Esta diversidade de infecções envolve as membranas das mucosas, a pele e os tecidos mais profundos, variando as mesmas entre o moderado e o extremamente severo. 1. Colonização- proliferação de um microorganismo após anexação aos tecidos do hospedeiro ou outras superfícies. Madigan, et al. (8).. 2.
(13) A faringite afecta as crianças em idade escolar, dos 5 aos 15 anos (5) e também adultos como os recrutas no Texas (10), devido à facilidade que Streptococcus apresenta em se espalhar por entre as multidões, nomeadamente através da tosse e espirros. A faringite associada à febre reumática ocorre na Primavera e Inverno, ao passo que as infecções da pele ocorrem no Verão, e podem estar associadas à glomerulonefrite aguda (5). Os serótipos mais frequentes de S. pyogenes associados à faringite e à febre reumática são os M1, 3, 5, 6, 14, 18, 19 e 24. Os serótipos M que causam impétigo (infecção da pele) são diferentes destes. Quanto às infecções da pele (impétigo, erisipela e celulite) podem ser causadas pelo GAS, mas no caso da erisipela e da celulite também podem ser causadas pelos grupos B, C e G. A erisipela afecta as camadas superficiais da pele, enquanto que a celulite afecta os tecidos sub-cutâneos. Nestes casos, a porta de entrada do GAS é as queimaduras e outros tipos de lesões na pele. Por outro lado, a fasceíte necrosante e a STSS são infecções invasivas. A primeira, destrói a gordura perimuscular e a fascia ou bainha muscular, apresentando elevada taxa de mortalidade, e a segunda, afecta vários órgãos (10). 1.3.2- Infecções não supurativas ou sequelas As sequelas deixadas por S. pyogenes são a febre reumática e a glomerulonefrite. A febre reumática pressupõe a presença de alguns dos seguintes sinais: poliartrite, lesão cardíaca. Os serótipos mais frequentes são M5, 6 e 18. Na glomerulonefrite aguda (GNA), os serótipos envolvidos diferem conforme a infecção tenha surgido na faringe ou na pele. A GNA pós-angina aparece em crianças e jovens, ao passo que a GNA pós-cutânea surge em todas as idades. Nas sequelas, o isolamento de GAS não é possível. O diagnóstico é feito com base na etiologia imunológica por síntese de anticorpos antiestreptocócicos que reconhecem antigénios semelhantes no coração ou no rim (10). 1.4- Constituintes celulares e factores de virulência de S. pyogenes. O conhecimento dos factores de virulência é o primeiro passo para a descoberta de vacinas e fármacos, daí a sua importância em os descrever (11). 1.4.1- Constituintes celulares intrínsecos: 1.4.1.1- Proteína M A proteína M é um factor major de virulência de Streptococcus do Grupo A e está associada a resistência à fagocitose e adesão às células (12). Durante décadas, a determinação serológica da proteína M foi o modelo adoptado, baseado na diversidade antigénica desta proteína. Em 1962, Lancefield desenvolveu um método para preparar extractos de Streptococcus para tipagem serológica. Desde então outros métodos têm sido desenvolvidos, o que permitiu identificar 124 genótipos M diferentes (7).. 3.
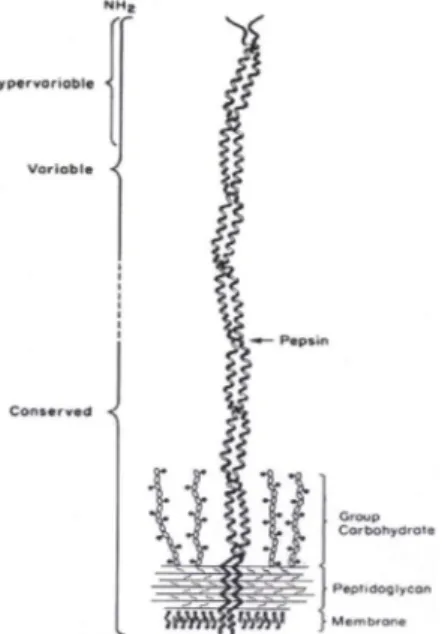
(14) A proteína M exibe na sua superfície fímbrias (5). Cada fímbria consiste numa só molécula dimera, com 50 a 60 nm de comprimento, não sendo composta por múltiplas sub-unidades como no caso dos pili das bactérias Gram negativas (13). Por outro lado, a sequência completa da proteína M6 (emm6) permitiu compreender a sequência amino acídica desta proteína. A proteína M é composta por duas cadeias polipeptídicas com uma conformação αhélice (7). O segmento C terminal é composto por animoácidos hidrofóbicos, ancorado na membrana celular que atravessa a camada de peptidoglicano, sendo este rico em prolina e glicina. Segue-se uma região central que tolera uma vasta variação de sequências de amino ácidos. A região N-terminal desempenha um papel importante na actividade biológica da molécula uma vez que leva à produção de anticorpos pelo hospedeiro. Esta região designa-se por hipervariável, como se pode observar na figura 2. É de referir ainda que a proteína M49 não possui o segmento N-terminal (14).. Figura 2- Modelo da proteína M6, da estirpe D471, baseado em dados da estrutura e da sequência da proteína. A sequência nucleotídica da região N-terminal tem aproximadamente 11 aminoácidos e denomina-se hipervariável por variar muito entre estirpes de S. pyogenes. Fischetti, V.A. (13).. No Homem, a proteína M liga-se a proteínas do hospedeiro (por exemplo, o factor H e o fibrinogénio), inactivando a via alternativa do complemento evitando assim a fogocitose e a morte por linfócitos polimorfonucleares (PMN) (7). 1.4.1.2- Proteína T A proteína T não está relacionada com a virulência, no entanto possui especificidade pelo que permite a diferenciação laboratorial dos diferentes serótipos (15). Dos constituintes celulares intrínsecos também fazem parte o ácido hialurónico, o ácido lipoteicóico, a proteína F e o factor de opacidade do soro (OF) que são responsáveis pela adesão, colonização e invasão das membranas das mucosas e da pele no Homem (16).. 4.
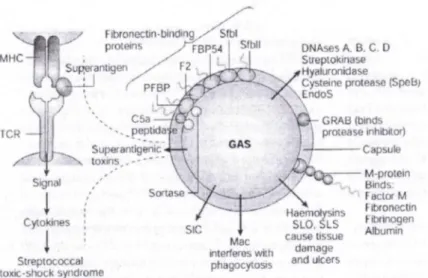
(15) 1.4.1.3- Ácido hialurónico A cápsula do GAS é composto por ácido hialurónico, um polímero de elevado peso molecular constituído por N-acetilglucosamina e ácido glucorónico. Também se sabe que o tamanho da cápsula não é o mesmo em todas as estirpes deste género. As colónias com um aspecto mucóide, quando cultivadas em agar de sangue, possuem uma “cápsula maior”. Assim, verificou-se que as estirpes encapsuladas resistem mais facilmente à fagocitose quando comparadas com as não capsuladas (7). Em 1992, Johnson, D. R. et al., (17) referem que o aspecto mucóide das colónias em 3% de Streptococcus do Grupo A verifica-se em casos com faringite, em 21% dos isolados com origem em doenças invasivas e em 42% dos isolados, em doentes com febre reumática aguda. 1.4.2- Proteínas extracelulares Deste grupo de proteínas extracelulares fazem parte: a estreptolisina O; a estreptolisina S; as DNAses A, B, C e D; a hialuronidase; a estreptoquinase; a exotoxina estreptocócica pirogénica B (Spe B- protease cisteina) e a C5a peptidase. O GAS, em geral produz as duas hemolisinas: a estreptolisina O e S, no entanto só a O é antigénica. Quer a estreptolisina O, quer a S danificam as membranas dos PMN, das plaquetas e dos organelos celulares. Têm ainda em comum o facto de não serem inibidas na presença de oxigénio. As DNAses degradam o DNA; a hialuronidase degrada o ácido hialurónico dos tecidos; a estreptoquinase dissolve os coágulos, através da conversão do plasminogénio em plasmina; a Spe B é uma protease e a peptidase C5a cliva a ligação deste factor do complemento aos PMN (7). A proteína Sic (“Streptococcal inhibitor of complement”) é secretada pelas estirpes M1 inibindo uma fracção do complemento e a lise da bactéria (18, 19). 1.4.3- As exotoxinas pirogénicas. As exotoxinas pirogénicas actuam como superantigénios capazes de se ligarem ao Complexo de Histocompatibilidade Major II e a receptores específicos das células T. Esta ligação desplota a activação rápida de enumeras células T, levando à secreção de citocinas (por exemplo, Interleucina-2, interferon γ), responsáveis por falência de múltiplos órgãos, o que caracteriza a STSS (19). Na figura 3 podem observarse os factores de virulência de S. pyogenes.. 5.
(16) Figura 3- Factores de virulência de S. pyogenes. Mitchell, T.J. (19).. 1. 5- Transmissão da infecção. A transmissão ocorre de humano para humano por contacto com secreções oriundas do nariz, da garganta, tosse, espirros ou por lesões na pele. Os portadores assintomáticos são menos contagiantes do que os que manifestam a infecção. O período de incubação é curto, entre 1 a 3 dias. O tratamento com antibiótico, por exemplo a penicilina, evita a propagação da infecção. Num estudo com o objectivo de detectar o tempo que demora a negativar a presença de S. pyogenes em crianças, os autores descobriram que 24 h após o início da terapêutica, menos de 20% das culturas eram positivas (20). 1.6- Objectivos deste estudo - Identificar os clones de Streptococcus pyogenes com mais de 80% de semelhança de perfis de macrorestrição do DNA obtidos por PFGE e linhagens genéticas com base em polimorfismos do gene emm (emm “typing”) e de genes nativos (“Multilocus Sequence Typing”MLST) - Avaliar a distribuição espaço-temporal dos clones e inferir a sua associação com “Day Care Centers-DCC” e estação do ano.. 6.
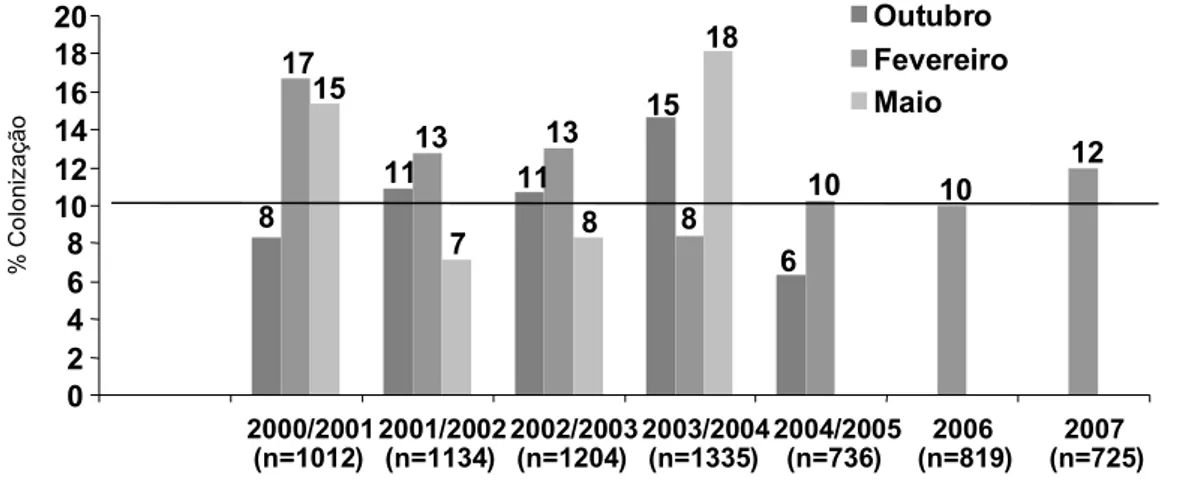
(17) 2 - Material e Métodos. 2.1- Participantes/Amostra Neste estudo foi colhido um total de 1026 exsudados orofaríngeos de portadores assintomáticos de S. pyogenes, provenientes de dezasseis Creches/Jardins-de-Infância do Concelho de Oeiras e duas Escolas do Distrito de Lisboa, abrangendo 10 578 participantes. Englobou funcionários, pais e crianças dessas Instituições. O período de recolha foi de 2000-2007. Os participantes responderam a um questionário e assentiram a confidencialidade dos dados (em anexo I).. % Colonização. A percentagem média de colonização foi de 9,7% (1026/10 578) (22), evidenciada na figura 4.. 20 18 16 14 12 10 8 6 4 2 0. 17 15. 15. 13 11. 13 11 8. Outubro Fevereiro Maio. 18. 7. 12 10 8. 10. 8 6. 2000/2001 2001/2002 2002/2003 2003/2004 2004/2005 2006 (n=1012) (n=1134) (n=1204) (n=1335) (n=736) (n=819). 2007 (n=725). Figura 4- Percentagem de colonização de S. pyogenes em crianças portadoras no Concelho de Oeiras (2000-2007). A linha a preto representa a percentagem média de colonização. Sanches, I., et al., 2009. (21).. Com uma taxa de colonização inferior a 10% registaram-se 307 isolados e com uma taxa entre os 10 e os 14%, registaram-se 299 isolados. Os dados apresentados nesta tese referem-se a 420 isolados com uma taxa de colonização igual ou superior a 14%. Estes isolados advêm de portadores assintomáticos de S. pyogenes pertencentes a 10 Creches/Jardins-de-Infância (codificados por DCC- “Day Care Centres”) do Concelho de Oeiras e a duas Escolas de Lisboa, colhidos entre 2000 e 2007. Os 420 isolados foram colhidos num total de 1822 participantes (anexo II). Das 420 estirpes em estudo, o operador B desenvolveu o seguinte trabalho laboratorial: 53 amostras para “Pulsed Field Gel Electrophoresis” (PFGE), 105 amostras para emm “typing” e 20 amostras para “Multilocus Sequence Typing” (MLST). O restante foi efectuado pelo operador A. 2.2- Conservação das estirpes bacterianas Todas as estirpes foram conservadas a -80ºC em tubos de criopreservação contendo 1,8 ml de meio líquido “Brain Heart Infusion” suplementado com 20% de glicerol.. 7.
(18) 2.3- Estirpes bacterianas e condições de crescimento. As estirpes bacterianas advêm de exsudados da cavidade orofaringea de humanos (crianças e adultos). As estirpes cresceram em meio sólido “Triptic Soy Agar” (TSA) suplementado com 5% de sangue desfibrinado de carneiro (Probiologica, U.E.). 2.4- Identificação presuntiva de Streptococcus pyogenes 2.4.1- Testes Serológicos. Os testes serológicos são testes de aglutinação em lâmina entre o composto/reagente e várias colónias de colónias de S. pyogenes, retiradas com uma ansa de 10 µl. 2.4.1.1- Hidrólise do L-pirrolidonil-β-naftil-amido (PYR). As estirpes que hidrolisam este composto exibem uma cor rosa, o que significa que possuem a enzima pirrolidonil peptidase. O teste do PYR efectuou-se a isolados resistentes à bacitracina. 2.4.1.2- Determinação do grupo A de Lancefield. O teste usado foi o de “Slidex. ®. Strepto A, bioMérieux®, Marcy l´Etoile, France”. A aglutinação significa a. presença do carbohidrato do grupo A e a não aglutinação significa ausência desse carbohidrato. 2.4.1.3- T-“typing”. Os soros utilizados foram comprados ao “ Swedish Institute for Infectious Disease- Smittskyddinstitutet”. Cinco eram polivalentes e vinte eram monovalentes. Quando há uma aglutinação com um dos soros polivalentes, passa-se aos soros monovalentes para determinar o(s) tipo(s) T. Quando não há nenhuma aglutinação com os soros polivalentes, a estirpe é considerada “non typeable” (NT). 2.4.2- Teste de susceptabilidade à bacitracina. O teste de susceptabilidade à bacitracina consiste no teste de difusão do disco com 0,04 U de bacitracinaBBLTM, “Becton Dickinson, Le Pont de Claix, France”. A ausência de halo de inibição à volta do disco de bacitracina corresponde a estirpes resistentes à bacitracina, enquanto que a presença do halo de inibição significa estirpes susceptíveis à bacitracina.. 8.
(19) 2.5- Métodos genotípicos 2.5.1- Electroforese em Gel de Agarose por Campo Pulsado- “Pulsed Field Gel Electrophoresis, PFGE” Este método consiste no corte do DNA cromossomal utilizando enzimas de restrição de corte raro. Os fragmentos obtidos são de massa molecular elevada, por isso são sujeitos a uma electroforese de campo pulsado de modo a separar os fragmentos de DNA produzidos pela restrição, obtendo-se um padrão característico para cada estirpe. O método utilizado foi adaptado de Chung, M., et al., (23), a que designámos Método A, executado pelo operador A. Como houve outro operador- operador B- que introduziu outras alterações, designámos por Método B. A descrição dos materiais usados nesta electroforese encontram-se no anexo III. 2.5.1.1- Propagação das culturas Os isolados conservados a -80ºC foram semeados em meio de “Trytic Soy Agar- TSA” com sangue de carneiro. Incubaram a 37ºC durante 24 h. Depois, inocularam-se algumas colónias em meio líquido “Todd-Hewitt” (Método A) ou meio líquido “Brain Heart Infusion” (Método B), a 37ºC durante 24h. 2.5.1.2- Lavagem celular e ajuste da concentração celular. Pipetar 2 ml de cada cultura anterior para tubos “eppendorf”. Centrifugar a 13000 rpm durante 20 min. Conversar as culturas em gelo. Decantar o sobrenadante. Ressuspender o “pellet” em 1 ml de PIV e centrifugar a 13000 rpm, durante 25 min. Decantar e adicionar 200 µl de PIV. Para ajustar a concentração das suspensões celulares de cada isolado, pipetou-se 1 ml de PIV para uma “cuvette” do espectrofotómetro e 5 µl da suspensão celular. Mediu-se a absorvância a 620 nm. Os valores de absorvância obtidos devem variar entre 0,025 cm-1 e 0,15 cm -1, caso contrário todas as etapas anteriores devem ser repetidas. Após a leitura da absorvância adicionou-se o volume de PIV necessário para que a densidade óptima final fosse 5,0. O volume de PIV adicionado foi calculado pela fórmula seguinte: Vadd (µl)= (DOx 40x 210) – 210 Vadd- volume (µl) de PIV a adicionar à suspensão celular DO- valor da densidade óptica a 620 nm 40- Quociente resultante da divisão do factor de diluição (200) pelo valor da densidade óptica óptima (5,0). A quantidade de PIV a adicionar é aferida tendo em conta que o volume de suspensão celular no tubo é de aproximadamente 210 µl e tendo em atenção que o valor da absorvância registado corresponde a uma diluição de 5 µl de suspensão em 1 ml de PIV.. 9.
(20) 2.5.1.3- Preparação dos discos de agarose. Colocar em “eppendorfs” 150 µl (Método A) ou 500 µl (Método B) da suspensão celular a 42ºC e adicionar 150 µl da solução de agarose “Seaplaque” (Método A) ou 500 µl da solução de agarose “Low Melting Point” (Método B), à mesma temperatura. No Método A, pipetar 12 vezes 20µl da mistura para uma placa de vidro (forrada com “parafilm” e pré-lavada com etanol a 70ºC). Colocar por cima dessas gotas uma lâmina de vidro (pré-lavada com etanol a 70ºC), achatando-as, obtendo-se os discos. Colocar este preparo 5 min a -20ºC e depois 10 min à temperatura ambiente. Retirar a lâmina e com o auxílio de uma ansa transferir os discos para um tubo “Falcon” de 15 ml (figura 5). No Método B, encher uma seringa de insulina (com a ponta cortada) com a mistura descrita. Colocá-la em cima de gelo, 10 min. Depois com uma lamela cortar os discos para um tubo “Falcon” previamente identificado (figura 6).. Figura 5- Discos de DNA obtidos pelo Método A.. Figura 6- Discos de DNA obtidos pelo Método B.. Chung, M., et al., (23 ).. Tenreiro, R. P. A. (24).. 2.5.1.4- Lise celular. Aos tubos Falcon contendo os discos, adicionar 1 ml de solução de lise (solução EC+ 1 mg/ml Lisozima+ 5 U/µl de Mutanolisina) (Método A) ou 4 ml no Método B. Incubar durante a noite a 37ºC. 2.5.1.5- Desproteinização. Decantar a suspensão anterior e adicionar 1 ml ou 4 ml de solução ESP (Tampão ES+ 1 mg/ml Proteinase K) (Método A ou Método B). Incubar a 50ºC durante 17 h (Método A) ou 24 h (Método B).. 10.
(21) 2.5.1.6- Lavagem para remoção da proteinase K. Decantar a solução ESP. Suspender em 12 ml de tampão TE 1X. Colocar os tubos em agitação, 30 min. Decantar e resuspender em TE 1X. Repetir este passo 8 vezes. Armazenar os discos a 4ºC em 1 ml ou 4 ml (Método A ou B) de TE 1X. 2.5.1.7- Restrição do DNA com a enzima SmaI. Este passo deve realizar-se, de preferência, no dia ou um dia antes da corrida electroforética. Incubar à temperatura ambiente durante 40 min um disco com 150 µl de tampão pré-SmaI (“Invitrogen”Método A, “Takara”- Método B). Decantar e adicionar 50 µl do tampão com 20 U/µl ou 10 U/µl de SmaI (Método A ou B). Para parar a restrição adicionar 5 µl de azul de bromofenol 6x e armazenar os discos. 2.5.1.8- Preparação do gel com os discos de DNA. Preparar um gel de agarose “Seakem” a 2% em TBE a 0,5X e guardar a 50ºC. Montar as peças do suporte do gel e de seguida colocar os discos nos dentes do pente com a ajuda de uma ansa e lamela. Nas extremidades do pente colocar um disco de marcador DNA lambda de massa molecular conhecida. Colocar o pente no suporte e cuidadosamente verter a agarose, arrefecida, sem que haja movimentação dos discos colocados no pente. Selar os poços com agarose e deixar solidificar. Por fim, retirar o aparato de moldagem e colocar o gel no aparelho de PFGE onde previamente se colocou 2 l de TBE a 0,5X. Iniciar a corrida electroforética com: temperatura 11,3ºC, tempo inicial de pulso 5 s, tempo final de pulso 35 s, voltagem 6V/cm, ângulo 120º, tempo de corrida 23 h. 2.5.1.9- Coloração do gel e digitalização da imagem. Retirar o gel do aparelho e colocá-lo na solução de brometo de etídeo, em agitação suave, durante 40 min. Depois descorar o gel em 300 ml de água bidestilada autoclavada, em agitação suave durante 10 min. Em seguida captar a imagem do gel num sistema fotográfico. 2.5.1.10- Análise dos perfis de PFGE. A análise dos padrões de bandas de DNA cromossomal foi por inspecção visual. Os DNAs produziram 9 ou mais fragmentos, pelo que, uma banda de diferença foi o critério para definir os diferentes padrões de PFGE (atribuindo uma letra diferente a um padrão diferente). Estes padrões também foram comparados através da construção de um dendrograma utilizando o “software BioNumerics 6.5- Applied Maths”. Os clusters foram definidos para grupos de estirpes que partilham entre si 80% ou mais de semelhança.. 11.
(22) 2.5.2- emm “typing” O protocolo do emm “type” encontra-se descrito no “site” do “Centers for Disease Control and Prevention” [online] (25). O primeiro passo deste protocolo consiste na preparação do lisado bacteriano. O operador B obteve o lisado de outra forma: colocou um disco guardado em TE 1X a 4ºC num “eppendorf” contendo 450 µl de TE 1X. Depois colocou-o num termobloco a 65ºC, 10 min (30). No entanto, em alguns casos foi necessário proceder conforme descrito no protocolo do CDC. Para visualizar os amplicões resultantes do PCR e os fragmentos de restrição por RFLP (“Restriction Fragment Length Polymorphism”), prepararam-se géis de agarose a 1% e a 3% em Tris Acetato EDTA, respectivamente. Depois da gelose derreter adicionou-se 25µl de “GelRed, Biotium”. Utilizaram-se dois marcadores: “1Kb DNA ladder, New England, Biolabs” para os amplicões de PCR e o “O´geneRuler 100 bp DNA ladder, Fermentas” para os fragmentos obtidos por RFLP. A atribuição do tipo emm foi estabelecida mediante comparação com padrões representativos, existentes no laboratório. Os que diferiam dos padrões representativos foram purificadas pelo “Kit Jet Quick PCR purification, Genomed” e sequenciados pela Empresa STABVida. As sequencias obtidas foram editadas no programa “BioEdit” e depois submetidas no “Blast 2.0 server” do CDC [online] (26) que nos indicou o tipo emm da estirpe, via correio electrónico. 2.5.3- MLST- “Multilocus Sequence Typing” Em 2006, (27) tinham sido publicados 35 esquemas de MLST, 33 dos quais acessível via “internet”. Assim, no “site” do MLST [Online] (28) encontram-se as Sequências Tipo (ST) de Streptococcus pyogenes, ou seja, as sequências nucleotídicas de múltiplos loci que codificam os 7 genes “housekeeping” (genes nativos) desta espécie: gki, gtr, mutI, mutS, recP, xpt, yqiL, com 434 a 498 bp. As condições de PCR encontram-se descritas no “site” MLST [online]. O marcador de massa molecular usado na electroforese foi o “O´geneRuler 100 bp DNA ladder, Fermentas”. Os amplicões foram purificados pelo “Kit Jet Quick PCR purification, Genomed” e sequenciados pela Empresa STABVida, no equipamento ABI3730XL (“AppliedBiosystems”), que utiliza o método de Sanger e a química “Bigdye Terminator V3.1, AppliedBiosystems”. É de notar que na sequenciação de produtos de PCR por MLST são usados ambos os “primeres”, sendo sequenciadas as duas cadeias de DNA. As sequências obtidas devem ter 100% de precisão, de forma a evitar que um alelo conhecido seja convertido num alelo novo. 2.5.3.1- Análise das sequências no “software DNAStar- SeqMen”. As sequências resultantes são analisadas pelo programa informático “DNAStar-SeqMen”. A sequência resultante designa-se sequência “consensus”2. Depois, aceder ao endereço electrónico do MLST [online] e. 2. Sequência “consensus”- sequência de ácidos nucleicos, na qual, a base numa dada posição é a base mais frequentemente encontrada quando se comparam sequências experimentalmente obtidas. Madigan, M. T., et al., (8).. 12.
(23) seleccionar “Locus Query” e escolher “Single Locus”. O passo seguinte consiste em copiar a sequência “consensus” do alelo pretendido, colá-lo no espaço destinado e o “site” atribui o número do alelo, caso haja 100% de correspondência entre o alelo e as sequências existente no “site”. Caso contrário, a sequência terá de ser re-analisada ou mesmo re-sequenciada. Seguidamente, para obter a sequência tipo (ST), clicar em “Profile Query” e depois em “Allelic”. Introduzir o número de cada um dos sete alelos e seleccionar “Query entire base” e o número da ST da estirpe é atribuído. 2.5.3.2- Análise dos dados de MLST- “eBURST”. O “eBURST” é um “software” que tem implícitas duas questões: como é que os clones de bactérias surgem e como se diversificam? O “eBURST” foca-se na identificação de grupos de isolados intimamente relacionados, dentro da população; na identificação do ancestral ou do “founder genotype” de cada grupo e nos caminhos evolutivos dos descendentes, desde o “founder” até aos restantes membros desse grupo (num curto espaço de tempo) (29) Assim, ao introduzirem-se os dados dos perfis alélicos obtidos por MLST no “eBUST”, a árvore resultante corresponderá, eventualmente, a variantes que diferem em um ou dois dos sete loci, designando-se “Single Locus Variant- SLV” ou “Double Locus Variant- DLV”, respectivamente. Outra definição importante é a de complexo clonal, um conjunto de isolados com origem no ancestral ou no “founder” da ST e com múltiplos “SLVs” (30). 2.5.4- Inferência Estatística. Para avaliar a distribuição dos clones e inferir a sua associação com os DCCs (hipótese 1) e com as estacões do ano (hipótese 2), elaboraram-se duas tabelas, uma com os clusters e os DCCs e a outra com os clusters e as estações do ano. O programa de tratamento de dados usado foi o SPSS versão 18.0 para Windows.. 13.
(24) 3 - Resultados 3.1- Amostra. Dos 420 isolados, 10 (2,4%) são de adultos femininos: 2 provêm de familiares (mães) cujos filhos frequentam os respectivos “Day Care Centers- DCC”, 6 provêm de Auxiliares de Acção Educativa, 1 provém duma Cozinheira e o outro de uma funcionária cuja actividade Não foi Determinada (ND) no questionário como se pode verificar na figura 7. 7 6. Nº de Adultos. 5 4. DCC 3. DCC 6. DCC 6. DCC 2. DCC 3. DCC 11. DCC 7. DCC 8. DCC 12. 3 2 1 0 Auxiliares de Acção Educativa. Cozinheira. Familiares. ND. Figura 7– Número de adultos (familiares/funcionários) por DCC.. As idades dos adultos oscilavam entre os 19 e os 68 anos de idade. 410 isolados advêm de crianças, sendo 168 (40,2%) do sexo feminino e 242 (59,0%) do sexo masculino. As idades variaram entre 1 e 6 anos de idade. A figura 8 ilustra o predomínio do sexo masculino em todos os DCCs, à excepção no DCC 2 em que o. Nº de Crianças. número de crianças do sexo masculino é igual ao do sexo feminino. 50 45 40 35 30 25 20 15 10 5 0. M. DCC 1. F. DCC 2. DCC 3. DCC 4. DCC 5. DCC 6. DCC 7. DCC 8. DCC 11 DCC 12. Figura 8– Prevalência do género das crianças em cada DCC, entre 2000 e 2007.. 3.2- Taxa de colonização No nosso estudo, a taxa de colonização por S. pyogenes é igual ou superior a 14%, como se pode observar na figura 9.. 14.
(25) 60. Taxa de Colonização (%). 50 40. DCC1. DCC2. DCC3. DCC4. DCC5. DCC6. DCC7. DCC8. DCC11. DCC12. 30 20 10. O ut .2 M 000 ar .2 Ab 001 r.2 M 00 ai 1 .2 Ju 001 n. 2 Ju 001 n. 20 Ju 0 n. 1 2 Fe 001 v. 2 Fe 002 v. 2 Fe 00 v. 2 M 20 ai 02 o. 2 O 002 ut .2 O 00 ut 2 .2 O 002 ut .2 Fe 003 v. 2 Fe 003 v M .20 ai 03 o. M 200 ai o. 3 M 200 ai o. 3 2 O 00 ut 3 .2 M ai 003 o M .200 aio 4 M .20 aio 04 .2 Fe 004 v. 2 M 005 ar .2 M 00 ar 6 .2 M 00 ar 6 .2 M 00 ar 7 .2 00 7. 0. Figura 9- Taxa de colonização nos diferentes DCCs, no período de 2000-2007. A linha a preto assinala os 14%.. 3.3- Teste de sensibilidade à bacitracina. O teste da susceptibilidade à bacitracina foi realizado em todos os isolados provenientes de adultos e de crianças. No entanto, em 2006, no DCC 11 identificaram-se dois isolados resistentes à bacitracina, em crianças com 2 e 4 anos de idade, com o padrão de PFGE-F e em 2007, no DCC 5, identificaram-se três isolados resistentes à bacitracina em crianças com 5 anos de idade e com o padrão de PFGE-AC. Os cinco isolados eram emm 28 e em relação à resistência a macrólidos por S. pyogenes o fenótipo encontrado foi o cMLSB (mecanismo de resistência a Macrólidos, Lincosamidas e Estreptogramina B constitutivo) em todos eles (anexo IV). A resistência diz-se constitutiva quando a bactéria produz metilases mesmo na ausência de um indutor. Essas metilases, designadas “Erm” são responsáveis pela metilação da adenina A2058 da sub-unidade 23S do rRNA alvo, comprometendo a ligação do antibiótico ao seu alvo e desta forma o microorganismo resiste (31). 3.4- T “typing”. Entre 2000 e 2006 foram analisados, por T-“typing”, 231 isolados provenientes de crianças e 7 provenientes de adultos. No total, os 238 isolados correspondem a 14 tipos T diferentes. Verificou-se que 53 (22,3%) isolados eram T 12 (2002-05), 44 (18,5%) eram NT (“Non Typeable”) (2001-05), 32 (13,4%) eram T 1 (200004) e 30 (12,6%) eram T 3/13/B3264 (2001-03), como se pode observar na figura 10.. 15.
(26) 2005 2004 2003 2002 2001. T6. T. 5/ T 11 4 /1 2/ 27 /4 T 4 8/ 25 /Im p1 9 T 4/ 28 T 5/ 27 /4 4. 3/ 13 3/ 13 /B 32 64. 3. T. T. T. 28 T. T2 /2 8. T2. 12 T. N. T. T. 2000. 1. Nº de Isolados. 2006. 60 55 50 45 40 35 30 25 20 15 10 5 0. Figura 10- Tipos T de 238 isolados de S. pyogenes (2000-2006).. 3.5- Critérios para avaliação dos métodos de tipificação A avaliação da eficiência dos métodos de tipagem baseia-se nos seguintes critérios: tipabilidade, reprodutibilidade e poder discriminatório (32, 33). 3.5.1- Tipabilidade A tipabilidade de um método é a percentagem de tipos atribuídos a diferentes isolados bacterianos (32). 3.5.2- Reprodutibilidade A reprodutibilidade reflecte a atribuição do mesmo tipo a um isolado testado em diferentes ocasiões. Para haver reprodutibilidade intra e inter-laboratorial tem de haver protocolos padrão e pessoal com treino. 3.5.3- Poder discriminatório/ Índice de Simpson/ Intervalos de Confiança O poder discriminatório de um método de tipificação indica a probabilidade de duas estirpes não relacionadas, extraídas de uma população, pertencerem a tipos diferentes (32). Hunter, P.R. & Gaston, M.A. (32), propuseram o índice de diversidade de Simpson´s (D) para calcular o poder discriminatório dos métodos de tipificação. O índice de diversidade de Simpson´s pode ser calculado pela seguinte fórmula:. Onde, N é o número total de isolados; S é o número total de tipos obtido e nj é o número de isolados pertencentes aos tipos j. Para os autores (32), índices discriminatórios superiores a 0,90 representam um valor aceitável na interpretação dos resultados. Para, Van Belkum, A., et al., (33), o valor de (D) deve ser, idealmente, 1 mas na prática deve ser na ordem de 0,95 para métodos de tipificação, tendo em conta que a probabilidade de erro é de 5%. Por isso, é importante determinar os Intervalos de Confiança (I.C.) para os índices de Simpson´s de cada método de tipificação, permitindo uma melhor avaliação dos mesmos (34). Os intervalos de confiança são usados para indicar a confiança que se pode depositar numa estimativa (http://pt.wikipedia.org/wiki/Intervalo_de_confian%C3%A7a).. 16.
(27) 3.6- PFGE- “Pulsed Field Gel Electrophoresis”. Os fragmentos de DNA do nosso estudo, obtidos por PFGE oscilaram entre 48,5 e 485 Kb e representam um “fingerprint” identificativo e de diferenciação de estirpes bacterianas. 3.6.1- Tipabilidade De acordo com o histograma apresentado na figura 11, os três padrões de PFGE com maior percentagem são: o padrão AB com 22,6% (n=95), o padrão X com 16,2% (n=68) e o padrão AP com 12,6% (n=53). Os. Padrões de PFGE. padrões restantes apresentam percentagens inferiores e iguais a 7%. AB X AP BG EF BK AD BH AJ BE BC EQ CZ BM EH EK FW DK CX AC F EL DX DM DF BI B A N GA FZ EX ET ES ER EP EO EN EM ED DN CY CE BL BJ BF BD AU AK 0 5 10 15 20 25 30 35 40 45 50 55 60 65 70 75 80 85 90 95 Nº de Isolados. Figura 11– Histograma de barras de 49 padrões de PFGE de 420 isolados.. 3.6.2- Prevalência dos padrões de PFGE nos DCCs e sua localização geográfica. Na figura 12-a) pode observar-se a prevalência dos 49 padrões de PFGE em cada “Day Care Center” e na figura 12-b) pode-se observar a sua distribuição geográfica.. 17.
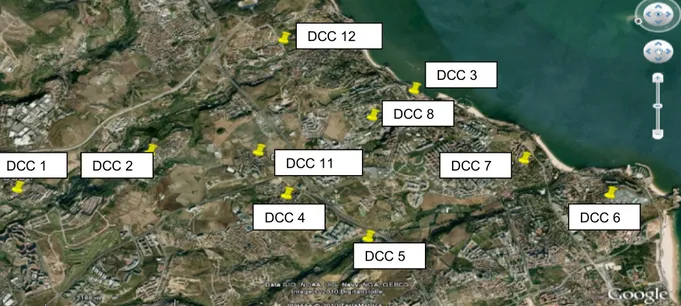
(28) DCC 1. DCC 2. 15 Nº de Isolados. Nº de Isolados. 25 20 15 10. 10 5 0. 5. AP. 0 X. AP. GA e FZ. BH. A. Heterogénio. Padrões de PFGE. Padrões PFGE 14. DCC 3. 25. Nº de Isolados. Nº de Isolados. 30 20 15 10 5. DCC 4. 12 10 8 6 4 2 0 AP. 0. AB. BM. X. EQ. Heterogénio. Padrões de PFGE. Heterogénio. Padrões de PFGE DCC 5. 15 Nº de Isolados. Nº de Isolados. 50 40 30 20 10. DCC 6. 10 5 0. 0 AB. X. BE. BC. BG. Heterogénio. Padrões de PFGE. Padrões de PFGE. 25. 30. DCC 7. 20. DCC 8. 25 Nº de Isolados. Nº de Isolados. Heterogénio. 15 10 5 0 AB. X. BG. AD. 20 15 10 5. Heterogénio. 0. Padrões de PFGE. 16 14 12 10. BG. X. Heterogénio. Padrões de PFGE DCC 12 20 Nº de Isolados. Nº de Isolados. AB DCC 11. 8 6 4 2 0. 15 10 5 0. BK. AJ Padrões de PFGE. Heterogénio. EF. AB Padrões de PFGE. Figura 12- a) Prevalência dos padrões de PFGE nos “Day Care Censtres”, entre 2000 e 2007.. 18.
(29) DCC 12 DCC 3 DCC 8. DCC 1. DCC 11. DCC 2. DCC 7. DCC 4. DCC 6 DCC 5. Figura 12- b) Localização geográfica dos DCCs no Concelho de Oeiras.. A heterogeneidade de padrões de PFGE pode ser apresentada pela seguinte por ordem decrescente: DCC 3 (n=26) > DCC 7 (n=21) > DCC 6 (n=13) > DCC 5 (n=11) > DCC 4 (n=8) > DCC 2 e 8 (cada com n=5) > DCC 11 (n=3). 3.6.3- Reprodutibilidade de PFGE Para o estudo da reprodutibilidade do PFGE testaram-se 9,3% (n=39/420) estirpes em duas ocasiões diferentes. Como referido no capítulo Material e Métodos, a preparação dos discos de agarose do PFGE diferiu: entre placa de vidro e lâmina (Método A executado pelo o operador A) ou através de uma seringa e lamela (Método B executado pelo o operador B). A cada isolado testado em duplicado corresponde uma percentagem de semelhança e quando esta se encontra acima de 80%, pode-se afirmar que houve reprodutibilidade do método de PFGE. Dos 39 isolados testados, houve dois cuja percentagem de semelhança foi inferior a 80%. Embora este resultado ocorresse no Método B, convém referir que a causa mais provável se deva a uma restrição incompleta do DNA com a enzima SmaI e não à forma de preparação dos discos de agarose. No que se refere aos demais isolados, os valores das percentagens de semelhança denotam ter havido reprodutibilidade do PFGE, quer pelo Método A quer pelo método B (tabela 2).. Tabela 2- Estudo da reprodutibilidade de PFGE para 39 estirpes testadas pelos métodos A/B. As percentagens de semelhança acima dos 80 % indicam haver reprodutibilidade para o Método de PFGE. % Semelhança (BioNumerics 6.5) < 80%. (a). Isolados testados em duplicado (n=39). Método (A/ B). 2. B. [80,90]%. 9. B. 100%. 1. B. 80%. 3. B. ]80,90]%. 8. B. ]90,100]%. 7. B. 95%. 4. A. 100%. 5. A. (a). Causa provável: restrição enzimática incompleta. 19.
(30) 3.6.4- Dendrograma. O dendrograma, elaborado no programa “BioNumerics versão 6.5- Applied Maths”, Bélgica, permite-nos distinguir 23 clusters (grupos clonais), correspondendo a 397 isolados (388 de crianças e 9 de adultos) clonalmente relacionados. Na figura 13, os clusters são representados por numeração romana, sendo o cluster I o que regista maior número de isolados (n=99) e o cluster XXIII menor número de isolados (n=2). Para tal, foi preciso ter em conta os critérios de tipificação de estirpes bacterianas, segundo os quais as estirpes que diferem 2 a 3 fragmentos de DNA são consideradas relacionadas e as que diferem entre 4 a 6 fragmentos são consideradas possivelmente relacionadas (35), ou seja, as estirpes são consideradas clonalmente relacionadas se apresentarem uma percentagem de semelhança superior a 80 % (linha azul da figura 13). Nota-se que há clusters característicos de dados DCCs. Por exemplo, o cluster X é característico do DCC 2, o cluster VIII é característico do DCC 12.. 20.
(31) 21.
(32) 3.7- emm-“typing” 3.7.1- Tipabilidade De acordo com o histograma apresentado na figura 14 os três tipos emm que apresentam maior percentagem são: emm 12 com 40,5% (n=170/420), emm 1 com 18,3% (n=77/420) e o emm 3 com 12,4% (n=52/420). Os restantes tipos emm apresentam percentagens inferiores a 8 %. emm 12 emm 1 emm 3 emm 75 emm 2 emm 6. Tipos emm. emm 89 emm 28 emm 4 emm 77 emm 22 emm 48 emm 78 emm 94 emm 64 st1815 st4050 0. 10. 20. 30. 40. 50. 60. 70. 80. 90. 100 110 120 130 140 150 160 170 180. Nº de Isolados. Figura 14- Histograma de barras de 420 isolados que ilustra a prevalência dos 17 tipos emm.. 3.7.2- Diversidade dos tipos emm A análise de 420 isolados de S. pyogenes revelou 17 tipos emm diferentes. A distribuição da diversidade destes tipos emm pelos 10 “Day Care Centers” (DCC) encontra-se na tabela 3.. 22.
(33) Tabela 3 - Distribuição dos tipos emm pelos 10 DCCs (2000-2007). Tipos emm 12 1 75 3 2 4 28 6 22 78 89 94 48 64 77 st40.50 st18.15. DCC 1. DCC 2. DCC 3. DCC 4. DCC 5. DCC 6. DCC 7. DCC 8. DCC 11. DCC 12. X. X. X. X. X. X. X. X. X. X. X. X. X. X. X. X. X. X. X. X. X. X. X. X. X. X. X. X. X X. X X. X. X. X X. X X. X X. X X. X X. X. X. X. X. X X. X. X. X. X X X X X. Tendo em conta os dados da figura 14 e os da tabela 3, verificou-se que o emm 12 foi identificado em todos os DCCs, o emm 1 em nove DCCs e o emm 3 em cinco DCCs. Passando à análise da tabela 3, a maior diversidade dos tipos emm registou-se no DCC 3 (nove tipos emm diferentes), seguido do DCC 6 e DCC 7 (ambos com oito tipos emm diferentes). O DCC 5 e o DCC 8 apresentam sete tipos emm diferentes. O DCC 4 apresenta seis tipos emm diferentes, o DCC 2 apresenta quatro tipos emm diferentes e os DCCs 1, 11 e 12 com três tipos emm diferentes. A diversidade dos tipos emm nos DCCs pode ser apresentada por ordem decrescente: DCC 3> DCC 6, DCC 7> DCC 5, DCC 8> DCC 4> DCC 2> DCC 1, DCC 11, DCC 12. 3.7.3- Distribuição dos tipos emm, de 2000 a 2007 A análise da figura 15 permite-nos afirmar que o tipo emm mais frequente (emm 12) teve o seu expoente máximo em 2003, com 59 isolados. No ano seguinte desceu para 21 isolados, mantendo-se dentro destes valores até 2007. Em relação ao emm 1 e emm 3, o maior número de tipos emm 1 (n=21) verificou-se em 2004 e o maior número de tipos emm 3 (n=27) verificou-se em 2001. Para os tipos emm 64, st1815, st4050 só se registou um caso de cada, em 2005, 2002 e 2003, respectivamente.. 23.
(34) 70 emm 1 emm 2. 60. emm 3 emm 4 emm 6. 50. Nº de Estirpes. emm 12 emm 22 40. emm 28 emm 48 emm 64. 30. emm 75 emm 77 20. emm 78 emm 89 emm 94. 10. st1815 st4050. 0 2000. 2001. 2002. 2003. 2004. 2005. 2006. 2007. Figura 15- Distribuição dos tipos emm de S. pyogenes em isolados de colonização, de 2000-07.. 3.7.4- Análise da distribuição dos tipos emm na Primavera, no Outono e no Inverno. Durante 8 anos foram colhidos 209 exsudados orofaringeos no Inverno, 122 na Primavera e 89 no Outono. No Inverno, os tipos emm com maior prevalência foram o emm 12 (n=99, predominante no DCC 8), o emm 75 (n=31, predominante no DCC 12), o emm 1 (n=25, predominante no DCC 1), o emm 3 (n=22, predominante no DCC 11) e o emm 6 (n=15, predominante no DCC 7) como se pode observar na tabela 4. Tabela 4- Tipos emm identificados no Inverno (2001-03 e 2005-07). 2001. emm 1. DCC2. DCC4. 1. 6. emm 3. 2002. DCC6. DCC7. DCC4. 5. DCC6. 2003. DCC8. DCC2. DCC3. 2005. DCC4. 2006. DCC7. DCC11. DCC12. 1. DCC1. Total n=isolados 25. 12 2. 22. 2. 3. emm 78. 1. emm 6. 3. emm 94. 2. 15. 1. 2. 3. 3. emm 75. 7. 2. 1. emm 12. 1. 21. emm 4. 1. 2 2. 19. 13. 8. 18. 16. 31. 1. 1. 18. 1. 1. emm 89. 1. 1 1. 9. 13. 10. 8. 5. 99 1. emm st1815. 1. 7 1. 10. 1. emm 2. emm 64 Total n=isolados. DCC5. 12. 10. emm 28. 2007. 23. 2. 15. 9. 29. 1 30. 20. 14. 21. 209. Na Primavera, os tipos emm com maior prevalência foram o emm 1 (n=34, predominante no DCC 8), o emm 12 (n=32, predominante no DCC 1), o emm 3 (n=30, predominante no DCC 8) e o emm 89 (n=10, no DCC 5), como se pode observar na tabela 5.. 24.
(35) Tabela 5- Tipos emm identificados na Primavera (2001-04 e 2007). 2001. 2002. DCC3. DDC4. emm 1. 3. 4. emm 3. 7. 10. emm 28. 2. 1. emm 78. 1. emm 6. 2003. 2004. 2007. Total DCC8 DCC4 DCC5 DCC6 DCC1 DCC5 DCC8 DCC12 n=isolados. DCC7. 8. 18. 1. 13. 3 1. 2. 2. emm 94. 0. emm 2. 1. emm 75. 1. emm 12. 3. 1. 2 1. 6. emm 89. 2. 21. 32. 10. emm 22 emm 4 Total n=isolados. 34 30. 10 4. 13. 6. 11. 17. 6. 10. 4. 6. 21. 2. 1. 11. 20. 3 1. 122. No Outono, os tipos emm com maior prevalência foram o emm 12 (n=40, predominante no DCC 5), o emm 1 (n=18, predominante no DCC 7) e o emm 2 (n=12, no DCC 2), como se pode observar na tabela 6.. Tabela 6- Tipos emm identificados no Outono (2000, 2002 e 2003). 2000 DCC2 emm 2. 12. emm 1. 2. emm 48. 2. emm 12. 2002 DCC2. 2003 DCC3. DCC5. DCC7. Total n=isolados 12. 4. 12. 18. 2. 40. 2 9. 29. emm 22. 1. 1. emm 4. 5. 5. emm 75. 1. emm st4050. 1. emm 77 Total n=isolados. 1. 1 8. 16. 9. 12. 2. 37. 8 15. 89. Também foram identificados dois novos sub-tipos emm, o emm 3.51 (cluster V) e o emm st4050 (cluster VI) (anexo V). 3.8- Determinação do Coeficiente de Wallace. O cálculo do coeficiente de Wallace visa comparar os tipos atribuídos e explorar a concordância entre os métodos de tipificação (41). Para comparar os métodos de PFGE e emm “typing” de 397 isolados clonalmente relacionadas, submeteram-se os padrões/tipos no “site” www.comparingpartitions.info e os resultados do coeficiente de Wallace (para um Intervalo de Confiança de 95%) demonstram que duas estirpes que pertencem ao mesmo cluster têm 99,7% de probabilidade de terem o mesmo tipo emm (Coeficiente de Wallace. PFGE→emm=. 0,997) e que duas estirpes com o mesmo tipo emm têm 47,9% de. probabilidade de pertencerem ao mesmo cluster (Coeficiente de Wallace emm→PFGE= 0,479) (anexo VI).. 25.
(36) 3.9- MLST- “Multilocus Sequense type” 3.9.1. Tipabilidade O esquema de MLST para Streptococcus pyogenes para os 7 genes nativos (consultar Material e Métodos) foi determinado para 44 isolados tendo-se obtido 12 sequências tipo diferentes, sendo a ST 36 a que apresenta maior prevalência, n=12 (27,3%) como se pode observar na figura 16. ST 408 ST 161 Sequências Tipo (MLST). ST 39 ST 406 ST 15 ST 52 ST 46 ST 55 ST 28 ST 382 ST 150 ST 36 0. 2. 4. 6. 8. 10. 12. Nº de Isolados. Figura 16- Histograma de barras de 44 isolados que ilustra a distribuição das 12 ST.. 3.9.2- “eBURST”. As ST do nosso estudo já eram conhecidas na base de dados do MLST [online] (28). Depois foram analisadas pelo “software eBURST”- uma abordagem da sua filogenia. No anexo VII encontra-se o relatório do “eBURST v3” (http://eburst.mlst.net/v3/mlst_datasets/) que corresponde à análise de 2098 isolados, agrupados em 77 grupos e com 527 ST. Um grupo é um conjunto de ST que partilham 6 ou mais alelos num total de 7, não podendo haver sobreposição das ST em grupos diferentes. No nosso estudo existe o complexo clonal 15, CC15, em que a ST 406 representa um SLV (“Single Locus Variant”) do “predicted founder”3 ST 15, porque a ST 406 apresenta uma alteração de posição do alelo gtr, ou seja, o alelo gtr da ST 406 ocupa a posição 29 e o da ST 15 ocupa a posição 6 (anexo VIII). As ST 150, 52, 36, 55, 39, 28 e 46 correspondem aos próprios ancestrais (“predicted founder”), respectivamente aos grupos G18, G13, G7, G41, G6, G2 e G8. Quanto à ST 382 e à ST 408 correspondem, respectivamente, a um SLV do ancestral ST 37 e a um DLV (“Double Locus Variant”) do ancestral ST 407. 3. “Predicted founder”- é a Sequência Tipo com maior número de SLVs e DLVs, ou seja, a ST “predicted founder” é aquela a partir da qual se diversificam as ST que diferem em um ou dois dos 7 alelos. Regra geral a ST “predicted founder” é a que apresenta maior prevalência, maior valor “bootstrap” e menor distância em relação a todas as outras ST do grupo.. Feil, E. J., et al., (29).. 26.
(37) Por último, a ST 161 corresponde a um “singleton”, ou seja, uma sequência tipo que não se enquadra na definição de grupo (figura 17). A frequência (n=isolados) com que as ST foram identificadas no nosso trabalho apresenta a seguinte ordem decrescente: ST 36 (n=12) > ST 150 (n=6) > ST 28 (n=5), ST 382 (n=5) > ST 55 (n=4) > ST 46 (n=3) > ST 15 (n=2), ST 52 (n=2), ST 406 (n=2) > ST 39 (n=1), ST 161 (n=1), ST 408 (n=1).. ST150/PFGE-ET/emm 75/Cluster XIII/ n=1 ST150/PFGE-EH,EQ/emm 75/Cluster XIV/ n=2 ST150/PFGE-EF/emm 75/Cluster VIII/ n=1. G18 G13 ST52/PFGE-ED,EL/emm 28/Cluster XIX/ n=2. ST36/PFGE-AB, AP, BK, FW/emm 12/Cluster I/ n=7 ST36/PFGE-AB,AP/emm 12/Cluster II/ n=2 Singleton ST36/PFGE-DM/emm 12/Cluster XXIII/ n=1. G7. G41. G28 ST15/PFGE-BM,EM/emm 3/Cluster VI/ n=2 G9 ST406/PFGE-BG/emm 3/Cluster V/ n=2 ST382/PFGE-AD/emm 1 e 6/Cluster IX/ n=2 ST382/PFGE-DX, N/emm 6/Cluster IX/n=2. CC 15 ST46/PFGE-A,B/emm 22/Cluster XVI/ n=2 ST46/PFGE-AU/emm 22/Cluster XXI/ n=1. G8 ST55/PFGE-BH/emm 2/Cluster X/ n=2 ST55/PFGE-DX/emm 2/Cluster XVIII/ n=1 ST55/PFGE-ES/emm 2/Cluster XXI/ n=1. G2. G1 ST408/PFGE-AJ/emm 3/Cluster VI/ n=1. G6. ST28/PFGE-EK/emm 1/Cluster III/ n=1 ST28/PFGE-X/emm 1/Cluster IV/ n=3 ST28/PFGE-CX/emm 1/Cluster XX/ n=1. ST39/PFGE-DF/emm 4/Cluster XII/ n=1. Figura 17 – Diagrama obtido pelo “software eBURSTv3“(http://eburst.mlst.net) que compara as Sequências Tipo da base de dados de MLST de Streptococcus pyogenes com os 44 isolados do nosso estudo. Os círculos maiores preenchidos a azul e a preto designam os “predicted founder” do grupo (G). Os círculos menores preenchidos a preto designam “SLV”. O círculo de cor amarelo designa um subgrupo. A seta a vermelho assinala as 12 ST encontradas no nosso estudo. CC15 designa o complexo clonal 15. Dos 44 isolados analisadas pelo eBURST apenas 39 apresentam mais de 80% de semelhança (dado obtido do dendrograma). As linhagens destes 39 isolados encontram-se apresentadas junto a cada grupo G correspondente.. 3.9.3- Comparação de isolados de colonização de S. pyogenes com os da base de dados do MLST. O aspecto mais importante é que as ST do nosso estudo correspondem a isolados da cavidade orofaringea de crianças portadoras assintomáticas de S. pyogenes e as ST de S. pyogenes descritas no “site” do MLST correspondem a isolados de outros produtos biológicos, como o sangue, as feridas, o tracto aéreo superior, o líquido cefalo raquidiano, entre outras, ocorrendo em crianças e/ou adultos (36). A tabela 7 resume a data, os locais e o tipo emm das ST encontradas no nosso estudo e no resto do mundo. Também se indicam os fenótipos/genotipos de resistência a macrólidos detectados em estudos portugueses.. 27.
(38) Assim, a ST 36, com maior número de isolados identificados no nosso estudo, encontra-se distribuída por 4 continentes (tabela 7), enquanto que a ST 39, embora se encontre disseminada pelos 5 continentes, surge num isolado do nosso estudo. A ST 408 é a única que se confina a Portugal e com diferentes tipos emm: um isolado de um líquido cefalo raquidiano (37) e outro encontrado no nosso estudo. Em algumas ST, os tipos emm alteram no contexto espaço temporal, caso das ST 28, 36, 39, 52, 382, 406 e 408, como se pode observar na tabela 7.. 28.
(39) Tabela 7- Localização geográfica das ST encontradas no nosso estudo.. Resistência a macrólidos (Portugal). ST. Localização Geográfica. Data/Local/emm "type" (1ª Descrição). Década 90. Séc. XXI. 15. Europa, América e Egipto. 1942/ EUA/ 3. Alemanha, Brasil, Egipto. RC, Portugal. 28. Euroásia e América do Norte. 1932/ RU/ 1. Alemanha, Suécia, Finlândia, China, Japão, Taiwan, EUA. Nepal, Portugal, Polónia, RC, Alemanha(emm3,4, 28). Fenótipo M. 1956/ EUA/ 12. Alemanha(emm77), Polónia, Bélgica, Irlanda, Finlândia, Suíça, Itália, Etiópia, Turquia, China, Japão, Taiwan, Coreia do Sul, Brasil, Argentina, Peru, Chile, Canadá. Portugal, Índia, Grécia, Polónia, RC (emm44, 75). Fenótipo/Genótipo c MLSB/ermB. 1994/ EUA/ 4. Canadá, Taiwan (emm sts104), Suécia, Finlândia, Polónia, Alemanha, Itália. Espanha, Portugal, RU, RC, Hungria, Japão, África do Sul, Coreia do Sul, Nepal. Fenótipo M. 1986/ EUA/ 22. Portugal, Alemanha, Bélgica, França, Itália, Canadá, Brasil, Taiwan, Irlanda, RU, México. Portugal, Espanha, RU, RC. Fenótipo MLSB. Alemanha, Canadá, Áustria, França, Hungria, Polónia, RC, Túrquia, Coreia do Sul. Portugal, Espanha(emm77), Grécia, RU, Alemanha, Polónia, b RC, Suécia. Fenótipo MLSB e resistência a bacitracina e isolados d colonização. 36. Euroásia, Oceania e América. 39. Euroásia, Oceania, América e África do Sul. 46. Eroásia, América e África do Sul. 52. Euroásia e América do Norte. 1995/ EUA/ 28. 55. Europa e América do Norte. 1938/ RU/ 2. Bélgica, Alemanha, EUA. Espanha, Portugal, RC,. Europa e 150 Oceania. 1993/ Espanha/ 75. Polónia, Alemanha, RU, RC, Austrália. Portugal, Espanha, RC. Península Ibérica e 161 México. 1967/ Taiwan/ 48. México, Espanha, Portugal. 382 Euroásia. 1999/Espanha,Áustria/Rússia/ 6. Portugal(emm1 e 3), RC, Alemanha(emm1). 406 Europa. 1999/ Alemanha/ 3. 408 Portugal. 2004/ Portugal/ 89. Portugal (emm107) e. Alemanaha, Espanha, Portugal Portugal (emm3). EUA- Estados Unidos da América; RU- Reino Unido; RC- Repúplica Checa; Portugal- detectado em Portugal, em crianças portadoras assintomáticas de S. pyogenes. a. b c d e. Silva-Costa, et al., 2006. Clin Microb Infect. 12: 513-518 Eriksson, BK, et al., 2003. Clin Infect Dis. 37:1189-1193 Siva-Costa, et al., 2008. Clin Microb Infect. 14: 1152-1159 Pires, R., et al., 2009. FEMS Microb Lett. 296: 235-240 Friães, A., et al., 2007. J Clin Microb. 45: 2044-2047. 29. a. a. a.
(40) 3.10- Linhagens Genéticas. Tendo por base 397 isolados com mais de 80% de semelhança dos perfis de macro-restrição do DNA, obtidos por PFGE e com base em polimorfismos do gene emm (emm “typing”) e de genes nativos (“Multilocus Sequence Typing”- MLST), definiram-se as linhagens genéticas, de acordo com a estação do ano. Assim, no Outono obtiveram-se cinco linhagens com mais de cinco isolados (n) nos respectivos DCCs: → PFGE-AB, AP/ emm 12/ ST 36, n=39, DCCs: 2, 5,7 → PFGE-X/ emm 1/ ST 28, n=18, DCCs: 2, 3, 7 → PFGE-BH/ emm 2/ ST 55, n=12, DCC 2 → PFGE-BC/emm 77, n=5, DCC 5 → PFGE-CZ, DF/ emm 4, n=5, DCC 3 E quatro linhagens, cada uma com um isolado, no respectivo DCC: → PFGE-B/ emm 22, DCC 3 → PFGE-BM/ emm st4050, DCC 3 → PFGE-DF/ emm 75/ ST 39, DCC 3 → PFGE-ET/ emm 75/ ST 150, DCC 7, No Inverno obtiveram-se sete linhagens com mais de três isolados (n) nos respectivos DCCs: → PFGE-AB, AP, BK, DM, FW, FZ/ emm 12/ ST 36, n=97, DCCs: 1, 2, 3, 4, 5, 6, 7, 8, 11, 12 → PFGE-EF, EH, EQ/ emm 75/ ST 150, n= 28, DCCs: 3, 4, 6, 8, 12 → PFGE-EK, X/emm 1/ ST 28, n= 25, DCCs: 1, 2, 4, 6, 7, 12 → PFGE-AJ, BG/ emm 3/ ST 408, n= 21, DCCs: 6, 11 → PFGE-AD, DX, N/ emm 6/ ST 382, n= 14, DCCs: 4, 6, 7 → PFGE-AC, EL/ emm 28/ ST 52, n= 5, DCCs: 5, 7 → PFGE-DK/ emm 2/ ST 55, n= 3, DCC 7 E quatro linhagens, cada uma com um isolado, no respectivo DCC: → PFGE-AD/ emm 1/ ST 382, DCC4 → PFGE-EH/ emm st1815, DCC 8 → PFGE-BE/ emm 89, DCC 4 → PFGE-EP/ emm 94, DCC 4, Na Primavera obtiveram-se oito linhagens com mais de dois isolados (n) nos respectivos DCCs: → PFGE-CX, X/ emm 1/ ST 28, n=33, DCCs: 3, 4, 5, 8 → PFGE-AB, AP, DM/ emm 12/ ST 36, n=32, DCCs: 1, 4, 6, 8 → PFGE-BG, BM, EM, ER/ emm 3/ ST 15, n=29, DCCs: 3, 7, 8 → PFGE-BE, BF/ emm 89, n= 10, DCC 5 → PFGE-A, AU, B/ emm 22/ ST 46, n=4, DCC 6 → PFGE-CZ/ emm 4, n= 3, DCCs: 5, 8 → PFGE-AD/ emm 6, n= 2, DCC 4 → PFGE-BJ, ES/ emm 2/ ST 55, n=2, DCCs: 5, 8. 30.
(41) E duas linhagens, cada uma com um isolado, no respectivo DCC: → PFGE-ED/ emm 28/ ST 52, DCC 7 → PFGE-EH/ emm 75, DCC 8. 3.11- Poder Discriminatório e Intervalos de Confiança de PFGE, emm “typing” e MLST A partir do “site” www.comparingpartitions.info é possível obter os histogramas, o índice de Simpson e os Intervalos de Confiança (I.C.) para cada um dos métodos de tipificação. Foram calculados os índices de diversidade de Simpson´s e os respectivos intervalos de confiança de 44 isolados de colonização para os três métodos de tipificação: PFGE, emm “typing” e MLST (tabela 8). A comparação dos intervalos de confiança, para um nível de confiança de 95%, indicam que o emm “typing” e o MLST apresentam um poder discriminatório muito semelhante, porque os intervalos de confiança quase que se sobrepõem, enquanto que o PFGE apresenta um I.C. maior e consequentemente um maior poder discriminatório. Comparando estes três métodos pode-se dizer que o emm “typing” e o MLST possuem índices de diversidade semelhantes (0,875 e 0,881, respectivamente), enquanto que o PFGE possui um índice de diversidade de Simpson´s maior (0,982). Quer isto dizer que duas estirpes, escolhidas aleatoriamente, têm 98% de hipóteses de pertencerem a tipos diferentes, no caso do PFGE e 88% no caso do emm “typing” e do MLST. Tabela 8– Índice de Diversidade de Simpson´s (D), Intervalos de Confiança (I.C) e número de tipos obtidos por PFGE, emm “typing” e MLST para 44 isolados de colonização por Streptococcus pyogenes. Métodos Genotípicos PFGE emm type MLST. D. I.C. (95%). 0,982 0,875 0,881. (0,967-0,997) (0,824-0,926) (0,828-0,933). Nº de tipos 33 11 12. 3.12- Inferência Estatística 3.12.1-Teste de independência do Qui-Quadrado- simulação de Monte-Carlo. Para responder a duas hipóteses diferentes: H1: não existe associação entre os clusters e os “Day Care Centers- DCCs”, H2: não existe associação entre os clusters e as estações Primavera, Outono e Inverno recorreu-se ao valor do teste exacto de Fisher que é consonante com a simulação de Monte-Carlo (38), uma vez que os pressupostos do Qui-quadrado não se encontravam satisfeitos, nomeadamente o pressuposto de que não deve haver mais do que 20% das células das frequências esperadas inferiores a 5 (anexo IX e X). Como referido anteriormente, o número total de estirpes clonalmente relacionadas foi de 397. Assim, constata-se que a distribuição dos clusters varia consoante os DCCs, χ2 = 604,503, p=0,000 (anexo IX) e que a distribuição dos clusters varia consoante as estações da Primavera, Outono e Inverno, χ2 = 275,313 p=0,000 (anexo X). A análise das diferenças foi efectuada com o apoio nos resíduos. 31.
Imagem

Documentos relacionados
Afinal de contas, tanto uma quanto a outra são ferramentas essenciais para a compreensão da realidade, além de ser o principal motivo da re- pulsa pela matemática, uma vez que é
Assim, propusemos que o processo criado pelo PPC é um processo de natureza iterativa e que esta iteração veiculada pelo PPC, contrariamente ao que é proposto em Cunha (2006)
Apresenta a Campanha Obra-Prima, que visa a mudança comportamental por meio da conscientização diante de algumas atitudes recorrentes nas bibliotecas da
Através da revisão de literatura e a análise crítica do autor, este trabalho apresenta uma proposta de “estrutura conceitual” para a integração dos “fatores humanos” ao
No Estado do Pará as seguintes potencialidades são observadas a partir do processo de descentralização da gestão florestal: i desenvolvimento da política florestal estadual; ii
É possível estabelecer um elo de ligação e comparação entre o sangue periférico e a saliva, no que concerne a diferentes estados fisiológicos e patológicos
O mecanismo de competição atribuído aos antagonistas como responsável pelo controle da doença faz com que meios que promovam restrições de elementos essenciais ao desenvolvimento