UNIVERSIDADE FEDERAL DO PARÁ INSTITUTO DE CIÊNCIAS BIOLÓGICAS
PROGRAMA DE PÓS-GRADUAÇÃO EM GENÉTICA E BIOLOGIA MOLECULAR
Perfil de microRNAs Hepáticos pode Regular Apoptose, Lesões Vasculares
e Inflamação na Dengue Hemorrágica
LAYANNA FREITAS DE OLIVEIRA
LAYANNA FREITAS DE OLIVEIRA
Perfil de microRNAs Hepáticos pode Regular Apoptose, Lesões Vasculares
e Inflamação na Dengue Hemorrágica
Tese submetida ao Programa de Pós-Graduação em Genética e Biologia Molecular da UFPA como requisito para obtenção do Doutorado em Genética e Biologia Molecular.
Orientador: Prof. Dr. Rommel Mário Rodriguez Burbano
INSTITUIÇÕES FINANCIADORAS
Instituto Evandro Chagas (IEC), Ananindeua –PA Universidade Federal do Pará (UFPA), Belém – PA
AGRADECIMENTO
Agradeço ao Dr. Márcio Nunes, por ter me incluído com tanta receptividade e generosidade no seu grupo do Centro de Inovações Tecnológicas (CIT) e ter me apoiado incondicionalmente na execução desse projeto, sempre apoiando minhas decisões.
Aos Profs. Dr. Eduardo Santos e Rommel Burbano, que me inspiraram e motivaram ao longo desses quatro anos de dificuldades e aprendizado.
Gratidão a todos integrantes do CIT, por serem excelentes no trabalho em equipe, pela honestidade e amizade, que torna o laboratório não só produtivo como divertido. Meu agradecimento especial ao João Vianez, Rodrigo, Daisy e Jedson, pela contribuição neste trabalho, seja diretamente em experimentos e análises ou me substituindo em minhas tarefas quando eu já não era capaz de cumpri-las sozinha. Obrigada a todos amigos do CIT, em especial Eduardo Lana, Luciano, Poliana, Rafael, Tibério e Gabriela.
Agradeço imensamente às minhas amigas, orientadoras e conselheiras Janaína e Maria Helena, por toda paciência, ajuda, companheirismo nos momentos difíceis e felizes; a parceria de vocês faz com que eu tenha o triplo da capacidade que teria sozinha, obrigada minhas irmãs. À minha família, obrigada por entender todas ausências nos aniversários e reuniões ao longo desses anos e continuar me incentivando e se orgulhando de cada passo avançado. Agradeço especialmente meus pais Alberto e Ivanete e minha irmã Laenna por serem meu alicerce e nunca me deixarem hesitar.
Gratidão em todos aspectos ao meu esposo João, pelas valiosas planilhas que me pouparam semanas de trabalho manual, por me dar força e entender de forma tão carinhosa minha excessiva dedicação ao doutorado nesses anos. Se eu já tive a soma direta dos amigos no trabalho, com minha família e marido, tive a força de sete, oito, nove pessoas para enfrentar todas dificuldades que apareceram neste trabalho, só posso dizer no plural, que conseguimos finalizar o nosso desafio!
RESUMO
ABSTRACT
LISTA DE ILUSTRAÇÕES
LISTA DE TABELAS
Tabela 1. Dados clínicos e dos resultados dos sequenciamentos por amostra. IAM: infarto agudo do miocárdio; Dhf: dengue hemorrágica...45
Tabela 2. Lista dos 30 microRNAs mais expressos nos grupos de amostras de tecido hepático de controles e dengue hemorrágica (DHF) com respectivos valores de média de expressão por grupo, normalizados em counts per million (CPM)...47
Tabela 3. Resumo dos resultados obtidos da análise de expressão diferencial, apresentados os 38 microRNAs com valor de p < 0,05 cujas expressões em CPM foram maiores que 5. Os microRNAs com diferença significante são os com FDR < 0,05 e logFC > 2...49
Tabela Suplementar 1. Resultados do teste para comparação entre os grupos de dengue hemorrágica (DHF) com menos e mais de 30 anos de idade. logFC: log do Fold Change; GLM: General Linear Model; FDR: False Discovery Rate...78
Tabela Suplementar 2. Dez grupos de anotação funcional dos genes alvo dos miRNAs regulados positivamente no tecido hepático de indivíduos que faleceram de dengue hemorrágica. Presente as vias detectadas e os genes relacionados a cada uma com seus respectivos escores de enriquecimento, valores de p com ajuste de Benjamini...79
LISTA DE ABREVIATURAS E SIGLAS
DENV _ Virus dengue EBV _ Virus Epstein-Barr HBV _ Virus da hepatite B HCV _ Virus da hepatite C
JEV _ Virus da encefalite japonesa RNA _ ácido ribonucleico
dsRNA _ RNA dupla fita miRNA _ MicroRNA mRNA _ RNA mensageiro ncRNA _ RNA não codificante piRNA _ RNA de interação com piwi RNAi _ interferência por RNA
sfRNA _ RNA subgenômico de flavivirus siRNA _ pequenos RNA de interferência ssRNA _ RNA simples fita
ALT _ alanina aminotransferase AST _ aspartato amino transferase C _ proteína do capsídeo
cDNA _ DNA (ácido desoxiribonucleico) complementar CPM _ counts per million
DC _ célula dendrítica DE _ Expressão diferencial E _ proteína do envelope FC _ Fold Change
FDR _ False Discovery Rate
DHF _ Febre Hemorrágica da Dengue GLM _ General Linear Model
GO _ Gene Ontology
hg_19 _ human genome versão 19 HLA _ antígeno leucocitário humano IAM _ infarto agudo do miocárdio IFN _ Infterferon
Ig _ Imunoglobulina
IH _ inibição da hemaglutinação
KEGG _ Kyoto Encyclopedia of Genes and Genomes LMA _ Leucemia Mielóide Aguda
M _ proteína de mebrana
MBL _ lecitina ligadora de manose MDS _ multidimensional scale
MRE _ elemento de reconhecimento de miRNA NS _ proteína não estrutural
ORF _ quadro aberto de leitura
PAGE _ eletroforese em gel de poliacrilamida PCR _ reação em cadeia da polimerase pH _ potencial hidrogeniônico prM _ proteína pré membrana
SCD _ síndrome do choque da dengue SNP _ polimorfismo de único nucleotídeo TGF _ fator de trtansformação do cresciemento TNF _ fator de necrose tumoral
UTR _ região não traduzida
SUMÁRIO
1 INTRODUÇÃO ... 16
1.1 Contextualização ... 16
1.2 O Vírus da dengue ... 17
1.3 Classificação, diagnóstico e tratamento ... 21
1.3.1 Classificação ... 21
1.3.2 Diagnóstico Laboratorial ... 23
1.3.3 Tratamento ... 25
1.4 Epidemiologia ... 25
1.5 Infecção e Imunopatogenia ... 30
1.6 RNAi: microRNAs ... 34
1.7 Regulação de miRNAs em doenças ... 36
1.8 Objetivos ... 40
1.8.1 Objetivo Geral ... 40
1.8.2 Objetivos Específicos ... 40
2 MATERIAL E MÉTODO ... 41
2.1 Amostras ... 41
2.2 Extração de miRNA e Sequenciamento ... 41
2.3 Análises dos Dados de Sequenciamento ... 42
2.4 Análise de miRNAs Diferencialmente Expressos ... 43
2.5 Predição de Alvos dos miRNAs ... 43
2.6 Análise Funcional ... 43
3 RESULTADOS ... 44
3.1 Sequenciamento ... 44
3.2 Identificação de miRNAs expressos ... 46
3.3 Análise de Expressão Diferencial: DHF e Controles ... 47
3.4 Análise de Expressão Diferencial: DHF < 30 anos e DHF > 30 anos. ... 54
3.5 Predição de Genes Alvo e Anotação Funcional ... 56
4 DISCUSSÃO ... 61
5 CONCLUSÃO ... 67
1 INTRODUÇÃO
1.1 Contextualização
A dengue é a arbovirose mais prevalente entre humanos, uma doença é caracterizada principalmente por febre, artralgia, mialgia e exantema. O Vírus dengue (Dengue virus – DENV) é transmitido pelos mosquitos Aedes aegypti e Ae. albopictus, que são vetores da doença e endêmicos em países tropicais e subtropicais. Uma vez no organismo do vetor, após o repasto sanguíneo em um indivíduo infectado, o DENV passa por um período de incubação extrínseca, de oito a doze dias, e a partir daí está apto a transmitir o vírus até o fim do seu ciclo de vida (de seis a oito semanas).
O DENV e seu mosquito vetor são amplamente distribuídos ao redor do mundo, estando atualmente presentes em todos os continentes. Existem quatro sorotipos virais que apresentam proteção cruzada parcial entre si, todos são capazes de causar doença. A dengue é endêmica em mais de 100 países e aproximadamente 300 milhões de pessoas no mundo são infectadas pelo Vírus dengue por ano, 25% dos infectados desenvolvem quadros de dengue (Bhatt et al. 2013). O período de incubação do vírus no hospedeiro humano é em média de cinco a seis dias, e a transmissão para o mosquito pode ocorrer durante o período de viremia, que começa um dia antes do aparecimento da febre, e vai até aproximadamente o quinto dia de doença. Foram identificados ciclos silvestres do DENV, tendo primatas não humanos como hospedeiros e mosquitos do gênero Aedes como vetores (Rico-Hesse 1990; Wang et al. 2000); há evidências de que na Ásia ou África, esses ancestrais silvestres tenham dado origem aos sorotipos DENV que hoje infectam humanos.
O quadro clínico varia desde formas mais brandas, chamadas Dengue Clássica, até quadros mais graves chamados Febre Hemorrágica da Dengue, caracterizada por uma síndrome de extravasamento plasmático que pode levar o indivíduo à morte se não tratado corretamente. Essas diferenças apresentadas entre infecções assintomáticas e diferentes níveis de gravidade da doença são alvos de extensas pesquisas, acredita-se que a causa seja imunopatológica, grande parte da sintomatologia da dengue deve-se à resposta do hospedeiro contra o vírus (Sun and Kochel 2013; Jain et al. 2014).
A presença de tipos diferentes de miRNA é altamente variável e sensível a muitas alterações no organismo, provocadas por exemplo por infecções, câncer e envelhecimento. Os microRNAs podem ser usados como marcadores de alterações (biomarcadores), possivelmente como mecanismos de tratamento, além de serem extremamente úteis no esclarecimento de mecanismos patogênicos das doenças.
1.2 O Vírus da dengue
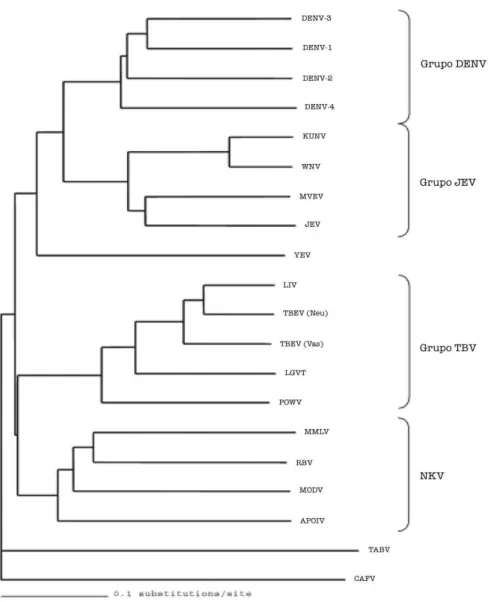
O Vírus dengue (DENV) pertence ao gênero Flavivirus, da família Flaviviridae. Existem 53 vírus que infectam humanos e animais, pertencentes ao gênero Flavivirus, que são classificados também como arbovírus, que são vírus transmitidos aos hospedeiros vertebrados através de vetores artrópodes como mosquitos e carrapatos (Figura 1).
Existem vários grupos sorológicos de arbovírus no gênero Flavirirus, distinguidos através de testes mediados por anticorpos, inibição da hemaglutinação (IH) e reação de detecção da proteína NS1 (método mais recente que os anteriores); para o Vírus dengue são determinados quatro grupos: DENV-1, DENV-2, DENV-3 e DENV-4.
Figura 1: Árvore neighbor-joining compilada com sequências de flavivírus disponíveis no Genbank, usando Kimura dois parâmetros como fórmula de distância. As abreviações e nomenclaturas são as mesmas do Comitê Internacional de Taxonomia de Vírus (ICTV). Vírus incluídos são: dengue 1-4 (DENV-1, 2, 3 e 4); Kunjin (KUNV); West Nile (WNV); Murray Valley encephalitis (MVEV); Japanese encephalitis (JEV); Yellow fever (YFV); Louping Ill (LIV); Tick-borne encephalitis (TBEV) linhagem Neudorlf (Neu); Tick-borne encephalitis (TBEV) linhagem Vasilchenko (Vas); Langat (LGTV); Powassan (POWV); Montana myotis leukoencephalistis (MMLV); Rio Bravo (RBV); Modoc (MODV); Apoi (APOIV); Tamana bat (TABV); Agente de fusão celular (CFAV). NKV - vetor artrópode desconhecido (Halstead 2008b).
Figura 2: Esquema da estrutura de proteínas de um flavivírus imaturo associado à célula (à esquerda) e a partícula madura à direita (Halstead 2008b).
A proteína E é uma proteína N-glicosilada de fusão da membrana, composta por três domínios envolvidos na virulência: domínio I e domínio II abrigam o peptídeo de fusão em sua extremidade distal e o peptídeo III é responsável pela ligação com receptor (Chen et al. 1996). Formando o capsídeo encontram-se heterotetrâmeros de E:M:M:E que ancoram a região subjacente à bicamada lipídica do envelope, através de hélices transmembrana E-T1, E-T2, M-T1 e M-T2 esse conjunto forma a estrutura viral e envolve o material genético (Zhang et al. 2013).
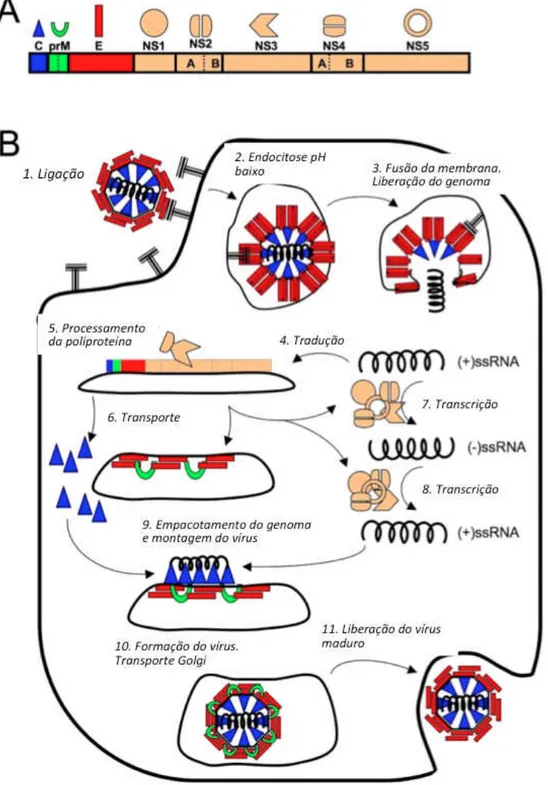
O genoma viral completo possui aproximadamente 11 kb e apenas uma matriz de leitura aberta (open reading frame – ORF), flanqueada por regiões não traduzidas (untranslated region - UTR) a 5’ de 100 nt, com um cap tipo I m7G5’ppp5’A no mRNA e a 3’ com 400 a 800 nt, essas regiões possuem várias sequências conservadas e estruturas de haste e laço que acredita-se estarem envolvidas nos processos de replicação, tradução e interação com o hospedeiro (Brinton et al. 1986; Hahn et al. 1987).
Figura 3: Esquema representativo do ciclo de replicação do Vírus dengue. A) Legenda das proteínas codificadas pelo vírus dengue em cada região do genoma. B) Passo-a-passo da entrada na célula, replicação, maturação e liberação da partícula madura final (Tomlinson et al. 2009).
2011; Jain et al. 2014). Como principal proteína exposta na superfície viral, a glicoproteína E deve executar funções de ligação com receptores celulares, fusão com membranas celulares do hospedeiro, direcionamento da montagem viral e apresentação de grupos antigênicos que induzem a resposta imune (Modis et al. 2004).
Depois de infectar o mosquito, o vírus exerce intensa replicação no esôfago, a partir daí que a infecção será disseminada para outros tecidos. Para que o vírus seja transmitido durante o repasto sanguíneo para um hospedeiro humano ou primata, o DENV deve chegar até as glândulas salivares e promover replicação para que seja secretado na saliva (Halstead 2008a).
1.3 Classificação, diagnóstico e tratamento
A dengue é uma doença aguda febril de amplo espectro de manifestações, desde uma infecção assintomática, na maioria dos casos, até casos muito graves que necessitam acompanhamento médico constante.
Depois do período de incubação, geralmente 3 a 6 dias após a inoculação do vírus, quadros de dengue tem início abrupto com febre alta (entre 39oC e 40oC). Em crianças pequenas, a fase de febre pode levar à desidratação e distúrbios neurológicos que podem causar convulsões. A doença é altamente incapacitante, causando grandes limitações para o indivíduo, devido a intensa cefaleia, dor retro-orbital, mialgia, artralgia, petéquias, exantema e leucopenia. O exantema está presente em aproximadamente 50% dos casos e é principalmente do tipo maculo-papular, atingindo face, tronco e membros (WHO 2009; Brasil 2013).
1.3.1 Classificação
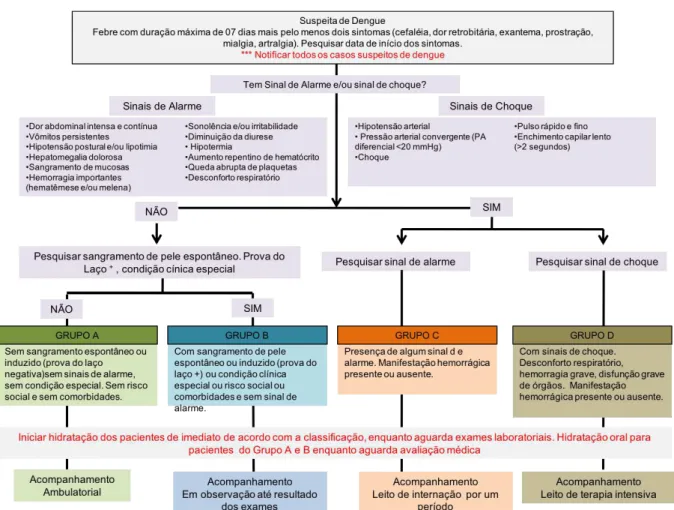
Figura 4. Fluxograma com a classificação atual de risco e manejo clínico de caso suspeito de dengue (Ministério Da Saúde 2016).
Apesar da classificação atual, a anterior e os termos Dengue Clássica (Dengue Fever - DF), Febre Hemorrágica da Dengue (Dengue Hemorrhagic Fever – DHF) e Síndrome do Choque da Dengue (Dengue Shock Syndrome – DSS) ainda são amplamente utilizados principalmente na literatura científica, por esse motivo mais o fato de que na época do diagnóstico das amostras utilizadas neste trabalho, a classificação adotada era a anterior, neste trabalho será utilizada a nomenclatura febre hemorrágica da dengue ou dengue hemorrágica (DHF – dengue hemorrhagic fever). A classificação anterior é detalhada a seguir:
a) Dengue Clássica (DC)
É o caso suspeito, confirmado laboratorialmente. Em epidemias, os primeiros casos da área devem ter confirmação laboratorial e os demais diagnósticos podem ser clínico-epidemiológicos;
b) Febre Hemorrágica da Dengue (DHF)
Caso confirmado laboratorialmente, com a presença dos seguintes critérios: - Febre ou história de febre recente de sete dias;
- Tendências hemorrágicas evidenciadas por: prova do laço positiva, petéquias, equimoses ou púrpuras, sangramentos de mucosas do trato gastrintestinal e outros;
- Extravasamento de plasma causado por aumento de permeabilidade capilar detectado por: hematócrito com aumento de 20% sobre o basal inicial; queda do hematócrito em 20%, após o tratamento adequado; derrame pleural, ascite ou hipoproteinemia.
A DHF pode ser classificada de acordo com o nível de gravidade apresentado:
- Grau I: febre acompanhada de sintomas inespecíficos, em que a única manifestação hemorrágica é a prova do laço positiva;
- Grau II: além das manifestações do grau I, hemorragias espontâneas leves (sangramento de pele, epistaxe, gengivorragia e outros);
- Grau III: Síndrome do Choque da Dengue (SCD), colapso circulatório com pulso fraco e rápido, estreitamento da pressão arterial ou hipotensão, pele pegajosa e fria e inquietação;
- Grau IV: Síndrome do Choque da Dengue (SCD), ou seja, choque profundo com ausência de pressão arterial e pressão de pulso imperceptível.
1.3.2 Diagnóstico Laboratorial
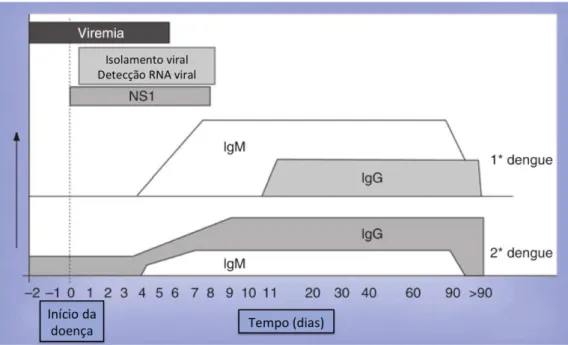
Figura 5: Janela de detecção de antígenos e anticorpos para o diagnóstico da dengue. NS1: proteína não estrutural 1; IgM: imunoglobulina M; IgG: imunoglobulina G. (Tang and Ooi 2012).
A viremia inicia 2 a 3 dias antes do aparecimento dos sintomas e perdura até 4 a 5 dias depois. Durante esse período, sangue, plasma ou soro podem ser usados para o isolamento do vírus. A técnica mais usada na rotina de diagnóstico é a inoculação em linhagens de células C6/36 derivadas de Ae. albopictus (Tesh 1979), o sucesso do isolamento pode ser verificado através do efeito citopático e por ensaios de imunofluorescência com anticorpos monoclonais.
Outro método utilizado é a reação em cadeia da polimerase (PCR) precedida de transcrição reversa (RT-PCR); realizada após extração de RNA viral a partir de sangue, soro ou plasma; é um método sensível e específico para confirmação de dengue. Vários primers e protocolos estão descritos para amplificação de regiões do genoma do DENV por meio de PCR convencional ou PCR em tempo real, utilizando SYBR Green ou sondas oligonucleotídicas.
resposta de anticorpos antidengue apresenta reação cruzada entre os quatro sorotipos e outros flavivírus; falsos positivos são encontrados em pacientes com infecções prévias de dengue e malária (Groen et al. 2000; Prince and Matud 2011).
1.3.3 Tratamento
O controle da reprodução do vetor é o único método de prevenção da dengue disponível. Ainda não existem vacinas ou drogas antivirais específicas para o DENV, mas existem alguns modelos candidatos em desenvolvimento, entre eles vacinas de vírus vivos atenuados, vírus vivos quiméricos (GUY et al., 2015), vírus inativados e de vírus vivos recombinantes (MURRELL; WU; BUTLER, 2011). O tratamento dos pacientes consiste em cuidado de apoio aos sintomas como repouso, antitérmicos e analgésicos. Em casos de choque hipovolêmico, é imprescindível hospitalização para reposição de fluidos perdidos com o extravasamento vascular. Aspirinas e outros salicilatos devem ser evitados devido o aumento de permeabilidade capilar (Tang and Ooi 2012).
Na ausência de sinais de alarme, a conduta utilizada é apenas o acompanhamento ambulatorial e hidratação oral. Drogas sintomáticas são recomendadas para pacientes com febre elevada e dor. Nos casos de dengue com sintomas clássicos sem sinais de alarme mas com a presença de sangramento espontâneo (petéquias) ou induzido (prova do laço) e condições clínicas especiais e/ou comorbidades, a conduta terapêutica consiste nas mesmas citadas anteriormente e além disso um acompanhamento de exames constantes, verificando sempre o estado do hemograma e hematócrito. Nos casos com presença de sinais de alarme, a hidratação venosa deve ser imediata, dependendo da gravidade do quadro e dos resultados dos exames, o paciente deve ser encaminhado para leito de internação por no mínimo 48h, nos casos de choque a internação deve ser em leitos de terapia intensiva com reposição volêmica e avaliação clínica a cada 15-30 minutos (Brasil 2013; Bäck and Lundkvist 2013).
1.4 Epidemiologia
que um indivíduo se deslocasse entre continentes diferentes dentro do período de incubação/viremia possibilitando a transmissão (Gubler 2002). O maior registro de pandemia ocorreu em 1998, quando 56 países foram atingidos e cerca de 1,2 bilhão de pessoas foram infectadas (Gurugama et al. 2010).
Cuba foi o primeiro país da América Latina a relatar uma grande epidemia de dengue no período de 1977-78, com o sorotipo DENV-1. A primeira epidemia de DHF ocorreu em 1981 com o sorotipo DENV-2. Com um programa de controle do vetor, Cuba alcançou um período de 16 anos sem dengue, até 1997, quando outra epidemia de DENV-2 afetou a região (Guzmán et al. 1999).
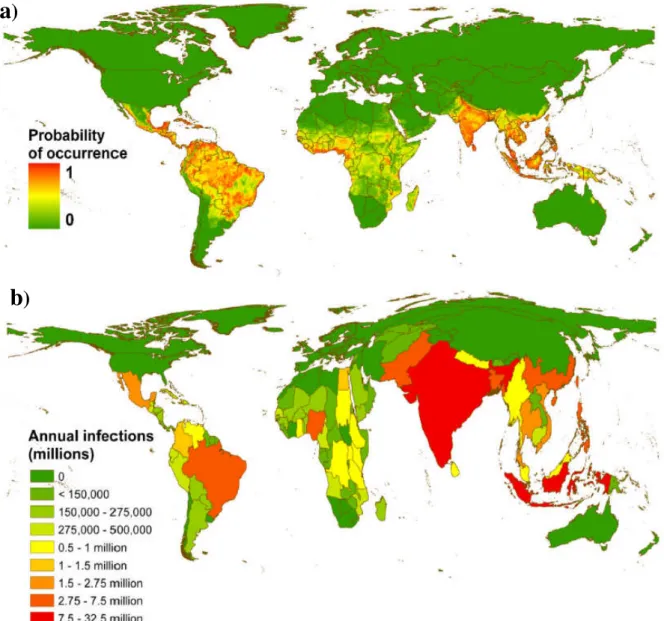
Figura 6: Presença e risco global de dengue em 2010. a, probabilidade de ocorrência de dengue com resolução espacial de 5Km. Áreas com alta probabilidade estão em vermelho e com baixas probabilidades estão em verde. b, mostra o cartograma do número anual de infecções para todas idades (Bhatt et al. 2013).
a
)
Os primeiros casos de dengue registrados no Brasil datam do século XIX. Em 1981-82 os vírus DENV-1 e DENV-4 foram isolados pela primeira vez durante a epidemia em Boa Vista, Roraima, região próxima da Venezuela; foram efetuadas ações de controle do mosquito vetor e não houveram epidemias no Brasil dentro dos quatro anos seguintes. Vieram ocorrer novamente epidemias de dengue causadas pelo vírus DENV-1, nos estados do Rio de Janeiro, São Paulo e estados do nordeste do Brasil nos anos 1986 e 1987.
Em 1990 e 1991 a epidemia foi provocada por vírus do sorotipo 2 e acometeu novamente os estados do Rio de Janeiro, São Paulo, além de Mato Grosso e Tocantins, DENV-1 também circulava nesse mesmo período em São Paulo e Minas Gerais (Dégallier et al. 1996; Nogueira et al. 2007).
Em 1992 foi identificada a reintrodução do Ae. aegypti no estado do Pará e em 1995 foram notificados os primeiros casos de dengue nas cidades de Redenção e Rondon do Pará. No ano de 1996, foram registrados em Belém oito casos de uma doença febril que viria a ser identificada como dengue semanas depois, após o isolamento do vírus DENV-1 e um ano depois o DENV-2 também foi isolado, a partir daí ambos sorotipos continuaram circulantes na cidade de Belém (Travassos da Rosa et al. 2000).
O sorotipo 3 foi predominante a partir de 2003, a partir de 2007, a prevalência foi do DENV-2 (Teixeira et al. 2013). Em julho de 2010 o sorotipo DENV-4 ressurgiu em Boa Vista (RR), após 28 anos ausente em epidemias no Brasil (Temporão et al. 2011). A Secretaria de Vigilância em Saúde do Ministério da Saúde (SVS) registrou um total de 1.484.899 casos prováveis de dengue no país até a semana epidemiológica 48 (30/12/2012 a 30/11/2013) de 2013. A Região Sudeste teve o maior número de casos (940.453 casos; 63,3%), seguida da Região Centro-Oeste (274.109 casos; 18,5%), Nordeste (151.012 casos; 10,2%), Sul (69.030 casos; 4,6%) e Norte (50.295 casos; 3,4%). A incidência global do país em 2013 foi de 765,5 casos por 100.000 habitantes e todas as regiões apresentaram incidência acima de 100 casos por 100.000 habitantes. Na análise comparativa em relação ao ano de 2012, observa-se aumento de 163% dos casos no país. No boletim epidemiológico de 2016, até a semana 16 os casos de dengue no Brasil já superam os números de 2015, e ambos já têm incidência maior que 2014 em pelo menos o dobro (Figura 7).
de mosquitos aumenta em períodos chuvosos, levando ao consequente aumento no número de casos, no Estado do Pará, o número máximo de casos registrados de dengue ocorre entre janeiro e março. O Ae. aegypti é altamente adaptado ao ambiente urbano e vive em proximidade com seres humanos, seu ciclo de alimentação permite com que um único mosquito consiga entrar em contato com múltiplos hospedeiros em um ciclo gonotrófico (Bäck and Lundkvist 2013).
Figura 7. Incidência de dengue no Brasil nos anos de 2014, 2015 e 2016 (até a 16a semana epidemiológica. (Fonte: Sinan Online em 25/04/2016).
Todos os quatro sorotipos do DENV (DENV-1, DENV-2, DENV-3 e DENV-4) são capazes de causar quadros graves de dengue, sendo que e a carga viral no decorrer da doença está fortemente associada ao desenvolvimento de dengue grave. Sorotipos 2 e DENV-4 aparecem menos frequentemente associados com quadros graves durante a infecção primária da dengue, enquanto que em infecções secundárias DENV-2 e DENV-3 são mais frequentemente associados a quadros graves (Fried et al. 2010).
É comum a ocorrência de oscilações na prevalência de cada sorotipo em um cenário de epidemia, normalmente um sorotipo é dominante durante um período de 2 a 4 anos, quando sua prevalência diminui e dá lugar a um outro sorotipo que o substitui (Adams et al., 2006). A causa para esse declínio seria o acúmulo de imunidade de rebanho em uma população, onde o número de indivíduos suscetíveis diminuiria ao longo desses anos (Whitehorn e Simmons, 2011).
em relação a adultos, provavelmente devido a uma maior permeabilidade do endotélio vascular (Gamble et al., 2000).
Durante epidemias em Cuba, foi observado que comorbidades pré-existentes tais como asma brônquica, diabetes melitus e anemia falciforme apresentavam altas frequências entre pacientes com quadros graves de dengue (Bravo et al., 1987; Díaz et al., 1988; Limonta et al., 2009). No Brasil, um estudo realizado nas cidades de Fortaleza e Salvador encontrou que entre os casos de DHF havia alta frequência de indivíduos com alergia e diabetes (Figueiredo et al., 2010). Também tem sido observado que condições clínicas crônicas estão presentes em 70% dos casos fatais de dengue (Rigau-Pérez e Laufer, 2006). Trabalhos em Singapura encontraram que em pacientes com hipertensão os quadros de dengue são geralmente benignos (Lye et al., 2010).
Evidências que a genética do hospedeiro influencia na suscetibilidade à dengue vêm de estudos caso-controle. Durante a infecção, os antígenos virais são apresentados aos linfócitos T através do complexo principal de histocompatibilidade, em humanos HLA (Human Leucocyte Antigen) de classe I e II. Diversos alelos diferentes do HLA, entre eles alelos de HLA-A e B são associados com diferentes fenótipos de dengue, alguns conferindo proteção ou suscetibilidade ao desenvolvimento de dengue e/ou DHF (Stephens et al., 2002; Coffey et al., 2009). Uma hipótese para a geração de suscetibilidade através de moléculas de HLA é que a natureza e a magnitude da resposta imune gerada apresentação de antígenos pelas moléculas de MHC de classe I poderia contribuir com a imunopatologia da dengue. (Kalayanarooj et al., 2007) foi o primeiro estudo a associar o grupo sanguíneo ABO com a dengue, este estudo encontrou que crianças com o tipo AB tinham maior probabilidade de desenvolver DHF do que crianças do tipo O, A ou B.
Vários polimorfismos de nucleotídeo único (Single Nucleotide Polymorphism – SNP) têm sido associados ao desenvolvimento da dengue e suas formas clínicas. Um SNP no gene que codifica receptores Fcγ presentes em células dendríticas (dendritc cell - DC) foi associado com proteção contra dengue grave (DG) em pacientes do Vietnã; polimorfismo não-sinônimo, alelo C no gene do receptor de vitamina D foi associado com proteção contra SCD; o alelo A na região promotora de TNF-α foi associado com risco de DG em pacientes da Venezuela; e polimorfismo na proteína de sinalização JAK1 foi associado com DG em população brasileira (Silva et al., 2010; Coffey et al., 2009; Stephens, 2010).
gene CD209 já foram identificados e associados a doenças em que o microrganismo utiliza esse receptor na infecção (Sakuntabhai et al. 2005; Wang et al. 2011; Oliveira et al. 2014).
1.5 Infecção e Imunopatogenia
No ponto inicial da infecção, durante o repasto sanguíneo (da fêmea do mosquito do gênero Aedes) em humanos, o DENV é injetado e infecta inicialmente as células de Langerhans (células dendríticas [dendritc cell - DC] epidérmicas) e queratinócitos (figura 8). As células infectadas migram do sítio de infecção para os linfonodos, onde monócitos e macrófagos são recrutados e se tornarão alvo da infecção, consequentemente o vírus replica-se e dissemina-se pelo sistema linfático. Em autópsias de casos fatais, foram encontrados vírus nos tecidos da pele, fígado, baço, linfonodos, rins, medula óssea, pulmão, timo e cérebro (Martina et al. 2009; Wan et al. 2013).
Figura 8: Resposta imonológica cutânea à infecção do Dengue virus (DENV). Célula dendrítica (DC).Modificado de(St John et al. 2013).
mediadores envolvidos na inflamação e homeostase do hospedeiro (Libraty et al. 2001; Fernandez-Gracia et al. 2009; Martina et al. 2009).
A grande variedade de tecidos e células infectadas do hospedeiro sugere que os receptores para o DENV sejam amplamente distribuídos. Atualmente, vários são os candidatos a receptores para o vírus, entre eles Lecitina Ligadora de Manose (MBL), Heparam Sulfato, Sulfato de Condroitina e CD209 (Libraty et al. 2001; Avirutnan et al. 2007; Miller et al. 2008). O DENV causa lesão em vários órgãos, sendo o fígado o mais atingido, é um sítio de intensa replicação e contribui significativamente para o aumento da carga viral. Os receptores hepáticos descritos são os proteoglicanos de heparam sulfato, via de entrada do DENV nos hepatócitos, que são as células mais atingidas durante a infecção junto com as células de Kupffer (Hilgard and Stockert 2000; Sreekanth et al. 2016). As alterações mais importantes no fígado durante a dengue são mediadas pela apoptose e necrose de hepatócitos, essas alterações acompanham insuficiência portal e infiltrado inflamatório intralobular de células mononucleares (Pagliari et al. 2014). Em análises histopatológicas de fígado de casos fatais por dengue, são encontrados adicionalmente hemorragia, edema, danos causados por alterações metabólicas e reações inflamatórias, esteatose, hiperplasia e destruição das células de Kupffer (Póvoa et al. 2014). Acredita-se que a inflamação no fígado se desenvolva com o objetivo de proteger os hepatócitos de dano, favorecer o reparo dos tecidos lesionados e para o reestabelecimento da homeostase (Brenner et al. 2013).
A infecção e inflamação no fígado leva ao desenvolvimento de um ambiente com várias células expressando IL-6 e TGF-beta, com ausência de células expressando Foxp3, identificado no estudo de Pagliari et al. (2014). A expressão de TGF-beta pode estar relacionada à apoptose de hepatócitos, caracterizada histologicamente pela presença dos corpúsculos de Councilman. Tanto IL-6 como TGF-beta são citocinas com funções importantes na inflamação e provavelmente estão associadas ao aumento da permeabilidade vascular, extravasamento plasmático e consequente agravamento dos sintomas da dengue (Pagliari et al. 2014). TNF-alfa, IFN-gama e IL-2 também são citocinas vasoativas que podem ser expressas durante a infecção, (Lühn et al. 2007) identificaram a capacidade de linfócitos Treg em inibir a produção dessas citocinas durante a dengue.
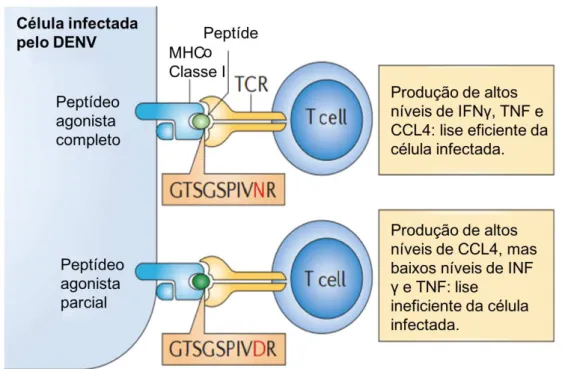
imunopatologia. De acordo com a teoria do “pecado original antigênico” células T de memória de baixa afinidade geradas na primeira infecção de DENV se expandem seletivamente durante a segunda infecção por outro sorotipo, isso acontece antes da ativação de células T naive de maior avidez (Figura 9). A reação cruzada leva a produção de grandes concentrações de citocinas pro-inflamatórias que contribuem para a patogênese e extravasamento plasmático na dengue, entretanto esse processo não é necessário para causar dengue graves em crianças (Wan et al. 2013; Weiskopf and Sette 2014).
Figura 9. Interação de linfócitos T com células infectadas e o efeito da ativação por peptídeos totalmente agonistas e agonistas parciais (Rothman 2011).
Tanto a resposta imune celular quanto a humoral contribuem para a eliminação do vírus. A inibição da infectividade viral por anticorpos de neutralização tem um importante papel na proteção contra a dengue. Assim como no modelo celular descrito, o papel da indução de anticorpos também pode apresentar dois lados, gerando proteção e contribuindo para o desenvolvimento de quadros mais graves da doença.
com epítopos E, a afinidade do anticorpo, a acessibilidade do epítopo na superfície viral e a interação anticorpo-complemento (Murphy and Whitehead 2011).
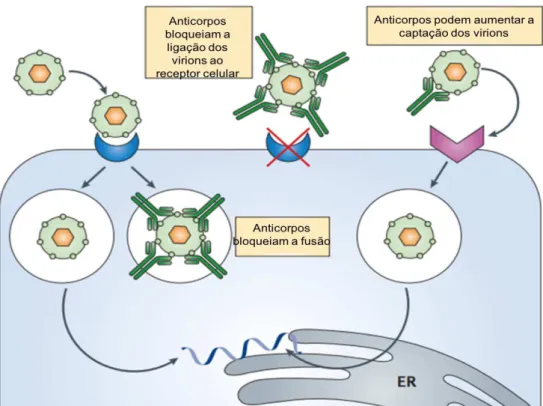
A infecção pode ser prevenida pelos anticorpos por vários mecanismos que incluem: bloqueio de ligação, inibição da fusão da membrana do vírus com a membrana do endossomo (o que impede a liberação do RNA viral no citoplasma) e lise de vírus cobertos por anticorpos pelo sistema complemento.
Assim como na resposta celular, o modelo de imunoamplificação dependente de anticorpo (ADE – antibody-dependent enhancement) é um mecanismo associado à gravidade da dengue (Halstead et al. 1977; Chareonsirisuthigul et al. 2007). Nesse modelo, os anticorpos heterólogos provenientes de uma infecção prévia por outro sorotipo poderiam exacerbar a infecção pelo Vírus dengue através de células que apresentam receptores Fcγ (Figura 10). Esse mecanismo pode aumentar a eficiência da infecção e suprimir a resposta anti-viral mediada por interferon (IFN) do tipo I (Halstead 2012; Ho et al. 2013).
Figura 10. Efeito da imunoamplificação dependente de anticorpo na infecção e replicação viral (Rothman 2011).
poderiam contribuir com o extravasamento vascular endotelial e trombocitopenia nos casos graves de dengue (Ho et al. 2013).
Entre estratégias de regulação associados à imunopatogenia da dengue, alguns mecanismos podem influenciar diretamente nas respostas imunológicas, como o mecanismo de interferência por RNA (RNAi) que pode determinar desde a eficiência da replicação viral até regular a expressão de citocinas como IFN, IL, TNF e TGF. Entender a dinâmica da infecção e o processo patogênico é indispensável para o desenvolvimento de métodos de proteção ou tratamento contra a dengue, os mecanismos de regulação da doença como resposta celular e humoral, além de mecanismos epigenéticos como RNA de interferência (RNAi) no hospedeiro humano e no mosquito, devem ser profundamente identificados. Muitos trabalhos têm investigado a função do RNAi nos mosquitos Aedes; no hospedeiro humano, o número total de publicações investigando influência de RNAi durante a dengue no hospedeiro é menor, portanto ainda é uma grande área a ser explorada.
1.6 RNAi: microRNAs
Interferência por RNA (RNAi) é um processo epigenético de controle da expressão gênica ao nível pós-transcricional e de alta especificidade, o termo RNAi é usado para descrever de forma ampla, a inibição da expressão gênica pelos pequenos RNAs não codificantes. Epigenética refere-se a mudanças na expressão gênica disparadas por estímulos e causadas por processos independentes de modificações na sequência do DNA, os mecanismos incluem: metilação do DNA e de histonas, remodelagem da cromatina e silenciamento por ncRNA (Gómez-Díaz et al. 2012).. A complementariedade do RNA associado a proteínas como endonucleases e argonautas, resulta na degradação do RNA mensageiro (mRNA) alvo. Esses RNA não codificantes (ncRNAs) são gerados dentro da célula, a partir de longas cadeias de RNA dupla fita (dsRNA) ou a partir de moléculas exógenas (Gavazzo et al. 2013). Os ncRNA não codificantes estão envolvidos em vários tipos de funções, além da regulação gênica por inibição direta do mRNA, exercem funções na defesa contra vírus, defesa contra atividade de transposons, orientação na modelagem da cromatina, metilação de RNA e formação do spliceossomo. Os principais tipos de RNAs regulatórios e mais estudados são: microRNA (miRNA), pequenos RNAs de interferência (siRNA) e RNA de interação com piwi (piRNA), estes últimos envolvidos no controle do desenvolvimento germinativo (Gavazzo et al. 2013).
Os siRNAs são pequenas moléculas dsRNA produzidas a partir de cadeias extensas de dsRNA de origem endógena ou exógena, enquanto os miRNAs são pequenas moléculas de RNA fita simples (ssRNA) produzidos de estruturas intracelulares dupla fita, os hairpins, de um RNA precursor ou transcritas de genes contendo palíndromos (Dogini et al. 2014).
Os miRNAs envolvidos na regulação da expressão gênica, possuem uma região entre a base 2 e 8, chamada região seed (semente), que é a sequência conservada na molécula e extremamente importante no reconhecimento do alvo, localizado em geral nas regiões 3’UTR de mRNAs, chamados de elementos de reconhecimento de miRNA (miRNA recognition element – MRE). Muitas vezes os genes para miRNA podem estar localizadas dentro ou sobrepostas a sequências de mRNA já anotadas, alguns deles possuem seus próprios promotores independente do “gene hospedeiro”.
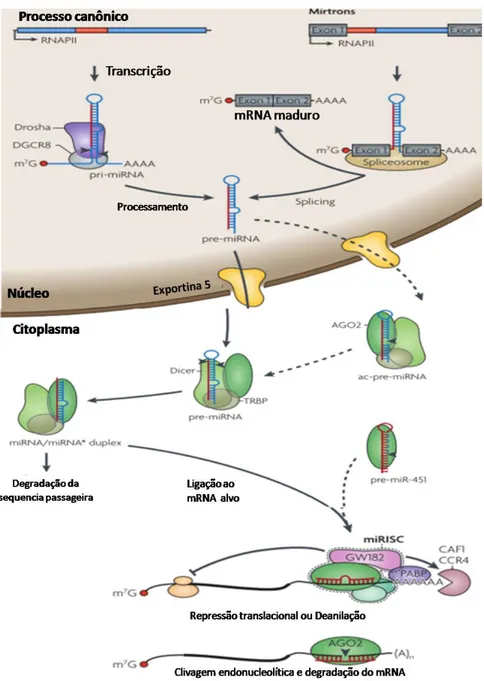
Os genes que codificam miRNAs são transcritos pela RNA polimerase II e ocasionalmente pela polimerase III; é produzido primeiramente o miRNA primário (pri-miRNA), uma molécula longa dobrada em um hairpin contendo modificações 5’ e 3’ idênticas as do mRNA (Krol et al. 2010). Ainda no núcleo, pri-miRNAs servem de substrato para duas enzimas da família das RNase III, as endonucleases Drosha e Dicer, o produto da clivagem da Drosha produz uma sequência de ~70 nt e é chamado pré miRNA (pre-miRNA) que é transportado para o citoplasma onde a Dicer processa e gera um duplex miRNA-3p/5p (anteriormente miRNA/miRNA*) de ~22 nt. Drosha e Dicer clivam respectivamente no ponto de início e término da sequência madura. Uma única fita do miRNA formado se liga ao complexo de silenciamento induzido por miRNA (RISC), esse complexo de proteínas ligado ao miRNA pareia com mRNA complementar levando a sua repressão traducional, deadenilação ou degradação (Bartel 2004; Krol et al. 2010). As proteínas argonautas (AGO) interagem com miRNA e atuam no sentido 3’ como efetoras da repressão, essenciais na montagem e função do RISC (Figura 11).
A nomenclatura oficial usada é a desenvolvida pelo miRBase, onde sequências numéricas indicam miRNAs diferentes e um prefixo de três a quatro letras indica a espécie, o termo “miR” indica que trata-se de uma molécula madura, enquanto os precursores são identificados por “mir”, sendo assim, hsa-miR-101 significa: Homo sapiens - miRNA maduro - código numérico de identificação individual (Griffiths-Jones et al. 2006).
1.7 Regulação de miRNAs em doenças
Os microRNAs apresentam expressão específica tecidual e temporal, sendo uma das principais funções associadas a regulação temporal do desenvolvimento (Lee et al. 1993). Até o momento, miRNAs foram associados à regulação da expressão de aproximadamente 1/3 dos genes humanos, executando importantes funções em vários processos biológicos como regulação do ciclo celular, apoptose, diferenciação celular e desenvolvimento embrionário (Bartel 2004). A regulação da função dos miRNAs também é finamente controlada por dezenas de enzimas e fatores de processamento; a regulação dos miRNAs ocorre através de RNases que degradam as sequências precursoras (por exemplo a RNase MCPIP1 degrada pre-miRNA antes que possa ser processado pela Dicer), pela degradação do miRNA maduro após sua ação, dissociando a proteína AGO do complexo (Graves and Zeng 2012) e ainda através de regulação por patógenos.
Figura 11: Biogênese de microRNA. Drosha processa o pri-miRNA no núcleo para produzir o pre-miRNA, em outra via de síntese, ela ocorre a partir de íntrons, chamados mirtrons, essa via dispensa a ação da Drosha pois a excisão do íntron por splicing já gera o precursor do miRNA. A partir daí as duas vias convergem para o transporte ao citoplasma, clivagem pela Dicer e ligação ao complexo de silenciamento induzido por miRNA – RISC (Krol et al. 2010).
Se tratando de infecções virais, existe uma lista extensa de associações, o miR-146a tem destaque entre os miRNAs regulatórios do sistema imune, sua regulação positiva já foi associada com aumento da taxa de replicação em cultivo de HCV, HBV e os arbovírus DENV e Virus da Encefalite Japonesa (JEV), pelo efeito de reprimir a liberação de mediadores inflamatórios e a resposta imune inata (Wu et al. 2013; Sharma et al. 2015; Bandiera et al. 2016; Ye and Steinle 2016). O miR-146a-5p foi encontrado sub-expresso no soro de indivíduos na fase virêmica da dengue, na contramão da tendência em associar sua expressão a prognósticos ruins. A suspeita levantada no estudo foi que ele poderia estar sendo produzido mas não excretado, dessa forma não ser identificado no perfil circulante. Os miRNAs superexpressos nessa coorte foram miR-21-5p, -590-5p, -188-5p e 152-3p, caracterizados como possíveis biomarcadores para dengue (Ouyang et al. 2016).
Nos perfis hepáticos, o miRNA mais marcante dentro do cenário de associação a doenças é o miR-122, que é específico do fígado e está relacionado à manutenção da homeostase, metabolismo de lipídeos, glicose e ferro. É fortemente associado a patologias hepáticas como infecções, esteato-hepatite não alcóolica, fibrose e câncer, apresenta em geral efeitos tanto na proteção quanto na progressão da doença, dependendo da etiologia e tipo celular envolvido (Brunetto et al. 2014; Tan et al. 2014a; Tsubota et al. 2014; Tan et al. 2014b; Castillo et al. 2015; Thakral and Ghoshal 2015; Dong et al. 2016; Liu et al. 2016).
Alguns miRNAs de origem viral e celular podem ter impacto positivo ou negativo na replicação viral, sequências produzidas por vírus podem regular diretamente a expressão gênica, podendo mimetizar miRNAs celulares e sequestrar redes regulatórias predefinidas (Wang 2009). Moléculas de RNA subgenômico em flavivirus (sfRNA), são ncRNAs resultantes da degradação incompleta do genoma de RNA, variam de tamanho entre espécies de flavivírus e podem possuir em torno de 500 nt. Esses sfRNAs são necessários na patogenicidade dos flavivirus em modelos animais, embora o mecanismo exato pelo qual isso ocorra não seja claro, foi demonstrado que essas moléculas também interferem na resposta por INF alfa e beta em camundongos e podem servir como a principal fonte de miRNA em células de mosquitos (Schnettler et al. 2012; Hussain and Asgari 2014; Pijlman 2014).
Vírus são parasitas intracelulares obrigatórios e dependem totalmente da maquinaria do hospedeiro para sobrevivência e propagação, por isso são altamente suscetíveis ao programa de regulação gênica da célula hospedeira. Além disso, também são capazes de modular a expressão gênica, que tem como alvo os genes do hospedeiro e seus próprios genes, utilizando ncRNAs é possível regular a reação do sistema imune e ampliar as possibilidades de escape.
O interesse nos miRNAs proporcionou um grande avanço de conhecimento nessa área desde a identificação de associação com doenças em 2002. O desenvolvimento de tecnologias de sequenciamento de nova geração que produzem resultados de alta profundidade, viabilizou um maior avanço das pesquisas associando miRNAs a diversas doenças e condições animais e vegetais, entre as doenças humanas o maior destaque está nas pesquisas em câncer, mas muita informação vem sendo produzida em estudo com microorganismos, incluindo vírus. A busca por biomarcadores para diagnóstico e oportunidades terapêuticas através da manipulação farmacológica de microRNAs são os principais objetivos das pesquisas.
A maioria dos estudos de miRNA em dengue descrevem e analisam aspectos da relação virus/mosquito ou miRNAs virais que tem alvos genes humanos. Em relação aos esforços para entender e aplicar a atuação de RNAi virais e no mosquito Aedes, podemos dizer que são poucos os trabalhos que tratam da expressão e regulação dos microRNAs no hospedeiro humano e sua relação com a modulação da resposta imune da dengue. Quanto ao fígado, as alterações bioquímicas durante a infecção são amplamente estudadas e descritas na literatura, assim como o perfil de citocinas expressas na dengue (Ahmed et al. 2014; Pagliari et al. 2014; Póvoa et al. 2014; Jain et al. 2014), entretanto, assim como em muitos tecidos, o perfil de expressão de microRNAs no ambiente hepático humano é desconhecido.
1.8 Objetivos
1.8.1 Objetivo Geral
Caracterizar o perfil de expressão diferencial de microRNAs do tecido hepático humano provenientes de casos fatais de DHF/dengue grave e propor os mecanismos de regulação dessas moléculas nas vias metabólicas relacionadas à patogenia da doença.
1.8.2 Objetivos Específicos
- Descrever o perfil de expressão de miRNA do fígado de indivíduos com óbito por DHF pelo método de miRNA-Seq;
- Descrever o perfil de expressão de miRNA do fígado de indivíduos controles sem dengue por miRNA-Seq;
- Verificar a influência da idade na expressão de miRNAs e como fator confundidor na análise comparativa de dengue e controle;
- Caracterizar a expressão diferencial de miRNA nos tecidos de dengue grave e de controles;
- Predizer os genes alvo dos miRNAs descritos associados;
2 MATERIAL E MÉTODO
2.1 Amostras
As amostras de de tecido hepático de pacientes com DHF (n=10) fixadas em parafina foram selecionadas dos arquivos do Departamento de Patologia do Hospital Guilherme Álvaro, município de Santos, São Paulo, Brasil. As espécies de tecido hepático foram obtidas durante viscerotomia de pacientes que faleceram por DHF durante surto no ano de 2010, todos procedimentos seguiram os padrões do Comitê de Ética Institucional (Pagliari et al. 2016). A idade dos pacientes variou de 19 a 62 anos com média de 39,5 anos distribuídos em 7 mulheres e 3 homens.
O diagnóstico de dengue foi confirmado por correlação entre dados clínicos, diagnóstico sorológico (anticorpos IgM e antígenos NS1 com detecção por ELISA) e exame histológico por coloração hematoxilina-eosina com detecção de antígenos virais através de ensaio imunohistoquímico (Pagliari et al. 2016). Esses indivíduos não apresentavam nenhuma comorbidade.
Os controles (n = 5) incluíram espécies de tecido hepático fixado em parafina que tiveram como causa de óbito infarto agudo do miocárdio, faleceram sem nenhuma infecção ou danos hepáticos, confirmados histologicamente (dados não apresentados). A idade média do grupo foi de 55 anos, com 1 mulher e 4 homens. Todas coletas de amostras teciduais foram realizadas em até 12 horas após o momento da morte.
2.2 Extração de miRNA e Sequenciamento
Os pequenos RNAs, incluindo microRNAs, das amostras foram extraídos de cortes de 5µm dos blocos de tecidos, utilizando o High Pure miRNA Isolation Kit (Roche 454 Life Sciences, Branford, CT, USA). Foi realizada uma incubação de 16 horas com proteinase K, seguida da extração conforme o protocolo do fabricante. A qualidade e quantidade de RNA extraído foi verificada usando respectivamente o Qubit 2.0 Fluorômetro (Thermo Scientific, Waltham, MA, USA) e Small RNA Analysis Kits no Agilent 2100 Bioanalyzer (Agilent Technologies, Santa Clara, CA, USA).
Diego, CA, USA), na primeira etapa é realizada a ligação dos adaptadores às extremidades 5’e 3’ dos fragmentos de RNA, em seguida a transcrição reversa por PCR (RT-PCR), para fragmentos de cDNA formados do RNA inicial mais os adaptadores 5’e 3’, essa etapa também enriquece a amostra selecionando as moléculas ligadas a adaptadores em ambas terminações. A biblioteca foi purificada por eletroforese em PAGE 6% e eluída em água ultrapura; as etapas seguintes foram a validação da biblioteca no Agilent 2100 Bioanalyzer (confirmação do tamanho, pureza e concentração da biblioteca) e normalização da concentração para 2nM. As bibliotecas geradas foram então agrupadas no mesmo volume e sequenciadas em duas corridas no Illumina MiSeq System (Illumina Inc., San Diego, CA, USA).
2.3 Análises dos Dados de Sequenciamento
Após o sequenciamento, as imagens geradas no instrumento foram analisadas e o primeiro processamento foi realizado no próprio sequenciador, o basecalling, efetuado pelo Off-Line Basecaller (OLB v1.8.0, Illumia Inc., San Diego, CA, USA). As leituras resultantes foram submetidas aos seguintes processos: (1) FastQC (Andrews, 2010. Disponível em: http://www.bioinformatics.babraham.ac.uk/ projects/fastqc) usado para visualizar os parâmetros das leituras do sequenciamento antes e depois dos tratamentos aplicados; (2) remoção de adaptadores 3’ e sequências contendo apenas N executado com o FastXToll Kit (http://hannonlab.cshl.edu/ fastx_toolkit/index.html); (3) as sequencias redundantes foram agrupadas e as leituras menores que 17nt eliminadas no mapper, script em Perl incluso no miRDeep2 (Friedländer et al. 2012), além disso o mapper realizou o alinhamentos das leituras contra o genoma humano v.hg19.
2.4 Análise de miRNAs Diferencialmente Expressos
Para comparar a expressão de miRNA nos grupos Controle e Pacientes DHF, foram utilizados os resultados de contagens gerados pelo miRDeep2, utilizando o pacote EdgeR - Bioconductor (Robinson et al. 2009), o fator de normalização por amostra foi obtido e as contagens foram normalizadas em counts per million (CPM), em seguida executada a análise de expressão diferencial (Differencial Expression – DE), foram aplicados os testes estatísticos Exato de Fisher e GLM (General Linear Model), com correção FDR (False Discovery Rate) usada para ajustar o valor de p. Os miRNAs com contagem abaixo de 5 em todas amostras foram excluídos nesse processamento. Os miRNA diferencialmente expressos foram determinados de acordo com o critério de fold change (FC) ≥ 2 associado à determinação de significância pelo valor FDR < 0,05.
2.5 Predição de Alvos dos miRNAs
Os genes alvo em potencial dos miRNAs diferencialmente expressos foram preditos com algoritmos de predição de alvos que usam três abordagens diferentes: (1) TargetScan (disponível em www.targetscan.org) método computacional, critério context++ score < -0.2; (2) miRDB (disponível em www.miRDB.org) que utiliza o método de inteligência artificial com critério de seleção para score > 80; e (3) miRTarBase (disponível em www.miRTarBase.mbc.nctu.edu.tw/), um banco de dados curado da literatura baseado em evidência experimental, com critério de seleção de que o alvo tenha sido identificado por no mínimo dois métodos diferentes.
2.6 Análise Funcional
3 RESULTADOS
3.1 Sequenciamento
A quantificação de miRNAs extraídos dos cortes de parafina foi em média 67ng/uL de DHF e 138ng/uL de amostras controle, devido já estar enriquecido apenas com os pequenos RNAs, essa quantidade de miRNA foi suficiente para o preparo da biblioteca, sem necessidade de enriquecimento adicional.
As 15 bibliotecas sequenciadas geraram total de 77.367.198 leituras em três corridas no MiSeq (Illumina), cada biblioteca gerou uma média de 5.157.813 leituras, todas com qualidade média Phred Score Q37. Após o tratamento dos dados brutos com remoção de adaptadores e exclusão das sequências com N, o número médio de leituras por amostra passou para 2.724.497, referente ao conjunto de leituras adequadas para os processamentos seguintes. Essa quantidade corresponde a média de 53,1% dos dados brutos sequenciados; em todas amostras o tamanho das leituras ficou em 22nt, coerente com o tamanho referente à maioria dos microRNAs maduros (Tabela 1).
Tabela 1. Dados clínicos e dos resultados dos sequenciamentos por amostra. IAM: infarto agudo do miocárdio; Dhf: dengue hemorrágica
Amostra_ID
Dados Clínicos Dados Sequenciamento
Idade Sexo Causa de óbito Leituras
brutas
Leituras filtradas
Leituras utilizáveis (%)
Qualidade média
Controle-63 44 M IAM, aterosclerose 1.565.618 724.999 46,3 37
Controle-81 48 M Edema agudo de pulmão, IAM, aterosclerose sistêmica 4.212.073 2.793.822 66,3 37
Controle-94 56 M IAM, aterosclerose 6.192.749 5.263.821 85,0 37
Controle-13 58 M Nulo 1.498.026 938.826 62,7 37
Controle-14 73 F Nulo 5.280.226 3.885.399 73,6 37
Dhf-140 19 F Derrame pleural, ascite, edema cerebral 5.086.952 3.627.180 71,3 37 Dhf-173 52 M Derrame pleural, hepatite transinfecciosa 7.644.988 3.787.025 49,5 37 Dhf-198 52 F Derrame pleural e peritoneal, hepatite transinfecciosa, hematêmese 3.001.348 864.836 28,8 37
Dhf-236 41 F Derrame pleural, ascite 6.079.568 2.349.387 38,6 37
Dhf-253 25 F Hemorragia esplênica, hepatite aguda 5.464.911 2.514.411 46,0 37
Dhf-255 62 M Esplenomegalia 7.225.149 3.923.213 54,3 37
3.2 Identificação de miRNAs expressos
Os dados processados foram mapeados de acordo com o banco de dados miRBase v21
humano (contendo 2588 miRNAs maduros e 1881 precursores hairpin). Utilizando o
mapper.pl, realizamos o mapeamento com o genoma humano hg19_UCSC e a seleção de
miRNAs de tamanho entre 18 a 26nt, as amostras tiveram em média 0,88% de miRNAs
mapeados dentro dos dados filtrados gerados. Utilizando miRDeep2, foi identificado o total de
1129 miRNAs maduros descritos no banco de dados e mais 52 alinhados com banco de hairpin,
entretanto todos tiveram menos que 100 leituras mapeadas, portanto neste trabalho não será
realizada essa abordagem. Os 30 miRNAs mais expressos em cada grupo são detalhados na
Tabela 2, os valores de expressão estão normalizados por CPM (counts per million) e
apresentam a média aritmética da expressão nos dois grupos. Observamos que o miR-122-5p,
específico do tecido hepático, tem a expressão extremamente elevada nas amostras sem dengue
e não aparece na lista dos mais expressos (top) 30 de DHF, enquanto o mais expresso no grupo
DHF, o miR-143-3p ainda aparece na lista de Controle na quinta posição, com valores de CPM
aparentemente muito semelhantes (CPM = 93.950,57 e 96.662,51, respectivamente). Entre
outras diferenças que merecem ser ressaltadas, estão os miRNAs miR-10b-5p e 146a-5p
aparecendo entre os top expressos nos Controles e os miR-126-5p e 133a-5p entre os top
expressos em DHF. Entre os outros listados, podemos identificar grande semelhança em quase
todas posições, como miR-192-5p, 22-3p, 10a-5p, 30a-5p, entre os 7 mais expressos em ambos
grupos. Entre os mais expressos podemos observar o grande número de miRNAs 5p
(anteriormente identificados com *), a sequência complementar à sequência guia 3p, que é a
canônica em regulação da expressão, hoje sabe-se que as sequencias 5p também podem ser
funcionais e cada vez mais os trabalhos tem identificado e analisado seus efeitos celulares.
Não foi realizado nenhum outro filtro prévio para exclusão de leituras, com alinhamento
contra bancos de dados de outras espécies de RNA (Rfam e piRNABank, por exemplo) ou
contra o genoma humano, o objetivo foi manter o máximo de sequencias disponíveis para
identificação com os miRNAs maduros já descritos, já que neste estudo não foi realizada a
Tabela 2. Lista dos 30 microRNAs mais expressos nos grupos de amostras de tecido hepático de controles e dengue hemorrágica (DHF) com respectivos valores de média de expressão por grupo, normalizados em counts per million (CPM).
Ranking miRNA Controle
(média CPM) miRNA
DHF (média CPM) 1 hsa-miR-122-5p* 225797,54 hsa-miR-143-3p 93950,57 2 hsa-miR-192-5p 142361,14 hsa-miR-192-5p 60231,03 3 hsa-miR-10b-5p* 134833,23 hsa-miR-27b-3p 42784,48 4 hsa-miR-22-3p 99530,51 hsa-miR-22-3p 42661,72 5 hsa-miR-143-3p 96662,53 hsa-miR-21-5p 37702,97 6 hsa-miR-10a-5p 87218,63 hsa-miR-10a-5p 26870,72 7 hsa-miR-30a-5p 65286,96 hsa-miR-30a-5p 23868,67 8 hsa-miR-26a-5p 46657,54 hsa-miR-126-5p* 22629,93 9 hsa-miR-27b-3p 39069,49 hsa-miR-26a-5p 22190,67 10 hsa-miR-148a-3p 32382,40 hsa-miR-451a 18251,66 11 hsa-miR-486-5p 29730,49 hsa-miR-486-5p 16815,22 12 hsa-miR-146b-5p 26265,61 hsa-let-7f-5p 12382,60 13 hsa-miR-21-5p 17553,78 hsa-miR-181a-5p 11090,36 14 hsa-miR-30e-5p 16693,45 hsa-let-7i-5p 10764,92 15 hsa-miR-451a 13438,83 hsa-miR-148a-3p 10506,86 16 hsa-let-7f-5p 13017,50 hsa-miR-30e-5p 9277,09 17 hsa-miR-30d-5p 12961,10 hsa-miR-16-5p 8057,48 18 hsa-miR-378a-3p 9132,45 hsa-miR-146b-5p 7199,33 19 hsa-miR-92a-3p 8745,08 hsa-miR-101-3p 6530,05 20 hsa-miR-146a-5p* 8548,37 hsa-miR-133a-3p* 6351,99 21 hsa-let-7i-5p 8317,40 hsa-miR-30d-5p 6051,11 22 hsa-let-7a-5p 7247,17 hsa-miR-186-5p 5738,95 23 hsa-miR-92a-3p 7076,30 hsa-miR-26b-5p 5689,11 24 hsa-miR-26b-5p 6166,08 hsa-miR-92a-3p 5455,07 25 hsa-miR-181a-5p 5357,57 hsa-let-7a-5p 5146,89 26 hsa-let-7g-5p 4898,23 hsa-miR-92a-3p 4914,85 27 hsa-miR-186-5p 4652,75 hsa-miR-126-3p 4757,55 28 hsa-miR-16-5p 4275,93 hsa-let-7g-5p 4154,16 29 hsa-miR-100-5p 3979,56 hsa-miR-142-5p 3984,70 30 hsa-miR-99a-5p 3712,07 hsa-miR-141-3p 3859,45 * miRNAs que estão diferencialmente expressos (análise detalhada a seguir).
3.3 Análise de Expressão Diferencial: DHF e Controles
A partir dos resultados de contagem de leituras gerados pelo miRDeep2, foi realizada a
análise de expressão diferencial com pacote edgeR, para identificar se as diferenças na
expressão de miRNAs entre os dois grupos seriam estatisticamente significantes, nessa etapa,
foram excluídos miRNAs identificados que tiveram alinhamento de menos que 5 leituras.
As primeiras análises foram executadas entre todas amostras juntas com dez DHF e
cinco controles, essa combinação de amostras já indicava um problema de subestruturamento
devido a grande diferença de idades no grupo DHF (de 19 a 62 anos) em contraste com a maior
diferenças significantes nos níveis de expressão, o problema de subestruturamento foi
confirmado na análise de MDS (Multidimensional scaling) bidimensional (Figura Suplementar
1), que não foi capaz de agrupar as amostras de nenhuma forma, seja pela idade ou pela
condição de óbito.
A mesma análise foi repetida, excluindo apenas os quatro indivíduos com idade menor
que 30 anos (Dhf_140, 253, 277, 884), o gráfico da análise MDS demonstrou um agrupamento
satisfatório, dividindo as amostras pela condição de óbito, controles e dengue hemorrágica.
Entretanto, uma das amostras de DHF, Dhf_299 destaca-se dos dois grupos formados (Figura
Suplementar 2), foi identificada nessa amostra uma importante característica que tornou seus
resultados dignos de dúvida, o fato do rendimento bruto do sequenciamento ter sido igual a
6.633.703 leituras e após o filtro, apenas 10% dos dados se mostraram úteis, com 89,2% de
sequencias formadas apenas por N, essa amostra teve 717.805 leituras analisadas nesse teste,
foi o pior sequenciamento entre todas 15 amostras e dessa forma, a opção foi remover a amostra
Dhf_299 devido aparente falha técnica.
Trabalhando com dez amostras de fígado, sendo cinco replicatas de controles e cinco
DHF, foi possível identificar um grupo de 38 miRNAs expressos diferencialmente,
considerando o valor de p (Tabela 3). Ao aplicar o ajuste do valor de p, os resultados de FDR
se mantém significantes em oito microRNAs: 133a-3p, 122-5p, 10b-5p,
miR-204-5p, miR-126-5p, miR-148a-5p, miR-146a-5p e miR-423-5p.
Entre esses oito miRNAs, dois estão regulados positivamente no fígado de DHF: o
primeiro, hsa-miR-133a-3p apresentou logFC = 6,77 e FDR < 0,00000001, valores totais de
CPM estão mais de 100 vezes aumentados no tecido hepático de dengue. Apesar desse aumento,
a significância encontrada é certamente devido a uma única amostra, a Dhf_255, que teve
expressão de 31.503,26, enquanto a segunda amostra com maior expressão desse grupo teve
CPM de 96,63. Independente disso, esse é um miRNA que merece atenção devido sua relação
já descrita com doenças infecciosas incluindo dengue e diversos cânceres.
O segundo superexpresso em tecido com DHF, o hsa-miR-126-5p, apresenta CPM mais
homogêneo no grupo DHF (logFC = 3,09; FDR = 0,00675) e está aumentado quase nove vezes
no fígado de dengue comparado com controle (Figura 12). Embora tenha um valor de p mais
miRNA ID logFC logCPM Teste GLM Controles (Expressão CPM) Dengue Hemorrágica (Expressão CPM)
PValue FDR Controle
63 Controle 81 Controle 94 Controle 13 Controle 14
Dhf_173 Dhf_198 Dhf_236 Dhf_255 Dhf_347
Figura 12. Expressão de hsa-miR-126-5p nos tecidos hepáticos dos grupos de dengue hemorrágica (Dhf) e controle. Mostrando evidente regulação positiva no grupo de Dhf (logFC = 3,09; FDR = 0,00675).
A maioria dos miRNAs detectados estão sub-expressos na dengue em relação ao
controle, entre eles está o hsa-miR-122-5p que também é a molécula com maior nível de
expressão em todo grupo controle (logFC = -6,59; FDR < 0,00000001), com média de CPM de
225.797,2 no grupo controle e 2.336,4 no grupo DHF, ou seja, mais de 100 vezes menos
expresso no grupo de dengue (Figura 13).
O miR-10b-5p é o segundo com maior nível de expressão entre o grupo controle,
diminuído quase 100 vezes na DHF, seguido do miR-146a-5p, ambos apresentam valores de
CPM altos e com logFC = -6,1663 (FDR < 0,00000001) e logFC = -2,6476 (FDR = 0,0387)
respectivamente (Figura 14).
Os miRNAs hsa-miR-204-5p, hsa-miR-148a-5p e hsa-miR-423-5p possuem CPMs
mais baixos em relação aos outros já descritos, mais ainda assim apresentam valores
significantes nos testes estatísticos e logFC que permitem afirmar sua associação não aleatória
com a condição estudada, resultados são respectivamente: logFC = -5,6317 e FDR < 0,000001;
Figura 13. Diferença na expressão do hsa-miR-122-5p entre grupos amostrais, evidenciando a diminuição no grupo de tecido hepático com dengue hemorrágica. Valores de expressão em log(CPM), logFC = -6,59; FDR < 0,00000001.
De forma geral, foram encontrados mais miRNAs sub-expressos no grupo DHF do que
superexpressos, de acordo com a análise de Heatmap, realizada com os valores de CPM por
amostra/por miRNA, podemos visualizar a formação de dois grupos distintos a partir dos dados
de expressão de miRNAs (Figura 15). Onde também pode ser observado a maior expressão dos
miRNAs citados anteriormente na região inferior da figura.
Essa apresentação evidencia outra discrepância na amostra Controle_63, que se mostra
com expressão claramente diferente das outras replicatas, aparecendo como um grupo externo
nos clados gerados; para resolver essa questão, foi realizada uma nova análise excluindo essa
amostra e todas outras que tiveram baixo rendimento no sequenciamento, e não foi identificada
qualquer alteração relevante na lista dos miRNAs significantes ou mesmo nos valores de FC,
apenas os valores de p e FDR tiveram um discreto aumento, provavelmente devido diminuição
do poder estatístico causado pelo menor número de replicatas, portanto essa amostra foi
mantida em todas análises.
Na análise MDS, o gráfico é construído com base no logFC (Figura 16), explicita a
dispersão de todas amostras na escala multidimensional (MDS), neste caso, bidimensional, e
evidencia a separação das amostras em dois grupos, o de DHF mostra-se bem consistente, com
uma proximidade maior entre si do que as amostras controle, que se apresenta mais disperso
denotando a heterogeneidade nesse grupo, apesar disso, é muito clara a separação entre as
amostras DHF e controle. Após a remoção da amostra Dhf_299, foi possível observar o
aumento na resolução do agrupamento gerado, produzindo essa diferença nas dispersões das
amostras no MDS com e sem a amostra Dhf_299 com problemas técnicos (Figura Sup. 4 e
É interessante observar que duas das amostras de DHF possuem uma maior proximidade
entre si na análise de MDS (Figura 16), que são Dhf_255 e Dhf_347. Foi possível identificar
uma variável comum entre essas amostras, ambas apresentaram acometimento esplênico
(esplenomegalia e congestão esplênica), em contraste com as outras amostras que não
apresentaram essa condição como causa de óbito.
Figura 16. Análise MDSPlot, bidimensional com base nos valores de logFC (fold change), destacando o agrupamento das amostras DHF (febre hemorrágica da dengue) separadas das amotras controle (Control).
3.4 Análise de Expressão Diferencial: DHF < 30 anos e DHF > 30 anos.
Devido às grandes diferenças nas idades dos indivíduos do grupo de dengue, foram
realizadas as análises de expressão diferencial dentro do grupo DHF, a fim de identificar qual
a extensão da influência e a relevância da idade na expressão dos miRNAs. Estes grupos foram
divididos entre menores que 30 anos, chamados “< 30” e grupos de indivíduos com mais de 30
anos, chamados “>30”, com o teste GLM foram identificados cinco miRNAs com diferença
(logFC = 7,6; FDR < 0,0000001) e outros quatro superexpressos no grupo < 30 anos:
miR-122-5p (logFC = -6,982; FDR < 0,0000001), miR-10b-miR-122-5p (logFC = -6,474; FDR < 0,0000001),
miR-204-5p (logFC = -5,896; FDR < 0,0000001) , miR-146a-5p (logFC = -2,578; FDR =
0,0185), todos estes miRNAs também são os diferencialmente expressos no grupo DHF em
relação ao controle, mas com uma diferença ainda mais acentuada entre estes grupos de idades
diferentes, que pode ser observada na diminuição dos valores FDR (Tabela Suplementar 1). Na
análise de MDS, ficou evidente o agrupamento das amostras >30, separadas do grupo de
amostras < 30 (Figura 17), entre estas se observa a amostra Dhf_884 bastante destacada dos
dois grupos, as variáveis que podem estar ocasionando essa separação podem ser o sexo
masculino e o número e diversidade de sintomas apresentados no atestado de óbito ser maior
que das outras.
Figura 17. Análise de MDS entre os grupos de dengue hemorrágica de indivíduos com menos de 30 anos (preto) e indivíduos com mais de 30 anos (vermelho).
A análise de Heatmap (Figura 18) demonstra os mesmos resultados obtidos por MDS,
Dhf_277 e Dhf_884 e no outro clado, as amostras com mais de 30 anos: Dhf_173, Dhf_198,
Dhf_255, Dhf_236 e Dhf_347.
Os microRNAs que aparecem repetidos na imagem são referentes a isoformas, ou seja,
sequencias similares provenientes do mesmo precursor pri-miRNA e com poucas substituições
de bases na região seed.
Figura 18. Análise de Heatmap baseada nos valores de CPM, mostrando os miRNAs diferencialmente expressos nos grupos de dengue hemorrágica (Dhf) com menos de 30 anos (Dhf_140, Dhf_253, Dhf_277 e Dhf_884) e mais de 30 anos (Dhf_173, Dhf_198, Dhf_255, Dhf_236 e Dhf_347).
3.5 Predição de Genes Alvo e Anotação Funcional
A predição de alvos para cada miRNA diferencialmente expresso foi realizada nas três
plataformas TargetScan, miRTarBase e miRDB, cada uma executa abordagem diferente para
predição, computacional, experimental e inteligência artificial, respectivamente. O valor limite