1
UNIVERSIDADE FEDERAL DO PARÁ NÚCLEO DE MEDICINA TROPICAL
PROGRAMA DE PÓS-GRADUAÇÃO EM MEDICINA TROPICAL
RÉGIS PILONI MAESTRI
ANALISE FILOGENÉTICA DE GENES DE PROVÁVEL ORIGEM NÃO HUMANA DE ROTAVÍRUS DO GRUPO A EM ESPÉCIMES FECAIS DE CRIANÇAS COM
GASTRENTERITE AGUDA PROVENIENTES DE BELÉM, BRASIL
2
RÉGIS PILONI MAESTRI
ANALISE FILOGENÉTICA DE GENES DE PROVÁVEL ORIGEM NÃO HUMANA DE ROTAVÍRUS DO GRUPO A EM ESPÉCIMES FECAIS DE CRIANÇAS COM
GASTRENTERITE AGUDA PROVENIENTES DE BELÉM, BRASIL
Dissertação de mestrado apresentada ao Programa de Pós-Graduação em Doenças Tropicais, do Núcleo de Medicina Tropical, da Universidade Federal do Pará, como requisito parcial para a obtenção do título de Mestre em Doenças Tropicais.
Orientadora: Profª Drª Joana D’Arc
Pereira Mascarenhas
3 Dados Internacionais de Catalogação–na-Publicação (CIP) –
Biblioteca do Núcleo de Medicina Tropical/UFPA, Belém-PA
___________________________________________________________
Maestri, Régis Piloni
Análise filogenética de genes de provável origem não humana de rotavírus do grupo A em espécimes fecais de crianças com gastrenterite aguda provenientes de Belém, Brasil / Régis Piloni Maestri; orientadora, Joana D’Arc Pereira Mascarenhas. – 2012.
Dissertação (Mestrado) – Universidade Federal do Pará. Núcleo de Medicina Tropical. Programa de Pós-Graduação
em Doenças Tropicais. Belém, 2012.
1. Gastrenterite - Crianças - Belém (PA). 2. Infecções por rotavírus – Crianças - Belém. 3. Rotavírus - Filogenia. I. Mascarenhas, Joana D’Arc Pereira, orient. II. Título.
CDD: 22. ed. 576.88
4
UNIVERSIDADE FEDERAL DO PARÁ NÚCLEO DE MEDICINA TROPICAL
PROGRAMA DE PÓS-GRADUAÇÃO EM DOENÇAS TROPICAIS
RÉGIS PILONI MAESTRI
ANALISE FILOGENÉTICA DE GENES DE PROVÁVEL ORIGEM NÃO HUMANA DE ROTAVÍRUS DO GRUPO A EM ESPÉCIMES FECAIS DE CRIANÇAS COM
GASTRENTERITE AGUDA PROVENIENTES DE BELÉM, BRASIL
Dissertação de Mestrado apresentada como requisito parcial para a obtenção do título de Mestre em Doenças Tropicais
Aprovada em:
Banca Examinadora
________________________________ Dra. Joana D’Arc Pereira Mascarenhas
Orientadora – Instituto Evandro Chagas
_________________________________ Prof. Dr. Evonnildo Costa Gonçalves - Titular
Universidade Federal do Pará, UFPA
__________________________________ Profª. Drª. Maisa Silva de Sousa - Titular
Universidade Federal do Pará, UFPA
____________________________________ Profª. Drª. Yvone Benchimol Gabbay - Titular
Instituto Evandro Chagas, MS, SVS
_____________________________________ Prof. Drª. Luana da Silva Soares - Suplente
5 AGRADECIMENTOS
A Deus, por todas as chances que Ele me proporcionou.
Aos meus pais, João Régis Dalla Maestri e Solange Piloni Maestri, que sempre acreditaram em mim e apoiaram minhas decisões e deram suporte para chegar até aqui.
Aos meus irmãos, Felipe P. Maestri, Alvino Maestri Neto e Mayra P. Maestri, que sempre estiveram e estão na mesma estrada lutando comigo.
A minha esposa, Karen Cristini Yumi Ogawa Maestri, a qual precisou de paciência para entender minha ausência.
A minha SUPER orientadora, Dra Joana D’Arc Pereira Mascarenhas, minha imensa
gratidão pela oportunidade a mim dada, os conhecimentos repassados e a paciência dedicada.
Ao Instituto Evandro Chagas na pessoa de sua diretora Drª. Elizabeth Santos.
A Seção de Virologia do IEC, representada pelo seu chefe Dr. Alexandre da Costa Linhares.
Ao Conselho Nacional de Desenvolvimento Científico e Tecnológico - CNPq pela bolsa fornecida.
A Dr José Paulo Gagliardi Leite do Laboratório de Virologia Comparada, Fiocruz –
RJ.
Ao Núcleo de Medicina Tropical da Universidade Federal do Pará pelo ensino.
A minha “mãe” Euzeni, que me ajudou como podia.
Aos amigos do laboratório de rotavírus Alessilva, Carolina, Daniel, Darivaldo, Delana, Jane Cecília, Jane Kaiano, Luana, René e Sylvia que sempre me deram suporte no desenvolvimento deste trabalho.
Aos amigos de seção de virologia, na, Darleise, Diele, Glicelia, Ivete, Ian Jefferson, Jessy, Jhones, Juliana, Kaio, Luiza, Mila, Samea.
6
“Se um problema é grande demais, não pense nele... E, se ele é pequeno demais, pra quê pensar nele?”
7
Trabalho realizado no Laboratório de Rotavírus da Seção de Virologia do Instituto Evandro Chagas (IEC), da Secretaria de Vigilância em Saúde (SVS/MS), sob a orientação da Dra. Joana
8 RESUMO
Os rotavírus (RVs) são a principal causa de gastrenterites viróticas agudas tanto em seres humanos, como em animais jovens de várias espécies, incluindo bezerros, equinos, suínos, caninos, felinos e aves. A diversidade genética dos RVs está associada a diferentes mecanismos de evolução. Nesse contexto registrem-se: mutação pontual, rearranjo genômico e reestruturação (reassortment). O objetivo do
presente estudo foi realizar a caracterização molecular dos genes que codificam para as proteínas estruturais e não-estruturais em amostras não usuais de RVs. Os espécimes clínicos selecionados para este estudo foram oriundos de projetos de pesquisa em gastrenterites virais conduzidos no Instituto Evandro Chagas e provenientes de crianças e neonato com gastrenterite por RVs. Os espécimes fecais foram submetidos à reação em cadeia mediada pela polimerase, para os genes estruturais (VP1-VP4, VP6 e VP7) e não estruturais (NSP1-NSP6), os quais foram sequenciados posteriormente. Oito amostras não usuais de RV oriundas de crianças e neonato com gastrenterite foram analisadas evidenciando a ocorrência de eventos de rearranjos entre genes provenientes de origem animal em 5/8 (62,5%) das amostras analisadas. Desta forma, o presente estudo demonstra que apesar de ser rara a transmissão de RVs entre espécies (animais – humanos), ela está ocorrendo na natureza, como o que possivelmente ocorreu nas amostras do presente estudo NB150, HSP034, HSP180, HST327 e RV10109. O estudo é pioneiro na região amazônica e reforça dados descritos anteriormente que demonstram o estreito relacionamento existente entre genes provenientes de origem humana e animal que possam representar um desafio às vacinas ora em uso introduzidas em escala progressiva nos programas nacionais de imunização.
9 ABSTRACT
Rotaviruses (RVs) are the main cause of acute viral gastroenteritis in both humans and young animals of species such as calves, horses, pigs, dogs, cats, and birds. The genetic diversity of RVs is related to a variety of evolutionary mechanisms, including point mutation, genome reassortment, and reassortment. The objective of this study was realized the molecular charaxterization of the genes that encode structural and nonstructural proteins in unusual RV strains. The clinical specimens selected for this study were obtained from children and newborns with RV gastroenteritis, who participated in research projects on viral gastroenteritis conducted at the Evandro Chagas Institute. Structural (VP1-VP4, VP6, and VP7) and
nonstructural (NSP1-NSP6) genes were amplified from stool samples by the
polymerase chain reaction and subsequently sequenced. Eight unusual RV strains isolated from children and newborns with gastroenteritis were studied. Reassortments between genes of animal origin were observed in 5/8 (62.5%) strains analyzed. These results demonstrate that, although rare, interspecies (animal-human) transmission of RVs occurs in nature, as observed in the present study in strains NB150, HSP034, HSP180, HST327, and RV10109. This study is the first of its kind conducted in the Amazon region and supports previous data showing a close relationship between genes of human and animal origin, representing a challenge to the large-scale introduction of RV vaccines in national immunization programs.
10 LISTA DE FIGURAS
Figura 1 Representação esquemática das proteínas dos capsídeos
externo, intermediário e interno da partícula viral de RVs... 22 Figura 2 Eletroferotipos de RVs demonstrando os sete grupos... 25 Figura 3 Modelo esquemático do ciclo de replicação dos RVs. As etapas de
replicação estão indicadas nas letras... 35 Figura 4 Descrição sucinta das amostras que foram objeto de análise... 43 Figura 5 Filograma representando a classificação das amostras do estudo
em diferentes genotipos G de VP7... 56 Figura 6 Filograma representando a classificação das amostras do estudo
em diferentes genotipos P[ ] de VP4... 57 Figura 7 Filograma representando a classificação das amostras do estudo
em diferentes genotipos I de VP6... 58 Figura 8 Filograma representando a classificação das amostras do estudo
em diferentes genotipos R de VP1... 59 Figura 9 Filograma representando a classificação das amostras do estudo
em diferentes genotipos C de VP2... 60 Figura 10 Filograma representando a classificação das amostras do estudo
em diferentes genotipos M de VP3... 61 Figura 11 Filograma representando a classificação das amostras do estudo
em diferentes genotipos A de NSP1... 64 Figura 12 Filograma representando a classificação das amostras do estudo
em diferentes genotipos N de NSP2... 65
Figura 13 Filograma representando a classificação das amostras do estudo
em diferentes genotipos T de NSP3...
66 Figura 14 Filograma representando a classificação das amostras do estudo
em diferentes genotipos E de NSP4... 67 Figura 15 Filograma representando a classificação das amostras do estudo
11 LISTA DE TABELAS
Tabela 1 Valores de cut-off de percentagem de identidade nucleotídica que diferem os diversos genotipos de RVs considerando os 11
segmentos genômicos... 24 Tabela 2 Distribuição do genotipo G RVs em várias espécies de animais... 28 Tabela 3 Distribuição do genotipo P[ ] RVs em várias espécies de animais... 29 Tabela 4 Características dos estudos sobre RVs realizados em Belém,
Pará, no período de 1992 a 2006, cujos espécimes fecais foram
utilizados nesta pesquisa... 45
Tabela 5 Reagentes utilizados para uma amostra, na obtenção do cDNA
por meio da transcrição reversa, na reação de RT-PCR... 47 Tabela 6 Reagentes utilizados para uma amostra, para a amplificação pela
PCR, da RT-PCR... 48 Tabela 7 Sequência dos iniciadores utilizados na RT-PCR e no
sequenciamento de nucleotídeos dos genes que codificam as
12 LISTA DE ABREVIATURAS E SIGLAS
°C Graus centígrados
µL Microlito
µM Micromolar
Aa Aminoácido
BLAST Basic local aligment search tool cDNA DNA complementar
dATP Desoxiadenina dCTP Desoxicitosina dGTP Desoxiguanina
DNA Ácido Desoxirribonucléico dNTP Desoxirinonucleotídeo
dsRNA Dupla fita de Ácido Ribonucléico dTTP Desoxitimina
EGPA Eletroforese em Gel de Poliacrilamida IEC Instituto Evandro Chagas
ME Microscópio eletrônico
mM Milimolar
NJ Neighbor-joining
NMT Núcleo de Medicina Tropical NSP Proteína não estrutural
OMS Organização Mundial de Saúde ORF Região de leitura aberta
pb Pares de Base
PCR Reação em Cadeia mediada pela Polimerase q.s.p. quantidade suficiente para
RNA Ácido Ribonucléico
RT-PCR Reação em Cadeia mediada pela Polimerase precedida de Trancrição Reversa
RVs Rotavírus
13
TA Temperatura Ambiente
14 SUMÁRIO
1. INTRODUÇÃO... 16
2. JUSTIFICATIVA... 18
3. OBJETIVOS... 19
3.1 OBJETIVO GERAL... 19
3.2. OBJETIVOS ESPECÍFICOS... 19
4 REFERENCIAL TEÓRICO... 20
4.1. GASTRENTERITE VIRAL... 20
4.2. O ROTAVÍRUS... 20
4.2.1 Histórico... 20
4.2.2. Partícula viral... 21
4.2.3. Classificação... 23
4.2.4. Genoma... 25
4.2.5. Epidemiologia Molecular dos RVs... 26
4.2.6. Proteinas Estruturais... 27
4.2.7. Proteinas Não-Estruturais... 30
4.2.8. Replicação Viral... 32
4.2.9. Mecanismo de Evolução Genetica... 34
4.2.10. Transmissão... 36
4.2.11. Patogenia... 37
4.2.12. Manifestaçoes Clínicas... 38
4.2.13. Imunização... 39
5. MATERIAL E MÉTODOS... 39
5.1. ASPECTOS ÉTICOS E DE BIOSSEGURANÇA... 39
5.2. PACIENTES E ESPÉCIMES CLÍNICOS... 40
5.3. SUSPENSÕES FECAIS... 45
5.4. EXTRAÇÃO DO GENOMA VIRAL... 45
5.5. REAÇÃO EM CADEIA MEDIADA PELA POLIMERASE PRECEDIDA DE TRANSCRIÇÃO REVERSA (RT-PCR)... 46
5.6. ELETROFORESE EM GEL DE AGAROSE... 49
15
FILOGENÉTICA... 50
6. RESULTADOS... 51
6.1. GENES ESTRUTURAIS:... 54
6.2. GENES NÃO-ESTRUTURAIS:... 62
7. DISCUSSÃO... 69
8. CONCLUSÕES... 74
9. PERSPECTIVAS... 75
REFERÊNCIAS... 76
APÊNDICE... 96
ANEXO... 135
16 1. INTRODUÇÃO
Considera-se diarreia aguda uma doença infecciosa, em geral de curso auto-limitado, que pode variar de algumas horas podendo perdurar por até 14 dias. É caracterizada pelo aumento do número de evacuações que se manifesta clinicamente por três ou mais evacuações em 24h, com fezes aquosas ou semi-sólidas, acompanhadas de febre, vômitos, cólicas e desidratação (OLIVEIRA, et al., 2007; BRASIL, 2006; MOTA et al., 1994; SOUZA et al., 2002).
A diarreia aguda pode ser ocasionada por vários patógenos, com destaque para os agentes bacterianos, virais e parasitários. Os agentes bacterianos são mais importantes nos países em desenvolvimento, com destaque para a Escherichia coli,
principalmente do grupo enteropatogênico clássico (EPEC), enquanto que os agentes virais são relevantes tanto em países desenvolvidos como em desenvolvimento, contribuindo para as elevadas taxas de hospitalização e pelos altos índices de morbi-mortalidade infantil, respectivamente (OLIVEIRA et al., 1998; 2007; PEREIRA, 2003).
Estima-se que os rotavírus (RVs) sejam responsáveis pela morte de 453 mil crianças menores de 5 anos, ou seja, de cada 260 crianças nascidas ao ano, uma morre de diarreia por RVs antes do seu quinto aniversário (ESTES E KAPIKIAN, 2007). Embora a proporção de RVs em crianças hospitalizadas com diarreia seja maior em países desenvolvidos, a maioria das mortes relacionadas aos RVs encontra-se nos países em desenvolvimento (TATE et al., 2011).
Os RVs são a principal causa de gastrenterite viral aguda tanto em seres humanos, como em animais jovens de várias espécies, incluindo bezerros, equinos, suínos, caninos, felinos e aves (ESTES E KAPIKIAN, 2007; MÜLLER E JOHNE, 2007).
A principal via de transmissão dos RVs é a fecal-oral, embora se postule a transmissão pela via respiratória, por meio de aerossóis, bem como por fômites (ESTES E KAPIKIAN, 2007; SATTAR et al., 1994; ZHENG et al., 1991).
17
ANTUNES, 2007; MILLER E MCANN, 2001; PARASHAR et al., 2003; 2006). Nos países desenvolvidos, onde as condições de saneamento e higiene são satisfatórias, os RVs se associam a ocorrências epidêmicas, embora o número de óbitos seja limitado (GLASS, 2006; GLASS E PARASHAR, 2006).
Os RVs são membros da família Reoviridae e seu genoma é constituído por
11 segmentos de RNA de dupla fita (dsRNA). Tais segmentos codificam 12 proteínas: seis estruturais (VP1, VP2, VP3, VP4, VP6 e VP7) e seis não estruturais: (NSP1, NSP2, NSP3, NSP4 e NSP5/6) (ESTES E KAPIKIAN, 2007).
A classificação do RVs do Grupo A é baseada em uma combinação binária envolvendo os genes VP4 (genotipos P) por revelar-se sensível à protease, e VP7
(genotipos G) de glicoproteína. Contudo, recentemente foi proposto por Matthijnssens et al. (2008a) uma nova classificação dos RVs com base na análise dos 11 genes virais, a fim de proporcionar um melhor entendimento acerca da função de cada proteína bem como compreender o relacionamento evolucionário entre espécies desses vírus. A análise das proteínas estruturais e não-estruturais de RVs se faz necessária principalmente no que se refere a uma possível transmissão envolvendo RVs de origem humana e animal e melhor compreensão acerca das relações genéticas e evolução desses vírus (MATTHIJNSSENS et al., 2008a).
Nos estudos conduzidos em Belém, Brasil em neonatos e crianças com diarreia foi observado similaridade com amostras de origem suína para os genes
VP4 e NSP4 (MASCARENHAS et al., 2007a, b). DA SILVA et al., (2011)
18 2. JUSTIFICATIVA
Até o momento, estudos sobre a caracterização molecular dos 11 genes ainda são escassos no mundo, principalmente no que tange a possível transmissão entre espécies envolvendo RVs de origem humana e animal (MUKHERJEE et al., 2009; SUBODH et al., 2006; VARGHESE et al., 2006).
Os resultados de vários estudos de vigilância epidemiológica tem relatado um número crescente de segmentos gênicos de origem animal, principalmente aqueles que codificam os genotipos G e P, vêm sendo identificados, em espécimes fecais humanos, em diferentes partes do mundo, fornecendo evidências de que os animais podem atuar como uma fonte de vírus para a diversificação gênica dos RVs humano (SANTOS E HOSHINO, 2005; GENTSCH et al., 2005; LEITE et al., 1996; GOUVEA E SANTOS, 1999; MASCARENHAS et al., 2002; MARTELLA et al., 2010).
Desta maneira, o conhecimento da heterogeneidade genética marcante nos 11 segmentos de RNA de dupla fita em diferentes cepas de RVs bem como evidências que dão suporte para a transmissão entre espécies, como resultado de múltiplos rearranjos, se reveste da maior importância visto que os RVs apresentam vários mecanismos de diversidade genética, levando a evolução desses vírus.
No Brasil estudos sobre os 11 genes de RVs estão sendo conduzidos no Rio de Janeiro, Vitória e Manaus (SILVA, 2009; TORT, 2009) e em amostras circulantes em Belém, Pará. Nesse sentido, a análise dos genes codificantes para as proteínas estruturais e não estruturais de RVs faz-se necessária a fim de ampliar o conhecimento sobre o papel dessas proteínas no estabelecimento da doença. Este estudo constitui a primeira pesquisa conduzida em Belém envolvendo a análise desses genes.
19 3. OBJETIVOS
3.1. OBJETIVO GERAL
Realizar a análise genética dos genes que codificam para as proteínas estruturais e não estruturais de rotavírus não usuais do grupo A em espécimes fecais de crianças e neonato com gastrenterite aguda em Belém, Pará.
3.2. OBJETIVOS ESPECÍFICOS
I. Realizar a padronização da RT-PCR dos genes VP1, VP2, VP3, NSP1, NSP2, NSP3 e NSP5/6
II. Verificar a possível quebra de barreira entre espécies;
20 4. REFERENCIAL TEÓRICO
4.1. GASTRENTERITE VIRAL
A diarreia aguda é caracterizada pelo aumento do número de evacuações, com fezes aquosas ou semi-sólidas acompanhadas de febre, vômitos e cólicas, podendo perdurar por até 14 dias. O impacto que esta doença ocasiona está relacionado principalmente aos danos à saúde da população infantil, bem como no contexto da sociedade em geral (BRASIL, 2006).
A diarreia aguda pode ser ocasionada por vários agentes, com destaque para os agentes bacterianos, virais e parasitários. Os agentes bacterianos são relativamente mais importantes nos países em desenvolvimento, com destaque para a Escherichia coli, principalmente do grupo enteropatogênico clássico (EPEC),
enquanto os agentes virais são relevantes em países desenvolvidos e naqueles em desenvolvimento, contribuindo para as elevadas taxas de hospitalização e pelos altos índices de morbi-mortalidade infantil, respectivamente (OLIVEIRA et al., 1998; 2007; PEREIRA, 2003; VRANJAC, 2004).
Os RVs são membros da família Reoviridae e seu genoma é constituído por
11 segmentos de RNA de fita dupla (dsRNA). São considerados a principal causa de gastrenterites viróticas agudas tanto em seres humanos, como em animais jovens de várias espécies, incluindo bezerros, equinos, suínos, caninos, felinos e aves (ESTES E KAPIKIAN, 2007; MÜLLER E JOHNE, 2007).
4.2. OS ROTAVÍRUS
4.2.1. Histórico
21
Em um estudo conduzido por Bishop et al. (1973) em Melbourne, Austrália, com crianças que apresentavam diarreia grave de origem não bacteriana, o RVs foi detectado pela primeira vez em finas seções do epitélio duodenal, utilizando a técnica de visualização direta por microscopia eletrônica, e concomitantemente identificado na Inglaterra por Flewett et al. (1973). Algumas de suas primeiras denominações foram Orbivirus-like (BISHOP et al., 1973), Reovirus-like agent (KAPIKIAN et al., 1974) e Duovírus (DAVIDSON et al., 1975), porém, por apresentar
na microscopia eletrônica o aspecto de uma “roda” Flewett e Woode, (1978) os denominaram de RVs.
No Brasil, a primeira detecção do RVs ocorreu no ano de 1976, em Belém, Pará, em um estudo-piloto conduzido no Hospital da Santa Casa de Misericórdia do Pará, envolvendo crianças com gastrenterite aguda sob atendimentos ambulatorial e hospitalar (LINHARES et al., 1977).
4.2.2. Partícula viral
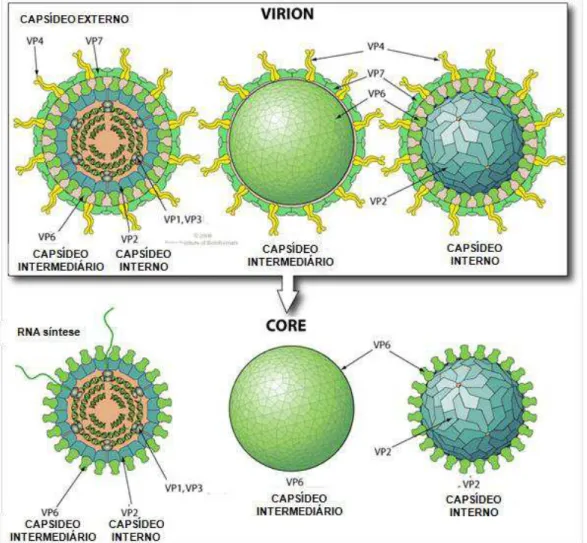
A partícula viral infecciosa (vírion) é não-envelopada, com nucleocapsídeo com simetria icosaédrica, medindo 70 nm a 90 nm de diâmetro. Estruturalmente os RVs são formados por três camadas protéicas concêntricas: capsídeo externo, capsídeo intermediário e o capsídeo interno (Figura 1), que contém genoma formado por ácido ribonucléico de dupla fita (dsRNA) de natureza segmentada (MERTENS, 2004). Associadas ao dsRNA no core, encontram-se além da enzima transcriptase, três proteínas estruturais: VP1, VP2 e VP3, codificadas pelos segmentos 1, 2 e 3, respectivamente. Essas proteínas representam em conjunto aproximadamente 18% das proteínas virais, sendo a proteína VP2, a mais abundante e a que mais interage com a proteína VP6 e com o genoma viral (JAYARAM et al., 2004). O capsídeo
intermediário é formado pela proteína VP6, codificada pelo segmento 6, sendo a mais abundante (51%), e que determina os antígenos grupo-específicos (A-G), sendo uma das mais importantes proteínas estruturais (ESTES E KAPIKIAN, 2007; JAYARAM et al., 2004).
22
por cerca de 10 a 12 nm que se projetam da matriz protéica, compreendendo 1,5% da partícula viral e desempenhado importante papel no mecanismo da penetração viral na célula. A proteína VP7 é uma Glicoproteína que compõe 30% da partícula viral. Forma a matriz do capsídeo externo e se associa à produção de anticorpos neutralizantes, determinante dos sorotipos/genotipos G.
A VP7 e VP4 atuam induzindo, separadamente, a produção de anticorpos neutralizantes, sendo que a VP4 possui propriedades adicionais como virulência e hemaglutinação (ESTES E KAPIKIAN, 2007; JAYARAM et al., 2004).
Outras cinco proteínas não-estruturais são codificadas, estando presentes apenas na célula infectada e exercendo funções vitais para a infectividade e replicação das partículas virais sendo assim denominadas: NSP1, NSP2, NSP3, NSP4 e NSP5/6 (ESTES E KAPIKIAN, 2007).
Figura 1 - Representação esquemática das proteínas dos capsídeos externo, intermediário e interno da partícula viral de RVs.
23 4.2.3. Classificação
Os RVs pertencem à família Reoviridae, gênero Rotavirus. Apresentam três
especificidades antigênicas, quais sejam, grupo, subgrupo e genotipo. Até o momento estão classificados em sete grupos sorologicamente distintos (A-G) dependendo dos diferentes epítopos presentes na proteína de capsídeo intermediário VP6. Os grupos A, B e C têm sido detectados acometendo seres humanos e animais, enquanto os demais foram identificados apenas em animais. Os integrantes do grupo A são os RVs mais amplamente dispersos, sendo predominantes na natureza e associados à diarreia no homem e em diversas espécies animais, apesar de o grupo C já ter sido detectado em surtos e casos esporádicos na região amazônica (ESTES E KAPIKIAN, 2007; GABBAY et al., 1999; 2008).
Dentro do grupo A são definidos subgrupos (SG) e sorotipos/genotipos segundo suas características sorológicas e moleculares. A proteína VP6 é responsável pela especificidade de diferentes SG, pela presença ou ausência de epítopos imunoreativos frente a determinados anticorpos monoclonais, denominados como SGI, SGII, SGI+II e SG não I e não II (ESTES E KAPIKIAN, 2007; GREENBERG et al., 1983;). O SGII é o mais frequente entre os humanos, enquanto o SGI é mais detectado entre as amostras de origem animal (DESSELBERGER, 1996; ITURRIZA-GÓMARA et al., 2002).
Os grupos designados B a G compreendem, em geral, amostras que infectam animais, com a proteína VP6 diferente daquela comum aos RVs integrantes do grupo A. Tais amostras não são detectadas por técnicas sorológicas convencionais, sendo sua identificação em geral efetuada por imunomicroscopia eletrônica, eletroforese do dsRNA em gel de poliacrilamida (EGPA) e metodologias moleculares (ESTES E KAPIKIAN, 2007).
Os sorotipos por sua vez, são determinados por anticorpos monoclonais específicos e testes de neutralização, enquanto que os genotipos são determinados por métodos de biologia molecular. Neste contexto são reconhecidos27 genotipos/sorotipos “G” e 35 diferentes genotipos “P”, com 14 sorotipos “P”
24
O tipo P é descrito com a letra P e entre colchetes o número do sorotipo e/ou genotipo correspondente, já o tipo G vem acompanhado do número (ESTES E KAPIKIAN, 2007).
MATTHIJNSSENS et al. (2008a), propos uma nova classificação genômica dos RVs, baseada nas características moleculares dos genes que codificam para as proteínas: VP7 - VP4 - VP6 - VP1 - VP2 - VP3 - NSP1 - NSP2 - NSP3 - NSP4 - NSP5/6, designando-se Gx - P[x] - Ix - Rx - Cx - Mx - Ax - Nx - Tx - Ex - Hx, respectivamente. Para cada um dos 11 segmentos genômicos são descritos valores de cut-off específicos de identidade nucleotídica, (Tabela 1), sendo esses valores
baseados na caracterização molecular e análise filogenética do genoma de 53 amostras que foram designados como modelo padrão (protótipos) para a caracterização dos RVs.
Tal classificação visa proporcionar um melhor entendimento acerca da função de cada proteína codificada por esses segmentos genômico bem como compreender o relacionamento evolucionário entre espécies desses vírus (MATTHIJNSSENS et al., 2008b).
Tabela 1 - Valores de cut-off de percentagem de identidade nucleotídica que diferem
os diversos genotipos de RVs considerando os 11 segmentos genômicos.
Gene
Valores cut-off de
identidade
nucleotídica (%)
Genotipo Designação do nome dos
genotipos
VP7 80 19G Glicoproteína
VP4 80 27P Sensível a Protease
VP6 85 11I Capsídeo Interno
VP1 83 4R RNA polimerase – RNA dependente
VP2 84 5C Proteína do Core
VP3 81 6M Metiltransferase
NSP1 79 14A Antagonista do Interferon
NSP2 85 5N NTPase
NSP3 85 7T Intensificador da Tradução
NSP4 85 11E Enterotoxina
NSP5/6 91 6H Fosfoproteína (pHosphoprptein)
25 4.2.4. Genoma
Os RVs possuem 11 segmentos de dsRNA, que codificam 12 proteínas, inseridos no core viral, reunindo aproximadamente 18.522 pares de base (pb), rico em adenina e uracila, sendo que os segmentos variam em tamanho de 667 a 3.302 pb, do menor para o maior gene. Desta maneira, cada segmento é responsável pela síntese de uma única proteína, com exceção do 11º que codifica duas proteínas (NSP5 e NSP6). Os RVs representam os únicos agentes virais conhecidos por possuir 11 segmentos de dsRNA infectando tanto mamíferos como aves (Figura 2) (ESTES E KAPIKIAN, 2007; RAMIG, 2004).
+
-Figura 2 – Eletroferotipos de RVs demonstrando os sete grupos. Fonte: adaptada de Kapikian et al. (2001).
26
foram relatados casos de diarreia associados a esses vírus. Já os dos grupos D, E, F e G infectam apenas animais. A configuração 4:2:3:2 é típica dos RVs do Grupo A, e a 5:1:3:2 caracteriza-se como a distribuição dos fragmentos genômicos observada tipicamente em aves. Os integrantes dos grupos B e D denotam padrão 4:2:2:3, e o C, por sua vez, 4:3:2:2 (ESTES E KAPIKIAN, 2007).
Os dsRNAs não são infecciosos, refletindo o fato de que as partículas virais contém suas próprias RNA polimerases dependente de RNA (RNA-dependente viral) para transcrever os segmentos individuais de RNA em RNA mensageiros (mRNA).
Os segmentos genômicos têm uma estrutura semelhante, sendo que no sentido 5’ 3’ a sequência dos genes inicia com uma guanina, seguida por região não
codificante. Logo após é encontrada uma longa “fase leitura aberta de” (ORF- open reading frame) para codificação da proteína, acompanhada de um códon de
“terminação” e novamente uma área não codificadora finalizada por citosina. Os segmentos de RNA não apresentam sinal de poliadenilação e são ricos em adenina e uracila (58% a 67%) (ESTES E KAPIKIAN, 2007).
4.2.5. Epidemiologia molecular dos RVs
Estudos têm demostrado a ampla diversidade genética de RVs circulantes no mundo (LEITE et al., 2008; SANTOS E HOSHINO, 2005; GURGEL et al., 2008). Relatos na literatura descrevem 27 genotipos/sorotipos “G” e 35 P [ ], sendo que os vários tipos G e P tendem a segregar de acordo com padrões espécie-específicos em diversas espécies animais, como demostrado nas Tabelas 2 e 3. (MATTHIJNSSENS et al., 2008b; MARTELLA et al., 2010).
No que tange às infecções nosocomiais por rotavírus no Brasil, a principal investigação ocorreu no período de 1992 a 1994 e envolveu 237 crianças atendidas em um hospital público de Belém. Os dados dessa investigação demonstraram que os Rvs foram responsáveis por cerca de 30% dos episódios diarréicos desenvolvidos no decorrer da internação, bem como 7% das infecções assintomáticas entre crianças menores de cinco anos (GUSMÃO et al., 1994).
27
mundo, sendo que alguns genotipos podem periodicamente ter maior relevância em determinadas localidades, como o G5P[8] no Brasil nos anos 90 (século XX) (SANTOS e HOSHINO, 2005; GENTSCH et al., 2005). As demais combinações, consideradas não usuais, são menos frequentes e representam uma média de 4,9% das infecções descritas em todo o mundo.
Os genotipos não usuais podem surgir a partir de infecções mistas, sugerindo uma constante troca de material genético entre as amostras de RVs humano ou entre amostras de RVs humano e animal (MASCARENHAS et al., 2002; ITURRIZA-GÓMARA et al., 2001; SANTOS e HOSHINO, 2005; MASCARENHAS et al., 2007a). Estudos envolvendo a caracterização molecular de RVs são importantes, tanto para o monitoramento de amostras circulantes quanto para identificar a presença de novos genotipos, principalmente no cenário pós implantação da vacina, o qual pode sofrer interferências a longo prazo devido a tal diversidade (RAMACHANDRAN et al., 1998; RAHMAN et al., 2005a; LEITE et al., 2008;
KHAMRIN et al., 2007; MATTHIJNSSENS et al., 2009).
4.2.6. Proteinas Estruturais
VP1. A proteína VP1, codificada pelo segmento 1 do dsRNA, é constituinte do core viral e integrante do complexo de replicação viral, atuando como uma RNA polimerase RNA dependente. Entre as proteínas do core é a única que possui uma sequência específica de reconhecimento ao RNA viral (ESTES E KAPIKIAN, 2007; MERTENS, 2004).
VP2. A proteína VP2, codificada pelo segmento 2, faz parte do complexo de replicação devido a sua capacidade de ligação com o RNA viral por meio dos resíduos N-terminais, sendo a proteína mais abundante no core (ESTES E
KAPIKIAN, 2007; JAYARAM et al., 2004).
VP3. A proteína VP3, codificada pelo segmento 3, possui atividade metiltransferase, guaniltransferase e participa do processo de transcrição viral juntamente com a proteína VP1, ligando-se ao RNA de fita simples (ESTES E KAPIKIAN, 2007).
28
28 Tabela 2 - Distribuição do genotipo G de RVs de várias espécies de animais.
● - Círculos cheios indicam que o genotipo G é epidemiologicamente relevante para a espécie.
○ - Círculos vazios indicam descrição esporádica ou relevância epidemiológica baixa do genotipo G.
Genotipo (G) 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19
Humano ● ● ● ● ○ ○ ○ ● ○ ○ ●
Suíno ○ ● ● ● ● ○ ○ ● ○ ● ○
Símio ● ○
Bovino ○ ○ ○ ● ● ● ○
Ovino / Caprino ○ ● ● ○ ● ●
Equino ● ● ○ ○ ● ●
Canino ●
Felino ●
Camundongo ●
Coelho ●
Aves ● ● ● ●
29 Tabela 3 - Distribuição do genotipo P[ ] de RVs de várias espécies de animais.
● - Círculos cheios indicam que o genotipo P[ ] é epidemiologicamente relevante para a espécie.
○ - Círculos vazios indicam descrição esporádica ou relevância epidemiológica baixa do genotipo P[ ].
Genotipo (P[ ]) 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27
Humano ○ ○ ● ○ ● ○ ● ● ● ● ● ○ ●
Suíno ○ ○ ● ● ○ ○ ● ● ● ● ●
Símio ○ ● ● ●
Bovino ● ○ ● ● ○ ● ○ ●
Ovino / Caprino ● ○ ● ○ ● ○ ● ●
Equino ○ ○ ○ ●
Canino ●
Felino ● ●
Camundongo ● ●
Coelho ● ●
Aves ●
30 VP6. Proteína viral mais abundante e a principal portadora dos determinantes antigênicos. Compõe o capsídeo interno e é formada por dois domínios, um interagindo com a proteína VP7 e outro com a VP2. Devido sua organização na partícula viral, está envolvida nas duas principais funções do vírus, quais sejam, o processo de entrada na célula hospedeira e a replicação viral, por meio de sua interação com a proteína VP2 e com as proteínas VP4 e VP7. Os resíduos de aminoáciods 296 a 259 e 305, são importantes no reconhecimento pelos anticorpos monoclonais (MAbs) de subgrupo (ESTES E KAPIKIAN, 2007; JAYARAM et al., 2004).
VP7. Glicoproteína mais imunogênica do capsídeo externo e responsável pela síntese de anticorpos neutralizantes. Acredita-se que esta proteína possa modular a atividade da VP4 no processo de adsorção e entrada do RVs na célula. Tem a capacidade de interagir com moléculas da superfície celular, uma vez que a proteína VP4 tenha iniciado o processo de adsorção (BEISNER et al., 1998; ESTES E
KAPIKIAN, 2007; JAYARAM et al., 2004; MENDEZ et al., 1996). Diferentes estudos
têm demonstrado que concentrações apropriadas de cálcio (Ca++) são necessárias para se manter a estabilidade da partícula, aparentemente pela estabilidade da glicoprotéina VP7. A VP7 apresenta uma ORF composta de 326 aminoácidos (aa) com dois códons de iniciação. Cada uma dessas regiões precede domínios hidrofóbicos designados de H1 (aa 6-23) e H2 (aa 33-44), que podem funcionar como uma sequência sinalizadora para dirigir a VP7 para o retículo endoplasmático (RE). Um terceiro códon de iniciação também está presente antes do segundo domínio hidrofóbico. Algumas amostras do vírus contêm até três sítios potenciais de glicosilação, contudo, somente dois sítios são aparentemente usados (KAPIKIAN et al., 2001).
4.2.7. Proteinas não-estruturais
31 NSP1. Proteína codificada pelo segmento 5 do dsRNA viral. Apresenta associações com o citoesqueleto celular favorecendo a ligação do vírus à célula. É a proteína viral menos conservada por apresentar elevada diversidade, mostrando maior variabilidade de sequência do que VP4 e VP7. Possui domínios relativamente conservados (zinc finger), contudo, evidenciou não ser uma proteína essencial à
replicação e virulência viral, já que não está presente em todas as variantes. Está envolvida no escape imune viral, atuando como antagonista do interferon tipo I (IFN) por diversos mecanismos (ESTES E KAPIKIAN, 2007; GRAFF et al., 2009; MERTENS, 2004).
NSP2. Proteína codificada pelo segmento 8 e altamente conservada. Esta proteína se expressa em altos níveis em células infectadas e está localizada nos viroplasmas. Sua associação com a NSP5 faz com que essas duas proteínas estejam envolvidas na replicação e “empacotamento” do RNA viral. A NSP2 possui
ainda atividade desestabilizadora de hélices de ácidos nucléicos e atividade NTPase (ESTES E KAPIKIAN, 2007; JAYARAM et al., 2004; MERTENS, 2004; TARAPOREWALA et al., 1999).
NSP3. É codificada pelo sétimo segmento do genoma viral e atua na regulação da tradução viral e previne a degradação do mRNA por nucleases celulares (JAYARAM et al., 2004).
NSP4. Codificada pelo segmento 10 é uma glicoproteína transmembrana, de localização no reticulo endoplasmático (RE), com 175 aa e massa molecular de 28 KDa, sendo a única proteína não-estrutural que não se liga ao RNA. Tem sido estudada por sua importância na morfogênese e virulência. Na morfogênese do vírus, a NSP4 atua como receptor intracelular na membrana do RE para as partículas incompletas ou double-layered particles (DLPs) durante o processo de
maturação além de ser um receptor para VP4 (ESTES E KAPIKIAN, 2007; HUANG et.al., 2004). Participa também no transporte dessas partículas através do RE e na
32
(JOHANSEN et al., 1999). Diferenças entre as sequências de NSP4 de RVs causando infecções sintomáticas e assintomáticas têm sido descritas em alguns estudos. Desta maneira a NSP4 tem sido considerada um determinante de virulência na infecção por RVs (KIRKWOOD et al., 1999; PAGER et al., 2000; ZHANG et al., 1998).
NSP5. Proteína codificada pelo décimo primeiro segmento. Possui atividade autoquinase e em células infectadas apresenta formas hipo e hiper fosforiladas (TARAPOREWALA E PATTON, 2004). Quando a NSP5 é expressa em células não infectadas a NSP2 induz a hiper fosforilação de NSP5, mostrando a interação dessas duas proteínas. Durante o processo de replicação a NSP2, NSP5 e NSP6 estão associadas na formação de viroplasmas (ESTES E KAPIKIAN, 2007; TARAPOREWALA E PATTON, 2004).
NSP6. A proteína NSP6 também é codificada pelo décimo primeiro segmento. É encontrada principalmente nos viroplasmas e interage com a NSP5, evidenciando sua participação nos processos de replicação e encapsidação do vírus. Tem sido proposto que algumas variantes dos grupos A e C de RVs não codifiquem esta proteína (ESTES E KAPIKIAN, 2007; TARAPOREWALA E PATTON, 2004).
4.2.8. Replicação viral
Após a entrada do RVs no organismo, as partículas virais migram para o intestino delgado, penetrando nas células epiteliais maduras que recobrem as microvilosidades por mecanismo ainda não plenamente elucidado (LUNDGREN E SVENSSON, 2003).
Somente partículas com triplo capsídeo conseguem aderir-se às células. A proteína VP4 tem função essencial no ciclo de replicação do vírus, incluindo a ligação ao receptor e a penetração celular. O capsídeo externo está diretamente envolvido no processo infeccioso, atuando na adesão celular, penetração da membrana e entrada na célula.
A infecciosidade dos RVs in vitro é aumentada pela presença da enzima
33
enterócitos (ESTES E KAPIKIAN, 2007; GUERRERO et al., 2010; HEWISH et al., 2000; ARIAS et al., 2004; SHAW et al., 1996).
Após a internalização, o vírion é transportado ao lisossomo onde ocorre a perda do seu revestimento externo por ação das enzimas lisossomais devido às baixas concentrações de íons cálcio, dando origem a partículas de dupla camada. Estudos têm demonstrado que concentrações apropriadas de íons Ca++ são necessárias para se manter a estabilidade da partícula viral, aparentemente pela estabilidade de VP7. A remoção dos íons Ca++ dissocia os trímeros de VP7 em monômeros, liberando a VP7 (AOKI et al., 2009).
34
maduras, são liberadas por meio da lise celular, de acordo com a Figura 3 (BALL et al., 2005; ESTES E KAPIKIAN, 2007; JAYARAM et al., 2004; RAMIG, 1997).
4.2.9. Mecanismo de evolução genética
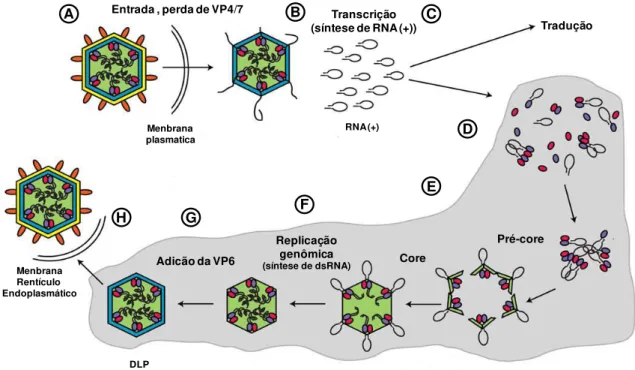
Os RVs apresentam elevada variação genética devido seu genoma ser segmentado. Desta maneira, nem sempre se observa o padrão eletroforético descrito nos protótipos virais. Um dos aspectos mais observados nos estudos de diversidade genética entre os RVs tem sido o polimorfismo eletroforético exibido pelos segmentos de dsRNA. A diversidade está relacionada com diferentes mecanismos genéticos como: a) acumulação de mutações pontuais; b) rearranjo genômico, em que dois vírus genotipicamente diferentes, após coinfecção, geram uma nova prole contendo seqüências derivadas dos vírus originais e c) recombinação intramolecular que são inserções, geralmente duplicações, ou deleções, na sequência de segmentos (ITURRIZA-GÓMARA et al., 2001; RAMIG, 1997).
35 Entrada , perda de VP4/7
Menbrana plasmatica
Transcrição (síntese de RNA (+))
Adicão da VP6
Pré-core Replicação
genômica
(síntese de dsRNA)
Tradução RNA (+) Core DLP Menbrana Rentículo Endoplasmático
A B C
D
E F
G H
A - Entrada nas células-alvo com perda da VP4 (laranja) e VP7 (amarelo);
B - VP1 (roxo) e VP3 (rosa) promovem a síntese de RNA (+) dos onze genes de RVs (elipses pretas);
C - Os RNA (+) servirão como molde para tradução bem como molde para a produção de novas fitas de RNA (-);
D - As proteinas virais e o RNA (+) se acumulam no viroplasma (cinza);
E - VP1, VP3 e o RNA (+) associam-se para formar o pré-core, seguido pela adição da VP2 (verde) para formar o core;
F - Replicação dos onze segmentos de dsRNA genômico;
G - VP6 (azul) é adicionada para amadurecer o core , formando as DLPs.
H - DLPs entram no RE, adquirem a VP4 e VP7 e por brotamento a particula viral é liberada
Figura 3 - Modelo esquemático do ciclo de replicação dos RVs. As etapas de replicação estão indicadas nas letras.
Fonte: adaptada de Guglielmi et al. (2010).
A permuta de material genético entre vírus de diferentes espécies é chamada de rearranjo, e possibilita a geração de novas variantes de RVs. Acredita-se que a
36
animais de diferentes espécies, fenômeno que é evidente em países em desenvolvimento. (MARTELLA et al., 2010; MASCARENHAS et al., 2007a; 2007b; GHOSH, et al. 2011).
Vários segmentos genômicos de RVs parecem estar envolvidos como potenciais determinantes de restrição ao hospedeiro e virulência. Contudo, o entendimento da base molecular na restrição das espécies e a virulência dos RVs, são ainda dificultados pela quantidade insignificante de dados de sequenciamento do genoma completo de amostras de RVs (MATTHIJNSSENS et al., 2006). Contudo, estudos de análise filogenética dos genes estruturais e não estruturais já realizados tem revelado um consistente padrão evolucionário entre RVs de origem humana e animal (ABE et al., 2011; ARORA E CHITAMBAR, 2011; BÁNYAI et al., 2011; MASCARENHAS et al., 2006; 2007b)
4.2.10. Transmissão
4.2.10. Transmissão
Altas concentrações de RVs são excretadas durante a evacuação, com eliminação de cerca de um trilhão de partículas virais por milímetro cúbico de espécime fecal, sendo que são necessários apenas dez vírions para desencadear uma infecção no homem (ESTES E KAPIKIAN, 2007). O período de incubação varia de um a três dias, e o pico máximo de excreção ocorre entre o terceiro e o quarto dias após o aparecimento dos primeiros sinais e sintomas. Tais parâmetros, associados à sua estabilidade físico-química, são os determinantes da elevada transmissibilidade deste vírus, principalmente onde há frequente contato pessoa a pessoa como creches e enfermarias pediátricas (ESTES E KAPIKIAN, 2007; MÜLLER E JOHNE, 2007).
37
A transmissão entre espécies tem sido registrada como sendo mais provável de ocorrer nos países em desenvolvimento, onde há influência das condições insalubres de higiene e do maior convívio dos homens com animais, principalmente bovino, suíno e aves (JAIN et al., 2001; MARTELLA et al., 2006; TEODOROFF et al., 2005).
Em Belém, em um estudo conduzido com espécimes fecais de neonatos e crianças com diarreia foi observado similaridade de RVs com genes provenientes de amostras de origem suína (MASCARENHAS et al., 2007a; 2007b).
4.2.11. Patogenia
4.2.11. Patogenia
Embora se tenha amplo conhecimento sobre a estrutura dos RVs e seus componentes, o mecanismo exato da entrada na célula ainda é pouco compreendido. A patogênese dos RVs é baseada em modelos animais, e ainda não está totalmente esclarecida, porem sabe-se que os RVs infectam os enterócitos diferenciados maduros do intestino delgado e por mecanismos desconhecidos migram para o trato gastrointestinal causando viremia (CRAWFORD et al., 2006; MATTHEW E VENKATARAM, 2010).
O efeito deletério viral é de natureza lítica, resultando num quadro diarréico de natureza osmótica devido ao efluxo de líquidos (KAPIKIAN et al., 2001). Existe ainda um componente de má-absorção da diarreia que parece estar relacionado ao dano primário do epitélio intestinal pela infecção viral e pela ação da enterotoxina viral NSP4 secretada. Tais efeitos são observados no epitélio e podem ser expressos
pelo comprometimento da “bomba” de Ca2+ nas células secretoras da cripta (BALL et
al., 1996; ESTES E KAPIKIAN, 2007; LUNDGREN E SVENSSON, 2003).
Aoki et al. (2009), demonstraram que o balanço eletrolítico de íons Ca2+ são necessários para manter a estabilidade da partícula viral e acredita-se que seja devido a estabilidade de VP7. A remoção destes íons dissocia os trímeros de VP7, liberando a VP7 do virion e iniciando a penetração na célula.
38
RVs pode incluir não somente a gastrenterite, mas também a viremia, indicando que os RVs poderiam causar uma doença sistêmica mais ampla em diferentes órgãos.
Um novo sistema in vitro foi estabelecido para o cultivo isolado de pequenas
vilosidades intestinais de camundongos recém-nascidos apresentando-se como perspectiva para um modelo útil e eficaz para estudos de infecção por RVs (GUERRERO et al., 2010).
4.2.12. Manifestaçoes clínicas
4.2.12. Manifestações clínicas
A maior incidência de infecção sintomática por RVs ocorre principalmente na faixa etária de 6 a 24 meses, e estudos demonstram que ele causa 12-71% dos episódios de gastrenterites graves nessa idade (O’RYAN et al., 2001). Adultos também podem ser infectados, embora a doença tenda a ser mais moderada (ROMERO et al., 2007).
As infecções por RVs podem ser assintomáticas ou se expressarem como gastrenterite moderada ou grave. O período de incubação é curto (um a três dias) e as manifestações clínicas mais frequentes são a tríade clássica, diarreia, febre e vômitos. A falta de intervenção terapêutica precoce pode culminar com desidratação e óbito. Os neonatos e as crianças com até quatro meses de idade são os que mais apresentam infecções assintomáticas, provavelmente devido à proteção conferida pelos anticorpos maternos (LINHARES et al.,1989; KAPIKIAN et al., 2001). A febre é registrada em mais da metade dos casos, com temperatura axilar não raro ≥ 39ºC.
Eventualmente, o quadro clínico envolve outros sintomas tais como náuseas, inapetência e dor abdominal. Outras condições clínicas atípicas podem ocorrer, incluindo as diarreias sanguinolentas. Também tem sido registrado o comprometimento respiratório, evoluindo com otite média e broncopneumonia, registrando-se detecção dos RVs, a partir das secreções respiratórias (ZAHN E MARSHALL, 2006).
39
4.2.13. Imunização
4.2.13. Imunização
Duas diferentes vacinas contra RVs vêm mostrando eficácia no cenário atual, uma preparação pentavalente de origem bovino-humana, denominada Rotateq® (Merck, West Point, Pennsilvania) e a outra vacina é a Rotarix®, representada por preparação monovalente, atenuada, de origem humana (especificidade P[8]G1), produzida pela GlaxoSmithKline Biologicals (Rixensart, Bélgica).
A vacina Rotarix® foi introduzida no calendário do Programa Nacional de Imunizações desde 2006, sendo administrada em duas doses pela via oral, aos dois e aos quatro meses de idade (BRASIL, 2006). Esta vacina apresenta proteção tanto homóloga quanto heteróloga, induzindo redução significativa na frequência de detecção de RVs em crianças com gastrenterite (GURGEL et al., 2008; LINHARES et al., 2006; SALVADOR et al.,2011).
No Brasil, a vacina Rotarix® tem apresentado proteção de aproximadamente 70% para todas as diarreias por RVs, de 86% a 98% para as formas graves e de 80% a 95%, para hospitalização por diarreia associada aos RVs, reforçando a eficácia da vacina o que influi diretamente nos indicadores de morbimortalidade por RVs (GILIO, 2009).
O Ministério da Saúde estabeleceu o indicador de cobertura vacinal para a Rotarix® em 90%. Dados do Ministério da Saúde revelam que, em 2009, da população menor de um ano, que totalizava 6.380.189 crianças, a vacina contra o RVs foi devidamente aplicada em 5.238.685 infantes, o que traduz uma cobertura vacinal de 82% (BRASIL, 2009).
40 5. MATERIAL E MÉTODOS
5.1. ASPECTOS ÉTICOS E DE BIOSSEGURANÇA
O estudo ocasionou riscos ou prejuízo mínimo para as crianças e neonatos e seus responsáveis. A presente investigação foi submetida e avaliada pelo Comitê de Ética em Pesquisa do Instituto Evandro Chagas em respeito às normas nacionais e internacionais que regulamentam tal atividade, sendo aprovado com o protocolo CEP/IEC – N° 004/2011 (ANEXO 1).
O Termo de consentimento livre e esclarecido não foi aplicado no presente estudo, devido o mesmo ser formado por projetos anteriormente desenvolvidos no IEC, portanto, tal termo foi aplicado no seu período de realização.
Em relação aos aspectos de biossegurança, no decorrer dos procedimentos foram utilizados equipamentos de proteção individual (EPIs) e todo material potencialmente contaminado foi manipulado em laboratório de segurança NB2 com o uso de cabine de contenção biológica nível 2.
5.2. PACIENTES E ESPÉCIMES CLÍNICOS
Os espécimes fecais analisados no estudo foram oriundos de projetos de pesquisa em gastrenterites virais conduzidos no Instituto Evandro Chagas (Estudo com neonatos; Hospital-Sentinela e Rede de Vigilância), descritos resumidamente na Tabela 4. A inclusão do espécime consistiu de: amostra positiva para RVs que apresentasse características genéticas com amostra de RVs animal e estar sob acondicionamento em freezer -20ºC. As amostras que não satisfizessem esses critérios, foram excluídas do estudo.
44 a. Estudo com neonatos
Essa investigação avaliou as características epidemiológicas das infecções por RVs envolvendo recém-nascidos hospitalizados em Belém, Pará. Foi realizada entre maio de 1994 a maio de 1998 e incluiu 437 neonatos com até 28 dias de idade. Um total de 614 espécimes fecais foi coletado, dos quais 51 foram associados à infecção por RVs (Linhares et al., 2002).
b. Estudo de vigilância da doença por RVs em Belém, Pará, antes e após a implementação de vacinas (Estudo Hospital-Sentinela)
Esse estudo objetivou medir a diversidade antigênica das amostras circulantes de RVs e o impacto da doença causada por esses agentes em Belém, Pará. Foi conduzido no período de maio de 1998 a maio de 2000, envolvendo crianças com diarreia aguda na faixa etária de 0 a 3 anos, atendidas a nível ambulatorial ou hospitalar em Belém, Pará. Das 834 amostras coletadas, 281 foram positivas para RVs por ELISA (MASCARENHAS et al., 2006; 2007).
c. Estudo de vigilância epidemiológica das gastrenterites por RVs no Brasil: determinação do impacto da doença (Rede Oficial de Vigilância de Rotavírus)
45 Tabela 4 - Características dos estudos sobre RVs realizados em Belém, Pará, no período de 1992 a 2006, cujos espécimes fecais foram utilizados nesta pesquisa.
Estudo, título abreviado Características
do estudo Período
Faixa etária Positivo para RV/amostras coletadas Amostras que apresentaram semelhança com RV animal Características não humana em estudos anteriores
Referência
A,Nosocomial/infecção
em comunidade Hospitalar
1992 - 1994
0–5 anos 48/290 (16%) NSC206++ NSC209++ NSC212++ Eletroferotipo de perfil aviário
Gusmão et al. (1994)
B, Projeto com
Neonatos Hospitalar
1996 - 1998
< 28 dias
51/614
(8%) NB150
++ VP4 e NSP4 origem suína
Mascarenhas, et al. (2007a)
C, Projeto Hospital Sentinela
Hospitalar / Posto de Saúde
1998 - 2000
0–3 anos 281/834 (34%) HST327+ HSP180+ HSP034+
VP4 e NSP4 origem suína
Mascarenhas, et al. (2007b)
D, Projeto Rede de
Vigilância Comunitário
Desde fevereiro de 2006 0-5 anos 66/370
(18%) RV10109
+ VP7 origem felina (Dados não publicados)
+ Community acquired diarrhea ++ Nosocomial diarrhea
5.3. SUSPENSÕES FECAIS
As suspensões fecais foram preparadas a 10% em tampão Tris-Ca++ 0,01M pH 7,2, visando à extração do RNA viral (BOOM et al., 1990). Em seguida foram homogeneizadas e centrifugadas a 3000 rpm por 10 min a 4ºC. O sobrenadante foi coletado e estocado a -20ºC.
5.4. EXTRAÇÃO DO GENOMA VIRAL
O genoma viral foi extraído a partir de 400 µL de suspensões fecais de acordo com o método descrito por Boom et al. (1990). Para cada amostra foi identificado um microtubo cônico com capacidade de 1,5 mL e adicionado 20 µL de proteinase K (20 mg/mL) e 800 µL de Tampão L6 e incubados em banho-maria a 56°C por 10 minutos. Em seguida se adicionou 200 µL de etanol absoluto gelado e 20 µL de sílica e se homogeneizou em agitador orbital.
46
banho-maria 56°C por 15 minutos, com a tampa do banho-maria e dos tubos abertas. Após a sedimentação se adicionou 60 µL de água livre de Rnase, levado ao banho-maria a 56°C por 15 minutos e posteriormente centrifuga a 14.000 rpm por 4 minutos. Em seguida foi coletado cuidadosamente o sobrenadante (30 a 40 µL) e transferido para um tubo previamente identificado e armazenado à -20°C.
5.5. REAÇÃO EM CADEIA MEDIADA PELA POLIMERASE PRECEDIDA DE TRANSCRIÇÃO REVERSA (RT-PCR)
A RT-PCR foi realizada, em duas etapas usando-se 2,5 µL do dsRNA. A primeira etapa consistiu na obtenção do DNA complementar (cDNA) por meio de transcrição reversa, e a segunda etapa consistiu na amplificação pela reação em cadeia mediada pela polimerase (PCR).
47 Tabela 5 - Sequência dos iniciadores utilizados na RT-PCR e no sequenciamento de nucleotídeos dos genes que codificam as proteínas estruturais e não-estruturais de RVs.
Iniciador Gene Sequencia Amplicon*
VP1F(+) 1
VP1 5’-GGC TAT TAA AGC TGT ACA ATG GG-3’ 686
VP1IR(-) 1 5’-TAA TCC TCA TGA GAA AAC ACT GAC-3’
VP2F(+) 1
VP2 5’-GGC TAT TAA AGG CTC AAT GGC G-3’ 686
VP2IR(-) 1 5’-CTT CAT CTT GAA ATA TAG CAA TCA C-3’
VP3F(+) 1
VP3 5’-GGC TAT TAA AGC AGT ACC AGT AG-3’ 702
VP3IR(-) 1 5’-GTA AAC ATA GAT TCA TTA CGC GGA CC-3’
Con4con3(+) 2
VP4 5’-TGG CTT CGC CAT TTT ATA GAC A-3' 876
Con4con2(-) 2 5’-ATT TCG GAC CAT TTA TAA CC–3’
VP6- F(+) 3
VP6 5’–GAC GGV GCR ACT ACA TGG T–3’ 376
VP6-R(-) 3 5’–GTC CAA TTC ATN CCT GGT GG–3`
9con1(+) 4
VP7
5’-TAT TAC ATT GCA TTT CTT TCC-3'
904
9con2(-) 4 5'-GTA TAA AAT ACT TGC CAC CA-3'
Beg9(+) 5 5´GGC TTT AAA AGA GAG AAT TTC CGT CTG G-3´
1062
End9(-) 5 5´-GGT CAC ATC ATA CAA TTC TAA TCT AAG-3´
NSP1F(+) 6
NSP1 5´-GGG CTT TTT TTT GAA AAG TC-3´ 1565
NSP1R(-) 6 5´-GGT CAC ATT TTA TGC TGC CTA-3´
K-NSP2F 7
NSP2 5´-GGC TTT TAA AGC GTC TCA GTC-3´ 1038
K-NSP2R 7 5´-GGT CAC ATA AGC GCT TTC TAT-3´
GEN-NSP3F(+) 8
NSP3 5´-GGC TTT TAA TGC TTT TCA GTG-3´ 1062
GEN-NSP3R(-) 8 5´- ACA TAA CGC CCC TAT AGC-3´
JRG30 F(+) 9
NSP4 5´-GGC TTT TAA AAG TTC TGT T-3´ 738
JRG31R(-) 9 5´-ACC ATT CCT TCC ATT AAC-3
GEN-NSP5F(+) 8
NSP5/6 5´-GGC TTT TAA AGC GCT ACA G-3´ 664
GEN-NSP5R(-) 8 5´-GGT CAC AAA ACG GGA GT-3´
* amplicon em pares de base (pb)
Fonte: 1Varghese et al. (2006); 2Gentsch et al. (1992); 3Iturriza-Gómara et al. (2002);
4Das et al. (1994); 5Gouvea et al. (1990) ; 6Nakagomi e Kaga, (1995); 7
48 Tabela 6 - Reagentes utilizados para uma amostra, na obtenção do cDNA por meio da transcrição reversa, na reação de RT-PCR.
Reagentes Concentração Volume para reação (µL)
H2O livre de DNAase / RNAase -- 17,25
dNTP* 25 mM 1
Tampão para PCR 10 X 2,5
MgCl2 50 mM 1
Par de iniciador 20 mM 2
RT** 20 U/µL 0,25
* Mistura dos quatrodesoxirribonucleotídeos trifosfato: dATP, dCTP, dGTP e dTTP. **II RNAse Reverse Transcriptase 10000U, 20 U/L
Após a obtenção do cDNA, foi realizada a reação para amplificação dos genes VP7, VP4, VP6, VP1, VP2, VP3, NSP1, NSP2, NSP3, NSP4 e NSP5/6 pela
PCR, adicionando ao cDNA obtido na primeira etapa uma mistura de reagentes (Tabela 7) nas seguintes condições: 1 ciclo de 94ºC por 5 minutos, seguido de 35 ciclos de 94ºC por 1 minuto, 45ºC por 2 minutos e 72ºC por 2 minutos e um ciclo de 72ºC por 10 minutos de extensão final. Para os genes VP4, VP6, VP7 e NSP4: 1
ciclo de 94ºC por 5 minutos, seguido de 35 ciclos de 94ºC por 30 segundos, 42ºC por 2 minutos e 72ºC por 2 minutos e um ciclo de 72ºC por 10. A RT-PCR foi padronizada para os genes VP1, VP2, VP3, NSP1, NSP2, NSP3 e NSP5/6, sendo
que para os genes VP7, VP4, VP6 e NSP4 a técnica já estava estabelecida e em
49 Tabela 7 - Reagentes utilizados para uma amostra, para a amplificação pela PCR, da RT-PCR.
Reagentes Concentração
Volume para reação (µL)
NSP1, 2,3 e 5 VP1, 2 e 3
Volume para reação (µL)
VP4, 6e 7 NSP4
H2O livre de DNAase / RNase --- 18,2 20,25
dNTP 25 mM 3 1
Tampão para PCR 10X 2,5 2,5
MgCl2 50 mM 1 1
Taq DNA Polimerase 5 U/µL 0,3 0,25
Os amplicons obtidos para os genes: VP1 (686 pb), VP2 (686 pb), VP3 (702
pb), VP4 (875 pb), VP6 (379 pb), VP7 (904 pb e 1062 pb), NSP1 (1565 pb), NSP3
(1062 pb), NSP4 (738 pb) e NSP5/6 (664 pb) foram submetidos ao sequenciamento
de nucleotídeos.
5.6. ELETROFORESE EM GEL DE AGAROSE
Os produtos obtidos na RT-PCR foram aplicados juntamente com o azul de bromofenol (corante), além do marcador de peso molecular de 123 pb. Os mesmos foram submetidos à eletroforese horizontal em gel de agarose a 1,5% em tampão tris-borato-EDTA (TBE) 1X, a 120 Volts (V) e 400 milli-Ampéres (mA) por 30 minutos. Posteriormente, o gel foi corado com brometo de etídio por 20 minutos em agitador orbital, lavado em água destilada, e visualizado e fotografado em aparelho para foto documentação de gel.
5.7. PURIFICAÇÃO E QUANTIFICAÇÃO DO cDNA
50
A quantificação do DNA, expressa em nanogramas (ng), foi realizada com o marcador de peso molecular Low DNA Mass Ladder, servindo de parâmetro para a reação de sequenciamento.
5.8. SEQUENCIAMENTO DE NUCLEOTÍDEOS E ANÁLISE FILOGENÉTICA
A reação de sequenciamento foi conduzida segundo o protocolo descrito pelo fabricante do kit Big Dye Terminator. As sequências obtidas foram alinhadas e
editadas utilizando o programa BioEdit (Version 7.0.5.2) (HALL, 1999) e comparadas com sequências de outras amostras de RVs disponíveis no banco de genes
“Genbank” (http://www.ncbi.nlm.nih.gov), a partir do programa BLAST. Os filogramas foram construídos empregando-se o programa MEGA v. 7.0.5.2., pelo método de
“Neighbor-Joining” (SAITOU e NEI, 1987), baseado nos parâmetros de Kimura-2-parametros (K2p) (Kimura, 1980), utilizando o teste de confiabilidade não paramétrico de bootstrap.
As amostras do presente estudo foram analisadas para o gene VP7 de RVs,
utilizando como parâmetro a classificação para G1, G2, G3 e G4 proposta por Jin et al. (1996), Page e Steele (2004), Bok et al. (2002) e Matthijnssens et al. (2008a), respectivamente. Para o gene VP4 foram considerados os genotipos P[4], P[6] e
P[9] e utilizou-se como parâmetro as classificações propostas por Arista et al. (2005), Martella et al. (2006) e Matthijnssens et al. (2008a), respectivamente. Os demais genes foram agrupados em genotipos conforme os valores de cut-off
51 6. RESULTADOS
52 Tabela 8 – Constelação de genotipos analisados neste estudo. O perfil genético dos genes está representado pelos códigos de cores: Humano (branco); Suíno (azul); Felino (roxo); Bovino (laranja); Origem não determinada (marrom); Sequencia não obtida devido esgotamento do espécime fecal (x). Foi utilizado para orientação gênica na tabela o protótipo de origem humana DS-1 descrito por Matthijnssens et al. (2008a).
VP7 VP4 VP6 VP1 VP2 VP3 NSP1 NSP2 NSP3 NSP4 NSP5/6 Genotipo
DS-1 G2 P[4] I2 R2 C2 M2 A2 N2 T2 E2 H2 G2-P[4]-I2-R2-C2-M2-A2-N2-T2-E2-H2
NSC206 G2 x x R2 x M2 A2 N2 T2 x H2 G2-R2-M2-A2-N2-T2-H2
NSC209 G2 P[4] x R2 C2 M2 A2 N2 T2 x H2 G2-P[4]-R2-C2-M2-A2-N2-T2-H2
NSC212 G2 P[4] x X x x x N2 T2 x H2 G2-P[4]-N2-T2-H2
53
Não foi possível a realização com sucesso da análise filogenética em 17 sequencias (Tabela 8), as quais foram insistentemente repetidas, porém, devido ao esgotamento dos espécimes fecais, visto se tratar de amostras já trabalhadas em projetos anteriores, não foi possível prosseguir com as análises.
No presente estudo foi verificada a ocorrência de eventos de rearranjo entre genes provenientes de animais em 5/8 (62,5%) das amostras, devido a alta similaridades das amostras de origem humana do presente estudo quando comparada com amostras de outras espécies.
A Tabela 8 apresenta resumidamente a relação genética das amostras analisadas, demonstrando que as amostras NSC 206, 209 e 212 foram incluídas na presente análise por apresentarem uma distribuição atípica dos clusters à
eletroforese em gel de poliacrilamida, configurando-se perfil similar ao descrito para RVs aviários. Contudo, as mesmas não evidenciaram genes sugestivos de eventuais permutas envolvendo RVs de origem animal quando analisados geneticamente os 11 genes de RVs.
Por outro lado, a análise para os outros RVs: NB150, HSP034, HSP180, HST327 e RV10109 demonstra que: a) há uma amostra (RV10109) sugerindo origem felina com a detecção do genotipo P[9]; b) duas (NB150 e HST327) que apresentam algumas características gênicas de vírus de suínos; c) outra (HSP180) aparentemente com genes oriundos de amostras bovina e suína; e d) uma quinta (HSP034) comportando algumas características gênicas relacionadas aos RVs suínos.
Na análise para os genes estruturais e não estruturais de RVs terá ênfase os genes que no presente estudo, evidenciaram similaridade com genes de origem animal.
As sequências parciais de nucleotídeos determinadas neste estudo foram depositados na base de dados GenBank (http://www.ncbi.nlm.nih.gov) e atribuído os


![Figura 6 – Filograma representando a classificação das amostras do estudo em diferentes genotipos P[ ] de VP4 1 (824 pb)](https://thumb-eu.123doks.com/thumbv2/123dok_br/16279462.716114/56.892.128.808.121.866/figura-filograma-representando-classificação-amostras-estudo-diferentes-genotipos.webp)




