Universidade Nova de Lisboa
Instituto de Higiene e Medicina Tropical
Papel das regiões C2-V3-C3 na indução de anticorpos
neutralizantes anti-VIH-2
Autor: Débora Joana Rodrigues Lopes de Almeida
Orientador: Investigador Doutor José Marcelino Coorientador: Prof. Doutor Celso Cunha
i
Em primeiro lugar, gostaria de agradecer à pessoa que tornou possível a escrita desta dissertação, por ter acreditado em mim e nas minhas capacidades desde o início, e por isso, ter permitido que eu ingressasse neste mestrado. Obrigada mãe por teres sido não só o meu suporte financeiro mas também por seres o meu suporte emocional e psicológico. Obrigada por puxares sempre por mim, por estares sempre do meu lado e por poder contar sempre contigo.
Ao meu orientador, Investigador Doutor José Marcelino, por me ter dado a oportunidade de trabalhar neste tema. Quero agradecer-lhe pelo apoio dado, pela compreensão e paciência que teve comigo. Obrigada por todos os comentários, sugestões e críticas construtivas que permitiram a elaboração desta dissertação.
Ao meu coorientador, Prof. Doutor Celso Cunha, por, primeiro que tudo, me ter aceite como aluna no mestrado e, em segundo, por me ter acolhido tão bem no laboratório, por todo apoio dado e por animar os meus dias com a sua boa disposição.
Ao João Tavanez por tudo. Obrigada por me ensinares tanto, desde a matemática mais básica aos procedimentos que me permitiram chegar ao fim de mais esta etapa. Obrigada também por me chamares fatalista e me dares tantas vezes na cabeça, incentivando-me a fazer mais e melhor, por estares sempre presente e por todo o apoio e, acima de tudo, pela tua (santa) paciência. Ah! E peço desculpa se este agradecimento tem muitas vírgulas mas há coisas que nunca mudam.
Ao David Mateus, pela amizade, pelo companheirismo, por aturar o meu mau humor no laboratório e fora dele. Obrigada por seres o Mr. Google do laboratório e por me teres ensinado tanta coisa. Obrigada por seres o meu muro das lamentações, por nunca me deixares desistir e por puxares sempre por mim.
ii
À Mariana Fernandes, por ter sido uma excelente colega nos trabalhos de grupo, por nunca desistir mesmo quando o cansaço era muito e por todas as discussões construtivas que tivemos. Obrigada por todas as vezes que me ajudaste durante o meu trabalho de bancada para que eu chegasse a horas ao meu emprego e por seres uma colega de corredor do laboratório tão boa, mesmo quando me acertavas com o armário ao saíres da tua secretária. Gostaria de poder continuar a sentar-me costas com costas contigo, em qualquer laboratório deste mundo porque além de uma excelente profissional e colega, és uma amiga espectacular.
À Ana Bolas, por ser uma amiga tão querida e uma colega de laboratório tão boa, cheia de energia positiva. Obrigada por seres tão prestável, por me teres ajudado sempre que precisei e por me animares em dias menos bons. Obrigada por me teres recebido tão bem no laboratório, a tua amizade e companheirismo foram muito importantes.
A todas as outras pessoas que faziam parte do laboratório e que com o seu companheirismo tornaram tudo melhor. Obrigada por me terem recebido tão bem, por me fazerem sentir integrada, por ouvirem as minhas dúvidas, as minhas queixas e os meus risos de alegria. Obrigada Armanda, Joana, Maria e Mafalda. Em especial, à Prof. Doutora Gabriela Santos-Gomes por me deixar fazer do seu gabinete consultório de psicologia, por me ter acolhido tão bem no laboratório e por todo o apoio. Percebo quase tanto de Leishmaniose como de VIH. Quase.
iii Rôxas que não escolheu de todo a ciência como modo de vida mas que já é quase cientista só de me ouvir falar tanto. Obrigada por toda a compreensão e apoio, por me incentivares e, acima de tudo, pela paciência com que me ouvias pensar alto, mesmo depois de teres estado tantas horas a trabalhar. Agradeço até as “piadinhas” que fazias, mesmo aquelas que eu fingia não achar piada nenhuma porque me relembraste que isto da ciência até pode ter graça. À Madalena Barbosa, pela amizade, por ser tão boa ouvinte, por todos os “força miúda!” que me disse e por ter sido uma excelente colega de trabalho. Se alguma vez te obriguei a trabalhar mais horas para que eu pudesser estar no laboratório, desculpa-me e obrigada. Um dia quando voltarmos a trabalhar juntas (como sócias, claro), eu deixo que tu descontes essas horas a mais.
v
A Síndrome da Imunodeficiência Adquirida (SIDA) é uma doença grave que se caracteriza pela destruição ou pelo desaparecimento de reações imunitárias do organismo, que permitem o surgimento de infeções oportunistas que podem levar à morte. O agente etiológico responsável pela SIDA é o Vírus da Imunodeficiência Humana (VIH) e existem dois tipos: o tipo 1 (VIH-1) e o tipo 2 (VIH-2). Quando comparados, o VIH-2 tem uma taxa de transmissão menor e uma progressão da doença mais lenta, o que faz com que os indivíduos infetados tenham um melhor prognóstico. Um dos fatores importantes nesta resposta imunológica é a presença de anticorpos neutralizantes (nAbs) e a pouca capacidade do VIH-2 na evasão a esta resposta. Os nAbs têm a capacidade de inibir a entrada do vírus nas células, através da sua ligação a regiões específicas presentes nas glicoproteínas do invólucro viral. A região V3 do domínio C2V3C3 existente na glicoproteína de superfície do invólucro do VIH-2 é um dos alvos preferenciais dos nAbs. No entanto, desconhece-se ainda de que forma as regiões adjacentes, C2 e C3, contribuem para uma resposta neutralizante. Este estudo teve como objetivos caracterizar o impacto das regiões C2, V3 e C3 na resposta de anticorpos não neutralizantes e neutralizantes na infeção por VIH-2. Para isso, recorreu-se à produção de péptidos recombinantes correspondentes a cada uma das regiões referidas anteriormente através da transformação e crescimento de bactérias competentes E. coli TOP10 com plasmídios clonados com fragmentos de ADN das regiões C2, V3 e C3, respetivamente. Após a sua produção, os péptidos recombinantes foram purificados por Fast Protein Liquid Chromatography (FPLC) e utilizados como antigénios em imunoensaios (ELISA), para estudar a reatividade de cada um dos péptidos com um painel de soros positivos para VIH-2. Observou-se maior serorreatividade contra o péptido recombinante V3 (prV3), seguido do prC3 e do prC2. Foram feitas correlações entre os valores obtidos neste estudo e valores obtidos em estudo realizado anteriormente sobre a potência da neutralização dos anticorpos (IC50) presentes em alguns dos soros utilizados. Verificou-se que não havia associação entre a neutralização e a serorreatividade contra cada região, o que pode indicar que os anticorpos neutralizantes produzidos por indivíduos infetados pelo VIH-2 não são específicos para nenhuma das respetivas regiões. No entanto, o número elevado de casos de serorreatividade pode ser indicativo de que as 3 regiões em conjunto possam ser responsáveis pela indução de anticorpos neutralizantes específicos para o domínio C2V3C3, tal como já foi observado anteriormente.
vi the appearance of opportunistic infections that can lead to death. The etiological agent responsible for AIDS is the Human Immunodeficiency Virus (HIV) and there are two types: type 1 (HIV-1) and type (HIV-2). When compared, HIV-2 has a lower transmission rate and a slower progression of the disease, which causes the infected individuals to have a better prognosis. One of the important factors in this immune response is the presence of neutralizing antibodies (nAbs) and the limited capacity of HIV-2 to escape. The nAbs have the ability to inhibit viral entry into cells, through its binding to specific regions present in the viral envelope glycoproteins. The V3 region existing in C2V3C3 domain present on surface glycoprotein of HIV-2 envelope is the main target of nAbs. However, it is still unknown how the adjacent regions, C2 and C3, contribute to a neutralizing response. This study aimed to characterize the impact of the regions C2, V3 and C3 in non-neutralizing and neutralizing antibody response in infection by HIV-2. Therefore, recombinant peptides were produced corresponding to each of the regions mentioned above by transformation and growth of competent bacteria E. coli TOP10 with plasmids
cloned with DNA fragments of the C2, V3 and C3 regions, respectively. After their production, the recombinant peptides were purified by Fast Protein Liquid Chromatography (FPLC) and used as antigens in imunoassays (ELISA) to study the reactivity of each peptide with a panel of positive sera for HIV-2. It was observed a higher serorreactivity against recombinant peptide V3 (prV3), followed by prC3 and prC2. Correlations were made between the values obtained in this study and values obtained in a previous study about the potency of neutralizing antibodies (IC50) present in some of
the used sera. It was found that there was no association between the neutralization and serorreactivity against each region, which may indicate that neutralizing antibodies produced by individuals infected with HIV-2 are not specific for any of the respective regions. However, the large number of cases of serorreactivity may be indicative that the three regions together may be responsible for the induction of neutralizing antibodies specific for C2V3C3 domain, as noted earlier.
Key-words: HIV-2 infection, C2V3C3 region, neutralizing antibodies,
vii
Agradecimentos ……… i
Resumo ………..………... v
Abstract ………...……… vi
Índice ………...………...vii
Índice de figuras ………x
Índice de tabelas ……….………..……...xii
Lista de abreviaturas ………..……....xiii
I. Introdução ………. 1
1. A SIDA e a descoberta do VIH ……….1
2. Classificação, origem e diversidade genética do VIH-2 ……….2
3. Epidemiologia ……….………..2
4. Transmissão do vírus ………...………..3
5. Patogénese do vírus ………..……….3
6. Organização estrutural e genómica do vírus ……….……….6
6.1. Invólucro viral: constituição e importância ………..8
7. Ciclo de replicação viral ………..………10
7.1. Utilização de co-recetores para a entrada do vírus na célula ………..12
7.2. Interação entre gp-SU e co-recetores ………..……….…..14
8. Respostas imunitárias ………...…………...14
8.1. Imunidade inata ……….14
8.2. Resposta celular ………...…………..15
viii
8.3.2.1. Região V3 da gp-SU ……….…….18
Enquadramento e objetivos do estudo ………...………..20
II. Material e Métodos ……….21
1. Origem e descrição do vetor plasmídico ……….……….21
2. Extração de ADN plasmídico ………..22
3. Origem e preparação dos primers ………..………..23
4. Amplificação de regiões do gene env do VIH-2 ………..……….25
5. Purificação dos produtos de PCR ……….26
6. Digestão dupla enzimática ………...……….27
7. Purificação dos produtos de digestão ……….………..28
8. Clonagem dos fragmentos de ADN obtidos por PCR no vetor ……….28
8.1. Obtenção do vetor a partir do gel de agarose ………28
8.2. Ligação dos produtos de PCR ao vetor plasmídico e transformação bacteriana………...………. 30
9. Produção de péptidos recombinantes ………..……….31
10. Purificação de péptidos recombinantes ………..32
11. Avaliação da reatividade dos anticorpos contra os péptidos recombinantes ……….32
11.1. Amostras biológicas ………32
11.2. Enzyme-Linked Immunosorbent Assay (ELISA) ……….33
11.3. Análise estatística ………34
III. Resultados ……….35
ix
1.3. Obtenção do vetor plasmídico ……….……….38
2. Purificação dos péptidos recombinantes ……….…………40
3. Avaliação das reatividades entre anticorpos e péptidos recombinantes ………...…. 42
3.1. Correlação entre serorreatividade e neutralização dos anticorpos ….………...…44
IV. Discussão e Conclusões ……….49
V. Referências Bibliográficas ………..55
x Figura 1: Evolução da infeção por VIH. ………..….5
Figura 2: Organização estrutural de uma partícula de VIH. ………..….…...7
Figura 3: Organização genómica do VIH-1 e do VIH-2. ……….…….7
Figura 4: Esquema representativo das regiões hipervariáveis (V1 – V5) e das regiões conservadas (C1 – C5) da gp120 do VIH-1. ………..……9
Figura 5: Esquema da estrutura primária da poliproteína precursora Env do VIH-1. ...9
Figura 6: Estrutura conformacional de um monómero da gp125 do VIH-2 e da gp120 do
VIH-1. ……….10
Figura 7: Esquema do ciclo de replicação viral. ……….12
Figura 8: Interação entre as glicoproteínas do VIH-1, o recetor CD4 e o co-recetor
(CXCR4 ou CCR5). ………13
Figura 9: Estrutura conformacional em trímero da gp125 e a ligação de um Fab anti-V3
do VIH-2. ………19
Figura 10: Esquema detalhado sobre o vetor pTrcHis e as suas características. ……….21
Figura 11: Sequência nucleotídica do gene env do VIH-2. ……….24 Figura 12: Representação esquemática da aplicação dos antigénios e dos soros a testar.
……….33
Figura 13: Sequência nucleotídica correspondente à região C2 do gene env(5’3’). ...35 Figura 14: Sequência nucleotídica correspondente à região V3 do gene env(5’3’). ...35 Figura 15: Sequência nucleotídica correspondente à região C3 do gene env(5’ 3’). ..35 Figura 16: Visualização dos produtos de amplificação do ADN por PCR, após eletroforese em minigel de agarose a 1%, em tampão TAE 1X e brometo de etídeo
(1:20000, stock 10%). ………..37
Figura 17: Visualização dos produtos obtidos após separação por eletroforese em minigel de agarose a 0,8%, em tampão TAE 1X e brometo de etídeo (1:20000, stock 10%). ……38
Figura 18: Visualização dos produtos resultantes da digestão dupla do ADN plasmídico dos vetores pTrcC2, pTrcV3 e pTrcC3, respetivamente, após separação por eletroforese
em minigel de agarose a 1%. ………39
Figura 19: Exemplo de um cromatograma obtido durante o processo de purificação de
xi
reatividade dos anticorpos que reagiram contra o péptido recombinante C2. …………..46
Figura 22: Relação entre a potência dos anticorpos neutralizantes heterólogos e a reatividade dos anticorpos que reagiram contra o péptido recombinante V3. …………..47
xii Tabela 1: Caracterização dos primers utilizados na amplificação de ADN por PCR. ….25 Tabela 2: Condições utilizadas durante o PCR para a amplificação do ADN das regiões
C2, V3 e C3. ………26
Tabela 3: Descrição das misturas de reação utilizadas na clonagem. ………..30
Tabela 4: Caracterização das regiões C2, V3 e C3, quanto ao tamanho do fragmento e
número de aminoácidos que originam. ………36
Tabela 5: Concentração de péptido recombinante (µg/mL) obtido nas alíquotas numeradas de 1 a 15, após quantificação no espectofotómetro NanoDrop™, com Abs=
280. ……….42
Tabela 6: Serorreatividade de cada soro testado contra todos os péptidos recombinantes
produzidos. ………..…43
Tabela 7: Potência da resposta neutralizante por anticorpos IgG, presentes em amostras
xiii aa – aminoácidos
Abs – absorvância
ADN –ácido desoxirribonucleico
ARN –ácido ribonucleico
ARNm – ácido ribonucleico mensageiro
Bregs –células B reguladoras, do inglês “regulatory B cells”
BSA –albumina de soro bovino, do inglês “bovine serum albumin” CA –cápside viral
CDC –Centro de Controlo de Doenças
CIAP –do inglês “calf intestine alkaline phosphatase”
CRF –formas recombinantes circulantes, do inglês “circulating recombinant form” DC –células dendríticas, do inglês “dendritic cells”
DNA –do inglês “deoxyribonucleic acid” DO –densidade óptica
dsDNA –do inglês “double strandeddeoxyribonucleic acid” ELISA –do inglês “enzyme-linked immunosorbent assay” EUA –Estados Unidos da América
Fab – do inglês “Fragment antigen binding”
FPLC –do inglês “fast protein liquid chromatography”
g –aceleração da gravidade g – grama
xiv HIV –do inglês “human immunodeficiency virus”
HTLV –do inglês “human T-lymphotropic virus” IC50–do inglês “fifty percent inhibitory concentrations”
IPTG –isopropil-β-D-tiogalactopiranósido
kDa –kilodalton
L – litro
LAV –do inglês “lymphadenopathy-associated virus”
mAbs –anticorpos monoclonais, do inglês “monoclonal antibodies” MCS –do inglês “multiple cloning site”
mL –mililitro
MM –marcador molecular
MPER –do inglês “membrane proximal external region” mRNA –do inglês “messenger ribonucleic acid”
nAbs –anticorpos neutralizantes, do inglês “neutralizing antibodies” NK –do inglês “natural killers”
nm –nanómetro
ON –do inglês “overnight” pb –pares de bases
PCR –reação de polimerase em cadeia, do inglês “polymerase chain reaction” pr –péptido recombinante
xv SIDA –síndrome da imunodeficiência adquirida
SIVcpz –do inglês “Simian Immunodeficiency Virus from chimpanzee”
SIVsmm –do inglês “Simian Immunodeficiency Virus from sooty mangabey monkeys” ssRNA –ácido ribonucleico de cadeia simples, do inglês “single-stranded ribonucleic acid”
TAE –tampão Tris-Acetato-EDTA
TBS –tampão Tris Salino, do inglês “Tris-Buffered Saline”
TRIM5α–do inglês“Tripartite motif protein isoform 5 alpha”
UNAIDS –do inglês “Joint United Nations Programme on HIV/AIDS” URF –forma recombinante única, do inglês “Unique Recombinant Form”
V – volt
1 I. Introdução
1. A SIDA e a descoberta do VIH
Em 1981 começaram a surgir e a serem descritos, em indivíduos homossexuais, casos de infeções oportunistas e de cancros raros que implicavam a deterioração do sistema imunitário para o seu desenvolvimento. A pneumonia causada por Pneumocystis
jirovecii (referida antigamente por Pneumocystis carinii) e o Sarcoma de Kaposi são
algumas dessas doenças (CDC, 1981). Devido ao aumento do número de indivíduos com o sistema imunitário debilitado, o Centro de Controlo de Doenças (CDC) atribuiu o nome de Síndrome da Imunodeficiência Adquirida (SIDA) à doença que inicialmente parecia só afetar homossexuais. No entanto, em 1983, começaram a ser conhecidos casos de SIDA em indivíduos que utilizavam drogas injetáveis, em mulheres por contacto sexual com homens infetados, em crianças por transmissão materna e em indivíduos imunocomprometidos, tais como os hemofílicos (CDC, 1983; Essex et al., 1983; Harris
et al., 1983; Oleske & Minnefor, 1983; Lazzarin et al., 1984).
Foi no mesmo ano de 1983 que foi isolado, a partir de uma amostra de sangue de um paciente com SIDA, por Françoise Barré-Sinoussi e por Luc Montagnier, do Instituto Pasteur em França, o vírus responsável pela SIDA, denominado primeiramente de Vírus T-linfotrópico Humano tipo III (do inglês Human T-Lymphotropic Virus type II, HTLV-III) ou Vírus Associado a Linfoadenopatia (do inglês Lymphadenopathy-Associated
Virus, LAV) e posteriormente de Vírus da Imunodeficiência Humana do tipo 1 (VIH-1
ou, do inglês Human Immunodeficiency Virus type 1, HIV-1) (Barré-Sinoussi et al., 1983; Gallo et al., 1984).
Três anos mais tarde, em 1986, doentes internados no hospital Egas Moniz em Lisboa, provenientes de África Ocidental, apresentavam sintomas semelhantes aos de SIDA. A partir de amostras recolhidas dos indivíduos doentes, foi isolado um novo tipo de retrovírus, que apresentava semelhanças morfológicas com o VIH-1. Contudo, os indivíduos apresentavam serologia negativa para o mesmo. Assim, o novo vírus foi denominado de LAV-II e também alterado posteriormente para Vírus da Imunodeficiência Humana tipo 2 (VIH-2 ou, do inglês Human Immunodeficiency Virus
2 2. Classificação, origem e diversidade genética do VIH
Ambos os tipos de VIH pertencem à família Retroviridae e à subfamília
Orthoretrovirinae, género Lentivirus. Os vírus pertencentes a este género
caracterizam-se por causarem infeções crónicas em espécies mamíferas (Sharp et al., 2011).
A origem do VIH-1 e do VIH-2 deve-se a transmissões zoonóticas sucessivas de dois vírus distintos, que causam imunodeficiência nos símios, sendo eles o Simian
Immunodeficiency Virus from chimpanzee (SIVcpz) no caso de VIH-1 e o Simian
Immunodeficiency Virus from sooty mangabey monkeys (SIVsmm) para o caso do
VIH-2 (Sharp et al., 2011; Nyamweya et al., 2013).
Com base na diversidade genética, o VIH-1 foi classificado em 4 grupos (M, N, O e P). Os vírus do grupo M são os responsáveis pela pandemia e podem ainda ser divididos em 9 subtipos (A, B, C, D, F, G, H, J e K) (Sharp et al., 2011) e 6 sub-subtipos (A1 – A4 e F1 – F2) (Barroso et al., 2011; Brun-Vézinet & Charpentier, 2013). Também já foram encontradas pelo menos 72 formas recombinantes circulantes (do inglês
Circulating Recombinant Form, CRF) (Los Alamos HIV database, 2015) e formas
recombinantes únicas (do inglês Unique Recombinant Form, URF) (Buonaguro et al., 2007).
O VIH-2 está dividido em 8 grupos genéticos (A – H). No entanto, os grupos A e B são os principais responsáveis pela epidemia, sendo o grupo A o mais prevalente (de Silva et al., 2008; Borrego & Taveira, 2013). Também já foi identificada uma CRF para o VIH-2 (Ibe et al., 2010).
3. Epidemiologia
Segundo um relatório de 2014 da Joint United Nations Programme on HIV/AIDS (UNAIDS), desde o início da pandemia em 1985 mais de 78 milhões de pessoas foram infetadas pelo VIH e 39 milhões de pessoas morreram (UNAIDS, 2014).
3
socioeconómicas a essa região de África, tais como, Portugal, França, Índia e Brasil. Uma explicação para a discrepância geográfica está nas taxas de transmissão de ambos os vírus, sendo que o VIH-1 é mais facilmente transmitido do que o VIH-2 (de Silva et al., 2008; Sharp et al., 2011).
4. Transmissão do vírus
O VIH pode transmitir-se por contacto sexual, por contacto com sangue infetado através de transfusões ou partilha/utilização de objetos cortantes ou perfurantes, por utilização de drogas injetáveis devido à partilha de seringas e, ainda, por transmissão vertical, ou seja, de mãe para filho, quer durante o parto, quer por amamentação (Cohen
et al., 2011; Shaw & Hunter 2012).
A taxa de transmissão do VIH-1 é mais elevada na fase aguda da infeção, em que a replicação viral é maior e, consequentemente, a carga viral é mais elevada (Cohen et
al., 2011). Esta correlação também pode explicar o facto de o VIH-2 ter uma taxa de
transmissão mais baixa ou quase nula no caso de transmissão vertical, já que a carga viral presente numa infeção por VIH-2 é baixa (Popper et al, 2000; Sharp et al., 2011).
A transmissão do vírus está também correlacionada com a presença de outras doenças sexualmente transmissíveis, tais como a sífilis e vários tipos de hepatites. Isto deve-se ao facto das vias de transmissão das infeções serem as mesmas, assim como as respostas imunitárias provocadas poderem potenciar o risco de coinfeções (Marschalkó
et al.,2015).
5. Patogénese do vírus
4
Mogensen et al., 2010; Moir et al., 2011). É nesta fase que os indivíduos infetados começam a sentir sintomas, normalmente muito semelhantes a uma gripe, e que os anticorpos produzidos contra o vírus podem ser detetados, 3 a 4 semanas após a infeção (Mogensen et al., 2010; Moir et al., 2011).
5
da quantidade de células T CD4+ circulantes e a quantidade de cópias de ARN viral presentes no sangue ao longo das várias fases da infeção.
Figura 1: Evolução da infeção por VIH (Adaptado de Fanales-Belasio et al., 2010). A linha a tracejado representa a quantidade de células T CD4+ circulantes e a linha contínua representa a quantidade de cópias de ARN viral presentes no sangue.
Os indivíduos infetados por VIH-2, quando comparados com os indivíduos infetados por VIH-1, apresentam uma virémia baixa ou inexistente no plasma (Popper et
al., 2000) mas também uma maior suscetibilidade ao controlo por parte do sistema
imunitário, embora a ativação imunitária seja mais reduzida, o que faz com que estes indivíduos consigam sobreviver durante mais tempo, sem surgimento de doenças oportunistas (Berry et al., 1998; Sharp et al., 2011). A carga viral mais baixa pode dever-se a diferenças na transcrição, já que as taxas de replicação do VIH-2 são mais baixas, comparativamente ao VIH-1, ainda que a carga de ADN proviral seja semelhante para ambos os vírus (Popper et al., 2000). Tudo isto, permite que a transmissibilidade do vírus do tipo 2 seja mais baixa. No entanto, os indivíduos infetados pelo VIH-2 também progridem para a fase de SIDA, ainda que a progressão ocorra de forma mais lenta (Nyamweya et al., 2013)
6
As diferenças na patogénese dos dois tipos de vírus não podem ser explicadas pelas cargas virais presentes no plasma, uma vez que indivíduos infetados por VIH-2 também evoluem para a fase de SIDA, mesmo quando a carga viral é muito baixa (Nyamweya et al., 2013). A utilização de um grande número de co-recetores pelo VIH-2 deveria representar uma patogenicidade maior, comparativamente ao VIH-1. Portanto, uma explicação pode ser a suscetibilidade do vírus ao controlo pelo sistema imunitário, que no caso do VIH-2 é maior, visto que é mais facilmente sinalizado e neutralizado devido à estrutura e conformação de algumas regiões das glicoproteínas do invólucro (Shi
et al., 2005; Nyamweya et al., 2013).
6. Organização estrutural e genómica do vírus
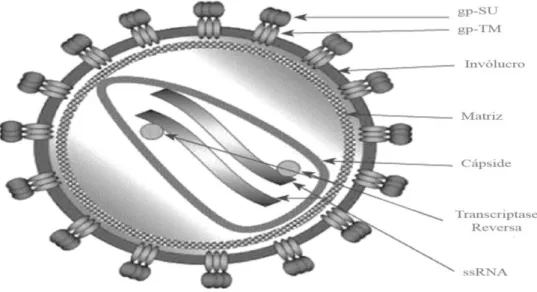
7 Figura 2: Organização estrutural de uma partícula de VIH (Adaptado de Fanales-Belasio et al., 2010).
Cada molécula de ARN tem 9 genes que codificam vários tipos de proteínas, sendo que quatro genes codificam as proteínas acessórias já anteriormente referidas, 2 codificam as proteínas de regulação tat e rev e os outros 3 genes codificam as proteínas de estrutura gag, pol e env (Turner & Summers, 1999; Freed, 2001; Benjamin et al., 2005). O gene env codifica uma poliproteína percursora, que ao ser glicolisada no complexo de Golgi e clivada pela protease do vírus, dá origem à glicoproteína do invólucro. Na figura 3 pode ser visto um esquema da organização genómica do VIH-1 e do VIH-2.
8 6.1. Invólucro viral: constituição e importância
Tal como referido anteriormente, os viriões do VIH têm na sua estrutura um invólucro, que por sua vez é constituído por gp-TM e por gp-SU. Ambos os vírus têm os dois tipos de glicoproteínas, embora sejam diferentes no peso molecular. O VIH-1 tem na sua constituição a gp-SU com 120kDa (gp120) e a gp-TM com 41kDa (gp41), enquanto o VIH-2 é constituído pela gp125 e a gp36 (Borrego & Taveira, 2013). As glicoproteínas são heterodímeros que, após maturação do virião, se encontram associadas como trímeros.
A gp-SU é constituída por cinco regiões hipervariáveis, de V1 a V5 e por cinco regiões conservadas que separam cada uma das regiões hipervariáveis e que são igualmente de C1 a C5 (Turner & Summers, 1999). As regiões hipervariáveis têm como característica a formação de loops.
No caso da gp120 do VIH-1, a região V1 e V2 estão separadas por uma ligação dissulfídica, sendo que os respetivos loops estão contidos num loop, formando assim o domínio V1/V2. Este é bastante variável em termos de tamanho do loop, assim como no número de locais de glicosilação. As regiões V3 e V4 também estão separadas por uma ligação dissulfídica. O domínio V3 mas também as regiões C2, C3 e C4 não variam muito em termos de tamanho. As regiões responsáveis pela ligação ao recetor CD4 são as regiões C1, C3 e C4, que têm em si domínios conservadas que são determinantes para a ligação, o que significa que as regiões hipervariáveis não têm um papel direto na ligação ao CD4. No entanto, a região V3 é a responsável pela fusão da membrana e pela seleção do coreceptor a utilizar, e é determinante na neutralização por anticorpos (Checkley et
al., 2011). A disposição das regiões constantes e das regiões hipervariáveis da gp120 do
9
Figura 4: Esquema representativo das regiões hipervariáveis (V1 – V5) e das regiões conservadas (C1 – C5) da gp120 do VIH-1 (adaptado de McCaffrey et al., 2004).
A gp41 é constituída por 3 domínios: um intracitoplasmático, um transmembranar e outro ectodomínio extracelular. É neste último que se encontra o péptido de fusão (Turner & Summers, 1999; Weiss, 2003; Checkley et al., 2011). É dentro do ectodomínio que se encontram os principais determinantes da fusão. Um dos mais conhecidos e estudados é a Membrane-proximal external region (MPER), constituída por 24 aminoácidos e necessária para a infetividade do vírus (Checkley et al., 2011).
A figura 5 esquematiza a estrutura primária da poliproteína precursora Env, a gp160, que após clivagem origina a gp120 e a gp41, ambas presentes no invólucro do VIH-1.
10
A gp125 do VIH-2 é igualmente constituída por 5 regiões hipervariáveis (V1/V2, V3, V4 e V5) e por 5 regiões conservadas (C1 – C5), com uma estrutura primária semelhante à estrutura do VIH-1. As regiões responsáveis pela ligação entre o vírus e o recetor CD4 são as regiões C2, C3, C4 e V4. A gp36 do VIH-2 é também constituída pelos 3 domínios referidos anteriormente (Taveira, 2014). Na figura 6 podem ser observadas as estruturas conformacionais da gp120 do VIH-1 e da gp125 do VIH-2.
Figura 6: Estrutura conformacional de um monómero da gp125 do VIH-2 e da gp120 do VIH-1 (Adaptado de Uchtenhagen et al., 2011).
É devido à interação entre as glicoproteínas do invólucro e recetores e co-recetores da célula hospedeira que a entrada do vírus acontece e todo o ciclo replicativo se dá (Borrego & Taveira, 2013). Consequentemente, a estrutura e conformação dos vários constituintes do invólucro vão ter um papel fundamental nas respostas imunitárias por parte do indivíduo.
7. Ciclo de replicação viral
11
forma específica e sequencial com o recetor CD4, que pode ser encontrado em vários tipos de células, tais como as células T ou os macrófagos. Após esta primeira ligação, a gp-SU interage também com um co-recetor, havendo mudanças a nível tridimensional gp-TM (Bour et al., 1995; Huang et al., 2005). Como consequência, uma região hidrofóbica desta glicoproteína - denominada de péptido de fusão - fica exposta e é introduzida na membrana citoplasmática da célula, levando à formação de um poro de fusão que permite a entrada da cápside do vírus (Turner & Summers, 1999; Markosyan
et al., 2009). Logo de seguida, dá-se a libertação do seu conteúdo no citoplasma da célula
12 Figura 7: Esquema do ciclo de replicação viral: primeiro dá-se a ligação, fusão e entrada da partícula viral na célula (1). Após a libertação do ARN viral, o mesmo sofre transcrição reversa, dando origem ao ADN proviral (2 e 3), para ser inserido no ADN da célula hospedeira (4). Após a transcrição o ARNm viral é transportado para fora do núcleo e é traduzido no citoplasma, dando origem às proteínas que irão constituir a nova partícula viral (5), que é libertada da célula por gemulação (6) (Adaptado de Fanales-Belasio et al.,
2010).
7.1. Utilização de co-recetores para a entrada do vírus na célula
13
Figura 8: Interação entre as glicoproteínas do VIH-1, o recetor CD4 e o co-recetor (CXCR4 ou CCR5), que permite a entrada do vírus na célula (Adaptado de Wilen et al., 2012).
Quando a infeção é feita pelo VIH-1 encontram-se, maioritariamente, variantes do vírus que utilizam o CCR5 (vírus R5) logo após a transmissão, durante a fase crónica assintomática e também em indivíduos em fase avançada de infeção (Azevedo-Pereira et
al., 2003; Santos-Costa et al., 2014). Isto pode ser um indicador de que os vírus R5
escapam melhor às respostas imunitárias ou que estes variantes conseguem infetar células que vão servir de reservatório (Santos-Costa et al., 2014). No entanto, num estado mais avançado da doença, podem surgir variantes do vírus que utilizam preferencialmente o CXCR4 (vírus X4) ou vírus que utilizem ambos os co-recetores (vírus X4R5). Esta mudança está associada a um decréscimo acentuado na contagem de células T CD4 e a uma rápida progressão da doença (Azevedo-Pereira et al., 2003; Shi et al., 2005).
No caso de infeção por VIH-2, este pode utilizar o CCR5 numa fase inicial, quando as contagens de células T CD4 ainda são elevadas, mas também pode utilizar alternativamente o CXCR4. Esta mudança foi observada em doentes cujas contagens de células T CD4 se encontravam abaixo de 200 células/µL, ou seja, em estado avançado de doença (Shi et al., 2005; Marcelino et al.; 2012).
14
APJ, ChemR23, CXCR7/RDC1, D6, BLTR e BOB (Azevedo-Pereira et al., 2003; Shi et
al., 2005; Santos-Costa et al., 2014).
Alguns isolados VIH-2 conseguem também infetar células utilizando apenas co-recetores, ou seja, sem utilizar o recetor CD4. Estes são mais facilmente neutralizados pelos anticorpos pois o local de ligação ao co-recetor está mais exposto e é uma região de ligação preferencial por parte de anticorpos neutralizantes (Thomas et al., 2003; Shi et
al., 2005)
7.2 Interação entre gp-SU e co-recetores
A utilização dos co-recetores é determinada por características presentes em certas regiões das glicoproteínas. No caso do VIH-1, a região V3 da gp-SU é a principal região responsável na seleção do co-recetor a ser utilizado (Hoffman & Doms, 1999). Quando se dá a interação entre a gp120 e o recetor CD4, a região V3 fica mais exposta, o que faz com que seja a mesma a ligar-se aos co-recetores, em particular ao CXCR4 e ao CCR5 (Gorny et al., 2004).
Relativamente ao VIH-2, estudos apontam igualmente para a influência da região V3 na escolha entre CCR5 e CXCR4 (Shi et al., 2005; Visseaux et al., 2012) mas também da região V1/V2 (Santos-Costa et al., 2014). Segundo o que foi observado por Shi e os seus colegas, a região V3 tem uma configuração de mais fácil acesso, quando comparado com o mesmo domínio no VIH-1, o que explica não só a maior sensibilidade à neutralização mas também a capacidade de utilizar co-recetores alternativos (Shi et al., 2005).
8. Respostas imunitárias
8.1. Imunidade inata
15
2010; Wilson et al., 2013). As DC mieloides e os monócitos produzem moléculas proinflamatórias, tais como as citocinas e as quimiocinas, e participam no recrutamento de células T que vão controlar a replicação viral (Cavaleiro et al., 2013).
Está provado que a resposta imune inata não atua de igual forma, dependendo se a infeção é por VIH-1 ou por VIH-2. Foi demonstrado por Nuvor e seus colegas que os níveis de citotoxicidade das células NK são significativamente mais elevadas em indivíduos infetados por VIH-2, quando a contagem de células T CD4 também é elevada. No entanto, quando há um decréscimo de células T CD4+ devido à progressão da infeção, a citotoxicidade das NK diminui (Nuvor et al., 2006).
Algumas moléculas são responsáveis nesta primeira fase de resposta pelo controlo da replicação viral. As DC exprimem uma proteína, a SAMDH1, que tem a capacidade de bloquear a replicação de alguns retrovírus, tais como o VIH-1. O mesmo não acontece quando a infeção ocorre por VIH-2, já que a sua proteína acessória Vpx vai ligar-se a SAMDH1, diminuindo assim a sua capacidade inibitória (Nyamweya et al., 2013; Zhang
et al., 2014). Outra proteína, a Tripartite motif protein isoform 5 alpha (TRIM5α), atua
como fator antiretroviral, bloqueando a infeção, antes do estabelecimento do provirus. A replicação do VIH-2 é mais facilmente controlada por esta proteína, comparativamente ao VIH-1 (Ylinen et al., 2005).
8.2. Resposta celular
A resposta celular realizada pelas células T CD4+ é diferente para cada tipo de VIH. No caso de infeção por VIH-1, a quantidade de células T CD4+ produtoras da citocina 2 e com capacidade proliferativa diminui. A diminuição da quantidade de IL-2 produzida leva a uma redução da quantidade destas células e a um aumento da apoptose. No caso de infeção crónica e assintomática por VIH-2, o número de células T CD4+ polifuncionais, produtoras de citocinas e com capacidade proliferativa é maior (Duvall et
al., 2008). De facto, a produção da citocina IL-2 é maior, o que permite uma maior
16
ocorre mais eficazmente, uma vez que a função tímica é preservada por mais tempo (Gautier et al, 2007).
A resposta imunitária celular realizada pelas células T CD8+ também se verifica e a sua capacidade proliferativa e a citotoxicidade associadas às mesmas são semelhantes, quer na infeção por VIH-1 ou por VIH-2 (Duvall et al., 2008). Estas células produzem moléculas efetoras, tais como citocinas (em especial IFN-gama e TNF-alfa) e perforinas. Contudo, a produção das últimas, assim como a citotoxicidade associada a estas células não estão associados ao controlo da virémia do VIH-2, contrariamente ao que acontece numa infeção por VIH-1. O controlo e a baixa carga viral devem-se à produção de várias citocinas e à magnitude da resposta das células T CD8+ polifuncionais (Leligdowicz et
al., 2007; Duvall et al., 2008; Silva et al., 2012).
A quantidade e o desempenho das células T CD4 e das células T CD8 podem ser influenciadas pela atividade imunossupressora que as proteínas do invólucro têm (Cavaleiro et al., 2000). Sabe-se que as proteínas do invólucro do VIH-2 vão fazer com que não haja ativação das células, através da interação com os monócitos. Assim a depleção das células T é menor e isso faz com que a progressão da doença aconteça mais lentamente (Cavaleiro et al., 2007).
A atividade e quantidade de células T é igualmente influenciada pelas células B reguladoras (do inglês regulatory B cells, Bregs). Estas conseguem controlar a proliferação das células T CD4+ e influenciam a atividade citotóxica das células T, medida pelas células T CD8+ (Siewe et al., 2014).
8.3. Resposta humoral
8.3.1. Anticorpos Neutralizantes
Os anticorpos neutralizantes (do inglês neutralizing antibodies, nAbs) são proteínas que se ligam a regiões específicas das glicoproteínas do invólucro, gp-SU ou gp-TM, e que impedem a entrada do vírus na célula.
17
vírus tem a capacidade de escapar à neutralização numa fase inicial da infeção, através da seleção de variantes resistentes (Wei et al., 2003; Bunnik et al., 2010). Shi e seus colegas concluíram também que não existe associação entre neutralização e preferência na utilização de co-recetor, embora tenham observado que a maioria dos isolados R5 eram sensíveis à neutralização autóloga. A resposta dada por nAbs heterólogos é igualmente diferente, já que é de grande amplitude numa infeção por VIH-2 (Shi et al., 2005). Estudos realizados anteriormente demonstram que vírus primários isolados de indivíduos com infeção crónica por VIH-2 são mais suscetíveis à neutralização. Esta suscetibilidade não se observa em vírus primários isolados de indivíduos infetados com VIH-1 (Kong et al., 2012; Ozkaya et al., 2012; de Silva et al., 2012). Também não foi encontrada associação entre a neutralização heteróloga e a escolha na utilização de co-recetor (Shi et al., 2005). No caso de infeção por VIH-1, nAbs heterólogos são encontrados em indivíduos considerados long-term non-progressor (Pilgrim et al, 1997). Em termos de respostas cruzadas, Weiss e os seus colegas demonstraram que os nAbs contra VIH-2 conseguem neutralizar VIH-1 mas o contrário não se observa (Weiss et al., 1988).
A sensibilidade à neutralização por anticorpos pode estar relacionada com a forma como certos epitopos são apresentados, o que depende se o vírus é R5 ou X4. Os vírus X4 são mais sensíveis à neutralização comparativamente aos vírus R5, no caso de infeção por VIH-1 (Bunnik et al., 2007). O mesmo não se verifica numa infeção por VIH-2, já que foi observado que os vírus R5 são mais sensíveis e o vírus X4 mais resistentes à neutralização (Marcelino et al., 2010). A potência e amplitude da resposta dada pelos nAbs depende do número de células T CD4+ presentes no plasma dos doentes, sendo que a sua diminuição e progressão para doença tinham como consequência a diminuição da atividade dos nAbs. Também foi demonstrado que alguns doentes infetados com VIH-2, numa fase crónica de doença, conseguem produzir nAbs autólogos. No entanto, a maioria dos indivíduos produzem nAbs heterólogos (Marcelino et al., 2012).
8.3.2. Determinantes da neutralização
18 al., 2014). Esta última região é bastante conservada e é por isso um dos alvos preferenciais
dos anticorpos neutralizantes (Checkley et al., 2011). No entanto, o vírus consegue escapar aos anticorpos neutralizantes através de uma série de estratégias tais como, a variação nas sequências nucleotídicas, alterações conformacionais ou adição de glicanos (Bunnik et al., 2010). Um exemplo é o aumento do tamanho do loop do domínio V1/V2 e do número de locais de glicosilação que promovem o escape à neutralização, o que pode ser correlacionado com a progressão da doença (Checkley et al., 2011).
No caso do VIH-2, foram descritos alguns epitopos alvo dos anticorpos neutralizantes, presentes na região terminal COOH da gp41 e nas regiões V1, V2, V4 e C5 na gp125, assim como a região V3 (McKnight et al., 1996; Marcelino et al., 2010; Kong et al., 2012; de Silva et al., 2012).
8.3.2.1. Região V3 da gp-SU
Estudos anteriores demonstraram que esta região, com estrutura e conformação em loop, é responsável não só na escolha do co-recetor a ser utilizado durante a ligação do vírus à célula mas também na indução de nAbs (Shi et al., 2005; Borrego et al., 2008; Visseaux et al., 2011). A região V3 do VIH-2 é rodeada pelas regiões C2 e C3, tal como acontece com o VIH-1. Foi observado que nAbs do tipo IgG têm como alvo preferencial a região C2V3C3. Isto não significa que estes anticorpos neutralizem por si o vírus mas podem servir de sinalização para que outros anticorpos específicos para outras regiões atuem em conjunto (Marcelino et al., 2012).
Estudos com anticorpos monoclonais (do inglês Monoclonal Antibodies, mAbs) permitiram perceber que os mesmos tinham capacidade de neutralizar isolados primários de VIH-1, tendo como alvo epitopos conformacionais presentes na região V3 (Conley et
al., 1994; Krachmarov et al., 2006 ). O mesmo foi provado para a neutralização do
VIH-19
2 e, consequentemente, como o vírus pode ser neutralizado (Sourial & Nilsson, 2008; Uchtenhagen et al., 2011). A figura 9 mostra a ligação entre um Fab anti-V3 do VIH-2 e a gp125.
Figura 9: Estrutura conformacional em trímero da gp125 e a ligação de um Fab anti-V3 do VIH-2 (Adaptado de Uchtenhagen et al., 2011).
Porém, e embora a região V3 seja a responsável pela sensibilidade à neutralização em ambos os vírus, a resposta dada por anticorpos neutralizantes não é igual para os dois tipos de vírus, sendo que o VIH-2 é mais facilmente neutralizado do que o VIH-1 (Chauveau et al., 2015). Este último tem uma maior capacidade de escapar à neutralização e uma das explicações para este fenómeno é a mudança no padrão de glicosilação da glicoproteína, já que os potenciais locais de glicosilação na região V3 do VIH-1 são 4 a 5, ou seja, em maior número do que para a mesma região do VIH-2, que são 2 a 3 (Shi et
al., 2005; Wei et al., 2003). Estudos anteriores permitiram observar que o número de
20 Enquadramento e objetivos do estudo
Tal como foi dito anteriormente, os anticorpos neutralizantes têm a capacidade de inibir a entrada do vírus nas células, contribuindo assim para o controlo da replicação viral. O seu alvo preferencial é a região V3 do domínio C2V3C3 existente na gp125 do VIH-2. No entanto, desconhece-se ainda de que forma as regiões C2 e C3, contribuem para a indução de uma neutralização eficaz.
Foi observado em um estudo anterior que as regiões C2 e C3 encontram-se bastante expostas no invólucro e podem conter em si epitopos neutralizáveis. Tal como acontece no VIH-1, as regiões C2 e C3 do VIH-2 são as mais variáveis no que toca à diversidade de aminoácidos (Barroso et al., 2011). Logo, é importante perceber qual o papel de cada uma das regiões do domínio C2V3C3 da gp125 na indução de uma resposta neutralizante, o que poderia permitir o desenvolvimento de uma vacina, através da produção de péptidos sintéticos.
21 II. Material e métodos
1. Origem e descrição do vetor plasmídico
O vetor plasmídico foi construído de forma a conter a região C2V3C3 do gene env do VIH-2, para transformação em bactérias competentes Escherichia coli (E. coli) TOP 10 (Invitrogen™, Estados Unidos da América (EUA)), como descrito anteriormente (Marcelino et al., 2005). O plasmídio gerado foi denominado de pTrcC2-C3.
Figura 10: Esquema detalhado sobre o vetor pTrcHis e as suas características. O local de inserção do fragmento C2V3C3 encontra-se assinalado na figura (Adaptado do manual do fabricante).
22
(IPTG) ao meio de crescimento bacteriano. De igual importância é o gene de resistência à ampicilina, que permite a seleção dos clones bacterianos. Outra característica é o fragmento de 6xHis, responsável pela formação de uma cauda de poli-histidinas que auxilia no processo de purificação das proteínas recombinantes.
2. Extração de ADN plasmídico
Numa primeira fase, foi necessário fazer a seleção dos clones bacterianos recombinantes. Para isso, colocou-se meio LB-Agar (Anexo 1), suplementado com ampicilina (100 µg/mL) (Sigma-Aldrich, Alemanha), em placas de Petri, para isolamento das colónias bacterianas de interesse. As placas foram colocadas a incubar a 37ºC, durante a noite (do inglês overnight, ON). As colónias foram repicadas e inoculadas em meio LB líquido (Anexo 1), igualmente suplementado com ampicilina (100 µg/mL) (Sigma-Aldrich, Alemanha), em agitador orbital, a 37ºC, 250 rpm, ON.
A extração foi realizada com recurso ao kit comercial GenElute™ Plasmid
Miniprep Kit (Sigma-Aldrich, Alemanha), seguindo as instruções do fabricante.
Resumidamente, recolheu-se o pellet de 1 mL da cultura líquida preparada anteriormente, após centrifugação (Centrifuge 5415 D, Eppendorf, Alemanha) a 12000 g, durante 1 minuto. O pellet foi completamente ressuspendido com 200 µL de solução de ressuspensão e homogeneizado no vórtex. De seguida, fez-se a lise celular com a solução de lise e misturou-se por inversão até a mistura ter um aspecto viscoso. Após 5 minutos, neutralizou-se com 350 µL de solução de neutralização, misturou-se por inversão 4 a 6 vezes e centrifugou-se (Centrifuge 5415 D, Eppendorf, Alemanha) a 12000 g, durante 10 minutos. O objetivo foi obter um lisado neutralizado, que foi transferido para uma coluna previamente preparada (a preparação da coluna fez-se num tubo de microcentrífuga, com adição de 500 µL de solução de preparação da coluna, seguida de centrifugação (Centrifuge 5415 D, Eppendorf, Alemanha) a 12000 g, durante 1 minuto, e descarte de sobrenadante). Após a transferência, foi feita uma centrifugação (Centrifuge 5415 D, Eppendorf, Alemanha) a 12000 g, durante 1 minuto. A lavagem da coluna foi feita com a adição de 750 µL de solução de lavagem diluída, seguida de centrifugação (Centrifuge 5415 D, Eppendorf, Alemanha) a 12000 g, durante 1 minuto. Descartou-se o sobrenadante e procedeu-se a nova centrifugação (Centrifuge 5415 D, Eppendorf, Alemanha) a 12000
23
a coluna para um novo tubo de microcentrífuga e adicionou-se 100 µL de solução de eluição. Centrifugou-se (Centrifuge 5415 D, Eppendorf, Alemanha) a 12000 g, durante 1 minuto. O ADN plasmídico eluído, que serviu de molde para a amplificação da região C2V3C3, foi quantificado com o espectrofotómetro NanoDrop™ 1000 (Thermo Fisher Scientific, EUA).
A visualização do ADN plasmídico fez-se por eletroforese em minigel de agarose (Sigma-Aldrich, Alemanha) a 1% (p/v), em tampão Tris-Acetato-EDTA (TAE) 1X (Anexo 1), com adição de brometo de etídio (1:20000, stock 10%). A amostra foi aplicada no gel, após mistura de 15 µL de ADN com 5 µL de loading dye 6X (Fermentas, Canadá). Foi também aplicado um marcador molecular (MM) GeneRuler™ DNA Ladder Mix (Fermentas, Canadá), para comparação dos tamanhos dos fragmentos, após migração no gel. A voltagem utilizada foi de 80V, durante 30 minutos. Todos os fragmentos obtidos foram visualizados com recurso a radiação ultravioleta.
3. Origem e preparação dos primers
A sequência do gene env do VIH-2 pode ser encontrada na base de dados
24 Figura 11: Sequência nucleotídica do gene env do VIH-2. As regiões C2, V3 e C3 encontram-se
sublinhadas a verde, a amarelo e a cinzento, respetivamente.
25
Os primers (Stabvida, Portugal) foram selecionados com base nas sequências conhecidas para cada uma das regiões descritas anteriormente e nas sequências para os locais de corte das enzimas de restrição Bam HI e HindIII. Este último pormenor é muito importante pois é o que permite que cada fragmento seja clonado exatamente dentro do
Multiple Cloning Site (MCS) do vetor plasmídico, que é cortado pelas enzimas de
restrição anteriormente referidas. A elaboração e seleção dos primers adequados foram feitas com recurso ao software Primer 3 (Koressaar & Remm, 2007; Untergrasser et al., 2012).
Tabela 1:Caracterização dos primers utilizados na amplificação de ADN por PCR.
Os liofilizados dos primers foram hidratados com Nuclease-free water (QIAGEN, Alemanha), para uma concentração de 100 M. Estes stocks serviram para fazer alíquotas de trabalho, por diluição também com Nuclease-free water (QIAGEN, Alemanha), com concentração final de 20 M, que foram mantidas a -20ºC.
4. Amplificação de regiões do gene env do VIH-2
O ADN das regiões C2, V3 e C3 foi amplificado, respetivamente, por Reação em Cadeia da Polimerase (do inglês Polymerase Chain Reaction, PCR). O ADN da região C2V3C3 foi também amplificada para servir de controlo. O PCR foi realizado com recurso a um termociclador GenPro (Bioer Technologies, China). As condições para a amplificação são descritas na tabela 2:
Região amplificada
Nome do
Primer Sequência 5' - 3' Tm (ºC)
C2F AAA AGG ATC CTT TGG CTT TAA TGG CAC TAG GG 61.9 C2R GGG CAA GCT TAT GCA TAG TAA GAT TAT ACT GT 58.2 V3F ACA GGA TCC TGT AAG AGG CCA GGA AAT AAG 60.9 V3R TGC AAG CTT TTC GCC TTT GAA CCA GCA CCA 66.0 C3F AAA GGA TCC TGG AGG GAA GCC ATG CAG 64.1 C3R CTC AAG CTT TCT GCA GTT AGT CCA CAT ATA TAC 58.3 C2V3C3F AAT GGC ACT AGG GCA GAA A 54.2 C2V3C3R CTG CAT CTG AGC CTC TTC CT 56.7 C2
V3
C3
26 Tabela 2:Condições utilizadas durante o PCR para a amplificação do ADN das regiões C2, V3 e C3. As mesmas condições foram utilizadas para a amplificação do ADN do domínio C2V3C3.
Para cada região a amplificar foi feita uma mistura de PCR que continha 10 µL de
GoTaq® buffer 5X (Promega, EUA), 0,50 µL de GoTaq® DNA polymerase (5U/µl)
(Promega, EUA), 1,5 µL de dNTP Mix 10mM (Promega, EUA), 1,5 µL de ADN plasmídico, 0,50 µL de primer forward e 0,50 µL de primer reverse (específicos para cada região a amplificar), e Nuclease-free water (QIAGEN, Alemanha) até perfazer um volume final de 50 µL.
Para garantir que os produtos de PCR estavam a ser amplificados de forma específica, foi feita eletroforese em minigel de agarose a 1% (p/v), nas condições descritas anteriormente. Foi feita uma mistura para cada fragmento amplificado, com 15 µL de ADN e 5 µL de loading dye 6X (Fermentas, Canadá). O MM aplicado foi o Mass Ruler™
DNA Ladder, Low Range (Fermentas, Canadá). A voltagem utilizada foi novamente de
80V mas durante 1h30. Todos os fragmentos obtidos foram visualizados com recurso a radiação ultravioleta.
5. Purificação dos produtos de PCR
Esta etapa foi feita com recurso ao illustra™ GFX™ PCR DNA and Gel Band
Purification Kit (GE Healthcare, Inglaterra). Este kit permite purificar ADN de soluções
ou reações enzimáticas mas também de géis de agarose. Neste caso, foi seguido o protocolo para a primeira opção, segundo as instruções do fabricante. Em suma, foi adicionado o Capture buffer type 3 a cada amostra de ADN, numa proporção de 1:5. Misturou-se e centrifugou-se (Centrifuge 5415 D, Eppendorf, Alemanha). Por cada purificação a fazer, foi colocada uma coluna GFX MicroSpin dentro de um tubo coletor.
Etapas Condições
Desnaturação 95ºC, 5 minutos
Amplificação 95ºC, 30 segundos; 56ºC, 30 segundos; 72ºC, 30 segundos (30 ciclos)
Extensão 72ºC, 5 minutos
27
A mistura anterior foi carregada na coluna, que foi a centrifugar (Centrifuge 5415 D, Eppendorf, Alemanha), ainda no tubo coletor, durante 30 segundos, a 16000 g. O sobrenadante presente no tubo coletor foi descartado. Foram adicionados 500 µL de Wash
buffer type 1 à coluna, que foi novamente a centrifugar (Centrifuge 5415 D, Eppendorf,
Alemanha), juntamente com o tubo coletor, a 16000 g, durante 30 segundos. O tubo coletor foi descartado e a coluna foi colocada em um novo tubo de microcentrífuga de 1,5 mL (DNAse-free). A eluição foi feita por adição de 50 µL de Elution buffer type 4 no centro da membrana da coluna e subsequente incubação a temperatura ambiente, durante 1 minuto. Centrifugou-se (Centrifuge 5415 D, Eppendorf, Alemanha) a coluna juntamente com o tubo, a 16000 g, durante 1 minuto, para recuperação do ADN purificado. O ADN purificado foi finalmente quantificado com o espectrofotómetro NanoDrop™ 1000 (Thermo Fisher Scientific, EUA).
6. Digestão dupla enzimática
Dadas as características dos primers desenhados, todos os produtos amplificados continham não só cada região de interesse (C2, V3, C3 e C2V3C3, respetivamente), mas também sequências para os locais de corte das enzimas de restrição Bam HI e Hind III. Ao fazer a digestão dupla com estas enzimas, o ADN amplificado passou a ter extremidades coesivas, essenciais para o passo de clonagem no vetor plasmídico.
28 7. Purificação dos produtos de digestão
A purificação do ADN obtido por digestão dupla foi feita por precipitação com etanol absoluto. Para cada fragmento de ADN das várias regiões foi preparado um tubo com 30 µL de ADN, com 70 µL de Nuclease-free water (QIAGEN, Alemanha). A cada um foi adicionado 100 µL de uma mistura de fenol/clorofórmio. Todos os tubos foram ao vórtex durante 1 minuto. Depois todos os tubos foram a centrifugar (Centrifuge 5415 D, Eppendorf, Alemanha) durante 5 minutos a velocidade máxima, para posterior remoção do sobrenadante para novos tubos. A estes foram adicionados 200 µL de etanol a 100%. Depois de misturados no vórtex, os tubos foram armazenados a -20ºC, ON. No dia seguinte e depois de retirados do congelador, os tubos foram sujeitos a centrifugação a velocidade máxima (Centrifuge 5415 D, Eppendorf, Alemanha), durante meia hora, a 4ºC. Removeram-se os sobrenadantes e foram adicionados 100 µL de etanol a 70% ao
pellet de cada tubo. Procedeu-se a nova centrifugação (Centrifuge 5415 D, Eppendorf,
Alemanha), durante 10 minutos, 4ºC, a velocidade máxima. Removeram-se os sobrenadantes e deixaram-se os tubos abertos a temperatura ambiente durante 10 minutos. Adicionou-se Nuclease-free water (QIAGEN, Alemanha) a cada tubo e ressuspendeu-se. Todo o ADN obtido e purificado foi quantificado com recurso ao espectrofotómetro NanoDrop™ 1000 (Thermo Fisher Scientific, EUA).
8. Clonagem dos fragmentos de ADN obtidos por PCR no vetor
8.1. Obtenção do vetor a partir do gel de agarose
O vetor utilizado nas clonagens teve origem no vetor plasmídico inicial, o pTrcC2-C3. Após extração do ADN plasmídico, como descrito anteriormente, fez-se nova digestão dupla com as enzimas de restrição Bam HI e Hind III, para obter o vetor sem a região C2V3C3, que serviu para fazer a ligação dos fragmentos de ADN obtidos anteriormente por amplificação e digestão enzimática.
29
2h30, e depois foi adicionado 1 µL de Calf-Intestinal Alkaline Phosphatase (CIAP; Fermentas, Canadá), tendo-se deixado a mesma a decorrer mais 30 minutos.
30
durante 1 minuto, para recuperação do ADN purificado. A sua concentração foi obtida por leitura em espectrofotómetro NanoDrop™ 1000 (Thermo Fisher Scientific, EUA).
8.2. Ligação dos produtos de PCR ao vetor plasmídico e transformação bacteriana das ligações
Foi feita uma mistura de reação para cada fragmento de ADN das regiões C2, V3 e C3, respetivamente. Foi feita também uma mistura em que foi colocado o vetor sem fragmento de ADN a ligar, para controlar a sua capacidade de re-ligação. A tabela 3 mostra o que foi colocado em cada tubo.
Tabela 3: Descrição das misturas de reação utilizadas na clonagem.
Quantidades de Reagentes (µL)
Vetor Fragmento(ADN
) Ligase buffer
T4 Ligase
Tris buffer 10 mM pH8
C2 1 3 2 2 12
Volume final de 20 µL
V3 1 1 2 2 14
C3 1 2 2 2 13
Vetor 1 0 2 2 15
31
Alemanha) que permite a seleção de clones recombinantes. As mesmas foram incubadas a 37ºC, ON.
Na manhã seguinte, as colónias presentes nas placas foram repicadas separadamente e inoculadas em 10 mL de meio LB líquido (Anexo 1), suplementado com ampicilina (100 µg/mL) (Sigma-Aldrich, Alemanha), como descrito anteriormente. Com estes inóculos foram feitos stocks de bactérias transformadas, em glicerol a 20% (Anexo 1), que foram armazenados a -80ºC. Estes inóculos permitiram também fazer a validação dos clones por sequenciação (Stabvida, Portugal), após extração do ADN plasmídico por Miniprep e digestões enzimáticas, como descritas anteriormente. Os produtos das digestões obtidos foram confirmados por electroforese em minigel de agarose a 1%, nas condições descritas anteriormente. Para facilitar a descrição dos novos plasmídios, os mesmos passaram a denominar-se de pTrcC2, pTrcV3 e pTrcC3.
9. Produção de péptidos recombinantes
Inicialmente foram preparados pré-inóculos de várias colónias de E. coli TOP10 transformadas com pTrcC2, pTrcV3 e pTrcC3, em 5 mL de meio LB líquido (Anexo 1), suplementado com ampicilina (100 µg/mL) (Sigma-Aldrich, Alemanha). Estes foram colocados a 37ºC, 250 rpm, ON.
Foram preparados Erlenmeyers com 500 mL de meio LB líquido (Anexo 1), suplementado com ampicilina (100 µg/mL) (Sigma-Aldrich, Alemanha). A cada um foi adicionado o respetivo pré-inoculo preparado anteriormente. Os Erlenmeyers foram colocados a incubar em agitador orbital, a 37ºC e 250 rpm. Foram feitas leituras de
DO600nmaté a mesma atingir o valor de 0,8. A indução da atividade da β-galactosidase
32
10 segundos de ultra-sons (UP200S, Hielscher Ultrasound Technology, Alemanha). Por fim, os lisados celulares foram centrifugados Centrifuge 5810 R, Eppendorf, Alemanha) durante 20 minutos, a 3000 rpm, e os sobrenadantes foram recolhidos.
10. Purificação de péptidos recombinantes
Este passo foi realizado com recurso a cromatografia líquida, a Fast Protein
Liquid Chromatography (FPLC), que consiste na separação de proteínas presentes em
misturas para sua análise e purificação. Como o vetor plasmídico permitiu a produção de péptidos recombinantes com caudas de poli-histidinas, a purificação foi realizada de acordo com a interação entre as histidinas e o níquel presente na resina de Sepharose e por eluição por variação do pH.
O sistema utilizado foi o GE Pharmacia FPLC system (GE Healthcare, Inglaterra) e a coluna cromatográfica foi a HisTrap™ HP, 5 mL (GE Healthcare, Inglaterra). Para cada péptido a purificar, foi filtrada uma amostra do sobrenadante recolhido anteriormente com um filtro de 0,45 µm. A amostra filtrada foi então diluída na proporção de 1:2 com tampão de ureia 8M pH 7.8 (Anexo 1) e aplicada na coluna. Foi feita uma primeira eluição com o tampão referido anteriormente, seguido de duas eluições com tampão de ureia 8M pH 6 e pH 5.3 (Anexo 1), respetivamente, para eliminação de contaminantes. A eluição do péptido recombinante foi feita com tampão de ureia 8M a pH 4 (Anexo 1). Foram recolhidas várias alíquotas para quantificação do péptido recombinante a Abs = 280, com recurso ao NanoDrop™ (Thermo Scientific, EUA).
11. Avaliação da reatividade dos anticorpos contra os péptidos recombinantes
11.1. Amostras biológicas
33 11.2. Enzyme-Linked Immunosorbent Assay (ELISA)
Este procedimento permitiu observar a antigenicidade de cada um dos péptidos purificados. O tipo de ELISA realizado foi pelo método indireto.
Com recurso a um módulo Nunc-Immuno™ com microtiras Maxisorp™ (Thermo Scientific, EUA), adsorveram-se 100 µL/poço de cada péptido recombinante diluído em tampão bicarbonato 0.05M pH 9.4 (Anexo 1). O módulo foi colocado a incubar a 4ºC, ON. A figura 12 esquematiza a forma como foi feita a adsorção dos antigénios (péptidos) e também a ordem pela qual se aplicaram os soros.
Figura 12: Representação esquemática da aplicação dos antigénios e dos soros a testar. SP significa soro positivo para VIH-2 e SN soro negativo para VIH-2.
Os péptidos C2, V3 e C3 foram adsorvidos com uma concentração de 25 µg/mL, e o péptido C2V3C3 foi adsorvido com uma concentração de 2,5 µg/mL. Foram feitas duas lavagens com 300 µL/poço com Tampão Tris Salino (do inglês Tris-Buffered Saline, TBS) a pH 7.4 (Anexo 1). Os micropoços foram bloqueados com Albumina de Soro Bovino (do inglês Bovine Serum Albumin, BSA) (Sigma-Aldrich, Alemanha) a 5% (Anexo 1) e incubados 1h, a 37ºC. Foram feitas mais duas lavagens com tampão TBS, nas condições anteriores. Em cada micropoço adicionaram-se 100 µL de cada soro diluído em TBS-Tween 20 (Anexo 1) (diluição 1:300) e fez-se nova incubação a 37ºC, durante 1h. Após 5 lavagens com TBS-Tween 20, adicionaram-se 100 µL/poço de anticorpo de cabra anti-IgG humano conjugado com fosfatase alcalina (Sigma-Aldrich, Alemanha), diluído em TBS-Tween 20 (1:4000), com 0,1% de BSA (Sigma-Aldrich, Alemanha). Os micropoços foram mais uma vez incubados a 37ºC, durante 1h. Depois de lavados 5 vezes nas mesmas condições descritas anteriormente, adicionaram-se 100 µL/poço do substrato cromogénico p-nitrofenilfosfato (SIGMAFAST™ p-Nitrophenil phosphate Tablets,
Soros SP1 SP2 SP3 SP4 SP5 SP6 SP7 SP8 SP9 SN
Poços 1 2 3 4 5 6 7 8 9 10 11 12
A SP1 SP2
B SP3 SP4
C SP5 SP6
D SP7 SP8
E SP9 SN
F G H C2 V3 C3 C2V3C3 Sem antigénio Antigénios (péptidos)
34
Sigma-Aldrich, Alemanha) e incubou-se no escuro durante 30 minutos. De seguida, foram feitas leituras da densidade óptica (DO) em leitor de microplacas (Sinergy™ HT, BioTek®, EUA), a 405 nm.
11.3. Análise estatística
35 III. Resultados
1. Clonagem
1.1. Caracterização dos primers utilizados e dos fragmentos de ADN a amplificar
Os primers utilizados na amplificação do ADN das regiões C2, V3 e C3 e do domínio C2V3C3, pertencentes ao gene env do VIH-2, foram desenhados e selecionados tendo em conta a sequência nucleotídica para cada região, como descrito no capítulo anterior. As imagens seguintes esquematizam a sequência nucleotídica de cada região e dos seus respetivos primers. As sequências nucleotídicas correspondentes aos locais de corte das enzimas de restrição que foram utilizadas para as posteriores digestões enzimáticas encontram-se igualmente assinaladas nas figuras seguintes.
Figura 13: Sequência nucleotídica correspondente à região C2 do gene env (5’3’). Por baixo e do lado
esquerdo, pode ser vista a sequência nucleotídica do primer C2F, com a sequência correspondente ao local
de corte pela enzima de restrição Bam HI; do lado direito encontra-se a sequência nucleotídica do primer
C2R, também com a sequência do local de corte pela enzima de restrição Hind III assinalada.
Figura 14: Sequência nucleotídica correspondente à região V3 do gene env (5’3’). Por baixo e do lado
esquerdo, pode ser vista a sequência nucleotídica do primer V3F, com a sequência correspondente ao local
de corte pela enzima de restrição Bam HI; do lado direito encontra-se a sequência nucleotídica do primer
V3R, também com a sequência do local de corte pela enzima de restrição Hind III assinalada.
Figura 15: Sequência nucleotídica correspondente à região C3 do gene env (5’ 3’). Por baixo e do lado
esquerdo, pode ser vista a sequência nucleotídica do primer C3F, com a sequência correspondente ao local
de corte pela enzima de restrição Bam HI; do lado direito encontra-se a sequência nucleotídica do primer