UNIVERSIDADE FEDERAL DO CEARÁ CENTRO DE CIÊNCIAS
DEPARTAMENTO DE QUÍMICA ORGÂNICA E INORGÂNICA PROGRAMA DE PÓS-GRADUAÇÃO EM QUÍMICA
PAULA KARINA SANTOS UCHÔA
ESTUDO DO POTENCIAL QUÍMICO-FARMACOLÓGICO DE METABÓLITOS SECUNDÁRIOS DE FUNGOS DA COSTA CEARENSE: Aspergillus niger
ESTUDO DO POTENCIAL QUÍMICO-FARMACOLÓGICO DE METABÓLITOS SECUNDÁRIOS DE FUNGOS DA COSTA CEARENSE: Aspergillus niger
Tese apresentada ao Programa de Pós-Graduação em Química da Universidade Federal do Ceará, como requisito parcial à obtenção do título de Doutor em Química. Área de concentração: Química Orgânica.
Orientador: Prof. Dra. Mary Anne Sousa Lima.
ESTUDO DO POTENCIAL QUÍMICO-FARMACOLÓGICO DE METABÓLITOS SECUNDÁRIOS DE FUNGOS DA COSTA CEARENSE: Aspergillus niger
Tese apresentada ao Programa de Pós-Graduação em Química da Universidade Federal do Ceará, como requisito parcial à obtenção do título de Doutor em Química. Área de concentração: Química Orgânica.
Aprovada em: 25 / 01 / 2017.
BANCA EXAMINADORA
________________________________________ Prof. Dra. Mary Anne Sousa Lima (Orientador)
Universidade Federal do Ceará (UFC)
_________________________________________ Prof. Dra. Fátima Miranda Nunes
Universidade Federal do Ceará (UFC)
_________________________________________ Prof. Dra. Nilce Gramosa Pompeu Viana de Sousa Brasil
Universidade Federal do Ceará (UFC)
_________________________________________ Prof. Dra. Antônia Fádia Valentim de Amorim
Universidade Estadual do Ceará (UECE)
_________________________________________ Prof. Dra. Selene Maia de Morais
À Deus acima de tudo, por me conceder força e fé para caminhar.
À Prof. Dra. Mary Anne Sousa Lima pela excelente orientação, compreensão e tempo
dedicado à produção dessa tese.
Ao professor Dr. Leslie Gunatilaka (University of Arizona), pela orientação durante o estágio de doutorado sanduíche.
Aos professores participantes da banca examinadora pelo tempo, pelas valiosas colaborações e sugestões.
Aos professores do programa de Pós-Graduação em Química pelos conhecimentos transmitidos.
Aos professores, Edilberto Rocha Silveira (UFC), Antônia Pimenta (UFC), Raimundo Braz-Filho (UENF), Leticia V. C. Lotufo (USP), Diego Wilke (UFC), pela contribuição no desenvolvimento desse trabalho, e em especial à Professora Conceição Oliveira por todo carinho, atenção e apoio.
Ao Dr. Kithsiri.Wijeratne, Dr. Yaming Xu e Dra. Patricia Espinosa pelo enorme ensinamento e
contribuição na realização dos experimentos de epigenética.
À Dra. Natália Nogueira pelos primeiros ensinamentos na parte microbiológica, além das
valorosas reflexões, críticas e sugestões recebidas.
Aos amigos e companheiros de laboratório, Honório Sousa, Regivaldo Gomes, Paulo Riceli, João Evangelista, Hélio Oliveira, por toda ajuda, ensinamentos e conselhos durante o curso. Aos colegas do Labs e Lafiplam, pela acolhida em seus laboratórios e suporte para a realização de muitos experimentos.
Aos operadores do infravermelho, espectrômetro de massa e de ressonância magnética nuclear pela obtenção dos espectros.
Às amigas Manuela e Macella pelo companheirismo nos momentos mais difíceis. À minha família por todo amor e apoio em todos os momentos.
Aos órgãos financiadores CNPq, CAPES, FUNCAP, FINEP E PRONEX. Em particular à CAPES e ao CNPq pelas bolsas de pesquisa a mim concedidas, proporcionando maior tempo de dedicação à pesquisa.
O extrato AcOEt da cepa BRF074 (Aspergillus niger), cultivada em BD e isolada a partir de sedimentos coletados na praia do Pecém (Fortaleza-CE), apresentou alta atividade citotóxica (77%; IC50 0,95) frente à linhagem de células tumorais HCT-116 (cólon). Desta forma, um
estudo bioguiado de cultivo foi conduzido para este micro-organismo utilizando quatro meios de culturas distintos: BD (batata, dextrose), BDL (batata, dextrose e levedura), MPD (malte, peptona e dextrose) e MntPL (manitol, peptona e levedura), em quatro períodos de crescimento: 07, 14, 21 e 28 dias. A partir do cultivo de A. niger em meio de cultura BD por 28 dias, foram isolados sete metabólitos secundários: hidroxi-L-Pro-L-Leu), ciclo(4-hidroxi-L-Pro-L-Phe), ciclo(L-Pro-L-Leu), ciclo(L-Pro-L-Phe), pseurotin D, pseurotin A e clovalicina. O meio de cultivo BDL (14 dias) levou ao isolamento da dicetopiperazina ciclo(L-Pro-L-Val) e dos ciclopeptídeos malformina A e malformina C, enquanto que o meio MPD durante 7 dias forneceu um ester furânico, de caráter inédito na literatura. As malforminas A e C e o composto inédito apresentaram-se ativas frente a linhagem de células HCT-116. Um método quantitativo de cromatografia líquida de alta eficiência (CLAE) foi desenvolvido e validado para a quantificação destes metabólitos citotóxicos em diferentes meios nutricionais e períodos de crescimento. A partir deste método, foi observado que no meio BDL, nos períodos de 21 e 28 dias as malforminas A e C foram produzidos em maior quantidade, enquanto que o ester furânico foi produzido em maiores teores no meio MPD em 28 dias. Em paralelo, foi realizado um estudo envolvendo epigenética utilizando o reagente SAHA e a cepa do fungo LG0949. A partir deste experimento foram isolados os compostos phomalactona, dihidrophomalactona e acetophomalactona, e os compostos acetanilida e dímero do reagente SAHA. O isolamento dos metabólitos secundários foi realizado através de cromatografia em fase reversa C18, utilizando cartucho para extração em fase sólida e cromatografia líquida de alta eficiência (CLAE). A caracterização estrutural foi realizada através do uso de métodos espectrométricos, tais como espectrometria de massas, ressonância magnética nuclear (1H e 13C) e infravermelho, além de comparação de dados da literatura.
EtOAc extract from the BRF074 strain (Aspergillus niger), cultivated in BD medium and isolated from sediments collected on Pecém beach (CE), showed high cytotoxic activity (77%, IC50 0,95) against the cancer cell line HCT-116 (colon). Thus, a bioguided study
aiming the optimization of the culturing conditions of A. niger was conducted in which four different culture media were evaluated: PD (potato, dextrose), PDY (potato, dextrose and yeast), MPD (malt, peptone and dextrose) and MntPY (mannitol, peptone and yeast), in four growth periods: 07, 14, 21 and 28 days. From the cultive of A. niger in BD culture medium during 28 days, seven secondary metabolites were isolated: cyclo(4-hydroxy-L-Pro-L-Leu), cyclo(4-hydroxy-L-Pro-L-Phe), cyclo(L-Pro-L-Leu), cyclo(L-Pro-L-Phe), pseurotin D, pseurotin A and chlovalicin. The BDL culture medium (14 days) led to the isolation of the diketopiperazine cyclo(L-Pro-L-Val) and cyclopeptides malformin A and malformin C, whereas the MPD medium for 7 days provided a new compound, an ester furanic first report in the literature. Malformins A and C and the new compound were active against the cancer cell line HCT-116. A quantitative method of high performance liquid chromatography (HPLC) was developed and validated for the quantification of these cytotoxic metabolites in different nutritional media and growth periods. This method reveal that in the BDL medium, within 21 and 28 days, greater amounts of malformin A and C were produced, whereas the furanic ester was produced in higher levels in the MPD medium with 28 days. In parallel, an epigenetic study was performed using SAHA as reagent and the fungus strain LG0949. This experiment led to the isolation of the compounds phomalactone, dihydrophomalactone and acetophomalactone, acetanilide and a dimer of the SAHA. Isolation of the secondary metabolites was conducted using C18 reverse phase chromatography using a solid phase extraction cartridge and high performance liquid chromatography (HPLC). The structural characterization was done using spectrometric techniques such as mass spectrometry, nuclear magnetic resonance (1H and 13C) and infrared, in addition to comparison with literature data.
Figura 1 Exemplos de fármacos obtidos a partir de micro-organismos ... 27
Figura 2 Metabólitos bioativos obtidos a partir de fungos de origem marinha ... 29
Figura 3 Aspergillus niger ... 30
Figura 4 Inibição do crescimento celular (%) dos extratos do cultivo de A. niger em meio BD com 7, 14, 21 e 28 dias de crescimento, testado na concentração de
50 g/mL ... 57
Figura 5 Cromatogramas da análise por CLAE correspondente a 7, 14, 21 e 28 dias de cultivo de A. niger em BDL ... 59
Figura 6 Cromatogramas da análise por CLAE correspondente a 7, 14, 21 e 28 dias de cultivo de A. niger em MPD ... 59
Figura 7 Cromatogramas da análise por CLAE correspondente a 7, 14, 21 e 28 dias de cultivo de A. niger em MntPL ... 60
Figura 8 Inibição do crescimento celular (%) dos extratos do cultivo de A. niger nos meios de cultivo BDL, MPD e MntPL com 7, 14, 21 e 28 dias de crescimento,
testado nas concentrações de 5 e 50 g/mL para a linhagem de células H CT-116 ... 61
Figura 9 Estrutura química de BRF074 (3) - ciclo(L-Pro-L-Leu) ... 64
Figura 10 Espectro de IV de BRF074 (3) ... 66
Figura 11 Espectro de massa de alta resolução (EM-IES) no modo positivo de BRF074 (3) ... 66
Figura 12 Espectro de RMN 1H de BRF074 (3) (500 MHz, CD
3OD) e expansões (B) e
(C) ... 67
Figura 13 Espectro de RMN 2D, 1H, 1H - COSY de BRF074 (3) (500 MHz, CD
3OD) ... 68
Figura 14 Espectro de RMN 13C - CPD de BRF074 (3) (125 MHz, CD
3OD) ... 69
Figura 15 Espectro de RMN 13C - DEPT 135º de BRF074 (3) (125 MHz, CD
3OD) ... 69
Figura 16 Espectro de RMN 2D, 1H, 13C - HSQC de BRF074 (3) (500 x 125 MHz,
CD3OD) ... 71
Figura 18 Estrutura de BRF074 (1) - ciclo(4-hidroxi-L-Pro-L-Leu) ... 73
Figura 19 Espectro de IV de BRF074 (1) ... 74
Figura 20 Espectro de massa de alta resolução (EM-IES) no modo positivo de BRF074 (1) ... 74
Figura 21 Espectro de RMN 1H de BRF074 (1) (500 MHz, CD
3OD) (A) e expansões (B)
e (C) ... 75
Figura 22 Espectro de RMN 13C - CPD de BRF074 (1) (125 MHz, CD
3OD) ... 76
Figura 23 Espectro de RMN 2D, 1H, 13C - HSQC de BRF074 (1) (500 x 125 MHz,
CD3OD) ... 76
Figura 24 Estrutura química de BRF074 (4) - ciclo(L-Pro-L-Phe) ... 78
Figura 25 Espectro de massa de alta resolução (EM-IES) no modo positivo de BRF074 (4) ... 79
Figura 26 Espectro de RMN 1H de BRF074 (4) (300 MHz, CD
3OD) (A) e expansões (B)
e (C) ... 79
Figura 27 Espectro de RMN 13C de BRF074 (4) (300 MHz, CD
3OD) ... 80
Figura 28 Espectro de RMN 13C - DEPT 135º de BRF074 (4) (75 MHz, CD
3OD) ... 80
Figura 29 Espectro de RMN 2D, 1H, 13C - HSQC de BRF074 (4) (300 x 75 MHz,
CD3OD) ... 81
Figura 30 Espectro de RMN 2D, 1H, 13C - HMBC de BRF074 (4) (300 x 75 MHz,
CD3OD) ... 82
Figura 31 Estrutura química de BRF074 (2) - ciclo(4-hidroxi-L-Pro-L-Phe) ... 83
Figura 32 Espectro de IV de BRF074 (2) ... 85
Figura 33 Espectro de massa de alta resolução (EM-IES) no modo positivo de BRF074 (2) ... 85
Figura 34 Espectro de RMN 1H de BRF074 (2) (300 MHz, CD
3OD) (A) e expansões (B)
Figura 36 Estrutura química de BRF074 (8) - ciclo(L-Pro-L-Tyr) ... 89
Figura 37 Espectro de massa de alta resolução (EM-IES) no modo positivo de BRF074 (8) ... 90
Figura 38 Espectro de RMN 1H de BRF074 (8) (500 MHz, C
5D5N) ... 90
Figura 39 Espectro de RMN 13C - CPD de BRF074 (8) (125 MHz, C
5D5N) ... 91
Figura 40 Espectro de RMN 2D, 1H, 13C - HSQC de BRF074 (8) (500 x 125 MHz,
C5D5N) ... 92
Figura 41 Espectro de RMN 2D, 1H, 13C - HMBC de BRF074 (8) (500 x 125 MHz,
C5D5N) ... 93
Figura 42 Estrutura química de BRF074 (6) - Pseurotina A ... 96
Figura 43 Espectro de IV de BRF074 (6) ... 98
Figura 44 Espectro de massa de alta resolução (EM-IES) no modo positivo de BRF074 (6) ... 98
Figura 45 Espectro de RMN 1H de BRF074 (6) (300 MHz, CD
3OD) (A) e expansões (B)
e (C) ... 99
Figura 46 Espectro de RMN 2D, 1H, 1H - COSY de BRF074 (6) (300 MHz, CD
3OD) ... 100
Figura 47 Espectro de RMN 13C - CPD de BRF074 (6) (75 MHz, CD
3OD) ... 101
Figura 48 Espectro de RMN 13C - DEPT 135º de BRF074 (6) (75 MHz, CD
3OD) ... 101
Figura 49 Espectro de RMN 2D, 1H, 13C - HSQC de BRF074 (6) (300 x 75 MHz,
CD3OD) ... 102
Figura 50 Espectro de RMN 2D, 1H, 13C - HMBC de BRF074 (6) (300 x 75 MHz,
CD3OD) ... 103
Figura 51 Estrutura química de BRF074 (5) - Pseurotina D ... 105
Figura 52 Espectro de IV de BRF074 (5) ... 108
e (C) ... 109
Figura 55 Espectro de RMN 2D, 1H, 1H - COSY de BRF074 (5) (500 MHz, CD
3OD) ... 110
Figura 56 Espectro de RMN 13C - CPD de BRF074 (5) (125 MHz, CD
3OD) ... 111
Figura 57 Espectro de RMN 13C - DEPT 135º de BRF074 (5) (125 MHz, CD
3OD) ... 111
Figura 58 Espectro de RMN 2D, 1H, 13C - HSQC de BRF074 (5) (500 x 125 MHz,
CD3OD) ... 112
Figura 59 Espectro de RMN 2D, 1H, 13C - HMBC de BRF074 (5) (500 x 125 MHz,
CD3OD) ... 113
Figura 60 Estrutura química de BRF074 (7) - clovalicina ... 117
Figura 61 Espectro de IV de BRF074 (7) ... 118
Figura 62 Espectro de massa de alta resolução (EM-IES) no modo positivo de BRF074 (7) ... 118
Figura 63 Espectro de RMN 1H de BRF074 (7) (500 MHz, CD
3OD) (A) e expansão
(B) ... 119
Figura 64 Espectro de RMN 2D, 1H, 1H - COSY de BRF074 (7) (500 MHz, CD
3OD) ... 120
Figura 65 Espectro de RMN 13C - CPD de BRF074 (7) (125 MHz, CD
3OD) ... 121
Figura 66 Espectro de RMN 13C - DEPT 135º de BRF074 (7) (125 MHz, CD
3OD) ... 121
Figura 67 Espectro de RMN 2D, 1H, 13C - HSQC de BRF074 (7) (500 x 125 MHz,
CD3OD) ... 122
Figura 68 Espectro de RMN 2D, 1H, 13C - HMBC de BRF074 (7) (500 x 125 MHz,
CD3OD) ... 123
Figura 69 Estrutura química de BRF074 (9) - Malformina A1 ... 126
Figura 70 Espectro de massa de alta resolução (EM-IES) no modo positivo de BRF074 (9) ... 128
Figura 71 Espectro de RMN 1H de BRF074 (9) (500 MHz, C
5D5N) (A) e expansões (B)
e expansão (B) ... 129
Figura 73 Espectro de RMN 13C - CPD de BRF074 (9) (125 MHz, C
5D5N) ... 130
Figura 74 Espectro de RMN 13C - DEPT 135º de BRF074 (9) (125 MHz, C
5D5N) ... 130
Figura 75 Espectro de RMN 2D, 1H, 13C - HSQC de BRF074 (9) (500 x 125 MHz,
C5D5N) (A) e expansão (B) ... 131
Figura 76 Espectro de RMN 2D, 1H, 13C - HMBC de BRF074 (9) (500 x 125 MHz,
C5D5N) (A) e expansão (B) ... 132
Figura 77 Estrutura química de BRF074 (10) - Malformina C ... 134
Figura 78 Espectro de massa de alta resolução (EM-IES) no modo positivo de BRF074 (10) ... 137
Figura 79 Espectro de RMN 1H de BRF074 (10) (500 MHz, C
5D5N) (A) e expansões (B)
e (C) ... 137
Figura 80 Espectro de RMN 2D, 1H, 1H - COSY de BRF074 (10) (500 MHz, C
5D5N) ... 138
Figura 81 Espectro de RMN 13C - CPD de BRF074 (10) (125 MHz, C
5D5N) ... 139
Figura 82 Espectro de RMN 13C - DEPT 135º de BRF074 (10) (125 MHz, C
5D5N) ... 139
Figura 83 Espectro de RMN 2D, 1H, 13C - HSQC de BRF074 (10) (500 x 125 MHz,
C5D5N) (A) e expansão (B) ... 140
Figura 84 Espectro de RMN 2D, 1H, 13C - HMBC de BRF074 (10) (500 x 125 MHz,
C5D5N) (A) e expansão (B) ... 141
Figura 85 Estrutura química de BRF074 (11) ... 144
Figura 86 Espectro de massa de alta resolução (EM-IES) no modo positivo de BRF074 (11) ... 146
Figura 87 Espectro de RMN 1H de BRF074 (11) (500 MHz, CD
3OD) ... 146
Figura 88 Espectro de RMN 2D, 1H, 1H - COSY de BRF074 (11) (500 MHz, CD
3OD) .... 147
Figura 89 Espectro de RMN 13C - CPD de BRF074 (11) (125 MHz, CD
3OD) ... 148
Figura 90 Espectro de RMN 13C - DEPT 135º de BRF074 (11) (125 MHz, CD
CD3OD) ... 149
Figura 92 Espectro de RMN 2D, 1H, 13C - HMBC de BRF074 (11) (500 x 125 MHz,
CD3OD) ... 150
Figura 93 Compostos citotóxicos BRF074 (9), (10) e (11) produzidos por Aspergillus niger (BRF074) ... 152
Figura 94 Curvas analíticas dos compostos bioativos produzidos por A. niger BRF074. Figura 94A: curva analítica de BRF074 (9); Figura 94B: curva analítica de BRF074 (10) e Figura 94C: curva analítica de BRF074 (11) ... 154
Figura 95 Cromatogramas HPLC dos compostos puros BRF074 (9), (10) e (11) (Figura 95A) e extratos obtidos a partir do cultivo durante 28 dias de A. niger (BRF 074) em meio MPD (Figura 95B), meio BDL (Figura 95C) e meio MntPL (Figura 95D) ... 158
Figura 96 Cromatograma do cultivo de LG0949 em meio de cultura BD sem adição de SAHA ... 160
Figura 97 Cromatograma do cultivo de LG0949 em meio de cultura BD com adição de SAHA na concentração de 500 µM ... 160
Figura 98 Cromatograma do cultivo de LG0949 em meio de cultura BD com adição de SAHA na concentração de 100 µM ... 161
Figura 99 Cromatograma do cultivo de LG0949 em meio de cultura BD com adição de SAHA na concentração de 200 µM ... 161
Figura 100 Cromatograma do cultivo de LG0949 em meio de cultura BD com adição de SAHA na concentração de 300 µM ... 161
Figura 101 Cromatograma do cultivo de LG0949 em meio de cultura BD com adição de SAHA na concentração de 400 µM ... 162
Figura 102 Cromatograma do cultivo de LG0949 em meio de cultura BD com adição de SAHA na concentração de 500 µM ... 162
BD com adição de SAHA na concentração de 200 µM ... 163
Figura 105 Espectros de RMN 1H de LG0949 (1) e LG0949 (2) (400 MHz, CDCl 3) ... 164
Figura 106 Espectros de RMN 1H de LG0949 (1) e LG0949 (3) (400 MHz, CDCl 3) ... 164
Figura 107 Espectros de RMN 1H de LG0949 (4) (400 MHz, DMSO-d 6) ... 165
Figura 108 Espectros de RMN 1H de LG0949 (5) e SAHA (400 MHz, CD 3OD) ... 165
Figura 109 Espectro de RMN 1H da fração LG0949-SAHA-A3 (400 MHz, CDCl 3) ... 166
Figura 110 Cromatograma dos extratos LG0949-SAHA e LG0949-controle ... 167
Figura 111 Cromatograma do fracionamento em CLAE de BRF074-BD-F3 ... 176
Figura 112 Cromatograma do fracionamento em CLAE de BRF074-BD-F4 ... 177
Figura 113 Cromatograma do fracionamento em CLAE de BRF074-BD-F5 ... 178
Figura 114 Cromatograma do fracionamento em CLAE de BRF074-BD-F6 ... 179
Figura 115 Cromatograma do fracionamento em CLAE de BRF074-BDL-F3 ... 181
Figura 116 Cromatograma do fracionamento em CLAE de BRF074-BDL-F6 ... 182
Figura 117 Cromatograma do fracionamento em CLAE de BRF074-MPD-F5 ... 184
Figura 118 Cromatograma do fracionamento em CLAE de LG0949-SAHA-A2-f3 ... 189
Figura 119 Cromatograma do fracionamento em CLAE de LG0949-SAHA-A3 ... 190
Gráfico 1 Classes de metabólitos secundários identificados em A. niger ... 53
Tabela 1 Relação nominal dos metabólitos secundários identificados em Aspergillus niger e origem do micro-organismo ... 33
Tabela 2 Estruturas dos metabólitos secundários identificados em Aspergillus niger ... 39
Tabela 3 Massa dos extratos obtidos no cultivo de A. niger em meio de cultura BD com 07, 14, 21 e 28 dias de crescimento ... 56
Tabela 4 Massa dos extratos obtidos no cultivo de A. niger em meio de cultura BDL com 07, 14, 21 e 28 dias de crescimento ... 57
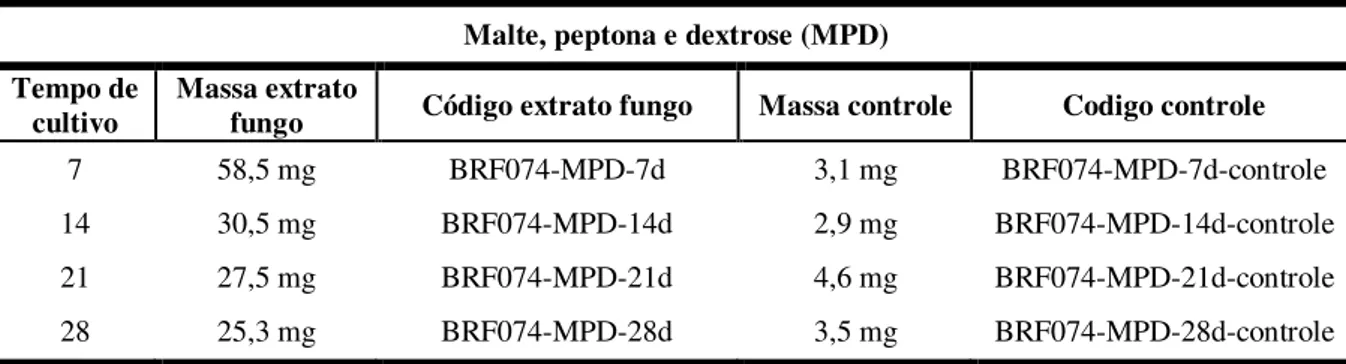
Tabela 5 Massa dos extratos obtidos no cultivo de A. niger em meio de cultura MPD com 07, 14, 21 e 28 dias de crescimento ... 58
Tabela 6 Massa dos extratos obtidos no cultivo de A. niger em meio de cultura MntPL com 07, 14, 21 e 28 dias de crescimento ... 58
Tabela 7 Dados de deslocamento químico (δ) de RMN 13C e padrão de hidrogenação de
BRF074 (3) ... 62
Tabela 8 Dados de RMN 1H e 13C com correlações de 1H, 13C - HSQC e 13C-HMBC nJ
(n = 2, n = 3) de BRF 074 (3) e comparação com os dados de RMN 13C da
literatura para o ciclo(L-Pro-L-Leu) (FURTADO et al., 2005) ... 65
Tabela 9 Dados de RMN 1H e 13C de BRF 074 (1) e comparação com os dados de RMN 1H e 13C da literatura para o ciclo(4-hidroxy-L-Pro-L-Leu) (FURTADO et al.,
2005) ... 73
Tabela 10 Dados de RMN 1H e 13C com correlações de 1H, 13C - HSQC e 13C-HMBC nJ
(n = 2, n = 3) de BRF 074 (4) e comparação com os dados de RMN 13C da
literatura para o ciclo(L-Pro-L-Phe) (FURTADO et al., 2005) ... 78
Tabela 11 Dados de RMN 1H e 13C de BRF 074 (2) e comparação com os dados de RMN 1H e 13C da literatura para ciclo(4-hidroxy-L-Pro-L-Phe) (FURTADO et al,
2005) ... 84
Tabela 12 Dados de RMN 1H e 13C com correlações de 1H, 13C - HSQC e 13C-HMBC nJ
(n = 2, n = 3) de BRF 074 (8) e comparação com os dados de RMN 13C da
(n = 2, n = 3) de BRF 074 (6) e comparação com os dados de RMN 13C da
literatura para Pseurotin A (WIEMANN et al., 2013) ... 97
Tabela 14 Dados de RMN 1H e 13C com correlações de 1H, 13C – HSQC e 13C-HMBC nJ
(n = 2, n = 3) de BRF 074 (5) ... 106
Tabela 15 Dados de RMN 1H e 13C de BRF 074 (5) e comparação com os dados de RMN 1H e 13C de BRF074 (6) - Pseurotin A ... 107
Tabela 16 Dados de deslocamento químico (δ) de RMN 13C e padrão de hidrogenação de
BRF074 (7) ... 114
Tabela 17 Dados de RMN 1H e 13C com correlações de 1H, 13C – HSQC e 13C-HMBC nJ
(n = 2, n = 3) de BRF 074 (7) e comparação com os dados de RMN 13C da
literatura para clovalicina (TAKAMATSU et al., 1996) ... 117
Tabela 18 Dados de RMN 1H e 13C com correlações de 1H, 13C – HSQC e 13C-HMBC nJ
(n = 2, n = 3) de BRF 074 (9) e comparação com os dados de RMN 13C da
literatura para Malformina A1 (KOJIMA et al., 2009) ... 127
Tabela 19 Dados de RMN 1H e 13C com correlações de 1H, 13C – HSQC e 13C-HMBC nJ
(n = 2, n = 3) de BRF074 (10) e comparação com os dados de RMN 13C da
literatura para Malformina C (VAROGLU et al., 2000) ... 135
Tabela 20 Dados de RMN 1H e 13C de BRF074 (10) e comparação com os dados de
RMN 1H e 13C de BRF074 (9) - Malformina A
1 ... 136
Tabela 21 Dados de deslocamento químico (δ) de RMN 13C e padrão de hidrogenação de
BRF074 (11) ... 142
Tabela 22 Dados de RMN 1H e 13C com correlações de 1H, 13C – HSQC e 13C-HMBC nJ
(n = 2, n = 3) de BRF 074 (11) ... 145
Tabela 23 Concentrações dos analitos, BRF074 (9), (10) e (11), injetados em triplicadas e as áreas correspondentes para cada curva analítica ... 153
Tabela 24 Parâmetros da curva analítica utilizados na quantificação dos compostos bioativos BRF074 (9), (10) e (11) produzidos por A. niger (BRF074) ... 153
encontrados nos extratos MPD produzidos por A. niger (BRF 074). Concentração dos extratos = 2000 µg.mL-1 ... 156
Tabela 27 Teores (µg.mL-1) dos compostos bioativos BRF074 (9), (10) e (11)
encontrados nos extratos BDL produzidos por A. niger (BRF 074). Concentração dos extratos = 2000 µg.mL-1 ... 157
Tabela 28 Teores (µg.mL-1) dos compostos bioativos BRF074 (9), (10) e (11)
encontrados nos extratos MntPL produzidos por A. niger (BRF 074). Concentração dos extratos = 2000 µg.mL-1 ... 157
Tabela 29 Rendimento fracionamento cromatográfico de BRF074-BD ... 175
Tabela 30 Massas das frações coletadas no fracionamento em CLAE da amostra BRF074-BD-F3 ... 177
Tabela 31 Massas das frações coletadas no fracionamento em CLAE da amostra BRF074-BD-F4 ... 178
Tabela 32 Massas das frações coletadas no fracionamento em CLAE da amostra BRF074-BD-F6 ... 179
Tabela 33 Rendimento fracionamento cromatográfico de BRF074-BDL ... 180
Tabela 34 Rendimento fracionamento cromatográfico de BRF074-MPD ... 183
Tabela 35 Rendimento fracionamento cromatográfico em Sephadex de LG0949-SAHA .. 188
Rotação óptica específica
Deslocamento químico
Diâmetro
© Copyright
® Marca Registrada
TM Trademark
% Porcentagem
AcOEt Acetato de etila
BD Batata Dextrose
BDA Batata Dextrose Ágar
BDL Batata Dextrose Levedura
BRF074-BD-7d Extrato de Aspergillus niger cultivado em BD durante 7 dias BRF074-BD-14d Extrato de Aspergillus niger cultivado em BD durante 14 dias BRF074-BD-21d Extrato de Aspergillus niger cultivado em BD durante 21 dias BRF074-BD-28d Extrato de Aspergillus niger cultivado em BD durante 28 dias BRF074-BDL-7d Extrato de Aspergillus niger cultivado em BDL durante 7 dias BRF074-BDL-14d Extrato de Aspergillus niger cultivado em BDL durante 14 dias BRF074-BDL-21d Extrato de Aspergillus niger cultivado em BDL durante 21 dias BRF074-BDL-28d Extrato de Aspergillus niger cultivado em BDL durante 28 dias BRF074-MntPL-7d Extrato de Aspergillus niger cultivado em MntPL durante 7
dias
BRF074-MntPL-14d Extrato de Aspergillus niger cultivado em MntPL durante 14 dias
BRF074-MntPL-21d Extrato de Aspergillus niger cultivado em MntPL durante 21 dias
BRF074-MntPL-28d Extrato de Aspergillus niger cultivado em MntPL durante 28 dias
BRF074-MPD-7d Extrato de Aspergillus niger cultivado em MPD durante 7 dias BRF074-MPD-14d Extrato de Aspergillus niger cultivado em MPD durante 14
dias
BRF074-MPD-28d Extrato de Aspergillus niger cultivado em MPD durante 28 dias
C Carbono não hidrogenado
C5D5N Piridina deuterada
CH Carbono metínico
CH2 Carbono metilênico
CH3 Carbono metílico
CHCl3 Clorofórmio
CH2Cl2 Diclorometano
CAPES Coordenação de Aperfeiçoamento de Pessoal de Nível Superior
CCD Cromatografia em Cama Delgada
CD3OD Metanol deuterado
CENAUREMN Centro Nordestino da Aplicação e Uso da Ressonância Magnética Nuclear
CH3CN Acetonitrila
CLAE Cromatografia Líquida de Alta Eficiência
COSY Correlation Spectroscopy
CPD Composite Pulse Decoupling
d Dupleto
DAD Detector De Arranjo De Diodos
dd Duplo dupleto
ddd Duplo dupleto dupleto
DEPT Distortionless Enhancement by polarization Transfer
DPR Desvio Padrão Relativo
DQOI Departamento de Química Orgânica e Inorgânica
dt Duplo tripleto
Ecotox Laboratório de Ecotoxicologia
EM Espectrometria de massa
EtOAc Ethyl acetate
HPLC High Performance Liquid Chromatography HSQC Heteronuclear Single Quantun Coherence
Hz Hertz
ICH International Conference on Harmonisation IDH Índice de Deficiência de Hidrogênio
IES Ionização por electrospray
IT Ion Trap
IV Infravermelho
J Constante de acoplamento
LaBBMar Laboratório de Bioprospecção e Biotecnologia Marinha LABOMAR Instituto de Ciências do Mar
LABS Laboratório de Biotecnologia e Síntese Orgânica LCMS Liquid chromatography - mass spectrometry
LEMANOR Laboratório de Espectrometria de Massa do Nordeste LG0949-SAHA Extrato do cultivo em grande escala de LG0949 em meio de
cultura BD com adição de SAHA na concentração de 200 µM LG0949-controle Extrato do cultivo em grande escala de LG0949 em meio de
cultura BD sem adição de SAHA
MeOH Metanol
MntPL Manitol Peptona Levedura MntPY Mannitol Peptone Yeast
MPD Malte Peptona Dextrose
m Multipleto
NPC Natural Products Center
ODS Octacedilsilano
PD Potato Dextrose
PDSE Programa de Doutorado Sanduíche no Exterior
PDY Potato Dextrose Yeast
p.f. Ponto de fusão
ppm Ponto de fusão
PTFE Politetrafluoretileno
qt Quarteto de tripleto
RMN 13C Ressonância Magnética Nuclear de Carbono-13
RMN 1H Ressonância Magnética Nuclear de Hidrogênio
s Simpleto
SAHA Ácido Hidroxâmico Suberoilanilida
sl simpleto largo
t Tripleto
td Triplo dupleto
tl Tripleto largo
TOF Time Of Flight
UFC Universidade Federal do Ceará UFLC Ultra Fast Liquid Chromatography UV-Vis Ultravioleta-Vísivel
1 INTRODUÇÃO ... 26 2 OBJETIVOS ... 31 2.1 Objetivos específicos ... 31 3 LEVANTAMENTO BIBLIOGRÁFICO ... 32 4 RESULTADOS E DISCUSSÕES ... 55 4.1 Otimização do cultivo de A. niger para a produção de metabólitos
secundários com atividade citotóxica ... 55
4.1.1 Cultivo em pequena escala em meio de cultura BD ... 55 4.1.1.1 Atividade citotóxica dos extratos do cultivo em pequena escala em meio de
cultura BD ... 56
4.1.2 Cultivo em pequena escala em meio de cultura BDL, MPD e MntPL ... 57 4.1.2.1 Atividade citotóxica dos extratos do cultivo em pequena escala em meio de
cultura BDL, MPD e MntPL ... 60 4.2 Determinações estruturais ... 61
4.2.1 Determinação estrutural de BRF074 (3) ... 62
4.2.2 Determinação estrutural de BRF074 (1) ... 72
4.2.3 Determinação estrutural de BRF074 (4) ... 77
4.2.4 Determinação estrutural de BRF074 (2) ... 83
4.2.5 Determinação estrutural de BRF074 (8) ... 88
4.2.6 Determinação estrutural de BRF074 (6) ... 94
4.2.7 Determinação estrutural de BRF074 (5) ... 104
4.2.8 Determinação estrutural de BRF074 (7) ... 114
4.2.9 Determinação estrutural de BRF074 (9) ... 124
4.2.10 Determinação estrutural de BRF074 (10) ... 133
4.2.11 Determinação estrutural de BRF074 (11) ... 142
5 DESENVOLVIMENTO E VALIDAÇÃO DE MÉTODO DE ANÁLISE
POR CLAE VISANDO A QUANTIFICAÇÃO DOS METABÓLITOS
SECUNDÁRIOS CITOTÓXICOS DE Aspergillus niger ... 151 5.1 Validação do método analítico ... 152
5.1.1 Linearidade ... 152
5.1.4 Exatidão ... 155 5.3 Aplicação do método - padrão de crescimento de Aspergillus niger
(BRF074) ... 156 6 EPIGENÉTICA ... 160 7 PARTE EXPERIMENTAL ... 168 7.1 Métodos cromatográficos ... 168
7.1.1 Cromatografia em camada delgada ... 168
7.1.2 Cromatografia por adsorção em fase reversa ... 168
7.1.3 Cromatografia líquida de alta eficiência (CLAE) ... 168 7.2 Métodos espectroscópicos ... 169
7.2.1 Espectroscopia na região do infravermelho ... 169
7.2.2 Espectroscopia de ressonância magnética nuclear ... 169
7.2.3 Espectrometria de massa (EM) ... 170 7.3 Métodos físicos ... 170
7.3.1 Rotação óptica ... 170 7.4 Micro-organismo ... 170
7.4.1 Obtenção e identificação ... 170
7.4.2 Preservação ... 171 7.5 Preparação dos meios de cultura ... 171
717.5.1 Esterilização ... 171
7.5.2 Água do mar sintética (AM) ... 171
7.5.3 Batata dextrose ágar (BDA) ... 171
7.5.4 Batata-dextrose (BD) ... 172
7.5.5 Batata-dextrose-levedura (BDL) ... 172
7.5.6 Manitol-peptona-levedura (MntPL) ... 172
7.5.7 Malte-peptona-dextrose (MPD) ... 172
7.5.8 Levedura malte - yeast malt (YM) ... 172 7.6 Produção de metabólitos secundários ... 172
7.6.1 Cultivo em meio líquido para obtenção dos extratos brutos ... 172
7.6.2 Procedimento geral de extração ... 173
7.6.3 Otimização do cultivo ... 173
7.6.6 Cultivo em grande escala de BRF074 em meio de cultura MPD ... 174 7.7 Estudo químico ... 175
7.7.1 Estudo químico do extrato BRF074-BD ... 175 7.7.1.1 Análises cromatográficas das amostras obtidas no fracionamento do extrato
BRF074-BD ... 175
7.7.1.2 Fracionamento em CLAE de BRF074-BD-F3 ... 176 7.7.1.3 Fracionamento em CLAE de BRF074-BD-F4 ... 177 7.7.1.4 Fracionamento em CLAE de BRF074-BD-F5 ... 178 7.7.1.5 Fracionamento em CLAE de BRF074-BD-F6 ... 179
7.7.2 Estudo químico do extrato BRF074-BDL ... 180 7.7.2.1 Análises cromatográficas das amostras obtidas no fracionamento do extrato
BRF074-BDL ... 180
7.7.2.2 Fracionamento em CLAE de BRF074-BDL-F3 ... 181 7.7.2.3 Fracionamento em CLAE de BRF074-BDL-F6 ... 181
7.7.3 Estudo químico do extrato BRF074-MPD ... 182 7.7.3.1 Análises cromatográficas das amostras obtidas no fracionamento do extrato
BRF074-MPD ... 183
7.7.3.2 Fracionamento em CLAE de BRF074-MDD-F5 ... 183 7.8 Teste citotóxico ... 184 7.9 Quantificação ... 185
7.9.1 Amostras ... 185
7.9.2 Análise quantitativa por cromatografia líquida de alta eficiência (análise por CLAE) ... 185 7.10 Epigenética ... 186
7.10.1 Cultivo em meio líquido para obtenção dos extratos brutos ... 186
7.10.2 Procedimento geral de extração ... 186
7.10.3 Metodologia geral de triagem ... 186
7.10.4 Cultivo em pequena escala utilizando diferentes concentrações de SAHA ... 187
7.10.5 Cultivo em grande escala de LG0949 em meio de cultura BD+SAHA ... 187
1 INTRODUÇÃO
Os produtos naturais possuem uma rica história no desenvolvimento de medicamentos. Muitas drogas sintéticas usadas na prática clínica foram desenvolvidas, ao menos parcialmente, a partir de fontes naturais. Há muito tempo, compostos farmacologicamente ativos têm auxiliado no tratamento de doenças humanas, podendo ser utilizados como antibióticos (penicilina), analgésicos (aspirina, opióides), imunossupressivos (rapamicina), dentre outras aplicações. (CHERBLANC et al., 2013; NEWMAN; CRAGG; KINGSTON, 2008).
O número de exemplos bem sucedidos, na química de produtos naturais microbianos, vem crescendo ao longo dos anos, com os micro-organismos sendo conhecidos por produzir metabólitos secundários com elevado potencial terapêutico, incluindo exemplos como as cefalosporinas (Cephalosporium), ciclosporinas (Trichoderma), rapamicinas (Streptomyces) e estatinas (Aspergillus e Penicillium) (FIGURA 1, p. 20) (CRAGG; NEWMAN, 2013; CHANG et al., 2011).
Figura 1 - Exemplos de fármacos obtidos a partir de micro-organismos
Fonte: CRAGG; NEWMAN, 2013.
cultivados em grande escala, tornando-se uma fonte ilimitada e contínua de matérias-primas necessárias para o desenvolvimento de drogas. Além disso, os produtos naturais microbianos representam um enorme e inexplorado recurso de estruturas químicas únicas, produzidas para fins de proteção, comunicação e em resposta a mudanças em seu habitat (BAILLY, 2009; GUNATILAKA, 2006; MARIENHAGEN; BOTT, 2013).
Dentre o universo dos micro-organismos, é importante ressaltar a química dos fungos marinhos, os quais têm sido encontrados associados a algas, plantas, invertebrados, moluscos, e ainda presentes em sedimentos de costões e regiões de mangue. (FELÍCIO; OLIVEIRA; DEBONSI, 2012). Em uma revisão bibliográfica realizada por Blunt e colaboradores (2014, p. 237), somente no ano de 2012, cerca de 217 novos compostos com atividades biológicas relevantes foram isolados a partir de fungos de origem marinha. Esse grande potencial na produção de metabólitos secundários bioativos pode ser explicado devido aos avanços obtidos no campo da biotecnologia, aliados ao emprego de técnicas modernas de
fracionamento químico, elucidação estrutural e “screening” na busca por novos protótipos (VIEGAS JR; BOLZANI; BARREIRO, 2006).
Em particular, os fungos provenientes de habitat marinho associados a sedimentos ou animais constituem uma fonte importante de novos produtos com atividades anticâncer, antibacteriana, antiplasmódica e anti-inflamatória (BHATNAGAR; KIM, 2010). Du e colaboradores (2007) isolaram um derivado de antraquinona denominado de aspergiolide A a partir de Aspergillus glaucus, que apresentou uma potente citotoxicidade contra a exposição das linhagens celulares K562 e P388. Gai e colaboradores (2007) revelaram a Fusarielin E como um novo agente antibiótico a partir de Fusarium sp. As dioxopiperazinas com potente atividade antibacteriana, desidroxibisdetiobis-metiltio-gliotoxina, bisdetiobis-metiltiogliotoxina e gliotoxina, foram relatadas a partir de fungo do gênero Pseudallescheria. Nenkep e colaboradores. (2010), descreveram o isolamento de benzoquinonas halogenadas, bromoclorogentisilquinonas A e B, com significativa atividade contra DPPH. Por outro lado, o isolamento da ahalimida, uma dicetopiperazina descoberta na década de 1990 que age como agente de despolimerização da tubulina, serviu como molécula-chave para a síntese do plinabulina, um análogo sintético que está sendo testado na fase I de ensaios clínicos no tratamento de câncer do pulmão (FENICAL et al., 1998; KINGSTON, 2009; AREN et al., 2010).
de Aspergillus fumigatus, que agem na fase G2/M do ciclo celular (ZHAO et al., 2002) (FIGURA 2, p. 29).
Figura 2 - Metabólitos bioativos obtidos a partir de fungos de origem marinha
Fonte: Elaborada pelo autor
Os estudos mais recentes relacionados ao entendimento do genoma microbiano revelam informações valiosas sobre os princípios da biossíntese de metabólitos secundários microbianos. Através da manipulação de fatores nutricionais e ambientais no cultivo do micro-organismo, a biossíntese dos metabólitos secundários produzidos pode ser completamente alterada, permitindo a produção de vários compostos a partir de uma única cepa. Essa particularidade, frequentemente citada na literatura como OSMAC (one strain many compounds), quando aliada ao uso de compostos denominados de modulares epigenéticos, permitem a indução ou inibição de certas rotas biosintéticas, oferecendo novas oportunidades para maximizar a diversidade química dos metabólitos produzidos e mostrando-se como alternativa promissora na descoberta de novas moléculas. (PARANAGAMA; WIJERATNE; GUNATILAKA. 2007; SCHERLACH; HERTWECK, 2009).
seriam capazes de controlar a expressão de genes silenciados e contribuir para o desenvolvimento de novas moléculas (WILLIAMS et al., 2008). Desde então, vários estudos similares têm sido descritos na literatura, os quais corroboram o emprego de moduladores epigenéticos como uma ferramenta promissora a produção de novos produtos naturais (CHERBLANC et al., 2013).
O Brasil é apresentado como um imenso patrimônio genético de valor econômico-estratégico inestimável, principalmente no campo do desenvolvimento de novos medicamentos. Com uma fantástica biodiversidade que abrange 20% do total de espécies do planeta, onde somente a Amazônia detém cerca de 26% das florestas tropicais remanescentes no mundo, e ainda contando com uma faixa litorânea com cerca de 7400 km, estima-se que o Brasil também possui uma grande diversidade microbiana, que ainda encontra-se totalmente desconhecida. Este patrimônio natural oferece grandes possibilidades para o desenvolvimento de pesquisas que propiciem a sua preservação e inovação, através da descoberta de novos compostos de interesse industrial. Como consequência, atualmente vários grupos de pesquisa nacionais estão investigando substâncias isoladas de algas, fungos e invertebrados marinhos e seu potencial contra várias doenças, como câncer, trombose, aids e outras (Ministério da Saúde, 2010).
Fungos do gênero Aspergillus são amplamente distribuídos na natureza e compreendem patogênicos humanos, animais e vegetais, além de fungos com uma gama de aplicações industriais. O gênero Aspergillus é rico em espécies, cerca de aproximadamente 250 já foram descritas na literatura, e estas espécies são capazes de produzir um grande número de extrólitos, incluindo metabólitos secundários, peptídeos/proteínas bioativos, enzimas, etc (BHETARIYA et al., 2011; FRISVARD; LARSEN, 2015; KANG et al., 2013).
Entre os vários grupos de fungos, as espécies do gênero Aspergillus são amplamente estudadas tanto do ponto de vista genômico quanto proteômico, visando desvendar os mecanismos biológicos e moleculares atuantes nesses fungos. Aspergillus fumigatus, Aspergillus flavus, Aspergillus niger, Aspergillus parasiticus, Aspergillus nidulans e Aspergillus terreus são algumas das espécies relevantes para aplicações humanas, agrícolas e biotecnológicas (BHETARIYA et al., 2011).
biotransformação, biorremediação e pré-tratamento de resíduos (NIELSEN et al., 2009; FRISVARD et al., 2011).
Figura 3 - Aspergillus niger
Fonte: Elaborado pelo autor
Estudos sobre a sequência genômica de A. niger revelaram a presença de 32 genes poliketídeos sintases (PKS), bem como 15 genes polipeptídios não ribossomais sintase (NRPS) e 9 genes híbridos PKS-NRPS, o que evidenciou a versatilidade e o potencial biossintético dessas espécies (CHIANG et al., 2011).
2 OBJETIVOS
Contribuir para o conhecimento químico do fungo Aspergillus niger BRF074
associado a amostra de sedimento proveniente da costa cearense e realização de experimentos de epigenética.
2.1 Objetivos específicos
Avaliar o potencial citotóxico de fungos provenientes de sedimentos da costa cearense frente à linhagem de células tumorais do carcinoma colorretal humano HCT-116 visando a seleção de uma cepa para produção de metabólitos secundários bioativos;
Otimizar as condições de cultivo microbiológico do fungo Aspergillus niger BRF074 em pequena escala variando-se os meios nutricionais e diferentes intervalos de tempo visando a produção de novos compostos bioativos;
Realizar fermentações em grande escala utilizando as condições dos experimentos que se mostraram promissores na produção de metabólitos secundários em pequena escala;
Isolamento e caracterização estrutural dos metabólitos secundários provenientes dos extratos produzidos nas fermentações em grande escala;
Investigar a atividade citotóxica de extratos, frações e metabólitos isolados empregando modelos "in vitro", com vistas à descoberta de novas moléculas potencialmente ativas;
Quantificação dos metabólitos secudários bioativos nos extratos provenientes das diferentes condições de cultivo de A. niger;
3 LEVANTAMENTO BIBLIOGRÁFICO
Uma revisão bibliográfica realizada por Nielsen e colaboradores (2009) acerca dos metabólitos secundários produzidos por A. niger revelou a presença de aproximadamente 60 compostos, pertencentes a diferentes classes estruturais, tais como: fumonisinas, nafto- -pironas, bicumarinas, malforminas e etc. Desta forma, tendo em vista a grande diversidade de metabólitos secundários, foi efetuada uma minuciosa busca visando uma atualização e complementação dos dados reportados.
A ferramenta de pesquisa empregada foi a base de dados Scifinder, que reúne informações registradas no Chemical Abstracts, utilizando-se como termos chave as palavras
“Aspergillus niger”, refinando os resultados para “secondary metabolites” e “natural products”, no período de 1λ66-2016.
Tabela 1 - Relação nominal dos metabólitos secundários identificados em Aspergillus niger e origem do micro-organismo
Composto Origem do
micro-organismo Referências bibliográficas Nafto--pironas (NGPs)
Aurasperona A (1) Fungo endofítico ZHANG; LI; WANG, 2007a
Solo AKIYAMA et al., 2003
Aurasperona B (2) Fungo marinho LEUTOU; YUN; SON, 2016
Fungo endofítico BOURAS et al., 2005
Aurasperona C (3) Fungo endofítico BOURAS et al., 2005
Aurasperona D (4) Fungo endofítico BOURAS et al., 2005
Aurasperona E (5) Fungo endofítico BOURAS et al., 2005
Aurasperona F (6) Fungo endofítico BOURAS et al., 2005
Aurasperona G (7) Fungo endofítico BOURAS et al., 2007
Aurasperona H (8) Fungo endofítico Li et al., 2016
Dianidroaurasperona C (9) Fungo endofítico ZHANG; LI; WANG, 2007a
Isoaurasperona (10) Fungo endofítico GHOSAL; BISWA;
CHAKRABARTI, 1979
Isoaurasperona A (11) Fungo marinho LI et al., 2016
Fungo endofítico LI et al., 2013 Isoaurasperona F (12) Fungo endofítico LI et al., 2013
Fonsecinona A (13) Fungo marinho LI et al., 2016
Fungo endofítico ZHANG; LI; WANG, 2007a
Solo AKIYAMA et al., 2003
Fonsecinona B (14) Fungo marinho LI et al., 2016
Fungo endofítico ZHANG; LI; WANG, 2007a
Fonsecinona C (15) Fungo marinho LI et al., 2016
Fungo endofítico ZHANG; LI; WANG, 2007a
Fonsecinona D (16) Fungo marinho LI et al., 2016
Fungo endofítico ZHANG; LI; WANG, 2007a
Asperpirona A (17) Fungo endofítico LI et al., 2013
Solo AKIYAMA et al., 2003
Asperpirona B (18) Fungo marinho LI et al., 2016
Solo AKIYAMA et al., 2003
Asperpirona C (19) Fungo marinho LI et al., 2016
Solo AKIYAMA et al., 2003
Fungo endofítico ZHANG; LI; WANG, 2007a
Asperpirona D (20) Fungo endofítico LI et al., 2013
Asperpirona E (21) Fungo endofítico LI et al., 2013
Nigerona (22) Fungo endofítico HAPPI et al., 2015
Nigerona ester metílico (23) - EHRLICH; DELUCCA;
CIEGLER, 1984 2-hidroxidihidronigerona (24) Fungo endofítico HAPPI et al., 2015
Nigerasperona B (25) Fungo endofítico ZHANG; LI; WANG, 2007a
Nigerasperona C (26) Fungo endofítico ZHANG; LI; WANG, 2007a
5,7-dihidroxi-2-[1-(4-metoxi-6-oxo-6H-
piran-2-il)-2-feniletilamino]-[1,4]naftoquinona (27)
Fungo endofítico ZHANG et al., 2007d
Nigerasperona A (28) Fungo endofítico ZHANG; LI; WANG, 2007a
TMC-256A1 (29) Fungo marinho LEUTOU; YUN; SON, 2016
Fungo endofítico FRISVAD et al., 2014
Solo SAKURAI et al., 2002
TMC-256C1 (30) Solo SAKURAI et al., 2002
Flavasperona (31) Fungo marinho LEUTOU; YUN; SON, 2016
Solo SAKURAI et al., 2002
6,9-dibromoflavasperona (32) Fungo marinho LEUTOU; YUN; SON, 2016
Fonsecina (33) Fungo marinho ZHOU et al., 2016
Fungo endofítico FRISVAD et al., 2014
Fonsecina B (34) Solo SAKURAI et al., 2002
Continuação
Tabela 1 - Relação nominal dos metabólitos secundários identificados em Aspergillus niger e origem do micro-organismo
Composto Origem do
micro-organismo Referências bibliográficas Nafto--pironas (NGPs)
Rubrofusarin (35) Fungo marinho ZHOU et al., 2016
Fungo endofítico GHOSAL; BISWA; CHAKRABARTI, 1979
Rubrofusarin B (36) Fungo marinho ZHOU et al., 2016
Solo SAKURAI et al., 2002
nor-Rubrofusarina (37) - TANAKA et al., 1966
Rubrofusarin-6-O-α-D-ribofuranosideo (38) Fungo endofítico LI et al., 2013 (R)-10-(3-succinimidil)-TMC-256A1 (39) Fungo endofítico LI et al., 2013
-pironas
Asnipirona A (40) Fungo endofítico LIU et al., 2011
Asnipirona B (41) Fungo endofítico LIU et al., 2011
Nafuredina (42) Fungo marinho UI et al., 2001
Campirona A (43) Fungo endofítico LI et al., 2015
Campirona B (44) Fungo endofítico LI et al., 2015
Campirona C (45) Fungo endofítico LI et al., 2015
Pirofeno (46) Fungo endofítico HAPPI et al., 2015
Fungo marinho VAROGLU; CREWS, 2000
Isopirofeno (47) Fungo endofítico ZHANG et al., 2010
Nigerapirona A (48) Fungo endofítico LIU et al., 2011
Nigerapirona B (49) Fungo endofítico LIU et al., 2011
Nigerapirona C (50) Fungo endofítico LIU et al., 2011
Nigerapirona D (51) Fungo endofítico LIU et al., 2011
Nigerapirona E (52) Fungo endofítico LIU et al., 2011
Nigerapirona F (53) Fungo endofítico LIU et al., 2011
Nigerapirona G (54) Fungo endofítico LIU et al., 2011
Nigerapirona H (55) Fungo endofítico LIU et al., 2011
Aspergillusol (56) Fungo endofítico ZHANG et al., 2010
Walterolactona A (57) Fungo endofítico LI et al., 2015 4-(hidroximetil)-5-hidroxi-2H-piran-2-ona
(58) Fungo endofítico HAPPI et al., 2015
4-hidroximetil-5,6-dihidro-piran-2-ona (59) Fungo endofítico LI et al., 2015
Bicumarinas
Bicumanigrina (60) Fungo marinho HIORT et al., 2004
Kotanina (61) Fungo marinho SCHLINGMANN et al., 2007
Demetilkotanina (62) Fungo marinho SCHLINGMANN et al., 2007
Funalenona (63) Solo INOKOSHI et al., 1999
Orlandin (64) Fungo marinho SCHLINGMANN et al., 2007
Azafilonas
Azanigerona A (65) - ZABALA et al., 2012
Azanigerona B (66) - ZABALA et al., 2012
Azanigerona C (67) - ZABALA et al., 2012
Azanigerona D (68) - ZABALA et al., 2012
Azanigerona E (69) - ZABALA et al., 2012
Azanigerona F (70) - ZABALA et al., 2012
Piranonogrinas
Piranonigrina A (71) Fungo marinho SCHLINGMANN et al., 2007
Piranonigrina B (72) Fungo marinho HIORT et al., 2004
Piranonigrina C (73) Fungo marinho HIORT et al., 2004
Piranonigrina D (74) Fungo marinho HIORT et al., 2004
Piranonigrina E1 (75) - YAMAMOTO et al., 2015
Piranonigrina E2 (76) - RIKO; NAKAMURA;
Continuação
Tabela 1 - Relação nominal dos metabólitos secundários identificados em Aspergillus niger e origem do micro-organismo
Composto Origem do
micro-organismo Referências bibliográficas Piranonogrinas
Piranonigrina F (77) - YAMAMOTO et al., 2015
Piranonigrina G (78) - YAMAMOTO et al., 2015
Piranonigrina H (79) - YAMAMOTO et al., 2015
Piranonigrina I (80) - YAMAMOTO et al., 2015
Piranonigrina J (81) - YAMAMOTO et al., 2015
Piranonigrina K (82) - YAMAMOTO et al., 2015
Piranonigrina S (83) Fungo marinho SCHLINGMANN et al., 2007
Ciclopeptídeos
Malformina A1 (84) Fungo endofítico LI et al., 2013
Malformina A2 (85) - KIM et al., 1993a
Malformina A3 (86) - KIM et al., 1993a
Malformina A4 (87) - KIM et al., 1993a
Malformina B1a (88) - KIM et al., 1993b
Malformina B1b (89) - KIM et al., 1993b
Malformina B2 (90) - KIM et al., 1993b
Malformina B3 (91) - KIM et al., 1993b
Malformina B4 (92) - KIM et al., 1993b
Malformina B5 (93) - KIM et al., 1993b
Malformina C (94) Fungo marinho ZHOU et al., 2016
Fungo endofítico MIKUŠUOVÁ et al., 2014
Beauvericin (95) - BOECKER et al., 2014
EnniantinaB (96) - BOECKER et al., 2014
Dicetopiperazinas
Asperazina (97) Fungo endofítico LI et al., 2015
Fungo marinho VAROGLU; CREWS, 2000
Asperazina A (98) Fungo endofítico LI et al., 2015
ciclo(D-Phe-L-Trp) (99) Fungo endofítico LI et al., 2015 ciclo-(L-Trp-L-Ile) (100) Fungo endofítico ZHANG et al., 2010 ciclo-(L-Trp-L-Phe) (101) Fungo endofítico ZHANG et al., 2010 ciclo(L-Trp-L-Trp) (102) Fungo endofítico LI et al., 2015 ciclo-(L-Trp-L-Tyr) (103) Fungo endofítico ZHANG et al., 2010
Derivados do ácido itacônico
Ácido hexilitaconico (104) Fungo marinho VAROGLU; CREWS, 2000
Ácido itaconico (105) - HOSSAIN et al., 2016
Ácido Tensyuico (106) Solo HASEGAWA et al., 2007
Ácido Tensyuico B (107) Solo HASEGAWA et al., 2007
Ácido Tensyuico C (108) Solo HASEGAWA et al., 2007
Ácido Tensyuico D (109) Solo HASEGAWA et al., 2007
Ácido Tensyuico E (110) Solo HASEGAWA et al., 2007
Ácido Tensyuico F (111) Solo HASEGAWA et al., 2007
Esteróides
Nigerasterol A (112) Fungo endofítico LIU et al., 2013 Nigerasterol B (113) Fungo endofítico LIU et al., 2013 (22E,24R)-5α,8α
-epidioxiergosta-6,22-dien-3-ol (114)
Fungo endofítico ZHANG et al., 2007b
(22E,24R)-ergosta-4,6,8(14),22-tetraen-3-ona (115)
Fungo endofítico ZHANG et al., 2007b
(22E,24R)-ergosta-5,7,22-trien-3-ol (116) Fungo endofítico ZHANG et al., 2007b (22E,24R)-ergosta-7,22-dien-3,5α,6-triol
(117)
Fungo endofítico ZHANG et al., 2007b
Ergosterimida (118) Fungo endofítico ZHANG et al., 2007b
Pigmentos
Continuação
Tabela 1 - Relação nominal dos metabólitos secundários identificados em Aspergillus niger e origem do micro-organismo
Composto Origem do
micro-organismo Referências bibliográficas Pigmentos
Asperenona (119) - JEFFERSON, 1967
Aspergillin (120) - RAY; EAKIN, 1975
Cicloleucomelon (121) Fungo marinho HIORT et al., 2004
Lipídeos
Ácido
14-hidroxi-13-(2-hidroxietoxi)-hentetetraconta-15,28-dienoico (122) Fungo marinho WU et al., 2008
Glicerolípideo (123) Fungo marinho WU et al., 2008
Asperamida A (124) Fungo endofítico ZHANG et al., 2007c
Asperamida B (125) Fungo endofítico ZHANG et al., 2007c
Asperiamida B (126) Fungo marinho WU et al., 2008
Asperiamida C (127) Fungo marinho WU et al., 2008
Ácido palmitico (128) Fungo endofítico SIDDIQUI et al., 2003 Ácido esteárico (129) Fungo endofítico SIDDIQUI et al., 2003
Micotoxinas
Fumonisina B2 (130) Fungo endofítico MIKUŠUOVÁ et al., 2014
Fumonisina B4 (131) - JØRGENSEN et al., 2011
Ochratoxin (132) - NIELSEN et al., 2009
Ochratoxin A (133) Fungo endofítico MIKUŠUOVÁ et al., 2014
Ochratoxin α (134) - SØRENSEN et al., 2009
Gliotoxina (135) - LEWIS et al., 2005
Yanutonas
Yanutona A (136) Fungo marinho BUGNI et al., 2000
Yanutona B (137) Fungo marinho BUGNI et al., 2000
Yanutona C (138) Fungo marinho BUGNI et al., 2000
Yanutona D (139) Fungo marinho BUGNI et al., 2000
Yanutona E (140) Fungo marinho BUGNI et al., 2000
Yanutona F (141) Fungo marinho BUGNI et al., 2000
Yanutona G (142) - HOLM et al., 2014
Yanutona H (143) - HOLM et al., 2014
Yanutona I (144) - HOLM et al., 2014
Yanutona J (145) - HOLM et al., 2014
Yanutona K (146) - PETERSEN et al., 2015
Yanutona L (147) - PETERSEN et al., 2015
Yanutona M (148) - PETERSEN et al., 2015
Yanutona X1 (149) - HOLM et al., 2014
Yanutona X2 (150) - PETERSEN et al., 2015
1-hidroxi-yanutona A (151) Fungo marinho BUGNI et al., 2000 1-hidroxi-yanutona C (152) Fungo marinho BUGNI et al., 2000 22-deacetilyanutona A (153) Fungo marinho BUGNI et al., 2000
7-deacetoxi-yanutona A (154) - HOLM et al., 2014
Terpenos
(1S,6S,10S)-6,10-dimetil
biciclo[4.4.0]dec-2-en-4-ona (155)
- PRIEGNITZ et al., 2015
(6S,10S)-6,10-dimetil
biciclo[4.4.0]dec-1-en-3-ona (156) - PRIEGNITZ et al., 2015 (8S,9R,10S)-8,10-dimetil-1-octalina (157) - PRIEGNITZ et al., 2015
2-metilisoborneol (158) - PRIEGNITZ et al., 2015
Geosmina (159) - PRIEGNITZ et al., 2015
Germacreno D (160) - PRIEGNITZ et al., 2015
Germacra-1(10)E,5E-dien-11-ol (161) - PRIEGNITZ et al., 2015
Asperrubrol (162) - RABACHE; NEUMANN;
LAVOLLAY, 1974
-copaeno (163) - PRIEGNITZ et al., 2015
Continuação
Tabela 1 - Relação nominal dos metabólitos secundários identificados em Aspergillus niger e origem do micro-organismo
Composto Origem do
micro-organismo Referências bibliográficas Terpenos
-ylangeno (164) - PRIEGNITZ et al., 2015
6,11-epoxi-isodaucano (165) - PRIEGNITZ et al., 2015
Outros
Aspernigerina (166) - SHEN et al., 2006
Aspernigrina A (167) Fungo marinho ZHOU et al., 2016
Atromentina (168) Fungo marinho SCHLINGMANN et al., 2007
Aspernigrina B (169) Fungo marinho ZHOU et al., 2016
Aspernigrina C (170) Fungo marinho ZHOU et al., 2016
Aspernigrina D (171) Fungo marinho ZHOU et al., 2016
FK17-P2a (172) - ZABALA et al., 2012
Antafumicina A (173) - FUJIMOTO et al., 1993
Antafumicina B (174) - FUJIMOTO et al., 1993
10,23-dihidro-24,25-desidroaflavinina (175) Fungo endofítico FRISVAD et al., 2014
Anominina (176) Fungo endofítico FRISVAD et al., 2014
Nigerloxina (177) - SURESHA; SRINIVASAN,
2013 (Z)-4-{[(Z)-1-(hidroximetil)-2-fenil-1-etenil]
amino}-4-oxo-2-butenoato de metila (178) - YUAN et al., 2006
Nigerazina A (179) Solo IWAMOTO et al., 1985
Nigerazina B (180) Solo IWAMOTO et al., 1985
Nigragillina (181) Fungo endofítico FRISVAD et al., 2014
Fungo marinho SCHLINGMANN et al., 2007
Averufina (182) Fungo marinho WU et al., 2008
Nidurufina (183) Fungo marinho WU et al., 2008
8,10,12-trihidroxi-9-metoxi-7α -metil-7,7α,1βα,13-tetrahidrobenzo
cicloheptaoxocin-6-ona (184)
Fungo endofítico ELFITA et al., 2012
Tensidol A (185) Fungo endofítico FRISVAD et al., 2014
Solo FUKUDA et al., 2006
Tensidol B (186) Fungo endofítico FRISVAD et al., 2014
Solo FUKUDA et al., 2006
Asperaldini (187) - SEKHAR et al., 2003
Metanoato de
6-formil-benzofuran-5-carboxila (188) Fungo endofítico SIDDIQUI
et al., 2003
Ácido 5,8-dihidroxioctadeca-9,12-dienóico
(5,8-diHODE) (189) - LEE et al., 2013
4-[(3R)-hidroxi]-3,5,5-trimetil-ciclohex-2-en-1-ona (190) Fungo endofítico SIDDIQUI et al., 2003
Ácido aspérico (191) Fungo marinho VAROGLU; CREWS, 2000
Anidrido 2-carboximetil-3-hexilmaleico
(192) - ALMASSI; GHISALBERTI; ROWLAND, 1994
Ácido 2-metileno-3-hexilbutanodióico (193) - ALMASSI; GHISALBERTI; ROWLAND, 1994
Agglomerin F (194) - YANG et al., 2014
Ácido carlósico (195) - YANG et al., 2014
Carlosiato de metila (196) - YANG et al., 2014
Ácido fenilacetico (197) Fungo endofítico NAIR; BURKE, 1988
Ácido fenoxiacetico (198) Fungo endofítico NAIR; BURKE, 1988
Ácido kojico (199) Fungo endofítico HAPPI et al., 2015
Continuação
Tabela 1 - Relação nominal dos metabólitos secundários identificados em Aspergillus niger e origem do micro-organismo
Composto Origem do
micro-organismo Referências bibliográficas Outros
2-feniletanol (202) Fungo endofítico WANI et al., 2010
4-[1-(4-hidroxifenil)-1-metiletil]-3-metilfenol (203) - SEKHAR et al., 2002
Ácido dihidrocarolico (204) Solo ALVI et al., 2000
Penitricina D (205) Solo ALVI et al., 2000
Ácido oxálico (206) - ANDERSEN; LEHMANN;
NIELSEN, 2009
Ácido cítrico (207) - ANDERSEN; LEHMANN;
NIELSEN, 2009
Ácido gluconico (208) - ANDERSEN; LEHMANN;
NIELSEN, 2009
Manitol (209) Fungo endofítico NAIR; BURKE, 1988
3-metil-8-hidroxi-4-decenoato de metila
(210) Fungo endofítico NAIR; BURKE, 1988
Oct-1-en-3-ona (211) - PRIEGNITZ et al., 2015
Oct-1-en-3-ol (212) - BORJESSON; STOLLMAN;
SCHNURER, 1993
2-metilpropan-1-ol (213) - BORJESSON; STOLLMAN;
SCHNURER, 1993
Dissulfeto de dimetila (214) - BORJESSON; STOLLMAN;
SCHNURER, 1993 Fonte: Elaborada pelo autor
Na Tabela 2 abaixo, encontram-se as respectivas estruturas químicas dos metabólitos secundários relacionados na Tabela 1 (p. 39).
Tabela 2 - Estruturas dos metabólitos secundários identificados em Aspergillus niger
Nafto--pironas (NGPs)
OH OCH3
H3CO
H3CO OCH3 O OH O CH3 O O CH3 (1) OH OCH3 H3CO H3CO OCH3 O OH O CH3 OH O O CH3 OH (2) OH OCH3 H3CO
HO OCH3
O OH O CH3 OH O O CH3 OH (3) OH OH
H3CO
H3CO OCH3 O OH O CH3 O O CH3 (4) OH OCH3 H3CO H3CO OCH3 O OH O CH3 O O CH3 OH (5) OH OCH3
H3CO
HO OCH3
O OH O CH3 O O CH3 OH (6) OH OCH3
H3CO
HO OCH3
O OH O CH3 O O CH3 HO HO (7) OCH3 H3CO O OH O CH3 O H3CO OCH3
OH
O
CH3
(8)
O CH3 O OCH3
H3CO
HO OCH3
Continuação
Tabela 2 - Estruturas dos metabólitos secundários identificados em Aspergillus niger
Nafto--pironas (NGPs)
O CH3 O OCH3 OH
H3CO
H3CO OH
OCH3
O O
CH3
(10)
O CH3 O OCH3
H3CO
H3CO OCH3
OH O O CH3 OH (11)
O CH3 O OCH3
H3CO
HO OCH3
OH
O O OH
H3C OH
(12)
O OCH3
H3CO
H3CO OCH3
OH O CH3 O OH O CH3 (13) O OH O OCH3
H3CO
OH
CH3
O H3CO OCH3
OH O CH3 (14) O O O OH CH3 OCH3 H3CO
OH
CH3
O OH OCH3 H3CO
(15)
OH OCH3
H3CO
H3CO OCH3 O OH O
CH3
O
H3C OH O
(16)
O OCH3
H3CO
HO OCH3
OH O CH3 O OH O CH3 (17) O O CH3 O OH OCH3 H3CO H3CO OCH3 OH O CH3 (18) O OH CH3 O OH OCH3 H3CO
H3CO OCH3
O CH3 O (19) O OH CH3 O OH OCH3
H3CO
HO OCH3
O CH3 O (20) O O CH3 O OH OCH3
H3CO
HO OCH3
OH O CH3
Continuação
Tabela 2 - Estruturas dos metabólitos secundários identificados em Aspergillus niger
Nafto--pironas (NGPs) O O O O OH OCH3
H3C H3C
OH OH (22) O O O O OH OCH3
H3C H3C
OCH3 OH
(23)
O
O CH3
O OH OCH3
OCH3 OH CH3 O OCH3 H3CO OH (24) OH H3CO
H3CO OCH3
OH OCH3 O O CH3 OH O
H3C OH
O
(25)
O CH3 O
H3CO
HO OCH3
OH
O O
CH3 OCH3 OH
OH (26) HN O O OCH3 O O OH OH (27) O O OH OH OCH3
H3CO
(28) O O CH3 OH OCH3 HO (29) O O CH3 OH HO OCH3 (30) O O CH3 OH H3CO OCH3 (31) O OCH3 Br
H3CO
Br OH O CH3 (32) OH HO OCH3 O O CH3 OH (33) OH
H3CO OCH3 O O CH3 OH (34) O
O CH3 H3CO
OH OH
(35)
O
O CH3 H3CO
OCH3 OH
(36)
O
CH3
Continuação
Tabela 2 - Estruturas dos metabólitos secundários identificados em Aspergillus niger
-pironas
H3C
CH3 O O OCH3 CH3 (40)
H3C
CH3 O O OCH3 (41) O CH3 CH3 CH3
CH3 O
OH O
H3C
(42)
O CH3 CH3 HN O CH3 H3CO O (43) O HN O CH3 H3CO
O
H3C CH3
(44) O HN O CH3 H3CO O CH3 CH3 (45) O O OCH3 NH O
H3C
(46) HN O CH3 O O OCH3 (47) O O OCH3 H3C
CH3
(48)
O O
OCH3 H3C
CH3 CH3 (49) O O OCH3 H3C
O CH3
(50) O O OCH3 CH3 H3C
O CH3
(51)
O O
OCH3 H3C
O CH3 (52) O O OCH3 CH3 CH3 CH3 (53) O O OCH3 CH3 CH3 (54) O O OCH3 H3C
CH3 (55) OH O OCH3 O (56) O O
Continuação
Tabela 2 - Estruturas dos metabólitos secundários identificados em Aspergillus niger
Bicumarinas
O
O OCH3
CH3 OCH3 O OH O CH3 HO (60) O O O
OCH3 CH3 OCH3
O OCH3 CH3
H3CO
(61)
O O CH3 OCH3
H3CO O
O OCH3 HO CH3 (62) HO CH3 HO
H3CO
O HO OH (63) O O CH3 HO OCH3 O OH CH3 O OCH3 (64) Azafilonas O O OH O O O CH3 O CH3 CH3 H3C
(65) O O O O CH3 O CH3 CH3 H3C
OH CH3 (66) O O O O CH3 O CH3 CH3 H3C
OH CH3 OH (67) N O OH O O O CH3 O CH3 CH3 H3C H (68) O O O HO CH3 OH CH3 (69) O O O O CH3 OH CH3 O
H3C
(70) Piranonogrinas O HN CH3 OH O O HO (71) O N CH3 OH O HO HO H3CO (72) O N CH3 OH O H3CO
HO HO