L')
UNIVERSIDADE FEDERAL DE UBERLÃNDIA CENTRO DE CIENCIAS BIOMÉDICAS
CURSODE CIENCIAS BIOLÓGICAS
Análise
da Segregação
Genérica
do
Cruzamento
entre
Phaseolus
vulgar/Zs
e Phaseolus
acufifo/ius
por
meio
de
Marcadores
Moleculares
e
Bioquímicos
Miriam
Gisele Gasparotto
Monografia
apresentada
à Coordenação do Curso- de Ciências Biológicas, da Universidade Federal de Uberlândia, para obtenção do grau de Bacharel em Ciências Biológicas.Uberlândia
—MGlà
UNIVERSIDADE FEDERAL DE UBERLANDIA CENTRO DE CIENCIAS BIOMÉDICAS
CURSODE CIÉNCIAS BIOLÓGICAS
Análise da
Segregação Genética
do
Cruzamento
entre
Phaseolus vulgaris
e
Phaseolus
acufifa/tbs
por
meio
de
Marcadores
Moleculares
e
Bioquímicos
Miriam
Gisele Gasparotto
Prof. Ph.D Luiz Ricardo GoulartFilho
(Orientador)
Monografia
apresentada
à Coordenação do Curso de Ciências Biológicas, da Universidade Federal de Uberlândia, paraobtenção
do grau de Bacharel emCiências Biológicas.
Uberlândia
— MG&
UNIVERSIDADE FEDERAL DE UBERLÁNDIA CENTRO DE CIENCIAS BIOMÉDICAS
CURSODE CIENCIAS BIOLÓGICAS
Análise
da Segregação
Genética
do
Cruzamento
enfre
Phaseolus
vulgaris
e Phaseolus acufifo/ius por
meio
de Marcadores
Moleculares e
Bioquímicos
Miriam
Gisele Gasparotto
Aprovada pela banca examinadora em
_l_/1998
Nota:7
Prof. Dr. Luiz RIcardo GoulartFilho
o—orientador)
Maraã/Qu
Prof. Dr. Warwick
EstevalrlKerr
(Co--orientador)
)
)
)
)))
))))))
)
))))))
)
)))))))
)
)
Ao meu noivoMarcos,
Que durante todos esses anos, apesar da distância, esteve aomeu lado
compartilhando problemas, respeitando minhas limitaçõeseaceitando minha ausência. A quem devograndeparte das
minhas conquistas e realizações.
))
)
))))))))))
))
"
Necessitamos de biólogos que nos digam o quepodemos edevemosfazer
para
sobrevivere o que não devemosfazer, se esperarmos manter emelhorara
qualidade de vida. Odestino domundo dependedaintegração,preservaçãoeextensão do conhecimento quepossui
um
número reduzido dehomens, que somenteagoracomeçam
a
sedarconta dopoder desproporcionado quepossuem e quão enormee'
a
tarefaa
realizar. “)
)
))
))))
)
)
agradecimentos especiais
Ao meuorientador Luiz,
Queme sempre incentivou acreditando naminha
capacidade, dando totalliberdade
para a
busca de novos conhecimentos, queapesardopoucotempodisponivel, sempreestevepronto
para
me atender eajudar.
A quem tenhograndeadmiração.
Aos meuspais,
Quemesmo sem conhecer este universoao
qual
faço
parte hoje, orgulharam-se do meutrabalho, ajudando-me superardificuldades
com suas orações e apoiando—menasminhas
escolhas. Com todo respeitoe carinho.
vii
agradecimentos
Agradeçoà Deus,
PelaVida, pela coragem, pelas realizaçõesepelas pessoas que até então cruzaram
omeu caminho,e quedequalquer maneira ajudaram-me subir os degraus rumoà
conquistados
meus objetivos.
Ao meuco—orientadorGismar,
Pela paciênciae atenção dispensada,o que muito contribuiu paraarealização deste trabalho.
Ao meu co-orientador Warwick Estevam Kerr,
Pelo simples exemplodeVida e de trabalho que nos passa, fazendo-nos acreditar
que o
sucesso pessoale profissional estãonabuscadenovos conhecimentos.
Ao meu irmão Júnioreminha cunhada Kristiane,
Pelo apoio,ereconhecimento da importância dessa conquista.
A minha irmã Bia,
Pela sinceridadede suas atitudes,e o carinhocom quemerecebeua cadavolta.
As minhas “irmãs” Andréia, Beatriz eCristiane,
Pela amizade, companheirismoepresença. Pelos momentos dificeise maravilhosos que
)
)
)
))
)
viii
Aos meus queridos amigos Gilberto, Isa, eRicardo
Pela confiançaeamizade sinceraque me dedicaramcom tantocarinho.
Aos meus amigos WarleieMarcelo,
Que sempre me ajudaram,seja pelas discussões científicas,oupela realizaçãodetarefas, a
quem devo muita admiraçãoe amizade.
Aos amigos do laboratório: Mauricio,Vivian, Elizângela, Bárbara, Graciele, Waldesse,
Rosana, Luciano, Kátia, Wanessa, Leonardo, André, Walter, Juliana, Wânia, Frederico, Ana Cândida, Ana Luiza, Adriana, SorayaeCristiane, pela amizadee momentosfelizes, os quais
deixarão muitassaudades.
Á
4lª
turma de Ciência Biológicas,Cristiane, Lisandra, Adriana, Katiere, Paula, Marta, Renato, Cristiana,Juliana, Évelin, RonaldoeLuciana.
AUniversidade Federalde Uberlândia,
SUMÁRIO
1 — INTRODUÇÃO
... 1
1.1 —A Cultura...
...
1
1.2— Genética
... 2 1.3—Cruzamentos lnterespecíficos ...
3 1.4—Marcadores Bioquímicos
e
Moleculares... 5 2— OBJETÍVOS
... 9 2.1 — Geral
... 9 2.2 —Específico...
9 3—MATERIAL E MÉTODOS
... 10 3.1 — Material Biológico
... 10 3.2—Análise Bioquímica
... 11
3.2.1 — Extração de Proteínas ...
11
3.2.1.1 — Faseolina ...
11
3.2.1.2 —Proteínas Solúveis em NaCl 0,5 M
...12 3.2.2— Eletroforese em Gel de Poliacrilamida
(SDS—PAGE)...12 3.3 —AnáliseMolecular
... 13 3.3.1 — Extração de DNA
... 13 3.3.2 —Dosagem do DNA
em Espectrofotômetro...14 3.3.3 —Amplificação do DNA via PCR pela Técnica
RAPD...14 3.3.4 —Eletroforese em Gel de Agarose
e
Visualização...15 3.4 —Estudos de Ligação por Análise
Bioquímica
e
Molecular...15 4— RESULTADOS... 16 4.1 — Faseolina ...
16 4.2 —Proteínas Solúveis em NaCl 0,5 M
... 16 4.3 —Marcadores Moleculares RAPD
... 19 4.4 — Estudos de Ligação...
21
4.5— Distãncia Genéticas ...
5
_
DISCUSSÓES... 24 5.1 — Faseolina
... 24 5.2 —Proteínas Solúveis em
NaCl 0,5 M
... 24 5.3—Marcadores Moleculares
... 27 5.4 —Estudos de Ligação
... 28 5.5 —Distância Genéticas
... 29 6 —CONCLUSÃO
... 31
7
-
REFERÉNCIAS BIBLIOGRÁFICAS... 32 8 —ANEXO
RESUMO
O feijão comum,
Phaseolus
vulgaris,representa
importante fonte de proteinaspara a
maioria da população depaíses
subdesenvolvidose
de baixa renda. Visando
essa
importância na alimentação humana, programas de melhoramento recorrem àcruzamentos entre
espécies
paraa
obtenção de cultivares maisadaptadas
à região de cultivo.Ofeijão—tepari,
Phaseolus
acutifolius,é
reconhecido como importante fonte deresistência a
seca, sendo
portanto um excelente genótipo para cruzamentos interespecificos com P. vulgaris.O monitoramento do
melhoramento clássico pela genética molecular pode reduzir
gastos e ganhar
tempoaté
o lançamento de novas cultivares.Com
a
intenção de avaliar a recombinação genética docruzamento entre P. vulgaris, cultivar lca Pijão
e
P. acutifo/ius, cultivar GL—O441, seguidos de dois retrocruzamentos com o pai recorrente P. vulgaris, avaliou-seas segregações
gênicas
em 39 indivíduos da progênie resultante R2C1.
Os resultados indicaram que uma
autofecundação
em R1C1 pode ter ocorrido,e
que possíveis falhas de pareamentoe
complexas interaçõesgênicas
ou cromossômicas estejam alterando a proporçãoesperada
3:1 de homozigotos :heterozigotos.
Dois grupos de ligação foram
construídos a partir dos marcadores bioquímicos e moleculares, por
este
mapa foi identificado ocorrência de1. INTRODUÇÃO
1. 1. A
Cultura
A cultura do feijoeiro,
é
de fundamental importância para o suprimento alimentar de grande parte da população mundial, principalmente em
países
subdesenvolvidose
de baixa renda, da AméricaLatina
e
África, onderepresenta
principal fonte protéica.O feijão
e
o4º
produto emárea
plantada,
e
o6º
em valor da produção agrícola no Brasil.Sua
importância social,como alimento substituto de proteínas
de feijão. Dentre eles, a quebra de tolerância
e
ou resistência.A
criação de novos cultivares, visando potencial produtivo elevado
e
adaptabilidade climática, é um dos principaiscomponentes
da estrutura de produçãodo feijão. A tecnologia
gerada através destes processos,
envolvendoanos
depesquisa e vastos
recursos financeiros, precisaser
levada aoagricultor, o que só poderá
ser
feito por meio dasemente
(VIEIRA,A variabilidade genética existente na natureza fornece elementos que
possibilitam a melhoria e
a adaptação
de uma determinadaespécie
a um determinado ambiente.Sendo
amplamente utilizada no melhoramneto genético, ela é abase
na qualse
fundamenta o trabalho de criação de novos cultivares(VIEIRA,
1988).
1. 2.
Genética
Segundo
Burkart (1952), o gêneroPhaseolus é
composto por aproximadamente 180
espécies,
distribuídasnas zonas quentes
de ambos hemisférios, mas com predominâncianas
Américas,sendo a
maioria diplóides (2n=22), de reprodução autogãmica, havendo raras exceções. Portanto, apresença
de diversas
espécies
silvestrese
a ampla distribuição geográfica que possui,o torna uma planta modelo para
estudos
de transferência genética,expressão
gênica,comportamento do genoma, mapeamento genético
e
evolução.A
espécie
mais cultivada do gêneroPhaseolus,
éo feijão comum
Phaseolus
vulgaris, amplamente distribuído e utilizado, além desua
importânciasocial, possui
características
como ploidiae
ciclo curto, que o torna, excelentebase
para o fluxo gênico comoutras espécies.
O P. vulgaris, em geral não
se adapta aos
trópicos úmidos, mas
cresce
bem emáreas
com chuvas regulares,desde
os trópicosaté as zonas temperadas
. É
muito sensível tanto
às geadas
quantoàs
altas temperaturas. Condições de
seca
duranteas épocas
críticas do florescimento ao enchimento da vagem,são
também muito prejudiciais.Da mesma maneira, o
excesso
de chuvacausa
aqueda
de florese aumenta
a ocorrência de enfermidades (ZlMMERMANN& TEIXEIRA, 1988).O feijão-tepari
(Phaseolus
acutifo/ius), tem distribuiçãolimitada,
e é
cultivado com alguma intensidadenas áreas
doSudoeste
da América do Norte. Verifica-se na literatura, queesta espécie
tem uma resistência considerável àseca, e
caracteriza-se
porapresentar
folhase sementes pequenas e
rendimentos muito baixo,mesmo
sob
condições favoráveis, quando comparada com o feijoeirocomum (P. vulgaris)
DJ
1. 3.
Cruzamentos lnterespecíficos
A exploração de
espécies
afins de plantas cultivadas e'uma
das
maneiras dese
introduzirgenes
adicionais emvariedades
cultivadas. Ahibridação
é
um importante instrumentopara
o melhoramento deespécies
de plantas cultivadas.A
variabilidade pode
ser
obtida tanto por meio da hibridação intervarietal (métodos convencionais), quanto entre diferentesgêneros
(intergenéricas), ou entre diferentesespécies
(interespecificas) (PRESTES & GOULART, 1995).Os cruzamentos de P. vulgariscom P. acutifo/ius,
apresentam
resultados ricos em informaçõesgenéticas
para fins agronômicos, morfológicos, bioquímicos e moleculares. Do ponto de vista básico,estas
informações têmsido utilizadas para
especulações
da origem, isolamento geográfico, compatibilidade de cruzamento e outros,e são
aplicados diretamente na orientação em programas de melhoramento.Segundo
Mejía-Jiménezet
al. (1994), osucesso
nas transferências degenes
da maioria
das
caracteristicas deinteresse
de teparipara
feijão comum, tem sido
poucas vezes
realizadas,e
entreas causas
paraesse
limitadosucesso
inclui: uso de poucos genótipos deambas espécies
para hibridação; ocorrência de híbridos F1 letais,associados a
certos genótipos deambas espécies;
ocorrência de abortodo embrião; esterilidade do híbrido e avaliação inadequada da progênie híbrida interespecifica.
A realização de cruzamentos entre
espécies
dogênero Phaseolus, tem sido
usado
como alternativa para incorporar ao feijão comum (P. vulgaris)determinadas
características
agronômicas importantes, como resistência adoenças e pragas e
resistência a
seca,
queestão presentes
nas demaisespécies
cultivadas do gênero. Sobre outroaspecto
a realização de tais cruzamentos,serve
para
estudar
a relação evolutiva entreespécies
(SMARTT, 1970).Crocomo & Cabral (1985)
destacaram a espécie
P.acutifo/ius como uma importante fonte de tolerância ao
estresse
hídricoe
térmico,e
resistente à algumasdoenças.
Thomas & Waines (1984) relataram a importância daespécie
em hibridação interespecifica com P. vulgaris, como fonte de tolerância aoestresse
hídricoe
a altas temperaturas.dificuldades de
sucesso
na hibridação. Astentativas de transferência de caracteristica para o feijão comum por hibridação interespecifica, não têm
sido bem sucedidas, devido
aos
diferentes níveisde
incompatibilidade(ClAT, 1990).
Guo
et
al. (1989) citam o melhordesempenho
de híbridos
resultantes
de retrocruzamentos entrePhaseolus
coccineus e P. vulgaris,quando
este
último foiusado
como pai recorrente. Da mesma forma,Mejía-Jiménez
et
al, (1994), relatamser
gradativamente dispensável ouso de cultura de embriões, para
resgatar
híbridos entre P. vulgarise
P. acutifo/ius, a medida que retrocruzamentoscom P. vulgaris recorrente eram realizados.
O principal procedimento para
a
transferência direta degenes e
por meio damanipulação sexual. Após a produção de híbridos faz-se o retrocruzamento com o genitores de
caracteres desejáveis
(GOULART, 1990).O método de retrocruzamento, e'
frequentemente empregado em cruzamentos divergentes, visando elevar
a
frequência degenes
favoráveis na população. Ele pode também
ser
aplicado naadaptação
de germoplasmaexótico, reduzindo
sua
contribuição genética na formação de populaçõessegregantes.
Mejia-Jiménez
et
al. (1994), citam que problemas com esterilidade dohíbrido F1, poderiam
ser
vencidos parcialmente pelo retrocruzamento recorrente com feijão comum, mas isso reduz a possibilidde de reter
genes
deinteresse
dotepari.O comportamento citogenético, pode muitas
vezes estar associado
a viabilidade do híbrido, onde o pareamento entre cromossomosé consequência
da uma complexa interação entre genes. Este
pareamento é essencial
para osucesso
de cruzamentos interespecificos (PRESTES & GOULART, 1995).Os efeitos deletérios dos
genes
envolvidos em cruzamentos entreindivíduos que não pertençam à mesma
espécie,
podemser
eliminadosse
os cromossomos forem homólogos, pois há recombinação, entretanto
se
os cromossomos forem homeólogos ou não homólogos, tais efeitos tornam-se difíceisde serem eliminados (PRESTES & GOULART, 1995).
caracteres
importantes de fontes primáriase secundárias
de genes,
são
alguns dos desafios que devemser
resolvidos (ClAT, 1990). Porém,muitas
das
limitaçõesapresentadas
àpesquisa
não podemser
resolvidas pelaexigindo assim, a aplicação de técnicas mais
avançadas
da biotecnologia (GOULART, 1996).1. 4.
Marcadores Bioquímicos e Moleculares
Os
marcadores
bioquímicos obtidos sejam por umareação
enzimática diferenciada entre indivíduos, ou por uma proteína constitutivapolimórfica,
são
caracteres
deinteresse
emestudo
de genéticae
evolução como:estudos de
diversidade genética,estudos
de ligaçãoassociadas
a características importantese
outros.
Dessa
forma, osmarcadores
bioquímicos, juntamente com outros têm contribuído para elucidação de vários
estudos
genéticos (VlElRA, 1997).O centro de origem do feijoeiro
comum (P. vulgaris)
e
do feijão-tepari (Pacutífo/ius) e' conhecido como Mesoamericano, compreendendoo sul do México,
e
América Central (VlElRA, 1967). Os primeirosautores
a estudarem os centros de origem do feijão,
basearam-se
emcaracterísticas
morfológicas distintas e comuns
observadas nas
plantas. A partir dadécada
de 70, o usode marcadores
bioquímicos começa a
ser
utilizado, adicionando informações para diferenciar os centros de origem de cultivarese espécies
do gêneroPhaseolus.
A faseolina
está
diretamente envolvidacom o centro de origem no
gênero
Phaseolus.
Dois centros conhecidos, denominados mesoamericanoe
andino, paraos quais há um padrão de faseolina conhecido
e
distinguível.A faseolina
é
um marcador bioquímicobastante
utilizado nosestudos
genéticos em feijoeiro comum. Ela é a principalproteína de
reseva
dasemente
de feijoeiro comum,e representa
(BG—46% dototal de proteína dasemente
(MA & BLlSS,1978).
Estudos genéticos envolvendo cruzamentos, mostraram que os
genes
que codificam os diferentes polipeptídeos componentes da faseolinaestão
intimamente ligados, formando um bloco gênioo, queé
herdado emcaráter
deherança
simples (ROMEROet
al., 1975)A biologia molecular tem sido uma
ótima ferramenta
para estudos
de diversidade genética. Informaçõessobre
oparentesco
oua
similaridade genética entre cultivares dentro de um banco de germoplasma, tem aplicações importantesA tecnologia da PCR
(Reação
em Cadeia da Polimerase), surgiu na
década
de 80,
e sua concepção
causou um verdadeira revolução nabiologia. A facilidade,
rapidez, versatilidade e sensibilidade da PCR, a torna
poderosa para estudos
genéticos moleculares envolvendo grande número de indivíduos de qualquer organismo vivo.A PCR
é
caracterizada porum ciclo de 3etapas: desnaturação,
aneiamentoe
extensão. A fita dupla de DNA alvo e'desnaturada,
a seguir ocorre o aneiamento deprimers,
e
por final, a enzima DNA polimerase sintetise nova fita,pela adição de nucleotídeos. Este ciclo e' repetido muitas
vezes
de formaa
produziro DNA alvo em
progressão
geométrica.A partir de 1990, um nova técnica PCR-RAPD
(Random Amplified PolymorphicDNA), foi desenvolvida com a idéia de
se
utilizar primers curtos(10 pb), de
sequência
arbitrária (WILLIAMS,et
al., 1990). Aprincipal vantagem da técnica
PCR—RAPD
é
a de nãorequerer
informaçõesgenéticas
prévias dasequência
deDNA (TlNGEY,
et
al., 1992).Além
dessa
importante caracteristica, Williams,
et
al., (1990), emseu
trabalho
destacam
comograndes vantagens
a rapideze
simplicidade da técnica,
e
a utilização dequantidades
mínimas de DNAnecessárias para
análise genômica de um indivíduo.Uma característica da técnica PCR-RAPD,
é
o fato dese
comportarem comomarcadores
genéticos dominantes, isto é, o indivíduoheterozigoto não é diferenciado do homozigoto dominante
para
amplificação. Éusada
em umavariedade de aplicações incluindo: construção de
mapas
genéticos, estabelecimento de relacionamentos filogenéticos, análise de estrutura e diversidade genética em populações, obtenção de fingerprints, marcas molecularesassociadas
àgenes
de interesse.Os
marcadores
molecularessão
aplicados em programas de melhoramento para: reduzirem o número de retrocruzamentos,e
ou cruzamentos dialélicos, ligações agenes
de resistência, controle de qualidade por fingerprints,incorporação de
genes,
e monitoração a introduções degenes
de plantas silvestres. A utilizaçãode
marcadores
molecularesapresenta
váriasvantagens
sobremarcadores
morfológicos convencionais, tais como: exibem neutralidade fenotípica sem influência ambiental;são herdados
em dominânciaexibem interações epistáticas ou pleiotrópicas, podendo
ser
detectado tanto em tecidos jovens como adultos,apresentam
resultados positivos quanto ao melhoramento decaracterísticas
quantitativas (BECKMAM &SOLLER, 1983).
A técnica RAPD utiliza um primer,
e este
tem
sequência
arbitrária de nucleotídeos. Os marcadores comportam—se com dominância, havendo a possibilidade dese
comportarem com codominãncia (individuos 2n). Os marcadores RAPDapresentam-se
de maneira muito eficienteno mapeamento de polimorfismo ligados
a
locos de resistência adoenças
(FERREIRA &GRATTAPAGLIA, 1996). Os
marcadores
moleculares podem maximizar a eficiência dos programas de retrocruzamentos, aumentando a probabilidade de conversão dos indivíduose
reduzindo o tempo requerido para obter umarecuperação
aceitável doprogenitor recorrente (BORÉM, 1997). Diversas aplicações de marcadores moleculares em melhoramento genético podem
ser
distribuídas em aplicações cujos resultadosapresentam
expectativas de curto, médioe
longoprazo. (FERREIRA &
GRATTAPAGLIA, 1996).
A proporção do genoma que pode
ser
monitorada commarcadores
molecularese
em função doseu
númeroe
dasua
distribuiçãono genoma.Para as
espécies
que dispõem demapas
commarcadores
que saturamo genoma em intervalos menores que 20 CM seria teoricamente possível selecionar
um individuo praticamente idêntico ao genitor recorrente com a avaliação de grande número de progênies da primeira
geração
de retrocruzamentos (BORÉM, 1997).As aplicações de curto prazo envolvem basicamente, a identificação
e
discriminação de genótipos. Nas aplicações de médio-longo prazo, os marcadores permitem quantificar a variabilidade genética ao nivel de
sequência
de DNAe
correlacioná-la com aexpressão
fenotípica em procedimentos de mapeamento genético (FERREIRA & GRATTAPAGLIA, 1996).A
análise
genética ao nivel de DNAestá se
tornandocada
vez mais eficiente. Com isso, uma grande quantidade de informaçõessobre
genomas de plantascultivadas
estão sendo geradas.
As perspectivassão
de queas
próximasdécadas
assistirão a uma explosãodeste
conhecimento, abrindo inúmeras oportunidadespara a
exploração mais eficiente da variabilidade genética existente tantonas
variedades
cultivadas como no germoplasma silvestre. Hoje,a identificação
e
localização degenes
de importância econômicaé
um objetivo)
)))))
)
)
nas
culturas tradicionais maisestudadas
e manipuladas geneticamente, mas também em
espécies essencialmente
nãodomesticadas
)
)))))
)
2. OBJETIVOS
2. 1. Geral
-Ana|isar
dasegregação
gênica resultante do cruzamento entreas espécies
Phaseolus
vulgarise Phaseolus
acutifolius.2. 2.
Específico
-Determinar
a divergência genética entreas espécies Phaseolus
vulgaris
e
Phaseolus
acutifolius, e caracteriza-Ia bioquímicae
molecularmente;- Realizar
estudos
de ligação entre osmarcadores
10
3. MATERIALE MÉTODOS
O
presente
trabalho foi realizado no Laboratório de GenéticaMolecular,
pertencente
ao Departamento de Genéticae
Bioquímica, da Universidade Federalde Uberlândia,no periodo de junho de 1997
a
junho de 1998.3. 1. Material
Biológico
Para
a realizaçãodeste
trabalho, foram obtidas junto ao CNPAF(Centro Nacional de
Pesquisa
de Arroze
Feijão, Goiãnia — GO ),sementes
de
Phaseolus
vulgaris (Feijão comum), cultivar lca Pijão,
Phaseolus
acutifo/lus (Feljão-tepari), Série GL-O441,e
uma progênie de 39 híbridos resultante de dois retrocruzamentos entreas espécies,
ondea espécie
P. vulgaris foi utilizadacomo genitor recorrente,
e
P. acutifolius como genitor doador ( Tabela 1).As
sementes
dos genitorese
a progênie R2C1 foram divididas em dois lotes comduas sementes
cada,sendo
umdeles usado
para análise eletroforética
das
frações
protéicase
o outro plantado emcasa
de vegetação, para o desenvolvimento11
Tabela1.Relação das espéciesehíbridos analisados
Genítores
Phaseolus vulgaris lcaPijão
Phaseolus acutlfJ/ius GL-0441
Progênie
chl
3262 3312 10867
10881
3267 10858 10868 10882
3272 10859 10869 10883 3277 10860 10871 10884
3282 10861 10872
10885
3287 10862 10874 10886 3292 10863 10875 10888 3297 10864 10877 10889 3302 10865 10878 10890
3307 10866 10880
3. 2.
Análise Bioquímica
3. 2. 1.
Extração de Proteínas
A principio retirou—se o tegumento
e
o embriãodas sementes,
os cotilédones foramentão macerados
em gral de porcelanaaté
a formação de um pó fino, que foi transferido e tubos de eppendorrf, para a realização da extração
das
frações protéicas.Estas
foramarmazenadas
emfreezer
—80ºCpara posterior análise eletroforética.
3. 2. 1. 1.
Faseolina
O procedimento para a extração de faseolina,
foi
segundo
o protocolo descritopor Romero
et
al. (1975), com modificações no tempo de extração.Foi adicionado a
60 mg de macerado, 1,2 ml de solução de extração
(NaCl 0,5 M, ácido ascórbico
0,25 M, pH 2,4). O extrato
permaneceu
em12
protegido de luminosidade, para a extração da proteína, em
seguida
foi centrifugadoa 13.000rpm por 20 minutos,
a
4ºC. Osobrenadante
foicoletado,
e
a ele adicionado 5xseu
volume deágua
desionizadagelada
(padronizou—sea retirada de 250 pl do
volumetotal do
sobrenadante
para quea
extraçãofosse
realizada em microtubos de 1,5 ml). Estaetapa
foi repetida porduas vezes para a
eliminação do ácidoascórbico,
e
o precipitadofinal obtido foi solubilizadoem 250 ul de NaCl 0,5 M.3. 2. 1. 2.
Proteínas Solúveis em
NaCl 0,5 M As proteínas foram extraídas de acordo com oprotocolo descrito por Echeverrigaray
et
al. (1993). Junto a 20 mg de macerado foi adicionado 200ul de
solução NaCl 0,5 M,
e
mantido sob agitação constante,a
4ºC. O extrato foientão centrifugado a 10.000rpm por 20 minutos,
e
osobrenadante
recolhido.3. 2. 2.
Eletroforese em
Gelde Poliacrilamida Desnaturante
(SDS-PAGE)
As
amostras
de proteínas foramdesnaturadas e dissociadas
em
seus
constituintes peptídeos, por aquecimento a 100ºC por 5 minutos em
presença
de tampão da amostra 2X SDS buffer (0,125 M Tris—HCl, pH = 6,8, SDS4%, glicerol 20%, B-mercaptoetanol 10 %), (LAEMMLI, 1970).
As
subunidades
foramseparadas
por eletroforese emgel de poliacrilamida
desnaturante
(SDS-PAGE),segundo
Laemmli (1970) na concentração de10%. As
dimensões
dos géis foram de 16 cm x 17 cm x 0,75mm.Como solução tampão, para a corrida eletroforética, foi utilizado Trizma
base
0,1M; EDTA7,8 mM; glicina 0,77 M e SDS 0,3% pH 8,3.
As eletroforeses foram conduzidas
a
20 mApor aproximadamente 5 h, tanto para a faseolina como para a fração solúvel em NaCl 0,5M.
Após
esta
etapa, os géis foramcorados
em solução de Coomassie Brilhant Blue R 250 (50% metanol; 0,125% Coomassie; 10%ácido acético), porum periodo de 12 h,
então
foram submetidosà
solução dedescoloração
(10%ácido acético), para retirada do
excesso
de corante, e por fim fixados em solução fixadora (ácido acético 7%).Os géis foram
secados baseado
no método dePopescu
(1983),adaptado para
secagem
em bastidores, onde eram envolvidos entreduas
folhas de papel celofaneporoso, e
esticados
no bastidor,permanecendo
assim por 24 h, aproximadamente.3. 3.
Análise
Molecular3. 3. 1.
Extração de
DNAO DNA genômico, foi extraído de acordo com o
procedimento descrito por Vilarinhos
et
al. (1994), com algumasadaptações
para microextração em microcentrífuga. Inicialmente folhas jovens foram coletadas, logo em
seguida
pesados
200 mg, emaceradas
empresença
de nitrogênio líquido. O macerado foitransferido para microtubos de 2 ml, onde foi adicionado 1 ml de tampão de extração
(Tris-HCI 100mM pH 8,0; EDTA 50 mM pH 8,0; NaCl 1 M; 1,5% de CTAB; 1% de
B-mercaptoetanol),
e
incubadas em banho-maria à temperatura de 65ºC por 60 a 90 minutos aproximadamente, fazendoagitações suaves
acada
15 minutos.Para
oprocesso
de desproteinização, 600 ul de clorofórmio—álcool isoamílico (24:1), foiadicionado ao tubo, realizando leves
agitações
por inversão, durante 5 minutos. Os microtubos foram centrifugados por 15 minutos a 4000 rpm. Osobrenadante
foi
transferido para um novo tubo,
e
submetidoà
uma novaetapa
de desproteinização com 900 ul de clorofórmio-álcool isoamílico(24:1), sob a mesma condição anterior. Aosobrenadante
final, foi adicionado 2/3 deseu
volume de isopropanol gelado,e
os tubos deixados emfreezer
o tempo suficiente paraa
precipitação do DNA. A seguir, foram centrifugados a 10000 rpm por 10 minutos, onde afase aquosa
foi removida,e
o precipitado incubado com etanol 80% a 37ºC por 20 minutos.Passada esta
etapa, o DNA foisecado
a vácuo eressuspendido
em 200 ul de tampão TE(Tris—HCI
10mM pH 8,0
e
EDTA1 mM pH 8,0) contendo RNAsena
concentração
de 10 ug/ml,14
3. 3. 2.
Dosagem do
DNAemEspectrofotômetro
O DNA foi quantificado por leitura de
absorbância
a 260 nm emespectrofotõmetro, Sambrook
et
al., (1989), utilizando uma alíquota de 10 D.] decada
amostra diluída100 vezes. A qualidade do DNA foi avaliada
submetendo-se
10 ul daamostra a eletroforese em gel de
agarose a
0,8%. Apósa
avaliaçãoas
amostra foram diluídas emágua
ultrapura, emconcentração
para trabalho de 10ng/ul.3. 3. 3.
Amplificação do
DNAvia PCRpela Técnica
RAPDA PCR é
caracterizada
por um ciclo de 3etapas: desnaturação,
anelamento e extensão. A fita dupla de DNAé desnaturada à
aproximadamente 95ºC, emseguida
a temperatura cai em torno de 45ºC, onde ocorre o anelamento de primers,
e
por final, ha' um novo aumento de temperatura para 72ºC, permitindo que a enzima DNApolimerase sintetiza nova fita, pela adição de nucleotídeos. Este ciclo
é
repetidomuitas
vezes
de forma a produziro DNA alvo emprogressão
geométrica.A
reação
emcadeia
da polimerase foi realizada de acordo com Williamset
al.(1990), com algumas modificações. Foram
testados
4 primers desequência
arbitrária com 10 pb de comprimento, da OPERON Technology (Tabela 2). Cadareação
de amplificação continha: 1,5 unidade de Taq DNA polimerase, tampão dereação
1X (10 mM Tris—HCI pH 8,0; 50 mM de KCl; 2,0 mM de MgCl2), 100 mM decada
(dCTP, dATP, dGTPe
(FTP), 8 pmoles decada
oligonuceotídeo (primers),e
20 ng de DNA genômico, completando comágua
ultrapura para um volume final de25ul.
As
reações
ocorreram em termociclador MJResearch
lNC, modelo PTC—100, programadopara
3 ciclos iniciais de 94ºC/1 min., 35ºC/2 min.e
72ºC/2 min., mais 34Tabela2.Sequência dos primers testados para análise de
divergência genética entreP. vulgaris e P. acutifo/íus
Primer Sequência(5'
ª
3')GPB-10
GPB—11
CPB-18
OPS ]]
CTGCTGGGAC GTAGACCCGT CCACAGCAGT AGTCGGGTGG
3. 3. 4.
Eletroforese em
Gelde Agarose e Visualização
dos
Fragmentos
Os produtos amplificados foram
corados
com brometo de etídeo (0,2 ug/ml), visualizados em transluminador de luz ultravioleta,e
fotografados em lmageMaster— VDS. Como suporte para
a separação das
regiões amplificadas foi utilizado gel de
agarose
1,2%e
tampão de gel de eletroforese de TBE 0,5X (Tris-HCl, EDTAe
ácido bórico), (SAMBROOKet
al., 1989).O tempopara a
corrida eletroforética,foi de 2 a 3
h,
até
queas bandas estivessem
nitidamenteseparadas
no gel.3. 4.
Estudos de Ligações por Análise Bioquímicae
Molecular
Por meio do padrão de
segregação
genética da progênie, umamatriz de
dados
foianalisada
considerandopresença
eausência
de bandas.
Para
acaracterização
dos grupos de ligação,e
percentual da distância genética dosindivíduos, foi utilizado o programa Mapmaker
e
“Análise de16
4.
RESULTADOS
4. 1.
Faseolina
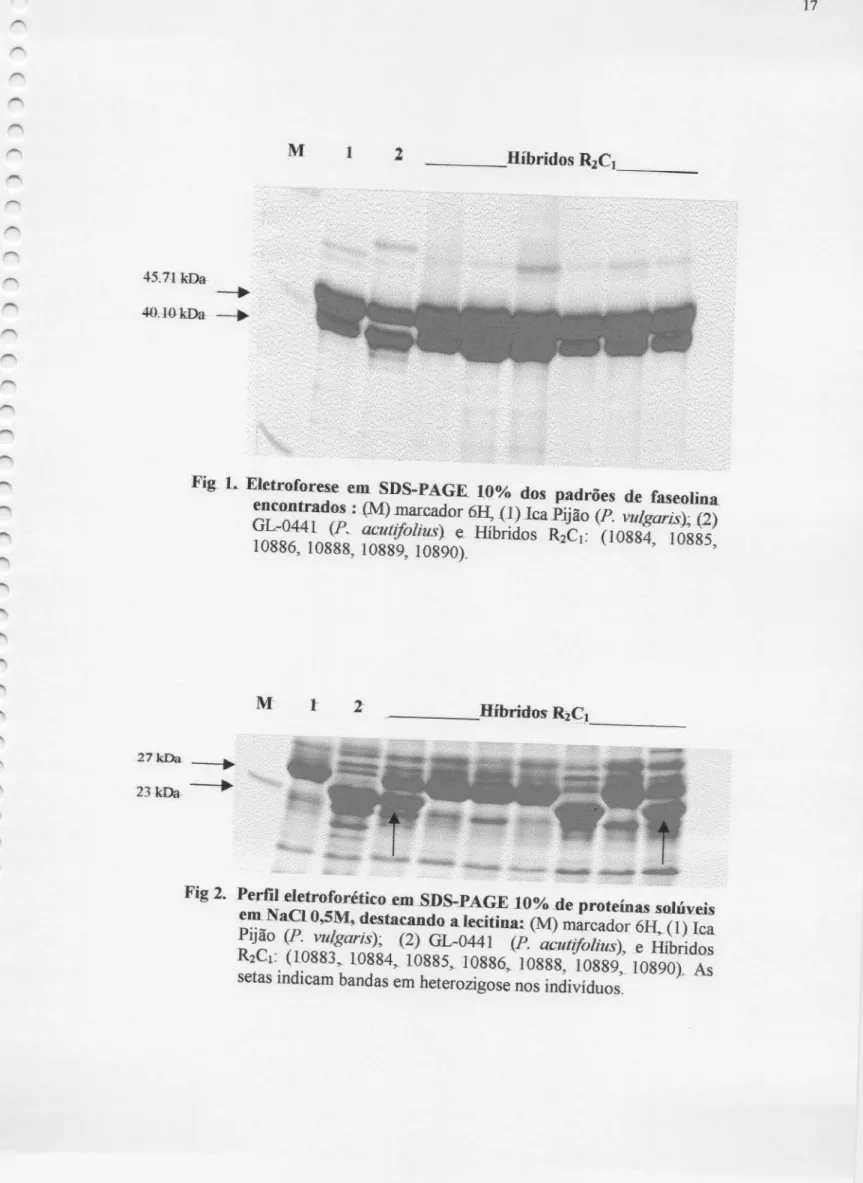
O perfil eletroforético da faseolina para os genitores
lca Pijão
e
GL—O441, nãoapresentou
diferenças. Duasbandas
depesos
moleculares45.71 kDa
e
40.10 kDa, foram identificadas em ambos. Os híbridos R2C1, da mesma forma, obtiveramo mesmo padrão de
bandas
encontrado nos genitores (Figura1).
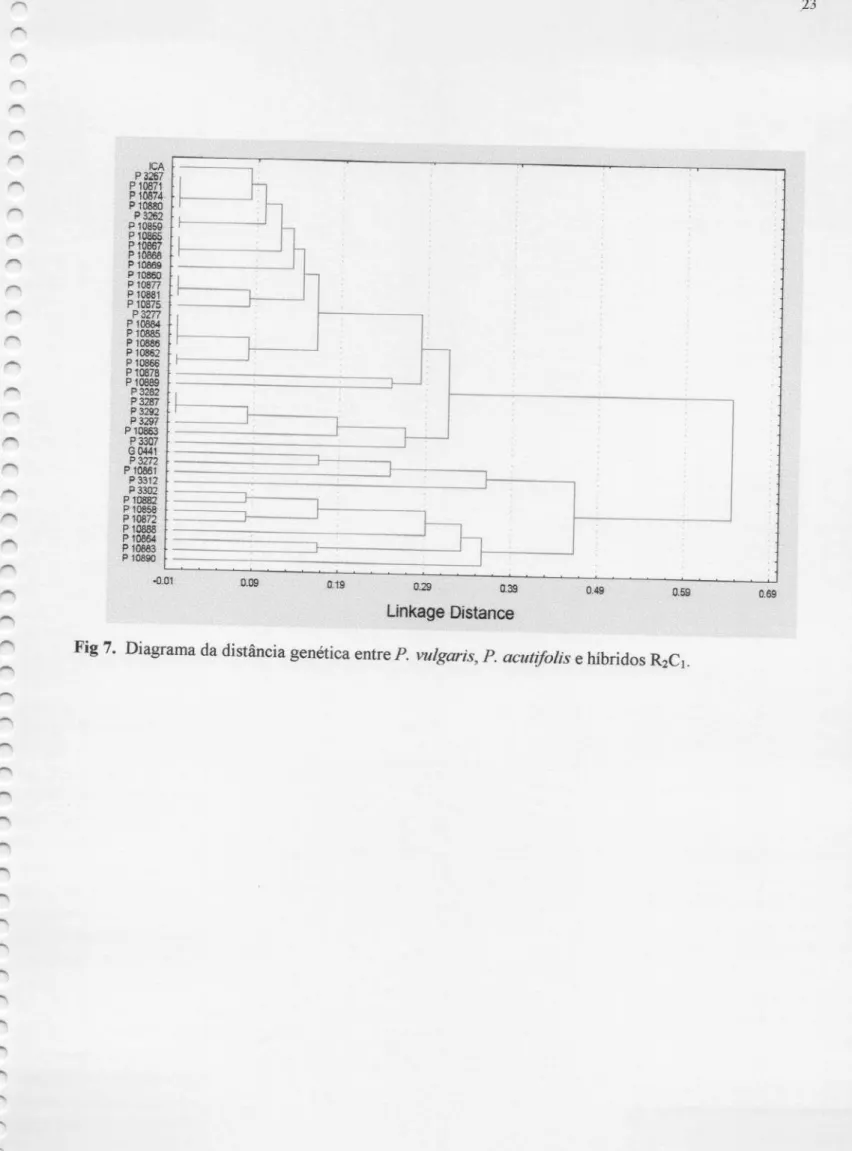
4. 2.
Proteínas Solúveis em
NaCl0,5 M
Dez
bandas
protéicas, obtidas pela eletroforese realizadaem
ambos
genitores
e
híbridos, lca Pijão (pai recorrente),GL-O441(pai doador),
e
R2C1, foramanalisadas
quanto àpresença
eausência
de bandas.A
presença
de heterozigose foidetectada
emapenas
2 bandas,as
da lecitina, de 27 kDae
23 kDa (Figura 2), com proporção de indivíduos heterozigotos de 7,69% (3casos
em 39). Em 7bandas,
os resultados apresentaram, uma proporçãode
3:1,sendo
75% de caracteristica homozigóticaherdada
do pai recorrente, lcaPijão,
e
25% de caracteristica homozigóticaherdada
do pai doador (Figura3). Emuma
das bandas a de
33.4 kDa, houvesegregação
3:1 com prevalência debandas herdadas
da cultivar GL—O441)))
)
)
)
)))))))))))))))))))))))))))))))))))
17
M 1 2 Híbridos
RZCI
45.715»
«neki»
—>
AFig
L
Eletroforese em SDS-PAGE10% dos padrões defaseolina encontrados: (M) marcador 6H, (1)IcaPijão (P. vuLgarlk);
(2) GL-0441 (P, acutzfolius) & Híbridos R2C1: (10884, 10885,
10886, 10888, 10889, 10890).
Fig2. Perfileletroforéticoem SDS-RAGE 10%.de
proteínassolúveis
em
NaCl
0,5M, destacandoa
lecitina:(M) marcador 6H, (1) [ea Pijão (P. vulgaris); (2) GL-0441 (P, a;:utzfolius),e
HíbridosR2C1: (10883, 10884, 10885, 10886, 10888,
10889, 10890). As
))))))))))))))))))))))')))))))))))))))))))))))))))
Fig
& Perfil eletroforético emSDS—PAGE,10% de proteínas solúveis emNaCl 0,5M:
(M) marcador 6H, (1) lca Pijão( P.
vulgairs); ,(2) GL—044J aº,awtg'folius)&Híbridos RzCLL (10869, 1.0871,,10872, 10874,.,10875,.,1,08,77,,
10818, 10880,10881, 10882),Asbandassinalizadas pelaseta—apresentaram. diefríhnínã'n 'ª']
4. 3.
Marcadores Moleculares
RAPDOs quatro primers testados, geraram um total de cinco
bandas
polimórficas (Tabela 3), que foram avaliadas quantoa presença e ausência
de bandas. Entre
essas
cinco, trêssão
resultado de amplificação peloGPB—10, uma pelo GPB-18
e
uma pelo OPS-11. Das três
bandas
polimórficasgeradas
peloGPB-10, uma (600pb)
apresentou segregação
de 3:1 (29bandas presentes
para 10ausentes), as
outrasduas bandas
polimón'icasgeradas,
530 pbe
1230 pb,apresentaram segregação
de372
(presentes
paraausentes)
e 33:65(ausentes
para presentes), respectivamente (Figura4).
Tabela3. Totaisde bandas amplificadase polimórt'lcaspor
primer Bandas
Primer Totais Polimórtícas
Tamanho pb
OPB-lO 12 03
380—1800
OPB-l 1 12
00 320—2072
OPB—l8 04 01
400-2000
OPS-11 08 01
300—1400
Outra banda a
apresentar segregação
polimórfica, foia de 1000 pb
gerada
pelo primer OPS-11, com 31presentes
para 8ausentes.
Oprimer GPB-18, gerou
banda
polimórfica de 800 pb, na proporção de 33:13,presentes
paraausentes.
Além
das bandas
polimórficas, foiconstatado
tambémbandas
queestão
presentes
em todos os indivíduos R2C1,e
queestão presentes apenas
no cultivar lca Pijão,geradas
pelos primers GPB-10 e11 (Figuras 4
e
5),e
OPS—11. Casosemelhante
não foi encontrado para a cultivar GL—0441, exceto
se
essa
bandaestiver
presente
emambos
genitores.Bandas
aparentemente
nãoencontradas
nos genitores foram
constatadas
nos híbridos R2C1, em amplicação pelo primer GPB-11 (Figura20
M
I
2Híbrídestcl
<—1230pb
' 600pb.
t
530 pbFig-A. Eletroforesede DNA, em gel de»
agarose1,2%. primerGPB-10. (M) marcador molecular 100 pb“; (1) Ica Pijâo (P. vulgaris); (2)
GL-0441(P. acutifolius)
e
Híbridos RZCI: (3262, 3267, 3272, 3277, 3282,3287, 3292, 3297, 3302, 3307, 3312,
10858, 10859, 10860, 10861, 10862, 10863).
Fig. S;.Eletroferesede DNA em gel
deagarose
1,2%. Pn'mer OPB-l—l.(M) Marcador MolecuIaf 100 pb;
(I)
103 Pijãe (P. vulgaris); (2)GL,-0441 (P. acutifolius), e“ Híbridos RZCI: (3262, 3267, 3272,
21
4. 4.
Estudos
de Ligação
Todas
as bandas
polimórficas obtidas de DNAe
proteínas, foramlançadas
em uma matriz
baseada
napresença e ausência
de bandas, e entãoanalisados
no programa Mapmaker,versão
3.0, paraestudos
de ligação. Foientão
obtido dois grupos de ligação, um maior composto pelos marcadores 27 kDa, 34kDa, 33.4 kDa, OPB-10b, OPB-10ae
OPS-11, totalizando 125,5 cM, e outro menor formado pelos marcadores 23 kDa, 29 kDa, 28 kDae
31.4 kDa, totalizando 32.2 cM. Quatromarcadores
121 kDa, 63.6 kDa, 65 kDa e OPB-10c nãoapresentaram
ligação (Figura6). No maiorgrupo, 33.4 kDa e o OPB-10b,representam
um ligação formada porum marcador de proteínae
outro de DNA,separados
por 21.8 cM.A maior distância entre os
marcadores
foi de 31.9 GM, e a menor de10.4 cM.
4. 5.
Distâncias Genéticas
A estimativa
das
distânciasgenéticas
envolvendo genitores
e
progêniesRZC1, realizada pelo programa de estatística, distinguiu
dois clusters, distantes em 65%,
sendo
um maior ao qualpertence a
cultivar lca Pijão com 29indivíduos,
e
outro menor com a cultivar GL—0441, com 12 indivíduos. As proporções em númerode indivíduos aproxima-se de
31
(Figura7). Dentrodocluster
do GL-0441,há ainda outra subdivisão,
separando este
em dois menoresclusters
distanciados por uma distância genética de 47%, O indivíduo mais próximo do GL—0441é
o 3262,distanciados em 17%. As distâncias
genéticas
entre os indivíduos domenor
cluster
são
maiores queas
distânciasgenéticas
entre os indivíduosdo maior cluster.No
cluster
ao qual pertencea
cultivar lca Pijão, verifica—semenores distâncias
genéticas
entreseus
indivíduos,sendo
que a maior distânciaobservada
foi de 31 %. Os indivíduos mais próximos de cultivar lca Pijão, 10880, 10874, 10871)
)
)))))
))))
)
Fig6.
Lec27kDa
__
31.9 cM34kDa
_—
25.0 cM33.4kDa
21.8 cM OPB-lOb———
25.0CM
OPB-IOa
——
21.8CMOPS-11
—_
125,5 cM
Lec 23kDa
_
29 kDa F—IO.4cM
28kDa
_—
21.8 cM31.4 kDa
%—
32,2 cMOPB-1Ob II
Os
OPB-lOa = 530pb
600 pb OPB-lOc
:
1230pbmarcadores:
121 kDa, 63.6 kDa, 65. kDa, OPB-lOc,
não estão ligados a
nenhum grupo de
ligação.
Mapa de ligação construído a partir dos marcadres bio
)
)))))))))
)
)))))
)))))))))
)
))))))))
)
)
)))
4101 DDB HTS 11.29 1159 (1:19
9.59
Linkage Distance
Fig 7. Diagrama da distância genética entre P. vulgaris, P. acutifolis
ehíbridosR2C1.
0.69
24
5. DISCUSSÓES
5. 1.
Faseolina
A faseolina é uma proteína extremamente conservada,
e está
diretamente relacionada com o centro de origem daespécie
(MA, & BLISS, 1978). A análise doperfil eletroforético de faseolina não indicou diferenças entre
as duas espécies,
provavelmente devido a origem comum,
mesoamericana
deambas
(VIEIRA ,1967). Esta hipóteseé
reforçada, pelo comportamentodessa
herança
obtido na progênie,onde
pode-se
verificar que todos os indivíduosapresentam
padrão idênticoao dos genitores (Figura1).
5. 2.
Proteínas Solúveis em
NaCl 0,5 MEstma—se que no retrocruzamento com o pai recorrente lca
Pijão, aproximadamente 87,5% do genoma já
esteja presente
na progênie R201,e
a proporção de indivíduos homozigotos para heterozigotos deveser
de 3:1(Figura 8).A característica em homozigose
herdada
pelos híbridos deve pertencer ao genitor recorrente.
As
análises das
10bandas
protéicasestudadas, neste
trabalho, revelou que a
segregação
dosgenes
paraessas
bandas estão
seguindo uma proporção de 3:1,
sendo
75% de características homozigotasherdadas
do genitor recorrente,NUi
de
características
homozigóticasherdadas
do genitor doador (Figura7). A
ocorrência de indivíduos heterozigotos, foi
constatada
emapenas duas das
dezbandas analisadas
(27 kDae
23 kDa),as
da lecitina, com frequência na progênie baixa (7,69%), não correspondendo com oesperado
de 25% de heterozigose, por banda analisada.
A
estes
resultados, muitashipóteses
tentam explicar,a
ocorrênciadessas
bandas
homozigóticas do genitor doador, e a não ocorrência debandas
heterozigõticas. Embora entre
espécies
próximas, a hibridação interespecífica já traz no próprio termo que diferençasgenéticas
deverãoser superadas para
osucesso
do cruzamento. Barreiras a nível de cromossomose genes, são esperadas,
devido à divergênciasgenéticas
ocorridas durante oprocesso
de evolução deambas
espécies.
As principaisespécies
de plantas cultivadas hibridizam facilmente comespécies
geneticamente mais próximas dela (KNOTT, 1987). Porém a substituiçãodo segmento cromossômico da
espécie
receptora por umsegmento
cromossômico de outraespécie
afim pode resultar em duplicações, emdeieções
ou emassociações
gênica indesejáveis,esses
efeitos são, particularmente, maiores emespécies
diplóides (PRESTES & GOULART, 1985).A substituição total de um cromossomo do P. vulgaris por
um do P. acutifo/ius pode ter ocorrido, originando assim
bandas
homozigóticas do P. acutifo/ius, entre os indivíduos da progênie.Outro fator a
se
considerar, é asupressão
cromossômica,demonstrada
por Gustafson & Ross (1990), onde aexpressão
gênica podeser
parcial ou totalmente inibida,dependendo
do genomapara
o qual osgenes
foram transferidos. Estasupressão
podeestar
ocorrendo quando em heterozigose, o cromossomo oua
região cromossômica do P. acutifo/ius, suprime o cromossomo ou a região cromossômica do P. vulgaris,
expressando apenas seu
produto de transcrição.Todas
as
possíveis hipóteses que justifiquem a falta de correlação entreas
frequências



![Fig. 8 Esquema do retrocruzamento autofecundação no RIC ], b) R2C1, &) e esquema de retrocruzamento seguido de](https://thumb-eu.123doks.com/thumbv2/123dok_br/16873447.754545/37.892.32.871.30.1208/fig-esquema-retrocruzamento-autofecundação-ric-esquema-retrocruzamento-seguido.webp)