Universidade de Brasília
Faculdade de Medicina
Programa de Pós-graduação em Patologia Molecular
Análise proteômica da fração nuclear de formas
epimastigotas de Trypanosoma cruzi
Agenor de Castro Moreira dos Santos Júnior
Brasília
2014
Universidade de Brasília
Faculdade de Medicina
Programa de Pós-graduação em Patologia Molecular
Análise proteômica da fração nuclear de formas
epimastigotas de Trypanosoma cruzi
Agenor de Castro Moreira dos Santos Júnior
Orientador: Prof. Dr. Carlos André Ornelas Ricart
Coorientadora: Profa. Dra. Beatriz Dolabela de Lima
Dissertação
apresentada
ao
programa de pós-graduação em
Patologia
Molecular
da
Universidade de Brasília como
requisito parcial à obtenção do grau
de mestre em Patologia Molecular.
Brasília
2014
Agradecimentos
Depois de muito trabalho e muitas alegrias eu gostaria de prestar meus
mais sinceros agradecimentos aos queridos amigos que tanto contribuíram para
minha formação intelectual durante estes dois anos de trabalho.
Ao querido professor Carlos André que com muita paciência e serenidade
me orientou. Sempre compreensivo, me incentivando e apoiando nas dificuldades
diárias. Um verdadeiro amigo.
Agradeço a minha coorientadora professora Beatriz Dolabela que abriu as
portas de seu laboratório. Pela confiança, paciência e incentivo. Sinto-me
privilegiado pela oportunidade de ser seu aluno.
Agradeço ao professor Dário Kalume pela imensa ajuda prestada.
Agradeço a todos os professores que participaram da construção desta
dissertação e da minha formação durante o mestrado. De maneira especial ao
professor Marcelo Valle, Sebastien, Cecília Favale, Bergmann, José Raimundo
Correa e Consuelo.
Agradeço a professora Fabiane e a amiga Marina Firmino que me
acompanham desde a graduação e foram responsáveis por minha vinda para a
UnB.
Agradeço ao Programa de Pós-graduação em Patologia Molecular e a
CAPES que me ofereceram uma bolsa de pós-graduação.
Ao meu irmão de ciência e de coração Ricardo Camargo, pelo apoio, pelas
conversas, pelas ideias, pelas caronas, mas, sobretudo pela amizade.
A Dra Diana que com seu jeito doce e carinhoso sempre estava disponível
para me ajudar.
Aos amigos da LBQP Adelson, Aline, Anne, Arthur, Carol, Hialy, Jaques,
Marina, Micaella, Nicholas, Nuno, Rayner, Samuel, Simone,... e de forma especial
ao grande amigo Agnelo.
Aos amigos do LABIOGENE, Fabiana, Marinez, Aurea, Anderson, Daniel,
Stenia.
Aos amigos da FHB que foram sempre muito compreensivos me apoiando
em todos os momentos.
Aos meus pais Cecília e Agenor, grandes exemplos da minha vida. Este
trabalho é dedicado a vocês.
Aos meus irmãos Marina e Moysés, e de forma especial a Jordana que me
encorajou a seguir este meu sonho e possibilitou realizá-lo.
Sumário
Lista de Figuras ... 7
Listas de Siglas e Abreviaturas ... 8
Resumo ... 9 Abstract ... 10 1. INTRODUÇÃO ... 11 1.1. Doença de Chagas ... 11 1.2. O Trypanosoma cruzi ... 14 1.3. O núcleo do T. cruzi ... 16 1.4. Proteoma e Proteômica ... 17 1.5. Proteômica em T. cruzi ... 19
1.6. Isolamento de frações nucleares de tripanossomatídeos. ... 20
2. OBJETIVO ... 21 2.1. Objetivos Geral ... 21 2.2. Objetivos Específicos ... 21 3. JUSTIFICATIVA ... 22 4. MATERIAIS E MÉTODOS ... 23 4.1. Materiais ... 23 4.1.1. Linhagem do T. cruzi ... 23
4.1.2. Soluções e Meios de Cultura ... 23
4.2. Métodos ... 25
4.2.1. Delineamento Experimental ... 25
4.2.2. Cultura das formas epimastigotas de T. cruzi CL-Brener. ... 26
4.2.3. Lise Celular ... 26
4.2.4. Separação de organelas de alta densidade ... 26
4.2.5. Purificação Nuclear ... 27
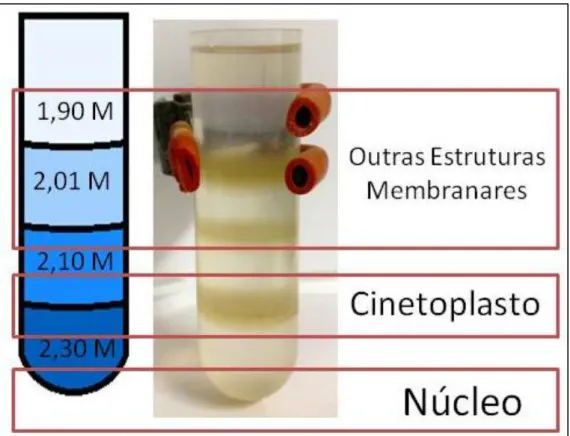
4.2.6. Identificação das frações da ultracentrifugação por microscopia ... 29
4.2.7. SDS – PAGE ... 29
4.2.8. Eletroforese Bidimensional de Proteínas (2-DE) ... 30
4.2.9. Identificação de proteínas ... 32
5. RESULTADOS E DISCUSSÃO ... 35
5.1. Fracionamento Celular de Formas Epimastigotas de T. cruzi ... 35
5.2. Identificação das frações celulares por microscopia. ... 36
5.3. Análise do isolamento nuclear por meio de SDS-PAGE... 41
5.5. Identificação das proteínas da fração nuclear por LC-MS/MS ... 44
6. CONCLUSÕES E PESPECTIVAS ... 65
7. REFERÊNCIAS BIBLIOGRÁFICAS ... 66
7
Lista de Figuras
Figura 1: Sintomatologia da doença de Chagas ... 12
Figura 2: Mapa de distribuição dos casos de doença de Chagas no mundo: separação por diferentes tipos de transmissão. ... 13
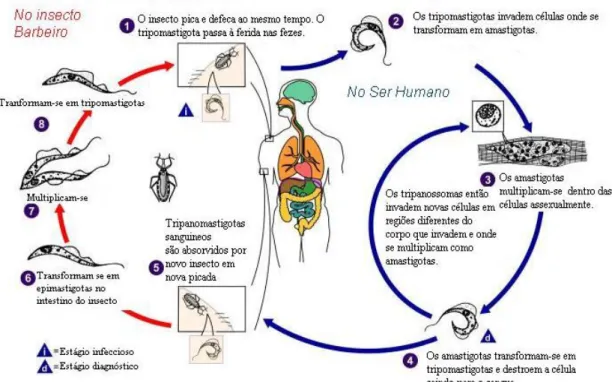
Figura 3: Ciclo de vida do Trypanosoma cruzi. ... 15
Figura 4: Dinâmica dos cromossomos recém sintetizados durante o ciclo celular do T. cruzi. ... 17
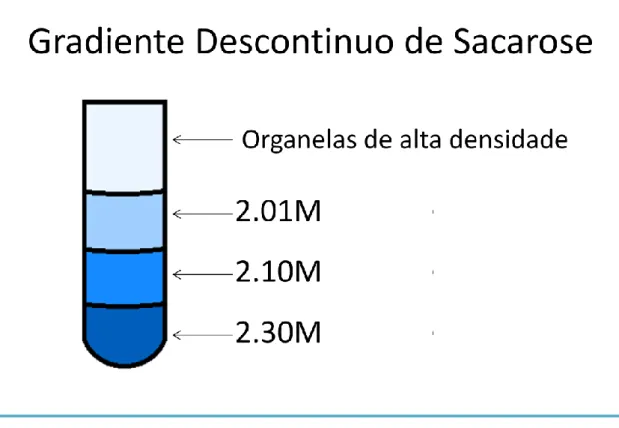
Figura 5: Representação da montagem do gradiente descontínuo de sacarose para separação da fração nuclear de T. cruzi. ... 27
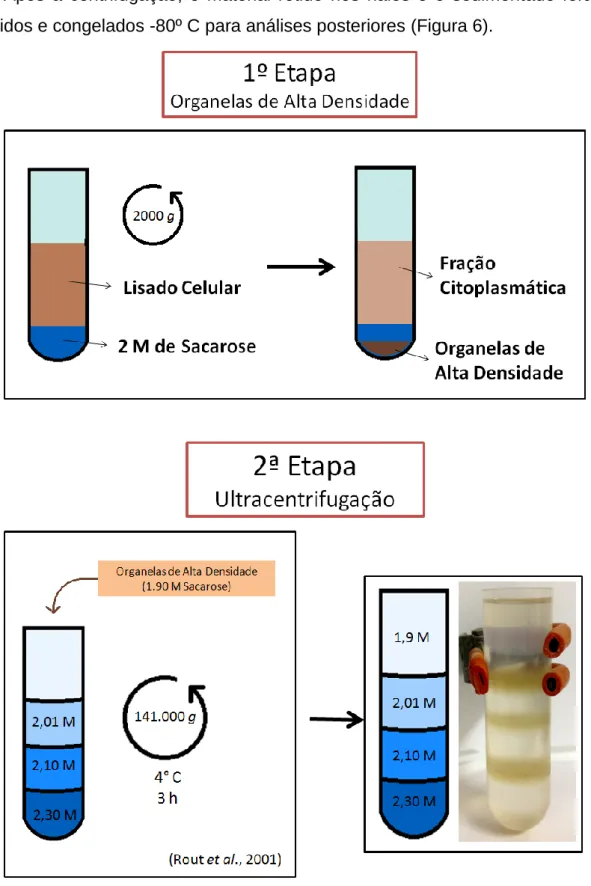
Figura 6: Metodologia do fracionamento celular de celular para obtenção de núcleos de T. cruzi isolados... 28
Figura 7: Fracionamento subcelular de extrato de formas epimastigotas de T. cruzi por ultracentrifugação em gradiente de sacarose. ... 35
Figura 8: Microscopia da fração nuclear. ... 37
Figura 9: Microscopia da fração enriquecida de cinetoplastos ... 38
Figura 10: Fracionamento celular de formas epimastigotas de T. cruzi. ... 39
Figura 11: Comparação das frações de núcleos e cinetoplastos marcados com DAPI por microscopia. ... 39
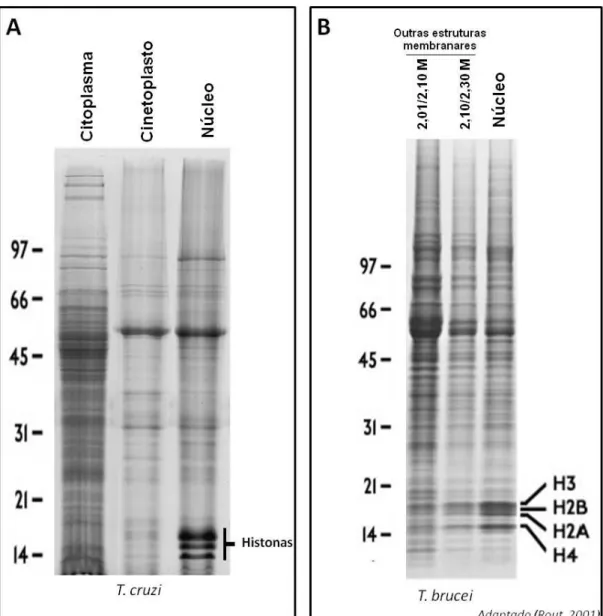
Figura 12: SDS-PAGE de amostras provenientes do fracionamento celular. ... 41
Figura 13: Eletroforese Bidimensional (2DE) da fração nuclear de T. cruzi: ... 43
Figura 14: Gráfico da distribuição celular das proteínas com localização celular única segundo por análise de Bioinformática (programa Blast2Go). ... 52
Figura 15: Gráfico das distribuição (em porcentagem) de proteínas identificadas por grupos de função GO, confeccionadas através de ferramentas de bioinformática (Blast2GO). ... 61
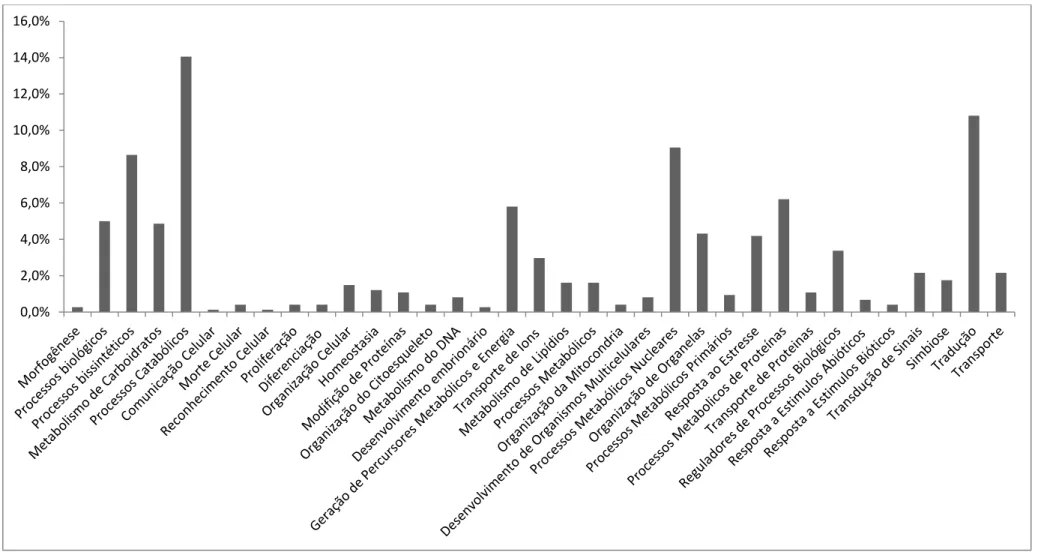
Figura 16: Gráfico da distribuição (em porcentagem) de proteínas identificadas por participação em processos biológicos GO, obtidos através de análise de bioinformática (Blast2GO). ... 63