U
NIVERSIDADE DE
L
ISBOA
F
ACULDADE DE
C
IÊNCIAS
D
EPARTAMENTO DE
B
IOLOGIA
V
EGETAL
S
TREPTOCOCCUS PYOGENES COMO AGENTE
DE INFECÇÃO DA PELE E TECIDOS MOLES
Catarina Teresa Condinho Pato
MESTRADO EM MICROBIOLOGIA APLICADA
2011
U
NIVERSIDADE DE
L
ISBOA
F
ACULDADE DE
C
IÊNCIAS
D
EPARTAMENTO DE
B
IOLOGIA
V
EGETAL
S
TREPTOCOCCUS PYOGENES COMO AGENTE
DE INFECÇÃO DA PELE E TECIDOS MOLES
Dissertação orientada por Professor Doutor Mário Ramirez
e Professora Doutora Manuela Carolino
Catarina Teresa Condinho Pato
M
ESTRADO EM
M
ICROBIOLOGIA
A
PLICADA
2011
S
TREPTOCOCCUS PYOGENES COMO AGENTE
DE INFECÇÃO DA PELE E TECIDOS MOLES
Catarina Teresa Condinho Pato
M
ESTRADO EM
M
ICROBIOLOGIA
A
PLICADA
2011
Esta tese foi realizada na Unidade de Microbiologia Molecular e Infecção do Instituto
de Medicina Molecular da Faculdade de Medicina da Universidade de Lisboa
(IMM-FML) sob a orientação directa do Professor Doutor Mário Ramirez. A Professora
Doutora Manuela Carolino foi a orientador interna designada no âmbito do Mestrado
AGRADECIMENTOS
Ao Professor Doutor Mário Ramirez, o meu orientador neste trabalho, agradeço toda a sua disponibilidade para orientar esta dissertação, pelos conhecimentos transmitidos e pela confiança em mim depositada.
Ao Professor Doutor José Melo-Cristino, Director do Instituto de Microbiologia da Faculdade de Medicina de Lisboa, agradeço a oportunidade de me ter integrado na sua equipa de laboratório.
À Professora Doutora Manuela Carolino, a minha orientadora interna, pela disponibilidade e pela revisão final do trabalho.
A todos os meus colegas do Instituto de Microbiologia, nomeadamente à Ana Friães e à Catarina Costa, pela orientação em laboratório e pelo esclarecimento de todas as dúvidas.
RESUMO
Em Portugal, os estudos epidemiológicos em Streptococcus pyogenes englobam sobretudo estirpes responsáveis por infecções do aparelho respiratório superior e por infecções invasivas. No entanto, não há registo de estudos focados em estirpes de S. pyogenes recolhidas de infecções de pele e tecidos moles. Este estudo baseou-se na caracterização de 320 estirpes recolhidas em vários hospitais do país, durante o período de 2003 a 2009, a partir de amostras de pus de lesões cutâneas, como feridas e abcessos. A caracterização das estirpes foi efectuada por diferentes métodos de tipagem, nomeadamente serotipagem T, tipagem emm, determinação dos perfis de toxinas e determinação dos perfis de macro-restrição obtidos por electroforese em campo pulsado (PFGE). Em todos os métodos utilizados verificou-se uma elevada diversidade da população (índice de diversidade de Simpson ≥0.86). As estirpes do tipo emm89 (n=62) foram as mais frequentes, seguidas por estirpes do tipo emm1 (n=55). Quando consideradas em conjunto, estas estirpes correspondem a 36,6% da população total e constituem os dois principais grupos definidos por comparação dos perfis de PFGE. Assim como estes, verificou-se que a maioria dos grupos definidos por PFGE são principalmente constituídos por estirpes do mesmo tipo emm. Relativamente aos perfis de toxinas, observou-se que estirpes com o mesmo tipo emm, normalmente, apreobservou-sentam um perfil igual ou semelhante. Também se determinou a resistência a alguns antimicrobianos de interesse clínico ou epidemiológico e os respectivos génotipos. No total, observou-se uma resistência aos macrólidos de 10,3%, com uma dominância de estirpes de fenótipo cMLSB em relação ao
fénótipo M. A resistência à tetraciclina foi observada em 47 estirpes, das quais 12 eram também resistentes aos macrólidos. Tendo em conta os diferentes tipos emm detectados e o número de estirpes correspondentes, a implementação da vacina experimental 26-valente em todas as faixas etárias poderia prevenir 67,8% das infecções de pele e tecidos moles em Portugal.
Palavras-chave: Streptococcus pyogenes; infecções da pele e tecidos moles; métodos de tipagem; antimicrobianos; vacinas.
ABSTRACT
In Portugal, epidemiological studies of Streptococcus pyogenes concentrate mostly on isolates recovered from pharyngitis and invasive infections. However, there is no record of studies focused on S. pyogenes isolates recovered from skin and soft tissue infections. This work aimed to characterize 320 isolates recovered in several hospitals across the country, from skin lesions, like wounds and abscesses. The typing methods used were T typing, emm typing, exotoxin profiling and pulsed field gel electrophoresis (PFGE) profiling. In all of these, a very diverse population was detected (Simpson’s index of diversity ≥ 0.86). The most prevalent
emm type was emm89 (n=62) followed by emm1 (n=55). Together, isolates of these emm
types accounted 36.6% of the population and composed the two major clusters defined by comparison of PFGE profiles. Like these clusters, the majority of clusters are composed mostly of isolates with the same emm type. Toxin profiling revealed that isolates with the same emm type, usually share the same or a similar toxin profile. We also determined the resistance to a panel of antibiotics of clinical and epidemiological interest, as well as the associated resistance genotypes. Erythromycin resistance was observed in 33 isolates (10%), of which 11 presented the M phenotype, while 22 were constitutively resistant to clindamycin (cMLSB phenotype).
Tetracycline resistance was detected in 47 isolates (14.7%), of which 12 were also resistance to erythromycin. According to the different emm types detected, the implementation in all age groups of the experimental 26-valent M protein-based vaccine could prevent 67.8% of skin and soft tissue infections in Portugal.
Key-words: Streptococcus pyogenes; skin and soft tissue infections; typing methods; antibiotics; vaccines.
ÍNDICE
1. INTRODUÇÃO ... 1
1.1. Descrição do agente etiológico e da doença ... 1
1.2. Identificação de S.pyogenes e condições de cultura... 2
1.3. Tipagem epidemiológica ... 2
1.3.1. Serotipagem T ... 3
1.3.2. Tipagem emm ... 3
1.3.3. Perfil de toxinas ... 4
1.3.4. Electroforese em campo pulsado ... 5
1.4. Susceptibilidade a antimicrobianos e respectivos genótipos de resistência ... 5
1.5. Vacinas ... 6
1.6. Objectivos ... 7
2. MATERIAIS E MÉTODOS ... 8
2.1. Informação das estirpes ... 8
2.2. Serotipagem T ... 8
2.3. Tipagem emm ... 8
2.3.1. Amplificação ... 8
2.3.2. Digestão do produto de PCR. ... 9
2.3.3. Extracção de DNA ... 10
2.4. Determinação do perfil de toxinas. ... 11
2.5. Efectroforese em campo pulsado ... 12
2.5.1. Extracção e imobilização do DNA bacteriano ... 12
2.5.2. Digestão ... 12
2.5.3. Separação ... 13
2.5.4. Análise dos perfis genómicos ... 13
2.6. Hibridação “Southern blot” ... 13
2.7. Determinação da susceptibilidade a antimicrobianos ... 15
2.8. Detecção dos genes de resistência aos macrólidos ... 16
2.9. Detecção dos genes de resistência à tetraciclina ... 16
2.10. Análise estatística ... 17
3. RESULTADOS ... 18
3.1. Idade e sexo dos indivíduos de onde foram recolhidas as estirpes ... 18
3.2.1. Prevalência de estirpes dos tipos emm1 e emm89 ... 19
3.2.2. Tipo emm vs idade dos indivíduos de onde foram recolhidas as estirpes. ... 19
3.3. Serotipagem T ... 20
3.4. Perfis de toxinas estreptocócicas ... 20
3.5. Resistência a antimicrobianos e respectivos génotipos associados. ... 21
3.6. Electroforese em campo pulsado... 23
3.7. Comparação dos métodos de tipagem. ... 25
4. DISCUSSÃO ... 26
6. CONCLUSÃO ... 35
1
1. INTRODUÇÃO
1.1. Descrição do agente etiológico e da doença
As bactérias da espécie Streptococcus pyogenes também designadas por estreptococos do grupo A (GAS) são agentes patogénicos de carácter Gram positivo que infectam exclusivamente o ser humano (Cunningham et al., 2000). São os principais agentes bacterianos responsáveis por faringo-amigdalites de crianças em idade escolar, sendo estimado que ocorram globalmente mais de 600 milhões de casos por ano (Carapetis et al., 2005). São também por ano, responsáveis por mais de 111 milhões de casos de infecções da pele e tecidos moles como impétigo, pioderma gangrenoso, erisipela e escarlatina (Carapetis et al., 2005; Cunningham et al., 2000). No entanto, a maior morbilidade e mortalidade resulta das infecções invasivas e das sequelas auto-imunes. Entre as infecções invasivas estão a celulite, a miosite e a fasceíte necrozante, estando esta última frequentemente associada a bacteriémia e síndrome de choque tóxico estreptocócico (STSS) (Cunningham et al., 2000). Estima-se que ocorram anualmente entre 8950-11500 casos de infecção invasiva nos Estados Unidos da América que resultam em cerca de 1300 mortes (O’Loughlin et al., 2007). Na Europa, a taxa de infecções invasivas por GAS durante o período 2003-2004 foi de 3 em cada 100 mil habitantes (Lamagni et al., 2008).
As sequelas mais comuns são a febre reumática e a glomerulonefrite aguda pós-estreptocócica (Cunningham et al., 2000). A febre reumática é uma sequela das faringo-amigdalites por GAS que se desenvolve quando estas não são devidamente tratadas. Trata-se de uma doença auto-imune que resulta do mimetismo molecular entre a proteína M e vários constituintes dos tecidos humanos. Este mecanismo resulta da activação não especifica das células T com proliferação das células T auto-reactivas e do facto dos anticorpos dirigidos à proteína M da superfície bacteriana reconhecerem também a miosina, tropomiosina, queratina e N-acetilglucosamina que constituem as válvulas cardíacas e membranas sinoviais (Dale et al., 1986). Embora se verifique um decréscimo na incidência de febre reumática nos países desenvolvidos, esta é a maior causa de mortalidade associada a doenças cardiovasculares em países em desenvolvimento. A Organização Mundial de Saúde estima que 12 milhões de pessoas sofram desta sequela, das quais 400000 morrem todos os anos (WHO report, 1992). Relativamente à glomerulonefrite aguda, não se sabe exactamente o mecanismo pelo qual a doença é despoletada, porém, assim como se verifica para a febre reumática, já foram descritas semelhanças entre os constituintes do rim e da superfície bacteriana de GAS (Bisno et al., 2003).
2
1.2. Identificação de S.pyogenes e condições de cultura.
Em laboratório, o crescimento de S. pyogenes é óptimo em meios de gelose enriquecidos com sangue. Após 24 horas de incubação a 37°C, podem observar-se colónias brancas de 1-2mm com grandes halos de β-hemólise (hemólise total dos eritrócitos presentes no meio). A identificação presuntiva faz-se utilizando a classificação de Lancefield, que permite a diferenciação entre espécies de estreptococos β-hemolíticos. Este método baseia-se nas diferenças imunológicas dos polissacarídeos da parede celular (grupo A, B, C, F e G) ou nos ácidos lipoteicoícos (Grupo D) (Murray, 2005). A maioria dos estreptococos isolados de infecções humanas que aglutinam com o soro A da classificação de Lancefield correspondem à espécie S. pyogenes. Sendo por este motivo que se designam estes cocos como estreptococos do grupo A. Porém, raramente, algumas estirpes de Streptococcus dysgalactiae supsb.
equisimilis e Streptcoccus anginosus recolhidas de humanos podem também apresentar o
carbohidrato A (Facklam, 2002). Nestes casos, se não se recorrer a outros métodos, a estirpe pode ser incorrectamente identificada.
A determinação da susceptibilidade à bacitracina é também frequentemente utilizada como teste para distinguir estirpes de S. pyogenes de outros estreptococos β-hemolíticos (Maxted, 1953). A maioria das estirpes de S.pyogenes são susceptíveis a este antimicrobiano, no entanto, já foram descritas estirpes resistentes (Silva-costa et al., 2006). Por esta razão, este método deve também ser complementado com outros.
1.3. Tipagem epidemiológica
Os surtos de doenças infecciosas, incluindo as provocadas por GAS, resultam normalmente da exposição a uma fonte comum do agente etiológico. Epidemiologicamente, os organismos envolvidos num surto são relacionados devido a uma origem comum, partilhando factores de virulência, propriedades bioquímicas e características genótipicas. Porém, quando recolhidos de diferentes produtos, em diferentes períodos e locais podem ser diferenciados através de vários métodos de tipagem.
Os métodos de tipagem regularmente utilizados podem basear-se em características fenotípicas ou moleculares. Os métodos fenotípicos tendem a ser mais simples, mais baratos e mais rápidos. Embora sejam vantajosos para a rápida identificação de estirpes responsáveis por surtos, não são os mais apropriados para estudos filogenéticos devido ao seu insuficiente poder discriminatório. Por exemplo, genes que codificam proteínas da superfície bacteriana têm tendência a evoluir rapidamente devido a uma maior pressão selectiva. Este fenómeno pode levar a que estirpes com um ancestral comum não sejam identificadas como tal ou então
3 Figura 1. Proteína M6
Adaptado de Bisno et al., (2003)
que estirpes que não possuem um ancestral comum, devido a fenómenos de transferência horizontal, possam ser consideradas proximamente relacionadas (Feil et al., 2004).
Os métodos moleculares ao contrário dos fenótipicos fornecem informação sobre a relação entre estirpes o que permite determinar os focos de infecção, detectar fenómenos de infecção cruzada, compreender a evolução das populações microbianas, permitir acompanhar a disseminação geográfica e temporal de clones de estirpes com determinadas características e monitorizar e avaliar programas de vacinação (Olive et al., 1999).
Neste estudo, o método fenotípico utilizado foi a serotipagem T. A nível molecular recorreu-se à tipagem emm, à determinação do perfil de toxinas e ainda à determinação dos perfis de macro-restrição pelo método de electroforese em campo pulsado (PFGE).
1.3.1. Serotipagem T
A serotipagem T é uma classificação serológica desenvolvida por Lancefield há mais de 50 anos (Lancefield et al., 1946). Esta técnica baseia-se nas diferenças estruturais de uma proteína presente na superfície bacteriana que por ser resistente à tripsina foi designada de proteína T. Apesar de não existirem soros suficientes para detectar todas as variedades existentes da proteína T, este método continua a ser frequentemente utilizado.
Recentemente verificou-se que este antigénio é um dos constituintes dos pili em S.
pyogenes (Mora et al., 2005). Pensa-se que os pili em S.pyogenes sejam importantes estruturas
no estabelecimento de infecção, nomeadamente no processo de adesão às células que revestem o epitélio respiratório da faringe e na capacidade de formação de biofilmes (Manetti
et al., 2007).
1.3.2. Tipagem emm
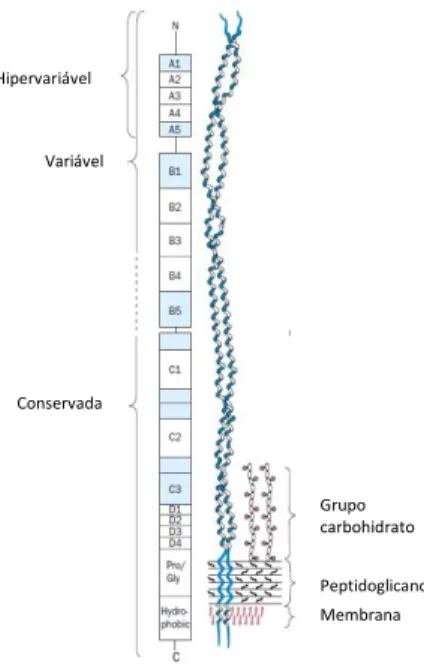
O método de tipagem emm consiste na determinação da sequência de DNA do gene emm que codifica a região N-terminal da proteína M. A proteína M localiza-se superfície celular e é considerada um dos principais factores de virulência em S.pyogenes principalmente devido à sua capacidade anti-fagocítica (Bisno et al., 1979). Esta proteína é composta por duas cadeias polipéptidicas ancoradas na membrana celular, que atravessam a parede celular e aparecem com fibrilhas na superfície celular. As cadeias contêm blocos (A-D), que diferem no tamanho e na sequência de aminoácidos (Figura 1). A extremidade C-terminal da
Variável Hipervariável Conservada Grupo carbohidrato Peptidoglicano Membrana
4 molécula localizada no interior da parede e membrana celular é altamente conservada entre diferentes estirpes de GAS. A porção N-terminal que se estende para o exterior, acaba em 11 aminoácidos que variam consoante as estirpes. Esta área, acompanhada do segmento adjacente da região A constitui a região hipervariável desta proteína (Bisno et al., 2003). Foi com base nas diferenças desta região entre estirpes que Lancefield desenvolveu um sistema de classificação serológica com o objectivo de distinguir estirpes de GAS (Lancefield, 1927). Contudo, esta técnica, tem algumas desvantagens, tal como a difícil preparação e manutenção dos soros e a detecção de um número cada vez maior de estirpes não tipáveis. Devido a estas limitações e com o avanço tecnológico começou-se a recorrer à amplificação e sequenciação da região hipervariável por PCR. A sequência obtida é comparada com outras da base de dados
do “Centers for Disease Control and Prevention"
(http:/www.cdc.gov/ncidod/bioyech/strep/strepblast.htm). Quando existe um elevado nível de semelhança (≥95%) com a extremidade 5’ de uma sequência conhecida, atribui-se à sequência o mesmo tipo emm.
1.3.3. Perfil de toxinas
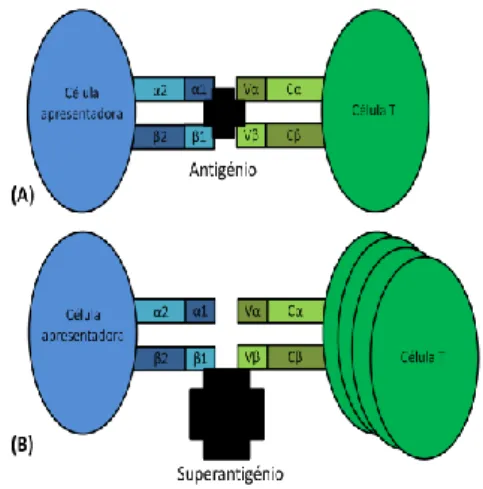
Entre os factores de virulência de S. pyogenes estão também as exotoxinas pirogénicas estreptocócicas (proteínas Spe) frequentemente associadas ao choque tóxico estreptocócico (STSS). Estas exotoxinas funcionam como superantigénios (SAg) pois apresentam a capacidade de se ligarem simultaneamente ao complexo maior de histocompatibilidade de classe II (MHC II) e aos receptores das células T (TCR) (Figura 2). A ligação leva à activação das células T independentemente da presença de antigénio, o que resulta numa secreção maciça de citoquinas inflamatórias como o interferão-γ, interleucina-1 e factor de necrose tumural α. A
libertação descontrolada destes moduladores pode levar à activação do complemento, das cascatas de coagulação e fribrinolíticas, que resulta em hipotensão e falência de vários órgãos, como se verifica em casos de STSS (Bisno et al., 2003).
No total já foram descritos em GAS, 11 superantigénios: SpeA, SpeC, SpeG, SpeH, SpeI, SpeJ, SpeK, SpeL, SpeM, superantigénio estreptocócico A (Ssa) e a exotoxina estreptocócica mitogénica Z (SmeZ). As proteínas SpeB e SpeF, apesar de inicialmente descritas como Figura 2. Interacção entre o MHC classe II do
hospedeiro das células apresentadoras de antigénios com os receptores das células T. (A) Na apresentação do antigénio após o processamento convencional. (B) Na presença de superantigénios. Adaptado de Sriskandan
5 exotoxinas devido a uma contaminação com SmeZ, são de facto uma protease e uma desoxirribonuclease, respectivamente (Gerlach et al., 2001; Kapur et al., 1993).
A maioria dos genes que codificam superantigénios estão associados a bacteriófagos, com excepção dos genes speG, speJ e smeZ. Consideram-se estes últimos genes como cromossomais, devido à sua presença em todas as estirpes analisadas em alguns estudos (Proft
et al., 2000; Proft et al., 2003) e à sequenciação do genoma de uma estirpe M1 (SF370), em
que não se detectou nenhuma associação destes genes com elementos móveis (Ferreti et al., 2001).
1.3.4. Electroforese em campo pulsado
A técnica de PFGE é um método de tipagem molecular que consiste na digestão do DNA total imobilizado em discos de agarose. A digestão é feita com uma endonuclease que fragmenta o DNA em locais de ocorrência pouco frequente. Os fragmentos, quando separados em geles de agarose por electroforese em campo pulsado, originam perfis específicos que através de um software adequado podem ser comparados e relacionados com elevada reprodutibilidade (Silva-Costa et al., 2008). Este método tem sido amplamente utilizado na tipagem de GAS devido à capacidade de distinguir estirpes com o mesmo tipo emm (Carriço et
al., 2006).
1.4. Susceptibilidade a antimicrobianos e respectivos genótipos de resistência
Os antimicrobianos são a opção mais utilizada no tratamento de infecções bacterianas. Desde o início da sua utilização que têm sido descritos casos de resistência e disseminação dos vários mecanismos associados. Tendo em conta que a escolha do antibiótico pode ser decisiva no curso da infecção, é essencial existir um controlo regular dos níveis de resistência para que a terapêutica empírica adoptada seja a mais adequada.
No caso de faringo-amigdalites causadas por GAS, o antibiótico mais utilizado na terapêutica continua a ser a penicilina. Porém, devido a alergias ou à facilidade de administração, os macrólidos e as lincosamidas são importantes escolhas alternativas. Em casos de infecções graves da pele e tecidos moles recorre-se à clindamicina (lincosamida) em associação com os β-lactâmicos para inibir a produção de exotoxinas (Allen et al., 2010). Quando se detectam estirpes resistentes aos macrólidos e às lincosamidas, as fluoroquinolonas são uma alternativa possível (Malhotra et al., 2009).
A azitromicina é o macrólido mais utilizado na terapêutica de infecções por GAS. Em laboratório utiliza-se a eritromicina para determinar a susceptibilidade aos macrólidos. Esta classe de antimicrobianos inibe a síntese proteica por ligação reversível ao RNA ribossomal 23S
6 da subunidade 50S dos ribossomas de organismos susceptíveis, o que resulta no bloqueio da reacção de translocação durante o alongamento da cadeia polipeptídica (Roberts et al., 1999).
Os mecanismos de resistência aos macrólidos mais frequentes são a modificação do local alvo e produção de uma bomba de efluxo (Leclercq et al., 2002). A modificação do local alvo é feita por uma metilase (proteína Erm) codificada pelos genes erm. Esta proteína metila um resíduo de adenina na subunidade 23S do ribossoma e impede a ligação dos macrólidos, lincosamidas e estreptograminas B (fenótipo MLSB). Em S. pyogenes, o fenótipo MLSB pode ser
mediado por dois genes erm: o gene erm(B) e o gene erm(TR), pertencente à classe erm(A). A expressão destes genes pode ser constitutiva ou indutível pela presença de eritromicina, dando origem aos fenótipos cMLSB ou iMLSB, respectivamente (Leclercq et al., 2002). O
segundo mecanismo que confere resistência aos macrólidos em GAS é a presença de uma bomba de efluxo associada à membrana. Esta bomba é codificada pelos genes mef [mef(A) ou
mef(E)] e exporta para o exterior as moléculas de antibiótico o que confere resistência aos
macrólidos (Roberts et al., 1999; Sutcliffe et al., 1996).
Relativamente às tetraciclinas, embora não sejam geralmente utilizadas na terapêutica, a resistência a este antimicrobiano é analisada em laboratório por estar frequentemente associada com a resistência à eritromicina. As tetraciclinas actuam por inibição da síntese proteica ao impedirem a associação do tRNA ao ribossoma bacteriano (Chopra et al., 2001). A resistência às tetraciclinas tem vindo a ser descrita por vários estudos em bactérias comensais e patogénicas maioritariamente devido a aquisição dos genes tet. Estes genes, à semelhança do que se verifica para os macrólidos, conferem resistência por dois mecanismos, efluxo do antibiótico e protecção ribossomal. Em bactérias de carácter Gram positivo os mais frequentes são os genes tet(K), tet(L), tet(M) e tet(O) (Chopra et al., 2001). Os genes tet(K) e tet(L) codificam bombas de efluxo, que se associam à membrana citoplasmatica e exportam as moléculas de tetraciclina para o exterior das células, o que reduz a sua concentração intracelular protegendo os ribossomas da acção dos antimicrobianos (Chopra et
al., 2001). Os genes tet(M) e tet(O) codificam proteínas de protecção ribossomal
citoplasmáticas com homologia aos factores de elongação EF-TU e EF-G. A ligação destas proteínas aos ribossomas provoca uma alteração conformacional que impede a ligação das tetraciclinas (Sanchez-Pescador et al., 1988).
1.5. Vacinas
Devido ao elevado número de infecções por GAS, foi proposto o desenvolvimento de uma vacina segura e eficaz que se espera vir a ter um grande impacto na saúde de milhões de pessoas. As vacinas propostas podem ser classificadas em duas categorias: as que contêm
7 determinantes de virulência conservados em diferentes serótipos (C5a-peptidase, SpeB, proteínas de ligação à fibronectina, região C-terminal da proteína M) e as que se baseiam na região variável N-terminal da proteína de superfície M (Bisno et al., 2005). Com base na extremidade N-terminal da proteína M de estirpes de diferentes tipos foi desenvolvida uma vacina 26-valente que se estima conferir protecção na América do Norte contra 85% das estirpes responsáveis por faringite, 92.5% das estirpes associadas a febre reumática e 87.6% das estirpes invasivas, incluindo 100% das estirpes associadas a fasceíte necrozante (McNeil et
al., 2005: Shulman et al., 2009). Esta vacina já completou a fase I e II dos ensaios clínicos em
adultos, sendo bem tolerada, imunogénica e não despoletando reacções cruzadas. Actualmente está a ser considerada para ensaios clínicos em crianças, que representam um dos grupos alvo desta vacina (Dale et al., 2008).
1.6. Objectivos
O conhecimento das características das estirpes mais prevalentes e o relacionamento entre estirpes de diferentes infecções é essencial para adequar a terapêutica utilizada e desenvolver novas medidas de prevenção. Devido à ausência em Portugal de estudos epidemiológicos focados em estirpes recolhidas de infecções de pele e tecidos moles, este estudo pretende:
Caracterizar estirpes de GAS recolhidas em Portugal entre 2003 e 2009 de amostras de pus de feridas e abcessos, através de métodos fenótipicos (serotipagem T) e moleculares (tipagem emm, determinação dos perfis por PFGE e determinação dos perfis de toxinas) a fim de avaliar diversidade da população bacteriana.
Avaliar e comparar as diversas metodologias utilizadas.
Determinar o número de estirpes resistentes aos macrólidos e analisar a distribuição destas e dos respectivos genótipos de resistência na população. Averiguar também a associação descrita entre estirpes do fenótipo cMLSB e a resistência à tetraciclina. Comparar as propriedades moleculares das principais linhagens com estudos que
8
2. MATERIAIS E MÉTODOS
2.1. Informação das estirpes
Este estudo englobou 320 estirpes de S. pyogenes recolhidas em vários hospitais do país, durante o período 2003-2009, que foram isoladas a partir de amostras de pús de lesões cutâneas, como feridas e abcessos.
A identificação das estirpes foi feita nos respectivos hospitais e confirmada no laboratório da Unidade de Microbiologia e Infecção, pela morfologia das colónias, β-hemólise e susceptibilidade à bacitracina. No caso de estirpes resistentes a este antibiótico verificou-se a presença do antigénio característico na parede celular (Streptococcal Grouping Kit, Oxoid, Basingstoke, UK).
Antes de se iniciar o processo de tipagem, as estirpes foram repicadas a partir de uma colónia em meio TSA (Tryptic Soy Agar) (Oxoid, Basingstoke, UK). Posteriormente foram conservadas a -80°C em meio TSB (Tryptic Soy Broth) (Oxoid, Basingstoke, UK) com 15% (V/V) de glicerol.
2.2. Serotipagem T
Para determinar o serótipo T das estirpes, inoculou-se cerca de 10µL de meio de preservação onde a estirpes estavam conservadas, em 5mL de Todd-Hewitt (Oxoid, Basingstoke, UK) e deixou-se a incubar a 30°C durante a noite. No dia seguinte, centrifugou-se a 3220g durante 15min à temperatura ambiente e desprezou-se o sobrenadante. Adicionaram-se 4 gotas de Todd-Hewitt (Oxoid, Basingstoke, UK) e recorrendo aos reagentes do kit (Auxiliary Reagents for Heamolytic Streptococcus Typing, Denka Seiken, Tóquio, Japão), adicionaram-se 4 gotas de extracto pancreático suíno, 1 gota de vermelho fenol e 2 gotas de solução de ajuste pH. Misturou-se e incubou-se a 37°C durante 1h. Finalmente, centrifugou-se novamente a 3220g durante 15min à temperatura ambiente e desprezou-se o sobrenadante. Fez-se uma nova suspensão em 500µL de PBS 1X a partir da qual se efectuou a aglutinação com os soros específicos. A aglutinação foi feita numa lâmina com 5 soros polivalentes (T, U, W, X e Y) e 19 monovalentes (1, 2, 3, 4, 5/27/44, 6, 8, 9, 11, 12, 13, 14/49, 18, 22, 23, 25, 28, B3264 e Imp.19) (Haemolytic Streptococcus Group A Typing–sera, Denka Seiken, Toquio, Japão).
2.3. Tipagem emm
2.3.1. Amplificação
A tipagem emm, como já foi referido, permite a identificação da região hipervariável do gene emm que codifica a proteína M. Este método consiste na amplificação, purificação, e
9 sequenciação da região N-terminal do gene emm. Todos os passos, incluindo a identificação do tipo emm, foram executados de acordo com os protocolos e recomendações do CDC (http://www.cdc.gov/ncidod/biotech/strep/protocols.htm).
A amplificação foi feita por reacção em cadeia de polimerase (PCR) num volume total de 50µL, utilizando 1,5 µL de sobrenadante de lisado bacteriano como fonte de DNA molde. Para preparar o lisado bacteriano recolheu-se, com uma ansa, algumas colónias a partir de placa e suspenderam-se em 300µL de solução de cloreto de sódio (NaCl 0,85%). Seguidamente, colocou-se o tubo a 70°C durante 15min. Centrifugou-se a amostra a 18000g durante 2min e desprezou-se o sobrenadante. Fez-se uma nova suspensão do sedimento em 50µL de TE 1X (Tris 10mM; EDTA 1mM, pH8), 6µL de mutanolisina (5 U/µL, Sigma-Aldrich, Steinheim, Alemanha) e 2µL de hialuronidase (12,3mg/mL, Sigma-Aldrich, Steinheim, Alemanha). Após 30min a 37°C, ferveu-se a 100°C durante 10min. Os lisados foram conservados a -20°C até se efectuar o PCR. Os restantes componentes do PCR foram adicionados de modo a obter uma concentração final de tampão de PCR “green GoTaq flexi”a 1X (Promega, Madison, WI, USA), 0.02 U/µL de DNA polimerase GoTaq (Promega, Madison, WI, USA), 0,2 mM de cada nucleotídeo (Fermentas, Vilnius, Lituania) e 1,5mM de MgCl2 (Promega, Madison, WI, USA). Os
primers utilizados foram o emm1 e emm2, (Quadro 1, em anexo) ambos a uma concentração final de 1,4pmol/µL. O ciclo de PCR consiste numa temperatura inicial de 94°C durante 1min, seguido de 10 ciclos de 15s a 94°C, 30s a 46.5°C e 1min e 15s a 72°C, 20 ciclos de 15s a 94°C, 30s a 46.5°C, e 1min e 15s a 72°C com um incremento de 10s para cada ciclo subsequente. No final deixa-se 10min a 72°C.
Os produtos de PCR foram purificados com o Kit “High Pure PCR product purification Kit” (Roche, Manheim, Alemanha) de acordo com as instruções do fabricante e depois enviados para sequenciar (Macrogen, Coreia do Sul e Holanda) com o primer emmseq2
(Quadro 1, em anexo). Utilizam-se as primeiras 240 bases da sequência para interrogar utilizando o BLAST disponível da base de dados do CDC (http://www.cdc.gov/ncidod/biotech/strep/strepblast.htm). Quando existe uma elevada semelhança (≥95%) com a extremidade 5’ de uma sequência conhecida, atribui-se o mesmo tipo emm.
2.3.2. Digestão do produto de PCR.
A digestão e separação dos produtos da reacção de PCR para amplificar a região do gene emm que é utilizada na tipagem, permite comparar de uma forma mais económica os tipos emm de diferentes estirpes. Considerando que perfis iguais correspondem ao mesmo tipo emm, é possível através da análise e comparação destes prever o tipo emm. A digestão
10 dos produtos de PCR foi feita simultaneamente com as endonucleases HincII (Fermentas, Vilnius, Lituânia) e HaeIII (Fermentas, Vilnius, Lituânia) [2,5U de cada para 5µL de produto de PCR num volume total de 6,1µL que se perfez com buffer Tango 10X (Fermentas, Vilnius, Lituânia)]. Os produtos desta digestão foram separados num gel de 3% (m/v) [2% de agarose NuSieve (Lonza, Rockland, USA) e 1% agarose normal (Bio-Rad, Munique, Alemanha)]. Das estirpes com perfis iguais, enviou-se uma para sequenciar.
2.3.3. Extracção de DNA
Por vezes, devido a uma qualidade insuficiente ou a uma rápida degradação do DNA, o lisado bacteriano preparado de acordo com as recomendações do CDC não permitiu a amplificação do gene emm. Nestes casos, recorreu-se à extracção de DNA por outro método. Adicionou-se 10µL de stock de congelação a 5mL de Todd-Hewitt e incubou-se a 37°C. No dia seguinte, centrifugou-se a cultura a 3220g durante 15min a 4°C. Desprezou-se o sobrenadante e fez-se uma nova suspensão em 468,2µL de TE1X (Tris 10 mM pH7,5; EDTA 1mM pH8,0). Após se transferir para um tubo de 2mL, adicionou-se 30µL de dodecil sulfato de sódio (SDS) (10%, Bio-Rad, Munique Alemanha), 3µL de proteinase K (20mg/mL; Roche, Manheim, Alemanha), 60µL de mutanolisina (5U/µL; Sigma-Adrich, Steinheim, Alemanha), 20µL de hialuronidase (12,3mg/mL; Adrich, Steinheim, Alemanha) e 18,8µL de lisozima (20mg/mL; Sigma-Adrich, Steinheim, Alemanha). Agitou-se no vortex e incubou-se durante 1h a 37°C. Seguidamente, adicionou-se 15µL de ribonuclease (20mg/mL; Sigma-Adrich, Steinheim, Alemanha) e incubou-se a 37°C durante 30min. Finalmente adicionou-se 100µL de NaCl 5M e 80µL NaCl CTAB [10% de brometo de hexadeciltrimetilamónio (CTAB) em NaCl 0,7M]. Levou-se ao vortex e após incubação durante 10min a 65°C, adicionaram-se 800µL clorofórmio/álcool isoamílico (24:1). Centrifugou-se durante 5min a 18000g e transferiu-se a fase aquosa para outro tubo de 2mL. Adicionaram-se 600µL fenol/clorofórmio/álcool isoamílico (25:24:1), levou-se ao vortex e voltou-levou-se a centrifugar a 18000g durante mais 5min. Novamente, transferiu-levou-se a fase aquosa para outro tubo, desta vez de 1,5mL, e neste caso adicionaram-se 600µL de isopropanol. Ao agitar é por vezes possível observar as fibras de DNA. Centrifugou-se, mais uma vez, durante 5min a 18000g e desprezou-se o sobrenadante. Em seguida, adicionaram-se 500µL de etanol 70% a -20°C. Repetiu-se este último passo e deixou-se secar ao ar. Por fim, fez-se a suspensão do DNA em 50µL de TE 1X. Com este método obteve-se concentrações de DNA muito superiores ao necessário para realizar o PCR, por isso foi necessário quantificar o DNA e diluir para 200ng/µL. Utilizando-se consoante as reacções entre 200 e 300ng (1-1,5µL).
11
2.4. Determinação do perfil de toxinas.
A presença dos genes speF, ssa, smeZ, speH, speK, speJ, speG, speC, speK, speL, speI,
speB, speA foi detectada através de 2 PCRs multiplex. Foram utilizados pares de primers
específicos para cada gene com capacidade de amplificar todas as variantes alélicas descritas de acordo com as 13 sequências genómicas publicadas (GenBank AE004092, AE009949, AE014074, AM295007, BA000034, CP000003, CP000017, CP000056, CP000259-CP000262, CP000829) e também com as sequências das variantes alélicas do speA, speC e smeZ disponíveis no GenBank.
A amplificação dos genes speF, ssa, smeZ, speH, speK, speJ e speG foi efectuada num volume total de 25 µL, com tampão de PCR 1X “green GoTaq flexi”(Promega, Madison, WI, USA), MgCl2 a 5mM, nucleotídeos a 0,2 mM, cada, 0.08U de DNA polimerase GoTaq
(Promega) e 1µL de DNA (lisado bacteriano preparado como descrito para a determinação do
emm). Os pares de primers utilizados foram speF1/speF2, ssa1/ssa2, smeZ-F/smeZ-R2,
speH1/speH3, speK-f/speK-r2, speJg-f2/speJg-r e speG-f/spe-r com uma concentração final de 0,8pmol/µL, 0,2pmol/µL, 1,8pmol/µL, 0,32pmol/µL, 0,4 pmol/µL, 0,12pmol/µL e 0,24pmol/µL, respectivamente (Quadro 1, em anexo).
A amplificação dos genes speC, speM, speL, speI, speB, speA foi também efectuada num volume total de 25 µL, com tampão de PCR “green GoTaq flexi” a 1X (Promega), MgCl2 a
5mM (Promega), nucleotídeos a 0,2 mM cada (Fermentas), 0.06U de DNA Polimerase GoTaq (Promega) e 1µL de DNA (lisado bacteriano preparado como descrito para a determinação do
emm). Os pares de primers utilizados foram speC-f2/speC-r2, speK-f/speM-r, speL-f/speL-r,
speI1/speI3, speB1/speB2, speA-f2/spe-r2 a uma concentração final de 0,2pmol/µL, 0,24pmol/µL, 0,24pmol/µL, 0,2pmol/µL, 0,1pmol/µL, 0,16pmol/µL, respectivamente (Quadro 1, em anexo).
O programa de PCR para o primeiro multiplex descrito consistiu numa temperatura inicial de desnaturação de 95°C durante 2min, seguido de 40 ciclos a 95°C durante 1min, 54°C, durante 30s e 72°C durante 1min e 30s. No segundo multiplex descrito, o programa utilizado foi o mesmo, com excepção da temperatura de “annealing”, que é de 62°C em vez de 54°C. Os produtos de PCR do primeiro e do segundo multiplex foram separados em géis de agarose de 1% e 2%, respectivamente. Como controlo positivo para cada reacção misturou-se o DNA de uma estirpe M1 (SF370) com o DNA de outras estirpes com os restantes genes que se estavam a testar. A estirpe M1 foi obtida da “Coleccion Española de Cultivos Tipo” (Valencia, Espanha, CECT 5109) e contém os genes speB, speF, speG, smeZ, speC, speH, speI e speJ.
12
2.5. Efectroforese em campo pulsado
2.5.1. Extracção e imobilização do DNA bacteriano
Inoculou-se 15µL do “stock” congelado em 5mL de Todd-Hewitt (Oxoid, Basigstoke, UK) e colocou-se, durante a noite na estufa a 37°C. Centrifugou-se a 3320g durante 15min a 4°C, desprezou-se o sobrenadante e fez-se uma nova suspensão em 5mL de TEN (Tris 100mM; EDTA 100mM; NaCl 150mM, pH7,5). Voltou-se a centrifugar nas mesmas condições e fez-se uma nova suspensão em 1mL de TEN. Seguidamente, transferiu-se para um tubo de 1,5mL e centrifugou-se novamente durante 15min a 3320g. Desprezou-se o sobrenadante e fez-se uma nova suspensão em 200µL de TEN.
Para adequar a concentração de DNA, leu-se a densidade óptica a 620nm e calculou-se o volume de TEN a adicionar a cada amostra (Vadd=ODx40x240-240).
Para fazer os discos, misturou-se 150µL de “low melting agarose” 1,5% (m/V) (Bio-rad, Munique, Alemanha) diluída em PIV (Tris 10mM pH8; NaCl 1M) a 150µL de suspensão bacteriana com a concentração adequada e equilibrada a 41°C. Após levar ao vortex, colocaram-se sucessivamente gotas de 20 µL numa superfície lisa forrada com parafilme, que solidificaram após 5 min a -20°C. Para a lise das células adicionou-se aos discos uma solução constituída por 0,67mg/mL de lisozima (Sigma-Aldrich, Steinheim, Alemanha), 16,67mg/mL de ribonuclease A Aldrich, Steinheim, Alemanha) e 16,67U/mL de mutanolizina (Sigma-Aldrich, Steinheim, Alemanha) e EC (Tris 6mM pH7,5; EDTA 100mM pH7,5; NaCl 1M, desoxicolato de sódio 5mM; N-lauril sarcosine de sódio 17mM; Brij 58 0,5% m/v; MgCl2 1mM).
Incubou-se a 37°C durante pelo menos durante 5h.
Seguidamente, colocaram-se os discos em tampão ES (EDTA 0,5mM pH7,5; N-Lauril sarcosina de sódio 34mM) com 0,5 mg/mL de proteinase K (Roche, Menheim, Alemanha) durante 24h a 50°C. Procedeu-se a lavagens com tampão TE (1X) (10mM Tris pH7,5; 1mM EDTA pH8). Até se fazer a digestão, os discos ficaram armazenados nesta solução a 4°C.
2.5.2. Digestão
Para a digestão, os discos foram colocados durante a noite ou com 7,5 U de SmaI (Fermentas, Vilnius, Lituania) a 30°C num volume final de 45µL, ou com 3,75U de Crf9I (Fermentas, Vilnius, Lituania) a 37°C num volume total de 20µL. Os tampões utilizados na digestão foram tango 10X (Fermentas, Vilnius, Lituania) no caso da SmaI e tampão Crf9I (Fermentas, Vilnius, Lituania) para a enzima Crf9I, respectivamente. Antes se de efectuar a digestão com as endonucleases fez-se um equilíbrio de uma hora à temperatura de digestão em 500µL (SmaI) ou 100µL (Crf9I) de tampão de restrição, tango 10X ou tampão Crf9I, respectivamente. A utilização da enzima Crf9I, restringiu-se a estirpes de fenótipo M. A
13 expressão deste fenótipo, resulta frequentemente da presença do gene mef(A) que se localiza no mesmo elemento genético de uma metiltransferase (Figueiredo et al., 2006). A SmaI ao ser sensível à metilação, não digere o DNA metilado.
2.5.3. Separação
Após digestão, adicionou-se “loading buffer” aos discos e colocaram-se num gel de agarose 1% (p/V) (BioRad, Munique, Alemanha) assim como o marcador de peso molecular “Lambda ladder PFGE marker” (New England BioLabs, UK). A corrida ocorreu durante 23h em tampão TBE 0.5X (Tris-borate-EDTA) (BioRad, Munique, Alemanha), num aparelho CHEF-DR III (BioRad, Munique, Alemanha), a 6,0 V/cm3 com um tempo inicial de 5s e um tempo final de 35s.
2.5.4. Análise dos perfis genómicos
Os perfis de macro-restrição obtidos para cada estirpe foram analisados e comparados com recurso ao programa informático BioNumerics (Applied-Maths, Sint-Martens-Latem, Bélgica). Desta forma construíram-se dendrogramas utilizando o coeficiente de Dice (com as definições de optimização e tolerância de 1,0 e 1,5%, respectivamente) e o método de agrupamento hierárquico UPGMA (“unweighted pair group method with arithmetic means”). Consideraram-se como grupos de PFGE, grupos de 5 ou mais estirpes com semelhança igual ou superior a 80%.
2.6. Hibridação “Southern blot”
Através desta técnica pretendeu-se confirmar a presença ou ausência dos genes speM,
speL, speB e speF. Começou-se por preparar o DNA genómico em discos de agarose, digeriu-se
com SmaI e separou-se por electroforese em campo pulsado nas condições descritas no ponto 2.5. Seguidamente, transferiu-se o DNA para uma membrana de nylon (Hybond-N+, Amersham Biociences, Bucks, UK) por sobreposição do gel de PFGE num sistema de vácuo (Pharmacia Biotech AB, Uppsala, Suécia). Ligou-se o aparelho a 40mmHg e cobriu-se o gel com solução de despurinação (HCL 0,25M) durante 10min. Seguidamente, removeu-se o restante da solução de depurinação e adicionou-se solução de desnaturação (NaOH 0,4M; NaCl 1,5M). Deixou-se nestas condições durante 2h e 30min com o cuidado de não deixar a superfície do gel secar. Após retirar a solução de desnaturação com uma pipeta retirou-se o gel de agarose e desligou-se o vácuo. Lavou-desligou-se a membrana com SSC 6X (Citrato de Sódio90mM; NaCl 0,9M; pH7) e deixou-se ao ar entre 5 a 15 minutos. De modo a fixar o DNA, expôs-se a membrana a radiações UV durante 20s no “UV crosslinker” (Alfagene, Cambridge, UK).
As sondas utilizadas correspondem aos produtos de PCR resultantes da amplificação de um fragmento interno de cada gene após purificação. Para a amplificação em separado dos
14 genes speM, speL e speF, utilizaram-se as mesmas condições. Os primers utilizados foram os mesmos que os utilizados nos multiplex para a detecção das exotoxinas (Quadro 1, em anexo). A concentração final dos componentes foi, tampão 1X “green Gotaq flexi” (Promega), DNA polimerase GoTaq a 0,02U (Promega), nucleótidos a 0,2mM (Fermentas), MgCl2 a 1,5mM
(Promega) e primers a 0,4 pmol/µL, num volume total de 50µL com 3µL de DNA (lisado bacteriano preparado como descrito para a determinação do tipo emm). O programa foi semelhante ao utilizado para amplificação das toxinas no multiplex. No caso dos gene speM e
speL, utilizou-se uma temperatura de hibridação de 62°C e no caso do gene speF de 54°C. As
condições para a amplificação do speB diferem das anteriores. Num volume total de 50µL com 3 µL de DNA (lisado bacteriano extraído como descrito na determinação do tipo de emm), utilizou-se uma concentração final de tampão “green GoTaq flexi” 1X (Promega), 0,02U/µL de DNA polimerase GoTaq (Promega), 0,2mM de nucleótidos (Fermentas) e 0,4 pmol/µL de primer. O programa começou a uma temperatura de 94°C durante 5 min, seguida de 35 ciclos de 1min a 94°C, 2min a 58°C e 1min a 72°C.
A marcação e hibridação foram efectuadas com o marcador de ácidos nucleícos ECL e os reagentes do sistema de detecção (GE Health care, Buckinghamshire, UK) de acordo com as instruções de fabricante. Começou-se por lavar a membrana com SSC 5X (Citrato de sódio 75mM; NaCl 0,75M; pH7) e coloca-la na garrafa de hibridação com cerca de 20mL do SSC 5X utilizado na lavagem. Incubou-se a 42°C com rotação durante 15min. Desprezou-se o SSC 5X e adicionou-se a solução de hibridação (tampão de Hibridação; 5% m/v de agente bloqueante; NaCl 0,5M). Incubou-se a 42°C com rotação durante 1 hora. Entretanto, diluiu-se a sonda para 10ng/µL em 15µL com água fornecida com o kit e adicionou-se 4 µL marcador “DNA controlo” (Kit). Ferveu-se durante 5min e colocou-se em gelo. De seguida, adicionou-se 19µL de “Labelling reagent” (Kit) e 19µL de glutaraldeído (Kit). Após incubação a 37ºC durante 20min, adicionou-se 1mL de solução de hibridação, misturou-se e colocou-se na garrafa de hibridação com a membrana. Incubou-se durante a noite a 42ºC com rotação. No dia seguinte, removeu-se a solução de hibridação e lavou-removeu-se a membrana 2 vezes com rotação durante 20min a 42°C com solução de lavagem primária (Ureia 6M; SDS 0,4%; SSC 0,5X). Retirou-se a membrana da garrafa de hibridação e lavou-se com SSC 2X (Citrato de Sódio 30mM; NaCl 0,3M; pH7) durante 5min à temperatura ambiente. Para a fase da detecção, misturou-se num tubo, 7,5mL de solução de detecção 1 com 7,5mL de solução de detecção 2 (Kit) e adicionou-se de imediato a mistura à membrana. Após 1 minuto a agitar a membrana de modo a cobri-la amplamente com a solução, guardou-se a solução num tubo e colocou-se a membrana numa mica de plástico com o DNA para cima, tendo o cuidado de eliminar as bolhas de ar. Sucessivamente colocou-se a mica numa cassete.
15 Numa câmera escura colocou-se uma chapa de autoradiografia dentro da cassete por cima da mica de modo a cobrir toda a membrana. O tempo de exposição variou de 30min a 5h. No final do procedimento, a membrana ficou armazenada a 4°C numa mica com a solução de detecção para uma eventual reutilização.
2.7. Determinação da susceptibilidade a antimicrobianos
Para determinar a susceptibilidade aos antimicrobianos utilizou-se a metodologia de Kirby-bauer de acordo com as recomendações do CLSI (Clinical and Laboratory Standards Institute, 2006). Este método consiste na difusão de antibióticos presentes em discos de papel que são colocados na superfície de uma placa com meio Mueller-Hinton (Oxoid, Basingstoke, UK), suplementada com 5% (V/V) de sangue de carneiro desfibrinado (ProBiológica, Belas, Portugal) e semeada com uma determinada estirpe. Para se semear a estirpe na placa onde se colocam os discos, ajustou-se visualmente a turbidez do inóculo numa suspensão de NaCl 0,85% a uma suspensão 0,5 da escala de Macfarland. Após aplicar os discos, colocou-se a placa na estufa durante a noite a 37°C com atmosfera de 5% CO2. A leitura efectuou-se de acordo
com os critérios de interpretação do “Clinical and Laboratory Standard Institute” (Quadro 1).
Quadro1. Critérios de interpretação dos antibióticos utilizados segundo as normas do CLSIa
Conteúdo disco (µg) Susceptível (mm) Intermédio (mm) Resistente (mm) Penicilina 10 ≥24 - - Eritromicina 15 ≥21 16-20 ≤15 Clindamicina 2 ≥19 16-18 ≤15 Tetraciclina 30 ≥23 19-22 ≤18 Cloranfenicol 30 ≥21 18-20 ≤17 Levofloxacina 5 ≥17 14-16 ≤13 Linezolide 30 ≥21 - - Vancomicina 30 ≥17 - - a
Clinical and Laboratory Standards Institute. Performance Standards for antimicrobial susceptibility testing, sixteenth informational supplement. CLSI document M100-S16. Wayne, PA: CLSI, 2006.
Para os resultados intermédios efectuou-se a determinação da concentração inibitória mínima (CIM) com E-teste de acordo com as instruções do fabricante (bioMérieux, Marcy-l’Etoile, França). O E-teste é uma tira com um gradiente de concentração de um determinado antibiótico. À semelhança do que se fez para os discos, utilizaram-se placas de meio Muller-Hinton com 5% de sangue, semeadas com um inóculo ajustado ao valor 0,5 da escala de McFarland.
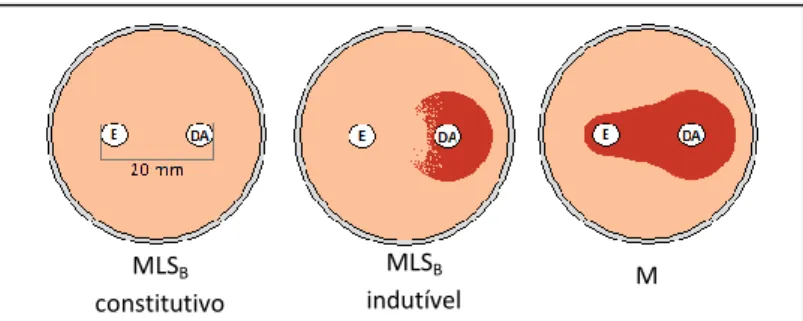
O fenótipo de resistência aos macrólidos foi determinado através do método de duplo disco, em que se colocam os discos de eritromicina e clindamicina a 20mm de distância (Figura 3). Estão descritos dois fenótipos distintos, M e MLSB. O fenótipo MLSB pode, por sua vez ser
16
Figura 3. Fenótipos de resistência aos macrólidos em Streptococcus pyogenes. (E), eritromicina, (DA),
clindamicina.
2.8. Detecção dos genes de resistência aos macrólidos
A detecção dos genes de resistência aos macrólidos, mef, erm(B) e erm(TR) foi feita por PCR multiplex num volume total de 10µL utilizando como molde o lisado bacteriano preparado por incubação com mutanolisina e hialuronidase de acordo com os protocolos e recomendações do CDC como descrito para a determinação do tipo emm (http://www.cdc.gov/ncidod/biotech/strep/protocols.htm). Os componentes utilizados e as suas concentrações finais foram respectivamente: tampão de PCR “Super Tth” (Enzime Technologies, UK) a 1X, DNA polimerase Super Tth a 0,0625U (Enzime Technologies, UK) e nucleótidos a 2mM cada (Fermentas, Vilnius, Lituânia). Os pares de primers utilizados foram mefA/E1/mefA/E2, ermB1/ermB2, ermTR1/ermTR2, com concentração de 0,05pmol/µL, 0,25pmol/µL e 0,4pmol/µL, respectivamente (Quadro 1, em anexo). O ciclo de PCR iniciou-se com um período de 5min a 94°C, ao qual se segue 40 ciclos, de 30s a 94°C, 30s a 51°C e 1min a 72°C.
Em todas as estirpes em que se detectou o gene mef fez-se um PCR adicional para distinguir o mef(A) do mef(E). Com 1µL dos mesmos moldes de DNA preparou-se uma reacção com um volume total de 20uL composta por tampão “green GoTaq flexi” a 1X (Promega, Madison, USA), 0.004U/µL de DNA polimerase GoTaq (Promega, Madison, USA), 0.125 mM de cada nucleotídeo (Fermentas, Vilnius, Lituania) e 3mM de MgCl2 (Promega, Madison, USA). Os
primers utilizados foram o mefR a 1,7pmol/µL em conjunto com mef1 e mef2, ambos a uma concentração final de 1pmol/µL. (Quadro 1, em anexo). O ciclo de PCR iniciou-se com um período de 4min a 94°C, ao qual se segue 30 ciclos, de 1mim a 94°C, 1mim a 56°C e 1min a 72°C. Para finalizar, 10min a 72°C.
2.9. Detecção dos genes de resistência à tetraciclina
Para a detecção dos genes que conferem resistência à tetraciclina [tet(L), tet(M), tet(K) e tet(O)] foi necessário realizar dois PCRs distintos.
A presença dos genes tet(L), tet(M) e tet(K) foi testada num PCR multiplex com um volume total de 50µL em que se adiciona 2µL de lisado bacteriano preparado por incubação
M MLSB indutível MLSB constitutivo C
17 com mutanolisina e hialuronidase de acordo com os protocolos e recomendações do CDC, como descrito para a determinação do tipo emm. A concentração final dos componentes utilizados foi tampão “green GoTat Flexi” 1X (Promega, Madison, Wi, USA), 0,2mM de cada nucleotídeo (Fermentas, Vilnius, Lituania), 2mM de MgCl2 (Promega) e 0,05U de DNA
polimerase GoTaq (Promega). Os 3 pares de primers utilizados foram tetL-up/tetL-rev, tetM-up/tetM-rev e tetK-up/tetK-rev, todos a uma concentração final de 0,2pmol/µL (Quadro 1, em anexo). O programa utilizado consiste em 35 ciclos de 1min a 95°C, 1 min a 50°C e 1min e 30s a 72°C. Por fim 5min a 72°C. A presença de tet(O) foi analisada à parte com os primers tetO-up/tetO-rev (Quadro 1, em anexo) em que a concentração final dos componentes no PCR e a quantidade de DNA molde foram iguais às utilizados no multiplex. O programa apenas diferiu na temperatura de hibridação, que é de 55°C em vez de 50°C.
2.10. Análise estatística
Para determinar a significância estatística entre resultados recorreu-se sempre que possível ao teste de Fisher,bilateral (http://www.graphpad.com/quickcalcs/contingency1.cfm). Utilizou-se o teste de Cochran-Armitage (Altman, 1999) de forma a avaliar se existia alguma tendência significativa para os valores de resistência aos macrólidos e às tetraciclinas ao longo dos anos.
Os resultados dos métodos de tipagem foram comparados através do cálculo do índice de diversidade de Simpson e do coeficiente de Wallace, assim como os respectivos intervalos de confiança a 95% (IC95%) (www.COMPARINGPARTITIONS.INFO). O índice de diversidade de Simpson (SID) indica a diversidade de uma população. Este índice é definido como a probabilidade de duas estirpes escolhidas aleatoriamente de uma amostra apresentarem classificações diferentes num determinado método de tipagem (Grudmann et al., 2001). Por exemplo, quanto maior o SID relativamente ao tipo emm, menor é a probabilidade de duas estirpes escolhidas ao acaso apresentarem igual tipo emm. O coeficiente de Wallace indica a probabilidade de duas estirpes consideradas relacionadas por um determinado método de tipagem ficarem também relacionadas quando se recorre a outro método de tipagem (Wallace
18
3. RESULTADOS
3.1. Idade e sexo dos indivíduos de onde foram recolhidas as estirpes
Das 320 estirpes de S.pyogenes estudadas, 154 (48,1%) foram recolhidas a partir de infecções em indivíduos do sexo feminino e 166 de infecções em indivíduos do sexo masculino (51,9%). As idades variaram dos 7 dias aos 97 anos. Através da análise por décadas de idades verificou-se que a maioria dos casos ocorreu em crianças entre os 0 e 9 anos de idade (21,3%) (informação não apresentada). Para analisar a relação entre idade e o sexo dos indivíduos dos quais foram recolhidos as estirpes,
separaram-se as estirpes em três classepararam-ses: crianças (<18), adultos (18-64) e idosos (>64) (Figura 4). Até aos 64 anos de idade, embora não seja estatisticamente significativo, indivíduos do sexo masculino foram os mais afectados (Teste de Fisher: p=0.32). Nos idosos (>64) a maioria das estirpes foram recolhidas de indivíduos do sexo feminino.
3.2. Tipagem emm
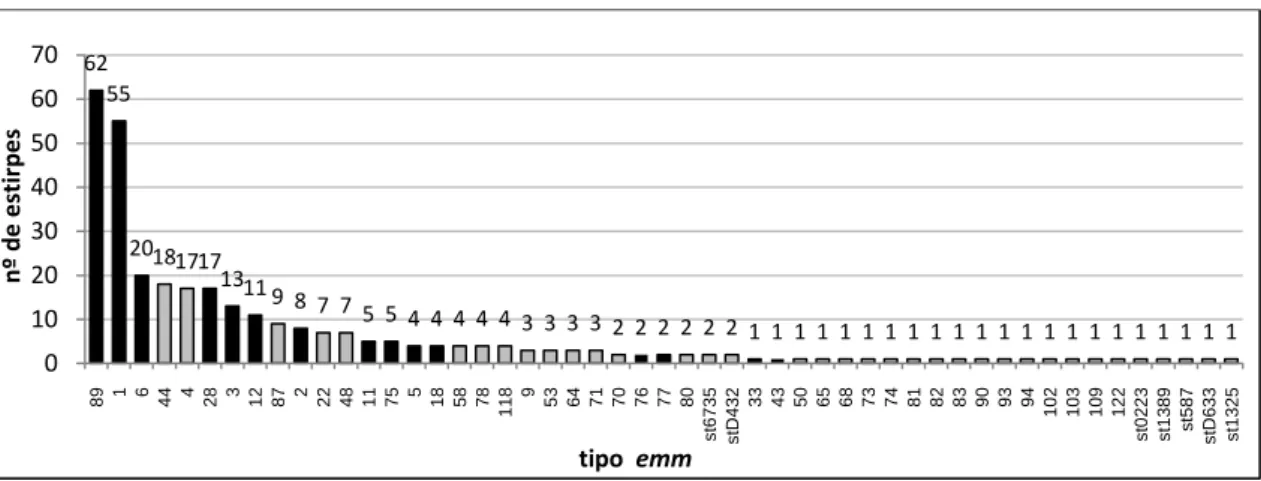
Neste estudo foram identificadas estirpes de 51 tipos emm (SID= 0,916 [IC 95% 0,899-0,933]). Duas estirpes apresentaram sequências novas que foram submetidas à base de dados: um novo tipo do gene emm (st1325) e um novo subtipo do gene emm87 (emm87.6). As estirpes dos tipos emm89 (n=62 estirpes) e emm1 (n=55 estirpes) foram as mais frequentes, constituindo em conjunto, 36,6% da população. Em seguida, as estirpes mais frequentes foram as dos tipos emm6 (n=20), emm44 (n=18), emm4 (n=17) e emm28 (n=17) que em conjunto com as dos tipos emm89 e emm1 correspondem a 59.1% do número total de estirpes (Figura
62 55 20181717 1311 9 8 7 7 5 5 4 4 4 4 4 3 3 3 3 2 2 2 2 2 2 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 0 10 20 30 40 50 60 70 89 1 6 44 4 28 3 12 87 2 22 48 11 75 5 18 58 78 118 9 53 64 71 70 76 77 80 st6 7 3 5 stD4 3 2 33 43 50 65 68 73 74 81 82 83 90 93 94 102 103 109 122 st0 2 2 3 st1 3 8 9 st5 8 7 stD6 3 3 st1 3 2 5 n º d e e sti rp e s tipo emm
Figura 4. Relação do número de estirpes por classes de
idades e sexo dos indivíduos infectados.
Figura 5. Tipos emm mais prevalentes na população. As barras a preto correspondem aos tipos emm incluídos na
futura vacina 26-valente. O rótulo por cima das barras corresponde ao número de estirpes observadas do respectivo tipo emm.
47 78 42 33 73 47 0 20 40 60 80 100 120 140 160 (<18) [18-64] (>64) n º d e e sti rp e s Classe de idade F M
19 5). Dos 26 tipos emm incluídos na futura vacina que está de momento em ensaios clínicos, 15 foram identificados neste estudo e correspondem a 67.8% do número total de estirpes (Figura 5).
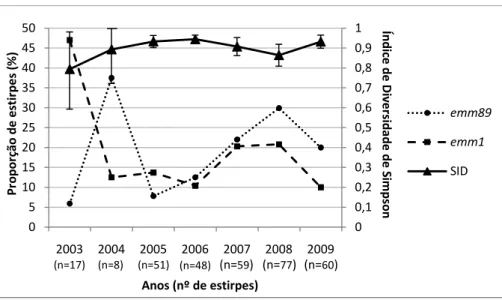
3.2.1. Prevalência de estirpes dos tipos emm1 e emm89
Como apresentado na figura 6, entre 2003 e 2004, existiram grandes alterações na proporção de estirpes dos tipos emm1 e emm89. Isto ocorre provavelmente devido ao reduzido número de estirpes recolhidas durante estes anos. Em 2005 o número de estirpes do tipo emm1 foi ligeiramente superior ao número de estirpes do tipo emm89. Nos restantes anos, estirpes do tipo emm89 foram sempre as mais prevalentes. De 2006 para 2007 verifica-se um aumento de estirpes com ambos os tipos, verifica-seguido em 2008 de uma estabilização no número de estirpes do tipo emm1 e um contínuo aumento no número de estirpes do tipo
emm89. Em 2009, embora não seja significativo, estirpes de ambos os tipos emm diminuiram
na população (teste de Fisher: emm1 [p=0,10]; emm89 [p=0,24]).
Figura 6. Relação entre a distribuição das estirpes dos dois tipos emm mais prevalentes e o índice de diversidade de
Simpson para todos os tipos emm da população.
3.2.2. Tipo emm vs idade dos indivíduos de onde foram recolhidas as estirpes.
De modo a avaliar se existia alguma relação entre o tipo emm das estirpes e a idade dos indivíduos de onde estas foram recolhidas, analisaram-se estas duas características em conjunto para as estirpes dos tipos emm mais prevalentes (≥5 estirpes). De facto, as estirpes da maioria dos tipos emm apresentados no gráfico da figura 7 causam infecções nas 3 classes de idades. Para as estirpes dos tipos emm28, emm22 e emm48 embora se tenham recolhido poucas ou nenhumas de crianças, estas diferenças não são significativas (teste de Fisher:
emm28 [p=0,08]; emm22 e emm48 [p=0,20]).
0 0,1 0,2 0,3 0,4 0,5 0,6 0,7 0,8 0,9 1 0 5 10 15 20 25 30 35 40 45 50 2003 2004 2005 2006 2007 2008 2009 Ín d ic e d e Di ve rsi d ad e d e S imp so n Pr o p o rç ão d e e sti rp e s (% ) Anos (nº de estirpes) emm89 emm1 SID (n=17) (n=8) (n=51) (n=48) (n=59) (n=77) (n=60)
20
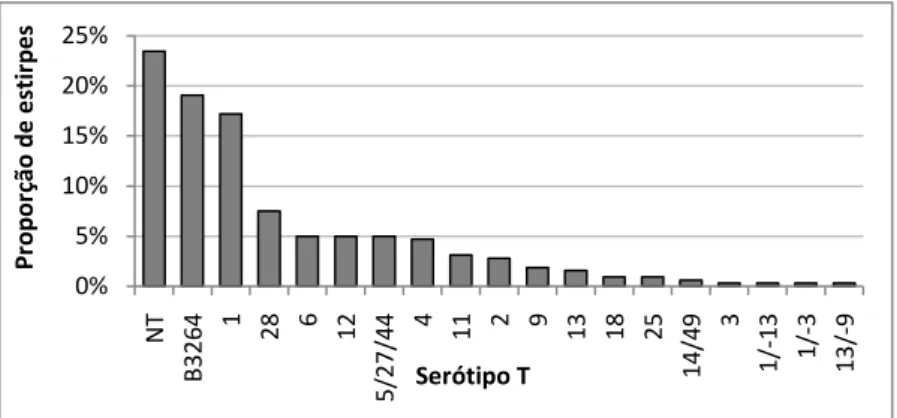
3.3. Serotipagem T
Relativamente à serotipagem T identificaram-se 19 serotipos ou conjugações de serotipos diferentes (SID=0,864 [IC 95% 0,846-0,882]) (Figura 8). No total, 23,4% das estirpes (n=75) foram consideradas não tipáveis. Em seguida, 19% (n=61) aglutinaram com o soro TB3264 sendo maioritariamente estirpes do tipo emm 89, e 17% aglutinaram com o soro T1, sendo estas maioritariamente do tipo emm1 (Quadro 2, em anexo).
3.4. Perfis de toxinas estreptocócicas
Para as 320 estirpes deste estudo foram identificados 50 perfis de toxinas distintos (SID=0,934 [IC 95% 0,922-0,946]) (Quadro 3, em anexo). Em todas, à excepção de uma, detectou-se a presença dos genes speB e speF. A ausência destes genes nessa estirpe foi confirmada através de hibridação por “Southern blot”. Os genes smeZ, speG, speJ, frequentemente considerados cromossomais, foram detectados em 95,9% (n=307), 94,1% (n=301), e 40,3% (n=129) do total de estirpes, respectivamente (Quadro 4, em anexo). Dos genes codificados em fagos, o mais prevalente foi o speC (57,8%, n=185), seguido do speA (32,8%, n=105), ssa (20,6%, n=66), speH (16,3%, n=52) e speK (16,3%, n=52), speI (14,1%, n=45), speM (6,9%, n=22) e speL (5,9%, n=19) (Quadro 4, em anexo).
0 10 20 30 40 50 60 70 89 1 6 44 4 28 3 12 87 22 48 11 75 n º d e e sti rp e s tipo emm >64 anos [18-64] anos <18 anos 0% 5% 10% 15% 20% 25% NT B3264 1 28 6 12 5/27/44 4 11 2 9 13 18 25 14/49 3 1/ -13 1/-3 13/ -9 Pr o p o rç ão d e e sti rp e s Serótipo T
Figura 8. Prevalência das estirpes com os diferentes serótipos T.
21 Ao comparar os perfis de toxinas das estirpes dos tipos emm mais prevalentes, verificou-se uma consistente presença ou ausência de determinados genes de toxinas em estirpes com o mesmo tipo emm (Quadro 3 e 4, em anexo).
Tendo em conta os resultados apresentados no quadro 4, em anexo, a presença do gene
speA foi detectada apenas em estirpes de 4 tipos emm distintos, sendo esta presença,
significativa em estirpes dos tipos emm1, emm6 e emm3. Em contraste com o gene speA, o gene speC está disperso por estirpes de vários tipos emm. No entanto, em nenhuma das estirpes do tipo emm3, foi detectada a presença deste gene. Os genes speG e smeZ, considerados cromossomais, foram amplamente detectados nas estirpes do estudo. Curiosamente, nenhuma estirpe do tipo emm4 apresenta o gene speG e nenhuma estirpe do tipo emm2 apresenta o gene smeZ. Relativamente ao gene speJ, com excepção das estirpes do tipo emm89, encontra-se presente em todas as estirpes dos outros tipos emm em que foi identificado. O gene speK parece ser característico das estirpes do tipo emm6, pois foi o único tipo emm em que este gene foi detectado em todas as estirpes. A presença do gene ssa foi detectada em todas as estirpes dos tipos emm4 e emm3.
Os genes speH e speI foram ambos detectados em 45 estirpes (14,1%) e considerados significativamente associados a estirpes dos tipos emm44, emm12, emm48 e emm11, embora tenham sido também detectados em estirpes de outros tipos. O gene speI nunca foi detectado na ausência do gene speH. A presença conjunta dos genes speL e speM foi detectada em 19 estirpes (5,9%) e estatisticamente associada a estirpes dos tipos emm2 e emm75. Em 3 estirpes, todas do tipo emm58, apenas se detectou o gene speM. A ausência do gene speL e a presença do gene speM foi confirmada por hibridação “Southern blot”. Pelo contrário, o gene
speL nunca foi detectado na ausência do gene speM.
3.5. Resistência a antimicrobianos e respectivos génotipos associados.
Todas as estirpes do estudo eram susceptíveis à penicilina, ao linezolide e à vancomicina. Relativamente ao cloranfenicol encontrou-se uma estirpe resistente. À levofloxacina detectaram-se duas estirpes não susceptíveis, cujos resultados foram confirmados através de E-teste (CIM=6 e 3 mg/mL).
A resistência aos macrólidos foi detectada em 10.3% do total de estirpes analisadas (n=33). Durante os anos incluídos no estudo verificou-se uma diminuição na proporção de estirpes resistentes (Figura 9), porém sem significância estatística (teste de Cocharan-Armitage, p=0.18). No primeiro ano (2003) foi quando se verificou o valor mais elevado de resistência (29,4%). Em 2004 ocorreu uma grande diminuição (12,5%), seguida de um aumento para 19,6% em 2005. De 2005 para 2007 os valores decresceram (19,6% em 2005, 16,7% em
22 2006, 5,1% em 2007), sendo esta diminuição mais acentuada de 2006 para 2007. Entre 2007 e 2008 houve uma estagnação (5,1% em 2007, 5,2% em 2008) e em 2009 voltou-se a verificar uma descida na resistência (3,3%) (Figura 9). Embora a diminuição dos valores ao longo dos anos não tenha sido significativa pelo teste de Cocharan-Armitage, se utilizarmos o teste de Fisher para comparar os períodos de 2003 a 2006 e 2007 a 2009, verifica-se que a diminuição foi significativa (teste de Fisher: p<0.0001). Esta discordância é provavelmente devida ao reduzido número de estirpes nos primeiros anos de estudo, nomeadamente em 2003 e 2004, que se traduz num elevado erro associado. Este factor pode também ser a explicação para o valor irregularmente baixo de resistência em 2004, relativamente a 2003 e 2005 (Figura 9). De facto, a diminuição dos valores de 2003 para 2004 e o subsequente aumento de 2004 para 2005 não foram significativos (teste de Fisher: p=0,62; p=1,00, respectivamente).
Figura 9. Resistência à eritromicina e respectivos fenótipos associados.
Das 33 estirpes resistentes aos macrólidos, 22 apresentaram o fenótipo cMLSB (66,7%)
e 11 apresentaram o fenótipo M (33.7%). O fenótipo cMLSB, com excepção nos anos 2003 e
2007, foi o mais comum (Figura 9). Entre 2003 e 2006 as alterações registadas para os valores de resistência aos macrólidos foram sobretudo devido às alterações no número de estirpes de fénotipo M. A partir de 2006, o número de estirpes de fenótipo cMLSB foi o principal
determinante na variação da resistência total (Figura 9). Em todas as estirpes de fenótipo cMLSB foi detectada a presença do gene erm(B). Na população bacteriana em estudo, estas
estirpes são maioritariamente dos tipos emm11 ou emm28. Das 11 estirpes que apresentavam fenótipo M, detectou-se em 10, a presença do gene mef(A). Noutra foi detectada a presença de mef(E), que curiosamente corresponde também à única estirpe resistente ao cloranfenicol. A resistência aos macrólidos por mefA (fenótipo M) parece não ser característica de nenhum tipo emm específico, estando distribuída por estirpes do tipo emm1, emm4, emm9, emm12,
emm18 e emm89 (Quadro 2, em anexo). 0 10 20 30 40 50 60 2003 2004 2005 2006 2007 2008 2009 Pr o p o rç ão d e e sti rp e s (% ) Anos Resistência aos Macrólidos fenótipo M fenótipo MLSb fenótipo cMLSB
23 Figura 10. Resistência à tetraciclina.
A resistência à tetraciclina foi observada em 47 estirpes (14,7%). No total dos anos não se verificou nenhuma tendência significativa na evolução dos valores (teste de Cochran-Armitage, p=0,48) (Figura 10). A diminuição dos valores de 2006 para 2007 (teste de Fisher: p=0,08), assim como o aumento em 2008 (teste de Fisher: p=0,12) não foram estatisticamente significativos. Na maioria destas estirpes, a resistência à tetraciclina foi devida à presença exclusiva do gene tet(M) (89,4%). Em 3 estirpes para além da presença do gene tet(M) foi também detectada a presença do gene tet(L) e em duas estirpes detectou-se apenas a presença do gene tet(O) (Quadro 2, em anexo).
Das 47 estirpes resistentes à tetraciclina, 12 apresentaram também resistência aos macrólidos: 10 de fenótipo cMLSB e 2 de fénotipo M. Os gene erm(B) (fenótipo cMLSB) e o gene tet(M) foram detectados em conjunto em todas as estirpes dos tipo emm11 e emm76. Embora
se tenha detectado o gene erm(B) em 70,6% das estirpes de emm28, apenas uma foi também resistente à tetraciclina.
Relativamente à bacitracina, detectaram-se 12 estirpes resistentes, todas do tipo emm28 e com fenótipo cMLSB.
3.6. Electroforese em campo pulsado
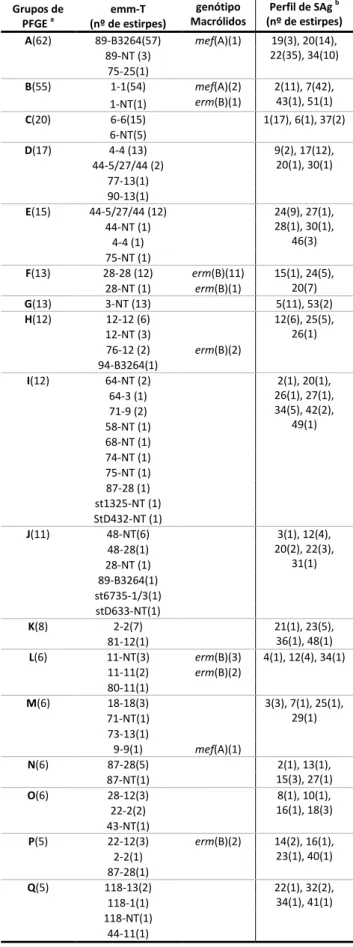
O dendrograma obtido com base nos perfis de macro-restrição das 320 estirpes por PFGE é constituído por 17 grupos (n≥5 estirpes; coeficiente de Dice≥80%) (SID=0,916 [IC95% 0,899-0,933]) que incluem 85% do total das estirpes (n=272) (Figura 11). As 48 estirpes restantes (15%), ou apresentaram perfis únicos ou estavam incluídas em grupos com menos de 5 estirpes. Os dois maiores grupos definidos por PFGE, A e B, em concordância com a tipagem
emm, são principalmente constituídos por estirpes TB3264/emm89 e T1/emm1,
respectivamente (Quadro 2). Apenas 5 dos 17 grupos de PFGE principais foram homogéneos no tipo emm (B, C, F, G e N) (Quadro 2). Todos os outros incluíram estirpes com pelo menos dois tipos emm distintos ainda que um fosse sempre predominante.
0 10 20 30 40 2003 2004 2005 2006 2007 2008 2009 Pr o p o rç ão d e e sti rp es (%) Anos Resistência à tetraciclina MLSb p=0.08 p=0.12 cMLSB