U
NIVERSIDADE
C
ATÓLICA DE
B
RASÍLIA
PRÓ-REITORIA DE PÓS- GRADUAÇÃO SCRICTU
SENSU EM CIENCIAS GENÔMICAS E BIOTECNOLOGIA
MESTRADO
Brasilia
2009
Variabilidade em locos associados ao
glaucoma primário de ângulo aberto
Autor (a): Erica Carine Campos Caldas
Erica Carine Campos Caldas
Variabilidade em locos associados ao
glaucoma primário de ângulo aberto
Dissertação apresentada ao programa
de Pós-Graduação
Stricto Sensu
em
Ciências Genômicas e Biotecnologia da
Universidade Católica de Brasília, como
requisito para obtenção do Título de
Mestre em Ciências Genômicas e
Biotecnologia.
Orientador:
Dr. Rinaldo Wellerson
Pereira
Ficha elaborada pela Biblioteca Pós-Graduação da UCB C145v Caldas, Erica Carine Campos
Variabilidade em locos associados ao glaucoma primário de ângulo aberto. / Erica Carine Campos Caldas. – 2009.
115f.; : il.; : 30 cm
Dissertação (mestrado) – Universidade Católica de Brasília, 2009. Orientação: Rinaldo Wellerson Pereira
1. Glaucoma. 2. Manifestações oculares de doenças. 3. Visão. I. Pereira, Rinaldo Wellerson, orient. II. Título.
DEDICATÓRIA
Ao meu Senhor Jesus Cristo por sua preciosa e doce presença em
minha vida e em todos os momentos de minha vida.
Aos meus pais (in memorian) pelo incentivo,
acesso a educação, formação e por terem
me ensinado a persistir e não desistir diante das difilculdades.
AGRADECIMENTOS
Ao meu orientador Prof Dr. Rinaldo Pereira pela confiança, orientação,
paciência, apoio e principalmente por me ensinar a ser mais independente e
criativa. Agradeço pela oportunidade e aprendizado.
Aos professores Dr Otávio Toledo e Dra Cristine Póvoa pela contribuição
prestada durante a avaliação desta Dissertação.
Ao Prof. MSc. Túlio Lins pela amizade, sugestões e apoio durante a realização
deste estudo.
As Profs(a) Dras Rosângela Vieira e Eliza Duarte pela confiança, incentivo,
amizade, sugestões e pelos momentos de descontração.
Ao Prof Dr. Ruy Caldas e a todo corpo Docente da Pós-Graduação em
Ciências Genômicas em especial ao Prof Dr. Dario Grattapaglia pelo
fornecimento das amostras para a realização deste estudo e uso de
equipamentos.
Aos alunos de iniciação cientifica, demais alunos e colaboradores do Prof
Rinaldo : Marcela, Haony, Amanda, Priscila, Paula,Rodrigo,Thales, Alause,
Tailce,Gustavo, Larissa, Kátia, Ana Paula, Carol,Rômulo,Willian,Carla,
Danielle, obrigada pelo apoio no desenvolvimento deste projeto e pelos
momentos de descontração.
As amigas Rosana Falcão, Franciele, Kelly Barreto e Janaína Fernandes pelo
apoio, incentivo nos momentos tranquilos, difíceis e felizes.
A todos os colegas e amigos do Laboratório de Biotecnologia pelos momentos
de descontração e aprendizado.
Ao Conselho Nacional de Desenvolvimento Científico e Tecnológico pela bolsa
de estudos concedida .
Aos funcionários do Programa de Pós- Graduação pela atenção e apoio
durante esta etapa.
As amigas Ilma Julinda e Hilda Ferraz pelo amor, carinho, atenção, incentivo e
por terem me recebido tão bem em suas casas e famílias. Obrigada por todo
apoio nos momentos mais difíceis e por fazerem parte de minha vida.
Aos meus pastores e discipuladores, pessoas que exerceram e exercem
autoridade sobre minha vida e aos que oram por mim de perto e de longe.
Obrigada pelas orações .
EPÍGRAFO
“
Meu coração exulta no Senhor,
o meu poder está exaltado no Senhor; porquanto
me alegro na sua salvação...
porque O Senhor é o Deus da sabedoria.
O Senhor dá a sabedoria e da sua boca vem
o conhecimento e entendimento.
O Senhor empobrece e enriquece, abaixa
e também exalta. Levanta o pobre do pó, exalta
o necessitado para o fazer assentar entre os principes
”.
Resumo
indicaram que a distribuição haplotípica e os dados de desequilíbrio na
população brasileira, quando comparados com aquelas das populações do
HapMap apresentaram padrão intermediários aos das populações de Europeus
e Africanos. A transferibilidade de tagSNPs entre as populações (Yoruba e
Européia) do HapMap e a brasileira se mostrou incerta possivelmente por
populações por não possuírem heterogeneidade genética e serem adequadas
para serem utilizadas em estudos de associação genética quando a população
alvo é miscigenada. Os SNPs que compuseram um painel para utilização em
estudos de MALD foram então minerados em populações Européias e
Africanas no HapMap das regiões flanqueadas por 1Mpb de 10 microssatélites
autossômicos. Foram selecionados marcadores que apresentaram valor de
associação acima de 0,5. Todos os SNPs selecionados foram comparados ao
banco de dados de Smith et al., 2004 que apresenta 3,011 SNPs já mapeados
por MALD. Um total de 291 SNPs informativos de ancestralidade foram
recuperados sendo estes candidatos a explicar a prevalência de GPAA em
populações africanas e afro-descendentes.
Abstract
The glaucoma is a group of eye diseases that are characterized by gradually
evolving damage typical in the optic nerve with consequent changes in
vision,The most frequent type whose incidence is higher in individuals
belonging to the black population group is the primary open-angle glaucoma
(GPAA) which corresponds to approximately 50% of cases. Currently,
mutations in at least three genes (MYOC, OPTN and WDR36). have been
related to the manifestation of glaucoma with Mendelian segregation. The role
of these genes in the incidence of glaucoma as a multifactorial disease is still
unknown. The Brazilian population presents high genetic heterogeneity and it is
therefore targets for studies of genetic association. The mixing is an important
factor to be considered in relation to GPAA, as studies of genetic association,
mapping by linkage disequilibrium generated by the mixing (MALDI) confirm
this fact. The search for SNPs that explain the incidence of a disease or are
informative of ancestry is the subject of research by the scientific community,
these SNPS can be found in a database world, the HapMap. In this sense this
work was to evaluate variability over the MYOC gene in a sample of the general
population as a preliminary to their use in investigations of a case-control and
recover from ancestry informative SNPs for use in association studies by
linkage dusequilibrium generated by mixing (MALDI) in loci linked to GPAA
using the HapMap data. For both 15 tagSNPs were selected along the gene
using tools and data available in the database of the HapMap project. Besides
HapMap the dbSNP were used for comparison of allele frequencies and
analyzes the linkage disequilibrium.The 15 tagSNPs were genotyped in a
sample of the population composed of 200 unrelated individuals, using PCR
and extension of the basic single reactions (Snapshot, Applied Biosystems) in
both systems with multiplex capillary electrophoresis in ABI3130xl equipment.
Analysis of patterns of linkage disequilibrium for the tagSNPs selected were
considered to be a statistical tool for association studies of candidate genes.
The results indicated that the distribution of haplotypes and the data imbalance
in the population when compared with those of the HapMap populations
showed intermediate standard to the populations of Europeans and Africans.
The portability of tagSNPs between populations (Yoruba and European) of the
HapMap and the Brazilian was uncertain possibly because people do not have
a genetic heterogeneity and are suitable for use in genetic association studies
where the population is mixed. The SNPs that composed a panel for use in
studies of MALDI were mined in European and African populations in the
HapMap regions flanked by 1Mpb of 10 autosomal microsatellites. We selected
markers that showed association value above 0.5. All selected SNPs were
compared to database of Smith et al., 2004 shows that 3.011 SNPs already
mapped by MALDI. A total of 291 SNPs informative for ancestry were
recovered which are candidates to explain the prevalence of GPAA in African
populations and african-descendants
LISTA DE FIGURAS
! "
! #$$%
#$
#&'()
(
*
+,-. /*
*
0
"1
! 2
(
! #$$3
#4
5&+
67 (
- 8
9
: 6;
(
* (
(
(
( <=
(
8 >!
(
! #$$?
5$
@& '
0
(
(
(
(
(
=
(
(
:
(
(
(
'
(#5%?3$!
(#5%?3A!. (A$545$ B ( (#54 ?!
%@
%& '
0
(
(
(
(
=
( "
(
:
(
( ( (
(A@#%5A5!
(3%5A$5!. (#$5#%%% B (##5A?3%
%@
A&'
0
(
(
(
(
( "
(
:
(
(
(
(3%@%A@A!
( $4 5553@!. ( $4 5??? B ( #$5%4A$
%%
3& '
0
(
(
(
(
( "
(
:
(
(
(
( #$3555A!
( #$3A 5@ . ( #$?#%35
%%
?&'
0
(
(
(
(
( (
+
=
( (
"
(
+,-.
(
0
; .
- (
.-+ $?5
(
(
; "
(
"'"
$A
%A
4&'
0
(
(
(
( (
+
= #
"
(
+,-.
(
0
;
(
'+%44
%A
$&
0
( 0 )CD
( E
(
+,-.
(
67 (
(
F +
.'G ,HI
A$
&B ( ) *
6;
( %
( (
(
+,-.
(
67 (
F +
,HI
.'G -( )
(
1 (
BJK
9-B L #M-(
: (
BJK
9-B N# -( )
(
=
BJ
A5
#&
;
B ( ) *
6;
( %
"
( (
(
6;
(
-( )
(
1
BJK
9-B L # -( : (
BJK
9-B O
# -( )
(
=
BJ
AA
5&
;
B ( ) *
6;
( %
"
(
(
67 ( ,HI
H
-( )
(
1
BJK
9-B L # -( : (
BJK
9-B O #
-( )
(
=
BJ
AA
@&
;
B ( ) *
6;
( %
"
(
+,-.
(
67 ( .'G
H
'
6; .'G '
-( )
(
1
BJK
9-B L # -( : (
BJK
9-B O # -( )
(
=
%&
;
( ) *
6;
6;
(
?
(
+,-. -( )
(
1
BJK
9-B L # -( : (
BJK
9-B
O # -( )
(
=
BJ
A3
A&
;
( )
6;
(
67 (
(
! ,
'
E
-( )
(
1
BJK
9-B L # -( : (
BJK
9-B O
# -( )
(
=
BJ
A?
3& I
(
(
7 (
B#"5A@
B#" 3
:
(
((
#
34
?& I
(
(
7 (
B#"5#%
B#"# ?4
:
(
((
#
?$
4& I
(
(
7 (
B5" 3A?
B%"@#@
:
((
5
%
(
?
LISTA DE TABELAS
8
&
(
((
(
#@
8
#&8
(
(
(
(
(
(
(
(
*
(
:
(
6;
@@
8
5& -
*
(
+,-.
:
(
.H
%$
8
@& I
(
6;
( )
%
8
%&8
1 (
( 0
( (
(
(
(
( %
"
(
+,-.
(
(
%5
8
A&
)CD
( E
(
( %
"
(
+,-.
(
67 (
(
!
,
'
E
%?
8
3&
)CD
(
*
(
( %
"
(
%4
8
?&P
(
F
:
(
(
(
E
(
(
(
0
(
=
(1
(
6;
(
,
(
:
(
F +
P
(
(
(
N$!$%
A
8
4&P
(
F
:
(
(
(
E
(
(
(
0
(
=
(1
(
6;
(
'
E
(
:
(
F +
P
(
(
(
N$ $%
A
8
$& F
( 0
(
(
(
+,-.
6; .'G
A5
8
& P
(
BJ!
(
((
1 6 !
(
0
6
(
>
"
(
+,-.
6;
(
A@
8
# & P
(
BJ!
(
((
1 6 !
(
0
6
(
>
"
(
+,-.
6; F +
Q,
A@
8
5&P
(
BJ&
(
((
1 6 !
(
0
6
(
>
"
(
+,-.
6; .'G
A%
8
@& F
(
(
( 0 )CD
(
(
"
(
+,-.
A4
8
%&
)
( 1
*
(
(
67 ( ,
!
(
'
E
3$
8
A& 9
(
(( E
(
7 (
(
34
8
3&"
( )
(
B
$!%
(
(
0
6
0 )CD
(
7 ( B#"
3 B#"5A@
?$
8
?&"
( )
(
B
$!%
(
(
8
4& "
( )
(
B
$!%
(
(
0
6
0 )CD
; B%"@#@
?#
8
#$&"
( )
(
B
$!%
(
(
0
6
0 )CD
; B%"#%$
?5
8
# &"
( )
(
B
$!%
(
(
0
6
0 )CD
; B4"#4$
?@
8
##&"
( )
(
B
$!%
(
(
0
6
0 )CD
; B $"#
?%
8
#5&"
( )
(
B
$!%
(
(
0
6
0 )CD
; B
"453
?A
8
#@&"
( )
(
B
$!%
(
(
Sumário
AGRADECIMENTOS ... vi
EPÍGRAFO ... viii
Resumo ... ix
Abstract... xi
LISTA DE TABELAS ... xiv
Sumário ... xvi
1. Introdução ... 18
1.1. Glaucoma ... 18
1.2. Glaucoma Primário de Ângulo Aberto ... 19
1.3. Mecanismos genéticos e susceptibilidade do Glaucoma do Adulto ... 22
1.4. Genes candidatos para o Glaucoma Primário de Ângulo Aberto (GPAA) ... 25
1.5. Polimorfismos de uma única base (SNPs) e estudos de associação ... 33
1.6. Transferibilidade de tagSNPs ... 34
1.7. SNPs informativos de ancestralidade ... 36
1.8. Estudos de associação na população brasileira ... 38
2. Hipótese de trabalho ... 41
3. Objetivos ... 41
3.1. Geral ... 41
3.2. Específicos ... 41
I-Análises de desequilíbrio de ligação para tagSNPs do gene MYOC na população brasileira . 42 4. Material e Métodos ... 43
4.1. Amostras biológicas utilizadas nesse estudo ... 43
4.2. Extração de DNA ... 43
4.3. Seleção de tagSNPs nos gene MYOC ... 43
4.4. Genótipos do HapMap ... 43
4.5. Desenho de sistemas de genotipagem por minisequenciamento por SNaPshot® Multiplex Single Base Extension Kit ... 45
4.6. Amplificação pela PCR e da reação de minisenquenciamento ... 46
4.7. Amplificação em multiplex ... 46
4.8. Purificação enzimática dos produtos da PCR ... 46
4.9. Genotipagem pela reação de minisequenciamento ... 47
4.10. Detecção de fragmentos e obtenção dos genótipos ... 47
4.11. Análise descritiva dos SNPs genotipados ... 48
4.12. Análise de Haplótipos e Desequilíbrio de Ligação ... 48
5. Resultados ... 49
5.1. tagSNPs selecionados no gene MYOC ... 49
5.2. Amplificação pela PCR e Genotipagem por minisenquenciamento... 52
5.4. Análises de desequilíbrio de ligação ... 62
6. Discussão ... 71
II-Mineração de SNPs informativos de ancestralidade em locos ligados ao Glaucoma primário de ângulo Aberto (GPAA) ... 77
7. Material e Métodos ... 78
7.1. Seleção de locos de microssatélites ligados ao GPAA ... 78
7.2. Recuperação de SNPs em regiões genômicas flanqueando microssatélites com pico de ligação com GPAA ... 78
7.3. Seleção de SNPs informativos de ancestralidade ... 78
7.4. Resultados ... 79
7.5. Discussão ... 89
8. Conclusões ... 91
9. Referências Bibliográficas ... 92
1.
Introdução
1.1. Glaucoma
Glaucoma é uma designação geral para um grupo de doenças que apresentam em comum neuropatia óptica degenerativa e a perda de células da retina, geralmente associadas à elevação da pressão intra-ocular (PIO) que levam a graves alterações no campo visual, fazendo do glaucoma uma das principais causas de cegueira em todo o mundo (Quigley, 1993; Povoa, T et al., 2001; Povoa, Santiago et al., 2006; Sakata, 2006).
O glaucoma pode ser classificado de acordo com sua etiologia, aspecto anatômico do seio camerular e idade de aparecimento. Entre os grupos de glaucoma há aqueles classificados como primário, onde os mecanismos da elevação da PIO não são bem definidos. O glaucoma primário de ângulo aberto (GPAA) é caracterizado pela não obstrução dos canais de saída da câmara interior do globo ocular. Uma vez que os canais forem obstruídos pela íris caracterizando uma falha da malha trabecular, o glaucoma é classificado como sendo primário de ângulo fechado (GPAF) (Vasconcellos, 2001). O glaucoma é classificado de secundário quando os mecanismos do aumento da PIO são conhecidos (Leske, Ederer et al., 1981; Sakata, 2006). No entanto, a etiologia do glaucoma é geralmente multifatorial. Processos isquêmicos, auto-imunes, intervenções cirúrgicas e fatores genéticos estão associados ao glaucoma (Wiggs, 2007).
O diagnóstico do glaucoma é feito após um exame oftalmológico apurado aferindo sempre a pressão intra-ocular e exames de fundo de olho como: tonometria de aplanação, gonioscopia, curva tensional diária, prova de sobrecarga hídrica, campimetria computadorizada, campimetria de freqüência duplicada (FDT), retinografia simples, estereofotografia de papila e topografia de disco óptico (HRT) (Lauretti e Filho, 1997; Netland, 2007).
Em geral a pressão intra-ocular acima de 24 mmHg é indicada como um dos fatores de risco para o desenvolvimento do glaucoma, mas outros fatores de risco como idade, etnia, hereditariedade, miopia, história familiar e diabetes melitus (DM) também contribuem para o desenvolvimento deste grupo de patologias (Vasconcellos, 2001; Betinjane, Silva et al., 2005; Fan, Wang et al., 2006).
com a alteração genética encontrada e o entendimento destas mutações pode ser aplicado no diagnóstico precoce da doença para melhor tratamento (Vasconcellos, 2001; Samples, 2003; Cella, 2005).
1.2. Glaucoma Primário de Ângulo Aberto
O GPAA pode ser subdividido em dois subtipos: o glaucoma de pressão intra-ocular alta e o glaucoma de pressão normal (GPN) (Tavares e Mello, 2005). Esta classificação se baseia no nível da pressão intra-ocular (PIO) cujo valor limitante é de 21 mmHg (Shields, Ritch et al., 1996; Sakata, 2006). O glaucoma de pressão normal (GPN) é uma neuropatia óptica caracterizada pela diminuição da camada de fibras nervosas da retina, aumento da relação escavação/disco e defeito de campo visual, porém sem evidência de elevação da pressão ocular. Alguns autores sugerem que o GPN seja na realidade uma neuropatia óptica hereditária, causada por uma disfunção mitocondrial (Buono, Foroozan et al., 2002). O diagnóstico do GPN deve ser feito por exclusão após extensa investigação de outras causas através de cuidadosa coleta da história familiar e exame ocular sistêmico (Shields, 1998).
O glaucoma de pressão alta é caracterizado pela evidência do aumento da pressão intra-ocular que apresenta sua relação causal com o desenvolvimento da doença (Armaly, Krueguer et al., 1980; David, Zangwill et al., 1987). O glaucoma primário de ângulo aberto de pressão alta pode ser subdividido em glaucoma Juvenil (JOAG) e glaucoma do adulto (Vasconcellos, 2001). O GPAA pode ser subdividido em dois grupos de acordo com a idade do seu aparecimento, com sua severidade e de acordo com o padrão de herança.
Quando a doença é diagnosticada após os 35 anos, tem curso insidioso e padrão de herança freqüentemente não definido sendo chamada de glaucoma crônico simples (GCS) ou GPAA do adulto. O glaucoma diagnosticado entre 3 e 35 anos com curso agressivo, pressões intra-oculares bastante elevadas e em geral padrão de herança autossômica dominante é chamado glaucoma juvenil (JOAG) ou glaucoma primário de ângulo aberto de início precoce. Estes dois quadros clínicos já foram considerados doenças diferentes, entretanto, atualmente, tende-se a admitir como a mesma doença (Morissette, Cote et al., 1995; Povoa, Santiago et al., 2006)
Doenças como o diabetes melitus e a hipertensão arterial além de outros fatores de risco como idade, grupo populacional, história familiar (hereditariedade) e uso de esteróidespodem contribuir para o desenvolvimento do glaucoma de ângulo aberto. Assim, o GPAA deve ser considerado como uma doença multifatorial (Becker, Kolker et al., 1966; Weinstein, Munnangi et al., 1985; Quigley e Broman, 2006).
Estudos relatam que o GPAA apresenta estimativas de prevalência e pior prognóstico em indivíduos de etnia negra quando comparados a asiáticos e europeus. Em Baltimore um estudo clássico avaliou populações com diferentes características raciais dentro de uma mesma amostra comparando a prevalência de GPAA entre brancos e negros e observou que os indivíduos negros apresentavam taxas de prevalência de GPAA 4,97% maiores do que os pacientes brancos (Tielsch, Katz et al., 1991).
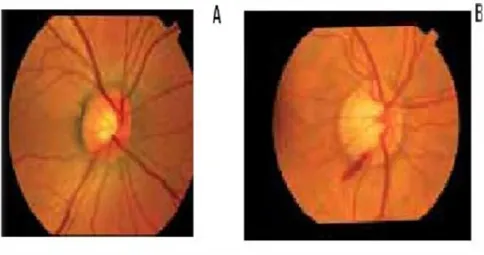
Figura 1.Nervo óptico. A - Nervo óptico normal. B-Nervo óptico com glaucoma primário de ângulo aberto (GPAA).Adaptado de (Betinjane, Silva et al., 2005)
.
Em um estudo realizado em uma população do sul do Brasil, a prevalência do GPAA em indivíduos de autodenominados brancos (2,03%, ajustada idade/grupo populacional) foi semelhante às taxas de prevalência observadas em estudos realizados em populações de origem européia (Tielsch, Katz et al., 1991; Sakata, 2006) e hispânica (Quigley, 2001).
A prevalência de GPAA em indivíduos do grupo populacional não-branca (3,58%,comparando idade/grupo populacional) foi menor do que a observada em investigações realizadas em populações negras, mas semelhante à prevalência do glaucoma primário de ângulo aberto na população mestiça do estudo de Barbados (Leske, Connell et al., 1994).
1.3. Mecanismos genéticos e susceptibilidade do Glaucoma do Adulto
O glaucoma, em suas várias formas de manifestação, apresenta padrões de herança que vão da segregação mendeliana dominante ou recessiva a formas de herança multifatorial ou complexa (Libby, Gould et al., 2005; Gjessing e Lie, 2008). O GPAA de início adulto é classicamente reconhecido como uma doença com padrão de herança complexa, enquanto que o glaucoma primário de ângulo aberto juvenil (JOAG) apresenta herança autossômica dominante e de alta penetrância (Lichter, 1994; Povoa, Santiago et al., 2006). Algo que se tem buscado é entender o papel de locos identificados em glaucomas com padrão de herança mendeliana na manifestação do glaucoma como fenótipo complexo.
A maioria dos casos de glaucoma juvenil apresenta um padrão autossômico dominante. Esse tipo de glaucoma está geneticamente ligado ao cromossomo (1q21-31) ou ao gene MYOC, ou à co-existência de variações alélicas nos genes MYOC e CYP1B1 do glaucoma congênito. Algumas variações alélicas severas específicas do gene MYOC como a mutação Gly368Stop, Pro370Leu e a Tyr437His, estão associadas a um início mais precoce com a pressão ocular elevada, com pouca ou nenhuma resposta ao tratamento clínico (Alward, Johnson et al., 1996; Nguyen, Chen et al., 1998; Alward, Kwon et al., 2002)
Seis locos cromossômicos foram localizados inicialmente para glaucoma primário de ângulo aberto: GLC1A, GLC1B, GLC1C, GLC1D, GLC1E, e GLC1F nos cromossomos 1q24-25, 2cen-q13, 3q21-24, 8q23, 10p, e 7q35-36 respectivamente (Alward, Johnson et al., 1996; Stone, Fingert et al., 1997; Fingert, Ying et al., 1998; Shepard, Jacobson et al., 2001; Fingert, Stone et al., 2002). Estes locos foram mapeados em estudos de ligação utilizando famílias onde o GPAA apresentava padrão segregação mendeliana. O loco GLC1A é aquele onde está mapeado o gene que codifica para a miocilina (TIGR/Myoc) (Alward, Fingert et al., 1998; Fingert, Stone et al., 2002) que é expressa em muitos tecidos oculares incluindo esclera, corpo ciliar, malha trabecular (TM), retina, bem como em tecidos não-oculares como o coração, pulmão e pâncreas (Sakata, 2006; Tink, Barnstable et al., 2008).
Locos genômicos identificados por mapeamento de ligação tornam-se candidatos para estudos de associação em amostras populacionais buscando entender a possível contribuição destes genes na variação do risco de desenvolvimento de glaucoma (Craig, Baird et al., 2001; Park, Tibudan et al., 2007).
Estudos em populações incluindo a brasileira realizados principalmente em famílias, indicaram que alterações no gene da miocilina podem levar ao desenvolvimento do glaucoma. Estudos de seqüenciamento da região codificante do gene MYOC dão conta de que mutações neste gene estão presentes em 1,1% - 4% dos casos de GPAA, dependendo da população (Nguyen, Chen et al., 1998; Monemi, Spaeth et al., 2005; Fan, Wang et al., 2006).
Tabela 1:Genes e loci associados ao Glaucoma.
Símbolo Loci Gene Tipo de Glaucoma
GLC1E 10p15-14 OPTN GPAA
GLC1A 1q23-25 MYOC/TIGR GPAA
9q22 GPAA
20p12 GPAA
1q21–31 MYOC/TIGR JOAG
6p25 FKHL7
GLC1B 2cen-q13 MYOC/TIGR GPAA
GLC1C 3q21-24 MYOC/TIGR GPAA
GLC1D 8q23 MYOC/TIGR GPAA
GLC1G 5q21.3-22. WDR36 GPAA
GLC1F 7q35-36
GLC1I 15q11-13
GLC1J 9q22
GLC1K 20p12
15q24. LOXL1
GPDS2 18q11-2
GLC3A 2p22-21 CYP1B1 Glaucoma congênito
RIEG1 4q25 PITX2
RIEG2 13q14
IRID1 11p13 PAX 6
IRID2 6p25 (FOXc1 FKHL7)
4q25 PITX
10q25 PITX3
4q25 PITX2
NNO 1 11p ? VMD2
NNO2 11q23.3 MFRP
AN2 11p13 PAX 6
PPCD1 20p11.21- VSX 1
PPCD2 q11.23 COL8A2
PPCD3 1p34.3-32.3 10p11.2
4q25-q26 PITX2
2p22-p21 CYP1B1
6p25 FOXC1
13q31-32 14q32 15q21
FBN1 (Fibril in)
5q13-14 WGN1
Fonte: OMIM- Online Mendelian Inheritance in Man:
1.4.
Genes candidatos para o Glaucoma Primário de
Ângulo Aberto (GPAA)
Estudos tem estabelecido que o glaucoma primário de ângulo aberto é geneticamente heterogêneo sendo causados por diferentes genes. Três genes (MYOC,OPTN e WDR36) foram identificados sendo estes associados diretamente na causa e desenvolvimento com GPAA (Monemi, Spaeth et al., 2005; Fan, Leung et al., 2006; Park, Tibudan et al., 2007). Estudos indicam que variações alélicas severas nestes genes estão bem associadas no desenvolvimento do glaucoma primário de ângulo aberto e com formas mendelianas (Monemi, Spaeth et al., 2005; Hauser, Sena et al., 2006; Hogewind, Gaplovska-Kysela et al., 2007).
O MYOC (miocilina) se constitui em um dos principais genes candidatos na patogênese do glaucoma primário de ângulo aberto (Stone, Fingert et al., 1997). Diversos estudos vem sendo realizados indicando sua associação com o GPAA. Mutações neste gene tem sido identificadas como uma das principais causas de glaucoma cujos fenótipos observados nos indivíduos com GPAA associados com o gene MYOC são heterogêneos(Jonhson, Alward et al., 1996; Libby, Gould et al., 2005)
O segundo gene associado ao GPAA é o OPTN (optineurina). Originalmente o gene OPTN foi identificado como FIP-2 e sabe-se que sua expressão é variada. A proteína opineurina é encontrada na retina, estrutura trabecular e em tecidos do globo ocular além de outros tecidos (Rezaie, Child et al., 2002; Hauser, Sena et al., 2006; Lopez-Martinez, Lopez-Garrido et al., 2007). A proteína optineurina é caracterizada pela indução de neuropatias. Alterações alélicas no gene OPTN causam atrofia do nervo óptico além de degeneração das células ganglionares da retina (Alexander, Samples et al., 1991; Yao, Cheng et al., 2006). Variações em seqüências do gene OPTN evidenciam uma forte correlação com o GPAA (Hauser, Sena et al., 2006; Liu, Schmidt et al., 2007). O gene mais recente descoberto associado ao glaucoma primário de ângulo aberto é o WDR36. Este gene está localizado no loco GLC1G. Embora a função protéica ainda seja desconhecida, estudos sugerem que mutações no gene WDR36 causam mudanças severas no desenvolvimento da doença. Em indivíduos afetados mutações na região promotora do gene WRD36 podem ocasionar o GPAA (Kramer, Samples et al., 2006; Wiggs, 2007).
Atualmente os pesquisadores estão começando a estudar os polimorfismos de nucleotídeos únicos (SNPs) para quantificar a percentagem da ancestralidade de Africanos e Europeus que apresentam o GPAA uma vez que a incidência desta patologia é significativamente maior em africanos quando comparada a populações européias (Tink, Barnstable et al., 2008).
1.4.1.1. O gene MYOC
O gene MYOC (OMIM 601652) e a proteína miocilina foram os primeiros a serem descobertos e estudados sendo estes associados ao glaucoma primário de ângulo aberto. O gene foi identificado em duas etapas de pesquisas: a primeira constituiu em análises de ligação em famílias com glaucoma primário de ângulo aberto juvenil (JOAG); Indivíduos diagnósticados com JOAG apresentam a forma mais severa da doença quando comparado com o GPAA em adultos sendo possível a identificação da região cromossomal 1q21–31 associada ao glaucoma (Fingert, Ying et al., 1998; Tamm e Fuchshofer, 2007; De Voogd, Wolfs et al., 2008)
A segunda etapa promoveu a descoberta da miocilina em um estudo em que a indução do corticoiesteróide dexametazona em culturas de células da malha trabecular foi avaliada, observou-se, aumento da produção da proteína miocilina associada ao glaucoma (Alward, Johnson et al., 1996; Sunden, Alward et al., 1996; Nguyen, Chen et al., 1998; Shepard, Jacobson et al., 2001; Fingert, Stone et al., 2002; Mackay, Kallberg et al., 2008).
A proteína miocilina tem localização extra e intracelular sendo encontrada principalmente nas células da malha trabecular com expressão em diversos tecidos oculares e não oculares. Alterações na malha trabecular podem estar implicadas na patogênese do glaucoma (Swiderski, Ross et al., 2000; Póvoa, 2002).
A utilização de corticosteróides como a dexametasona induz a expressão da proteína miocilina expressa especificamente em humanos. Outros genes específicos observados com a indução da dexametasona são bons candidatos ao glaucoma. Os efeitos do aumento da pressão intra-ocular também foram observados na expressão do MYOC (Lo, Rowlette et al., 2003). O gene MYOC está localizado no braço longo cromossomo 1 dentro do intervalo recombinante de D1S194 e D1S238, entre regiões 2 e 3 (Sunden, Alward et al., 1996; Stone, Fingert et al., 1997; Fingert, Stone et al., 2002; Mackey, Healey et al., 2003).
A associação entre o MYOC e o GPAA foi descoberta em 1997 onde 49 mutações foram identificadas em diferentes grupos populacionais (Stone, Fingert et al., 1997; Shepard, Jacobson et al., 2001; Fingert, Stone et al., 2002). O seqüenciamento do éxon 3 do gene MYOC permitiu identificar mutações em 4,4% dos 227 pacientes diagnosticados com glaucoma que possuíam história familiar, em 0,3% dos 380 pacientes da população geral, em nenhum dos 91 voluntários normais com idade acima de 40 anos e sem história familiar de glaucoma e em ,9% dos 103 pacientes com glaucoma independente da história familiar (Ritch, Steinberger et al., 1993; Nguyen, Chen et al., 1998; Fingert, Stone et al., 2002; Mackey, Healey et al., 2003)
associação genética foram realizados em pacientes de diferentes populações como a Chinesa, Japonesa, Coreana e Indiana e mostraram a base molecular do gene candidato MYOC na causa do GPAA (Mukhopadhyay, Hogewind et al., 2007). O nível protéico da miocilina está fortemente associado ao estágio progressivo da doença uma vez que estudos mostraram que deficiências no gene MYOC têm sido associadas com o glaucoma primário de ângulo aberto. A função específica da proteína miocilina em glaucomas está em ativa investigação.
A região promotora do gene MYOC/TIGR apresenta dois sítios de transição que incluem dois resíduos de citocina adjacentes ás seqüência TATA e CAT boxes (Nguyen, Chen et al., 1998). O gene MYOC apresenta em sua estrutura genômica 3 regiões exônicas separadas por 2 regiões intrônicas e uma região promotora de aproximadamente 5 kilobases (Nguyen, Chen et al., 1998). A miocilina é uma glicoproteína (Figura 2) que apresenta peso molecular aproximado entre 55 e 57 kDa com expressão aumentada na íris, corpo ciliar,córnea, esclera , malha trabecular, músculo ciliar, humor vítreo e nervo ótico (Adam, Belmouden et al., 1997; Tomarev e Malyukova, 2003).
Os estudos têm focado na expressão e localização da miocilina em humanos. Em sua localização intracelular é possível observar a sua associação com microtúbulos, mitocôndrias, cadeias regulatórias de miosina, actina e vimentina (Kubota, Noda et al., 1997; Mertts, Garfield et al., 1999; Ueda, Wentz-Hunter et al., 2000; Ueda e Yue, 2000). Os efeitos químicos da chaperonina foram avaliados sobre a secreção de miocilina mutante no glaucoma primário de ângulo aberto (Yam, Gaplovska-Kysela et al., 2007).
Estudos de polimorfismos em famílias vêm sendo realizados e tem revelado um completo desequilíbrio de ligação entre asiáticos e outros grupos populacionais (Mukhopadhyay, Hogewind et al., 2007). Estudos comprovam que mutações no gene MYOC causam o GPAA. Investigações em pacientes de 30 famílias com GPAA demonstraram que a patogênese da doença está fortemente ligada a mutações no éxon 3 do gene MYOC (Hogewind, Gaplovska-Kysela et al., 2007).
Figura 2:Esquema da estrutura da proteína MYOC. Zíper de leucina e domínio da olfatomedina.(Adaptado de (Shepard, Jacobson et al., 2007)
1.4.1.2. O gene OPTN
O gene OPTN (neuropatia óptica induzida ou proteína OPTN) tem sido descrito em diferentes populações com glaucoma e que suas seqüências gênicas estão associadas com o fenótipo característico da doença (Leung, Fan et al., 2003; Yao, Cheng et al., 2006). O gene OPTN (OMIM 602432) foi descrito originalmente no cromossomo 10 na região 10p14-15 (GLC1E) com mutações em indivíduos que apresentavam pressão ocular normal (Rezaie, Child et al., 2002; Tink, Barnstable et al., 2008).
A proteína OPTN tem peso molecular de 66-kDa e está presente no humor aquoso e em tecidos oculares no segmento anterior, retina e nervo óptico de humanos, macacos e ratos (Rezaie, Child et al., 2003; Rezaie, Waitzman et al., 2005). mutações nas seqüências do gene OPTN vêm sendo relatadas em estudos do tipo caso- controle em populações e estão associadas na causa do glaucoma primário de ângulo aberto. Estas mutações são diretamente relacionadas com essa forma de glaucoma uma vez que estão associadas com um aumento da pressão ocular e danos significativos no nervo óptico em pacientes diagnosticados com glaucoma primário de ângulo aberto (Rezaie, Child et al., 2002; Morrisson e I P, 2003; Lopez-Martinez, Lopez-Garrido et al., 2007).
Estudos em diferentes populações evidenciaram extensas variações nas seqüências do gene OPTN e estas mutações estão associadas com o glaucoma em suas diferentes formas. Os processos moleculares de mutações do gene OPTN contribuem para as causas de neuropatias ópticas relacionadas com glaucomas(figura 3) (Tink, Barnstable et al., 2008).
Análises das mutações no gene OPTN tendem a complementar a evidencia entre o envolvimento deste gene na apresentação do fenótipo característico de glaucoma primário de ângulo aberto. Estudos recentes encontraram a mutação E50K exclusivamente associada com forma familiar de glaucoma. Estudos clínicos em pacientes evidenciaram que a mutação OPTN-E50K está associada com a forma mais severa do GPAA (Alward, Kwon et al., 2003; Aung, Ebenezer et al., 2003; Hauser, Sena et al., 2006). Investigações sobre mutações do gene OPTN em diversos grupos de pacientes com glaucoma têm sido realizadas para elucidar os mecanismos biológicos das formas mutantes das proteínas envolvidas na patogênese do glaucoma (Rezaie, Child et al., 2002; Tink, Barnstable et al., 2008).
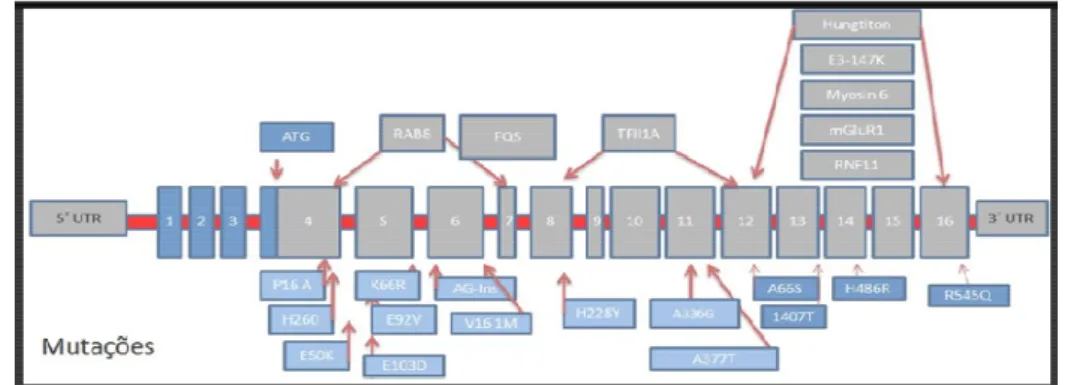
Figura 3:Mutações ao longo do gene OPTN. Localização das proteínas nos respectivos Éxons. Adaptado de (Tink, Barnstable et al., 2008).
A associação da proteína OPTN a interação com outras proteínas sugere que esta proteína tem significante função biológica no desenvolvimento normal do olho, nos processos de interação proteina-proteina além do possível tráfego e secreção de outras proteínas intracelulares.As informações obtidas das características básicas da proteína OPTN bem como o processo molecular nas mutações ocorridas no gene OPTN contribuem para neuropatias glaucomatosas do nervo óptico (Tink, Barnstable et al., 2008).
1.4.1.3. O gene WDR36
Além do gene OPTN, o gene WDR36 (OMIM *609669) é o mais recente descoberto e associado ao glaucoma primário de ângulo aberto. Utilizando estudos de ligação (Monemi, Spaeth et al., 2005), identificou o loco GLC1G. Mutações neste gene foram identificadas e relacionadas com o glaucoma primário de ângulo aberto (Kramer, Samples et al., 2006). O gene WDR36 ou repetição protéica 36 está localizado no cromossomo 5 na região 5q21.3 (Monemi, Spaeth et al., 2005; Kramer, Samples et al., 2006).
em grupos com GPAA sugerem a ativação de células T que medeiam à imunidade na rota do glaucoma (Schwartz, 2004). Duas mutações (I604V e A449T) apresentaram freqüências de 2,54% e 1,3% em pacientes com glaucoma. Diversos estudos indicam que mutações no gene WDR36 são responsáveis entre 10% a 17% de casos de glaucoma (Raymond e Rodrigue, 2005{Pasutto, 2008 #778).
Estudos identificaram que há diferenças nos níveis de expressão dos genes MYOC, OPTN e WDR36 em culturas de células (Rozsa, S et al., 2008). Estes genes estão associados com diferentes formas de glaucoma (Weinstein, Munnangi et al., 1985; Fan, Wang et al., 2006). Outras Estudos em populações no oeste da África sugerem que mutações no gene WDR36 estão relacionadas na regulação do controle dos níveis de pressão intra-ocular (Tink, Barnstable et al., 2008).
Por ser um novo gene descoberto na patogênese do glaucoma, as informações ainda são limitadas. Estudos de expressão gênica em tecidos oculares e não-oculares de humanos foram realizados e transcritos de RNAm foram observados no coração, placenta, fígado, músculo esquelético e pâncreas. Em tecidos oculares foi possível verificar os níveis dos transcritos no corpo ciliar,esclera, íris e músculo ciliar (Monemi, Spaeth et al., 2005).
O mecanismo molecular do gene WDR36 na patogênese das diferentes formas de glaucoma está sendo investigado por ser um gene considerado um importante contribuinte no desenvolvimento do glaucoma (Tink, Barnstable et al., 2008). Mutações neste gene estão associadas na causa do GPAA. Em casos com história familiar de glaucoma 50% destes são ocasionados por mutações sendo o GPAA transmitido por herança autossômica dominante. Em caos esporádicos, a presença da mutação no gene WDR36 somado com a idade avançada pode aumentar o risco da ocorrência do glaucoma em 100% na população (Tink, Barnstable et al., 2008).
Estudos do tipo caso-controle em pacientes com glaucoma de diferentes populações (Inglesa, Americana, Canadense, Alemã e Francesa) estão sendo realizados em busca do maior entendimento sobre as mutações envolvidas nos genes associados com o glaucoma primário de ângulo aberto (Raymond e Rodrigue, 2005; Hauser, Sena et al., 2006; Pasutto, Mardin et al., 2008). Um numero de mutações no gene WDR36 e variações na seqüência de DNA tem sido observadas em diferentes populações. A mutação A449T tem sido identificada em populações nos estudos do tipo caso-contole.
1.4.1.4. O gene CYP1B1
Estudos de ligação gênica identificaram outro gene além dos que já foram mencionados o CYP1B1 localizado no loco GLC3 (2p22-p21) vem sendo descrito como mais um gene candidato na causa do glaucoma (Vincent, Billingsley et al., 2002; Melki, Colomb et al., 2004; Kumar, Basavaraj et al., 2007). O gene CYP1B1 é pertencente à família 1 e subfamília B1 do citocromo P450 (CYP) e super família de enzimas envolvidas no metabolismo oxidativo de uma variedade de compostos endógenos ou xenobióticos. A expressão do gene CYP1B1 é visualizada no epitélio ciliar, córnea e retina e na estrutura da câmara anterior do olho (Choudhary, Jansson et al., 2006; Doshi, Marcus et al., 2006).
O CYP1B1 (OMIM 601771) é um gene comumente associado com glaucoma congênito primário (PCG), mas mutações neste gene também têm sido associadas com glaucoma de ângulo aberto juvenil (JOAG) (Acharya, Mookherjee et al., 2006). Em alguns pacientes que apresentaram mutações neste gene e no MYOC, a etiologia desta doença foi considerada. Em outras populações as mutações no gene CYP1B1 ocorrem entre 20% a 100% dos casos de pacientes com glaucoma (Bayat, Yazdani et al., 2008).
1.5. Polimorfismos de uma única base (SNPs) e estudos de associação
Os polimorfismos de uma única base são marcadores bialélicos resultantes de substituições de bases nitrogenadas durante a replicação do DNA, devido a processos mutacionais espontâneos e induzidos. Estes marcadores de estrutura simples são abundantes no genoma humano e constituem uma ferramenta valiosa para estudos de associação genética e identificação de genes que possam explicar a variação de fenótipos complexos (Risch e Merikangas, 1996; Goodwin, 2007).
A utilização de marcadores do tipo SNP no entendimento de regiões genômicas importantes para a variação de fenótipos complexos experimentou um grande avanço nos últimos 10 anos. Em 1998 foi publicado o primeiro estudo onde métodos de larga escala foram utilizados para identificação e mapeamento de cerca de 2500 SNPs (Wang, Fan et al., 1998). Atualmente o dbSNP, banco de dados público, dispõe de aproximadamente 9 milhões de SNPs submetidos, dos quais quase 3 milhões são validados, ou seja, são realmente polimórficos (http://www.ncbi.nlm.nih.gov/projects/SNP/snp_summary.cgi, acesso em 11/11/2008).
Os desenhos experimentais para a condução de estudos de associação, sejam os fenótipos em questão qualitativos ou quantitativos, são atualmente quase todos baseados no princípio do desequilíbrio de ligação, tanto em desenhos com regiões candidatas ou por varredura genômica do genoma inteiro (GWAS, do inglês Genome-wide association studies) (Cardon e Bell, 2001; Hirschhorn e Daly, 2005).
O desequilíbrio de ligação pode ser entendido como um desvio na freqüência na ocorrência conjunta de alelos em dois ou mais locos adjacentes (haplótipos) em relação à freqüência esperada em uma segregação independente (Lewontin, 1988; Strachan e Read, 2002). O DL é uma medida estatística que pode estimar os valores de associação entre alelos e estes são calculados de acordo com métodos estatísticos específicos (Lewontin, 1964; 1995; Maniatis, Morton et al., 2005). A quantificação do desequilíbrio de ligação pode ser realizada de diversas maneiras. A mensuração do DL pode então ser realizada usando grau de associação entre pares de marcadores bialélicos definidos comumente de D´ (calcula o desequilíbrio gamético) e r2 (normalizada em função de suas freqüências alélicas) (Lewontin, 1964; Ohta e Kimura, 1969; Lewontin, 1988 ).
A demonstração de que o genoma humano é dividido em blocos de alto e baixo desequilíbrio de ligação levou a racionalização de que o número de SNPs necessários para o desenvolvimento de estudos de associação por varredura do genoma total poderia ser menor que os 1 milhão previstos caso o desequilíbrio de ligação fosse homogêneo ao longo do genoma com extensão máxima de 3000 pares de base (Kruglyak, 1999; Zhang, Calabrese et al., 2002).
Com o objetivo de oferecer maior racionalidade na escolha de SNPs que permitissem capturar a maior diversidade haplotípica ao longo do genoma foi iniciado o projeto HapMap (Hapmap, 2003). A segunda fase do HapMap caracterizou cerca de 3,1 milhões de SNPs genotipados em 270 indivíduos de 4 populações de diferentes regiões (Yoruba, Européia e regiões asiáticas) com 25-35% de variações comuns (Frazer, Ballinger et al., 2007; Manolio, Brooks et al., 2008).
Os tagSNPs são SNPs que apresentam alta capacidade de capturar a diversidade da variedade haplotipica ao longo de todo genoma. Os tagSNPs apresentam como vantagem a captura da variedade haplotipica com menor número de SNPs a ser genotipados.Com estes dados, empresas como Illumina e Afymetrix desenvolveram sistemas para a genotipagem de 500 mil a 650 mil tagSNPs podendo predizer padrões de desequilíbrio de ligação (Ke, Hunt et al., 2004; Gonzalez-Neira, Ke et al., 2006)
No ano de 2007 foram publicados os primeiros resultados de grandes estudos de associação genômica ampla envolvendo grupos amostrais na casa dos milhares. Até o momento os principais estudos de associação genômica ampla são conduzidos no desenho casos e controles. A partir da genotipagem de milhares de SNPs, a utilização de um valor de p 0,05 como nível de significância para descartar a hipótese nula de não associação, o número potencial de associações simplesmente ao acaso seria da ordem de 5000. Para reduzir o número de associações espúrias o consenso é o de se utilizar a correção de Bonferroni para múltiplos testes. Assumindo valor de p 0,05 para um único teste, com a genotipagem de 100000 SNPs, o valor de p corrigido seria de 5 x 10-7. No entanto, para que se mantenha alto poder de detecção da associação nestas condições são necessários grupos de casos e controles na ordem de milhares (Manolio, Brooks et al., 2008).
1.6. Transferibilidade de tagSNPs
A escolha de tagSNPs se constitui um dos focos do projeto HapMap por permitir estudos de transferibilidade entre as populações inseridas no projeto e as populações alvo. Estudos comparativos das amostras populacionais dentro e entre as regiões geográficas tem ampliado o rigor da seleção destes marcadores para garantir maiores informações sobre a transferibilidade (Liu, Sawyer et al., 2004; Gu, Yu et al., 2008). O projeto HapMap por sua vez vêm sendo utilizado como referência para escolha de tagSNPs uma vez que este identifica padrões comuns na seqüência de DNA e a correlação entre estes (Tihp, 2003; Tantoso, Yang et al., 2006)
A aplicação dos estudos de desequilíbrio de ligação permite a obtenção de informações sobre tagSNPs de populações do HapMap e população alvo em estudos de associação indireta (Mueller, Lohmussaar et al., 2005; Mahasirimongkol, Chantratita et al., 2006). No entanto, sabe-se que a história demográfica, padrão de desequilíbrio de ligação (DL) para as quatro populações do HapMap é similar (De La Vega, Isaac et al., 2005). Entretanto, a extensão do desequilíbrio de ligação, haplótipos e freqüências alélicas de áreas com elevado DL, podem ser diferentes (Sawyer, Mukherjee et al., 2005)
A eficiência destes marcadores pode ser avaliada com base nos dados do HapMap através de estudos de transferibilidade entre populações e doenças complexas por detectar sinais de associação e por sua vez reduzirem custos de genotipagem (Doris, 2002; Lim, Kim et al., 2006; Ribas, Gonzalez-Neira et al., 2006).
Estudos tem sugerido a portabilidade do uso destes marcadores através de populações por serem marcadores altamente informativos quando utilizados para estudos de transferibilidade entre cada grupo continental (Gonzalez-Neira, Ke et al., 2006) por sua propriedade de captura de variação de haplótipos, um tagSNP de uma população pode ser altamente informativo para outra população em estudo.
Outros estudos reportam variações significantes entre populações em blocos estruturais e tagSNPs. Investigações entre as populações africanas e européias utilizando 16 marcadores encontraram variações quantitativas e qualitativas significativas do desequilíbrio de ligação entre e nos grupos populacionais utilizados (Gu, Pakstis et al., 2005; Sawyer, Mukherjee et al., 2005).
1.7. SNPs informativos de ancestralidade
Os marcadores informativos de ancestralidade(AIM- Ancestry Informative Markers) tem sido utilizados para investigar a estrutura genética de populações humanas com base nas diferenças de freqüências alélicas. Os AIM´s podem estimar as diferenças genéticas entre os grupos étnicos e susceptibilidade e resistência a doenças (Shriver, Frudakis et al., 2005; Frudakis, 2007).
Estes marcadores podem medir as proporções de populações parentais em populações miscigenadas((Shriver, Smith et al., 1997; Rosenberg, 2002; Shriver e Kittles, 2004). As populações miscigenadas são importantes no mapeamento de regiões nas populações parentais e para estudos de associação por mapas de miscigenação de desequilíbrio de ligação (Admixture mapping by linkage disequilibrium ) (Coffey, Reidy et al.). (Shriver, Parra et al., 2003) (Parra, Marcini et al., 1998).
Marcadores que apresentem diferenças alélicas em duas ou mais populações podem ser considerados como informativos de ancestralidade. Neste contexto os microssatélites(Short Tandem Repeats) são pontenciais marcadores devido a sua carateristica multi-alélica entre populações. Os SNPs e Indels (Inserção/Deleção) são marcadores que fornecem informações para somente duas populações (Rosenberg, Pritchard et al., 2002). No entanto os SNPs apresentam maior quantidade de informação do que os microssatélites sendo vantajosos com relação a quantidade de informação sobre ancestralidade e também pela carateristica de apresentar grandes diferenciais de freqüências alélicas entre populações distintas e por conseqüência altos níveis de Fst e baixos níveis de heterozigosidade (Shriver, Smith et al., 1997; Rosenberg, Li et al., 2003; Shriver, Parra et al., 2003)
A busca por SNPs que expliquem a incidência de uma patologia ou que sejam informativos de ancestralidade tem sido alvo de pesquisas por parte da comunidade científica. Painéis de marcadores utilizando os AIMs vem sendo propostos para Africanos, Europeus , Afro-Americanos, Hispânicos e Nativo- Americanos. O projeto HapMap constitui-se em um banco de dados que abriga informações sobre marcadores sendo possível fazer comparações entre as sequencias genéticas sendo possível identificar regiões genômicas variantes entre as populações CEPH constituindo-se portanto em uma importante ferramenta para pesquisas na área biológica (Smith, Lautenberger et al., 2001; Halder e Shriver, 2003; Mao, Bigham et al., 2007; Price, Patterson et al., 2007).
O glaucoma primário de ângulo aberto, por ser considerado uma doença complexa cuja maior incidência, é em individuos pertencententes ao grupo populacional de negros e /ou afrodescendentes é portanto uma patologia considerada importante para estudos de associação genética(Sommer, Tielsch et al., 1991; Rotimi, Chen; et al., (2006)). Estes estudos visam detectar associação entre um ou mais SNPs que pode representar uma carateristica quantitativa discreta ou doença (Cordell e Clayton, 2005).
A miscigenação pode interferir na ligação por desequilíbrio, aumentando a sua freqüência e incidência. O MALD tem maior chance de sucesso em populações Afro-descendentes. A incidência de doenças complexas se mostra maior nesse grupo, quando comparado com Europeus. Os maracdores do tipo SNPs de populações Africanas chegam a ser cinco vezes mais informativos quando comparados aos de populações Européias ou Hispânicas (Smith, Lautenberger et al., 2001; Smith e O'brien, 2005). Doenças complexas como a hipertensão arterial, câncer de próstata, esclerose sistêmica foram identificadas com o uso de MALD (Smith et al., 2001). Por ser uma doença complexa, o GPAA pode ser um potencial candidato a mapeamento por MALD.
Em um estudo realizado com uma família de Barbados mostrou alguns marcadores que poderiam estar ligados ao GPAA(Nemesure, Jiao et al., 2003). Dentre eles, quatro maracdores do tipo microssatelites apresentaram valor de associação considerável (considerando para este trabalho LOD score acima de 2: D10S548, D10S211, D2S2189 e D2S325 . Outra investigação para análise de ligação para locos ligados ao GPAA mostrou marcadores com LOD score maiores (> 1, 5) nos cromossomos D5S2501,D14S3587,D5S424 e D3S1786 respectivamente (Rotimi, Chen; et al., (2006)).
O Brasil se torna um potencial grupo de estudo para a técnica do MALD por apresentar uma população altamente miscigenada. Além disso, estudos de dados genotípicos apresenta a contribuição de ancestralidade genética africana em cerca de 15% para o Brasil (Callegari-Jacques, Grattapaglia et al., 2003) (Lins, 2007). Mesmo com a miscigenação é necessário ter cautela ao dizer que o Brasil pode ser mais incidente em relação ao GPAA.
1.8. Estudos de associação na população brasileira
A população brasileira é considerada como a mais heterogênea do mundo resultantes da mistura entre Europeus, Africanos e Ameríndios (Pimenta, Zuccherato et al., 2006). A distribuição dos grupos populacionais encontrados no Brasil não se deu de forma homogênea durante o período de colonização ocorrendo assim mistura de genes e conseqüentes características genotípicas e fenotípicas foram adicionadas á nossa população (Alves-Silva, Da Silva Santos et al., 2000; Carvalho-Silva, Santos et al., 2001).
A caracterização da população brasileira se deu pelo cruzamento direcional entre mulheres africanas e ameríndias e homens europeus (Alves-Silva, Da Silva Santos et al., 2000; Carvalho-Silva, Santos et al., 2001; Abe-Sandes, Silva et al., 2004; Ferreira, 2006). Devido a esta heterogeneidade na sua formação, esta população está propensa a um alto grau de polimorfismo ou a uma distribuição diferenciada, conforme a contribuição étnica original, constituindo um alvo importante para o conhecimento sobre a distribuição alélica e genotípica, com relação aos marcadores genéticos.
Nos últimos anos, tem sido crescente o interesse por marcadores que possibilitam a obtenção de informações nos estudos de mapeamento de loci ligados a doenças complexas por desequilíbrio de ligação (LD) e populações miscigenadas para estimar o nível de mistura em populações recentes (Hoggart, Shriver et al., 2004).
Este desequilíbrio de ligação é particularmente alto entre marcadores polimórficos que apresentam alelos com grandes diferenças de freqüência entre as populações base. Este método foca em fenótipos que tenham incidência diferenciada nas populações ancestrais, a partir das quais foi gerada a população miscigenada. A lógica é a de que em um grupo de indivíduos miscigenados afetados, tem-se um maior compartilhamento de regiões genômicas com a mesma origem. Ou seja, a população onde o fenótipo é mais incidente. (Seldin, Tian et al., 2007).
A limitação no conjunto de regiões genômicas que podem ser identificadas pode ser compensada por uma redução no número de marcadores necessários para a condução de estudos de associação genômica ampla por MALD. Estima-se que com 2000 a 5000 marcadores informativos de ancestralidade bem distribuídos pelo genoma seja possível conduzir com sucesso estudos de MALD (Hoggart, Shriver et al., 2004).
moleculares e arcabouço analítico estabelecido para condução de tais estudos (Mckeigue, 2005; Smith, Thomas et al., 2005).
Painéis de marcadores informativos de ancestralidade distribuídos ao longo do genoma foram identificados e caracterizados para utilização em MALD em populações com ancestralidade euro-afiricana (Smith, Lautenberger et al., 2001; Collins-Schramm, Phillips et al., 2002; Carlson, Eberle et al., 2004) e populações com considerável ancestralidade ameríndia (Tian, Hinds et al., 2006).
A partir de 2005 aparecem na literatura os primeiros trabalhos de mapeamento de regiões genômicas utilizando os conceitos de MALD. Estes trabalhos mapearam regiões associadas à esclerose múltipla, hipertensão, câncer de próstata e níveis de IL6 (interleucina 6) e IL6Rs (receptor de interleucina 6 solúvel), asma e calcificação da artéria coronária (Choudhry, Coyle et al., 2006; Freedman, Haiman et al., 2006; Kidd, Pakstis et al., 2006).
Estudos de associação ampla recentes (GWAs) têm demonstrado fortes evidencias entre variantes genéticas e doenças complexas. Estes estudos têm sido aplicados em investigações do tipo caso-controle em populações de ancestralidade Européia cujo conhecimento sobre o risco e freqüência de alelos em outras populações e a transferibilidade dos SNPs, associações e diferenciação destas doenças na população humana estão sendo amplamente realizados (Myles S, Davison D et al., 2008). Estes estudos relacionados com doenças sugerem que o desequilíbrio de ligação contribui para o entendimento de associações entre a variante genética e a doença. Estudos sobre câncer de cólon-retal cerca de 30% dos casos estão associados com raras variantes multifatoriais.esta hipótese propõe que há susceptibilidade de doenças humanas comuns podendo sofrer a ação conjunta de variantes de diferentes genes que conferem um moderado aumento no risco da doença. Cada variante é especifica para cada população pelos efeitos de deriva genética que cada população está sujeita (Bodmer, 1999; Bodmer e Bonilla, 2008).
Variações em nível da seqüência de DNA são comuns em populações humanas, sendo que a maior parte destas variações são neutras. Algumas variantes são naturais, outras são específicas em muitas doenças. Estudos indicam que mais de 1800 locos foram identificados e associados com variantes de doenças raras e comuns . Investigações a nível de seqüência destes locos variantes que causam doenças tem sido identificadas gene a gene. Para algumas desordens, fenótipos similares causados por mutações que afetam partes de alguns genes (heterogeneidade alélica), estas mutações podem contribuir em pequena proporção no padrão alélico (Smith e Lusis, 2002; Haines e Pericak-Vance, 2006).
comuns(common disease) além de múltiplas variantes raras (Smith e Lusis, 2002; Bodmer e Bonilla, 2008; Collins, 2008). O entendimento sobre a arquitertura alélica de doenças humanas comuns se torna uma ferramenta para identificação de genes e alelos associados a doenças complexas. Estudos de desqequilibrio de ligação tem sido utillizados como um método para mapeamento de genes associados a doenças complexas. O métodos baseados nos estudos de DL melhor observados quando há um único alelo que apresente suscetibilidade em determinada doença sendo difícil o estudo quando há substancial heterogeneidade alélica mas estudos sobre a hetergeneidade alélica apresentam implicações importantes para investigações de mapeamento de genes e estudos de associação (Pritchard e Cox, 2002; Smith e Lusis, 2002).
2.
Hipótese de trabalho
TagSNPs selecionados a partir da amostra Africana no HapMap são a melhor estratégia para transferir informações do HapMap para a população brasileira quando do desenho de estudos de associação genética para o fenótipo complexo Glaucoma
3.
Objetivos
3.1. Geral
Estudar a transferibilidade de tagSNPs selecionados a partir da amostra Africana do HapMap para a população brasileira nos gene MYOC e identificar SNPs para estudos de associação em locos ligados ao GPAA.
3.2. Específicos
• Selecionar um conjunto de SNPs da amostra africana do HapMap para os
gene MYOC.
• Genotipar um conjunto de tagSNPs selecionados da amostra Africana do
HapMap para o gene MYOC em uma amostra da população brasileira.
• Determinar o padrão de desequilíbrio de ligação e haplótipos do gene MYOC
em uma amostra da população brasileira utilizando TagSNPs.
• Verificar a utilização dos conjuntos de tagSNPs dos genes, MYOC e OPTN
para estudos de associação indireta.
•
Estudar a transferibilidade de tagSNPs entre as populações do HapMap e abrasileira
.
• Recuperar genótipos de SNPs com 1 Mpb a 5´ e 3´ de locos microssatélites
ligados ao GPAA em famílias de afro-descendentes do HapMap
• Eleger SNPs informativos de ancestralidade utilizando LOD score de p 0,5
4.
Material e Métodos
4.1. Amostras biológicas utilizadas nesse estudo
Amostras de sangue de 200 indivíduos não correlacionados de diferentes regiões do Brasil (Sul, Centro Oeste, Nordeste e Norte) foram obtidas de sujeitos que se submeteram a testes de paternidade. Os indivíduos assinaram um termo de consentimento informado da utilização de suas amostras de DNA em estudos de diversidade genética.
4.2.
Extração de DNA
O DNA genômico das amostras da população brasileira utilizadas neste estudo foi extraído dos leucócitos periféricos com modificações do método salting out (Miller, Dykes et al., 1988). Para utilização do DNA nas reações de PCR, as amostras foram quantificadas em gel de agarose a 1,0% corado com brometo de etídio (1 mg/mL) utilizando padrões de DNA de concentrações conhecidas cuja visualização se procedeu em luz ultravioleta. A partir da concentração observada, as amostras foram diluídas para a concentração de trabalho a 10 ng/ L.
4.3. Seleção de tagSNPs nos gene MYOC
A seleção dos tagSNPs para o gene MYOC foi realizada através do banco de dados International HapMap Consortium (http://www.hapmap.org) pelo algoritmo HapMart obedecendo ao critério de freqüência alélica mínima (MAF) igual ou maior que 5% da população YRI (povo Yoruba de Ibadan, Nigéria) (Carlson, 2004).
4.4. Genótipos do HapMap