tt
Pró – Reitoria de Graduação
Curso de Bacharelado em Ciências Biológicas
Trabalho de Conclusão de Curso
CLONAGEM DE FRAGMENTOS DO PROMOTOR DE
ALGODÃO UCEA1.7EM VETOR DE TRANSFORMAÇÃO DE
PLANTAS.
PHILIPPE DE CASTRO LINS
Brasília, novembro de 2012
Pró-Reitoria Acadêmica
Escola de Saúde
Programa de Pós Graduação Stricto Sensu em Biotecnologia
CARACTERIZAÇÃO FUNCIONAL DO PROMOTOR DE SOJA
UCES8.3
Autor: Philippe de Castro Lins
Orientadora: Prof
aDr
aMaria Fátima Grossi de Sá
PHILIPPE DE CASTRO LINS
CARACTERIZAÇÃO FUNCIONAL DO PROMOTOR DE SOJA UCES8.3
Dissertação apresentada ao Programa de Pós-Graduação Stricto Sensu em Biotecnologia da Universidade Católica de Brasília, como requisito parcial para a obtenção do Título de Mestre em Biotecnologia.
Orientador: Profa Dra Maria Fátima Grossi de Sá
7,5cm
Ficha elaborada pela Biblioteca Pós-Graduação da UCB
L759c Lins, Philippe de Castro.
Caracterização funcional do promotor de soja UCES8.3. / Philippe de Castro Lins – 2015.
62 f.; il.: 30 cm
Dissertação (Mestrado) – Universidade Católica de Brasília, 2015. Orientação: Profa. Dra. Maria Fátima Grossi de Sá
1. Biotecnologia. 2. Promotor. 3. Elementos cis. 4. Tecido-específico. 5. Expressão gênica. I. Sá, Maria Fátima Grossi de, orient. II. Título.
AGRADECIMENTOS
Agradeço primeiramente a Deus por esse dom de viver e por sempre estar presente na minha vida! Agradeço à minha família por todo apoio e por toda ajuda durante esses anos, aos meus amigos por estarem do meu lado e por me acompanharem nas disciplinas, por tantos trabalhos e momentos divertidos.
À Capes pelo programa de bolsas PROSUP que possibilitou a realização do meu mestrado.
Ao grupo do laboratório LIMPP Embrapa-Cenargen que me auxiliou nesse trabalho, em especial: à minha orientadora Maria Fátima Grossi de Sá, à Isabela Tristan Lourenço, Antônio Américo Barbosa Viana, Rodrigo Fragoso, Leonardo Pepino, Ísis e Thuanne.
RESUMO
Referência: LINS, Philippe de Castro. Caracterização funcional do promotor de soja UCES8.3. 2015. 62 páginas. Mestrado em Ciências Genômicas e Biotecnologia. Pró-Reitoria de Graduação e Pesquisa Programa de Pós-Graduação Stricto Sensu em Biotecnologia Universidade Católica de Brasília. Brasília, 2015.
Promotores gênicos regulam a expressão de genes, quantitativamente e qualitativamente. As sequências regulatórias possuem elementos cis e trans atuantes que vão direcionar e posicionar corretamente a RNA polimerase para que haja o processo de transcrição do DNA. A engenharia genética de plantas, por meio da transformação de plantas, expressando genes de interesse, vem utilizando, na maioria dos estudos, o promotor CaMV35S de caráter constitutivo. Um novo promotor isolado de soja, denominado uceS8.3 é também um promotor constitutivo demonstrando conduzir a expressão em diferentes tecidos vegetais. A análise da expressão do gene uidA, GUS, demonstrou que o perfil de expressão controlada pelo promotor uceS8.3 é comparável ou superior ao promotor CaMV35S em tecidos vegetais, como botão floral e raiz. Foram identificados, na sequência do promotor uceS8.3, elementos cis regulatórios que podem ser responsáveis pelo perfil de expressão gênica controlada por esse promotor. Os módulos desse promotor, quando comparados com o promotor CaMV35S, e o próprio uceS8.3 demonstraram uma diferença de expressão nos tecidos vegetais. Elementos cis regulatórios como ROOTMOTIFPABOX1, POLLEN1LELAT52, MRE, Sp1 e I-box. O conjunto de elementos cis específicos, localizados nos módulos do promotor uceS8.3, podem ser os elementos mínimos necessários para controlar a expressão em tecidos de folha e botão floral. A análise de expressão quantitativa e de ensaios fluorimétricos de GUS nos tecidos folha e raiz, demonstraram uma correlação entre os níveis de transcrito e os níveis de atividade específica da enzima β-glucuronidase no módulo 2 (720pb), porém há uma diferença na correlação destes níveis entre o promotor uceS8.3 e o módulo 4 (170pb). Os estudos aqui apresentados demonstraram que o promotor uceS8.3 e seus módulos possuem alta capacidade de conduzir expressão em tecidos florais e raízes, podendo ser utilizado e aplicado biotecnologicamente para diferentes tipos de estresses bióticos, incluindo insetos-praga e nematóides.
ABSTRACT
Gene promoters regulate gene expression quantitatively and qualitatively. The regulatory sequences have cis elements and with trans acting that will direct and correctly position the RNA polymerase which is the process of DNA transcription. Genetic engineering of plants by transforming plants expressing genes of interest, use in most studies, constitutive CaMV35S promoter character. A new promoter isolated from soybeans, which is called uceS8.3 showing a constitutive promoter driving expression in different plant tissues. The analysis of the expression of the uidA gene, GUS, demonstrated that the expression profile uceS8.3 controlled by the promoter is comparable or superior to the CaMV35S promoter in plant tissues such as flower buds and roots. Were identified following the uceS8.3 promoter cis regulatory elements that may be responsible for gene expression profile controlled by this promoter. Modules of this promoter, when compared with the CaMV35S promoter and the uceS8.3 itself demonstrated a difference in expression in plant tissues. Cis regulatory elements as ROOTMOTIFPABOX1, POLLEN1LELAT52, MRE, Sp1 and I-box. The set of specific cis elements located in the promoter uceS8.3 modules may be the minimum necessary elements to control expression in leaf and flower bud tissues. The quantitative expression analysis and fluorometric GUS assays leaf and root tissues have demonstrated a correlation between transcript levels and the
specific activity levels of a β-glucuronidase enzyme in module 2 (720pb), but there is a difference in the balance of these levels between uceS8.3 promoter and Module 4 (170pb). The studies presented here demonstrated that the promoter and its modules has high ability to drive expression in flower tissues and roots, may be used and applied to different types of biotechnologically biotic stresses, including insect pests and nematodes.
SUMÁRIO
1.INTRODUÇÃO ... 9
1.1 PROMOTORES GÊNICOS ... 9
1.2 ELEMENTOS CIS E TRANS ... 10
1.3 UTILIZAÇÃO DE PROMOTORES NA TRANSFORMAÇÃO DE PLANTAS ... 11
1.3.1 Promotor constitutivo CaMV35S ... 12
1.3.2 Promotores tecido específicos ... 14
1.3.3 Promotores induzidos por fatores externos ... 14
1.4 PROMOTORES DE UBIQUITINA ... 15
1.5 PROMOTOR DE SOJA UCES8.3 ... 16
2 JUSTIFICATIVAS ... 26
3. OBJETIVO ... 26
3.1 OBJETIVOS ESPECÍFICOS ... 26
4. METODOLOGIA ... 27
4.1 DEFINIÇÃO E AMPLIFICAÇÃO DOS FRAGMENTOS DO PROMOTOR UCES8.3 ... 27
4.2 QUANTIFICAÇÃO DO PRODUTO DE PCR ... 27
4.3 CLONAGEM NO VETOR INTERMEDIÁRIO PDONR 221 ... 28
4.4TRANSFORMAÇÃO DE CÉLULAS COMPETENTES E SELEÇÃO DAS COLÔNIAS POSITIVAS ... 29
4.5 EXTRAÇÃO DE DNA PLASMIDIAL ... 29
4.6 RECOMBINAÇÃO LR DO VETOR PDONR 221 COM PKGWFS7 ... 30
4.7 CONFIRMAÇÃO DA SUBCLONAGEM NO VETOR PKGWFS7 ... 31
4.8 TRANSFORMAÇÃO DE Agrobacterium tumefaciens ... 31
4.9 TRANSFORMAÇÃO DE Arabidopsis thaliana E SELEÇÃO DAS SEMENTES TRANSFORMADAS ... 31
4.10 ENSAIO HISTOQUÍMICO DE GUS ... 32
4.11 EXTRAÇÃO DE RNA E SÍNTESE DE CDNA ... 33
4.12 PCR EM TEMPO REAL ... 34
4.13 ENSAIOS FLUORIMÉTRICOS DE GUS... 34
5.1 AMPLIFICAÇÃO E CLONAGEM DOS MÓDULOS DO PROMOTOR UCES8.3
NO VETOR PDONR 221 ... 36
5.2 SUBCLONAGEM LR DAS CONSTRUÇÕES DOS MÓDULOS COM O VETOR DE TRANSFORMAÇÃO DE PLANTAS PKGWFS ... 38
5.3 TRANSFORMAÇÃO DE CÉLULAS COMPETENTES DE AGROBACTÉRIA . 40 5.4 TRANSFORMAÇÃO DE Arabidopsis thaliana E SELEÇÃO DE PLANTAS POSITIVAS IN VITRO ... 41
5.5 ENSAIO HISTOQUÍMICO DE GUS ... 43
5.6 PCR EM TEMPO REAL DE GUS E GFP ... 45
5.7 ENSAIO FLUORIMÉTRICO DE GUS ... 46
6. DISCUSSÂO ... 48
6.1 ANÁLISE DOS ELEMENTOS CIS NOS MÓDULOS DO PROMOTOR UCES8.3 PELO ENSAIO HISTOQUÍMICO ... 48
6.2 ANÁLISE DA EXPRESSÃO RELATIVA DE MRNA E ATIVIDADE ESPECÍFICA DE GUS ... 51
7. CONCLUSÃO ... 53
8. PERSPECTIVAS DE ESTUDOS FUTUROS ... 54
1. INTRODUÇÃO
1.1 PROMOTORES GÊNICOS
Os promotores gênicos ou sequências regulatórias são responsáveis pela regulação da expressão de genes, havendo em sua cadeia de nucleotídeos elementos cis, que são sítios para a ligação de proteínas específicas, fatores de transcrição (TFs), ou fatores trans, e o posicionamento da RNA polimerase e orientação 5’ 3’ (PEREMARTI et al, 2010). Em eucariotos, é conhecido proteínas controlarem a transcrição em regiões a jusante da sequência promotora. Esse conjunto de elementos, contidos nas sequências de DNA determina o ponto correto para o início do processo de transcrição, podendo determinar ainda o padrão temporal e espacial em que o processo irá acontecer (HERNANDEZ-GARCIA; FINER, 2014). Promotores gênicos são fragmentos de DNA, que ficam a jusante do gene, nos quais a RNA polimerase se liga para iniciar o processo transcricional e, portanto, são essenciais e relacionados a expressão fenotípica do material genético, representando assim um importante processo para a correta transcrição do gene (BATUT et al 2013; BANG, 2013)
O início da transcrição do gene (TSS, transcription start site) determina a numeração dada às sequências regulatórias a montante (-) ou a jusante (+). Em organismos eucariotos há sequências conservadas que auxiliam no posicionamento da RNA polimerase, em uma distância de aproximadamente - 30 pb do TSS, conhecida como TATA box, muitas vezes constituída da sequência de nucleotídeos TATA (BANG, 2013). Outra sequência localizada aproximadamente a - 100 pb é o
CAAT box e a - 200 pb o GC box, o que caracteriza o core promoter (BATUT et al.
2013), ou promotor basal. Dentre os elementos conservados em eucariotos, o TATA box é o melhor caracterizado no promotor basal. O promotor basal determina a posição e orientação correta do início da transcrição do gene (BUTLER; KADONAGA, 2002; STEPHEN; JAMES, 2003).
1.2 ELEMENTOS CIS E TRANS
Próximo ao promotor, para o reconhecimento da RNA polimerase II, existe uma variedade de sequências curtas, conhecidas como elementos cis atuantes, no qual cada elemento é reconhecido por um fator atuante trans. Esses elementos, que coordenam as proteínas para o posicionamento correto para o início da transcrição, são localizados acima do TATA box, podendo estar presentes em diferentes orientações e distâncias do TSS, como esquematizado na figura 1 (STEPHEN; JAMES, 2003; PEREMARTI et al, 2010).
Os elementos cis do DNA recrutam proteínas com motivos de ligação ao DNA, que são os fatores de transcrição. Os fatores de transcrição básicos de um promotor eucarioto incluem: TFIID, TFIIB, TFIIF, TFIIE e TFIIH, que se agrupam no complexo de pré-iniciação da transcrição. O TBP (TATA box-binding protein) reconhece o elemento TATA box, que é uma subunidade do TFIID, e também interage com o fator TFIIB, um polipeptídeo (BUTLER; KADONAGA, 2002).
Figura 1: A figura esquematiza a posição de alguns elementos do DNA, fundamentais para o início da transcrição. O elemento BRE é reconhecido pelo fator de transcrição TFIIB; o TATA box é reconhecido pela proteína ligante de TATA; o INR é o elemento de iniciação reconhecido por TFIID; e DPE (do inglês-downstream promoter element), reconhecido por TFIID. Juntos, formam elementos essenciais do promotor basal para garantir o correto posicionamento da RNA polimerase para o início do processo de transcrição.
hormônios da planta ou por fatores climáticos que, então, de forma combinada, vão diminuir ou aumentar a expressão do gene que controlam (BUTLER; KADONAGA, 2002).
Dessa forma, promotores gênicos são de suma importância e essenciais para garantir a expressão de genes de interesse, que apresentam potencial de serem empregados biotecnologicamente (ZOU et al, 2011). Dentro desse aspecto, o promotor do vírus mosaico da couve-flor (CaMV35S) é um dos promotores mais utilizados em pesquisas e manipulação de plantas geneticamente modificadas (GM), por sua característica de expressar constitutivamente na maior parte dos tecidos vegetais (ODELL et al, 1985).
1.3.UTILIZAÇÃO DE PROMOTORES NA TRANSFORMAÇÃO DE PLANTAS
Existem vários promotores que são utilizados na transformação de plantas, porém o promotor 35S do vírus mosaico da couve-flor (CaMV35S), isolado e descrito em ODELL (1985) , é o mais utilizado na geração de plantas GM por induzir um nível elevado de expressão de transgenes e, portanto, bastante utilizado como modelo para a análise da expressão de genes de plantas (Figura 2) (FANG et al, 1989).
1.3.1 Promotor constitutivo CaMV35S
O promotor CaMV35S é capaz de promover a expressão constitutiva em plantas dicotiledôneas. Contudo, em plantas monocotiledôneas, a expressão controlada pelo promotor é menos efetiva (ELMAYAN; TEPFER, 1995; SCHENK et al, 2001).
Esse promotor possui regiões ativadoras que são responsáveis pelo alto nível de expressão controlada e direcionada pelo CaMV35S. Essas regiões já descritas em ODELL (1985) são localizadas na posição -343 a -46 do promotor CaMV35S, determinando a maior parte do controle da expressão do gene. Porém, relatos posteriores demonstraram que deleções internas a esse intervalo de nucleotídeos pode ser subdividido em três regiões funcionais (RONG-XIANG FANG et al, 1989). As regiões localizadas entre -343 até -208 e -208 até -90 são as responsáveis por potencializar a atividade transcricional, quando comparado com o promotor CaMV35S. A terceira região localizada entre -90 até -46 demonstrou conduzir pouca expressão gênica, porém exerce um importante papel na regulação das outras regiões, como demonstrado na figura 3 (RONG-XIANG FANG et al, 1989).
Figura 3: Representação esquemática das regiões modulares do promotor do vírus mosaico da couve flor (CaMV35S). As regiões em cinza representam os motivos ativadores do promotor CaMV35S, sendo respectivamente: -343/-208, -208/-90 e -90/-46. As duas primeiras regiões são responsáveis por potencializar a expressão do promotor. Já a terceira região desempenha um papel regulatório com as duas primeiras regiões. Em destaque vermelho, estão as sequências de CAAT-box e em amarelo, a sequência do TATA-box no promotor. Adaptada de (RONG-XIANG FANG, 1989)
Figura 4: Representação esquemática de prováveis elementos cis atuantes reconhecidos pela célula vegetal do promotor CaMV35S. Conjunto de elementos cis baseado no banco de dados PlantCARE e PLACE (LESCOT et al, 2002).
Por se tratar de uma sequência de DNA exógena, processos celulares de metilação, remodelação da cromatina e remoção do DNA exógeno fazem com que o promotor seja silenciado pela planta, encerrando dessa forma os níveis de expressão direcionados e controlados pelo promotor CaMV35S (RAWAT et al, 2011; YU et al 2011). Este efeito pode ser minimizado utilizando-se promotores endógenos (VIANA et al, 2011; LI et al, 2012).
1.3.2 Promotores tecido específicos
Promotores que possuem a característica de controlar e direcionar a expressão de genes de interesse em tecidos específicos ou, ainda, que sejam induzidos por algum tipo de estresse, passam a ter um interesse maior para a produção e desenvolvimento de produtos biotecnológicos (CHEN et al, 2011; CALLIS et al, 2014).
Exemplos de relatos sobre promotores com característica tecido-específica são
os promotores que controlam a expressão dos genes β-faseolina de feijão e α -globulina de algodão, testados em plantas de Arabidopsis e tabaco (VAN DER GEEST; HALL, 1997; RAWAT et al, 2011). O promotor do gene β-faseolina é específico em sementes, expressando de maneira significativa o gene no
endosperma e embrião da semente. A atividade do promotor α-globulina é direcionada em sementes de tabaco e Arabidopsis, e em algodão, nos cotilédones na fase de expansão (VAN DER GEEST; HALL, 1997; RAWAT et al, 2011).
Foram descritos recentemente relatos sobre a caracterização de promotores tecido-específicos de botão floral isolados de algodão. Esses promotores isolados dos genes GhPME-like1, GhβGal-like1 e GhPL-like1 demonstraram ser altamente expressos em botão floral (ARTICO et al, 2013). As análises dos elementos cis atuantes desses promotores revelaram a presença de motivos tecido-específicos que direcionam a expressão para o botão floral, incluindo elementos que estão envolvidos na expressão de pólen, frutos e grãos (ARTICO et al, 2013).
1.3.3 Promotores induzidos por fatores externos
Alguns promotores podem ser ativados em resposta a fatores externos. Estes são chamados de promotores induzíveis, expressando genes após a percepção de sinais ambientais, químicos ou lesões (ZUO et al, 2000). Em tecidos vegetais, estímulos bióticos ou abióticos provocam a expressão de genes para defesa ou para suportar o tipo de estresse que a planta está sofrendo (MARUYAMA et al, 2012).
expressão de moléculas (peptídeos e/ou proteínas) de defesa ou mecanismos de reparo. Promotores que atuam no controle de genes induzíveis possuem alto valor biotecnológico, pois são ferramentas de grande utilidade por direcionarem a expressão do transgene diretamente ao tecido lesado (YAMAGUCHI-SHINOZAKI e SHINOZAKI, 1994; GREFEN et al, 2010).
É necessário identificar promotores cuja expressão temporal dos genes de interesse seja ativada de maneira pontual já que, se o gene é expresso em situações desnecessárias há um desperdício de energia da planta para suprir uma falsa demanda, ou seja, gasto energético desnecessário (DUBEY et al, 2013).
Tendo em vista possibilidades de diferentes ferramentas biotecnológicas, as novas gerações de plantas GM utilizarão promotores tecido-específicos e/ou induzíveis. (XU et al, 2011; MAFRA et al, 2013; WEI et al, 2013).
1.4. PROMOTORES DE UBIQUITINA
Os genes relacionados com a ubiquitinação participam de funções muito importantes no sistema intracelular (VIANA et al., 2011). Dentre estas, a mais destacada é a de controle transcricional, por meio da ubiquitinação de histonas. Outras funções que a ubiquitina realiza são as de controle da tradução, regulação da endocitose, tráfego de proteínas e reparo do DNA (HERNANDEZ-GARCIA et al, 2009; MURATANI, 2003).
semelhantes (CHIERA et al, 2007; MANN et al, 2011).
A alta expressão desses promotores é diretamente influenciada quantitativamente pela presença de íntrons, localizados na 5’ UTR (untranslated region) (PARRA et al, 2011). As sequências íntrons próximas do códon de tradução são elementos cis responsáveis pelo ativador IME (intron-mediated enhancement) de expressão gênica em plantas (LU et al, 2008; SAMADDER et al, 2008).
Tendo em vista o isolamento e a caracterização de promotores de ubiquitina em vários grupos de plantas, este estudo teve como objetivo a caracterização de diferentes módulos do promotor de soja uceS8.3, isolado anteriormente em GROSSI DE SA (2010).
1.5 PROMOTOR DE SOJA UCES8.3
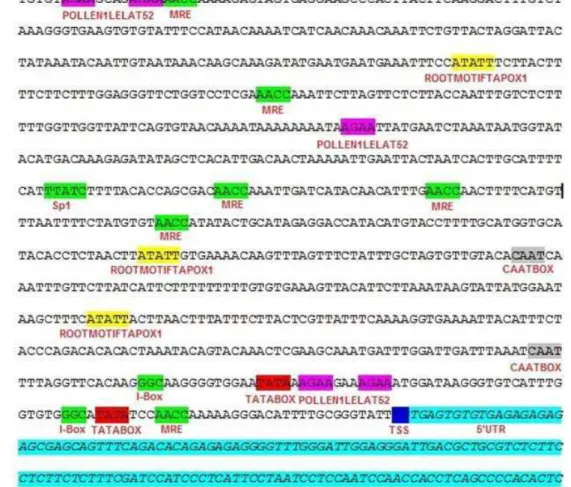
Estudos realizados através do software PlantCARE revelaram a existência de possíveis elementos cis, presentes no promotor uceS8.3. A figura 6 mostra a posição de alguns dos elementos encontrados pelo software, bem como a nomenclatura e função de cada motivo.
O promotor de soja uceS8.3 demonstrou capacidade de expressar altos níveis de expressão em diferentes tecidos de A. thaliana. Essa característica de expressão está diretamente ligada ao fato do promotor uceS8.3 ser constitutivo, direcionando assim altos níveis de expressão em tecidos vegetais como raiz, caule, folha e botão floral, quando comparado ao promotor CaMV35S.
Já foi relatado que o promotor uceS8.3 possui esse alto padrão de expressão devido a presença de íntron localizado na região +160 a +132 do gene E2 (BOURDON; LONSDALE, 2001). Essa região é conhecida como 5´UTR e, segundo trabalhos anteriores GROSSI DE SA (2010), já foi verificado que a ausência desse íntron diminui consideravelmente a expressão do gene repórter GUS em todos os tecidos vegetais (MIRANDA, 2011; VIANA et al., 2011; HERNANDEZ-GARCIA et al, 2009).
Figura 6: Posição de elementos cis atuantes encontrados pelo software PlantCARE e PLACE (LESCOT, 2002) no promotor de soja UceS8.3. Os motivos estão representados por cores e suas respectivas disposições ao longo do promotor. Em verde estão destacados os elementos específicos de folha, em amarelo os elementos específicos de raiz, em roxo os elementos específicos de botão floral e em azul claro a sequência 5’UTR. Elementos básicos do promotor como TATAbox e CAATbox estão destacados em vermelho e cinza, respectivamente. A distância entre o 5´UTR e o ATG corresponde a 24 nucleotídeos.
A análise desse promotor revelou um perfil de alta expressão do gene repórter
uidA (GUS) em plantas de A. thaliana. A expressão do gene repórter controlada pelo
Segundo GROSSI DE SA (2010), apesar da semelhança de expressão analisada em folha pelos promotores 35SdAMV e uceS8.3, os níveis de expressão em raiz, caule e tecidos florais são maiores se comparados ao 35Sd (Figura 7). Baseado nos dados obtidos é indicativo que a diferença obtida quanto aos níveis de expressão exista devido à ausência, em Arabidopsis, de elementos cis e fatores
trans atuantes de soja em Arabidopsis. E de maneira alternativa, o uceS8.3 não
possui elementos cis enhancers presentes na sequências regulatórias do gene da proteína de conjugação ubiquitina.
Os elementos cis atuantes vão orientar e posicionar corretamente a RNA polimerase para que se dê início ao processo de transcrição. A compreensão dos elementos cis é importante para caracterizar funcionalmente as regiões do promotor, bem como identificar os elementos necessários para a expressão gênica em diferentes tecidos vegetais (CHEN et al, 2012; ZHAO et al, 2012).
2. JUSTIFICATIVAS
Tendo em vista a crescente utilização de tecnologias do DNA recombinante e de transformação de plantas para a obtenção de eventos de plantas GM e a constante busca por novos genes de interesse a serem inseridos nessas cultivares, faz-se necessária também a prospecção e compreensão de sequências regulatórias da expressão gênica em sistemas vegetais.
Essa abordagem permite a geração de novas plantas GM expressando genes de interesse em tecidos específicos, criando novas perspectivas para obtenção de plantas resistentes a pragas que atendam às demandas específicas de expressão.
A utilização de promotores isolados de sistemas vegetais para geração de plantas GM, possuem preferência em relação a promotores derivados de outros organismos. O órgão responsável pela liberação de eventos de plantas transgênicas, CTNBio, entende a utilização de promotores isolados de vegetais em plantas GM possui riscos de biossegurança reduzidos em comparação a promotores derivados de outros organismos.
Dentro desse contexto, um fator importante é a preocupação com a proteção intelectual de módulos do promotor uceS8.3, a fim de diminuir o custo de geração de plantas GM, produzidas em território brasileiro e a valorização dos royalties nacionais em âmbito mundial. Além, da liberdade de desenvolvimento de produtos por toda empresa sem a necessidade da utilização de promotores patenteados por outros órgãos vinculados a pesquisa.
3. OBJETIVO
Caracterizar funcionalmente os módulos do promotor do gene da proteína de conjugação ubiquitina em plantas de A. thaliana.
3.1 OBJETIVOS ESPECÍFICOS
Definir os fragmentos das regiões regulatórias do promotor uceS8.3.
Amplificar os módulos do promotor de soja uceS8.3.
Clonar os módulos no vetor de transformação de plantas PKGWFS7.
Transformar A. tumefaciens com os vetores construídos.
4. METODOLOGIA
4.1 DEFINIÇÃO E AMPLIFICAÇÃO DOS FRAGMENTOS DO PROMOTOR UCES8.3 A fim de gerar os fragmentos do promotor uceS8.3 funcionais, foi necessário garantir a presença de elementos essenciais cis atuantes em cada módulo. Portanto, elementos do núcleo do promotor como o TATABOX e CAATBOX foram mantidos e todas as construções. Em um segundo momento, a definição dos tamanhos variados de cada módulo foi determinada de forma aleatória, a fim de que se possa estudar a atividade regulatória do promotor com um conjunto de motivos sendo removidos de maneira gradual. Para obtenção dos módulos do promotor uceS8.3 foi realizada a amplificação destes fragmentos pela reação em cadeia da polimerase (PCR) utilizando-se primers desenhados em nosso laboratório. A reação continha o volume total de 20 µL, sendo 1 µL de DNA molde (uceS8.3) a partir da concentração de 1 µg/µL, 0,5 µL de primer forward a 12,5 µM, 0,5 µL de primer reverse a 12,5 µM, 0,5 µL de Platinum Taq DNA Polimerase High Fidelity 5U/µL, 0,5 µL de dNTP a 10 mM, 2 µL tampão High Fidelity Reaction Buffer 10X, que contém 600mM de Tris - SO4 (pH 8.9) e 180mM de (NH4)2SO4, 0,8 µL de MgSO4 a 50mM e 14,2 µL de água Milli-Q.A PCR foi incubada a 95°C por 5 minutos, 35 ciclos de 1 minuto a 95°C, 1 minuto a 55°C e 1 minuto e 30 segundos a 68°C. Depois dos 35 ciclos, uma incubação final de 5 minutos a 68°C, finalizando a reação em 4°C. Foram feitas quatro reações, cada uma contendo um par diferente de primers para cada módulo desejado. Uma alíquota de 5 µL de cada PCR foi utilizada para a confirmação da amplificação em gel de agarose 1%.
4.2 QUANTIFICAÇÃO DO PRODUTO DE PCR
4.3 CLONAGEM NO VETOR INTERMEDIÁRIO PDONR 221
Os fragmentos da PCR foram clonados no vetor intermediário pDONR 221 (Invitrogen) (Figura 12) seguindo a reação utilizada: 6 µL de DNA amplificado na concentração de 20 ng/ µL, 2 µL(120 ng/ µL) do vetor pDONR 221, 2 µL da enzima BP clonase. Incubando a reação em temperatura ambiente (25°C) over night (aproximadamente 16h), logo após foi adicionado 1 µL de proteinase K e incubado a 37°C por 15 minutos.
Figura 12: Mapa do vetor pDONR 221 indicando o sítio de clonagem dos módulos do promotor
4.4 TRANSFORMAÇÃO DE CÉLULAS COMPETENTES DE E. COLI E SELEÇÃO DAS COLÔNIAS POSITIVAS
Células competentes do kit de clonagem foram utilizadas para a transformação. 50 µL de células competentes One Shot TOP10 foram incubados no gelo por 20 minutos, aplicando-se o choque térmico a 42 °C por 90 segundos e retornando as células para o gelo por 2 minutos. Dentro do fluxo laminar foram adicionados 150 µL de meio SOC, deixando as células por 1 hora a 37 °C na estufa. Após esse período, todo o conteúdo foi plaqueado numa placa de meio LB sólido contendo o antibiótico canamicina (50 mg/mL) e incubado na estufa por 16 h.
Das colônias crescidas na placa foram feitos inóculos em meio LB líquido de 5 mL contendo o antibiótico canamicina (50 mg/mL) em agitador a 37 °C numa rotação de 200 rpm por 16 horas.
4.5 EXTRAÇÃO DE DNA PLASMIDIAL
no freezer a -20°C.
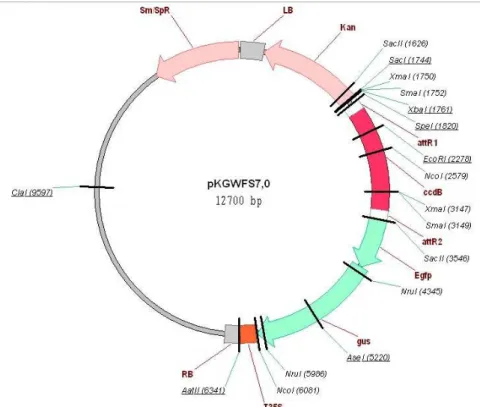
4.6 RECOMBINAÇÃO LR DO VETOR PDONR 221 COM PKGWFS7
Figura 13: Vetor pKGWFS7 indicando o sítio de recombinação LR em vermelho. Em verde, os genes repórteres cuja expressão será controlada pelos módulos do promotor uceS8.3. Em rosa, os genes de seleção de transformantes, tanto em E.coli quanto em plantas. Disponível em:
http://gateway.psb.ugent.be/vector/show/pKGWFS7/search/index/
4.7 CONFIRMAÇÃO DA SUBCLONAGEM NO VETOR PKGWF7 POR PCR
Após a extração do DNA plasmidial, foi realizada uma reação de PCR utilizando o mesmo protocolo já descrito anteriormente com os pares de primers para cada módulo do promotor. Para a reação de digestão foi utilizado o seguinte protocolo: 5 µL do DNA de interesse, 2 µL de tampão NEB 4 (New England Biolabs), 2 µL da enzima de restrição SacII e para completar o volume 11 µL de água MilliQ, totalizando o volume final de 20 µL de reação.
4.8 TRANSFORMAÇÃO DE AGROBACTERIUM TUMEFACIENS
gelo. Após esse tempo, as células foram colocadas na cuveta e dado o choque elétrico. Rapidamente foi adicionado 1 mL de meio LB estéril à cuveta. O volume foi transferido a um microtubo e incubado na estufa a 28°C por 2 horas. Posteriormente as células transformadas foram plaqueadas em placas de LB contendo os antibióticos rifampicina 50 mg/mL gentamicina 50 mg/mL e espectinomicina 50 mg/mL e incubadas por 48h a 28 °C. Para a seleção das colônias positivas foi realizada uma PCR de colônia.
4.9 TRANSFORMAÇÃO DE ARABIDOPSIS THALIANA E SELEÇÃO DAS SEMENTES TRANSFORMADAS
Das colônias positivas confirmadas por PCR, células foram inoculadas em 125 mL de meio LB contendo os respectivos antibióticos descritos anteriormente, crescidas sob agitação de 180 rpm a 28 °C. O meio contendo as bactérias foi centrifugado a 4000 rpm por 10 minutos em temperatura ambiente. O pellet de células foi ressuspendido numa solução de 5% de sacarose e 0,05% de Silwet L-77 para que a OD600 chegasse a 0,8. As Arabidopsis com flores foram imersas na solução de agrobactéria e deixadas por 5 segundos para a infiltração. Para cada construção foram transformadas 45 plantas (3 plantas em cada copo de cultivo totalizando 15 copos). Posteriormente, foram ensacadas com saco plástico e deixadas numa sala sem luz por 24 h. Após esse período, os plásticos foram removidos e as plantas encaminhadas para a sala climatizada (20 °C a 26 °C) e com fotoperíodo (dia 16 h/ noite 8 h) para que se recuperassem e produzissem as sementes. Tal evento leva de 30 a 45 dias até a coleta (CLOUGH, 1998). Posteriormente as sementes foram esterilizadas para seleção de transformantes com etanol 70% por 10 minutos. No fluxo de plantas retira-se o álcool e adiciona-se 1ml de solução de esterilização (0,05% Triton X-100 e álcool 70%) por 5 minutos agitando levemente. Retira-se a solução de esterilização e as sementes foram lavadas com etanol 100% por 3 vezes e deixadas para secar.
são aclimatadas em casa de vegetação para Arabidopsis com temperatura controlada entre 20 °C e 26 °C e fotoperíodo (dia 16 h/noite 8 h).
4.10 ENSAIO HISTOQUÍMICO DE GUS
Com a planta em estado de desenvolvimento um pouco mais avançado, foi possível coletar umas amostras de folhas para confirmação das plantas transformadas. Após a confirmação e seleção das plantas positivas foram feitos cortes nos tecidos vegetais tais como: raiz, folha, caule, e botão floral. Esses tecidos foram incubados na solução x-gluc (NaH2PO4.H2O 100mM, KNaH2PO4.H2O 100mM, K4Fe(CN)6.3H2O 0,5Mm, Na2EDTA.2H2O 10mM, Triton® X-100 0,1%, x-Gluc 50mg/mL 1mM e H2O destilada para o volume de solução desejado, mantendo as concentrações finais), incubadas a 37°C no escuro por 16 horas para avaliação da expressão de GUS em diferentes tecidos da planta. Após a incubação os tecidos foram lavados com etanol 70% para interromper a reação e retirar a clorofila, permitindo melhor visualização da cor azul nos tecidos.
4.11 EXTRAÇÃO DE RNA E SÍNTESE DE CDNA
adicionado 500 µL de tampão RPE centrifugado por 15 segundos a 10.000rpm. Novamente foi descartado o líquido coletado e adicionados 500 µL de tampão RPE na coluna e centrifugado por 2 minutos na velocidade máxima para secagem da membrana. A coluna foi transferida para um microtubo novo e nela adicionada 30 µL de água Rnase-free. Os tubos foram centrifugados por 1 minuto a 10.000rpm para eluição do RNA.
A partir do RNA extraído foi iniciado a síntese de cDNA no mesmo dia da extração de RNA. O kit utilizado foi o Super Script III First-Strand Synthesis Super Mix for a qRT-PCR da INVITROGEN. Para a síntese de cDNA o RNA extraído de cada tecido da planta e de cada construção foi diluído 1:20 e a partir dessa diluição foram utilizados 3 µL para a reação, seguindo o protocolo do fabricante. Para cada reação foram adicionados 3 µL do RNA , 10 µL da solução 2X RT Reaction Mix, 2 µL de RT enxime MIX e 5 µL de DEPC-treated ucater, totalizando 20 µL de reação. Todos os reagentes foram misturados gentilmente e incubados no termociclador em um ciclo de 25°C por 10 minutos, 50°C por 30 minutos e a 85°C por 5 minutos. Após esse ciclo, as amostras foram incubadas no gelo por 5 minutos e adicionado 1 µL (2U) de RNAse H e incubadas a 37°C no termociclador por 20 minutos. Posteriormente as amostras foram estocadas a -20°C.
4.12 PCR EM TEMPO REAL
A partir dos cDNAs sintetizados, foram realizadas diluições de 1:20 para cada cDNA e estas foram utilizadas como molde na PCR em tempo real. As reações de qRT-PCR foram conduzidas no termociclador 7500 fast real-time PCR System da Applied Biosystems, com o software 7500 software v2.3. A PCR continha Syber Green Rox Plus Master Mix 2X(LGC), 200nM de cada primer (Actina, GAPDH, GUS e GFP) e 2 µL de cDNA dos tecidos de cada construção. Todos os experimentos foram analisados em triplicata experimental, e o programa utilizado na máquina foi: 95° por 15 minutos, 95°C por 20 segundos, 55° por vinte segundos, e 72° por vinte segundos repetidos por 40 ciclos. O método estatístico utilizado pelo software foi o
4.13 ENSAIOS FLUORIMÉTRICOS DE GUS
Plantas crescidas em placas contendo meio MS com aproximadamente 2 semanas de crescimento foram utilizadas para a realização dos ensaios. Somente puderam ser utilizados os tecidos vegetais folhas, uma vez que o estágio de desenvolvimento das plantas ainda era prematuro e estas se encontravam in vitro.
Aproximadamente 100mg do tecido de cada construção foram macerados em nitrogênio líquido e transferidos para tubos de micro centrífuga. Foram adicionado 500µL de tampão de extração (50 ml de Tampão fosfato 0,2M pH 7,0- concentração final de 100mM; 2 mL EDTA 500mM pH 8 - concentração final de 10nM; 330µL Sarcosil 30% - concentração final de 0,1%; 100 µL de Triton X-100 - concentração final de 10nM; 70 µL β-mercaptoetanol - concentração final de 10mM e H2O - completar o volume para 100mL). Os extratos foram centrifugados a 12.000rpm por 10 minutos e o sobrenadante transferidos para novos tubos. Para a quantificação de proteínas, um ensaio de Bradford foi realizado e, a partir das concentrações obtidas, os extratos foram diluídos para a concentração final de 1µg de proteína.
Para a reação enzimática, foram distribuídos 150 µL de tampão de reação (MUG 2mM) em placa de elisa para triplicatas de cada amostra e esta foi incubada a 37°C para pré aquecimento. Foram adicionados 50 µL de cada extrato para a reação. A cada 15 minutos foram coletados 20 µL da reação e transferidos para 1980 µL de tampão de parada (Na2CO3 0,2M). Foram coletados 4 pontos, totalizando 1 hora de reação enzimática.
5. RESULTADOS
5.1 AMPLIFICAÇÃO E CLONAGEM DOS MÓDULOS DO PROMOTOR UCES8.3 NO VETOR PDONR221
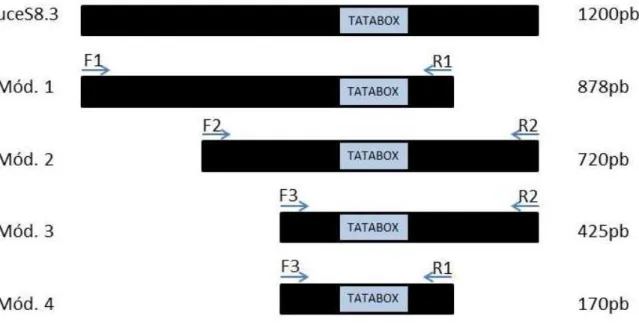
Visando estudar os níveis de expressão conduzidos pelos módulos do promotor de soja uceS8.3, cada construção foi subclonada em vetor de transformação de plantas contendo os genes repórteres GUS e GFP. A amplificação dos módulos do promotor de soja uces8.3 foi realizada a reação em cadeia da polimerase (PCR), utilizando-se os respectivos primers de cada módulo, esquematizado na Tabela 1, além do tamanho, em pb, do promotor uceS8.3 e seus respectivos módulos. Na figura 14 estão esquematizados o promotor uceS8.3 e as diferentes deleções de cada um dos módulos gerados. Os produtos da PCR foram analisados em gel de agarose, como consta na Figura 15.
Tabela 1: Quadro indicando a relação de cada módulo com o par de oligonucleotídeos
correspondente para sua amplificação e seu tamanho em pb.
Fragmentos do promotor Par de primers Tamanho do fragmento (pb)
uceS8.3 (promotor inteiro) - 1200
1 F1 e R1 878
2 F2 e R2 720
3 F3 e R2 425
Figura 14: Representação esquemática das deleções de nucleotídeos de cada par de oligonucleotídeos, a partir do promotor uceS8.3 contendo 1200pb.
O DNA dos módulos foram quantificados para serem clonados no vetor intermediário pDONR 221. Os produtos da PCR foram então utilizados para a recombinação no vetor pDONR 221. As células competentes de E.coli foram transformadas. A confirmação da clonagem foi realizada por PCR de colônia, como mostrado na figura 16.
Figura 16: PCR de colônia confirmando a clonagem dos módulos 1 a 4 do promotor uceS8.3 no vetor pDONR 221. MW- Marcador molecular 1 kb plus, amplicon do módulo 1 possui 878 pb, o módulo 2 possui 720 pb, o módulo 3 possui 425 pb, e o módulo 4 possui 170 pb.
.
5.2 SUBCLONAGEM DAS CONSTRUÇÕES DOS MÓDULOS NO VETOR DE TRANSFORMAÇÃO DE PLANTAS PKGWFS7
Figura 18: Análise por PCR dos fragmentos dos módulos 3 e 4 do promotor uceS8.3 no vetor pKGWFS7 recombinado. MW - 1kb plus. Tamanho dos módulos em pb respectivamente: 425 e 170.
5.3 TRANSFORMAÇÃO DE CÉLULAS COMPETENTES DE AGROBACTÉRIA
Figura 19: PCR de colônia de Agrobactéria para a confirmação da transformação com o vetor pKGWFS7. MW- 1 kb plus. Para os módulos 1, 2 e 3 foram adquiridas duas colônia de Agrobactéria PCR positiva para a clonagem, já para os módulos 4 foram obtidas três colônias. Tamanho dos módulos em pb respectivamente: 878, 720, 425 e 170.
5.4 TRANSFORMAÇÃO DE A. THALIANA E SELEÇÃO DE PLANTAS POSITIVAS in
vitro
Figura 20: Fotos do processo de transformação de Arabidopsis thaliana. Em 1) células de agrobactéria em solução de transformação. Em 2) imersão dos botões florais na solução de agrobactéria. Em 3) aclimatação em ambiente úmido. E em 4) plantas sendo organizadas em bandejas para serem levadas a um ambiente com luminosidade baixa ou, se possível, sem luz, para melhorar eficiência de transformação.
Foram realizados onze eventos de transformação de plantas, considerando que em cada copo de cultivo havia um total de três plantas. Logo, para cada módulo foram transformadas 33 plantas (Tabela 2). As sementes então foram coletadas e por porções de sementes, foram esterilizadas e plaqueadas em meio MS contendo o antibiótico canamicina para a seleção dos transformantes.
Tabela 2: Relação da quantidade de plantas transformadas por módulos do promotor uceS8.3
Módulos do promotor uceS8.3 Número de plantas transformadas Plantas positivas obtidas
uceS8.3 (promotor inteiro) 33 (3 plantas por copo) 10
Módulo 1 33 (3 plantas por copo) Em andamento
Módulo 2 33 (3 plantas por copo) 10
Módulo 3 33 (3 plantas por copo) Em andamento
Para a identificação das plantas transformadas na placa com meio MS contendo o agente seletivo canamicina, foi necessário um período de 2 a 3 semanas. Inicialmente todas as sementes germinaram, porém no decorrer dos dias as plantas que não possuíam o vetor com o gene de seleção começaram a ficar descoloridas (Figura 21). Somente as plantas GM contendo o gene de seleção ao meio desenvolveram raiz. Devido ao escape na seleção dos transformantes, se faz necessária a espera de 2 a 3 semanas de incubação na sala de cultivo de plantas.
Figura 21: Seleção de plantas transformadas em meio MS contendo o agente seletivo canamicina 50mg/ml. As plantas positivas crescem e desenvolvem raiz, ao contrário das plantas não transformadas que não desenvolvem coloração e pouca raiz. Plantas não transformadas na área destacada.
5.5 ENSAIO HISTOQUÍMICO DE GUS
Obtidas as plantas positivas para cada construção, foi realizado ensaio histoquímico de GUS nos tecidos: folha, caule, raiz e botão floral. Os tecidos foram separados e incubados na solução x-gluc. Após o período de incubação, as amostras foram descoloridas e fotografadas.
Como mostra a figura 22, a atividade do gene repórter GUS, sob controle da promotor 35Sd não foi tão elevada em botão floral, quando comparada com a atividade GUS sob controle das demais construções. No caule da construção do promotor uceS8.3 há a presença de uma pequena coloração azul, não sugerindo, de fato, atividade de GUS, sendo, possivelmente, um background causado pelo corte.
5.6 PCR EM TEMPO REAL DE GUS E GFP
A partir da síntese de cDNA foi realizada a reação de PCR em tempo real para as amostras de plantas não transformadas, com o promotor CaMV35S, o promotor uceS8.3, módulo 2 e 4 do promotor uceS8.3. Os dados brutos foram analisados no próprio software da PCR em tempo real e a partir desses dados foi elaborado um gráfico para cada gene repórter (Figuras 23 e 24).
Apesar da existência de transcritos para o gene repórter GFP, não é possível visualizar a proteína ativa nos tecidos vegetais transformados. A sequência de GFP desse vetor de transformação de plantas (pKGWFS7) não possui a sinalização da protéina para o retículo endoplasmático, por isso não há a possibilidade de se visualizar a fluorescência da proteína.
Figura 24: Quantificação relativa da expressão do gene Repórter GFP em diferentes tecidos da planta. Em vermelho está representada a expressão relativa do promotor CaMV35S; em verde, a expressão do promotor uceS8.3; em roxo, a expressão do módulo 2 e em laranja, a expressão do módulo 4.
.
5.7 ENSAIO FLUORIMÉTRICO DE GUS
Com os extratos brutos de proteína foram realizados ensaios fluorimétricos para avaliar a atividade específica de GUS em tecidos de folha. Com os dados obtidos no fluorímetro, foram realizados cálculos para identificar a atividade específica de GUS sob o substrato, tendo o resultado obtido por pmol/min/ug de proteína, como mostram as figuras 25 e 26.
uceS8.3 e pelos módulos do uceS8.3, módulo 2 e módulo 4 em folha. A capacidade do módulo 2 de conduzir a expressão de GUS foi menor em folha comparado ao promotor uceS8.3 e o módulo 4. O módulo 4 mostrou níveis de expressão semelhantes ao uceS8.3.
6. DISCUSSÃO
6.1 ANÁLISE DOS ELEMENTOS CIS NOS MÓDULOS DO PROMOTOR
UCES8.3 PELO ENSAIO HISTOQUÍMICO
Os módulos do promotor uceS8.3, juntamente com os controles de expressão pelo promotor CaMV35S e próprio promotor de soja uceS8.3, foram avaliados em A.
thaliana. Planta de A. thaliana tem sido bastante utilizada como um sistema
heterólogo para caracterização dos elementos cis de promotores de plantas dicotiledôneas (ARTICO et al, 2013; KOORNEEF, 2010; KOIA et al, 2013)
O promotor de soja uceS8.3 demonstrou conduzir alta expressão em folha, raiz, caule e botão floral, como relatado por GROSSI DE SA (2010). A expressão constitutiva desse promotor ocorre devido a elementos cis atuantes que o controlam e direcionam para diferentes tecidos vegetais (MIRANDA, 2011). Já os altos níveis de expressão gênica, controlados por esse promotor, são relacionados à presença de um íntron na região 5´UTR. Há relatos de que a ausência do íntron localizado nessa região do gene E2 diminui os níveis de expressão dos promotores de poliubiquitina, demonstrando sua importância para controlar altos níveis de expressão gênica (HERNANDEZ-GARCIA et al, 2009; VIANA et al, 2011; WHALLEY et al, 2011).
Os bancos de dados PlantCARE, PLACE e Plant-PAN (W.C. CHANG, 2008) identificaram os prováveis elementos cis como o Sp1(CC(G/A)CCC), MRE (AACCTAA), I-box (GATAAGGGT) que estão envolvidos na expressão em folha, pois estes estão relacionados ao processo de fotossíntese (TRAN THANH et al, 2012; SAFRANY et al, 2008; WANG et al, 2013). O destaque para esses elementos é devido a ocorrência destes em todos os módulos do promotor uceS8.3, aqui caracterizados. Para o caule, não foram encontrados elementos cis específicos.
sugere que as deleções de suas repetições não interferem na expressão do gene (SUN, 2010). Promotores isolados de algodão como o GhPME-like1 e GhβGal-like1, demonstraram ser específicos de pólen, direcionando a expressão do gene para o botão floral (ARTICO et al, 2013; SUN, 2010)
Elementos específicos de raiz foram encontrados na sequência do promotor e seus módulos. O elemento cis envolvido na expressão da raiz foi o ROOTMOTIFTAPOX. Este motivo é encontrado pelo menos uma vez no promotor uceS8.3 e em seus módulos (WITTKOPP, 2012; ROSE, 2002). Os prováveis motivos encontrados nos módulos e no promotor uceS8.3 estão esquematizados na figura 27.
Figura 27: Representação esquemática da presença de prováveis elementos cis atuantes no promotor de soja uceS8.3 e seus módulos. Os elementos basais CAAT-box e TATAbox estão representados pelos triângulos. Os elementos específicos estão representados por: círculo (Folha), retângulo (Raiz), círculo e quadrado preenchidos (Botão floral, Pólen e Flor).
6.2 ANÁLISE DA EXPRESSÃO RELATIVA DE mRNA E ATIVIDADE ESPECÍFICA DE GUS
Analisando os dados obtidos pela PCR em tempo real, a atividade específica de GUS e os ensaios histoquímicos é possível perceber a diferença entre níveis de transcritos com a proteína em sua forma ativa. O ensaio histoquímico de GUS é um ensaio qualitativo, ou seja, apenas indica a presença do DNA exógeno na planta. Os dados da PCR em tempo real, por outro lado, indicam os níveis de transcrito do gene controlado pelos promotores. Já o ensaio fluorimétrico mostra a atividade específica da enzima β-glucuronidasesobre o substrato MUG.
De fato, a ausência de alguns elementos cis caracterizam os níveis de expressão avaliados (HERNANDEZ-GARCIA et al, 2014). Os elementos que são responsáveis pela expressão nos tecidos foram identificados e relacionados à expressão de transcritos em PCR em tempo real, confirmando a sua importância no promotor uceS8.3 e em seus respectivos módulos.
Ao analisar os resultados da PCR em tempo real e do ensaio fluorimétrico de GUS nos tecidos folha e raiz, pode-se notar uma correlação entre os níveis de
transcrito e os níveis de atividade específica da enzima β-glucuronidase no módulo 2, porém há uma diferença na correlação destes níveis entre o promotor uceS8.3 e o módulo 4. Os níveis de transcritos do promotor uceS8.3 em folha e do módulo 4 em raiz são mais elevados, porém, ao analisar o ensaio fluorimétrico de GUS, pode-se notar que os níveis de atividade específica da enzima β-glucuronidase entre as duas construções são semelhantes para os dois tecidos. Pode-se supor que essa diferença entre os níveis de transcrito possa estar relacionada a algum fator abiótico ou a uma correlação entre mRNA e proteína, não totalmente esclarecida (DOV GREENBAUM et al, 2003; VELEZ-BERMUDEZ; SCHMIDT et al, 2014).
(KRISTENSEN et al, 2013; VELEZ-BERMUDEZ; SCHMIDT, 2014).
De acordo com DE SOUSA ABREU (2009), a diversidade celular contribui para a diferença entre mRNA e abundância de proteínas. Relatos em raízes de A.
thaliana demonstram que o tecido responde à deficiência de fosfato no solo
regulando os níveis de transcritos com a abundância de proteínas produzidas, o que pode justificar a diferença de resultados encontrados nos ensaios fluorimétricos com a expressão relativa de mRNA (LAN; SCHMIDT, 2012; VÉLEZ-BERMÚDEZ; SCHMIDT, 2014). Em plantas, a eficiência no processo de tradução pode mudar em resposta a efeitos abióticos, levando a um viés no número de mRNA que serão efetivamente traduzidos (TOBIAS; SERRANO, 2009; BAERENFALLER et al, 2012; WALLEY et al, 2013).
7. CONCLUSÃO
De acordo com os objetivos propostos, o presente estudo demonstra novas informações a respeito dos módulos do promotor uceS8.3 por meio das análises histoquímicas e fluorimétricas de GUS e PCR em tempo real em nível transcricional.
Foram caracterizados dois módulos dos quatro propostos. Estes módulos demonstraram conduzir níveis de expressão semelhantes ao promotor uceS8.3. Dentro desses módulos, foram encontrados alguns elementos cis regulatórios necessários para conferir níveis de expressão do gene repórter, como os: Sp1, MRE, IBox, ROOTMOTIFTAPOX e POLLEN1LELAT52. A análise quantitativa dos níveis de transcritos pela PCR em tempo real revela que o padrão de expressão controlado pelo módulo 4 é semelhante a do promotor uceS8.3. Já o padrão de expressão controlado pelo módulo 2 demonstra níveis inferiores quando comparado ao promotor uceS8.3.
A análise do ensaio fluorimétrico de GUS demonstrou que a quantidade de transcritos não está diretamente relacionada aos níveis de atividade específica da enzima. Isso demonstra que o processo de tradução é independente do processo de transcrição, ou seja, o número de mRNA não necessariamente corresponde ao número de proteínas ativas.
8. PERSPECTIVAS DE ESTUDOS FUTUROS
Este trabalho tem como estudos futuros a realização de ensaios fluorimétricos de GUS, Elisa quantitativo para avaliar a relação entre mRNA e proteínas na sua forma ativa, além da compreensão dos outros módulos do promotor. Há também o intuito de analisar mecanismos de regulação e identificar módulos que dirijam altos níveis de expressão, quando submetidos a estresses bióticos e abióticos, visando identificar a resposta desses módulos nos tecidos específicos, relacionando com a presença dos elementos cis nos módulos do promotor.
9. REFERÊNCIAS BIBLIOGRÁFICAS
AMARASINGHE, B., FAIVRE-NITSCHKE, E, ET AL. Genomic approaches to the discovery of promoters for sustained expression in cotton (Gossypium hirsutum L.) under
field conditions: expression analysis in transgenic cotton and Arabidopsis of a Rubisco small
subunit promoter identified using EST sequence analysis and cDNA microarrays. Plant Biotechnology Journal. n. 23, p. 437-450, 2006.
ARTICO, S., JULIA LAMBRET-FROTTÉ, SARAH MUNIZ NARDELI, OSMUNDO BRILHANTE OLIVEIRA-NETO, MARIA FÁTIMA GROSSI-DE-SA, MARCIO ALVES-FERREIRA. Isolation and Characterization of Three New Promoters from Gossypium
hirsutum that Show High Activity in Reproductive Tissues. Plant Mol Biol Rep. 2013.
BAERENFALLER, K., MASSONNET, C., WALSH, S., BAGINSKY, S., BÜHLMANN, P., HENNIG, L., HIRSCH-HOFFMANN M, HOWELL, K. A., KAHLAU, S., RADZIEJWOSKI, A., RUSSENBERGER, D., RUTISHAUSER, D., SMALL, I., STEKHOVEN, D., SULPICE, R., SVOZIL, J., WUYTS, N., STITT, M., HILSON, P., GRANIER, C., GRUISSEM, W. Systems based analysis of Arabidopsis leaf growth reveals adaptation to water deficit. Mol. Syst. Biol. n. 8, 2012.
BANG, S.W., PARK, S. H., JEONG, J.S., KIM, Y.S., JUNG, H., HÁ, S.H., KIM, J.K. Characterization of the stress-inducible OsNCED3 promoter in different transgenic rice organs and over three homozygous generations. Planta. p. 211-224, 2013
BATUT, P., DOBIN, A., PLESSY, C., CARNINCI, P., GINGERAS, T.R. High-fidelity promoter profiling reveals widespread alternative promoter usage and transposon-driven developmental gene expression. Genome Res. p.169-180, 2013
BOURDON, V. H., A.; LONSDALE, D, M. Introns and their positions affect the translational activity of mRNA in plant cells. EMBO Rep. n. 2, p. 394-398, 2001.
BUTLER, J., KADONAGA, JT. The RNA polymerase II core promoter: a key component in the regulation of gene expression. Genes & Development. n. 6, p. 2583-2592, 2002.
CALLIS, J., RAASCH, J.A., VIERSTRA, R.D. Ubiquitin extension proteins of Arabidopsis
thaliana. Structure, localization, and expression of their promoters in transgenic tobacco. J
Biol Chem. 1990
CALLIS, J. The ubiquitination machinery of the ubiquitin system. The Arabidopsis book.
2014.
CHEN, L. M. Y., WANG, C., SU, P., LI, T., WANG, R., HAO, X.,YANG, G., HEG, G. A. O. C. Characterization of a novel pollen-specific promoter from wheat (Triticum Aestivum L.).
CHEN, Z.H., GREFEN, C., DONALD, N., HILLS, A., BLATT, M. A bicistronic,ubiquitin-10 promoter-based vector cassette for transient transformation and functional analysis of membrane transport demonstrates the utility of quantitative voltage clamp studies on intact
Arabidopsis root epidermis. Plant Cell Environ. n.34, p. 224-564, 2011
CHIERA, J. M., B. R., DORSEY SL, PARK EH, BUENROSTRO-NAVA MT, LING PP, FINER JJ. Isolation of two highly active soybean (Glycine max (L.) Merr.) promoters and
their characterization using a new automated image collection and analysis system. Plant Cell
Rep. n. 26, p. 1501-1509, 2007.
CHRISTENSEN, A. H., Q. P. Ubiquitin promoter-based vectors for high levels of selectable and/or screenable marker genes in monocotyledonous plants. Transgenic Res. n. 5, p.
213-218, 1996.
CLOUGH, S. J., BENT, A.F. Floral dip: a simplified method for Agrobacterium-mediated
transformation of Arabidopsis thaliana. Plant J. n. 16, p. 735-743, 1998.
DE SOUSA ABREU, R., PENALVA, L. O., MARCOTTE, E. M.,; AND VOGEL, C. Global signatures of protein and mRNA expression levels. Mol. Biosyst. n. 5, p. 1512-1526, 2009.
DENNIS, A. P., O´MALLEY, B. W. Rush hour at the promoter: How the ubiquitin-proteasome pathway polices the traffic flow os nuclear receptor-dependent transcription. J
Steroid Biochem Mol Biol. n. 93, p. 139-151, 2005.
DOV GREENBAUM, C. C., KENNETH WILLIAMS, MARK GERSTEIN. Comparing protein abundance and mRNA expression levels on a genomic scale. Genome Biol. 2003.
DUBEY, N., GOEL, R, RANJAN, A, IDRIS, A, SINGH, SK, BAG, SK, CHANDRASHEKAR, K, PANDEY, KD, SINGH, PK, SAWANT, SV. Comparative transcriptome analysis of Gossypium hirsutum L. in response to sap sucking insects: aphid and
whitefly. BMC Genomics. n 14, p. 241, 2013.
ELMAYAN, T., TEPFER, M. Evaluation in tobacco of the organ specificity and strength of the rolD promoter, domain A of the 35S promoter and the 35S2 promoter. Transgenic Res. n.
4, p. 388-396, 1995.
FANG, R. X. et al. Multiple cis regulatory elements for maximal expression of the cauliflower mosaic virus 35S promoter in transgenic plants. Plant Cell. n. 1, p. 141-150, 1989.
GREFEN, C., DONALD, N., SCHUMACHER, K., BLATT, M. A ubiquitin-10 promoter-based vector set for fluorescent protein tagging facilitates temporal stability and native protein distribution in transient and stable expression studies. Plant J. n. 64, p. 355-365, 2010
HERNANDEZ-GARCIA, C. M., FINER, J. J. Identification and validation of promoters and cis-acting regulatory elements. Plant Science. p. 109-119, 2014.
HERNANDEZ-GARCIA, C., M.; MARTINELLI, A. P.; BOUCHARD, R. A.; FINER, J. J. . A soybean (Glycine max) polyubiquitin promoter gives strong constitutive expression in
transgenic soybean. Plant Cell Rep. n. 28, p. 837-849, 2009.
INGOLIA, N. T. Ribosome profiling: new views of translation, from single codons to genome scale. Nat. Rev. Genet. n. 15, 2014.
JUNTAWONG, P., GIRKE, T., BAZIN, J., AND BAILEY-SERRES, J., SMITH, L. G., ET AL. Translational dynamics revealed by genome-wide profiling of ribosome footprints in
Arabidopsis. Proc. Natl. Acad. Sci. n. 111, p. 203-212, 2014.
KOHLI, A. Molecular characterization of transforming plasmid rearrangement in transgenic rice reveals a recombination hotsport in the CaMV promoter and confirms the predominace of microhomology mediated recombination. The Plant Journal. n. 17, p. 591-601, 1999.
KOIA, J. M. R., HENDRY, C., LIM, L., BOTELLA, J.R. Pineapple translation factor SUI1 and ribosomal protein L36 promoters drive constitutive transgene expression patterns in
Arabidopsis thaliana. Plant Mol Biol. n. 81, p. 327-336, 2013.
KOORNEEF M, M. D. The development of Arabidopsis as a model plant. Plant J. n. 61, p.
909-921, 2010.
KRISTENSEN, A. R., GSPONER, J., AND FOSTER, L. J. Protein synthesis rate is the predominant regulator of protein expression during differentiation. Mol. Syst. Biol. n. 689,
2013.
KWON, S. C., YI, H., EICHELBAUM, K., FOHR, S., FISCHER, B., YOU, K. T., ET AL. The RNA-binding protein repertoire of embryonic stem cells. Nat. Struct. Mol. Biol. n. 20, p.
1122-1130, 2013.
LAN, P., LI, W., AND SCHMIDT, W. . Complementary proteome and transcriptome profiling in phosphate-deficient Arabidopsis roots reveals multiple levels of gene regulation. Mol. Cell. Proteomics. n. 11, p. 1156-1166, 2012.
LESCOT, M., DÉHAIS, P., MOREAU, Y., DE MOOR, B., ROUZÉ ,P.,AND ROMBAUTS, S. PlantCARE: a database of plant cis-acting regulatory elements and a portal to tools for in silico analysis of promoter sequences. Nucleic Acids Res., Database issue. n. 30, p. 325-327,
2002.
LI, Z. T., KIM, K. H., JASINSKI, J. R., CREECH, M. R., GRAY, D. J. Large-scale characterizationof promoters from grapevine (Vitis spp.) using quantitative anthocyanin and
GUS assay systems, Plant Science. n. 196, 2012
MAFRA, V., MARTINS, PK, FRANCISCO, CS, RIBEIRO-ALVES, M, FREITAS-ASTÚA, J, MACHADO, MACKAY, I. M., MACKAY, J.F., NISSEN, M.D., SLOOTS, T.P. Candidatus Liberibacter americanus induces significant reprogramming of the transcriptome of the susceptible citrus genotype. BMC Genomics. n. 14, p. 247, 2013.
MAHALIK, S., SHARMA, A. K., MUKHERJEE, K.J. . Genome engineering for improved recombinant protein expression in Escherichia coli. Microb Cell Fact. v. 13, n. 1, p. 177,
2014
MANN, D. G., KING, Z. R., LIU, W., JOYCE, B. L., PERCIFIELD, R. J., HAWKINS, J. S., LAFAYETTE, P. R., ARTELT, B. J., BURRIS, J. N., MAZAREI, M., BENNETZEN, J. L., PARROTT, W. A., STEWART, C. N. JR. Switchgrass (Panicum virgatum L.) polyubiquitin
gene (PvUbi1 and PvUbi2) promoters for use in plant transformation. BMC Biotechnol. v.
11, n. 11, p. 74, 2011
MARUYAMA, K., TODAKA, D., MIZOI, J., YOSHIDA, T., KIDOKORO, S., MATSUKURA, S., TAKASAKI, H., SAKURAI, T., YAMAMOTO, Y.Y., YOSHIWARA, K., KOJIMA, M., SAKAKIBARA, H., SHINOZAKI, K., YAMAGUCHI-SHINOZAKI, K. Identification of cis-acting promoter elements in cold- and dehydration-induced transcriptional pathways in Arabidopsis, rice, and soybean. DNA Res. n. 3749, 2012
MIRANDA, V. D. J. Caracterização da expressão do gene codificador da enzima de conjugação a ubiquitina (E2) em soja inoculada com Meloidogyne incognita e infestada com
Anticarsia gemmatalis. 2011. (Mestrado). Departamento de Biologia Celular, Universidade
de Brasília
MOABBI, A. M., AGARWAL, N., EL KADERI, B., ANSARI, A. Role for gene looping in intron-mediated enhancement of transcription, Proceedings of the National Academyof Sciences of the United States of America. n. 109, 2012
MURATANI M, T. W. How the ubiquitin-proteasome system controls transcription. Nat Rev Mol Cell Biol. n. 4, p. 192-201, 2003.
ODELL, J. T.; NAGY, F.; CHUA, N. H. Identification of DNA sequences required for activity of the cauliflower mosaic virus 35S promoter. Nature. n. 313, p. 810-812, 1985.
PARRA, G., BRADNAM, K., ROSE, A.B., KORF, I. Comparative and functional analysisof intron-mediated enhancement signals reveals conserved features amongplants. Nucleic Acids Research. n. 39, 2011
PONNALA L, W. Y., SUN Q, VAN WIJK, K.J. Correlation of mRNA and protein abundance in the developing maize leaf. Plant J. n. 3, p. 424-440, 2014.
RAWAT, P., SINGH, AK, CHAUDHARY, B, KUMAR, S, GAUTAM, T, KANORIA, S, KAUR, G, KUMAR, P, PENTAL, D, BURMA, PK. Detrimental effect of expression of Bt endotoxin Cry1Ac on in vitro regeneration, in vivo growth and development of tobacco and cotton transgenics. Journal of Biosciences. n. 36, p. 363-376, 2011.
ROLLFINKE IK, S. M., PFITZNER UM. Characterization and expression of a heptaubiquitin gene from tomato. Genes & Development. n. 211, p. 267-276, 1998.
RONG-XIANG FANG, F. N., SHANTHI SIVASUBRAMANIAM AND NAM-HAI CHUA Multiple cis Regulatory Elements for Maximal Expression of the Cauliflower Mosaic Virus 35S Promoter in Transgenic Plants The Plant Cell. n. 1, p. 141-150, 1989.
ROSE, A. B. Requirements for intron-mediated enhancement of gene expression in
Arabidopsis. RNA. n. 8, p. 1444-1453, 2002.
RUSSELL, J.D., SCALF, M., BOOK, A.J., LADROR, D.T., VIERSTRA, R.D., SMITH, L.M., COON, J.J. Characterization and quantification of intact 26S proteasome proteins by real-time measurement of intrinsic fluorescence prior to top-down mass spectrometry. PLoS ONE, 2013
SAFRANY J, H. V., MATE Z, CIOLFI A, FEHER B, ORAVECZ A, STEC A, DALLMANN G, MORELLI G, ULM R, NAGY F. Identification of a novel cis-regulatory element for UV-B-induced transcription in Arabidopsis. Plant J. n. 54, p. 402-414, 2008.
SAMADDER P, S. E., LU J, LI X, QU R. Transcriptional and post-transcriptional enhancement of gene expression by the 5' UTR intron of rice rubi3 gene in transgenic rice cells. Mol Genet Genomics. n. 279, p. 429–439, 2008.
SCHENK, P., REMANS, T, SAGI, L, ELLIOTT, AR, DIETZGEN, RG, SWENNEN, R, EBERT, PR, GROF, CPL, MANNERS, JM. Promoters for pregenomic RNA of banana streak badnavirus are active for transgene expression in monocot and dicot plants. Plant Mol. Biol.
n. 47, p. 399-412, 2001.
STEPHEN, T., JAMES T. The RNA Polymerase II Core promoter. Annu. Rev. Biochem. n.
72, p. 449-479, 2003.
SUN L, N. S. Analysis of promoter activity of members of the PECTATE LYASE-LIKE (PLL) gene family in cell separation in Arabidopsis. BMC Plant Biol. n. 1, 2010.
TOBIAS MAIER, M. G., LUIS SERRANO. Correlation of mRNA and protein in complex biological samples. FEBS Letters. n. 583, p. 3966-3973, 2009.
2676-2691, 2012.
VAN DER GEEST, A., HALL, TC. The β-phaseolin 5' matrix attachment region acts as an
enhancer facilitator. Plant Mol. Biol. n. 33, p. 553-557, 1997.
VÉLEZ-BERMÚDEZ, I.C., SCHMIDT, W. The conundrum of discordant protein and mRNA expression. Are plants special? Front Plant Sci., 2014.
VENTER, M. Synthetic promoters: genetic control through cis engineering. Trends Plant Sci. n. 12, p. 118-24, 2007.
VIANA, A. A., FRAGOSO, R. R., GUIMARÃES, L. M., PONTES, N., OLIVEIRA-NETO, O. B., ARTICO, S., NARDELI, S. M., ALVES-FERREIRA, M., BATISTA, J. A. N., SILVA, M. C. M., GROSSI DE SA, M. F. Isolation and functional characterization of a cotton ubiquitination-related promoter and 5'UTR that drives high levels of expression in root and flower tissues. BMC Biotechnol. n. 11, p. 115, 2011.
W.C. CHANG, T. Y. L., H.D. HUANG, H.Y. HUANG AND R.L. PAN. PlantPAN: Plant Promoter Analysis Navigator, for identifying combinatorial cis-regulatory elements with distance constraint in plant gene group. BMC Genomics. n. 9, 2008.
WALLEY, J. W., SHEN, Z., SARTOR, R., WU, K. J., OSBORN, J., SMITH, L. G., ET AL. . Reconstruction of protein networks from an atlas of maize seed proteotypes. Proc. Natl. Acad. Sci. n. 110, 2013.
WANG B, S. Y., SONG N, WANG XJ, FENG H, HUANG LL, KANG ZS. Identification of UV-B-induced microRNAs in wheat. Genet Mol Res. n. 4, p. 4213-4221, 2013.
WANG J, O. J. Rice ubiquitin promoters: deletion analysis and potential usefulness in plant transformation systems. Plant Cell Rep. n. 22, p. 129-134, 2003.
WEI HR, W. M., MORE PH, ALBERT HH. Comparative expression analysis of two sugarcane polyubiquitin promoters and flanking sequences in transgenic plants. J Plant Physiol. n. 160, p. 1241-1251, 2003.
WEI, X., ZHANG, X, SHEN, D, WANG, H, WU, Q, LU, P, QIU, Y, SONG, J, ZHANG, Y, LI, X. Transcriptome Analysis of Barbarea vulgaris Infested with Diamondback Moth (Plutella
xylostella) Larvae. PlosOne. n. 8, 2013.
WHALLEY, H.J., SARGEANT, A.W., STEELE, J.F., LACOERE, T., LAMB, R., SAUNDERS, N.J., KNIGHT, H., KNIGHT, M.R. Transcriptomic analysis reveals calcium regulation of specific promoter motifs in Arabidopsis. Plant Cell. n. 11, p. 4079-95, 2011
WITTKOPP, P., KALAY, G. Cis-regulatory elements: molecular mechanisms and evolutionary processes underlying divergence. Nature Review Genetics. n. 13, p. 59-69,
2012.
analysis of light-regulated alternative splicing mediated by photoreceptors in Physcomitrella
patens. Genome Biol. n. 15, 2014.
XU, L., ZHU, L, TU, L, LIU, L, YUAN, D, JIN, L, LONG, L, ZHANG, X. Lignin metabolism has a central role in the resistance of cotton to the wilt fungus Verticillium dahliae as revealed
by RNA-Seq-dependent transcriptional analysis and histochemistry. Journal of Experimental Botany. n. 15, p. 5607-5621, 2011.
YAMAGUCHI-SHINOZAKI, K., SHINOZAKI, K. novel cis-acting element in a Arabidopsis
gene is involved in responsiveness to drought, low-temperature, or highsalinity stress. Plant Cell. n. 6, p. 251-264, 1994.
YU, H., LI, YH, WU, KM. Risk Assessment and Ecological Effects of Transgenic Bacillus
thuringiensis Crops on Non-Target Organisms. Journal of Integrative Plant Biology,. n. 53,
p. 520-538, 2011.
ZHAO Y, S. S., LI X, ZHAI Y, ZHANGQ, QIAN D,WANG Q. Isolation and activity analysis of a seed-abundant soyAP1 gene promoter from soybean. Plant Mol Biol Rep. n. 30, p.
1400-1407, 2012.
ZOU, C., SUN, K., MACKALUSO, J.D., SEDDON, A.E., JIN, R. THOMASHOW, M.F., SHIU, S.H. Cis-regulatory code of stress-responsive transcription in Arabidopsis thaliana.
Proc Natl Acad Sci USA. v. 108, n. 36, p. 14992-14997, 2011