U
NIVERSIDADE DE
L
ISBOA
Faculdade de Ciências
Departamento de Informática
APLICAÇÕES COM BASE EM ONTOLOGIAS
BIOMÉDICAS: UMA ABORDAGEM WEB
SEMÂNTICA
Bruno Filipe Gomes Feteira da Costa Tavares
Mestrado em Tecnologias da Informação Aplicadas as
Ciências Biológicas e Médicas
U
NIVERSIDADE DE
L
ISBOA
Faculdade de Ciências
Departamento de Informática
APLICAÇÕES COM BASE EM ONTOLOGIAS
BIOMÉDICAS: UMA ABORDAGEM WEB
SEMÂNTICA
Bruno Filipe Gomes Feteira da Costa Tavares
Trabalho orientado pelo Prof. Doutor Francisco José Moreira Couto
Mestrado em Tecnologias da Informação Aplicadas as
Ciências Biológicas e Médicas
Agradecimentos
Em primeiro lugar quero agradecer à minha mãe por me aturar e pelos sacrifícios que faz. Ao meu irmão que me ajudou a crescer. Aos meus primos e amigos pelas palhaçadas todas. À Cláudia por ter estado sempre ao meu lado.
Ao Professor António Ferreira pelas minhas primeiras aulas de programação, o que me levou a seguir este caminho. Ao Professor Francisco Couto que me trouxe para este curso e me tem ajudado neste projecto, bem como os conselhos que sempre me foi dando. Aos meus colegas de laboratório que me ajudaram e ensinaram. Quero também agradecer ao grupo LaSIGE pela bolsa que me ofereceu, tornando possível a realização deste projecto.
Dedicatória:
i
Resumo
Este relatório apresenta o trabalho centrado na construção da BOA (Biomedical Ontology Applications) framework. A actividade de investigação BOA do grupo XLDB do LASIGE visa investigar e desenvolver aplicações para eficientemente explorar a informação contida nas ontologias biomédicas, por exemplo Gene Ontology (GO) e Chemical Entities of Biological Interest (ChEBI).
A actividade de investigação BOA disponibilizava três ferramentas web para a comunidade: ProteInOn, CMPSim e CESSM. O objectivo desta tese foi a integração destas ferramentas numa adordagem web semântica, com base numa framework composta por: i) back-end, onde os scripts correm, ii) mediador, um conjunto de serviços web que permite a comunicação entre os diferentes componentes da framework e iii) front-end, páginas web que permitem interagir e visualizar os resultados das ferramentas.
Os serviços web foram desenvolvidos por forma a poderem ser utilizados internamente, por ferramentas criadas pelo grupo ou pelas páginas web das ferramentas, e externamente, de uma forma directa por utilizadores ou por integração noutras ferramentas ou páginas web.
É de salientar que foram implementadas duas arquitecturas de serviços web de modo a cumprir todos os objectivos propostos. Uma arquitectura RESTful, facilitando assim a utilizadores sem conhecimentos de programação, e uma arquitectura RPC, que supera as limitações encontradas na arquitectura RESTful.
Palavras Chave:
iii
Abstract
This report presents the work focused on building a BOA (Biomedical Ontology Applications) framework. The BOA research activity of XLDB group of LASIGE aims to investigate and develop applications to efficiently exploit the information contained in biomedical ontologies, such as Gene Ontology (GO) and Chemical Entities of Biological Interest (ChEBI).
BOA offers three web tools for the community: ProteInOn, and CMPSim CESSM. The aim of this thesis was to integrate these tools into a semantic web approach, based on a framework consists of: i) back-end, where the scripts run, ii) a mediator, a set of web services that allows communication between the different components of the framework and iii) front-end, web pages that allow to interact and view the results of the tools.
The web services were developed so that they can be used internally, by tools created by the group or the web pages of tools, and externally, in a direct manner by users or integration into other tools or web pages.
It is noteworthy that two web services architectures were implemented in order to fulfill all objectives. A RESTful architecture, making it easier for users without programming skills, and an RPC architecture that overcomes the limitations found in the RESTful architecture.
Keywords:
v
Índice
Lista de Figuras ... vii
Lista de Tabelas ... viii
Capítulo 1 Introdução ... 1
1.1 Contextualização ... 1
1.2 Motivação ... 2
1.3 BOA ... 3
1.3.1 BOA Framework Inicial ... 3
1.4 Objectivos ... 4
1.5 Metodologia ... 5
1.6 Planeamento ... 7
1.7 Contribuições ... 8
1.8 Organização do Documento ... 8
Capítulo 2 Trabalho Relacionado ... 11
2.1 Arquitecturas de Serviços Web ... 11
2.1.1 RESTful ... 12
2.1.2 RPC ... 12
2.1.3 REST-RPC hybrid ... 13
2.1.4 Vantagens e Desvantagens ... 14
Capítulo 3 BOA Framework ... 17
3.1 Serviços Web ... 19
3.1.1 RESTful ... 19
3.1.2 RPC ... 20
3.2 Back-End ... 22
vi
4.1 RESTful ... 23
4.2 RPC ... 25
4.3 Parâmetros ... 26
4.4 Resultados ... 27
Capítulo 5 Back-End e Front-End ... 37
5.1 Back-End ... 37 5.1.1 ProteInOn ... 37 5.1.2 CMPSim ... 37 5.1.3 Guia ... 38 5.2 Front-End ... 38 5.2.1 ProteInOn ... 39 5.2.2 CMPSim ... 39 5.2.3 Exemplos ... 39 Capítulo 6 Conclusão ... 43 6.1 Framework do BOA ... 43 6.1.1 Mediador ... 43 6.1.2 Back-End ... 44 6.1.3 Front-End ... 44 6.2 Planeamento ... 45 6.3 Trabalho Futuro ... 45 Capítulo 7 Bibliografia ... 47
vii
Lista de Figuras
Figura 1 – Diagrama inicial da BOA Framework. ... 3
Figura 2 – Exemplo de um pedido RPC do site ... 13
Figura 3 – Arquitectura da BOA Framework proposta. ... 17
Figura 4 – Estrutura base do envelope SOAP para o ... 21
Figura 5 – Exemplo do envelope SOAP para o cálculo ... 22
Figura 6 – Estrutura base do envelope SOAP para o ... 25
Figura 7 – Exemplo do envelope SOAP para o cálculo ... 26
Figura 8 – Exemplo dos resultados base para o cálculo ... 28
Figura 9 – Exemplo dos resultados base para o cálculo ... 29
Figura 10 – Exemplo dos resultados base para o cálculo... 30
Figura 11 – Exemplo dos resultados base para o cálculo... 30
Figura 12 – Exemplo dos resultados base para o cálculo... 30
Figura 13 – Exemplo dos resultados base para o cálculo... 31
Figura 14 – Exemplo dos resultados base para o cálculo... 31
Figura 15 – Exemplo dos resultados base para o cálculo... 31
Figura 16 – Exemplo dos resultados base para o cálculo... 31
Figura 17 – Exemplo dos resultados base para o cálculo... 31
Figura 18 – Exemplo dos resultados base para o cálculo... 31
Figura 19 – Exemplo dos resultados base para o cálculo... 32
Figura 20 – Resultados para o cálculo da SSM. ... 32
Figura 21 – Resultados para o cálculo da Characterization ... 35
Figura 22 – Exemplo de um pedido XMLHTTPResquest ... 40
viii
Lista de Tabelas
Tabela 1 – Indicação das tarefas e início e fim da sua execução. ... 7
Tabela 2 – RESTful API para as ferramentas existentes. ... 23
Tabela 3 – Valor de <query> e os respectivos parâmetros ... 25
Tabela 4 – Valor de <query> e os respectivos parâmetros ... 25
Tabela 5 – Condições para a utilização de cada um... 27
1
Capítulo 1 Introdução
Devido às técnicas da era da genómica houve um aumento da informação disponível na área das ciências biomédicas, como se pode constatar pelo número de sequências de proteínas e informações funcionais no UniProt1 que crescem exponencialmente2. Com o aumento da informação é necessário recorrer a sistemas computacionais para ajudar na análise e estruturação dessa informação. Desta forma, foram criadas diversas bases de dados e ontologias nesta área, tais como o GO3 (Gene Ontology), que tem como objectivo padronizar a descrição funcional de genes e produtos de genes; o ChEBI4 (Chemical Entities of Biological Interest) uma ontologia de compostos químicos com interesse biológico; e o CAZy5 (Carbohydrate-Active enZYmes) uma base de dados de enzimas activas em carbohidratos.
1.1 Contextualização
Neste contexto foi criada uma nova linha de investigação do LASIGE (Large-Scale Informatic Systems Laboratory), Informática Biomédica, onde a equipa do XLDB está empenhada em desenvolver ferramentas web sob a actividade de investigação BOA
1 http://www.uniprot.org/ 2 http://www.ebi.ac.uk/uniprot/TrEMBLstats/ 3 http://www.geneontology.org/ 4 http://www.ebi.ac.uk/chebi/ 5 http://www.cazy.org/
2
(Biomedical Ontology Applications). BOA visa investigar e desenvolver aplicações para eficientemente explorar a informação contida nas ontologias biomédicas.
No início deste trabalho, BOA disponibilizava três ferramentas web para a comunidade: i) ProteInOn: uma ferramenta que pode ser usada para encontrar anotações GO de proteínas, calcular a semelhança semântica entre proteínas, obter o info content e calcular a semelhança semântica dos termos GO; ii) CESSM, utilizado para a avaliação automática de medidas de semelhança semântica com base no GO; iii) CMPSim fornece uma medida de semelhança funcional entre compostos químicos e vias metabólicas usando medidas de semelhança semântica baseadas no ChEBI. No entanto, BOA está também a desenvolver outras ferramentas de web, nomeadamente o GRYFUN, para a visualização, filtragem e análise de perfis de anotação funcional GO de famílias de proteínas.
1.2 Motivação
Apesar dos contributos para a comunidade científica destas ferramentas [1,2,3,4,5], estas não seguem uma abordagem de web semântica, não tirando partido das tecnologias de serviços web ou AJAX (Asynchronous Javascript And XML).
A criação de uma BOA Framework com o recurso a serviços web iria fornecer à comunidade um modo de integrar de uma forma automática as nossas ferramentas e as suas funcionalidades nos seus projectos ou páginas. Uma abordagem baseada na web semântica permitiria que a actividade BOA tivesse uma melhor interligação com outros sistemas nesta área, nomeadamente o Linked Data Project6. Por outro lado, com o AJAX é possível criar páginas web mais dinâmicas sem com isso aumentar a carga de
6
3
download para o utilizador, permite fazer pedidos ao servidor sem que se tenha de carregar a página por completo (através dos serviços web).
1.3 BOA
As várias ferramentas desenvolvidas no âmbito da actividade de investigação BOA são geridas pelos diversos investigadores da equipa. Cada um dos investigadores faz as suas ferramentas na linguagem que mais gosta ou sabe (Java, Python, PHP e Perl entre outras) o que torna a comunicação entre ferramentas mais complexa, dificultando assim a possibilidade de uma nova ferramenta utilizar uma funcionalidade de uma ferramenta já existente.
1.3.1 BOA Framework Inicial
No início deste trabalho, o BOA Framework tinha a arquitectura apresentada na Figura 1.
Figura 1 – Diagrama inicial da BOA Framework
Front-End
Internet
Serviços Web Externos, Sistemas de Informação e base de dados (UniProt, Gene Ontology, KEGG, etc.)
Back-End Base de dados local Página web ProteInOn ProteInOn script Update BD CMPSim script Nova Ferramenta script Página web CMPSim Página web N.F.
4
A framework era composta pela i) base de dados, que contem a GO, GOA (Gene Ontology Annotation), ChEBI e KEGG7 (Kyoto Encyclopedia of Genes and Genomes), ii) scripts de actualização da base de dados local, que comunica com as bases de dados das ontologias (GO por exemplo) obtendo as versões mais actuais destas e actualizando a nossa base de dados local com essa informação, iii) os diferentes scripts das ferramentas, recebem uma lista de identificadores (de proteínas, termos GO, compostos químicos ou vias metabólicas) e um conjunto de parâmetros, comunicando depois com a base de dados para calcular a semelhança semântica ou fazer uma caracterização dessa lista, enviando os resultados para a página web, num formato HTML. Esta framework revelava diversos problemas, nomeadamente, i) a dificuldade da interacção entre as diversas ferramentas, já que as diferentes ferramentas estão implementadas em diferentes linguagens, ii) a impossibilidade de terceiros poderem integrar as nossas ferramentas nas suas páginas web ou nas suas ferramentas de uma forma automática, a única possibilidade para esta integração é através do scrapping das páginas web de cada ferramenta; por último iii) a dificuldade de actualizar as ferramentas e as respectivas páginas web, devido à ligação directa entre as ferramentas e as páginas web, a actualização dos scripts ou da página web frequentemente gerava incompatibilidades.
1.4 Objectivos
O principal objectivo deste trabalho é o desenvolvimento de uma nova framework, que com recurso a tecnologias de web semântica e web 2.0 possam minimizar os problemas atrás indicados. Neste sentido este trabalho irá criar serviços web, que irão
7
5
servir de mediador entre as nossas ferramentas e as respectivas páginas web, permitido ainda a comunicação entre uma nova ferramenta e as já existentes, bem como a possibilidade de integração das nossas ferramentas com outras ferramentas ou páginas web externas.
Como objectivos secundários neste trabalho serão feitas modificações nas actuais ferramentas BOA de modo a poderem disponibilizar os resultados em diferentes formatos (XML, JSON, TSV e CSV) e, serão actualizadas as páginas web já criadas para as diferentes ferramentas de modo a incorporar estes serviços web com base na tecnologia AJAX.
1.5 Metodologia
De modo a cumprir os objectivos propostos irei implementar uma arquitectura de serviços web RESTful. A arquitectura RESTful é uma arquitectura orientada para os recursos (ROA), em que a informação necessária ao pedido é enviada no URL. Foram identificados os diferentes recursos, sendo escolhidos um conjunto de conceitos de modo a poder representa-los:
ontology – Tal como foi descrito anteriormente, as nossas ferramentas têm como objectivo explorar ontologias médicas, podendo, neste momento, serem escolhidas as ontologias GO ou ChEBI.
concept ou entity – Nestas ontologias existem dois tipos de elementos, conceitos e entidades. Os conceitos são definidos a partir de um vocabulário controlado. Os conceitos são usados para descrever (anotar) as entidades, no caso do GO os conceitos são os termos (term) e as entidades são as proteínas (protein), no caso do ChEBI os conceitos são os compostos químicos (compound) e as entidades são as vias metabólicas
6
(pathways). Uma anotação é, nesta tese, considerada como um par conceito x entidade.
inputList – uma lista de identificadores separada por vírgulas, consoante as escolhas feitas anteriormente, podemos ter: Q13263, P35222 (para GO/protein); GO:0050681, GO:0030331 (para GO/term); 15377, C00011 (para ChEBI/compound) e map00910, map00473 (para ChEBI/pathway). feature – Consoante a lista de identificadores e as escolhas feitas
anteriormente é possível fazer dois tipos de operações, calcular a semelhança semântica entre os diversos conceitos ou entidades, ou caracterizar (Characterization) o conjunto de entidades (GO/protein ou ChEBI/pathway).
parameter – Consoante as escolhas feitas anteriormente existem três parâmetros possíveis:
o measure – para calcular a semelhança semântica é possível utilizar diferentes medidas, neste momento encontram-se implementadas as seguintes: para ambas as ontologias, pode-se utilizar simGIC ou simUI; para a ontologia GO ainda se podem utilizar as medidas ResnikDCA, Resnik, LinDCA, Lin, JiangConrathDCA ou JiangConrath.
o goType – A ontologia GO está dividida em três sub-ontologias (aspects) sendo assim, no caso da GO/protein é possível escolher em que aspecto queremos trabalhar, tendo como argumento: molecular_function, biological_process ou cellular_component. o IEA – Nas ontologias algumas das anotações são geradas por
7
forma, para a ontologia GO, podemos optar pela utilização de tais anotações, tendo como argumento: true ou false.
1.6 Planeamento
De modo a cumprir os objectos propostos, irei seguir o seguinte o planeamento apresentado na Tabela 1. Este planeamento é composto por sete tarefas, duas de escrita (tarefas 1 e 7), duas de testes e integração (tarefas 3 e 5) e uma para cada parte da framework, serviços web (tarefa 2), back-end (tarefa 4) e front-end (tarefa 5).
Tabela 1 – Indicação das tarefas e início e fim da sua execução
Tarefa Início Fim
1 – Escrita relatório preliminar 1 Setembro 31 Outubro
2 – Serviços web 1 Novembro 31 Dezembro
3 – Teste e integração 15 Dezembro 15 Janeiro
4 – Back-end 1 Janeiro 28 Fevereiro
5 – Teste e integração 15 Fevereiro 15 Março
6 – Front-end 1 Março 30 Abril
7 – Escrita tese 1 Junho 1 Julho
A tarefa 1 corresponde a escrita do relatório preliminar e leitura de bibliografia, com a duração de dois meses. A tarefa 2 é a implementação dos serviços web, também com duração de dois meses. A tarefa 3 vai servir de teste dos serviços criados anteriormente e integração destes com o back-end, parte desta tarefa será feita ao mesmo tempo da execução das tarefas 2 e 4, e tem uma duração de quatro semanas. A tarefa 4 corresponde a modificação do back-end tendo a duração de dois meses. A tarefa 5 vai verificar novamente os serviços web e o back-end juntamente com a integração destes no front-end, tal como na tarefa 3, parte da tarefa será executada ao mesmo tempo da execução das tarefas 4 e 6, tendo uma duração de quatro semanas. A tarefa 6 corresponde às modificações no front-end e tem a duração de dois meses. A tarefa 7 é a escrita da tese e tem duração de 3 meses. Este projecto tem assim a duração de doze meses. De salientar que não está planeada uma tarefa de testes e verificações depois da tarefa 6 (front-end) pois durante o processo qualquer alteração é imediatamente visionada, e no caso do ProteInOn parte das alterações necessárias já foram executadas.
8
1.7 Contribuições
Este trabalho contribuiu com uma nova BOA framework baseada numa abordagem web semântica. Esta framework foi descrita em duas publicações científicas:
Hugo Bastos, Bruno Tavares, Cátia Pesquita, Daniel Faria, Francisco Couto: Application of Gene Ontology to Gene Identification. In Silico Tools for Gene Discovery: Methods in Molecular Biology. Springer, 2011.
Bruno Tavares, Hugo Bastos, Daniel Faria, João Ferreira, Tiago Grego, Cátia Pesquita and Francisco Couto. The Biomedical Ontology Applications (BOA) framework. Software Demonstration. International Conference on Biomedical Ontology, USA, 2011.
1.8 Organização do Documento
Este documento está organizado da seguinte forma:
Capítulo 2 – Trabalho Relacionado
Neste capítulo são apresentadas as tecnologias usadas e os trabalhos relacionados.
Capítulo 3 – BOA Framework
Neste capítulo são apresentadas todas as arquitecturas escolhidas para este trabalho de modo a cumprir os objectivos propostos.
9 Capítulo 4 – Mediador
Neste capítulo aborda-se em detalhe a implementação dos serviços web, nomeadamente as arquitecturas seleccionadas, RESTful e RPC. São também discutidos os URI’s base e todas as estratégias escolhidas.
Capítulo 5 – Back-End e Front-End
Neste capítulo são apresentadas as modificações feitas nos Scripts das ferramentas. Também é apresentado um tutorial para a criação de futuras ferramentas e posterior integração na BOA. São também apresentadas as modificações feitas nas páginas das ferramentas. Sendo ainda apresentados exemplos do código em Javascript utilizado, para integrar os serviços web numa página web.
Capítulo 6 – Conclusão
Neste último capítulo é discutido o planeamento e o cumprimento dos objectivos propostos. É também discutido resumidamente as principais decisões de implementação tomadas e é também apresentado trabalho futuro no âmbito do BOA.
11
Capítulo 2 Trabalho Relacionado
Este capítulo apresenta três arquitecturas de Serviços web, RESTful, RPC e REST-RPC hybrid, e as respectivas vantagens e desvantagens de cada uma delas. O objectivo é dar a entender as escolhas feitas neste trabalho e que irão ser detalhadas no capítulo seguinte.
2.1 Arquitecturas de Serviços Web
Os serviços web seguem três passos [6]: 1) recolher toda a informação necessária para fazer o pedido HTTP; 2) formatar essa informação e enviar para o respectivo servidor; 3) processar a resposta. Estes passos podem ser implementados através de três diferentes Arquitecturas de serviços web, para tal, basta verificar de que modo é informado ao servidor “o que se quer fazer” (method) e “onde se quer fazer” (scoping information) no pedido HTTP.
A arquitectura RESTful (Representational State Transfer) utiliza os métodos HTTP (GET, POST, etc...) como método do pedido (“o que se quer fazer”), enquanto o scoping information (“onde se quer fazer”) é enviado no URI. Na arquitectura RPC (Remote Procedure Call) envia ambos (método e scoping information) no corpo da mensagem do pedido HTTP. A arquitectura REST-RPC hybrid, normalmente confundida com a arquitectura RESTful, envia ambos (método e scoping information) no URI.
12
2.1.1 RESTful
A arquitectura RESTful é uma arquitectura orientada para os recursos (ROA) que segue quatro características principais: Endereçamento (Addressability), sem estado (Stateless), representação (Representation) e uma interface uniforme (Uniform Interface). Um dos princípios da arquitectura RESTful é que a informação que se quer disponibilizar para os utilizadores esteja dividida em recursos (daí ser uma ROA) e que seja possível ter uma, ou mais, representações de cada recurso, a isto chama-se Representation. Cada recurso vai ter pelo menos um indentificador, isto é conseguido devido ao facto do scoping information ir no URI, tendo assim um URI para cada recurso (Addressability). Ser Stateless significa que toda a informação necessária a cada pedido vai toda nesse pedido, o servidor não tem de guardar nenhum tipo de informação sobre pedidos anteriores, esta é uma das características mais fáceis de implementar, visto o protocolo HTTP ser ele próprio Stateless. Embora seja possível criar um serviço web com estado (State), mas este não será RESTful. Como a arquitectura RESTful utiliza os métodos HTTP, diz-se que tem uma Uniform Interface, pois são métodos padrão e sabe-se o que estes devem fazer.
Um bom exemplo de uma arquitectura RESTful é o sistema de pesquisa do Google, caso se queira procurar por bioinformática basta escrever o seguinte URI:
http://www.google.pt/search?q=bioinformatica.
2.1.2 RPC
Uma arquitectura RPC é uma arquitectura orientada para os serviços (SOA) que expõe um URI (endpoint) para cada processo capaz de lidar com chamadas de procedimento remoto [6]. Nesta arquitectura tanto a scoping information como o
13
method vão no corpo da mensagem do pedido HTTP. Pode ser usado qualquer tipo de formato, mas é usual a utilização de XML, ou uma especificação de XML chamada envelope SOAP ou mesmo texto simples. É muitas vezes confundida a utilização de envelope SOAP como arquitectura SOAP, este conceito não existe. A chamada do serviço web continua a ser um pedido HTTP, dentro do envelope HTTP, encontra-se um envelope SOAP que contem o método e a scoping information.
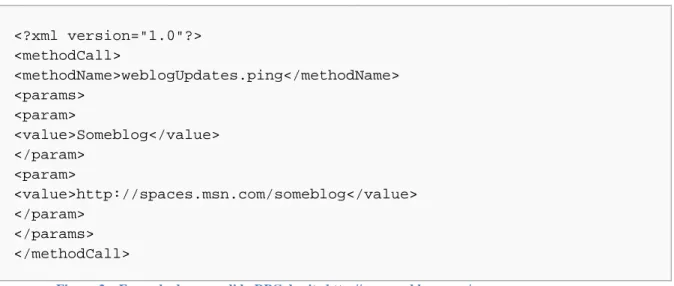
O site http://www.weblogs.com/ é um exemplo onde existe uma implementação RPC, tendo o seguinte endpoint: http://rpc.weblogs.com/RPC2. O Weblogs também tem uma implementação RESTful. Na Figura 2 está um exemplo de um pedido XML deste serviço. <?xml version="1.0"?> <methodCall> <methodName>weblogUpdates.ping</methodName> <params> <param> <value>Someblog</value> </param> <param> <value>http://spaces.msn.com/someblog</value> </param> </params> </methodCall>
Figura 2 – Exemplo de um pedido RPC do site http://www.weblogs.com/
2.1.3 REST-RPC hybrid
No caso da arquitectura REST-RPC hydrib o scoping information vai no URI, tal como na arquitectura RESTful, mas ao contrário desta, não são utilizados os métodos HTTP sendo então especificados, normalmente no URI. Para melhor se perceber esta arquitectura vai ser mostrado um exemplo, “REST” API do flickr. Neste exemplo toda a informação é enviada no URI:
14
http://www.flickr.com/services/rest?api_key=xxx&method=flickr.photos.search&tags= penguin, e pode ser interpretado como “search photos with etiqueta penguin”. Assim parece que é uma arquitectura RESTful, onde temos a scoping information no URI e é utilizado o método HTTP, GET. Mas isto seria verdade caso fosse um serviço web apenas de leitura (read-only), o que não é o caso. Para apagar uma fotografia seria este URI:
http://www.flickr.com/services/rest?api_key=xxx&method=flickr.photos.delete&tags=p
enguin, que tem como scoping information “photos with etiqueta penguin”, mas o
método “deixou” de ser o método HTTP, que continua a ser o método GET, passando a ser “flickr.photos.delete”.
2.1.4 Vantagens e Desvantagens
As diferenças entre estas três arquitecturas, faz com que cada uma tenha as suas vantagens e desvantagens. As arquitecturas RESTful e REST-RPC hybird são as mais fáceis de utilizar devido ao facto de ambas terem a característica de Addressability, basta para isso, escrever um URI num browser e aceder assim à representação desse recurso. Mas no caso da arquitectura RESTful isso só é verdade na utilização do método HTTP GET (no caso de serviços web read-only não existe uma diferença prática entre uma arquitectura RESTful e REST-RPC hybrid), pois é impossível modificar o método HTTP no browser, ao contrário da arquitectura REST-RPC hybrid, em que o método também vai no URI. Esta vantagem da arquitectura REST-RPC hybrid gera também uma desvantagem, a não utilização de uma Uniform Interface, deixando assim de ser Safety e Idempotence. Mas a Addressability tem limitações, embora alargadas, por exemplo o Microsoft Internet Explorer não consegue manipular URI com mais de 2083
15
caracteres, enquanto o Apache não consegue responder a pedidos com URI maiores do que 8Kb [6]. Tendo assim a arquitectura RPC vantagem, pois não tem qualquer tipo de limite, devido ao facto de só se usar um URI endpoint e toda a informação ir no corpo da mensagem do pedido HTTP.
Devido ao facto de esta framework não ter um sistema de registo de utilizadores, nem a informação utilizada nos serviços web ser de algum modo confidencial, a segurança destas arquitecturas não foram objecto de estudo no âmbito desta tese.
17
Capítulo 3 BOA Framework
De modo a resolver os problemas da BOA framework inicial, i) a dificuldade da interacção entre as diversas ferramentas, ii) a impossibilidade de terceiros poderem integrar as nossas ferramentas nas suas páginas web ou nas suas ferramentas de uma forma automática e iii) a dificuldade de actualizar as ferramentas e as respectivas páginas web, foi proposto uma nova arquitectura para o BOA Framework, apresentada no diagrama da Figura 3.
Figura 3 – Arquitectura da BOA Framework proposta
Nesta nova arquitectura a comunicação entre as páginas (Front-End) e os scripts (Back-End) é feita através de um novo componente, o Mediador. Os serviços web do Mediador facilitam a comunicação entre as diferentes ferramentas, e caso uma nova
Front-End
Internet
Serviços Web Externos, Sistemas de Informação e Base-de-Dados (UniProt, Gene Ontology, KEGG, etc.)
Back-End Página ProteInOn Aplicações Externas Mediador Base-de-Dados Serviços Web ProteInOn Página CMPSim Página N.F. Update BD CMPSim Nova Ferramenta
18
ferramenta necessite de utilizar uma funcionalidade de uma das ferramentas existentes, apenas tem de fazer uma chamada ao respectivo serviço web. Do mesmo modo, os serviços web podem também ser utilizados por terceiros para integrar as funcionalidades das nossas ferramentas nas suas páginas ou sistemas. Esta nova arquitectura facilita também a actualização dos diversos componentes, por exemplo, é possível actualizar o ProteInOn de modo a que este disponibilize o link de determinada proteína para o GO, sem que com isso tenha de ser alterado a página do ProteInOn. Apesar de que isto não implique que não se possa depois actualizar a página de modo a esta mostrar essa nova informação.
Os resultados enviados pelos serviços web podem ser obtidos em vários formatos: XML, JSON, CSV e TSV. Foram escolhidos estes formatos por diversas razões: i) o formato XML permite uma eficiente integração com outros sistemas, nomeadamente o Linked Data Project; ii) o formato JSON foi escolhido por ser um formato que é directamente interpretado por linguagens de programação, nomeadamente Javascript e Python, permitindo assim uma eficiente integração dos serviços web em páginas web e scripts; iii) os formatos Comma-separated values (CSV) e Tab-Separated Values (TSV) são formatos facilmente interpretados por humanos, sendo possíveis de abrir em editores de texto, como por exemplo notepad, e em folhas de cálculo, como o excel. Este ponto é relevante no projecto porque alguns dos nossos utilizadores estão habituados apenas a este tipo de programas e formatos.
É ainda possível ao utilizador pedir informação extra. Por exemplo neste momento é possível incluir os nomes dos termos ou compostos para além dos seus identificadores para a Characterization. Esta possibilidade foi implementada de modo a poder conter outras informações extra. Assim, se houver uma actualização das ferramentas, disponibilizando novas informações, estas podem ser acedidas pelos
19
utilizadores, mas sem obrigar a actualizações nas ferramentas que não necessitam dessa nova informação.
3.1 Serviços Web
Da análise feita aos diferentes tipos de arquitecturas e das respectivas vantagens e desvantagens, foi decidido implementar duas arquitecturas, RESTful e RPC, ao contrário do que tinha sido proposto inicialmente (apenas a implementação da arquitectura RESTful). A arquitectura RESTful foi escolhida devido ao facto de para ser utilizada apenas é preciso escrever um URI, e este ponto é importante pois alguns dos nossos utilizadores não têm conhecimentos de programação. Mas esta facilidade de utilização acarreta uma limitação no tamanho do input. As nossas ferramentas podem receber até mil proteínas como entrada, sendo que cada proteína é representada pelo seu ACC (conjunto de 6 caracteres), podendo assim o URI atingir mais de 6000 caracteres. Por esse facto foi então decidido complementar a implementação dos serviços web com uma arquitectura RPC. Como os nossos serviços web são só read-only não iríamos ter vantagens em implementar uma arquitectura REST-RPC hybrid, em detrimento da arquitectura RESTful.
3.1.1 RESTful
Tal como foi dito anteriormente, a escolha de implementar a arquitectura RESTful deve-se ao facto de não ser necessário conhecimentos informáticos para escrever um URI e começar a utilizar o serviço web. De modo a potenciar esta facilidade de
20
utilização, foi pensada uma estrutura hierárquica para o URI base, como se pode verificar aqui:
/Ontology/concept|entity/inputList/feature?parameter1=param Value1&..., inicialmente é escolhida a ontologia em que se quer trabalhar, posteriormente é indicado se se trata dos conceitos dessa ontologia ou das entidades.
Depois é introduzido a lista de identificadores com a respectiva característica (SSM ou Characterization) que se deseja calcular, e os seus parâmetros, os parâmetros não se encontram separados por barras pois não têm uma hierarquia entre eles e estão relacionados directamente com a feature. Assim, se o utilizador quiser calcular a semelhança semântica entre os GO terms “estrogen receptor binding” (GO:0030331) e “androgen receptor binding” (GO:0050681) só teria de escrever /GO/term/GO:0030331,GO:0050681/SSM?measure=simUI&IEA=false.
De modo a separar a informação da escolha do recurso e da representação deste, a escolha do formato dos resultados e da informação extra (nomes dos termos ou composto na feature Characterization) é feita no header do pedido HTTP. Para a escolha do formato basta utilizar o campo Content-type, um campo normalmente utilizado neste sentido, enquanto para a informação extra é utilizado o campo include. A decisão desta separação foi tomada para manter o URI o mais simples e intuitivo possível, mantendo assim a sua facilidade de utilização. Também a pensar na facilidade de utilização foi escolhido para padrão de formato dos resultados, o formato TSV. Assim, caso não seja indicado o formato desejado, os resultados serão enviados no formato TSV.
3.1.2 RPC
De modo a superar as limitações (limite de caracteres de URI) da arquitectura RESTful foi implementada, em alternativa, a arquitectura RPC, podendo assim o utilizador escolher a que melhor lhe convém. Nesta arquitectura é utilizada a tecnologia
21
SOAP, visto ser uma das mais usadas com este tipo de arquitectura, e como tinha referido anteriormente em alguns casos é confundida com a própria arquitectura. Sendo uma arquitectura orientada para os serviços (SOA) foram identificados os diferentes serviços que disponibilizávamos, SSM e Characterization, sendo criado um endpoint para cada um destes /BOA/SSM e /BOA/Characterization. O scoping information e o method vão então no envelope SOAP, no corpo da mensagem do pedido HTTP, este envelope tem a estrutura apresentada na Figura 4. Sendo as etiquetas: i) <query>, que é utilizado como método deste pedido; ii) <acc>, que é a lista de identificadores e iii) <parameter>, que podem ser a <measure>, <goType> e <IEA>.
<?xml version="1.0" encoding="utf-8"?> <soap:Envelope xmlns:xsi="http://www.w3.org/2001/XMLSchema-instance" xmlns:xsd="http://www.w3.org/2001/XMLSchema" xmlns:soap="http://schemas.xmlsoap.org/soap/envelope/"> <soap:Body> <BOArequest xmlns="http://www.webserviceX.NET/"> <query>{query}</query> <acc>{inputList}</acc> <parameter1>paramValue1</parameter1> ... </BOArequest> </soap:Body> </soap:Envelope>
Figura 4 – Estrutura base do envelope SOAP para o pedido HTTP na arquitectura RPC
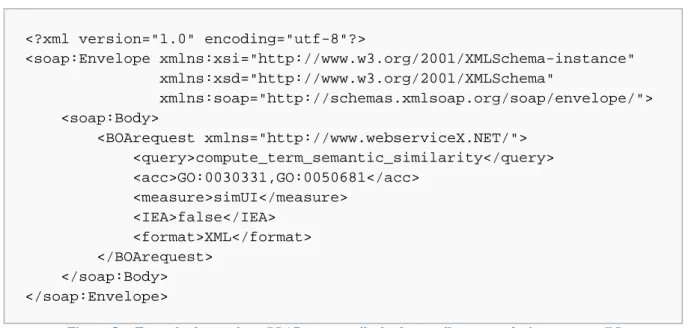
Assim, utilizando o exemplo apresentado para a arquitectura RESTful, calcular a semelhança semântica entre os GO terms “estrogen receptor binding” (GO:0030331) e “androgen receptor binding” (GO:0050681), teria de ser mandado o envelope SOAP apresentado na Figura 5 para o endpoint /BOA/SSM.
Como a implementação da arquitectura RPC está mais pensada para a integração com outras ferramentas e páginas web, o formato padrão da resposta é o JSON. Sendo possível alterar este formato, enviando no envelope SOAP, dentro da etiqueta BOArequest, uma etiqueta “format“ com o respectivo formato desejado (como se pode verificar na Figura 5). E do mesmo modo, é possível introduzir uma etiqueta “include” indicando que informação extra se pretende.
22 <?xml version="1.0" encoding="utf-8"?> <soap:Envelope xmlns:xsi="http://www.w3.org/2001/XMLSchema-instance" xmlns:xsd="http://www.w3.org/2001/XMLSchema" xmlns:soap="http://schemas.xmlsoap.org/soap/envelope/"> <soap:Body> <BOArequest xmlns="http://www.webserviceX.NET/"> <query>compute_term_semantic_similarity</query> <acc>GO:0030331,GO:0050681</acc> <measure>simUI</measure> <IEA>false</IEA> <format>XML</format> </BOArequest> </soap:Body> </soap:Envelope>
Figura 5 – Exemplo do envelope SOAP para o cálculo da semelhança semântica entre os GO terms GO:0030331 e GO:0050681
3.2 Back-End
Com os resultados dos serviços web a serem disponibilizados em diferentes formatos, tinha duas possibilidades de o fazer, ou convertia o formato dos resultados no serviço web ou directamente nos scripts das diversas ferramentas. Optei por fazer a conversão nos scripts pois evita que os resultados sejam percorridos por duas vezes, uma vez nos scripts e outra nos serviços web. Embora o percorrer dos resultados e a sua formatação não seja o passo limitante das nossas ferramentas, esta decisão foi tomada porque, para um input grande o número de resultados pode ser bastante elevado, se, por exemplo, tivermos um input de 1000 proteínas (sendo este o limite máximo permitido para o input) para calcular a semelhança semântica, o resultado são 499500 pares de proteínas8 com as respectivas semelhanças semânticas.
8
Matriz triangular superior (1000 1000 1000
23
Capítulo 4 Mediador
Tal como foi indicado anteriormente, foram implementadas duas arquitecturas de serviços web, neste capítulo vão ser detalhados os diferentes URI’s (arquitectura RESTful) e envelopes SOAP (arquitectura RPC).
4.1 RESTful
Na arquitectura RESTful podemos identificar duas partes distintas no URI base: /ontology/concept|entity/inputList/feature?parameter1=param Value1&... Existe uma hierarquia entre os conceitos, onde se indica em que ontologia se está a trabalhar (ontology) e se se está a trabalhar com os conceitos dessa ontologia ou com as entidades (concept|entity) introduzindo depois uma lista referente ao par ontology/concept|entity e é indicado a feature que se pretende calcular dessa lista. Relativamente a feature é ainda indicado os diferentes parâmetros, caso seja aplicável. Os parâmetros não estão estruturados hierarquicamente, como os outros conceitos, pois não têm relação entre eles. Por exemplo, a escolha da medida (measure) em nada influência a escolha do aspect do GO (goType).
Na arquitectura RESTful o método utilizado é o método do pedido HTTP, no nosso caso o único método utilizado é o método GET devido ao caso de estes serviços web serem read-only, só é possível receber informação, não é possível introduzir nova informação ou alterar a existente.
Na Tabela 2 estão apresentados os diferentes URI’s para representar os diferentes recursos que disponibilizamos.
24
URI Método HTTP Operação
/GO/protein/<inputList>/SSM?measure=<me asure>&goType=<gotype>&iea=<true|false>
GET Obter os valores de semelhança semântica entre todas as proteínas introduzidas
/GO/protein/<inputList>/Characterization?go type=<gotype>&iea=<true|false>
GET Obter até 100 termos GO associados às proteínas introduzidas
/GO/term/<inputList>/ssm?measure=<measu re>&iea=<true|false>
GET Obter os valores de semelhança semântica entre todos os termos GO introduzidos
/Chebi/pathway/<inputList>/ssm GET Obter os valores de semelhança semântica entre todas as vias metabólicas introduzidas /Chebi/pathway/<inputList>/Characterization GET Obter até 100 compostos ChEBI associados às vias metabólicas introduzidas /Chebi/compound/<inputList>/ssm?measure=
<measure>
GET Obter os valores de semelhança semântica entre todos os compostos ChEBI introduzidos
A escolha do formato dos resultados é feita no header do pedido HTTP, no campo Content-type, existindo quatro formatos aceites. Para obter os resultados em XML o contect-type terá de ser “text/xml”, para JSON é utilizado “application/json”, para TSV usa-se “text/tab-separated-values” e para CSV utiliza-se “text/comma-separated-values”. Por omissão, e tal como já foi referido, o formato enviado é o TSV, pois é um formato de fácil interpretação por humanos, sendo possível abrir o resultado numa folha de cálculo, como por exemplo no excel.
Em relação a informação adicional, tal como foi dito, esta é indicada no campo “include” do header do pedido HTTP. Neste momento o único valor aceite para este
25
campo é “name” para incluir assim os nomes dos termos ou compostos na feature Characterization. Caso sejam implementadas outras informações adicionais é possível adicionar os respectivos indicadores, neste campo, separados por vírgulas.
4.2 RPC
Tal como já foi referido anteriormente, a arquitectura RPC envia o scoping information e o method dentro do corpo da mensagem do pedido HTTP, neste caso, num envelope SOAP. A estrutura base do envelope SOAP é apresentada na Figura 6.
<?xml version="1.0" encoding="utf-8"?> <soap:Envelope xmlns:xsi="http://www.w3.org/2001/XMLSchema-instance" xmlns:xsd="http://www.w3.org/2001/XMLSchema" xmlns:soap="http://schemas.xmlsoap.org/soap/envelope/"> <soap:Body> <BOArequest xmlns="http://www.webserviceX.NET/"> <query>{query}</query> <acc>{inputList}</acc> <parameter1>paramValue1</parameter1> ... </BOArequest> </soap:Body> </soap:Envelope>
Figura 6 – Estrutura base do envelope SOAP para o pedido HTTP na arquitectura RPC
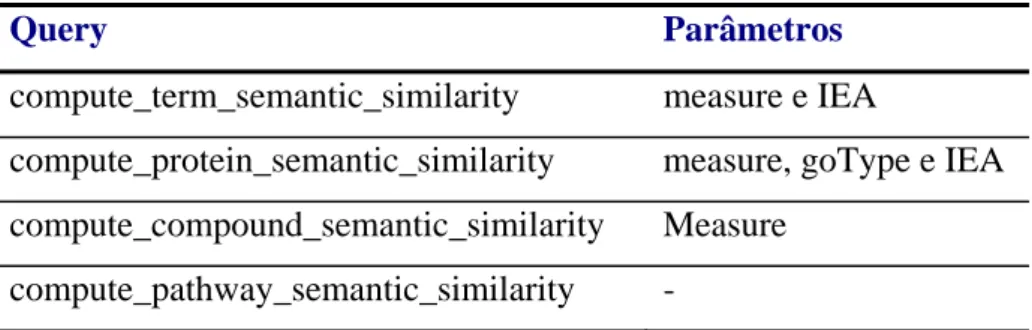
Para os diferentes endpoint existem diferentes valores para os conceitos. Na Tabela 3 estão indicados que valores de <query> e que parâmetros se utilizam no endpoint /SSM e, na Tabela 4 para o endpoint /Characterization.
Tabela 3 – Valor de <query> e os respectivos parâmetros para o endpoint /SSM
Query Parâmetros
compute_term_semantic_similarity measure e IEA
compute_protein_semantic_similarity measure, goType e IEA compute_compound_semantic_similarity Measure
compute_pathway_semantic_similarity -
26
Query Parâmetros
find_GO_terms_representativity goType e IEA find_compounds_representativity -
A escolha do formato dos resultados é na etiqueta format do envelope SOAP, como se pode ver o exemplo na Figura 7. Podendo ter os seguintes valores para os respectivos formatos: i) “text/xml” para XML; ii) “application/json” para JSON; iii) “text/tab-separated-values” para TSV e iv) “text/comma-separated-values” para CSV. Tal como foi referido anteriormente, é também possível, para o endpoint Characterization, adicionar uma etiqueta “include”, dentro da etiqueta “BOArequest”, com o valor “name” de modo a incluir os nomes dos termos ou compostos na resposta, e tal como na arquitectura RESTful, caso haja mais que uma informação adicional que se pretenda incluir (neste momento só é possível adicionar os nomes) esta é também indicada nesta etiqueta separando as diferentes informações adicionais que se deseja por vírgulas. <?xml version="1.0" encoding="utf-8"?> <soap:Envelope xmlns:xsi="http://www.w3.org/2001/XMLSchema-instance" xmlns:xsd="http://www.w3.org/2001/XMLSchema" xmlns:soap="http://schemas.xmlsoap.org/soap/envelope/"> <soap:Body> <BOArequest xmlns="http://www.webserviceX.NET/"> <query>compute_term_semantic_similarity</query> <acc>GO:0030331,GO:0050681</acc> <measure>simUI</measure> <IEA>false</IEA> <format>XML</format> </BOArequest> </soap:Body> </soap:Envelope>
Figura 7 – Exemplo do envelope SOAP para o cálculo da semelhança semântica entre os GO terms GO:0030331 e GO:0050681 recebendo os resultados no formato XML
4.3 Parâmetros
Em ambas as arquitecturas de serviços web, RESTful e RPC, o funcionamento dos parâmetros é igual: i) o parâmetro measure é utilizado para calcular a SSM na ontologia GO em ambos os casos, proteínas e termos, e na ontologia ChEBI para os compostos; ii) o parâmetro goType só é utilizado na ontologia GO para as proteínas,
27
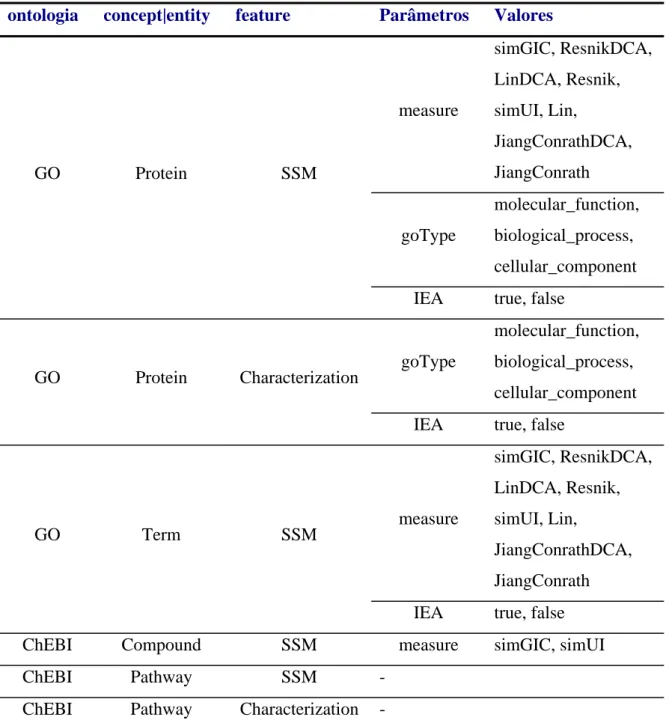
quer para o cálculo da SSM quer para o Characterization e iii) o parâmetro IEA é utilizado no cálculo da SSM na ontologia GO, tanto para proteínas como para termos, e no cálculo do Characterization. Na Tabela 5 estão indicadas as condições para usar cada um dos parâmetros e os respectivos valores.
Tabela 5 – Condições para a utilização de cada um dos parâmetros e os respectivos valores
ontologia concept|entity feature Parâmetros Valores
GO Protein SSM measure simGIC, ResnikDCA, LinDCA, Resnik, simUI, Lin, JiangConrathDCA, JiangConrath goType molecular_function, biological_process, cellular_component IEA true, false
GO Protein Characterization goType
molecular_function, biological_process, cellular_component IEA true, false
GO Term SSM measure simGIC, ResnikDCA, LinDCA, Resnik, simUI, Lin, JiangConrathDCA, JiangConrath IEA true, false
ChEBI Compound SSM measure simGIC, simUI
ChEBI Pathway SSM -
ChEBI Pathway Characterization -
4.4 Resultados
Tal como já foi referido, os resultados são disponibilizados em quatro formatos, CSV, TSV, JSON e XML, não havendo diferenciação dos resultados entre as diferentes
28
arquitecturas de serviços web, RESTful e RPC. Todos os formatos têm indicado que informação contêm: i) nos formatos CSV e TSV a primeira linha é constituída por um conjunto de termos identificadores dos diferentes elementos dos resultados, sendo separados por vírgulas ou tabulações, respectivamente, e cada linha seguinte contém os resultados correspondentes; ii) no formato JSON os resultados estão numa lista em que cada elemento da lista está no formato JSON, tendo um termo que identifica cada elemento do resultado; iii) no formato XML existe um cabeçalho com informação sobre os resultados (ontologia utilizada, feature calculada, etc), tendo depois uma etiqueta de resultados que contém uma lista de elementos com os resultados, dentro destes elementos estão um conjunto de termos (tags) com os respectivos elementos dos resultados. Da Figura 8 à Figura 21 estão representados os diferentes resultados base e alguns exemplos. <?xml version="1.0"?> <rdf:RDF xmlns:rdf="http://www.w3.org/1999/02/22-rdf-syntax-ns#" xmlns:xsd="http://www.w3.org/2001/XMLSchema#" xmlns:ssm="http://xldb.di.fc.ul.pt/ssm#"> <ssm:SemanticSimilarity> <ssm:ontology rdf:datatype='http://www.w3.org/2001/XMLSchema#string'>ontology</ssm:o ntology> <ssm:measure rdf:datatype='http://www.w3.org/2001/XMLSchema#string'>measure</ssm:me asure> <ssm:Sim> <ssm:entity> <concept|entity rdf:about="link"> <concept|entity:accession>acc</go:accession> </concept|entity> </ssm:entity> <ssm:entity> <concept|entity rdf:about="link"> <concept|entity:accession>acc</go:accession> </concept|entity> </ssm:entity> <ssm:score rdf:datatype='http://www.w3.org/2001/XMLSchema#float'>value</ssm:score > </ssm:Sim> <ssm:Sim> ... </ssm:Sim> </ssm:SemanticSimilarity> </rdf:RDF>
29 <?xml version="1.0"?> <rdf:RDF xmlns:rdf="http://www.w3.org/1999/02/22-rdf-syntax-ns#" xmlns:xsd="http://www.w3.org/2001/XMLSchema#" xmlns:char="http://xldb.di.fc.ul.pt/characterization#"> <char:Characterization> <char:ontology rdf:datatype='http://www.w3.org/2001/XMLSchema#string'>ontology</char: ontology> <char:Char> <char:entity> <concept rdf:about="link"> <concept:accession>acc</go:accession> </concept> </char:entity> <char:entityNum rdf:datatype='http://www.w3.org/2001/XMLSchema#int'>value</char:entity Num> <char:Precent rdf:datatype='http://www.w3.org/2001/XMLSchema#percent'>value</char:Pr ecent> <char:e-value rdf:datatype='http://www.w3.org/2001/XMLSchema#scientific-decimals'>value</char:e-value> <char:infoContent rdf:datatype='http://www.w3.org/2001/XMLSchema#float'>value</char:info Content> <char:entityList> <entity rdf:about="link"> <entity:accession>acc</entity:accession> </entity> <entity rdf:about="link"> ... </entity> </char:entityList> </char:Char> <char:Char> ... </char:Char> </char:Characterization> </rdf:RDF>
Figura 9 – Exemplo dos resultados base para o cálculo da Characterization no formato XML
<?xml version="1.0"?>
<rdf:RDF xmlns:rdf="http://www.w3.org/1999/02/22-rdf-syntax-ns#" xmlns:xsd="http://www.w3.org/2001/XMLSchema#"
xmlns:char="http://xldb.di.fc.ul.pt/characterization#"> <char:Characterization>
30 <char:ontology rdf:datatype='http://www.w3.org/2001/XMLSchema#string'>ontology</char: ontology> <char:Char> <char:entity> <concept rdf:about="link"> <concept:accession>acc</concept:accession> <concept:name>name</concept:name> </concept> </char:entity> <char:entityNum rdf:datatype='http://www.w3.org/2001/XMLSchema#int'>value</char:entity Num> <char:Precent rdf:datatype='http://www.w3.org/2001/XMLSchema#percent'>value</char:Pr ecent> <char:e-value rdf:datatype='http://www.w3.org/2001/XMLSchema#scientific-decimals'>value</char:e-value> <char:infoContent rdf:datatype='http://www.w3.org/2001/XMLSchema#float'>value</char:info Content> <char:entityList> <entity rdf:about="link"> <entity:accession>acc</entity:accession> </entity> <entity rdf:about="link"> ... </entity> </char:entityList> </char:Char> <char:Char> ... </char:Char> </char:Characterization> </rdf:RDF>
Figura 10 – Exemplo dos resultados base para o cálculo da Characterization no formato XML, com a inclusão dos nomes
{'sim':[{'entity':[acc,acc],'score':value},...]}
Figura 11 – Exemplo dos resultados base para o cálculo da SSM no formato JSON
{'Char':[{'entity':{'accession':acc},'entityNum':value,'Precent':value
,'e-value':value,'infoContent':value,'entityList':entity},...]}
31
{'Char':[{'entity':{'name':name,'accession':acc},'entityNum':value,'Pr
ecent':value,'e-value':value,'infoContent':value,'entityList':entity},
...]}
Figura 13 – Exemplo dos resultados base para o cálculo da Characterization no formato JSON, com a inclusão dos nomes
entity,entity,score
acc,acc,value
...
Figura 14 – Exemplo dos resultados base para o cálculo da SSM no formato CSV
Accession,entityNum,Precent,e-value,infoContent,entityList
Acc,value,value,value,value,entity
...
Figura 15 – Exemplo dos resultados base para o cálculo da Characterization no formato CSV
Accession,name,entityNum,Precent,e-value,infoContent,entityList
Acc,name,value,value,value,value,entity
...
Figura 16 – Exemplo dos resultados base para o cálculo da Characterization no formato CSV, com a inclusão dos nomes
Entity entity score
Acc acc value
...
Figura 17 – Exemplo dos resultados base para o cálculo da SSM no formato TSV
accession entityNum Precent e-value infoContent entityList
acc value value value value entity
...
32
accession name entityNum Precent e-value infoContent entityList
acc name value value value value entity
...
Figura 19 – Exemplo dos resultados base para o cálculo da Characterization no formato TSV, com a inclusão dos nomes
{'sim':[{'entity':[{'accession':'P55058'},{'accession':'P54136'}],'sco re':0.139},{'entity':[{'accession':'P27918'},{'accession':'P55058'}],' score':0.086},{'entity':[{'accession':'P27918'},{'accession':'P54136'} ],'score':0.083},{'entity':[{'accession':'P55058'},{'accession':'Q9HB6 3'}],'score':0.05},{'entity':[{'accession':'P27918'},{'accession':'Q9H B63'}],'score':0.031},{'entity':[{'accession':'Q9HB63'},{'accession':' P54136'}],'score':0.03}]}
Figura 20 – Resultados para o cálculo da SSM no formato JSON, para as proteínas P27918, P55058, Q9HB63, P54136 com IEA igual a true, goType igual a biological_process e a measure igual a simUI
<?xml version="1.0"?> <rdf:RDF xmlns:rdf="http://www.w3.org/1999/02/22-rdf-syntax-ns#" xmlns:xsd="http://www.w3.org/2001/XMLSchema#" xmlns:chebi="http://www.ebi.ac.uk/chebi/init.do#" xmlns:kegg="http://www.genome.jp/kegg/pathway.html/#" xmlns:char="http://xldb.di.fc.ul.pt/characterization#"> <char:Characterization> <char:ontology rdf:datatype="http://www.w3.org/2001/XMLSchema#string">chebi_ontology< /char:ontology> <char:Char> <char:entity> <chebi:compound rdf:about="http://www.ebi.ac.uk/chebi/searchId.do?chebiId=CHEBI:15705" > <chebi:accession>CHEBI:15705</chebi:accession> </chebi:compound> </char:entity> <char:ProtNum rdf:datatype='http://www.w3.org/2001/XMLSchema#int'>2</char:ProtNum> <char:Precent rdf:datatype='http://www.w3.org/2001/XMLSchema#percent'>100.0%</char:P recent> <char:e-value rdf:datatype='http://www.w3.org/2001/XMLSchema#scientific-decimals'>5.8e-02</char:e-value>
33 <char:infoContent rdf:datatype='http://www.w3.org/2001/XMLSchema#float'>0.2500</char:inf oContent> <char:PathwayList> <kegg:pathway rdf:about="http://www.genome.jp/dbget-bin/www_bget?pathway:map00473"> <kegg:accession>map00473</kegg:accession> </kegg:pathway> <kegg:pathway rdf:about="http://www.genome.jp/dbget-bin/www_bget?pathway:map00910"> <kegg:accession>map00910</kegg:accession> </kegg:pathway> </char:PathwayList> </char:Char> <char:Char> <char:entity> <chebi:compound rdf:about="http://www.ebi.ac.uk/chebi/searchId.do?chebiId=CHEBI:35757" > <chebi:accession>CHEBI:35757</chebi:accession> </chebi:compound> </char:entity> <char:ProtNum rdf:datatype='http://www.w3.org/2001/XMLSchema#int'>2</char:ProtNum> <char:Precent rdf:datatype='http://www.w3.org/2001/XMLSchema#percent'>100.0%</char:P recent> <char:e-value rdf:datatype='http://www.w3.org/2001/XMLSchema#scientific-decimals'>1.3e-01</char:e-value> <char:infoContent rdf:datatype='http://www.w3.org/2001/XMLSchema#float'>0.1828</char:inf oContent> <char:PathwayList> <kegg:pathway rdf:about="http://www.genome.jp/dbget-bin/www_bget?pathway:map00473"> <kegg:accession>map00473</kegg:accession> </kegg:pathway> <kegg:pathway rdf:about="http://www.genome.jp/dbget-bin/www_bget?pathway:map00910"> <kegg:accession>map00910</kegg:accession> </kegg:pathway> </char:PathwayList> </char:Char> <char:Char> <char:entity> <chebi:compound rdf:about="http://www.ebi.ac.uk/chebi/searchId.do?chebiId=CHEBI:25384" > <chebi:accession>CHEBI:25384</chebi:accession> </chebi:compound> </char:entity>
34 <char:ProtNum rdf:datatype='http://www.w3.org/2001/XMLSchema#int'>2</char:ProtNum> <char:Precent rdf:datatype='http://www.w3.org/2001/XMLSchema#percent'>100.0%</char:P recent> <char:e-value rdf:datatype='http://www.w3.org/2001/XMLSchema#scientific-decimals'>3.4e-01</char:e-value> <char:infoContent rdf:datatype='http://www.w3.org/2001/XMLSchema#float'>0.0953</char:inf oContent> <char:PathwayList> <kegg:pathway rdf:about="http://www.genome.jp/dbget-bin/www_bget?pathway:map00473"> <kegg:accession>map00473</kegg:accession> </kegg:pathway> <kegg:pathway rdf:about="http://www.genome.jp/dbget-bin/www_bget?pathway:map00910"> <kegg:accession>map00910</kegg:accession> </kegg:pathway> </char:PathwayList> </char:Char> <char:Char> <char:entity> <chebi:compound rdf:about="http://www.ebi.ac.uk/chebi/searchId.do?chebiId=CHEBI:25703" > <chebi:accession>CHEBI:25703</chebi:accession> </chebi:compound> </char:entity> <char:ProtNum rdf:datatype='http://www.w3.org/2001/XMLSchema#int'>2</char:ProtNum> <char:Precent rdf:datatype='http://www.w3.org/2001/XMLSchema#percent'>100.0%</char:P recent> <char:e-value rdf:datatype='http://www.w3.org/2001/XMLSchema#scientific-decimals'>3.9e-01</char:e-value> <char:infoContent rdf:datatype='http://www.w3.org/2001/XMLSchema#float'>0.0825</char:inf oContent> <char:PathwayList> <kegg:pathway rdf:about="http://www.genome.jp/dbget-bin/www_bget?pathway:map00473"> <kegg:accession>map00473</kegg:accession> </kegg:pathway> <kegg:pathway rdf:about="http://www.genome.jp/dbget-bin/www_bget?pathway:map00910"> <kegg:accession>map00910</kegg:accession> </kegg:pathway> </char:PathwayList> </char:Char>
35 </char:Characterization>
</rdf:RDF>
Figura 21 – Resultados para o cálculo da Characterization no formato XML, para as vias metabólicas map00910 e map00473
37
Capítulo 5 Back-End e Front-End
Neste capítulo vão ser apresentadas as modificações efectuadas no back-end e no front-end para a integração do mediador na framework.
5.1 Back-End
Um dos objectivos secundários deste trabalho era a actualização dos scripts das ferramentas de modo a estas formatarem os resultados num formato pedido pelo utilizador, TSV, CSV, JSON ou XML. Para ambas as ferramentas, ProteInOn e CMPSim, a estratégia tomada foi idêntica, adicionar um novo argumento na chamada destas ferramentas, que iria indicar qual o formato pretendido, e, no ciclo onde os resultados eram formatados, verificar qual era o formato pretendido, e assim, devolver os resultados nesse formato. Para além destas modificações, foi também adicionado um argumento opcional de modo a incluir a informação adicional desejada pelo utilizador.
5.1.1 ProteInOn
No caso do ProteInOn esta actualização quase que foi automática, pois já tinha sido feita uma actualização prévia do ProteInOn de modo a este receber um argumento correspondente ao formato pedido pelo utilizador e, os resultados eram formatados em dois formatos, XML e numa tabela HTML. Assim, foi só necessário actualizar o formato XML e introduzir os outros três formatos, JSON, CSV e TSV. De seguida foi acrescentado um argumento que indicasse que informação adicional o utilizador deseja. Este argumento foi introduzido no final da chamada da ferramenta. Assim, não só não era necessário fazer alterações em termos de código, como se torna opcional.
5.1.2 CMPSim
Para o CMPSim esta actualização sofreu uns passos adicionais, visto esta ferramenta só permitir o cálculo da semelhança semântica entre dois compostos ou duas
38
vias metabólicas, usando duas medidas implementadas, simGIC e simUI. Por esta razão foi modificado a chamada da ferramenta de modo a receber como argumentos, para além do formato desejado, qual a medida para o cálculo da semelhança semântica e, utilizado no mesmo modo que para o ProteInOn, um argumento opcional para a informação adicional. Por outro lado, foi adicionado um ciclo de modo a percorrer o input de compostos ou vias metabólicas e, assim calcular a semelhança semântica entre os diferentes pares, sendo formatados no formato pedido pelo utilizador. No caso da funcionalidade Characterization, e vista a estrutura da ontologia ChEBI ser idêntica ao GO, foi aproveitado o código do ProteInOn para, com as devidas adaptações, se poder ter esta funcionalidade no CMPSim.
5.1.3 Guia
Para as futuras ferramentas ou funcionalidades é necessário ter em conta dois pontos: i) a formatação dos resultados em quatro formatos diferentes, TSV, CSV, JSON e XML; ii) e a possibilidade de informação adicional pedida pelo utilizador. É então sugerido que: i) para novas funcionalidades (ontologias biomédicas já em uso) seja primeiro feito um resultado base para cada um dos formatos (TSV, CSV, JSON e XML), de modo a ser o mais transversal possível para as diferentes ontologias biomédicas, e só depois especificar todos os formatos base para cada ontologia em uso; ii) no caso de novas ferramentas, com utilização de outras ontologias biomédicas e as mesmas funcionalidades (as que sejam aplicáveis a essas ontologias), sejam utilizados os formatos base apresentados no Capítulo 4 (4.4 Resultados). Para as novas ferramentas é ainda sugerido seguir a estratégia tomada quer no ProteInOn como no CMPSim, de ter um argumento opcional (como último argumento da chamada dos scripts) para as informações adicionais pedidas pelo utilizador.
5.2 Front-End
Tal como já foi dito, a actualização do Front-End teve dois propósitos : i) por um lado, fazer com que as páginas web das ferramentas usassem os serviços web criados; ii) por outro lado, dar ao utilizador mais funcionalidades e tornar a página dinâmica não aumentando a carga de informação trocada com o servidor.
39
5.2.1 ProteInOn
No caso do ProteInOn a actualização do Front-End foi um processo facilitado, pois já tinha sido feito por mim uma actualização deste, anteriormente. Esta actualização teve assim dois passos: i) trocar os serviços web anteriormente utilizados pelos que foram criados (arquitectura RPC) e ii) processar os resultados para visualização destes. Em relação ao primeiro ponto, como todo o Javascript (AJAX) já tinha sido escrito, foi só alterar o URL do XMLHTTPRequest pelos respectivos endpoints (SSM e Characterization) e o conteúdo do envelope (SOAP). Em relação ao segundo ponto implementei dois processos: i) criei um ciclo que percorre todos os resultados e introduz estes numa tabela HTML e ii) criei um pequeno módulo Javascript que permite a páginação da tabela de resultados e a ordenação destas.
5.2.2 CMPSim
A actualização do Front-End do CMPSim foi um processo que se fez em mais passos. i) foi modificar o Front-End de modo a receber mais que dois compostos ou vias metabólicas, para tal, foram substituídas as duas etiquetas “input” por uma “text-area” e incluído uma “select” (drop-down list), ii) foi também necessário escrever todo o código Javascript, pois esta ainda era uma simples página estática, onde se inclui a utilização dos serviços web criados no XMLHTTPRequest, iii) por último, foi utilizado o pequeno módulo criado anteriormente (ProteInOn) para manipular a tabela de resultados.
5.2.3 Exemplos
Tal como indiquei, a integração dos serviços web no Front-End é a partir da função XMLHTTPRequest do Javascript, na Figura 22 encontra-se um exemplo de como fazer essa chamada.
function Request( url , query , param , measure , goType , IEA ) { xmlhttp = null;
if ( window.XMLHttpRequest ) { xmlhttp = new XMLHttpRequest( ); }
else {
xmlhttp = new ActiveXObject( "Microsoft.XMLHTTP" ); }
40
if (xmlhttp.readyState==4 && xmlhttp.status==200) { respostaJSON = eval('(' + xmlhttp.responseText + ')'); for( var i in respostaJSON['Char'] ) {
... } } }
xmlhttp.open( "POST" , url+"/BOA/SSM/" , true );
xmlhttp.setRequestHeader( 'SOAPAction' , url+'/BOA/SSM/' ); xmlhttp.setRequestHeader( 'Content-Type' , 'application/json' ); var xml = '<?xml version="1.0" encoding="utf-8"?>' +
'<soap:Envelope xmlns:xsi="http://www.w3.org/2001/XMLSchema-instance" ' + 'xmlns:xsd="http://www.w3.org/2001/XMLSchema" ' + 'xmlns:soap="http://schemas.xmlsoap.org/soap/envelope/">' + '<soap:Body> ' + '<BOArequest xmlns="http://www.webserviceX.NET/"> ' + '<query>' + query + '</query> ' +
'<acc>' + param + '</acc> ' +
'<measure>' + measure + '</measure> ' + '<goType>' + goType + '</goType> ' + '<IEA>' + IEA + '</IEA> ' +
'<include>name</include> ' + '</BOArequest> ' + '</soap:Body> ' + '</soap:Envelope>'; xmlhttp.send( xml ); }
Figura 22 – Exemplo de um pedido XMLHTTPResquest para os serviços web arquitectura RPC
Mas não é só a partir de Javascript que se pode aceder aos serviços web, e também não é obrigatório que sejam utilizados os serviços web de arquitectura RPC, na Figura 23 está um exemplo de como aceder aos serviços web de arquitectura RESTful a partir de um script em Perl.
#!/usr/bin/perl use strict; use warnings;
# Create a user agent object use LWP::UserAgent;
my $ua = LWP::UserAgent->new; $ua->agent("MyUserAgent");
#array containing the ids to be processed
my @id = (28385,28386,28387,28388,28389,28390); my $ids = join(',',@id);
41 my $req = HTTP::Request->new(GET =>
"http://xldb.fc.ul.pt/biotools/BOA/Chebi/compound/$ids/SSM/measure/sim GIC");
$req->content_type('text/tab-separated-values');
# Pass request to the user agent and get a response back my $res = $ua->request($req);
# Check the outcome of the response if ($res->is_success) {
my $result = $res->content;
my @result = split(/\n/, $result); my $index=0;
while ($index < @result) {
if ($result[$index] =~ m/\d+\t*\d+\t*\d+.\d+\s*/ig) { print "$index:\t$result[$index]\n"; } $index++; } } else { print $res->status_line, "\n"; }