Análise da influência do kernel de transição na estimativa de parâmetros em
modelo aplicado ao processo de produção de etanol por saccharomyces cerevisiae
Analysis of the influence of transition kernel in the estimation of parameters in
model applied to the process of ethanol production by saccharomyces cerevisiae
DOI:10.34117/bjdv6n6-087
Recebimento dos originais: 01/05/2020 Aceitação para publicação: 03/06/2020
Camila Santana Dias
Mestre em Engenharia Química pela Universidade Federal do Pará Instituição: Universidade Federal do Pará
Endereço: R. Augusto Corrêa, 01 - Guamá, Belém - PA, Brasil E-mail: com.camiladias@gmail.com
Berilo Costa de Matos Junior
Bacharel em Engenharia de Controle e Automação pelo Instituto Federal de Educação, Ciência e Tecnologia do Pará
Instituição: Universidade Federal do Pará
Endereço: R. Augusto Corrêa, 01 - Guamá, Belém - PA, Brasil E-mail: berilojunior7@hotmail.com
Bruno Marques Viegas
Doutor em Engenharia de Recursos Naturais pela Universidade Federal do Pará Instituição: Universidade Federal do Pará
Endereço: R. Augusto Corrêa, 01 - Guamá, Belém - PA, Brasil E-mail: bviegas@ufpa.br
Nielson Fernando da Paixão Ribeiro
Doutor em Engenharia Química pelo Instituto Alberto Luiz Coimbra-UFRJ Instituição: Universidade Federal do Pará
Endereço: R. Augusto Corrêa, 01 - Guamá, Belém - PA, Brasil E-mail: nielson@ufpa.br
Emanuel Negrão Macêdo
Doutor em Engenharia Mecânica pela Universidade Federal do Rio de Janeiro Instituição: Universidade Federal do Pará
Endereço: R. Augusto Corrêa, 01 - Guamá, Belém - PA, Brasil E-mail:enegrao@ufpa.br
Diego Cardoso Estumano
Doutor em Engenharia Mecânica pela Universidade Federal do Rio de Janeiro Instituição: Universidade Federal do Pará
Endereço: R. Augusto Corrêa, 01 - Guamá, Belém - PA, Brasil E-mail:dcestumano@ufpa.br
RESUMO
Este trabalho avalia um modelo de cinética microbiana de produção de etanol por processo fermentativo, ponderado pela concentração máxima do microrganismo Saccharomyces cerevisiae. Empregou-se o método de Monte Carlo via Cadeias de Markov, com algoritmo de Metropolis-Hastings, para estimação dos parâmetros, bem como para obtenção do perfil das variáveis de estado do sistema analisado. Diferentes kernels de transição foram avaliados ao utilizar as distribuições de probabilidade: uniforme, t-Student e normal. O kernel com distribuição t-Student alcançou o equilíbrio em menor período de aquecimento. Já o de kernel uniforme, necessitou de mais estados da cadeia de Markov para alcançar a distribuição estacionária. A distribuição normal, apresentou elevada taxa de rejeição de parâmetros candidatos, fazendo com que a cadeia não se movesse por vários estados. Os valores mostrados pelo RMS indicam boas aproximações entre o obtido com a estimação e a referência.
Palavras chave: Kernel de transição, MCMC, Produção de etanol, Saccharomyces cerevisiae. ABSTRACT
This work is available in a microbial model of microbial ethanol production by a fermentation process, weighted by the maximum concentration of microorganism Saccharomyces cerevisiae. The Monte Carlo method via Markov Chains, with a Metropolis-Hastings algorithm, was used to estimate the most sensitive parameters, as well as to obtain the profile of the state variables of the analyzed system. Different transition kernels were evaluated used: uniform, t-Student and normal probability distributions. The kernel with t-Student distribution reached equilibrium in a shorter warm-up period. The uniform kernel, however, required more states in the Markov chain to achieve stationary distribution. The normal distribution showed a high rate of rejection of candidate parameters, causing the chain not to move through several states. The values shown by the RMS indicated good approximations between the one obtained with the estimation and the reference.
Keywords: Transition kernel, MCMC, Ethanol production, Saccharomyces cerevisiae. 1 INTRODUÇÃO
O etanol é produzido pela fermentação de biomassa de várias origens. Os microrganismos comumente utilizados em processos fermentativos para obtenção de etanol, são as leveduras. A
Saccharomyces cerevisiae é uma das mais conhecidas e aplicadas nessa rota (El-gendy et al, 2013,
Izmirlioglu e Demirci, 2010).
A modelagem deste processo, seja em batelada e batelada alimentada, é formulada como um sistema de equações algébrico-diferenciais obtido a partir balanços de massa para substratos, biomassa e produtos, expressões cinéticas, equações de projeto (Ochoa et al 2016).
Estas equações incluem vários parâmetros, muitos dos quais relacionados à cinética da reação e cujos valores são geralmente incertos. O estudo de modelos cinéticos pode contribuir para o controle e redução de custos do processo, aumentar a qualidade do produto, podendo ser utilizados para descrever o fenômeno reacional em diferentes condições (Dodić et al, 2012).
Nesse sentido, este trabalho visa utilizar as distribuições de probabilidade uniforme, t-Student e normal no kernel de transição, ou distribuição auxiliar, do algoritmo de Metropolis-Hastings. No
intuito de verificar a maneira como influenciam a etapa de geração de parâmetros candidatos e a evolução das cadeias de Markov no processo de estimação de parâmetros de um modelo empregado em processos fermentativos para produção de etanol.
2 MODELO DIRETO
O modelo direto (MD) utilizado neste trabalho, refere-se ao balanço de massa em reator batelada realizado com o objetivo de descrever as concentrações de Biomassa (X), Sacarose (S), Glicose (S1) e Etanol (P), conforme apresentado nas Equações (1) – (5)1. Os valores utilizados como
informação a priori para os parâmetros µmáx; K1, K, YXS e YXP são mostrados na Tabela 1, encontrados
no estudo feito por (Silva et al, 2016).
dX X dt = (1) XS dS X dt Y = − (2) 1 1 1 dS K S KS dt = − (3) XP dP X dt Y = − (4) X (1 ) X máx máx = − (5)
Tabela 1: Valores utilizados como informação a priori para os parâmetros.
Parâmetro (hmáx -1)
1
K (h-1)
K (h-1) Y XS Y XP
Valor 5,1164 37,5556 20,5343 0,02795 0,04872
Fonte: Silva et al., 2016.
XS
Y é o fator de conversão do substrato em biomassa (YXS =dX dS), Y o fator de conversão de XP substrato em produto (YXP =dX dP), Ke K são constantes cinéticas, 1 éa velocidade específica de crescimento celular proposta por Dodić et al (2012) e Xmáx é a máxima concentração celular atingida.
O vetor que contém as variáveis de estado Biomassa (X), Sacarose (S), Glicose (S1) e Etanol
(P) está representado conforme a Equação (6) e o que contém os parâmetros µmáx, K, K1, YXS, YXP
[X S S1 P] Y (6) 1 XS XP [ máx, K , K, Y , Y ] P (7) 3 ANÁLISE DE SENSIBILIDADE
É importante identificar os parâmetros para os quais as variáveis de estado do modelo são mais sensíveis, o que é alcançado em geral por uma análise de sensibilidade (AS) (Ochoa et al., 2016). Em problemas cujos parâmetros apresentam diferentes ordens de magnitude o coeficiente de sensibilidade pode apresentar diferentes ordens de grandeza, dificultando a identificação e comparação de possível dependência linear entre eles (Ozisik & Orlande, 2000). Esta dificuldade pode ser amenizada utilizando-se o coeficiente de sensibilidade reduzido, conforme mostrado na Equação (8).
O método de aproximação por diferenças finitas, é utilizado no cálculo deste coeficiente, por apresentar fácil implementação (Kirsch et al. 2005) e, principalmente, por não haver solução analítica para determinar o coeficiente de sensibilidade reduzido (ver Equação (8)).
i ij j j J Y P P (8)
Neste trabalho a análise de sensibilidade local foi aplicada com objetivo de indicar quais entre os parâmetros apresentaria sensibilidade significativa à cinética reacional estudada. Além disso, fazer um estudo de dependência linear uma vez que caso os coeficientes de sensibilidade sejam linearmente dependentes é necessário que escolha apenas um dentre o conjunto que a dependência linear foi observada (Ozisik & Orlande, 2000).
4 INFERÊNCIA BAYESIANA
A análise por inferência bayesiana fornece uma estrutura onde o conhecimento da distribuição de probabilidade a priori dos parâmetros e os dados experimentais são considerados simultaneamente. Essa estrutura permite obter inferências na forma de distribuição de probabilidade a posteriori a espeito dos parâmetros do modelo que se considera descrever o sistema analisado (Pasqualette et al, 2016, Estumano et al., 2014, Galagali e Marzouk, 2014, Gamerman, 1996).
A combinação dessas informações é dada pelo teorema de Bayes, conforme mostrado pela Equação (9). π( | )π( ) π( | ) = π( ) Y P P P Y Y (9)
Em que π( | )P Y é a distribuição de probabilidade a posteriori, π( )P a distribuição de probabilidade a priori dos parâmetros, π( | )Y P a verossimilhança que contém a informação das medidas e o denominador π( )Y , que funciona apenas como uma constante de normalização.
Estimativas de parâmetros e variáveis de estado através de técnicas bayesianas vem sendo aplicadas em diferentes pesquisas de engenharia química como cinética química (Dias et al., 2020, Baia et al., 2020, Dias et al., 2019, Monteiro et al., 2019), balanço populacional (Moura et al., 2020a, Moura et al., 2010b), adsorção (Amador et al., 2020, Coutinho et al., 2020, Amador et al., 2019, Ferreira et al., 2019).
Métodos iterativos são utilizados para extrair informações da posteriori com o propósito de gerar amostras aleatórias dessa distribuição (Gelman et al., 2004). Nestetrabalho o método utilizado para este fim foi o de Monte Carlo via Cadeias de Markov.
4.1 MONTE CARLO VIA CADEIAS DE MARKOV (MCMC)
Método de Monte Carlo via Cadeias de Markov (MCMC) é um processo estocástico em que cada valor simulado, 𝑷𝒊, dado todos os valores anteriores 𝑷0, … , 𝑷𝑖−1 , depende apenas do seu
antecessor 𝑷𝑖−1 (Orlande et al., 2011).
Metropolis-Hastings (MH) é um algoritmo de aceitação-rejeição que utiliza o kernel de transição q
(
P*|Pi-1)
e deste retira amostras aleatórias com objetivo de gerar parâmetros candidatos*
P . Tais parâmetros são aceitos ou não de acordo com a probabilidade dada pela razão de Hastings
( α ) conforme mostrado na Equação (10) (Metropolis et al., 1953, Hastings, 1970; Naveira-cotta, 2009).
(
i-1 *)
(
(
*) (
) (
i-1 *)
)
i-1 * i-1 π | q | α , = min π | q | P Y P P P P P Y P P (10)A estimativa dos parâmetros, realizada, com o método de Monte Carlo via Cadeia de Markov (MCMC), foi implementado segundo o algoritmo de Metropolis-Hastings e seguiu a sequência apresentada na Figura 1.
Figura 1: Fluxograma do algoritmo utilizado para estimativa dos parâmetros. Início Pare i = 1 Especificar P0 i = i +1 Pi-1 = P* Cálculo de α (Pi-1, P*) u ~ U(0,1) Verifica-se aceitação u ≤ α u > α i ≤ N P* candidato a partir de q(P*|Pi-1) i ≤ N i = i + 1 5 RESULTADOS
A informação a priori dos parâmetros foi assumida como normalmente distribuída. O modelo direto foi solucionado obtendo-se o perfil de referência (YRef) para as variáveis de estado. A partir
deste perfil, medidas simuladas foram geradas com diferentes níveis de incerteza (σmed = 5 e 10%)
conforme mostrado pela Equação 11.
med Ref
med
= +σ
Y Y ε (11)
onde ε é uma variável aleatória N(0,1).
A etapa de geração do parâmetro candidato P*, deu-se conforme as distribuições auxiliares
(kernel de transição) mostradas nas Equações (12a) – (12c).
* i-1 i-1 N = + wr P P P Normal (12a) * i-1 i-1 T = + wr P P P t-Student (12b) * i-1 i-1 U = + w r P P P Uniforme (12c)
onde rN, rT e rU representam números aleatórios de uma distribuição normal, t- Student e uniforme
respectivamente, com média zero e desvio um. Em que w é o passo de procura e a este não devem ser atribuídos valores muito pequenos a fim de que a convergência da cadeia seja muito lenta ou muito grandes sendo necessário um número maior de iterações, neste trabalho o valor de w foi de 0,003. Visando verificar se o parâmetro candidato P seria aceito ou não, um número randômico u *
foi gerado a partir de uma distribuição uniforme u~U(0,1) e comparado com o resultado da Equação (10).
Sendo u ≤ α o novo valor é aceito e atualizado o parâmetro Pi−1=P*. Caso contrário a cadeia não se move e um novo valor candidato é gerado. Este processo iterativo se repete até que o número de estados da cadeia de Markov (N) seja atingido.
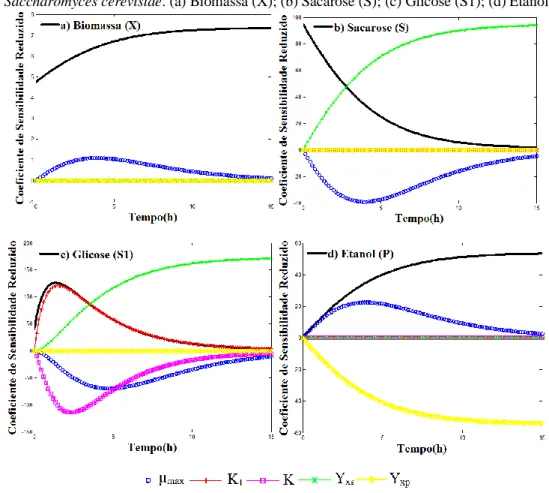
A análise gráfica (Figura 2) da sensibilidade dos parâmetros em relação às variáveis de estado, resultados estes sumarizados na Tabela 1. Assim como as estimativas para cada cenário avaliado, conforme mostrado pelas Tabelas 3 - 5.
É possível observar na Figura 2 o coeficiente de sensibilidade reduzido, cuja magnitude dos parâmetros, indica a sensibilidade que estes exercem em relação a cada espécie envolvida no processo de fermentação para obtenção de etanol. Graficamente foi possível perceber que o único parâmetro sensível para biomassa (X) foi µmax. Já para a sacarose (S) µmax e Yxs. Em relação a glicose (S1), µmax,
K1, K, Yxs apresentaram sensibilidade, por fim, para o etanol (P) µmax e Yxp, foram os parâmetros
influentes.
Figura 2: Coeficiente de sensibilidade reduzido para todas as variáveis de estado envolvidas na produção de etanol com
Saccharomyces cerevisiae. (a) Biomassa (X); (b) Sacarose (S); (c) Glicose (S1); (d) Etanol
A (in)dependência linear entre os parâmetros está indicada na Tabela 2 por uma simbologia, no qual se em uma mesma coluna os marcadores forem diferentes, trata-se de parâmetros linearmente
independentes. Se forem iguais, indicam dependência linear. A escala de cinza serve para apontar onde é mais acentuada a magnitude dos parâmetros.
Tabela 2: Sensibilidade dos parâmetros em relação as variáveis de estado.
Variáveis de Estado Parâmetros X S S1 P µmax ▲ ▲ ▲ ▲ K1 - - x - K - - x - Yxs - ● ● - Yxp - - - ●
A análise da Tabela 2 mostra que para a maioria das espécies (X, S e P) os parâmetros apresentaram independência linear. Entretanto, a dinâmica simétrica em relação aos coeficientes K e K1 observado para a glicose (S1), indica que são linearmente dependentes. A Figura 3 apoia o que foi
observado na AS e mostra a forte correlação que há entre eles.
Figura 3: Correlação entre os parâmetros K e K1.
Sutton et al., (2016) atribuem que o efeito da correlação paramétrica gera sensibilidade local, onde alterações em um parâmetro afeta outros, o que viola a suposição de independência entre eles. A fim de que o processo de estimação não seja prejudicado, apenas um entre esses dois parâmetros, foi estimado com os demais indicados pela sensibilidade.
Jayawardhana et al. (2008) aplicaram algumas perturbações no processo de estimação de parâmetros com MCMC buscando gerar alguns casos e observar os efeitos provocados. No mesmo sentido, este trabalho avaliou perturbações na média (µ) e desvio dos parâmetros (σp) ao utilizar priori
gaussiana. Bem como atribuir diferentes níveis de incerteza às medidas simuladas (σm).
Este trabalho utilizou as distribuições uniforme, t-Student e normal para representar números aleatórios empregados na etapa de geração do parâmetro candidato com a finalidade de avaliar a influência do kernel de transição. Bem como, verificar a influência de cada uma no processo de
estimação. As Figuras 4, 5 e 6 exibem estados das cadeias de Markov para os diferentes kernels e diferentes valores de estimativa inicial para os parâmetros. A ampliação aplicada às Figuras, serve apenas para uma visualização mais detalhada da evolução das cadeias, e Pref refere-se ao valor
assumido como referência para os parâmetros.
Nas Figuras 4 – 6, diferentes valores de estimativa inicial foram empregados (PRef, 0,5PRef e
1,5PRef) no intuito de verificar se o algoritmo desenvolvido possuía viés e se convergiria para uma
distribuição de equilíbrio mesmo que a cadeia partisse de diferentes pontos.
Figura 4: Cadeias de Markov com kernel uniforme para os parâmetros µmax (a), K (b) e Yxp (c) com diferentes valores de
estimativa inicial. Valor de referência (PRef) (linha vermelha); a partir do valor de referência (linha azul); 50% abaixo do
valor de referência (linha rosa); e 50% acima do valor de referência (linha preta). (a)
Figura 5: Cadeias de Markov com kernel t-Student para os parâmetros µmax (a), K (b) e Yxp (c) com diferentes valores de
estimativa inicial. Valor de referência (PRef) (linha vermelha); a partir do valor de referência (linha azul); 50% abaixo do
valor de referência (linha rosa); e 50% acima do valor de referência (linha preta). (c)
(a)
Figura 6: Cadeias de Markov com kernel normal para os parâmetros µmax (a), K (b) e Yxp (c) com diferentes valores de
estimativa inicial. Valor de referência (PRef) (linha vermelha); a partir do valor de referência (linha azul); 50% abaixo do
valor de referência (linha rosa); e 50% acima do valor de referência (linha preta).
(c)
(a)
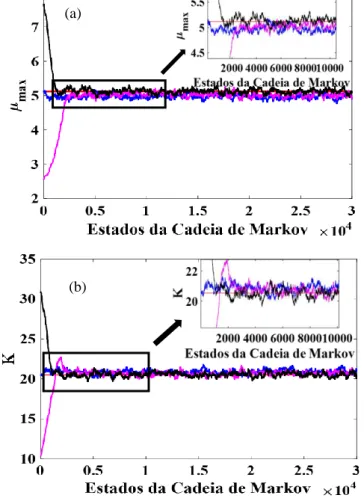
Ao utilizar o kernel de transição uniforme, 30.000 estados da cadeia de Markov foram necessários para que uma distribuição de equilíbrio pudesse ser alcançada. Bem como um período maior de aquecimento, ou seja, estados iniciais da cadeia que precedem a distribuição estacionária. Apesar disso, pode-se obter bons resultados no processo de estimação que utilizou este kernel.
A mesma análise foi realizada para o kernel que utilizou a t-student. Observou-se que as cadeias convergiram para uma distribuição de equilíbrio, sendo necessário menor número de estados de aquecimento, em torno de 2.000 de um total de 10.000, se comparado com o uniforme, que necessitou de cerca de 5.000 estados para que o aquecimento da cadeia ocorresse e o equilíbrio fosse atingido.
Apesar de a distribuição normal ser amplamente aplicada para representação da distribuição auxiliar geradora de parâmetro candidato, outras distribuições com caudas mais densas, como é o caso da t-Student, podem ser empregadas (Cabras et al., 2015). Segundo os resultados obtidos neste trabalho, o kernel que utiliza esta distribuição mostrou-se eficaz para estimar parâmetros do modelo aplicado à produção de etanol com Saccharomyces cerevisiae.
O cenário com kernel de distribuição normal, apresentou baixa taxa de aceitação de parâmetros candidatos, o que conferiu à cadeia uma convergência lenta, conforme evidenciado para o parâmetro Yxp na Figura 6. Em estudos como o de Hjorth e Vadeby (2002), é possível encontrar mais detalhes a
respeito de convergência de cadeias de Markov.
Na tentativa de contornar este efeito, analisou-se os casos com um número maior de estados da cadeia de Markov 30.000, não obtendo modificações significativas no comportamento das cadeias. Entretanto, os resultados de RMS apontaram boas aproximações entre o perfil exato em relação ao estimado, já para o menor número de estados avaliados (10.000). Ou seja, apesar da elevada taxa de
rejeição de parâmetros candidatos, os aceitos foram bons o suficiente para obter resultados satisfatórios.
Na Figura 7 são demonstrados os perfis para as variáveis de estado do estudo em questão, observou-se que todos ficaram compreendidos pelo intervalo de credibilidade de 99% (linha pontilhada). Indicam a solução exata (linha preta), obtida a partir da solução do modelo direto, a linha sólida vermelha representa a média das soluções com os parâmetros que foram estimados e o marcador representa as medidas simuladas. O ajuste dos demais perfis podem ser analisados mediante o valor do RMS
Figura 7: Média das estimativas das variáveis de estado (linha vermelha); solução exata do modelo direto (linha preta); medidas simuladas (marcador cinza) para Biomassa (a), Sacarose (b), Glicose (c) e Etanol (d). Com kernel de distribuição normal, σp = 10%, σm = 5%, N = 10.000 e intervalo de credibilidade de 99%.
As Tabelas 3 – 5 expõe todos os casos com diferentes kernels de transição avaliados neste trabalho, assim também o erro RMS como forma de verificar o quão satisfatórias foram as estimativas e a capacidade destas em representar o perfil das variáveis de estado de interesse.
(a) (b)
A Tabela 3 apresenta bons ajustes no caso em que tanto o desvio do parâmetro (σp) como o de
medida (σm) equivaliam a 5% do Pref. Tal resultado era previsto, uma vez que pequenas incertezas
representam cenários ótimos e, em alguns casos, são difíceis de serem alcançados experimentalmente.
Tabela 3: Casos analisados para kernel uniforme e erro RMS.
Est.Ini µ σp σm X S S1 P Kernel Uniforme RMS 1 Pref Pref 5% 5% 0,0028 0,1021 7,6022 1,4514 2 10% 0,2029 7,2576 2,6661 0,6992 3 10% 10% 0,0323 1,1550 9,5994 1,8370 4 1,2Pref 5% 5% 0,1269 4,5412 4,3713 1,2851 5 10% 0,2006 7,1785 6,9144 1,6942 6 10% 10% 0,0154 0,54974 2,8863 3,1031 7 0,5xPref Pref 10% 5% 0,1308 4,6812 7,9715 3,1085 8 1,5xPref 0,0192 0,6853 12,7115 3,0341
A média das soluções utilizando o valor estimado para os parâmetros aproximou-se do perfil exato obtido com a solução do modelo direto, conforme ratificado pelos baixos valores de erro mostrados pelo RMS. Entretanto o número de cadeias de Markov utilizados para que as estimativas fossem mais acuradas foi superior, 30.000 no total, quando comparado ao necessário para os demais kernels.
É possível observar que para cada perturbação ou incerteza inserida no processo de estimação, as variáveis de estado respondem de maneiras diferentes. O que torna possível selecionar qual cenário em que determinada componente de interesse possa ser favorecida em sua produção, comportamento semelhante ocorreu para os demais kernels.
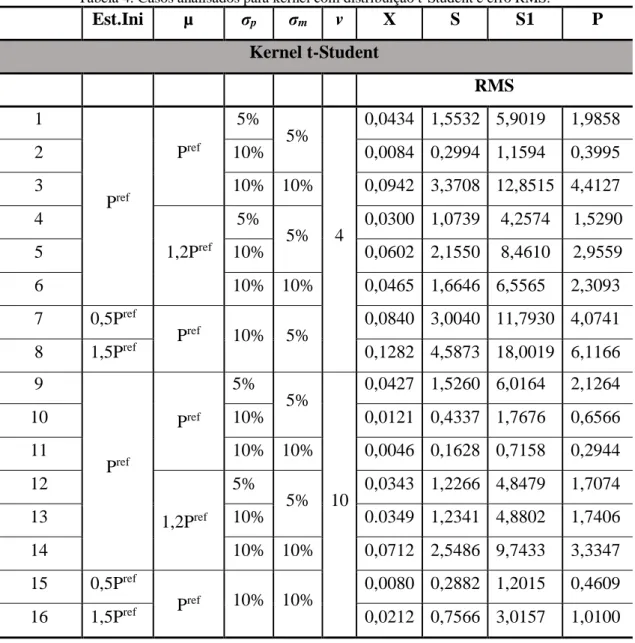
Para a construção dos casos mostrados na Tabela 4, a métrica graus de liberdade foi inserida, uma vez que a distribuição t-Student estava sendo avaliada. Entre os cenários observados para um grau de liberdade igual a 4, o caso 2 apresentou menor valor de RMS para todas as variáveis de estado.
Já para um grau de liberdade igual a 10 o caso 11 foi o que apresentou menor valor de RMS para todas as variáveis de estado. Este comportamento pode ser atribuído ao efeito que o grau de liberdade exerce sobre a cauda da distribuição t-Student, aumentando a probabilidade de que mais valores sejam selecionados, o que pode favorecer a escolha de um valor ótimo para o parâmetro candidato.
Tabela 4: Casos analisados para kernel com distribuição t-Student e erro RMS. Est.Ini µ σp σm v X S S1 P Kernel t-Student RMS 1 Pref Pref 5% 5% 4 0,0434 1,5532 5,9019 1,9858 2 10% 0,0084 0,2994 1,1594 0,3995 3 10% 10% 0,0942 3,3708 12,8515 4,4127 4 1,2Pref 5% 5% 0,0300 1,0739 4,2574 1,5290 5 10% 0,0602 2,1550 8,4610 2,9559 6 10% 10% 0,0465 1,6646 6,5565 2,3093 7 0,5Pref Pref 10% 5% 0,0840 3,0040 11,7930 4,0741 8 1,5Pref 0,1282 4,5873 18,0019 6,1166 9 Pref Pref 5% 5% 10 0,0427 1,5260 6,0164 2,1264 10 10% 0,0121 0,4337 1,7676 0,6566 11 10% 10% 0,0046 0,1628 0,7158 0,2944 12 1,2Pref 5% 5% 0,0343 1,2266 4,8479 1,7074 13 10% 0.0349 1,2341 4,8802 1,7406 14 10% 10% 0,0712 2,5486 9,7433 3,3347 15 0,5Pref Pref 10% 10% 0,0080 0,2882 1,2015 0,4609 16 1,5Pref 0,0212 0,7566 3,0157 1,0100
A Tabela 5 traz os resultados dos testes efetuados, onde houve alteração nos valores de estimativa inicial, na média e no desvio da distribuição a priori e incerteza das medidas. Mesmo com diversas alterações nesses critérios, as soluções de RMS para cada variável de estado apresentaram valores satisfatórios.
Apesar de terem sido realizadas análises para uma quantidade maior de estados da cadeia de Markov (30.000), os valores de RMS apontaram que 10.000 estados seria o suficiente para a obtenção de boas aproximações para as estimativas.
Tabela 5: Casos analisados para kernel com distribuição normal e erro RMS. Est.Ini µ σp σm X S S1 P Kernel Normal - N = 10.000 RMS 1 Pref Pref 5% 5% 0,0606 2,1694 0,6902 2,5901 2 10% 0,0196 0,7022 5,2763 0,7346 3 10% 10% 0,0346 1,2359 1,2491 2,9716 4 1,2*Pref 5% 5% 0,0057 0,2052 3,4966 0,8011 5 10% 0,0113 0,4029 0,4127 2,4306 6 10% 10% 0,0362 1,295 8,8865 15,0214 7 0,5Pref Pref 5% 5% 0,0206 0,7364 0,5054 4,3359 8 10% 0,0062 0,2215 8,8713 2,1432 9 10% 10% 0,0335 1,1975 0,7403 8,3981 10 1,5Pref Pref 5% 5% 0,0278 0,9929 1,6792 1,1832 11 10% 0,0285 1,02056 5,1892 2,7182 12 10% 10% 0,0713 2,5521 3,1967 4,6128 N = 30.000 Est.Ini µ σp σm X S S1 P RMS 1 Pref Pref 5% 5% 0,0966 3,4549 0,2552 3,6747 2 10% 0,078 2,7898 1,3295 5,1806 3 10% 10% 0,0864 3,0907 0,0439 1,7007 4 1,2Pref 5% 5% 0,0887 3,1755 2,208 3,8377 5 10% 0,0341 1,2212 4,4674 0,9511 6 10% 10% 0,0504 1,8033 3,4408 0,8036 7 0,5Pref Pref 5% 5% 0,0149 0,5319 3,6764 6,625 8 10% 0,0137 0,4919 1,0068 2,7247 9 10% 10% 0,0297 1,0639 0,5269 4,1131 10 15Pref Pref 5% 5% 0,0142 0,5066 0,8191 2,9098 11 10% 0,0707 2,5285 4,4592 2,9674 12 10% 10% 0,0444 1,5876 5,2661 2,339
A Figura 8 mostra uma representação esquemática das distribuições uniforme, t-Student e normal, aqui utilizadas.
Figura 8: Representação esquemática das distribuições de probabilidade uniforme (linha vermelha), t-Student (linha azul) e normal (linha preta).
Em resumo, os resultados permitiram observar a maneira como as distribuições aplicadas como auxiliares na etapa de geração de parâmetros candidatos afetaram o processo de estimação. O kernel com distribuição uniforme nesse estudo, apresentou maior limitação na procura de um novo parâmetro que pudesse ser aceito, sendo necessários mais estados da cadeia de Markov.
A cauda menos densa da normal, se comparada a t-Student, conferiu menor região de probabilidade onde o parâmetro candidato pudesse ser encontrado. Isto pode justificaro fato de que por vários estados a cadeia não se movia até que um novo parâmetro candidato fosse aceito.
O processo de estimação com a t-Student foi satisfatório ao atingir a distribuição estacionária sem necessitar de mais estados da cadeia de Markov. A cauda mais densa desta distribuição, possibilitou ou mesmo ampliou a área de busca na distribuição auxiliar, onde gera-se novo parâmetro candidato. Os valores 4 e 10 atribuídos à métrica graus de liberdade teveram influência significativa no peso da cauda da referida distribuição.
Apesar dos efeitos sobre os estados de aquecimento, comportamento das cadeias ou mesmo o número total de estados da cadeia de Markov, pode-se concluir que todos os kernels avaliados apresentaram resultados satisfatórios no processo de estimação, podendo ser aplicados em outros contextos de estudo. Neste em particular de produção de etanol por processo fermentativo, o kernel com distribuição t-Student pode ser destacado em relação aos demais.
6 CONCLUSÃO
O kernel de transição que utilizou a distribuição uniforme necessitou de um número maior de estados da cadeia de Markov para que a distribuição de equilíbrio pudesse ser alcançada. Já o que utilizou a t-Student alcançou a distribuição estacionária com boa mistura da cadeia de Markov, com menos estados de aquecimento e bons ajustes apontados pelo RMS.
O kernel com distribuição normal demonstrou elevada taxa de rejeição de parâmetros candidatos, o que fez com que a cadeia permanecesse vários estados sem aceitar um novo parâmetro. Tal fato atribuiu à cadeia uma característica de lenta convergência, como ficou mais evidente para o parâmetro YXP. Contudo, os resultados de RMS demonstraram bons ajustes, ou seja, apesar da
elevada taxa de rejeição os parâmetros aceitos foram bons o suficiente para alcançar aproximações satisfatórias. Nesse sentido, foi comprovado que estimativas de parâmetros do modelo aplicado em produção de etanol por Saccharomyces cerevisiae através da Técnica Bayesiana Monte Carlo via Cadeia de Markov foram obtidas com acurácia e precisão.
REFERÊNCIAS
AMADOR, I. C. B., VIEGAS, B. M., ESTUMANO, D. C., MACÊDO, E. N., RIBEIRO, N. F. Da P. Estimativa da curva de ruptura do processo de adsorção através do algoritmo de reamostragem por importância. Avanços das Pesquisas e Inovações na Engenharia Química 2. 1ed.: Atena Editora, p. 192-204, 2020.
AMADOR, I. C. B., VIEGAS, B. M, ESTUMANO, D. C., Ribeiro, N. F. P. Aplicação de filtros de partículas para estimativa de curva de ruptura do processo de adsorção. In: XXXIX Congresso Brasileiro de Sistemas Particulados, 2019, Belém. XXXIX Congresso Brasileiro de Sistemas
Particulados - ENEMP, 2019.
BAIA, R. T., LOBATO, S. L. A., OLIVEIRA, K. B., MACEDO, E. N., BRAGA, E. M., ESTUMANO, D. C. Inverse problem for estimating and optimization of parameters of batch ethanol fermentation process using Bayesian techniques. Brazilian journal of development, v. 6, p. 26496-26516, 2020. CABRAS, S., NUEDA, M. E. C., RULI, E. Approximate Bayesian computation by modelling summary statistics in a quasi-likelihood framework. Bayesian Analysis. v. 10, n. 2, p. 411-439. 2015. COUTINHO, J. P. de S., AMADOR, I. C. B., VIEGAS, B. M., RIBEIRO, N. F. da P., ESTUMANO, D. C. Aplicação do método de monte carlo via cadeia de markov para estimativa de parâmetros em modelos de curvas de ruptura. Avanços das Pesquisas e Inovações na Engenharia Química 2. 1ed.:
Atena Editora, p. 85-99, 2020.
COSTA Jr, J. M., NAVEIRA-COTTA, C. P. Estimation of kinetic coefficients in micro-reactors for biodiesel synthesis: Bayesian inference with reduced mass transfer model. Chemical Engineering
Research and Design. v. 141. p. 550-565. Nov. 2019.
DIAS, C. S., MONTEIRO, L. S., VIEGAS, B. M., ESTUMANO, D. C.; Ribeiro N.F.P . Filtro de partícula sir aplicado ao estudo da hidrólise de matérias graxas. In: XXXIX Congresso Brasileiro de Sistemas Particulados, 2019, Belém. XXXIX Congresso Brasileiro de Sistemas Particulados - ENEMP, 2019.
DIAS, C. S., MONTEIRO, L. S., VIEGAS, B. M., ESTUMANO, D. C., RIBEIRO, N. F. Da P. Estimativa de variáveis de estado em modelo de hidrólise de matérias graxas. Avanços das Pesquisas e Inovações na Engenharia Química 2. 1ed.: Atena Editora, p. 44-58, 2020.
DODIĆ, J. M., VUČUROVIĆ, D. G., DODIĆ, S. N., GRAHOVAC, J. A., POPOV, S. D., NEDELJKOVIĆ, N. M. Kinetic modelling of batch ethanol production from sugar beet raw juice,
Applied energy, v. 99. p. 192-197. Jun. 2012.
EL-DALATONY, M. M.; KURADE, M. B., ABOU-SHANAB, A. I. KIM, H., SALAMA, E. S., JEON, B.H. Long-term production of bioethanol in repeated-batch fermentation of microalgal biomass using immobilized Saccharomyces cerevisiae, Bioresource technology. v. 219. p. 98 – 105. Jul. 2016.
ESTUMANO, D. C., HAMILTON, F. C., COLACO, M. J., LEIROZ, A. J., ORLANDE, H. R. B., CARVALHO, R. N., DULIKRAVICH, G. S. Bayesian estimate of mass fraction of burned fuel in internal combustion engines using pressure measurements. Engineering Optimization IV, p. 997-1003, 2014.
FERREIRA, J. L., ESTUMANO, D. C., SOUZA, M. de M. V. M., MACÊDO, E. N. Application of the markov chain monte carlo method to estimation of parameters in a model of adsorption-enhanced reaction process for mercury removal from natural gas. Impactos das Tecnologias na Engenharia Química 3. 3ed.: Atena Editora, p. 315-321, 2019.
FIRTH, S. K., LOMAS, K. J., WRIGHT, A. J. Targeting household energy-efficiency measures using sensitivity analysis, Building Research & Information, v. 38. n. 1. p. 25 – 41. 2010.
GALAGALI, N.; MARZOUK, Y.M. Bayesian inference of chemical kinetic models from proposed reactions, Chemical Engineering Science, v. 123. p. 170–190. 2014.
GAMERMAN, D. Simulação Estocástica via Cadeias de Markov. 1. ed. São Paulo: ABE, 1996 GELMAN, A.; CARLIN, J.B.; RUDIN, D.B.; Bayesian data analysis. 2 ed. Nova York: CRC, 2004. HASTINGS, W.K., Monte Carlo Sampling Methods Using Markov Chains and Their Applications.
Biometrika. v. 57, pp.97-109, 1970.
HJORT, U., VADEBY, A. The empirical KL-measure of MCMC convergence. 2013.
IZMIRLIOGLU, G., DEMIRCI, A. Ethanol Production from Waste Potato Mash by Using
Saccharomyces Cerevisiae. v. 2. p. 738 – 753. Out. 2012.
JAYAWARDHANA, B., KELL, D. B., RATTRAY, M. Bayesian inference of the sites of perturbations in metabolic pathways via Markov chain Monte Carlo. Bioinformatics. v. 24. n.9. p. 1191-1197. Mar. 2008.
KIRSCH, P., ESSLINGER, C., CHEN, Q., MIER, D., LIS, S., SIDDHANTI, S., MEYER-LINDENBERG, A. Oxytocin modulates neural circuitry for social cognition and fear in humans.
Journal of Neuroscience, v. 25. n. 49, p. 11489-11493. 2005.
METROPOLIS, N., ROSENBLUTH, A. W., ROSENBLUTH, M. N., TELLER, A. H.,
TELLER, E. Equations of state calculations by fast computing machines. J. Chem. Phys. v.21. pp.1087-1092, 1953.
MOURA, C. H. R. de, ESTUMANO, D. C., QUARESMA, J. N. N., MACÊDO, E. N., VIEGAS, B. M., MONTEIRO, L. S., LOPES, D. S. Aplicação da técnica de monte carlo via cadeia de markov para estimativa de parâmetros de modelos de balanço populacional para sistemas particulados. Avanços das Pesquisas e Inovações na Engenharia Química 2. 1ed.: Atena Editora, p. 59-72, 2020a.
MOURA, C. H. R. de, ESTUMANO, D. C., QUARESMA, J. N. N., MACÊDO, E. N., VIEGAS, B. M., MONTEIRO, L. S., LOPES, D. S. Aplicação da técnica de monte carlo via cadeia de markov para estimativa de parâmetros em modelo de balanço populacional de cristalização de gibbsita com cinética constante. Avanços das Pesquisas e Inovações na Engenharia Química 2. 1ed.: Atena
Editora, p. 73-84, 2020b,
MONTEIRO, L. S., DIAS, C. S., MOURA, C. H., VIEGAS, B. M., ESTUMANO, D. C., Ribeiro N.F.P . Estimativa de parâmetros da hidrogenação por transferência catalítica do óleo de soja via mcmc. In: XXXIX Congresso Brasileiro de Sistemas Particulados, 2019, Belém. XXXIX Congresso
Brasileiro de Sistemas Particulados - ENEMP, 2019.
NAVEIRA-COTTA, C.P., Problemas Inversos de Condução de Calor em Meios Heterogêneos: Análise Teórico-Experimental Via Transformação Integral, Inferência Bayesiana e Termografia por Infravermelho. (Doutorado em Engenharia Mecânica). Rio de Janeiro, 2009.
OCHOA, M. P., ESTRADA, V., MAGGIO, J. DI, HOCH, P. M. Dynamic global sensitivity analysis in bioreactor networks for bioethanol production, Bioresource Technology, v. 200. p. 666 – 679. Out. 2016.
ÖZISIK, M.N., ORLANDE, H.R.B., Inverse Heat Transfer, New York: Taylor & Francis, 2000.
PASQUALETTE, M. A., ESTUMANO, D. C., HAMILTON, F. C., COLAÇO, M. J.,
SILVA, C. L., SANTANA, P. L., SILVA, C. F., PAGANO, R. L. Modelagem e estimação de parâmetros do processo de produção de etanol em reator batelada por Saccharomyces cerevisiae.
Scientia Plena. v. 12. n. 5. 2016.
SUTTON, J. E., GUO, W., KATSOULAKIS, M. A., VLACHOS, D. G. Effects of correlated parameters and uncertainty in electronic-structure-based chemical kinetic modelling. Nature