RÉGIA MARIA FELTRIN DAMBROS
AMPLIFICAÇÃO, CLONAGEM E EXPRESSÃO DE PROTEÍNA
RECOMBINANTE DO VÍRUS DA DOENÇA DE AUJESZKY
EM SISTEMA DE BACULOVÍRUS PARA UTILIZAÇÃO
EM PROGRAMA DE CONTROLE E ERRADICAÇÃO
LAGES – SC
UNIVERSIDADE DO ESTADO DE SANTA CATARINA – UDESC
CENTRO DE CIÊNCIAS AGROVETERINÁRIAS - CAV
MESTRADO EM CIÊNCIAS VETERINÁRIAS
RÉGIA MARIA FELTRIN DAMBROS
AMPLIFICAÇÃO, CLONAGEM E EXPRESSÃO DE PROTEÍNA
RECOMBINANTE DO VÍRUS DA DOENÇA DE AUJESZKY
EM SISTEMA DE BACULOVÍRUS PARA UTILIZAÇÃO
EM PROGRAMA DE CONTROLE E ERRADICAÇÃO
Dissertação apresentada ao Curso de Pós-graduação em Ciências Veterinárias, da Universidade do Estado de Santa Catarina, como requisito parcial para obtenção do grau de Mestre em Ciências Veterinárias.
Orientadora: Profª. Drª. Janice Reis Ciacci
Zanella
RÉGIA MARIA FELTRIN DAMBROS
AMPLIFICAÇÃO, CLONAGEM E EXPRESSÃO DE PROTEÍNA
RECOMBINANTE DO VÍRUS DA DOENÇA DE AUJESZKY
EM SISTEMA DE BACULOVÍRUS PARA UTILIZAÇÃO
EM PROGRAMA DE CONTROLE E ERRADICAÇÃO
Dissertação apresentada ao Curso de Pós-graduação em Ciências Veterinárias, da Universidade do Estado de Santa Catarina, como requisito parcial para obtenção do grau de Mestre em Ciências Veterinárias.
Banca Examinadora:
Orientadora: _____________________________________________ Profª Drª Janice Reis Ciacci Zanella
EMBRAPA Suínos e Aves Concórdia SC
Co-orientadora: _____________________________________________ Profª Drª Eliana Knackfuss Vaz
CAV – UDESC Lages SC
Membro: _____________________________________________ Profª Drª Vânia Helena Techio
UnC Concórdia SC
AGRADECIMENTOS
A minha orientadora, Drª Janice Reis Ciacci Zanella, que por confiar no meu trabalho, permitiu que eu desse esse grande passo e pudesse crescer sob tantos aspectos. Considero-a um ser humano fantástico, sempre pronta para o apoio didático, mas sobretudo sempre aberta para as lamentações e por reconfortar com palavras de incentivo. E a grande verdade, “que tudo vai dar certo ao seu tempo, cada coisa na sua vez”. Obrigada pela orientação, confiança, oportunidade e amizade.
Agradecer a minha co-orientadora, Drª Eliana Knackfuss Vaz, pelo apoio e suporte técnico nas inúmeras vezes que solicitamos sua ajuda. Agradecer pelo empréstimo das literaturas, pelas excelentes ‘dicas’, sempre nos alertando dos entraves a serem vencidos, e pelo exemplo de profissional dedicado e extremamente capacitado. Muito abrigada.
Aos professores do CAV-UDESC, em especial aos da pós-graduação, por me fazerem descobrir que ainda sou capaz de aprender. Obrigada por tudo, em especial pela paciência e pela amizade.
Agradeço a coordenação do curso de pós-graduação, na pessoa do professor Dr. Alceu Mezzalira e atualmente na do professor Dr. Aury Nunes de Moraes e em seus nomes agradecer ao colegiado e funcionários da pós-graduação e a toda direção do CAV-UDESC de Lages.
Aos colegas e amigos encontrados durante o período em que fiquei em Lages. Vocês moram em meu coração e sempre passeiam em meus pensamentos.
Aos professores Sandra Ferraz Catagna (com quem tive o prazer de morar por alguns dias) e Ubirajara Costa (membro suplente de nossa banca), competentes profissionais, mas sobretudo pessoas maravilhosas que nos contagiam com sua garra e semblantes sempre felizes.
Agradecer aos membros da minha banca de avaliação da dissertação, à Drª Janice Zanella (minha orientadora), à Drª Eliana K. Vaz (minha co-orientadora) e a Drª Vânia H. Techio (membro externo da banca) da UnC de Concórdia. Vocês contribuíram imensamente com suas avaliações, questionamentos, sugestões e correções para a melhoria da produção escrita dos trabalhos realizados e para o nosso crescimento científico-profissional. Muito obrigada.
Em Lages, tive a oportunidade de ser vizinha da família da Gorete. Família maravilhosa que me fez inúmeros favores e sempre estava pronta a me socorrer. Muito obrigada, Gorete. Sua família tem um lugar bem especial em meu coração.
Tive o privilégio de encontrar uma amiga-irmã em Lages. É interessante como as pessoas se cruzam, como as afinidades se atraem ou até pelas diferenças. Nossa diferença de idade me fez tua irmã de coração mais velha, entretanto eras tu que por muitas e muitas vezes, me escutaste e me viste chorar de saudades de meus filhos, do meu lar. Encontrei amparo em tua família. A adorável Carol (minha amiguinha), em teu filho Alan, em tua mãe (dona Odete) e em ti, querida amiga. Os fatos que se sucederam, as fatalidades que ocorreram. Sei lá! Será que tudo isso já estava escrito? Olha! Tenho em ti um sincero carinho e uma eterna amiga. Muito obrigada por tudo, querida amiga e irmãzinha, Carine Rüsche.
Agradeço ao professor Dr. Leonardo José Richtzenhain, VPS/FMVZ/USP (Lab. Biologia Molecular Aplicada e Sorologia da USP), que além do profissional e pesquisador de renome, sempre demonstra ser um ser humano com um coração enorme e de uma dedicação para com os trabalhos e com as pessoas sem igual. Obrigada pelo incentivo e principalmente por acreditar na nossa capacidade de trabalho.
Sou grata ao destino, pois através dele, encontrei a Simone (Perecmanis) e por seu intermédio conheci o professor Bergmann.
6
À pessoa fantástica, de uma sabedoria e serenidade imensa, por tudo que me ajudou. Pela oportunidade de participar do seu curso em biologia molecular e de trabalhar em seu laboratório. De nos acolher de forma tão despretensiosa, estendendo uma mão tão profissional e tão amiga, nos fazendo sentir em casa. Pela paciência para me ensinar e poder em seu laboratório ter conseguido desenvolver uma das etapas mais importantes deste trabalho que ora finalizo. Muito obrigada, prof. Bergmann Morais Ribeiro, e em seu nome agradeço a todos professores do Laboratório de Microscopia Eletrônica e Virologia do Departamento de Biologia Celular da Universidade de Brasília (DF).
Um abraço bem grande e toda minha gratidão a todos do Laboratório de Microscopia Eletrônica e Virologia da UnB (DF) que me ajudaram em todos os aspectos durante minhas estadas em Brasília. Um forte abraço aos colegas/amigos do laboratório, a Susane, a Ana Maria, ao Roberto, ao Marcelo (Spicer man), a todos os ‘meninos’, a Mariana, a Thati, a Maria, a Danielle, a Érica, a Aline e a tantos outros não mencionados aqui, meu muito obrigada. Minha eterna gratidão.
Toda minha admiração pela obstinação e coragem ao meu braço direito no laboratório da virologia molecular da UnB Raimundo Wagner. Sou muito grata por tudo que me ensinou e ajudou. Pela presença constante no laboratório, de segunda a segunda, do início da manhã até noite adentro para vencermos as etapas de trabalho. Muito, muito obrigada.
Agradecer a Nayara (UnB) que sempre estava pronta a me socorrer, ser humano generoso e acolhedor, de um coração imenso. Tecnicamente não preciso nem citar todas as suas qualificações. Parabéns pela tua garra e que Deus te mantenha assim tão especial, tão comprometida em ajudar o mundo a ser melhor.
Aos colegas (e muitos amigos) do laboratório de virologia da EMBRAPA Suínos e Aves: a Marisete, a Tânia, ao seu Nene (que Deus permita que retorne em breve com muita saúde), a Magda, a Neide, ao ex-bolsista Kleithon, a bolsista Michele, ao Marcelo (o Japa), a Danielle (a Gava) e a Lauren.
Agradecer a todos do laboratório de bacteriologia e micoplasma da EMBRAPA Suínos e Aves, em especial aos pesquisadores Laurimar, Cátia, Virgínia e Jalusa. Agradecer a Marni (que por inúmeras vezes me auxiliou e me ensinou), ao Bordin, ao Vizotto (quantas vezes você me ajudou com os preparos dos reagentes, pH e tudo mais?), a Susana (da mesma forma me auxiliou muitíssimo com os géis de poliacrilamida), a Beatriz, a Daiane e a todos os bolsistas e estagiários.
Meus sinceros agradecimentos a todos do laboratório de sanidade: a Fran, ao Altair, ao Djalmo, ao Maximino, ao Gerson, a Salete, ao Alexandre, a Sílvia, ao Armando, ao Dhamer, ao Idelsino, a Maria, ao Jair e a tantos outros que não citei ou que já estão em outras pradarias.
Obrigada aos pesquisadores do laboratório de sanidade da EMBRAPA Doralice, Carlos Costa, Mônica, Fátima e Morés.
Um abraço especial para a pesquisadora Fátima, minha amiga desde 1991, dos bons tempos das aulas de inglês, lembra? Como é revitalizante as boas gargalhadas que damos juntas. A gente ri à toa. Rimos por qualquer motivo e isso é muito bom. Obrigada.
Aos amigos Lauren, Marisete, Marcelo, Tânia, Michele e Neide que além de todo suporte profissional me apoiaram em tudo. Obrigadaaaaa!!! Foi muito bom tê-los por perto, sem a sua colaboração muito, com certeza, não teria sido alcançado. E tudo foi bom. Os momentos de trabalho, os intervalos para o café, as quintas-feiras com os lanches da Leoni, e muito, muito trabalho.
Um agradecimento especial à Neide, que foi meu braço direito e muitas vezes o esquerdo. Ajudou-me sob inúmeros aspectos. Você é uma pessoa maravilhosa e uma profissional de muita competência, apesar de sua pouca idade. Pois é “Alemoa”, você faz idéia de quanto me ajudou? E ainda por cima fazíamos brincadeiras, fazendo-a ficar zangada e vermelhinha? Ah! São essas coisas e principalmente as pessoas que fazem a vida ser tão especial. Muito abrigada!
Obrigada a você, Marcelo (Japa), sobretudo pela paciência para me explicar o que me parecia tão difícil e complicado, socorrendo-me com dicas fantásticas na área de informática, nas dúvidas com a PCR, técnicas recombinantes e outros testes da bio-molecular. (E lembre que está devendo ‘aquela’ costela de gado assada no forno).
pessoas. Foi muito bom ter você por perto. Sempre incentivando e se colocando à disposição. Foram muitos papos legais e muitas gargalhadas gostosas. Às vezes ríamos do nada, já em outras, em virtude de meus ‘achados espetaculares’. Obrigada pelas palavras reconfortantes e pelo incentivo constante.
Aos funcionários do laboratório CEDISA: a Eliane, a Franciele, a Nara, a Andréia, a Marna, a Keli, a Loni, a Simaia, a Tatiane, a Áuria e a Kellen. Quero reforçar aqui a imensa importância que vocês tiveram para que hoje eu alcance este objetivo. Com certeza vocês me ajudaram em muito.
A você Áuria, cara amiga, só agradecer e agradecer. Saiba que você é exemplo de sucesso. Que com tudo que a vida ofereceu, você transformou em uma saborosa e refrescante limonada. Pessoa batalhadora e uma profissional dedicada e comprometida com a qualidade do produto final.
Aos colegas da CIDASC, técnicos e amigos do laboratório CEDISA Ricardo Volcato, José Luiz Marques e Helder Machado (atualmente em área administrativa), nosso gerente regional da CIDASC de Concórdia (SC). Pessoas que comprometidas com o nosso objetivo, não mediram esforços em permitir que pudéssemos nos afastar de nossa bancada de trabalho para poder desenvolver este projeto profissional e, sobretudo de vida. Muito obrigada.
A CIDASC, aqui representada pelo presidente da empresa, Sr. Vilmar Carelli e atualmente Dr. Hamilton Ricardo Farias e pelo comitê de Pós–graduação, por ter aprovado meu projeto de pós-graduação e ter confiado no meu potencial. Com certeza retornarei um pouco melhor e cheia de vontade de ajudar, de colaborar com o engrandecimento de nossa empresa. Muito obrigada.
Agradeço a EMBRAPA Suínos e Aves de Concórdia por ter me recebido tão bem e me oportunizado desenvolver meu projeto de mestrado. Agradecer na pessoa do chefe geral Dr. Élsio Figueiredo, e em seu nome estender os agradecimentos à toda a direção e funcionários. Muito obrigada.
Aos meus irmãos, Rosmary de Fátima (a Fata), Robson José (o Bob), Rosane Aparecida (a Ane), Ricarda Malena (a Kada), Rosilda Helena (a Zida) e o Renan André (Renan), nosso meio-irmão e meu afilhado. É de vocês que busco referências. Obrigada por compartilharem comigo de toda essa história.
E quase por fim, à minha família. José Aníbal, minha outra metade. Marina, meu anjo da guarda e José Raul, criativo e cheio de pegadinhas, me faz rir com freqüência. Obrigada! Vocês representam o tudo de bom, o sol do amanhecer, a chuva do entardecer e a brisa de viver. Vocês simplesmente são tudo para mim.
Ao responsável por tudo isso. O dono de toda obra. Muito obrigada por permitir que eu faça parte da sintonia universal. De ter encontrado momentos e pessoas tão maravilhosas. Por ter me mostrado que é na humildade que encontramos os maiores sábios, que na serenidade se obtém o oceano cheio de respostas e que todas as qualificações estão exemplificadas na vivência do homem, o ser racional, com o livre arbítrio.
“Se vi mais longe do que os outros, é porque estava apoiado nos ombros de gigantes”.
ISAAC NEWTON
RESUMO
A doença de Aujeszky (DA) é uma enfermidade infecto-contagiosa que causa graves prejuízos econômicos ao produtor e à agroindústria suinícola. Com o objetivo de desenvolver insumos e aprimorar tecnologias que sejam mais rápidas, sensíveis e específicas de diagnóstico para uso em regiões livres ou em erradicação da DA, a seqüência codificadora da glicoproteína E (gE) do vírus da doença de Aujeszky (VDA) foi amplificada, clonada e expressada. Através da engenharia genética a seqüência do gene da gE foi propagada em um organismo hospedeiro. A gE foi amplificada pela técnica da reação em cadeia da polimerase (PCR) , clonada no vetor pGem®-T Easy e transformada em células competentes de Escherichia coli, DH-5α™. O clone obtido foi subclonado no plasmídeo de expressão pFastBac™1, o qual possui o sítio promotor do gene da poliedrina. O subclone obtido foi analisado para certificação de sua orientação correta dentro do plasmídeo com as endonucleases de restrição BamH I e EcoR I. O subclone com a orientação correta teve seu DNA extraído e usado para a transposição dentro do “bacmid” (baculovírus recombinante e plasmídeo “helper” em célula competente DH10Bac™). As colônias com inserto gE foram selecionadas pelo fenótipo da colônia, a qual expressa cor branca quando clonada. As colônias brancas recombinantes tiveram seu DNA extraído e usado para a co-transfecção em células do inseto Trichoplusia ni (BTI-Tn5B1-4). O baculovírus gE-recombinante ao ser inoculado em cultivo celular, expressou a gE recombinante, comprovada pela técnica de PCR e “Western blotting”. O baculovírus gE-recombinante contendo apenas o gene da gE do VDA será utilizado para produção de antígeno e de anticorpos monoclonais, o que auxiliará no desenvolvimento de um teste de diagnóstico mais sensível, específico e mais seguro para uso em áreas livres do VDA.
ABSTRACT
Aujeszky’s disease (AD) is an infect-contagious illness that causes serious economical damages to the producer and the swine industry. Aiming to develop mechanisms and to improve technologies that are faster, more sensitive and more specific for diagnosis and for use in free areas or in AD’s eradication programs, the sequence codifier of the glycoprotein E (gE) of Aujeszky’s disease virus (ADV) was amplified, cloned and expressed. Through genetic engineering the sequence of the gE gene was propagated in an host organism. The gE was amplified by the technique of polimerase chain reaction (PCR), cloned in the vector pGem®-T Easy and transformed in competent cells of Escherichia coli, DH-5α™. The clone obtained was sub-cloned in the expression plasmid pFastBac™1, which contains the promoter gene of the polyhedrin. The obtained subclone was analyzed inside for certification of its correct plasmid orientation with the restriction endonucleases BamH I and EcoR I. The sub-clone with the correct orientation had its DNA extracted and used for transposition inside the bacmid (recombinant baculovirus and “helper” plasmid with competent DH10Bac™ cell). Colonies with inserted gE were selected by the phenotype of the colony, which expresses white color when cloned. White recombinant colonies had their DNA extracted and used for cotransfection in insect cells Trichoplusia ni (BTI-Tn5B1-4). The recombinant-gE baculovirus was inoculated in cultured cells and expressed the recombinant gE by PCR and “Western blotting”. The recombinant-gE baculovirus containing only the gE gene of the VDA will be used for antigen and monoclonal antibodies production, which will aid in the development of a more sensitive, specific and safer for the use in VDA free regions.
LISTA DE ILUSTRAÇÕES
Figura 01 - Mapa da organização do genoma VDA... 31
Figura 02 - Nucleocapsídeo típico de herpesvírus………... 32
Figura 03 - Partícula de herpesvírus... 33
Figura 04 - Representação esquemática do ciclo de replicação do VDA... 35
Figura 05 - Representação esquemática de tipos genômicos do VDA (I, II e III) com base na migração dos três primeiros fragmentos obtidos com BamH I... 40
Figura 06 - Cultura de células BTI-Tn5B1-4 infectadas como vírus AcMNPV selvagem... 44
Figura 07 - Monocamada de cultura celular do inseto BTI-Tn-5B1-4... 47
Figura 08 - Diferentes fases do ciclo de vida da Trichoplusia ni………. 48
Figura 09 - Esquema da produção da glicoproteína gE do VDA... 53
Figura 10 - DNA genômico do vírus da doença de Aujeszky... 80
Figura 11 - DNA genômico do VDA digerido com BamH I... 80
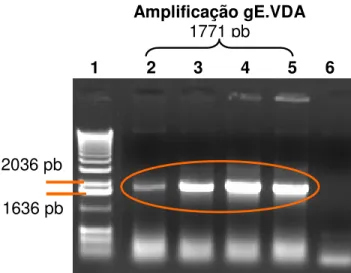
Figura 12 - Gel de agarose 0,8% da PCR gE.VDA... 81
Figura 13 - Teste da qualidade e concentração do DNA gE.VDA purificado e do plasmídeo pGem®-T Easy... 83
Figura 14 - DNAs da minipreparação da clonagem pGem®-T Easy com a seqüência codificadora gE do VDA... 84
Figura 15 - Digestão de DNAs plasmideais transformantes com EcoR I ... 86
Figura 17 - Teste da qualidade e da concentração de DNA do fragmento gE. VDA recuperado e purificado... 88 Figura 18 - Digestão do DNA gE.VDA com a enzima Bsr I em gel de agarose 2%.
88 Figura 19 - Simulação mapa de restrição da glicoproteína E com os dois
“primers” com sítios de restrição EcoR I e BamH I... 89 Figura 20 - Gel de agarose 0,8% de amostras de DNA daminiprepreparação
dos subclones recombinante pFastBac-gE.VDA... 90 Figura 21 - Gel de agarose 0,8% do DNA plasmideal recombinante
pfastBac-gE.VDA... 92 Figura 22 - Teste da qualidade e concentração do DNA plasmideal
recombi-nante pFastBac -gE.VDA em gel de agarose 1%...
93
Figura 23 - Análise em gel de agarose 0,5% da minipreparação de DNAs
“bacmid” da transposição... 96 Figura 24 - Gel de agarose das reações da PCR com amostras de DNAs de
colônias da transposição dos plasmídeos recombinantes pFastBac -gE.VDA com o “bacmid”... 97 Figura 25 - Células Trichoplusia ni BTI-TN5B1-4 infectadas com o vírus
AcMNPV selvagem... 98 Figura 26 - Células Trichoplusia ni BTI-Tn-5B1-4 infectadas com o vírus
recombinante bacmid-pFastBac-gE.VDA... 199 Figura 27 - Gel de agarose das reações de PCR com amostras de DNA dos
vírus recombinantes “bacmid” da co-transfecção... 100 Figura 28 - Gel SDS-PAGE da co-transfecção dos vírus recombinantes bac-
mid. pFastBac-gE.VDA... 101 Figura 29 - “Western blotting” de vírus recombinantes bacmid.pFastBacgE.
VDA da co-transfecção... 102 Figura 30 - Gel SDS-PAGE de infecção transiente do vírus recombinante
bacmid.pFastBac-gE.VDA...
103 Figura 31 - “Western blotting” de vírus recombinantes bacmid.pFastBac-gE.
VDA da infecção em células de inseto... 104 Figura 32 - “Western blotting” de recombinantes bacmid.pFastBac-gE.VDA da
infecção em células de inseto... 105 Figura 33 - Análise por “Western blotting” do extrato celular da produção de
LISTA DE ABREVIATURAS
α Alfa
aa Aminoácido
AcMNPV Autographa californica nuclear polyhedrosis virus (baculovírus) APS Persulfato de amômia
β Beta
bacmid.pFastBac-gE.VDA Vírus recombinante bacmid-plasmídeo com a gE do VDA BHV-1 Herpesvírus bovino tipo 1
BV “Budded virus” (vírus que brota extracelular)
CIAP “Calf Intestinal Alkaline Phosphatase” (fosfatase alcalina) cm2 Centímetro quadrado
D Dálton
DMSO Dimetil sulfóxido de sódio DNA Ácido desoxirribonucléico dNTP Deoxinucleotídeos trifosfatos D.O. Densidade ótica
ECP Efeito citopático
EDTA Ácido etilediamino tetracético EHV-1 e 4 Herpesvírus eqüino tipo 1 e 4
g Glicoproteína gD Glicoproteína D gE Glicoproteína E gI Glicoproteína I gM Glicoproteína M
gP I Glicoproteína gP I (antigo nome da gE) gx Força da gravidade
º Graus
ºC Graus Celsius ou centígrados
HSPG “Heparan Sulfate Proteoglycan” (receptor celular) HSV-1 Herpes simplex vírus tipo 1
I% Percentagem de inibição IF Imunofluorescência Ig Imunoglobulina
IPTG Isopropiltio-β-D-Galactoside IRS Seqüência Repetida Interna k Kilo (103)
kbp kilo pares de base kDa kilo Dáltons
LB Meio de crescimento bacteriano "Luria – Bertani"
LMP “Low Melting Point” (gel de agarose de baixo ponto de aquecimento) µ Micro (10-6)
M Molar ou molaridade (mol/L) mA miliamperes
mL Mililitro mM Milimolar
MOI Multiplicidade de infecção
MOPS 3-[N-N orpholino] propanesulfonic acid η Nano (10-9)
nm Namômetro
OB “Occlusion body” (corpo de oclusão = poliedro)
occ Corpos oclusos de poliedro do baculovírus (múltiplos e grandes poliedros) ORF “Open Reading Frame” (fase aberta de leitura)
OV “Occluded virus” (vírus ocluso: corpos de oclusão poliédricos) PBS Tampão salina com fosfatos
PCR “Polimerase Chain Reaction” (reação em cadeia da polimerase) PS “Primer sense” (Iniciador “forward”)
pFastBac-gE.VDA Plasmídeo recombinante de expressão com a gE do VDA pfu Unidade formadora de placa
pGem-gE.VDA Plasmídeo recombinante de clonagem com a gE do VDA pH Potencial hidrogeniônico
pmol Picomoles polh Poliedrina
PA “Primer antisense” (Iniciador reverso) % Porcentagem
RI Seqüência Repetida Interna (região repetida invertida interna do genoma viral) RFLP “Restriction Fragment Lenght Polimorfism” (Análise por polimorfismo de
tamanho determinado por perfil de restrição enzimática) rpm Rotações por minuto
RNA Ácido ribonucléico
RT Seqüência Repetida Terminal (região repetida invertida terminal do genoma viral) SDS-PAGE Sódio Duodecil Sulfato – Eletroforese em Gel de Poliacrilamida
SFB Soro fetal bovino
SK-6 “Swine Kidney” (células de linhagem, fibroblastos de rim suíno) SPF “Specific Patogen Free”
Taq “Thermus aquaticus”
TCID50 Dose infectante para 50% dos cultivos celulares Tris Tris (hidroximetil) aminometano
TRS Seqüência Repetida Terminal
UL “Unique Long” (região única longa do genoma viral) US “Unique Short” (região única curta do genoma viral) UV Ultravioleta
VDA Vírus da doença de Aujezsky VZV Vírus da varicella-zoster
SUMÁRIO
INTRODUÇÃO ... 24
1 REVISÃO DE LITERATURA... 28
1.1 HERPESVÍRUS... 28
1.2 HERPESVÍRUS SUÍNO TIPO 1 (VDA)... 29
1.2.1 Genoma... 30
1.2.2 Capsídeo... 31
1.2.3.Tegumento... 32
1.2.4 Envelope... 32
1.2.5 Glicoproteínas... 33
1.2.6 Ciclo de replicação do VDA... 34
1.2.7 Latência... 35
1.3 DETECÇÃO DO AGENTE INFECCIOSO OU DE ANTICORPOS ANTI VDA... 36
1.4 CLONAGEM E EXPRESSÃO... 41
1.4.1 Baculovírus... 42
1.4.1.1 Infecção e replicação viral... 44
1.4.1.2 Expressão gênica em baculovírus... 44
1.4.1.3 Baculovírus como vetor de expressão... 45
1.4.2 Cultura de células de inseto... 47
1.4.3 Poliedrina, promotor e lacZ ... 48
1.4.4 Plasmídeos e transposons... 49
2 MATERIAIS E MÉTODOS... 53
2.1 CÉLULAS... 54
2.2 AMOSTRAS DE VÍRUS DA DOENÇA DE AUJESZKY... 54
2.3 EXTRAÇÃO DE DNA GENÔMICO VIRAL... 54
20
2.4 REAÇÕES EM CADEIA DA POLIMERASE (PCR)... 56 2.4.1 Desenho dos iniciadores da seqüência do gene gE do VDA... 56 2.4.2 Padronização do protocolo da PCR para amplificar a seqüência
codificadora da gE do VDA... 56 2.4.2.1 Purificação das reações da PCR que amplificaram a seqüência
codificadora gE do VDA... 57 2.5 PREPARO DE CÉLULAS COMPETENTES... 57 2.5.1 Preparo de células competentes E. coli DH5α™ com cloreto de cálcio
(CaCl2)... 58 2.5.2 Preparo de células competentes E. coli DH5α™ com cloreto de rubídio
(RbCl2)... 59 2.6 TRANSFORMAÇÃO DE CÉLULAS COMPETENTES DH5α™ – CLONA
GEM E SUBCLONAGEM... 60 2.6.1 Ligação inserto gE.VDA com o plasmídeo pGem®-T Easy e clonagem
por transformação das células competentes... 60 2.6.1.1 Isolamento do DNA plasmideal recombinante pGem-gE.VDA por mini
preparação... 62 2.6.1.2 Análise do DNA plasmideal recombinante pGem-gE.VDA com enzi-
mas de restrição... 63 2.6.1.2.1 Liberação do fragmento gE.VDA do DNA plasmideal recombinante
pGem-gE.VDA... 63 2.6.1.2.2 Digestão do fragmento gE.VDA com a endonuclease de restrição
Bsr I e análise eletroforética do fragmento gE digerido... 64 2.6.2 Subclonagem do fragmento gE.VDA e do plasmídeo de expressão
pFastBac™1…... 64 2.6.2.1 Digestão do plasmídeo de expressão pFastBac™1... 65 2.6.2.2 Ligação do fragmento gE.VDA com o plasmídeo de expressão pFast-
Bac™1 linearizado e subclonagem por transformação das células competentes... 65 2.6.2.2.1 Isolamento do DNA plasmideal recombinante pFastBac-gE.VDA
por minipreparação... 66 a) Análise do DNA plasmideal recombinante pFastBac-gE.VDA com enzimas
de restrição... 66 2.7 TRANSPOSIÇÃO DO DNA PLASMIDEAL RECOMBINANTE pFAST-
BAC-gE.VDA COM O BACULOVÍRUS RECOMBINANTE “BACMID” ATRAVÉS DA TRANSFORMAÇÃO DE CÉLULAS COMPETENTES E. coli DH10BAC™... 67 2.7.1 Transposição do DNA plasmideal recombinante pFastBac-gE.VDA por
21
2.7.1.1 Seleção dos recombinantes bacmid.pFastBac-gE.VDA pelo fenótipo das colônias bacterianas……….. 68 2.7.1.2 Isolamento do DNA viral recombinante bacmid.pFastBac-gE.VDA de
colônias bacterianas DH10Bac™ transformadas – Isolamento de plasmídeos grandes... 69 2.7.1.3 Análise do DNA viral recombinante bacmid.pFastBac-gE.VDA isolado
de colônias bacterianas DH10Bac™ transformadas... 70 2.7.1.3.1 Análise do DNA viral recombinante bacmid.pFastBac-gE.VDA por
eletroforese em gel de agarose... 70 2.7.1.3.2 Análise do DNA viral recombinante bacmid.pFastBac-gE.VDA pela
técnica da PCR... 70 2.8 CO-TRANSFECÇÃO DE CÉLULAS DE INSETO COM O DNA VIRAL
RECOMBI-NANTE BACMID.pFASTBAC-gE.VDA... 70 2.8.1 Preparo das células do inseto Trichoplusia ni em placas de
cultivo... 70 2.8.2 Preparo do DNA viral recombinante bacmid.pFastBac-gE.VDA... 71 2.8.3 Co-transfecção em células de inseto do DNA recombinante bacmid.
pFast Bac-gE.VDA com lipofectina... 71 2.8.3.1 Isolamento do DNA viral recombinante bacmid.pFastBac-gE.VDA
das co-transfecções... 72 2.8.3.1.1 Análise do DNA viral recombinante bacmid.pFastBac-gE.VDA das
co-transfecções pela técnica de PCR... 73 2.8.4 Infecção de culturas celulares de inseto com vírus recombinante
bacmid.pFastBac-gE.VDA para avaliação dos níveis de expressão e tempo de infecção... 73 2.8.5 Preparo das amostras de células co-transfectadas e infectadas com
vírus recombinante bacmid.pFastBac-gE.VDA... 74 2.9 ANÁLISE DA EXPRESSÃO DA GLICOPROTEÍNA E POR ELETRO-
FORESE EM GEL DE POLIACRILAMIDA (SDS-PAGE) EM CONDIÇÕES DESNATURANTES... 74 2.9.1 Preparo dos géis de poliacrilamida... 75 2.9.2 Análise dos géis de poliacrilamida... 75 2.9.3 Revelação dos géis... 75 2.10 ANÁLISE DA EXPRESSÃO DA GLICOPROTEÍNA E POR “WESTERN
3 RESULTADOS E DISCUSSÃO... 79 3.1 PRODUÇÃO DE DNA GENÔMICO VDA... 79 3.2 REAÇÃO EM CADEIA DA POLIMERASE (PCR)... 81 3.2.1 Purificação e análise da seqüência gE.VDA amplificada pela PCR... 82 3.3 TRANSFORMAÇÃO DE CÉLULAS COMPETENTES DH5α™ -
CLONAGEM E SUBCLONAGEM DA GLICOPROTEÍNA E DO VDA... 83 3.3.1 Ligação inserto gE.VDA com o plasmídeo pGem®-T Easy e clonagem
por transformação das células competentes... 83 3.3.1.1 Análise do DNA plasmideal recombinante pGem-gE.VDA isolado por
minipreparação... 84 3.3.1.2 Análise do DNA plasmideal recombinante pGem-gE.VDA com
enzima de restrição... 85 3.3.1.2.1 Análise do DNA gE.VDA liberado com EcoR I, recuperado em LMP
e purificado... 87 3.3.1.2.2 Digestão do fragmento gE.VDA com a enzima de restrição Bsr I... 88 3.3.2. Ligação e subclonagem do gene gE.VDA e do plasmídeo de
expressão pFastBac™1... 90 3.3.2.1 Análise do DNA plasmideal recombinante pFastBac-gE.VDA com
enzimas de restrição... 91 3.4 TRANSPOSIÇÃO DO DNA PLASMIDEAL RECOMBINANTE pFAST-
BAC-gE.VDA COM O BACULOVÍRUS RECOMBINANTE “BACMID” ATRAVÉS DA TRANSFORMAÇÃO DE CÉLULAS COMPETENTES E. coli DH10BAC™... 94 3.4.1 Resultado da seleção dos “bacmid” recombinantes pelo fenótipo das
colônias bacterianas... 94 3.4.1.1 Isolamento e análise do DNA viral recombinante
bacmid.pFastBac-gE.VDA de colônias bacterianas DH10Bac™ transformadas por minipreparação de plasmídeos grandes... 95 3.4.1.1.1 Análise do DNA viral recombinante bacmid.pFastBac-gE.VDA por
eletroforese... 95 3.4.1.1.2 Análise do DNA viral recombinante bacmid.pFastBac-gE.VDA pela
técnica de PCR... 96 3.5 CO-TRANSFECÇÃO DE CÉLULAS DE INSETO COM O DNA
RECOMBINANTE BACMID.pFASTBAC-gE.VDA E EXPRESSÃO DO GENE HETERÓLOGO... 97 3.5.1 Análises dos vírus recombinantes “bacmid” expressos em células de
3.5.1.1 Análise do DNA viral recombinante bacmid.pFastBac-gE.VDA das co-transfecções pela técnica de PCR... 100 3.5.1.2 Análises eletroforéticas SDS-PAGE e “Western blotting” da expressão
INTRODUÇÃO
A região Sul do Brasil, em particular o Estado de Santa Catarina (SC), é caracterizada por pequenas propriedades agrícolas. O Estado de SC é constituído por 293 municípios e representa 1,13% da superfície do território brasileiro com uma área de 95.318,3 km2 (ICEPA, 2006). De acordo com o IBGE, em 2000 o estado possuía uma população de 5.333.284 habitantes, dos quais aproximadamente 21% (~1.119.989 habitantes) viviam no campo (IBGE, 2006).
Conforme levantamento agropecuário de Santa Catarina (LAC, 2003) realizado entre o período de 01/09/2002 e 31/08/2003, o Estado de SC possui um total de 230.157 estabelecimentos agrícolas, dos quais 54.730 são estabelecimentos suinícolas com um total de 5.579.975 suínos. A suinocultura catarinense possui o melhor nível de produtividade do país, tanto no campo como na indústria e está concentrada, principalmente, no Meio Oeste e Oeste catarinense (SANDRIN, 2000).
Ainda segundo o instituto CEPA (2006), no ano de 2005, o Estado de SC foi responsável por 24,3% da produção de carne suína no Brasil e, quanto à formação do valor bruto da produção agropecuária estadual, a suinocultura é a segunda principal atividade econômica, com 19% do total. Emprega diretamente em torno de 65 mil e, indiretamente, mais de 140 mil pessoas. A região Oeste do estado concentra 70% do rebanho e 90% da produção suinícola (ICEPA, 2006).
O crescimento mundial e a modernização da indústria suinícola nas últimas duas décadas evidenciam a necessidade de uma maior e mais detalhada atenção no que diz respeito à saúde dos plantéis (SESTI, 2003).
A DA é uma doença infecto-contagiosa causada por um herpesvírus que causa graves prejuízos econômicos ao produtor e à agroindústria (ROMERO et al., 1989; TOOD e McFERRAN, 1985). A significância econômica da DA baseia-se, sobretudo, nas perdas por transtornos reprodutivos; mortalidade, geralmente de 100%, de leitões recém-nascidos; transtornos respiratórios em animais da terminação, levando, geralmente à infecções secundárias; estabelecimento de latência em suínos (KLUGE et al., 1999; METTENLEITER, 2000), o que dificulta seu controle. As granjas registradas com diagnóstico da infecção perdem a certificação para a venda de reprodutores e de material genético (sêmen e embriões) e a DA impõe restrições às exportações (ZANELLA, 2002). A DA está classificada junto ao Código Zoosanitário da Oficina Internacional de Epizootias (O.I.E., 2006), e a sua notificação é compulsória.
No Brasil, o primeiro relato da DA data de 1912 (CARINI e MACIEL, 1912) em gado bovino e, em Santa Catarina desde 1983 em suínos (ROMERO et al., 1984). Segundo Sandrin (2000), após os primeiros focos da DA em SC, adotou-se como forma de controle da doença somente o uso de vacinação dos plantéis suínos diagnosticados com a doença, sem adotar outras medidas sanitárias para evitar a difusão do vírus. A ausência de programas formais de erradicação aumenta a incidência da doença, gastos com vacinações por tempo indefinido e mortes de outras espécies domésticas que vêm a se infectar. O impacto econômico anual da DA sobre a atividade suinícola estadual foi estimado, no ano de 2001, em R$ 931.224,00 (ZANELLA, 2002).
rebanhos onde a rastreabilidade indicou aquisição de animais de granjas infectadas e rebanhos localizados num raio de 2500 metros de um rebanho com surto da doença, também foram amostrados sorologicamente. Além da sorologia, medidas de biosseguridade e controle da doença foram adotadas, conforme preconizado no programa de erradicação. Como resultado preliminar dos trabalhos desenvolvidos pelo programa de erradicação da DA, o último caso de infecção pelo VDA, em SC, ocorreu em julho de 2004 (MORÉS, 2005).
A vacina para DA aprovada pelo Ministério da Agricultura, Pecuária e Abastecimento (MAPA) desde 1995 para uso no Brasil, é uma vacina inativada com uma deleção de todo o gene que codifica a glicoproteína E (gE) do vírus da DA (VDA). Isto permite identificar e diferenciar animais infectados com vírus de campo daqueles vacinados com esta vacina, através de um teste ELISA diferencial para a glicoproteína gE.
Os testes sorológicos de ELISA são necessários para monitorar, controlar ou detectar infecções suínas (TOMA, MOUTOU e FORTIER, 1979). Para tanto, é necessário dispor de testes para a detecção de anticorpos contra o VDA que sejam específicos, realizados em série e de rápida execução na obtenção do resultado (TOMA, MOUTOU e FORTIER, 1979) visando auxiliar os programas de controle e erradicação da DA em suínos.
Uma ferramenta que vem sendo atualmente empregada para se obter bons testes de diagnóstico é a biologia molecular. Uma vantagem destas técnicas é poder trabalhar com apenas partes não essenciais de microorganismos patógenos. Uma característica importante da manipulação genética é poder propagar moléculas de ácido nucléico de um determinado organismo em um outro hospedeiro diferente através da habilidade de atravessar as barreiras naturais da espécie (WOLD e PRINROSE, 1991).
Objetiva-se amplificar, clonar e expressar a proteína recombinante do vírus da DA, utilizando o sistema de baculovírus recombinante para que possa ser utilizado em programas de controle e erradicação da DA em suínos. Ao utilizar somente uma porção do VDA, uma glicoproteína não essencial para a replicação viral em cultura celular (KLUGE et al., 1999; SPEAR, 1993; ZUCKERMANN, 1988), obtêm-se um antígeno direcionado à anticorpos anti-VDA com grande especificidade e sensibilidade.
1 REVISÃO DE LITERATURA
1.1 HERPESVÍRUS
Os herpesvírus são membros da família Herpesviridae e estão amplamente disseminados na natureza (FENNER et al., 1993). Possuem um genoma de fita dupla de DNA, tamanho do vírion e a estrutura (capsídeo, tegumento e envelope) semelhantes entre si e sofrem uma fase latente em seu ciclo de vida (ROIZMAN e SEARS, 1991).
A família Herpesviridae tem uma extraordinária diversidade biológica e com base em suas propriedades biológicas, na organização e conteúdo genômico a maioria dos herpesvírus pode ser subdividido em três principais subfamílias Alphaherpesvirinae, Betaherpesvirinae e Gammaherpesvirinae (MINSON et al., 2000; ROIZMAN, 1991) em conformidade com o Comitê Internacional de Taxonomia Viral (ICTV, 2006). Essas subfamílias diferem no tipo celular onde a latência é estabelecida e na duração do seu ciclo produtivo de replicação.
Os membros da subfamília Alphaherpesvirinae têm um maior número de hospedeiros, tendem a se reproduzir rapidamente com efeitos citopáticos e produzir partículas virais em poucas horas. Além disso, podem estabelecer latência nos gânglios nervosos próximos à região infectada (ROIZMAN, 1991) e em neurônios (KLUGE et al., 1999; METTENLEITER, 2000). Os membros da subfamília Betaherpesvirinae estão restritos a um espectro menor de hospedeiros em relação aos da subfamília Alfaherpesvirinae e possuem um ciclo longo de replicação (ROIZMAN, 1991). Os membros da subfamília Gamaherpesvirinae estão limitados à família ou ordem a que seus hospedeiros naturais pertencem e a latência viral é freqüentemente demonstrada em tecido linfóide (ROIZMAN, 1991).
raros relatos de VDA humano não foram substanciais e refletem, provavelmente, uma baixa reação cruzada de anticorpos gB do HSV-1 com anticorpos anti-gB do VDA (ROBBINS, et al., 1987).
1.2 HERPESVÍRUS SUÍNO TIPO 1 (VDA)
O vírus da doença de Aujeszky (VDA), também denominado de vírus da pseudo-raiva (PRV), ou herpesvírus suíno tipo 1 (POMERANZ, REYNOLDS e HENGARTNER, 2005) é o agente patogênico da DA e pertence à família Herpesviridae, subfamília Alphaherpesvirinae, gênero Varicelovirus. Os Alfaherpesvírus são vírus DNA de cadeia dupla, com envelope, e que possuem várias propriedades biológicas em comum. Infectam várias espécies animais, replicam rápido e liticamente em células de cultivos celulares, possuem neurotropismo e estabelecem infecções latentes em gânglios do sistema nervoso periférico e em outros tecidos nervosos (KLUGE et al., 1999; METTENLEITER, 2000; ROIZMAN, 1991). O sítio predominante da infecção latente é o gânglio trigeminal (POMERANZ, REYNOLDS e HENGARTNER, 2005).
A DA é uma doença de grande impacto econômico sobre a produção de suínos por causar inúmeros sinais clínicos, incluindo transtornos reprodutivos, respiratórios e do sistema nervoso central (KLUGE et al., 1999; METTENLEITER, 2000), causando alta mortalidade de leitões, com estabelecimento de infecção latente em suínos e recorrência viral (KLUGE et al., 1999), levando à restrição na movimentação de animais destinados à reprodução (ROMERO et al., 1989). Enquanto esforços para erradicar o VDA nos Estados Unidos e na Europa mostram grande progresso, este patógeno ainda é um problema endêmico em muitos países (POMERANZ, REYNOLDS e HENGARTNER, 2005).
(ELIOT et al., 1989, KIT e KIT, 1991; VAN OIRSCHOT et al., 1990). Portanto, a base das vacinas diferenciais consiste na inativação ou deleção de um gene que codifica uma glicoproteína não essencial, e o uso desse vírus mutante como vacina (KIT e KIT, 1991). A engenharia genética permitiu que um ou mais genes de glicoproteínas não essenciais fossem construídos e aplicados na produção de vacinas (PENSAERT et al., 1992).
1.2.1 Genoma
O genoma de VDA é constituído por uma molécula de DNA dupla-fita, com 143.461 nucleotídeos (143,461Kpb), 72 ORF (“Open Reading Frame”) homólogas com alfaherpesvírus relacionados (KLUPP et al., 2004).
As 72 ORFs codificam 70 diferentes proteínas, pois os genes que codificam as proteínas IE180 e US1 são encontrados duas vezes: uma vez na seqüência repetida interna (RI) e outra na seqüência repetida terminal (RT). Todo o genoma do VDA foi seqüenciado usando fragmentos de seqüências provenientes de seis diferentes cepas. As cepas foram sempre seqüenciadas e identificadas, tendo como base principal, a sua homologia com os genes encontrados em outros alfaherpesvírus (KLUPP et al., 2004). Aproximadamente metade dos genes alfaherpesvírus são considerados como não essenciais, isto é, são dispensáveis para a replicação viral, pelo menos em cultura de células (METTENLEITER, 2000; SPEAR e LONGNECKER, 2003).
Figura 1 – Mapa da organização do genoma VDA. A) Diagrama esquemático do genoma VDA: a seqüência única longa (UL) e a seqüência única curta (US), as seqüências repetidas invertidas interna (RI) e terminal (RT). B) Mapa dos fragmentos liberados com enzima de restrição BamH I. C) Ampliação do fragmento 7 BamH I localizado na seqüência US, com sítios de clivagem importantes: ) BamH I; St) StuI; Sp) SphI).
Fonte: Brack et al., 2000 (Adaptado).
1.2.2 Capsídeo
Figura 2 – Nucleocapsídeo típico de herpesvírus. Detalhe digitalizado de um penta-peplômero axial, cercado por hexa-peplômeros.
Fonte: Zhou et al., 1999.
1.2.3 Tegumento
A camada de tegumento do VDA (Figura 3) é uma estrutura eletrondensa amorfa que preenche o espaço entre o capsídeo e a membrana do envelope de partículas maduras de herpesvírus (ROIZMAN, 1991). As proteínas do tegumento têm papel importante durante a entrada e a morfogênese do vírion (METTENLEITER, 2002).
1.2.4 Envelope
Figura 3 – Partícula de herpesvírus. N) nucleocapsídeo; T) tegumento; E) envelope; G) glicoproteínas.
Fonte: Stannard, 2003.
1.2.5 Glicoproteínas
As glicoproteínas dos herpesvírus são encontradas em grande quantidade em todas as membranas das células infectadas, assim como no envelope viral. As glicoproteínas formam protusões ou espículas no envelope viral, que nos herpesvírus são mais curtas e em maior número do que aquelas que aparecem nas superfícies de outros vírus envelopados (ROIZMAN, 1991). Essas proteínas de membrana desempenham funções vitais no processo de infecção viral, mediando os processos de reconhecimento e adsorção das células do hospedeiro, penetração do vírion, fusão e disseminação do vírus célula-a-célula em cultivos celulares (POMERANZ, REYNOLDS e HENGARTNER, 2005). As proteínas também modulam a resposta imune e promovem a formação de sincício. Devido à sua localização na superfície do envelope viral e nas membranas das células infectadas, as glicoproteínas são o principal alvo do sistema imune do hospedeiro em resposta à infecção (TODD, HULL e McNAIR, 1987).
O genoma do VDA codifica 16 proteínas de membrana e onze dessas proteínas, durante sua passagem pelo trans Golgi, são modificadas por glicosilação N- ou O-ligado por adição de cadeias laterais de açucares e, então, denominadas de glicoproteínas gB, gC, gD, gE, gG, gH, gI, gK, gL, gM e gN (METTENLEITER, 2000), com peso molecular entre 50 kDa a 130 kDa (HAMPL, 1984). Quatro proteínas adicionais trans-membrana que não são glicosiladas (UL20, UL43, US9 e provavelmente UL24) são encontradas no envelope viral
G
(FUCHS et al., 2002; KLUPP, GRANZOW e METTENLEITER, 2000) e a UL34, uma proteína de membrana encontrada no envelope primário do vírion. Durante a entrada, as glicoproteínas gC, gB, gD, gH e gL são responsáveis pela fixação à superfície da célula hospedeira e a subseqüente fusão do envelope viral com a membrana plasmática (FAVOREEL et al., 1997; HAMPL, et al., 1984). As glicoproteínas gH, gL, gM, gN, gE e gI, interagem genicamente formando os complexos não-cavalentes: gH/gL, gMgN e gE/gI (METTENLEITER, 2002).
Zuckermann et al. (1988) e Whealy et al. (1993) relataram que a glicoproteína E (gE) do VDA forma uma ligação complexa não-covalente com a glicoproteína I (gI), e que este complexo é uma entidade funcional e importante para a correta apresentação dos descontínuos epítopos imunodominantes na gE. O complexo antigênico gE/gI é mais bem reconhecido pelo anticorpo monoclonal que o antígeno gE sozinho (JACOBS et al., 1995 apud GUT et al., 1999).
A nomenclatura padrão das glicoproteínas de envelope do VDA e do HSV foi adaptada no ano de 1993, durante o “18th International Hespesvirus Workshop”. Trabalhos publicados antes do ano de 1995, nominavam as glicoproteínas gB, gC, gD, gE, gG e gI do VDA como gII, gIII, gp50, gI, gX e gp63, respectivamente.
1.2.6 Ciclo de replicação do VDA
Figura 4 – Representação esquemática do ciclo de replicação do VDA.
Fonte: Pomeranz, Reynolds e Hengartner, 2005.
1.2.7 Latência
Membros da subfamília Alphaherpesvirinae podem estabelecer infecções latentes em gânglios nervosos, onde o DNA viral pode persistir em forma epissomal (não integrado ao DNA do genoma da célula) nos núcleos das células
Binding
infectadas. Portanto, animais com infecção latente servem de reservatório natural para o vírus durante toda a vida do animal e são um fator constante de risco de ocorrer uma reativação e liberação viral e a conseqüente disseminação do vírus para uma população susceptível. A latência é definida como um status no qual o DNA viral persiste, mas a infecção viral não é produzida (METTENLEITER, 2000).
Segundo Mettenleiter (2000) e Pomeranz, Reynolds e Hengartner (2005), durante a infecção aguda, partículas virais do VDA replicam primeiro nos tecidos epiteliais da mucosa orofaringeana e então entram diretamente nos terminais nervosos do nervo sensorial da região infectada. Após a primeira replicação no epitélio, a progênie viral é produzida abundantemente levando a um incremento da infecção primária dos neurônios. Os principais sítios de latência do VDA são o gânglio trigeminal, o bulbo olfatório e a tonsila. Nesses órgãos, o DNA viral pode ser detectado mesmo na ausência de uma infecção viral produtiva e transcritos associados à latência (LAT -“latency asociated transcripts”) podem ser demonstrados por PCR (METTENLEITER, 2000). Portanto, durante a latência, a expressão do gene viral fica restrita à transcrição de uma parte distinta do genoma viral, denominada LAT.
O vírus pode ser reativado e liberado de animais com infecção latente após serem submetidos a fatores estressantes. Esses fatores incluem transporte, condições precárias de criação, outras enfermidades concomitantes, partos, trocas na dieta, condições extremas e variáveis de temperatura e umidade, tratamento com agentes imunossupressivos (corticosteróides), sendo um dos fatores responsáveis pela perpetuação e transmissão do vírus no rebanho (KLUGE et al., 1999; ROIZMAN, 1991). Ao final do processo de reativação, o vírion é transportado através dos axônios ao local onde ocorreu a infecção original.
1.3 DETECÇÃO DO AGENTE INFECCIOSO OU DE ANTICORPOS ANTI VDA
moleculares de diagnóstico. O isolamento viral é a técnica padrão para a detecção do VDA (ROMERO et al., 1986). Para sua execução, suspensões de tecidos ou secreções são colocadas sobre cultivos de células, as quais podem ser cultivos primários ou linhagens celulares contínuas. Após um período variável de incubação, a presença do vírus é detectada pelo efeito citopático (ECP) causado nas células do cultivo (ROMERO et al., 1986).
A técnica de reação em cadeia da polimerase (PCR) promove a amplificação de uma região selecionada da molécula de interesse do DNA. Qualquer região da molécula pode ser selecionada, contanto que as seqüências das bordas da região sejam conhecidas (ALBERTS, et al., 1999; PASSAGLIA e ZAHA, 1996). Apresenta como vantagem a possibilidade de alcançar altos índices de sensibilidade e especificidade, além da rapidez de execução (ALBERTS, et al., 1999; PASSAGLIA e ZAHA, 1996; WOLD e PRINROSE, 1991). Nesse caso, podem ser utilizados tanto tecidos como secreções para a extração do DNA viral.
Uma técnica usada para visualizar e quantificar proteínas e DNAs é a eletroforese em gel. Moléculas de DNA têm carga negativa, e sob uma corrente elétrica, as moléculas de DNA migram através do gel a uma razão que dependerá do tamanho do DNA. Uma molécula pequena de DNA poderá atravessar/passar através do gel facilmente e, portanto migrar mais facilmente que as moléculas maiores (WOLD e PRINROSE, 1991). Ainda, segundo Wold e Prinrose (1991), o gel de eletroforese não é somente usado como um método analítico, mas também para purificar fragmentos específicos de DNA. O gel é composto por acrilamida ou agarose. O uso da agarose é conveniente para separar fragmentos de DNA que podem variar de tamanho desde poucas centenas a 20 kb, enquanto que para fragmentos pequenos de DNA, é preferível utilizar a acrilamida.
A análise genômica com enzimas de restrição é um tipo de análise voltada para a caracterização genômica de amostras virais já isoladas. Após a multiplicação viral em cultivo celular, o DNA da amostra viral é extraído e posteriormente clivado com enzimas de restrição. A análise dos fragmentos do DNA digerido é feita por eletroforese em gel de agarose e revelado com brometo de etídeo (GIELKENS e BERNS, 1982).
da hélice em lugares determinados (BEN-PORAT et al., 1984; PASSAGLIA e ZAHA, 1996). São enzimas indispensáveis na análise da estrutura dos cromossomos, no sequenciamento de moléculas longas de DNA, no isolamento de genes e na criação de moléculas novas de DNA que podem ser clonadas (PASSAGLIA e ZAHA, 1996). O número e tamanho do fragmento gerado de populações diferentes de moléculas fornecem informações sobre a constituição genética das mesmas. Assim, se a clivagem do DNA de duas cepas diferentes de vírus por uma determinada enzima gerar fragmentos que diferem no seu número e tamanhos, confirma-se a existência de heterogeneidade genética entre essas cepas. Estas diferenças são úteis em estudos epidemiológicos (BEN-PORAT et al., 1984).
Análises do genoma de vários isolados de VDA recuperados de diferentes casos epidemiológicos demonstraram que seus padrões de fragmentos de restrição são diferentes. Essas diferenças no padrão de migração podem ser devidas a uma perda dos sítios de clivagem (gerando fusão de fragmentos), deleção ou inserção de seqüências de nucleotídeos (gerando assim fragmentos maiores ou menores) ou translocação de seqüências de uma região do genoma para outra (mudando, assim, também o tamanho do fragmento) (BEN-PORAT et al., 1984). Ficinska et al. (2003), caracterizaram cepas do VDA com virulência reduzida, com mutações na região única curta (US
)
, focando principalmente os genes gE e gI e, detectaram que o peso molecular da gE e/ou gI, podem variar entre as cepas do VDA.A localização genômica dos principais fragmentos de DNA gerados pelas enzimas de restrição BamH I e Kpn I foram descritas por Ben-Porat, Rixon e Blakenship (1979) e Rixon e Ben-Porat (1979), os quais publicaram o mapa físico dos sítios de clivagem usando uma cepa do VDA.
Os resultados obtidos por Herrmann, Heppner e Ludwig (1984) mostraram que os quatro tipos de genomas são correlatos com certas áreas geográficas. Os vírus do grupo I são similares ou idênticos com o padrão de DNA BamH I publicado por Rixon e Ben-Porat (1979). As cepas do grupo II não possuem o fragmento número 2 do grupo I, mas possuem dois novos fragmentos acima e abaixo do fragmento 4. Esses dois grupos representam vírus encontrados na Europa Central (Alemanha e Bélgica). A freqüência do genoma tipo II junto com seus variantes foi de aproximadamente 75% em todos os isolados. O Grupo III (genoma tipo III) e IV (genoma tipo IV) são estritamente limitados aos isolados originários do Norte Europeu (Dinamarca e Suécia) e Tailândia, respectivamente. O grupo III difere ligeiramente do grupo I, quando analisado com Bam HI, mas foi bem distinto no padrão de digestão com BstE II. O Grupo genômico IV, porém, diferiu amplamente na distribuição dos sítios de clivagem com ambas as enzimas (HERMANN et al., 1984).
Muitos outros estudos com endonucleases têm sido conduzidos ou para análise do DNA após várias passagens em cultura celular ou animais (MENGELING et al., 1983; WATHEN e PIRTLE, 1984) ou para comparar DNAs virais de diferentes regiões ou países (CHRISTENSEN, SOERENSEN e LEI, 1987; PIATTI et al., 2001; SCHAEFER et al., 2005; TODD e McFERRAN, 1985).
No Brasil, um trabalho que analisou o perfil de restrição com BamH I de 30 amostras isoladas do VDA, dentre os anos de 1982 a 1996, de diferentes regiões do país, encontrou 28 amostras com perfil genômico do grupo II e somente duas amostras do grupo I (PIATTI et al., 2001). Recentemente, Schaefer e colaboradores (2005) analisaram através da digestão com BamH I, 38 amostras de DNA genômico de isolados VDA dos últimos 20 anos (1983 a 2003) provenientes da região Sul do Brasil. Trinta e seis amostras de DNA apresentaram perfil grupo II e somente duas do grupo I.
Figura 5 – Representação esquemática de tipos genômicos do VDA (I, II e III) com base na migração dos três primeiros fragmentos obtidos com BamH I.
Fonte: Piatti et al., 2001.
Inúmeros testes sorológicos laboratoriais podem ser conduzidos para detectar anticorpos anti-VDA. Este tipo de diagnóstico pode ser realizado através de técnicas como a imunoperoxidase, imunofluorescência, soroneutralização (SN) e ensaios imunoenzimáticos. A SN é considerada a técnica padrão para detecção de anticorpos neutralizantes contra VDA (ROMERO et al., 1986). Entretanto, é uma técnica trabalhosa dependente da existência de um estoque de vírus e de cultivos celulares adequados. O resultado da prova é usualmente obtido em três a cinco dias e a SN não permite uma diferenciação precisa entre animais infectados com vírus de campo de animais vacinados contra o VDA.
Os testes imunoenzimáticos do tipo ELISA (enzyme linked immunosorbent assay) são muito utilizados atualmente, devido à sua alta sensibilidade, especificidade, capacidade de processamento de grande número de amostras, relativa facilidade e rapidez de execução e, geralmente, os resultados de provas tipo ELISA, podem ser quantificados através do uso do espectrofotômetro (VAN OIRSCHOT, 1989).
Segundo Gut et al. (1999), o sucesso de um programa de controle e erradicação utilizando vacinas gene-deletadas para a glicoproteína gE do VDA com testes sorológicos diferenciais depende da habilidade do teste diferencial em detectar, com precisão, suínos infectados, uma vez que, a diferenciação entre suínos infectados e vacinados para a DA é muito importante para o controle da doença em países que praticam a vacinação (VAN OIRSCHOT e OEI, 1989).
Com o surgimento da cepa VDA Bartha, com uma deleção natural do gene gE, Van Oirschot e Oei (1989) desenvolveram um teste sorológico que permitia distinguir animais vacinados (sem anticorpos anti-gE mas com anticorpos contra outras glicoproteínas do VDA) dos animais com anticorpos anti-gE (infectados com vírus selvagem). A partir de então, o conceito de “vacinas marcadas” surgiu e o sucesso de seu uso nos programas de erradicação contribuiu para reforçar este conceito (METTENLEITER, 2000).
Com o advento da engenharia genética, testes de diagnóstico podem ser melhorados sob o aspecto de sensibilidade e especificidade e quanto a não difusão de organismos patogênicos, pois a engenharia genética permite manipular apenas partes antigênicas específicas, garantindo maior sensibilidade antígeno-anticorpo e inocuidade quanto a infectividade viral (WOLD e PRINROSE, 1991). Atualmente, vários trabalhos com organismos recombinantes expressando proteínas virais têm sido publicados (ESHAGHI et al., 2004; GUT et al., 1999; JARVIS, HOWE e AUMILLER, 2001; KIMMAN et al., 1996; MORENKOV et al., 1997).
1.4 CLONAGEM E EXPRESSÃO
hospedeira, onde poderá se replicar. O processo de introdução de DNA em células é chamado de transformação.
Plasmídeos são moléculas de DNA circular, de fita dupla, extracromossômicos, que existem naturalmente em bactérias e em alguns organismos eucariotos unicelulares, como as leveduras. Possuem capacidade de replicação autônoma e freqüentemente, transportam genes que conferem resistência a antibióticos (PASSAGLIA e ZAHA, 1996), o que permite distinguir células hospedeiras que receberam o inserto com o vetor plasmideal daquelas que não receberam.
Quando se tem a confirmação da presença do inserto de interesse na célula hospedeira, o produto pode ser co-cultivado com um vírus (selvagem ou recombinante) em cultura celular, através do processo denominado de co-transfecção. A co-transfecção ocorre quando dois ou mais produtos são inoculados conjuntamente para gerar um só produto final (PASSAGLIA e ZAHA, 1996; O’REILLY, MILLER e LUCKOW, 1992).
Segundo O’Reilly, Miller e Luckow (1992), após a co-transfecção, o produto é purificado em placa de cultivo celular, e a glicoproteína expressa (baculovírus em células do inseto) poderá ser identificada sob vários métodos, incluindo triagem visual, por hibridização do DNA, amplificação por PCR, imunodetecção e teste da atividade enzimática (enzimas de restrição) do produto gênico expresso. Após confirmar a identidade e pureza do produto obtido, este poderá ser utilizado como insumo para produzir outros produtos (O’REILLY, MILLER e LUCKOW, 1992), tais como anticorpos monoclonais e antígenos de interesse.
1.4.1 Baculovírus
entretanto nenhum relato em plantas, mamíferos ou outros vertebrados (WEIGHTMAN e BANKS, 1999).
Os Nucleopolyedrovirus (NPV) induzem a formação de poliedrose nuclear (poliedros) enquanto que os Granulovírus (GVs) têm pequenos corpos de oclusão chamados de grânulos, que geralmente contém um único vírion (FUNK, BRAUNAGEL e ROHRMANN, 1997). O baculovírus produtor de poliedros pode ser utilizado para co-transfecção de transformantes com inserto de interesse nas células de inseto (RIBEIRO, SOUZA e KITAJIMA, 1998).
O tamanho do genoma para diferentes membros da família Baculoviridae pode variar de 80 a 180 kbp, composto por um DNA circular de fita dupla (VAN REGENMORTEL et al., 2000). Segundo Ribeiro, Souza e Kitajima (1998), a família Baculoviridae é muito importante em entomologia aplicada, pelo uso como bioinseticidas virais, vetores de expressão de proteínas heterólogas e de terapia gênica. O capsídeo em forma de bastonete pode acomodar, teoricamente, até 100.000 pares de base (pb) de DNAs adicionais de inserções montadas de material genético de outros organismos (POSSEE e ROHRMANN, 1997).
Figura 6 – Cultura de células BTI-Tn-5B1-4 infectadas como vírus AcMNPV. A seta identifica núcleos celulares repletos de poliedros.
1.4.1.1 Infecção e replicação viral
Durante a replicação viral dos baculovírus, os vírions podem assumir duas formas bioquímica e morfologicamente distintas. Em uma delas o vírion brota da membrana citoplasmática da célula hospedeira para o meio extracelular de forma polarizada, sendo envelopado individualmente e é denominado “budded virus” (BV) ou de “extracelular vírus” (ECV). A outra forma é denominada “polyhedra-derived virus” (PDV) ou de “occluded virus” (OV) em que o vírion adquire a membrana sintetizada no núcleo da célula infectada, podendo ser encontrado mais de um nucleocapsídeo por vírion, oclusos em cristais protéicos denominados corpos de oclusão (GRANADOS e WILLIAMS, 1986).
1.4.1.2 Expressão gênica em baculovírus
regulam a expressão gênica seguinte. Uma vez iniciado o processo de infecção, a expressão gênica ocorre ordenadamente em cascata (O’REILLY, MILLER e LUCKOW, 1992). Analogicamente à síntese de proteínas virais, os RNAs mensageiros virais também são sintetizados de uma maneira temporal em células infectadas (FRIESEN, 1997).
A expressão de genes virais pode ser dividida em duas fases gerais: a primeira (“early”), que ocorre até 7 horas pós-infecção (hpi), antes da replicação do DNA viral, e a segunda (“late”), que ocorre a partir do começo ou após a replicação. Durante a fase inicial da infecção, dois grupos de genes podem ser diferenciados: os “immediate early” e os “delayed early”. Os genes “late” também podem ser subdivididos em genes “late” e “very late”, que são expressos a partir ou após o começo da replicação do DNA viral (18 hpi em diante) quando ocorre a síntese de proteínas envolvidas na oclusão das partículas virais, como a poliedrina e a p10, expressas em grande quantidade (O’REILLY, MILLER e LUCKOW, 1992; RIBEIRO, SOUZA e KITAJIMA, 1998). A partir de 60 horas a lise celular já pode ser observada.
1.4.1.3 Baculovírus como vetor de expressão
Os baculovírus têm sido largamente usados como vetores de expressão gênica desde 1980. Os primeiros relatos do uso de baculovírus como vetores de expressão foram publicados por Smith, Summers e Fraser (1983) e Pennock, Shoemaker e Miller (1984), que usaram o AcMNPV (O’REILLY, MILLER e LUCKOW, 1992).
Ribeiro e Crook (1998), relatam algumas vantagens para a utilização do sistema de expressão de proteínas heterólogas, como por exemplo: a alta especificidade dos baculovírus, o que torna o sistema seguro de ser utilizado; existência de promotores fortemente ativos durante a fase tardia da infecção (não interferindo no ciclo viral); altos níveis de expressão das proteínas heterólogas; facilidade de purificação das proteínas heterólogas; capacidade para inserção de grandes quantidades de DNA e a co-expressão de dois ou mais genes.
é altamente transcrito durante o estágio tardio da infecção. O genoma circular de fita dupla do AcNPV tem mais de 130 kpb com múltiplos sítios de reconhecimento para muitas endonucleases de restrição. A proteína recombinante é freqüentemente expressa em altos níveis em larvas ou cultura de células de inseto infectadas (LUCKOW e SUMMERS, 1988; MILLER, 1997; O’REILLY, MILLER e LUCKOW, 1992) e a proteína de interesse é detectada por meio de várias técnicas, como a eletroforese em gel de poliacrilamida, Western blotting, ensaios enzimáticos (RIBEIRO, SOUZA e KITAJIMA, 1998) e PCR.
A tecnologia para a construção de baculovírus vetores é feita com base em plasmídeos de transferência (JARVIS, 1997), inserindo uma seqüência que codifica uma proteína de interesse dentro de vetor de transferência. Após a inserção do gene de interesse no vetor plasmideal, esse é introduzido em células de inseto juntamente com o DNA viral, e espera-se que através da recombinação homóloga entre seqüências virais no vetor e o DNA viral, ocorra a troca entre o gene viral não-essencial e o gene de interesse presente no vetor. A freqüência de recombinantes é de aproximadamente 0,1% e a progênie é usualmente selecionada por testes de placa, o qual pode levar de 4 a 6 semanas. O clone recombinante é identificado por microscopia através do fenótipo distinto (corpos de oclusão / occ) nas células infectadas (LUCKOW et al., 1993). Segundo Kost, Condreay e Jarvis (2005) e Zhao, Chapman e Jones (2003), a seleção por testes de placa de vírus recombinantes além de morosa é difícil de identificar quando se usa o fenótipo sem corpos oclusos (sem poliedros = occ-).
Outra vantagem do sistema de expressão, baseado em baculovírus, é que esses não são infecciosos a vertebrados (FEDERICI, 1997), assim, são considerados suficientemente seguros para manipulação e produção industrial de proteínas de importância biotecnológica. As aplicações dessas proteínas podem ser citadas para uso na produção de vacinas, montagem de testes de diagnóstico, análise tri-dimensional de estruturas protéicas, produção de bio-inseticidas (JARVIS, 1997; RIBEIRO, SOUZA e KITAJIMA, 1998), pesquisas biomédicas entre outros.
de placa. Este método envolve transposição sítio-específica de um gene exógeno de um plasmídeo doador para um DNA baculovírus recombinante, ou “bacmid” de tal forma que o gene exógeno é controlado pelo promotor da poliedrina (LUCKOW et al., 1993).
1.4.2 Cultura de células de inseto
Uma linhagem celular de insetos foi desenvolvida por Granados et al. (1994), denominada BTI-Tn-5B1-4 (Figura 7). As células BTI-Tn5B1-4 têm morfologia esférica e são preparadas de ovos do lepdóptero Trichoplusia ni pertencente à família de insetos Noctuidae da ordem Lepidoptera, conhecida popularmente como lagarta mede-palmo (Figura 8 A a D). As células de ovos da Trichoplusia ni têm como principal propriedade a alta densidade de expressão de proteínas de baculovírus.
A. B.
C. D.
Figura 8 - Diferentes fases do ciclo de vida da Trichoplusia ni. a) ovo; b) larva; c) e d) adulto.
Fonte: Fotos A e C: Mau e Kessing, 1992; Foto B: Amaral, 2005; Foto D: Capinera e Castner, 2005.
1.4.3 Poliedrina, promotor e lacZ
Ao final do ciclo de replicação do baculovírus (JARVIS, 1997) o núcleo ocupa o maior volume da célula infectada do hospedeiro e está preenchido com poliedros. O principal componente dos poliedros é uma proteína denominada poliedrina (O’REILLY, MILLER e LUCKOW, 1992), que compreende pelo menos 25% da proteína total em células infectadas com baculovírus durante a fase “very late” da infecção (SMITH, SUMMERS e FRASER, 1983). O desenvolvimento dos baculovírus como vetores de expressão se fundamentou na propriedade de produzir grandes quantidades de poliedrina durante a infecção (JARVIS, 1997) sob o comando do promotor da poliedrina (polh), um promotor muito forte e de tamanho pequeno (50 pb) que pode ser removido para outros locais do genoma (O’REILLY, MILLER e LUCKOW, 1992).