Pró-Reitoria de Pós-Graduação e Pesquisa
Stricto Sensu
em Ciências Genômicas e Biotecnologia
CONSTRUÇÃO DE BIBLIOTECA METAGENÔMICA DE DNA
MICROBIANO DO SOLO AMAZÔNICO E PROSPECÇÃO DE
ENZIMAS DE INTERESSE BIOTECNOLÓGICO
Brasília - DF
2011
Shelly de Fátima Paluan
Construção de Biblioteca Metagenômica de DNA
Microbiano do Solo Amazônico e Prospecção de
Enzimas de Interesse Biotecnológico
Brasília, DF 2011
Orientadora: Dra. Betania Ferraz Quirino Dissertação apresentada ao Programa de Pós-Graduação Stricto Sensu em
A Deus, aos meus mentores espirituais, aos meus pais, ao meu marido, as minhas doggies e aos
AGRADECIMENTOS
Primeiramente, agradeço a Deus e seus benfeitores de luz por sempre cuidarem de mim e guiar o meu caminho;
Agradeço aos meus pais, Alcides e Shirley, por sempre acreditarem, me apoiarem e darem seu sangue por mim;
Agradeço ao meu irmão Max por todo o apoio e admiração;
Agradeço ao meu marido Rodrigo que, mesmo com todas as dificuldades, me ajudou da melhor maneira possível;
Agradeço a minha orientadora Betania, por confiar em mim e me guiar neste longo e árduo processo,
Agradeço a minha tia Sueli, minha segunda mãe, por sempre confiar em mim e fazer com que eu acreditasse em meu potencial (e por sempre fazer aquele “bifão” e feijão deliciosos!);
Agradeço ao restante de minha família, que sempre me apoiou e acreditou em mim;
Agradeço aos amigos do lab, principalmente aos do meu grupo, com quem dividi tantos momentos inesquecíveis e com quem pude aprender muito;
Agradeço aos meus amigos-irmãos: Débora, Tainá, Karla, Andréa, Chris, Larissa, Isabel, Tiago, André, Gerson, Anselmo, Renato e Rodrigo por sempre me apoiarem tanto e me darem forças em todos os aspectos necessários;
Agradeço em especial aqueles que me ajudaram durante todas as etapas do desenvolvimento deste projeto:
- a Thaís, por me ensinar como construir uma biblioteca;
- ao Rodrigo, por coletar o solo utilizado e me ensinar muitas coisas; - a Betulia, por me ajudar e me ensinar tantas coisas;
- a Ohana e ao Samuel por sempre me ajudarem desde o começo e me ensinarem muitas coisas;
- as minhas filhas científicas Raquel Bispo e Ana Carolina Bitencourt, por me ajudarem tanto em momentos tão difíceis,
- ao Ricardo, por toda ajuda com meus géis,
- ao Daniel, meu “quase filho”, por me ajudar com os resultados de quitinases; - ao William por me ajudar tanto na parte de bioinformática; (te devo muitos “toddynhos”);
A CAPES, CNPq, e FAP-DF pelo apoio financeiro,
A UCB por ceder seu laboratório;
A todo o staff e professores da UCB, em especial os Professores Octávio, Cristine,
Chico, Beatriz, Ruy, aos secretários Fábio e Ana;
Agradeço a minha banca de defesa, Profs. Cristine Barreto e Fernando Araripe, pelas dicas valiosas;
RESUMO
Problemas relacionados à diminuição dos estoques de combustíveis fósseis, políticos e ambientais têm levado à busca de alternativas renováveis. A biomassa vegetal é abundante e barata, sendo candidata ideal para produção de biocombustíveis; entanto, o material lignocelulósico presente na parede celular das plantas possui estrutura complexa e atualmente não existem métodos eficazes que sejam economicamente viáveis para que este seja utilizado como matéria-prima na produção de bioetanol. Enzimas microbianas têm amplo histórico de aplicação industrial e são capazes de romper as ligações complexas da parede celular de plantas. Devido à quantidade de matéria orgânica na Floresta Amazônica, existe um grande número de microrganismos participando sua biodegradação. A microbiota associada ao bioma Amazônia é relativamente pouco explorada biotecnologicamente. Além disso, já que apenas 1% dos microrganismos pode ser cultivado no laboratório, técnicas moleculares que dispensam o cultivo são as mais adequadas para aplicações biotecnológicas. Para ultrapassar essa barreira, pode-se utilizar a técnica do metagenoma, onde a fração bacteriana da microbiota completa de uma amostra é explorada por técnicas moleculares. Neste trabalho, foi construída uma biblioteca metagenômica de expressão de pequenos insertos (de 3 a 8 kb) a partir do DNA microbiano encontrado no solo Amazônico, contendo 70.000 clones clonados em E. coli EPI300. Ela foi dividida em
duas partes: biblioteca 1, com 30.000 clones, e biblioteca 2, com 40.000 clones. Esta biblioteca foi triada para atividades enzimáticas de amilase, β-glicosidase, CMCase, lipase, protease, quitinase e xilanase, tendo sido encontrados um clone de amilase, doze β-glicosidases, onze CMCases, três quitinases e onze xilanases. Somente três clones foram escolhidos para serem estudados em detalhe e sequenciados: AmAmi01 (amilase), AmEndo02 (CMCase) e AmEndo03 (CMCase). Após o sequenciamento e análise destes, descobriu-se que o clone AmAmi01 tem uma ORF para amidase e não amilase. Este fato apresenta possivelmente uma nova estratégia de triagem para a atividade de amidase. O clone AmEndo02 não tem nenhuma ORF conhecida para atividade celulolítica e o AmEndo03 tem uma ORF para uma β-manosidase. É fato que algumas enzimas apresentam promiscuidade com substratos. Com isso, os clones AmEndo02 e AmEndo03 foram inoculados em meio LB ágar contendo diferentes tipos de CMC (0,1%, m/v), xilana de birchwood (0,5%, m/v) e quitina coloidal (2%, m/v), onde
portanto, confirmar a veracidade do modelo gerado), o inibidor foi testado na concentração de 0,1M em meio LB ágar com CMC a 0,1% (m/v). Porém, este inibidor provou-se tóxico para a E. coli EPI300, enfatizando a necessidade de obtenção da
proteína de interesse do clone AmEndo03 purificada para a prática de ensaios enzimáticos. Foram feitos estudos filogenéticos com as sequências protéicas da ORFs de interesse dos clones AmAmi01 (amidase) e AmEndo03 (β-manosidase), provando que o DNA do inserto de ambos provém de bactérias. Este trabalho é somente um esforço inicial para a exploração biotecnológica sustentável da riqueza metabólica da microbiota do solo Amazônico. Conclui-se que a metagenômica é uma técnica que permite a exploração biotecnológica de microorganismos ainda desconhecidos, mas também há uma grande necessidade de melhoramento nas técnicas de bioensaios para atividades enzimáticas.
ABSTRACT
Political and environmental problems, as well as the diminishing available fuel-stocks, have pushed for the search of renewable alternatives. Plant biomass is abundant and cheap, making it the ideal candidate for the production; however, the lignocellulosic matter in plant cell walls has a very complex structure and, so far, there are not any effective methods that are economically viable for the production of bioethanol using the latter. Microbial enzymes have a long history of industrial utilization and they are capable of breaking the complex bonds in plant cell walls. Due to the amount of organic matter in the Amazon Forest, there are a great number of microorganisms participating in its biodegradation. The microbiota associated to the Amazon biome is relatively underexplored biotechnologically. Moreover, since only 1% of microorganisms can be readily cultured in a lab, molecular techniques that are not dependent on culturing are more adequate for biotechnological applications. The metagenome, where the whole bacterial composition of a sample can be explored by molecular techniques, is an option to surpass this limitation. For this study, a small insert – 3 to 8 kb – metagenomic library of 70,000 clones was constructed using microbial DNA found in Amazon soil. E. coli
EPI300 was used as the host and the library was constructed in two parts: library 1, with 30,000 clones, and library 2, with 40,000 clones. This library was screened for enzymatic activities of amylase, β-glucosidase, CMCase, lipase, protease, chitinase and xylanase, where one amylase, twelve β-glucosidases, seven CMCases, three chitinases and eleven xylanases clones were isolated. Three clones were chosen to be further studied and sequenced: AmAmi01 (amylase), AmEndo02 (CMCase) and AmEndo03 (CMCase). After sequencing and analyzing these clones, it was verified that clone AmAmi01 had an ORF coding for amidase and not amylase, possibly presenting a new strategy for screening for amidase activity. Clone AmEndo02 does not have any ORF coding for cellulolytic activity and AmEndo03 has an ORF coding for a β-mannosidase. Enzyme substrate promiscuity is a known fact. Therefore, clones AmEndo02 and AmEndo03 were inoculated on LB agar medium containing different types of CMC (0.1%, m/v), birchwood xylan (0.5%, m/v) and colloidal chitin (2%, m/v), where they presented positive activity for all substrates. Primers for amplifying AmEndo03’s ORF coding for the β-manosidase were constructed. In the future, these primers will aid in sub-cloning and expressing this ORF in order to obtain the purified protein of interest. This protein will be used for more specific and sensitive enzymatic assays, excluding the need for a host. Due to the similarity of the sequence from this AmEndo03’s ORF to proteins that have already been experimentally modelled and deposited on PDB database, it was decided to also model such protein by a comparative approach. A model was generated containing a ligand in it that is possibly an inhibitor. In order to test in vitro if this inhibitor indeed binds to the protein’s catalytic site (and, therefore, to
enzymatic assays. Phylogenetic studies were done with the protein sequences of the ORFs of interest from clones AmAmi01 (amidase) and AmEndo03 (β-manosidase), proving that the DNA present in both clones is microbial. This study is just an initial effort for the sustainable biotechnological exploration of the metabolic richness from the Amazon soil microbiota. It was concluded that metagenomics is a technique that allows for the biotechnological exploration of unknown microorganisms, but there is also a great need for improvement on the techniques to assay for enzymatic activities. Also, even though the library was screened for various enzymatic activities, the focus of this study was the isolation of new glycosyl hydrolases that can be used in the production of second generation ethanol.
ÍNDICE DE FIGURAS
Figura 1 – Solo Amazônico brasileiro na área onde foram coletadas as amostras. Nota-se uma espessa camada de matéria orgânica preNota-sente no solo. ... 20 Figura 2 - Método de amostragem do solo coletado. Foram coletados cinco pontos com 30 metros de distância. Dentro de cada ponto, foram coletados cinco sub-pontos com 10 centímetros de distância e estes foram juntados na mesma amostra. ... 42 Figura 3 – Localização da área amostrada para este trabalho. O solo foi coletado pela parte da manhã na Estação Experimental do Distrito Agropecuário da Suframa, uma área de floresta nativa cujo solo é do tipo Latossolo amarelo, no município de Manaus – AM, dia 27 de maio de 2009. As amostras utilizadas foram A1#1 e A1#2. ... 42 Figura 4 – Mapa do vetor utilizado na construção da biblioteca metagenômica de
pequenos insertos, pCF430 (NEWMAN e FUQUA, 1999). ... 44
Figura 5 – Processamento da amostra de solo amazônico brasileiro em trama de 2mm para remover o material vegetal e desfazer os grumos de solo. ... 58 Figura 6 – Extração de DNA total do solo Amazônico Brasileiro. Aqui está sendo
apresentada a extração do DNA total do solo com fenol e clorofórmio. A primeira
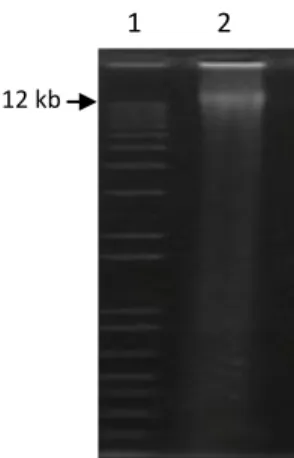
camada representa a parte aquosa, onde se encontra o DNA de interesse. ... 59 Figura 7 – Gel de agarose low-melting 1% (tampão TAE) com amostra do DNA total
extraído e utilizado para construir a biblioteca – 1. 1kb plus DNA ladder (Invitrogen); 2. Amostra do DNA extraído. ... 59 Figura 8 – Gel de agarose 1% (tampão TBE) com clones aleatórios das duas frações da biblioteca digeridos com PstI. ... 60
Figura 9 – Digestão em duplicata do clone AmAmi01 com PstI em gel de agarose 0,8%
(TBE). 1 – 1kb plus DNA ladder (Invitrogen); 2 –pCF430 + PstI; 3,4 – AmAMI01 + PstI.
... 63 Figura 10 - Teste de re-transformaçãodo clone AmAmi01. Meio LB com 0,5% amido (m/v) corada com vapor de iodo metálico após 6 dias a temperatura ambiente. ... 64 Figura 11 – Meio LB ágar com 1% esculina (m/v), citrato de ferro 0,05% (m/v),
tetraciclina a 20 μg/mL e arabinose a 0,02% para triagem de clones com atividade de β-glicosidase (3 dias a temperatura ambiente). As setas indicam os clones com
atividade positiva. ... 65 Figura 12 –Gel de agarose 1% (tampão TBE) com os clones de β-glicosidase digeridos com PstI (1 – 1kb plus DNA ladder (Invitrogen); 2 a 13 –AmβG02, 03, 04, 05, 06, 09,
10, 11, 12, 15, 16 e 18 digeridos com PstI). ... 65
Figura 13 –Gel de agarose 1% (tampão TBE) com os clones de β-glicosidase digeridos com PstI e EcoRI (1 – 1kb plus DNA ladder (Fermentas); 2 a 13 –AmβG02, 03, 04, 05,
06, 09, 10, 11, 12, 15, 16 e 18 digeridos com PstI e EcoRI). ... 66
Figura 14 – Gel de agarose 0,8% (tampão TBE) dos clones de CMCase (AmEndo02 e AmEndo03) e pCF430 digeridos com PstI. 1. 1kb plus DNA ladder (Fermentas); 2.
AmEndo02 + PstI; 3. AmEndo03 + PstI; 4. pCF430 + PstI. ... 67
0,1% CMC pré-hidratado (m/v), tetraciclina a 20 μg/mL e arabinose 0,02% (m/v) corado com Congo red após 15 dias a temperatura ambiente. ... 67 Figura 16 – Clones AmEndo02 (A), AmEndo03 (B) e pCF430 (C) em meio LB ágar
com CMC pré-hidratado a 0,1% (m/v) (1), CMC de baixa viscosidade a 0,1% (m/v) (2), CMC de média viscosidade a 0,1% (m/v) (3) e xilana de birchwood a 0,5% (m/v)(4), tetraciclina a 20 μg/mL e arabinose a 0,02% (m/v) corados com Congo red após 5 dias a temperatura ambiente. ... 68 Figura 17 – Meio LB com CMC de alta-viscosidade a 0,1% (m/v) com tetraciclina a 20 μg/mL e arabinose a 0,02% (m/v). Aqui estão apresentados os clones pCF430,
AmEndo02 e AmEndo03 (em ordem) nas placas coradas com Congo red após 15 dias a temperatura ambiente. ... 68 Figura 18 – Clones pCF430, 22E7 (AmQit01), 23B9 (AmQit02) e 41A2 (AmQit03)
plaqueados em meio LB ágar com tetraciclina a 20 µg/mL, arabinose a 0,02% (m/v) e quitina coloidal a 2% (m/v) corado com Congo red após 7 dias a temperatura ambiente (POLLA, 2011). ... 69 Figura 19 – Ensaio em placa (0,5% amido + ágar ) com lisado e sobrenadante dos clones pCF430 e AmAmi01 (1. pCF430; 2. AmAmi01; 3. Enzima industrial; A = lisado; B
= sobrenadante) corado com vapor de iodo metálico após 3 dias a temperatura
ambiente. ... 70 Figura 20 – Mapa do inserto do clone de AmAmi01. O clone apresenta três ORFs em diferentes fases de leitura. As fases de leitura estão indicadas entre parênteses, assim como as bases onde as ORFs começam e terminam. As setas indicam o sentido de leitura da ORF. ... 71 Figura 21 – Mapa do inserto do clone AmEndo02. O clone apresenta três ORFs em diferentes fases de leitura. As fases de leitura estão indicadas entre parênteses, assim como as bases onde as ORFs começam e terminam. As setas indicam o sentido de leitura da ORF. ... 72 Figura 22 – Mapa do inserto do clone AmEndo03. O clone apresenta quatro ORFs em diferentes fases de leitura. As fases de leitura estão indicadas entre parênteses, assim como as bases onde as ORFs começam e terminam. As setas indicam o sentido de leitura da ORF. ... 73 Figura 23 – Antibiograma com antibióticos em disco (A. E. coli EPI300 + pCF430; B. E. coli EPI300 + AmAmi01). Meio Mueller-Hinton com tetraciclina a 20 μg/mL e arabinose
a 0,02% (m/v) após 2 dias a temperatura ambiente. As concentrações dos antibióticos utilizados são apresentadas na tabela 9. ... 75 Figura 24 – Árvore filogenética da AmAmi01-AM. A árvore foi construída pelo método
Maximum-likelihood utilizando o software MEGA 5.05 com o modelo
Jones-Taylor-Thornton (JTT). Foi calculada a significância estatística para cada clado utilizando 1.000 repetições de bootstrap. Utilizou-se as 26 sequências proteicas não-hipotéticas
que apresentaram similaridade a AmAmi01-AM após análise no Blastp. Organismos do mesmo filo foram agrupados e as sequências foram identificadas pelo número de identificação do NCBI (gi). ... 77 Figura 25 – Árvore filogenética da AmEndo-BM. A árvore foi construída pelo método
Maximum-likelihood utilizando o software MEGA 5.05 com o modelo
apresentaram similaridade a AmEndo03-BM com bit-score acima de 500 após análise
no Blastp. Como outgroup foi utilizada uma sequência proteica de β-manosidase de
fungo (Penicillium marneffei),pois todas sequências utilizadas eram provenientes de
bactérias. Organismos do mesmo filo foram agrupados e as sequências foram
identificadas pelo número de identificação do NCBI (gi). ... 78 Figura 26 – Resultado da análise da sequência da AmEndo03-BM no Blastp utilizando o banco de dados PDB. A seta indica a sequência utilizada como padrão (2JE8) para a modelagem molecular da AmEndo03-BM. ... 80 Figura 27 – Estrutura tridimensional da enzima BtMan2A. Esta enzima contém cinco
domínios: Domínio 1 (vermelho), domínio 2 (verde), domínio 3 (azul claro), contendo o sítio catalítico, domínio 4 (amarelo) e domínio 5 (roxo). As esferas em cinza
representam o inibidor da enzima, Bis-tris propano (B3P). ... 81 Figura 28 – Alinhamento das sequências de amino ácidos das estruturas BtMan2A e
AmEndo03-BM feito pela ferramenta ClustalW (THOMPSON; GIBSON; HIGGINS,
2002). Os amino ácidos selecionados em amarelo são os resíduos ativos de cada estrutura. Os asteriscos significam que o amino ácido é idêntico em ambas sequências. A presença de pontos significa que os amino ácidos são similares. Por fim, a ausência de qualquer marcação indica que os amino ácidos não têm qualquer similaridade. Cálculos indicam que as duas estruturas têm 40,26% de identidade (número de asteriscos dividido por número total de amino ácidos) e 73,91% de similaridade
(número de pontos dividido por número total de amino ácidos). ... 82 Figura 29 – Modelo Molecular da AmEndo03-BM. A enzima contém quatro domínios: Domínio 1 (vermelho), domínio 2 (verde), domínio 3 (azul claro), contendo o sítio catalítico, e domínio 4 (amarelo). As esferas em cinza representam o possível inibidor da enzima, Bis-tris propano (B3P). ... 83 Figura 30 – Sobreposição entre a estrutura da BtMan2A (verde) e o modelo molecular
da AmEndo03-BM (azul). Este alinhamento apresenta um desvio de raiz quadrada média (RMSD) de 0,4. ... 83 Figura 31 – Diagrama de Ramachandran para o modelo molecular criado para a
AmEndo03-BM. ... 84 Figura 32 – Resultado da análise da estrutura da AmEndo03-BM no ProSA-Web. O ponto negro indica a localização e Z-score do modelo da AmEndo03-BM, provando que a estrutura é semelhante a alguma outra estrutura resolvida por cristalografia. ... 85 Figura 33 – Tríades catalíticas. A – Resíduos ativos da BtMan2A; B – Resíduos ativos
da AmEndo03-BM. Existem algumas diferenças entre as duas estruturas: há um
resíduo de asparagina a mais, na posição 284 da AmEndo03-BM, uma substituição de um triptofano por um acido glutâmico e uma tirosina que está em uma posição diferente (resíduo 537 na BtMan2A e 377 na AmEndo03-BM). ... 86
ÍNDICE DE TABELAS
Tabela 1 – Primers utilizados para sequenciar o inserto do clone AmAmi01. ... 52 Tabela 2 – Primers utilizados para sequenciar o inserto do clone AmEndo02. ... 52 Tabela 3 – Primers utilizados para sequenciar o inserto do clone AmEndo03. ... 53 Tabela 4 – Concentrações e classes dos antibióticos utilizados no antibiograma com antibióticos no meio... 54 Tabela 5 – Concentrações e classes dos antibióticos em disco utilizados no
antibiograma. ... 55 Tabela 6 – Resultado do sequenciamento das extremidades de clones aleatórios da biblioteca. ... 61 Tabela 7 – Resultados das triagens realizadas com as bibliotecas (B1 – Biblioteca 1; B2 – Biblioteca 2). ... 62 Tabela 8 – Resultado da análise das ORFs do clone AmAmi01 no Blastp. As massas moleculares foram arredondadas. ... 72 Tabela 9 – Resultado da análise das ORFs do clone AmEndo02 no Blastp. As massas moleculares foram arredondadas. ... 73 Tabela 10 – Resultados das análises das ORFs do clone AmEndo03 no Blastp. As massas moleculares foram arredondadas. ... 74 Tabela 11 – Resultado do antibiograma com antibióticos em disco (E. coli EPI300). ... 75
SUMÁRIO
AGRADECIMENTOS ... 04
RESUMO ... 06
ABSTRACT ... 08
ÍNDICE DE FIGURAS ... 10
ÍNDICE DE TABELAS ... 13
SUMÁRIO ... 324
ÍNDICE DE ABREVIAÇÕES ... 357
1 INTRODUÇÃO ... 188
O Etanol no Brasil e no Mundo ... 18
2 REVISÃO DE LITERATURA ... 20
Matérias-prima para Biocombustíveis ... 20
A Técnica da Metagenômica ... 22
O Solo Amazônico Brasileiro ... 25
Estrutura da Parede Celular de Plantas ... 28
A Desconstrução da Parede Celular ... 30
Outras Enzimas para a Produção de Bioenergia ... 33
3 JUSTIFICATIVA ... 37
4 HIPÓTESES... 39
5 OBJETIVOS ... 40
Geral ... 40
Específicos ... 40
6 MATERIAIS E MÉTODOS ... 41
Coleta do solo ... 41
Biblioteca Metagenômica ... 43
Extração do DNA Total ... 43
Biblioteca Metagenômica de Pequenos Insertos ... 44
Triagem Funcional para Enzimas de Interesse Biotecnológico ... 46
Caracterização Enzimática Primária dos Clones Encontrados ... 50
Sequenciamento dos Clones Positivos ... 47
Análise das Sequências dos Clones ... 52
Teste de Resistência a Antibióticos ... 53
Modelagem Molecular de Sequências Protéicas ... 55
Confirmação Experimental da Modelagem Molecular do AmEndo03 ... 56
7 RESULTADOS ... 58
Processamento da Amostra ... 58
Extração de DNA ... 58
Construção da Biblioteca ... 59
Validação da Biblioteca ... 60
Triagem Funcional para Enzimas de Interesse Biotecnológico ... 61
Triagem para Atividade de Amilase ... 62
Triagem para Atividade de β-Glicosidase ... 64
Triagem para Atividade de CMCase ... 66
Triagem para Atividade de Quitinase ... 69
Triagem para Atividade de Xilanase ... 69
Caracterização Enzimática Primária dos Clones Encontrados ... 70
AmAmi01 ... 70
AmEndo02 e AmEndo03 ... 70
Sequenciamento dos Clones Positivos ... 71
Análise das sequências ... 71
Testes de Resistência a Antibióticos ... 74
Árvores Filogenéticas das ORFs Possivelmente Codificadoras para as Atividades Enzimáticas de Interesse Observadas ... 76
AmAmi01 ... 76
AmEndo03 ... 76
Modelagem Molecular ... 79
Descrição do Modelo Molecular do AmEndo03 ... 79
Avaliação do Sítio Ativo da AmEndo03-BM ... 85
8 DISCUSSÃO ... 87
Biblioteca Metagenômica de Pequenos insertos ... 87
Clones Isolados para Atividades Enzimáticas de Interesse ... 88
Clone AmAmi01 ... 88
Clone de AmEndo02 ... 92
Clone AmEndo03 ... 94
9 CONCLUSÕES ... 99
10 PERSPECTIVAS FUTURAS ... 101
12 APÊNDICE ... 111
Sequências dos Clones ... 111
AmAmi01 ... 111
AmEndo02 ... 113
ÍNDICE DE ABREVIAÇÕES
%– porcentagem aa– amino ácidos
β– beta
C°– graus Celsius
CMC– carboximetil celulose DNA – ácido desoxirribonucléico DNS – ácido dinitrosalicílico
EDTA – ácido etilenodiamino tetra-acético
g– constante de gravidade g– gramas
kg– quilogramas kb– kilo bases kDa– kilo Daltons LB – Luria Bertani µg– microgramas µL– microlitros mg– miligramas mL– mililitros
MUC – metilumbeliferil β-D-celobiosídeo MUG –metilumbiliferil β-D-glicosídeo m/v– massa por volume
ORF –open reading frame RNA – ácido ribonucléico
18 1 INTRODUÇÃO
O Etanol no Brasil e no Mundo
O Brasil foi o primeiro país no mundo a utilizar o etanol como combustível produzido a partir de cana-de-açúcar como matéria-prima a nível industrial (SOCCOL et al., 2009) e o seu sistema de agroenergia é considerado o mais eficiente (GOLDEMBERG, 2007). Apesar de hoje a tecnologia da produção do etanol e de veículos movidos pelo mesmo estar bastante avançada e bem estabelecida no país, este processo demorou anos para atingir o estado atual e espera-se que evolua ainda mais com a produção de biocombustíveis de segunda geração.
Até o fim da década de 70, somente o álcool anidro era utilizado no Brasil como combustível. Nesta época, o país estava passando por dificuldade econômica devido a sua dependência da importação de petróleo. Visando melhorar a situação do país, o governo brasileiro se aliou à indústria automotiva para promover o desenvolvimento tecnológico de veículos que utilizassem o álcool como combustível único. Portanto, no começo da década de 80, o governo brasileiro estabeleceu que o álcool fosse vendido a 60% do valor da gasolina, uma ação que criou vantagem para o álcool na relação entre preço e poder calorífico comparado à gasolina. Isto resultou que entre os anos de 1983 e 1988, cerca de 90% dos veículos vendidos no país eram movidos a etanol (EMPRESA BRASILEIRA DE PESQUISA AGROPECUARIA, 2006).
19
ano de 1997. Daí até o ano de 2000, a redução do consumo de álcool hidratado superou o aumento do consumo de álcool anidro (EMBRAPA, 2006).
A utilização total de álcool voltou a crescer a partir de meados de 2001, quando o uso de álcool anidro foi maior do que a queda absoluta no consumo de álcool hidratado. Neste período, ao mesmo tempo em que a indústria alcooleira aumentava de importância novamente no mercado, o preço do petróleo começou a subir novamente. Isso fez com que as indústrias buscassem por veículos ainda mais econômicos, criando então os veículos flex – veículos bicombustíveis movidos a álcool e/ou gasolina – e lançando-os no mercado em março de 2003. Estes veículos disseminaram-se de forma tão rápida que até junho de 2010, 81,6% dos veículos vendidos no país eram carros flex (SOUTO e QUIRINO, 2011). Acredita-se que este fato favoreça bastante o
consumo de biocombustíveis no futuro. Atualmente, o álcool lidera o mercado de combustíveis no país, com participação superior a 40% dos mesmos (EMBRAPA, 2006).
Apesar do sucesso histórico da utilização do etanol como combustível renovável no Brasil, outros países começaram a investir na área e o quadro mundial atual é de alta competitividade entre eles. A produção mundial de etanol para 2010 foi de 85.8 bilhões de litros, sendo os Estados Unidos o líder em produção com 45 bilhões de litros, seguido do Brasil e da União Européia. A previsão para 2011 é ainda maior, com uma estimativa de 88.7 bilhões de litros produzidos mundialmente (GLOBAL RENEWABLE FUELS ALLIANCE, 2011). Todavia, estes países mencionados não são os únicos buscando maneiras eficientes de se produzir biocombustíveis através de material lignocelulósico. Por exemplo, na Coréia existe um grande problema com a falta de matéria-prima para se produzir álcool de primeira geração. Porém, o país tem uma grande quantidade de resíduos orgânicos, florestais, municipais, animais e agriculturais. Todos estes resíduos podem ser utilizados na produção de etanol de segunda geração e o país está investindo bastante na pesquisa científica para aperfeiçoar a produção do mesmo (JUN-SEOK et al., 2010).
20
utilização de amido para a produção do mesmo pode interferir na produção de produtos alimentícios e gerar futuros problemas éticos (MABEE e SADDLER, 2009; SOCCOLet al., 2009). Por fim, estima-se que a produção de etanol mundial em 2010 eliminou cerca de 101 milhões de toneladas de gases de efeito estufa da atmosfera, e a previsão para 2011 é que sejam eliminadas 105 milhões de toneladas destes gases (GLOBAL RENEWABLE FUELS ALLIANCE, 2011).
2 REVISÃO DE LITERATURA
Matérias-prima para Biocombustíveis
Existem controvérsias sobre a escolha da matéria-prima a ser utilizada na produção de bioetanol, ou seja, a escolha entre utilizar matéria lignocelulósica ou amido. O primeiro problema está na dificuldade de bioconversão do material escolhido. Quando se utiliza o material lignocelulósico, primeiro é necessário separar a celulose e a hemicelulose da lignina. Em seguida, este material passa por um processo de hidrólise que é muito complexo, pois envolve cinco açúcares diferentes, os quais estão presentes nos materiais lignocelulósicos: glicose, galactose, manose, xilose e arabinose (WYMAN, 2003). Por outro lado, o processo de bioconversão do amido é menos complexo, pois este material é muito mais simples de ser processado, seguido da hidrólise enzimática que gera somente um açúcar (WYMAN, 2003), dependendo de qual enzima é utilizada (AKOH et al., 2008).
21
Mesmo com toda a eficiência do Brasil na produção de etanol, nem toda a biomassa produzida é utilizada. Por exemplo, atualmente somente um terço da cana-de-açúcar é utilizado na produção de bioenergia, um terço é bagaço (que é queimado para produzir energia) e um terço é jogado nos campos para serem decompostos por microrganismos. O Brasil produz anualmente cerca de 186 milhões de toneladas de bagaço (SOCCOL et al., 2009), ou cerca de 270-280 kg de bagaço por tonelada de
cana-de-açúcar (RODRIGUESet al., 2003).
O bagaço é um resíduo poroso do colmo da cana que sobra após esta ser esmagada para extrair o seu suco (PANDEY et al., 2000) e é morfologicamente heterogêneo e composto de fibras e outros elementos estruturais, tal como vasos, parênquima, e células epiteliais (SANJUAN et al., 2001). Ele contém de 19-24% de lignina, 27-32% de hemicelulose, 32-44% de celulose e 4.5-9% de cinzas, onde o restante contém outros compostos tais como minerais e ceras (JACOBSEN e WYMAN, 2002). Há uma grande quantidade de energia potencial no bagaço da cana que poderia ser utilizada para se produzir etanol de segunda geração, mas ainda não existem maneiras eficientes que sejam economicamente viáveis de se fazer isso.
Além do material lignocelulósico da cana-de-açúcar, existem diversas outras fontes de matéria lignocelulósica no Brasil que podem ser transformadas em bioenergia. Estas incluem palha de milho, arroz e trigo, resíduos do processamento de citros, biomassa de coco, gramíneas, resíduos da indústria de papel, resíduos da extração de óleo de mamona e girassol, resíduos municipais sólidos celulósicos e outros (SOCCOLet al., 2009). Uma alternativa interessante, proposta por Champagne, seria a utilização de esterco como matéria-prima para a produção de etanol de segunda geração (CHAMPAGNE, 2008).
22
A Técnica da Metagenômica
A técnica da metagenômica permite estudar e explorar a diversidade microbiana proveniente de amostras ambientais sem a necessidade de cultivo dos mesmos (HANDELSMAN, 2004). Para criar uma biblioteca metagenômica, o DNA total de uma amostra ambiental é extraído, clonado em um vetor adequado e posteriormente inserido em um hospedeiro para que este seja triado para diversas finalidades, tal como marcadores gênicos (STEIN et al., 1996), expressão de atividades enzimáticas de interesse e outros (RONDON et al., 2000; MACNEIL et al., 2001; GILLESPIE et al., 2002; LORENZ et al., 2002; LORENZ e SCHLEPER, 2002; COURTOIS et al., 2003; DIAZ-TORRES et al., 2003; KNIETSCH et al., 2003; SCHLOSS e HANDELSMAN,
2003). Esta técnica revolucionou os estudos ambientais de microrganismos, já que somente cerca de 1% dos microrganismos existentes no planeta podem ser cultivados e as enzimas de organismos não cultivados podem ter características diferentes das enzimas conhecidas e/ou utilizadas atualmente (TORSVIK, GOKSOYR e DAAE, 1990; TORSVIK e OVREAS, 2002). Vários trabalhos metagenômicos já foram realizados utilizando diversas amostras, incluindo variados tipos de solo, águas provenientes de lagos, mares e outros, lama, rúmen e água potável (SCHMEISSER, STEELE e STREIT, 2007).
23
Quanto ao hospedeiro a ser empregado na criação da biblioteca metagenômica, geralmente se utiliza a bactéria Escherichia coli, mas existem controvérsias sobre a
utilização desta como o único hospedeiro na construção de uma biblioteca metagenômica, pois um único hospedeiro para expressar genes de outros organismos poderia implicar em problemas de expressão (DANIEL, 2004). Por outro lado, a E. coli
já foi utilizada com sucesso como hospedeiro em vários trabalhos (HENNE et al., 1999; HENNE et al., 2000; RONDON et al., 2000; ENTCHEVA et al., 2001; MACNEIL et al., 2001; MAJERNIK, GOTTSCHALK e DANIEL, 2001; GILLESPIE et al., 2002; KNIETSCH et al., 2003; VOGET et al., 2003).
A vantagem de se trabalhar com E. coli é por este ser um hospedeiro já utilizado
em indústrias, onde foram estabelecidos procedimentos para trabalhar com o mesmo, facilitando seu manuseio (DANIEL, 2004). Há evidencias também de que a expressão em E. coli não será limitada a genes derivados de Proteobacteria (filo ao qual a E. coli
faz parte), pois genes de diversos outros procariotos já foram expressados na mesma sem a necessidade de engenharia genética (HANDELSMAN et al., 1998; RONDON et al., 2000; COURTOIS et al., 2001; HURT et al., 2001; SANTOSA, 2001; Stokes et al., 2001; DANIEL, 2002; COURTOIS et al., 2003). Além do mais, uma biblioteca do DNA de Bacillus cereus utilizando BACs (bacterial artificial chromosomes, em inglês) foi
construída e expressada em E. coli sem nenhum problema, onde os genes de B. cereus foram expressados em alta quantidade (trabalho feito por M.R.R., S.J., Raffel,
R.M.G., e J.H. não publicado).
Um fator interessante é que a distância entre E. coli e B. cereus se compara a
distância genética entre humanos e paramécios (HANDELSMAN et al., 1998). Por último, para enriquecer a biblioteca e garantir que não existem problemas de expressão por utilizar um único hospedeiro, pode-se também utilizador outros hospedeiros tal como Streptomyces, Bacillus e Archaea (HANDELSMAN et al., 1998).
24
genes clonados serão expressos em hospedeiros diferentes, podendo gerar problemas de expressão. Por outro lado, a biblioteca pode também ser triada por semelhança de sequências, um processo que não seria limitado pela expressão de genes em um hospedeiro diferente, como já fora mencionado. Neste tipo de triagem, são criadas sondas de DNA ou primers, os quais são derivados de sequências de genes ou proteínas já identificados por outros trabalhos (DANIEL, 2004). Esta técnica não é seletiva para genes completos (KNIETSCH, 2003) e requer muito trabalho para analisar cada clone encontrado (KNIETSCH, 2003). Além do mais, só serão encontrados genes que sejam similares aos já identificados e, portanto, genes com sequências não-homólogas não serão identificados (DANIEL, 2004; SCHMEISSER, STEELE e STREIT, 2007).
Para construir a biblioteca metagenômica, podem ser utilizados pequenos ou grandes insertos, dependendo do que se espera conseguir com a biblioteca; ou seja, se a idéia é conseguir genes individuais ou produtos de genes (pequenos insertos), ou se é esperado detectar operons e clusters de genes que codificam vias degradativas ou
biosintéticas completas (grandes insertos). A primeira coisa a se pensar quando se constrói qualquer tipo de biblioteca é a integridade do DNA que precisa ser extraído da maior quantidade de organismos possíveis e precisa ser purificado sem ser degradado, pois há necessidade de que o DNA tenha alto peso molecular, principalmente no caso das bibliotecas de grandes insertos (SCHMEISSER, STEELE e STREIT, 2007). As bibliotecas de grandes insertos são necessárias para analisar o tamanho, complexidade e diversidade do metagenoma. Para construí-las são utilizados cosmídeos (ENTCHEVA et al., 2001; COURTOIS et al., 2003), BACs (RONDON et al., 2000) ou fosmídeos (QUAISER et al., 2002; QUAISER et al., 2003; TREUSCH et al., 2004). Por outro lado, as bibliotecas de pequenos insertos, ao longo prazo, são mais adequadas para gerar mais informações sobre sequências de DNA do que as bibliotecas de grandes insertos (VENTER et al., 2004; BANFIELD et al., 2005).
Existem vários trabalhos realizados com a técnica da metagenômica. De acordo com Schmmeisser et al., até 2007 já haviam sido encontrados mais de 20 clones com
25
solo, onde somente um foi purificado e caracterizado (VOGET, STEELE e STREIT, 2006). Apesar de este clone ter sido isolado de um ambiente não-extremo, a enzima produzida por ele tinha alto nível de estabilidade em um largo espectro de valores de pH que chegava até 9, além de ficar estável a uma temperatura de 40°C por várias horas e numa concentração de NaCl a 3M. Clones celulolíticos também já foram identificados em bibliotecas metagenômicas de ambientes extremos. Em uma biblioteca de lagos extremamente alcalinos (Soda lakes, em Inglês) no Egito e na
África, foram encontrados mais de 12 destes clones e alguns produziam enzimas que demonstravam halotolerância características ao ambiente (REES et al., 2003; GRANT et al., 2004). Outro estudo feito em 1995 revelou um clone anaeróbico termofílico com atividade celulolítica, a qual era estimulada pela presença de lignocelulose (HEALY et al., 1995; VOGET, STEELE e STREIT, 2006).
Em um estudo feito por Yi Feng et al. (2007), foi construída uma biblioteca metagenômica com cosmídeos utilizando DNA microbiano do ceco de coelhos. Neste trabalho foram isolados onze clones com atividade celulolítica, onde quatro eram endo-β-1,4-glucanases e sete eram β-glicosidases. Todos estes clones tinham menos de 50% de identidade e 70% de similaridade de resíduos de aminoácidos com as celulases do banco de dados. As quatro endo-β-1,4-glucanases pertenciam à família de glicosil hidrolases 5 (GHF5) e as sete β-glicosidases pertenciam à família de glicosil hidrolases 3 (GHF3), além de formarem dois ramos separados na árvore filogenética (FENG, 2007). Deste modo, novas celulases podem ser encontradas em diversos ambientes. Geralmente, bibliotecas metagenômicas voltadas à identificação de celulases são construídas com DNA microbiano obtidas em ambientes extremos, mas há evidencias de que enzimas celulolíticas com alta estabilidade e apropriadas para utilização em aplicações industriais podem ser encontradas em ambientes não-extremos que tenham alta diversidade genética (SCHMEISSER, STEELE e STREIT, 2007).
O Solo Amazônico Brasileiro
26
por uma camada espessa de matéria orgânica que é constantemente degradada por microrganismos ainda não explorados (Figura 1). Sabe-se que o CO2 disponível na
atmosfera é proveniente do processo de respiração de plantas e animais, mas a grande maioria deste gás – cerca de 90 a 95% – provêm da biodegradação microbiana de materiais orgânicos (EMBRAPA, 2002). A grande maioria da matéria orgânica disponível no meio ambiente – de 40 a 60% – existe na forma de celulose e vários microrganismos atuam nesta biomassa. Por isso, acredita-se que a hidrólise desta está na base da biodegradação e age estrategicamente na preservação do meio ambiente. Se não existissem microrganismos degradando o material lignocelulósico, a camada da Terra estaria coberta por restos vegetais e animais, tornando a vida inviável (LEVY, 1987). O solo carrega em si a maior população de microrganismos existente no planeta (HANDELSMAN et al., 1998; WHITMAN, COLEMAN e WIEBE, 1998) e foi estimado que em cada grama de solo existam cerca de 1.000 a 10.000 espécies de procariotos ainda desconhecidos (TORSVIK, GOKSOYR e DAAE, 1990). Portanto, a exploração de microrganismos presentes no solo Amazônico brasileiro pela técnica da metagenômica é promissora na identificação de genes codificando para enzimas que possam ser utilizadas na produção de bioenergia.
27
Em 1997, Borneman e Triplett fizeram o primeiro estudo sobre a diversidade microbiana do solo Amazônico de uma floresta intacta e uma área que foi desmatada e utilizada para criação de gado, utilizando um método independente de cultivo. Neste estudo foram analisadas 100 amostras totais de solo (50 de cada área) através de RISA e da amplificação de 16S rRNA. Pelas análises de 16S rRNA, 98 sequências eram provenientes de Bacteria e 2 de Archaea (BORNEMAN e TRIPLETT, 1997).
O fato de aparecerem 2 sequências de Archaea em amostras de solo foi
bastante interessante, pois somente uma sequência parcial de Archaea havia sido
identificada anteriormente neste habitat. A maioria das sequências provenientes de
Bacteria tinha pouca similaridade com sequências encontradas em estudos anteriores,
além de nenhuma sequência ser repetida, demonstrando que o solo Amazônico tem grande diversidade ainda não explorada. Pelas análises de RISA, foi comprovado que há uma grande divergência na diversidade microbiana entre o solo da floresta intacta e o solo utilizado para criação de gado, o que demonstra que o desmatamento de áreas naturais afeta seriamente a biodiversidade microbiana do solo. Por fim, a amplificação de somente 100 sequências de 16S rRNA não é o bastante para se caracterizar a biodiversidade microbiana do solo Amazônico e estudos mais aprofundados precisam ser realizados (BORNEMAN e TRIPLETT, 1997).
Em 2007, Fierer et al. realizaram outro estudo inovador na caracterização de microorganismos presentes no solo. Neste estudo, os pesquisadores utilizaram técnicas de análise de sequências provenientes de metagenoma e 16S rRNA para comparar a diversidade filogenética de bactérias, arquéias, fungos, e vírus em solos provenientes de uma pradaria de gramas altas, um deserto árido e floresta Amazônica no Peru. Foram analisadas 1.129 sequências para bactérias, 527 sequências para arqueias, 929 sequências para fungos e 4.577 sequências para vírus (números totais de sequências de cada grupo incluindo todas as amostras ambientais). As sequências de bactérias, arquéias e fungos foram estudadas por análise de 16S rRNA (procariotos) e 18S rRNA (eucariotos). Estas foram agrupadas em OTUs (Operational Taxonomic Unit) com 97% de similaridade e foram feitas curvas de rarefação para cada grupo em
28
a distribuição de OTUs dos mesmos foi estimada por um modelo matemático (FIERER et al., 2007).
Nenhuma curva de rarefação atingiu uma assíntota, indicando que as diversidades das bactérias, arquéias e fungos não foram amostradas o suficiente e que métodos não-paramétricos não podem ser utilizados para estimar o número total de OTUs de cada comunidade. Portanto, foram utilizadas técnicas paramétricas baseadas na distribuição de abundância de OTU para predizer a diversidade destes grupos em nível de comunidade, assumindo que a abundância da distribuição de OTU é igual para bibliotecas e comunidades. Para os vírus, foi utilizado um modelo matemático para predizer a abundância da distribuição de OTUs. Neste estudo pode-se comprovar que a riqueza em nível de OTU de todos os grupos – bactérias, arquéias, fungos e vírus – são muito altos em todos os locais amostrados, onde a riqueza das arquéias, fungos e vírus são iguais ou até mais altas do que a riqueza de bactérias de solo em todas as amostras (FIERER et al., 2007).
Também foi concluído que há uma grande diversidade de bactérias, arquéias, fungos e vírus ao nível local e global, e que nenhuma das OTUs de arquéias, fungos ou bactérias foram encontradas em mais de um local. Quanto aos vírus, somente uma OTU foi encontrada em dois locais diferentes. Nenhum dos solos estudados continha a comunidade de microrganismos mais diversa. Por fim, os autores concluíram que a diversidade de microrganismos do solo somente está começando a ser explorada e que a maioria da diversidade microbiana do planeta continua não sendo descoberta (FIERER et al., 2007).
Estrutura da Parede Celular de Plantas
A parede celular de plantas é complexa e composta por vários polímeros, incluindo a celulose que é o polissacarídeo mais abundante no planeta. Não obstante, a celulose é extremamente difícil de ser degradada, pois tem uma estrutura quase cristalina e insolúvel, além de estar envolvida pelos outros polímeros da parede celular, o que dificulta ainda mais o seu acesso (WILSON, 2009).
29
celular de plantas. Na parte externa de todas as células de plantas, existe uma parede celular primária produzida no começo do ciclo de vida da célula, que começa quando a célula-mãe ainda está se dividindo, e se estende até a fase de crescimento (CARPITA e GIBEAUT, 1993). Geralmente, esta parede é uma cadeia de microfibrilas de celulose interligadas e cobertas por hemiceluloses reticuladas. As duas juntas estão localizadas dentro de uma matriz de polissacarídeos ácidos que contém várias enzimas e proteínas estruturais que auxiliam na construção e degradação da parede celular (VERGARA e CARPITA, 2001).
Existem dois tipos de parede celular primárias em angiospermas: tipo I, compostas de xiloglucanas incorporadas em um gel de pectinas reticuladas com proteínas estruturais (dicotiledôneas e monocotiledôneas liliáceas) e tipo II, compostas de glucurono-arabinoxilanas e ligações (1,3), (1,4) β-D-glucanas diferenciadas, as quais são incorporadas em uma cadeia de polissacarídeos ácida com glucorono-arabinoxilanas altamente substituídas com quantidades pequenas de pectina e proteína (poales e comelinoides-monocotiledôneas) (CARPITA e GIBEAUT, 1993; CARPITA; MCCANN e ROBERTS 2008).
30
de paredes celulares secundárias ricas em xilana e celulose (MELLEROWICZ et al., 2001).
Entender bem a estrutura desta parede celular secundária é de suma importância, pois o material lignocelulósico que será utilizado na produção de biocombustíveis provavelmente será proveniente de gramíneas senescentes ou madeiras de rápido crescimento, as quais mantêm grande parte de seus polissacarídeos guardados nos tecidos da parede celular secundária (HARRIS e DEBOLT, 2010).
A parede celular serve como um dispositivo para a construção da planta. Ela envolve células novas da planta fornecendo suporte estrutural e este suporte é ligado por uma matriz de polissacarídeos flexível e composta de hemicelulose e polissacarídeos ácidos ou pectina, os quais provêm flexibilidade para a expansão da célula (Willats et al., 2001). Nas paredes celulares envolvendo células de tecidos mais maduros de plantas herbáceas ou de troncos e galhos de árvores, a celulose está cercada de lignina que proporciona mais força, resistência a água e proteção contra patógenos (CAMPBELL e SEDEROFF, 1996).
Para produzir os açúcares contidos nas paredes celulares, as plantas captam energia do sol e o CO2 da atmosfera e convertem-nos em energia através do processo
de fotossíntese. Este fato é o que torna o material lignocelulósico o candidato ideal como fonte de energia para a produção de biocombustíveis de segunda geração, já que a disponibilidade de raios solares e dióxido de carbono que são fixados pelas plantas são abundantes e infinitos. Além do mais, as plantas são adaptadas para sobreviverem em qualquer bioma do planeta (WRIGHT et al., 2004). Estima-se que 1011 toneladas de biomassa vegetal, na qual a grande parte vem de parede celular, são hidrolisadas anualmente por enzimas provenientes de microrganismos e a energia liberada equivale a 640 bilhões de barris de petróleo (BOUDET et al., 2003; RAGAUSKAS et al., 2006).
A Desconstrução da Parede Celular
31
quebram ligações β-1,4 que ligam duas glicoses adjacentes na celulose. Existem três tipos de celulases: endo-β-1,4-glicanases, exo-β-1,4-glicanases e β-1,4-glicosidases (NIDETZKY et al., 1994; WILSON e IRWIN, 1999).
As endoglicanases hidrolisam sítios internos amorfos da celulose, gerando oligossacarídeos de tamanhos variados e com terminais diferentes. Já as exoglicanases hidrolisam os terminais da celulose microcristalina em um processo onde cadeias celulosídicas são removidas da estrutura cristalina, gerando glicose e celobiose como produtos finais. Por fim, as β-glicosidases transformam celodextrinas e celobioses solúveis em glicose. A maioria das celulases tem uma estrutura modular com atividade catalítica e de ligação a carboidratos denominada CBM ( carbohydrate-binding module, em inglês). Esta estrutura se liga a superfície da celulose, o que traz o
domínio catalítico da enzima mais próximo à superfície do substrato, um fato de suma importância para a atividade das exoglicanases. Por fim, as celulases são muito diversas: elas têm onze famílias diferentes classificadas por similaridade de sequências, onde cada família pode conter um ou mais tipos de celulase (WILSON, 2009).
Os organismos aeróbicos degradam a celulose através de enzimas que são secretadas ou localizadas na membrana externa dos mesmos (TOMME, WARREN e GILKES, 1995; WARREN, 1996). Por outro lado, microrganismos anaeróbicos degradam a celulose utilizando complexos enzimáticos chamados celulossomos (BEGUIN e LEMAIRE, 1996) que são bastante eficazes na degradação da parede celular de plantas (UVERSKY e KATAEVA, 2006). A estrutura básica de um celulossomo tem duas partes: a primeira é composta por uma proteína de arcabouço não-enzimática com sítios de ligação de enzimas chamadas cohesins e um CBM; a
segunda parte contém enzimas variáveis com dockerins que ligam as enzimas às cohesins (DOI, 2008). Em 2003, foi publicado por Xu que esta proteína de arcabouço
pode ligar até 96 enzimas (XU et al., 2003). As enzimas que fazem parte deste complexo incluem celulases, xilanases, pectinases, mananases, esterases, liquenases e outras (DOI, 2008), todas utilizadas na degradação da parede celular de plantas.
32
chamadas expansinas (CAREY e COSGROVE, 2007), proteínas de fungos homólogas a expansinas chamadas “swollenins” (YAO et al., 2008) e uma proteína parecida a uma expansina que é encontrada em Bacillus subtilis (KERFF et al., 2008). Por fim, as
celulases já foram encontradas em fungos, bactérias, plantas, protistas e em vários invertebrados que a produzem internamente, incluindo insetos, crustáceos, anelídeos, moluscos e nematóides (WATANABE e TOKUDA, 2001; DAVISON e BLAXTER, 2005). Para o processo de bioconversão do material lignocelulósico em bioenergia, é necessário fazer o pré-tratamento do material para quebrar a estrutura da lignina e romper a estrutura cristalina da celulose, melhorando a acessibilidade das enzimas celulolíticas durante o passo de hidrólise (MOSIER et al., 2005). Apesar de ser um passo crucial na conversão de material lignocelulósico em bioetanol, o pré-tratamento da biomassa representa um dos custos econômicos mais altos no processo (MOSIER et al., 2005).
Diferentes materiais lignocelulósicos têm características físico-químicas diferentes e por isso existe a necessidade de utilizar tecnologias de pré-tratamento adequadas para cada tipo de biomassa (ALVIRA et al., 2009). Além do mais, a escolha do pré-tratamento tem alto impacto nos subsequentes passos do processo de bioconversão (GALBE e ZACCHI, 2007). Um pré-tratamento efetivo gera alto rendimento do produto final com poucos resíduos tóxicos e custo mais baixo (YANG e WYMAN, 2008).
33
isso, há uma grande necessidade de se encontrarem enzimas que sejam tolerantes a situações de alto estresse envolvidas na fase de pré-tratamento da biomassa.
Depois do pré-tratamento da biomassa, é então feita a hidrólise enzimática para quebrar a celulose e hemicelulose em monômeros que posteriormente serão fermentadas por outros microrganismos para gerar álcool. Geralmente é utilizado um coquetel de enzimas –endoglicanases, exoglicanases e β-glicosidases – para que este processo ocorra eficientemente. Estes processos de hidrólise e fermentação podem ocorrer juntos (SFF – simultaneous saccarification and fermentation, em Inglês) ou
separados (SHF – separate hydrolysis and fermentation, em Inglês) (SOCCOL et al.,
2009). Esta decisão é feita baseada no material a ser utilizado; ou seja, se os dois processos são feitos simultaneamente, o material a ser escolhido terá que ser mais específico, mas se os processos ocorrem separadamente, haverá mais opções sobre o que utilizar (MABEE e SADDLER, 2009).
Recentemente, no ano de 2010, a empresa Novozymes lançou um coquetel de enzimas chamado Cellic® CTec2. Este complexo de enzimas contém alta atividade de β-glicosidase, além de ter hemicelulases e um intensificador da atividade celulolítica para que ocorra a hidrólise completa do material, gerando assim mais etanol. O fato inovador deste produto é que ele pode ser utilizado em menor quantidade do que outras enzimas que já existiam no mercado, gerando o mesmo tanto de etanol ou até mais do que os concorrentes, e ele também pode ser utilizado em conjunto com qualquer tipo de pré-tratamento, sendo tolerante a inibidores produzidos durante diferentes tipos de pré-tratamentos (NOVOZYMES, 2010).
Outras Enzimas para a Produção de Bioenergia
34
com alto grau de ramificações conectadas por ligações glicosídicas α,1-4 e α,1-6 (AKOHet al., 2008).
Atualmente, o processo industrial de hidrólise do amido se baseia em dois passos. Primeiramente, o amido é liquidificado, ou seja, seus grânulos são solubilizados em dextrinas de menor peso molecular. O segundo passo do processo é a sacarificação, onde os oligossacarídeos são hidrolisados por uma β-amilase (maltose como produto final) ou uma glucoamilase (glicose como produto final). O produto final da sacarificação pode então ser fermentado para a obtenção do bioetanol ou então utilizado na preparação de diversos alimentos. Dentre as matérias-primas ricas em amido que podem ser utilizadas para a produção do bioetanol estão o arroz, milho, mandioca, sorgo, trigo e batata (AKOH et al., 2008).
Para que as amilases tenham acesso aos grânulos de amido, este carboidrato precisa ser separado da matriz proteica, ou glúten, durante o pré-tratamento do material a ser utilizado. Este processo pode ser pela via química ou pela via enzimática, ou pela combinação de ambos, onde as proteases – enzimas que degradam proteínas – podem ser utilizadas. Por outro lado, este processo de proteólise não pode ser extensivo a ponto de degradar totalmente o glúten, pois há interesse na recuperação deste para outros usos, a exemplo da indústria alimentícia. Portanto, nem todas as proteases são passíveis de serem utilizadas no processo. Por último, a utilização das proteases diminui o uso de sulfitos e o tempo de saturação dos grânulos de amido, tornando o processo mais rápido e possivelmente mais barato (JOHNSTON e SINGH, 2005; RAMIREZ et al., 2009).
Outras enzimas que podem ser utilizadas na produção de biocombustíveis são as lipases. As lipases, ou triacilglicerol acil hidrolases (EC 3.1.1.3), são enzimas que sintetizam ésteres alquílicos de ácidos graxos, hidrolisam triacilglicerídeos para produzir ácidos graxos livres e glicerídeos, e também esterificam ácidos carboxílicos com o auxílio de alcoóis (NIELSEN, BRASK e FJERBAEK, 2008). Devido a sua afinidade a óleos e seu produto final, as lipases podem ser utilizadas na produção de biodiesel através de óleos vegetais.
35
(utilizando bactérias como biocatalizadores), todos utilizando matérias-primas renováveis (ADAMCZAK, BORNSCHEUER e BEDNARSKI, 2009). Em comparação com o diesel, o biodiesel não é toxico e contém capacidade de combustão aprimorada. Atualmente, os óleos mais utilizados para produzir o biodiesel são derivados de palmeira, soja e colza (ROTTIG et al., 2010). Na indústria, o biodiesel é produzido através da transesterificação de triacilglicerídeos de óleos vegetais com o uso de metanol, mas outros alcoóis podem ser utilizados, como o etanol (AL-ZUHAIR, 2007). Para o Brasil, a utilização de etanol para produzir biodiesel seria uma boa alternativa, pois no país este álcool é produzido por um custo muito menor (HAAS et al., 2006). Além do mais, o etanol apresenta toxidade muito mais baixa que o metanol (ALBUQUERQUE et al. , 2009).
As primeiras tentativas para produzir biodiesel pela via enzimática utilizavam enzimas livres e imobilizadas. A obtenção de enzimas livres é a mais barata, mas por outro lado, sua utilização não é economicamente viável porque elas são mais difíceis de reutilizar e necessitam de um tempo prolongado para concluir a reação (NIELSEN, BRASK e FJERBAEK, 2008). Por outro lado, as enzimas imobilizadas custam mais para se obter, mas podem ser reutilizadas várias vezes e ficam estáveis a temperaturas mais altas, aumentando a velocidade da reação. No fim, as enzimas imobilizadas acabam se tornando mais economicamente viáveis (DU et al., 2008).
36
37 3 JUSTIFICATIVA
O Brasil foi o primeiro país no mundo a produzir etanol a partir de cana-de-açúcar em larga escala e utilizá-lo como combustível, uma ação que diminuiu a dependência do país na importação de petróleo (EMBRAPA, 2006). Apesar deste sistema brasileiro de produção de etanol ser considerado o mais eficiente (GOLDEMBERG, 2007), a quantidade de etanol produzido ainda não é suficiente para substituir o uso de gasolina e preencher a demanda total de combustíveis no país. Além do mais, a utilização da cana-de-açúcar sozinha não supre a quantidade de matéria-prima necessária para produzir todo o etanol demandado. Uma opção interessante seria a utilização de material lignocelulósico para produzir o bioetanol de segunda geração, onde poderia ser utilizado como matéria-prima o bagaço da cana, dejetos municipais lignocelulósicos, restos industriais, dentre outros (SOCCOL et al., 2009). Por outro lado, ainda não foram desenvolvidas técnicas que sejam ambas eficazes e economicamente viáveis para se produzir etanol de segunda geração, pois a parede celular de plantas é muito complexa e de difícil acesso para ser degradada.
Para catalisar a hidrólise destes materiais lignocelulósicos, as enzimas de microrganismos seriam uma alternativa atrativa, pois estas já têm sido utilizadas em processos industriais por anos. Uma técnica de sucesso para a identificação de novas enzimas é a metagenômica, onde os microrganismos de amostras ambientais podem ser estudados sem a necessidade de cultivo (HANDELSMAN, 2004), já que somente 1% destes pode ser cultivado (TORSVIK, GOKSOYR e DAAE, 1990; TORSVIK e OVREAS, 2002). Já foram feitos estudos metagenômicos de diversas amostras ambientais, incluindo rúmen, águas de rios e mares, lama, e outros, onde enzimas de atividades variadas foram identificadas (ENTCHEVA et al., 2001; GILLESPIE et al., 2002; HENNE et al., 1999; HENNE et al., 2000; KNIETSCH et al., 2003; MACNEIL et al., 2001; MAJERNIK, GOTTSCHALK e DANIEL, 2001; RONDON et al., 2000; VOGET et al., 2003).
38
microrganismos e as enzimas produzidas pelos mesmos poderiam ser utilizadas na produção de bioenergia. O primeiro estudo de diversidade com o solo Amazônico brasileiro foi realizado em 1997 por Borneman e Triplett, onde foi estabelecido que neste solo existisse uma grande biodiversidade de microrganismos ainda não explorada (BORNEMAN e TRIPLETT, 1997). Em 2007, Fierer e parceiros também fizeram um estudo com diversos solos, incluindo o solo Amazônico do Peru, onde também foi concluído que a diversidade microbiana dos solos ainda está longe de ser completamente caracterizada (FIERER et al., 2007).
39 4 HIPÓTESES
A metagenômica já foi empregada com sucesso em vários trabalhos anteriores, onde genes codificando novas enzimas foram encontrados. O solo é o habitat para milhares de microrganismos desconhecidos que fazem parte da base da cadeira alimentar, cujas enzimas estão constantemente degradando matéria lignocelulósica e outros compostos. Ao realizar um estudo metagenômico com o solo Amazônico brasileiro, material ainda inexplorado por esta perspectiva, novas enzimas poderão ser identificadas e futuramente serem utilizadas em processos biotecnológicos.
40 5 OBJETIVOS
Geral
Construir uma biblioteca metagenômica de pequenos insertos com DNA proveniente de microrganismos presentes no solo Amazônico brasileiro para a obtenção de genes que codifiquem para enzimas de interesse biotecnológico, com foco nas celulases para a produção de bioenergia.
Específicos
Construir biblioteca metagenômica de pequenos insertos com o DNA de microrganismos presentes no solo Amazônico brasileiro;
Buscar genes que codifiquem para atividades enzimáticas de interesse
biotecnológico através de triagem funcional da biblioteca, focando na obtenção de celulases;
Sequenciar os genes encontrados;
41 6 MATERIAIS E MÉTODOS
Coleta do solo
42
Figura 2 - Método de amostragem do solo coletado. Foram coletados cinco pontos com 30 metros de distância. Dentro de cada ponto, foram coletados cinco sub-pontos com 10 centímetros de distância e estes foram juntados na mesma amostra.
43
Biblioteca Metagenômica
Extração do DNA Total
O DNA total da amostra foi extraído de acordo com o protocolo desenvolvido pelo Dr. Alexandre Rosado (comunicação pessoal). Foram adicionadas 3 g de glass beads de aproximadamente 200 µm juntamente com 5 mL de tampão fosfato de sódio
100 mM à uma amostra de 2 g do solo e agitado rigorosamente 3 vezes por 90 segundos com intervalos de 10 segundos. Logo após, a mistura passou por extração com 4 mL de fenol:clorofórmio (25:24) e foi centrifugada a 4.500 g por 5 minutos. Em
seguida, foi feita outra extração utilizando clorofórmio:álcool isoamílico (24:1) e foi centrifugado a 4.500 g por outros 5 minutos. O DNA contido na parte aquosa foi então
precipitado com 500 µL de cloreto de sódio 5 M e etanol 100% a -20°C por cerca de 1 hora. A mistura foi então centrifugada a 18.000 g por 10 minutos e depois lavada com
etanol 70% duas vezes. O pellet resultante foi ressuspendido a 55 °C por 5 minutos e
depois aliquotado em tubos eppendorf de 1,5 mL contendo 0.1 g de cloreto de césio
cada. A mistura foi homogeneizada e repousada por 1 hora à temperatura ambiente. Seguido disso, as amostras foram centrifugadas a 16.000 g por 20 minutos e o DNA
contido no sobrenadante foi precipitado com 300 µL de isopropanol e 400 µL de água milli-Q por 5 minutos à temperatura ambiente e centrifugado a 16.000 g por 15 minutos.
O sobrenadante foi descartado e o processo anterior foi repetido. Posteriormente, o
pellet de DNA foi lavado duas vezes com etanol 70%. Assim que o pellet secou, ele foi
ressuspendido com água milli-Q. Para purificar o DNA, este foi corrido em gel de ágar ose low melting 1% (m/v) feito com tampão TAE sem brometo de etídeo por um período overnight a 30 V com o marcador 1kb plus DNA ladder (Invitrogen). A parte do gel com
o marcador foi cortada e corada com brometo de etídeo para servir de referência sobre a localização do DNA de interesse. Para eluir o DNA do gel, foi utilizado o kit de eluição
44
Biblioteca Metagenômica de Pequenos Insertos
- Vetor pCF430
O vetor utilizado na construção da biblioteca de pequenos insertos foi o pCF430
(Figura 4), um vetor desenvolvido para clonagem em E. coli (NEWMAN; FUQUA,
1999). Seu promotor controlando a expressão do DNA clonado é induzido pela presença de L-arabinose na concentração de 0,02% (m/v) e seu antibiótico de seleção é tetraciclina na concentração de 20 µg/mL (SAMBROOK, FRITSCH e MANIATIS, 1989; NEWMAN e FUQUA, 1999).
Figura 4 – Mapa do vetor utilizado na construção da biblioteca metagenômica de pequenos insertos, pCF430 (NEWMAN e FUQUA, 1999).
- Construção da Biblioteca
O vetor pCF430 foi digerido com a enzima de restrição PstI (Promega) overnight
a 37°C. Este foi purificado em gel de ágar ose 0,8% (m/v) com tampão TBE e eluído do
gel utilizando o kit de purificação caseiro Ultraclean. Após eluído, o vetor foi
desfosforilado utilizando a enzima SAP (Promega) de acordo com as especificações do fornecedor.
Concomitantemente, o DNA total da amostra de solo também foi digerido com a enzima de restrição PstI (Promega) e analisado em gel de ágar ose low-melting 1%
(m/v) com tampão TAE sem brometo de etídio. Com base no marcador 1Kb plus DNA ladder, que também foi corrido no gel e depois corado com brometo de etídio, a região
45
inserido no pCF430 (digerido com PstI e desfosforilado) utilizando a enzima T4 DNA ligase (Promega) de acordo com as especificações do fornecedor. O vetor com inserto
foi inserido em E. coli eletrocompetente cepa EPI300 (Epicentre) por meio de
eletroporação. O eletroporador (Biorad) foi ajustado com capacitância de 25µF, resistência a 200Ω e 700Ω e voltagem a 2,5Kv (proporção DNA:célula competente de 250ng : 50 µL), e as células eletroporadas foram adicionadas a 1 mL de meio SOC e incubadas a 37°C por uma hora. Essa transformação foi plaqueada em meio LB ágar com tetraciclina a 20 µg/mL e os clones resultantes foram raspados das placas e guardados em glicerol com concentração final de 20% (v/v) a -80°C. A construção da biblioteca teve duas etapas e as frações da biblioteca foram denominadas “biblioteca 1” – proveniente da amostra A1#1 –e “biblioteca 2” – proveniente da amostra A1#2 – com 30.000 e 40.000 clones respectivamente. Os números dos clones foram estimados contando o número de colônias de metade das placas e multiplicando este por dois.
- Isolamento de Clones Aleatórios em Placas Deep-well
Alguns tipos de bioensaios para identificar atividades enzimáticas são destrutivos; portanto, existe a necessidade de clones guardados que possam ser acessados caso apresentem atividade enzimática de interesse. Como as duas frações da biblioteca foram guardadas em criotubos contendo todos os clones, foi necessário isolar alguns clones e armazená-los em placas deep-well para a realização de triagens
destrutivas. As duas frações da biblioteca foram diluídas e plaqueadas em meio LB ágar com tetraciclina a 20 µg/mL. Posteriormente, foram adicionados 120 μL de caldo LB com tetraciclina a 20 µg/mL em cada poço de placas deep-well e as colônias
isoladas foram inoculadas neles. As placas foram mantidas a 37°C por um período
overnight e no dia seguinte foram adicionados 80 μL de glicerol 50% (v/v) em cada
poço e as placas foram acondicionadas a -80°C para uso posterior. Cada placa deep-well acondiciona 96 clones. Por motivos de espaço e tempo, não foi possível guardar