Il convient également de noter que jusqu’à présent, peu de travaux ont examiné les effets de l’hybridation interspécifique et de la duplication du génome sur la dynamique évolutive des allopolyploïdes. Le chapitre cinq explore les effets de l'hybridation et de la duplication du génome sur le transcriptome de Spartina.
Spéciation dichotomique et divergence transcriptomique
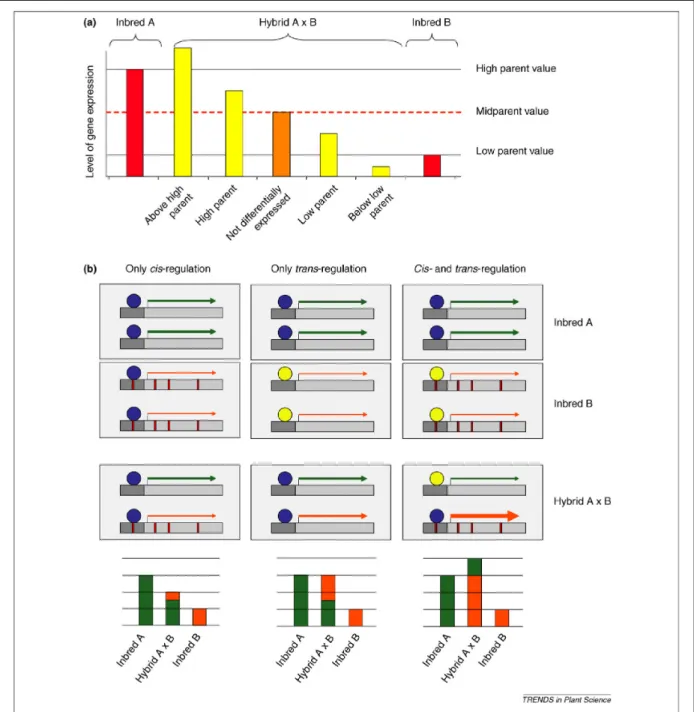
La variation de l'expression des gènes résulte de nombreux mécanismes génétiques impliquant des polymorphismes dans les régions codantes, promotrices ou régulatrices (Wittkopp et al. 2004) et des mécanismes épigénétiques (Jaenisch & Bird 2003). Différentes catégories de gènes chez les Brassicaceae présentent une divergence transcriptomique corrélée à la distance phylogénétique (d'après Broadley et al. 2008).

Evolution réticulée, polyploïdie et évolution du transcriptome
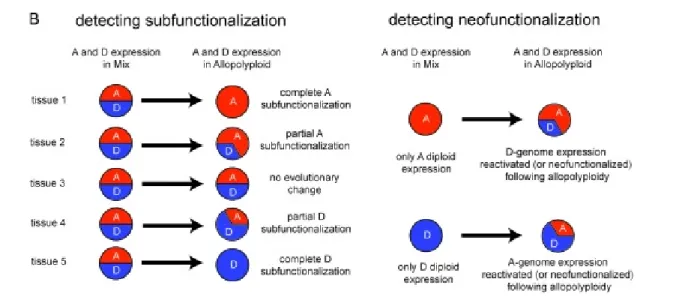
Les profils d'expression de l'hybride Helianthus deserticola ont été comparés à ceux des parents par micropuce (Lai et al. 2006). Les autopolyploïdes ont été relativement peu étudiées dans le contexte de l'évolution du génome (pour revue Parisod et al. 2010b).

Les spartines sont caractérisées par un métabolisme photosynthétique C4 (Long et al. 1975, Maricle et al. 2009). Le rôle de cette molécule chez les plantes est encore débattu (Otte et al. 2004). On pense qu’il est impliqué dans divers mécanismes de tolérance au stress.

Evolution génétique
Ces dynamiques semblent également s'observer dans l'environnement d'éléments transposables (Parisod et al. 2009, Ainouche et al. 2009). Ces changements épigénétiques semblent encore plus importants dans les régions adjacentes aux éléments transposables (Parisod et al. 2009).
Matériel biologique
Analyses du transcriptome 1. Choix méthodologiques
Une puce anonyme de sonde d'ADNc de tournesol a permis de montrer l'effet de l'hybridation chez l'hybride homoploïde Helianthus deserticola (Lai et al. 2006). Cette méthode a également été utilisée pour étudier les conséquences de l'autopolyploïdie chez la pomme de terre (Solanum tuberosum) (Stupar et al. 2007).
Microarrays have become a central tool in the comparison of genome-wide expression patterns (Lockhart & Winzeler 2000; Ji et al. 2004). These approaches are now increasingly being used in wild species (reviewed in Kammenga et al. 2007) to understand evolutionary and adaptive processes: e.g. Spartina is composed of 13–17 perennial species exhibiting different ploidy levels (basic chromosome number x = 10), ranging from tetraploid to dodecaploid (Ainouche et al. 2004a and references therein).
Spartina species have a C4 photosynthetic system that uses the ATP-dependent phosphoenolpyruvate carboxykinase (PCK) as a decarboxylating enzyme, like other Chloridoideae species (Christin et al. 2009; Maricle et al. 2007). It is introduced to the Pacific coast of California, western Europe (southern England, western France and north-eastern Spain) and China, where it has experienced spectacular expansion (An et al. 2007). All data replicates were normalized using the glyceraldehyde-3-phosphate dehydrogenase (GA3PDH), gene amplified with the primer sequences AGAGTGCCTCGTCAAGGAGA (F) and CTCCCAAGCAATCCTCATGT (R) (Baisakh et al. 2006).
Sequence quality files were generated using PHRED software (Ewing et al.1998; Ewing & Green 1998).
RESULTS
Introduction générale et démarche suivie
Par conséquent, ils représentent une région majeure susceptible d'être affectée par l'hybridation et la duplication du génome lors de la spéciation allopolyploïde (Parisod et al. 2010a). Plusieurs études ont montré que l'allopolyploïdisation est généralement associée à des changements structurels et fonctionnels rapides dans les génomes des régions adjacentes aux ET (Matzke & Matzke 1998, Comai et al. 2003). L’analyse du génome d’espèces polyploïdes plus ou moins anciennes révèle que des changements peuvent être provoqués par des éléments transposables sur une base génétique (par exemple Zhao et al. 1998 ; Petit et al. 2010), épigénétique (par exemple Parisod et al. 2009) ou transcriptionnelle. (par exemple Kashkush et al. 2003).
Les dynamiques d'amplification-délétion (« turnover ») des éléments transposables peuvent contribuer à la divergence plus ou moins rapide des génomes polyploïdes par rapport aux espèces ancestrales (Ozkan et al. 2003, Grover et al. 2007). Chez le blé allotétraploïde (AABB), les données de séquençage des régions génomiques du chromosome 3B dans les génomes A et B ont montré que l'allopolyploïdisation n'entraînait pas la propagation d'éléments transposables (Charle et al. 2008). Sur la corde, des éléments transposables commencent à être explorés au niveau structural et épigénétique, grâce à diverses méthodes de « transposon display » telles que l'IRAP (Enhanced Inter-Retrotransposon Polymorphism), REMAP (Enhanced Retrotransposon-Microsatellite Polymorphism) de Baumel et al. 2002b), et MSTD (Méthyl-Sensitive Transposon Display) de Parisod et al.
Nous avons comparé nos résultats avec ceux obtenus dans l'équipe (1) sur l'ADN génomique ciblant les mêmes éléments dans le brin (Parisod et al. 2009) et (2) à partir d'AFLP d'ADNc dans les mêmes échantillons (Dauvergne et Pourtau, données non publiées) .

Résultats
Les effets de l'hybridation ont été étudiés en comparant les profils de restriction obtenus chez l'hybride F1 (en SSAP de l'ADNc et de l'ADN génomique) avec ceux des deux parents. L'écart à l'additivité des profils parentaux chez l'hybride est calculé en comparant les fragments observés avec un profil « théorique » résultant de l'addition des fragments (monomorphes et polymorphes) présents chez les deux espèces parentales observées. L'additivité des profils parentaux est significativement plus élevée (82 à 92 %) dans le cas de l'ADN génomique (par rapport à l'ADNc), l'élément Ins2 présentant l'additivité la plus élevée.
L'effet d'hybridation dans l'environnement des trois éléments transposables (en sommant les trois profils des 3 ET) est statistiquement beaucoup plus important au niveau transcriptomique (écart à l'additivité de 35%) qu'au niveau génomique (écart à l'additivité de 13,6%). ) (rapport Z = 5,72). Ceci indique un effet plus marqué de l'hybridation sur le transcriptome dans les régions flanquant ces éléments transposables. Additivité avec profil parental attendu (%) Additivité avec "PAT" (%). a) Additivité des fragments parentaux dans l'hybride pour les 3 TE au niveau transcriptomique (SSAP de l'ADNc) et génomique (SSAP de l'ADN génomique) (b) Comparaison de l'additivité des fragments parentaux dans l'hybride et de l'allopolyploïde dans l'hybride l'environnement des trois ET (SSAP de l'ADNc) et sur l'ensemble du transcriptome (AFLP de l'ADNc).
Contrairement à l'événement d'hybridation, une différence significative est détectée au niveau génomique entre les profils AFLP et les régions adjacentes des ET (rapport z = 3,92).

Discussion
Au niveau du génome global, la différence d'additivité entre l'hybride et le profil additif théorique des deux parents est de 6 % (Salmon et al. 2005). Cet écart est doublé (13,6 %) au voisinage des trois ET étudiés par SSAP génomique selon Parisod et al. Nos analyses SSAP ont permis d'étudier le polymorphisme de 179 fragments d'ADNc pour les trois éléments, contre 338 fragments obtenus pour les mêmes éléments avec l'ADN génomique (Parisod et al. 2009).
L'effet de l'hybridation au voisinage des éléments transposables semble être plus important au niveau du transcriptome qu'au niveau génomique, comme Parisod et al. Ces résultats sont en accord avec les analyses du transcriptome réalisées par microarray (Chelaifa et al. 2010), qui révèlent une dominance de l'expression des gènes du parent maternel (S. alterniflora) dans l'hybride S, selon la revue déjà réalisée par Parisod et al. déjà 2010a), des modifications épigénétiques et une activité transcriptionnelle ont été détectées dans le blé allohexaploïde.
En revanche, dans le coton allotétraploïde, apparu il y a environ 2 millions d'années (MA), le taux de suppression des éléments transposables semble avoir dépassé le taux de duplication (Grover et al. 2008).

Introduction
La méiose des deux hybrides est irrégulière, mais le taux d'appariement des chromosomes (sous forme de trivalents) est plus élevé chez S. Cette différence de comportement des chromosomes, outre les différences morphologiques, renforçait l'idée d'une origine par croisement réciproque des espèces parentales, hypothèse qui fut réfutée par l'analyse des génomes chloroplastiques, qui révéla que les deux les hybrides avaient un génome chloroplastique identique à celui de S. Les analyses de la diversité des populations d'espèces parentales en Europe occidentale ont montré très peu de variation génétique entre les individus de S.
À ce jour, le compartiment transcriptomique de ces deux hybrides n’a jamais été analysé et nous émettons donc l’hypothèse qu’une différence de régulation (génétique ou épigénétique) de l’expression des gènes pourrait expliquer la divergence morphologique et écologique des deux hybrides. Quelle est la proportion et la nature des gènes différentiellement exprimés entre les deux hybrides, et pouvons-nous relier ces différences au niveau phénotypique ?
Matériel et méthodes
Résultats
En revanche, il est à noter que les gènes transgressifs détectés chez les deux hybrides sont complètement différents. Ces données suggèrent des différences transcriptomiques significatives entre les deux hybrides dans l'environnement des trois ET étudiés. La comparaison des gènes transgressifs et différentiellement exprimés entre les deux hybrides montre un nombre significatif de gènes transgressifs en cas d'activation (68) chez S.x neyrautii.
Il est à noter que les gènes différentiellement exprimés entre les deux hybrides représentent environ 1/3 des gènes différentiellement exprimés entre les deux parents S. Les gènes différentiellement exprimés entre les deux hybrides ont été identifiés à l'aide de Gene Ontology (GO ) en référence à la catégorie fonctionnelle des processus biologiques. Comme le montre le graphique (Figure 28), les gènes différentiellement exprimés entre les deux hybrides ont une répartition quasiment égale entre les différentes catégories.
Profils d'expression de gènes exprimés différentiellement entre les deux hybrides par rapport aux deux parents.
Discussion
À ce jour, la plupart des changements liés aux éléments transposables chez les hybrides ont été rapportés au niveau transcriptomique (Kashkush et al Parisod et al. 2010a). Nous avons encore peu de recul sur les comparaisons de la divergence de l'expression globale du génome estimée par microarrays entre taxons (voir discussion Chelaifa et al. Chapitre IV). À titre de comparaison, la divergence d'expression mesurée selon la même technique entre les espèces de spartine plus anciennes (qui divergeaient il y a environ 2 à 3 ans) a été estimée à 13 % (Chelaifa et al. 2010, chapitre V).
Chez les polyploïdes sauvages, Hegarty et al. 2005) ont évité les difficultés liées à l'absence de ressources génomiques du séneçon pour examiner les changements d'expression avec un tableau anonyme contenant de l'ADNc. Ce caractère transgressif joue un rôle important dans les capacités adaptatives des génomes hybrides (Lai et al. 2006, Hegarty et al. 2009). Ces régions du génome semblent également être plus affectées par les changements de méthylation de CpG après hybridation qu'après duplication (Parisod et al. 2009).
Conformément aux analyses phylogénétiques précédentes (Baumel et al. 2002, Fortuné et al. 2007), nous l'avons fait.