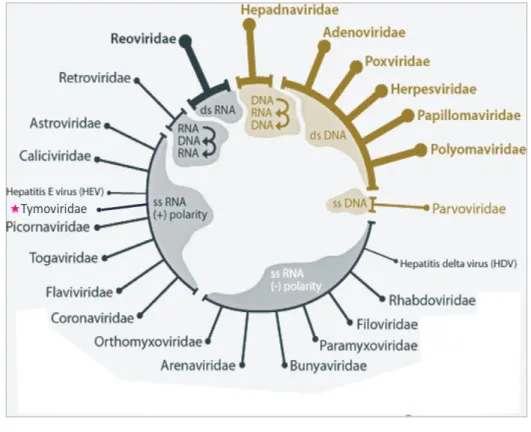
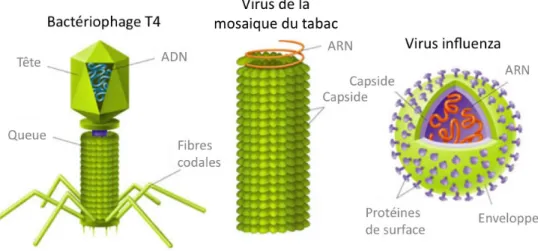
Le virus de la mosaïque jaune du navet (TYMV) est un excellent modèle pour la réplication des virus à ARN simple brin de sens positif. L'un des acteurs clés de la réplication est la polymérase qui permet la synthèse de nouveaux génomes. Le virus de la mosaïque du tabac est un virus en forme de spirale constitué simplement d'une nuclécapside cylindrique.
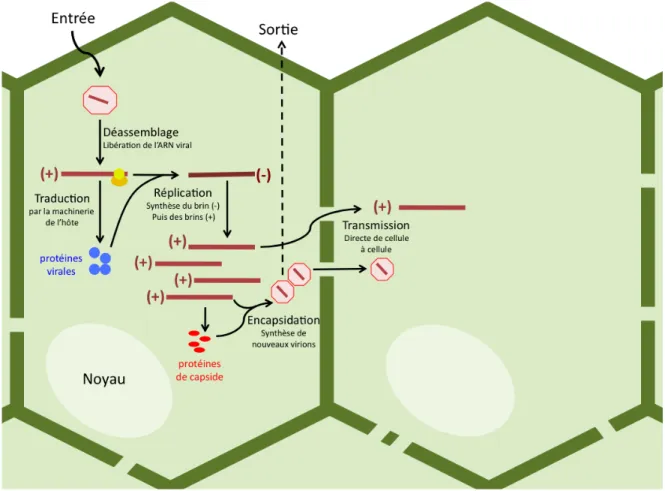
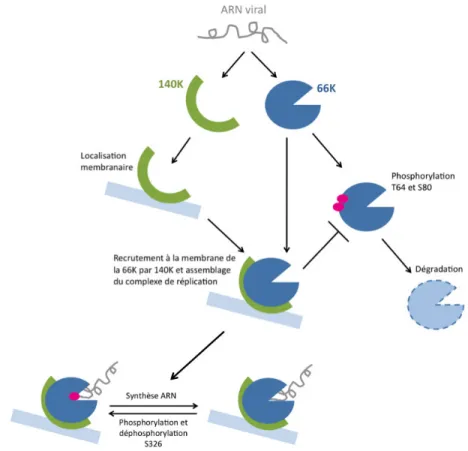
Ce complexe se retrouve toujours au niveau de structures membranaires spécifiques lors de la réplication des virus à ARN(+). Ceci sera directement traduit par la machinerie cellulaire hôte (en jaune) pour fournir les protéines virales nécessaires à la réplication (en bleu). Dans la deuxième partie, est représenté le mécanisme de protection lié à la protéase (en vert) qui peut inhiber cette dégradation en recrutant RdRp au niveau des structures membranaires chloroplastiques (en bleu clair) lors de la formation du complexe de réplication.
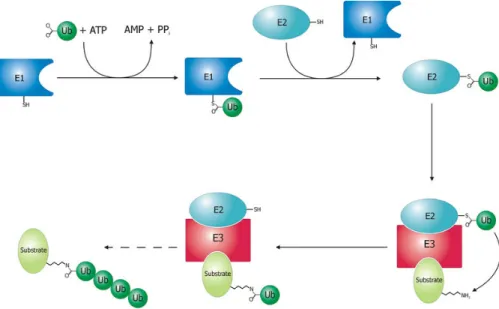
Stabilisation de la β-cathénine dans les cellules infectées par latence Augmentation de l'activité de la ribonucléotide réductase. Si nous disposons d’un tel modèle, nous pouvons utiliser sa structure comme première approximation de celle de la protéine étudiée. Dans le premier cas, les méthionines de la protéine en question sont remplacées par des sélénométhionines.
En utilisant la diffusion anormale, nous surmontons l’isomorphisme car nous n’avons besoin que d’un cristal contenant un atome lourd.
CRISTALLISATION ASSISTEE PAR UN CONTAMINANT (ARTICLE 1)
En résolvant la structure du domaine TYMVpro, nous avons réalisé qu'un contaminant était également présent dans l'unité asymétrique. Ce travail a été partiellement soutenu par l'Agence nationale de la recherche (contrat ANR-06-BLAN-0062) à IJ et SB. Dans cet article, la cristallogenèse et la résolution des structures vous ont été expliquées.
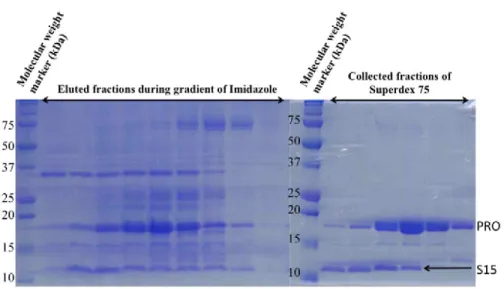
Lorsque les cristaux sont apparus, nous avons eu des problèmes avec un projet parallèle concernant la dissolution des gels de polyacrylamide. Nous avons donc analysé sur ces gels le rendement de la filtration sur gel utilisé pour les tests de cristallisation pour voir s'il était le même à ce moment-là (Figure 1, Article 1). Cela pourrait correspondre soit à un peptide dégradant la protéase, soit au contaminant observé lors de la purification.
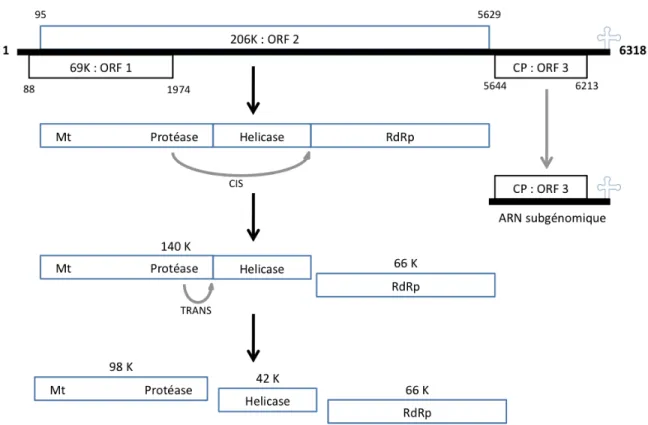
Cependant, un bug technique de la plateforme a retardé ce résultat de plusieurs semaines et deux jours avant que nous l'obtenions. Nous avons finalement résolu les phases par MIRAS, confirmant la présence de S15 dans l'empilement cristallin, comme expliqué dans l'article 1. Néanmoins, à titre purement informatif, nous avons essayé de savoir si nous aurions pu résoudre le problème des phases, à partir de la structure résolue de S15, par remplacement moléculaire. Considérant la masse moléculaire du domaine TYMVpro (18 kDa) et celle du contaminant S15 (10 kDa), nous avons décidé d'utiliser le module de remplacement moléculaire automatique de Phaser 2.3.0 inclus dans la suite Phoenix avec deux molécules S15 et également 2 possibles Molécules TYMVpro dans l’unité asymétrique.
Nous avons utilisé la chaîne O de la structure portant le code pdb 2QAL pour que le modèle de S15 utilisé corresponde à la sous-unité ribosomale présente dans E. Le programme a pu retrouver sans ambiguïté le groupe spatial P3121 et attribuer le modèle à 31% de l'électron asymétrique. densité unitaire. À partir de la carte calculée et de la structure partiellement reconstruite de S15, nous avons ensuite utilisé la fonction de reconstruction automatique du package phenix pour voir si le domaine TYMVpro pouvait également être reconstruit facilement et avec précision.
La contamination, généralement redoutée par les biocristallographes, montre ici que dans notre cas elle peut favoriser la cristallisation mais aussi la dissolution de la structure. Le contaminant décrit dans ces études s'est en réalité cristallisé seul, au détriment de la protéine d'intérêt (Kefala 2010, Cámara-Artigas 2006). Parfois, un contaminant ayant le même poids moléculaire que la protéine étudiée peut nous induire en erreur avant la cristallisation.
ETUDE STRUCTURALE DE LA PROTEASE DU TYMV (ARTICLE 2)
In the crystal, one molecule's C-terminus inserts into the catalytic cleft of the next, providing a view of the N-terminal product complex in replication polyprotein processing. This structure sheds light on both the two documented functions of PRO: First, one PRO's C-terminus sticks into the catalytic cleft of the next in the crystal. This provides us with a detailed overview of the N-terminal product complex in replication polyprotein processing.
Similarly, the long loop (residues 771-782) connecting the N-terminal lobe to the central lobe comes from the other side of the a3-b6 interface to helix a3. This feature is unusual in the closest relatives of the known structure of PRO (see below) and contributes to the exposure of the catalytic dyad at the exit of the catalytic site. The infinite spirals of PRO in the crystal are formed by inserting the C-terminus of one molecule into the catalytic site of the next.
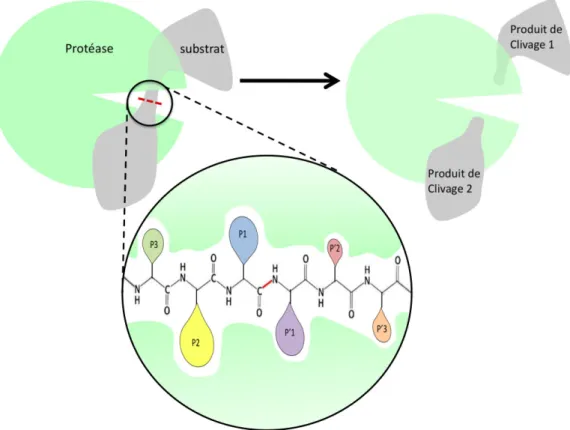
The residues involved in the interface are located mostly, but not exclusively (Fig. 4A), in and around the entrance to the catalytic cleft of the peptidase and in the C-terminus of the substrate. In our crystal structure, the side chain hydroxyl of Sers879 hydrogen bonds to the main chain nitrogen of the catalytic Cysp783. The relaxed G/A/S specificity at P1 largely arises from solvent exposure at the exit of the catalytic cleft (see above).
Similarly, the constriction of the cleft active site at the S2 pocket is less pronounced than in the closest relatives of the known structure (see below). Molecular recognition of the substrate by the peptidase further involves patches that are recessed relative to the C-terminus of the substrate (Fig. 4A). A second patch in this lobe is centered on Asnp760 at the end of the extended a2-b2 loop.
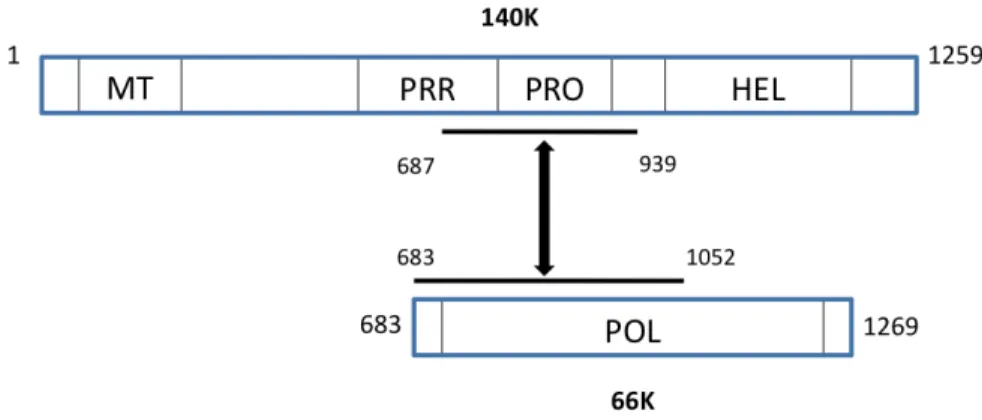
Our structure rules out the possibility that the C-terminus of PRO loops back to the entrance of the catalytic cleft. Therefore, cleavage of the TYMV replication polyprotein at the PRO/HEL junction (Fig. 1) occurs only in trans. It occurs in the replication complex that includes the two products of the first cleavage: the 66K RdRp and the 140K protein.
Crystal intermediate of the N-terminal complex of this trans cleavage reveals molecular determinants for peptidase sequence specificity. The first element specifically recognizes a characteristic bulge of two consecutive cis-prolines just before the catalytic His in the PRO substrate molecule.
OBTENTION D’UN FRAGMENT SOLUBLE DE LA RDRP DU TYMV
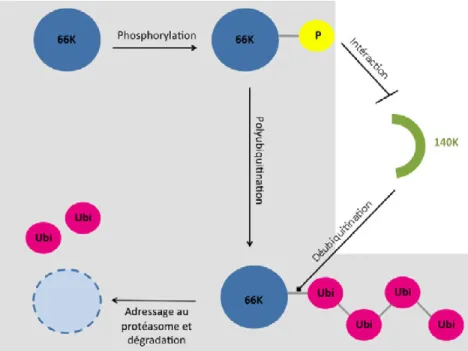
Situées dans la séquence PEST N-terminale, la thréonine 64 et la sérine 80, dans leur état phosphorylé, sont un signal d'ubiquitination 66K. On sait notamment que la polymérase du virus de la dengue de type 2 (Kapoor 1995), celle du virus de la mosaïque du concombre (Kim 2002) et du norovirus (Eden 2011) est phosphorylée. Ainsi, le domaine « TYMVpol » choisi est constitué des 585 derniers résidus de la polyprotéine et correspond à 66K au total.
Malheureusement, toute la polymérase TYMV exprimée se trouvait dans la fraction insoluble. Aucun de ces paramètres ne nous a donné de solution concernant la solubilité du 66K et l'ensemble était encore insoluble (Tableau 3). Là encore, aucune variation n'a permis de résoudre ne serait-ce qu'une partie de la protéine d'intérêt.
Après analyse par spectrométrie de masse de la bande correspondante, nous avons confirmé notre capacité à produire et purifier un E . Par dilution en série, nous n'avons pas pu obtenir du 66K stable en fin de renaturation. Enfin, pour garantir le repliement correct de la protéine, nous avons réalisé une expérience de dichroïsme circulaire ainsi que des tests d'activité.
Le spectre obtenu ici est celui résultant de la soustraction du signal correspondant au buffer. Les problèmes que nous avons rencontrés lors de nos tentatives de production de la polymérase TYMV peuvent s’expliquer par la biologie associée au virus. Naturellement, nous avons tenté de caractériser la séquence N-terminale désordonnée de notre domaine TYMVpro afin de déterminer.
Dans notre cas, nous connaissons effectivement un partenaire de la polymérase TYMV qui permet sa stabilisation : la protéase. L’étude de cette dernière a permis de mieux comprendre comment se produit cette maturation lors de la réplication virale. A partir de la protéase produite et purifiée, nous avons développé la formation d'un tel complexe ainsi que sa purification.
Le rôle de l’ubiquitination réversible de la polymérase et de l’effet protecteur de la protéase au cours du cycle viral n’est pas encore clair. Nous émettons d’abord l’hypothèse que l’ubiquitination est un moyen de défense de la cellule hôte.