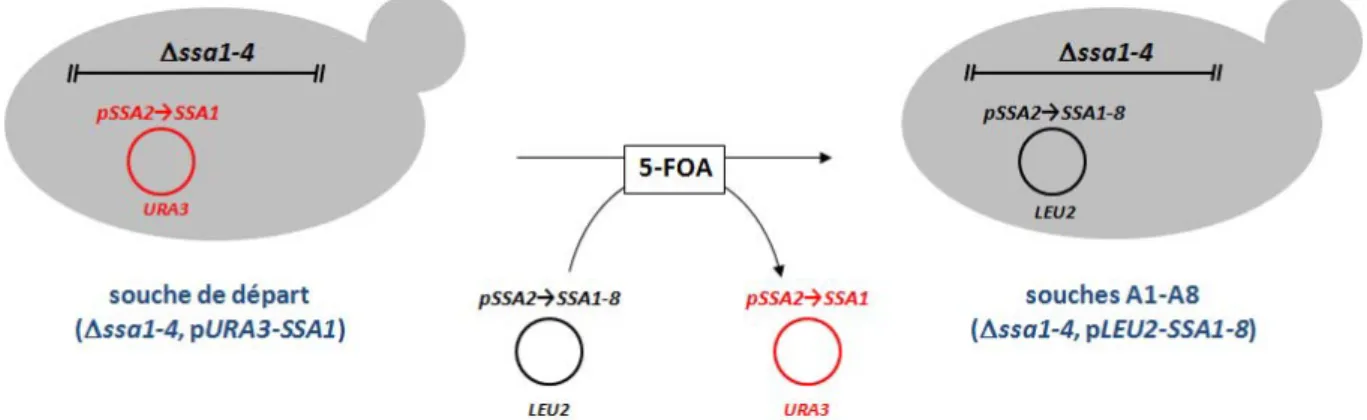
Je la remercie pour la qualité de son enseignement sur Yarrowia, mais aussi de m'avoir montré la voie de ses impressionnantes capacités d'organisation. La protéostasie consiste en un réseau de régulation qui s'étend de la transcription à la synthèse, au repliement, à l'assemblage, au trafic, à la désagrégation et à la dégradation des protéines. Les régulateurs de la protéostasie (cercles roses) interviennent à tous les niveaux du réseau et régulent le bon repliement des protéines et la dégradation des espèces allochtones.
B.1. De l’hypothèse thermodynamique aux paysages énergétiques de repliement
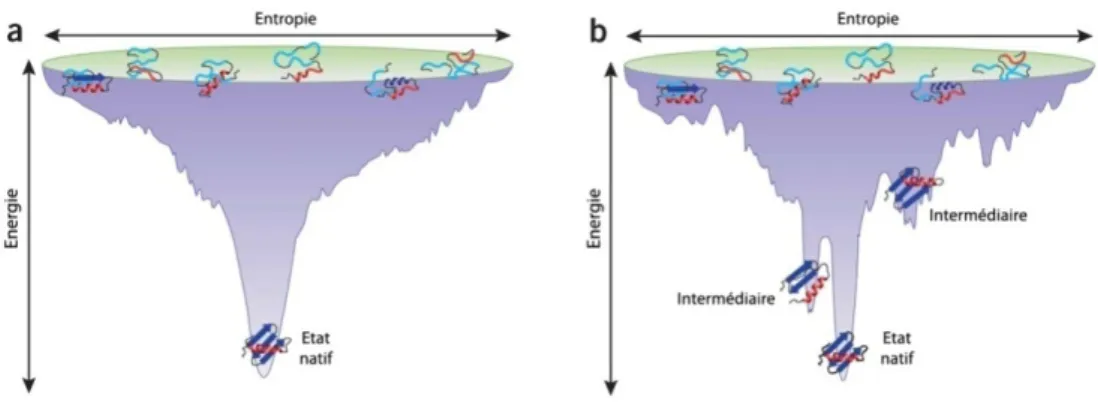
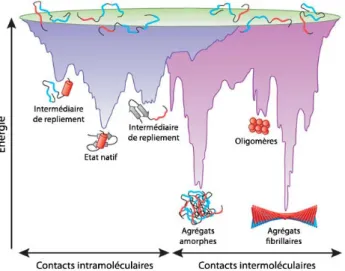
Les chaînes polypeptidiques forment des intermédiaires de repliement stables (vallées), puis passent par des états de transition (collines) pour atteindre leur conformation native, un état où l'énergie libre est la plus faible (Figure 2.b). Les chaînes polypeptidiques dénaturées représentent un large ensemble de structures résultant d’interactions natives et non natives. Exemple d'un paysage énergétique relativement lisse où les chaînes polypeptidiques ont tendance à réduire l'énergie libre interne pour atteindre leur conformation native.
B.2. Contraintes liées au repliement des protéines et agrégation
Exemple d'un paysage énergétique plus dur dans lequel les chaînes polypeptidiques passent par un ou plusieurs intermédiaires de repliement avant d'atteindre leur conformation native. Pendant ce temps, les chaînes polypeptidiques peuvent être sujettes à un mauvais repliement et à une agrégation si elles n'acquièrent pas une conformation stable (Hartl et Hayer-Hartl, 2009 ; Kramer et al., 2009). La région bleue représente le paysage énergétique du repliement des chaînes polypeptidiques vers leur conformation native.
C. Les chaperons moléculaires 19
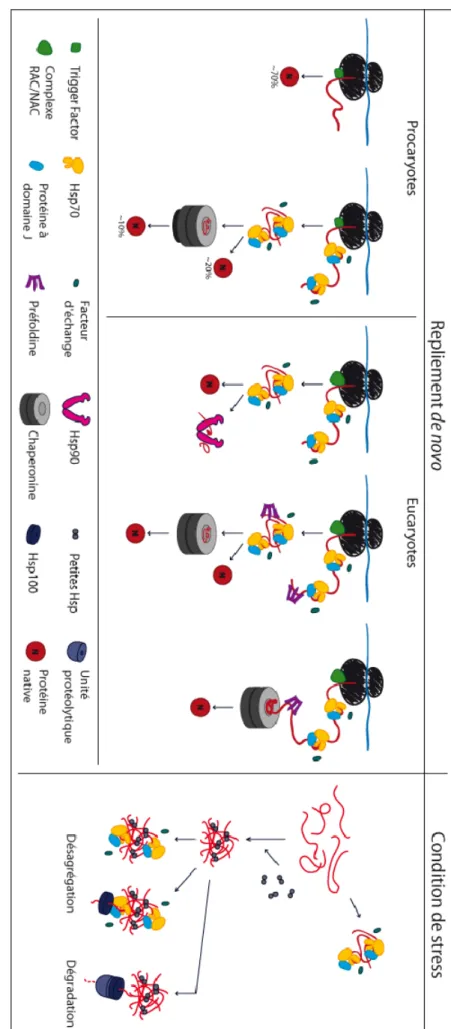
Enfin, un arsenal de chaperons moléculaires est au cœur de l’appareil qui permet le repliement natif des protéines et le maintien de la protéostasie. Chez les procaryotes, on pense que les mêmes chaperons moléculaires participent à la fois au repliement et à l'assemblage de protéines nouvellement synthétisées ou dénaturées suite à un stress cellulaire (Bukau et al., 1996). Chez les eucaryotes, les travaux de Judith Frydman et ses collègues ont mis en évidence l'existence de deux réseaux distincts de chaperons moléculaires (Albanese et al., 2006).
C.2. Fonctions et organisation des chaperons moléculaires dans la cellule
Chez Escherichia coli, il a été démontré que la plupart des chaperons moléculaires (Hsp70 DnaK et chaperonine GroEL/GroES) ainsi que leurs chaperons interagissent avec un grand nombre de protéines nouvellement synthétisées (Deuerling et al., 1999). ; Ewalt et coll., 1997 ; Teter et al., 1999), mais que leur expression est également induite par le stress et qu'ils sont nécessaires au traitement des protéines dénaturées par choc thermique (Parsell et Lindquist, 1993). En complément de ces travaux réalisés selon une approche globale, des spécificités fonctionnelles au sein de différents paralogues de chaperons moléculaires eucaryotes ont été décrites (Daugaard et al., 2007 ; Kabani et Martineau, 2008b). Fonctions et organisation des chaperons moléculaires dans la cellule Différentes familles de chaperons moléculaires coexistent dans la cellule.
C.2.a. Synthèse des protéines et repliement de novo
La liaison de la NAC au ribosome se produit uniquement via la sous-unité β, et les deux sous-unités interagissent avec la chaîne polypeptidique naissante (Beatrix et al., 2000). Ce couplage de chaperons moléculaires facilite le repliement co-traductionnel et évite la libération de protéines non natives dans le cytosol (Siegers et al., 1999 ; Thulasiraman et al., 1999. Elles sont transloquées dans le réticulum endoplasmique (RE), pt. dans la voie de sécrétion, où ils subissent des modifications co- et post-traductionnelles (par exemple formation de ponts disulfure, glycosylation) (Buck et al., 2007 ; van Anken et Braakman, 2005.
C.2.b. Contrôle qualité des protéines
Seules les protéines correctement repliées ou assemblées quittent le réticulum endoplasmique, les autres restent dans le RE et sont dégradées (Bukau et al., 2006 ; Schroder, 2008). Dans un deuxième temps, l'interaction directe des protéines mal repliées avec Ire1 provoquerait l'activation de son activité ribonucléase au niveau de son domaine cytoplasmique (Kimata et al., 2007). C'est notamment le cas de l'ubiquitine ligase CHIP (Carboxyl terminus of Hsp70-Interacting Protein ; protéine qui interagit avec la région C-terminale de Hsp70) qui participe à l'équilibre entre le repliement et la dégradation des protéines (Ballinger et al., 1999 ; Esser et al. al., 2004).
C.2.c. Agrégation des protéines
Par exemple, CHIP et le facteur d'échange Bag1, en complexe avec Hsp70, inhibent son activité ATPase et orientent sa fonction vers le processus de dégradation protéasomale (Ballinger et al., 1999 ; Demand et al., 2001 ; Meacham et al., 2001. À l'inverse, l'activité ubiquitine ligase de CHIP est inhibée lorsqu'elle est complexée avec Hsp70 et le facteur d'échange HspBP1, et la dégradation par le protéasome est altérée (Alberti et al., 2004).
C.3. Acteurs majeurs du repliement et du dépliement des protéines
C.3.a. Famille des chaperonines
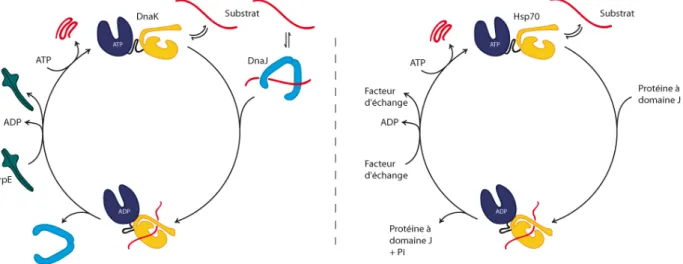
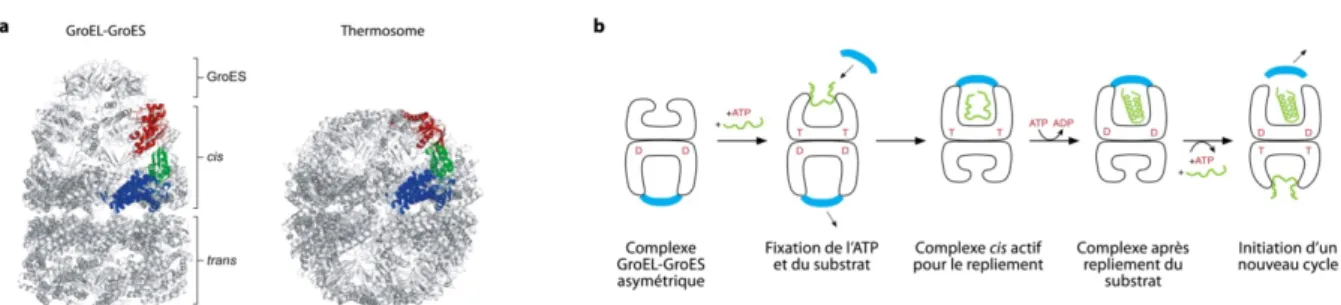
Les chaperonines fonctionnent selon un cycle de réaction dépendant de l'ATP (Figure 5b) (Horwich et al., 2009 ; Horwich et al., 2007). Cependant, la fonction globale de ce chaperon moléculaire en ce qui concerne le repliement des protéines semble conservée (Booth et al., 2008). En revanche, chez les eucaryotes, TRiC/CCT interagit avec les Hsp70, qui peuvent fournir un substrat à replier directement dans la cavité chaperonine ( Cuellar et al., 2008 ).
C.3.b. Hsp90
Dans le cas de la kinase Cdk4, elle est liée asymétriquement au domaine M et probablement au domaine N de l'un des deux monomères (Vaughan et al., 2006). Cette coopération est médiée par le cofacteur Hop, qui interagit principalement via ses domaines de répétition tétratricopeptide (TPR) avec le motif EEVD C-terminal de Hsp70 et Hsp90 (Onuoha et al., 2008 ; Scheufler et al., 2000 Communication entre les deux chaperons). les systèmes permettraient à Hsp70 de délivrer des substrats spécifiques à Hsp90 (Wegele et al., 2004 ; Wegele et al., 2006).
C.3.c. Hsp100
C.3.d. Petites Hsp
D. Conclusion : les Hsp70 au cœur de la protéostase 33
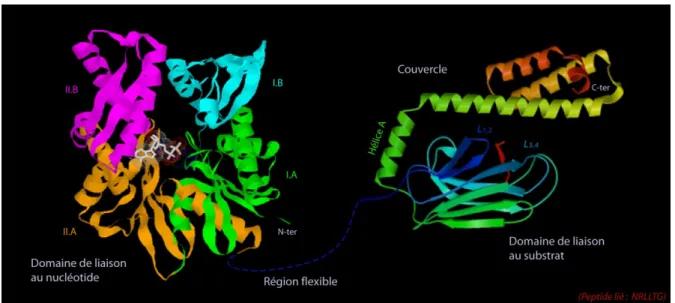
La structure du domaine de liaison au substrat est présentée en relation avec le peptide NRLLTG. Le domaine de liaison aux nucléotides ou domaine ATPase est extrêmement conservé (alignement à la page 66). Le domaine de liaison au substrat est coiffé d'un couvercle mobile constitué de trois hélices α (Zhu et al., 1996).
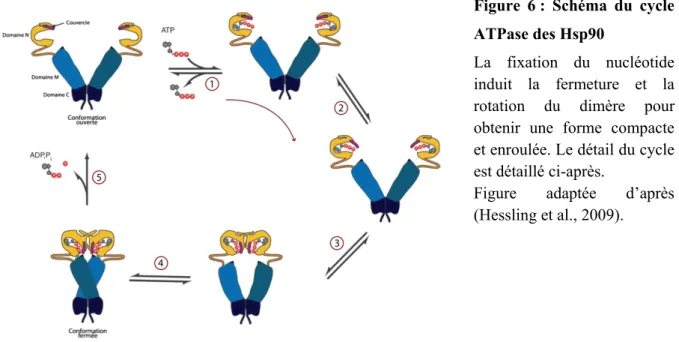
A.2. Cycle ATPase
La liaison de l'ATP au NBD provoque une courbure et une insertion de la région flexible entre le NBD et le SBD. Sous la forme ATP, le domaine de liaison aux nucléotides aurait une conformation étendue, la région intermédiaire flexible serait repliée entre le NBD et le SBD et le couvercle serait ouvert sur le SBD. La liaison au substrat et l'interaction avec une protéine du domaine J favoriseraient le mouvement de la région intermédiaire.
A.3. Mécanismes d’action des Hsp70
L'activité de la DnaK ATPase est ensuite stimulée de manière synergique par la liaison au substrat et par la DnaJ (Laufen et al., 1999). Après hydrolyse de l'ATP, le NBD aurait une conformation plus fermée après rotation de ses sous-domaines, la région intermédiaire serait plus exposée et l'hélice A du couvercle agirait au niveau du sillon présent au fond du NBD. . Hsp70 semble être capable de solubiliser certains agrégats protéiques sans l'aide de Hsp100 (Skowyra et al., 1990).
B. Co-chaperons des Hsp70 39
Leur mécanisme de dissolution peut s'expliquer par le modèle d'attraction entropique (De Los Rios et al., 2006 ; Goloubinoff et De Los Rios, 2007).
B.1.a. Structure et classification
B.1.b. Fonctions
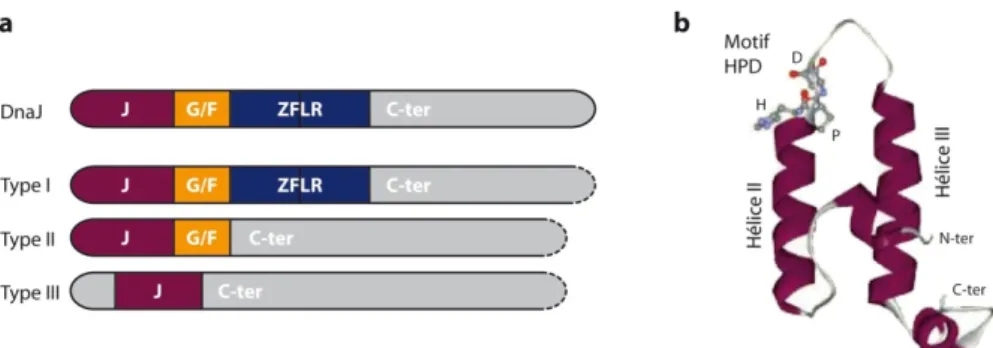
Les protéines du domaine J de type III ont un domaine J dont la position est variable et des domaines fonctionnels différents pour assurer des fonctions spécifiques. Dans le cas des protéines à domaine J de classe I et II, le domaine C-terminal est un domaine de liaison au substrat et d'homodimérisation. Les protéines du domaine J de type I et II ont une fonction similaire dans la reconnaissance et le transfert de substrats non natifs vers les Hsp70 (Kelley, 1998).
B.2. Facteurs d’échange
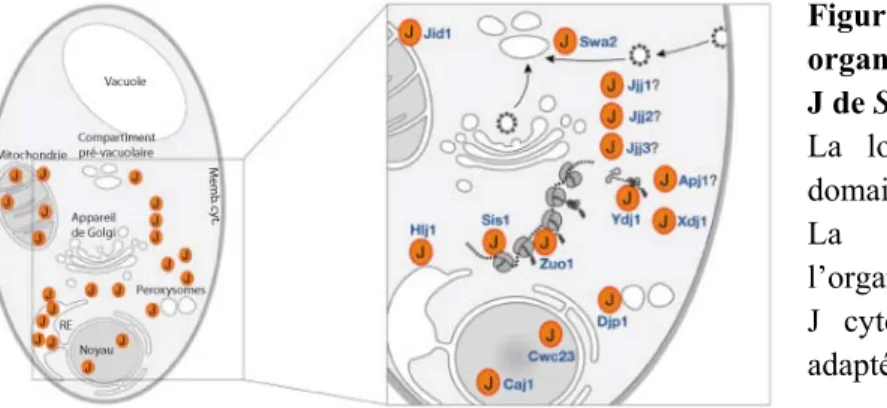
À l'inverse, les orthologues de Hsp70 n'interagissent pas avec toutes les protéines du domaine J dans le même compartiment cellulaire (Figure 10). Chez Saccharomyces cerevisiae certains sont impliqués dans des fonctions spécifiques comme Djp1 dans la biogenèse des peroxysomes (Hettema et al., 1998) ou Sis1 dans l'initiation de la traduction (Zhong et Arndt, 1993). Ils ne participent pas au pliage, mais plutôt aux processus de montage et de démontage des complexes.
B.2.a. GrpE
Brehmer et al., 2004) sont plus ou moins contradictoires, mais tous soulignent le rôle du GrpE dans la dégradation du substrat. De plus, il a été proposé que le domaine N-terminal de GrpE puisse servir de leurre pour le domaine SBD de DnaK (Brehmer et al., 2004 ; Chesnokova et al., 2003. À des températures élevées, ils adoptent une hélice réversible. conformation qui diminue l'activité du facteur d'échange GrpE (Grimshaw et al., 2001).
B.2.b. Protéines à domaine BAG
GrpE et Bag1 induisent tous deux une ouverture comparable du NBD via différents mécanismes d'action (Harrison et al., 1997 ; Sondermann et al., 2001) (Figure 12). De plus, Bag1 ne peut pas catalyser la dissociation de l'ATP et la dissociation de l'ADP est affectée par la présence de phosphate inorganique (Gassler et al., 2001). GrpE et Bag1 montrent une sélectivité pour leurs partenaires en raison de différences structurelles dans les sites de liaison nucléotidiques entre Hsc70 eucaryote et DnaK (Brehmer et al., 2001).
B.2.c. HspBP1 et protéines BAP
Cette interaction provoquerait le déplacement du sous-domaine IB du fait de l'encombrement stérique généré, et donc une distorsion des deux lobes du NBD, permettant la libération du nucléotide (Figure 13).
B.2.d. Famille Hsp110
En revanche, DnaK ne semble pas affecter HscB, DjlB et DjlC (Genevaux et al., 2007). Masison (NIH, Bethesda, USA), qui a développé le système expérimental que nous avons utilisé (Tutar et al., 2006). Mehdi Kabani a montré que la souche 1278b fes1 est incapable de former des biofilms (Martineau et al., 2007).
The hyper-invasiveness of the mutant Δydj1 was known and linked to a deregulation of the expression of FLO11 (Palecek et al., 2000). We previously reported that the cytosolic Hsp70 machinery plays crucial roles in mat formation ( Martineau et al., 2007 ). Indeed, deletion of the genes encoding Ydj1p or Sse1p dramatically affected mat formation and caused a hyperinvasive phenotype by increasing the expression of Flo11p on the cell surface ( Martineau et al., 2007 ).
Mutations in the cytosolic Hsp70s Ssa1p and Ssa2p had milder effects on mat formation (Martineau et al., 2007). The Flo11-HA protein migrated as a high molecular weight fuzzy band, reflecting its glycosylated state (Martineau et al., 2007). Point mutations in the TPR (G388R) or in the J domain (HPDAAA) had much milder effects than complete deletion of the corresponding domains (Xiao et al., 2006).
However, the combination of these two mutations abolished Swa2p function in vivo, similar to the entire deletion of the SWA2 gene ( Xiao et al., 2006 ). Deletion of the SWA2 gene results in pleiotropic phenotypes, including slow growth and defects in protein trafficking (Pishvaee et al., 2000). We faced the same dilemma when we identified FES1 as a gene required for mat formation ( Martineau et al., 2007 ).
Such modifications can include the addition of a GPI anchor, N-glycosylation, and O-mannosylation (Douglas et al., 2007). After purification and concentration, the protein was retained on the aggregation gel and considered aggregated (Douglas et al., 2007).