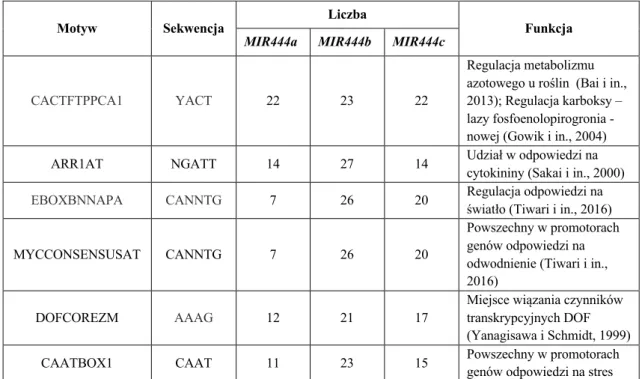
Mutations introduced into the sequence coding for PEP444a have been shown to be highly lethal. Furthermore, pep444c transgenic plants with mutations in the sequence coding for PEP444c showed reduced levels of microRNA444c in barley crops.
Wstęp
Biogeneza roślinnych mikroRNA
W przypadku mikroRNA zlokalizowanego w eksonie zaobserwowano spadek poziomu ekspresji microRNA402 po inaktywacji miejsca donorowego 5' ss (Knop et al., 2017). Dane literaturowe wskazują, że w procesie tym bierze udział białko związane z retikulum endoplazmatycznym AMP1 (ALTERED MERISTEM PROGRAM 1) (Li et al., 2013).
Rodzina genów MIR444
Te czynniki transkrypcyjne wiążą się bezpośrednio z sekwencjami regulatorowymi CArG w promotorze genu OsRDR1 (Wang i in., 2016). Kolejne badanie przeprowadzone przez Jiao i in., 2020 wykazało, że w ryżu jony amonowe (NH4+) stymulują biosyntezę brassinosteroidów (BR) poprzez zależną od mikroRNA444 regulację czynników transkrypcyjnych MADS-box.
Spadek poziomu ekspresji HAM i HAM2, które biorą udział w rozwoju systemu korzeniowego, prowadzi do zmniejszenia liczby korzeni bocznych (Lauressergues i in., 2015; Lv i in., 2016). Badania przeprowadzone na Arabidopsis wykazały obecność czterech peptydów kodowanych przez geny MIR: miPEP164a, miPEP165a, miPEP319a i miPEP858a (Lauressergues i in., 2015; Sharma i in., 2020).
Cel pracy
Materiały
Materiał roślinny
Oligonukleotydy
Wektory
Enzymy restrykcyjne Fast Digest ThermoFisher Scientific Inhibitor fosfataz (Pierce™ Mini tablete) ThermoFisher Scientific Inhibitor proteaz Complete Mini EDTA - brez Roche. Polimeraza DNA DreamTaq (5U/μl) ThermoFisher Scientific Polimeraza DNA Q5® High-Fidelity New England BioLabs SYBR® Green PCR Master Mix ThermoFisher Scientific.
Materiały do hodowli bakterii W pracy wykorzystano następujące szczepy bakterii
W celu przygotowania pożywki stałej do mieszaniny dodano 15 g agaru i po ochłodzeniu pożywki do 50°C dodano odpowiednie antybiotyki.
Materiały do analiz białkowych
Bufory i roztwory wykorzystane do pracy z DNA i RNA
Materiały do przeprowadzenia transformacji, selekcji i regeneracji zarodków jęczmiennych
100% stężony roztwór witamin, roztwór Dikamby i roztwór CuSO4,5 H2O dodawano po sterylizacji, po schłodzeniu pożywki, bezpośrednio przed wylaniem na płytki. 100% stężony roztwór witamin, 2,4 - D, roztwór CuSO4,5 H2O, roztwór BAP, timentynę i higromycynę dodawano po sterylizacji, po ochłodzeniu pożywki, bezpośrednio przed jej wylaniem na płytki.
Materiały wykorzystane w eksperymencie profilowania polisomów
Do probówek wirówkowych (Seton Scientific, open-top polyclearTM, 7030) odpipetowano 1,65 ml 60% sacharozy i umieszczono w temperaturze -80°C na jedną godzinę.
Metody
- Uprawa roślin
- Eksperymenty z wykorzystaniem kwasów nukleinowych
- Profilowanie polisomów
- Izolacja i transformacja zarodków jęczmiennych Izolacja zarodków jęczmiennych
- Analizy bioinformatyczne
Po wybraniu odpowiednich kolonii, które zawierały żądaną wstawkę, wybrane kolonie Agrobacterium tumefaciens zaszczepiono płynną pożywką hodowlaną MG/L z odpowiednimi antybiotykami i inkubowano w 28°C z wytrząsaniem 200 obrotów na minutę. Fragmenty żelu rozbito na mniejsze fragmenty, dodano 450 μl buforu EBR i inkubowano w temperaturze 25°C przez noc z wytrząsaniem przy 300 obr./min. 1 ml buforu ekstrakcyjnego dodano do 300 mg materiału roślinnego roztartego w ciekłym azocie i inkubowano w temperaturze 4°C przez 1 godzinę z wytrząsaniem przy 1000 obr./min.
Po zablokowaniu błonę inkubowano w temperaturze pokojowej w roztworze przeciwciał pierwszorzędowych (1:000 HORVU6 anty-PEP444a, 1:5000 F2DAQ5 anty-PEP444c, 1:3000 anty-aktyna) przygotowanym w 2% roztworze odtłuszczonego mleka. Błonę następnie przemyto trzykrotnie buforem TBST (3 razy przez 10 minut) i inkubowano przez jedną godzinę w temperaturze pokojowej w roztworze drugorzędowego przeciwciała skoniugowanego z peroksydazą chrzanową. Kolonię bakteryjną Agrobacterium tumefaciens szczep AGL1 niosącą właściwy plazmid zaszczepiono w 10 ml pożywki MG/L z odpowiednimi antybiotykami i inkubowano przez 40 godzin w temperaturze 28°C z wytrząsaniem przy 200 obrotach na minutę.
W przeddzień planowanej transformacji część inokulum zaszczepiono do 10 ml pożywki MG/L bez antybiotyków i inkubowano w temperaturze 28°C przez 18 godzin z wytrząsaniem przy 200 obr./min. Zarodki następnie przenoszono do nowych szalek z pożywką indukującą kalus i inkubowano w temperaturze 23°C w ciemności przez 3 dni.
Wyniki
Analiza elementów regulatorowych w promotorach genów MIR444
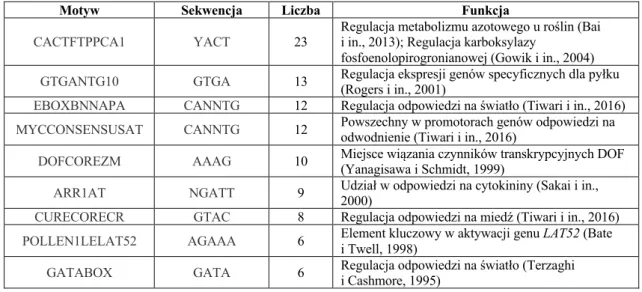
MYBCOREATCYCB1 AACGG 1 2 8 Miejsce wiązania czynnika transkrypcyjnego MYB, regulacja odpowiedzi ABA (Abe i in., 2003) MYB2CONSENSUSA. EBOXBNNAPA CANNTG 12 Regulacja odpowiedzi na światło (Tiwari i in., 2016) MYCCONSENSUSAT CANNTG 12 Powszechne u promotorów genów odpowiedzi na światło. ROOTMOTIFTAPOX1 ATATT 6 Element związany z regulacją ekspresji genów w komórkach korzenia (Zhang et al., 2019) RAV1AAT CAACA 5 Miejsce wiązania czynnika transkrypcyjnego.
MYBCORE CNGTTR 4 Miejsce wiązania białka MYB (Chen i in., 2019) CIACADIANLELHC CAANNNNATC 4 Element rytmu okołodobowego (Piechulla i in., 1998). RHERPATEXPA7 KCACGW 2 Element związany z regulacją ekspresji genów w komórkach korzenia (Ahmadi et al., 2018) MYB2CONSENSUSAT YAACKG 2 Miejsce wiązania czynnika transkrypcyjnego. 10PEHVPSBD TATTCT 2 Regulacja odpowiedzi na światło (Yazaki i in., 2004) ACGTATERD1 ACGT 2 Obecny w promotorze genu ERD1 u Arabidopsis.
INRNTPSADB YTCANTYY 2 Miejsce inicjacji transkrypcji w promotorach z niedoborem TATA box (Xi i in., 2007) WBOXATNPR1 TTGAC 2 Miejsce wiązania czynnika transkrypcyjnego. Słabszy sygnał odnotowano również w próbkach kontrolnych (przy braku induktora), co może wskazywać na wyciek plazmidu (Golda i in., 2007).
Zaprojektowano dwa pojedyncze kierujące RNA Cas9 (sgRNA) dla sekwencji kodującej PEP444a, dwa kierujące RNA dla sekwencji kodującej miPEP444b i dwa kierujące RNA dla sekwencji kodującej PEP444c. Dwa RNA ukierunkowane na sekwencję kodującą PEP444a (sgRNA1 i sgRNA2) wprowadzono do pojedynczego konstruktu, który transformowano do niedojrzałych zarodków jęczmienia przy użyciu Agrobacterium tumefaciens. W przeciwieństwie do tego, dwa RNA prowadzące do sekwencji kodującej miPEP444b (sgRNA1 i sgRNA2) i PEP444c (sgRNA3 i sgRNA4) zostały jednocześnie wprowadzone do drugiego konstruktu użytego do transformacji.
Do transformacji użyto konstruktu zawierającego dwa RNA nakierowane na sekwencję kodującą miPEP444b (sgRNA1 i sgRNA2) i dwa RNA nakierowane na sekwencję kodującą PEP444c (sgRNA3 i sgRNA4). Wybrano linie pep444c-332.10, które miały insercję 1 pz i delecję 9 pz w regionie sekwencji kodującej miPEP444c oraz linie pep444c-336.4. Niewielki wzrost wartości QY max, określającej maksymalną wydajność kwantową fotosystemu II, zaobserwowano w roślinach pep444c-332.10 w porównaniu z roślinami WT.
Aby określić, czy mutacje wprowadzone do sekwencji kodującej PEP444c wpływają na akumulację mikroRNA444c, przeprowadzono mały eksperyment głębokiego sekwencjonowania RNA. Wyniki analizy wykazały statystycznie istotny spadek poziomów mikroRNA444c w korzeniach w roślinach pep444c-332.10 w porównaniu z roślinami typu dzikiego (ryc. 41).
Dyskusja
Rodzina genów MIR444 w jęczmieniu zwyczajnym
Eksperymenty przeprowadzone przez Szarzyńską i in., 2009 wykazały również obecność ośmiu różnych pA w prekursorze miR166a w roślinach WT i sześciu w analizowanym prekursorze w mutantach hyl1. Wykazano, że wybór miejsca poliadenylacji może wpływać na lokalizację mRNA, jego stabilność i wydajność translacji (Wu i in., 2011; Guo i in., 2016). Obserwacje te zostały potwierdzone badaniami nad miR399, którego ekspresja jest indukowana w warunkach niedoboru fosforu (Bari i in., 2006).
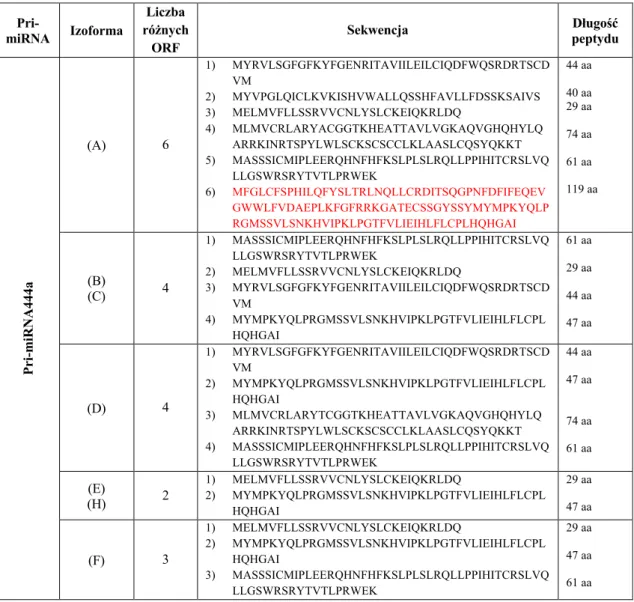
Dane te wskazują, że transfer miR399 z pędu do korzenia sygnalizuje pobieranie Pi przez korzenie (Lin i in., 2008; Chen i Rechavi, 2022). W mutancie białka HEN1 Arabidopsis (hen1-1) zaobserwowano obniżenie poziomu ekspresji kilku mikroRNA, w tym miR395 i miR399, w wyniku przerwania biogenezy mikroRNA (Buhtz i in., 2010). Analiza sekwencji kodującej miPEP444b wykazała podobieństwo (100% identyczności sekwencji aminokwasowej) do niezidentyfikowanego białka jęczmienia zwyczajnego (Matsumoto i in., 2011).
Badania przeprowadzone na Arabidopsis wykazały, że niektóre transkrypty MIR zawierające w swojej sekwencji ORF (pri-miR156a, pri-miR159a, pri-miR160a, pri-miR165a, pri-miR166b, pri-miR167a, pri-miR171a, pri-miR172a, miR319b) również związany z rybosomami (Lauressergues i in., 2022). Pozostaje to jednak niezgodne z badaniem Ormancey i in., 2020, które wykazało, że analizowany miPEP165a u Arabidopsis nie jest transportowany z korzeni do pędów.
Wykorzystanie technologii CRISPR/Cas9 do edycji genów MIR444
Region szóstego eksonu MIR444a, w którym zaprojektowano dwa kierujące RNA do edycji sekwencji kodującej PEP444a, w odwrotnej nici odpowiada sekwencji drugiego intronu genu MADS57. Zatem ta sekwencja nie jest częścią sekwencji kodującej (CDS) czynnika transkrypcyjnego MADS57. Uzyskane wyniki nie pozwalają na wyjaśnienie, czy możliwy efekt śmiertelny mógł być spowodowany mutacjami wprowadzonymi w regionie sekwencji kodującej PEP444a lub w obrębie genu MADS57.
Ze względu na brak potwierdzenia obecności miPEP444b w roślinach jęczmienia przy użyciu swoistego przeciwciała i techniki Western blot, zidentyfikowano transgeniczne linie z mutacjami tylko w obrębie sekwencji kodującej PEP444c (pep444c-332.10; pep444c-336.4; pep444c-336.6). wybrano do dalszej analizy. Natomiast u roślin linii pep444c-336.4 i pep444c-336.6 wprowadzone mutacje spowodowały zmianę 28 aa, nie zaobserwowano zmiany długości całego peptydu. Jednak analiza poziomu ekspresji mRNA MADS27 przeprowadzona w ramach tej pracy nie wykazała statystycznie istotnych zmian między WT a pep444c-332.10, pep444c-336.4 i pep444c-336.6.
Konieczne jest zatem przeprowadzenie dalszych eksperymentów wyjaśniających, w jaki sposób wprowadzone przez nas mutacje w regionie sekwencji kodującej PEP444c wpływają na strukturę korzeni i pędów jęczmienia. Jednak w roślinach z wyprowadzonych linii transgenicznych zaobserwowano statystycznie istotny spadek poziomu primiRNA444c, co sugeruje wpływ mutacji wprowadzonych w regionie sekwencji kodującej PEP444c na akumulację pri-miRNA444c.
Znaczenie PEP444c w biogenezie mikroRNA444c
Dane literaturowe wskazują, że mikroRNA156 reguluje poziom ekspresji genów z rodziny SPL (SQUAMOSA PROMOTER BINDING PROTEIN-LIKE) zaangażowanych we wzrost i rozwój roślin (Yamasaki et al., 2004; Xing et al., 2010). Ponadto mikroRNA156 i SPL3 odgrywają ważną rolę w odpowiedzi stresowej niedoboru Pi u Arabidopsis (Lei i in., 2016). Ciekawą obserwacją jest również spadek poziomu mikroRNA444c w roślinach pep444c-332.10 w przeciwieństwie do osa-miR827, bdi-miR156h-5p i bdi-miR156f-3p, zaangażowanych w regulację szlaków sygnałowych Pi.
Jednak doniesienia literaturowe wskazują na ważną rolę mikroRNA444c w odpowiedzi na Pi i NO3- w ryżu (Yan et al., 2014). Uzyskane przez nas wyniki mogą wskazywać na związek między procesami regulacji szlaków sygnałowych NO3-, Pi, co jest zgodne z innymi danymi literaturowymi (Wang et al., 2021). Nasz zidentyfikowany PEP444c, kodowany przez gen MIR444c, znacząco wpływa na akumulację mikroRNA444c w korzeniach jęczmienia.
Ze względu na rolę mikroRNA444c we wzroście i rozwoju roślin oraz jego udział w szlakach sygnałowych NO3 i Pi, niezwykle istotne jest prowadzenie dalszych eksperymentów w celu pogłębienia wiedzy w tym zakresie. Na podstawie wyników niniejszej pracy oraz dostępnych danych literaturowych można zaproponować model pokazujący możliwy udział PEP444c kodowanego przez gen MIR444c w biogenezie mikroRNA (ryc. 43).
Wnioski
ATCAGCGCTCGCTCGCACGCACCAACCCAACCCAACCCAACCCTACCCTACTCCGTTCG CGCCCGGTTGGTGGCTGGAGCCCGCGGAGGACTCGAGAATATCTGATGCTTATGGTCTG TCGCTTGGCCAGATACGCATGTGGCGGCACCAAGCATGAGGCAACAACTGCAGTACTTG TGGGGAAGGCACAAGTAGGACACCAACATTACCTGCAAGCAAGACGCAAAATTAATAGA ACATCACCATACTTGTGGCTTTCTTGCAAGTCGTGCAGTTGCTGCCTCAAGCTTGCTGC CTCCCTTTGCCAAAGCTATCAGAAAAAAACATAAAGTTGTGTTGTGTTAGTTTCCCTAA ACTGTAACACATGTTTTACAAATTGATCTAGAAATGATTGGCTCACTGTTAATTTAAAC CAATAATTACTCGATAACTGCATATGCACATGTTATGGAAGTAAGGCGGTAGAATACTT ATGTTTTCATCTACTGGCAGGTAGCAACACGTATAGGTATATCACTCTGATTTGAATTA AGGTCCAAACATTCAGAACTGCTTACTTTGCTGGCACATCCCATCGGTGAAATTACATA TAGGTTTATGTACAGAGTACTCTCAGGGTTTGGATTCAAATATTTTGGTGAAAACAGAA TTACTGCTGTAATAATTTTAGAAATATTATGCATACAGGATTTTTGGCAATCACGAGAC AGGACATCATGTGATGTGATGTGATGGTGCGGCTGCACACCTATAAGCCTTCACCTTGC CCTTACCCTCTATTCTAACCATTTAAGTAATCCGAACTAGTTCAAGCCTTACTGTACAT CAACATGGCCTCATCAAGTATTTGCATGATTCCTTTGGAAGAAAGACAACATAACTTTC ATTTTAAGTCACTTCCCCTCAGTCTTAGGCAGCTACTCCCTCCGATCCATATTACTTGT CGCAGTTTAGTACAACTTCTTGGAAGCTGGCGCAGCAGGTATACAGTCACCTTGCCTCG CTGGGAAAAATAGGGAGGTAAGATACTTCAACAGAATCTGAGGTTCAGTATGTGTTGTG CGCTAATTTTGGTTGTCCTTGTGCTACAGTATCTTCAGAGTAGCAGGGTGTAGCAGAAC AGTGGAGCTCTTAAACAGGAGCTGCTGCGTCAAATGCTATGTATGTCCCAGGCTTGCAG ATCTGCTTAAAAGTCAAAATATCTCATGTTTGGGCTCTGCTTCAGTCCTCACATTTTGC AGTTCTACTCTTTGACTCGTCTAAATCAGCTATTGTGTCGTGATATCACTTCACAGGGT CCCAACTTTGACTTTATTTTCGAACAAGAGGTGGGGTGGTGGCTGTTCGTTGATGCCGA GCCGCTGAAGTTTGGCTTCAGGAGGAAGGGTGCAACGGAGTGTTCTTCTGGTTATTCCA GTTACATGTACATGCCCAAGTACCAGCTACCAAGGGGCATGTCTTCAGTCTTGAGTAAC AAGCATGTTATTCCAAAGCTGCCTGGCACTTTTGTACTAATAGAAATTCATTTATTTTT GTGTCCACTACACCAGCATGGTGCAATTTAGAAGGAAAATATTGAGAAGCTGCCTAACT TAGGGTATGTCCAATAGTCTTATCTCAACCGTATGTAAGGATTGTTAGTTAGGAAAGAA CAAGAAAGGAGAGCTATAGTTGTATGGAGCTGATGGTATTTTTGCTCTCGTCTCGTGTT GTCTGTAACTTGTACTCCCTTTGTAAAGAAATACAAAAGCGTTTGGATCAGTAATTTAG ATCATGATCTAAATGCTCTTATATTTCTTCACGGAAGGAATATCT. 114 ATGGAAGTAAGGCGGTAGAATACTTATGTTTTCATCTACTGGCAGGTAGCAACACGTAT AGGTATATCACTCTGATTTGAATTAAGGTCCAAACATTCAGAACTGCTTACTTTGCTGG CACATCCCATCGGTGAAATTACATATAGGTTTATGTACAGAGTACTCTCAGGGTTTGGA TTCAAATATTTTGGTGAAAACAGAATTACTGCTGTAATAATTTTAGAAATATTATGCAT ACAGGATTTTTGGCAATCACGAGACAGGACATCATGTGATGTGATGTGATGGTGCGGCT GCACACCTATAAGCCTTCACCTTGCCCTTACCCTCTATTCTAACCATTTAAGTAATCCG AACTAGTTCAAGCCTTACTGTACATCAACATGGCCTCATCAAGTATTTGCATGATTCCT TTGGAAGAAAGACAACATAACTTTCATTTTAAGTCACTTCCCCTCAGTCTTAGGCAGCT ACTCCCTCCGATCCATATTACTTGTCGCAGTTTAGTACAACTTCTTGGAAGCTGGCGCA GCAGGTATACAGTCACCTTGCCTCGCTGGGAAAAATAGGGAGGTAAGATACTTCAACAG AATCTGAGGTTCAGTATGTGTTGTGCGCTAATTTTGGTTGTCCTTGTGCTACAGTATCT TCAGAGTAGCAGGGTGTAGCAGAACAGTGGAGCTCTTAAACAGGAGCTGCTGCGTCAAA TGCTATGGTCCCAACTTTGACTTTATTTTCGAACAAGAGGTGGGGTGGTGGCTGTTCGT TGATGCCGAGCCGCTGAAGTTTGGCTTCAGGAGGAAGGGTGCAACGGAGTGTTCTTCTG GTTATTCCAGTTACATGTACATGCCCAAGTACCAGCTACCAAGGGGCATGTCTTCAGTC TTGAGTAACAAGCATGTTATTCCAAAGCTGCCTGGCACTTTTGTACTAATAGAAATTCA TTTATTTTTGTGTCCACTACACCAGCATGGTGCAATTTAGAAGGAAAATATTGAGAAGC TGCCTAACTTAGGGTATGTCCAATAGTCTTATCTCAACCGTATGTAAGGATTGTTAGTT AGGAAAGAACAAGAAAGGAGAGCTATAGTTGTATGGAGCTGATGGTATTTTTGCTCTCG TCTCGTGTTGTCTGTAACTTGTACTCCCTTTGTAAAGAAATACAAAAGCGTTTGGATCA GTAATTTAGATCATGATCTAAATGCTCTTATATTTCTTCACGGAAGGAATATCT (C). ATCAGCGCTCGCTCGCACGCACCAACCCAACCCAACCCAACCCTACCCTACTCCGTTCG CGCCCGGTTGGTGGCTGGAGCCCGCGGAGGACTCGAGATACGCATGTGGCGGCACCAAG CATGAGGCAACAACTGCAGTACTTGTGGGGAAGGCACAAGTAGGACACCAACATTACCT GCAAGCAAGACGCAAAATTAATAGAACATCACCATACTTGTGGCTTTCTTGCAAGTCGT GCAGTTGCTGCCTCAAGCTTGCTGCCTCCCTTTGCCAAAGCTATCAGAAAAAAACATAA AGTTGTGTTGTGTTAGTTTCCCTAAACTGTAACACATGTTTTACAAATTGATCTAGAAA TGATTGGCTCACTGTTAATTTAAACCAATAATTACTCGATAACTGCATATGCACATGTT ATGGAAGTAAGGCGGTAGAATACTTATGTTTTCATCTACTGGCAGGTAGCAACACGTAT AGGTATATCACTCTGATTTGAATTAAGGTCCAAACATTCAGAACTGCTTACTTTGCTGG CACATCCCATCGGTGAAATTACATATAGGTTTATGTACAGAGTACTCTCAGGGTTTGGA TTCAAATATTTTGGTGAAAACAGAATTACTGCTGTAATAATTTTAGAAATATTATGCAT ACAGGATTTTTGGCAATCACGAGACAGGACATCATGTGATGTGATGTGATGGTGCGGCT GCACACCTATAAGCCTTCACCTTGCCCTTACCCTCTATTCTAACCATTTAAGTAATCCG AACTAGTTCAAGCCTTACTGTACATCAACATGGCCTCATCAAGTATTTGCATGATTCCT TTGGAAGAAAGACAACATAACTTTCATTTTAAGTCACTTCCCCTCAGTCTTAGGCAGCT ACTCCCTCCGATCCATATTACTTGTCGCAGTTTAGTACAACTTCTTGGAAGCTGGCGCA GCAGGTATACAGTCACCTTGCCTCGCTGGGAAAAATAGGGAGTATCTTCAGAGTAGCAG GGTGTAGCAGAACAGTGGAGCTCTTAAACAGGAGCTGCTGCGTCAAATGCTATGGTCCC AACTTTGACTTTATTTTCGAACAAGAGGTGGGGTGGTGGCTGTTCGTTGATGCCGAGCC GCTGAAGTTTGGCTTCAGGAGGAAGGGTGCAACGGAGTGTTCTTCTGGTTATTCCAGTT ACATGTACATGCCCAAGTACCAGCTACCAAGGGGCATGTCTTCAGTCTTGAGTAACAAG CATGTTATTCCAAAGCTGCCTGGCACTTTTGTACTAATAGAAATTCATTTATTTTTGTG TCCACTACACCAGCATGGTGCAATTTAGAAGGAAAATATTGAGAAGCTGCCTAACTTAG GGTATGTCCAATAGTCTTATCTCAACCGTATGTAAGGATTGTTAGTTAGGAAAGAACAA GAAAGGAGAGCTATAGTTGTATGGAGCTGATGGTATTTTTGCTCTCGTCTCGTGTTGTC TGTAACTTGTACTCCCTTTGTAAAGAAATACAAAAGCGTTTGGATCAGTAATTTAGATC ATGATCTAAATGCTCTTATATTTCTTCACGGAAGGAATATCT.
ATCAGCGCTCGCTCGCACGCACCAACCCAACCCAACCCAACCCTACCCTACTCCGTTCG CGCCCGGTTGGTGGCTGGAGCCCGCGGAGGACTCGAGAATATCTGATGCTTATGGTCTG TCGCTTGGCCAGATACACATGTGGCGGCACCAAGCATGAGGCAACAACTGCAGTACTTG TGGGGAAGGCACAAGTAGGACACCAACATTACCTGCAAGCAAGACGCAAAATTAATAGA ACATCACCATACTTGTGGCTTTCTTGCAAGTCGTGCAGTTGCTGCCTCAAGCTTGCTGC CTCCCTTTGCCAAAGCTATCAGAAAAAAACATAAAGTTGTGTTGTGTTAGTTTCCCTAA ACTGTAACACATGTTTTACAAATTGATCTAGAAATGATTGGCTCACTGTTAATTTAAAC CAATAATTACTCGATAACTGCATATGCACATGTTATGGAAGTAAGGCGGTAGAATACTT ATGTTTTCATCTACTGGCAGGTAGCAACACGTATAGGTATATCACTCTGATTTGAATTA AGGTCCAAACATTCAGAACTGCTTACTTTGCTGGCACATCCCATCGGTGAAATTACATA TAGGTTTATGTACAGAGTACTCTCAGGGTTTGGATTCAAATATTTTGGTGAAAACAGAA TTACTGCTGTAATAATTTTAGAAATATTATGCATACAGGATTTTTGGCAATCACGAGAC AGGACATCATGTGATGTGATGTGATGGTGCGGCTGCACACCTATAAGCCTTCACCTTGC CCTTACCCTCTATTCTAACCATTTAAGTAATCCGAACTAGTTCAAGCCTTACTGTACAT CAACATGGCCTCATCAAGTATTTGCATGATTCCTTTGGAAGAAAGACAACATAACTTTC ATTTTAAGTCACTTCCCCTCAGTCTTAGGCAGCTACTCCCTCCGATCCATATTACTTGT CGCAGTTTAGTACAACTTCTTGGAAGCTGGCGCAGCAGGTATACAGTCACCTTGCCTCG CTGGGAAAAATAGGGAGTATCTTCAGAGTAGCAGGGTGTAGCAGAACAGTGGAGCTCTT AAACAGGAGCTGCTGCGTCAAATGCTATGGTCCCAACTTTGACTTTATTTTCGAACAAG AGGTGGGGTGGTGGCTGTTCGTTGATGCCGAGCCGCTGAAGTTTGGCTTCAGGAGGAAG GGTGCAACGGAGTGTTCTTCTGGTTATTCCAGTTACATGTACATGCCCAAGTACCAGCT ACCAAGGGGCATGTCTTCAGTCTTGAGTAACAAGCATGTTATTCCAAAGCTGCCTGGCA CTTTTGTACTAATAGAAATTCATTTATTTTTGTGTCCACTACACCAGCATGGTGCAATT TAGAAGGAAAATATTGAGAAGCTGCCTAACTTAGGGTATGTCCAATAGTCTTATCTCAA CCGTATGTAAGGATTGTTAGTTAGGAAAGAACAAGAAAGGAGAGCTATAGTTGTATGGA GCTGATGGTATT. ATCAGCGCTCGCTCGCACGCACCAACCCAACCCAACCCAACCCTACCCTACTCCGTTCG CGCCCGGTTGGTGGCTGGAGCCCGCGGAGGACTCGAGATACGCATGTGGCGGCACCAAG CATGAGGCAACAACTGCAGTACTTGTGGGGAAGGCACAAGTAGGACACCAACATTACCT GCGAGCAAGACGCAAAATTAATAGAACATCACCATACTTGTGGCTTTCTTGCAAGTCGT GCAGTTGCTGCCTCAAGCTTGCTGCCTCCCTTTGCCAAAGCTATCAGAAAAAAACATAA ATCTTAGGCAGCTACTCCCTCCGATCCATATTACTTGTCGCAGTTTAGTACAACTTCTT GGAAGCTGGCGCAGCAGGTATACAGTCACCTTGCCTCGCTGGGAAAAATAGGGAGTATC TTCAGAGTAGCAGGGTGTAGCAGAACAGTGGAGCTCTTAAACAGGAGCTGCTGCGTCAA ATGCTATGGTCCCAACTTTGACTTTATTTTCGAACAAGAGGTGGGGTGGTGGCTGTTCG TTGATGCCGAGCCGCTGAAGTTTGGCTTCAGGAGGAAGGGTGCAACGGAGTGTTCTTCT GGTTATTCCAGTTACATGTACATGCCCAAGTACCAGCTACCAAGGGGCATGTCTTCAGT CTTGAGTAACAAGCATGTTATTCCAAAGCTGCCTGGCACTTTTGTACTAATAGAAATTC ATTTATTTTTGTGTCCACTACACCAGCATGGTGCAATTTAGAAGGAAAATATTGAGAAG CTGCCTAACTTAGGGTATGTCCAATAGTCTTATCTCAACCGTATGTAAGGATTGTTAGT TAGGAAAGAACAAGAAAGGAGAGCTATAGTTGTATGGAGCTGATGGTATTTTTGCTCTC GTCTCGTGTTGTCTGTAACTTGTACTCCCTTTGTAAAGAAATACAAAAGCGTTTGGATC AGTAATTTAGATCATGATCTAAATGCTCTTATATTTCTTCACGGAAGGAATATCT. CTCCGACTTTTCTTTCCCTCGCCGAAAAACCAGCTCCAGGCTCCAGCAGTAGCCAGTAG GCAACTTCCAATTTCATTATACTATTGCTCCCGCTAGGGTTCCAGCCTCCTCCATGGAG TCCTCGTCCGCTCGTAGATCCTTCACCAGCCGCCTTTTAGTTTTAGGTCCGCCTCTCCT CCCGCAGCCGGGAGTGCTTGGTCTTGGGCGCGCCTGGCCGCCTGCATGCGCCGAGGGTT TGAGTTATTGCATGTGGTGGCACCAAGCATGAGGCAACAACTGCATTACTTTCAAGGAA GTTACAAAATCTATGGGTCTTCATAATCGTGACTTTCTTGCAAGTTGTGCAGTTGCTGT CTCAAGCTTGCTGACTCCGTCTGCCAGACCGCAATGTGAAATAGGAAATTTAGTCAGTT TTAGCGAATGAATGATCATACGAATAAGTTTGAGGAGACATTCGACTCTAAACTATTCA CTCTTGTAAATACTGGAAACATGGACCCATGGAGCATTTCTTCACCGCGTCACCCCTCC ACACCTTAAAAAGAACTCATTTGAAGGCTAGTCCAGCCTCCCTGAAACTTACATTCACA GACATTCTCAACTGAGATGAAGCCACAGATCTTTGTATGTTGTATGTGGACGAAGAACA AACAGATTAACTCTTGTTCACGTCCCTCCTCATGTACACTTGTAGTTCTATTCTAGGAT TTCAACTCTGCACTCCATCTGCATCCTGAATATTCAGAGTCTCAAATTTGCACAACTCT CGATTTGATTCAGTGAACAAGTCAATAAGATAGATTTTTGGTCGAGGATAGGTTGAATA GGTCTGTACTTGCTTAATGATGCTATTCTTTTGACTTTTGAGTATGGATGACACGCTGT GTCAGTCTTTGATCAAAATGAGAAGATGCGTGATTGA.
CTCCGACTTTTCTTTCTCTCGCGAAAAACCAGCTCCAGGCTCCAGCAGTAGCCAGTAGCAACTTCCAATTTCATTATACTATTGCTCCCGCCTAGGGTTCCAGCCTCCTCCATGGAG TCCTCGTCCGCTCGTAGATCCTTCACCAGCCGCCCTTTTAGTTTTAGGTTCCGCCTCTCTCCTCCCGCCAGCCGTCCGGGTGGGG TGAGTT ATTGCATGTGGTGGCACCAAGCATGGAAACA ACTGCATTACTTTCAAGGAAA GTTACAAAAATCTATGGGTCTTCATATCGTGACTTTCTTGCAAGTTGTGCAGTTGCTGACTCCGTCTGCCAGACCGCAATGTGAAATAGGAAATTTAGTCAGGATTGATCCGTCAGTCAGTCCGTCAGTCGT ACTGGAAACATGGACCCATGGAGCATTCTTCACCGCGTCACCCCTCCACACCTTAAAAA AGAACTCAATTGAAGGCTAGTCC AGCCTCCCTGAAACTTACATTCAACAGACATTTCTCAG CTGAGATGAAGCCACAGATCTTTGTATGTTGTATGTGGACGAAAAACAAACAAACAGCCATTAAC TCTTGTTCACTACTACTCATTACT CTGCATCCTGAATATTCAGGTC TCAAATTTGCACAACTCTCGATTTGATTC GGTAAACAAGTCAATAAGATAAGATTTTTTGGTCGAGGATAGGTTGAATAGGTCTGGTACTT GCTTAATGATGCTATT CTTTTGACTTTTGAGTATGGATGACACGCTGTGTCAGTCTTTG ATCAAAATGAGAAGATGCGTGATTG. CTCCGACTTTTCTTTCTCTCGCGAAAAACCAGCTCCAGGCTCCAGCAGTAGCCAGTAGCAACTTCCAATTTCATTATACTATTGCTCCGCTAGGGTTCCAGCCTCCTTCATGGAG TCCTCGTCCGCTCGTAGATCCTTCACCAGCCGCCCTTTTAGTTTTAGGTTCCGCCTCTCTCCTCCCGCCAGCCGTCCGGGGGGGGGGGGTGAGTT ATTGCATGTGGTGGCACCAAGCATGAGGC AACAACTGCATTACTTTCAAGGAAA GTTACAAAAATCTATGGGTCTTCATATCGTGACTTTCTTGCAAGTTGTGCAGTTGTCTGTCTCAAGCTGCTGACTCCGTCTGCCAGACCGCAATGTGAAATAGGAAAATTGAAATTTAGTCAGGTACATTACTACCCCCCCAAAAGAACTCATTTGAAGGCTAGTC CAGCCTCCCTGAAACTTACATTCACAGACATTC TCAACTGAGATGAAGCCACAGATCTTTGT ATGTTGTATGTGGACGAAAAACAAACAGAT TAACTCTTGTTCACGTCCCTCTCTCTCATGTACTACTTGTAGTTTCTATTTCTAGGATTCAACT CTGCAGTCACTCAATTCAATTCAATTGAGT AAGTCAATAAGATAAGATTTTTTGGTCGAGG ATAGGTTGAATAGGTCGTGT ACTTGCTTAATGATGCTATTCTTTTGACTTTTTGAGTATGGATGACACGCTGTGTCAGTC TTTGATCAAAATGAGAAGATGCTGATT GA.
Literatura
Specific interactions between Dicer-like proteins and HYL1/DRB family dsRNA-binding proteins in Arabidopsis thaliana. Dual roles of the nuclear cap-binding complex and SERRATE in pre-mRNA splicing and microRNA processing in Arabidopsis thaliana. Modulation of the phosphate-deficient responses by MicroRNA156 and its targeted SQUAMOSA PROMOTERBINDING PROTEIN-LIKE 3 in Arabidopsis.
Functional analysis of cis-regulatory elements within the tobacco late pollen gene g10 promoter. The primary transcript of miR858 encodes the regulatory peptide and controls flavonoid biosynthesis and development in Arabidopsis. MicroRNA171c-targeted SCL6-II, SCL6-III, and SCL6-IV genes regulate shoot branching in Arabidopsis.
Suppression of MYBL2 by both microRNA858a and HY5 leads to activation of the anthocyanin biosynthetic pathway in Arabidopsis. Stress-induced alternative splicing provides a mechanism for regulating microRNA processing in Arabidopsis thaliana.