FACULDADE DE CIÊNCIAS AGRÁRIAS E VETERINÁRIAS
CÂMPUS DE JABOTICABAL
ANÁLISE DA EXPRESSÃO GÊNICA GLOBAL EM
MUTANTES DE Xanthomonas citri subsp. citri.
Elaine Costa Souza
Bióloga
FACULDADE DE CIÊNCIAS AGRÁRIAS E VETERINÁRIAS
CÂMPUS DE JABOTICABAL
ANÁLISE DA EXPRESSÃO GÊNICA GLOBAL EM
MUTANTES DE Xanthomonas citri subsp. citri.
Elaine Costa Souza
Orientador: Prof. Dr. Jesus Aparecido Ferro
Co-orientador: Prof. Dr. Marcelo Luiz de Laia
Tese apresentada à Faculdade de Ciências Agrárias e Veterinárias – Unesp, Câmpus de Jaboticabal, como parte das exigências para a obtenção do título de Doutor em Agronomia (Genética e Melhoramento de plantas)
JABOTICABAL – SÃO PAULO – BRASIL
Souza, Elaine Costa
S729a Análise da expressão gênica global de mutantes de Xanthomonas citri subsp. citri / Elaine Costa Souza. – – Jaboticabal, 2010
xvi, 128 f. ; i.l; 28 cm
Tese (doutorado) - Universidade Estadual Paulista, Faculdade de Ciências Agrárias e Veterinárias, 2010
Orientador: Jesus Aparecido Ferro Co-orientador: Marcelo Luiz de Laia
Banca examinadora: Agda Paula Facincani, José Belasque Junior, Haroldo Alves Pereira Junior, Eliana Gertrudes de Macedo Lemos
Bibliografia
1. Xanthomonas citri subsp. citri. 2. Mutantes. 3. Microarranjo. Título. II. Jaboticabal-Faculdade de Ciências Agrárias e Veterinárias.
CDU 631.52:632.3
DADOS CURRICULARES DA AUTORA
Elaine Costa Souza- Nascida em Ilha Solteira (SP), em 15 de outubro de 1979, com Licenciatura Plena em Ciências Biológicas pela Universidade Federal de Mato Grosso do Sul – Campus de Três Lagoas em 2003. Concluiu o mestrado em Agronomia – Sistema de Produção pela Faculdade de Engenharia de Ilha Solteira – UNESP – Campus de Ilha Solteira (2006), com ênfase em trabalhos de pesquisa relacionados a fungos patogênicos de plantas e genética sob orientação do Prof. Dr. Paulo Cezar Ceresini. Durante o Doutorado desenvolveu as atividades científicas do projeto intitulado Análise da expressão gênica global em mutantes de Xanthomonas citri subsp. citri, sob orientação do Prof. Dr. Jesus Aparecido
Dedico aos meus pais Elpídio
Celestino de Souza e Agnésia Costa de
Souza pelo apoio, compreensão, carinho, se
não fossem eles eu não chegaria até aqui,
AGRADECIMENTOS
Agradeço a Deus em primeiro lugar por chegar até aqui.
Ao Prof. Dr. Jesus Aparecido Ferro pela orientação, e oportunidade e confiança em mim depositada para realização deste trabalho.
Ao Prof. Dr. Marcelo Luiz de Laia pela co-orientação, pelos ensinamentos durantes esses anos e amizade.
A Profa. Maria Inês T. Ferro pela oportunidade concedida, ensinamentos e amizade
Ao grupo de pesquisa do LBM: Agda, André (Splinter), Andréia Fujimoto Daniele Jovino, Fabrício Edgar, Flávia, Flavinha, Gustavo, Júlio Bortolossi, Karina Dabbas, Mônica Cestari, Rafael Marini, Renata Tezza e Juliana Vantini pela amizade, apoio, carinho e compreensão.
A todos os docentes e funcionários do Departamento de Tecnologia da UNESP – Jaboticabal pelo carinho e amizade.
Aos meus amigos Jean de Oliveira, Gislayne Bitencourt, Márcia Stech, Mariângela Alves (uma grande amiga que fiz logo que cheguei em Jaboticabal), Vanessa Sato, Juliana Rossi e Janaína Gonçalves.
As minhas amigas Claudia Mara, Márcia Helena, Walkiria e Renata Moura pela amizade, carinho e apoio.
SUMÁRIO
LISTA DE ABREVIATURAS………....vii
LISTA DE FIGURAS………..ix
LISTA DE TABELAS………....xiv
RESUMO...xv
SUMMARY...xvi
CAPÍTULO 1 –CONSIDERAÇÕES GERAIS...1
1.INTRODUÇÃO...1
2. REVISÃO DE LITERATURA...3
2.1. Citricultura e o cancro cítrico ...3
2.2. Xanthomonas citri susbp. citri...6
2.3. Interação Planta-patógeno...8
2.4. Mutagênese aleatória...12
2.5. Técnica de microarranjos...13
Referências...16
CAPÍTULO 2 – Análise da expressão gênica global in vivo do mutante do gene xrvA de Xanthomonascitri subsp. citri (Xac)...22
RESUMO...22
SUMMARY...23
INTRODUÇÃO...24
MATERIAL E MÉTODOS...26
Linhagens bacterianas e condições de cultivo...26
Inoculação da bactéria em planta hospedeira...26
Extração de RNA total de Xanthomonascitri subsp. citri....27
Síntese de cDNA marcado com fluoróforos Cy3 e Cy5...27
Hibridização dos microarranjos com os cDNAs marcados...28
Análise das imagens e estatística dos dados...29
RESULTADOS...30
Sistema de secreção do tipo II (Degradação da parede celular)...48
Gene hrpX...50
Genes envolvidos na regulação de fatores de patogenicidade (rpf)...54
Genes do sistema de secreção do tipo IV...55
Transporte de Ferro...57
Transportadores ABC...58
Quimiotaxia e motilidade celular... 59
Elementos genéticos móveis...63
CONCLUSÕES...64
REFERÊNCIAS...65
CAPÍTULO 3 – Análise da expressão gênica global in vivo do mutante de uma histidina quinase de Xanthomonascitri subsp. citri (Xac)...75
RESUMO...75
SUMMARY...76
INTRODUÇÃO...77
MATERIAL E MÉTODOS...80
Linhagens bacterianas e condições de cultivo...80
Inoculação da bactéria em planta hospedeira...80
Extração de RNA total de Xanthomonascitri subsp. citri...81
Síntese de cDNA marcado com fluoróforos Cy3 e Cy5...81
Hibridização dos microarranjos com os cDNAs marcados...82
Análise das imagens e estatística dos dados...83
RESULTADOS...84
DISCUSSÃO...99
Sistemas de transdução de sinal de dois componentes (TCSTS)...101
Transportadores ABC...102
Quimiotaxia e motilidade celular...104
Elementos genéticos móveis...105
Sistema de secreção do tipo II (SST2)...105
Biossíntese de pequenas moléculas...109
CONCLUSÕES...110
REFERÊNCIAS...111
CAPÍTULO 4 - CONSIDERAÇÕES FINAIS...116
Validação do microarranjo...121
LISTA DE ABREVIATURAS
AraC Ativador transcricional
avr Gene de Avirulência
CVC Clorose Variegada dos Citros
DO Densidade Óptica,
DSF ("diffusible signal factor”) Fator de sinalização difusível
EPS polissacarídeo extracelular
g grama(s)
h hora(s)
HK Histidina quinase
HR Resposta de Hipersensibilidade
hrp Resposta de hipersensibilidade e patogenicidde
M Molar,
MarR “Multiple antibioic resistance regulator”
mg/mL miligramas por mililitros
min. Minuto(s)
ORF Fase ou Quadro Aberto de Leitura
pb pares de bases,
Proteína PR Proteína Relacionada à Patogênese
R Gene de Resistência
rpcF Fator de regulação de patogenicidade(rpc associado)
rpcG Fator de regulação de patogenicidade(rpc associado)
rpf Fatores de regulação de patogenicidade
RR Regulador de resposta
SST2 Sistema de Secreção do Tipo Dois
SST4 Sistema de Secreção do Tipo Quatro
SSTT Sistema de Secreção do Tipo Três
TCS “Two-component signal”
Xac Xanthomonas citri subsp. citri
Xcc Xanthomonas campestris pv. campestris
Xoc Xanthomonas oryzae pv. oryzicola
LISTA DE FIGURAS
CAPÍTULO 1 – Considerações Gerais………...
Figura 1. Sintomas de cancro cítrico causado por Xac em folhas, ramos e frutos (Fundecitrus)...6
CAPÍTULO 2 – Análise da expressão gênica global in vivo do mutante do gene
xrvA de Xanthomonas citri subsp. citri (Xac).
Figura 1. Genes diferencialmente expressos da categoria VIII (Proteínas hipotéticas) distribuídos nas subcategorias (VIII.A: Proteínas hipotéticas; VIII.B: Proteínas hipotéticas (inclui “no hits” e “hits” com baixo escore) e VIII.C: Proteínas hipotéticas conservadas em Xanthomonas)...31
Figura 2. Genes diferencialmente expressos da categoria I (Metabolismo intermediário) distribuídos nas subcategorias (I.A I: Degradação de polissacarídeos e oligossacarídeos, IB.10: Pool, multipurpose conversions, I.C.1: Respiração aeróbica, I.C.3: Transporte de elétrons, I.C.7: Ciclo dos ácidos tricarboxílicos (TCA), I.D.1: Sistemas de dois componentes, I.D.3: Quinases-Fosfatases)...32
Figura 3. Genes diferencialmente expressos da categoria VI. (Elementos genéticos móveis) distribuídos nas subcategorias (V.I.A: Funções relacionadas a fagos e profagos, V.I.B: Funções relacionadas à plasmídeos e V.I.C: Funções relacionadas à transposon e íntron)...33
Figura 4. Genes diferencialmente expressos da categoria V (Estruturas celulares) distribuídos nas subcategorias (V.A.1: Aminoácidos, aminas , V.A.2: Ânions, V.A.3: Carboidratos, ácidos orgânicos, álcoóis, V.A.4: Cátions, V.A.7: Outros, V.B: Divisão celular, V.C.: Quimiotaxia e mobilidade, V.D. Adaptação osmótica)... ...33
Figura 5. Genes diferencialmente expressos da categoria III distribuídos nas subcategorias (III.A.1: Replicação, III.A.3: Recombinação, III.A.4.: Reparo III.A.5: Restrição, modificação,III.B.4.: Aminoacil tRNA sintetases, modificação de tRNA, III.C.1: Tradução e modificação, III.C.3: Degradação de proteína, III.D.1: Polissacarídeos, III.D.2: Fosfolipídeos)... ...34
Figura 7. Genes diferencialmente expressos da categoria II (Biossíntese de pequenas moléculas) distribuídos nas subcategorias (II.A: Biossíntese de aminoácidos, II.A.2: Família do aspartato, família do piruvato, II.A.5: Histidina, II.B.2: Ribonucleotídeos de pirimidina, II.D.2: Ácido fólico, II.D.4: Molibdopterina,II.D.6: Piridoxina, II.D.8: Tiamina, II.F: Biossíntese de poliaminas)...35
Figura 8. Genes diferencialmente expressos da categoria IV (Estrutura Celular) distribuídos nas subcategorias (IV.A.1 Membrana interna:, IV.A.2: Constituintes da membrana externa, IV.B: Rede de mureína, peptidoglicano, IV.C: Polissacarídeos de superfície, lipopolissacarídeos e antígenos)... ...36
Figura 9. Genes diferencialmente expressos na categoria IX (Outros)... ...36
Figura 10. Representação esquemática do domínio presente na proteína
codificada pela ORF XAC1495 (http://pfam.sanger.ac.uk/). A proteína contém 134 resíduos de aminoácidos e um domínio H-NS...47
Figura 11. Esquema do sistema de secreção do tipo II. (Fonte: Homem, 2008) A seta vermelha indica o produto do gene xcsI que foi menos expresso no mutante
8B7...50
Figura 12. Agrupamento gênico hrp de Xanthomonas citri subsp. citri constituído
por 25 ORFs (Fonte: Carvalho, 2006).hpa2 corresponde à ORF XAC0417 e hpaF
corresponde a ORF XAC0393. hrpX não pertence ao cluster hrp e corresponde a
ORF XAC1994, a qual está separada de hpa2 por 1.836.622 bases do genoma da
Xac...52
Figura 13. Esquema das vias regulatórias que controlam a expressão de genes de virulência em Xanthomonas campestris pv. campestris (Xcc). RpfF está envolvido
na síntese de DSF, o qual se acumula no meio extracelular no início da fase estacionária de crescimento. DSF ativa o sistema de dois componentes RpfC/RpfG, o qual atua na hidrólise do di-GMP cíclico. Um segundo sistema de dois componentes que contribui para a hidrólise do di-GMP cíclico é constituído pelo sensor quinase RavS e pelo regulador de resposta RavR. RavS contêm dois domínios PAS que permite sua ativação por baixos níveis de oxigênio. Altos níveis e di-GMP cíclico promove a formação de biofilme e reprime a ligação do regulador transcricional Clp aos promotores de seus genes alvos. Clp induz a síntese de enzimas extracelulares e ativa a expressão dos genes regulatórios fhrR e zur. O ativador transcricional Zur e o regulador de resposta HrpG induzem a expressão de hrpX, o qual codifica um ativador transcricional tipo-AraC e é crucial para a
expressão de genes hrp. A expressão dos operons hrpE e hrpC de Xcc é também
incluindo hpaR, o qual codifica um regulador trascricional do tipo MarR.
(BÜTTNER & BONAS, 2010)...52
Figura 14. Representação esquemática do sistema flagelar em bactéria, onde o corpo basal é composto pelas proteínas FliG, FliM e FliN. Múltiplas cópias da proteína Flgl forma o Anel P, o qual , juntamente com o anel L forma uma estrutura cilíndrica rígida que segura a rotação da haste.( http://www.genome.jp/kegg/)...61
Figura 15. Representação do esquema de quimiotaxia em bactérias, na qual os receptores de sinal extracelular endereçam para o sistema de tradução de sinais, onde a proteína CheA recebe o sinal da MCP fosforilando CheY, que se liga ao corpo basal (complexo de proteínas FliG, FliM e FliN) e assim inicia o movimento rotacional dos flagelos. Este sistema para quando MCP sobre modificação pela ação das proteínas CheC e CheD, alterando a sensibilidade na percepção do sinal. (http://www.genome.jp/kegg/)...63
CAPITULO 3 – Análise da expressão gênica global in vivo do mutante de uma histidina quinase de Xanthomonascitri subsp. citri (Xac).
Figura 1. Genes diferencialmente expressos da categoria VIII (Proteínas hipotéticas) distribuídos na subcategorias (VIII.A: Proteínas hipotéticas, VIII.B: Proteínas hipotéticas (inclui “no hits” e “hits” com baixo escore) e VIII.C: Proteínas hipotéticas conservadas em Xanthomonas)...85
Figura 2. Genes diferencialmente expressos da categoria I (Metabolismo intermediário) distribuídos nas subcategorias (I.A I: Degradação de polissacarídeos e oligossacarídeos,I.A.2: Degradação de moléculas pequenas, IB.10: Pool, multipurpose conversions, I.C.2: Respiração anaeróbica e fermentação, I.C.3: Transporte de elétrons, I.C.7: Ciclo dos ácidos tricarboxílicos, I.C.8: Interconversão da força motriz ATP-próton, I.D.1: Sistemas de dois componentes, I.D.2: Ativatores-Repressores I.D.3: Quinases-Fosfatases...86
Figura 3. Genes diferencialmente expressos da categoria III distribuídos nas subcategorias (III.A.1: Replicação, III.A.3: Recombinação,III.A.5: Restrição, modificação,III.B.2: Proteínas Ribossomais, III.B.4.: Aminoacil tRNA sintetases, modificação de tRNA, III.C.1: Tradução e modificação, III.C.2:Chaperonas, III.C.3: Degradação de proteína, III.D.1: Polissacarídeos, III.D.2: Fosfolipídeos)...86
Figura 4. Genes diferencialmente expressos da categoria VII (Patogenicidade, virulência e adaptação). distribuídos na subcategorias (VII.C: Produção de toxina e detoxificação, VII.D: Degradação da parede da célula hospedeira , VII.F: Proteínas de superfície VII.G: Adaptação, condições atípicas, VII.H.:Outras)...87
profagos, V.I.B: Funções relacionadas a plasmídeos e V.I.C: Funções relacionadas a transposon e íntron)...88
Figura 6. Genes diferencialmente expressos da categoria II (Biossíntese de pequenas moléculas). distribuídos na subcategorias (II.A.2: Família do aspartato, família do piruvato,II.A.4: Família dos aminoácidos aromáticos,II.A.5: Histidina, II.D.1:Biotina, II.D.6: Piridoxina, II.D.11 Menaquinona, ubiquinona: II.D.14:Cobalamina)...88
Figura 7. Genes diferencialmente expressos da categoria IV (Estrutura Celular) distribuídos nas subcategorias (IV.A.1 Membrana interna:, IV.A.2: Constituintes da membrana externa, IV.B: Rede de mureína, peptidoglicano, IV.C: Polissacarídeos de superfície, lipopolissacarídeos e antígenos, IV.D: Estruturas de superfície)...89
Figura 8. Genes diferencialmente expressos da categoria V (Estruturas celulares) distribuídos nas subcategorias (V.A.1: Aminoácidos, aminas , V.A.4: Cátions, V.A.7: Outros, V.B: Divisão celular, V.C.: Quimiotaxia e mobilidade)...90
Figura 9. Genes diferencialmente expressos da categoria IX (Outros)...90
Figura 10- Sistema osmorregulatório de E. coli. Neste sistema ocorre um único
evento de transferência de grupo fosforil entre a proteína histidina quinase (HK) EnvZ e a sua proteína regulatora de resposta (RR) OmpR...100
Figura 11- Representação esquemática dos domínios presentes na proteína codificada pela ORF XAC3673 (http://pfam.sanger.ac.uk/). A proteína contém 579 resíduos de aminoácidos e três domínios: domínio fosforeceptor histidina quinase A (HisKA) próximo ao N-terminal; domínio ATPase histidina quinase (HATPase) e domínio regulador sensor (REC) receptor de fosfato presente no C-terminal. O local de inserção do Transposon no mutante 18D6 ocorreu no domínio REC logo após o resíduo 533 (indicado pela ponta de seta)...100
Figura 12. Representação esquemática de uma proteína transpotadora ABC na sua forma ativada pela hidrólise de uma molécula de ATP. Uma proteína específica periplasmática carrega a substância a ser transportada pelo transportador de membrana...103
sinal. (http://www.genome.jp/kegg/)...105
Figura 14. Representação esquemática do sistema de secreção do tipo II (SST2). Fonte: HOMEM, 2008. A seta vermelha indica o produto do gene xcsI que foi
menos expresso no mutante 18D6...107
CAPÍTULO 4 – Considerações Gerais
Figura 1.– Diagrama de Venn mostrando o número total de genes diferencialmente expressos comuns aos dois mutantes de Xanthomonas citri subsp. citri e o número
de genes diferencialmente expressos e exclusivos para cada um deles...117
Figura 2. Diagrama de Venn mostrando o número de genes da categoria VIII
diferencialmente expressos comuns aos dois mutantes de Xanthomonas citri
subsp. citri e o número de genes diferencialmente expressos exclusivos para cada
LISTA DE TABELAS
CAPÍTULO 2 – Análise da expressão gênica global in vivo do mutante do gene
xrvA de Xanthomonas citri subsp. citri (Xac).
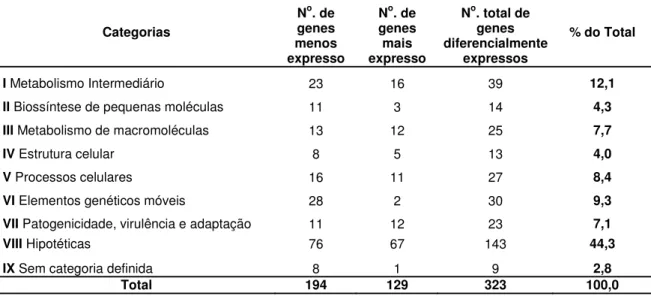
Tabela 1. Genes diferencialmente expressos no mutante 8B7 quando multiplicado por 72 horas em folhas de laranjeira Pêra e sua distribuição pelas grandes categorias funcionais...31
Tabela 2. Genes diferencialmente expressos no mutante 8B7 quando multiplicado por 72 horas em folhas de laranja Pêra e sua distribuição pelas categorias funcionais...37
CAPITULO 3 – Análise da expressão gênica global in vivo do mutante de uma histidina quinase de Xanthomonascitri subsp. citri (Xac).
Tabela 1. Genes diferencialmente expressos no mutante 18D6 quando multiplicado por 72 horas em folhas de laranjeira Pêra e sua distribuição pelas grandes categorias funcionais... 85
Tabela 2. Genes diferencialmente expressos no mutante 18D6 quando multiplicado por 72 horas em folhas de laranja Pera e sua distribuição pelas categorias funcionais...91
CAPÍTULO 4. Considerações gerais...
Tabela 1. Tabela comparativa de alguns dos genes diferencialmente expressos que apareceram no mutante 8B7 e 18D6...117
Tabela 2. Sequências gênicas (ORFs) presentes mais de uma vez (na forma de pares) no microarranjo e que se mostraram diferencialmente expressas no mutante 8B7...123 .
Tabela 3. Sequências gênicas (ORFs) presentes mais de uma vez (na forma de pares) no microarranjo e que se mostraram diferencialmente expressas no mutante18D6...123
Apêndice
Tabela Suplementar I. Categorias de genes da Xanthomonas citri subsp. citri
(Xac) utilizadas para anotação do genoma (SILVA et al.,
Análise da expressão gênica global em mutantes de Xanthomonas citri subsp. citri
RESUMO - O cancro cítrico é uma das principais doenças da cultura do citros, provocando lesões nas folhas, ramos e frutos, tendo como consequência a queda dos frutos e folhas, o que leva à perdas significativas na produção. A partir do
sequenciamento completo do genoma da bactéria gram-negativa Xanthomonas
citri subsp. citri (Xac), agente causal do cancro cítrico, abriu-se a possibilidade da
utilização de estratégias de análise genômica funcional no estudo da função de genes da bactéria relacionados com a infecção na planta e com o desenvolvimento da doença. Uma das estratégias utilizadas foi a obtenção de mutantes de Xac contendo genes relacionados à patogenicidade e virulência interrompidos pelo método de mutagênese insercional aleatória utilizando o
transposon Tn5 (LAIA et al., 2009). No presente trabalho a técnica de
microarranjos de DNA foi utilizada para avaliar a expressão global de genes de dois mutantes de Xac 72 h após a infecção in planta. Em um dos mutantes (8B7) o
gene interrompido foi o xrvA, um regulador de virulência, e no outro mutante
(18D6) o gene interrompido codifica uma histidina quinase híbrida sensora que faz parte de um sistema de transdução de sinal de dois componentes. Os resultados das hibridizações revelaram um total de 553 genes diferencialmente expressos para os dois mutantes estudados quando comparado com o genótipo selvagem (Xac 306), sendo 323 no mutante 8B7 e 230 no mutante 18D6. Esses genes foram divididos em diferentes categorias funcionais e uma análise funcional comparativa revelou que eles podem desempenhar um papel importante no processo de patogenicidade.
Palavras-chaves: Xanthomonas citri subp. citri, RNA, microarranjo,
Analysis of global gene expression in mutants of Xanthomonas citri subsp.
citri
SUMMARY – Citrus canker is a major disease affecting citrus crops worldwide, causing lesions on leaves, branches and fruits that results in the falling of fruit and leaves, leading to significant losses in orange production. The complete genome sequencing of Xanthomonas citri subsp. citri (Xac), a Gram-negative bacteria and
the causal agent of citrus canker, allowed the possibility of using functional genomic strategies to study the function of genes related to plant infection and disease development in this bacteria. One strategy was to produce mutants for phatogenicity and virulence genes by random insertional mutagenesis using Tn5 Transposon (LAIA et al., 2009). In the present work DNA microarray analysis was
used to evaluate the global gene expression profile of two Xac mutants after 72 hours of plant infection. One mutant (8B7) carry a mutation in the xrvA gene
(XAC1495), a virulence regulator, and the other (18D6) carry a mutation in a hybrid histidine quinase sensor of a two-component signal transduction system. The results revealed a total of 553 differentially expressed genes for the two mutant strains compared with Xac wild type, with 323 in the mutant 8B7, and 230 in the mutant 18D6. These genes were allocated into several functional categories and a comparative functional analysis showed that they can play an important role in the pathogenicity and virulence of Xac.
Keywords: Xanthomonas citri subsp. citri, RNA, microarray, pathogenicity, xrvA
CAPÍTULO 1 – CONSIDERAÇÕES GERAIS
1. INTRODUÇÃO
Desde a década de 80 que o Brasil tornou-se o maior produtor mundial de laranja. Com mais de 1 milhão de hectares de plantas cítricas em seu território, onde a maior parte da produção destina-se à indústria do suco, produz em média 1,7 milhões de toneladas e arrecadou aproximadamente 3,5 bilhões de reais em 2003. No Estado de São Paulo, na safra 2008/2009, a produção de laranja foi de 352,57 milhões de caixas (IEA). Portanto, a citricultura é muito importante para o país e para o estado de São Paulo, em particular.
O cancro cítrico é uma doença altamente destrutiva da cultura de citros, pois ocorre mundialmente e afeta a maioria das variedades comerciais de citros. Esta doença é causada pelos fitopatógenos Xanthomonas citri subsp. citri, X.
fuscan subsp. aurantifolli e X. alfalfae subsp. citrumelonis (SCHAAD, et al., 2006).
No Brasil, a forma asiática do fitopatógeno (X. citri subsp. citri - Xac) é a mais
severa, atacando todas as variedades de citros. (WHITESIDE et al., 1988).
A erradicação continua a ser utilizada porque o controle químico não é eficaz em barrar adequadamente o desenvolvimento da doença. Muito pelo contrário, seu uso pode ocasionar o surgimento de isolados altamente resistentes. Assim, a erradicação é um método adotado em todas as regiões produtoras. No entanto, novos métodos de controle devem ser buscados. Consonante com esse pensamento, o estudo das vias metabólicas utilizadas por Xac durante o processo infeccioso pode revelar novos alvos para o desenvolvimento de drogas que possam eliminar ou controlar o patógeno ou, até mesmo que possam ser utilizados para o desenvolvimento de tecnologia de controle baseado na transgenia.
Considerando a importância mundial da doença, especialmente no Brasil, pesquisadores brasileiros, financiados pela FAPESP, sequenciaram o genoma completo de Xac (isolado 306), agente causal da cancrose A, e o da Xanthomonas
campestris pv campestris (Xcc), agente causal da podridão negra em crucíferas
(SILVA et al., 2002). A Xac isolado 306 apresentou 4.313 ORFs, das quais 62,8%
estavam associadas a funções putativas quando da anotação do genoma. Dentre os demais genes, 29,5% mostraram-se conservados entre as duas bactérias e 331 (7,7%) são exclusivos de Xac. Em adição, Xac possui dois plasmídeos com 115
genes, sendo que a 55 deles não foi possível atribuir nenhuma função no processo de anotação.
inserção aleatória de Transposon, quando foram identificados diversos genes implicados no processo de patogênese e virulência nessa fitobactéria (LAIA et al.,
2009). Dentre esses mutantes, oito se mostraram avirulentos (ausência de sintoma) quando inoculados em folhas de limão cravo.
Apesar desses resultados serem altamente relevantes, a interferência desses genes no processo global de expressão gênica, modulando outros genes importantes, continua desconhecido. Esse conhecimento é de suma importância para a tomada de decisão em utilizar tais genes como possíveis alvos de controle. Portanto, o uso de outras abordagens, como a técnica de microarranjo de DNA, se presta à análise genômica funcional visando à elucidação de vias de transdução de sinais, permitindo identificar como se dá a modulação gênica durante o processo infeccioso. Desta maneira, não somente os genes, mas as vias envolvidas no processo assim determinadas poderão ajudar futuramente no desenvolvimento de drogas para o controle curativo da doença.
2. REVISÃO DA LITERATURA
2.1. Citricultura e o cancro cítrico
A citricultura brasileira é um dos setores mais competitivos do agronegócio mundial. O Brasil detém 40% da produção mundial de laranja e 60% da produção de suco de laranja, movimentando 9 bilhões por ano (CANAL RURAL, 2008). Desde a década de 80 o Brasil é o maior produtor mundial de laranja. No Estado de São Paulo, na safra 2008/2009, a produção de laranja foi de 352,57 milhões de caixas (IEA).
Apesar do grande potencial produtivo dos citros, que pode chegar a mais de 40 t de frutos por hectare (DONADIO, 1988), esses são suscetíveis a um grande número de doenças, dentre as quais algumas com alto potencial de destruição, como a tristeza (CTV) causada pelo vírus Citrus tristeza virus, a clorose variegada
PALAZZO, 1999), o cancro cítrico causado pela bactéria Xanthomonas citri subsp.
citri (Xac) (NAMEKATA et al., 1996) e, mais recentemente, o Huanglongbing (HLB)
ou Greening, causado pela bactéria Candidatus Liberibacter asiaticus e
Candidatus Liberibacter americanus (TEIXEIRA et al., 2005).
O cancro cítrico foi relatado pela primeira vez no Brasil em 1957, inicialmente na região de Presidente Prudente, SP, introduzido a partir de mudas importadas por imigrantes japoneses e se disseminou rapidamente para outros Estados. A doença foi posteriormente constatada em regiões do Estado de São Paulo, Mato Grosso do Sul, Paraná, Santa Catarina e Rio Grande do Sul (NAMEKATA et al., 1996). Desde que o primeiro relato da doença foi constatado
no Brasil, diversas medidas de controle vem sendo empregadas no intuito de reduzir os prejuízos causados por esta doença. No entanto nenhum controle curativo é eficaz, sendo que a única maneira de eliminar a doença do pomar é a exclusão ou erradicação da planta doente, processo que encarece substancialmente a produção, pois há a necessidade de eliminação de todas as plantas ao redor do foco em um raio de 30 m, sendo proibido o cultivo de citros no local pelos próximos dois anos sem que o citricultor tenha direito a qualquer indenização. Contudo, a erradicação das árvores contaminadas não garante a eliminação total da bactéria causadora do cancro (FUNDECITRUS). Além disso, Xac é praga quarentenária no Brasil (BRASIL, 1999) e em vários países produtores e consumidores, o que torna a presença dessa bactéria uma das principais barreiras fitossanitárias ao livre comércio de frutas frescas, limitando tanto o comércio internacional como o regional.
Além da erradicação outros métodos de controle do cancro cítrico são utilizados, como localização dos viveiros de mudas em áreas livres de cancro cítrico, estabelecimento de “quebra ventos” nas áreas de produção e uso de pulverizações de cobres e de controle da larva minadora dos citrus (ROBERTS, 2002).
O cancro cítrico é uma doença causada pela bactéria gram-negativa
uniflagelado e aeróbico obrigatório, que forma colônias amarelas e mucóides em meios de cultura artificiais. Na literatura são descritos três tipos de cancro cítrico: tipos A, B e C (MOREIRA et al., 2010). O tipo A, que causa mais dano econômico
para a citricultura, é originário da Ásia e é causado pela Xac, a qual é encontrada praticamente em todas as regiões onde já foi detectado o cancro cítrico (FEICHTENBERGER, 1998) e ataca todos os citros. Os tipos B e C são encontrados apenas na América do Sul. A cancrose B, presente na Argentina, Paraguai e Uruguai, tem como agente causal a X. fuscans subsp.aurantifolii type
B, a qual infecta Citrus limon mas também é encontrada em C. sinensis e C.
paradisi. A cancrose C, presente no Estado de São Paulo, é causada pela X.
fuscans subsp. aurantifolii type C, a qual ataca apenas C. aurantifolia (limão
galego).
A disseminação de Xac em plantas de citros ocorre através das chuvas, restos de colheita, pessoas ou veículos transitando pela lavoura (PALAZZO et al.,
1984).
Os sintomas característicos de cancro cítrico envolvem a formação de lesões corticosas e salientes em toda a parte aérea da planta, incluindo folhas, ramos e frutos, que se manifestam de uma semana a dois meses após a infecção (AMORIM & FILHO, 1999). A indução da formação de tecido hiperplástico (divisões mitóticas excessivas calogênicas), que resulta em lesões do tipo cancro, é a sintomatologia característica e essencial para o diagnóstico desta doença (Figura 1).
SILVA et al. (2002) publicaram o sequenciamento e a anotação do genoma
Figura 1. Sintomas de cancro cítrico causado por Xac em folhas, ramos e frutos (Fonte: Fundecitrus)
2.2. Xanthomonas citri subsp. citri
O gênero Xanthomonas contém uma ampla variedade de espécies,
linhagens e hospedeiros (124 monocotiledôneas e 286 dicotiledôneas) (LEYNS et
al., 1984) e representa o maior grupo de bactérias fitopatogênicas, com uma
grande diversidade patogênica e importância econômica (VAUTERIN et al., 2000;
VICENTE et al.; 2001). Além disso, o agente causal do cancro cítrico está entre as
Xanthomonas como a bactéria que causa maior impacto na agricultura mundial
(SCHUBERT et al., 2001).
Existem duas características morfológicas comumente encontradas no gênero Xanthomonas: 1) a produção de goma xantana (exopolissacarídeo que
A reclassificação das espécies de Xanthomonas patogênicas de citros foi
realizada por estudos das análises de sequências de regiões ITS (“internal transcribed spacer”), por AFLP (“amplified fragment lenght polymorphism”), por testes de re-associação DNA-DNA e por características fenotípicas (SCHAAD et
.al., 2006). Anteriormente todas pertenciam a uma mesma espécie bacteriana,
Xanthomonas axonopodis, e eram separadas por patovares (citri, aurantifolli e
citrumelonis) (VAUTERIN et al., 1995). A recente classificação considera esses
organismos como sendo pertencentes a três espécies diferentes: Xanthomonas
citri subsp. citri (antes Xanthomonas axonopodis pv. citri - Xac), Xanthomonas
fuscans subsp. aurantifolii (antes Xanthomonas axonopodis pv. aurantifolli) e
Xanthomonas alfalfae subsp. citrumelonis (antes Xanthomonas axonopodis pv.
citrumelonis) (SCHAAD et al., 2006).
Xac apresenta respiração aeróbica, metabolismo oxidativo e nunca fermentativo, não é fixadora de nitrogênio, não forma esporos, apresenta morfologia de bastonetes, possui flagelo polar, está adaptada ao ambiente das plantas cítricas (crescimento endofítico), sendo raramente encontrada livre no solo (SWINGS & CIVEROLO, 1993; WELLS, 1987).
A penetração de Xac na planta hospedeira ocorre nos tecidos vegetais de citros através de aberturas naturais como os estômatos, hidatódios e lenticelas ou por ferimentos (GOTTWALD & GRAHAM, 1992; GRAHAM et al., 1992). Assim,
2.3. Interação Planta-patógeno
O mecanismo de interação entre patógeno e planta é caracterizado por trocas de sinais que envolvem um sistema de “ataque e defesa contra o ataque” (STAHL & BISHOP, 2000). De um lado a planta tenta eliminar o patógeno no sítio de infecção, ocorrendo suicídio das células atacadas, e do outro, o patógeno tenta desativar o sistema de defesa da planta para sobreviver. Assim a presença do patógeno ativa na planta a produção de substâncias para evitar a instalação dos microrganismos. No entanto, se o patógeno conseguir se instalar no tecido vegetal, um segundo mecanismo de defesa da planta é ativado para impedir a proliferação do patógeno. Se o patógeno ganha a batalha, a doença se instala e diz-se que a planta é sensível; se a planta ganha a batalha, seu mecanismo de defesa é eficiente contra aquele patógeno e diz-se que a planta é resistente.
A proteção das plantas contra a maioria dos patógenos é adquirida por meio de barreiras pré-formadas, tais como cutículas espessas e presença de compostos antimicrobianos (HAMMOND-KOSACK & JONES, 2000).
A defesa da planta é induzida por um caminho básico de reconhecimento e transdução de sinais para ativar as respostas de defesas, tais como: geração de espécies ativas de oxigênio (DIXON et al., 1994; BOLWELL et al., 1995), morte
programada de células no sítio de infecção (resposta de hipersensibilidade - HR) (HAMMOND-KOSACK & JONES, 1996), deposição de glicoproteínas ricas em hidroxiprolinas (CORBIN, et al., 1987), lignificação da parece celular (GRAND et
al.,1987), síntese e acúmulo de fitoalexinas (GUSTINE et al., 1990) e síntese de
proteínas PR antimicrobianas.
Muitas bactérias fitopatogênicas colonizam o espaço intercelular (apoplasto) de plantas hospedeiras. Este nicho é nutricionalmente pobre, carregado de substâncias antimicrobianas e acídicas. No entanto, a fim de garantir a sua sobrevivência, o patógeno necessita desenvolver estratégias para conseguir o máximo de nutrientes do hospedeiro para sua consequente colonização e multiplicação. O estabelecimento do patógeno bacteriano no hospedeiro e sua habilidade em causar doenças dependem de um processo complexo que requer a atividade coordenada de muitos genes bacterianos ligados à patogenicidade e virulência. Os produtos destes genes são, em última análise, os responsáveis pela virulência e patogenicidade do patógeno e, na grande maioria dos casos, a identidade e o seu modo de ação ainda são obscuros.
As interações entre planta e patógeno são denominadas como compatíveis ou incompatíveis. Na interação compatível, a bactéria é capaz de mascarar ou reprimir os mecanismos de defesa da planta hospedeira, desenvolvendo-se nos tecidos vegetais, manifestando a doença por meio de mecanismos que envolvem a produção de toxinas, secreção de enzimas líticas extracelulares e liberação de fatores de virulência nos meios extra e intracelular da planta hospedeira (WHITE
et al., 2000).
Logo, a interação compatível implica na doença propriamente dita, na qual ocorre a invasão e colonização do patógeno na planta hospedeira, sem que esta consiga disparar seu arsenal de defesa o suficiente em intensidade e tempo para barrar o patógeno.
Na interação incompatível, ocorre uma restrição ao sítio de infecção, pois ocorre um rápido e intenso disparo dos mecanismos de defesa da planta em resposta ao reconhecimento específico entre uma molécula elicitora do patógeno e o receptor específico da planta.(CAMARGO, 1995). Este tipo de interação prediz que o gene de avirulência (avr) do patógeno codifica uma proteína elicitora (Avr)
capaz de interagir especificamente com um receptor na planta (proteína R) codificada pelo gene de resistência R (GABRIEL et al., 1993; KEEN, 1990; BUEL,
R da planta, leva a uma rápida ativação das vias de transdução de sinais que resulta na resposta de resistência, muitas vezes associada a uma necrose localizada também denominada reação de hipersensibilidade (HR, “Hipersensitivity Response”)
A resposta de hipersensibilidade é caracterizada por uma morte celular programada das células vegetais infectadas pelo patógeno e células adjacentes ao sítio de infecção, que leva ao bloqueio e eliminação deste sem que haja manifestação da doença (GOODMAN & NOVACKY, 1994; STASKAWICZ et al.,
1995). Os eventos fisiológicos associados à HR envolvem um aumento intracelular dos níveis de espécies ativas de oxigênio (O2-, H2O2 e OH-), perda de íons
potássio (K+) e ganho de íons hidrogênio (H+) pelas células, destruição de compartimentos e fortalecimento das paredes celulares e da cutícula vegetal, produção de agentes antimicrobianos (fitoalexinas) e indução da expressão de proteínas de defesa (conhecidas como proteínas PR, de “pathogenesis related”),
que limitam a colonização de tecidos vegetais pelo agente invasor (HAMMOND-KOSAK & JONES, 1996). A clonagem e caracterização de diversos genes avr de
origem bacteriana revelaram uma grande diversidade entre os produtos destes genes, o que impediu que fossem feitas generalizações sobre a estrutura de moléculas elicitoras (VIVIAN & GIBBON, 1997).
Estudos com mutantes para os genes avr mostraram que a presença deles
confere uma vantagem seletiva para o patógeno na ausência do gene R
correspondente (LAUGE et al.,1998; KJEMTRUP et al., 2000; WHITE et al., 2000;
STASKAWICZ et al.; 2001), o que explica sua manutenção na população do
patógeno. Contudo, postula-se que os genes avr poderiam agir, primeiramente,
como fatores de virulência que, durante a co-evolução do patossistema, se tornaram alvos dos genes R da planta (COLLMER, 1998). No entanto, apesar de importantes para o patógeno, os genes avr sofrem frequentes mutações, o que
Além dos genes avr, um agrupamento gênico denominado hrp
(“hipersensibility response and pathogenicity”) foi evidenciado em estudos com mutantes de bactérias fitopatogênicas, para os quais tem se verificado um papel tanto na patogenicidade em plantas suscetíveis quanto na indução de hipersensibilidade (HR) em plantas contendo o gene de resistência (R) correspondente ou em planta não-hospedeiras (BONAS & VAN DEN ACKERVEKEN, 1997).
Assim, a verificação de que os genes hrp são essenciais para a
patogenicidade e indução de HR indica que a funcionalidade dos genes avr
depende dos genes hrp (DANGL., 1994). Os genes hrp foram primeiramente
identificados em Pseudomonas spp e em bactérias fitopatogênicas gram-negativas
dos gêneros Xanthomonas spp., Rasltonia solanocearum e Erwinia spp. (ALFANO
& COLLMER, 1997). Algumas proteínas destes agrupamentos de genes apresentaram homologia com vários componentes do sistema de secreção do tipo III (SSTT) de patógenos bacterianos de animais (Yersinia, Shigella, Salmonella),
onde as proteínas codificadas por genes hrp formam uma estrutura tubular do tipo
“pilus” capazes de conectar as células bacterianas e animal, permitindo a translocação de proteínas da bactéria relacionadas à virulência, através da membrana plasmática, diretamente para o citoplasma da célula hospedeira animal (VAN GIJESEGEM et al., 1993).
RAHME et al. (1992), em estudos de controle da expressão de genes hrp,
mostrou que há uma complexidade inerente à função indispensável que eles exercem para o estabelecimento tanto de interações compatíveis como de interações incompatíveis, no confronto bactéria-planta. A regulação dos genes hrp
é dirigida por sinais da planta, osmolariedade, pH e disponibilidade de nutrientes, evidenciando uma complexa gama de sinais aos quais a fitobactéria é confrontada nos diferentes estádios de seu desenvolvimento na região apoplástica do tecido vegetal.
spp., Ralstonia spp. e Ewinia spp. permitiram evidenciar características peculiares
a cada tipo de doença (ALFANO JR. & COMMER A., 1997). Portanto, estudos sobre o mecanismo capaz de mediar a injeção de proteínas secretadas por bactérias diretamente na célula vegetal vêm possibilitando uma melhor compreensão dos diversos aspectos que envolvem a indução de HR em plantas resistentes.
2.4. Mutagênese aleatória
O sequenciamento completo do genoma de Xanthomonas citri subsp. citri
(Xac) e de Xanthomonas campestris pv. campestris (Xcc) e a anotação dos genes
presentes nestes genomas permitiram a identificação de genes putativos envolvidos na patogenicidade e virulência destas fitobactérias (SILVA et al., 2002).
A disponibilidade da sequência do genoma de vários organismos levou ao surgimento de técnicas de análise de expressão gênica em larga escala, tais como os macro e microarranjos de DNA e o estudo do proteoma diferencial, as quais permitem uma análise global da expressão gênica em diferentes condições experimentais. Essas abordagens também estão sendo utilizadas nas pesquisas com bactérias patogênicas de plantas e devem revelar novos genes e mecanismos envolvidos na patogenicidade e virulência .
Uma outra abordagem que permite identificar em larga escala genes ligados a patogenicidade e virulência é a mutagênese aleatória por meio da inserção de transposon no genoma in vivo. Esta técnica tem sido amplamente
utilizada com sucesso em vários microrganismos patogênicos e não patogênicos (BROWN & HOLDEN, 1998; FERREIRA et al., 2001; HUDSON et al.; 2006). LAIA
et al. (2009), utilizando esta técnica, obteve uma biblioteca de mutantes de Xac
inoculados em folhas de plantas de limoeiro cravo para confirmação da alteração da patogenicidade, 122 apresentaram algum tipo de alteração na capacidade de causar doença ou em elicitar sintomas característicos de cancro, sendo que em 8 deles houve ausência total de sintomas (LAIA et al., 2009).
A utilização de mutantes para estudos de análise da expressão gênica em larga escala em comparação com as cepas selvagens irá revelar os genes diferencialmente expressos em cada caso, o que levará a uma maior compreensão do processo infeccioso na interação planta-patógeno.
2.5. Técnica de microarranjos
Com o sequenciamento completo do genoma humano e de outros
organismos, surgiu um novo desafio que é compreender como esses genes atuam. A disponibilidade da estrutura de alguns genes abre caminhos para uma difícil tarefa que é a de atribuir funções biológicas a cada gene individualmente, sendo mais desafiante ainda explicar as redes altamente complexas de regulação dentro das células vivas, onde genes, proteínas e metabólitos se interagem. Técnicas tradicionais de estudos de expressão gênica como Northen blotting e
Diferencial display permitem a análise de um ou alguns genes simultaneamente,
oferecendo uma compreensão limitada dentro dos processos celulares. Todavia, a
técnica de microarranjo (microarray) de DNA é uma ferramenta muito mais
poderosa para estudos de expressão gênica em larga escala. Assim, essa disposição global da técnica de microarranjo de DNA permite elucidar um conjunto de genes e redes metabólicas.
observadas não são resultados de eventos aleatórios ou biológicos com variabilidade inerente à metodologia experimental utilizada (DHARMANDI et al.,
2004).
Apesar do primeiro artigo de análise de expressão gênica utilizando microarranjo de DNA ter sido publicado em 1995 (SCHENA et.al, 1995), o conceito
miniaturizado de análise de hibridização foi desenvolvido em meados dos anos 80 (EKINS & CHU, 1999). Por exemplo, estudos imunoquímicos utilizando composto fluorescente foram descritos por Ekins em 1989, e o uso de hibridização com marcação fluorescente foi avaliado por concentração analítica de proteínas. Embora não seja exatamente equivalente, o uso de sondas fluorescentes para avaliar hibridização de ácidos nucléicos é o suporte principal de muitos estudos de arranjos e é o início de uma extensão lógica de estudos imunoquímicos.
O microarranjo tem sido utilizado em vários estudos de expressão gênica, e um exemplo de sua utilização é nos estudos de microrganismos patogênicos, tanto de plantas como em animais.
Por décadas pesquisadores tem utilizado uma ampla variedade de métodos para estudar tanto a interação de microorganismos patogênicos com seus hospedeiros como os mecanismos de defesa utilizados pelos hospedeiros. Estas informações são cruciais para um esboço seguro e efetivo de diagnóstico, terapêutica e vacinas (MAEDA et al., 2001)
O presente estudo teve por objetivo avaliar a expressão gênica global in
planta, por meio da técnica de microarranjo de DNA, de mutantes de
Xanthomonas citri sbsp. citri (Xac) que apresentam patogenicidade alterada ou
REFERÊNCIAS
ALFANO, J. R.; COLLMER, A. The type III (Hrp) secretion pathway of plant pathogenic bacteria: trafficking harpins, avr proteins, and death. Journal of Bacteriology, Washington, v.179, p.5655-5662, 1997
AMORIM, L. e BERGAMIN FILHO, A. A epidemiologia do cancro cítrico. In: L. ZAMBOLIM e A. J. A. MONTEIRO (Ed.). 3º. Encontro de fitopatologia: Doenças de fruteiras tropicais. Viçosa: Universidade Federal de Viçosa, 1999. A epidemiologia do cancro cítrico, p.1-17.
BEBENDO, I. P. Manchas foliares. In: Bergamin Filho, A., Kimati, H.; Amorin, L. eds. Manual de Fitopatologia. Volume 1: Princípios e conceitos, 3. ed. São Paulo: Agronômica Ceres, p. 848-858, 1995.
BOLWELL, G. P. et al. The origin of the oxidative burst in plants. Free Radical
Research, Yverdon, v. 23, n. 6, p. 517–532, 1995.
BONAS, U.; LAHAYE, T. Plant disease resistance triggered by pathogen-derived
molecules:refined models of specific recognition. Current Opinion in
Microbiology,London,v. 5, n. 1, p. 44–50, 2002.
BONAS, U.; & VAN DEN ACKERVEKEN, G. Recognition of bacterial avirulence proteins occurs inside the plant cell: ageneral phenomenon in resistance to bacterial diseases? The Plant Journal, v. 12, n. 1, p.1-7, 1997.
BRASIL. Ministério da agricultura e do abastecimento –secretaria de defesa agropecuária. Instrução Normativa-SDA nº38/1999 de 14 de outubro de 1999. Brasília - DF, 1999.
BROWN, J. S.; HOLDEN, D. W. Insertional mutagenesis of pathogenic fungi.
Current Opinion in Microbiology,London, v. 1, n. 4, p. 390–394,1998.
BUEL, C.R. Genes Involved in Plant-Pathogen Interactions. Induced Plant
Defensed Against Pathogens and Herbivores. 2nd. End. APS PRESS. The
American Phytopathological Society, St. Pul Minnesota, 1999.
CAMARGO, L. E. A. Análise genética da resistência e da patogenicidade. In: BERGAMIN FILHO, A., KIMATI, H.; AMORIM, L. (Eds.). Manual de Fitopatologia, v.1, p.470-492, 1995.
COLLMER, A. The Pseudomonas syringae pv. tomato HrpW protein has domains
similar to harpins and pectate lyases and can elicit the plant hypersensitive responseand bind to pectate. Journal of Bacteriology, Washington, v.180, p.5211-5217, 1998.
CORBIN, D. R.; SAUER, N.; LAMB, C. J. Differential regulation of a hydroxyproline-rich glycoprotein gene family in wounded and infected plants.
Molecular and Cellular Biology, Washington, v. 7, n. 12, p. 4337–4344, 1987.
DANGL, J. L. The enigmatic avirulence genes of phytopathogenic bacteria.
Current topics in microbiology and immunology, Berlin, v.192, p.99-118, 1994.
DHARMANDI, Y and GONZALEZ, R. DNA Microarrays: Experimental Issues, Data Analysis, and Application to Bacterial Systems. Biotechnology progress, New York, v.20, n. 5, p. 1309-1324, 2004.
DIXON, R. A.; HARRISON, M. J.; LAMB, C. J. Early events in the activation of plantdefense responses. Annual Review of Phytopathology, Palo Alto, v. 32, p. 479–501, 1994.
DONADIO, L. C. Produtividade dos citros em Israel. In: L. C. DONADIO (Ed.).
Produtividade de citros. Jaboticabal: FUNEP, 1988. Produtividade dos citros em Israel, p.47-62
EKINS, R. and CHU, F.W. Microarrays: theirs origins and aplications. Trends Biotechnology, Amsterdam, v.17, p. 217-218, 1999.
FEICHTENBERGER, E. Manejo ecológico das principais doenças fúngicas e
bacterianas dos citros no brasil. In: DONADIO, L. (Ed.). Anais do V Seminário Internacional de Citros – Tratos Culturais. Bebedouro: Fundação Cargill, 1998. p. 517.
FERREIRA, M. C. D. et al. Transposon mutagenesis reveals novel loci affecting
tolerance tosalt stress and growth at low temperature. Current Genetics,New York, v. 40, n. 1, p. 27–39, 2001
FUNDECITRUS. Fundo de Defesa da Citricultura. 2010.Disponível em:<
http://www.fundecitrus.com.br>. Acesso em: 02 Fev. 2010.
GABRIEL, D.W.; KINGSLEY, M.T.; YANG, Y.; CHEN, J.; e ROBERTS, P. Host-especific virulence genes of Xanthomonas. Molecular mechanisms of bacterial
virulence. Klumer Academics Publishers, Dordrecht, Netherlands. p. 141-158. In C.I. Kado and J. Crosa (ed.), 1993.
pathogens: A resistance phenomenon. St. Paul: APS Press, 1994
GOTTWALD, T. R.; GRAHAM, J. H. Compendium of Citrus Diseases (eds
TIMMER, L. W.; GARNSEY, S. M.; GRAHAM, J. H.), Am. Phytopathol. Soc., SP, 2000. p.5–7.
GRAHAM, J.H.; GOTTWALD, T.R.; RILEY, T.D.; ACHOR, D. Penetration through leaf stomata and growth of strains of Xanthomonas campestris in citrus cultivars
varying in susceptibility to bacterial diseases. Phytopathology, St. Paul, v. 82, n. 11, p. 1319-1325, 1992
GRAND, C.; SARNI, F.; LAMB, C. J. Rapid induction by fungal elicitor of the synthesisof cinnamyl-alcohol dehydrogenase, a specific enzyme of lignin synthesis. European Journal of Biochemistry,Berlin, v. 169, n. 1, p. 73–77, Nov 16 1987.
GUSTINE, D. L.;SHERWOOD, R.T.; LUKEZIC F. L.; MOYER B. G. AND DEVLIN W.S. Metabolites from pseudomonas-corrugata elicit phytoalexinbiosynthesis in white clover. Phytopathology,St. Paul, v. 80, n. 12, p. 1427–1432, 1990
HAMMOND-KOSACK, K.; JONES, J. D. G. Responses to plant pathogens. In:
.Biochemistry & molecular biology of plants. Rockville: American Society of Plant Physiologists, 2000. cap. 21, p. 1102–1156.
HAMMOND-KOSACK, K. E.; JONES, J. D. G. Resistance gene-dependent plant defense responses.The Plant Cell, Rockville, v. 8, n. 10, p. 1773–1791, 1996.
HUDSON, P. et al. Identification of a virulence-associated determinant,
dihydrolipoamide dehydrogenase (lpd), in Mycoplasma gallisepticum through in vivo screening of transposonmutants. Infection and immunity, Bethesda, , v. 74, n. 2, p. 931–939, 2006.
IEA. Instituto de Economia Agrícola. 2010. Disponível em:
http://www.iea.sp.gov.br/out/verTexto.php?codTexto=1043
KEEN, N. T. Gene-for-gene complementarity in plant-pathogen interactions.
Annual Review.Genetic. Palo Alto, v.24, p.447-463, 1990.
KJEMTRUP, S.; NIMCHUK, Z.; DANGL, J. L., Effector proteins of phytopathogenicbacteria: bifunctional signals in virulence and host recognition.
Current Opinion Microbiology, London, n.3, v.1, p.73-78, 2000.
LAIA, M.L.; MOREIRA, L.M.; DEZAJACOMO, J.; BRIGATI, J.B.; FERREIRA, C.B.; FERRO, M.I.T.; SILVA, A.C.R.; OLIVEIRA, J.C.F.; FERRO, J.A. New genes of
a transposon-based mutant library. BMC Microbiology, London, v.9, n.12, p.1-17, 2009.
LAUGE, R.; DE WIT, P. Fungal avirulence genes: Structure and possible functions.Fungal Genet. Biol, Orlando, n.24, v.3, p.285-297, 1998.
LEYNS, F.; DE CLEENE, M.; SWINGS J. G.; DE LEY, J. The host range of the genus Xanthomonas. Botanical Review, New York, v.50, p.308-356, 1984.
MAEDA, K.M.; GAO, Q.; SMALL, P.M. Microarray analysis of pathogens and their interaction with hosts. Cellular Microbiology,Oxford, v.3, p. 1-8, 2001
Leandro M Moreira, Nalvo F Almeida Jr, Neha Potnis, Luciano A Digiampietri. Novel insights into the genomic basis of citrus canker based on the genome sequences of two strains of Xanthomonas fuscans subsp. Aurantifolii. BMC
Genomics,London,11:238, 2010.
NAMEKATA, T., ROSSI, A. C. e CERAVOLO, L. C. Avaliação de novos métodos de erradicação de cancro cítrico. Laranja, v.17, n.1, p.67-78. 1996.
PALAZZO, D. A., MALAVOLTA JÚNIOR, V. A. e NOGUEIRA, E. M. C. Effect of some climatic factors on the index of infection of citrus canker caused of
Xanthomonas campestris pv. citri on Valencia orange (Citrus sinensis) in
Bataguassu, MS. Fitopatologia Brasileira,Brasilia, v.9, n.2, p.283-290, 1984.
PADMANABHAM, D.; VIDHYASEKARAN, P.; RAJAGOPALAN, C.K.S.; Chances in photosynthesis and carbohydrate content in canker and halo regions in
Xanthomonas citri infected citrus leaves. Indian Journal of
Phytopathology,Berlin, v. 26, p. 215-217, 1973.
ROBERTS, P. D. Citrus Canker Management Strategies. Indian river citrus
seminar. Southwest Florida Research and Education Center. 2002.
RAHME, L. G.; MINDRINOS, M. N.; PANOPOULOS, N. J. Plant and
environmentalsensory signals control the expression of hrp genes in
Pseudomonas syringae pv. phaseolicola. Journal of. Bacteriology, Washington, v.174, n.11,p.3499-3507,1992.
SCHAAD, N.W.; POSTNIKOVA, E.; LACY, G.; SECHLER. A.; AGARKOVA, I.; STROMBERG. P.E. Stromberg VK, Vidaver AK: Emended classification of
xanthomonad pathogens on citrus. Systematic and Applied Microbiology,
SCHENA, M. l.;SHALON, D.;DAVIS, R.W.; BROWN, P.O. Quantitative monitoring of gene expression patterns with a complementary DNA microarray. Science, New York, v.270, p. 467-470, 1995.
SCHUBERT, T.S.; RIZVI, S.A.;SUN, X.;GOTWALD, T.R.;GRAHAM, J.H.; DIXON,
W.N.. Meeting the challenge of eradicating citrus canker in Florida Again. Plant Disease, St. Paul, v.5, p.340-356, 2001.
SMYTH, G. K. Limma: linear models for microarray data. In: . Bioinformatics and Computational Biology Solutions using R and Bioconductor. New York: Springer, 2005. p.397–420.
STAHL, E.A. & BISHOP, J.G. Molecular evidence for plant-patogen arms races.
Current Opinion in Plant Biology,London,v.3, n.4, p. 299-304, 2000.
STASKAWICZ, B. J.,, MUDGETT, M.B.; DANGL J.L.; GALAN, J.E. Common and
contrasting themes of plant and animal diseases. Science, New York, n.292, v.5525, p.2285-2289, 2001.
STASKAWICZ, B. J.; AUSUBEL, F.M.; BAKER, B.J.; ELLIS, J.G.; JONES,. J.D.; Molecular genetics of plant disease resistance. Science, New York, v.268, p.661-667, 1995.
SWINGS, J. G.; CIVEROLO, E. L. Xanthomonas. Chapman and Hall, New York,
NY, 1993.
SILVA, A. C. R. DA.; FERRO, J.A.; REINACH, F. C. et al.Comparison of the
genomes of two Xanthomonas pathogens with differing host specificities.
Nature,London, v.417, p.459-463, 2002.
TEIXEIRA, D.C., DANET, J.L., EVEILLARD, S., MARTINS E.C., CINTRA, W.C.J.; YAMAMOTTO, P.T., LOPES, S.A., BASSANEZZI, R.B., AYRES, A.J., COLETTE, S., BOVÉ, J. Citrus huanglongbing in São Paulo State, Brazil: PCR detection of the Candidatus Liberibacter species associated with the disease. Molecular and Cellular Probes, Londres, v.19, n.3, p.173-179, 2005.
VAUTERIN, L.; RADEMAKER, J.; SWINGS, J. Synopsis on the taxonomy of the genus Xanthomonas. Phytopathology, St. Paul, v.90, p.677-682, 2000.
VAUTERIN, L.; SWINGS, J.; KERSTERS, K. Grouping of campestris pathovars by
SDS-PAGE of proteins. Journal of General Microbiology, Reading, ondon,
v.137, p.1307-16,1996.
VAUTERIN, L.; HOSTE, B.; KERSTERS, K.; and SWINGS, J. Reclassification of
n.3, p.472-489,1995.
VICENTE, J. G.; CONWAY, J.; ROBERTS, S, J.; TAYLOR, J. D. Identification and origin of Xanthomonas campestris pv. campestris races and related pathovars.
Phytopathology, St. Paul, v.91, p.492-499, 2001.
VIVIAN, A.; E GIBBON, M. J. Avirulence genes in plant-pathogenic bacteria: signal or weapons? Microbiology, Reading,Washington, v.143, p.693-704, 1997.
WELLS, J. et al. Xylella fastidiosa gen nov, sp nov Gram-negative, xylem
limitedfastidious plant bacteria related to Xanthomonas ssp., International
Journal Of Systematic Bacteriology,Reading, Ames,v.37, p.136-143, 1987.
WHITE, F. F.; YANG, B.; JOHNSON, L. B. Prospects for understanding avirulence gene function. Current Opinion in Plant Biology,London, v. 3, n. 4, p. 291–298, 2000.
WHITESIDE, J. O.; GARNSEY, S. M; TIMMER, L. W. Compendium of citrus
CAPÍTULO 2 – Análise da expressão gênica global in vivo do mutante do gene xrvA de Xanthomonascitri subsp. citri (Xac).
RESUMO – Em um trabalho recente do laboratório foi demonstrado que a interrupção do gene xrvA (XAC1495) de Xanthomonas citri subsp. citri (Xac), um
regulador de virulência, fez com que a bactéria mutante (8B7) deixasse de
apresentar alguns dos sintomas característicos do cancro cítrico, tais como
encharcamento, necrose e hiperplasia. O gene xrvA codifica uma proteína
contendo um domínio semelhante ao domínio H-NS (“histone-like nucleoid
structuringprotein”) e proteínas contendo domínio H-NS e H-NS-like são proteínas
modulares associadas ao nucleóide bacteriano. Além disso, alguns estudos tem sugerido que a proteína codificada pelo gene xrvA pode estar envolvida em
processos de replicação, recombinação e transposição. O objetivo do presente trabalho foi o de verificar, por meio da técnica de microarranjo de DNA, se a interrupção desse gene afeta a expressão de outros genes. Para isso, avaliou-se a expressão diferencial global de genes do mutante 8B7 após 72 h de infecção in
planta comparado com Xac selvagem). Os resultados obtidos mostraram que 323
genes foram diferencialmente expressos, sendo 194 mais expressos e 129 menos expressos em comparação com Xac selvagem. Esses genes estão distribuídos em 56 categorias funcionais e, embora a grande maioria codifique proteínas hipotéticas, a mutação no gene xrvA levou a uma redução na expressão de genes
relacionados a patogenicidade e virulência, incluindo xcsI, hrpX e rpfC. Além
desses, genes do sistema de secreção do tipo IV (SST4), transportadores ABC, transportadores de ferro, genes relacionados a quimiotaxia e motilidade celular e elementos genéticos móveis também se mostraram diferencialmente expressos no mutante.
Palavras-Chaves: Xanthomonas citri subsp. citri, mutante, Microarranjo de DNA,
CAPíTULO 2 – In vivo Global gene expression analysis of a Xanthomonas
citri subsp. citri (Xac) mutant carrying a xrvA knockouted gene
SUMMARY – Recently was demonstrated that mutation in the xrvA Xac gene
(XAC1495), a virulence regulator, results in the abolishment or mildness of citrus canker symptons in the mutant (8B7), such abolishment of moist spots and necrose and reduction of hyperplasia. The xrvA gene codes for a histone-like
nucleoid-structuring (H-NS) protein that belongs to a family of bacterial proteins that play a role in the formation of nucleoid structure and affect gene expression under certain conditions. In the present work DNA microarray analysis was used to verify which other genes had their expression profile altered by the disruption of the
xrvA gene. A global gene expression analysis was performed in the mutant 72
hours after plant infection in comparison with Xac wild type. The results showed that 323 genes were differentially expressed in the mutant, with 194 being more expressed and 129 being less expressed in relation to Xac wild type. These genes are distributed um 56 functional categories, and although the majority code for hypothetic proteins, genes coding for proteins related to virulence and pathogenicity showed a marked alteration in expression, including xcsI, hrpX and
rpfC. Besides these genes, others genes also related to virulence and
pathogenicity mechanisms also showed a differencial expression in the mutant, including genes belonging to type IV secretion system (T4SS), ABC transporters, iron transporters, chemotaxis and cell motility related genes and mobile elements
Keywords: Xanthomonas citri subsp. citri, mutant, DNA microarray, differentially
INTRODUÇÃO
A metodologia de microarranjo de DNA vem sendo muito utilizada para detecção de alterações de expressão gênica em bactérias em respostas a diversas condições ambientais e de estresse (LUCCHINI et al., 2001). Esta
metodologia também tem sido utilizada em diversos estudos de análise comparativa de genomas de bactérias patogênicas e na obtenção do perfil de expressão destas bactérias em meios de cultura que simulam o microambiente do hospedeiro (SCHOOLNIK, 2002). O objetivo final dos estudos de expressão gênica em larga escala de bactérias fitopatogênicas é a identificação dos genes da bactéria que são regulados diferencialmente pelo hospedeiro, estando dentro desta classe de genes aqueles que são responsáveis pela adaptação do patógeno aos microambientes do hospedeiro ou que codificam determinantes de virulência (SCHOOLNIK, 2002).
SEIB et al. (2007) compararam dois isolados de Neisseria gonorrhoeae,
agente causal da gonorréia, um selvagem e outro mutante, por meio da técnica de microarranjos de DNA. Os autores observaram no mutante para o gene oxyR a
perda da atividade do gene gor e prx e um aumento na expressão do gene katA,
que codifica uma catalase. HAN et al. (2006) verificaram diferenças metabólicas
relacionadas ao tamanho de colônias em um modelo murino por meio dessa mesma técnica. Células mutantes para o gene Tk (timidina quinase) apresentaram
dois tipos de fenótipos quanto ao crescimento, colônias pequenas e colônias grandes. As colônias maiores têm uma taxa de crescimento normal, enquanto os menores apresentam uma baixa taxa de crescimento. Essa diferença é relacionada a mutações pontuais ou cromossomais. A comparação por meio de microarranjos de DNA entre esses dois tipos de colônias possibilitou identificar genes relacionados, e possivelmente responsáveis, por aqueles fenótipos (HAN et
al., 2006). Igualmente, FADL et al. (2007) analisaram a expressão diferencial de
genes em Aeromonas hydrophila selvagem versus um mutante para os genes act
enterotoxina citotóxica secretada do tipo II, respectivamente. Eles observaram que 434 genes tiveram a expressão alterada no modelo murino e 80 tiveram a expressão alterada em células epiteliais humanas.
O mutante 8B7 (XAC1495), com inserção do Transposon no gene xrvA, um
regulador de virulência, apresentou ausência de encharcamento, leve hiperplasia e ausência de necrose. Sabe-se que o gene xrvA codifica uma proteína contendo
um domínio semelhante ao domínio H-NS (“histone-like nucleoid structuring
protein”) e que proteínas contendo domínio H-NS e H-NS-like são proteínas
modulares associados ao nucleóide bacteriano. H-NS é uma das proteínas mais abundantes em DNA de enterobactérias. Além disso, tem sido sugerido um possível papel para H-NS em muitos processos biológicos como replicação, recombinação e transposição (USSERY et al., 1994).
A proteína H-NS atua como um fator regulatório global em linhagens de E.
coli não patogênicas e estudos de expressão gênica global em um mutante do
gene hns em E. coli patogênica mostrou que a ausência do gene tem um grande
impacto no padrão de expressão gênica da E. coli e que o mesmo regula também
a expressão dos fatores de virulência clássicos (MÜLLER et al., 2006). Vários
trabalhos têm indicado que o gene xrvA é um regulador de virulência em várias
bactérias patogênicas. Recentemente foi mostrado que uma linhagem de Xylella
fastidiosa que causa a doença de Pierce (PD) em videira estimula a expressão do
regulador de virulência xrvA quando crescida em xilema de um cultivar de videira
susceptível, mas não quando crescida em xilema de citros, o qual não é seu
hospedeiro (SHI et al., 2010). Também recentemente foi mostrado que em
Xanthomonas oryzae pv. oryzae (Xoo), bactéria que causa a murchidão do arroz,
mutação no gene xrvA leva a uma redução significativa na virulência na planta
hospedeira e um retardamento na resposta HR em planta não hospedeira (FENG
et al., 2009)
avaliar a expressão gênica global, por meio da técnica de microarranjo de DNA, do
mutante do gene xrvA de Xac (8B7 - XAC1495), que apresenta fenótipo
parcialmente avirulento, comparada com a da Xac 306 selvagem, após 72 horas de multiplicação in planta, afim de elucidar a diferenciação na expressão de genes
relacionados à patogenicidade e virulência no patossistema Xac-citros.
MATERIAL E MÉTODOS
Linhagens bacterianas e condições de cultivo
As linhagens de Xac 306 (linhagem que teve o genoma sequenciado por DA SILVA et al., 2002) e do mutante 8B7 foram mantidas no laboratório em meio
TSA (10 g/L triptona, 10g/L de sacarose, 1 g/L de glutamato monossódio) líquido suplementado com canamicina (100 mg/mL) para o mutante e sem canamicina para Xac selvagem e acrescido de glicerol para concentração final de 25% e armazenadas em ultrafreezer à -80 °C. Para a multiplicação das bactérias a partir
do estoque em glicerol utilizou-se o meio de cultura sólido TSA (10 g/L triptona, 10g/L de sacarose, 1 g/L de glutamato monossódio, e 15 g/L de Ágar) e incubação a 28 °C por 72 h.
Inoculação da bactéria em planta hospedeira
Após as 72 horas de cultivo em meio de cultura sólido TSA, as células foram coletadas e a concentração celular foi padronizada com água bi-destilada gelada, para D.O600nm = 0,3 (108 cfu/mL), em tubos Falcon de 50 mL contendo
água bidestilada gelada. Em seguida, essa suspensão de células foi infiltrada, com o auxílio de uma seringa, em todo o limbo de folhas jovens de laranjeira Pêra. Foram utilizadas um total de 8 plantas, sendo 4 para o mutante e 4 para a Xac selvagem.