UNIVERSIDADE FEDERAL DE SÃO CARLOS CENTRO DE CIÊNCIAS BIOLÓGICAS E DA SAÚDE
PROGRAMA DE PÓS-GRADUAÇÃO EM GENÉTICA E EVOLUÇÃO
“ESTRUTURA GENÉTICA DAS POPULAÇÕES DE ABELHAS
AFRICANIZADAS (Apis mellifera L.) DO BRASIL E URUGUAI DETERMINADA POR MEIO DE POLIMORFISMOS DO DNA MITOCONDRIAL”
THAÍS COLLET
UNIVERSIDADE FEDERAL DE SÃO CARLOS CENTRO DE CIÊNCIAS BIOLÓGICAS E DA SAÚDE
PROGRAMA DE PÓS-GRADUAÇÃO EM GENÉTICA E EVOLUÇÃO
“ESTRUTURA GENÉTICA DAS POPULAÇÕES DE ABELHAS
AFRICANIZADAS (Apis mellifera L.) DO BRASIL E URUGUAI DETERMINADA POR MEIO DE POLIMORFISMOS DO DNA MITOCONDRIAL”
THAÍS COLLET
Dissertação de Mestrado apresentada ao Programa de Pós-Graduação em Genética e Evolução do Centro de Ciências Biológicas e da Saúde da Universidade Federal de São Carlos, como parte dos requisitos para a obtenção do título de Mestre em Genética e Evolução, área de concentração: Genética e Evolução.
Ficha catalográfica elaborada pelo DePT da Biblioteca Comunitária da UFSCar
C698eg
Collet, Thaís.
Estrutura genética das populações de abelhas
africanizadas (Apis mellifera L.) do Brasil determinada por meio de polimorfismos do DNA mitocondrial / Thaís Collet. -- São Carlos : UFSCar, 2004.
66 p.
Dissertação (Mestrado) -- Universidade Federal de São Carlos, 2004.
1. Genética de populações. 2. Abelha - africanizada. 3. Apis mellifera. 4. DNA mitocondrial. I. Título.
Orientador
“Mas tu ó servo meu, não temas porque eu sou contigo; não te assombres
porque eu sou o teu Deus; te tomo pela tua mão direita, te fortaleço, te ajudo
e te sustento com a minha destra fiel”.
Isaías 41: 10 e 13
Todo o meu agradecimento Àquele que nos tem fortalecido.
Agradecimentos
Muitas pessoas tiveram participações fundamentais na realização deste trabalho, estando direta ou indiretamente envolvidas nele, ou realizando os papéis que fazem toda a diferença na minha vida: o de família e o de amigos.
Portanto, deixo aqui os meus agradecimentos a algumas dessas pessoas:
Aos meus pais, Nelita e Martim, e aos meus irmãos André, Ulisses e Rodrigo, que são a melhor base e referência da minha formação pessoal, por me proporcionarem todas as oportunidades para a formação profissional.
Ao professor Dr. Marco Antonio Del Lama, pela presença sempre constante no desenvolvimento deste trabalho, demonstrando orientação com segurança, além dos melhores exemplos de princípios que um aluno em formação pode obter de um orientador.
Aos amigos do Laboratório de Genética Evolutiva de Himenópteros: Isabel, Daniele, Mariana, Margarita e Otávio, por fazerem do laboratório um ambiente tão agradável e por se revelarem não apenas colegas de trabalho, mas sim amigos.
Ao Rogério Oliveira Souza, que de forma amiga me ensinou todas as técnicas desde o começo do trabalho, além de ajudar na análise e discussão dos resultados, e ao Carlos Prada Quiroga, pelo grande companheirismo, ajuda e incentivos durante a realização do trabalho.
À professora Dra. Maria Cristina Arias, por me receber em seu laboratório no Instituto de Biociências da USP e pelas valiosas discussões a respeito dos resultados. Agradeço também a todo o pessoal do seu laboratório por terem me recebido tão bem durante o período que permaneci junto deles.
Aos professores do Programa de Pós-Graduação em Genética e Evolução, com alguns dos quais, por meio das disciplinas cursadas, muito aprendi.
Aos funcionários e amigos, tanto do Departamento de Genética e Evolução quanto da Pós-Graduação, pela convivência tão saudável.
Aos apicultores ou pesquisadores do Brasil e demais países, pela maneira cordial que nos forneceram as amostras de abelhas analisadas neste trabalho.
Às professoras Dras. Maria Claudia C. R. Takasusuki e Sandra A. O. Collet pela grande amizade e incentivos para que eu realizasse o Mestrado.
A todas as pessoas que conheci nesse período em São Carlos, principalmente algumas cujos nomes eu não poderia deixar de lembrar: Priscila, Elisângela, Milena, Marcilene, Patrícia, Lídia, Franciéli, Tatiana, Joel, Sônia, Nelson, Martha, Rogério, Larissa, Leu, Marcos, Liana, Júnior e Fernando. Estas são algumas das pessoas as quais considero muito valiosas, estando junto de mim todos os dias ou em cidades distantes e até mesmo em outro país, todas são muito importantes na minha vida.
À Luciana Oliveira Souza e à Noêmia Mie Orii pela forma acolhedora com que me receberam em São Paulo.
i
Índice
LISTA DE FIGURAS ... iii
LISTA DE TABELAS ... iv
RESUMO GERAL ... v
ABSTRACT... vii
1. Introdução Geral ... 1
1.1 Origem e Evolução de Apis mellifera ... 1
1.2 O processo de africanização... 3
1.3 Estudos populacionais realizados com as subespécies de Apis mellifera ... 7
1.3.1 Morfometria ... 7
1.3.2 Alozimas ... 8
1.3.3 Polimorfismos do DNA nuclear ... 9
1.3.4 Padrões do DNA mitocondrial ... 11
2. Justificativa e Objetivos ... 15
3. Referências Bibliográficas ... 17
4. Padrões de PCR-RFLP da região 16S do DNA mitocondrial diferenciam ramos evolutivos de Apis mellifera... 23
4.1. Resumo ... 24
4.2. Introdução ... 25
4.3. Material e Métodos ... 28
4.3.1 Amostras e extração de DNA ... 28
4.3.2. Amplificação por PCR e digestão com endonucleases ... 28
ii
4.3.3. Clonagem e seqüenciamento do DNA ... 30
4.4. Resultados ... 31
4.5. Discussão ... 37
4.6. Referências Bibliográficas ... 40
5. Estrutura genética das populações de abelhas africanizadas (Apis mellifera L.) do Brasil determinada por meio do polimorfismo da região tRNAleu_COII do DNA mitocondrial ... 43
5.1. Resumo ... 44
5.2. Introdução ... 45
5.3. Material e Métodos ... 47
5.3.1. Amostras e extração de DNA ... 47
5.3.2. Amplificação por PCR e digestão com endonuclease ... 47
5.3.3. Clonagem e seqüenciamento do DNA ... 47
5.4. Resultados ... 49
5.5. Discussão ... 57
5.6. Referências Bibliográficas ... 62
iii
LISTA DE FIGURAS
Padrões de PCR-RFLP da região 16S do DNA mitocondrial diferenciam ramos evolutivos de Apis mellifera
FIGURA 1. Padrões de amplificação e restrição das subespécies analisadas para a região 16S ... 32
FIGURA 2. Sítios de variação do fragmento 16S ... 33
FIGURA 3. Padrões da digestão dupla comEco RI e Vsp I ... 36
Estrutura genética das populações de abelhas africanizadas (Apis mellifera
L.) do Brasil determinada por meio do polimorfismo da região tRNAleu_COII do DNA mitocondrial
FIGURA 1. Mitótipos observados a partir da digestão com Dra I para a região intergênica tRNAleu_COII ... 52
FIGURA 2. Mapas de restrição e comprimento dos fragmentos em pares
de base ... 53
FIGURA 3. Seqüências P e Q da região tRNAleu-COII ... 54
iv
LISTA DE TABELAS
Padrões de PCR-RFLP da região 16S do DNA mitocondrial diferenciam ramos evolutivos de Apis mellifera
TABELA 1. Número de colônias de Apis mellifera e seus respectivos países de coleta ... 29
TABELA 2. Padrões discordantes observados nos diferentes marcadores
utilizados na identificação das subespécies ... 34
Estrutura genética das populações de abelhas africanizadas (Apis mellifera
L.) do Brasil determinada por meio do polimorfismo da região tRNAleu_COII do DNA mitocondrial
v
RESUMO GERAL
Os ramos evolutivos dentro dos quais as subespécies de Apis mellifera são classificadas estão originalmente distribuídos nos continentes africano (ramos A e Y), europeu (ramos M e C) e asiático (ramo O). Essa distribuição vem sendo progressivamente alterada devido às introduções de subespécies em várias outras regiões do mundo, ocasionadas principalmente por atividades apícolas. Dessa forma, a genética de populações vem ganhando um amplo campo de estudos relacionados às mudanças na composição das populações já estabelecidas e das recém introduzidas. A introdução das abelhas africanas no continente americano, que resultou no processo conhecido como africanização, se tornou um dos eventos mais estudados, dentre aqueles relacionados às introduções de Apis mellifera.
Uma das primeiras abordagens nos estudos que envolvem introduções de subespécies é a caracterização das diferentes linhagens que contribuem para a formação das populações. Nesse sentido, são descritos neste trabalho os diferentes padrões de restrição obtidos a partir de um fragmento da região 16S do DNA mitocondrial que, em relação às metodologias utilizadas até então, permitem uma melhor diferenciação das subespécies pertencentes aos ramos evolutivos A, M e C.
vi
freqüentes foram os africanos A1 e A4, provavelmente introduzidos no Brasil via
vii
ABSTRACT
The Apis mellifera subspecies are classified into evolutionary branches originally distributed in the African (branches A and Y), European (branches C and M) and Asian (branch O) continents. This distribution has been changing due to the subspecies introductions to other countries, mainly occurred by beekeeping activities. Therefore, population genetics studies on the already established and newly introduced populations have increased. The introduction of the African honeybee in American continent, known as Africanization, became one of the most studied events related to Apis mellifera introductions.
The characterization of different lineages that contribute to hybrid populations is the first approach of the studies related to subspecies introductions. In this way, we describe the restriction patterns obtained from a 16S region fragment of the mitochondrial DNA. These patterns enables the differentiation among races from the three evolutionary branches using only one region of the mitochondrial genome, without the requirement of amplification of other regions.
The determination of the racial composition of the hybrid product of
Introdução Geral 1
1. Introdução Geral
1.1. Origem e Evolução de Apis mellifera
Evidências fósseis são escassas, mas as abelhas provavelmente evoluíram
juntamente às fanerógamas no período Cretáceo (cerca de 146 a 74 milhões de
anos atrás). Alguns autores, como Gauld e Bolton (1996), sugerem que regiões de
clima tropical, como a África, seriam os possíveis locais de origem do gênero Apis.
Porém, os primeiros registros fósseis do gênero foram descobertos na Alemanha e
datam do Eoceno, ou seja, por volta de 50 milhões de anos atrás. De acordo com
Milner (1996), esse fato pode ser explicado por registros que apontam um clima
tropical para a Europa naquela época, e conforme o clima se tornava frio, as
abelhas, que ainda não teriam desenvolvido o controle da temperatura do ninho,
migraram para regiões tropicais.
Dois atributos foram essenciais para a evolução e biologia das abelhas: o
comportamento de agrupar-se e a habilidade de controlar a temperatura do ninho.
Estas características permitiram a regulação da temperatura interna da colônia
independentemente da temperatura externa. Dessa forma, o gênero Apis foi capaz
de colonizar uma ampla variedade de ambientes, desde os tropicais até os
temperados. A subfamília Meliponinae, por exemplo, que não apresenta essa
capacidade de regulação da temperatura do ninho, está restrita às regiões
tropicais (Milner, 1996).
A grande capacidade de Apis mellifera colonizar diferentes ambientes, da
mesma forma que as demais espécies do gênero Apis, levou essa abelha a
enfrentar muitas condições ecológicas diferentes. Este fato, associado ao
isolamento geográfico de algumas populações, permitiu a evolução de numerosas
subespécies adaptadas aos seus ambientes específicos.
Em 1978, Ruttner et al. agruparam as diferentes subespécies de A.
mellifera por meio da análise multivariada de caracteres morfométricos e estes
Introdução Geral 2
Atualmente são reconhecidos cinco ramos evolutivos que agrupam as 26
subespécies de A. mellifera descritas. A seguir são apresentados os ramos
juntamente com a distribuição original de cada um deles.
1) Ramo A: Característico das subespécies africanas A. m. adansonii, A. m.
capensis, A. m. intermissa, A. m. lamarckii, A. m. litorea, A. m. major, A. m.
monticola, A. m. sahariensis, A. m. scutellata, A. m. siciliana e A. m. unicolor.
2) Ramo M: Característico das seguintes subespécies localizadas na Europa
Ocidental: A. m. iberiensis e A. m. mellifera.
3) Ramo C: Compreende as subespécies A. m. carnica, A. m. cecropia, A. m.
macedonica, A. m. ligustica e A. m. sicula, que ocupam o leste europeu e norte do
Mediterrâneo.
4) Ramo O: Agrupa as subespécies do Oriente Médio A. m. adami, A. m.
anatoliaca, A. m. armeniaca, A. m. caucasica, A. m. cypria, A. m. meda e A. m.
syriaca.
5) Ramo Y: Este ramo compreende apenas a subespécie A. m. yemenitica,
localizada na região africana da Etiópia.
A distribuição original dos ramos evolutivos de Apis mellifera tem sido
progressivamente alterada devido às sucessivas introduções realizadas por
apicultores, de forma que, atualmente, as abelhas pertencentes às diferentes
linhagens podem ser encontradas em várias partes do mundo. De acordo com
Schneider et al. (2004), o transporte em larga escala de populações de A. mellifera
deu início a uma “mistura” de linhagens anteriormente distribuídas de forma
alopátrica. Dessa forma, vêm se tornando comum ao longo dos anos as
investigações que visam diferenciar as linhagens de abelhas envolvidas nas
introduções, a fim de compreender as mudanças na composição genética das
populações residentes e introduzidas, como resultado do transporte para
diferentes áreas.
Estudos dessa natureza foram realizados com as populações da ilha de
Introdução Geral 3
população relativamente uniforme, mas, de acordo com as freqüências de três
alelos de um loco MDH, recentes importações da subespécie ligustica têm
aumentado a variabilidade genética na região (Cornuet, 1979).
Oldroyd et al. (1995), estudaram processosde hibridização entre mellifera e
ligustica na Tasmânia (Austrália), introduzidas respectivamente em 1831 e 1884, e
observaram que o fluxo gênico entre as subespécies ocorria na costa, mas era
restrito nas regiões montanhosas mais frias, onde populações ferais de mellifera
eram predominantes, provavelmente devido ao fitness reduzido dos híbridos
mellifera/ligustica nas regiões frias.
Nas ilhas Baleares, foram observados haplótipos pertencentes à linhagem
M, resultado de importações de rainhas da Península Ibérica, o que demonstra
substituições de rainhas nativas (De la Rúa et al., 2001).
No entanto, o impacto mais marcante resultante da introdução de diferentes
subespécies de Apis mellifera em uma área ocorreu nas Américas e será discutido
no tópico a seguir.
1.2. O Processo de Africanização
O processo de expansão das abelhas africanas Apis mellifera scutellata
pelo continente americano é denominado de Africanização e teve início no Brasil
em 1956. De acordo com Kerr (1967), estas abelhas foram trazidas da África
(região de Tanganica e África do Sul) ao Brasil pelo próprio autor com o objetivo
de realizar o melhoramento e posterior distribuição de rainhas selecionadas aos
apicultores. O objetivo era aumentar a produção nacional de mel, uma vez que
dados da literatura internacional apontavam a subespécie A. m. scutellata como
grande produtora (Gonçalves, 1998).
Em 1957 ocorreu o escape acidental de 26 rainhas que se encontravam em
quarentena em apiário próximo a Rio Claro (SP), iniciando as enxameações
dessas rainhas e o cruzamento na natureza com as subespécies européias
Introdução Geral 4
Na América do Sul, as primeiras introduções ocorreram por meio dos
colonizadores espanhóis e portugueses, provavelmente com A. m. mellifera e A.
m. iberica. No Brasil, por volta do século XIX, imigrantes alemães e italianos
começaram a introduzir A. m. mellifera e A. m. ligustica respectivamente. Mais
recentemente, atividades apícolas comerciais em toda a América têm importado A.
m. ligustica.
Portanto, a abelha resultante do cruzamento entre as subespécies
européias já existentes com a africana é um polihíbrido que, de acordo com
Gonçalves (1998), por volta de 1974 recebeu a denominação “africanizada” devido
à dominância das características das abelhas africanas sobre as demais
européias.
Os enxames africanizados se expandiram pela América a uma velocidade
de cerca de 480 km/ano (Taylor, 1985), de forma que atualmente são encontrados
em grande parte do continente, incluindo várias áreas dos EUA (Kunzmann et al.,
2002). A colonização das Américas se deu, inclusive, com a substituição dos
enxames europeus preexistentes pela abelha africanizada. Alguns fatores, muitas
vezes combinados, são responsáveis por permitir essa expansão, bem como a
preservação das características africanas, seja nas colônias ferais ou nas
mantidas em apiários. A seguir, são descritos alguns desses fatores.
- Taxa de crescimento da colônia e enxameagem: As colônias africanas apresentam uma taxa de crescimento mais acelerada quando comparadas
com as européias, que se deve à maior habilidade na coleta de pólen e sua
rápida conversão às crias (Page et al., 2000). Com isso, a densidade das
colônias africanas é rapidamente aumentada, resultando na produção de um
enxame com maior capacidade de dispersão, que abandona as colônias e dá
início à enxameagem.
- Invasão das colônias européias pelas africanas: Neste caso, enxames africanos invadem colônias européias, substituem as rainhas e causam a
Introdução Geral 5
ocorre permanece pouco entendido, porém, de acordo com Danka et al.
(1992), fatores relacionados a feromônios dos enxames invasores, ausência
de rainha nas colônias invadidas ou ainda uma colônia debilitada, podem estar
envolvidos.
- Linhagens africanas se sobrepõem durante a substituição de rainhas: Quando abelhas africanas invadem uma nova área, as rainhas se acasalam
com zangões africanos e europeus, resultando numa colônia com operárias de
origem africana e européia. Pelo fato das rainhas que emergem primeiro
eliminarem aquelas que ainda permanecem nas células, as de origem africana
possuem uma vantagem competitiva, já que estas se desenvolvem mais
rápido e emergem antes das rainhas européias, possuindo mais
oportunidades de eliminá-las (DeGrandi-Hoffman et al., 1998).
- Vantagens adaptativas da abelha africana em ambientes neotropicais: Rinderer (1988) destaca que as abelhas africanas possuem vantagens em
relação às européias, tanto com relação à colonização quanto na capacidade
de sobreviver nos ambientes tropicais e subtropicais do continente americano.
O comportamento higiênico mais acentuado diante de doenças ou parasitas,
como o ácaro Varroa jacobsoni (responsável por atacar as crias), torna as
africanizadas mais tolerantes que a européias. Outras características, como
um forrageamento mais eficiente nas áreas tropicais, maior capacidade
defensiva diante de inimigos naturais ou a flexibilidade na escolha dos locais
de nidificação, contribuem para o estabelecimento bem sucedido desta abelha
na América.
Com relação aos impactos causados pela expansão das abelhas
africanizadas pelo continente, temia-se inicialmente que a entrada dessas abelhas
em novas áreas seria tanto perigosa para a população devido à sua
agressividade, quanto desvantajosa para os apicultores e a indústria de
polinização. No começo da década de 80, o Departamento de Agricultura dos EUA
Introdução Geral 6
anual de 19 milhões de dólares em atividades agrícolas que dependiam da
polinização por abelhas, além de um prejuízo ainda maior para a apicultura
(McDowell, 1984).
Porém, é possível que tais impactos sejam menores do que se imaginava
inicialmente (Schneider et al., 2004) pois a baixa taxa de dispersão das abelhas
africanizadas nos climas temperados americanos, juntamente com políticas de
controle por parte do governo, podem ter amenizado essas estimativas. Na
América do Sul, essas abelhas se mostraram superiores às européias na
polinização de certas culturas, e, por apresentarem maior resistência a doenças,
além de reduzida susceptibilidade a alguns pesticidas, têm sido incorporadas em
práticas agrícolas.
No Brasil, desde a década de 60, grupos de pesquisa vêm se
estabelecendo em programas de cruzamentos entre abelhas africanizadas e
européias com o objetivo de diluir os genes africanos, já que essas abelhas são
caracterizadas por um acentuado comportamento defensivo. O objetivo inicial de
introduzir as abelhas africanas no Brasil, ou seja, o incremento na produção
nacional de mel, foi alcançado, conforme constatado por Kerr (1967) com dados
que apontavam as abelhas africanas produzindo duas vezes mais que as italianas
e quatro vezes mais que as alemãs. Em 2001, o país produziu em torno de 22 mil
toneladas de mel, destacando-se em sexto lugar na produção mundial.
(www.cnptia.embrapa.br/sistemasdeprodução). O uso das abelhas africanizadas
na apicultura comercial vem sendo cada vez mais freqüente, havendo inclusive
apicultores no Brasil e outros países americanos que preferem estas abelhas
devido à sua alta produtividade (Gonçalves, 1998).
Apis mellifera scutellata é um excelente modelo animal para estudos de
fatores que influenciam o estabelecimento bem sucedido de espécies em novos
ambientes (Schneider et al., 2004). A colonização da maior parte do hemisfério
ocidental em menos de 50 anos por uma única subespécie é uma das mais
Introdução Geral 7
Dessa forma, a expansão da abelha africanizada por novos ambientes e
seu cruzamento com as demais subespécies têm possibilitado a realização de
vários estudos que visam diferenciar entre as subespécies, investigar o grau de
hibridização entre as abelhas européias existentes e as africanas introduzidas
posteriormente, bem como os mecanismos envolvidos no estabelecimento dessas
novas populações.
1.3. Estudos populacionais realizados com as subespécies de Apis mellifera
Vários trabalhos têm relatado estudos populacionais realizados com as
subespécies de Apis mellifera em praticamente todos os continentes. Em tais
análises, destaca-se a utilização de morfometria, alozimas, polimorfismos do DNA
nuclear e os padrões do DNA mitocondrial. A seguir, serão relatados alguns
trabalhos que se utilizaram desses marcadores para analisar populações de A.
mellifera e o seu emprego nas questões que envolvem as abelhas africanizadas
das Américas.
1.3.1. Morfometria
As subespécies de abelhas têm sido diferenciadas por meio de análises
morfométricas desde o início do século XX (Ruttner, 1988). Alpatov (1929, 1948)
introduziu medidas biométricas de partes do corpo das abelhas, como cor e
venação das asas, dando início às primeiras classificações.
Em 1978, Ruttner et al. utilizaram dados de morfometria para sugerir que A.
mellifera evoluiu em três principais ramos ou linhagens evolutivas (M, C e A),
classificando as subespécies descritas até então dentro desses ramos.
Atualmente, aproximadamente 40 caracteres morfométricos das abelhas podem
ser utilizados em análises multivariadas. A utilização desses caracteres é possível
devido aos padrões de variação morfológica encontrados em A. mellifera,
Introdução Geral 8
De acordo com Diniz-Filho (1994), os valores médios da asa anterior, por
exemplo, podem ser utilizados na diferenciação dos grupos de A. mellifera e na
estimativa de variação entre populações, pois esta característica apresenta
elevada herdabilidade e está altamente relacionada a gradientes morfológicos.
Abelhas africanizadas da América do Sul foram estudadas por Buco et al.
(1987) utilizando 25 caracteres morfológicos. Os autores constataram que essas
abelhas apresentavam tanto origem da linhagem parental européia quanto da
africana.
Colônias africanizadas do México foram analisadas por meio de três
métodos morfométricos (Guzman-Novoa et al., 1994). De acordo com os autores,
os métodos discriminam corretamente as amostras africanas e européias,
indicando que menos de 45% dos híbridos eram africanizados.
Lobo (1995) utilizou dados de morfometria das asas em seus estudos com
abelhas africanizadas da Costa Rica e observou uma origem predominantemente
africana em todas as localidades. O autor comparou a população africanizada da
Costa Rica com grupos de referência, como abelhas européias, africanizadas do
Brasil e européias existentes na Costa Rica antes do processo de africanização. O
comprimento e a largura das asas das abelhas costarriquenhas foram, em média,
menores quando comparadas com as européias, sendo mais similares ao padrão
africanizado utilizado como referência.
Dados obtidos com medidas da asa anterior e posterior de populações de
abelhas africanizadas do Brasil demonstraram uma grande semelhança dessas
abelhas com as africanas. Esta semelhança aumenta nas abelhas que ocupam as
regiões mais próximas ao Equador (Rotta, 1999).
1.3.2. Alozimas
Numerosos estudos têm sido realizados empregando-se alozimas para
estimar níveis de variabilidade genética em populações, além de ajudar a resolver
Introdução Geral 9
mellifera, alguns locos polimórficos, como MDH1, PGM, HK1, EST-3 e ADH,
apresentam os maiores níveis de polimorfismo, sendo muito utilizados em estudos
de parentesco entre grupos de populações dessa espécie (Lobo, 1995).
Vários autores destacam os locos MDH1 e HK1 na diferenciação de
populações européias e africanizadas (Lobo et al., 1989; Del Lama et al., 1988,
1990, 2004; Rotta, 1999). Tal fato de deve às diferentes freqüências para três
alelos de MDH1 apresentadas pelas três principais subespécies envolvidas no
processo de africanização, A. m. scutellata, A. m. mellifera e A. m. ligustica. A. m.
mellifera exibe altas freqüências do alelo Mdh180 , enquanto as africanas possuem
o alelo Mdh1100 praticamente fixado e A. m. ligustica apresenta grandes
proporções de Mdh165 (Sheppard e Berlocher, 1984; Badino et al., 1984) . Com
relação HK1, Del Lama et al. (1988) demonstraram que o alelo Hk1100 está
presente nas abelhas européias e Hk187 é característico das abelhas africanas.
Resultados obtidos com amostras de abelhas africanizadas da América do
Sul e Central demonstram uma grande contribuição das abelhas africanas na
formação das populações africanizadas, com uma composição de genes europeus
cuja freqüência varia de acordo com a região geográfica (Lobo et al., 1989; Del
Lama et al. 1990; Lobo e Krieger, 1992; Rotta, 1999; Diniz et al., 2003). Os
trabalhos apontam a predominância de genes africanos e, com relação aos
europeus, a maioria da contribuição se deve aos pertencentes a Apis mellifera
mellifera.
1.3.3. Polimorfismos do DNA nuclear
Muitas seqüências do genoma nuclear constituem uma fonte rica de
marcadores genéticos devido ao alto grau de variabilidade que possuem. Dessa
forma, esses marcadores vêm proporcionando avanços nos estudos de genética
de populações, além de muitos genes nucleares serem utilizados para inferências
Introdução Geral 10
Além dos polimorfismos gerados pelos tamanhos dos fragmentos de
restrição (RFLP) do DNA nuclear, os microsatélites também são considerados
efetivos em avaliações de variabilidade genética. Estes últimos marcadores
constituem seqüências curtas de nucleotídeos, repetidas em tandem e bastante
variáveis (Parker et al., 1998). Os marcadores nucleares constituem bons
indicadores da composição genética das abelhas e, de acordo com McMichael e
Hall (1996), comparando-se com a variabilidade morfométrica e bioquímica
(alozimas), são mais abundantes e não requerem expressão gênica para sua
detecção.
Estudos empregando RFLPs e microsatélites têm sido desenvolvidos no
sentido de elucidar a composição genética das populações de abelhas
africanizadas das américas. Contribuindo para isso, marcadores característicos de
abelhas européias e africanas vêm sendo identificados (Hall, 1986, 1990;
McMichael e Hall, 1996; Hall e McMichael, 2001).
Nas colônias ferais americanas africanizadas, os marcadores nucleares
característicos de abelhas do leste europeu (A. m. ligustica) foram observados
com baixas freqüências, indicando haver limitada introgressão paternal de
colônias européias manejadas nas populações ferais africanizadas (Hall, 1990).
Entretanto, como as freqüências de alelos africanos de outros locos foram
menores nas populações ferais americanas em relação a populações sul africanas
(Hall, 1992; 1998), provavelmente estaria havendo maiores níveis de hibridização
africana-européia do que o observado no estudo anterior.
Uma introgressão paternal a partir de abelhas do oeste europeu (A. m.
mellifera) estaria ocorrendo nas populações africanizadas, uma vez que Hall e
McMichael (2001) demonstraram que embora os alelos do leste europeu
estivessem praticamente ausentes em relação aos africanizados, havia de 26 a
31% de freqüência dos alelos característicos de A. m. mellifera.
De acordo com Suazo e Hall (2002) e Suazo et al. (2002), as freqüências
dos alelos africanos nas populações da América Central e do Sul aumenta do
Introdução Geral 11
aquelas estabelecidas a mais tempo. Estes autores também demonstraram
freqüências mais elevadas de alelos pertencentes a A. m. mellifera em relação aos
de A. m. ligustica.
Clarke et al. (2002), utilizando dados de microsatélites, descreveram que a
maioria das colônias mexicanas e venezuelanas por eles estudadas era híbrida
africana/A. m. mellifera.
1.3.4. Padrões do DNA mitocondrial
O DNA mitocondrial apresenta algumas características que tornam esta
molécula uma ferramenta bastante utilizada em estudos de filogenia, de relações
evolutivas entre espécies ou análises populacionais, como introduções de
populações e modos de dispersão.
Dentre as características que tornam o DNA mitocondrial um marcador
atrativo estão a herança uniparental (geralmente materna e sem recombinação),
as altas taxas evolutivas quando comparado ao genoma nuclear (provavelmente
devido a uma baixa eficiência do sistema de reparo na mitocôndria), a grande
quantidade, o que facilita seu isolamento, além de uma estrutura de organização
relativamente simples, constituindo-se de moléculas de fita dupla, compactas, com
um tamanho que em A. mellifera varia de 16 a 17 kb (Crozier e Crozier, 1993),
com poucas regiões intergênicas e sem íntrons.
Dados obtidos por meio da análise do DNA mitocondrial têm trazido
importantes esclarecimentos para os estudos com A. mellifera, contribuindo, por
exemplo, para o entendimento da evolução de suas subespécies. Algumas
endonucleases utilizadas em regiões específicas do DNA mitocondrial produzem
fragmentos capazes de distinguir entre as linhagens dessa espécie.
Crozier et al. (1991) relataram que a amplificação de um fragmento do gene
citocromo b e sua digestão com Bgl II era capaz de discriminar as abelhas da
Introdução Geral 12
Amplificando três fragmentos de diferentes regiões (subunidade ribossomal 16S,
citocromo oxidase I e região intergênica COI-COII) e digerindo-os com enzimas de
restrição específicas, Hall e Smith (1991) distinguiram diferentes padrões, os quais
foram atribuídos aos ramos A, M e C.
Em 1993, Garnery et al. descreveram uma metodologia que requer a
digestão do fragmento tRNA leu_ COII com a endonuclease Dra I. Esta técnica se
tornou amplamente utilizada em trabalhos sobre biogeografia e introgressões de
A. mellifera, seja na Europa (Garnery et al., 1995; Franck et al., 1998, 2000a;
Palmer et al., 2000), Ilhas Canárias (De la Rúa et al., 1998, 2001, 2002), África
(Moritz et al., 1994; Hepburn et al., 2000; Franck et al., 2001) ou Oriente Médio
(Franck et al., 2000b). A ampla utilização dessa metodologia se deve à
diferenciação de diversos haplótipos pertencentes aos ramos evolutivos, de
acordo com a distribuição geográfica das subespécies. Dessa forma, segundo
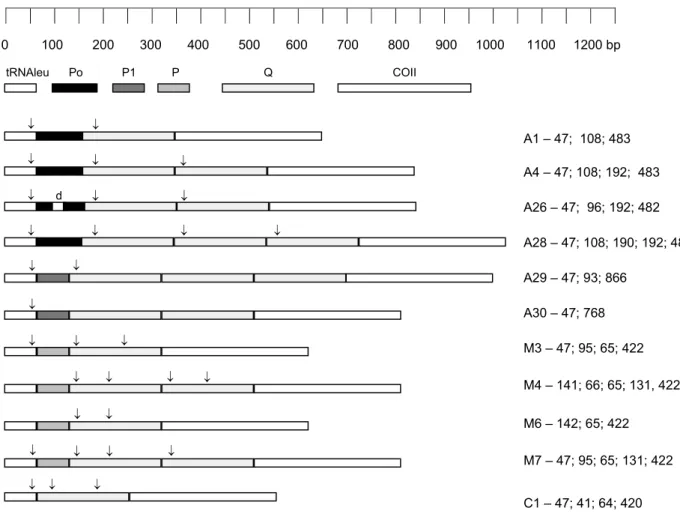
Garnery et al. (1993), cada ramo evolutivo possui uma variante de comprimento
característica para seqüências denominadas de P0 (ramo A), P (ramo M) ou
ausência de P (ramo C). A combinação das variantes P com até três repetições de
seqüências Q (P0Q, P0QQ, P0QQQ, PQ, PQQ, PQQQ e Q) é que caracteriza os
diferentes haplótipos dentro de cada ramo.
Os polimorfismos observados com os marcadores de DNA mitocondrial têm
se mostrado efetivos não somente nos estudos com populações africanas ou
européias, mas também nos trabalhos com as abelhas africanizadas do continente
americano.
De acordo com Smith et al. (1989), 97% das colônias de abelhas
africanizadas do Brasil, Venezuela, Honduras e México foram classificadas como
possuindo DNA mitocondrial africano. Hall e Muralidharan (1989) e Hall e Smith
(1991), sugerem, por meio de resultados obtidos com Bgl II (citb), uma origem via
A. m. scutellata para as abelhas africanizadas, tendo os enxames africanos
Introdução Geral 13
Sheppard et al. (1991), estudando abelhas coletadas no estado de São
Paulo, descreveram que o mtDNA possuía forte influência da subespécie
scutellata.
Na Costa Rica, Lobo (1995) estudou a composição genética de populações
de A.mellifera, analisando os sítios de restrição para Eco RI (16S) e Hinc II (COI).
O autor verificou que os padrões do DNA mitocondrial não indicavam origem
européia das abelhas, dados que contrastavam com os alelos característicos de
populações européias, observados com o marcador bioquímico MDH.
Colônias provenientes do México e Honduras apresentaram mtDNA
africano, porém com uma freqüência de 26 a 31% de alelos de DNA nuclear
europeu (Hall e McMichael, 2001). Segundo os autores, estes resultados sugerem
que as rainhas descendentes das africanas introduzidas no Brasil se acasalaram
com zangões europeus, permitindo a incorporação de marcadores neutros que
têm sido mantidos na população africanizada em expansão.
No Brasil, as abelhas africanizadas analisadas por Rotta (1999)
apresentaram padrões predominantemente africanos, de acordo com os
fragmentos originados pela endonuclease Bgl II para o loco citb, a despeito dos
resultados obtidos com as alozimas que indicaram uma freqüência de mais de
20% de alelos europeus nas abelhas brasileiras de certas áreas.
A utilização dos padrões gerados para o fragmento da região tRNA leu_ COII
tem sido observada em trabalhos realizados não somente com subespécies da
Europa, África ou Oriente Médio, como também têm contribuído com os estudos
que envolvem o processo de africanização na América.
Amostras de abelhas africanizadas da Costa Rica (Segura, 2000),
Venezuela (Clarke et al., 2001) e México (Clarke et al., 2001; Franck et al., 2001)
apresentaram uma predominância dos padrões A1 e A4 para a região tRNA leu_
COII. Tais padrões são característicos de populações de abelhas africanas.
Padrões característicos das subespécies européias ou mesmo outros padrões
Introdução Geral 14
Embora mitótipos africanos sejam predominantes nas populações
africanizadas, Diniz et al. (2003) relataram a presença de um padrão denominado
“português” quando analisaram colônias provenientes do Brasil e Uruguai. Tal
denominação se deve ao fato de haver evidências que apontam Portugal como
possível origem desse padrão. Da mesma forma, Sheppard et al. (1991) já haviam
descrito o mesmo padrão em amostras de abelhas da Argentina.
Amostras de colônias africanizadas da Argentina também apresentaram
resultados com a região tRNA leu_ COII que levaram Sheppard et al. (1999) a
concluir que mais de 25% do DNA mitocondrial africanizado daquele país possuía
origem diferente de A.m. scutellata.
Analisando amostras brasileiras e uruguaias, Ferreira (2002) sugeriu que
seria possível esclarecer a verdadeira origem das abelhas africanizadas por meio
da região tRNA leu_ COII. A autora se baseou nos polimorfismos gerados pela
enzima Dra I para inferir uma possível origem diferente de A.m. scutellata para as
Justificativa e Objetivos 15
2. Justificativa e Objetivos
De acordo com o exposto na introdução desse trabalho, a despeito dos
resultados obtidos a respeito do processo de africanização utilizando as diferentes
abordagens metodológicas, é possível verificar que alguns pontos ainda exigem
maior atenção:
1. Pelo fato de que os padrões Bgl II diferenciam somente entre a linhagem
européia e africana, uma possível origem mais diversa das colônias africanizadas
não pode ser detectada com este marcador. Portanto, os resultados obtidos por
meio de Bgl II não contribuíram para a caracterização de possíveis linhagens
diferentes na formação das populações africanizadas. Polimorfismos gerados por
outras regiões do DNA mitocondrial de abelhas africanizadas da América sugerem
uma herança mitocondrial mais diversa para essas populações. Portanto, a
análise dos padrões gerados por Dra I na região tRNA leu_ COII podem auxiliar no
esclarecimento da origem mitocondrial das abelhas africanizadas, devido ao
grande número de padrões relacionados às diferentes subespécies pertencentes
aos ramos A, M e C envolvidos no processo de africanização.
2. Resultados gerados pelos polimorfismos nucleares (morfometria,
alozimas e DNA nuclear) apontam uma significativa contribuição de genes
europeus na formação das populações africanizadas. Por outro lado, o DNA
mitocondrial dessas populações tem se mostrado composto quase que
exclusivamente da linhagem africana. Portanto, essa assimetria que
aparentemente existe entre os marcadores nucleares e mitocondriais merece ser
confirmada a partir dos resultados obtidos de um marcador mitocondrial mais
informativo.
Dessa forma, o trabalho entitulado “Estrutura genética das populações de
Justificativa e Objetivos 16
polimorfismo da região tRNAleu_COII do DNA mitocondrial” tem como objetivo
apresentar os padrões mitocondriais obtidos com a endonuclease Dra I na região
intergênica tRNAleu_COII de amostras do Brasil e Uruguai. Será discutida a relação
desses padrões com a origem européia ou africana das populações de Apis
mellifera das Américas e o quanto esses padrões são informativos para a
compreensão dos processos que levaram à formação dessas populações. A
contribuição desse marcador para o entendimento da provável assimetria entre os
resultados obtidos com marcadores nucleares e mitocondriais também é
abordada.
Durante o desenvolvimento do trabalho proposto, foi possível estabelecer o
uso de nova metodologia, tanto para os estudos das abelhas africanizadas das
Américas como para as demais introduções ocorridas mundialmente. Portanto, o
trabalho “Padrões de PCR-RFLP da região 16S do DNA mitocondrial diferenciam
ramos evolutivos de Apis mellifera” apresenta os resultados obtidos a partir desta
região mitocondrial, que permite identificação mais rápida, barata e segura das
subespécies pertencentes aos três ramos evolutivos de A. mellifera mais
Referências Bibliográficas 17
3. Referências Bibliográficas
ALFENAS, A.C. Eletroforese de isoenzimas e proteínas afins; fundamentos e
aplicações em plantas e microrganismos, Viçosa – MG. Ed.UFV, 1998, 574 p.
ALPATOV, V.V. Biometrical studies on variation and the races of honeybee. Q. Rev. Biol.,
v. 4, p. 1-58, 1929.
ALPATOV, V.V. The races of honeybees and their use in agriculture. Sredi Prirody, v. 4, p.
1-65, 1948.
ARIAS, M.C. & INFANTE-MALAQUIAS, M.E. RFLP: O emprego de enzimas de restrição
para detecção de polimorfismos no DNA. In: MATIOLI, S.R. Biologia molecular e
evolução. Ribeirão Preto: HOLOS, 2001. 202p.
BADINO, G., CELEBRANO, G., MANINO, A.. Population genetics of Italian honeybees
(Apis mellifera ligustica Spin) and its relationship with neighboring subspecies. Bull.
Mus. Reg. Sci. Nat. Torino, v. 2, p. 571-584, 1984.
BUCO, S.M., RINDERER, T.E., SYLVESTER, H.A., COLLINS, A.M., LANCASTER, V.A.
& CREWE, R.M. Morphometric differences between South American Africanized and
South African (Apis mellifera scutellata) honey bees. Apidologie, v. 18, p. 217-222,
1987.
CLARKE, K.E., RINDERER, T.E., FRANCK P., QUEZADA-EUÁN, J.G. & OLDROYD, B.P.
The Africanization of honey bees (Apis mellifera L.) of the Yucatán: a study of a
massive hybridization event across time. Evolution, v. 56, p. 1462-1474, 2002.
CORNUET, J.M. The MDH system in honeybees of Guadaloupe. J. Hered., v. 70, p.
223-224, 1979.
CROZIER, Y.C., KOULIANOS, S. & CROZIER, R.H. An improved test for Africanized
Referências Bibliográficas 18
CROZIER, R.H. & CROZIER, Y.C. The mitochondrial genome of the honeybee Apis
mellifera: complete sequence and genome organization. Genetics, v. 133, p. 97-117,
1993.
DANKA, R.G., HELLMICH, R.L. & RINDERER, T.E. Nest usurpation, supersedure and
colony failure contribute to Africanization of commercially managed European honey
bees in Venezuela. J. Apic. Res., v. 31, p. 119-123, 1992.
DEGRANDI-HOFFMAN, G., COLLINS, A., MARTIN, J.H., SCHMIDT, J.O. & SPANGLER,
H.G. Nest defense behavior in colonies from crosses between Africanized and
European honey bees (Apis mellifera L.). J. Insect Behav., v. 11, p. 37-45, 1998.
DE LA RÚA, P., GALIÁN, J., SERRANO, J. & MORITZ R.F.A.Molecular characterization
and population structure of the honeybees from the Balearic islands (Spain).
Apidologie, v. 32, p. 417-427, 2001.
DE LA RÚA, P., SERRANO, J. & GALIÁN, J. Biodiversity of Apis mellifera populations
from Tenerife (Canary Islands) and hybridization with East European races. Biodiv.
Conserv., v. 11, p. 59-67, 2002.
DEL LAMA, M.A., FIGUEIREDO, R.A., SOARES, A.E.E. & DEL LAMA, S.N. Hexoquinase
polymorphism in Apis mellifera and its use for Africanized honeybee identification.
Rev. Bras. Genet., v. 11, p. 287-297, 1988.
DEL LAMA, M.A., LOBO, J.A., SOARES, A.E.E. & DEL LAMA, S.N. Genetic differentiation
estimated by isozymic analysis of Africanized honeybee populations from Brazil and
from Central America. Apidologie, v. 21, p. 271-280, 1990.
DEL LAMA, M.A., SOUZA, R.O., DURAN, X.A. & SOARES A.E.E. Clinal variation and
selection on MDH allozymes in honeybees in Chile. Hereditas, v. 140, p. 149-153,
Referências Bibliográficas 19
DINIZ-FILHO, J.A.F. Variação geográfica de abelhas africanizadas (Apis mellifera L.)
no Brasil. 1994. 125p. Tese de Doutorado, Instituto de Biociências, UNESP, Rio
Claro, Brasil.
FERREIRA, K.M. O padrão africano do DNA mitocondrial das abelhas africanizadas
do Brasil tem origem diversa de Apis mellifera scutellata? 2002. 83p. Dissertação
de Mestrado – Programa de Pós-Graduação em Genética e Evolução. Universidade
Federal de São Carlos, SP, Brasil.
FRANCK, P., GARNERY, L., CELEBRANO, G., SOLIGNAC, M. & CORNUET, J.M. Hybrid
origins of honeybees from Italy (Apis mellifera ligustica) and Sicily (A. m. sicula). Mol.
Ecol., v. 9, p. 907-921, 2000a.
FRANCK, P., GARNERY, L., SOLIGNAC, M. & CORNUET, J.M. Molecular confirmation of
a fouth lineage in honey bees from the Near East. Apidologie, v. 31, p. 167-180,
2000b.
GARNEY, L., MOSSHINE, E.H. & CORNUET, J.M. Mitochondrial DNA variation in
Moroccan and Spanish honey bee populations. Mol. Ecol., v. 4, p. 465-471, 1995.
GAULD, I. & BOLTON, B. (eds.) The Hymenoptera. 2º Ed. Oxford University Press.
London, 1996.
GONÇALVES, L.S. Principais impactos biológicos causados pela africanização das
abelhas Apis mellifera e perspectivas da apicultura brasileira. Anais do III Encontro
sobre Abelhas. Ribeirão Preto, SP, Brasil, p. 31-36, 1998.
GUZMÁN-NOVOA, E., PAGE, R.E. & FONDRK, M.K. Morphometric techniques do not
detect intermediate and low levels of Africanization in honey bee (Hymenoptera:
Apidae) colonies. Ann. Entomol. Soc. Am., v. 85, p. 507-515, 1994.
HALL, H.G. DNA differences found between Africanized and European honeybees. Proc.
Referências Bibliográficas 20
HALL, H.G. Parental analysis of introgressive hybridization between African and European
honeybees using nuclear DNA RFLPs. Genetics, v. 125, p. 611-621, 1990.
HALL, H.G. & MCMICHAEL, M.A. Frequencies of restriction fragment-length
polymorphisms indicate that Neotropical honey bee populations have African and
West European origins. Ann. Entomol. Soc. Am., v. 94, p. 670-676, 2001.
HALL, H.G. Further characterization of nuclear DNA RFLP markers that distinguish African
and European honeybees. Arch. Insect Biochem. Physiol., v. 19, p. 163-175, 1992.
HALL, H.G. PCR amplification of a locus with RFLP alleles specific to African honey bees.
Biochem. Genet., v. 36, p. 351-361, 1998.
HEPBURN, H.R.; RADLOFF, S.E. & OGHIAKHE, S. Mountain honeybees of Africa.
Apidologie, v.31, p. 205-221, 2000.
KERR, W.E. The history of introduction of African bees to Brazil. South Afric. Bee J., v.
39, p. 3-5, 1967.
KUNZMANN, M.R., BUCHMANN, S.L., EDWARDS, J.F., THOENES, S.C. & ERICKSON
E.H. Africanized bees in North America, www.biology.usgs.gov/s+t/noframe, 2002.
LOBO, J.A. Morphometric, isozymic and mitochondrial variability of Africanized honey
bees in Costa Rica. Heredity, v. 75, p. 133-141, 1995.
LOBO, J.A. & KRIEGER, H. Maximum likelihood estimates of gene frequencies and racial
admixture in Apis mellifera L. (Africanized honey bees). Heredity, v. 68, p. 441-448,
1992.
McDOWELL, R. The Africanized honey bee in the Unites States: What will happen to the
US beekeeping industry? Agricultural Economic Report (US Department of
Referências Bibliográficas 21
McMICHAEL, M.A. & HALL, H.G. DNA RFLPs at a highly polymorphic locus distinguish
European and African subspecies of the honey bee, Apis mellifera L., and suggest
geographical origins of New World honey bees. Mol. Ecol., v. 5, p. 403-416, 1996.
MILNER, A. An introduction to understanding honeybees, their origins, evolution and
diversity. Bee improvement and bee breeders’ association.
www.angus.co.uk/bibba/bibborig, 1996.
OLDROYD, B.P., CORNUET, J.M., ROWE, D., RINDERER, T.E. & CROZIER R.H. Racial
admixture of Apis mellifera in Tasmania, Australia: similarities and differences with
natural hybrid zones in Europe. Heredity, v. 74, p. 315-325, 1995.
PAGE, R.E.JR, FONDRK, M.K., HUNT, G.J., GUZMÁN-NOVOA, E., HUMPHRIES, M.A.,
et al. Genetic dissection of honey bee (Apis mellifera) foraging behavior. J. Hered., v.
91, p. 474-479, 2000.
PALMER, M.R., SMITH, D.R. & KAFTANOGLU, O. Turkish honey bees: genetic variation
and evidence for a fourth lineage of Apis mellifera mtDNA. J. Hered., v. 91, p. 42-46,
2000.
PARKER, P.G., SNOW, A. A., SCHUG, M.D., BOOTON, G.C. & FUERST, P.A. What
molecules can tell us about populations: choosing and using a molecular marker.
Ecology, v. 79, p. 361-382, 1998.
ROTTA, I.T. Análise aloenzimática, morfométrica e dos padrões do DNA
mitocondrial das abelhas africanizadas do Brasil. 1999. 110p. Tese de Doutorado
– Programa de Pós-Graduação em Genética e Evolução. Universidade Federal de
São Carlos, SP, Brasil.
RUTTNER, F., TASSENCOURT, I. & LOUVEAUX, J. Biometrical-statistical analysis of the
geographic variability of Apis mellifera L. Apidologie, v. 9, p. 363-381, 1978.
RUTTNER, F. Biogeography and taxonomy of honeybee. New York-Berlin,
Referências Bibliográficas 22
SHEPPARD, W.S. & BERLOCHER, S.H. Enzyme polymorphism in Apis mellifera from
Norway. J. Apic. Res., v. 23, p. 64-69, 1984.
SHEPPARD, W.S., RINDERER, T.E., MAZOLLI, J.A., STELZER, J.A. & SHIMANUKI, H.
Gene flow between African-and European – derived honeybee populations in
Argentina. Nature, v. 349, p. 7882-7884, 1991.
SUAZO, A. & HALL, H.G. Nuclear DNA PCR-RFLPs that distinguish African and European
honey bee groups of subspecies. I. Comparison of long PCR and standard PCR to
screen for polymorphisms. Biochem. Genet., v. 40 p. 225-239, 2002.
SUAZO, A. McTIERMAN, R. & HALL, H.G. A locus with restriction fragment-length
polymorphisms characteristic of African and European honey bee (Hymenoptera:
Apidae) groups of subespecies. Ann. Entomol. Soc. Am., v. 95, p. 115-124, 2002.
TAYLOR, O.R. African bees: potential impact in the United States, Bull. Entomol. Soc.
4. Região 16S 23
4.
Padrões de PCR-RFLP da região 16S do DNA mitocondrial diferenciam
ramos evolutivos de Apis mellifera
Thaís Colleta, Maria Cristina Ariasb, Marco Antonio Del Lamaa
a Departamento de Genética e Evolução, Universidade Federal de São Carlos, São
Carlos, SP, Brasil
b Departamento de Biologia, Instituto de Biociências, Universidade de São Paulo, São
Paulo, SP, Brasil
Palavras-chave: Apis mellifera / linhagens evolutivas / diferenciação / DNA mitocondrial /
4.1. Resumo 16S 24
4.1. Resumo
Análises de padrões de restrição das regiões do DNA mitocondrial podem ser
utilizadas para classificar as subespécies de Apis mellifera nos ramos evolutivos
M, C ou A, propostos por Ruttner et al. (1978) com dados de morfometria. Neste
trabalho, são descritos diferentes padrões de restrição do mtDNA 16S para A. m.
mellifera, A. m. ligustica e A. m. scutellata. Embora distintos padrões tenham sido
obtidos com Eco RI, Alu I, Hinc II and Taq I, a diferenciação mais evidente entre os
três haplótipos foi obtida com Dra I e Vsp. Análises das seqüência nucleotídica do
fragmento do gene 16S revelou 10 sítios de substituição de base (1,35%) entre as
três subespécies, além de duas inserções em A. m. scutellata. A identificação
molecular de subespécies de origem européia tem se baseado no padrão de
restrição do gene mitocondrial CO I com a enzima Hinc II. Entretanto, foram
observados alguns resultados inconsistentes na utilização deste método,
4.2. Introdução 16S 25
4.2. Introdução
De acordo com evidências filogeográficas e morfométricas, Ruttner et al.
(1978) agruparam as subespécies de Apis mellifera em três linhagens ou ramos
evolutivos: M (Europa Ocidental), C (leste europeu) e A (África). A distribuição
original dos ramos tem sido progressivamente alterada devido às sucessivas
introduções realizadas por apicultores, de forma que, atualmente, as abelhas
pertencentes a essas linhagens podem ser encontradas em várias partes do
mundo. O transporte em larga escala de populações de A. mellifera deu início a
uma “mistura” de linhagens. A compreensão das mudanças na composição
genética das populações residentes, como resultado de introduções em diferentes
áreas geográficas, requer o uso de ferramentas que diferenciam as várias
linhagens de A. mellifera.
A composição genética de populações introduzidas tem sido investigada ao
longo dos últimos 25 anos. Estudos desta natureza foram realizados com as
populações da ilha de Guadalupe (Cornuet, 1979), da Argentina (Sheppard et al.,
1991), da Tasmânia, na Austrália (Oldroyd et al.,1995), nas ilhas Baleares (de la
Rua et al., 2001), Yucatan, no México (Clarke et al., 2001), sul do Brasil e Uruguai
(Diniz et al., 2003), Peru (Quezada-Euán et al., 2003) e Chile (Del Lama et al.,
2004), entre outros. O exemplo mais marcante resultante da introdução de
diferentes subespécies de Apis mellifera em uma área ocorreu na América do Sul.
O evento chave deste processo ocorreu na década de 50, quando a subespécie
africana A. m. scutellata foi introduzida no Brasil com o objetivo de melhorar a
produção de mel no país (Kerr, 1967). Após o escape de algumas rainhas
africanas em 1957 de um apiário onde se encontravam em quarentena, rainhas
africanas iniciaram um processo de intercruzamento com subespécies européias
residentes, iniciando assim o processo de africanização das abelhas
sul-americanas e, posteriormente, da América Central e do Norte. Atualmente,
abelhas Africanizadas são encontradas em grande parte do continente americano,
4.2. Introdução 16S 26
populações têm sido realizados para investigar o grau de hibridização entre
abelhas européias e africanas em diferentes regiões (Lobo et al., 1989; Rinderer et
al., 1991;Sheppard et al., 1991; Hall e McMichael, 2001).
A necessidade de caracterizar geneticamente as populações e identificar a
origem materna das colônias levou ao desenvolvimento de métodos capazes de
realizar tal caracterização. Dentre os métodos propostos, os marcadores
mitocondriais têm sido bastante utilizados, pois têm se mostrado efetivos na
diferenciação de populações envolvidas em processos de hibridização (Hall e
Smith, 1991; Garnery et al., 1993).
A metodologia mais utilizada atualmente na identificação da origem materna
das colônias que constituem as populações africanizadas consiste na amplificação
do gene para citocromo B (citB), seguida da restrição com Bgl II. Este teste
determina o padrão europeu (mellifera ou ligustica/carnica) caso um sítio de
restrição esteja presente ou o padrão Africano (scutellata) na ausência deste sítio
(Crozier et al., 1991). Na presença do padrão europeu, o protocolo segue com a
amplificação dos genes CO I e Ls rRNA e a digestão com Hinc II e Eco RI,
respectivamente (Hall e Smith, 1991). O sítio Hinc II para COI caracteriza o
haplótipo mellifera, característico das populações da Europa Ocidental, enquanto
o sítio Eco RI para Ls rRNA constitui o padrão das populações do leste europeu
(ligustica, carnica e caucasica).
Embora os atuais métodos permitam a distinção entre estas subespécies,
fica evidente as vantagens de novos protocolos que minimizem o tempo e os
custos para se obter tal diferenciação. Neste trabalho, nós descrevemos os
padrões de amplificação e restrição da região 16S do DNA mitocondrial em
amostras de Apis mellifera mellifera, Apis mellifera ligustica e abelhas
Africanizadas (Apis mellifera scutellata). Foi observada a ocorrência de ligeiras
diferenças na mobilidade eletroforética do fragmento amplificado em cada
subespécie. Estas diferenças podem ser mais facilmente visualizadas com o uso
de diferentes endonucleases, produzindo padrões de restrição distintos entre
4.2. Introdução 16S 27
identificação entre raças dos três ramos evolutivos propostos por Ruttner et al.
(1978) empregando uma única região do genoma mitocondrial, eliminando a
necessidade de amplificação de outras regiões. Análises comparativas entre o
método aqui proposto e os procedimentos anteriores (Crozier et al., 1991; Hall e
Smith, 1991) demonstraram que o novo método evita certas inconsistências que o
4.3. Material e Métodos 16S 28
4.3. Material e Métodos
4.3.1. Amostras e Extração de DNA
Foi amostrado um total de 290 colônias, sendo 39 de A. m. mellifera, 86 de
A. m. ligustica e 165 de abelhas africanizadas (AHB). Uma operária foi analisada
para cada colônia e as localidades de coleta estão descritas na Tabela I. As
abelhas, coletadas de colônias ferais ou apiários, foram mantidas a –200 C. O
DNA total foi extraído do tórax de cada indivíduo, de acordo com o método
fenol-clorofórmio descrito por Sheppard e McPheron (1991).
4.3.2. Amplificação por PCR e Digestão com Endonucleases
As regiões do DNA mitocondrial amplificadas e seus respectivos primers
(Crozier et al., 1991; Hall & Smith, 1991) foram: 485 pb do gene citocromo b (Cit b)
(F: 5’ – TAT GTA CTA CCA TGA GGA CAA ATA TC – 3’ e R: 5’ – ATT ACA CCT
CCT AAT TTA TTA GGA AT – 3’), 1044 pb do gene citocromo oxidase I (COI) (F:
5’- TTA AGA TCC CCA GGA TCA TG – 3’ e R: 5’- TGC AAA TAC TGC ACC TAT
TG – 3’) e parte do gene 16S compreendido entre os primers F: 5’ – TTT TGT
ACC TTT TGT ATC AGG GTT G – 3’ e R: 5’ – CTA TAG GGT CTT ATC GTC CC
– 3’. A PCR foi realizada num volume total de 25µl, contendo tampão de reação
10x, 250µM de cada dNTP, 2,5mM de MgCl2, 1µM dos primers F e R, 1µl de DNA,
1 U de Taq Polymerase (Promega) e 16µl de água esterilizada. A amplificação por
PCR para Cit b e 16S foi realizada da seguinte forma: 900 C por 1 min, 540 C por
45s e 620 C por 2 min. A reações para COI foram submetidas a uma denaturação
inicial de 3 min a 940 C seguida por 3 ciclos de 940 C por 1 min, 500 C por 2 min,
720 C por 3 min; 35 ciclos de 940 C por 1 min seguido por 2 min a 500 C, 1,5 min a
720 C e uma extensão final de 5 min a 720 C. Depois da amplificação, 2 µl dos
produtos de PCR foram submetidos a eletroforese em géis de poliacrilamida 8% e
4.3. Material e Métodos 16S 29
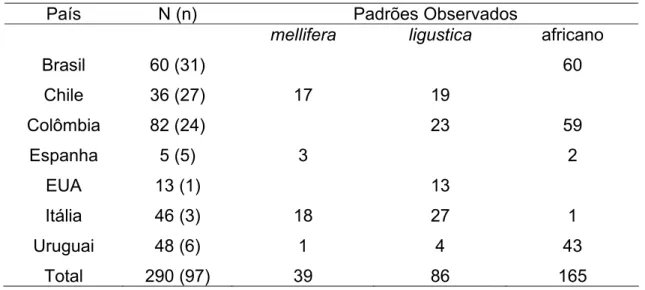
Tabela I. Número de colônias de Apis mellifera e seus respectivos países de coleta. As subespécies foram determinadas de acordo com os padrões do mtDNA.
N: número de colônias e n: número de localidades amostradas.
País N (n) Padrões Observados
mellifera ligustica africano
Brasil 60 (31) 60
Chile 36 (27) 17 19
Colômbia 82 (24) 23 59
Espanha 5 (5) 3 2
EUA 13 (1) 13
Itália 46 (3) 18 27 1
Uruguai 48 (6) 1 4 43
4.3. Material e Métodos 16S 30
restrição: Eco RI, Alu I, Hinc II, Taq I, Vsp I e Dra I para o fragmento 16S; Bgl II
para Cit b e Hinc IIpara COI. As reações de digestão foram mantidas a 370 C por
4h (exceto para Taq I que necessita de 600 C por 4h). Os fragmentos de restrição
foram separados em géis de poliacrilamida 10%.
Análises de restrição das regiões mitocondriais citocromo b (citb) e COI
foram realizadas para a caracterização molecular comparativa das amostras de
abelhas. Portanto, esta caracterização prévia se deu como um controle para o
padrão da linhagem africana (ausência do sítio Bgl II na região cit b) e para a
diferenciação dentro das subespécies européias (apenas A. m. mellifera possui o
sítio Hinc II na região COI).
Uma digestão dupla foi feita com as enzimas de restrição Eco RI e Vsp I
para as subespécies de A. mellifera a fim de se obter uma diferenciação mais
acentuada entre as três linhagens evolutivas.
4.3.3. Clonagem e Seqüenciamento do DNA
Fragmentos de PCR da região 16S, representando os três padrões de
restrição obtidos, foram clonados utilizando o kit de clonagem T-Easy (Promega) e
utilizados para transformar células competentes DH-5α de E. coli. Os clones
positivos foram selecionados e os vetores recombinantes recuperados e
seqüenciados de acordo com protocolos sugeridos pela Applied Biosystem
(www.appliedbiosystems.com). O seqüenciador automático ABI-3100 (Applied
Biosystems) foi utilizado para seqüenciar as amostras. Dois clones de cada
padrão foram seqüenciados a partir de ambas as direções. Os dados de
seqüência foram analisados por alinhamento múltiplo com o auxílio do programa
4.4. Resultados 16S 31
4.4. Resultados
As duas subespécies européias e a africanizada apresentaram diferentes
padrões de restrição para a região 16S. Os padrões de Eco RI, Alu I, Hinc II, Taq I,
Vsp Ie Dra I foram característicos para cada subespécie e para as africanizadas.
(Figura 1). Entre os 290 indivíduos analisados, não observou-se variação para os
padrões de amplificação e restrição dentro de cada subespécie.
O tamanho total do fragmento amplificado variou de acordo com a origem
do mtDNA, sendo de 740 pb em A. m. ligustica e A. m. mellifera e 742 em A. m.
scutellata.
Comparações por alinhamento múltiplo das seqüências de DNA revelaram
substituições nucleotídicas em 10 sítios (1,35%), sendo oito transições (1 A↔G e
7 C↔T) e duas transversões (1 A↔T e 1 C↔A) (Figura 2). Além disso, A. m.
scutellata apresentou duas inserções (T e A) nas posições 390 e 391,
respectivamente.
Algumas substituições de base ou inserções ocorreram em sítios de
restrição. O sítio Vsp I na posição 391 foi perdido em A. m. scutellata, devido às
duas inserções. Foi observado também a perda de um sítio Dra I na posição 477
de A. m. ligustica, ocasionado por uma substituição.
A origem racial identificada por Vsp I, Dra I e Eco RI (região 16S) foi
comparada com aquela que se utiliza dos padrões de Bgl II (Cit b) e Hinc II (COI).
Para a maioria das amostras, as duas abordagens moleculares estavam de
acordo. Entretanto, algumas exceções foram observadas com o sítio Hinc II no
gene COI. Por exemplo, duas colônias (que corresponde a 5,55%) do Chile foram
identificadas como mellifera de acordo com Hinc II (COI), mas apresentaram
padrão ligustica por Vsp I, Dra I e Eco RI(16S). Um resultado discordante também
foi obtido em 13% (ou seja, seis colônias) das amostras italianas. Além disso, uma
colônia da Itália (C5) foi tipada com o padrão africano de acordo com Vsp I, Dra Ie
Eco RI (16S) e Bgl II (Cit b), mas o padrão Hinc II (COI) foi característico de
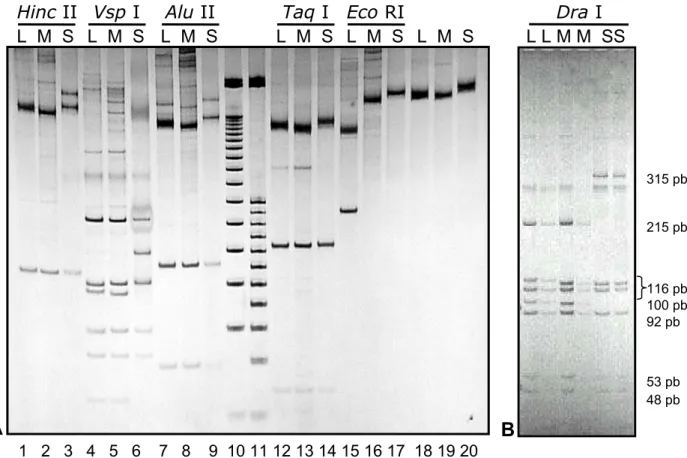
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20
A
B
L M S L M S L M S L M S L M S L M S
Hinc II Vsp I Alu II Taq I Eco RI
L L M M SS Dra I
315 pb
215 pb
116 pb 100 pb 92 pb
53 pb 48 pb 4.4. Resultados 16S 32
Figura 1. Padrões de amplificação e restrição do fragmento da região 16S do DNA
mitocondrial de Apis mellifera em géis de poliacrilamida 10% corados com prata. As
enzimas de restrição utilizadas e seus respectivos padrões estão indicados acima. L:
A. m. ligustica, M: A. m. mellifera e S: A. m. scutellata. A: Linhas 10 e 11correspondem aos marcadores de peso molecular de 50pb e 25 pb respectivamente. Linhas 18, 19 e
20 representam os fragmentos amplificados. B: Padrões de restrição obtidos com Dra I
4.4. Resultados 16S 33
13770 13811 13961 13990 14018 14025 14159 14182 14190 14352 14383 14405
A. m. ligustica* T A A C C C A C T C T C
A. m. ligustica . . . T G . . .
A. m. mellifera C T G A . T . T C T C T
A. m. scutellata C . G . T T . T C . . T
Figura 2. Sítios de variação do fragmento 16S. Os números correspondem às posições nucleotídicas de A. m. ligustica
(*) publicada por Crozier e Crozier (1993). As posições 14025 e 14159 representam diferenças entre as seqüências
descritas neste trabalho e as de Crozier e Crozier (1993). Os pontos indicam identidade nucleotídica. As abelhas
africanizadas (padrão scutellata) apresentaram duas inserções nas posições 14097 e 14098 (respectivamente 390 e
4.4. Resultados 16S 34
Tabela II. Padrões discordantes observados nos diferentes marcadores utilizados na identificação das subespécies.
País Colônia
Cit B COI 16S
Bgl II Hinc II Vsp I Dra I Eco RI
Itália C5 A M A A A
Itália M3 NA NM M M M
Itália MA NA M L L L
Itália MD2 NA NM M M M
Itália MD7 NA M L L L
Itália MD10 NA M L L L
Chile Coyhaique M L
Chile Olmoé M L L
A: padrão africano;
NA: não africano;
M: padrão mellifera;
NM: não mellifera;
4.4. Resultados 16S 35
A digestão dupla com as enzimas de restrição Eco RI e Vsp I para as
subespécies de A. mellifera permitiram uma diferenciação mais acentuada entre
L M S
161 pb
82 pb
Figura 3. Padrões de digestão dupla obtidos com Eco RI e Vsp I no
fragmento da região 16S do DNA mitocondrial de Apis mellifera. L: A.
m. ligustica; M: A. m. mellifera e S: A. m. scutellata.
243 pb
184 pb
148 pb 129 pb
91 pb
76 pb
53 pb