Pró-Reitoria de Pós-Graduação e Pesquisa
Stricto Sensu em Ciências Genômicas e
Biotecnologia
Trabalho de Conclusão de Curso
ANÁLISE DA EXPRESSÃO DE GENES
RELACIONADOS À TOTIPOTÊNCIA CELULAR EM
DIFERENTES ESTÁGIOS DO DESENVOLVIMENTO
EMBRIONÁRIO EM Bos taurus indicus
Autor: Anderson Napoleão Winckler Colatto
Orientador: Dr. Rodolfo Rumpf
ANDERSON NAPOLEÃO WINCKLER COLATTO
ANÁLISE DA EXPRESSÃO DE GENES RELACIONADOS À TOTIPOTÊNCIA CELULAR EM DIFERENTES ESTÁGIOS DO DESENVOLVIMENTO
EMBRIONÁRIO EM Bos taurus indicus.
Dissertação apresentada ao Programa de Pós-Graduação Strictu Senso em Ciências
Genômicas e Biotecnologia da Universidade Católica de Brasília, como requisito parcial para obtenção do Título de Mestre em Ciências Genômicas e Biotecnologia.
Orientador: Dr. Rodolfo Rumpf
7,5 cm
Ficha elaborada pela Biblioteca Pós-Graduação da UCB
12/07/2011
C697a Colatto, Anderson Napoleão Winckler
Análise da expressão de genes relacionados à totipotência celular em diferentes estágios do desenvolvimento embrionário em bos taurus indicus /
Anderson Napoleão Winckler Colatto – 2010. 66f. : il.; 30 cm
Dissertação (mestrado) – Universidade Católica de Brasília,2010. Orientação: Rodolfo Rumpf
1. Células-tronco. 2. Expressão gênica. 3. Clonagem molecular. 4. Bovino. 5. Biotecnologia I. Rumpf, Rodolfo, orient. II. Título.
Dissertação de autoria de Anderson Napoleão Winckler Colatto, intitulada “ANÁLISE DA EXPRESSÃO DE GENES RELACIONADOS À TOTIPOTÊNCIA CELULAR EM DIFERENTES
ESTÁGIOS DO DESENVOLVIMENTO EMBRIONÁRIO EM Bos taurus indicus.”, apresentada como
requisito parcial para obtenção do grau de Mestre em Ciências Genômicas e Biotecnologia da Universidade Católica de Brasília, em “data de aprovação”, defendida e aprovada pela banca examinadora abaixo assinada.
Prof. Dr. Rodolfo Rumpf Orientador
Empresa Brasileira de Pesquisa Agropecuária (EMBRAPA-CENARGEN) Curso de pós-graduação em Ciências Animais (UnB)
Prof. DR. Maurício Machaim Franco
Empresa Brasileira de Pesquisa Agropecuária (EMBRAPA-CENARGEN) Curso de pós-graduação em Ciências Animais (UnB)
Prof. Dra. Rosângela Vieira de Andrade
Programa de Pós-Graduação em Ciências Genômicas e Biotecnologia ( UCB/DF)
AGRADECIMENTO
Agradeço ao Dr. Rodolfo Rumpf pelo longo e atribulado período dedicado a minha orientação.
Agradeço aos técnicos, doutores, mestres e estudantes que compõe ou compuseram a equipe do Laboratório de Reprodução Animal da EMBRAPA CENARGEN, em especial: Regivaldo Vieira, Carlos Frederico Martins e Marcelo Tigre.
Agradeço aos técnicos, doutores, mestres e estudantes que compõe ou compuseram a equipe de Reprodução Animal da Fazenda Sucupira da EMBRAPA.
Agradeço a Danielle Cordeiro, companheira no laboratório e nos vários momentos de alegria e/ou crise experimental.
Agradeço à Dra. Rosangela Vieira de Andrade pelo apoio e imprescindíveis ensinamentos nos momentos finais deste trabalho.
Agradeço à Equipe do Laboratório de Biotecnologia do Campus II da Universidade Católica, aos alunos de iniciação científica que colaboraram no projeto e demais colaboradores do projeto, em especial ao Luis Henrique Sakamoto.
RESUMO
Referência: COLATTO, N. W. Anderson. Título: Análise da expressão de genes relacionados à totipotência celular em diferentes estágios do desenvolvimento embrionário em Bos taurus indicus.. 2010. 66 folhas. Dissertação de Mestrado em Ciências Genômicas e Biotécnologia)
– Universidade Católica de Brasília, Brasília, 2010.
A pesquisa com células-tronco é relatada a partir do inicio da década de 70. Reguladas por sinais intrínsecos e pelo ambiente externo, as células-tronco têm a habilidade de optar entre auto-renovação prolongada e a diferenciação. O termo totipotência é definido como o potencial ilimitado de células primordiais formarem tecidos embrionários, membranas e tecidos extra-embrionários. Dada as referências da literatura, este trabalho objetivou o estudo da expressão de genes relacionados como marcadores específicos para células tronco embrionárias durante fases específicas do desevovimento embrionário préimplantacional na raça Nelore, da subespécie zebuína (Bos taurus indicus). Foram utilizados pools de embriões
de 5 fases do período pré-implantacional: 4 células ( 48 horas da fecundação); 16 células (72 horas após a fecundação); mórula (64 células, 120 horas após a fecundação); blastocisto (Bl) (156 horas após a fecundação); blastocisto expandido (BX), (aproximadamente 168 horas após a fecundação). Através da técnica de Real Time-PCR quantitativo foi quantificada a expressão dos genes OCT4, NANOG, TRA1, e SSEA1, que estão relacionados ao grau de indiferenciação e a totipotência destas células. Durante as análises realizadas, NANOG, OCT4, SSEA1, e TRA1, utilizados para identificar células totipotentes, foram localizados com diferentes níveis de expressão em todas as fases descritas. Assim, infere-se que na espécie estudada existam células intermediárias transitórias que co-expressam tanto as características de células indiferenciadas quanto de seus derivados diferenciados, o que poderia explicar os resultados descritos neste trabalho. Embora NANOG, OCT4, SSEA1, e TRA1 sejam amplamente utilizados individualmente para identificar células-tronco humanas e de camundongos, sua utilização como marcadores individuais pode ser inconsistente para identificar células totipotentes de bovinos da raça Nelore. Foi observada a expressão do gene NANOG em embriões de 4 células, o que não é demonstrado na literatura para humanos e camundongos. Ainda foi possível observar a expressão de SSEA1 em todos os estágios estudados, o que contrasta com os relatos anteriores em humanos e camundongos de sua localização exclusiva em blastocistos. Destaca-se ainda que relatos anteriores demonstravam a ausência de expressão de SSEA1 em embriões bovinos. Ainda foi descrita a expressão de TRA1 em bovinos principalmente nos estágios BX, BL e 4C, e sua sub-expressão no estágio16C, o que sugere que exista na fase 4C resíduos da presença de TRA1 provenientes das geradoras.Portanto, com o estudo da expressão individual de cada um destes genes, não foi possível confirmar a existência de mecanismos únicos que garantam a identificação de linhagens puras de células-tronco embrionárias bovinas da subespécie Bos taurus indicus e a
confirmação de sua totipotência. Assim, até que se realizem estudos complementares, associando um maior conjunto de genes, as linhagens celulares oriundas destas células deverão manter sua definição como células-tronco “Tipo” embrionárias (embrionary stem like cells) de bovinos da subespécie Bos taurus indicus.
RESUMO EM LÍNGUA ESTRANGEIRA
Research with stem cells is reported from the early 70's. Regulated by intrinsic signals and the external environment, stem cells have the ability to choose between prolonged self-renewal and differentiation. The term totipotency is defined as the unlimited potential of primordial cells to form embryonic tissues, membranes and extra-embryonic tissues. Given the references in the literature, this work was to study the expression of related genes as specific markers for embryonic stem cells at specific stages of embryonic pre-implantation development in Nellore, subspecies zebu (Bos indicus). We used pools of embryos of five
stages of pre-implantation period: 4-cell (48 hours of fertilization), 16 cells (72 hours after fertilization), morula (64 cells, 120 hours after fertilization), blastocyst (Bl) (156 hours after fertilization), expanded blastocyst (BX), (approximately 168 hours after fertilization). Through a Real-Time quantitative PCR was quantified the expression of genes OCT4, NANOG, TRA1 and SSEA1, which are related to the degree of differentiation and totipotency of these cells. During the analysis, NANOG, OCT4, SSEA1 and TRA1 used to identify totipotent cells, were located at different levels of expression in all the phases. Thus, it appears that the species studied there are transitional intermediate cells co-express both the characteristics of undifferentiated and differentiated its derivatives, which could explain the results described in this paper. Although NANOG, OCT4, SSEA1 and TRA1 are widely used individually to identify human stem cells and mice, their use as individual markers may be inconsistent for identifying totipotent cells of Nelore cattle. We observed the NANOG gene expression in embryos of four cells, which is not shown in the literature for humans and mice. Although it was possible to observe the expression of SSEA1 at all stages studied, which contrasts with previous reports in humans and mice from its exclusive location in blastocysts. It is also emphasized that previous reports demonstrated the absence of expression in bovine embryos SSEA1. Although the expression has been described in cattle TRA1 mainly in stages BX, BL and 4C, and its sub-expression in estágio16C, which suggests that there is waste in Phase 4C TRA1 from the presence of the generators. Therefore, the study of individual expression of each of these genes was not possible to confirm the existence of unique mechanisms that ensure the identification of inbred lines of embryonic stem cells from cattle
Bos taurus indicus and confirmation of their totipotency. Thus, until further studies are
conducted, involving a larger set of genes, cell lines derived from these cells must maintain its definition as stem cells, "type" embryonic (stem embrionary like cells) from cattle Bos taurus indicus.
LISTA DE GRÁFICOS
Gráfico 01: Gráfico de CT comparativo por gene
Gráfico 02: Expressão relativa entre os Genes estudados nos diferentes estágios de desenvolvimento
Gráfico 03: Expressão relativa entre os Genes estudados nos diferentes estágios de desenvolvimento
Gráfico 04: Expressão relativa de H2A nos diferentes estágios de desenvolvimento Gráfico 05: Expressão relativa de NANOG nos diferentes estágios de desenvolvimento Gráfico 06: Expressão relativa de NANOG nos diferentes estágios de desenvolvimento utilizando logN
LISTA DE FIGURAS
Figura 01: Hierarquia de Células-tronco
Figura 02: Localização do gene OCT4 (PO5F1_BOVIN) no cromossomo 23
Figura 03: Localização do gene NANOG_BOVIN no cromossomo 05
Figura 04: Localização do gene FUT4_BOVIN (SSEA1) no cromossomo 15
LISTA DE TABELAS
Tabela 01: Momento de Coleta dos Embriões /Nº de embriões coletados Tabela 02: Referências dos genes utilizados
Tabela 03: Expressão de cada Gene X Estágio de desenvolvimento comparando com os padrões GAPDH e Estágio BX
LISTA DE SIGLAS
AGE - ativação do genoma embrionário BL - blastocisto
BX - blastocisto expandido cDNA-
CE - carcinoma embrionário
CEC - células embrionárias provenientes de carcinoma COC - complexos cummulus ovócito
CT -
CTE - células-tronco embrionárias DIA- atividade de diferenciação DNA - ácido desoxiribonucleico
EMBRAPA - Empresa Brasileira de Pesquisa Agropecuária FIV - fertilização in-vitro
ICM - inner cells mass (Massa Celular Interna) LIF - fator inibidor de leucemia
M - mórula
MZT - transição materno-zigótica PIVE - produção in vitro de embriões
RT-PCR - reação de polimerase em cadeia em tempo real RNA -
SUMÁRIO
1. INTRODUÇÃO . . . .15
2. REVISÃO DE LITERATURA . . . .17
2.1. CÉLULAS- TRONCO . . . .17
2.2. CÉLULAS- TRONCO EMBRIONÁRIAS . . . . .18
2.3. CÉLULAS- TRONCO EMBRIONÁRIAS EM BOVINOS. . . .21
2.4. GENES QUE CARACTERIZAM CÉLULAS TRONCO EMBRIONÁRIAS .22 2.4.1. OCT4 . . . .22
2.4.2. NANOG. . . .24
2.4.3. SSEA1. . . . .25
2.4.4. TRA1. . . . .26
2.5. DESENVOLVIMENTO EMBRIONÁRIO BOVINO, PERÍODO PRÉ-IMPLANTACIONAL . . . .27
3. OBJETIVOS . . . .30
4. MATERIAL E MÉTODOS . . . .31
4.1. LOCAL . . . .31
4.2. MATERIAL BIOLÓGICO . . . .31
4.3. PROCEDIMENTOS EXPERIMENTAIS . . . . .31
4.3.1. PRODUÇÃO DE EMBRIÕES BOVINOS IN VITRO PARA DOAÇÃO DE “CÉLULAS TRONCO EMBRIONÁRIAS”. .31 4.3.1.1. RECUPERAÇÃO DE OVÓCITOS E MATURAÇÃO IN VITRO .31 4.3.1.2. SELEÇÃO ESPERMÁTICA . . . .32
4.3.1.3. FECUNDAÇÃO IN VITRO E CULTIVO DE EMBRIÕES .32 4.3.2. FORMAÇÃO DE POOLS DE EMBRIÕES OU GRUPOS EXPERIMENTAIS . . . .32
4.3.3. CONGELAMENTO DE EMBRIÕES EM RNA LATTER . .33 4.3.4. PARÂMETROS DE AVALIAÇÃO . . . . .34
4.3.4.1. AVALIAÇÃO MOLECULAR DA TOTIPOTÊNCIA E GRAU DE INDIFERENCIAÇÃO DE EMBRIÕES BOVINOS EM DIFERENTES ESTÁGIOS DE DESENVOLVIMENTO PRÉ-IMPLANTACIONAL. .34 4.3.4.2. EXTRAÇÃO DE RNA . . . .34
4.3.4.4 TRATAMENTO DE AMOSTRAS COM DNASE . . .35 4.3.4.5. PREPARO DOS CDNA DAS AMOSTRAS . . .36 4.3.4.6 TAQMAN – REAL TIME PCR . . . . .37 4.3.4.7 ANÁLISE DE DADOS . . . .39
5. RESULTADOS E DISCUSSÃO .40
5.1. ANÁLISE DE CT . . . .40
5.2. EXPRESSÃO RELATIVA . . . .41
5.2.1. VARIAÇÃO DA EXPRESSÃO DO GENE H2A . . .43 5.2.2. VARIAÇÃO DA EXPRESSÃO DO GENE NANOG. . .45 5.2.3. VARIAÇÃO DA EXPRESSÃO DO GENE OCT4 . . .47 5.2.4.VARIAÇÃO DA EXPRESSÃO DO GENE SSEA1 . . .50 5.2.5.VARIAÇÃO DA EXPRESSÃO DO GENE TRA1 . . .51 5.3.VARIAÇÃO DA EXPRESSÃO GÊNICA EM CADA ESTÁGIO DE
DESENVOLVIMENTO . . . .53
6. CONCLUSÃO . . . .57
15
1. INTRODUÇÃO
A pesquisa com células-tronco é relatada a partir do inicio da década de 70. Neste período, células embrionárias provenientes de carcinoma (CEC), ou seja, células de linhagens germinativas de tumores chamados teratocarcinomas tiveram pela primeira vez as suas linhagens celulares estabelecidas (Anna etal, 2005).
Linhagens estáveis de células-tronco embrionárias têm sido estabelecidas em várias espécies como galinha, hamster, mink, suínos, macacos rhesus, incluindo humanos (Wang et al, 2005). Em bovinos a produção de supostas células-tronco embrionárias foi relatada pela
primeira vez por Saito et al, em 1992.
Reguladas por sinais intrínsecos e pelo ambiente externo, as células-tronco têm a habilidade de optar entre auto-renovação prolongada e a diferenciação. Ao removê-las de seu ambiente embrionário normal, as células-tronco podem proliferar e se renovar indefinidamente se cultivadas sob circunstâncias apropriadas in- vitro (Odorico et al, 2001).
Diversas chaves de regulação do metabolismo de células-tronco foram identificadas como essenciais para a formação da Massa Celular Interna (ICM) durante o desenvolvimento de embriões em pré-implantação e auto-renovação de células-tronco embrionárias pluripotentes. Entre estes reguladores incluem os genes Oct4, Sox2, e Nanog (Chew et al,
2005).
Dada às referências da literatura quanto à expressão de genes das células-tronco embrionárias bovinas, este trabalho objetivou o estudo da expressão de genes relacionados como marcadores específicos para células-tronco embrionárias durante fases específicas do desevovimento embrionário pré-implantacional, na raça Nelore, da subespécie zebuína (Bos taurus indicus).
A subespécie zebuína Bos taurus indicus foi escolhida em função do importante papel
econômico e social dos rebanhos zebuínos na pecuária brasileira e do interesse nacional e internacional na aquisição e multiplicação de animais de elevado valor genético (Zanenga, 1993).
Foi quantificada a expressão dos genes OCT4, H2A, NANOG, TRA-1, SSEA-1 por meio de Real Time-PCR quantitativo (Reação de transcriptase reversa em cadeia de polimerase em tempo real).
16 Deste modo, este trabalho amplia o estudo do período pré-implantacional bovino com o objetivo de detectar e quantificar a expressão destes genes, demonstrando a necessidade de determinar estágios específicos do desenvolvimento embrionário para a realização do isolamento das células tronco embrionárias em bovinos da raça Nelore.
A fim de explorar este potencial notável das células-tronco embrionárias bovinas é necessário um maior desenvolvimento das tecnologias atualmente utilizadas, buscando assegurar a manipulação adequada destas células. Apesar dos grandes avanços alcançados no estudo de células-tronco, pouco ainda se sabe sobre o isolamento, condições de cultivo, monitoramento, e principalmente sobre os mecanismos celulares e moleculares que mantém essas células indiferenciadas em cultivo ou que estimulam a sua diferenciação nos mais diversos tipos de tecido.
Estas tecnologias, incorporadas aos demais protocolos previamente desenvolvidos no Laboratório de Reprodução Animal da EMBRAPA - Recursos Genéticos e Biotecnologia servirão como ferramentas para a execução de novas técnicas de reprodução animal. Este trabalho traz como perspectiva futura a utilização de células-tronco cultivadas em processo de transferência nuclear, bem como a manutenção de cultivos celulares de células tronco embrionárias por períodos indeterminados, a produção de uma infinita fonte de células doadoras de núcleo e também a capacidade de manter in vitro células contendo linhagens
17
2. REVISÃO DA LITERATURA
2.1. Células-Tronco
O conceito de células-tronco foi originado no final do século XIX como um postulado teórico para descrever a habilidade de certos tecidos (sangue, pele, etc.) realizarem a auto-renovação por toda a vida, mesmo que eles sejam formados por células de vida curta (Bianco
et al, 2008).
Para melhorar as definições de células-tronco foram criadas sub-classificações com o objetivo de demonstrar a capacidade de diferenciação destas células. O termo totipotência foi definido como um potencial ilimitado de células primordiais (células de linhagens embrionárias primarias) formarem tecidos embrionários, membranas e tecidos extra-embrionários. Enquanto que as células pluripotentes, apesar de poder se diferenciar em células precursoras para as três linhagens germinativas, não são capazes de formar estruturas extra-embrionárias, ou seja, não são capazes de originar um embrião completo (Gerecht-Nir et al, 2004).
A maioria dos cientistas usa o termo pluripotentes para descrever células-tronco que podem dar origem a células derivadas de todos os três folhetos embrionários, mesoderme, endoderme e ectoderme. Estas três camadas germinativas são a origem embrionária de todas as células do corpo. Todos os muitos tipos diferentes de células especializadas que compõem o corpo são derivadas de uma dessas camadas germinativas. Células pluripotentes têm potencial para dar origem a qualquer tipo de célula, sendo esta uma propriedade observada no decurso natural do desenvolvimento embrionário e sob certas condições em laboratório (Slack, J.M, 2000; Anna et al.,2005).
Célula-tronco unipotente é um termo normalmente aplicado a uma célula em organismos adultos e significa que as células em questão são capazes de se diferenciar em apenas uma linhagem. Tecidos danificados são tipicamente unipotentes e dão origem a apenas um tipo de célula em condições normais. Este processo permite um estado constante de auto-renovação do tecido. Contudo, se o tecido é danificado e necessita que vários tipos de células sejam substituídas, células-tronco pluripotentes são ativadas para reparar os danos (Slack, J.M., 2000).
18 células que compõem o organismo. Isto é, as células-tronco têm o potencial de desenvolver células maduras com características, formas e funções especializadas. Muitos dos termos utilizados para definir células-tronco dependem do comportamento das células no organismo intacto (in vivo), em condições laboratoriais específicas (in vitro), ou após transplante in vivo,
mesmo quando realizado para tecido diferente daquele a partir do qual as células-tronco foram derivadas, conforme demonstrado na figura 01. Os tecidos de mamíferos adultos, inclusive humanos, são compostos de mais de 200 tipos de células diferentes. Podem ser descritas células nervosas (neurônios), células musculares (miócitos), células epiteliais, células sanguíneas (eritrócitos, monócitos, linfócitos, etc), células ósseas (osteócitos), cartilagem, entre outras (Chandross, K.J. and Mezey, E., 2001).
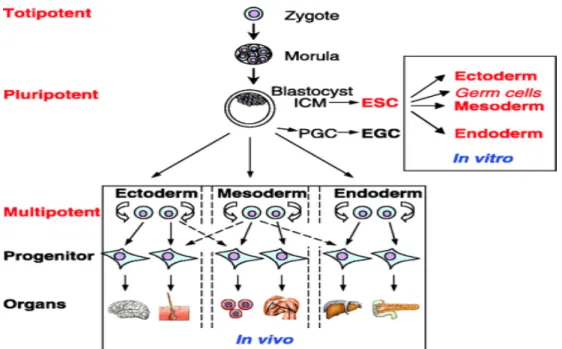
Figura 1: Hierarquia de Células tronco
Figura 1: Hierarquia de Células tronco. Esta Figura representa o desenvolvimento embrionário a partir do Zigoto e as possíveis diferenciações celulares até a formação de órgãos. (Adaptado de Anna et al.,2005)
2.2. Células-tronco Embrionárias
19 cuidadosa (tripsina/EDTA) e com a criação de subculturas (Robertson, 1987). Uma vez estabelecidas linhagens de células-tronco murinas (MES) foi demonstrada uma capacidade quase ilimitada de proliferação in vitro (Smith, 2001) mantendo a capacidade de contribuir
para todas as linhagens celulares. Em laboratório, foi demonstrado que as células MES mantêm o cariótipo normal e relativamente estável, mesmo com a continuação das passagens. Células-tronco também foram caracterizadas por um tempo de geração relativamente curto de 12 a 15 horas, com uma fase do ciclo celular G1 curta (Rohwedel et al, 1996).
CTE foram derivadas de ICM de embriões de ratos por Martin, em 1981, e por Evans e Kaufman, também em 1981. As células-tronco embrionárias têm sido derivadas de primatas não humanos e humanos, porém, até pouco tempo, somente algumas poucas linhagens haviam sido estudadas e caracterizadas (Francis Pau and Wolf, 2004). As células-tronco embrionárias podem ser obtidas através de ICM de blastocistos intactos, através de imunocirurgia, dissociação química, mecânica, ou outros métodos de isolamento de células pluripotentes que constituem o ICM (Tanaka et al, 2006).
O termo células-tronco embrionárias (CTE) foi introduzido para diferenciar células derivadas de carcinomas embrionários de outro grupo de células derivadas de embriões que demonstraram pluripotência. As células-tronco embrionárias são derivadas de células totipotentes de embriões em estágios iniciais de desenvolvimento. Estas linhagens celulares podem ser mantidas indiferenciadas em cultura indefinidamente, podendo, em certas condições, se diferenciar em vários tipos celulares. Sua principal característica é a capacidade de se diferenciar em células primordiais que darão origem as três linhagens germinativas (ectoderme, endoderme e mesoderme) in vivo ou in vitro (Thomson et al, 1998; Saito et al,
2003; Gerecht-Nir et al, 2004; Shufaro, 2004).
O interesse em células-tronco pluripotentes em camundongos foi estabelecido a partir de 1967. Foi demonstrado o isolamento de células-tronco a partir de teratocarninomas, mistura de tipos de células diferenciadas, incluindo os três folhetos embrionários (endoderme, mesoderme e ectoderme). Também foi demonstrada esta associação em células indiferenciadas que lembram células embrionárias, denominado carcinoma embrionário (CE) (Martin e Evans, 1975).
20 demonstrados, as CE não são consideradas ideais para o estudo de células de pluripotência devido ao seu cariótipo aneuploide. Entretanto, estes estudos com CE são considerados a base para o isolamento de células-tronco embrionárias (Martin, 1980; Evans, 1981).
Células-tronco embrionárias são derivadas de blastocistos durante o período pré-implantacional. O blastocisto é composto por uma camada externa do trofoblasto que são células que dão origem à placenta. Em seu interior está localizada uma população de células chamada massa celular interna (ICM). A ICM está destinada a dar origem a todos os tecidos do corpo, bem como, alguns tecidos extra-embrionários. É a partir destas células que as células-tronco embrionárias são derivadas (Martin, 1980; Evans, 1981).
O fator capaz de promover a auto-renovação inibindo a diferenciação prevista pelas células de nutrição foi identificado por volta de 1980. Originalmente denominado inibidor da atividade de diferenciação (DIA) (Smith and Hooper, 1987), esse fator mais tarde foi identificado como fator inibidor de leucemia (LIF), um membro da família das citocinas (IL-6) (Smith et al, 1988; Williams et al, 1988). Esta descoberta permitiu a propagação das
células-tronco embrionárias a partir da utilização de culturas de células de nutrição criopreservadas na presença de soro e LIF recombinante. Desta forma, as células-tronco embrionárias podem ser amplamente propagadas in vitro. Portanto, o papel do LIF é manter a pluripotência da população de células-tronco fora do seu desenvolvimento padrão (Nichols et al, 2001).
Semelhantes às células murinas, células-tronco embrionárias humanas apresentam potencial proliferativo mantido por períodos prolongados de cultura, mantendo um cariótipo normal mesmo em derivados clonais. Várias diferenças importantes são descritas entre células de camundongo e de origem humana. No final de 2001, linhagens de células-tronco embrionárias humanas foram estabelecidas usando camadas de nutrição de fibroblastos embrionárias de camundongo (Anna, 2005).
Linhagens de células-tronco pluripotentes têm sido estabelecidas para diversas espécies domésticas, como por exemplo, bovinos, ovinos, e suínos. Em bovinos, linhagens de células-tronco pluripotentes derivadas de embriões ou de células germinativas primordiais são consideradas uma fonte ilimitada de células pluripotentes. No entanto, o desenvolvimento da placenta de fetos provenientes de transferência nuclear de células-tronco é incompleto e várias abordagens para superar este problema estão sendo descritas (Strelchenko, 1996).
A criação de células-tronco de embriões de bovinos tem sido problemática. A pluripotência in vitro e in vivo foram descritas em várias linhagens de células-tronco
21 marcadores que são normalmente associados a linhagens de CTE geradas a partir de outras espécies (Mitalipova et al, 2001, Yindee et al, 2000).
2.3 Células-tronco embrionárias em bovinos
Alguns trabalhos destacam que linhagens de CTE bovinas podem manter um cariótipo normal através de passagens numerosas e podem diferenciar in vitro dando forma aos tecidos
do embrião. Dado seu potencial de auto-renovação, combinado com sua potencialidade para diferenciar-se em tipos específicos de células, estas células podem fornecer uma fonte ilimitada de células para transplantes (Stice et al, 1996). Estudos utilizando a exposição de
embriões bovinos de 2 e 4 células à [3H] uridina seguida por autoradiografia revelaram que a atividade transcricional poderia ser detectada no início do desenvolvimento (Viuff et al,
1996).
Avanços na tecnologia molecular facilitaram os estudos da transcrição de genes em embriões. Usando um display diferencial de RT-PCR, a sensibilidade à amanitina foi detectada em embriões entre 2 e 5 células, mas tornou-se predominante após os estágios de desenvolvimento de 6 a 8 células. (Natale, 2000, Kanka, 2003). Com a utilização de microarranjos, a expressão de alterações globais no gene foi caracterizada durante o desenvolvimento pré-implantacional de camundongos. Estas análises revelaram mudanças no perfil de transcrição em embriões de 2 células, maiores do que era descrito previamente (Zeng Schultz, 2004).
Em um estudo semelhante, os autores geraram e caracterizaram uma biblioteca de cDNA rica em transcrições embrionárias. Muitos destes transcritos são expressos em embriões de 6 e 8 células. Esses trabalhos seguem observações semelhantes às obtidas em camundongos e sugerem que há um baixo nível de atividade transcricional de embriões bovinos em estágios de 1 a 4 células, seguido pela ativação do genoma no estágio de 8 células (Kañka et al, 2008). Nesta fase, a ativação global do genoma do embrião bovino também foi
22
2.4. Genes que caracterizam células-tronco embrionárias
Apesar do potencial valor da utilização de células-tronco embrionárias bovinos para pesquisas com transgênicos e estudos de linhagem, os principais aspectos da criação de células-tronco embrionárias em bovinos, como a identificação específica de marcadores de pluripotência, precisam ser esclarecidos para alcançar bons resultados. Embora os genes SSEA4, TRA-1-60, TRA-1-81 e NANOG sejam amplamente utilizados para identificar células tronco embrionárias em camundongos e humanos, a sua utilização como marcadores foi considerada inadequada para identificar células pluripotentes em bovinos (Muñoz et al,
2008). Em 2009, foi demonstrado por Pant e Keefer que NANOG, POU5F1 e SOX2 são expressos por um período curto em cultivos de células bovinas em sistemas de cultivo com co-cultivo.
2.4.1 OCT4
O gene OCT4 é um membro da família POU de transcrição e desempenha um papel crucial durante a embriogênese. É localizado tanto em ICM quanto em TE em humanos (Gauffman et al, 2005), e em ICM de embriões de camundongos (Kirchhof et al, 2000).
Dados controversos descrevendo a expressão de OCT4 apenas no epiblasto (Vejlsted et al,
2005), ou na ICM e TE de embriões de bovinos (Kirchhof et al, 2000) também já foram
relatados. Estas discrepâncias entre espécies e a ausência de marcadores de células-tronco específicas de bovinos fazem necessários maiores estudos para validar marcadores de pluripotência antes da sua utilização heteroespecífica.
A proteína POU5F1 (OCT4) foi localizada tanto em ICM quanto em trofectoderma em bovinos, em caprinos, blastocistos bovinos e suínos (Pant and Keefer, 2009; He et al., 2006;
Kirchhof et al., 2000; van Eijk et al., 1999). Assim, enquanto POU5F1 de forma isolada não
pode servir como um marcador definitivo para CTE em bovinos, ele deve fazer parte do painel de marcadores de pluripotência, pois sua ausência seria indicativa de uma perda da pluripotência (Pant and Keefer, 2009).
Diversos genes alvo de OCT4 têm sido identificados em células-tronco embrionárias. Esse grupo inclui Fgf4, Utf1, Opn, Rex1/Zfp42, Fbx15, e Sox2 (5, 6, 8, 12, 27, 46, 47, 53). As
23 tronco embrionárias. O aumento ou diminuição do nível da expressão OCT4 resultam em diferenciação de células-tronco em endoderme primitivo e trofectoderme e, por conseqüência, gera a perda de pluripotência (Kurosaka et al, 2004; Chew et al, 2005).
A expressão de OCT4 é considerada um marcador chave para a identificação de células pluripotentes de camundongos e potencialmente em outras espécies de mamíferos. O OCT4 ortólogo de bovinos tem homologia de 90.6% e 81.7% em relação ao mesmo gene em humanos e ratos respectivamente. Avaliando a conservação da seqüência genômica e de regiões regulatórias, a proteína Oct4 dos bovinos não é restrita aparentemente às células embrionárias pluripotentes no estágio de pré-implantação, estando presentes no ICM e em trofectoderma, ainda em todas as células provenientes de blastocistos no 13º dia de cultivo (Kurosaka et al, 2004; Chew et al, 2005).
Os transcritos de OCT4 estão presentes em baixa concentração em oocitos bovinos. A transcrição do OCT4 bovino começa a partir do primeiro ao segundo ciclo celular após a ativação do genoma zigótico, seguido por um aumento na transcrição subseqüente à compactação. Os transcritos de OCT4 estão indubitavelmente em todas as células de embriões em estágio de mórula; entretanto, em blastocistos de bovinos no 7° dia, o sinal OCT4 não é visível no trofectoderma pela hibridização in situ, isso indica que a regulação transcricional de
OCT4 em diferenciação é similar às observadas em ratos e em outros mamíferos (Kurosaka et al, 2004).
Em bovinos, foi demonstrada uma variabilidade de expressão de oito genes em embriões individuais. Porém, esta diferenciação não ocorreu entre embriões produzidos por Transferência Nuclear –TN e Fertilização in-vitro – FIV. Ou seja, o nível de variação de
expressão de proteínas de cada um dos genes quando se compara embrião com embrião, não apresentam diferença significativa entre embriões produzidos por TN e FIV. A variabilidade dos níveis de expressão parece ser intrínseca aos genes, com um número de genes altamente expressos geralmente, mas nem sempre, exibindo variação menor em seus níveis. (Pfeffer et al, 2007).
Blastocistos bovinos no 8º dia também expressam proteína de Oct4 em ICM e células TE, mas a expressão da proteína Oct4 é restrita para as células de epiblasto e não para as células de embriões bovinos TE de 12 dias, sugerindo que OCT4 também é um marcador de pluripotência de ICM e de linhagens de epiblasto em células de bovinos (Cao et al, 2009).
Em Bos taurus o gene OCT4 fica localizado no cromossomo 23, possui 1083
24
Figura 02: Localização do gene OCT4 (PO5F1_BOVIN) no cromossomo 23. Código de acesso Ensembl: ENSBTAT00000028122
Nome PO5F1_BOVIN (UniProtKB/Swiss-Prot)
Em resumo, OCT4 é descrito como octâmero codante de transcrição fator 4, (encoding octamer-binding transcription factor-4). Está ligado ao complexo maior de histocompatibilidade no cromossomo 23, tendo sua ligação a estágios pré-implantacionais confirmada por imunofluorescencia indireta. Também foi observada em microscopia a laser sua presença em ICM e trofoblasto de blastocistos de 10 dias de desenvolvimento (van Eijk et al, 1999).
2.4.2 NANOG
Durante o desenvolvimento embrionário e diferenciação de célula-tronco, as células progenitoras perdem gradualmente a sua diferenciação potencial em paralelo com o aumento da perda de capacidade replicativa, uma característica importante do envelhecimento. Diferenças foram observadas entre marcadores de indiferenciação celular presentes ao comparar embriões de camundongos, humanos e bovinos. NANOG, que seria um fator de transcrição relacionado à pluripotência foi encontrado tanto em ICM (Massa Celular Interna) e trophectoderma (TE) de blastocistos bovinos (Degrelle et al, 2005). Porém, sua expressão
está restrita ao ICM em blastocistos humanos e de camundongos (Chambers et al, 2003;
Adjaye et al, 2005).
O gene NANOG é um fator crítico para a pluripotência encontrada em células embrionárias de camundongos (Chambers et al., 2003; Mitsui et al., 2003) e também foi
25 Em Bos taurus o gene NANOG fica localizado no cromossomo 05, possui 1622
nucleotídeos e 300 aminoácidos. A figura abaixo foi retirada do Ensebl genome browser e
demonstra a correta localização do gene NANOG dentro do cromossomo 05 (Kocabas et al,
2005).
Figura 03: Localização do gene NANOG_BOVIN no cromossomo 05. Código de acesso Ensembl: ENSBTAT00000027863
Nome NANOG_BOVIN (UniProtKB/Swiss-Prot)
NANOG é um regulador de transcrição envolvido na proliferação e auto-renovação de ICM e células tronco embrionárias. Impõe pluripotência em células-tronco embrionárias e impede sua diferenciação em relação à endoderme extraembrionária e linhagens de trofectoderma. Atua como um ativador e repressor transcricional. Vincula-se perfeitamente à seqüência consenso do DNA 5'-TAAT [GT] [GT] -3 'e 5'-[CG] [GA] [CG] C [GC] ATTAN [GC] -3'. Quando super-expressa, promove a entrada das células em fase S e proliferação (Kocabas et al, 2005).
2.4.3. SSEA1
26 Embora SSEA4, TRA-1-60, TRA-1-81 e NANOG sejam amplamente utilizados para identificar células tronco humanas e de camundongo, a sua utilização como marcadores pode ser inadequada para identificar células pluripotentes bovinas. Muitas pesquisas ainda são necessárias para identificar marcadores confiáveis de pluripotência para esta espécie (Muñoz
et al, 2007). Ainda destaca-se que antígenos embrionários de fase específica, como SSEA1,
SSEA4, TRA-1-60 e TRA 1-81, têm sido amplamente utilizados como marcadores para identificar células-tronco embrionárias de camundongos e humanos (Henderson et al, 2002).
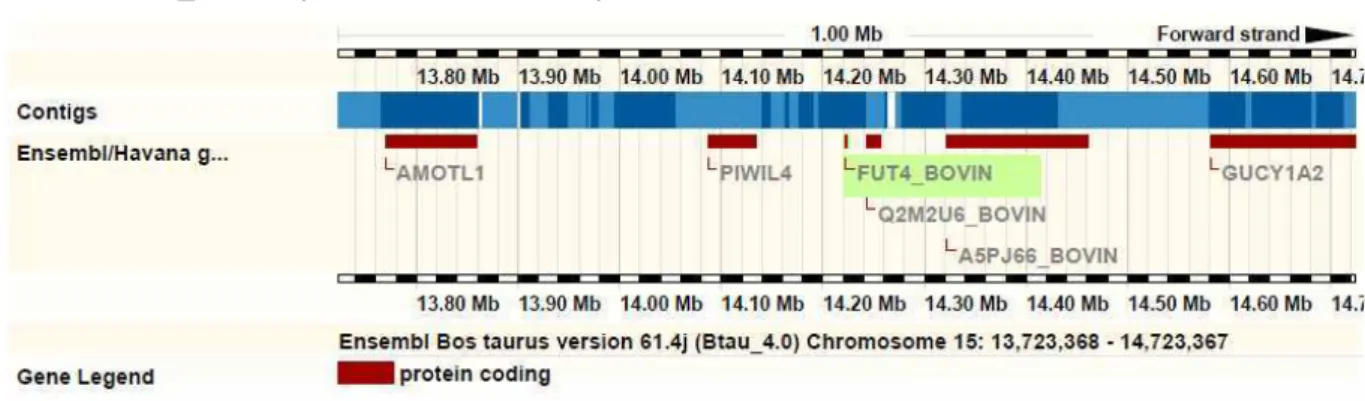
Em Bos taurus o gene FUT4_BOVIN (SSEA1) fica localizado no cromossomo 15,
possui 1410 nucleotídeos e 398 aminoácidos. A figura abaixo foi retirada do Ensebl genome browser e demonstra a correta localização do gene FUT4_BOVIN dentro do cromossomo 15
(Zimin et al, 2009).
Figura 04: Localização do gene FUT4_BOVIN (SSEA1) no cromossomo 15. Código de acesso Ensembl: ENSBTAG00000038062
Nome: FUT4_BOVIN (UniProtKB/Swiss-Prot)
FUT4 é considerado um catalizador de ligações alpha-1,3 glycosidica e também está envolvido na expressão de antígenos Lewis X/SSEA-1 e VIM-2. Estes dados foram obtidos por similaridade, pois ainda não existem dados experimentais que comprovem estes resultados (Javaud C. e Julien R., 2002).
2.4.4. TRA1
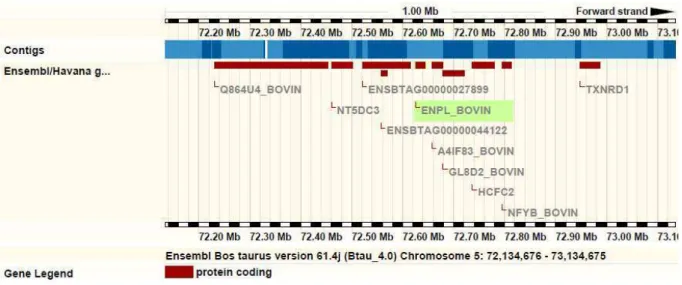
O gene TRA1 é também conhecido como HSP90B1, ou antígeno de rejeição tumoral (gp96) 1 [ Bos taurus ], ou ainda ENPL_BOVIN, precursor de endoplasmina. Apresenta
27 endoplasmático associada à degradação, além de possuir atividade de ATPase (Watanabe et al., 2001).
Em Bos taurus o gene TRA1 (ENPL_BOVIN) está localizado no cromossomo 5,
possui 2791 nucleotídeos e 806 aminoácidos. A figura abaixo foi retirada do Ensebl genome browser e demonstra a correta localização do gene TRA1 dentro do cromossomo 5 (Watanabe et al., 2001).
Figura 05: Localização do gene TRA1 (ENPL_BOVIN) no cromossomo 5. Código de acesso Ensembl: ENSBTAG00000003362
Nome: ENPL_BOVIN (UniProtKB/Swiss-Prot)
2.5. Desenvolvimento Embrionário Bovino, período pré-implantacional
Nos mamíferos, o período pré-implantação está compreendido entre a formação do zigoto até pouco além do estágio de blastocisto. Este período do desenvolvimento é caracterizado por quatro etapas biológicas distintas que ocorrem após a fertilização do ovócito e incluem desde a (I) primeira clivagem, (II) transição materno-zigótica (MZT) ou ativação do genoma embrionário (AGE), (III) a compactação e conseqüente formação da polarização do epitélio embrionário e (IV) a diferenciação de mórula em blastocisto (Wrenzycki et al, 2005),
28 da informação genética derivada do genoma do embrião (Kanka, 2003; Bettegowda et al,
2008).
Estudos com camundongos demonstraram que é possível isolar células-tronco embrionárias em um período limitado de seu desenvolvimento inicial, porém linhagens não validadas de CTE foram isoladas de embriões em estágios de pré-blastocisto ou pós-implantação. Outras linhagens celulares foram obtidas de embriões compactados de camundongo em estágio de mórula. As células-tronco embrionárias receberam interesse adicional porque as CTE humanas relatam diferenciação in-vitro em linhagem de
trofectoderma extra-embrionário (Tesar, 2005).
O zigoto é um dos estágios iniciais da divisão celular (blastômeros) compreendido até a fase de mórula, período em que são definidos como totipotentes. No estágio de blastocisto, apenas as células da massa celular interna (ICM) mantém a capacidade de gerar as três camadas germinativas, a endoderme, mesoderme e ectoderme, bem como as células germinativas primordiais, sendo, portanto, consideradas pluripotentes. Em tecidos adultos, células multipotentes existem em tecidos e órgãos para substituir as células perdidas ou lesadas. Células-tronco embrionárias, derivadas do ICM, têm a capacidade de desenvolvimento por diferenciação in vitro em células de todas as linhagens somáticas, bem como células germinativas (Anna et al.,2005).
A produção in vitro de embriões (PIVE) expandiu-se nas diferentes espécies animais
pouco tempo após o nascimento de Louise Brown, em 1978, na Inglaterra. Brown foi o primeiro bebê de proveta do mundo (Steptoe e Edwards, 1978). Em 1982, nasceu o primeiro bezerro bovino produzido por fecundação in vitro nos Estados Unidos (Brackett et al., 1982),
o que só foi possível graças às pesquisas iniciais desenvolvidas com animais de laboratório. O nascimento do primeiro bezerro por fecundação in vitro ocorreu no dia 9 de junho
de 1981. Neste trabalho, foram utilizadas 22 doadoras e sete receptoras e, para a fecundação, foram utilizadas amostras de sêmen fresco e congelado. As amostras de sêmen eram de animais pré-selecionados para inseminação artificial, o que indicava uma boa qualidade seminal. A coleta dos ovócitos foi realizada por via cirúrgica e foram recuperados 177 ovócitos já maturados dos quais 52% foram fecundados. Embora grande quantidade de embriões tenha sido transferida, a gestação só foi alcançada em uma receptora, a qual havia recebido apenas um embrião no estágio de quatro células. O primeiro bezerro de fecundação
in vitro nasceu pesando 45 kg e, após alguns meses de observação, não foram constatadas
29 Atualmente, o processo de fecundação in vitro na espécie bovina é o que apresenta
maior sucesso dentre todas as espécies, sendo que 40% ou mais dos ovócitos maturados e fecundados in vitro podem se desenvolver até blastocisto, e muitos bezerros já nasceram após
a utilização destas técnicas (Hasler, 1998; Bavister, 2002).
Após o maior entendimento do processo de fecundação em mamíferos, meios capazes de favorecer o crescimento embrionário até o estádio de blastocisto começaram a ser desenvolvidos. Em 1967, um primeiro trabalho demonstrou que zigotos de camundongos evoluíam até o estádio de duas células embrionárias na presença de lactato e piruvato (Whittinham e Biggers, 1967). Um ano depois, Whitten e Biggers obtiveram desenvolvimento de embriões em um meio simples, sem o aporte de substâncias macromoleculares de origem materna (Whitten e Biggers, 1968).
O cultivo pode se estender até o 7º dia após a fecundação in vitro, quando é realizada a
seleção e a avaliação dos embriões para a transferência ou criopreservação. Para avaliação da taxa de eclosão ou da qualidade embrionária, principalmente pela determinação do número e viabilidade de blastômeros, o cultivo in-vitro pode se estender até o 8º ou 9º dia após a
fecundação in vitro (Gonsalves et al., 2002).
Geralmente, é esperado que, após a maturação in vitro, aproximadamente 90% dos
ovócitos submetidos à maturação atinjam a metáfase II com expulsão do primeiro corpúsculo polar. Destes, 80% são fecundados e dividem-se pelo menos até o estádio de duas a quatro células. No entanto, apenas 25 a 40% destes embriões alcançam o estádio de blastocisto ou blastocisto expandido (Bavister et al., 1992). Este fato mostra que o cultivo in vitro é o
principal passo a determinar a eficiência do sistema e que muito deve ser feito para a melhoria dos resultados (Galli et al., 2003).
30
3. OBJETIVOS
Objetivo Geral
Analisar a expressão gênica de células embrionárias bovinas em diferentes estágios do desenvolvimento.
Objetivos Específicos
Padronizar metodologia de PCR em Tempo Real para embriões bovinos em diferentes estágios de maturação.
31
4. MATERIAIS E MÉTODOS
4.1. Local
O presente trabalho foi desenvolvido no Laboratório de Reprodução Animal da EMBRAPA - Recursos Genéticos e Biotecnologia, Laboratório de Reprodução Animal da Fazenda Sucupira da EMBRAPA-DF, e Laboratório de Biotecnologia do Campus II da Universidade Católica de Brasília, Brasília-DF.
4.2. Material Biológico
Para a execução deste trabalho foram utilizados embriões bovinos em 5 estágios de desenvolvimento pré-implantacional. Para sua produção destes embriões foram utilizados ovócitos bovinos obtidos de ovários de 12 fêmeas doadoras da espécie zebuína (Bos taurus indicus) da raça Nelore do rebanho da Fazenda Sucupira da EMBRAPA e sêmem,
previamente congelado, proveniente do ejaculado de touro da espécie zebuína (Bos taurus indicus) da raça Nelore. Para a produção dos embriões foi utilizado o protocolo de fertilização in vitro (PIVE) do Laboratório de Reprodução Animal da EMBRAPA - Recursos Genéticos e
Biotecnologia, Brasília-DF.
4.3. Procedimentos Experimentais
4.3.1. Produção de embriões bovinos “in-vitro” para avaliação da expressão de gênica
Buscando obter embriões bovinos potencialmente doadores de células-tronco embrionárias a produção de embriões foi realizada a partir da técnica de fertilização in vitro
(PIVE), utilizando o protocolo padronizado de fertilização in vitro do Laboratório de
Reprodução Animal da EMBRAPA Recursos Genéticos e Biotecnologia.
4.3.1.1. Recuperação de ovócitos e maturação in vitro
Complexos cummulus ovócito (COC) foram aspirados de folículos de 2-8 mm com
32 ml. Após a sedimentação, os COCs foram recuperados e selecionados com auxílio de estereomicroscópio.
Somente COCs que apresentavam citoplasma homogêneo e com pelo menos três camadas de células do cummulus, foram utilizados. Os COCs selecionados foram lavados e
transferidos, em número de 30-35, para uma gota de 200 µL de meio de maturação sob óleo de silicone e incubados por 22 h à 39ºC em uma concentração de 5% de CO2 em ar.
4.3.1.2. Seleção espermática
O sêmen do touro selecionado foi descongelado e as células espermáticas foram selecionadas por centrifugação em gradiente descontínuo de Percoll (45-90%). O gradiente de Percoll 45% foi preparado com 1 mL de Percoll 90% e 1 mL de SP-TALP. Após a
centrifugação, o sobrenadante foi descartado e o pellet foi centrifugado por 5 min em
SP-TALP. Posteriormente, o pellet resultante foi ressuspendido com 80 µL de SP-TALP e
utilizado para FIV.
4.3.1.3. Fecundação in vitro e cultivo de embriões
Os ovócitos maturados foram transferidos para gotas de 200 µL de meio de fecundação. Foi utilizado sêmen congelado de touro Nelore (B. indicus) e os espermatozóides
móveis foram separados por gradiente de densidade em Percoll 45% e 90%. A concentração
final de espermatozóides vivos foi de 1 x 10.000.000 espermatozóides/mL. Ovócitos e espermatozóides foram co-incubados por 20 horas em atmosfera de 5% CO2 em ar e temperatura de 39,0ºC com umidade máxima. Após a co-incubação, os possíveis zigotos foram lavados e transferidos para gotas de meio SOF suplementado com 5% de soro fetal bovino (SFB), e cultivados a 39ºC e 5% CO2 em ar por até 8 dias.
Os embriões foram acompanhados durante 8 dias, e neste período foram realizadas avaliações quanto à clivagem e formação de blastocisto.
4.3.2. Formação de Pools de Embriões ou Grupos Experimentais
33 aos embriões contendo 4 células, este grupo foi coletado aproximadamente 48 horas após a fecundação); Fase 2 – Fase 16C (O segundo grupo (Fase 2) corresponde aos embriões contendo 16 células, este grupo foi coletado aproximadamente 72 horas após a fecundação); Fase 3 – Fase Mórula (corresponde aos embriões contendo aproximadamente 64 células, em estagio de mórula. Este grupo foi coletado aproximadamente 120 horas após a fecundação); Fase 4 – Fase BL (corresponde aos embriões em estagio de blastocisto (Bl). Este grupo foi coletado aproximadamente 156 horas após a fecundação); Fase 5 – Fase BX (corresponde aos embriões em estágio de blastocisto expandido (BX). Este grupo foi coletado aproximadamente 168 horas após a fecundação).
Na tabela abaixo estão descritos os números de embriões coletados e o número de coletas realizadas para cada fase selecionada.
Tabela 01: Momento de Coleta dos Embriões/Nº de embriões coletados
Momento de Coleta dos Embriões /Nº de embriões coletados Pool Estágio de Desenvolvimento Número de Embriões
Coletados
Número de Coletas
1 4C (4 células) 22 5
2 16C (16 Células) 25 6
3 M (Mórula) 20 5
4 BL (Blastocisto) 24 5
5 BX (Blastocisto Expandido) 25 6
Total 116 27
A cada manipulação, grupos de aproximadamente 5 embriões de cada um dos estágios citados foram separados e congelados em RNA later conforme descrito a seguir.
4.3.3. Congelamento de Embriões em RNA later
Para o congelamento de embriões foi utilizada solução de estabilização de RNA (Stabilization Solution RNAlater® Tissue Collection) da Applied Biosystems. Grupos de até
34
4.3.4. Parâmetros de Avaliação
4.3.4.1. Avaliação molecular de embriões bovinos em diferentes estágios de desenvolvimento pré-implantacional.
Foi avaliada a expressão dos genes OCT4, NANOG, TRA-1, SSEA-1, GAPDH, H2A, utilizando técnica de PCR quantitativo em diferentes estágios do desenvolvimento embrionário pré-implantacional.
4.3.4.2. Extração de RNA
O RNA dos embriões foi extraído e purificado com o Kit Mini Spin Cell (Invitek®) conforme descrito abaixo.
Extração do RNA total pelo Kit Mini Spin Cell (Invitek®)
Para cada extração, 350µL de Solução de Lise R foram colocadas em ependorff de 1,5mL. Foram acrescentados aos ependorffs 3,5µL de DTT 0,1M. Depois de acrescentar a Solução de Lise R com o DTT em um ependorff com os embriões, foi pipetado o lisado diretamente no filtro DNA binding, acoplado em um tubo coletor com tampa. Até este momento todos os procedimentos foram realizados com as amostras conservadas em gelo. A partir deste ponto, todos os procedimentos são realizados a temperatura ambiente.
Em seguida as amostras foram incubadas por 1 minuto e centrifugadas a 12.000 rpm (rotações por minuto) por 2 minutos, após este passo o filtro foi descartado. Então foram acrescentados 350µL de Etanol 70% e misturados por pipetagem. A seguir foi pipetado todo o conteúdo do RNA diretamente no filtro RNA spin. As amostras foram incubadas por 1 min e centrifugadas a 10.000 rpm por 30 segundos; após descartar o filtrado (líquido), foram reutilizados tanto o filtro RNA spin quanto o tubo coletor. Foram Acrescentados 600 µL de tampão de lavagem (Wash Buffer R1) diretamente no filtro RNA spin. As amostras foram centrifugadas a 10.000 rpm por 30 segundos; em seguida foi descartado o filtrado novamente reutilizando tanto o filtro RNA spin quanto o tubo coletor.
35 foi Centrifugado a 12.000 rpm por 5min. Para obter a uma eluição total do RNA foi transferido o filtro RNA spin para o tubo de eluição, Adicionador 20µL de tampão de eluição (Elution Buffer R) diretamente sobre a membrana. Foram incubados por 3 minutos, centrifugados em 11.000 rpm por 90 segundos, obtendo volume final de 20µL.
4.3.4.3. Quantificação do RNA em NanoDrop
Após a extração, procedeu-se à avaliação da quantidade do RNA total através da
quantificação espectofotométrica a 260/230nm no espectofotómetro NanoDrop ND-100 (NanoDrop technologies, Wilmington, USA), de acordo com as instruções do fabricante. Foi utilizado 1 µl de cada amostra para esta avaliação. As amostras de RNA foram armazenadas a -80ºC.
4.3.4.4. Tratamento de Amostras com DNAse
O RNA de cada um dos pools de embriões foi tratado com DNase I recombinante, RNase-free (From bovine pancreas, expressed in Pichia pastori, Enzyme solution with 10×
Incubation Buffer, Deoxyribonuclease I, EC 3.1.21.1,Cat. No. 04 716 728 001, ROCHE®), conforme descrito abaixo.
A DNase I recombinante é uma forma recombinante da DNase I de pancreas bovino, expressa em Pichia pastoris. É uma glicoproteína com peso molecular de 39kD (Kilo
Daltons). A enzima recombinante é produzida sem o uso de células animais ou outros materiais derivados de animais. A DNase I é uma endocluease específica de DNA que hidrolisa o fosfodiester de fitas duplas e simples de DNA em uma mistura de oligo e mononucleotídeos.
A reação foi preparada da seguinte forma. 20µl de cada uma das amostras são adicionados à uma mistura composta por 2µl de Tampão (10X), 1 µl de DNaseI recombinante.
36
4.3.4.5. Preparo dos cDNA das amostras
O cDNA das amostras deste experimento foram produzidos utilizando High-Capacity cDNA Reverse Transcription Kit da Apllied Biosystems® conforme descrito abaixo.
O kit High-Capacity cDNA contêm todos os reagentes necessários para transcrição reversa (RT), a partir do total de RNA para uma fita simples de cDNA usando uma reação de 20 µl.
Inicialmente foi preparado o Master Mix para a reação (2X Reverse Transcription Master Mix). Em seguida foram adicionados os RNA de cada uma das amostras ao Master Mix de Concentração 1X. Após esta etapa a transcrição reversa foi realizada em termociclador. As amostras de cDNA foram retiradas do termociclador e armazenadas em Freezer -20º C .
Preparo do (Master Mix)
Todos os componentes da reação foram mantidas em gelo até o início da mistura. 4 µl de Tampão RT 10X são misturados com 1,6 µl de dNTP Mix, 4,0 µl de 10x RT Randon Primers, 2 µl de “Multiscribe” transcriptase reversa, 2 µl de inibidor de Rnase, 6,4 µl de H2O nuclease-free, performando um total de reação final de 40 µl para cada amostra.
Preparo da Reação
Foram pipetados 20 µL of 2✕ RT da master mix em cada eppendorf onde foram incluídos 20 µL de amostra RNA em cada eppendorf, estes foram selados e centrifugados em um spin para eliminar bolhas. As amostras foram mantidas em gelo até o momento de iniciar a termociclagem.
Condições de Termociclagem
37 até sua transferência para o Freezer com temperatura de -20º C onde foram mantidas até o preparo das reações de PCR em Tempo Real.
4.3.4.6. TaqMan - Real Time PCR (Reação de Polimerase em Cadeia em Tempo Real)
Antes da reação de qRT-PCR com o cDNA recém-síntetizado, as seguintes etapas devem ser realizadas: Seleção de um padrão interno adequado; Desenho de primers e sondas de genes alvo; Validação da eficiência da amplificação do gene alvo e padrão interno; Otimização de primers;
Preparação dos reagentes
A TaqMan ® Gene Expression (20 ✕) e as amostras de cDNA foram descongeladas em gelo, ressuspendidas gentilmente em vortex, e centrifugados brevemente. Os reagentes foram misturados para formar um Master Mix.
Cálculo do número de reações
Ocálculo do número de reações para cada ensaio foi realizado conforme orientação da Applied Biosystems, executando três repetições de cada reação.
Preparação da Reação Mix de PCR
Para cada amostra (para ser executado em triplicata), na seqüência da pipetagem em um tubo nuclease-free de microcentrífuga de 1,5 mL.
Para uma reação final de 20µL a reação de PCR foram utilizados por reação 1µL de 20X TaqMan Gene Expression Assays, 10 µL de 2X TaqMan Gene Expression Master Mix, 5µL de H20 Rnase-Free, e 4 µL de cDNA. Após inclusão de todos os ítens, os tubos foram invertidos várias vezes para misturar os componentes da reação e centrifugadas brevemente.
Após estes passos, 20 µL da reação de PCR mix em cada um dos poços da placa de 96 poços. A placa foi selada e centrifugada antes de ser levada ao equipamento.
38 motivo foram utilizados 2 µL do volume de cada uma das amostras em reações em uma concentração 2:20 µL. A concentração escolhida foi 1:1.
Após esta padronização foi preparada uma placa com 90 poços onde foram avaliadas as expressões dos Genes em Triplicata para cada uma das fases pré-implantacionais selecionadas.
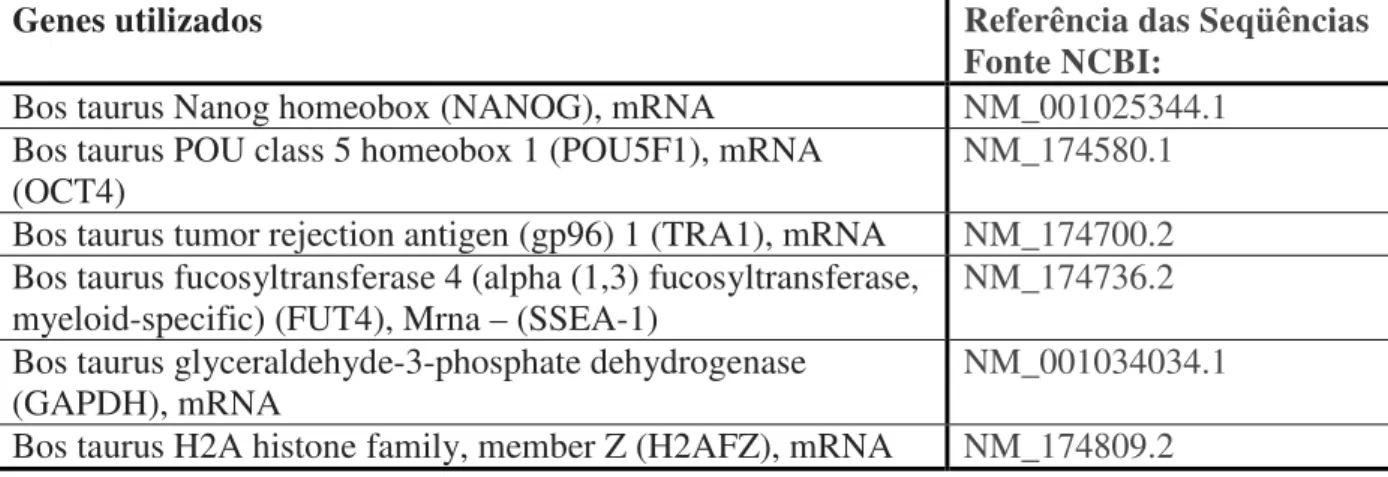
Na tabela 02, abaixo, estão descritas as referências dos genes que foram utilizados durante as avaliações.
Tabela 02: Referências dos genes utilizados
Genes utilizados Referência das Seqüências
Fonte NCBI:
Bos taurus Nanog homeobox (NANOG), mRNA NM_001025344.1
Bos taurus POU class 5 homeobox 1 (POU5F1), mRNA (OCT4)
NM_174580.1
Bos taurus tumor rejection antigen (gp96) 1 (TRA1), mRNA NM_174700.2
Bos taurus fucosyltransferase 4 (alpha (1,3) fucosyltransferase, myeloid-specific) (FUT4), Mrna – (SSEA-1)
NM_174736.2
Bos taurus glyceraldehyde-3-phosphate dehydrogenase (GAPDH), mRNA
NM_001034034.1
Bos taurus H2A histone family, member Z (H2AFZ), mRNA NM_174809.2
Reação de MIX
As reações de Mix foram preparadas conforme cálculos abaixo padronizados pelo sistema StepOnePlus™ -Real-Time PCR Systems.
Para cada amostra foram incluídas em tubos ependorf específicos: 5,0 µL de Máster Mix (20X); 0,50 µL de Mix de Ensaio; 1 µL da amostra; 3,50 µL de H2O perfazendo um volume final de 10 µL.
Diluição de amostras
39
4.3.4.7. Análise de Dados
40
5. RESULTADOS E DISCUSSÃO
5.1. Análise de CT
Apesar de não se esperar obter quantificação de RNA em amostras de pools de embriões bovinos, foi possível observar em NANODROP a presença de volume reduzido de RNA nas 5 amostras. Tendo em vista o aumento progressivo do volume de RNA extraído, as análises de CT demonstram a existência de variações entre os genes e as fases selecionadas ao compará-los de forma cruzada. O gráfico abaixo demonstra que os 6 genes escolhidos apresentam graus diferentes de expressão em cada uma das fases 5 fases de coleta de embriões.
Gráfico 01: Gráfico de CT comparativo por gene
Graficos de CT comparativo por gene cada um dos 5 grupos de amostras. As cores azul escuro, vermelho, verde, roxo e azul claro representam as 5 fases de seleção amostral (4C, 16C, M, BL, BX)
41 4 (BL)de amostras o menor Ct com o Gene OCT4 e maior Ct com o gene SSEA-1. E no grupo 5 (BX) de amostras o menor Ct com o Gene OCT4 e maior Ct com o gene SSEA-1.
Estes dados demonstram a existência de uma grande variação da expressão de cada gene durante as 5 fases experimentais. Assim cada um destes genes apresenta maior ou menor expressão gênica, conforme a mudança de estágio de desenvolvimento embrionário.
Como o ciclo limite (CT) é determinado a partir de um log-linear do sinal de PCR versus o número de ciclos, o CT é exponencial e não um termo linear. Por esta razão, qualquer apresentação estatística utilizando o CT pode ser utilizada para comparar a variação entre amostras em replicata (Livak and Schmittgen, 2001).
Em 2009, Ushijima e colaboradores criaram uma classificação de mudanças morfológicas de embriões bovinos segundo o número de clivagens. Neste trabalho, descreveu-se o aumento proporcional do número de células durante o dedescreveu-senvolvimento embrionário. Assim, durante a Fase 1 (4C) os embriões podem apresentar em média 3 a 4 células, enquanto que na Fase 2 (16C) apresentam em média 8 a 16 células. Na Fase 3 (M) apresentam em média 16 a 32 células, na Fase 4 (BL) os embriões possuem em média 97 a 129 células, e na Fase 5 (BX) seriam classificados embriões de em média 142 a 193 células (Ushijima et al.,
2009).
A partir da obtenção de CT as demais análises foram realizadas para comparação da expressão de genes alvo ao gene de referência. A analise de expressão de genes alvo é realizada comparando o desvio entre amostras padrões estabelecidas e os genes de referência e genes alvo, ou amostras de referência e amostras alvo trabalhando com genes de referência (Pfaffl, 2001).
5.2. Expressão Relativa
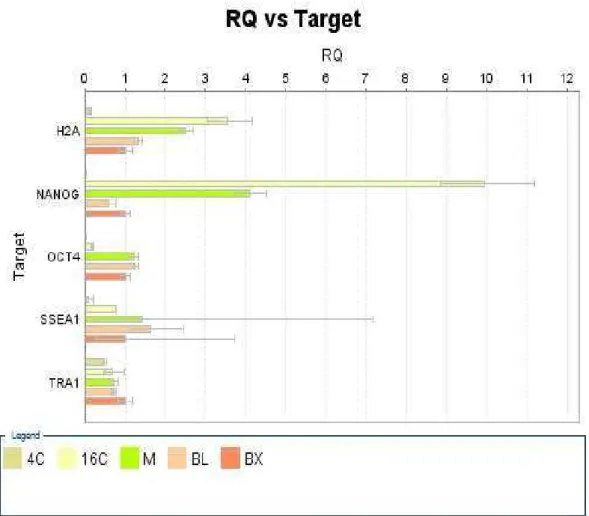
Para escolha dos padrões foi avaliada a Expressão Relativa das amostras entre os genes alvo durante as fases embrionárias, conforme a tabela abaixo. O gene GAPDH foi escolhido como Gene Padrão devido ao seu padrão de expressão ao comparar-se com os demais genes, garantindo a possibilidade de comparação em expressão relativa. O gene H2A, também devido às características descritas, foi utilizado com controle experimental devido a sua capacidade de expressão nas diversas fases experimentais.
42 observado que: o grupo de amostras NANOG apresentou expressão relativa em todos os 5 estágios de desenvolvimento escolhidos.
OCT4, SSEA1 e TRA1, também tiveram sua expressão relativa em maior ou menor grau expressas em todas as 5 fases experimentais. Desta forma, será possível observar no decorrer da discussão que existem diferenças significativas ao observar a expressão de cada gene individualmente em cada uma das fases, em bovinos da raça Nelore.
Gráfico 02: Expressão relativa entre os genes estudados nos diferentes estágios de desenvolvimento.
Gráfico de expressão relativa comparativo por gene cada um dos 5 grupos de amostras. As cores verde amarelo, verde limão, laranja e vermelho representam as 5 fases de seleção amostral (4C, 16C, M, BL, BX)
O GAPDH tem sido utilizado generalizadamente como padrão interno de RNA (Bustin, 2000), incluindo estudos da expressão gênica hepática em bovinos (Kobayashi et al.1999, Butler et al., 2003). O GAPDH também tem sido implicado em outros processos,
43 O GAPDH foi validado como gene de referência mais adequado para estudos em bovinos utilizando a metodologia de real-time PCR, podendo ser utilizado individualmente ou combinado como um índice para normalização dos dados (Robinson et al., 2007).
Na quantificação abaixo, realizada em logN, é possível observar com maior clareza a existência da expressão dos genes alvo em todos os 5 estágios selecionados.
Gráfico 03: Expressão relativa entre os genes estudados nos diferentes estágios de desenvolvimento (logN)
Gráfico de expressão relativa em logN comparativo por gene em cada um dos 5 grupos de amostras. As cores verde amarelo, verde limão, laranja e vermelho representam as 5 fases de seleção amostral (4C, 16C, M, BL, BX).
5.2.1. Variação da Expressão do Gene H2A
44 Gráfico 04 : Expressão relativa de H2A nos diferentes estágios de desenvolvimento
Gráfico de expressão relativa comparativo por gene cada um dos 5 grupos de amostras. As cores verde amarelo, verde limão, laranja e vermelho representam as 5 fases de seleção amostral (4C, 16C, M, BL, BX)
O grupo de amostras H2A 4C é o que apresentou a menor expressão relativa (ER). O grupo H2A 4C é regulado com menor expressão em comparação ao grupo controle. O grupo H2A 16C é regulado com maior expressão em comparação ao grupo controle. O grupo H2A M é regulado com maior expressão em comparação ao grupo controle. O grupo H2A BL é regulado com maior expressão em comparação ao grupo controle. Ou seja, a expressão de H2A foi inferior ao grupo controle BX somente no estágio 4C.
McGraw et al (2003) demonstraram, em um estudo da expressão gênica de H2A, que
durante o período pré-implantação em bovinos, ovócitos, embriões de 2 e 8 células e blastocistos mantém o DNA do gene H2A. Cada gene foi testado em um RT único, composto de 20 oócitos ou embriões em diferentes estágios, realizados em triplicata. Após a amplificação em RT, foram realizadas avaliações em PCR quantitativo utilizando o equivalente a dois oócitos ou embriões em cada estágio de divisão (McGraw et al., 2003).
45 sido utilizado em embriões produzidos por FIV (Corcoran et al., 2006) ou Transferência
Nuclear (Amarnath et al., 2007).
5.2.2. Variação da Expressão do Gene NANOG
A Expressão Relativa do gene NANOG nas amostras de cada uma das fases embrionárias selecionadas está descritas no gráfico abaixo. Sua expressão foi avaliada comparando os 5 estágios, gerando sua expressão relativa. Para as análises que seguem, a fase BX foi considerada padrão. Foi avaliada a expressão relativa entre as demais fases.
O grupo de amostras NANOG 4C foi considerado com menor expressão em comparação ao grupo controle. O grupo NANOG 16C é regulado com maior expressão em comparação ao grupo controle. O grupo NANOG M é regulado com maior expressão em comparação ao grupo controle. O grupo NANOG BL é regulado com menor expressão em comparação ao grupo controle.
Gráfico 05 : Expressão relativa de NANOG nos diferentes estágios de desenvolvimento
Gráfico de expressão relativa comparativo por gene cada um dos 5 grupos de amostras. As cores verde amarelo, verde limão, laranja e vermelho representam as 5 fases de seleção amostral (4C, 16C, M, BL, BX)
46 e epiblasto de embriões de 6.5 e 7.5 dias. Outro resultado também obtido durante a fase experimental deste trabalho demonstrou diferenças da literatura que até então afirmava que embriões não fertilizados, embriões de 2 a 8 células e culturas não expressariam estes genes (Mitsui et al., 2003; Chambers et al., 2003; Wang et al., 2003; Hart et al., 2004; Hatano et al., 2005).
A expressão avaliada nas fases iniciais, de 4 a 8 células, pode ser observada no gráfico 06. Para esta análise, a expressão relativa foi observada em logN.
Gráfico 06: Expressão relativa de NANOG nos diferentes estágios de desenvolvimento utilizando logN.
Gráfico de expressão relativa comparativo por gene cada um dos 5 grupos de amostras. As cores verde amarelo, verde limão, laranja e vermelho representam as 5 fases de seleção amostral (4C, 16C, M, BL, BX)
O gene NANOG é conhecido por caracterizar células pluripotentes em humanos e camundongos. Embriões de camundongos sem a presença de NANOG consistem em tecidos extraembrionários e sem epiblasto discernível (Mitsui et al., 2003). Além disso, a baixa
regulação de NANOG pode induzir, em camundongo, a diferenciação de linhagens extra-embrionárias (Hyslop et al, 2005, Hough et al., 2006). Assim, NANOG tem sido descrito