Programa de Pós-Graduação em Ciências Genômicas e Biotecnologia
ANÁLISE DE GENES DIFERENCIALMENTE EXPRESSOS NAS VIAS DE SINALIZAÇÃO (HEDGEHOG E WNT) EM CARCINOMAS BASOCELULARES HUMANOS
Brasília - DF
2012
Aluna: Natalia Gurgel do Carmo
NATALIA GURGEL DO CARMO
ANÁLISE DE GENES DIFERENCIALMENTE EXPRESSOS NAS VIAS DE SINALIZAÇÃO (HEDGEHOG E WNT) EM CARCINOMAS BASOCELULARES
HUMANOS
Dissertação apresentada ao Programa de Pós-Graduação Stricto Sensu em Ciências Genômicas e Biotecnologia da Universidade Católica de Brasília, para obtenção do título de Mestre em Ciências Genômicas e Biotecnologia.
Orientadora: Drª. Rosângela Vieira de Andrade Co-orientador: Dr. Robert Edward Pogue
Brasília
Ficha elaborada pela Biblioteca Pós-Graduação da UCB C287a Carmo, Natalia Gurgel do.
Análise de genes diferencialmente expressos nas vias de sinalização (Hedgehog e Wnt) em carcinomas basocelulares humanos. / Natalia Gurgel do Carmo – 2012.
96f. ; il.: 30 cm
Dissertação (mestrado) – Universidade Católica de Brasília, 2012. Orientação: Profa. Dra. Rosângela Vieira de Andrade
Coorientação: Robert Edward Pogue
1. Câncer de pele. 2. Genes. 3. Biotecnologia. I. Andrade, Rosângela Vieira de, oriente. II. Pogue, Robert Edward, coorient. III. Título.
Dissertação de autoria de Natalia Gurgel do Carmo, intitulada “Análise de genes diferencialmente expressos nas vias de sinalização (Hedgehog e Wnt) em carcinomas basocelulares humanos”
apresentada como requisito parcial para obtenção do grau de
Mestre em Ciências Genômicas e Biotecnologia da Universidade Católica de Brasília, em 16 de Abril de 2012, defendida e aprovada pela banca examinadora abaixo assinada:A professora Rosângela Vieira de Andrade por me orientar e estar sempre ao meu lado.
Ao professor Robert Edward Pogue pela paciência e pelos conhecimentos a mim compartilhados durante todo esse tempo.
A médica Simone Karst Passos pela parceria, pela ajuda, pelos conhecimentos compartilhados. A você meu muito obrigada!
Aos médicos Guilherme Fonseca e Roula Kozac pela parceria e atenção dados a minha pesquisa. Aos residentes, aos técnicos e funcionários do centro de Dermatologia do HRAN pela ajuda, pelo aprendizado e atenção dados a minha pessoa e ao meu trabalho.
Ao médico Luiz Sakamoto pelos conhecimentos e atenção.
Aos colegas do curso e da pós-graduação que me auxiliaram e me ajudaram a complementar meus conhecimentos aqui adquiridos. Meus sinceros agradecimentos em especial a Larissa Mendanha Lemos Cavalcante, Laureane Pavanelli, Nina Jardim Gasparini.
Aos meus alunos de Iniciação Científica, Luciana, Éster e Davi, pela ajuda prestada.
A Izavane Machado, pela ajuda e disposição em obter toda a parte experimental deste projeto, independente dos problemas que tivemos ao longo desses dois anos.
Ao Julien Correia, por todo suporte técnico, pela paciência para comigo e pelo esforço em fazer esse projeto dar certo.
Ao Luis Henrique Ferreira do Vale, por ser o motivo do meu caminho no campo acadêmico, obrigada por tudo que passamos!
A minha família, obrigada pelo apoio.
Ao CNPq, ao Laboratório de Ciências Genômicas e Biotecnologia da UCB, ao Centro de Nanotecnologia (CeNano) da UnB e a FAP-DF pelo suporte técnico e financeiro.
“Se a verdade revelada pelo DNA puder ser aceita sem temor, não há por
que não depositar esperanças naqueles que nos sucederão.”
CARMO, NG. Análise de genes diferencialmente expressos nas vias de sinalização (Hedgehog e Wnt) em carcinomas basocelulares humanos. 2012. 94 Folhas. Mestrado em Ciências Genômicas e Biotecnologia. Universidade Católica de Brasília, Brasília.
As vias de sinalização Hedgehog (HH) e Wingless-Int (Wnt) estão relacionadas com os processos de proliferação celular, diferenciação, angiogênese, remodelamento da matriz celular e homeostase de células tronco. Na via de sinalização HH várias mutações têm sido identificadas, incluindo mutações por perda de função no gene
ptch1 e mutações por ganho de função no gene smo. O carcinoma basocelular (CBC) é
o câncer de pele mais incidente entre os cânceres conhecidos e apresenta múltiplos fatores de risco. Contudo, ainda não é possível associar a ativação das vias de sinalização HH e Wnt com os diferentes subtipos histológicos do CBC. Como a progressão tumoral é um processo de várias etapas, o estudo de genes relacionados a estas vias de sinalização permitirá um melhor entendimento da carcinogênese do CBC. Neste contexto, o objetivo deste trabalho foi analisar genes diferencialmente expressos, relacionados às vias de sinalização HH e Wnt, em amostras de pacientes com CBC nodular e superficial comparados entre si e com o tecido de pele normal (sem neoplasia). As amostras foram coletadas a partir de cirurgias realizadas no centro de Dermatologia do Hospital Regional da Asa Norte. Primeiramente foi necessário padronizar a extração do RNA, uma vez que as amostras apresentaram peso inferior a 0,03g e as técnicas descritas para a extração não foram eficientes para uma obtenção de RNA de qualidade. Para a análise da expressão gênica, 84 genes foram selecionados e validados por PCR Array. Destes, 36 genes apresentaram um perfil de expressão gênica alterada quando comparados os três grupos. Para a análise estatística, o valor-P <0,005, a correlação e o número de cópias foram considerados. Comparado às expressões gênicas diferenciais nas amostras de CBC nodular em relação ao CBC superficial 8 genes (bmp5, hhip, otx2, prky, ptch1, ptch2, wnt2, zic1)
apresentaram-se superexpressos e 9 genes (csnk1a1l, dhh, ihh, ptchd2, wnt16, wnt3a, wnt7a, wnt8a, wnt9b) apresentaram-se reprimidos; comparado às amostras de CBC
nodular em relação ao controle sem neoplasia, 8 genes (foxe1, c6orf138, hhip, lrp2, otx2, prky, ptch1, ptch2) apresentaram-se superexpressos e 26 genes (bmp2, bmp4, cdon, csnk1a1l, dhh, erbb4, fgfr3, ihh, kctd11, ptchd1, ptchd2, shh, wif1, wnt10a, wnt10b, wnt16, wnt2b, wnt3, wnt3a, wnt4, wnt7a, wnt8a, wnt9b, zic2) apresentaram-se
reprimidos; e, comparado as amostras de CBC superficial em relação ao controle sem neoplasia, 3 genes (foxe1, lrp2, ptch2) apresentaram-se superexpressos e 14 genes
(bmp2, bmp4, bmp5, cdon, erbb4, fgfr3,wif1, wnt10a, wnt16, wnt2, wnt2b, wnt3, wnt4, wnt7b) apresentaram-se reprimidos.Uma representação gráfica localizando e inferindo
Hedgehog (HH) and Wingless-Int (Wnt) signaling pathway are related to the process of cellular proliferation, differentiation, angiogenesis, matrix remodeling and homeostasis of stem cells. Over the years, several mutations in HH signaling pathway have been identified, including the loss of function mutations in ptch1 and the gain of
function mutations in smo. Basal cell carcinoma (BCC) is the most common cancer and
has multifactorial risks. Nevertheless, it is still not possible to associate the activation of HH signaling pathway for BCC’s different histological subtypes. As tumor progression is a multistep process, the study of genes related to those pathways will allow a better understanding of BCC’s carcinogenesis. In this context, the objective of this work was to analyze differential genes expression in HH and Wnt signaling pathway in patients with superficial and nodular basal cell carcinoma compared between them and with a normal skin tissue. The samples were collect from surgery at the center of Dermatology at Hospital Regional da Asa Norte. Firstly, it was necessary standardize RNA extraction. The weight of the samples was less than 0,03g and the techniques described for the extraction were not efficient to obtaining a good RNA. Gene expression analysis was done by 84 genes that were selected and validate by PCR Array. From those, 36 genes showed differential gene expression when it was compared the 3 groups. For the statistic analysis were considered p-value, correlation method and fold regulation. Comparing nodular BCC to superficial BCC, 8 genes (bmp5, hhip, otx2, prky, ptch1, ptch2, wnt2, zic1) were upregulated and 9 genes (csnk1a1l, dhh, ihh, ptchd2, wnt16, wnt3a, wnt7a, wnt8a, wnt9b) were downregulated; comparing nodular BCC to control
without cancer, 8 genes (foxe1, c6orf138, hhip, lrp2, otx2, prky, ptch1, ptch2) were
upregulated and 26 genes (bmp2, bmp4, cdon, csnk1a1l, dhh, erbb4, fgfr3, ihh, kctd11, ptchd1, ptchd2, shh, wif1, wnt10a, wnt10b, wnt16, wnt2b, wnt3, wnt3a, wnt4, wnt7a, wnt8a, wnt9b, zic2) were downregulated; and comparing superficial BCC to control
without cancer, 3 genes (foxe1, lrp2, ptch2) were upregulated and 14 genes (bmp2, bmp4, bmp5, cdon, erbb4, fgfr3,wif1, wnt10a, wnt16, wnt2, wnt2b, wnt3, wnt4, wnt7b)
were downregulated. After these findings, a graphic representation localizing and inferring a possible regulation of the differential gene expression was made. The data reinforces literature of basal cell carcinoma and its subtypes and corroborate it. Studies on genetic alterations involved in tumorigenesis are important for the molecular characterization of a tumor. It may provide useful tools for the development and the identification of molecular markers aimed at Hedgehog and Wnt signaling pathways.
FIGURA 1.SUBTIPOS DE CARCIONOMA BASOCELULAR: ... 20 FIGURA 2.VIA DE SINALIZAÇÃO HEDGEHOG. ... 24 FIGURA 3.REPRESENTAÇÃO DA ATIVAÇÃO E REPRESSÃO DA VIA DE SINALIZAÇÃO HEDGEHOG 25 FIGURA 4.ILUSTRAÇÃO GERAL DA VIA WNT. ... 28 FIGURA 5.ANÁLISE DA QUALIDADE DO RNA. ... 43 FIGURA 6. DETERMINAÇÃO DO NÚMERO DE ALVOS DE REFERÊNCIA A SEREM UTILIZADOS NAS ANÁLISES UTILIZANDO O SOFTWARE GENORM (QBASEPLUS). ... 46 FIGURA 7.GRÁFICO DE DISPERSÃO ENTRE GRUPO CBC NODULAR EM RELAÇÃO AO CONTROLE (SEM NEOPLASIA) ... 48 FIGURA 8.GENES SUPEREXPRESSOS EM CBC NODULAR EM RELAÇÃO AO CONTROLE. ... 50 FIGURA 9.GENES MENOS EXPRESSOS EM CBC NODULAR EM RELAÇÃO AO CONTROLE. ... 52 FIGURA 10. GRÁFICO DE DISPERSÃO ENTRE GRUPO CBC SUPERFICIAL EM RELAÇÃO AO CONTROLE. ... 54 FIGURA 11.GENES SUPEREXPRESSOS EM CBC SUPERFICIAIS EM RELAÇÃO AO CONTROLE. ... 55 FIGURA 12.GENES REPRIMIDOS EM CBC SUPERFICIAIS EM RELAÇÃO AO CONTROLE. ... 57 FIGURA 13. GRÁFICO DE DISPERSÃO ENTRE GRUPO CBC NODULAR EM RELAÇÃO AO CBC
SUPERFICIAL.. ... 59 FIGURA 14.GENES SUPEREXPRESSOS EM CBC NODULAR EM RELAÇÃO AO CBC SUPERFICIAL.
... 60 FIGURA 15.GENES MENOS EXPRESSOS EM CBC NODULAR EM RELAÇÃO AO CBC SUPERFICIAL.
... 62 FIGURA 16.GENES COM VALOR-P SIGNIFICATIVOS EM TODOS OS GRUPOS ESTUDADOS. ... 63 FIGURA 17.VIA GÊNICA DO CARCINOMA BASOCELULAR. ... 78 FIGURA 18. ESQUEMA REPRESENTANDO GENES DIFERENCIALMENTE EXPRESSOS EM CBC
TABELA 1. INCIDÊNCIA DE CASOS NOVOS DE DIVERSOS TIPOS DE CÂNCER PARA HOMENS, SEGUNDO ESTIMATIVAS DO INCA(2012). ... 17 TABELA 2. INCIDÊNCIA DE CASOS NOVOS DE DIVERSOS TIPOS DE CÂNCER PARA MULHERES, SEGUNDO ESTIMATIVAS DO INCA(2012). ... 17 TABELA 3.DESORDENS HUMANAS ASSOCIADAS COM A DESREGULAÇÃO DA VIA HEDGEHOG. ... 26 TABELA 4. DESENHO REFERENTE À PLACA PCR ARRAY DA VIA DE SINALIZAÇÃO HH E DOS
GENES QUE CODIFICAM PROTEÍNAS ASSOCIADAS. EM ROSA, GENES RELACIONADOS À VIA SONIC HEDGEHOG E A VIA DE SINALIZAÇÃO DE HH. ... 39 TABELA 5.INTERPRETAÇÃO DO COEFICIENTE DE CORRELAÇÃO... 40 TABELA 6. DADOS CLÍNICOS DAS AMOSTRAS SELECIONADOS DOS PACIENTES SUBMETIDOS À
CIRURGIA. ... 42 TABELA 7.CONCENTRAÇÃO RNA(QUBIT). ... 43 TABELA 8. ANÁLISE DOS CDNA DAS AMOSTRAS SELECIONADAS PARA O PCR ARRAY POR
QPCR. ... 45 TABELA 9.GENES SUPEREXPRESSOS NO GRUPO CBC NODULAR EM RELAÇÃO AO CONTROLE. ... 49 TABELA 10.GENES MENOS EXPRESSOS DO GRUPO CBC NODULAR EM RELAÇÃO AO CONTROLE. ... 51 TABELA 11. GENES COM VALOR-P SIGNIFICATIVO (P<0,05) EM AMOSTRAS DE CBC NODULAR
EM RELAÇÃO AO CONTROLE. ... 53 TABELA 12. GENES SUPEREXPRESSOS DO GRUPO CBC SUPERFICIAL EM RELAÇÃO AO
CONTROLE. ... 55 TABELA 13. GENES MENOS EXPRESSOS DO GRUPO CBC SUPERFICIAL EM RELAÇÃO AO
CONTROLE. ... 56 TABELA 14.GENES COM VALOR-P SIGNIFICATIVO (P<0,05) EM CBC SUPERFICIAL EM RELAÇÃO
AO NORMAL. ... 58 TABELA 15. GENES SUPEREXPRESSOS COM VALOR-P SIGNIFICATIVO (P<0,05) EM CBC NODULAR EM RELAÇÃO AO CBC SUPERFICIAL. ... 60 TABELA 16. GENES MENOS EXPRESSOS COM VALOR-P SIGNIFICATIVO (P<0,05) EM CBC NODULAR EM RELAÇÃO AO CBC SUPERFICIAL. ... 61 TABELA 17.GENES COM VALOR-P SIGNIFICATIVO (P<0,05) EM CBC NODULAR EM RELAÇÃO AO
µg - Microgramas µL – Microlitros
CBC – Carcinoma basocelular
CBCnod – Carcinoma basocelular nodular CBCsup – Carcinoma basocelular superficial cDNA – DNA complementar
Ci – Fator de transição em Drosophila melanogaster
Ct – Cycle Threshold (ciclo limiar) DNA – Ácido desoxirribonucléico
DHH – Desert Hedgehog – morfogene de Hedgehog gDNA – DNA genômico
GLI – Proteína glioma
IHH – Indian Hedgehog – morfogene de Hedgehog INCA – Instituto Nacional do Câncer
HH – Proteína Hedgehog
HRAN – Hospital Regional da Asa Norte Kb – Kilo base
NRT – Controle da não-transcrição reversa PCR - Reação em cadeia da polimerase PPC – Controle positivo da PCR
PTCH – Receptor patched qPCR – PCR quantitativo RNA – Ácido ribonucléico
RTC – Controle da transcrição reversa
SHH – Sonic Hedgehog – morfogene de Hedgehog SMO – Proteína smoothened
1. CÂNCER DE PELE NÃO MELANOMA ________________________________ 16
2. CARCINOMA BASOCELULAR ______________________________________ 18
3. A VIA DE SINALIZAÇÃO HEDGEHOG ________________________________ 22
4. A VIA DE SINALIZAÇÃO WINGLESS-INT _____________________________ 27
5. JUSTIFICATIVA __________________________________________________ 30
6. OBJETIVOS _____________________________________________________ 31
6.1. GERAL ___________________________________________________________ 31
6.2. ESPECÍFICOS _____________________________________________________ 31
7. DELINEAMENTO EXPERIMENTAL ___________________________________ 32
8. MATERIAL E MÉTODOS ___________________________________________ 33
8.1. COLETAS DE AMOSTRAS ___________________________________________ 33
8.2. PADRONIZAÇÃO E EXTRAÇÃO DE RNA _______________________________ 33
8.3. ANÁLISE DA QUALIDADE E QUANTIFICAÇÃO DO RNA __________________ 34
8.4. IDENTIFICAÇÃO DE CONTAMINAÇÃO POR DNA GENÔMICO (gDNA) _______ 35
8.5. SÍNTESE DE DNA COMPLEMENTAR (cDNA) ____________________________ 35
8.6. CONTROLE DE QUALIDADE DO cDNA ________________________________ 35
8.7. REPLICATA EXPERIMENTAL ________________________________________ 36
8.8. PCR Array – VIA DE SINALIZAÇÃO HEDGEHOG ________________________ 36
8.9. ANÁLISE DOS DADOS ______________________________________________ 39
9. RESULTADOS E DISCUSSÃO ______________________________________ 41
9.1. ANÁLISE DA EXPRESSÃO GÊNICA ___________________________________ 45
9.1.1. Seleção dos melhores candidatos dos genes constitutivos _________________________ 45 9.1.2. Análise diferencial de genes da via de sinalização Hedgehog e Wnt _________________ 47 9.1.3. Comparação dos genes diferencialmente expressos nos grupos CBC nodular, CBC
superficial e controle _____________________________________________________________ 63 9.1.4. Genes diferencialmente expressos das vias HH e WNT ___________________________ 66 9.1.5. Via de sinalização HH e Wnt em CBC _________________________________________ 77
–
PCR Array Hedgehog Humana _________________________________________ 86
1. CÂNCER DE PELE NÃO MELANOMA
O câncer de pele não melanoma é uma doença que acomete mais as populações de pele clara e possui uma forte correlação com a latitude uma vez que a exposição solar é um fator etiológico (Telfer, 1999; Bath-Hextall, 2007; Van der Pols, 2011). Outros fatores de risco como a sensibilidade da pele ao sol, as doenças imunosupressoras, a exposição ocupacional e os pacientes imunocomprometidos (como os transplantados renais) apresentam um maior risco para o desenvolvimento do câncer da pele não melanoma. Isto porque esses fatores exibem uma diminuição no controle carcinogênico da pele (Xu et al., 2012). O câncer de pele não melanoma é considerado o mais frequente entre todos os tipos de câncer que acometem o ser humano, inclusive no Brasil é o tipo de câncer mais incidente (INCA, 2012).
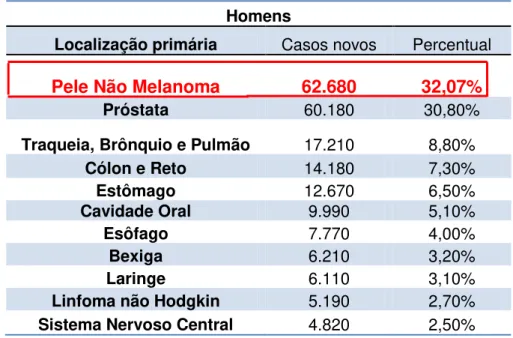
Em 2012, o Instituto Nacional de Câncer (INCA) estimou a quantidade de casos novos para os diversos tipos de câncer. O câncer de pele não melanoma é o que mais se destaca. Estimou-se para o Brasil 62.680 casos novos de câncer de pele não melanoma entre homens e 71.490 em mulheres, totalizando mais de 134.170 mil novos casos. Esses valores correspondem a um risco estimado de 65 casos novos a cada 100 mil homens e 71 para cada 100 mil mulheres. O câncer de pele não melanoma é o mais incidente em homens nas regiões Centro-Oeste (124/100 mil), Sul (80/100 mil) e Norte (38/100 mil), enquanto que nas regiões Sudeste (73/100 mil) e Nordeste (39/100 mil) é o segundo mais frequente. Nas mulheres é o mais frequente em todas as regiões, com um risco estimado de 109/100 mil na região Centro-Oeste, 91/100 mil na região Sudeste, 68/100 mil na região Sul, 43/100 mil na região Norte e 42/100 mil na região Nordeste (Tabela 1 e 2). No ano de 2010, segundo o INCA, o número de casos de câncer de pele não melanoma estimado pelo INCA foi de 53.410 entre homens e de 60.440 nas mulheres, totalizando mais de 113.850 casos. Observa-se assim o aumento desse câncer (mais de 30.000 mil novos casos) entre os anos de 2010 a 2012 (INCA, 2012).
subregistro devido ao subdiagnóstico. É recomendado, portanto, que as estimativas das taxas de incidência e dos números esperados de casos novos em relação a esse tipo de câncer sejam consideradas como estimativas mínimas (INCA, 2012).
Tabela 1. Incidência de casos novos de diversos tipos de câncer para homens, segundo estimativas do INCA (2012).
Tabela 2. Incidência de casos novos de diversos tipos de câncer para mulheres, segundo estimativas do INCA (2012).
Homens
Localização primária Casos novos Percentual
Pele Não Melanoma 62.680 32,07%
Próstata 60.180 30,80%
Traqueia, Brônquio e Pulmão 17.210 8,80%
Cólon e Reto 14.180 7,30%
Estômago 12.670 6,50%
Cavidade Oral 9.990 5,10%
Esôfago 7.770 4,00%
Bexiga 6.210 3,20%
Laringe 6.110 3,10%
Linfoma não Hodgkin 5.190 2,70%
Sistema Nervoso Central 4.820 2,50%
Mulheres
Localização primária Casos novos Percentual
Pele Não Melanoma 71.490 37,86%
Mama Feminina 52.680 27,90%
Colo do Útero 17.540 9,30%
Cólon e Reto 15.960 8,40%
Glândula Tireoide 10.590 5,60%
Traqueia, Brônquio e Pulmão 10.110 5,30%
Estômago 7.420 3,90%
Ovário 6.190 3,30%
Corpo do Útero 4.520 2,40%
Sistema Nervoso Central 4.450 2,40%
Classificam-se como câncer de pele não melanoma os tipos carcinoma basocelular e carcinoma epidermóide. O carcinoma basocelular pode ser do subtipo nodular, superficial, esclerosante, infiltrativo, micronodular, basoescamoso, infundibulocístico, fibroepitelioma e queratótico. O carcinoma epidermóide pode ser do subtipo invasivo, acantolítico, de células fusiformes, verrucoso e carcinoma epidermóide in situ ou Doença de Bowen (Colgan, Cappel, Pittelkow, 2011).
2. CARCINOMA BASOCELULAR
O carcinoma basocelular (CBC) é também conhecido por epitelioma basocelular ou basalioma. O CBC se origina das células basais da epiderme e surge predominantemente em áreas expostas a radiação ultravioleta, principalmente nos dois terços superiores da face, orelhas, nariz, lábios, sobrancelhas, não ocorrendo nas palmas das mãos, plantas dos pés e mucosas. Apresenta malignidade local e evolução crônica, podendo invadir e destruir tecidos adjacentes, incluindo ossos dependendo do subtipo do CBC (Sampaio, 2000; Schwartz, 2008). Apesar de apenas uma pequena proporção se tornar letal e vir a óbito, quando há a demora no diagnóstico podem ocorrer ulcerações e deformidades físicas graves em alguns casos. É mais frequente a partir da meia-idade (>40 anos) e em idosos (Sampaio, 2000). O CBC é o tipo mais incidente, representando 70-80% dos cânceres de pele não melanoma (Colgan, Cappel, Pittelkow, 2011).
compararam o CBC nodular com o do micronodular, sugerindo para o segundo um maior potencial destrutivo.
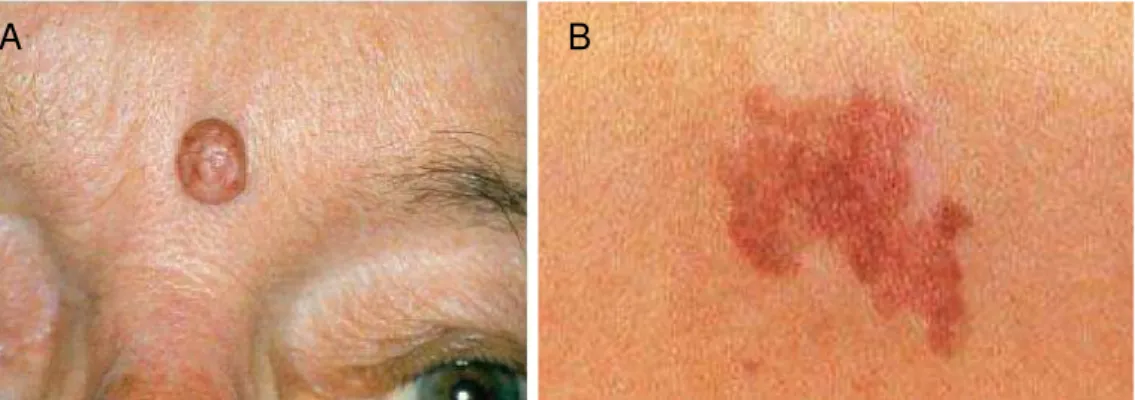
O CBC nodular é o subtipo mais comum, representando aproximadamente 78% de todos os CBC biopsiados (Colgan et al., 2011; Miot e Chinem, 2011). Geralmente é uma lesão única e frequentemente surge em áreas como cabeça e pescoço. Acomete em maior número homens e pessoas idosas. Caracteriza-se como pápula ou nódulo com aspecto perolado, muitas vezes com telangiectasias de padrão característico à dermatoscopia, que posteriormente se ulcera, podendo invadir os tecidos subjacentes (Miot e Chinem, 2011). Histologicamente, tem estrutura bem definida, com contornos precisos e massa neoplásica compacta, limitada por células dispostas em paliçadas. Em torno da lesão, observa-se uma retração da mucina, e o estroma tende a ser fino ao redor do tumor (Santos, 2010). As lesões de CBC nodular apresentam uma taxa de crescimento lento com seu tamanho limitado e quando tratadas prontamente apresentam baixa recorrência. Contudo, esse tipo de câncer pode ser localmente destrutivo quando não tratado, principalmente em áreas mais sensíveis do rosto como nos olhos e orelhas por apresentar maior capacidade de penetração no tecido (Colgan et al., 2011) (Figura 1 A).
Figura 1. Subtipos de Carcionoma Basocelular: (A) Carcinoma basocelular nodular. (B) carcinoma basocelular superficial (Schwartz, 1982; Disponível em http://www.neon.med.br, respectivamente).
A escolha do procedimento terapêutico para o tratamento destas neoplasias depende do tipo histológico, da localização, do tamanho e da profundidade do tumor. A excisão e sutura, conhecida também por exérese cirúrgica, geralmente é a primeira escolha no tratamento dessas neoplasias (Sampaio, 2000; Schwartz, 2008). Sexton et al. (1990), em estudo histológico com 1.039 CBC, estudaram as margens cirúrgicas dos CBC e chegaram à conclusão de que os tipos nodular e superficial, por terem arquitetura mais compactada, obtêm maior sucesso durante a excisão cirúrgica. No campo da cirurgia, uma opção para o tratamento de CBC nodular é a cirurgia micrográfica de Mohs que é uma técnica meticulosa (cirurgia com controle microscópico de margens). Curetagem e eletro-cirurgia são técnicas utilizadas para lesões pequenas e superficiais (Hacker et al., 1993). A criocirurgia é utilizada também nesses casos, porém é considerada como tratamento paliativo em tumores inoperáveis, pacientes com múltiplas lesões em face e tronco, doentes que não apresentam condições clínicas e/ou que se recusam a submeter-se à cirurgia (Sampaio, 2000). Pode-se indicar o uso de imunomoduladores como Imiquimod ou 5-fluorouracil tópico e também radioterapia. Outras terapias alternativas, como a terapia fotodinâmica, vêm ganhando espaço no sistema de saúde para o tratamento de ceratoses actínicas e CBC superficiais.
Vários fatores de risco estão associados ao CBC, como fatores genéticos, sexo, radiação solar, fatores ligados à dieta, radiação ionizante, exposição ao arsênico, pacientes imunocomprometidos, queimaduras, tabaco, ulcerações crônicas, locais de punção venosa, cicatriz de varicela, entre outras (van der Pols 2011; Colgan et al.,
2011; Gailani e Bale, 1997; Einspahr, Stratton et al., 2002; Iwasaki, Srivastava et al
2010). Os CBC esporádicos causados por eventos extrínsecos representam a maioria dos casos diagnosticados (Martinez, 2006). A predisposição hereditária ao CBC é mais rara e faz parte de algumas síndromes, como os indivíduos com albinismo, xeroderma pigmentoso, síndrome do nevo basocelular ou síndrome de Gorlin, síndrome Rasmussen, síndrome Rombo, síndrome Bazex-Christol-Dupre e doença de Darier (de Zwaan, Haass, 2010).
Segundo a patogênese do CBC é sugerido que os CBC emergem de células-troncos precursoras de queratinócitos geneticamente alteradas no folículo piloso, na glândula sebácea ou em células basais interfoliculares. Essas mutações aparecem espontaneamente, contudo elas podem ser associadas à predisposição genética como no caso das síndromes. O mecanismo mais frequente de indução de mutação somática é causado pela luz solar. A radiação ou exposição solar é o principal fator ambiental implicado na indução deste tipo de câncer, possui ação deletéria e cumulativa na pele (Xu et al., 2012).
Para o CBC, as mutações melhores caracterizadas são no gene p53 e ptch,
ambos possuem papel importante na supressão tumoral (Colgan et al., 2011). Outras alterações gênicas associadas à susceptibilidade em desenvolver o CBC são vistas nos genes smo, gli1, gpr49, cyp2d6, gst-t1, receptor da vitamina D, tnf e 14-3-3sigma, a
qual é controlada pelo p53 (O’Driscoll et al., 2006; Athar, Tang et al., 2006; Tanese,
Fukuma et al., 2008; Iwasaki, Srivastava et al., 2010). A via de sinalização melhor
associada à patogênese do CBC é a via sonic-hedgehog-patched-smoothened, ou simplesmente, via de sinalização Hedgehog. Recentemente, tem-se evidenciado que a via TGF-β/Smad está relacionada também na patogênese do CBC (Boukamp, 2011).
3. A VIA DE SINALIZAÇÃO HEDGEHOG
A via de sinalização Hedgehog (HH) é uma via de transdução de sinal importante na fase embrionária. Entretanto, na fase adulta, a via é geralmente quiescente e a sua ativação está implicada em diversos tipos de cânceres, como do trato digestivo, pancreático e de pulmão, atingindo tecidos de diferentes origens, por exemplo, pele, cérebro e músculo (Daya-Grosjean e Couve-Privat, 2005; Athar, Tanget al., 2006; Maun et al., 2010). O nome hedgehog é derivado da aparência do embrião
mutante da mosca de fruta Drosophila melanogaster, a qual é coberta por cerdas e,
neste sentido, se assemelha a um pequeno mamífero conhecido por ouriço (do inglês,
hedgehog) (Bale, 2002). Os componentes da via de sinalização HH foram
primeiramente caracterizados em Drosophila, e são eles: um ligante secretado (Hh),
um receptor (Ptc) e um fator de transcrição (Ci). As proteínas Hh de Drosophila
trabalham em conjunto com outras moléculas para estabelecer as estruturas básicas de um embrião (Jenkins, 2009).
Evolutivamente, estes componentes descritos acima são conservados dos insetos até os mamíferos. Em mamíferos, os componentes principais da via, diferentemente da Drosophila, são: a família HH, constituídos pelos genes Indian
Hedgehog (ihh), Desert Hedgehog (dhh) e Sonic Hedgehog (shh) (Iwasaki e Srivastava
et al., 2010); por dois receptores de membrana patched 1 e patched 2; pelo transdutor
de sinal de membrana smoothened (smo) e três fatores de transcrição gli1, gli2 e gli3
(Bale, 2002; Sanchez-Camacho, Rodriguez et al., 2005; Shimokawa, Svard et al.,
2007). O mecanismo de ativação da via de sinalização HH em Drosophila se diferencia
dos mamíferos devido à presença desses outros componentes apresentados.
Os três morfogenes de HH (ihh, dhh e shh) são intercambiáveis para a função de
induzir a transdução de sinal de HH, embora eles se distingam na forma em que se ligam ao PTCH1, e, consequentemente, no potencial para induzir uma resposta também (Pathi et al., 2001). Segundo Mann e Beachy (2010), as proteínas HH são
processadas, modificadas e liberadas de forma similar. Como funções, o gene ihh
regula a diferenciação de cartilagem, coordenando a proliferação e maturação de condrócitos durante o desenvolvimento do esqueleto endocondral (Lai e Mitchell, 2005). O gene dhh participa na espermatogênese e no desenvolvimento de células de
padronização do tubo neural e somitos-ventral adjacentes na linha média e atua concomitantemente no desenvolvimento da simetria esquerda-direita, participa da atividade de polarização nos membros e também desempenha um papel na morfogênese de várias partes do corpo (cérebro, esqueleto axial, pulmão, membros, cabelo, pele, dentes) (Bale e Yu, 2001; Athar, Tang et al., 2006; Iwasaki, Srivastava et
al., 2010). Na pele, a via de sinalização por SHH é crucial para a manutenção da população de células-tronco (Athar, Tanget al., 2006), além de ser o responsável pelo
controle do desenvolvimento dos folículos pilosos e de glândulas sebáceas (Iwasaki, Srivastavaet al., 2010).
Existem diversos homólogos patched em humanos, contudo os mais estudados são os genes ptch1 e ptch2. Entre 30% e 75% dos casos relatados de CBC
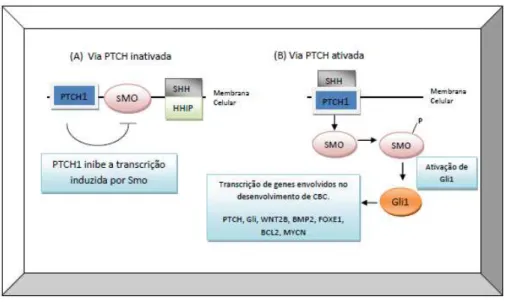
esporádicos, estes são associados à mutação no gene patched (Donavan, 2009). A proteína smoothened (SMO) também é fator essencial para ativação da via por ser um regulador positivo (Alcedo et al. 1996; van den Heuvel and Ingham, 1996). A liberação de SMO resulta na ativação dos fatores de transcrição Gli (Glioma), e na expressão de diversos outros genes. Na família Gli, temos o Gli1, Gli2 e Gli3. O Gli1 funciona como um efetor positivo da via de HH. O Gli2 pode funcionar tanto como um regulador transcricional positivo quanto negativo, dependendo do comando pós-transcricional e modificações pós-traducionais emitido. Já o Gli3 age como um repressor transcricional. O funcionamento da via HH acontece quando ocorre a ligação de proteínas HH ao receptor de membrana PTCH. Este sinal libera a proteína inibidora SMO, permitindo que a transdução do sinal ocorra através da membrana da célula, no contexto do cílio primário (estrutura celular específica), o qual resulta na ativação de fatores de transcrições zinc-finger da família Gli e na expressão de genes que regulam HH. Na
Figura 2. Via de sinalização Hedgehog. (A) Na ausência de SHH, SMO é “sequestrada” por PTCH1, receptor de membrana das células basais da epiderme, não ocorrendo a sinalização intracelular. (B) Na presença de SHH, esta se liga ao PTCH1, o que por sua vez libera SMO. A liberação de SMO ativa Gli1, que age na transcrição de diversos genes envolvidos na progressão tumoral de CBCs (Adaptado de Martinez et al., 2006).
A sinalização de HH é modulada por receptores de membrana celular como os antagonistas Cdon, Boc e GAS1, bem como de HHIP, uma proteína que interage com HH (Maun, 2010). O PTCH tem como mecanismo alternativo a possibilidade de sequestrar proteínas HH e limitar sua difusão, permitindo assim, o controle do alcance espacial da atividade de HH. O grande agente supressor dependente de HH é o supressor de fuso Su(fu), o qual retém proteínas Gli no citoplasma. Quando ocorre a perda da função de Su(fu), a via é potencialmente ativada devido a ligação independente à via (Bale, 2002; Jenkins, 2009).
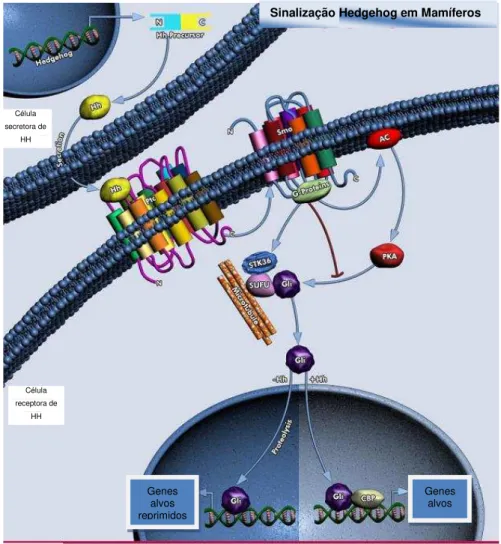
Os mecanismos reguladores da via de sinalização HH são múltiplos, destacando-se as modificações lipídicas covalentes nas proteínas HH. É importante ressaltar que a localização celular dos componentes na via possuem um papel importante na ativação na via de sinalização HH. Por exemplo, em Drosophila, o SMO
adentra em direção ao cílio onde interage com proteínas Gli que subsequentemente entram no núcleo(Figura 3)(Kim, J. et al., 2010).
Figura 3. Representação da ativação e repressão da via de sinalização Hedgehog (adaptado do SABiosciences).
A partir da elucidação da via de sinalização de HH em Drosophila, a sinalização
por HH tem sido correlacionada com diversos processos do desenvolvimento em vertebrados (Jenkins, 2009). Contudo, relata-se que não se sabe claramente como os sinais são transmitidos de HH para o PTCH e para o SMO. Novos estudos indicam que a via de sinalização HH não deve ser conservada totalmente entre vertebrados e invertebrados (Kim, J. et al., 2010). Denef et al., (2000) observou que a habilidade de PTCH em inibir o excesso estequiométrico de SMO sugere que a inibição não ocorra através do contato direto entre PTCH e SMO (Denef et al., 2000; Ingham et al., 2000; Taipale et al., 2000).
Sinalização Hedgehog em Mamíferos
Célula secretora de
HH
Célula receptora de
HH
Genes alvos reprimidos
Daya-Grosjean e Couve-Privat (2005) evidenciaram superexpressões de ptch
em CBC esporádicos, em pacientes com a Síndrome de Gorlin-Goltz e pacientes com xeroderma pigmentoso (XP), indicando que a ativação da via de sinalização HH por mutação ou atividade alterada de um ou mais membros devem ocorrer em CBC. Outras malformações severas congênitas estão relacionadas com mutações na via de sinalização HH e são observadas em fenótipos como holoprosencefalia ou na síndrome de Gorlin-Goltz (Tabela 3) (Daya-Grosjean e Couve-Privat, 2005).
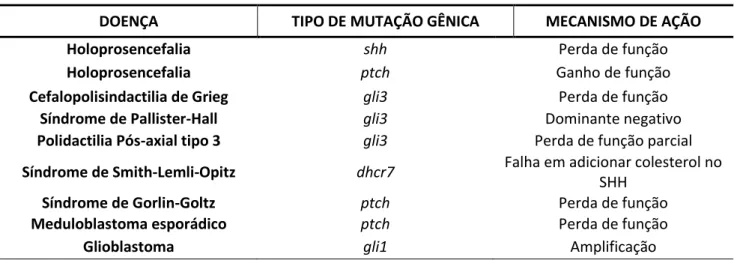
Tabela 3. Desordens humanas associadas com a desregulação da via hedgehog.
DOENÇA TIPO DE MUTAÇÃO GÊNICA MECANISMO DE AÇÃO
Holoprosencefalia shh Perda de função
Holoprosencefalia ptch Ganho de função
Cefalopolisindactilia de Grieg gli3 Perda de função
Síndrome de Pallister-Hall gli3 Dominante negativo
Polidactilia Pós-axial tipo 3 gli3 Perda de função parcial
Síndrome de Smith-Lemli-Opitz dhcr7 Falha em adicionar colesterol no SHH
Síndrome de Gorlin-Goltz ptch Perda de função
Meduloblastoma esporádico ptch Perda de função
Glioblastoma gli1 Amplificação
Muitas proteínas estão sendo estudadas visando bloquear a via de sinalização HH como uma alternativa de parar a progressão tumoral em tecidos neoplásicos. Entre elas, para se exemplificar, tem-se a proteína 5E1, que age bloqueando o sinal de HH e é utilizada como ferramenta em diversos estudos para identificar funções de HH em estudos in vitro e in vivo (Ericson et al., 1996; Fuse et al., 1999; Pepinsky et al., 2000).
Outras proteínas descritas por se ligar nas proteínas HH incluem Megalin, Vitronectina, Perlecan, Scube2 e Shifted, contudo as interações entre elas não são bem caracterizadas (Maun et al., 2010). Von Hoff et al., 2009, testou em pacientes em fase clínica I um inibidor de SMO chamado GDC-0449, para avaliar o perfil de segurança e os efeitos adversos da administração oral diária de GDC-0449 em pacientes metastáticos ou com carcinoma basacelular localmente avançado e outros tumores sólidos. O GDC-0449 foi descoberto por escaneamento de bibliotecas com tecnologia de alto rendimento (high-throughput screening) e logo após otimizando os
pâncreas. O trabalho de Von Hoff et al., 2009 mostrou que o GDC-0449 apresentou atividade anti-tumoral em 2 pacientes com carcinoma basocelular.
Atualmente, a ativação da via de sinalização HH está sendo melhor caracterizada e estudada para o CBC, porém ainda não foi possível associar em nível de expressão gênica os diferentes subtipos histológicos do CBC nem seus diferentes comportamentos in vivo (Martinez, 2006). Sabendo que o desenvolvimento tumoral é
um processo multifatorial, espera-se que a alteração de outros genes esteja envolvida na carcinogênese do CBC e de seus subtipos.
Em 2002, Tojo et al. avaliaram o padrão de expressão dos genes hip, shh e ptch
em CBC humanos em comparação aos mesmos genes em pele humana normal e outros tipos de tumores. Foi visto que a expressão de RNA mensageiro (RNAm) tanto em hip quanto em ptch estava aumentada em todos os CBC, enquanto que nenhuma
outra amostra de tumor de pele testada demonstrou um nível aumentado de tais RNAm quando comparado à pele normal. A transcrição de shh em contrapartida estava no
nível basal na maioria dos CBC. Esses resultados obtidos indicaram que tanto a expressão de hhip quanto de ptch está envolvida especificamente no desenvolvimento
de CBC. O trabalho também indica que a produção de hhip está conectada com a
expressão de ptch, mas não de SMO. Concluiu-se que os morfogenes de HH, o ptch, o smo e o hhip são capazes de modularem uma resposta ao sinal de HH através da
interação de proteínas HH em um loop de retroalimentação negativa regulatória. Yang (2008) corrobora com esses dados e relata que quase todos os CBC apresentam uma alta atividade na via de sinalização HH e estudos em modelos animais corroboram com a noção de que a ativação da sinalização de HH é suficiente para dirigir a tumorigênese em camundongos com CBC e células similares a CBC.
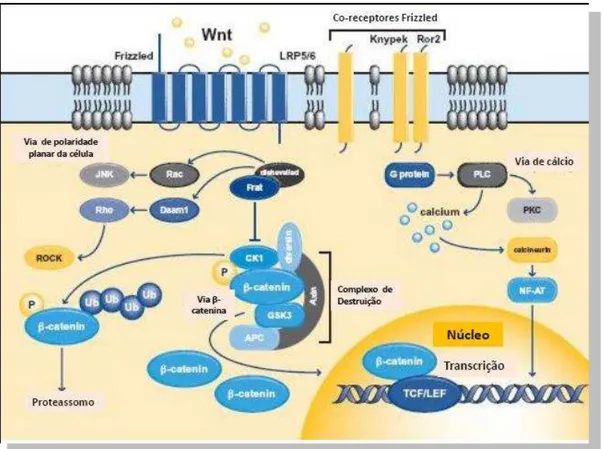
4. A VIA DE SINALIZAÇÃO WINGLESS-INT
receptores Frizzled (FZD) e pelo co-receptor de baixa densidade de proteína relacionada com lipoproteína (PRL)(Lustig,2003).
Os ligantes Wnt podem ser classificados em canônicos ou não-canônicos. Os Wnt canônicos, como exemplo o WNT3A, se ligam a receptores do tipo FZD, bem como a co-receptores LRP5/6, seguidos pelo recrutamento de complexas proteínas heteroméricas incluindo as Dishevelled, Axinas e receptores do complexo GSK3β. Os Wnt não-canônicos, como exemplo o WNT5A, se ligam a receptores FZD em conjunto com co-receptores alternados, por exemplo ROR1/2 e Ryk, o que causa mudanças independentes na β-catenina como a ativação de PKC. Esses dois tipos de ligantes, canônicos e não-canônicos, agem como antagonistas competitivos dos receptores compartilhados, como ilustrado na Figura 4 (Pourreyron et al, 2012). Além da via canônica, a via WNT pode ocorrer por meio da via de polaridade planar da célula e pela via Wnt/cálcio (Lustig, 2003).
A ativação da via Wnt, geralmente por mutações de proteínas dentro da própria via, conduz ao aumento da transcrição de genes importantes para o crescimento, proliferação, diferenciação, apoptose, estabilidade genética, migração e angiogênese nos tecidos (Pourreyron et al, 2012).
Durante o processo de desenvolvimento, a secreção de todos os ligantes Wnt, incluindo WNT5A está sujeita ao controle temporal e espacial preciso. Existe uma evidência crescente que a ativação da sinalização Wnt/β-catenina está associada com o desenvolvimento do tumor e/ou progressão tumoral (Lustig, 2003). A via de sinalização Wnt é uma das vias de sinalização que sofrem alterações importantes no CBC e auxiliam na progressão tumoral deste.
As vias de sinalização Wnt e Hedgehog são conhecidas por estarem envolvidas com o crescimento direto e padronização durante o desenvolvimento embrionário. Sabe-se que estas vias estão implicadas na regulação pós-embrionário de células-tronco em epitélios. Um papel patológico para as vias Wnt e HH emergiu a partir de estudos que mostraram uma elevada frequência de cânceres humanos específicos associados com mutações que constitutivamente ativam a resposta de transcrição de genes destas vias (Taipale, 2001). Neste trabalho, 84 genes foram selecionados e validados por PCR Array, destes, 36 genes (brtc, csnk1a1, csnl1a1l, csnk1d, csnk1e, csnk1g1, csnk1g2, ctnbb1, fbw11, prkaca, prkacb, prkacg, prkx, prky, sfrp1, siah1, wif1,
wnt1, wnt10a, wnt10b, wnt1, wnt 16, wnt2, wnt2b, wnt3, wnt3a, wnt4, wnt5a, wnt5b,
wnt6, wnt7a, wnt7b, wnt8a, wnt8b, wnt9a, wnt9b) participam da via de sinalização
5. JUSTIFICATIVA
Estudos sobre alterações genéticas envolvidos na tumorigênese são importantes para a caracterização molecular tumoral e podem oferecer ferramentas úteis para o desenvolvimento e identificação de marcadores moleculares. Como a progressão tumoral é um processo de várias etapas, o estudo de genes relacionados às vias de sinalização Hedgehog e Wnt permitem um melhor entendimento da carcinogênese do carcinoma basocelular e seus subtipos. Genes da via de sinalização Hedgehog, como
ptch e smo, além da família Gli têm contribuído substancialmente para a compreensão
genética do desenvolvimento destes carcinomas.
Portanto, um estudo envolvendo um número maior de genes envolvidos no funcionamento da via de sinalização Hedgehog e Wnt permitiria uma melhor compreensão do processo de proliferação tumoral, da invasão tumoral, do ciclo celular entre outras funções biológicas de importância para o desenvolvimento neoplásico. Por meio deste estudo, visa-se no futuro a identificação de potenciais marcadores moleculares para o diagnóstico dos subtipos dos carcinomas basocelulares.
6. OBJETIVOS
6.1. GERAL
Avaliar a expressão diferencial dos genes da via de sinalização Hedgehog e Wnt em pacientes com carcinoma basocelular nos subtipos nodular e superficial.
6.2. ESPECÍFICOS
1. Padronizar o método de extração de RNA a partir de tecido de pele humana.
2. Determinar o perfil de expressão dos genes envolvidos na via de sinalização Hedgehog e Wnt em carcinoma basocelular nodular e superficial por PCR Array.
7. DELINEAMENTO EXPERIMENTAL
Análise da qualidade e quantificação do RNA
Teste de contaminação com gDNA por qPCR
Coleta das amostras dos CBC
Extração de RNA
Síntese de cDNA Teste da Qualidade do cDNA
PCR Array das Vias de sinalização
8. MATERIAL E MÉTODOS
8.1. COLETAS DE AMOSTRAS
Primeiramente os pacientes foram avaliados e diagnosticados em uma consulta clínica, em seguida foram encaminhados para a marcação cirúrgica visando a exérese cirúrgica da lesão no centro de dermatologia de pequenas cirurgias do Hospital Regional da Asa Norte (HRAN). Após a cirurgia, as lesões foram seccionadas em duas partes: uma parte foi enviada para a confirmação histopatológica no Laboratório de Anatomia e Histopatologia do HRAN e a outra parte foi estocada em RNAlater
(QIAGEN) e armazenada a -80ºC no Laboratório de Ciências Genômicas e Biotecnologia da UCB para estudo molecular. Após confirmação histopatológica, acessada por meio eletrônico através do portal da Secretaria de Estado da Saúde do
Distrito Federal por meio do sistema TrakCare (em
www.portaldeexames.saude.df.gov.br/trakcareprod/web), pacientes confirmados para CBC nodular e superficial tiveram suas amostras selecionadas para o desenvolvimento deste trabalho. Devido à dificuldade de coletar amostras de tecido normal dos mesmos pacientes da cirurgia, amostras de tecido epitelial sem neoplasia de outro banco de tumor foram utilizadas como amostra controle neste trabalho. Este trabalho foi submetido e aprovado pelo Comitê de Ética em Pesquisa (Anexo I), sob o registro No. CEP/UCB 112/2009.
8.2. PADRONIZAÇÃO E EXTRAÇÃO DE RNA
Para padronizar o método de extração do RNA foram feitos diversos testes com material de pele humana. Dentre as técnicas mais comuns, como a utilização de cadinho e pistilo, nitrogênio líquido, TissueRuptor e o TissueLyser II, o equipamento TissueLyser II demonstrou melhor potencial para a extração. Entre os kits de extração,
Portanto, para este estudo, para o rompimento das células do tecido coletado foi utilizado o aparelho TissueLyser II (Retsch®, QIAGEN) com bolas de aço de 5mm, livre de RNAses, por três tempos de 3 minutos à 20Hz. Para a extração e purificação do RNA das amostras foi necessária uma padronização do protocolo, uma vez que o tecido epitelial possui várias proteínas associadas como colágeno e queratina, o que dificulta a obtenção de uma boa qualidade e elevada concentração de RNA. Para a extração foi utilizado o kit RNeasy Mini (QIAGEN) com algumas modificações: utilização da solução de 1mL do reagente TRIzol (Invitrogen) - o reagente TRIzol garante a integridade do RNA durante a fase de homogeneização e de lise celular por ser uma solução monofásica de fenol e isotiocianato de guanidina - ao invés do tampão de extração contendo β-mercaptoetanol recomendado pelo kit; e adição de DNase I e tampão de lise (buffer RDD). Após esta etapa e para assegurar a descontaminação total de DNA genômico, as amostras foram novamente submetidas ao tratamento com DNAse I, conforme recomendação do fabricante.
8.3. ANÁLISE DA QUALIDADE E QUANTIFICAÇÃO DO RNA
Para avaliar a integridade das amostras de RNA total extraído, 1 µL da amostra foi analisado em gel de agarose 1% contendo brometo de etídeo (10mg/mL) e visualizado no transiluminador.
8.4. IDENTIFICAÇÃO DE CONTAMINAÇÃO POR DNA GENÔMICO (gDNA)
O primeiro passo para a realização da PCR Array foi a verificação da contaminação de gDNA nas amostras de RNA. Foi realizado a técnica da PCR quantitativa (qPCR) utilizando sonda SYBER® Green e primer específico de gDNA (QIAGEN). Para cada amostra foi utilizado 1µL do RNA eluído, 12,5µL de SYBER® Green, 1µL de primer gDNA e 10,5µL de água livre de RNases. O ciclo de amplificação utilizado foi 1 ciclo a 95ºC/10min, 40 ciclos a 95ºC/15seg e 60ºC/1min, e uma etapa de
Melting Curve incluída automaticamente pelo próprio sistema operacional.
8.5. SÍNTESE DE DNA COMPLEMENTAR (cDNA)
O kit RT2 First Strand foi utilizado para síntese de cDNA das amostras de RNA extraídas de acordo com o protocolo recomendado pelo fabricante. Todas as amostras de RNA convertidas em cDNA apresentavam concentração final de 1µg.
8.6. CONTROLE DE QUALIDADE DO cDNA
Após a síntese de cDNA, a qualidade do cDNA foi analisada utilizando uma placa controle chamada RT2 RNA QC PCR Array, a qual é desenhada para avaliar a
qualidade de 12 amostras de cDNA simultaneamente. Esta etapa fornece uma completa confiabilidade do resultado de análise da expressão de genes, permitindo a exclusão de amostras que apresentem qualquer tipo de contaminação e a continuidade do experimento com total segurança. A placa contém controles para a integridade do RNA, presença de inibidores da transcrição reversa e presença de gDNA ou qualquer outra contaminação por DNA. A contaminação é visualizada por meio de sinais falso-positivos e curvas de dissociações de múltiplos picos. Além destes controles, dois genes constitutivos, actβ (β-actina) e hprt1 (hipoxantina fosforribosiltransferase 1),
tipicamente expressados em diferentes níveis em relação um ao outro, permitem uma previsão da faixa de valores esperado para o limiar (threshold) em reações
subseqüentes e permitem estimar a integridade do RNA.
O limiar de detecção, que é denominado “threshold”, foi configurado para 0,24
da amostra, permite determinar o número de ciclos necessários para o início da amplificação da sequência gênica-alvo presente no cDNA de cada amostra. Este valor é denominado Ct (“Cycle Threshold”), o qual é calculado pela intersecção da linha do
limiar de detecção da reação, com a linha da amplificação gênica de cada amostra. O valor da linha da base (baseline), o qual indica o nível de ruído nos ciclos iniciais e é
calculada substraindo-se da fluorescência obtida dos produtos de PCR, foi calculado automaticamente pelo próprio software para todas as amostras, segundo recomendação do fabricante. Para a amplificação, o programa de ciclagem utilizada foi o de 1 ciclo a 95ºC/10min e 40 ciclos a 95ºC/15seg e 60ºC/1min utilizando o aparelho Step One Plus (Applied Biosystems).
8.7. REPLICATA EXPERIMENTAL
Para este estudo não foram feitas triplicatas biológicas uma vez que as placas de PCR Array garantem uma confiabilidade por meio de controles endógenos presentes na mesma. Foi feita uma análise para cada tipo de amostra coletada com programa específico, sendo três amostras diferentes analisadas em cada grupo (controle – tecido normal sem neoplasia, CBC nodular, CBC superficial) em três placas de PCR Array.
8.8. PCR Array – VIA DE SINALIZAÇÃO HEDGEHOG
A análise dos dados obtidos no qPCR seguiram o modelo mínimo de informação para publicação de experimentos quantitativos por PCR em Tempo Real (MIQE guidelines) (Bustin, 2009).
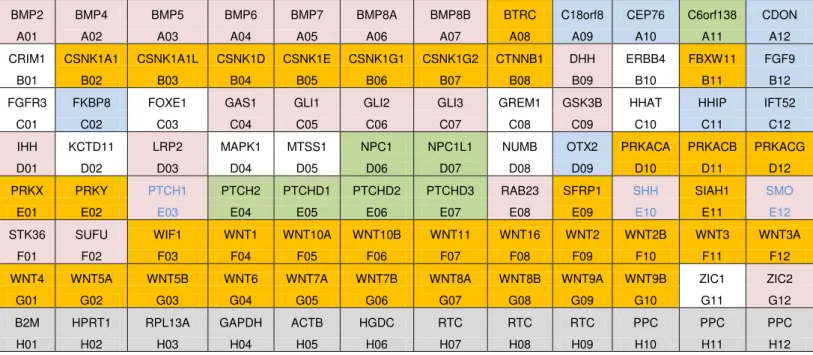
Além dos genes referentes à via, constam também cinco genes de referência (b2m - β-2 microglobulina, hprt1 - hipoxantina fosforribosiltransferase 1, rpl13a –
proteína ribosomal L13a, gapdh – gliceraldeído-3-fosfato dehidrogenase e actβ – β
-actina), um controle para contaminação com gDNA, três controles para a transcrição reversa e três controles positivos para amplificação da PCR (Tabela 4).
Os 84 genes referentes à via HH estão descritos abaixo. Alguns destes estão repetidos na descrição abaixo por terem mais de uma função associada ou participarem de mais de uma via:
- Membros da família Hedgehog e proteínas associadas: dhh, hhat, hhip, ihh, shh, siah1.
- Receptores Hedgehog: c18orf8, c6orf138, npc1, npc1l1, ptch1, ptch2, ptchd1, ptchd2, ptchd3.
- Via Sonic Hedgehog (Shh): gli1, gli2, gli3, gsk3b, ptch1, shh, smo, sufu.
- Via de sinalização Smoothened: cdon, cep76 (c18orf9), fgf9, fkbp8, gli1, gli2, gli3, hhip, ift52, otx2, ptch1, shh, smo.
- Via de sinalização de receptor WNT: btrc, csnk1a1, csnk1a1l, csnk1d, csnk1e, csnk1g1, csnk1g2, ctnnb1, fbxw11, gsk3b, prkaca, prkacb, prkacg, prkx, prky, sfrp1, siah1, wif1, wnt1, wnt10a, wnt10b, wnt11, wnt16, wnt2, wnt2b, wnt3, wnt3a, wnt4, wnt5a, wnt5b, wnt6, wnt7a, wnt7b, wnt8a, wnt8b, wnt9a, wnt9b.
- Outros genes envolvidos na via de sinalização Hedgehog: bmp2, bmp4, bmp5, bmp6, bmp7, bmp8a, bmp8b, dhh, gas1, ihh, lrp2, rab23, smo, stk36, zic2.
- Reguladores de diferenciação celular: bmp2, bmp4, bmp6, bmp7, bmp8a, bmp8b, ctnnb1, fgf9, gli1, gli3, ihh, mapk1, sfrp1, shh, siah1, smo, wnt1, wnt3a, wnt7a, zic1, zic2.
- Desenvolvimento do sistema nervoso: bmp4, crim1, ctnnb1, gli2, gli3, grem1, lrp2, mtss1, otx2, rab23, shh, siah1, wnt8b, zic1, zic2.
- Desenvolvimento Camera-Type Eye:bmp4, fkbp8, shh. - Desenvolvimento esquelético:ctnnb1, fgfr3, gli2, ihh, sufu.
- Desenvolvimento do coração:bmp4, ctnnb1, gli2, gli3, shh.
- Desenvolvimento do pulmão: bmp4, ctnnb1, gli1, gli2, gli3, hhip, shh, wnt5a. - Desenvolvimento da cartilagem:bmp2, bmp4, bmp5, bmp6, bmp7.
Tabela 4. Desenho referente à placa PCR Array da via de sinalização HH e dos genes que codificam proteínas associadas. Em rosa, genes relacionados à via sonic hedgehog e a via de sinalização de HH. Em azul, genes relacionados à via de sinalização SMO. Em laranja, genes relacionados à via de sinalização de receptor WNT. Em verde, genes relacionados a receptores de HH. Em branco, genes relacionados ao desenvolvimento de diversos sistemas e reguladores de diferenciação celular. Em cinza, genes relacionados ao controle de qualidade da placa.
BMP2 BMP4 BMP5 BMP6 BMP7 BMP8A BMP8B BTRC C18orf8 CEP76 C6orf138 CDON
A01 A02 A03 A04 A05 A06 A07 A08 A09 A10 A11 A12
CRIM1 CSNK1A1 CSNK1A1L CSNK1D CSNK1E CSNK1G1 CSNK1G2 CTNNB1 DHH ERBB4 FBXW11 FGF9
B01 B02 B03 B04 B05 B06 B07 B08 B09 B10 B11 B12
FGFR3 FKBP8 FOXE1 GAS1 GLI1 GLI2 GLI3 GREM1 GSK3B HHAT HHIP IFT52
C01 C02 C03 C04 C05 C06 C07 C08 C09 C10 C11 C12
IHH KCTD11 LRP2 MAPK1 MTSS1 NPC1 NPC1L1 NUMB OTX2 PRKACA PRKACB PRKACG
D01 D02 D03 D04 D05 D06 D07 D08 D09 D10 D11 D12
PRKX PRKY PTCH1 PTCH2 PTCHD1 PTCHD2 PTCHD3 RAB23 SFRP1 SHH SIAH1 SMO
E01 E02 E03 E04 E05 E06 E07 E08 E09 E10 E11 E12
STK36 SUFU WIF1 WNT1 WNT10A WNT10B WNT11 WNT16 WNT2 WNT2B WNT3 WNT3A
F01 F02 F03 F04 F05 F06 F07 F08 F09 F10 F11 F12
WNT4 WNT5A WNT5B WNT6 WNT7A WNT7B WNT8A WNT8B WNT9A WNT9B ZIC1 ZIC2
G01 G02 G03 G04 G05 G06 G07 G08 G09 G10 G11 G12
B2M HPRT1 RPL13A GAPDH ACTB HGDC RTC RTC RTC PPC PPC PPC
H01 H02 H03 H04 H05 H06 H07 H08 H09 H10 H11 H12
8.9. ANÁLISE DOS DADOS
A análise de dados foi realizada utilizando o teste t de Student através do portal Web-based PCR ARRAY Data Analysis Software (SABiosciencies, QIAGEN) para RT2
Profiler PCR Arrays como recomendado pelo fabricante. Para a seleção dos melhores constitutivos foi feita uma análise utilizando o programa GeNorm, integrado ao software da qbasePLUS (disponível em: <http://medgen.ugent.be/~jvdesomp/genorm/>).
O Web-based PCR ARRAY Data Analysis Software foi utilizado para análise dos
resultados obtidos no qPCR. Os resultados foram representados em formato de tabelas, gráfico de dispersão e perfil em três dimensões. O programa executa automaticamente a quantificação usando o método ΔΔCt, baseados em cálculos de fold-change, o que permitiu selecionar os genes diferencialmente expressos entre o
partir da distribuição de teste t de Student, a qual identifica a probabilidade desta
distribuição ser um evento aleatório ou não. Se a probabilidade for pequena, pode-se concluir que o resultado observado é estatisticamente relevante. O valor-P está ligado a um grau de confiança previamente estabelecido pelo teste (neste caso, o grau de confiança foi de 95%) para definir se a hipótese nula deve ser rejeitada ou não. Se o valor-P encontrado for maior que o valor-P tabelado, a hipótese nula é rejeitada (Callegari-Jacques, 2003). Para este trabalho o valor-P adotado foi P<0,05.
Para os casos do valor-P não significativo foi utilizado o método estatístico de medida da correlação interclasse. Este método permite obter a estimativa do coeficiente da correlação, a qual é resultado da raiz quadrada do produto do coeficiente da regressão de y em x e de x em y (Mather, 1994). A correlação indica a força e a direção do relacionamento linear entre duas variáveis aleatórias. Este método permite mostrar que há pares de variáveis aleatórias com forte dependência estatística (Shikamura, 2002). Podem ser utilizados vários coeficientes dependendo da situação, sendo o mais conhecido, o coeficiente de correlação de Pearson (ρ). Este é obtido dividindo a covariância de duas variáveis pelo produto de seus desvios padrão e é definido como:
Onde X e Y são duas variáveis aleatórias, com valores esperados μX e μY e desvios padrão σX e σY. Este coeficiente ρ assume valores entre -1 e 1. Zero indica completa falta de correlação. Os valores obtidos são interpretados da seguinte forma:
9. RESULTADOS E DISCUSSÃO
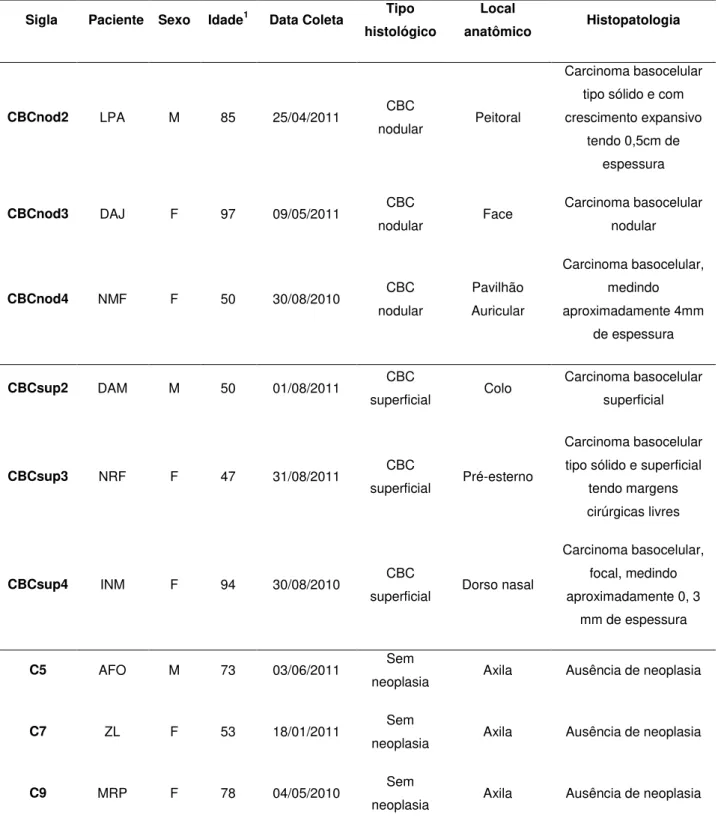
Tabela 6. Dados clínicos das amostras selecionados dos pacientes submetidos à cirurgia.
Sigla Paciente Sexo Idade1 Data Coleta Tipo
histológico
Local
anatômico Histopatologia
CBCnod2 LPA M 85 25/04/2011 CBC
nodular Peitoral
Carcinoma basocelular tipo sólido e com crescimento expansivo
tendo 0,5cm de espessura
CBCnod3 DAJ F 97 09/05/2011 CBC
nodular Face
Carcinoma basocelular nodular
CBCnod4 NMF F 50 30/08/2010 CBC
nodular Pavilhão Auricular Carcinoma basocelular, medindo aproximadamente 4mm de espessura
CBCsup2 DAM M 50 01/08/2011 CBC
superficial Colo
Carcinoma basocelular superficial
CBCsup3 NRF F 47 31/08/2011 CBC
superficial Pré-esterno
Carcinoma basocelular tipo sólido e superficial
tendo margens cirúrgicas livres
CBCsup4 INM F 94 30/08/2010 CBC
superficial Dorso nasal
Carcinoma basocelular, focal, medindo aproximadamente 0, 3
mm de espessura
C5 AFO M 73 03/06/2011 Sem
neoplasia Axila Ausência de neoplasia
C7 ZL F 53 18/01/2011 Sem
neoplasia Axila Ausência de neoplasia
C9 MRP F 78 04/05/2010 Sem
neoplasia Axila Ausência de neoplasia
Devido à dificuldade de extrair RNA da pele e da pequena quantidade de amostra obtida foi necessário padronizar o método de extração do RNA para a obtenção da qualidade de RNA necessária (1µg de RNA total). Como pode ser visto na Figura 5, o RNA obtido após padronização apresentou uma boa qualidade. Observa-se na figura a integridade dos RNA ribossomais 18S e 28S, e não é possível constatar contaminação por gDNA.
Figura 5. Análise da qualidade do RNA. Gel de Agarose 1% corado com brometo de etídeo contendo as seguintes amostras: Poço 1: Marcador de massa molecular 1Kb (Invitrogen). Poço 2: Paciente normal sem neoplasia C5. Poço 3: CBC nodular N2. Poço 4: CBC nodular N3. Poço 5: CBC nodular N4. Poço 6: CBC nodular N2. Poço 7: CBC nodular N3. Poço 8: CBC nodular N4.
Os valores obtidos da quantificação do RNA utilizando o Qubit estão demonstrados na Tabela 7.
Tabela 7. Concentração RNA (Qubit).
Amostra Quantificação (ng/µL)
CBC Nodular 2 3.920 CBC Nodular 3 3.970 CBC Nodular 4 2.930 CBC Superficial 2 996 CBC Superficial 3 1.300 CBC Superficial 4 972
Controle 5 140
Controle 7 150
Controle 9 78
Após a quantificação pelo aparelho Qubit, foi possível observar que as amostras controle (sem neoplasia) apresentaram valores quantitativos menores do que as amostras com neoplasia, tendo em vista que as amostras com neoplasia apresentam um maior número de células devido à característica intrínseca da célula tumoral. A célula tumoral apresenta fatores biológicos alterados como a proliferação celular, que auxiliam a entender esse resultado da quantificação. Logo, consideramos que é possível retirar uma quantidade maior de RNA devido à presença de um maior número de células nessas amostras.
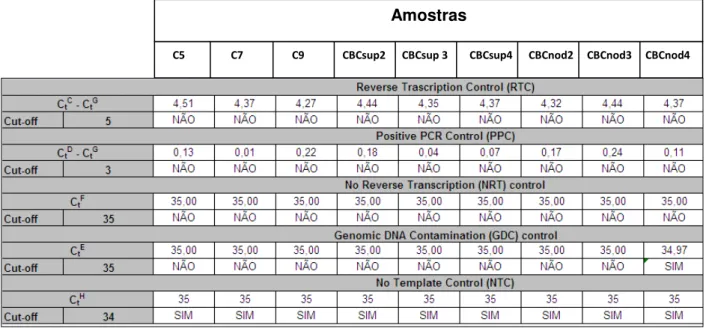
O RNA das 9 amostras selecionadas (3 controles normal, 3 CBC nodular e 3 CBC superficial) foram analisadas quanto à contaminação por gDNA por meio da qPCR. Após esta análise, foi feita a síntese de cDNA destas 9 amostras selecionadas por meio de kit específico para a síntese e elas foram testadas em uma placa específica para controle de qualidade (chamada RT2 RNA QC PCR Array). Para todas
Tabela 8. Análise dos cDNA das amostras selecionadas para o PCR Array por qPCR.
9.1. ANÁLISE DA EXPRESSÃO GÊNICA
9.1.1. Seleção dos melhores candidatos dos genes constitutivos
Para análise dos genes diferencialmente expressos obtidos na placa de PCR Array é fundamental o uso de genes de referência. Estes serão utilizados como controles internos para a normalização dos dados da expressão diferencial nos experimentos de qPCR. Na placa de PCR Array estão presentes 5 genes (b2m, hprt1, rpl13a, gapdh, actb) como potenciais normalizadores. O programa geNorm (qbasePLUS,
disponível por Biogazelle), que foi desenvolvido para determinar os genes mais estáveis de referência a partir de um conjunto de genes candidatos testados em um grupo experimental, foi utilizado para fazer esta seleção. A partir dele, um fator normalizador pode ser calculado para cada amostra a partir da média geométrica de um número definido para o gene de referência. Os resultados obtidos pelo programa geNorm indicaram 3 genes como potenciais normalizadores e são eles o actβ, gapdh e hprt1. A estabilidade do alvo de referência foi dada como média (geNorm M ≤ 1.0).
Valor normalmente visto quando se avalia candidatos alvos de referências em grupos de amostras heterogêneas (por exemplo, biópsias de câncer ou amostras de tecidos
C5 C7 C9 CBCsup2 CBCsup 3 CBCsup4 CBCnod2 CBCnod3 CBCnod4
diferentes), como pode ser visto na Figura 6. Após a seleção desses genes de referência, os mesmos foram utilizados na plataforma Web-Based PCR Array. Esta plataforma permite que o usuário escolha a melhor opção para o método de análise da normalização de genes no experimento. Uma vez que os genes actb, gapdh e hprt1
foram anteriormente selecionados pelo GeNorm, a análise de dados foi feita selecionando a opção manual. A normalização dos dados foi feita por meio do valor geométrico (opção selecionada na plataforma Web-Based PCR Array) para o cálculo
da expressão gênica, uma vez que a opção do valor geométrico fornece valores mais apurados.
Figura 6. Determinação do número de alvos de referência a serem utilizados nas análises utilizando o software geNorm (qbasePlus).
ACTB
GAPDH
9.1.2. Análise diferencial de genes da via de sinalização Hedgehog e Wnt
Para realizar a análise das vias, os seguintes grupos foram comparados:
9.1.2.1. Análise de genes do Primeiro Grupo (CBC nodular em relação ao grupo controle)
O primeiro grupo comparado e analisado foi o grupo do CBC nodular (média dos Ct de 03 amostras) em relação ao grupo controle (média dos Ct de 03 amostras). A partir dos valores de (2^-ΔCt) 1 dos dados do grupo do CBC nodular e do grupo controle foi possível obter o seguinte gráfico de dispersão representado na Figura 7, onde o grupo 1 é a representação das amostras de CBC nodular.
1 2^-DeltaCt
–é a elevação negativa do ΔCt na base 2; Ct é sigla para o CycleThreshold, que é
o ponto que detecta o ciclo na qual a reação atinge o limiar da fase exponencial, esse ponto permite a quantificação exata e reprodutível baseado na fluorescência.
Primeiro Grupo Segundo Grupo Terceiro Grupo
3 amostras de CBC nodular em relação a 3 amostras do grupo normal
3 amostras de CBC superficial em relação a 3 amostras do grupo normal
Figura 7. Gráfico de dispersão entre grupo CBC nodular em relação ao controle (sem neoplasia). O eixo y é representado pelos valores de Log10(Grupo1 2^-DeltaCt), onde o Grupo 1 é o grupo CBC nodular. O eixo x é representado pelos valores de Log10(Grupo Controle 2^-DeltaCt). Em vermelho estão representados os genes superexpressos. Em verdes estão representados os genes reprimidos. Em preto estão representados os genes com expressão basal.
Os genes identificados acima foram representados pelo símbolo “*” (asterisco), na cor vermelha indicando os genes superexpressos, e na cor verde indicando os genes reprimidos. Os 8 genes superexpressos representados acima obtidos foram o
c6orf138, foxe1, hhip, lrp2, otx2, prky, ptch1 e ptch2.
A diferença da expressão gênica de cada um desses genes e seu respectivo valor-P na análise proposta está representada na Tabela 9 abaixo. Apenas o gene
c6orf138, destacado em negrito, apresentou o valor-P significativo. O teste t de Student
aplicado na plataforma Web-Based PCR Array, o qual fornece o valor-P, não é o
A Figura 8 abaixo representa um gráfico de barras dos dados obtidos dos genes superexpressos em análise, na qual o eixo “y” representa o valor de fold change dos
genes em análise e o eixo “x” representa os genes do grupo controle e do grupo CBC nodular. Para os genes no grupo controle, o valor do fold change é igual a um por ser o
grupo normalizador. No grupo CBC nodular, os valores obtidos dos genes superexpressos foram: para o gene c6orf138 igual a 5,3223; para o gene foxe1 igual a
13,181; para o gene hhip igual a 46,526; para o gene lrp2 igual a 5,1179; para o gene otx2 igual a 4,0863; para o gene prky igual a 4,4172; para o gene ptch1 igual a 11,2305
e para o gene ptch2 igual a 93,8009.
Tabela 9. Genes superexpressos no grupo CBC nodular em relação ao controle. Destacando em negrito o gene com valor-P significativo.
Genes superexpressos CBC nodular em relação aogrupo controle Gene Diferença de expressão gênica Valor-P
C6orf138 5,3223 0,03887
FOXE1 13,181 0,20316
HHIP 46,526 0,253372
LRP2 5,1179 0,167554
OTX2 4,0863 0,357217
PRKY 4,4172 0,241498
PTCH1 11,2305 0,221019
Figura 8. Genes superexpressos no CBC nodular em relação ao controle.
Os 26 genes reprimidos representados no gráfico de dispersão (Figura 7) em cor verde foram: bmp2, bmp4, cdon, csnk1a1l, dhh, erbb4, fgfr3, ihh, kctd11, ptchd1, ptchd2, shh, rpl13a, hgdc, wif1, wnt10a, wnt10b, wnt16, wnt2b, wnt3, wnt3a, wnt4, wnt7a, wnt8a, wnt9b e zic2.