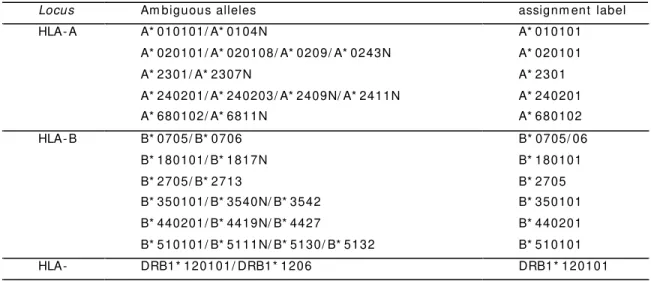
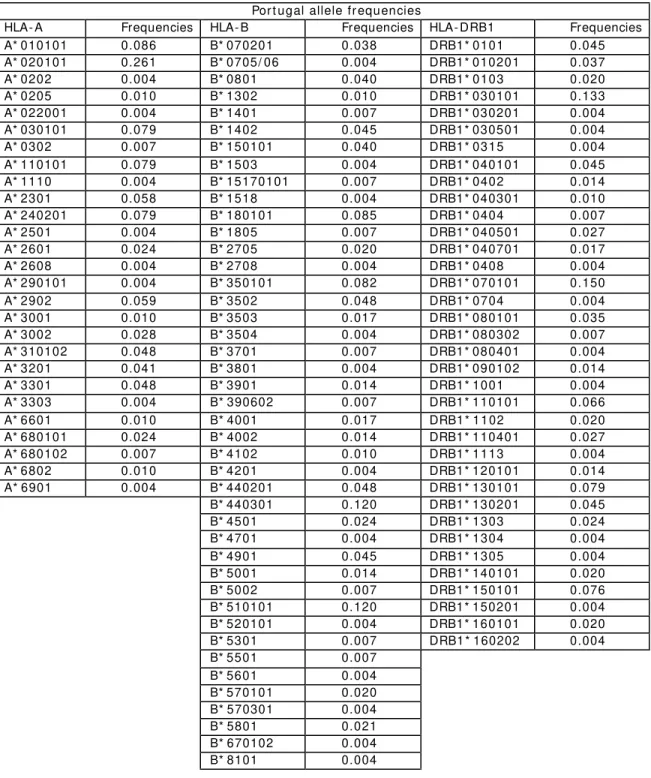
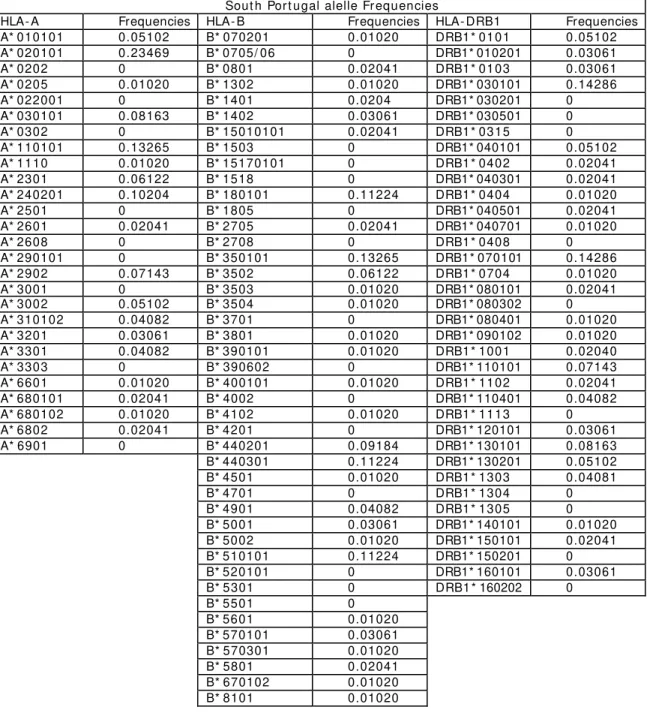
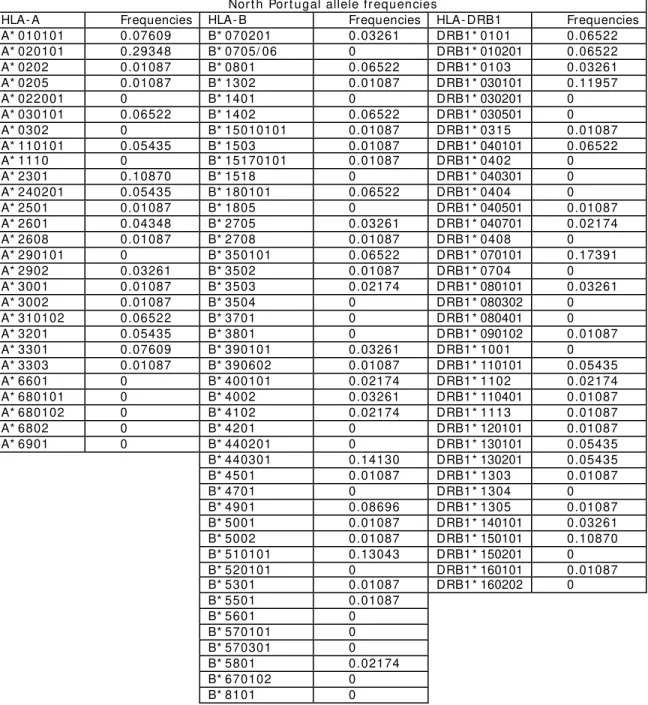
Diversidade Genét ica do Sist em a HLA
em Port ugal, Cabo Verde e Guiné- Bissau
H élde r Spínola de Fre it a s
Tese apresent ada na Universidade da Madeira para a obt enção
do grau de Dout or
D ive r sida de Ge n é t ica do Sist e m a H LA e m Por t u g a l, Ca b o V e r d e e
Guiné- Bissa u
por
Hélder Spínola de Freitas
Tese subm etida de acordo com o regulam ento em vigor na Universidade da
Madeira para obtenção do grau de
Doutor em Ciências Biológicas, especialidade de Citogenética e Biolo gia
Molecular
Universidade da Madeira
D iv e r sid a d e Ge n é t ica d o Sist e m a H LA e m Por t u g a l, Ca b o V e r d e e
Guiné- Bissa u
Hélder Spínola de Freitas
Supervisor Científico:
Professor Catedrático António Brehm
Departam ento de Biologia, Universidade da Madeira
A todos aqueles que,
independentem ente de alcançarem ou não
os seus obj ect ivos, encont ram força
Í ndice
Agradecim entos ... xv
Abreviaturas ... xvi
Resum o ... xvii
Abstract ... xix
1. I ntrodução ... 1
1.1. A descoberta do sistem a HLA... 1
1.2. Nom enclatura do sistem a HLA ... 2
1.3 . Técnicas de caracterização do sistem a HLA ... 3
1.4 . Estrutura e função do sistem a HLA ... 7
1.5. Evolução e diversificação do sistem a HLA ... 10
1.6. I m portância m édica do sistem a HLA ... 12
1.7. Aplicação dos genes HLA nas disputas de paternidade... 16
1.8. I m portância dos genes HLA em estudos populacionais ... 17
1.9. História das populações em estudo ... 20
1.10. Obj ectivos da tese ... 22
2. Material e m étodos... 25
2.1. Am ostragem das populações ... 25
2.2. Genes estudados... 27
2.3. Métodos utilizados ... 27
2.4. Análise de dados... 31
3. Distribution of HLA alleles in Portugal and Cabo Verde. Relationships with the slave trade route... 35
3.1. Abstract... 35
3.3. Materials and m et hods ... 39
3.3.1. Population sam ples ... 39
3.3.2. HLA typing and statistics ... 40
3.4. Results and discussion ... 41
3.4.1. HLA class I and I I allele frequencies ... 41
3.4.1.1. HLA- A ... 45
3.4.1.2. HLA- B ... 46
3.4.1.3. HLA- DRB1 ... 47
3.4.2. Haplotype frequencies ... 48
3.4.2.1. West European haplotypes ... 48
3.4.2.2. Mediterranean haplotypes ... 50
3.4.2.3. I berian- North African haplotypes ... 50
3.4.2.4. The sub- Saharan com ponent ... 50
3.4.2.5. The ‘Oriental’ com ponent ... 51
3.4.3. Principal Com ponent Analysis ... 52
3.4.4. Population com parisons ... 54
4. HLA genes in Portugal inferred from sequence- based typing: in the crossroad between Europe and Africa ... 57
4.1. Abstract ... 57
4.2. I ntroduction ... 58
4.3. Materials and m et hods ... 60
4.3.1. Population sam ples and HLA typing ... 60
4.3.2. Data analysis ... 62
4.4. Results ... 63
4.4.1. Data for HLA- A ... 68
4.4.3. Data for HLA- DRB1 ... 71
4.4.4. Haplotype frequencies ... 72
4.4.5. Phylogenetic analyses ... 78
4.5. Discussion ... 82
5. HLA class I and I I polym orphism s in Azores show different settlem ents in Oriental and Central islands ... 87
5.1. Abstract ... 87
5.2. Introduction ... 88
5.3. Materials and m et hods ... 90
5.3.1. Population sam ples and HLA typing ... 90
5.3.2. Data analysis ... 91
5.4. Results ... 93
5.4.1. HLA- A locus ... 97
5.4.2. HLA- B locus ... 97
5.4.3. HLA- Cw locus ... 97
5.4.4. HLA- DRB1 locus ... 98
5.4.5. HLA- DQA1 locus ... 98
5.4.6. HLA- DQB1 locus ... 98
5.4.7. Haplotype frequencies ... 99
5.4.8. Phylogenetic analyses ... 107
5.5. Discussion ... 111
5.5.1. Diversity between Azores islands ... 114
6. HLA polym orphism s in Cabo Verde and Guiné- Bissau inferred from sequence- based typing ... 117
6.1. Abstract ... 117
6.3. Materials and m et hods ... 120
6.3.1. Population sam ples and HLA typing ... 120
6.3.2. Data analysis ... 121
6.4. Results ... 123
6.4.1. Guiné- Bissau allele frequencies ... 128
6.4.2. Cabo Verde allele frequencies ...
129
6.4.3. Haplotype frequencies ... 129
6.4.4. Phylogenetic analyses ... 136
6.5. Discussion ... 139
7. HLA genes in Madeira island (Portugal) inferred from sequence-based typing: footprints from dif ferent origins ... 143
7.1. Abstract ... 143
7.2. I ntroduction ... 144
7.3. Materials and m et hods ... 145
7.3.1. Population sam ples and HLA typing ... 145
7.3.2. Data analysis ... 146
7.4. Results ... 146
7.4.1. Allele frequencies ... 146
7.4.2. Haplotype frequencies ... 148
7.5. Discussion ... 150
8. Discrepancies on HLA typing by SSOP and SBT techniques: a case study ... 155
8.1 Abst ract ... 155
8.2 I ntroduction ... 156
8.3 Materials and m ethods ... 157
9. Resultados globais ... 161
9.1. Parâm etros gerais ... 161
9.2. HLA- A ... 163
9.3. HLA- B ... 164
9.4. HLA- DRB1 ... 165
9.5. Frequências haplotípicas ... 166
9.6. Análises filogenéticas ... 167
10. Discussão geral ... 169
10.1. Distribuição dos alelos HLA em Portugal e Cabo Verde. I nfluências do com ércio de escravos ... 169
10.2. Caracterização dos genes HLA na população Portuguesa por sequenciação: na encruzilhada entre a Europa e África ... 171
10.3. Polim orfism os dos genes HLA m ostram diferenças no povoam ent o dos grupos Cent ral e Oriental do arquipélago dos Açores ... 190
10.3.1. Diversidade entre as ilhas Açorianas ... 192
10.4. Caracterização do sistem a HLA em Cabo Verde e Guiné-Bissau por sequenciação nucleotídica ... 194
10.5. Os genes HLA na ilha da Madeira (Portugal) caracterizados por sequenciação nucleotídica: m arcas de diferentes origens ... 196
10.6. Discrepâncias na caracterização do sistem a HLA por SSOP e SBT: um caso de estudo ... 199
10.7. Discussão global ... 200
10.8. Perspectivas futuras ... 204
11. Referências bibliográficas ... 207
Ag r a d e cim e n t os
Apresento os m eus m ais sinceros agradecim entos a António Brehm pelo
acom panham ent o e apoio que dispensou ao longo do desenvolvim ent o
dest e t rabalho. Agradeço t am bém a Derek Middlet on e a t odos os m em bros
do Laborat ório de Histocom patibilidade e I m m unogenética da I rlanda do
Norte pelo acolhim ento e apoio na abordagem à caracterização dos genes
HLA nas populações estudadas.
Agradeço ainda às m inhas colegas do Laboratório de Genética Hum ana pelo
apoio prestado. Agradeço a t odos os m em bros do Depart am ent o de Biologia
da Universidade da Madeira que directa ou indirectam ente prestaram apoio
ao trabalho desenvolvido. Agradeço ao Governo Regional da Madeira que,
através do Fundo Social Europeu, apoiou financeiram ente este proj ect o de
doutoram ento e ao program a I nterreg I I I B Madeira- Açores- Canárias que
financiou parte da investigação desenvolvida no âm bito deste trabalho.
Por últim o, agradeço a toda a m inha fam ília e am igos pelo apoio m oral, e
não só, que sem pre deram no desenvolvim ento deste trabalho,
nom eadam ente o Paulo Lucas na concepção da capa deste m anuscrito.
Destaco particularm ente a m inha esposa pela form a incondicional com o
sem pre m e incentivou e am parou, principalm ente nos m om entos m ais
Ab r e v ia t u r a s
a . C.- Antes de Cristo
Bp- Base pairs (pares de bases)
d . C.- Depois de Cristo
D C- Doença celíaca ( celiac desease)
D N A- Deoxyribonucleic acid (ácido desoxiribonucleíco)
HI V- Hum an im m unodeficiency virus
H LA - Hum an leukocyte antigens
Kb- Kilobases
M b- Megabases
M H C - Major histocom patibility com plex
P CA- Principal com ponent analysis
P CO- Principal coordinates analysis
P CR- Polym erase chain react ion
SBT- Sequence based typing
RFLP- Restriction fragm ent length polym orphysm
SSO P- Sequence specific oligonucleotide probes
SSP- Sequence specific priming
Re su m o
O polim orfism o dos genes HLA- A, HLA- B e HLA- DRB1 foi est udado em t rês
populações Portuguesas (Portugal continental, Madeira e Açores) e em duas
Africanas (Guiné- Bissau e Cabo Verde) . Os dados em alta resolução, obtidos
por sequenciação ( SBT) , foram com parados com a caract erização efect uada
pelo m étodo SSOP revelando 4,6% de incongruências entre os resultados
dest es dois m ét o dos.
Os alelo s m ais frequentes em cada um dos loci foram : HLA- A* 0201 em
t odas as populações ( 13,5- 26% ) ; HLA- B* 5101 em Portugal continental
( 12% , o m esm o par a o B* 440301) , Madeira ( 9,7% ) e Açores ( 9,8% ) e
B* 350101 na Guiné- Bissau ( 14,4% ) e Cabo Verde ( 13,2% );
HLA-DRB1* 0701 em Portugal continental ( 15% ) , Madeira ( 15,7% ) e Açores
( 18,3% ) , DRB1* 1304 na Guiné- Bissau ( 19,6% ) e DRB1* 110101 em Cabo
Verde ( 10,1% ) .
Os haplotipos 3-loci m ais predom inantes em cada população foram :
A* 020101- B* 440301- DRB1* 070101 em Port ugal continental (3,1% ) ,
A* 020101- B* 510101- DRB1* 130101 na Madeira ( 2,7% ) , A* 2902-B*
4403-DRB1* 0701 nos Açores ( 2,4% ) , A* 2301- B* 1503- 4403-DRB1* 110101 na
Guiné-Bissau ( 4,6% ) e A* 3002- B* 350101- DRB1* 1001 em Cabo Verde ( 2,8% ) .
O presente trabalho revela que a população continental Port uguesa t em
sido influenciada geneticam ente por Europeus e Norte Africanos devido a
várias im igrações históricas. O Norte de Portugal parece concentrar,
provavelm ente devido à pressão da expansão Árabe, um antigo pool
Os dados obt idos nos genes do sist em a HLA corroboram as font es hist óricas
que confirm am que o povoam ent o dos Açores t eve o cont ribut o de out ros
Europeus, essencialm ente Flam engos, para além dos Portugueses. As
frequências alélicas e haplotípicas neste arquipélago não apresentam um a
distribuição hom ogénea entre as ilhas do grupo Oriental e Central. O grupo
Central revela um a influência clara da Europa Central e um a m uit o m enor
afinidade a Portugal continental.
As frequências alélicas e haplotípicas m ostram que a ilha da Madeira foi
povoada por Europeus, a m aioria Portugueses, m as tam bém por
sub-Saharianos devido ao com ércio de escravos.
Cabo Verde não é um a população tipicam ente sub- Sahariana pois revela
um a im portante influência genética Europeia, para além da base genética
Africana. A análise dos haplotipos e dendrogram as m ost ram uma influência
genética Caucasiana no actual pool genético Cabo- Verdiano. Os
dendrogram as e a análise das coordenadas principais m ostram que os
Guineenses são m ais sem elhantes aos Norte Africanos do que qualquer
outra população sub- Sahariana já estudada ao nível do sistem a HLA,
provavelm ent e devido a cont act os hist óricos com out ros povos,
Abst r a ct
HLA- A, HLA- B, and HLA- DRB1 genetic polym orphism was exam ined in three
Portuguese (m ainland Portugal, Madeira and Azores) and two African
populations (Guiné- Bissau and Cabo Verde). Data obtained at
high-resolution level, using sequence- based typing, were com pared with m edium
resolution typing previously obtained by SSOP, showing 4.6% incongruence
bet ween t he t wo m et hods.
The m ost frequent allele at each loci was: HLA- A* 0201 ( 13.5 - 26% ) in all
populations; HLA- B* 5101 in m ainland Portugal ( 12% , the sam e as
B* 440301) , Madeira ( 9.7% ) , and Azores ( 9.8% ) and B* 350101 in
Guiné-Bissau ( 14.4% ) and Cabo Verde ( 13.2% ) ; HLA- DRB1* 0701 in mainland
Portugal ( 15% ) , Madeira ( 15.7% ) , and Azores ( 18.3% ) , DRB1* 1304 in
Guiné- Bissau (19.6% ), and DRB1 * 110101 in Cabo Verde ( 10.1% ) .
The predom inant three-loci haplotype found in each population was:
A* 020101- B* 440301- DRB1* 070101 in m ainland Portugal (3.1 % ),
A* 020101- B* 510101- DRB1* 130101 in Madeira ( 2.7% ) , A* 2902- B*
4403-DRB1* 0701 in Azores ( 2.4% ) , A* 2301- B* 1503- 4403-DRB1* 110101 in
Guiné-Bissau (4.6% ), and A* 3002- B* 350101- DRB1* 1001 in Cabo Verde ( 2.8% ) .
The present study dem onstrates that the mainland Portuguese population
has been genetically influenced by Europeans and North Africans, via
several historic im m igrations. North Portugal seem s to concentrate,
probably due the pressure of Arab expansion, an ancient genetic pool
originated from several North Africans and Europeans influences throughout
HLA data corroborat es hist orical sources t hat say t he Azores were populat ed
not only by Portuguese but also by other Europeans, m ostly Flem ish people.
Haplotype and allele frequencies in this archipelago show no hom ogeneous
distribution between Oriental and Central islands. The Central group clearly
show s an influence of central Europeans, with m uch less affinity to m ainland
Portugal.
HLA allele and haplotype frequencies show that Madeira island were
populated by Europeans, m ostly Portuguese, but also by sub- Saharan
Africans due to slave trade.
Cabo Verde is not a typical sub- Saharan population showing an im portant
European genetic influence besides the African basis. Haplotypes and
dendrogram analysis shows a Caucasian genetic influence in today’s gene
pool of Cabo Verdeans. Dendrogram s and principal coordinates analysis
show t hat Guineans are m ore sim ilar to North Africans than other HLA
studied sub- Saharans, probably due to ancient and recent historical links
Ca pít u lo 1
1 . I n t r odu çã o
1 .1 . A d e scob e r t a d o sist e m a H LA
O Com plexo Maior de Histocom patibilidade ( MHC - Maj or
Histocom patibility Com plex) , existente em todos os vertebrados, é
constituído, entre outros, por genes com im portantes funções im unológicas
e foi descobert o em 1937 por Pet er Gorer durante o estudo de transplantes
em rat os ( Gorer, 1937) . Mais tarde Jean Dausset ( 1958) publicou as suas
observações e conclusões sobre a capacidade do soro de pessoas
subm etidas a transfusões sanguíneas aglutinar leucócitos de outros
indivíduos. Dausset descobriu assim o prim eiro antigénio hum ano,
denom inado MAC ( agora HLA- A* 02) , um dos produt os dos genes do
sistem a hum ano de antigénios leucocít icos ( HLA - Hum an Leukocyte
Antigens) , o correspondente hum ano do MHC. Mais tarde, Jon van Rood e
van Leeuwen ( 1963) publicaram a descoberta de outro gene do sistem a
HLA a que deram o nom e de FOUR ( agora HLA- B) . Com a invenção da
técnica de cultura linfocitária m ista em 1964 (Bach e Hirschhorn, 1964;
descobert a de novos genes, nom eadam ent e o HLA- DR na década de 70 do
século XX ( Yunis e Am os, 1971; Tosi et al. 1978) .
O envolvim ent o do MHC na resposta im unitária do organism o só foi
confirm ada no início da década de setenta do século XX (Shevach et al.,
1972) m as act ualm ent e conhece- se j á em detalhe a estrutura e função dos
genes classe I e classe I I do sistem a HLA (Bj orkm an et al., 1987; Brown et
al. 1993) . Apesar de actualm ente desconhecerm os ainda o m ecanism o
exacto que associa determ inados alelo s e haplotipos ( conj unto de alelos de
diferentes loci t ransm it idos em bloco de geração em geração) do sist em a
HLA a um elevado risco de desenvolver determ inadas doenças autoim unes,
j á em 1967 est a associação era conhecida ( Am iel, 1967) .
Devido às suas m últ iplas funções, nom eadam ente ao nível da
histocom patibilidade e regulação im unitária, a investigação em torno do
sistem a HLA despertou o interesse de várias equipas de investigação
possibilitando a descoberta de largas centenas de alelos nos diferentes
genes.
1 .2 . N om e n cla t u r a do sist e m a H LA
Esta enorm e diversidade genética tornou necessário, desde m uito cedo,
o estabelecim ento de regras claras ao nível da nom enclatura utilizada,
t endo sido organizado em 1968 por Bernard Am os o prim eiro encont ro do
Com it é de Nom enclatura da Organização Mundial de Saúde (
WHO-Nom enclature- Com m it t ee, 1968) .
Um código de 4 dígitos que distingue os alelos que diferem ao nível das
proteínas codificadas foi im plem entado pela prim eira vez no Relatório de
Histocom patibilidade que decorreu em Nova I orque nesse m esm o ano
( Dupont, 1988) . Desta form a foi possível acom odar os diferentes alelos,
identificados através de m étodos m oleculares, que constituem cada grupo
serológico. A utilização de um asterisco depois da designação do locus
perm itiu distinguir a caracterização alélica por m étodos m oleculares da
caract erização por m ét odos serológicos. Em 1990 foi adicionado um quint o
dígito para distinguir as sequências que diferem apenas por substituições
nucleotídicas sinónim as (m utações silenciosas) (Bodm er et al., 1991) . No
entanto, este sistem a de nom enclatura j á está a atingir, para certos grupos
de alelos (e.g HLA- A* 02 e HLA- B* 15) , o nível m áxim o de alelos que
consegue com port ar pelo que já est ão a ser adopt adas novas convenções.
Assim , para acom odar novos alelos nos grupos m ais polim órficos, assim que
estes atinjam 99 alelos, um a segunda série será utilizada para continuação.
A série B* 95 está assim reservada para acom odar novos alelos quando o
B* 1599 for atingido, da m esm a form a que a série A* 92 dará continuidade
aos alelos do grupo A* 02 ( Marsh et al., 2002) .
1 .3 . Té cn ica s de ca r a ct e r iz a çã o do sist e m a H LA
A evolução das técnicas utilizadas na identificação e caracterização das
diferentes form as dos genes que com põem o sist em a HLA const it uiu um dos
factores cruciais no desenvolvim ento dos conhecim entos nesta área. Os
prim eiros passos na descobert a do sist em a HLA foram dados com recurso
às técnicas de leuco- aglutinação com as quais foi possível definir vários
antigénios codificados por este conj unto de genes ( Berah e Dausset, 1963) .
At é m eados dos anos 80 do século XX as t écnicas serológicas const it uíam a
O teste linfocitotóxico foi am plam ente divulgado na abordagem
serológica para identificação dos antigénios do sistem a HLA, existindo
actualm ente m últiplas variações desta técnica base. Esta técnica consiste na
m istura de linfócitos B com soro contendo anticorpos específicos para
determ inados antigénios do sistem a HLA. Os anticorpos, quando na
presença de linfócitos B contendo antigénios com plem entares, ligam- se
provocando , com a adição de um com plem entar, a destruição m em branar,
o que permit e a sua identificação por coloração ( Kippax et al., 1985;
Zachary et al. 1995).
Devido a problem as de reacção cruzada e indisponibilidade de certos
anticorpos, m uitos laboratórios adoptaram técnicas de genética m olecular
para a caract erização do sistem a HLA. No entanto, estes m ét odos
serológicos continuam a ser am plam ente utilizados em testes de
crossm at ching com o obj ectivo de validar potenciais dadores seleccionados
para transplantes de órgãos e tecidos. Este procedim ento é desenvolvido
com o salvaguarda à ocorrência de rej eição aguda do transplante pois alguns
pacientes poderão ter sido previam ente sensibilizados contra determ inados
antigénios HLA. Mesm o que os alelos do dador e do receptor sej am
com patíveis, em sequência de transfusões de sangue, da realização de
outros transplantes ou de um a gravidez, o paciente poderá ter desenvolvido
anticorpos contra alguns dos antigénios do dador levando a um a reacção
violenta logo no início do transplante, pondo em causa o seu sucesso
( Moor e et al., 1997)1.
Os m ét odos de rest rição enzim át ica ( RFLP- Restriction Fragm ent Length
Polym orphysm ) perm itiram dar os prim eiros passos na identificação
1
genética destes alelos no início da década de 80 do século XX perm itindo
um a m aior precisão com parativam ente aos m ét odos serológicos. Com o
advent o da PCR ( Polym erase Chain Reaction) (Saiki et al., 1985, Mullis e
Faloona, 1987), particularm ente após a sua autom atização, depressa os
m étodos de caracterização alélica do sistem a HLA por restrição enzim ática
evoluíram e m ais t arde foram m esm o ult rapassados por out ros
procedim ent os m ais m odernos ( Howell et al., 1989; Ot a et al., 1992) . A
associação entre a PCR e a restrição enzim ática perm itiu a adopção de um a
técnica de caracterização alélica do sistem a HLA m ais eficiente e em alta
resolução. Após a am plificação dos exões polim órficos do locus HLA em
estudo, o DNA obtido por PCR é digerido com enzim as de rest rição
específicas e o perfil dos fragm entos produzidos é analisado após separação
por electroforese ( Mitsunaga et al., 1998) .
Vários m ét odos foram desenvolv idos com b ase na PCR. A caracterização
alélica por SSOP (Sequence Specific Oligonucleotide Probes) recorre a
prim ers (oligonucleotidos) com plem ent ares de zonas conservadas para
am plificar os exões polim órficos dos loci HLA ( exões 2 e 3 nos genes HLA
classe I e exão 2 nos genes classe I I ). O DNA am plificado é fixo a um a
m em brana e é hibridado com sondas oligonucleotídicas m arcadas com
fluorescência. As sondas com plem entares às zonas polim órficas do DNA
am plificado produzem um padrão de hibridação cuja análise perm ite
identificar os alelos presentes do sistem a HLA (Middleton, 2000). Esta
técnica perm ite a caracterização do sistem a HLA em baixa ou alta resolução
consoante o painel de sondas utilizado e é adequada para grandes
O teste SSP (Sequence Specific Prim ing) utiliza directam ente a técnica
de PCR para identificar os alelos do sistem a HLA com recurso a
oligonucleotidos com plem entares a sequências nucleotídicas polim órficas
conhecidas ( Olerup e Zetterquist, 1992; Olerup e Zetterquist, 1993; Bunce
et al., 1995). Esta técnica baseia- se no fact o de um prim er com plem entar à
cadeia de DNA ser m ais eficiente na am plificação por PCR do que os prim ers
com um ou m ais nucleótidos não coincidentes. Prim ers específicos para a
am plificação de determ inados alelos, com separação e visualização através
de electroforese em géis de agarose, perm item caracterizar em baixa ou
alta resolução os genes HLA (Middleton e William s, 1997).
A caracterização alélica dos genes do sistem a HLA através da
sequenciação nucleotídica (SBT- Sequence Based Typing) é o m étodo
m olecular actualm ente m ais fiável, o qual perm ite inclusive a identificação
directa de novos alelos (McGinnis et al., 1995; Middleton e William s, 1997;
Kurz et al., 1999). Esta técnica desenvolve- se com a am plificação, por PCR,
dos fragm ent os cont endo os exões polim órficos a sequenciar. As técnicas e
tecnologias m ais recentes perm item a sequenciação directa do fragm ento
sendo que a sua natureza heterozigótica im plica a utilização de um
program a inform ático próprio para a descrim inação dos alelos presentes em
cada am ost ra.
A escolha do m étodo a utilizar na caracterização alélica do sistem a HLA
depende das especificidades de cada laboratório e dos obj ectivos do
trabalho. Desde a rapidez necessária at é ao núm ero de am ost ras, passando
pelo equipam ento existente, orçam ento disponível e experiência dos
recursos hum anos, inúm eras são as variáveis que influenciam a escolha do
nom eadam ente se pretendem baixa ou alta resolução, poderão optar por
utilizar m ais de um m étodo. No entanto, em qualquer dos casos, qualquer
um dos m étodos disponíveis necessita de ser continuam ente actualizado por
form a a integrar as alterações que perm item a evolução das técnicas e
considerar os novos alelos que continuam ente são descobertos ( Middleton e
William s, 1997).
1 .4 . Est r u t u r a e f u n çã o d o sist e m a H LA
Os loci HLA est ão localizados no braço m ais pequeno do crom ossom a 6
na banda 6p21.3 form ando um com plexo sistem a de genes funcionalm ente
relacionados entre si (Figura 1) (Bender et al. 1983) . O sistem a HLA é
const it uído por 224 genes, dos quais 128 são genes funcionais e 96 são
pseudogenes ( MHC sequencing consortium , 1999) .
Os genes funcionais do sistem a HLA são os m ais variáveis do genom a
hum ano com capacidade de expressão, 40% dos quais desem penham um
im portante papel em funções im unológicas. Para além destes genes existem
ainda no sistem a HLA elem entos repetitivos que com preendem quase 50%
da sequência do sistem a HLA (MHC sequencing consortium , 1999; Garrigan
& Hedrick, 2003) .
O sistem a HLA, com cerca de 4 Mb ( m egabases) de ext ensão, est á
subdividido em três regiões: classe I , classe I I e classe I I I , de acordo com a
estrutura e função dos seus genes. Os genes classe I, localizados na porção
m ais telom érica do sistem a HLA, são const it uídos por vários exões,
intercalados por intrões, que codificam para a form ação da cadeia
polipeptídica alfa de receptores glicoproteicos da m em brana das células
nucleadas, constituídos por um a região extracelular de ligação a antigénios,
um a região transm em branar e um segm ento citoplasm ático ( MHC
sequencing consortium , 1999) . Os polim orfism os nos genes classe I
localizam - se essencialm ente nos exões 2 e 3, os quais codificam para as
estruturas m oleculares alfa 1 e alfa 2 do receptor m em branar onde se
localiza o sítio de ligação aos antigénios ( Malissen et al., 1982) . Estes
receptores m em branares interagem com linfócitos T CD8 positivos que
detectam alterações de expressão nas células, alterações estas que podem
ocorrer devido a infecções ou ao desenvolvim ento de células tum orais.
Desta form a os linfócitos T ( CD8+ ) reconhecem as células que deverão
atacar para com bater o avanço de um a determ inada infecção ou de um
t um or ( Doherty & Zinkernagel, 1975).
Os genes classe I I localizam - se na região m ais centrom érica do sist em a
que se expressam nas células que apresentam antigénios, nom eadam ente
linfócitos B e células dendríticas. O exão 2, que codifica para a estrutura
extracelular do receptor m em branar, responsável pela ligação aos
antigénios, concentra a m aior parte dos polim orfism os nestes genes. Estes
receptores m em branares, constituídos por um a porção extracelular, um a
região transm em branar e um segm ento citoplásm ático, reconhecem a
presença de elem entos estranhos e estim ulam os linfócitos T CD4 positivos,
reforçando a resposta im unitária contra agentes infecciosos ( Doherty &
Zinkernagel, 1975; Gat t i & Pierre, 2003) .
A região classe I I I do sistem a HLA localiza- se entre os genes classe I e
classe I I e, apesar de não ser tão polim órfico com o estes últim os, constitui
o segm ent o do genom a hum ano com m aior densidade de genes ( Xie et al.,
2003)1. Ao contrário da região classe I e classe I I onde existem dezenas de
pseudogenes, a região classe I I I possui apenas dois ( MHC sequencing
consort ium , 1999) . Os genes presentes nesta região não são, na sua
m aioria, relacionados entre sim nem com os genes da classe I e I I e não
possuem um padrão com um de expressão. Os genes classes I I I dos sistem a
HLA possuem funções várias, destacando- se o seu papel na codificação de
proteínas solúveis im portantes na m odelação e regulação da resposta
imunitária ( Xie et al., 2003)1.
Os genes clássicos A, B, Cw (classe I ), DRB1,
HLA-DPB1 e HLA- DQB1 (classe I I ), co- dom inantes, são os m ais polim órficos e
m ais est udados do sist em a HLA (Mizuki et al., 1996) . Por out ro lado, os
genes não clássicos, nom eadam ente HLA- E, HLA- F e HLA- G ( classe I ) , com
funções ainda m al conhecidas, possuem um a distribuição m uito restrita,
baix os níveis de expressão e são pouco polim órficos com parat ivam ent e aos
genes clássicos, com os quais partilham um a estrutura sim ilar ( Le
Bouteiller, 1997).
Os genes classe I e I I constituem no seu conj unto 1998 alelos
diferentes, sendo o m ais polim órfico o locus HLA- B com 627 alelos,
seguindo- se o HLA- DRB1 com 394, o HLA- A com 349, o HLA- Cw com 182 e
o HLA- DPB1 com 116 ( Marsh et al., 2005) .
1 .5 . Evolu çã o e dive r sifica çã o do sist e m a H LA
O MHC, cuj o correspondente hum ano é o sistem a HLA, surgiu através de
repetidas duplicações e conversões de genes ao longo de m ilhões de ano no
decorrer da evolução dos vertebrados desde a divergência dos ciclóstom os
(Kasahara et al., 1996; Kasahara et al., 1997). A análise do MHC de alguns
vertebrados m ostra diferenças notórias, nom eadam ente ao nível da
inclusão, ordem e sequências nucleotídicas dos genes m as, por outro lado,
revela a existência de um a estrutura com um principalm ente entre os
m am íferos ( Yuhki et al. 2003)1. Ao cont rário do que sucede nos m am íferos,
nout ros vert ebrados com o aves e peixes, os loci MHC nem sem pre est ão
reunidos num único conjunto, dispersando- se no m esm o ou em
crom ossom as diferent es ( Miller et al., 1994; Bingulac- Popovic et al., 1997) .
A grande quantidade e diversidade de inform ação disponível
relativamente à variabilidade, estrutura e função dos genes do sistem a HLA
apresentam padrões de variação que m ostram sinais evidentes de selecção.
A frequência de hom ozigóticos em vários loci HLA é significativam ente
inferior ao esperado em condições neutrais em inúm eras populações
1
hum anas. Vários loci do sistem a HLA subm etidos ao teste de neutralidade
Ewens- Watterson têm rejeitado a neutralidade em inúm eras populações
( Tiercy et al., 1992; Ellis et al., 2000; Mack et al., 2000; Renquin et al.,
2001; Piancatelli et al., 2004; Cao et al., 2004) . A variação nos loci HLA
está essencialm ente concentrada nas regiões envolvidas na codificação dos
sítios de ligação aos antigénios dos receptores m em branares, situação que
não é susceptível de ter sido originada a partir de um a evolução neutral
( Parham et al., 1988; Hedrick et al., 1991; Valdes et al., 1999). A m aior
acum ulação de substituições não sinónim as nas regiões que codificam para
os sítios de ligação aos antigénios revela tam bém a actuação da selecção
natural, privilegiando o aum ento da variação que neste caso é
funcionalm ente relevante ( Nei & Goj obori, 1986; Hughes & Nei, 1988) .
O m ecanism o que parece ter m aiores responsabilidades na m anutenção
dos elevados níveis de polim orfism o encontrados nos genes do sistem a HLA,
e que fazem deles os m ais variáveis do genom a hum ano, é a selecção
oscilante, decorrente da acção de diferentes parasitas (Hedrick & Thom son,
1983; Hedrick, 1999) .
A selecção oscilante refere- se a m ecanism os de selecção natural que
conduzem à m anutenç ão de polim orfism os genéticos num a população, em
oposição à selecção direccional que favorece um único alelo. Entre os
diferentes m ecanism os inerentes à selecção oscilante destacam - se com o os
m ais estudados a “ vantagem dos heterozig ót icos” e a “ selecção dependente
da frequência” . A “ vantagem dos heterozigóticos” é baseada na ideia de que
os het erozig ót icos são capazes de reconhecer o dobro dos parasit as, um por
cada alelo, apresentando um a vantagem adaptativa em relação aos
frequência” baseia- se no pressuposto de que os alelos m ais raros possuem
um a forte vantagem selectiva relativam ente aos m ais com uns para os quais
os parasitas podem ter j á desenvolvido resistência ( Bodm er, 1972).
1 .6 . I m por t â n cia m é dica do sist e m a H LA
I núm eros est udos t êm dem onst rado que os genes do sist em a HLA est ão
associados a m ais de 500 doenças aut oim unes ou provocadas por agent es
externos. Cerca de 4% da população em países industrializados são
afect ados por doenças autoim unes, núm ero este com tendência para
aum entar. A predisposição para o desenvolvim ento destas doenças é m uitas
vezes o result ado de um a com binação em rede de vário s alelos do sistem a
HLA. Desta form a, a caracterização alélica do sistem a HLA pode ser usada
para identificar indivíduos com elevado risco de desenvolver determ inadas
doenças assim com o auxiliar no seu diagnóstico. Tendo em conta as
evidências actuais do envolvim ento directo dos loci HLA e os progressos no
conhecim ento da estrutura e função dos produt os dest es genes na respost a
im unitária, poderão, a curt o prazo, ser m elhor com preendidos os
m ecanism os responsáveis pelo desenvolvim ento destas doenças. Assim ,
com esta inform ação disponível, será possível actuar de um a form a
específica ao nível da prevenção e com bat e a est as enferm idades ( Thorsby,
1997; Thorsby and Lie, 2005) .
A e spondilit e a nquilosa nt e, um a doença reum ática crónica e
inflam atória, m uitas vezes progressiva, que afecta a coluna vertebral e as
articulações sacroilíacas, está fortem ente associada à presença do alelo
HLA- B* 27. O HLA- B* 27 contribui em 16 a 50% para o risco genético de
dependente de outros factores genéticos e am bientais ainda desconhecidos
(Reveille et al., 2001; Calin, 2002). A espondilite anquilosant e t em um a
prevalência de 0,2 a 0,8% em populações Caucasianas e cerca de 90% dos
doent es são HLA- B* 27 posit ivos, caract eríst ica que só acont ece em
aproxim adam ente 2% da população Portuguesa ( Braun et al., 1998;
Marker- Herm ann & Hoehler, 1998; Khan, 2000; Gonzalez et al., 2002;
Spínola et al., 2005) . As propriedades do HLA- B* 27 que determ inam esta
susceptibilidade à doença são ainda desconhecidas m as algum as
possibilidades têm sido aventadas, nom eadam ente a hipótese do “ péptido
artritogénico” . Esta hipótese propõe que um a infecção bacteriana provoca
um a resposta dos linfócitos T contra um péptido estranho ao organism o
m as estruturalm ente sim ilar a outro derivado do tecido articular norm al,
envolvendo os recept ores HLA- B* 27. Desta form a, o sistem a im unitário
at aca os t ecidos norm ais do próprio organism o não os reconhecendo com o
seus, levando ao desenvolvim ento da espondilite anquilosante (Benjam in &
Parham , 1990; Ram os & Lopez de Cast ro , 2002) .
A a r t r it e r e u m a t óide é um a doença autoim une que pode assum ir
vários graus de desenvolvim ento e que causa inflam ações crónicas das
articulações, podendo afectar tam bém outros tecidos do organism o
( Cornelia & Goronzy, 2000) . Apesar de ser ainda desconhecida a causa
exacta que origina est a doença, sabe- se que a inflam ação da m em brana
sinovial das articulações ocorre pela reacção autoim une das células T (Cope
& Sonderst rup, 1998) . Vários alelos do gene HLA- DRB1 ( DRB1* 0101,
DRB1* 0102, DRB1* 0401, DRB1* 0404, DRB1* 0405, DRB1* 0408,
DRB1* 1001 e DRB1* 1402) est ão associados a um risco elevado de
que um destes alelos eleva ainda m ais esse risco, suportando a ideia do
envolvim ento das células T CD4 positivas, através do reconhecim ento de
determ inado antigénio artritogénico, no desenvolvim ento desta doença
( Weyand et al., 1992; Schm idt et al., 1996; Gibert et al., 2003) .
A dia be t e s t ipo 1, um a doença autoim une específica de apenas um
órgão, resulta da destruição das células beta do pâncreas, responsáveis
pela produção de insulina. A destruição destas células ocorre pela reacção
autoim une das células T CD4 positivas despoletada por m ecanism os ainda
desconhecidos ( Kat z et al., 1995; Healey et al., 1995) .
A susceptibilidade para o desenvolvim ento da diabetes tipo 1 está
associada a várias haplotipos do sistem a HLA, havendo outros que conferem
prot ecção e out ros que aparent em ent e são neut rais (Singal & Blajchm an,
1973; Larsen & Alper, 2004) . A susceptibilidade para o desenvolvim ento de
diabetes tipo 1 é determ inada principalm ente pela com binação de alguns
alelos HLA- DQ e HLA- DR existentes no indivíduo. Os alelos HLA- DQA1* 03,
HLA- DQB1* 0201 e HLA- DRB1* 0405 são os que conferem m aior
susceptibilidade ao desenvolvim ento da doença enquanto que os alelos
HLA- DQA1* 0102, HLA- DQB1* 0602, HLA- DRB1* 0403 e HLA- DRB1* 0406 são
os que conferem m aior prot ecção ( Thorsby, 1997).
A doe n ça ce lía ca (DC) é a enteropatia m ais com um induzida pela
ingestão de alim entos e resulta de um a intolerância perm anente às
proteínas glúten, presentes nos cereais, que provocam um a reacção
autoim une contra as vilosidades da m ucosa intestinal. Cerca de 95% dos
doent es com DC apresent am o haplot ipo HLA- DQA1* 0501- HLA- DQB1* 0201
que quando na presença do haplotipo HLA- DQA1* 0201- DQB1* 0202 agrava
estes alelos, ligam - se aos péptidos glúten e apresentam - nos às células T
induzindo um a resposta inflam atória que causa a doença ( Sollid et al.,
1989; van de Wal et al., 1996; Vartdal et al., 1996).
A esclerose m últ ipla é um a doença inflam atória que afecta o sistem a
nervoso central através da desm ielinização dos neurónios, levando a um a
deficiência acentuada ao nível sensorial, m otor e cognitivo (McFarlin &
McFarland, 1 982a; McFarlin & McFarland, 1982b) .
A presença do haplotipo HLA- DRB1* 1501- HLA- DQB1* 0602 é um fact or
de risco no desenvolvim ento de esclerose m últipla, em bora sejam ainda
desconhecidos os m ecanism os responsáveis pelo desenvolvim ento desta
doença autoim une m ediada pelas células T ( Hillert & Olerup, 1993;
Barcellos et al., 2003; Sospedra & Martin, 2005) .
Out ras doenças t êm revelado um a fort e associação aos genes do sist em a
HLA, nom eadam ente: o desenvolvim ento de psor ía se, um a doença
inflam atória crónica que afecta a pele, o couro cabeludo, unhas e
articulações, e que est á fort em ent e associada à presença do alelo
HLA-Cw * 0602 ( Elder et al., 1994) ; a na rcolepsia, um a doença de perturbação
do sono, está associada à presença do alelo HLA- DQB1* 0602 ( Chabas et
al., 2003) ; a h e m ocr om a t ose h e r e dit á r ia, um a desordem ao nível do
m etabolism o que provoca acum ulação excessiva de ferro em alguns órgãos,
est á fort em ent e associada a um a m ut ação do gene HFE localizado na região
classe I do sist em a HLA (Lyon & Frank, 2001) ; e o desenvolvim ento de
a le r gia s, nom eadam ente ao polén e a alim entos, associadas a alguns
alelos de loci HLA classe I I (Boehncke et al., 1998) .
Pelo seu papel central na estrutura e funcionam ento do sistem a
particular na resistência ou progressão de doenças infecciosas. A título de
exem plo, a progressão da infecção por HI V é condicionada pela presença
de determ inados alelos HLA classe I e classe I I e o alelo HLA- B* 53 confere
prot ecção ao desenvolvim ento da m alária em crianças do Oest e Africano
( Hill et al., 1991; Al Jabri, 2002).
A com patibilidade nos genes HLA- A, HLA- B e HLA - DRB1 entre o dador e
recept or no t r a n spla n t e de ór gã os e t e cidos é fundam ental para o seu
sucesso por dim inuir significativam ente a probabilidade de rejeição
( Cicciarelli, 2 0 0 4) . Apesar de ser m ais provável encontrar um dador entre
indivíduos aparentados nem sem pre isso é possível pelo que tem surgido
em vários países regist os de dadores de órgãos e t ecidos, nom eadam ente
de m edula óssea.
1 .7 . Aplica çã o dos g e n e s H LA n a s d isp u t a s d e p a t e r n id a d e
Os genes do sistem a HLA têm sido tam bém utilizados em testes de
paternidade, tendo só por si um poder de exclusão superior a 90% (Ejele &
Nwauche, 2004) . Por out ro lado, a taxa de m utação nos HLA é inferior à
que ocorre nos loci STR ( Short Tandem Repeat) pelo que a com binação
destes dois sistem as na resolução de disputas de paternidade oferece
resultados m ais fiáveis, conferindo m aior inform ação e dim inuição das
1 .8 . I m por t â n cia dos ge n e s H LA e m e st u dos popu la cion a is
O ext enso polim orfism o e o linkage desiquilibria existentes entre
diferentes loci do sistem a HLA têm sido am plam ente utilizados com o
m arcadores genéticos em estudos antropológicos. As frequências alélicas e
haplotípicas dos loci HLA variam entre as diferentes populações e grupos
étnicos. Esta variabilidade nos genes HLA tem sido am plam ente investigada
em diferentes populações de form a a clarificar as suas origens e relações
evolutivas ( Pim tanothai et al., 2001; Luo et al., 2002; Piancatelli et al.,
2004; Ayed et al., 2004) .
A caracterização da diversidade alélica e haplotípica nos principais genes
do sistem a HLA constitui um poderoso instrum ento em estudos
populacionais. Estes genes extraordinariam ente polim órficos têm - se
revelado, a par dos m icrosatélites STR e do D- loop do DNA m itocondrial,
m uito im portantes no estudo da historia das m igrações hum anas (Arnaiz
-Villena et al., 2001a; Góm ez- Casado et al., 2001) . Pela sua função
im unitária, os genes HLA sofrem ao longo do tem po a influência da selecção
natural, predom inantem ente um a selecção oscilante devido à irregularidade
geográfica e tem poral das influências dos elem entos patogénicos. Por est e
fact o, est e conj unt o de genes pode não ser t ão út il no est udo de event os
dem ográficos m uito antigos ocorridos há algum as dezenas de m ilhares de
anos ( Hedrick, 1999).
As frequências alélicas nos genes do sistem a HLA têm dem onstrado ser
m ais sim ilares entre populações com origens com uns. Determ inados alelos
são m esm o caract eríst icos de algum as populações hum anas com
determ inadas origens geográficas (Middleton et al. 2004) . Desta form a, as
específicos perm item inferir sobre a origem e relações passadas das
populações.
A título de exem plo, frequências elevadas do alelo HLA- A* 0201 ( acim a
de 20% ) é um a característica m uito com um nas populações Caucasianas
enquanto que as populações da África sub- Sahariana apresentam outros
alelos com o os m ais frequent es, nom eadam ent e o A* 2301 e A* 3001
( Middleton et al. 2004; Cao et al. 2004) . O alelo A* 0201 corresponde a
m ais de 95% dos alelos do grupo A* 02 em populações Europeias
Caucasianas enquanto que nos Chineses de Singapura corresponde apenas
a 25% e m enos de 3% em Hindus I ndianos (Tiercy, 2002) . O A* 0211, raro
noutras populações m undiais, é o alelo m ais frequente do seu grupo no
Norte e Oeste da Í ndia ( Shankarkum ar et al. 2003) .
As populações Europeias apresent am no grupo A* 68 o alelo A* 6801
com o o m ais predom inante enquanto que nas populações sub- Saharianas o
A* 6802 é o m ais prevalent e ( Middleton et al. 2004; Cao et al. 2004).
Tam bém os alelos A* 3001 e A* 3002 apresent am frequências m uit o m ais
elevadas nas populações Africanas do que nas Europeias (Middleton et al.
2004; Cao et al. 2004) . O alelo A* 0206 aparece com frequências elevadas
apenas em populações Asiáticas e Am eríndias, podendo aparecer em
quantidades residuais noutras populações por in fluência das prim eiras
( Middleton et al. 2004) . Os alelos A* 1101, A* 2402 e A* 3303 aparecem nos
Asiáticos com frequências significativam ente m ais elevadas do que noutras
populações. Por out ro lado, os alelos B* 0801 e B* 4402 aparecem nos
Caucasianos com frequências m uito m ais elevadas do que noutras
populações (Cao et al., 2001) . Ent re os Caucasianos o alelo DRB1* 0401 é o
prevalent e é o DRB1* 0405 ( Jaini et al., 2002). Alguns alelos são
considerados específicos do continente Africano ( e.g. A* 0202, A* 0225,
B* 1503, B* 1516, B* 2703, B* 4202 e B* 5703) e a sua presença em
populações de outras origens reflecte m isturas ocorridas no passado ( Cao et
al. 2004) .
O grupo HLA- B* 27 constitui m ais um exem plo das diferenças na
distribuição alélica entre populações. O B* 2705 é o alelo que possui um a
distribuição m ais alargada no globo apresentando frequências m ais
elevadas nas regiões Circum polar e sub- Árctica da Eurásia e Am érica do
Nort e. O B* 2702 possui distribuição restrita às populações Caucasianas,
sendo particularm ente predom inante nalgum as populações Caucasianas do
Médio Orient e (Judeus) e do Norte de África. O B* 2703 é o alelo deste
grupo que predom ina nas populações sub- Saharianas e o B* 2704 o m ais
frequente nas populações Asiáticas (Blanco- Gelaz et al., 2001; Birinci et al.,
2005 ) .
Os alelos dos diferentes genes do sistem a HLA são frequentem ente
t ransm it idos em bloco de geração em geração e cert as com binações
( haplotipos) são encontradas m ais frequentem ente do que o esperado pelas
respectivas frequências alélicas. Este fenóm eno conhecido com o linkage
desiquilibrium resulta de um a localização fisicam ente próxim a dos loci
envolvidos e do efeito das forças selectivas ( Huttley et al. 1999) . Têm sido
identificados inúm eros haplotipos do sistem a HLA cuja distribuição
geográfica coincide com a existência de relações passadas entre as
populações ( Gom éz - Casado et al., 2000; Arnaiz - Villena et al., 2001ª ;
Por exem plo, os haplotipos A* 010101- B* 0801- DRB1* 030101 e A*
2902-B* 4403- DRB1* 0701 são de origem Europeia, considerando- se o prim eiro de
influência Celta ( Muro et al., 2001). O haplotipo A* 020101- B*
440301-DRB1* 070101 t em sido encont rado nas populações do Nort e e Oest e
Europeu (Sanchez- Velasco et al., 2003) . Já os haplot ipos A* 3301- B*
1402-DRB1* 010201 e A* 020101- B* 180101- 1402-DRB1* 1104 são considerados de
origem Mediterrânica (Gom éz - Casado et al., 2000) . O haplot ipo A*
020101-B* 070201- DRB1* 150101 t em sido encont rado em populações Europeias e
Nort e Africanas e os haplot ipos A* 020101- B* 510101- DRB1* 130101,
A* 010101- B* 5801- DRB1* 070101 e A* 3002- B* 180101- DRB1* 030101 têm
sido apontados com o de origem I bérica e Berbere (Gom éz - Casado et al.,
2000; Sanchez- Velasco et al., 2003) .
Desta form a, as frequências haplotípicas do sistem a HLA, associadas às
frequências alélicas, perm item identificar a influência genética nas
populações hum anas das m igrações e contactos existentes entre elas ao
longo do período hist órico e m esm o pré- histórico.
1 .9 . H ist ór ia da s popu la çõe s e m e st u do
O território continental Português é habitado deste o Paleolítico, tendo
sido o receptáculo de várias m igrações de inúm eros povos com diferentes
origens: Celtas, Germ ânicos, Fenícios e um a grande diversidade de povos
da bacia Mediterrânica. Árabes e Norte Africanos ocuparam a m aior parte
deste território durante m ais de 700 anos (Arnaiz - Villena et al., 1997) . No
século XV, devido ao tráfico de escravos, m uitos indivíduos da costa da
Guiné, entre o Senegal e o golfo da Guiné, foram trazidos para Portugal
resultado de um a com plexa e cont ínua rede de m igrações, invasões e
cruzam ento de povos de diferentes origens que se iniciou há 40 m il anos
( Alim en, 1987) . O arquipélago de Cabo Verde, descobert o por volt a de
1460, foi povoado por alguns Europeus e, principalm ente, por escravos
originários da costa Oeste Africana, onde se situa a actual Guiné- Bissau
( Carreira, 1983) . Cabo Verde funcionou durante três séculos com o um
interpost o de escravos Africanos que eram enviados para Portugal
continental, Madeira, Açores e continente Am ericano (Russell- Wood, 1998) .
A ilha da Madeira foi descoberta pelos Port ugueses em 1420 e com eçou
a ser povoada por nobres Portugueses, Judeus, exilados, condenados e
escravos do Nort e de África. Depois da descoberta de Cabo Verde, a
Madeira com eçou a receber escravos da cost a Oest e Africana, os quais
chegaram m esm o a constituir, em m eados do século XVI , 10% da
população ( Pereira, 1989) . A partir do m om ento em que a Madeira se
t ransform ou num pont o im port ant e para o com ércio no At lânt ico Nort e,
inúm eros indivíduos de várias origens Europeias t ais com o Espanhóis,
Italianos, Franceses e Ingleses tam bém se fixaram na ilha ( Russell- Wood,
1998) .
O arquipélago dos Açores foi descobert o na prim eira m et ade do século
XV e oficialm ente povoado em 1439. Este arquipélago constituído por nove
ilhas desem penhou um papel im portante no com ércio m arítim o entre a
Am érica e a Europa durante os séculos XV e XVI ( Monod, 1991). A sua
posição estratégica atraiu povoadores de diferentes origens, a m aior parte
Port ugueses m as t am bém Judeus, escravos Africanos, Flam engos,
Franceses, Italianos, Ingleses e Espanhóis ( Guill, 1993) . O povoam ento do
Flam engos pois j á em 1490 estavam instalados nestas ilhas cerca de 2000
indivíduos provenientes de Flandres (Rogers, 1979).
1 .1 0 . Ob j e ct iv os d a t e se
Esta tese teve por obj ectivo caracterizar em alta resolução a po pulação
Portuguesa relativam ente aos principais genes do sistem a HLA (HLA- A,
HLA- B e HLA- DRB1), distinguindo entre o Norte, Centro e Sul de Portugal
continental, Açores e Madeira. No sentido de obter a inform ação necessária
para m elhor distinguir a influência decorrent e do com ércio de escravos em
que Portugal esteve envolvido nos séculos XV a XVI I , foram tam bém
caracterizadas geneticam ente ao m esm o nível duas populações Africanas,
Guiné- Bissau e Cabo Verde.
Este trabalho pretende contribuir para a clarificação da origem e
influências genéticas da população Portuguesa, em particular das
sub-populações consideradas, e de Cabo Verde e Guiné- Bissau. Através do
est udo do ext enso polim orfism o exist ent e nos genes do sist em a HLA e do
linkage desiquilibria que ocorre ent re os seus loci, pretende- se identificar
m arcadores genéticos (frequências alélicas e haplotípicas) que clarifiquem
as suas origens e relações evolutivas. É ainda obj ectivo deste estudo
relacionar os m arcadores genéticos encontrados com a influência das
m igrações hum anas que envolveram ou afectaram as populações
consideradas.
Assim , a inform ação agora obtida para os loci HLA- A, HLA- B e HLA- DRB1
pretende constituir um contributo im portante, a par dos dados disponíveis
ao nível dos m icrosatélites STR e do D- loop do DNA m itocondrial (Brehm et
Fernandes et al., 2003; Gonçalves et al., 2003; Rosa et al., 2004;
Gonçalves et al., 2005) , para o estudo e caracterização genética das
populações Portuguesa, Cabo- Verdiana e Guineense.
Pelo seu im portante papel ao nível do sistem a im unitário, os genes HLA
estão sujeitos à influência da selecção natural ( Hedrick, 1999), pelo que, no
âm bito deste trabalho, foram procurados sinais da sua actuação.
Os dados obtidos pretendem ainda constituir um a base de inform ação
relevante para o desenvolvim ento de futuros estudos nas populações
Portuguesa, Cabo- Verdiana e Guineense relativam ente à associação dos
genes HLA a m últiplas doenças genéticas. No entanto, desde já, os dados
obtidos pretendem tam bém proporcionar inform ação sobre a prevalência na
população dos alelos e haplot ipos considerados associados a essas doenças
( Thorsby & Lie, 2005) e sobre a m aior ou m enor probabilidade de encontrar
dadores com pat íveis com det erm inados fenotipos para transplantes de
órgãos e t ecidos.
É tam bém propósito deste trabalho com parar a eficiência de duas
técnicas de caracterização genética dos loci HLA ( SSOP e SBT) de form a a
Ca pít u lo 2
2 . M a t e r ia l e m é t od os
2 .1 . Am ost r a ge m da s popu la çõe s
A caracterização do sistem a HLA na população Portuguesa foi
desenvolvida num a am ostra de 432 indivíduos não relacionados entre si,
saudáveis e do sexo m asculino cuj os pais e avós nasceram e viveram na
m esm a região, subdividindo- se de acordo com essa inform ação em Port ugal
continental (n= 145), arquipélago dos Açores ( n= 102) e ilha da Madeira
(n= 185) (Figura 2). Portugal continental foi subdividido em três diferentes
regiões, com lim ites coincidentes com as barreiras naturais form adas pelos
rios Tej o e Douro; Norte de Portugal ( n= 46) , Centro de Portugal ( n= 50) e
Sul de Portugal ( n= 49) . A am ostragem no arquipélago dos Açores
considerou a necessidade de garantir a representatividade das diferentes
ilhas, principalm ente as do grupo Central e Oriental. Na análise do sistem a
HLA na população Açoriana foram tam bém incluídos dados de um a am ostra
de 129 indivíduos da ilha Terceira anteriorm ente publicados (Arm as et al.,
De form a a obter in form ação, não disponível anteriorm ente, essencial na
análise com parativa da população Portuguesa, foram incluídos neste estudo
189 indivíduos saudáveis do sexo m asculino de dois países Africanos;
Guiné- Bissau e Cabo Verde (Figura 2). A am ostra da Guiné- Bissau consistiu
em 62 indivíduos pertencentes a sete diferentes grupos étnicos; balanta
( n= 10) , papel (n= 11), m andinga (n= 9), felupe (n= 5), bijagós (n= 10), fula
( n= 10) e m ancanha ( n= 10) . A am ost ra do arquipélago de Cabo Verde
reuniu um total de 124 indivíduos cuj os pais e avós nasceram e viveram na
m esm a ilha. De acordo com esta inform ação a am ostra foi subdividida em
duas; indivíduos das ilhas do Barlavento (n= 62) e indivíduos das ilhas do
Sot avent o ( n= 62) .
2 . 2 . Ge n e s e st u d a d os
Com o propósito de possibilitar a caracterização do sistem a HLA e inferir
sobre as influências genéticas e origens da população Portuguesa, foram
escolhidos para este estudo os genes HLA- A, HLA- B e HLA- DRB1. A escolha
dest es t rês loci prende- se com o fact o de serem os m ais polim órficos do
sistem a HLA e dos m ais diversos no genom a hum ano, características que
lhes conferem enorm es potencialidades em estudos antropológicos (Mizuki
et al. 1996; Marsh et al. 2005) . Apesar das evidências de que estes genes
estão suj eitos à pressão selectiva, vários estudos em inúm eras populações
m undiais têm perm itido identificar as influências genéticas resultantes de
m igrações e contactos entre populações ocorridos há vários m ilhares de
anos (Arnaiz - Villena et al., 1997, 1999; Gom ez- Casado et al., 2001;
Sánchez- Velasco et al., 2003) .
Por out ro lado, t orna- se im portante a caracterização destes três genes
do sistem a HLA por serem cruciais para o transplante de órgãos e tecidos,
est arem associados ao desenvolvim ent o de inúm eras doenças do foro
im unológico e possuírem grandes potencialidades forenses ( Boehncke et al.,
1998; Gibert et al., 2003; Cicciarelli, 2004; Ej ele & Nwauche, 2004).
2 .3 . M é t odos u t iliz a dos
A caracterização dos genes HLA- A, HLA- B e HLA- DRB1 nas populações
em estudo foi desenvolvida, num a prim eira fase, em m édia resolução com
recurso à técnica SSOP ( Sequence Specific Oligonucleotide Probes)
( Middleton, 2000) . A extracção do DNA foi concretizada através de um
cont endo EDTA (Walsh et al., 1991). O Chelex é um a resina que se agrega
aos diversos com ponentes celulares, excepto o m aterial genético,
perm itindo a extracção de DNA de form a rápida e com boa qualidade para
am plificação por PCR (Vigilant, 1999) . Foram am plificados os exões 2 e 3
dos loci HLA- A e HLA- B e o exão 2 do locus HLA- DRB1 através da técnica
PCR utiliz ando oligonucleotidos específicos. O DNA am plificado foi utilizado,
com recurso à técnica SSOP, para identificar os grupos de alelos do sistem a
HLA. O DNA am plificado foi im obilizado em m em branas e posteriorm ente
hibridado com sondas oligonucleotidicas m arcadas com plem ent ares a
sequências específicas ( Middleton, 2000) .
A caract erização em alt a resolução dos loci HLA- A, HLA- B e HLA- DRB1
em todas as populações em estudo foi desenvolvida através de
sequenciação nucleotídica (SBT- Sequence Based Typing), o m étodo
actualm ente m ais fiável que inclusive perm ite a identificação directa de
novos alelos. A extracção do DNA para os procedim entos de sequenciação
foi efectuada através de um a m etodologia clássica utilizando
fenol-clorofórm io a partir de am ostras de sangue total contendo EDTA. O fenol e
o clorofórm io são solventes orgânicos que possibilitam a rem oção de
proteínas e estruturas celulares da solução aquosa deixando apenas o DNA
( Sam brook et al., 1989) .
Os exões 2 e 3 dos loci HLA- A e HLA- B e o exão 2 do locus HLA- DRB1
foram am plificados por PCR recorrendo a oligonucleotidos específicos ( Kurz
et al., 1999; Williams et al., 2004) (Tabela 1). Os fragm entos am plificados
foram purificados e sequenciados em am bas as direcções num a ABI PRISM
310 GENETI C ANALYZER (Applied Biosystem s) , fazendo uso do kit ABI
Loci Pr im er s F.
( bp) P.
Ex ão 2 Sense AI 5 : 5 ’ GAG GGT CGG GC( G/ A) GGT CTC AGC CA 3 ’
Ant isense 3 AI n 2 SQR2 : 5 ’ CTC GGA CCC GGA GAC TGT 3 ’ 390 ( 1 )
H
LA
-A
Ex ão 3 Sense 3 AI n 2 SQF2 : 5 ’ TTA GGC CAA AAA TCC CCC 3 ’
Ant isen se 3AI n- 6 6 : 5 ’ TGT TGG TCC CAA TTG TCT CCC CTC 3 ’ 419 ( 1 )
Ex ão 2 Sense Bx 1 : 5 ’ GGG AGG AGC GAG GGG ACC ( G/ C) CA G 3 ’
Ant isense Bex 2 R: 5 ’ GGT CAC TCA CCG ( G/ T) C CTC G 3 ’ 363 ( 2 )
H
LA
-B
Ex ão 3 Sense Bex 3 F: 5 ’ GGG GCC AGG GTC TCA CA 3 ’
Ant isense Bin t 3 : 5 ’ GGA GGC CAT CCC CGG CGA CCT AT 3 ’ 344
( 2 )
DRB1* 01 Sense AMP1: 5’ TTC TTG TGG CAG CTT AAG TT 3’
Ant isense AMPB( n) : 5’ CCG CTG CAC ( T/ C) GT GAA ( G/ T) CT CT 3’ 261 ( 3 )
DRB1* 02 Sense DR2 : 5 ’ TTC CTG TGG CAG CCT AAG AGG 3 ’
Ant isense AMPB( n) : 5’ CCG CTG CAC ( T/ C) GT GAA ( G/ T) CT CT 3’ 261
( 3 )
DRB1* 03a Sense 3/ 11/ 6GF: 5’ GT TTC TTG GAG TAC TCT ACG TC 3’
Ant isense DR3R: 5’ GTA GTT GTG TCT GCA GTA ( G/ A) T 3’ 232 ( 4 )
DRB1* 04b Sense AMP4: 5’ GT TTC TTG GAG CAG GTT AAA C 3’
Ant isense AMPB CCG CTG CAC TGT GAA GCT CT 3 ’ 263
( 3 )
DRB1* 07 Sense DR7 : 5 ’ A CGT TTC CTG TGG CAG GG 3 ’
Ant isense AMPB( n) : 5’ CCG CTG CAC ( T/ C) GT GAA ( G/ T) CT CT 3’ 265 ( 3 )
DRB1* 08/ 12c Sense 8/ 12 GF: 5’ GT TTC TTG GAG TAC TCT ACG GG 3 ’
Ant isense AMPB( n) : 5’ CCG CTG CAC ( T/ C) GT GAA ( G/ T) CT CT 3’ 263 ( 4 )
DRB1* 09 Sense DR9 : 5 ’ CGT TTC TTG AAG CAG GAT AAG TT 3 ’
Ant isense AMPB( n) : 5’ CCG CTG CAC ( T/ C) GT GAA ( G/ T) CT CT 3’ 262 ( 3 )
DRB1* 10 Sense DR10 : 5 ’ ACC AGA CCA CGT TTC TTG GAG G 3 ’
Ant isense AMPB( n) : 5’ CCG CTG CAC ( T/ C) GT GAA ( G/ T) CT CT 3’ 259 ( 3 )
DRB1* 11/ 13d Sense 3/ 11/ 6GF: 5’ GT TTC TTG GAG TAC TCT ACG TC 3’
Ant isense 11/ 6R: 5’ GTA GTT GTG TCT GCA GTA GG 3’ 232 ( 5 )
DRB1* 14e Sense 3/ 11/ 6GF: 5’ GT TTC TTG GAG TAC TCT ACG TC 3 ’
Ant isense DR1 4 R: 5 ’ CC GTA GTT GTG TCT GCA A 3 ’ 234 ( 6 )
H LA -D R B 1
Am plifica t am bém os alelos aDRB1* 1107, DRB1* 1127, DRB1* 1333; bDRB1* 1122, DRB1* 1410;
cDRB1* 1105, DRB1* 1317, DRB1* 14( 04/ 11/ 15/ 28/ 31) ; dDRB1* 14 (alelos não amplificados pelos
out ros pr im er s) ; eDRB1* 1113, DRB1* 1117. F. (bp)= tam anho do fragm ento em pares de bases.
P= pr ogr am as de PCR:
( 1) 94º C/ 20 s; 66º C/ 50 s; 72º C/ 22 s ( 10 ciclos) e 94º C/ 20 s; 65º / 50 s; 72º C/ 22s ( 22 ciclos) . ( 2) 96º C/ 20 s; 68º C/ 30 s; 72º C/ 30 s ( 35 ciclos) .
( 3) 96º C/ 1 m ; 58º C/ 1 m ; 72º C/ 1 m ( 38 ciclos) .
( 4) 96º C/ 1 m ; 64º C/ 1 m ; 72º C/ 1 m ( 15 ciclos) e 96º C/ 1 m ; 56º C/ 1 m ; 72º C/ 1 m ( 25 ciclos) . ( 5) 96º C/ 1 m ; 65º C/ 1 m ; 72º C/ 1 m ( 15 ciclos) e 96º C/ 1 m ; 62º C/ 30 s ; 72º C/ 1 m ( 25 ciclos) . ( 6) 96º C/ 1 m ; 60º C/ 1 m ; 72º C/ 1 m ( 38 ciclos) .
Ta bela 1 .Prim ers e r espect iv os pr ogr am as de PCR ut ilizados na am plificação e
Biosyst em s, Fost er Cit y, CA, USA) e dos prim ers utilizados na am plificação.
Antes da sua utilização na reacção de sequenciação, o produto de PCR foi
convenientem ente purificado por centrifugação em colunas de Sephadex
G-50 (Pharm acia) para rem oção do excesso de prim ers e nucleótidos. A
reacção de sequenciação, através da técnica de PCR, perm itiu a obt enção
de inúm eros fragm ent os em cadeia sim ples com vários t am anhos, t ant os
tam anhos quantos nucleótidos existentes na sequência. Estes fragm entos
foram obtidos através da utilização de nucleótidos m arcados com
fluorescência (um a m arcação diferente para cada um dos quatro nucleótidos
do código genético) , os quais term inam a polim erização da nova cadeia
sem pre que são incluídos. Após rem oção do excesso de nucleótidos
m arcados, a utilização do sequenciador autom ático para a separação dos
fragm entos e a identificação do nucleótido inserido no fim de cada um deles
perm ite a obtenção da sequência nucleotídica do fragm ento analisado1.
As sequências obtidas foram analisadas com o program a Matchtools
Allele I dentification (Applied Biosystem s) de form a a identificar os alelos
present es. Os casos em que os polim orfism os no codão 86 do exão 2 do
locus HLA- DRB1 resultaram em am biguidades na identificação dos alelos,
foram utilizados os prim ers ant isense Dr86GR ( 5’ CTG CAC TGT GAA GCT
CTC AC 3’) e Dr86VR ( 5’ CTG CAC TGT GAA GCT CTC CA 3’) juntam ente
com os prim ers sense (Tabela 1) em duas am plificações separadas. Nestes
casos o program a de PCR utilizado foi o seguinte: 96º C/ 1 m ; 66º C/ 1 m ;
72º C/ 1 m ( 15 ciclos) e 96º C/ 1 m ; 58º C/ 1 m ; 72º C/ 1 m ( 25 ciclos)
(Williams et al., 2004) .
2 .4 . An á lise de da dos
Vários parâm etros genéticos foram calculados ou estim ados através da
aplicação do program a Arlequin v2.000 ( Schneider et al., 2000) aos
resultados obtidos: frequências alélicas, diversidade genética, equilíbrio
Hardy- Weinberg, frequências haplotípicas, linkage desiquilibrium, testes de
neutralidade selectiva (Ewes- Watterson, Slatkin e Chakraborty) e análise de
variância m olecular (AMOVA).
As frequências alélicas são calculadas pela divisão do núm ero de vezes
que cada alelo for encontrado pelo t ot al exist ent e (equivalente ao núm ero
de crom ossom as analisados) . Com um a am ostra adequada, as frequências
alélicas calculadas perm item um a boa aproxim ação à real prevalência dos
diferentes alelos na população. Esta inform ação é fundam ental para a
caract erização das populações em estudo e desenvolvim ento de análises
com parativas. As frequências alélicas nos genes em estudo não possuem
um a distribuição hom ogénea nas populações hum anas, sendo m uito
dependente da sua origem geográfica t ransform a- se num a característica
específica das populações (Middleton et al., 2004) .
A diversidade genética, equivalente à heterozigotia, corresponde à
probabilidade de dois alelos escolhidos ao acaso, num determ inado locus,
não estarem presentes no m esm o indivíduo ( Nei, 1987) . Com o
desenvolvim ento e aplicação das técnicas m oleculares na caracterização
alélica dos genes do sistem a HLA é cada vez m ais im provável a identificação
errada de indivíduos hom ozigóticos, tornando este parâm etro de
variabilidade genética cada vez m ais robust o.
O equilíbrio de Hardy- Weinberg estipula que um a população em