UNIVERSIDADEFEDERALDO RIO GRANDE DO NORTE
UNIVERSIDADEFEDERAL DORIOGRANDE DO NORTE
CENTRO DETECNOLOGIA
PROGRAMA DEPÓS-GRADUAÇÃO EMENGENHARIAELÉTRICA E DECOMPUTAÇÃO
Contribuições a técnicas de agrupamento e
visualização de dados multivariados utilizando
mapas auto-organizáveis
Leonardo Enzo Brito da Silva
Orientador: Prof. Dr. José Alfredo Ferreira Costa
Dissertação de Mestrado apresentada ao
Programa de Pós-Graduação em Engenharia Elétrica e de Computação da UFRN (área de concentração: Engenharia de Computação) como parte dos requisitos para obtenção do título de Mestre em Ciências.
UFRN / Biblioteca Central Zila Mamede Catalogação da Publicação na Fonte
Silva, Leonardo Enzo Brito da.
Contribuições a técnicas de agrupamento e visualização de dados multivariados utilizando mapas auto-organizáveis. / Leonardo Enzo Brito da Silva. – Natal, RN, 2013.
114 f.: il.
Orientador: Prof. Dr. José Alfredo Ferreira Costa.
Dissertação (Mestrado) – Universidade Federal do Rio Grande do Norte. Centro de Tecnologia. Programa de Pós-Graduação Engenharia Elétrica e da Computação.
1. Mapas auto-organizáveis – Redes neurais - Dissertação. 2. Mineração de dados - Dissertação. 3. Técnicas de visualização - Dissertação. 4. Algoritmos de agrupamento - Dissertação. I. Costa, José Alfredo Ferreira. II. Universidade Federal do Rio Grande do Norte. III. Título.
Agradecimentos
Agradeço a Deus por me guiar sempre na jornada da minha vida.
À minha família por multiplicar as alegrias e enfrentar comigo os desafios, me dando inabalável suporte em todos os momentos.
Ao meu orientador, professor Alfredo, pelos inúmeros ensinamentos e por confiar no nosso trabalho.
Aos membros do Laboratório de Sistemas Adaptativos e demais colegas de pós-graduação, que contribuíram direta ou indiretamente no desenvolvimento deste trabalho.
Ao CNPq, pelo apoio financeiro durante o mestrado.
Os mapas auto-organizáveis (SOM) são redes neurais artificiais amplamente utiliza-das no campo da mineração de dados, principalmente por se constituírem numa técnica de redução de dimensionalidade dada a grade fixa de neurônios associada à rede. A fim de particionar e visualizar adequadamente a rede SOM, os diversos métodos existentes na literatura devem ser aplicados em uma etapa de pós-processamento nos seus neurô-nios, visando inferir características relevantes do conjunto de dados. Em geral, tal pro-cessamento efetuado sobre os neurônios da rede, ao invés do conjunto de dados em sua totalidade, reduz o custo computacional, dada a quantização vetorial.
Este trabalho propõe pós-processamentos dos neurônios da rede SOM nos espaços de entrada e de saída, aliando técnicas de visualização a algoritmos baseados na força gravi-tacional e na procura do menor caminho com maior recompensa. Tais métodos levam em consideração forças de ligação entre neurônios vizinhos e características de distâncias e densidade de padrões, ambas associadas a posição que o neurônio ocupa no espaço dos dados após o treinamento da rede. Dessa forma, busca-se definir mais nitidamente a dis-posição dos agrupamentos presentes nos dados. Experimentos foram realizados para ava-liar os métodos propostos utilizando diversos conjuntos de dados gerados artificialmente, assim como conjuntos de dados do mundo real. Os resultados obtidos foram comparados com aqueles provenientes de alguns métodos bem conhecidos existentes na literatura.
Palavras-chave: Mapas Auto-Organizáveis, Mineração de Dados, Técnicas de
Abstract
Self-organizing maps (SOM) are artificial neural networks widely used in the data mining field, mainly because they constitute a dimensionality reduction technique given the fixed grid of neurons associated with the network. In order to properly the partition and visualize the SOM network, the various methods available in the literature must be applied in a post-processing stage, that consists of inferring, through its neurons, relevant characteristics of the data set. In general, such processing applied to the network neurons, instead of the entire database, reduces the computational costs due to vector quantization. This work proposes a post-processing of the SOM neurons in the input and output spaces, combining visualization techniques with algorithms based on gravitational for-ces and the search for the shortest path with the greatest reward. Such methods take into account the connection strength between neighbouring neurons and characteristics of pattern density and distances among neurons, both associated with the position that the neurons occupy in the data space after training the network. Thus, the goal consists of defining more clearly the arrangement of the clusters present in the data. Experiments were carried out so as to evaluate the proposed methods using various artificially gene-rated data sets, as well as real world data sets. The results obtained were compared with those from a number of well-known methods existent in the literature.
Keywords: Data Mining, Self-Organizing Maps, Visualization Techniques,
Sumário i
Lista de Figuras v
Lista de Tabelas xiii
Lista de Símbolos e Abreviaturas xv
1 Introdução 1
1.1 Justificativa e Motivação . . . 2
1.2 Problema . . . 3
1.3 Metodologia . . . 3
1.4 Objetivos do trabalho . . . 4
1.4.1 Objetivo Principal . . . 4
1.4.2 Objetivos Específicos . . . 4
1.5 Organização do texto . . . 5
2 Mapas Auto-Organizáveis 7 2.1 Introdução . . . 7
2.2 Algoritmos de Treinamento . . . 9
2.3 Configuração dos parâmetros da rede . . . 10
2.4 Qualidade do SOM . . . 11
3 Técnicas de Visualização usando SOM 13 3.1 Introdução . . . 13
3.2 Visualizações baseadas em distância de protótipos . . . 14
3.2.1 Matriz U . . . 14
3.2.2 Planos de componentes . . . 15
3.2.3 Matriz GC . . . 16
3.2.4 DISTvis . . . 17
3.3 Visualizações baseadas em densidade de padrões . . . 18
3.3.1 Histograma de hits . . . 18
3.3.2 Histograma de dados suavizado . . . 18
3.3.3 Matriz P . . . 19
3.3.4 CONNvis . . . 20
3.4 Técnicas Híbridas . . . 22
3.4.1 Matriz U* . . . 22
3.4.2 CONNDISTvis . . . 22
3.5 Comentários Finais . . . 23
4 Forças de conexão entre neurônios 25 4.1 Introdução . . . 25
4.2 Sistema Proposto . . . 28
4.2.1 Bloco SOM treinado . . . 28
4.2.2 Blocos dos especialistas . . . 29
4.2.3 Bloco combinador . . . 31
4.3 Resultados e Discussões . . . 31
4.4 Conclusões . . . 36
5 Métodos de visualização derivados 37 5.1 Introdução . . . 37
5.2 Abordagem Proposta . . . 37
5.3 Resultados . . . 39
5.4 Conclusões . . . 53
6 Espaço de Características 55 6.1 Introdução . . . 55
6.2 Abordagem Proposta . . . 56
6.3 Experimentos . . . 57
6.4 Conclusões . . . 64
7 Algoritmos de agrupamento gravitacionais 65 7.1 Introdução . . . 65
7.2 Algoritmos de agrupamento gravitacionais . . . 66
7.2.1 Variantes do algoritmo de agrupamento gravitacional . . . 66
7.2.2 Mapa auto-organizável gravitacional - gSOM . . . 68
7.2.3 Comentários Finais . . . 69
7.3 Abordagem Proposta . . . 70
7.4 Resultados . . . 73
7.5 Conclusões . . . 78
8 Identificação do Núcleo de Regiões Neurais 81 8.1 Introdução . . . 81
8.2 Método Proposto . . . 81
8.3 Experimentos . . . 86
8.3.1 Parâmetros de configuração . . . 86
8.3.2 Resultados e discussões . . . 86
8.4 Comparações com outros métodos . . . 92
10 Publicações 105
Referências bibliográficas 106
Lista de Figuras
1.1 A motivação do trabalho consiste em unir informações relativas a den-sidade e distâncias entre neurônios do SOM, de modo a propiciar uma
melhor observação da estrutura dos dados. . . 3
2.1 A rede SOM faz um mapeamento do espaço n-dimensional dos dados
(es-paço de entrada
A
) para a grade fixa p-dimensional de neurônios (espaçode saída
B
). Dessa forma, o i-ésimo neurônio do mapa representado pelovetor de pesoswié projetado na grade pelo seu vetor posiçãori. . . 7
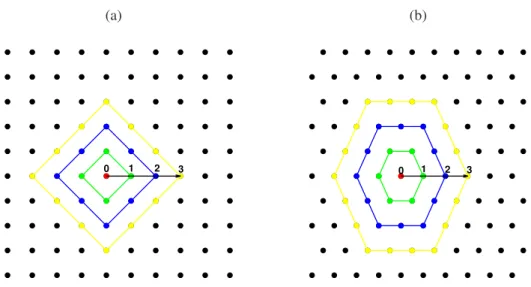
2.2 Para o neurônio ressaltado em vermelho, são ilustradas as vizinhanças 0 (o próprio neurônio), 1 (neurônios em verde), 2 (neurônios em azul) e 3 (neurônios em amarelo) considerando as topologias (a) retangular e
(b)hexagonal. . . 8
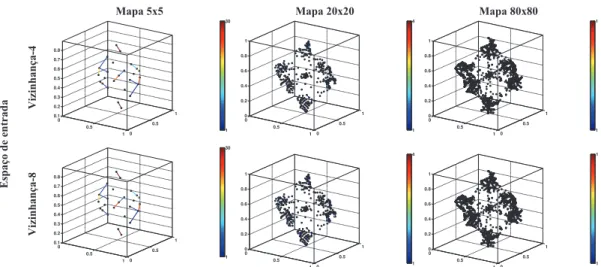
3.1 Redes SOM treinadas com a base de dadosHeptae utilizadas para
exem-plificar as técnicas de visualização. Os neurônios estão representados pe-los pontos em preto e os padrões em vermelho. Os mapas têm os
tama-nhos 5×5, 20×20 e 80×80. . . 14
3.2 Grade da rede SOM e posições relativas dos neurônios na matriz U. . . . 15 3.3 Matrizes U relativas aos mapas da figura 3.1. . . 15 3.4 Planos de componentes relativas aos mapas 5x5, 20x20 e 80x80 da figura
3.1, na primeira, segunda e terceira linhas, respectivamente. . . 16 3.5 Matrizes GC relativas aos mapas da figura 3.1. . . 17
3.6 Histogramas dehitsrelativas aos mapas da figura 3.1. . . 18
3.7 Histogramas de dados suavizados relativos aos mapas da figura 3.1. . . . 19
3.8 Matrizes P relativas aos mapas da figura 3.1. . . 20 3.9 CONNvis no espaço dos dados considerando vizinhanças 4 e 8, relativas
aos mapas da Figura 3.1. . . 21 3.10 CONNvis no espaço matricial de saída considerando vizinhanças 4 e 8,
relativas aos mapas da Figura 3.1. . . 21 3.11 Matrizes U* relativas aos mapas da figura 3.1. . . 22
neurônioswiewj. (a) Padrões representados pelos pontos azuis e verdes
formam o conjuntos
H
ieH
j, respectivamente. (b) Padrões representadospelos pontos azuis e verdes formam os conjuntosCRi,j eCRj,i,
respecti-vamente. (c) Padrões representados pelos pontos azuis e verdes formam
os conjuntos
M
ieM
i, respectivamente. Distâncias entre os centros de (d)H
ieH
j, (e)CRi,jeCRj,i, e (f)M
ieM
i. . . 274.2 (a) Utilização de neurônios (Nu) com tamanhos crescentes da rede SOM,
enquanto se lida com o mesmo conjunto de dados. (b) Número de ligações não-nulas entre os neurônios quando se utiliza a CONN (baseada nas re-giões de Voronoi) e CONNsphere (baseada em hiperesferas). Para
peque-nos mapas com grandePNRa diferença não é relevante, contudo, quando
se aumenta o tamanho do mapa, o número de conexões da CONNsphere é visivelmente maior do que a CONN. . . 28 4.3 Diagrama de Blocos da máquina de comitê. . . 29 4.4 Parte do bloco de um especialista. A entrada de cada bloco especialista é
uma matriz de similaridade (MS) que contém os valores correspondentes da medidas de dissimilaridade entre todos os neurônios. Para cada limiar o número de componentes conectados (CCL) é determinado por rotula-ção, pequenas regiões conectadas são rejeitadas e o número de agrupa-mentos é armazenado. . . 29
4.5 Segmentando a base de dadosD1. Na primeira coluna tem-se as ligações
provenientes das matrizes de similaridade definidas pelas distâncias de 1 a 3. Na segunda coluna, são mostrados os intervalos de estabilidade do número de agrupamentos negligenciando aqueles menores do que o
percentual α. Na terceira coluna tem-se o grafo podado no espaço de
dados. Embora os pequenos aglomerados não entrem na contagem do número de grupos, se as suas ligações são menores do que o limiar elas permanecem na saída de um determinado especialista. . . 32
4.6 Segmentando a base de dadosD1. Na primeira coluna tem-se as ligações
provenientes das matrizes de similaridade definidas pelas distâncias de 4 a 6. Na segunda coluna, são mostrados os intervalos de estabilidade do número de agrupamentos negligenciando aqueles menores do que o
percentual α. Na terceira coluna tem-se o grafo podado no espaço de
dados. Embora os pequenos aglomerados não entrem na contagem do número de grupos, se as suas ligações são menores do que o limiar elas permanecem na saída de um determinado especialista. . . 33
4.7 (a) Grafo do SOM treinado com o conjunto de dadosD1. (b) e (c) saída
do bloco combinador antes e depois da segunda filtragem, respectivamente. 34 4.8 Número de agrupamentos encontrados na determinação do limiar por
4.9 SOMs rotulados após a segmentação. Os mapas foram treinados com os
seguintes conjuntos de dados: (a)D1, (b)D2, (c) D3, (d)Chainlink, (e)
Tetra, (f)Twodiamonds, (g)Wine, e (h)Iris. . . 35
5.1 Rede SOM 30×30 treinada com as bases (a)D1, (b)Target, (c)Lsun, (d)
Atom, (e)Chainlink, (f)Tetra, (g)Hepta, (h)Irise (i)Wine. Os mapas dos
itens (h) e (i) foram obtidos utilizando análise de componentes principais (PCA). . . 40
5.2 As visualizações referem-se ao mapa treinado com a baseD1ilustrado na
Figura 5.1. (a) Histograma dehitsgerado com hisperesfera de raio igual
a 0.1094. (b) Matriz de interseção. (c) Matriz XOR. (d) Matriz Jaccard.
As matrizes de distâncias Euclidianas formadas com os conjuntos inter-seção são mostradas considerando (e) distâncias mínimas, (f) distâncias
máximas, (g) distâncias médias e (h) distâncias medianas. MatrizeskNN
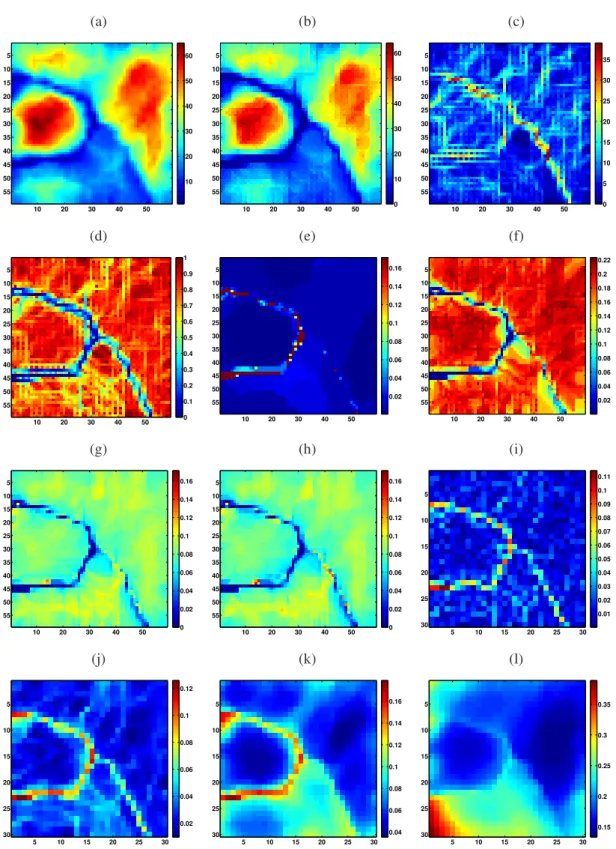
considerando (i)k=1 , (j)k=5 , (k)k=25 e (l)k=125. . . 41
5.3 As visualizações referem-se ao mapa treinado com a baseHeptailustrado
na Figura 5.1. (a) Histograma de hits gerado com hisperesfera de raio
igual a 0.1824. (b) Matriz de interseção. (c) Matriz XOR. (d) Matriz
Jac-card. As matrizes de distâncias Euclidianas formadas com os conjuntos interseção são mostradas considerando (e) distâncias mínimas, (f) distân-cias máximas, (g) distândistân-cias médias e (h) distândistân-cias medianas. Matrizes
kNN considerando (i)k=1 , (j)k=5 , (k)k=25 e (l)k=125. . . 42
5.4 As visualizações referem-se ao mapa treinado com a baseLsunilustrado
na Figura 5.1. (a) Histograma de hits gerado com hisperesfera de raio
igual a 0.1143. (b) Matriz de interseção. (c) Matriz XOR. (d) Matriz
Jac-card. As matrizes de distâncias Euclidianas formadas com os conjuntos interseção são mostradas considerando (e) distâncias mínimas, (f) distân-cias máximas, (g) distândistân-cias médias e (h) distândistân-cias medianas. Matrizes
kNN considerando (i)k=1 , (j)k=5 , (k)k=25 e (l)k=125. . . 43
5.5 As visualizações referem-se ao mapa treinado com a baseAtomilustrado
na Figura 5.1. (a) Histograma de hits gerado com hisperesfera de raio
igual a 0.2012. (b) Matriz de interseção. (c) Matriz XOR. (d) Matriz
Jac-card. As matrizes de distâncias Euclidianas formadas com os conjuntos interseção são mostradas considerando (e) distâncias mínimas, (f) distân-cias máximas, (g) distândistân-cias médias e (h) distândistân-cias medianas. Matrizes
kNN considerando (i)k=1 , (j)k=5 , (k)k=25 e (l)k=125. . . 44
5.6 As visualizações referem-se ao mapa treinado com a baseChainlink
ilus-trado na Figura 5.1. (a) Histograma de hitsgerado com hisperesfera de
raio igual a 0.1881. (b) Matriz de interseção. (c) Matriz XOR. (d) Matriz
Jaccard. As matrizes de distâncias Euclidianas formadas com os con-juntos interseção são mostradas considerando (e) distâncias mínimas, (f) distâncias máximas, (g) distâncias médias e (h) distâncias medianas.
igual a 0.1865. (b) Matriz de interseção. (c) Matriz XOR. (d) Matriz
Jac-card. As matrizes de distâncias Euclidianas formadas com os conjuntos interseção são mostradas considerando (e) distâncias mínimas, (f) distân-cias máximas, (g) distândistân-cias médias e (h) distândistân-cias medianas. Matrizes
kNN considerando (i)k=1 , (j)k=5 , (k)k=25 e (l)k=125. . . 46
5.8 As visualizações referem-se ao mapa treinado com a base Tetrailustrado
na Figura 5.1. (a) Histograma de hits gerado com hisperesfera de raio
igual a 0.1264. (b) Matriz de interseção. (c) Matriz XOR. (d) Matriz
Jac-card. As matrizes de distâncias Euclidianas formadas com os conjuntos interseção são mostradas considerando (e) distâncias mínimas, (f) distân-cias máximas, (g) distândistân-cias médias e (h) distândistân-cias medianas. Matrizes
kNN considerando (i)k=1 , (j)k=5 , (k)k=25 e (l)k=125. . . 47
5.9 As visualizações referem-se ao mapa treinado com a baseIrisilustrado na
Figura 5.1. (a) Histograma de hitsgerado com hisperesfera de raio igual
a 0.2574. (b) Matriz de interseção. (c) Matriz XOR. (d) Matriz Jaccard.
As matrizes de distâncias Euclidianas formadas com os conjuntos inter-seção são mostradas considerando (e) distâncias mínimas, (f) distâncias
máximas, (g) distâncias médias e (h) distâncias medianas. MatrizeskNN
considerando (i)k=1 , (j)k=5 , (k)k=25 e (l)k=125. . . 48
5.10 As visualizações referem-se ao mapa treinado com a baseWineilustrado
na Figura 5.1. (a) Histograma de hits gerado com hisperesfera de raio
igual a 0.4890. (b) Matriz de interseção. (c) Matriz XOR. (d) Matriz
Jac-card. As matrizes de distâncias Euclidianas formadas com os conjuntos interseção são mostradas considerando (e) distâncias mínimas, (f) distân-cias máximas, (g) distândistân-cias médias e (h) distândistân-cias medianas. Matrizes
kNN considerando (i)k=1 , (j)k=5 , (k)k=25 e (l)k=125. . . 49
5.11 As visualizações referem-se ao mapa 5×5 treinado com a baseHepta. (a)
Histograma dehitsobtida com a hiperesfera considerando o raio mínimo.
(b) Matriz de interseção. (c) Matriz XOR. (d) Matriz Jaccard. As matrizes de distâncias Euclidianas formadas com os conjuntos interseção são mos-tradas considerando (e) distâncias mínimas, (f) distâncias máximas, (g)
distâncias médias e (h) distâncias medianas. MatrizeskNN considerando
(i)k=1 , (j)k=5 , (k)k=25 e (l)k=125. . . 50
5.12 As visualizações referem-se ao mapa 20×20 treinado com a baseHepta.
(a) Histograma de hitsobtida com a hiperesfera considerando o raio
mí-nimo. (b) Matriz de interseção. (c) Matriz XOR. (d) Matriz Jaccard. As matrizes de distâncias Euclidianas formadas com os conjuntos inter-seção são mostradas considerando (e) distâncias mínimas, (f) distâncias
máximas, (g) distâncias médias e (h) distâncias medianas. MatrizeskNN
5.13 As visualizações referem-se ao mapa 80×80 treinado com a baseHepta.
(a) Histogramahitsobtida com a hiperesfera considerando o raio mínimo.
(b) Matriz de interseção. (c) Matriz XOR. (d) Matriz Jaccard. As matrizes de distâncias Euclidianas formadas com os conjuntos interseção são mos-tradas considerando (e) distâncias mínimas, (f) distâncias máximas, (g)
distâncias médias e (h) distâncias medianas. MatrizeskNN considerando
(i)k=1 , (j)k=5 , (k)k=25 e (l)k=125. . . 52
6.1 As três regiões coloridas mostradas no gráfico correspondem as regiões que consistem em neurônios interpoladores (vermelho), de fronteira (verde) e de agrupamento (azul). As linhas r, s e t simbolizam hiperplanos gené-ricos que dividem o espaço de características. . . 56 6.2 (a) Matriz U, (b) Matriz P, e (c) Matriz H (com raio definido pela Equação
6.2.) da rede SOM treinada com o conjunto de dadosDS3. . . 58
6.3 (a) Espaço de características dividido em duas regiões utilizando o algo-ritmo k-means. (b) Os dois grupos de neurônios vistos no espaço dos da-dos. Neurônios da rede SOM são mostrados em vermelho (classificados no subconjunto de neurônios interpoladores) e em azul (classificados no subconjunto dos neurônios de agrupamento). Neurônios do mesmo grupo são representados com as mesmas cores nos itens ‘a’ e ‘b’. (c) Conjunto
de dadosDS3filtrado. . . 59
6.4 Imagens o mesmo tamanho que a grade da rede. As partições do espaço
de características são mostradas para (a)k=2, (b)k=3, e (c) k=4, em
que cada cor corresponde a neurônios do mesmo grupo. . . 59 6.5 (a) Neurônios rotulados da rede SOM usando CCL. (b) Dados rotulados. . 59 6.6 (a) Neurônios da rede SOM rotulados através de: (a) k-means no espaço
de características com posterior CCL, (b) k-means no espaço dos dados sobre neurônios do SOM, sendo o DBI o critério para a escolha do
parâ-metrok, e (c) algoritmo watershed sobre a matriz U daFigura 3a. . . . . 60
6.7 Acurácia de classificação média e desvio padrão para a tarefa de agru-pamento realizada pelo k-means e pela rede competitiva tanto no espaço dos dados quanto no espaço de características, bem como o algoritmo watershed aplicado à matriz U gerada pelos SOMs treinados. . . 61 6.8 Eficiência de classificação para a tarefa de agrupamento executada pelo
k-means e pela rede competitiva tanto no espaço dos dados quanto no espaço de características, bem como para o algoritmo watershed aplicado à matriz U gerada pelos SOMs treinados. . . 63
7.1 Caso ilustrativo onde a atração entre wj (ponto vermelho) e wi (ponto
verde) é penalizada por pi,j (média de massa do grupo de neurônios
den-tro do círculo de raioα). Todos os neurônios cujas distâncias awjsão
me-nores ou igual aαsão consideradas como pertencentes ao mesmo grupo
coeso (pontos pretos) e, portanto, o parâmetro pi,j relacionados às suas
indicam qual é o padrão mais próximo para cada neurônio. (b) Gráfico das distâncias Euclidianas do padrão mais próximo para cada neurônio. . 74
7.3 (a) Os pontos vermelhos e pretos correspondem aos padrões da base de
dadosD1e aos neurônios, respectivamente. O ponto amarelo é um
neurô-nio que é o centro da sua hiperesfera correspondente, ilustrada como a circunfência azul. Todos os padrões dentro desta circunferência estão li-gados ao neurônio por linhas azuis. (b) Gráfico do número de padrões
dentro da hiperesfera de cada neurônio. (c) Histograma de hits gerado
com o número de padrões dentro das hiperesferas. (d) Os pontos
ver-melhos e pretos correspondem aos padrões da base de dados D1 e aos
neurônios, respectivamente. Os pontos verdes consistem nos padrões que fazem parte da interseção entre as circunferências de dois neurônios
vizi-nhos. (e) Gráfico do número de padrões na interseção dos neurônios wi
e wj. (f) Matriz de Jaccard cujos valores são calculados entre neurônios
que são 4-vizinhos na grade (espaço de saída). . . 74
7.4 Posições dos neurônios entre as épocas 5 a 30. Os neurônios estão se
reunindo nas regiões mais densas do conjunto de dadosD1. . . 75
7.5 (a) Os pontos vermelhos e pretos correspondem às posições iniciais e fi-nais dos neurônios, respectivamente. A evolução de cada parâmetro ao longo do tempo é mostrada em: (b) O número máximo possível de vizi-nhos (c) a distância máxima para o qual os neurônios são consideradas como pertencentes ao mesmo grupo (d), o raio da hiperesfera de um
de-terminado neurôniowj. . . 75
7.6 (a) Imagem da Matriz contendo o número efetivo de vizinhos para cada neurônio ao longo do tempo (b) Imagem ’a’ vista em forma de superfície. Os neurônios em regiões com menos padrões são vistos como estrias ou vales, tal como é esperado. O número efetivo dos vizinhos é uma fração
dekmax que é proporcional à densidade da região na qual o neurônio está
localizado. . . 76
7.7 Neurônios da rede SOM 10×10 treinada com as bases de dadosTetra(a),
Hepta(b), eD1(c). Os neurônios que resultaram da aplicação do método
proposto em “a”, “b” e “c” estão representados em (d), (e) e (f), respec-tivamente. Neurônios com a mesma cor nos gráficos na primeira linha convergiram para o mesmo ponto em seus respectivos gráficos associados na segunda linha. . . 76
7.8 Matriz U (a) e matriz U* (b) dos mapas 10×10 treinados. Matriz-U (c),
da rede SOM cujos neurônios foram submetidos à aplicação do método proposto. Os índices de 1 a 4 correspondem aos seguintes conjuntos de
8.1 Ilustração da formação do vetor de características fi para o neurônio i
no grid da rede SOM. O componente f11i é obtido a partir da matriz H
que transporta informação de densidade, enquanto f21i é obtido a partir da
matriz U que transporta informação sobre distâncias. . . 82
8.2 Ilustração de neurônios adjacentesie jna rede SOM. . . 83
8.3 Ilustração de (a) vizinhança-4 e (b) vizinhança-8 para o neurônio na grade SOM que está representado como um ponto vermelho. . . 84 8.4 Dinâmica do sistema: (a) movimento dos neurônios ao longo do tempo.
A cada iteração os pontos em cinza representam posições vazias na grade, enquanto que os pontos pretos representam as posições ocupadas. Os ta-manhos dos pontos negros são proporcionais ao número de neurônios no momento naquele ponto. (b) A movimentação dos neurônios ao longo do tempo vista em 2-D. Pontos vermelhos representam os marcadores (neurônios) que são a saída da última iteração, e as setas azuis o movi-mento dos neurônios considerando todas as iterações. . . 87 8.5 Número de neurônios em cada posição da grade da rede SOM no espaço
de saída: (a) após o treinamento com a base de dadosHeptae (b) após a
aplicação do algoritmo proposto. . . 88 8.6 Saída do algoritmo de poda de ligações. (a) Apenas as conexões abaixo
do limiar (força mínima entre dois marcadores) são cortadas. (b) Grupos de neurônios cujas ligações são acima da mínima mas não tem pelo menos
uma percentagemαmarcadores são removidos. As conexões que foram
retiradas estão destacadas nos retângulos verdes. . . 88
8.7 (a) SOM treinado com o conjunto de dadosHepta. (b) SOM rotulado após
a segmentação (saída do Algoritmo 8.3). (c) O SOM rotulado é utilizado para classificar os padrões dos conjuntos de dados. . . 88 8.8 Resultados obtidos para alguns conjuntos de dados: (a) SOM treinado,
(b) SOM rotulado após a segmentação e (c) padrões rotulados. . . 89 8.9 Resultados obtidos para alguns conjuntos de dados: (a) SOM treinado,
(b) SOM rotulado após a segmentação e (c) padrões rotulados. A PCA
foi aplicada para a visualização das basesIriseWine. . . 90
8.10 Os resultados obtidos para o agrupamento hierárquico do conjunto de
da-dosD4: (a) SOM treinado, (b) SOM rotulado após a segmentação e (c)
padrões rotulados. . . 91
A.1 Ilustração dos padrões pertencentes aos seguintes conjuntos de dados: (a)
Atom, (b) Chainlink, (c)DS0, (d) DS3, (e)DS4, (f) D1, (g) D2, (h)D3,
(i) D4, (j)Target, (k) Tetra, (l) Twodiamonds, (m) Engytime, (n) Hepta,
(o) Wingnut, (p)Iris, (q)Wine, e (r) Lsun. Os itens (g), (p) e (q) foram
4.1 Parâmetros utilizados para treinar as redes SOM. . . 29
4.2 Parâmetros utilizados nos experimentos. . . 31
4.3 Sumário dos resultados . . . 35
5.1 Propriedades das matrizes relativas às técnicas de visualização. . . 39
5.2 Avaliação qualitativa dos resultados obtidos com as visualizações propostas. 53 6.1 Parâmetros utilizados nos experimentos. . . 60
6.2 Frequência de Classificação. . . 62
7.1 Resumo do desempenho. . . 78
8.1 Parâmetros comuns usados para treinar as redes SOM. . . 86
8.2 Parâmetros utilizados nos experimentos. . . 87
8.3 Parâmetros utilizados para o agrupamento das da base de dadosD4. . . . 92
8.4 Resumo dos resultados. . . 92
8.5 Parâmetros comuns usados para treinar as redes SOM. . . 93
8.6 Acurácia de classificação para mapas retangulares comσf =0. . . 94
8.7 Acurácia de classificação para mapas retangulares comσf =1. . . 95
8.8 Acurácia de classificação para mapas quadrados comσf =0. . . 96
8.9 Acurácia de classificação para mapas quadrados comσf =1. . . 97
8.10 Sumário dos resultados. . . 99
9.1 Parâmetros definidos a priori. . . 101
A.1 Características das bases de dados. . . 114
Lista de Símbolos e Abreviaturas
kNN: k-Nearest Neighbors
BMU: Best Matching Unit
CCL: Connected Component Labeling
DBI: Davies-Bouldin Index
ESOM: Emergent Self-Organizing Maps
GMM: Gaussian Mixture Models
LVQ: Learning Vector Quantization
MBSAS: Modified Basic Sequential Algorithmic Scheme
MCE: Minimum Classification Error
PNR: Pattern-to-Neuron Ratio
SDH: Smoothed Data Histogram
SOM: Self-Organizing Maps
ViSOM: Visualization-Induced Self-Organizing Maps
WTA: Winner Takes It All
FCPS: Fundamental Clustering Problems Suite
PCA: Principal Component Analysis
Introdução
Atualmente, os avanços tecnológicos têm levado à coleta e ao armazenamento de uma infinidade de dados das mais variadas origens, tais como processos industriais, diagnósti-cos médidiagnósti-cos, comportamento de consumidores, dados financeiros, perfis de redes sociais, entre tantos outros [Larose 2005] e, com ainda mais intensidade, se for considerado o
fenômeno conhecido comoBig Data. Essa crescente quantidade de dados produzidos no
mundo moderno vem maximizando a necessidade de compreender a informação existente nos mesmos, e, simultaneamente, superar os problemas relacionados com a sua qualidade. Em outras palavras, tem-se posto em evidência a necessidade da exploração de dados, como uma forma de extrair informações valiosas. A mineração de dados é justamente um dos campos da ciência que visa transformar tais informações contidas nos dados em co-nhecimento útil [Tan et al. 2006], sendo, de modo geral, utilizada para realizar tarefas de predição (classificação, regressão, etc.) ou descrição (agrupamento, regras de associação, etc.).
A visualização é um recurso fundamental para a compreensão da estrutura dos da-dos utilizada-dos na descoberta de conhecimento e mineração de dada-dos, tanto nas aplicações científicas quanto comerciais. Ela consiste na conversão dos atributos dos dados numa estrutura visual a fim de observar suas características e propriedades. Existem diversas técnicas de visualização de dados, entre elas tem-se histogramas, gráficos de caixa, grá-ficos de dispersão, curvas de nível, coordenadas paralelas, entre outros. Frequentemente, a redução da dimensionalidade tem sido uma operação essencial para viabilizar a visua-lização de dados multidimensionais, baseado-se na aplicação de transformações sobre os dados e, posteriormente, projetando-os em espaços de dimensão mais baixa, sem que isso provoque distorções inadmissíveis na sua estrutura original.
A complexidade do espaço de atributos em algumas bases de dados e os problemas computacionais daí derivados quase sempre não permitem aproximações dedutivas e ba-seadas em modelos estatísticos tradicionais [Yin 2008]. Fatores como tamanho das bases de dados, a dimensionalidade, problemas de escalonamento dos atributos, bem como a
heterogeneidade, a qualidade (presença de ruído eoutliers, ausência de determinados
2 CAPÍTULO 1. INTRODUÇÃO
II 2005].
Nesse contexto, as técnicas na mineração de dados que visam resolver as tarefas de agrupamento podem ser genericamente definidas como a procura de grupos inerentes aos conjuntos de dados. Tradicionalmente, um método de agrupamento se destina a separar os dados de tal forma que as semelhanças intra-agrupamento e inter-agrupamentos sejam máximas e mínimas, respectivamente [Wright 1977]. Proporcionando, portanto, uma dis-posição apropriada dos dados, levando em consideração simultaneamente a sua estrutura e as distribuições dos padrões.
Uma grande variedade de métodos para realizar esta tarefa foram propostos e deriva-dos na literatura [Jain et al. 1999]. Um deriva-dos modelos matemáticos largamente difundideriva-dos são as redes neurais artificiais competitivas, e englobam um grupo de algoritmos utili-zados para a codificação e visualização de dados, bem como para realizar as tarefas de agrupamento. Entre os modelos de redes neurais, os mapas auto-organizáveis (SOM) [Kohonen 1982] são comumente utilizados no campo de mineração de dados, principal-mente devido ao mapeamento de um espaço de entrada de dimensão superior (espaço de dados) para um espaço de saída de menor dimensionalidade (grade fixa de neurônios), ao mesmo tempo preservando a topologia de dados e comprimindo informação [Costa & Yin 2010], devido ao fato ser um algoritmo de quantização vetorial. Neste sentido, a rede SOM pode ser vista como uma generalização não-linear da análise de componentes principais [Ritter 1995].
Por isso, além do fato de que o pós-processamento pode ser efetuado com os neurônios da rede ao invés de todo o conjunto de dados, a pronta possibilidade do uso de algoritmos de agrupamento clássicos aplicados aos neurônios da rede [Vesanto & Alhoniemi 2000] juntamente com várias técnicas de visualização que associam características dos dados à grade de neurônios topologicamente ordenada, permitindo que uma idéia inicial da dis-tribuição de dados seja obtida [Vesanto 1999], transforma os mapas auto-organizáveis em uma poderosa ferramenta para análise exploratória de dados. Não por acaso, desde que foi proposta, a rede SOM tem sido utilizada em uma vasta gama de aplicações [Kohonen 1990], dentre elas o reconhecimento de padrões, processamento de imagens (sensoriamento remoto, compressão de imagens, etc.), sistemas de controle e monitora-mento de processos (incluindo detecção e tolerância à falhas).
1.1
Justificativa e Motivação
A maioria das técnicas de agrupamento e visualização de dados aplicadas aos mapas auto-organizáveis levam em consideração somente métricas de distância ou então somente métricas de densidade espacial de padrões, de forma que a motivação deste trabalho con-siste em unir ambas informações para um melhor particionamento das bases de dados (Figura 1.1).
Densidade
Distâncias
Melhor
compreensão
dos
agrupamentos:
Forma/Estrutura
Figura 1.1: A motivação do trabalho consiste em unir informações relativas a densidade e distâncias entre neurônios do SOM, de modo a propiciar uma melhor observação da estrutura dos dados.
1.2
Problema
Com base no exposto, o problema foco da pesquisa consiste em estudar de que forma integrar propriedades de distâncias e densidades de padrões associadas aos neurônios da rede SOM em um algoritmo de agrupamento.
1.3
Metodologia
A fim de atuar sobre o problema descrito na seção anterior, este trabalho estabelece diferentes tipos de forças de conexão entre neurônios e as suas classificações relativas pela projeção em um espaço de características, as quais são extraídas em termos de padrões associados a eles e distâncias locais.
Para avaliar as forças de conexão, uma máquina de comitê funcionando como um sistema que objetiva particionar os dados analisa as forças de conexão entre neurônios através de medidas de similaridades com base em operações sobre subconjuntos de pa-drões associados aos neurônios. Estes subconjuntos são considerados como uma pequena partição dos dados para cada neurônio. Essas mesmas forças de ligação são utilizadas para gerar um conjunto de visualizações.
A seguir, para avaliar o espaço de características, o trabalho concentra-se em reali-zar a tarefa de filtragem de ruído, agrupamento e visualização através de particionamento do mapa auto-organizável de acordo com a distribuição de vetores de características as-sociados a cada neurônio da rede. Estes vetores englobam propriedades relacionadas à densidade de padrões e as distâncias entre os neurônios.
4 CAPÍTULO 1. INTRODUÇÃO
da rede SOM. O primeiro deles atua no espaço de entrada (espaço dos dados) e visa in-tensificar o contraste entre os agrupamentos proporcionado pela visualização da matriz U através da aplicação de um algoritmo baseado nos princípios gravitacionais sobre os neurônios do SOM, como forma de aumentar as distâncias inter-agrupamentos e diminuir as distâncias intra-agrupamentos, proporcionando uma visualização em que a separação entre os grupos é mais nítida. O segundo algoritmo de agrupamento concentra-se em par-ticionar o SOM através da aplicação de um algoritmo baseado em desconto-recompensa, isto é, na busca pelo menor caminho (baseado nas forças de conexão) com maior recom-pensa (baseado no espaço de características) como uma forma de identificar os neurônios do núcleo dos agrupamentos, de forma que os neurônios movam-se geodesicamente sobre a grade no espaço de saída.
1.4
Objetivos do trabalho
1.4.1
Objetivo Principal
O objetivo do trabalho consiste em analisar forças de conexão entre neurônios da rede SOM e sua relativa importância de modo a implementar algoritmos de agrupamento e prover uma visualização em que os agrupamentos estejam mais claros.
1.4.2
Objetivos Específicos
a) Implementar um algoritmo em que os neurônios movam-se no espaço de entrada (ba-seado no algoritmo gravitacional).
• Determinar a regra de atualização dos pesos.
• Definir um critério de parada para o algoritmo.
b) Implementar um algoritmo em que os neurônios movam-se no espaço de saída (base-ado em algoritmos do tipo desconto-recompensa).
• Determinar as funções de custo e recompensa.
• Definir um critério de parada para o algoritmo.
c) Avaliar os métodos através de simulações com bases de dados sintéticas e do mundo real.
d) Verificar a sensibilidade das heurísticas propostas aos seus parâmetros.
1.5
Organização do texto
Capítulo 2
Mapas Auto-Organizáveis
Este capítulo apresenta considerações de ordem geral sobre a rede neural artificial SOM.
2.1
Introdução
Os mapas auto-organizáveis (SOM, do inglêsself-organizing maps) são formados por
um conjunto de neurônios1 topologicamente ordenados dispostos numa grade. A grade
é geralmente referida como o espaço de saída
B
, enquanto que o espaço do conjunto dedados é conhecida como o espaço de entrada
A
(espaçoRndos padrõesx). Cada neurôniotem um vetor de pesos associado (w) no espaço de entrada, de modo que um mapeamento
de um espaço dimensional superior contínuo para um espaço dimensional inferior discreto é alcançado (Figura 2.1).
w,x
Espaço de Entrada Espaço de Saída
ri
wi
Figura 2.1: A rede SOM faz um mapeamento do espaço n-dimensional dos dados (espaço
de entrada
A
) para a grade fixa p-dimensional de neurônios (espaço de saídaB
). Dessaforma, o i-ésimo neurônio do mapa representado pelo vetor de pesoswi é projetado na
grade pelo seu vetor posiçãori.
A grade de neurônios pode ter topologia retangular ou hexagonal (Figura 2.2), dife-rindo quanto ao número de vizinhos imediatos (4 ou 6 respectivamente, vide os neurônios em verde que correspondem à vizinhança 1 da Figura 2.2). A princípio não há nenhuma vantagem em utilizar a vizinhança hexagonal [Ultsch & Herrmann 2005] em detrimento da retangular. Em geral utiliza-se redes com grades de saída unidimensional ou bidimen-sional, redes de dimensionalidades maiores são possíveis, mas em geral não são utiliza-das, haja visto a dificuldade da sua visualização. A rede SOM pode ser encarada como uma técnica de redução da dimensionalidade ao mapear os neurônios de espaço contínuo de dimensão superior (espaço de entrada) para um espaço discreto de dimensão inferior (espaço de saída).
A aprendizagem de redes SOM é não supervisionada, e o processo de treinamento é uma quantização vetorial de modo que, para cada padrão de entrada, há um neurônio
vencedor, o qual é aquele que tem a menor distância Euclidiana2 entre seu vetor de pesos
associado e o padrão de entrada. O vencedor é também denotado como BMU - best
matching unit, sendo designado com o índicec, como a seguir:
||xi−wc||=min
l ||xi−wl||, l= (1,2, ...,m) (2.1)
onde || · || é a distância Euclidiana, xi é o i-ésimo padrão do conjunto de dados
(n-dimensional) e w é um vetor de pesos (também n-dimensional). Assim, o espaço de
entrada é dividido em mregiões de Voronoi, correspondentes aos mneurônios da rede.
Os padrões dentro de uma região de Voronoi
H
j (j=1,2, ...,m)constituem umsubcon-junto do consubcon-junto de dados. A distância Euclidiana de qualquer padrão de
H
j para seuneurônio associadowjé mínima em relação a todos os outros neurônios.
(a)
2 3
1 0
(b)
0 1 2 3
Figura 2.2: Para o neurônio ressaltado em vermelho, são ilustradas as vizinhanças 0 (o próprio neurônio), 1 (neurônios em verde), 2 (neurônios em azul) e 3 (neurônios em amarelo) considerando as topologias (a) retangular e (b)hexagonal.
2.2. ALGORITMOS DE TREINAMENTO 9
Como já foi mencionado, cada neurônio tem um vetor de pesos associado. Treinar uma rede através de determinado tipo de aprendizado consiste em fazer o ajuste desses pesos. Para cada padrão apresentado à rede, os neurônios competem entre si de modo que o vencedor é o mais próximo em função de uma dada métrica de similaridade, no caso, a distância Euclidiana. As redes SOM apresentam os seguintes princípios [Haykin 2001]:
1. Competição
Ao apresentar um padrãoxià rede, o neurônio vencedor é tomado como aquele que
produz a menor distância Euclidiana.
2. Cooperação
Não somente o neurônio vencedor, mas toda a vizinhançahde neurônios é excitada,
fato que diferencia a rede SOM de uma rede meramente competitiva WTA (winner
takes it all).
3. Adaptação
Todos os neurônios da regiãohcentrada no neurônio vencedor participam do
pro-cesso de aprendizagem.
Os neurônios vencedores se deslocam em direção aos padrões levando consigo neurô-nio de sua vizinhança. Devido ao fator de magnificação da rede, existe um maior povo-amento de neurônios em áreas onde há uma maior quantidade de padrões, isto é, regiões mais densas.
2.2
Algoritmos de Treinamento
A fim de treinar a rede SOM, existem dois algoritmos: sequencialelote. No
treina-mento sequencial, a cada padrão apresentado ocorre adaptação dos protótipos, enquanto que no treinamento em lote após o final de cada época tem-se a atualização dos pesos pela resultante dos vetores associados ao movimento relacionado a cada padrão da base.
Em relação às características do aprendizado na rede SOM, suponha um vetor de entrada
xi= [x1,x2,···,xn] (2.2)
e o vetor de pesos para um neurônio j
wj= [w1,w2,···,wn]. (2.3)
No algoritmo de treinamento sequencial, cada padrão é apresentado à rede, um por
um. Para cada padrãoxio neurônio vencedorwc é determinado através da Equação 2.1.
Em seguida, o BMU e os seus vizinhos são movidos no sentido do padrão de entrada de acordo com a seguinte regra de atualização:
wj(t+1) =wj(t) +η(t)hc,j(t)
xi−wj(t)
(2.4)
ondet indica a iteração,wj(t)é o vetor de pesos associado ao neurônio j, xi é o padrão
η(t)representa a taxa de aprendizagem que regula a intensidade da atração para tal
pa-drão e hc,j(t) é a função de vizinhança (que está centrada no BMU de xi). A taxa de
aprendizagem deve ser uma função que diminui monotonicamente com o tempo.
No algoritmo de treinamento em lote, o conjunto de dados é apresentado ao mesmo tempo. Em cada época, os BMUs para todos os padrões são determinados. Então, todos
vetores de pesoswj(t)são atualizados simultaneamente, como uma média ponderada de
todos os padrões do conjunto de dados:
wj(t+1) = PN
i=1hj,c(t)xi PN
i=1hj,c(t)
(2.5)
ondeNé a cardinalidade do conjunto de dados, ehj,c(t)é o valor da função de vizinhança
do neurônio jna posição do BMU do padrãoxi.
Em ambos os algoritmos de treinamento, a função de vizinhança, genericamente
de-finida comohna equação 2.9, é uma função monotonicamente decrescente do número de
iterações e depende da distância matricial entre os neurônios jecna grade SOM:
h= f(||rc−rj||,t) (2.6)
onde rj e rc indicam a posição do neurônio j e do neurônio vencedor (para um dado
padrão xi), na grade mapa, respectivamente. Normalmente, a função de vizinhança é
definida como uma função gaussina:
h=exp
−||rc−rj||
2
2σ2(t)
(2.7)
onde σ é o raio da vizinhança (tamanho da vizinhança) que diminui monotonicamente
com respeito ao tempo.
Durante o período de treinamento, a rede SOM se comporta como uma rede elástica que se molda à forma intrínseca dos dados. O posicionamento dos neurônios no espaço de entrada reflete a densidade da distribuição do conjunto de dados, de modo que o número de neurônios de uma certa região do espaço de entrada está relacionada com o número de padrões nessa mesma a região, o que é conhecido como o fator de ampliação. Isto é, devido ao fator de ampliação a localização dos neurônios reproduz a distribuição do conjunto de dados: as regiões mais densas atraem mais neurônios.
2.3
Configuração dos parâmetros da rede
Conforme mencionado na seção anterior, a vizinhança e a taxa de aprendizagem de-vem ser monotonicamente decrescentes em função do tempo, isto é, dede-vem diminuir à medida que o algoritmo de treinamento progride. Sendo assim, para a definição e o trei-namento de uma rede SOM, é necessário definir os seguintes parâmetros:
a) A função de vizinhança (bolha, degrau, gaussiana, gaussiana ceifada, etc.);
2.4. QUALIDADE DO SOM 11
c) O tipo de treinamento (sequencial ou em lote);
d) A função da taxa de aprendizagem caso o treinamento seja sequencial (decaimento linear, exponencial, etc.);
e) O número de épocas para o treinamento.
f) Inicialização das posições dos neurônios no espaço de entrada.
Existem diversas abordagens para definir o tamanho do mapa, uma boa prática [Kohonen
2001] consiste em fazer as suas dimensõesaebproporcionais aos dois maiores
autova-lores da matriz de covariância dos dados,λ1eλ2respectivamente:
a
b ≈
s
λ1
λ2 (2.8)
Como regra geral, o número total de neurôniosm=a×bem uma rede SOM é
tipica-mente definido para cerca de:
m≈5√N (2.9)
ondeN é o número de padrões da base de dados.
A inicialização das posições dos neurônios no espaço de dados pode ser aleatório ou linear. Neste último caso, os neurônios são distribuídos através do hiperplano gerado
pelos autovetores associados a λ1 e λ2, isto é, em função da direção dos autovetores
correspondentes aos maiores autovalores da matriz de covariância dos dados.
2.4
Qualidade do SOM
A questão do tamanho do mapa e por conseguinte a razão de padrões por neurônios in-fluencia diretamente a qualidade de algumas técnicas de visualização, o que será abordado
nas seções seguintes. O termo razão de padrões por neurônios (PNR) pode ser definido
por:
PNR= N
m (2.10)
que está intimamente relacionado com a figura de mérito utilização de neurônios [Cheung & Law 2007]:
Nu=
1
m
m X
k=1
uk (2.11)
ondeuk é igual a um, se neurônioktem um padrão associado, e zero caso contrário. Em
geral, valores deNupróximos de 1 são desejados.
Nesse contexto relacionado ao número de protótipos, caso a rede seja pequena, por exemplo um número próximo ao valor real do número de agrupamentos, ela se assemelha a técnicas tradicionais como o k-means [Ultsch 2006]. Existem vertentes que promovem
o uso do ESOM (emergent SOM) [Ultsch & Mörchen 2005] e ViSOM (
afloram quando há um número de neurônios muito grande, maior inclusive do que a quan-tidade dos dados.
A rede SOM porém é um algoritmo de quantização vetorial, portanto vários padrões podem ser representados por um único neurônio. Existem dois erros que medem quanti-tativamente o mapeamento dos dados realizado pela rede, que são o erro de quantização
(qe) [Kohonen 2001] e o erro de topologia (te) [Kiviluoto 1996]:
qe=
1
N
N X
k=1
||xk−wxck|| (2.12)
te=
1
N
N X
k=1
f(xk) (2.13)
ondexk é o k-ésimo padrão retirado do conjunto de dados ewxck é o BMU do padrão xk.
A função f(xk)assume dois valores: é igual a zero, se e apenas se o primeiro e segundo
BMU do padrão xk são adjacentes no espaço de saída, caso contrário é igual à unidade.
O erro de quantização revela a resolução da rede. O erro de topologia ocorre quando há divergência entre a vizinhança de neurônios no espaço de entrada e no espaço de saída.
São desejados valores de te e qe pequenos. Uma extensa discussão sobre topologia em
Capítulo 3
Técnicas de Visualização usando SOM
Este capítulo apresenta considerações de ordem geral sobre algumas técnicas de pós-processamento relacionadas à visualização de características dos neurônios da rede SOM no espaço de saída.
3.1
Introdução
Os mapas auto-organizáveis são redes neurais artificiais amplamente utilizadas no campo da mineração de dados, principalmente por se constituírem numa técnica de re-dução de dimensionalidade não linear, que tenta, via treinamento não supervisionado, manter ao máximo as relações topológicas dos dados. Para visualizar adequadamente os agrupamentos, técnicas de visualização devem ser aplicadas em uma etapa de pós-processamento que consiste em inferir, utilizando os neurônios, características relevantes do conjunto de dados.
Em outras palavras, a rede SOM não é propriamente um algoritmo de agrupamento
[Ultsch 2005a], uma vez que é necessário um pós processamento voltado à visualização
utilizando características dos protótipos para inferir características dos dados. Existem na literatura diversos métodos para tal fim. Normalmente essas técnicas levam em conta não mais de uma métrica na sua definição, como por exemplo as distâncias (Euclidiana) entre protótipos, como na matriz U, nos planos de componentes, e na matriz GC, ou ainda apenas a densidade de padrões, tal qual a matriz P e a CONNvis. Existem alguns modelos híbridos que levam em conta tanto densidade de padrões como distância entre neurônios: CONNDISTvis e Matriz U* são as mais conhecidas. Essas técnicas estão descritas nas seções a seguir. Existem casos em que a métrica de distância é mais relevante do que a métrica de densidade de padrões e vice versa. Serão utilizadas, a título de exemplificação, redes SOM geradas com o SOMToolbox [Vesanto et al. 1999] e visualizações obtidas
com o pacote SOMVIS1, ambos no ambiente do MATLABR. Os mapas serão treinados
com o conjunto de dadosHepta(Figura 3.1), para em seguida serem aplicadas as técnicas
de visualização. A descrição das bases de dados se encontra no apêndice A.
1O pacote SOMVIS está disponível sob a Licença Pública Geral do GNU em:
(a)
0 0.5
1 0 0.5
1 0
0.2 0.4 0.6 0.8 1
Mapa 5x5
(b)
0 0.5
1 0 0.5
1 0
0.2 0.4 0.6 0.8 1
Mapa 20x20
(c)
0 0.5
1 0 0.5
1 0
0.2 0.4 0.6 0.8 1
Mapa 80x80
Figura 3.1: Redes SOM treinadas com a base de dadosHeptae utilizadas para
exemplifi-car as técnicas de visualização. Os neurônios estão representados pelos pontos em preto
e os padrões em vermelho. Os mapas têm os tamanhos 5×5, 20×20 e 80×80.
3.2
Visualizações baseadas em distância de protótipos
3.2.1
Matriz U
A matriz U [Ultsch & Siemon 1990] é uma das técnicas mais populares de visu-alização, e consiste em uma matriz em cujas posições são colocadas as distâncias no espaço de entrada entre os protótipos vizinhos no espaço de saída. Considere que um
mapa com grade retangular tenha um tamanho a×b, então a matriz U terá o tamanho
(2a−1)×(2b−1). As posições relativas aos neurônios na matriz U são obtidas por uma
função f dos valores das distâncias dos neurônios vizinhos, onde f pode ser a média, a
mediana, entre outros. A Figura 3.2 ilustra as posições(i,j)dos protótipos(w)e a suas
respectivas posições na matrizU(u).
Sendo assim o procedimento para obter os valores da matriz U nas posiçõesP1 aP9é
mostrado nas equações 3.1, 3.2, e 3.3. Para a posiçãoP1da Figura 3.2, tem-se:
U(P1) =||w(i,j)−w(i−1,j)|| (3.1)
onde|| · ||constitui distância euclidiana entre os vetores.
Deve-se proceder de modo análogo para as posições ,P3, P5e P7. Para a posiçãoP2
tem-se:
U(P2) =||w(i,j)−w(i−1,j+1)||+||w(i−1,j)−w(i,j+1)||
2√2 (3.2)
Deve-se proceder de modo análogo para as posiçõesP4, P6 e P8. Para a posiçãoP9
tem-se:
U(P9) = f(P1,P3,P5,P7) (3.3)
onde a função f, como já mencionado anteriormente pode ser, por exemplo a mediana.
3.2. VISUALIZAÇÕES BASEADAS EM DISTÂNCIA DE PROTÓTIPOS 15
(a)
w(i,j−1) w(i,j) w(i,j+1)
w(i−1,j)
w(i+1,j)
(b)
P9
P8 P1 P2
P7 P3
P4 P5 P6
Figura 3.2: Grade da rede SOM e posições relativas dos neurônios na matriz U.
(a)
Mapa 5x5
2 4 6 8 1
2 3 4 5 6 7 8
9 0.06
0.08 0.1 0.12 0.14 0.16 0.18 0.2 0.22 0.24 0.26
(b)
Mapa 20x20
5 10 15 20 25 30 35 5
10 15 20 25 30
35 0.05
0.1 0.15 0.2 0.25
(c)
Mapa 80x80
20 40 60 80 100 120 140 20
40 60 80 100 120
140 0.05
0.1 0.15 0.2 0.25 0.3 0.35 0.4 0.45
Figura 3.3: Matrizes U relativas aos mapas da figura 3.1.
enquanto em mapas grandes ela torna a definição de agrupamentos mais nítida, quando a métrica de distância for relevante.
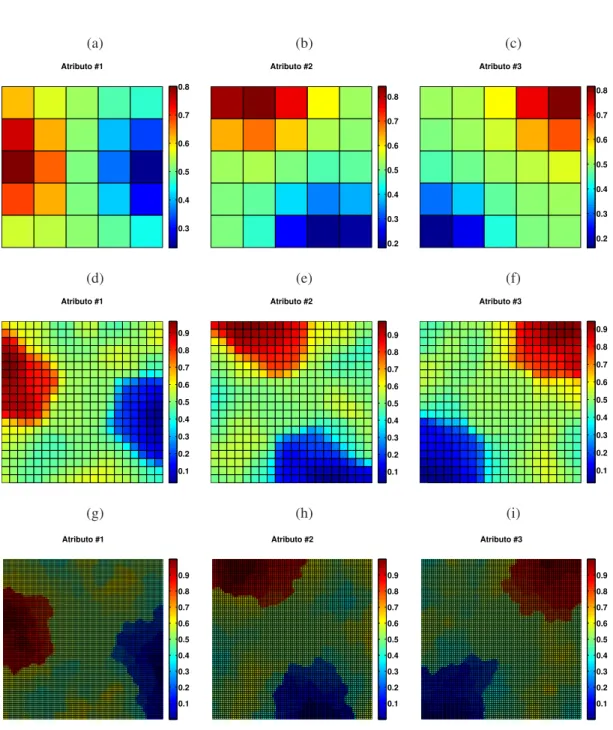
As distâncias Euclidianas na matriz U podem ser calculadas em função de todos os atributos ou em função de atributos específicos com a utilização de máscaras. O caso específico em que é calculada uma matriz U para cada atributo dos dados consiste nos planos de componentes, que será detalhado na próxima seção.
3.2.2
Planos de componentes
Os planos de componentes [Vesanto 1999] são uma variação da matriz U, onde são aplicadas máscaras no cálculo das distâncias. Dessa forma, se uma base de dados contiver
padrões noRn, existirá um plano de componente para cada atributo da base, isto é , haverá
nplanos de componentes e, para cada plano, a distância euclidiana será calculada somente
(a)
Atributo #1
0.3 0.4 0.5 0.6 0.7 0.8
(b)
Atributo #2
0.2 0.3 0.4 0.5 0.6 0.7 0.8
(c)
Atributo #3
0.2 0.3 0.4 0.5 0.6 0.7 0.8
(d)
Atributo #1
0.1 0.2 0.3 0.4 0.5 0.6 0.7 0.8 0.9
(e)
Atributo #2
0.1 0.2 0.3 0.4 0.5 0.6 0.7 0.8 0.9
(f)
Atributo #3
0.1 0.2 0.3 0.4 0.5 0.6 0.7 0.8 0.9
(g)
Atributo #1
0.1 0.2 0.3 0.4 0.5 0.6 0.7 0.8 0.9
(h)
Atributo #2
0.1 0.2 0.3 0.4 0.5 0.6 0.7 0.8 0.9
(i)
Atributo #3
0.1 0.2 0.3 0.4 0.5 0.6 0.7 0.8 0.9
Figura 3.4: Planos de componentes relativas aos mapas 5x5, 20x20 e 80x80 da figura 3.1, na primeira, segunda e terceira linhas, respectivamente.
3.2.3
Matriz GC
A matriz de gradientes de componentes [Costa 2011] consiste em uma técnica de
pro-cessamento digital de imagens. Considere que os dados estão noRn e que o mapa
con-sista em uma grade bidimensional de tamanhoa×b. Sendo assim, podem ser formadas
n matrizesa×b em cujas posições(i,j) estão as n-ésimas componentes dos protótipos
localizados nas posições(i,j)da grade de saída. A seguir aplica-se gradientes nas
3.2. VISUALIZAÇÕES BASEADAS EM DISTÂNCIA DE PROTÓTIPOS 17
Equação 3.4 :
GC(i,j) =
v u u t
n X
k=1 "
∂
Fk(i,j)
∂x
2
+
∂
Fk(i,j)
∂y
2#
(3.4)
A matriz e consequentemente a superfície obtida é mais suave do que a matriz U, mas apresenta o mesma dependência do tamanho do mapa (vide Figura 3.5).
(a)
Mapa 5x5
2 4 6 8
1 2 3 4 5 6 7 8
9 0.18
0.19 0.2 0.21 0.22 0.23 0.24
(b)
Mapa 20x20
5 10 15 20 25 30 35
5 10 15 20 25 30 35
0.05 0.1 0.15 0.2 0.25
(c)
Mapa 80x80
20 40 60 80 100 120 140
20 40 60 80 100 120
140 0.05
0.1 0.15 0.2 0.25 0.3
Figura 3.5: Matrizes GC relativas aos mapas da figura 3.1.
3.2.4
DISTvis
O DISTvis [Tasdemir 2010] é uma técnica de visualização baseada em grafos e con-siste no cálculo das distâncias entre neurônios vizinhos na grade levando em consideração os seus respectivos campos receptivos (vide a subseção 3.3.4). Seja a distância Euclidiana
entre os dois neurônioswiewj:
DIST(i,j) =||wi−wj|| (3.5)
Os valores das arestas do grafo DISTvis consistem na normalização de DIST da se-guinte forma:
DIST vis(i,j) = DIST(i,j)
maxi,j(DIST(i,j) | (CONN(i,j)6=0)∧(|i−j|=1))
(3.6)
caso DISTvis(i,j) seja maior do que 1, o seu valor é fixado na unidade.
O DISTvis pode ser visualizado considerando 4 ou 8 vizinhos na grade (considerando topologia retangular). A diferença entre considerar vizinhança-4 ou vizinhança-8, re-side no fato de que com 8 vizinhos existem mais conexões intra-agrupamento enquanto a quantidade de conexões inter-agrupamento permanece praticamente inalterada em re-lação à utilização de 4 vizinhos. Além disso, as distâncias entre neurônios que possuem
CONN(i,j)nula podem ser removidas com o intuito de facilitar a visualização das
no espaço de entrada (quando factível), através de linhas em tons de cinza, onde as mais escuras representam neurônios mais próximos.
3.3
Visualizações baseadas em densidade de padrões
3.3.1
Histograma de hits
O histograma dehitsconsiste em uma matriz de acumulação com o mesmo tamanho
do mapa, em cujas posições(i,j)são mostrados o número de padrões para os quais aquele
neurônio é o vencedor, como mostrado na Figura 3.6. Em outras palavras, trata-se de um
histograma 2D onde cada bin é a posição do neurônio e soma-se 1 unidade para cada
padrão para o qual aquele neurônio é o BMU.
(a)
Mapa 5x5
1 2 3 4 5
0.5 1 1.5 2 2.5 3 3.5 4 4.5 5
5.5 0
5 10 15 20 25 30
(b)
Mapa 20x20
5 10 15 20
2 4 6 8 10 12 14 16 18
20 0
0.5 1 1.5 2 2.5 3
(c)
Mapa 80x80
20 40 60 80
10 20 30 40 50 60 70
80 0
0.1 0.2 0.3 0.4 0.5 0.6 0.7 0.8 0.9 1
Figura 3.6: Histogramas dehitsrelativas aos mapas da figura 3.1.
Dessa forma, tem-se uma maneira de observar a densidade de padrões no mapa em
função dohits associados a cada neurônio. O histograma dehits exibe comportamento
inverso da matriz U em se tratando de dependência do tamanho do mapa: o fenômeno
da dissolução dos hits. Quanto maior o mapa, menor o PNR (número de padrões por
neurônios), de forma que a matriz (histograma 2D) começa a ficar esparsa. Esse problema é superado em parte pelo histograma de dados suavizado (descrito na seção a seguir), que observa não somente o neurônio vencedor, como também o segundo, o terceiro, até o s-ésimo, sendo este um parâmetro previamente definido.
3.3.2
Histograma de dados suavizado
Conforme foi mencionado no final da última subseção, com o aumento do mapa ocorre
o fenômeno da dissolução dos hits. Dessa forma, para a observação de uma matriz de
hits mais suave, procura-se considerar até o s-ésimo neurônio vencedor de uma dado
padrão, ondesé um parâmetro de entrada do algoritmo, haja visto que dado padrão pode
muito bem ser representado por mais de um neurônio desde que o mesmo esteja quase
tão próximo quanto o BMU. Sendo assim, cada neurônio é considerado como um bine
3.3. VISUALIZAÇÕES BASEADAS EM DENSIDADE DE PADRÕES 19
dado por:
s−(k−1)
cs
(3.7)
onde o parâmetrocsé dado por:
cs= s−1 X
i=0
(s−i) (3.8)
e o grau de pertinência aos demaisbinsé nulo [Pampalk et al. 2002].
Como foi mencionado, o SDH (smoothed data histogram) propõe um número fixo s
de neurônios vencedores a serem considerados, o que poderia variar em função de cada
padrão: um padrão xj pode possuir um quinto neurônio vencedor muito próximo por
uma distância ε do primeiro vencedor, enquanto um padrão xi pode possuir um quinto
neurônio vencedor próximo por uma distanciaγdo primeiro, ondeγpode ser muito maior
do que ε. Além disso, o valor s é um parâmetro de entrada que modifica o resultado
do método, sendo necessário vários testes para determinar o ideal. De modo geral ums
muito pequeno, e no limite igual a 1, leva o resultado a ser idêntico a um histograma de
hitstradicional. Ao se elevar o valor desas características dos dados começam a aflorar,
porém, com valores muito elevados, observa-se somente a formação de um único grande agrupamento, o que mostra que o algoritmo tem traços hierárquicos. A Figura 3.7 ilustra a aplicação do método em questão.
(a)
Mapa 5x5
0 10 20 30 40 50 60 70 80 90
(b)
Mapa 20x20
0 2 4 6 8 10 12
(c)
Mapa 80x80
0 0.5 1 1.5 2 2.5 3
Figura 3.7: Histogramas de dados suavizados relativos aos mapas da figura 3.1.
3.3.3
Matriz P
A matriz P [Ultsch 2003a] tem estrutura similar à matriz U. Essa técnica visa estimar
a densidade de probabilidade dos dados através da estimação de densidade de Pareto
[Ultsch 2005b] [Ultsch 2002]. Em cada posição(i,j) da matriz P, relativa ao neurônio
wcuja posição é também(i,j)na grade da rede SOM, é colocado o número de padrões
contidos em uma hiperesfera de raior centrada nesse neurônio. O raio é fixo para todos