Título: Identificação de genes envolvidos na degradação de xilana por meio de abordagens genômicas e metagenômicas.
Agradecimentos Resumo
Abstract Sumário
Pró-Reitoria de Pós-Graduação e Pesquisa
Programa de Pós-Graduação Stricto Sensu em Ciências Genômicas e
Biotecnologia
Identificação de genes envolvidos na degradação de xilana por meio
de abordagens genômica e metagenômica
Brasília - DF
2014
LUÍS FELIPE SCHROEDER
IDENTIFICAÇÃO DE GENES ENVOLVIDOS NA DEGRADAÇÃO DE XILANA POR MEIO DE ABORDAGENS GENÔMICA E METAGENÔMICA
Dissertação apresentada ao programa de Pós- Graduação Stricto Sensu em Ciências
Genômicas e Biotecnologia da Universidade Católica de Brasília, como requisito para a obtenção do Título de Mestre em Ciências Genômicas e Biotecnologia.
Orientadora: Drª. Cristine Chaves Barreto Co-orientadora: Drª. Betania Ferraz Quirino
Dissertação de autoria de Luís Felipe Schroeder, intitulada PROSPECÇÃO DE GENES ENVOLVIDOS NA DEGRADAÇÃO DE XILANA POR MEIO DE ABORDAGENS GENÔMICA E METAGENÔMICA, apresentada como requisito parcial para obtenção do grau de Mestre em Ciências Genômicas e Biotecnologia da Universidade Católica de Brasília, em 30 de Setembro de 2014, defendida e aprovada pela banca examinadora abaixo assinada:
_____________________________________________________________________ Profª. Drª. Cristine Chaves Barreto
Orientadora
Programa de Pós-graduação em Ciências Genômicas e Biotecnologia - UCB
_____________________________________________________________________ Profª. Drª. Betania Ferraz Quirino
Co-orientadora
Programa de Pós-graduação em Ciências Genômicas e Biotecnologia - UCB/ Embrapa Agroenergia
_____________________________________________________________________ Prof. Dr. Octávio Luiz Franco
Examinador Interno
Programa de Pós-graduação em Ciências Genômicas e Biotecnologia - UCB
_____________________________________________________________________ Prof. Dr. Edivaldo Ximenes Ferreira Filho
Examinador Externo
inclusive eu. Por tudo o que fez, faz e ainda fará em minha vida.
Agradeço a minha família, meus pais Jaime Alberto Schroeder e Marli Cristina Hamester Schroeder por todo amor, carinho, educação, força, dedicação por todo o apoio que me deram durante toda esta jornada (graduação e mestrado). Agradeço por todos os momentos felizes que me proporcionaram, me dando uma irmã Priscila Lisane Schroeder e um irmão André Guilherme Schroeder, que me ajudaram a ser quem eu sou hoje, apesar de não termos o controle de todas as coisas.
Agradeço aos meus professores que me ensinaram e me ajudaram na minha carreira profissional. Agradeço a professora doutora Cristine Chaves Barreto, pela oportunidade de ter trabalhado com você, no início como iniciação científica na graduação e agora durante o mestrado. Agradeço por ter me aturado durante todo este tempo apesar das minhas limitações e dificuldades. Agradeço por suas ideias muitas vezes geniais, mas difíceis de serem colocadas em prática, pelos ensinamentos, por todo o apoio, ajuda e incentivo concedido durante estes quase cinco anos. Aprendi muito com você.
Agradeço a professora doutora Betânia Ferraz Quirino, minha co-orientadora, pela oportunidade de ter trabalhado com você, pela confiança depositada em mim e por toda a ajuda que pode proporcionar durante todo o trabalho. Primeiramente como DTI e depois no mestrado. Pela oportunidade de participar do projeto da prospecção de enzimas em rúmen de caprinos, no qual este trabalho esta inserido.
Agradeço a banca examinadora, professor doutor Edivaldo Ximenes Ferreira Filho e professor doutor Octávio Luiz Franco por todas as sugestões e correções, que tornaram este trabalho melhor.
Agradeço ao diretor do Programa de Pós-graduação em Ciências Genômicas e Biotecnologia Rinaldo Pereira, por proporcionar e sempre tentar melhorar a qualidade do programa. E a todos os professores que fazem parte dele, pelas disciplinas ministradas, conversas e momentos de aprendizagem que pude ter com vocês.
Agradeço a Mara por todo o apoio na resolução das questões burocráticas, relativas ao curso, que muitas vezes são difíceis de serem resolvidas sem ajuda.
Agradeço também a Universidade Católica de Brasília e Embrapa Agroenergia pela estrutura fornecida para as pesquisas e disciplinas realizadas durante todo este projeto.
Agradeço a todos os meus colegas e amigos do mestrado que fizeram as disciplinas comigo. Apesar das dificuldades conseguimos completar esta etapa.
Agradeço aos meus colegas e amigos de bancada, vocês foram as pessoas com as quais eu passei mais tempo. Agradeço pela ajuda, pelas brincadeiras, pelos momentos de descontração e pelas conversas e desabafos. Muitos não continuaram ou estão em outros grupos. Rosiane, Érica, Flávio, Cassimira, Maria, Priscila, Otávio, Vinícius, Rayana, Laiane, Ailla, Flávia, Cristiane, Ramon, Juliana, Virgílio. Vocês foram muito importantes.
Agradeço aos amigos e colegas do laboratório. Pois a cooperação é necessária, sem essa interação entre os grupos, o laboratório seria muito chato e complicado de se trabalhar. Além dos momentos de descontração, conversas, almoços. Ohana, Carol, Jéssica, Leane, Betúlia, Stephan, Fábio, Lucas, Polyne, Flávia, Gabriela, Kamila, Nadyelle e muitos outros que de alguma forma participaram comigo deste mestrado.
Agradeço as pessoas que trabalharam e trabalham no laboratório a fim de facilitar e ajudar para que os projetos ocorram da melhor forma. Idaucy, Davi e Adevilton. Agradeço a Alessandra por tentar organizar e manter o laboratório funcionando.
Referência: SCHROEDER, Luís Felipe. Identificação de genes envolvidos na degradação de xilana por meio de abordagens genômica e metagenômica. 2014. 105. Dissertação de Mestrado em Ciências Genômicas e Biotecnologia - Universidade Católica de Brasília, Brasília -DF, 2014.
Existem diversos processos em desenvolvimento para a produção de etanol celulósico, havendo diferentes possíveis pré-tratamentos, com temperaturas e pH variados, além das diversas biomassas que podem ser utilizadas como fonte de açúcares fermentáveis. Dentre as enzimas importantes para a desconstrução de biomassa vegetal, destacam-se as xilanases. Estas enzimas são responsáveis pela desconstrução da estrutura hemicelulósica presente na parede celular das plantas. Há diversas maneiras para alcançar a identificação destas enzimas: purificação a partir de micro-organismo isolado sendo uma delas. No presente trabalho, foram utilizadas abordagens genômica e metagenômica a fim de realizar a prospecção dos genes responsáveis pela codificação para estas enzimas. Foram utilizados clones oriundos de duas bibliotecas para a detecção e avaliação da atividade em meio sólido suplementado com xilana e bagaço de cana-de-açúcar pré-tratado com ácido. Dezenove clones de uma biblioteca metagenômica de rúmen de caprinos e cinco clones de uma biblioteca genômica da bactéria AB60, com 15.000 clones construída no presente trabalho, foram selecionados inicialmente. Quatorze clones da biblioteca metagenômica foram sequenciados completamente e tiveram as suas ORFs analisadas. Quatro clones da biblioteca genômica foram parcialmente sequenciados e um clone teve a sua sequência completa determinada e as ORFs analisadas. Das 104 ORFs obtidas de todos os clones completamente ou parcialmente sequenciados, onze ORFs apresentaram alguma similaridade com genes de importância para a degradação de polissacarídeos complexos. Dentre as ORFs de maior importância e com maior probabilidade de estarem relacionadas com a atividade detectada, estão genes codificantes para β -glicosidase, α-xilosidase e β-glicuronidase. Além disso, também foram identificadas outras ORFs com menor probabilidade de relação com a atividade ou necessidade de sequenciamento completo de alguns dos clones para uma análise mais conclusiva. Cerca de 40% das ORFs presentes nos clones selecionados de rúmen e 37,8% das ORFs presentes nos clones selecionados de Acidobacteria apresentaram similaridade com proteínas hipotéticas ou proteínas não caracterizadas que podem ter importância na atividade detectada. Um gene de β-glicuronidase detectado em um clone da biblioteca metagenômica de rúmen de caprino foi sintetizado, otimizando-se sua sequência para expressão em Escherichia coli . Entretanto, não foi possível a sub-clonagem, expressão e purificação desta enzima no presente trabalho. Algumas ORFs detectadas podem ser utilizadas para estudos futuros de expressão e caracterização a fim de aprimorar o conhecimento a respeito do potencial biotecnológico presente no rúmen e na Acidobactéria AB60, além do papel ecológico destes micro-organismos em seu ambiente.
Referência: SCHROEDER, Luís Felipe. Identificação de genes envolvidos na degradação de xilana por meio de abordagens genômica e metagenômica. 2014. 105. Dissertação de Mestrado em Ciências Genômicas e Biotecnologia - Universidade Católica de Brasília, Brasília -DF, 2014.
There are different process being developed for cellulosic ethanol production, with possible different pretreatments with varying temperatures and pH, in addition to several biomasses can be used as the source of fermentable sugars. Among the important enzymes for deconstruction of plant biomass, stand out xylanases. These enzymes are responsible for deconstruction of the hemicellulose present in the structure of the plant cell walls. There are several ways to accomplish the identification of these enzymes: purification from an isolated microorganism is one. In this study, genomic and metagenomic approaches were used to carry out the prospection of the genes responsible for coding these enzymes. Clones from two libraries were used for detection and evaluation of activity on solid medium, supplemented with xylan and acid pretreated sugarcane bagasse. Nineteen clones of a goat rumen metagenomic library and five clones from an AB60 bacterium genomic library, with 15,000 clones constructed in this study, were selected initially. Fourteen clones from the metagenomic library were completely sequenced and their ORFs were analyzed. Four clones from the genomic library were partially sequenced and one clone had its sequence completely determined and 104 ORFs were obtained for all clones completely or partially sequenced ORFs were analyzed. Eleven ORFs showed some similarity to genes of importance for the degradation of complex polysaccharides. Among the most important ORFs and most likely to be related to the detected activity, are genes coding for β-glucosidase, α-xylosidase and β -glucuronidase. Furthermore, also other ORFs with lower probability of relation with the activity or necessity to full sequencing of the clones for a few more conclusive analysis were identified. About 40% of the ORFs present in the rumen clones and 37.8% of the ORFs present in Acidobacteria clones showed similarity with hypothetical or uncharacterized proteins, which could be important in the activity detected. A β-glucuronidase gene detected in a clone from the goat rumen metagenomic library was synthesized, its sequence was optimized for expression in Escherichia coli. However, in the present work, it was not possible to sub-cloning, made expression and purification of this enzyme. Some ORFs detected can be used for future studies of expression and characterization in order to improve knowledge about biotechnological potential present in the rumen and acidobacteria AB60, besides the ecological role of these microorganisms in their environment.
Figura 22 - Alinhamento da ORF 1 do clone D8-76. ... 53
Figura 23 - Etapas de preparação para a ligação do gene sintetizado no vetor de expressão pET21(a). ... 54
Figura 24 - Digestão com a enzima EcoRI dos clones da biblioteca genômica da AB60 selecionados ... 55
Figura 25 - Confirmação da atividade em xilana dos clones de Acidobacteria após a retransformação em ensaio em placa. ... 56
Figura 26 - Confirmação da atividade em bagaço de cana-de-açúcar dos clones de Acidobacteria após a retransformação em ensaio em placa. ... 56
Figura 27 - Esquema representativo do sequenciamento do clone D10-2. ... 58
Figura 28 - Dendrograma mostrando a relação da ORF 2F do clone D10-2 com ORFs similares encontradas em análises do programa Blastx... 59
Figura 29 - Dendrograma mostrando a relação da ORF 1R do clone D10-2 com ORFs similares encontradas em análises do programa Blastx... 60
Figura 31 - Dendrograma mostrando a relação da ORF 1F do clone E4-7 com ORFs similares encontradas em análises do programa Blastx. ... 61
Figura 32 - Dendrograma montado pelo programa Uniprot. ... 61
Figura 33 - Alinhamento da ORF 1(F) do clone E4-7. ... 62
Figura 34 - Dendrograma mostrando a relação da ORF 1R do clone E4-7 com ORFs similares encontradas em análises do programa Blastx. ... 63
Figura 35 - Dendrograma mostrando a relação da ORF 2R do clone E4-7 com ORFs similares encontradas em análises do programa Blastx. ... 64
Figura 36 - Esquema representativo do sequenciamento do clone G4-5. ... 64
Figura 37 - Dendrograma mostrando a relação da ORF 1R do clone G4-5 com ORFs similares encontradas em análises do programa Blastx. ... 65
Figura 38 - Esquema representativo do sequenciamento do clone A7-34. ... 98
Figura 39 - Esquema representativo do sequenciamento do clone B2-34. ... 98
Figura 40 - Esquema representativo do sequenciamento do clone C2-74. ... 99
Figura 41 - Esquema representativo do sequenciamento do clone C5-95. ... 99
Figura 42 - Esquema representativo do sequenciamento do clone C9-1. ... 99
Figura 43 - Esquema representativo do sequenciamento do clone D3-9. ... 100
Figura 47 - Esquema representativo do sequenciamento do clone F10-6. ... 101
Figura 48 - Esquema representativo do sequenciamento do clone G2-56. ... 101
Figura 49 - Esquema representativo do sequenciamento do clone H4-8. ... 102
Figura 50 - Esquema representativo do sequenciamento do clone H7-102. ... 102
Figura 51 - Esquema representativo do sequenciamento do clone H10-51. ... 102
Figura 52 - Esquema representativo do sequenciamento do clone D10-2. ... 103
Figura 53 - Esquema representativo do sequenciamento do clone F10-5. ... 103
Figura 54- Esquema representativo do sequenciamento do clone G4-5. ... 104
Figura 55 - Esquema representativo do sequenciamento do clone E4-7. ... 104
2 JUSTIFICATIVA ... 16
3 REVISÃO BIBLIOGRÁFICA ... 17
3.1 ENZIMAS HIDROLÍTICAS DEGRADADORAS DE XILANA ... 17
3.2 MICRORGANISMOS PRODUTORES DE XILANASES ... 21
3.3 ACIDOBACTERIA - GENOMAS E ENZIMAS HIDROLÍTICAS ... 22
3.4 METAGENÔMICA PARA PROSPECÇÃO DE ENZIMAS ... 24
3.5 XILANASES DE RÚMEN ... 25
3.6 SINERGISMO ENTRE ENZIMAS ... 28
4. OBJETIVOS ... 30
4.1 OBJETIVOS ESPECÍFICOS ... 30
4.1.1 Abordagem metagenômica ... 30
4.1.2 Abordagem genômica ... 30
5. MATERIAL E MÉTODOS ... 31
5.1 MONTAGEM BIBLIOTECA GENÔMICA DE ACIDOBACTERIA ... 32
5.2 DETECÇÃO DE CLONES COM ATIVIDADE EM XILANA OU BAGAÇO DE CANA-DE-AÇÚCAR PRÉ-TRATADO. ... 33
5.3 PREPARAÇÃO DE DNA PLASMIDIAL ... 35
5.4 CONFIRMAÇÃO DA ATIVIDADE DOS CLONES PRÉ-SELECIONADOS ... 35
5.5 ANÁLISE DO INSERTO DOS CLONES POSITIVOS ... 35
5.6 SEQUENCIAMENTO E ANÁLISE DAS ORFS PRESENTES NO INSERTO DOS CLONES SELECIONADOS ... 36
5.7 SÍNTESE DO GENE DE B-GLICURONIDASE IDENIFICADO NO CLONE B2-34 ... 37
6. RESULTADOS ... 39
6.1 CLONES DA BIBLIOTECA METAGENÔMICA DE RÚMEN DE CAPRINO ... 39
6.1.1 Seleção dos clones usados no presente trabalho ... 39
6.1.2 Confirmação da atividade dos clones selecionados ... 40
6.1.3 Retransformação dos clones diferentes ... 40
6.1.4 Análise da sequência dos clones selecionados ... 43
6.1.4.1 Clone B2-34 ... 44
6.1.4.1.1 ORF 1 ... 44
6.1.4.3 Clone D3-9 ... 49
6.1.4.3.1 ORF 1 ... 49
6.1.4.4 Clone D8-76 ... 51
6.1.4.4.1 ORF 1 ... 51
6.1.5 Sub-clonagem do gene sintetizado em vetor de expressão ... 54
6.2 GENÔMICA FUNCIONAL DE ACIDOBACTERIA... 55
6.2.1 Montagem da biblioteca e seleção dos clones positivos para atividade hidrolítica em bagaço de cana-de-açúcar e xilana ... 55
6.2.2 Análise do sequenciamento parcial dos clones selecionados na biblioteca genômica de Acidobactéria ... 57
6.2.2.1Clone D10-2 ... 58
6.2.2.1.1ORF 2(F) ... 58
6.2.2.1.2 ORF 1(R) ... 59
6.2.2.2 Clone E4-7 ... 60
6.2.2.2.1 ORF 1(F) ... 61
6.2.2.2.2 ORF 1(R) e 2(R) ... 62
6.2.2.3 Clone G4-5 ... 64
6.2.2.3.1 ORF 1(R) ... 64
7. DISCUSSÃO ... 66
7.1 ESTRATÉGIA DE TRIAGEM ... 66
7.2 BIBLIOTECA METAGENÔMICA ... 69
7.3 BIBLIOTECA GENÔMICA ... 74
8. CONCLUSÃO ... 78
9. PERSPECTIVAS FUTURAS ... 79
REFERÊNCIAS BIBLIOGRÁFICAS ... 80
APÊNDICE A - IDENTIFICAÇÃO DAS ORFS DOS CLONES DA BIBLIOTECA METAGENÔMICA ... 90
APÊNDICE B - IDENTIFICAÇÃO DAS ORFS DOS CLONES DA BIBLIOTECA GENÔMICA ... 96
1 INTRODUÇÃO
As enzimas são catalisadoras de diversas reações industriais, sendo aplicadas nas indústrias de alimentos, cosméticos, medicamentos, têxtil e biocombustíveis. O consumo mundial de enzimas aumenta a cada ano, incentivando a busca por novas enzimas, pois cada aplicação industrial tem suas particularidades, como diferentes tipos de substratos, além de temperaturas e pHs específicos de cada processo industrial.
A produção mundial de enzimas está atualmente concentrada em três grandes companhias: Novozymes (Dinamarca), DuPont (Estados Unidos) e Roche (Suíça). Enzimas hidrolíticas são provavelmente as mais produzidas mundialmente, sendo estimado que essa produção corresponda a 75% do total de enzimas produzidas industrialmente (LI, S. et al., 2012).
Para melhorar a produção de etanol a partir da biomassa lignocelulósica, as grandes empresas que tem a liderança da produção enzimática no planeta tem investido na produção de coquetéis enzimáticos de glicosídeo hidrolases. O uso destes coquetéis traz uma melhoria na produção do etanol em diversos aspectos: redução do custo total de produção, redução na quantidade de enzimas utilizada, redução no tempo de hidrólise, redução na severidade dos pré-tratamentos, redução do impacto ambiental e maior eficiência na conversão da biomassa em açúcares usados na fermentação. Caso essa produção não seja viável economicamente, existe a possibilidade de que as indústrias produtoras do etanol não façam o uso desta biomassa para a produção de etanol de segunda geração, além disso, é importante que o produto final chegue ao consumidor a um preço acessível e viável para que este venha a substituir o uso de outros combustíveis já existentes (gasolina, diesel, etanol de primeira geração) e faça o uso do etanol celulósico evitando a poluição do meio ambiente e o uso excessivo de combustíveis fósseis (DAMHUS et al., 2008).
(C1, C4 e C6), utilizado principalmente na liberação de glicose. Um segundo coquetel enzimático nomeado Cellic® HTec3, composto por hemicelulases, é composto por endo-xilanases e β-xilosidases, podendo ser usado juntamente com o coquetel anterior, ou separadamente para substratos ricos em hemicelulose, auxiliando na liberação de monômeros de xilose, que podem em alguns casos ser fermentados a etanol, butanol, acetato, xilitol e l-lactato (TANAKA et al., 2002; DAMHUS et al., 2008; BETTIGA et al., 2009; LEVASSEUR et al., 2010; LIU et al., 2012; NOVOZYMES, 2014).
As enzimas hidrolíticas são tradicionalmente obtidas de microrganismos, especialmente fungos e bactérias Gram positivas (VAN DIJL; HECKER, 2013). Esses microrganismos secretam enzimas diretamente em tanques de fermentação. Esses microrganismos são originários de diversos ambientes terrestres e aquáticos, incluindo ambientes considerados extremos para a vida, como no caso de microrganismos termófilos ou acidófilos. Essa abordagem, embora bem sucedida, apresenta algumas limitações, como a dificuldade de se cultivar a maioria dos microrganismos existentes no planeta. Além disso, uma vez cultivados e seu potencial de produção enzimática avaliado, cada microrganismo apresenta particularidades de crescimento o que pode influenciar no custo da produção, como é o caso dos organismos termófilos. Assim, a expressão heteróloga de genes que codificam enzimas de interesse tem sido uma alternativa utilizada na indústria. Esta expressão pode ser realizada utilizando microrganismos diversos como: bactérias (Escherichia coli, Bacillus subtilis), leveduras (Sacharomyces cerevisae, Pichia pastoris, Kluyveromyces lactis), e fungos filamentosos (Trichoderma reesei) entre outros (LI, J. et al., 2012; LISBOA et al., 2012; VAN DIJL; HECKER, 2013; ROSANO; CECCARELLI, 2014).
é difícil, por ser um organismo já cultivado e com uma análise prévia realizada no seu genoma. Utilizando a abordagem metagenômica funcional o intuito é buscar enzimas novas, desconhecidas, de organismos ainda não cultivados, e não somente de um organismo específico, mas de um conjunto de organismos presentes no ambiente. Esta abordagem também pode ser realizada por meio da triagem dos genes por bioinformática e tentativa de amplificação dos genes a partir do DNA ambiental, ou produção sintética da enzima (normalmente inviável economicamente), além da montagem de bibliotecas e triagem da mesma forma que na genômica funcional. Entretanto, a triagem dos clones na maioria das vezes deve ser de um número muito maior de clones. Além disso, há a possibilidade de falsos positivos e pode haver dificuldade de se conseguir chegar à produção da enzima de interesse em ambas abordagens, causada pela dificuldade das outras etapas para a expressão da enzima de interesse (HANDELSMAN, 2004).
2 JUSTIFICATIVA
As xilanases são enzimas de grande importância utilizadas em diversas áreas como: produção de papel, panificação, ração para animais, sucos, vinho e produção de etanol de segunda geração, a partir da desconstrução da biomassa lignocelulósica (hemicelulose) e liberação de açúcares para a fermentação.
A busca por enzimas com diferentes características para o uso na produção de etanol de segunda geração, tem sido cada vez maior, pelos diferentes pré-tratamentos pelos quais a biomassa lignocelulósica pode passar antes de chegar a indústria, ácido, básico entre outros. A busca por novos processos de produção tem sido intensificada e cada vez mais tem se buscado enzimas específicas a serem utilizadas em cada processo. Se houver um processo cujo pré-tratamento do bagaço de cana-de-açúcar é ácido e tem altas temperaturas, essas são as características importantes que devem ser consideradas na busca por enzimas que vão atuar neste processo. Se houver a possibilidade do uso de um coquetel enzimático específico para o processo a ser utilizado na indústria, e sua produção for viável economicamente, não existe a necessidade de usar coquetéis criados para o uso em processos com características variadas, que possuem ampla abrangência de atividade, entretanto podem ter uma eficiência menor.
3 REVISÃO BIBLIOGRÁFICA
3.1 ENZIMAS HIDROLÍTICAS DEGRADADORAS DE XILANA
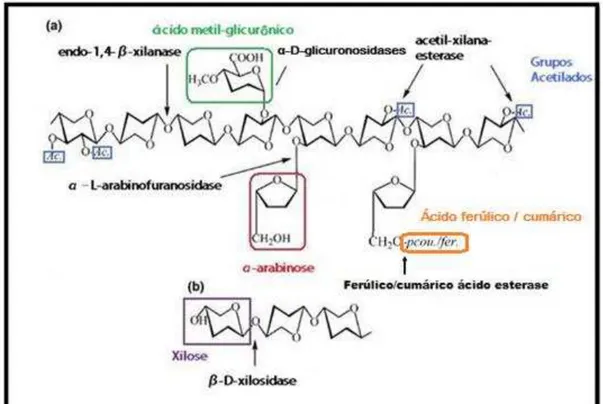
A xilana é o polissacarídeo hemicelulósico mais abundante na maioria das plantas, e pode ser encontrada também em algumas algas. Com exceção da espécie Plantago, que possui ligações β-1,3 e β-1,4, a xilana presente nas plantas superiores possui ligações β-1,4 de xilopiranose (forma cíclica da xilose representada segundo a concepção de Haworth), usualmente sendo substituída por outros açúcares, e adicionado à cadeia principal grupos acetilados. Na parede das plantas, a xilana está inserida entre as cadeias de celulose e a lignina. Ela reveste as fibras de celulose, enquanto suas ramificações interagem com a lignina. A xilana de plantas é formada principalmente por monossacarídeos dentre eles estão: D-xilose (principal monossacarídeo componente da xilana, açúcar pentose), α-arabinose, ácido metil-glicurônico, grupos acetilados, ácido p-cumárico e ferúlico. É formada a partir de ligações β -1,4 de xiloses formando sua cadeia principal e ligadas a estas xiloses existem ramificações dos outros grupos que podem compor esta estrutura em quantidades menores. Por outro lado, a xilana presente na parede celular de algas verdes do gênero Caulerpa sp. é considerada uma homoxilana por não possuir ramificações, sendo constituída por ligações β-1,3 de monômeros de xilose, enquanto em algas vermelhas dos gêneros Palmariales e Nemaliales a xilana presente é constituída de uma combinação de ligações β-1,3 e β-1,4 (EBRINGEROVA; HEINZE, 2000).
A xilana é o principal componente da hemicelulose, sendo o segundo recurso renovável mais abundante no planeta; o primeiro é a celulose. O termo hemicelulose foi instituído por Schulze (1891) como a fração isolada ou extraída de plantas com diluição alcalina, a classificação das frações hemicelulósicas se dá pelo tipo de açúcar presente em cada fração. As hemiceluloses incluem xilana, arabinoxilana, glicuronoxilana, xiloglicana, e glicomanana. Os principais açúcares que compõem as hemiceluloses são xilose (xilana), D-glicose, D-manose, D-galactose e L-arabinose. As hemiceluloses compõem cerca de 20 a 30% das paredes celulares das plantas, mas esta composição é variável de acordo com a planta. A hemicelulose dificulta o acesso de celulases às cadeias celulósicas na parede celular das plantas, durante o processo de degradação do material lignocelulósico para a liberação de açúcares (monossacarídeos) usados na fermentação (BASTAWDE, 1992; BEG et al., 2001).
entre um açúcar e um outro grupo. Estas enzimas possuem diversas formas de classificação. A classificação pode ser relacionada à posição na qual a enzima é capaz de clivar o substrato, nas extremidades (exo), ou no meio da cadeia do substrato (endo). Podem também ser classificadas de acordo com a União Internacional de Bioquímica e Biologia Molecular (IUBMB), em sua forma mais simples de classificação a partir da reação catalisada pela enzima, de modo que se duas enzimas diferentes catalisam a mesma reação bioquímica elas recebem a mesma classificação, caracterizada por um número correspondente, denominado EC – Enzyme Comission. Podem ser classificadas quanto ao seu mecanismo de reação, os mais comuns são por inversão e retenção, algumas famílias necessitam de um co-fator NAD para realizar a hidrólise (GEBLER et al., 1992; MCCARTER; WITHERS, 1994; DAVIES; HENRISSAT, 1995).
Existe um método de classificação baseado na sequência de aminoácidos no qual algoritmos são utilizados para atribuir famílias para as sequências, de modo a classificar as enzimas existentes em famílias baseando-se nas sequências. Outra forma de classificação tem como base a estrutura terciária das enzimas, sendo assim classificadas em "Clans". Cada Clan é correspondente a uma estrutura terciária de enzimas, classificando desta forma, mais de uma família de glicosídeo hidrolases em cada Clan. Todas estas formas de classificação, baseado na sequência de aminoácidos, na posição no qual o substrato é clivado pela enzima, a reação catalisada pela enzima, mecanismo de reação utilizado, e a estrutura terciária que estas enzimas possuem, são utilizadas na classificação de enzimas com atividade em carboidratos e estão presentes na base de dados (Carbohidrates Active Enzymes database - CAZY - http://www.cazy.org). Atualmente as Glicosídeo Hidrolases (GH) estão divididas em 131 famílias. (HENRISSAT, 1991; HENRISSAT; BAIROCH, 1996; CANTAREL et al., 2008).
Figura 1 - Esquema representativo das enzimas necessárias na desconstrução da xilana e o seu produto final liberado. (a) Cadeia principal com as ramificações. (b) Enzima necessária para a liberação do monômero de xilose, açúcar de cinco carbonos usado na fermentação por algumas leveduras.
Fonte: Adaptado de (COLLINS, GERDAY; FELLER, 2005).
Figura 2 - Estrutura da parede celular de cana-de-açúcar. Mostrando alguns dos seus principais componentes: celulose, hemicelulose e lignina.
Fonte: (TUPINAMBA et al., 2013).
O conhecimento que se tem a respeito da estrutura da parede celular dos diversos substratos que podem ser utilizados para a obtenção de açúcares fermentáveis é de suma importância. Uma nova forma de se realizar estes estudos é utilizando o “código glicômico”. De modo a detectar a cadeia principal dos vários componentes das paredes celulares (celulose, hemicelulose e pectina). Além disso estudar quais são as ramificações compostas por açúcares que poderão ser utilizados na fermentação e quais são as ramificações que não são açúcares (BUCKERIDGE; DE SOUZA, 2014).
levando a uma mudança no processo de produção do bioetanol (BUCKERIDGE; DE SOUZA, 2014).
3.2 MICRORGANISMOS PRODUTORES DE XILANASES
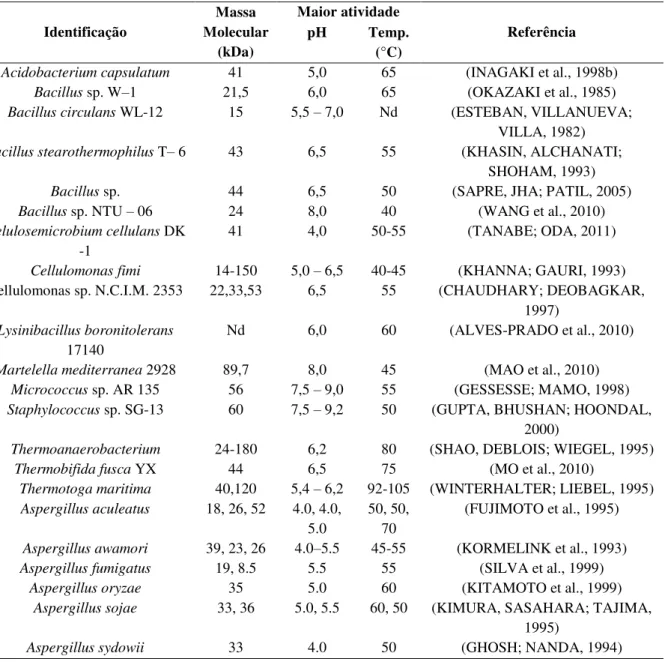
Dentre os microrganismos produtores de xilanases encontram-se os fungos filamentosos e as bactérias (Tabela 1).
Tabela 1 - Características de xilanases produzidas por bactérias e fungos filamentosos
Identificação Massa Molecular (kDa) Maior atividade Referência pH Temp.
(°C)
Acidobacterium capsulatum 41 5,0 65 (INAGAKI et al., 1998b) Bacillus sp. W–1 21,5 6,0 65 (OKAZAKI et al., 1985) Bacillus circulans WL-12 15 5,5 – 7,0 Nd (ESTEBAN, VILLANUEVA;
VILLA, 1982) Bacillus stearothermophilus T– 6 43 6,5 55 (KHASIN, ALCHANATI;
SHOHAM, 1993)
Bacillus sp. 44 6,5 50 (SAPRE, JHA; PATIL, 2005)
Bacillus sp. NTU – 06 24 8,0 40 (WANG et al., 2010) Celulosemicrobium cellulans DK
-1
41 4,0 50-55 (TANABE; ODA, 2011)
Cellulomonas fimi 14-150 5,0 – 6,5 40-45 (KHANNA; GAURI, 1993) Cellulomonas sp. N.C.I.M. 2353 22,33,53 6,5 55 (CHAUDHARY; DEOBAGKAR,
1997) Lysinibacillus boronitolerans
17140
Nd 6,0 60 (ALVES-PRADO et al., 2010)
Martelella mediterranea 2928 89,7 8,0 45 (MAO et al., 2010) Micrococcus sp. AR 135 56 7,5 – 9,0 55 (GESSESSE; MAMO, 1998) Staphylococcus sp. SG-13 60 7,5 – 9,2 50 (GUPTA, BHUSHAN; HOONDAL,
2000)
Thermoanaerobacterium 24-180 6,2 80 (SHAO, DEBLOIS; WIEGEL, 1995)
Thermobifida fusca YX 44 6,5 75 (MO et al., 2010)
Thermotoga maritima 40,120 5,4 – 6,2 92-105 (WINTERHALTER; LIEBEL, 1995) Aspergillus aculeatus 18, 26, 52 4.0, 4.0,
5.0
50, 50, 70
(FUJIMOTO et al., 1995)
Aspergillus awamori 39, 23, 26 4.0–5.5 45-55 (KORMELINK et al., 1993) Aspergillus fumigatus 19, 8.5 5.5 55 (SILVA et al., 1999)
Aspergillus oryzae 35 5.0 60 (KITAMOTO et al., 1999) Aspergillus sojae 33, 36 5.0, 5.5 60, 50 (KIMURA, SASAHARA; TAJIMA,
1995)
Aspergillus sydowii 33 4.0 50 (GHOSH; NANDA, 1994)
É possível verificar que existe uma grande variação na massa molecular das xilanases demonstradas na tabela acima, entre 8,5 e 180 kDa, mas em média essa massa molecular está entre 40 e 50 kDa. Em relação ao pH de maior atividade destas enzimas é possível verificar que o das enzimas de fungos é mais ácido variando entre 4 e 5,5, enquanto o pH de maior atividade das enzimas de bactérias varia entre 4,0 e 9,2. A temperatura de maior atividade também é variável, em fungos vai de 45 a 70ºC e nas xilanases de bactérias descritas acima esta temperatura varia entre 40 e 105ºC, e a média de temperatura está entre 50 e 60ºC das xilanases de bactérias listadas acima.
3.3 ACIDOBACTERIA - GENOMAS E ENZIMAS HIDROLÍTICAS
Atualmente existem nove genomas de bactérias pertencentes ao filo Acidobacteria sequenciados, publicados e disponíveis para comparações no banco de dados. Acidobacterium capsulatum, a espécie tipo do filo, foi isolada de drenagem ácida de mina no Japão, Candidatus Koribacter versatilis e Candidatus Solibacter usitatus foram isoladas de solo de pastagem na Austrália, Terriglobus saanencis, Granulicella mallensis e Granulicella tundricola isoladas de solo de tundra na Finlândia, Holophaga foetida isolada de amostra de lama de água doce em Konstanz na Alemanha, Candidatus Chloracidobacterium thermophilum isolado de fontes termais no Parque Nacional de Yellowstone, Terriglobus roseus, espécie tipo do gênero Terriglobus, isolada de solos agrícolas e Geotrix fermentans isolada de uma zona sedimentada contaminada por petróleo que possui o genoma parcialmente sequenciado. Os tamanhos dos genomas são variáveis e vão desde 3,7 Mb (Candidatus Chloracidobacterium thermophilum) a 9.0 Mb (Solibacter usitatus). O genoma da espécie tipo tem 4,1 Mb (WARD et al., 2009; ANDERSON et al., 2012; GARCIA COSTAS et al., 2012; RAWAT, MANNISTO, BROMBERG, et al., 2012; RAWAT, MANNISTO, STAROVOYTOV, et al., 2012).
sequências e 79 sequências da K. versatili. Por meio de relações filogenéticas, Ward (2009) demonstrou que os genes das enzimas α-glicuronidases presentes nos primeiros genomas sequenciados têm maior proximidade com fungos do que com bactérias. E ao final do trabalho concluiu-se que eram necessários mais estudos de fisiologia (isolamento e caracterização) para validar experimentalmente se estas bactérias possuem um papel importante na qualidade do solo através da decomposição da matéria orgânica, na ciclagem de nutrientes e na formação de agregados no solo, utilizando-se destas enzimas para a realização do seu papel no ecossistema no qual estão presentes. Na época em que tiveram os seus genomas sequenciados, estas bactérias haviam sido classificadas entre os 5% de genomas de bactérias que mais possuíam genes codificadores para glicosídeo hidrolases. Mais tarde, com a análise do sequenciamento dos outros três genomas foi possível prever que os novos genomas sequenciados possuíam ainda mais sequências de genes que codificam para estas enzimas, sendo que G. mallensis possui 166, G. tundricola 103 e T. saanensis 110 sequências codificadoras para glicosídeo hidrolases. Entretanto não se sabe se todos estes genes estão ativos (WARD et al., 2009; GARCIA COSTAS et al., 2012; RAWAT, MANNISTO, BROMBERG, et al., 2012; RAWAT, MANNISTO, STAROVOYTOV, et al., 2012).
Utilizando como base o genoma da A. capsulatum, algumas enzimas foram isoladas por meio da técnica de genômica funcional. Dentre elas estão uma xilanase ácida pertencente à família GH10, cujo pH e temperatura de maior atividade são 5,0 e 65ºC, respectivamente, e a massa molecular é de 41 kDa. Além disso, também foi isolada uma hidrolase da família GH79 β-Glicuronidase que foi caracterizada tanto bioquímica quanto estruturalmente, cujo pH e temperatura de maior atividade são pH 3,0 e 50ºC, respectivamente. Uma β-glicosidase ácida da espécie tipo também foi isolada, tendo massa molecular 90 kDa e pH e temperatura de maior atividade 3,0 e 55ºC, respectivamente. Outra enzima, uma trealose da família GH28, também foi caracterizada e tem pH e temperaturas de maior atividade 2,5 e 65ºC e massa molecular de 57 kDa (KISHIMOTO et al., 1991; INAGAKI et al., 1998a; INAGAKI et al., 2001; MICHIKAWA et al., 2012).
3.3.1 Acidobacteria AB60
Os genomas sequenciados de bactérias pertencentes ao filo Acidobacteria representam um grande potencial enzimático, não só para xilanases como para celulases, quitinases e outras enzimas de interesse biotecnológico. Existem poucas enzimas de Acidobacteria purificadas e caracterizadas. Desta forma, a busca por enzimas diferentes presentes nestas bactérias isoladas de solo de Cerrado é um potencial a ser explorado (WARD et al., 2009; GARCIA COSTAS et al., 2012; RAWAT, MANNISTO, STAROVOYTOV, et al., 2012).
A Acidobacteria AB60, foi isolada do solo de Cerrado. Esta bactéria pertence à subdivisão 1 deste filo, a subdivisão que possui o maior número de isolados. Este isolado possui 94,8% de similaridade no gene rRNA 16S com a espécie tipo Acidobacterium capsulatum, entretanto até o momento o genoma da AB60 ainda não foi sequenciado. Esta bactéria foi isolada em um meio cuja única fonte de carbono era xilana. Além disso, a AB60 tem capacidade de utilizar diversos monossacarídeos como fonte de carbono, como por exemplo arabinose, xilose, glicose e frutose. Outros polissacarídeos complexos foram testados como carboximetilcelulose e quitina, entretanto não houve crescimento em meio líquido (DE CASTRO, 2011; DE CASTRO et al., 2013).
3.4 METAGENÔMICA PARA PROSPECÇÃO DE ENZIMAS
Cerca de 99% dos micro-organismos do planeta ainda não são cultiváveis de forma isolada em laboratório. O uso da abordagem metagenômica veio para possibilitar o acesso a diversidade genética destes micro-organismos. Assim, a metagenômica possibilitou o aumento do conhecimento sobre estes micro-organismos e seu papel no ecossistema do qual fazem parte (HANDELSMAN et al., 2002; TORSVIK; ØVREÅS, 2002; HANDELSMAN, 2004; SABREE, RONDON; HANDELSMAN, 2009).
A prospecção de enzimas na abordagem metagenômica está dividida em três grupos principais: identificação baseada em sequenciamento, triagem baseada na expressão e visualização da função (metagenômica funcional) e "SIGEX" que é uma triagem baseada na indução do gene pelo substrato (HANDELSMAN, 2005).
A triagem baseada na expressão e visualização da atividade é baseada na expressão heteróloga de proteínas, na grande maioria das vezes realizada em Escherichia coli, havendo a necessidade de detecção visual da atividade procurada e criação de métodos de triagem específicos para a atividade a ser detectada (UCHIYAMA; MIYAZAKI, 2009).
Existe um terceiro método, denominado "SIGEX" que utiliza um sistema diferenciado com um gene repórter ligado a um conjunto de genes codificantes de vias biodegradativas, e este método é baseado na ideia de que o operon a ser detectado é induzido pelo substrato da via. De modo que, quando o inserto possui o operon induzido pelo substrato o clone ascende e os estudos na sequência levam à detecção dos genes responsáveis pela atividade. Entretanto, esta abordagem também possui algumas limitações, apesar da facilidade de visualização da atividade quando o método é utilizado (UCHIYAMA et al., 2005; UCHIYAMA; WATANABE, 2007).
Apesar das dificuldades, diversos trabalhos já foram realizados e enzimas pertencentes a micro-organismos de ambientes variados como solo, água, rúmen, intestino humano, com características variadas relativas a pH, temperatura e atividade funcional (celulases, xilanases, amilases, lipases) foram identificadas por abordagem metagenômica, sem a necessidade do cultivo destes organismos. Aliada a exploração do potencial biotecnológico dos micro-organismos presentes nestes ambientes, a metagenômica tem ajudado a desvendar um pouco da diversidade presente nestes ambientes. Além disso, estudos de metagenômica têm trazido maior conhecimento a respeito destes, ajudando também no seu isolamento para estudo e exploração usando abordagem tradicional. Assim, as abordagens metagenômica e tradicional complementam-se uma a outra (HANDELSMAN et al., 2002; BRENNAN et al., 2004; HANDELSMAN, 2004; VENTER et al., 2004; YUN et al., 2004; DANIEL, 2005; LECLEIR et al., 2007; DUAN et al., 2009; ARUMUGAM et al., 2011; FAORO et al., 2012).
3.5 XILANASES DE RÚMEN
desconstruir esta biomassa é um ponto que vem sendo explorado por diversos trabalhos. Entretanto, a grande maioria destes trabalhos é realizado com bovinos, ovinos e alguns trabalhos realizados com caprinos. Os caprinos da raça moxotó, raça muito presente no nordeste brasileiro, tem um grande potencial a ser explorado, por viverem em um ambiente com alimentação escassa onde a total ou grande parte da sua alimentação (plantas da catinga brasileira) tem de ser aproveitada, aproveitamento este feito pelos micro-organismos presentes no rúmen destes animais, além de o potencial biotecnológico presente neste ambiente ser pouco explorado (KAMRA, 2005; OLIVEIRA, ZANINE; SANTOS, 2007; CUNHA, KRUGER; QUIRINO, 2009; CUNHA, 2010; CUNHA et al., 2011).
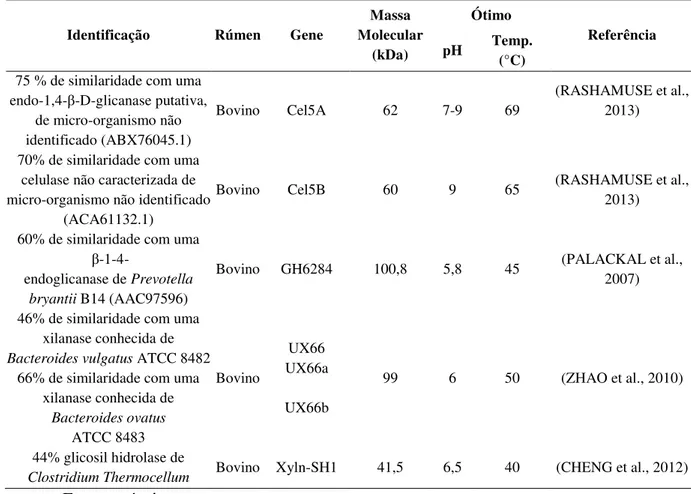
Tabela 2 - Trabalhos de metagenômica de rúmen onde enzimas com atividade xilanolítica foram isoladas.
Identificação Rúmen Gene
Massa Molecular
(kDa)
Ótimo
Referência pH Temp.
(°C)
75 % de similaridade com uma endo-1,4-β-D-glicanase putativa,
de micro-organismo não identificado (ABX76045.1)
Bovino Cel5A 62 7-9 69
(RASHAMUSE et al., 2013)
70% de similaridade com uma celulase não caracterizada de micro-organismo não identificado
(ACA61132.1)
Bovino Cel5B 60 9 65 (RASHAMUSE et al.,
2013) 60% de similaridade com uma
β-1-4-
endoglicanase de Prevotella bryantii B14 (AAC97596)
Bovino GH6284 100,8 5,8 45 (PALACKAL et al., 2007) 46% de similaridade com uma
xilanase conhecida de Bacteroides vulgatus ATCC 8482
66% de similaridade com uma xilanase conhecida de
Bacteroides ovatus ATCC 8483 Bovino UX66 UX66a UX66b
99 6 50 (ZHAO et al., 2010)
44% glicosil hidrolase de
Clostridium Thermocellum Bovino Xyln-SH1 41,5 6,5 40 (CHENG et al., 2012)
Fonte: próprio autor.
Existem algumas xilanases já descritas que possuem tanto ação xilanolítica, quanto celulósica como a Cel5A, Cel5B e GH6284. Isto é um aspecto importante a ser levado em consideração para uso industrial, já que por possuir as duas atividades a ação na desconstrução da biomassa lignocelulósica pode ser aumentada, aumentando a liberação de açúcares disponíveis para a fermentação. A capacidade da enzima de estar ativa em diferentes substratos pode estar relacionada à presença do domínio para a atividade nos diferentes substratos presente em sua estrutura, ou também à presença de um sítio ativo sem grande especificidade que tem a capacidade de se ligar a diferentes substratos (PALACKAL et al., 2007; VAN DYK; PLETSCHKE, 2012; RASHAMUSE et al., 2013).
As xilanases são muito diversas em diversos níveis de classificação como: estruturalmente (presença de domínio catalítico, presença ou ausência de CBMs), características bioquímicas (pH e temperatura ótimos e atividade, sensibilidade a variação destes), catálise (seleção de substrato, eficiência catalítica e sensitividade a inibidores). Existem duas famílias de grande importância relativas a xilanases, dentre elas estão a família das glicosideo hidrolases 10 e glicosideo hidrolases 11, além disso estão entre as famílias de enzimas mais estudadas relacionadas a xilanases (PAËS, BERRIN; BEAUGRAND, 2012).
Tanto na família GH10 quanto na família GH11 estão presentes enzimas pertencentes a bactérias e fungos consideradas xilanases, devido a alta especificidade das enzimas pertencentes a família GH11 alguns classificam estas enzimas como xilanases verdadeiras (PAËS, BERRIN; BEAUGRAND, 2012).
3.6 SINERGISMO ENTRE ENZIMAS
Sinergismo é a relação da quantidade de produto resultante de uma determinada reação em um determinado substrato, comparando o uso de duas ou mais enzimas juntas em relação à atividade destas enzimas separadamente neste mesmo substrato. Mostrando que o valor de produto liberado é maior com as enzimas trabalhando em conjunto em relação ao uso separado destas enzimas (COUGHLAN et al., 1993; VAN DYK; PLETSCHKE, 2012).
A produção de coquetéis enzimáticos é baseada em grande parte no que se sabe a respeito do sinergismo entre enzimas, além de testes confirmatórios de que em determinada quantidade a capacidade de catálise em relação a substratos determinados é aumentada, utilizando mais de uma enzima em conjunto.
Em estudos com hemicelulases, é necessária a quantificação dos diversos açúcares a serem liberados, além de outras substâncias que também estão presentes em sua cadeia e podem ser liberadas através da ação em sinergismo das enzimas. Isso deve ser realizado por meio da separação, identificação e quantificação destas substâncias resultantes da atividade das enzimas em determinado substrato. Três tipos de sinergismos foram identificados e nomeados: homosinergismo, heterosinergismo e anti-sinergismo. O homosinergismo é o sinergismo que ocorre entre enzimas do mesmo tipo que possuem a mesma função, de modo que ao serem usadas em conjunto verifica-se um aumento na liberação de produtos, em relação ao uso separado. Heterosinergismo ocorre entre as enzimas que clivam a cadeia principal e as enzimas que clivam as ramificações, podendo levar a um aumento significativo não só de um dos produtos liberados, por exemplo ao realizar a hidrólise de arabinoxilana fazendo o uso somente de uma α-arabinofuranosidase, obtem-se uma certa quantidade de arabinose liberada (açúcar redutor), entretanto se for adicionado juntamente uma endo-xilanase que vai agir na cadeia principal, além de um aumento na liberação de arabinose, pode verificar-se um aumento na xilose e xilobiose liberados como produtos desta reação. Quando a ação de uma enzima inibe a ação de outra enzima, isto é denominado anti-sinergismo, esta relação só pode ocorrer in vitro, quando uma enzima necessita de um substituinte específico para a realização da reação e de algum modo a outra enzima utilizada também necessita deste mesmo substituinte para a sua reação, desta forma as enzimas acabam competindo e uma delas é incapaz de realizar a sua reação levando ao anti-sinergismo (COUGHLAN et al., 1993; VAN DYK; PLETSCHKE, 2012).
4. OBJETIVOS
O objetivo geral deste trabalho é a busca de genes que codifiquem para enzimas com atividade xilanolítica, que possam ser utilizados na conversão de biomassa lignocelulósica a açúcares fermentáveis, utilizando abordagens genômica e metagenômica.
4.1 OBJETIVOS ESPECÍFICOS
4.1.1 Abordagem metagenômica
- Confirmação da atividade de clones positivos para atividade xilanolítica de uma sub-biblioteca metagenômica de rúmen de caprino da raça moxotó;
- Sequenciamento dos clones com atividade xilanolítica confirmada;
- Análise da sequência dos clones em busca dos genes responsáveis pela atividade detectada;
- Tentativa de sub-clonagem da ORF selecionada e identificada pela análise da sequência dos clones em vetor de expressão;
4.1.2 Abordagem genômica
- Montagem de biblioteca genômica de insertos entre 4 e 8 kb utilizando o DNA da bactéria Acidobacteria AB60 isolada de solo de Cerrado;
- Triagem da biblioteca genômica para detecção clones com atividade xilanolítica; - Sequenciamento dos clones positivos para a atividade detectada;
5. MATERIAL E MÉTODOS
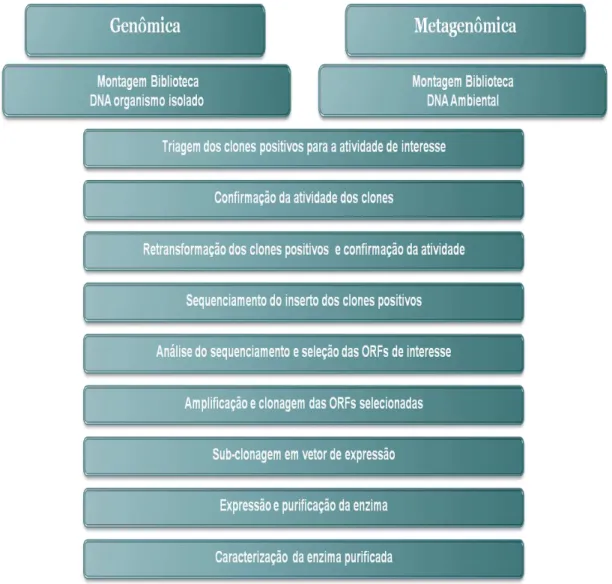
As abordagens genômica e metagenômica são duas abordagens possíveis de serem utilizadas na prospecção de enzimas. As duas abordagens se utilizam de técnicas em comum. Por exemplo, em ambas as abordagens são construídas bibliotecas, entretanto, na abordagem genômica utiliza-se DNA genômico de um organismo isolado e na abordagem metagenômica utiliza-se um conjunto de DNAs (metagenômicos) presentes no ambiente de interesse. As etapas subsequentes podem ser as mesmas para se atingir o objetivo final de obter-se o gene isolado, e a enzima codificada por ele purificada e caracterizada (Figura 3).
Figura 3 - Etapas básicas utilizadas nas abordagens genômica e metagenômica, mostrando as suas semelhanças apesar das particularidades das técnicas.
5.1 MONTAGEM BIBLIOTECA GENÔMICA DE ACIDOBACTERIA
A bactéria AB60, pertencente ao filo Acidobacteria, utilizada para a montagem da biblioteca genômica no presente trabalho foi obtida em trabalho anterior em 2009 durante o desenvolvimento de uma dissertação de mestrado no programa de Ciências Genômicas e Biotecnologia da Universidade Católica de Brasília (DE CASTRO, 2011; DE CASTRO et al., 2013).
A extração de DNA genômico bacteriano foi realizada após o crescimento de 4 semanas em 100 mL de meio VL55 líquido, para um litro de meio de cultura utilizou-se 3,9 g de ácido 2-[N-morfolino] etanosulfônico (MES), sulfato de magnésio (MgSO4 0,4 mM), cloreto de cálcio (CaCl2 1,2 mM), fosfato de amônia dibásico ((NH4)2HPO4 0,4 mM), solução de tungstato e selenito (2 mL), solução de vitaminas (2 mL), solução de elementos traço (2 mL) com xilana na concentração 0,05% como única fonte de carbono. Para a extração foi utilizado o protocolo baseado em (Wilson, 1989) (WILSON, 1989; SAIT, HUGENHOLTZ; JANSSEN, 2002).
A análise da qualidade e quantidade do DNA extraído foi realizada por eletroforese em gel de agarose na concentração de 1%.
O kit utilizado para clonagem foi o CopyControl Bac Cloning Kit (Epicentre) e a cepa utilizada para a transformação foi a Escherichia coli EPI300 (Epicentre). O CopyControl Bac Cloning kit possui três sítios de clonagem (EcoRI, BamHI e HindIII), de modo que o sítio escolhido para a clonagem no presente trabalho foi o da enzima EcoRI. O DNA genômico preparado foi digerido da seguinte forma: para cada 500 ng de DNA foram utilizadas 2 unidades de enzima EcoRI, incubando-se a 37ºC por 4 horas. Os fragmentos de DNA provenientes da digestão do DNA foram separados em gel de agarose low melting point (Invitrogen) a 1% por 16 horas a 30 volts. Fragmentos de DNA genômico entre 4 a 8 kb foram identificados com o auxílio de marcador molecular 1 kb Ladder (Invitrogen) e removidos do gel cortando-se a região de interesse com um bisturi.
A transformação foi realizada por eletroporação de acordo com o protocolo do Gene Pulser II (Biorad). Foram utilizados os parâmetros definidos para Escherichia coli, capacitância 25 µF, cuveta 0,1 cm e 1,8 kV de passagem de eletricidade no eletroporador. Foram utilizados 2 µL de ligação e 50 µL de célula competente da cepa EPI300.
As células transformadas foram incubadas por 1 hora a 37°C em 1 mL de meio SOC, sob agitação de 220 rpm. Depois de recuperadas, as células transformadas foram plaqueadas em meio Luria-Bertani (LB) adicionado de cloranfenicol (12,5 microgramas/mL), 5-bromo-4-chloro-3-indolil-β-D-galactopiranosideo (X-Gal 80 μg/mL) e isopropil-β -D-thiogalactopiranosideo (IPTG 0,5 mM). As placas foram incubadas por 16 horas a 37°C para o crescimento das colônias transformadas. As colônias com coloração branca foram selecionadas por “blue-white screening” e estocadas em placas de 96 poços em glicerol a 20% em freezer -80°C. Colônias aleatórias foram selecionadas para a análise do tamanho do inserto, por meio de mini-extração de DNA plasmidial e eletroforese do DNA em gel de agarose para a validação da biblioteca.
Para determinação do número de clones necessários para a montagem da biblioteca genômica em questão, a fim de o gene de interesse pudesse estar inserido na biblioteca, foi utilizada a seguinte equação: N=ln(1-P)/ln(1-[I/GS]), onde P é a probabilidade de o gene de interesse estar representado na biblioteca (normalmente usa-se 99%), I é o tamanho do inserto inserido no seu vetor neste caso 4 a 8 kb (cálculo realizado com 4 kb). GS é o tamanho do genoma clonado na biblioteca a ser montada (4.1 Mb genoma completo da Acidobacterium capsulatum espécie tipo do filo Acidobacteria). N é o número de clones necessário para se atingir estes parâmetros (4.610 clones).
5.2 DETECÇÃO DE CLONES COM ATIVIDADE EM XILANA OU BAGAÇO DE CANA-DE-AÇÚCAR PRÉ-TRATADO
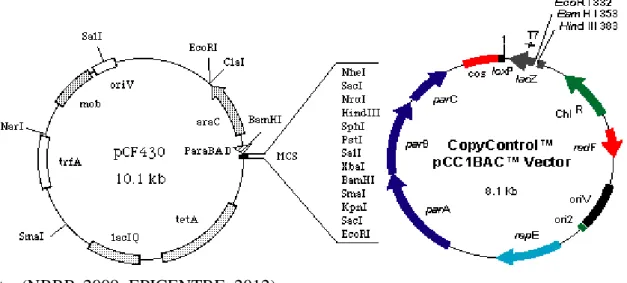
de seleção relativo a cada biblioteca (tetraciclina - gene tetA - clones da biblioteca metagenômica de rúmen de caprino) ou (cloranfenicol- gene ChlR - clones da biblioteca genômica de acidobactéria), e o indutor correspondente a cada biblioteca (arabinose - biblioteca de rúmen devido ao gene regulador araC e o gene promotor pBAD) ou (IPTG - biblioteca genômica de acidobactéria devido ao gene lacZ. A arabinose também foi utilizada na triagem com o vetor da biblioteca genômica, pois como no pCC1 FOS ela é utilizada para aumentar o número de cópias do vetor) presentes nos clones. O vetor utilizado na biblioteca metagenômica de rúmen de caprinos foi o pCF430, enquanto o vetor utilizado na biblioteca genômica da AB60 foi o pCC1BAC (Figura 4) (NEWMAN; FUQUA, 1999; SABREE, RONDON; HANDELSMAN, 2009; EPICENTRE, 2012).
Figura 4 - Mapa dos vetores utilizados na construção das bibliotecas. Vetor pCF430 - biblioteca metagenômica de rúmen de caprino e vetor CopyControl pCC1BAC - biblioteca genômica de Acidobacteria.
Fonte: (NBRP, 2009; EPICENTRE, 2012).
5.3 PREPARAÇÃO DE DNA PLASMIDIAL
O protocolo utilizado para a extração de DNA plasmidial foi baseado no protocolo descrito por Azevedo (2003). A preparação dos plasmídeos se deu a partir da extração de DNA plasmidial por lise alcalina das células. O cultivo inicial das células foi realizado em 100 mL de meio LB com o antibiótico adequado por 16h a 37ºC. Depois de crescidas, as células foram centrifugadas, o sobrenadante foi descartado, e ressuspendidas em STE (solução utilizada para retirada de excesso de meio de cultura) por duas vezes. Após a lavagem, as células foram centrifugadas novamente e as soluções I, II e III foram adicionadas, para a lise das células e liberação dos plasmídeos. Após esta etapa o isopropanol foi utilizado para a sedimentação do DNA plasmidial e depois o DNA foi lavado com uma solução de etanol 70% (AZEVEDO, 2003).
5.4 CONFIRMAÇÃO DA ATIVIDADE DOS CLONES PRÉ-SELECIONADOS
Os clones pré-selecionados a partir da detecção da atividade, tiveram o seu DNA plasmidial extraído e foram retransformados. Após a retransformação eles passaram por um novo processo para a confirmação da atividade como descrito anteriormente. Os clones com a atividade confirmada após a retransformação foram estocados em glicerol 20% no freezer a -80ºC e a sequência de nucleotídeos do insertos foi determinada.
5.5 ANÁLISE DO INSERTO DOS CLONES POSITIVOS
5.6 SEQUENCIAMENTO E ANÁLISE DAS ORFS PRESENTES NO INSERTO DOS CLONES SELECIONADOS
O sequenciamento foi realizado utilizando a técnica de primer walking. No início do sequenciamento foram usados primers (iniciadores) conhecidos pertencentes aos próprios vetores onde foram clonados os insertos (Tabela 3) nos dois sentidos 5’3’ para frente
(forward) e no sentido reverso 3’5’ (reverse) e com a continuidade do sequenciamento outros iniciadores foram montados utilizando padrões como: tamanho (ente 18 e 22 bases), temperatura de anelamento (entre 55º e 60ºC), e proporção do conteúdo de GC presente no primer (entre 45 e 55% de conteúdo GC). Estes novos iniciadores tinham como base a sequência anterior e foram montados utilizando o programa Primer3Plus (Untergasser et al., 2007), para a formação de contigs (sequências de DNA de tamanhos maiores, utilizando sequências menores alinhadas por bases em comum) e montagem da sequência dos clones.
Tabela 3 - Lista dos primers iniciais utilizados no sequenciamento dos clones
Primer Sequência Referências
ID pCC1TM 1 5'GGATGTGCTGCAAGGCGATTAAGTTGG3' (EPICENTRE, 2011) IR pCC1TM 2 5'CTCGTATGTTGTGTGGAATTGTGAGC3' (EPICENTRE, 2011) pCF430(D)3 5'CTGTTTCTCCATACCCGTT3' (CASTRO et al., 2011) pCF430(R)4 5'TGCAAGGCGATTAAGTTGG3' (CASTRO et al., 2011) Fonte: próprio autor. 1. ID: Iniciador direto; 2. IR: Iniciador reverso; 3. (D) Direto; 4. (R) Reverso.
sequências de DNA foram feitos utilizando-se o programa Muscle (EDGAR, 2004; TAMURA et al., 2013).
5.7 SÍNTESE DO GENE DE B-GLICURONIDASE IDENIFICADO NO CLONE B2-34 No clone B2-34 (clone da biblioteca de rúmen de caprinos) foi detectada uma ORF de interesse, potencialmente codificadora de uma enzima B-glicuronidase, enzima pertencente a família 2 das glicosil hidrolases.
Este gene foi selecionado para a síntese, por ser o gene que possuía o alinhamento mais próximo com sequências curadas de proteínas e mais completo em relação aos presentes no banco de dados. A empresa na qual a síntese foi realizada foi Life TechnologiesTM.
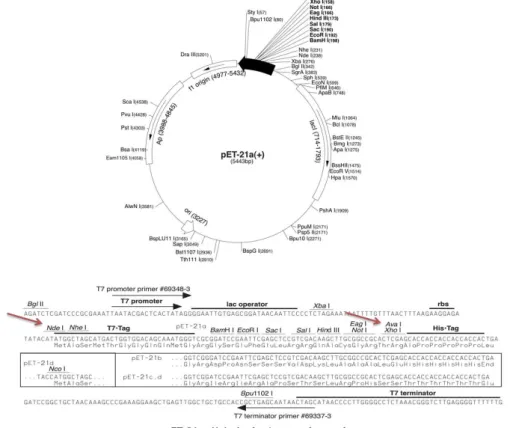
O gene sintetizado possuía 1824 pb e foi otimizado para a expressão em Escherichia coli além de serem adicionados sítios de restrição das enzimas NdeI e XhoI, fazendo com que o gene sintético tenha 1830 pb. A imagem representa o gene sintetizado enviado pela empresa no vetor pMK-RQ (Figura 5) (Tabela 11) (Apêndice E).
Figura 5 - Figura representativa do gene sintetizado em vetor pMK-RQ, vetor que apresenta um gene de resistência ao antibiótico canamicina.
O gene sintético do Clone B2-34 contém 1830 pb e possui em suas extremidades o sítio de restrição das enzimas NdeI e XhoI.
Fonte: (GENEART, 2014).
clonagem. Após a confirmação a célula transformada foi estocada em freezer a -80ºC em glicerol a 20%.
Figura 6 - Mapa do vetor de expressão utilizado na tentativa de clonagem do gene sintetizado.
Fonte: (NOVAGEN, 2014).
6. RESULTADOS
6.1 CLONES DA BIBLIOTECA METAGENÔMICA DE RÚMEN DE CAPRINO
6.1.1 Seleção dos clones usados no presente trabalho
Uma biblioteca metagenômica de DNA de micro-organismos do rúmen de caprinos da raça Moxotó foi montada anteriormente (CUNHA, 2010). Essa biblioteca contém fragmentos de DNA nos tamanhos entre 5 e 8 kb clonados em vetor pCF430 (NEWMAN; FUQUA, 1999). Um total de 463 clones com atividade hidrolítica em celulose foram inicialmente selecionados (RAMOS, 2011).
Uma sub-biblioteca contendo os clones selecionados na primeira triagem foi montada e estes clones foram posteriormente avaliados quanto à atividade hidrolítica em outros substratos como: CMC de alta viscosidade, CMC de baixa viscosidade, xilana, avicel e bagaço de cana-de-açúcar pré-tratado com ácido e macerado. Essas atividades foram avaliadas em presença ou ausência de arabinose, pois o vetor possui um promotor induzível por esse açúcar na região 5’ do fragmento clonado. A intensidade de coloração exibida pelos clones no ensaio em placas de petri foi avaliada sendo a menor atividade observada representara por (+); e a maior atividade por (+++) (RODRIGUES, 2014).
A partir desta segunda triagem foram escolhidos 16 clones com atividade em xilana e/ou em xilana combinada ao bagaço de cana-de-açúcar pré-tratado e 3 clones que possuíam atividade em CMC, xilana e bagaço de cana-de-açúcar (Tabela 4) para o presente trabalho.
Tabela 4 - Atividade inicial dos clones de biblioteca metagenômica de rúmen caprino em diversos substratos com e sem o uso do indutor arabinose
CMC de alta Viscosidade Bagaço de cana-de-açúcar
pré-tratamento ácido Xilana beechwood
Clones Arabinose +
Arabinose - Arabinose +
Arabinose - Arabinose + Arabinose -
F10-6 +++ - +++ - +++ -
H4-8 +++ - +++ + +++ -
D3-9 +++ - +++ - +++ -
A7-34 - - - - + -
B1-23 - - - - + -
B12-1 - - - + - +
B2-34 - - + - ++ +
B8-62 - - +++ - + -
C2-74 - - - + - +
C5-95 - - - +
C9-1 - - - + - ++
D3-54 - - ++ - + -
D8-76 - - + - + -
E1-5 - - - - + -
F1-7 - - ++ - ++ -
G2-56 - - ++ - ++ -
H10-51 - - - - + -
H4-51 - - - - + -
H7-102 - - - - + -
Fonte: Adaptado de (RODRIGUES, 2014). Atividade apresentada pelos clones sendo (+++) a maior atividade, (++) atividade média, (+) menor atividade e (-) sem atividade.
6.1.2 Confirmação da atividade dos clones selecionados
Para a execução do presente trabalho, foi realizada inicialmente a confirmação da atividade dos 19 clones selecionados, utilizando-se bagaço de cana-de-açúcar com pré-tratamento ácido e xilana como substrato com adição de arabinose. Em cada placa havia 5 colônias, de um mesmo transformante, de cada clone para verificar a atividade e todos os clones permaneceram com a atividade anteriormente descrita na (Tabela 4).
Após a confirmação da atividade, os 19 clones selecionados tiveram o seu plasmídeo extraído e digerido com enzima PstI para a diferenciação e seleção dos clones diferentes para a retransformação.
6.1.3 Retransformação dos clones diferentes
sequenciados (Figuras 8 e 9). Antes de serem sequenciados, os clones foram novamente digeridos com a enzima PstI, para a verificação de clones contendo insertos distintos, além da análise do tamanho esperado para o sequenciamento (Figura 7).
Figura 7 - Perfil de restrição dos clones de rúmen para seleção de insertos distintos. Clones retransformados digeridos com enzima PstI para a diferenciação e análise do tamanho. Todos os clones tiveram o inserto intacto e digerido apresentados de forma sequencial. 1, 8, 15, 22, 29 e 36: Marcador molecular Ladder 1 kb plus (Invitrogem); 2 e 3: clone B12-1; 4 e 5: clone C9-; 6 e 7: clone E1-5; 9 e 10: clone F10-6; 11 e 12: clone F1-7; 13 e 14: clone H4-8; 16 e 17: clone D3-9; 18 e 19: clone B2-34; 20 e 21: clone A7-34; 23 e 24: clone H4-51; 25 e 26: clone H10-51; 27 e 28: clone D3-54; 30e 31: clone G2-56; 32 e 33: clone B8-62; 34 e 35: clone C2-74; 37 e 38:clone D8-76; 39 e 40: clone C5-95; 41 e 42: clone H7-102.
Fonte: próprio autor.
Figura 8 - Confirmação da atividade em xilana dos clones de rúmen em ensaios em placas após a retransformação. Os clones com atividade apresentam um escurecimento no local do inóculo. 1. Clone C9-1; 2. Clone E1-5; 3. Clone F10-6; 4. Clone H4-8; 5. Clone D3-9; 6. Clone B2-34; 7. Clone A7-34; 8. Clone H10-51; 9. Clone D3-54; 10. Clone G2-56; 11. Clone C2-74; 12. Clone D8-76; 13. Clone C5-95; 14. Clone H7-102, 15. Vetor pCF 430 sem inserto (controle negativo). Circulado em vermelho estão os clones que tiveram a atividade continuada.
Figura 9 - Confirmação da atividade em bagaço de cana-de-açúcar dos clones de rúmen em ensaios em placas após a retransformação. Os clones com atividade apresentaram escurecimento no local do inóculo. 1. Clone C9-1; 2. Clone E1-5; 3. Clone F10-6; 4. Clone H4-8; 5. Clone D3-9; 6. Clone B2-34; 7. Clone A7-34; 8. Clone H10-51; 9. Clone D3-54; 10. Clone G2-56; 11. Clone C2-74; 12. Clone D8-76; 13. Clone C5-95; 14. Clone H7-102, 15. Vetor pCF 430 sem inserto (controle negativo). Circulado em vermelho estão os clones que tiveram a atividade continuada.
Fonte: próprio autor.
Alguns dos clones perderam a atividade após a retransformação como é possível verificar nas figuras 8 e 9. Os clones que tiveram a atividade continuada em xilana foram: C9-1, F10-6, D3-9, B2-34, A7-34, C2-74, D8-76, C5-95 e H7-102. Em cana-de-açúcar uma quantidade menor de clones permaneceu com atividade dentre eles: C9-1, B2-34 e D8-76. Existe a possibilidade que durante as várias etapas de seleção pelos quais os clones passaram, alguns deles realmente eram falsos positivos, ou alguns perderam a atividade após a retransformação. Apesar disso todos estes clones foram sequenciados.
Além dos 14 clones representados acima, os clones F1-7 e H4-51 tiveram o seu sequenciamento iniciado, mas foram descontinuados por serem iguais aos clones H4-8 e H10-51 respectivamente.
Tabela 5 - Tamanho estimado e sequenciado para cada clone selecionado.
Clone Tamanho estimado (pb) Tamanho sequenciado (pb)
B12-1 4300 -
C9-1 6000 5923
E1-5 4400 4151
F10-6 4150 4313
F1-7 6000 -
H4-8 6000 6423
D3-9 3000 3271
B2-34 10300 6318
A7-34 3400 4015
H4-51 4200 -
H10-51 3000 2956
D3-54 7300 7486
G2-56 5200 5379
B8-62 3000 -
C2-74 6000 5681
D8-76 5500 5363
C5-95 6300 6396
H7-102 4000 3608
Fonte: próprio autor. (-) clone não sequenciado.
6.1.4 Análise da sequência dos clones selecionados
Um total de 123 ORFs maiores que 300 pb identificadas para os 14 clones completamente sequenciados. Foram analisadas apenas as que apresentaram o valor de corte de e-value até 1e-4 (0,0183), desta forma 40,65% das ORFS as que possuíam valor acima
deste foram descartadas. Das 73 ORFs restantes (Apêndice A) apenas 5 ORFs foram inicialmente identificadas contendo possíveis enzimas relacionadas à degradação de polissacarídeos e foram analisadas em detalhes (Tabela 6).
Tabela 6 - Identificação das ORFs dos clones da biblioteca metagenômica de rúmen de caprino sequenciados que podem conter genes de importância relacionados à degradação de polissacarídeos.
Clone ORF Tamanho Descrição da ORF Identidade E-value
B2-34 1 1824 Β-glicuronidase [Prevotella sp. P6B4] 69% 0.0
B2-34 3 693 Hidrolase Alfa-Beta [Listeria weihenstephanensis FSL R9-0317]
50% 1e-70 C2-74 4 981 Proteína hipotética [Prevotella brevis] 52% 7 e-98 D3-9 1 1803 Proteína Hipotética [Prevotella sp. Sc00028] 59% 0.0 D8-76 1 2151 Glicosil Hidrolase da Família 31
[Prevotella sp. CAG:604]
70% 0.0
Os esquemas representando o sequenciamento dos clones serão mostrados somente para os clones que possuem ORFs que têm alguma relação com a degradação de polissacarídeos, entretanto, o esquema para todos os clones sequenciados foi montado e está disponível (Apêndice C). As ORFs presentes na tabela tiveram uma análise mais detalhada. Todas estas ORFs tiveram uma árvore filogenética montada e duas ORFs que possuíam o melhor e-value foram escolhidas para mostrar o alinhamento proteico.
6.1.4.1 Clone B2-34 6.1.4.1.1 ORF 1
O clone B2-34 possui duas ORFs (ORF 1 e ORF 3) as quais podem ter alguma relação com a atividade visualizada nos testes de atividade com o clone. A ORF 1 é potencialmente uma β-glicuronidase, pertencente a uma bactéria do gênero Prevotella sp. Este foi o gene sintetizado, pois o seu alinhamento proteico sugeriu que estava completo e apresentava algumas similaridades relacionadas ao sítio ativo de uma β-glicuronidase já curada de Escherichia coli. (Tabela 6) (Figuras 10 e 11).
Figura 10 - Figura 10: Esquema representativo do sequenciamento do clone B2-34. Análise das ORFs da sequência completa. As ORFS em cinza escuro são as possíveis ORFs responsáveis pela atividade do clone. As outras ORFs em cinza claro terão a sua informação apresentada em forma de tabela no apêndice A. A região em branco representa a região sequenciada.
Figura 11 - Dendrograma mostrando a relação da ORF 1 do clone B2-34 com ORFs similares encontradas em análises do programa Blastx. Alinhamento e construção realizados utilizando os programas Muscle e Mega 6. Dendrograma obtido pelo método Maximum Likelihood Tree. Os valores dos clados são referentes ao bootstrap de 1000 repetições.
Fonte: próprio autor.
Figura 12 - Alinhamento da ORF 1 do clone B2-34. Em branco com fundo verde (431aa) está o resíduo conservado glutamato (E) potencialmente parte do sítio ativo e as regiões com fundo azul são as regiões que apresentam a similaridade com a sequência alinhada (D5EVR4_PRER2 – Glicosil hidrolase da família 2 de Prevotella Ruminicola). O resíduo que possivelmente faz parte do sítio ativo foi baseado em alinhamento anterior com a sequência curada de Escherichia coli cuja nomenclatura é (BGLR_ECOLI). As regiões cujo aminoácidos estão apresentados com o fundo vermelho representam aminoácidos com propriedades negativas, enquanto as regiões cujo aminoácidos estão apresentados com o fundo verde representam aminoácidos com propriedades positivas.