UNIVERSIDADE DE SÃO PAULO
FACULDADE DE CIÊNCIAS FARMACÊUTICAS DE RIBEIRÃO PRETO
Estudo genético e molecular da disseminação da resistência aos
β
-lactâmicos em Pseudomonas aeruginosa
Renata Galetti
UNIVERSIDADE DE SÃO PAULO
FACULDADE DE CIÊNCIAS FARMACÊUTICAS DE RIBEIRÃO PRETO
Estudo genético e molecular da disseminação da resistência aos
β
-lactâmicos em Pseudomonas aeruginosa
Tese de Doutorado apresentada ao Programa de Pós-Graduação em Biociências Aplicadas à Farmácia para obtenção do Título de Doutor em Ciências
Área de Concentração: Biociências Aplicadas à Farmácia.
Orientada: Renata Galetti
Orientadora: Profa. Dra. Ana Lúcia da Costa Darini
AUTORIZO A REPRODUÇÃO E DIVULGAÇÃO TOTAL OU PARCIAL DESTE TRABALHO, POR QUALQUER MEIO CONVENCIONAL OU ELETRÔNICO, PARA FINS DE ESTUDO E PESQUISA, DESDE QUE CITADA A FONTE.
Galetti, Renata
Estudo genético e molecular da disseminação da resistência aos β-lactâmicos em Pseudomonas aeruginosa
60p. : il. ; 30cm.
Tese de Doutorado, apresentada à Faculdade de Ciências Farmacêuticas de Ribeirão Preto/USP – Área de concentração: Biociências Aplicadas à Farmácia.
Orientadora: Darini, Ana Lúcia da Costa.
FOLHA DE APROVAÇÃO
Renata Galetti
Estudo genético e molecular da disseminação da resistência aos β-lactâmicos em
Pseudomonas aeruginosa
Tese de Doutorado apresentada ao Programa de Pós-Graduação em Biociências Aplicadas à Farmácia para
obtenção do Título de Doutor em Ciências
Área de Concentração: Biociências Aplicadas à Farmácia.
Orientadora: Profª Drª Ana Lúcia da Costa Darini
Aprovado em:
Banca Examinadora
Prof. Dr. ____________________________________________________________
Instituição: ___________________________ Assinatura: _____________________
Prof. Dr. ____________________________________________________________
Instituição: ___________________________ Assinatura: _____________________
Prof. Dr. ____________________________________________________________
Instituição: ___________________________ Assinatura: _____________________
Prof. Dr. ____________________________________________________________
Instituição: ___________________________ Assinatura: _____________________
Prof. Dr. ____________________________________________________________
Dedico este trabalho à minha amada família, meus pais Marco e Angela,
meus irmãos Roberta e Rafael e meu namorado Vittor. Obrigada por todo amor
e carinho, pela força e companheirismo em todos os momentos. Sem vocês,
AGRADECIMENTOS
Agradeço a Deus, força maior, presente em todos os momentos da minha vida, por me guardar e iluminar, sempre.
Agradeço a minha mãe, Angela Montanini Galetti, por estar sempre ao meu lado, incondicionalmente. Ao meu pai, Marco Antonio Galetti, por acreditar e me proporcionar sempre o apoio necessário para tudo o que eu realizei; a vocês, meu amor e carinho.
Agradeço a minha irmã, Roberta Galetti, e meu irmão, Rafael Galetti, por estarem sempre presente em minha vida, a vocês minha eterna amizade e amor.
Agradeço a meu querido Vittor Ribeiro Brito, pela amizade, amor e companheirismo, pelo apoio e compreensão. Todo o tempo de espera valerá a pena!
Agradeço as minhas amigas Gisele Augusto Rodrigues de Oliveira e Natalícia de Jesus Antunes por dividir comigo a longa caminhada na pós-graduação. Obrigada por estarem sempre comigo.
Agradeço à minha orientadora, Profª Drª Ana Lúcia da Costa Darini, pela oportunidade concedida, ensinamentos diários e pela confiança em mim depositada. Minha admiração, respeito e enorme gratidão.
Aos amigos do LEBEM, Carolina P. C. Capizzani, Joseane Cristina Ferreira, Leonardo Neves de Andrade, Ludmilla Tonani, Natália Candido Caçador, Rubens Eduardo da Silva e Victor Hugo Pacagnelli Infante pela amizade, ótimos momentos, ajudas e sugestões. Aprendi com vocês que o trabalho em equipe é sempre prazeroso.
Agradeço a todos os pesquisadores que, gentilmente, cederam linhagens bacterianas utilizadas como controle neste trabalho.
Agradeço à Dra. Alessandra Carattoli pela oportunidade, supervisão e grande colaboração ao meu amadurecimento profissional durante estágio em seu laboratório. Agradeço também às pesquisadoras do Istituto Superiore di Sanità, Roma-Itália, Daniela Fortini, Laura Villa, Aurora Garcia-Fernandez e Claudia Feudi por todos os ensinamentos e por tornarem meus dias longe de casa mais curtos e prazerosos.
Agradeço à FAPESP pela bolsa de doutorado concedida (Processo nº 2010/51892-0).
i
RESUMO
GALETTI, R. Estudo genético e molecular da disseminação da resistência aos β -lactâmicos em Pseudomonas aeruginosa. 2014. 60f. Tese (Doutorado). Faculdade de Ciências Farmacêuticas de Ribeirão Preto – Universidade de São Paulo, Ribeirão Preto, 2014. A presença de plasmídeos conjugativos como IncP, IncU e IncFII carreando genes de resistência em Pseudomonas aeruginosa é de grande importância, pois podem ser trocados entre diferentes bactérias gram-negativas, disseminando a resistência aos antibióticos. Conhecer estes genes de resistência bem como os elementos genéticos que os carreiam é importante para entender os fatores que contribuem para a disseminação da resistência, auxiliando no controle da disseminação da resistência aos antibióticos. Ainda hoje não existe esquema para a tipagem de plasmídeos de P. aeruginosa, e são encontrados poucos trabalhos sobre estes plasmídeos. O objetivo deste estudo foi identificar os genes de resistência a antibióticos, o ambiente genético em que estes genes estão inseridos e a clonalidade dos isolados produtores de genes bla. No período do estudo, foram estudados 293 isolados de P. aeruginosa resistentes às cefalosporinas de 3ª e/ou 4ª gerações e/ou aos carbapenêmicos isoladas de pacientes de hospitais de Ribeirão Preto-SP, Belo Horizonte-MG, Franca-SP, Cuiabá-MT, de Barretos-SP e de Rio Branco-AC. Genes de resistência foram pesquisados por PCR. O perfil clonal dos isolados produtores de genes bla foi determinado por PFGE e MLST. A tipagem de plasmídeos foi feita por PFGE-S1 nuclease, hibridações com sondas específicas e tipagem de replicons (PBRT). Foram identificados 12 isolados carreando o gene
blaSPM-1, 16 isolados carreando o gene blaCTX-M-2 e 3 isolados carreando o gene blaKPC-2. Em
todos os 12 isolados produtores de SPM-1 foram identificadas duas cópias do elemento de inserção ISCR4, sendo uma cópia upstream e uma cópia downstream ao gene blaSPM-1
inseridos no cromossomo bacteriano. Em 13 dos 16 isolados produtores de CTX-M-2 o gene
blaCTX-M-2 foi encontrado associado ao elemento de inserção ISCR1 e em 3 ao elemento de
inserção ISEcp1 também inseridos no cromossomo bacteriano. Em 2 isolados o gene blaKPC-2
é carreado por um plasmídeo de ~3kb não tipável por PBRT e um em está inserido no cromossomo bacteriano. O ambiente genético do gene blaKPC-2 nos isolados estudados é
diferente daqueles encontrados na literatura. Os isolados produtores de genes bla citados apresentaram diversidade clonal, tanto por PFGE quanto MLST demonstrando que vários clones estão envolvidos na disseminação desses genes. Este trabalho identificou e caracterizou 31 isolados produtores de -lactamases, o ambiente genético destes genes e a clonalidade de isolados de várias cidades do Brasil e em períodos diferenciados, demonstrando a disseminação desses genes em diferentes hospitais brasileiros. Esses dados auxiliam no conhecimento dos fatores que estão envolvidos na disseminação da resistência aos antibióticos e podem auxiliar as CCIHs dos hospitais a definirem estratégias para controlar a disseminação desses microrganismos prevenindo surtos de bactérias multirresistentes.
ABSTRACT
GALETTI, R. Genetic and molecular study of β-lactams resistance dissemination in
Pseudomonas aeruginosa. 2014. 60f. Thesis (Doctoral). Faculdade de Ciências
Farmacêuticas de Ribeirão Preto – Universidade de São Paulo, Ribeirão Preto, 2014.
The presence of conjugative plasmids as IncP, IncU and Inc FII carrying resistance genes in
Pseudomonas aeruginosa is very important because t these plasmids can be shared among different bacteria, spreading antibiotic resistance. Knowledge of these genes as well as genetic elements carrying these genes it is important to understend the factors that contribute to the spread of resistance, helping to control the spread of antibiotic resistance. Today there is no plasmid typing scheme to P. aeruginosa and few papers are found about this subject. The purpose of this study was to investigate resistance genes, genetic environment of these genes and clonal relationship of the isolates carrying these resistance genes. In the period of this study was studied 293 P. aeruginosa resistant to third and fourth generations of cephalosporins and/or carbapenens isolated of patients from hosptital from Ribeirão Preto, Belo Horizonte-MG, Franca-SP, Cuiabá-MT, Barretos-SP and Rio Branco-AC. Resistance genes were investigated by PCR. Twelve isolates were identified carrying blaSPM-1 gene, 16 isolates carrying blaCTX-M-2 gene and 3 isolates carrying blaKPC-2 gene. Clonal profiles of
isolates producing resistance genes were investigated by PFGE and MLST. Plasmid typing was performed by PFGE-S1 nuclease, specific hybridizations and PCR replicon typing (PBRT). Two isolates presented a 3kb plasmid non-typeable by PBRT carrying blaKPC-2 gene. In all isolates SPM-1-producers were identified two copies of insertion sequence ISCR4, a copy upstream and a copy downstream to blaSPM-1 gene inserted in chromosomal DNA. In 13
of 16 isolates CTX-M-2-producers the blaCTX-M-2 gene was found associated to insertion sequence ISCR1 and in 3 isolates was associated to insertion sequence ISEcp1 also inserted in chromosomal DNA. Genetic environment of blaKPC-2 gene in the isolates studied it is different
from those found in the literature. Isolates producing bla genes are clonally diversified using both PFGE and MLST showing that various clones are responsible to spread these resistance genes. This work identified and characterized 31 P. aeruginosa- -lactamase-producing, the genetic environment of these genes and the clonal relationship of isolates collected from different periods from different cities of Brazil. These data can help us to understand the factors that are involved in the spread of antibiotics resistance and to help the Hospital Infection Control Committee to define strategies to control the spread of these microorganisms preventing outbreaks of resistant.
iii
RESUMEN
GALETTI, R. Estudio genético y molecular de la diseminación de resistencia a los β -lactámicos en Pseudomonas aeruginosa. 2014. 60f. Tesis (Doctorado). Facultad de Ciencias Farmacéuticas de Ribeirão Preto – Universidad de Sao Paulo, Ribeirão Preto, 2014.
La presencia de plásmidos conjugativos como IncP, IncU e IncFII carreando genes de resistencia en Pseudomonas aeruginosa es de grande importancia, una vez que pueden ser cambiados entre distintas bacterias, diseminando la resistencia a los antibióticos. Conocer a estos genes de resistencia, así como los elementos genéticos que los carrean es importante para entender los factores que contribuyen a la propagación de la resistencia, auxiliando en el control de la propagación de la resistencia a los antibióticosTodavía no existen esquemas para la tipificación de plásmidos de P.aeruginosa y se encuentran pocos trabajos sobre estos plásmidos. El periodo de ese estudio se estudiaron293 P.aeruginosa resistentes a las cefalosporinas de 3ª y/o 4ª generaciones y/o a los carbapenemicos aislados de pacientes de los hospitales de Ribeirão Preto-SP, Belo Horizonte-MG, Franca-SP, Cuiabá-MT, Barretos-SP y Rio Branco-AC. Los genes de resistencia fueron investigados por PCR. Fueron identificados 12 aislados carreando el gene blaSPM-1, 16 aislados carreando el gene blaCTX-M-2 y 3 aislados carreando el gene blaKPC-2. El perfil clonal de los aislados productores de -lactamases fue realizado por PEGE y MLST. La tipificación de plásmidos se hizo por PFGE-S1 nuclease, hibridaciones con sondas específicas y tipificación de replicones (PBRT). En 2 aislados el gene blaKPC-2 es carreado por un plásmido de ~3kb no tipable por PBRT. En todos los 12 aislados productores de SPM-1 fueron identificadas dos copias del elemento de inserción ISCR4, siendo una copia upstream al gene blaSPM-1 y una copia downstream al gene inseridos
al cromosoma bacteriano. En 13 de los 16 aislados productores de CTX-M-2 el gene
blaCTX-M-2 fue encontrado asociado al elemento de inserción ISCR1 y en 3 al elemento de inserción ISEcp1 también inseridos al cromosoma bacteriano. El ambiente genético del gene blaKPC-2 en
los aislados estudiados todavía no fue descrito en la literatura. Los aislados productores de genes bla presentaron diversidad clonal, tanto para PGFE cuanto MLST demostrando que varios clones son capaces y responsables por la diseminación de esos genes. Este trabajo identificó y caracterizó 31 aislados productores de -lactamases, así como el ambiente genético de esos genes y la clonalidad de aislados de varias ciudades de Brasil y en periodos diferenciados, demostrando la diseminación de esos genes en distintos hospitales brasileños. Esos datos pueden auxiliar la comprensión de los factores que están involucrados en la diseminación de la resistencia a los antibióticos y pueden ayudar a los hospitales a idear estrategias para controlar la propagación de estos microorganismos que impidan los brotes de bacterias resistentes.
LISTA DE FIGURAS
Figura 1 - Ambiente genético do gene blaSPM-1 nos isolados estudados... 22
Figura 2 - Ambiente genético do gene blaCTX-M-2 nos isolados estudados ... 23 Figura 3 - Dendrograma dos perfis de PFGE apresentado pelos isolados de P.
aeruginosa carreando genes bla, dado pelo coeficiente de Dice ge-rado pelo método UPGMA utilizando o programa BioNumerics 5.01 a partir do perfil de macrorrestrição do DNA genômico clivado com enzima SpeI apresentado na PFGE. Os perfis estão representados por letras maiúsculas do alfabeto... 25 Figura 4 - Filmes fotográficos demonstrando resultado de hibridação com sondas
v
LISTA DE TABELAS
Tabela 1 - Linhagens-controle utilizadas no estudo ... 11 Tabela 2 - Pares de primers utilizados para de genes de resistência e ambiente
genético... 16 Tabela 3 - Perfil de sensibilidade aos antibióticos e caracterização genética e
LISTA DE ABREVIATURAS, SÍMBOLOS E SIGLAS
µg Micrograma (s)
µl Microlitro (s)
°C Graus Centígrados
% Porcentagem
A Adenina
AIM Australia Imipenemase
ATCC American Type Culture Collection
ATM Aztreonam
Arg Arginina
Asp Aspartato
BHI Brain Heart Infusion
C Citosina
CAZ Ceftazidima
CCIH Comissão de Controle de Infecção Hospitalar CIM Concentração inibitória mínima
CLSI Clinical and Laboratory Standards Institute
cm Centímetros
CPM Cefepime
Cr Cromossomo
CTI Centro de Terapia Intensiva DDS Double Disc Synergism
DIM Dutch Imipenemase
DNA Ácido desoxirribonucléico dNTP Desoxinucleotídeos trifosfatados EDTA Ácido etileno diamino tetracético
ES EDTA sarcosil
Etest Epsilomer test
vii
FCFRP Faculdade de Ciências Farmacêuticas de Ribeirão Preto
G Guanina
GIM German imipenemase
Glu Glutamina
HCFMRP Hospital das Clínicas da Faculdade de Medicina de Ribeirão Preto Hyp Protein hipotética
IMP Imipenemase
IPM Imipenem
IS Insertion sequence
KHM Kyorin university Hospital Metallo-β-lactamase
LEBEM Laboratório Especial de Bacteriologia e Epidemiologia Molecular
M Molar (es)
mA Miliamper (es)
MBL Metalo-beta-lactamase
MER Meropenem
mg Miligrama (s)
MgCl2 Cloreto de magnésio
mL Mililitro (s)
mM Milimolar (es)
mm Milímetro (s)
MPA Ácido mercaptopropiônico NaCl Cloreto de sódio
NDM New Delhi Metallo-β-lactamase
ng Nanograma (s)
ORF Open reading frame
pb Pares de base
PBP Penicillin-binding proteins
PCR Polymerase chain reaction
PFGE Pulsed-field gel electrophoresis
PIV Tris NaCl
Pl Plasmídeo
pmol Picomol (es)
POL Polimixina
q.s.p. Quantidade suficiente para
s Segundo (s)
SADT Serviço de Apoio Diagnóstico e Tratamento
Ser Serina
SIM Seoul imipenemase
SPM São Paulo Metalo- -lactamase
T Timina
Taq Thermus aquaticus
TBE Tris borato EDTA
TE Tris, EDTA e água
U Unidade
UETDI Unidade de Emergência e Tratamento de Doenças Infecciosas UPGMA Unweighted Pair Group Method with Arithmetic Mean
UTI Unidade de Terapia Intensiva USP Universidade de São Paulo
V Volts
VIM Verona imipenemase
X Vezes
Zn++ Íon zinco
SUMÁRIO
RESUMO...i
ABSTRACT ...ii
RESUMEN ...iii
LISTA DE FIGURAS...iv
LISTA DE TABELAS ... v
LISTA DE ABREVIATURAS, SÍMBOLOS E SIGLAS ...vi
1 INTRODUÇÃO ... 1
1.1 Mecanismos de resistência aos antibióticos em Pseudomonasaeruginosa... 2
1.2 -lactamases ... 2
1.3 Plasmídeos ... 4
1.4 Fatores de virulência em P. aeruginosa... 5
2 OBJETIVOS ... 7
2.1 Objetivo geral ... 8
2.2 Objetivos específicos... 8
3 MATERIAIS E MÉTODOS ... 9
3.1 Isolados bacterianos... 10
3.2 Identificação bacteriana e armazenamento das bactérias ... 12
3.3 Teste de sensibilidade aos antibióticos... 12
3.4 Tipagem molecular dos isolados ... 12
3.4.1 Eletroforese em Campo Pulsado ... 12
3.4.2 Tipagem por seqüenciamento de multilocus... 14
3.5 Detecção molecular de genes codificadores de -lactamases ... 14
3.5.1 Extração de DNA genômico bacteriano ... 14
3.5.2 Amplificação de genes de resistência... 15
3.6 Sequenciamento... 17
3.7 Análise de plasmídeos ... 18
3.7.1 Perfil plasmideal ... 18
3.7.2 Localização de genes de resistência ... 18
3.7.2.2 Preparo da sonda genética ... 19
3.7.2.3 Reação de hibridização... 19
3.7.3 Tipagem dos plasmídeos por grupos de incompatibilidade... 20
3.7.4 Caracterização do ambiente genético dos genes de resistência ... 20
3.8 Pesquisa de genes de fatores de virulência... 20
4 RESULTADOS ... 21
4.1 Isolados bacterianos e Concentração Inibitória Mínima ... 22
4.2 Genes codificadores de -lactamases ... 22
4.3 Investigação do ambiente genético... 22
4.4 Clonalidade dos isolados de P. aeruginosa... 23
4.4.1 Pulsotipos ... 23
4.4.2 Tipo de Sequência gerada por MLST... 25
4.5 Pesquisa e caracterização de plasmídeos... 27
4.6 Pesquisa de genes codificadores de fatores de virulência ... 28
5 DISCUSSÃO ... 30
6 CONCLUSÕES... 37
7 REFERÊNCIAS ... 39
APÊNDICE ... 48
1.1 Mecanismos de resistência aos antibióticos em Pseudomonasaeruginosa
O gênero Pseudomonas sp. compreende grande número de espécies, sendo 25% dos microrganismos relacionados com infecções humanas. Na realidade, 70 a 80% dos bacilos gram-negativos não fermentadores isolados de espécimes clínicos humanos correspondem à espécie Pseudomonas aeruginosa, que está entre a maioria dos microrganismos resistentes aos antibióticos responsáveis por infecções hospitalares (HILL; HENRY; SPEERT, 2007). Geralmente, o tratamento dessas infecções é difícil devido à resistência natural desta espécie aos antimicrobianos, bem como à sua habilidade em adquirir mecanismos de resistência adicionais a múltiplas classes de antibióticos.
P. aeruginosa é intrinsecamente resistente a agentes antimicrobianos não relacionados entre si devido a vários fatores, como: produção de enzimas que degradam ou inativam o antibiótico (como a ocorrência natural de cefalosporinases), alteração da permeabilidade da membrana externa, expressão constitutiva de várias bombas de efluxo de antibióticos e alteração do sítio alvo do antibiótico (BONOMO; TOLMASKY, 2007).
Embora todos esses mecanismos de resistência sejam frequentemente encontrados em
P. aeruginosa, notadamente, o mecanismo de resistência a antibióticos mais importante e que causa mais preocupação em P. aeruginosa envolve a produção de -lactamases (HAWKEY; JONES, 2009).
1.2 β-lactamases
As enzimas -lactamases agem catalisando a hidrólise do anel β-lactâmico, inativando, assim, a ação de vários antibióticos pertencentes a esse grupo (BUSH, 2010).
Vários esquemas foram propostos para a classificação das -lactamases, porém as duas classificações mais utilizadas atualmente são as de Ambler e Bush-Jacoby-Medeiros. Este último esquema de classificação foi recentemente revisado e atualizado por Bush e Jacob (BUSH; JACOBY, 2010).
Genes codificadores de -lactamases podem ser mobilizados por vários elementos genéticos, como elementos de inserção (IS), integrons (In) ou transposons (Tn), que podem ou não, estar inseridos em plasmídeos conjugativos (HAWKEY; JONES, 2009). As principais -lactamases encontradas em P. aeruginosa são as ESBLs e as carbapenemases.
Introdução | 3
Com a emergência das ESBLs houve uso abusivo de carbapenêmicos, não surpreendentemente, culminando na emergência de carbapenemases (GUPTA et al., 2011; WALSH, 2010). As carbapenemases constituem o grupo mais versátil de β-lactamases, com espectro de atividade mais amplo que as ESBLs. Embora conhecidas como carbapenemases esse grupo de enzimas hidrolisa praticamente todos os antibióticos β-lactâmicos, com destaque para a classe dos carbapenêmicos (QUEENAN; BUSH, 2007).
As carbapenemases são divididas em dois grandes grupos, as metalo-beta-lactamases (MBL) a as serina-carbapenemases (CROWDER et al., 1998; QUEENAN; BUSH, 2007).
Atualmente são conhecidas nove subclasses de MBL adquiridas: IMP, VIM, SPM, GIM, SIM, AIM, KHM, NDM-1 e DIM-1 (POIREL et al., 2009; QUEENAN; BUSH, 2007; SEKIGUCHI et al., 2008; YONG et al., 2009)., porém somente IMP, VIM, SPM, NDM, AIM e GIM foram identificadas em P. aeruginosa (CORNAGLIA; GIAMARELLOU; ROSSOLINI, 2011).
As serina-carbapenemases mais identificadas e disseminadas são: OXA, GES e KPC. As MBL mais relatadas e disseminadas mundialmente são IMP e VIM. SPM é de destaque no Brasil, uma vez que, por motivos ainda não muito bem conhecidos, essa enzima é quase restrita (endêmica) ao território brasileiro, detectada frequentemente em P. aeruginosa
(QUEENAN; BUSH, 2007). Apenas um relato dessa enzima ocorreu na Europa, após o regresso de um turista suíço que visitou o Brasil, não ocorrendo disseminação (SALABI et al., 2010). As MBL adquiridas são codificadas por genes contidos em cassetes gênicos localizados no cromossomo ou plasmídeo bacteriano. No entanto, blaSPM-1 não é encontrado
como cassete gênico, e sim associado à uma sequência de inserção (ISCR4). Este gene foi encontrado em cromossomo e também em plasmídeo grande em diferentes linhagens de P. aeruginosa (TOLEMAN; BENNETT; WALSH, 2006).
NDM, a mais recente MBL relatada, se disseminou rapidamente da Índia para a Inglaterra, outros países da Europa, Ásia, Estados Unidos e Canadá e mais recentemente em enterobactérias no Brasil (CARVALHO-ASSEF et al., 2013).
Em P. aeruginosa as carbapenemases mais disseminadas são PER, GES e KPC. Assim como ocorrido com as enzimas ESBL CTX-M (CANTON; COQUE, 2006)(CANTÓN; COQUE, 2006), que apresentaram rápida disseminação pandêmica, o mesmo está acontecendo com a enzima KPC, pois os genes blaKPC possuem o suporte genético necessário para a transferência horizontal e rápida disseminação entre diferentes gêneros e espécies de bactérias. Esse gene está mundialmente disseminado em bactérias da família
Brasil (ANDRADE et al., 2011),e em 2006 foi relatado pela primeira vez em P. aeruginosa
na Colômbia (VILLEGAS et al., 2007). Jacome et al., (2012) fez o primeiro relato de blaKPC
em P. aeruginosa no Brasil, mostrando o potencial de disseminação desta enzima por diferentes gêneros e espécies bacterianas .
1.3 Plasmídeos
Como elementos genéticos móveis mais importantes, os plasmídeos possuem genes essenciais para a iniciação e controle de replicação e genes acessórios que podem ser úteis para as células hospedeiras como genes de virulência para a produção de bacteriocinas e genes que conferem resistência a antibióticos (CARATTOLI, 2011).
A classificação dos plasmídeos pode ser realizada baseando-se em diferentes critérios: número de cópias na célula, tamanho do plasmídeo, faixa de bactérias hospedeiras, grupo de incompatibilidade e capacidade de transferências entre as células (CARATTOLI, 2011).
A capacidade que um plasmídeo possui de se transferir de uma célula bacteriana para outra permite diferenciá-lo em: conjugativo, mobilizável e não-conjugativo. Os plasmídeos conjugativos são assim denominados porque possuem a capacidade intrínseca de se transferir de uma célula para outra (apresentam o sistema tra). Os mobilizáveis não possuem o sistema
tra, responsável pela capacidade de conjugação, mas possuem o sistema mob que lhes propiciam a capacidade de transferirem-se à outra célula na dependência de um plasmídeo conjugativo. Já os não-conjugativos são carentes das características de conjugação e mobilização (FROST et al., 2005).
A classificação mais utilizada é baseada na propriedade universal dos plasmídeos, que é a replicação. Esta classificação utiliza o princípio que plasmídeos com mesmo controle de replicação não podem estar presentes em uma mesma célula, o que foi chamado de "incompatibilidade de plasmídeos" (DATTA; HEDGES, 1971). Com base nessa informação, plasmídeos pertencentes ao mesmo grupo de incompatibilidade (Inc) não podem ser propagados em uma mesma linhagem celular (DATTA; HUGHES, 1983). Sendo assim, plasmídeos que possuem o mesmo controle de replicação são considerados “incompatíveis” enquanto que plasmídeos com diferente controle de replicação são “compatíveis” (COUTURIER et al., 1988; DATTA; HUGHES, 1983).
Carattoli et al;. (2005) propuseram a detecção de plasmídeos baseada em amplificação de segmentos específicos do DNA plasmideal (pDNA), para pesquisa de replicons
Introdução | 5
realização de 5 PCRs em multiplex e 3 PCRs simples, reconhecendo os replicons FIA, FIB, FIC, HI1, HI2, I1-I , L/M, N, P, W, T, A/C, K, B/O, X, Y, FrepB e FIIA, responsáveis pelos
principais grupos Inc de plasmídeos circulando entre enterobactérias (CARATTOLI, 2009). Alguns plasmídeos, pertencentes aos grupos acima citados, possuem a capacidade de se replicar tanto em enterobactérias quanto em P. aruginosa, mas a maioria dos plasmídeos que circulam em P. aruginosa é diferente daqueles que normalmente circulam entre enterobactérias, sendo que até o momento não existe um esquema de tipagem para esses plasmídeos, como acontece para enterobactérias e Acinetobacter spp. (BERTINI et al., 2010; CARATTOLI et al., 2005).
Plasmídeos IncN têm sido encontrados em amostras ambientais. Este fato alerta para a possibilidade da disseminação de espécies produtoras de enzimas CTX-M que não façam parte da família Enterobacteriaceae e que possam ser responsáveis pela presença de genes
blaCTX-M em P. aeruginosa e Acinetobcter sp. (CANTON; COQUE, 2006).
Atualmente, crescentes relatos relacionaram a disseminação de genes de resistência por plasmídeos, de diferentes tamanhos e origens, em bactérias negativas e gram-positivas, no hospital, na comunidade e em outros ambientes, por todo o mundo (CARATTOLI, 2011; JENSEN et al., 2010).
Plasmídeos pertencentes aos grupos IncP, IncW, IncN e IncQ podem ser transferidos e são capazes de se manter estáveis tanto em enterobactérias quanto em Pseudomonas spp. Em particular, plasmídeos IncP são amplamente distribuídos em bactérias gram-negativas como Escherichia coli, Pseudomonas spp., Enterobacter aerogenes e Sphingomonas spp. (POPOWSKA; KRAWCZYK-BALSKA, 2013). Plasmídeos pertencentes ao grupo IncW também podem ser encontrados em uma grande variedade de espécies bacterianas, incluindo gêneros Escherichia, Salmonella, Shigella e Pseudomonas sendo, por este motivo, considerados “broad host range”(FERNANDEZ-LOPEZ et al., 2006).
Almeida et al. (2012) encontraram IncFI carreando gene blaKPC-2 em Pseudomonas putida no Brasil. Em 2013, na Colômbia, Naas e colaboradores identificaram plasmídeos IncP6 (pCOL-1) e IncU (pPA-2) carreando o gene blaKPC em P. aeruginosa (NAAS et al., 2013), demonstrando o papel de plasmídeos pertencentes a grupos Inc na disseminação de genes de resistência tanto em enterobactérias como também em P. aeruginosa.
1.4 Fatores de virulência em P. aeruginosa
(FINNAN et al., 2004). Em pacientes com fibrose cística é o patógeno líder em morbidade e mortalidade. A investigação de fatores de virulência em P. aeruginosa é muito comum nesse grupo de pacientes, porém em P. aeruginosa resistentes a antibióticos, essa informação é escassa. Os principais fatores de virulência pesquisados são alginato (algD) citotoxina (exoS e
exoY), protease (apr) e fímbria (pilA), além de dois sistemas quorum sensing (QS) las e rhl
2.1 Objetivo geral
Estudar plasmídeos carreadores de genes bla em Pseudomonas aeruginosa de amostras clínicas isoladas em hospitais de diferentes regiões brasileiras e investigar possível presença desses plasmídeos em clones epidêmicos
2.2 Objetivos específicos
• Investigar a diversidade de genes codificadores de -lactamases presentes em P. aeruginosa estudadas;
• Detectar a presença de possíveis mutações, descritas ou não, nos genes encontrados;
• Identificar o ambiente genético em que estes genes estão inseridos e os elementos que os carreiam como integrons (In) ou transposons (Tn) e elementos de inserção (IS);
• Tipar o plasmídeo que carreia os genes bla de acordo com os grupos de incompatibilidade descritos para enterobactérias;
• Avaliar a clonalidade por eletroforese em campo pulsado entre P. aeruginosa que apresentarem genes de resistência;
• Avaliar a relação clonal entre P. aeruginosa que apresentarem genes de resistência por tipagem por sequenciamento de multilocus (MLST);
• Avaliar a relação de plasmídeos com genes de resistência encontrados e comparar os resultados com os dados conhecidos na literatura;
3.1 Isolados bacterianos
Foram enviadas ao Laboratório Especial de Bacteriologia e Epidemiologia Molecular (LEBEM), P. aeruginosa isoladas de diferentes hospitais isoladas de pacientes não repetidos e resistentes a cefalosporinas de 3ª e 4ª gerações e/ou carbapenêmicos. O envio, número de isolados e o período em que foram coletados foram de acordo com adisponibilidade de cada hospital/laboratório. Foram enviadas P. aeruginosa isoladas no Hospital das Clínicas da Faculdade de Medicina de Ribeirão Preto (HCFMRP-USP) (n=158 no período de abril a agosto de 2007 e 1° semestre de 2013), em hospitais de Belo Horizonte-MG (n=09 no período de março a julho de 2011), Franca-SP (n=07 no período de setembro a dezembro de 2010) e Barretos-SP (n=65 durante o ano de 2013)1, no Hospital Universitário Julio Muller de Cuiabá-MT (n=50 no período de janeiro/2011 a abril/2012) e Laboratório Bionorte, Rio Branco-AC (n=04 no período de novembro/2012 a fevereiro/2013), totalizando 293 isolados.
P.aeruginosa isoladas no HCFMRP-USP no período de 2007 foram enviadas ao laboratório no começo do estudo, os isolados do hospital de Belo Horizonte e do hospital de Franca foram enviados ao laboratório em meados de 2012 e os isolados de Cuiabá, Rio Branco, Barretos e HCFMRP-USP (2013) foram enviados ao LEBEM em janeiro e fevereiro de 2014.
As bactérias isoladas foram coletadas de placas de cultivo de isolamento primário de exames bacteriológicos, realizados mediante solicitações de rotina, no Laboratório de Microbiologia dos respectivos hospitais. Cada bactéria possui um grupo de dados catalogados fornecidos pelos hospitais constando: número de identificação LEBEM, data de entrada, sigla dos pacientes, clínica, gênero, cidade e Estado de isolamento, amostra clínica e perfil de resistência a antibióticos que estão apresentados no Anexo 1.
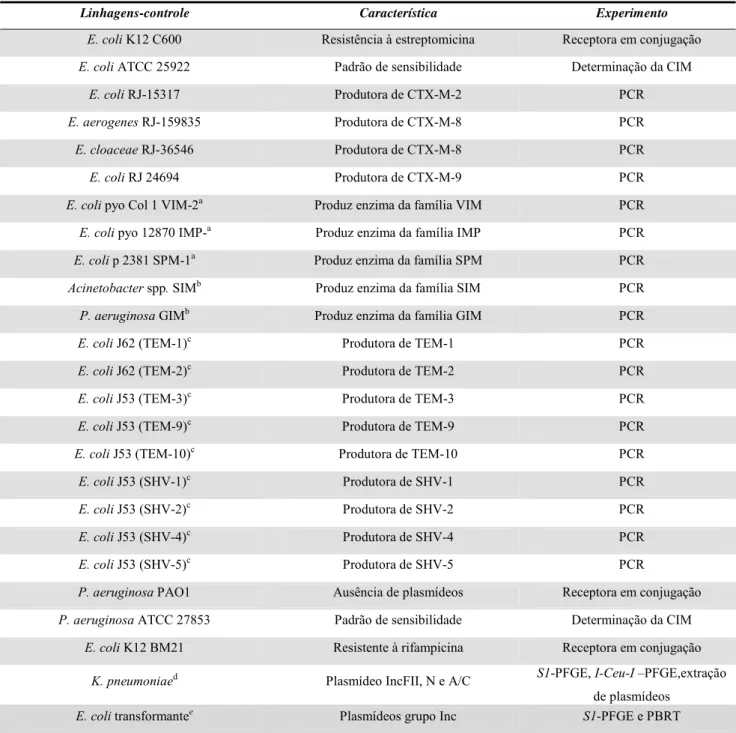
As linhagens-controle, suas características e o experimento em que estão sendo utilizadas estão demonstrados na Tabela 1.
1
Materiais e Métodos | 11
Tabela 1. Linhagens-controle utilizadas no estudo
Linhagens-controle Característica Experimento
E. coli K12 C600 Resistência à estreptomicina Receptora em conjugação
E. coli ATCC 25922 Padrão de sensibilidade Determinação da CIM
E. coli RJ-15317 Produtora de CTX-M-2 PCR
E. aerogenes RJ-159835 Produtora de CTX-M-8 PCR
E. cloaceae RJ-36546 Produtora de CTX-M-8 PCR
E. coli RJ 24694 Produtora de CTX-M-9 PCR
E. coli pyo Col 1 VIM-2a Produz enzima da família VIM PCR
E. coli pyo 12870 IMP-a Produz enzima da família IMP PCR
E. coli p 2381 SPM-1a Produz enzima da família SPM PCR
Acinetobacter spp. SIMb Produz enzima da família SIM PCR
P. aeruginosa GIMb Produz enzima da família GIM PCR
E. coli J62 (TEM-1)c Produtora de TEM-1 PCR
E. coli J62 (TEM-2)c Produtora de TEM-2 PCR
E. coli J53 (TEM-3)c Produtora de TEM-3 PCR
E. coli J53 (TEM-9)c Produtora de TEM-9 PCR
E. coli J53 (TEM-10)c Produtora de TEM-10 PCR
E. coli J53 (SHV-1)c Produtora de SHV-1 PCR
E. coli J53 (SHV-2)c Produtora de SHV-2 PCR
E. coli J53 (SHV-4)c Produtora de SHV-4 PCR
E. coli J53 (SHV-5)c Produtora de SHV-5 PCR
P. aeruginosa PAO1 Ausência de plasmídeos Receptora em conjugação
P. aeruginosa ATCC 27853 Padrão de sensibilidade Determinação da CIM
E. coli K12 BM21 Resistente à rifampicina Receptora em conjugação
K. pneumoniaed Plasmídeo IncFII, N e A/C S1-PFGE, I-Ceu-I –PFGE,extração de plasmídeos
E. coli transformantee Plasmídeos grupo Inc S1-PFGE e PBRT
a
Linhagem gentilmente cedida pelo Professor Dr. Laurent Poirel, Hospital de Bicêtre, Le Kremlin-Bicêtre, Paris, França.
b
Linhagem gentilmente cedida pela Professora Dr. Ana Cristina Gales, Laboratório ALERTA, Universidade Federal de São Paulo, São Paulo, Brasil.
c
Linhagens gentilmente cedidas pelo Professor Drº David Livermore, Antibiotic Resistance Monitoring and Reference Laboratory, Londres, Inglaterra.
d
Andrade et al, 2011 e
3.2 Identificação bacteriana e armazenamento das bactérias
P. aeruginosa isoladas no HCFMRP-USP, no hospital de Belo Horizonte-MG e no Hospital do Câncer de Barretos foram identificadas por provas fenotípicas convencionais e/ou por automação utilizando o aparelho Vitek®1 ou Vitek®2, no Laboratório de Microbiologia de cada hospital. P. aeruginosa isoladas no hospital de Franca-SP foram identificadas utilizando o sistema API (bioMérieux) para bacilos gram-negativos não fermentadores e as isoladas no Laboratório Bionorte-AC foram identificadas por provas fenotípicas convencionais (HILL et al., 2007).
Para confirmação da pureza e identificação da espécie, todos os isolados recebidos foram cultivados em ágar MacConkey (Oxoid, Inglaterra). Em seguida, foram cultivados em ágar Müller-Hinton (Oxoid, Inglaterra) por 18 a 24 horas a 37ºC, sendo, então, transferidas para caldo BHI (Brain Heart Infusion - Oxoid, Inglaterra) com glicerol a 15%, e mantidas constantemente a -80ºC. Para reativação gradativa do metabolismo bacteriano, confirmação da pureza e viabilidade das colônias, alíquotas das culturas estocadas foram cultivadas novamente em BHI, incubadas a 37ºC, durante 18 – 24 horas e depois transferidas para ágar MacConkey (Oxoid, Inglaterra) antes de serem submetidas aos experimentos.
3.3 Teste de sensibilidade aos antibióticos
O teste de sensibilidade aos antibióticos foi realizado por disco difusão de acordo com o padronizado pelo (CLSI, 2011)CLSI 2010 e/ou por automação utilizando o aparelho Vitek®1 ou Vitek®2, no Laboratório de Microbiologia de cada hospital. A determinação da concentração inibitória mínima (CIM) dos antimicrobianos foi realizada para os isolados que apresentaram genes de resistência, utilizando fitas de Etest® seguindo orientação do fabricante, para os seguintes antibióticos -lactâmicos: cefepime (CPM), ceftazidima (CAZ), imipenem (IMP), meropenem (MER), aztreonam (ATM) e polimixina B (POL). A interpretação dos valores de CIM obtidos foi baseada nas padronizações do CLSI (2013).
3.4 Tipagem molecular dos isolados
3.4.1 Eletroforese em Campo Pulsado
Materiais e Métodos | 13
centrifugada por 2 minutos a 15.000 g. As células bacterianas foram lavadas com 500µL da solução PIV (10mM Tris, pH 8,0, 1,0M NaCl), centrifugadas e o sobrenadante descartado. O precipitado foi suspenso em 200 µL de solução de suspensão (PIV) sendo 150 µL transferidos para um novo tubo, colocados em banho de água a 48ºC, durante 5 a 10 minutos. Em seguida foram adicionados 150 µL de agarose (1,5% Agarose Ultra Pure, Gbco BRL) para a confecção dos blocos de agarose.
Os blocos de agarose foram colocados em tubos contendo 1mL de solução de lise EC (6 mM Tris, pH 8,0, 1 M NaCl, 100 mM EDTA pH 8,0; 0,2% deoxycolato de sódio, 0,5%, 0,5% N-laurylsarcosine), acrescentando-se RNAse (Invitrogen) à concentração final de 20 µg/mL e incubadas durante 4 a 5 horas a 37º C. Em seguida a solução de lise foi retirada, e foi adicionando 1mL de solução tamponante ES (EDTA, pH9,0; sarcosil 1%) acrescida de 1 mg/mL de proteinase K e os tubos foram incubados a 50º C durante 18 a 24 horas. Os blocos foram lavados 5 vezes com 13mL da solução tamponante TE 1X (10 mM Tris, pH 7,5; 1 mM EDTA, pH8,0) com intervalo de 30 minutos para cada lavagem. Os blocos foram mantidos em solução tamponante TE, a 4º C, até o momento do uso.
Para a fragmentação do DNA, cada bloco foi colocado em 100 µL de solução tamponante 1X concentrada da enzima SpeI e mantida a 37º C por 30 minutos. Essa solução foi desprezada e uma nova solução, preparada no momento do uso, acrescida de 27 U de enzima de restrição SpeI (Fermentas) foi adicionada. A reação foi incubada em banho de água a 37º C por 18 a 20 horas.
Para a corrida eletroforética, foram preparados 150 mL de gel de agarose 1% (Agarose Ultra Pure, Gibco BRL) em TBE 0,5X (0,89 M Tris; 0,89 M ácido bórico; 0,25 M EDTA; pH 8,0). A eletroforese foi realizada em TBE 0,5X à temperatura de 14º C, voltagem de 6 V por e pulsos de 5-40 por 24 horas no aparelho CHEF DRIII (BioRad).
Após a eletroforese, o gel foi corado com brometo de etídeo a 1µg/mL, por 30 minutos, e descorado em água por mais 30 minutos, sendo, em seguida, observado e fotografado utilizando o sistema AlphaImager® (Alpha Innotech).
3.4.2 Tipagem por seqüenciamento de multilocus
Para P. aeruginosa produtoras de genes bla foi realizada a tipagem molecular por seqüenciamento de multilocus (MLST), segundo o esquema proposto por Curran et al. (2004) Este método é baseado na amplificação e sequenciamento de fragmentos internos de 7 genes
housekeeping de P. aeruginosa: acsA (Acetyl coenzyme A synthetase), aroE (Shikimate dehydrogenase), guaA (GMP synthase), mutL (DNA mismatch repair protein), nuoD (NADH dehydrogenase I chain C, D), ppsA (Phosphoenolpyruvate synthase) e trpE (Anthralite synthetase component I).
A metodologia foi realizada segundo as condições descritas no website
<http://pubmlst.org/paeruginosa/info/primers.shtml>. Para os genes que não foram amplificados utilizando o protocolo cedido pelo website, por indicação do curador do banco de dados Elli Pinock, foi utilizado o protocolo descrito por -Mansfeld e colaboradores (VAN MANSFELD et al., 2009).
Os alelos e STs (sequence typing) obtidos em cada amostra estudada foram comparados com os alelos e STs de todas as linhagens disponibilizadas no banco de dados de MLST para P. aeruginosa.
3.5 Detecção molecular de genes codificadores de β-lactamases
3.5.1 Extração de DNA genômico bacteriano
Materiais e Métodos | 15
horas e depois foram centrifugados durante 15 minutos, a 4 ºC. O sobrenadante foi desprezado e o sedimento foi seco em Speed-Vac (Eppendorf) à temperatura ambiente. Em seguida, o sedimento foi suspenso em água W-3500 (Sigma) adicionando-se RNase (Invitrogen) e deixado no banho de água, durante 1 hora, a 37 ºC.
A quantificação e avaliação da pureza do DNA foram analisadas em espectrofotômetro de luz ultravioleta com comprimentos de onda (λ) de 260nm e 280nm utilizando o aparelho NanoDrop2000 (Thermo Scientific), sendo a concentração do DNA fornecida em µg/mL. O DNA foi diluído em água W-3500 (Sigma) à concentração de 30ng/µL e armazenado a -20 ºC.
3.5.2 Amplificação de genes de resistência
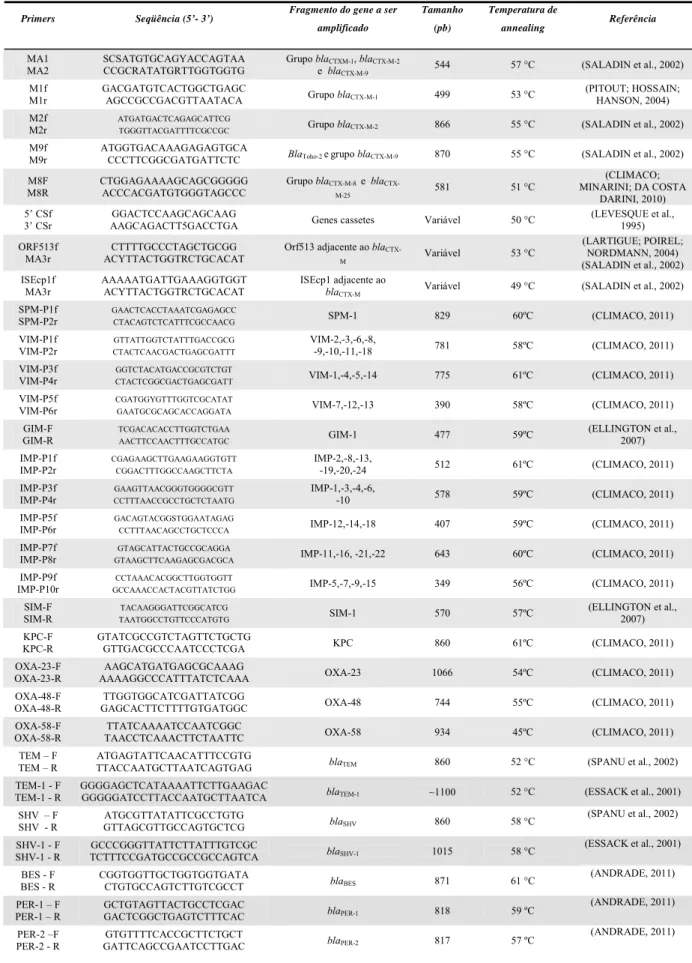
A detecção dos genes codificadores de resistência aos -lactâmicos: ESBL (CTX-M, TEM, SHV, BES, PER, VEB, GES e OXA), carbapenemases (SPM, IMP, VIM, GIM, SIM, KPC e OXA), ampC plasmideal (CMY) e às quinolonas (qnr e qep-A) foi feita por PCR. Foram utilizados pares de primers específicos, conforme exposto na Tabela 2.
A PCR foi realizada utilizando os seguintes componentes para o volume de reação de 50µL: produto de extração de DNA 60ng, tampão para PCR 1X (Plantinum ® taq DNA Polymerase High Fidelity (Invitrogen – Life Technologies), dNTP (mistura dos 4 nucleotídeos) 0,2mM (Eppendorf), primer mix 25pmol, taq-DNA polimerase 0,625 U (Plantinum ® taq DNA Polymerase High Fidelity [Invitrogen – Life Technologies]), MgCl2
2mM, e água para PCR (W-3500, Sigma)
O DNA foi inicialmente desnaturado, por aquecimento, a 95ºC, por 5 minutos. Em seguida a reação foi submetida a 30 ciclos térmicos das etapas de desnaturação, annealing e extensão, nas seguintes condições: Desnaturação – 1 minuto a 95 ºC; Annealing – 1 minuto à temperatura de annealing específica de cada par de primers (Tabela 2) e extensão – 1 minuto a 72 ºC. Após os 30 ciclos, foi procedida uma etapa de extensão final de 10 minutos a 72 ºC. O termociclador utilizado para os experimentos foi o Mastercycler Gradiente (Eppendorf). Os produtos de PCR foram analisados em gel de agarose 1%. Depois da eletroforese completa, o gel foi corado com brometo de etídeo (1µg/mL) e observado sob a luz ultravioleta sendo então fotografado utilizando o sistema de fotodocumentação AlphaImager (Alpha Innotech, EUA).
Tabela 2. Pares de primers utilizados para genes de resistência e ambiente genético
Primers Seqüência (5’- 3’) Fragmento do gene a ser
amplificado
Tamanho
(pb)
Temperatura de
annealing Referência
MA1 MA2
SCSATGTGCAGYACCAGTAA CCGCRATATGRTTGGTGGTG
Grupo blaCTXM-1, blaCTX-M-2
e blaCTX-M-9 544 57 °C (SALADIN et al., 2002)
M1f M1r
GACGATGTCACTGGCTGAGC
AGCCGCCGACGTTAATACA Grupo blaCTX-M-1 499 53 °C
(PITOUT; HOSSAIN; HANSON, 2004) M2f
M2r
ATGATGACTCAGAGCATTCG
TGGGTTACGATTTTCGCCGC Grupo blaCTX-M-2 866 55 °C (SALADIN et al., 2002)
M9f M9r
ATGGTGACAAAGAGAGTGCA
CCCTTCGGCGATGATTCTC BlaToho-2 e grupo blaCTX-M-9 870 55 °C (SALADIN et al., 2002) M8F
M8R
CTGGAGAAAAGCAGCGGGGG ACCCACGATGTGGGTAGCCC
Grupo blaCTX-M-8 e bla
CTX-M-25 581 51 °C
(CLIMACO; MINARINI; DA COSTA
DARINI, 2010) 5’ CSf
3’ CSr
GGACTCCAAGCAGCAAG
AAGCAGACTT5GACCTGA Genes cassetes Variável 50 °C
(LEVESQUE et al., 1995) ORF513f
MA3r
CTTTTGCCCTAGCTGCGG ACYTTACTGGTRCTGCACAT
Orf513 adjacente ao bla
CTX-M Variável 53 °C
(LARTIGUE; POIREL; NORDMANN, 2004) (SALADIN et al., 2002) ISEcp1f
MA3r
AAAAATGATTGAAAGGTGGT ACYTTACTGGTRCTGCACAT
ISEcp1 adjacente ao
blaCTX-M Variável 49 °C (SALADIN et al., 2002) SPM-P1f
SPM-P2r
GAACTCACCTAAATCGAGAGCC
CTACAGTCTCATTTCGCCAACG SPM-1 829 60ºC (CLIMACO, 2011)
VIM-P1f VIM-P2r
GTTATTGGTCTATTTGACCGCG CTACTCAACGACTGAGCGATTT
VIM-2,-3,-6,-8,
-9,-10,-11,-18 781 58ºC (CLIMACO, 2011) VIM-P3f
VIM-P4r
GGTCTACATGACCGCGTCTGT
CTACTCGGCGACTGAGCGATT VIM-1,-4,-5,-14 775 61ºC (CLIMACO, 2011)
VIM-P5f VIM-P6r
CGATGGYGTTTGGTCGCATAT
GAATGCGCAGCACCAGGATA VIM-7,-12,-13 390 58ºC (CLIMACO, 2011)
GIM-F GIM-R
TCGACACACCTTGGTCTGAA
AACTTCCAACTTTGCCATGC GIM-1 477 59ºC
(ELLINGTON et al., 2007) IMP-P1f IMP-P2r CGAGAAGCTTGAAGAAGGTGTT CGGACTTTGGCCAAGCTTCTA IMP-2,-8,-13,
-19,-20,-24 512 61ºC (CLIMACO, 2011) IMP-P3f
IMP-P4r
GAAGTTAACGGGTGGGGCGTT CCTTTAACCGCCTGCTCTAATG
IMP-1,-3,-4,-6,
-10 578 59ºC (CLIMACO, 2011) IMP-P5f
IMP-P6r
GACAGTACGGSTGGAATAGAG
CCTTTAACAGCCTGCTCCCA IMP-12,-14,-18 407 59ºC (CLIMACO, 2011)
IMP-P7f IMP-P8r
GTAGCATTACTGCCGCAGGA
GTAAGCTTCAAGAGCGACGCA IMP-11,-16, -21,-22 643 60ºC (CLIMACO, 2011)
IMP-P9f IMP-P10r
CCTAAACACGGCTTGGTGGTT
GCCAAACCACTACGTTATCTGG IMP-5,-7,-9,-15 349 56ºC (CLIMACO, 2011)
SIM-F SIM-R
TACAAGGGATTCGGCATCG
TAATGGCCTGTTCCCATGTG SIM-1 570 57ºC
(ELLINGTON et al., 2007) KPC-F
KPC-R
GTATCGCCGTCTAGTTCTGCTG
GTTGACGCCCAATCCCTCGA KPC 860 61ºC (CLIMACO, 2011) OXA-23-F
OXA-23-R
AAGCATGATGAGCGCAAAG
AAAAGGCCCATTTATCTCAAA OXA-23 1066 54ºC (CLIMACO, 2011) OXA-48-F
OXA-48-R
TTGGTGGCATCGATTATCGG
GAGCACTTCTTTTGTGATGGC OXA-48 744 55ºC (CLIMACO, 2011) OXA-58-F
OXA-58-R
TTATCAAAATCCAATCGGC
TAACCTCAAACTTCTAATTC OXA-58 934 45ºC (CLIMACO, 2011) TEM – F
TEM – R
ATGAGTATTCAACATTTCCGTG
TTACCAATGCTTAATCAGTGAG blaTEM 860 52 °C (SPANU et al., 2002) TEM-1 - F
TEM-1 - R
GGGGAGCTCATAAAATTCTTGAAGAC
GGGGGATCCTTACCAATGCTTAATCA blaTEM-1 ∼1100 52 °C (ESSACK et al., 2001) SHV – F
SHV - R
ATGCGTTATATTCGCCTGTG
GTTAGCGTTGCCAGTGCTCG blaSHV 860 58 °C
(SPANU et al., 2002)
SHV-1 - F SHV-1 - R
GCCCGGGTTATTCTTATTTGTCGC
TCTTTCCGATGCCGCCGCCAGTCA blaSHV-1 1015 58 °C
(ESSACK et al., 2001)
BES - F BES - R
CGGTGGTTGCTGGTGGTGATA
CTGTGCCAGTCTTGTCGCCT blaBES 871 61 °C
(ANDRADE, 2011)
PER-1 – F PER-1 – R
GCTGTAGTTACTGCCTCGAC
GACTCGGCTGAGTCTTTCAC blaPER-1 818 59 ºC
(ANDRADE, 2011)
PER-2 –F PER-2 - R
GTGTTTTCACCGCTTCTGCT
GATTCAGCCGAATCCTTGAC blaPER-2 817 57 ºC
(ANDRADE, 2011)
Materiais e Métodos | 17
...Conclusão
Primers Seqüência (5’- 3’) Fragmento do gene a ser
amplificado
Tamanho
(pb)
Temperatura de
annealing Referência
VEB – F VEB - R
CGGTAATTTAACCAGATAGGAGT
CAACATCATTGATGGCTGCT blaVEB 792 55 ºC (ANDRADE, 2011) GES - F
GES - R
GCGCTTCATTCACGCACTATTA
CTATTTGTCCGTGCTCAGGATG blaGES 862 58 ºC (ANDRADE, 2011) QNRA-F
QNRA-R
AGAGGATTTCTCACGCCAGG
TGCCAGGCACAGATCTTGAC qnrA 580 54°C (CATTOIR et al., 2007)
QNRB-F QNRB-R
GGMATHGAAATTCGCCACTG
TTTGCYGYYCGCCAGTCGAA qnrB 264 54°C (CATTOIR et al., 2007)
QNRC-F QNRC-R
GGGTTGTACATTTATTGAATC
TCCACTTTACGAGGTTCT qnrC 440 50°C (WANG et al., 2009) QNRD-F
QNRD-R
CGAGATCAATTTACGGGGAATA
AACAAGCTGAAGCGCCTG qnrD 218 50°C (CAVACO et al., 2009) QNRS-F
QNRs-R
GCAAGTTCATTGAACAGGGT
TCTAAACCGTCGAGTTCGGCG qnrS 428 54°C (CATTOIR et al., 2007)
QEPA-F QEPA-R
GCA GGT CCA GCA GCG GGT AG
CTT CCT GCC CGA GTA TCG qepAA 300 61°C (BAUDRY et al., 2009)
CMY-Fw CMY-Rv
GGGCCCGGACACCYTTTTGC
TAAGTGTAGATGACARCAGG CMY-like 1250 37°C
(D'ANDREA et al., 2006) Int1-F
Int1-R
GGTTCGTGCCTTCATCCGTTT
GCTTCGTGATGCCTGCTTGTT intl1 698 59°C (CLIMACO, 2011) Int2-F
Int2-R
GCTACCCTCTGTTATCTCTGC
CGTTATACGGGTGGTTGCAGA intl2 393 59°C (CLIMACO, 2011) Int3-F
Int3-R
GGAGGTTCAGACGTTGCTTTC
CCAGTGCATGAGGCAGATACA intl3 290 59°C (CLIMACO, 2011) UP-SPM
DW-SPM
GCCCGAAAAGGGCGTTTG
GATCTGGATGCCGTGGTT ISCR4 1000 60°C (CLIMACO, 2011) AD55FW2 GTTCTACAACAGTATTTGGT Plasmídeo KPC variável 54°C Este estudo
AD63FW1 ATCTGAACAGCAAGATATCCG Plasmídeo KPC variável 58°C Este estudo
KPC2Mfw KPC2mRV
TCATCGCCGCGCGCCGTAAC
TTGCCGTAACGGATGGGTGT Plasmídeo KPC 2500 65°C Este estudo
S=C ou G; Y=C ou T; R= A ou G.
3.6 Sequenciamento
Os produtos amplificados foram purificados com o kit de purificação GFX-TM PCR (Amershan Bioscience) para seqüenciamento. Para o seqüenciamento foi utilizado o Kit DYEnamicTM ET Dye Terminator (GE) e os procedimentos foram realizados conforme orientação do fabricante.
3.7 Análise de plasmídeos
3.7.1 Perfil plasmideal
A investigação da presença de plasmídeos (pDNA) foi realizada segundo Barton et al. (1995). A análise do pDNA foi realizada por PFGE após utilização da enzima S1 nuclease
(Fermentas), S1-PFGE. A avaliação baseia-se na ação seletiva da S1 nuclease que cliva totalmente o DNA cromossômico (crDNA) e converte o pDNA circular em pDNA linear, proporcionalmente ao seu tamanho. A reação foi realizada segundo recomendações do fabricante da enzima.
Os blocos contendo os DNA foram aplicados no gel de agarose 1%. A corrida eletroforética foi realizada a 14°C e 6 V, com pulsos de 5-50 por 3 horas e 30-45 por 12 horas, totalizando 15 horas no aparelho CHEF-DRIII (BioRad). Após a corrida eletrofética o gel foi corado com brometo de etídio (1 µg/mL) durante 20 minutos, observado e fotografado utilizando o sistema de fotodocumentação AlphaImager® (Alpha Innotech, EUA).
Os isolados nos quais foram visualizados plasmídeos pela PFGE-S1 nuclease o pDNA foi extraído utilizando o PureLinkTM – HiPure Plasmid Filter Midiprep Kit (Invitrogen, Germany) para realização de PCRs, sequenciamento, restrição com enzima BAM-H1 e transformação.
3.7.2 Localização de genes de resistência
A localização dos genes de resistência detectados por PCR foi realizada em membrana de náilon após transferência dos pDNA contidos no gel de PFGE-S1nuclease-ePFGE-I-Ceu-I. A enzima de restrição I-Ceu-I cliva o DNA cromossômico em fragmentos grandes mantendo o 16S rDNA intacto.
3.7.2.1 Transferência do DNA plasmideal para a membrana de náilon
Foi realizada a transferência do pDNA, a partir do gel de S1-PFGE, para uma membrana de náilon Hybond N+ (Amershan Bioscience).
Após o gel de S1-PFGE ser fotodocumentado, foi imerso em solução de HCl 0,25M para a fragmentação do DNA plasmideal. Em seguida, o gel foi mantido, durante 90 minutos, em solução de NaOH 0,4 N, para desnaturar o DNA plasmideal.
Materiais e Métodos | 19
FRITSCH; MANIATS, 1989), durante 24 horas, utilizando NaOH 0,4 N como solução tamponante de transferência.
Após a transferência para a membrana, a mesma foi mantida em solução de SSC 2X (citrato de sódio 30 mM, NaCl 0,3 M) durante 5 minutos, posteriormente, mantida em TrisHCl 0,5 M, pH=7,0 durante 2 minutos e, seca à temperatura ambiente. Finalizando, o DNA foi fixado na membrana por exposição ao calor de 80 °C durante 2 horas em forno de hibridação Hybridization oven/shaker (Amersham Pharmacia). As membranas foram armazenadas, ao abrigo da luz e à temperatura de 4 a 8 °C. A membrana foi usada em experimento de hibridação com sondas específicas para o gene de resistência interesse.
3.7.2.2 Preparo da sonda genética
Para os genes de interesse (blaKPC-2), foram sintetizadas sondas genéticas utilizando o
kit AmershamTM AlkPhos Direct Labelling Reagents (GE Healthcare), segundo recomendações do fabricante.
A síntese consiste em utilizar o produto de PCR purificado e quantificado da linhagem controle do respectivo gene de interesse. Esse produto foi diluído na concentração de 10 ng/µL, desnaturado à temperatura de 99 °C por 5 minutos, mantido em gelo durante outros 5 minutos. Posteriormente, foi adicionada uma solução de marcação de DNA, que contém fosfatase alcalina, incubada a 37 °C por 30 minutos. A sonda, assim sintetizada, foi utilizada imediatamente ou mantida à temperatura de 4-8 °C por até 2 horas.
3.7.2.3 Reação de hibridização
A membrana contendo o pDNA foi submetida à pré-hibridação, com líquido de hibridação, em forno de hibridação Hybridization oven/shaker (Amersham Pharmacia), à temperatura de 55-60 ºC durante 15 minutos. O líquido de hibridação utilizado foi preparado segundo recomendações do kit AmershamTM AlkPhos Direct Labelling Reagents (GE Healthcare).
Após os 15 minutos de pré-hibridação, a sonda específica para o gene de interesse foi adicionada ao líquido de hibridação, já em contato prévio com a membrana.
a filmes de autorradiografia Hyperfilm-ECL (GE Healthcare) após um período de exposição de 1-3 horas, a temperatura de 4 °C. A revelação da hibridação foi realizada manualmente, com a utilização de reagentes para revelação/fixação do sinal no filme de autorradiografia, com posterior análise visual dos resultados.
3.7.3 Tipagem dos plasmídeos por grupos de incompatibilidade
A PCR baseada na tipagem dos replicons (PBRT, do inglês PCR Based Replicon Typing) foi realizada como descrito por Carattoli et al., (2005). Dezoito pares de primers
foram utilizados para realizar 5 reações de PCR multiplex e 3 reações de PCR simples para reconhecer os replicons FIA, FIB, FIC, HI1, HI2, I1-I , L/M, N, P, W, T, A/C, K, B/O, X, Y, FrepB e FII.
3.7.4 Caracterização do ambiente genético dos genes de resistência
O ambiente genético, integrons (In), transposons (Tn) e elementos de inserção (IS), onde os genes de resistência encontrados provavelmente estão inseridos, foram pesquisados utilizando primers específicos (Tabela 2). Foram pesquisados integrons (In) classes 1, 2 e 3, transposon Tn4401 utilizando esquema proposto por Curião et al., (2010) colaboradores e elementos de inserção (IS), como ISCR4, ISCR1 e ISEcp1.
3.8 Pesquisa de genes de fatores de virulência
A pesquisa de genes de virulência (alginato-algD, citotoxinas-exoS e exoY, quorum sensing-lasI/lasR e rhlI/rhlR, protease-arp e fímbria-pilA) foi realizada para P. aeruginosa
4.1 Isolados bacterianos e Concentração Inibitória Mínima
O total de 293 isolados de P. aeruginosa resistentes a cefalosporinas de 3ª e/ou 4ª gerações e/ou carbapenêmicos foram enviadas ao LEBEM no decorrer deste estudo (Anexo 1), e foram pesquisados genes de resistência citados no item 3.5, por PCR em todos os isolados. Os valores da CIM, obtidos pelo Etest®, de cada antibiótico testado para as bactérias que apresentaram gene bla
pesquisados estão demonstrados na Tabela 3. Como esperado, todos os isolados produtores de carbapenemases são resistentes ao carbapenêmicos testados (imipenem e meropenem), cefalosporinas já os isolados produtores de ESBLapresentaram CIM variadas para os carbapenêmicos e cefalosporinas. Todos os isolados foram sensíveis à Polixina B.
4.2 Genes codificadores de β-lactamases
Dos 293 isolados de P. aeruginosa estudados, 31 apresentaram algum gene de resistência. Oito isolados (P. aeruginosa HC58, HC84, HC103, HC289, HC313, HC367, HC371 e Pa128Rp) do HCFMRP-USP apresentaram o gene blaSPM-1 e cinco (HC305, HC326, HC375, HC402 e HC408) apresentaram o gene blaCTX-M-2 totalizando 13 isolados produtores de enzimas -lactamases. Três isolados de Belo Horizonte (BH6, BH9 e BH11) apresentaram o gene blaKPC-2 e um (P. aeruginosa BH7) o gene blaSPM-1. Três isolados de Cuiabá (MT 598,
MT 55 e MT 276) apresentaram o gene blaSPM-1 e 11 isolados (MT138-03, MT447, MT448,
MT450, MT142, MT222, MT279, MT140, MT137, MT138-08 e MT141) apresentaram o gene blaCTX-M-2 (Tabela 3). Nenhum isolado dos hospitais de Franca, Barretos e Rio Branco
apresentou fragmentos amplificados para os genes de resistência pesquisados.
4.3 Investigação do ambiente genético
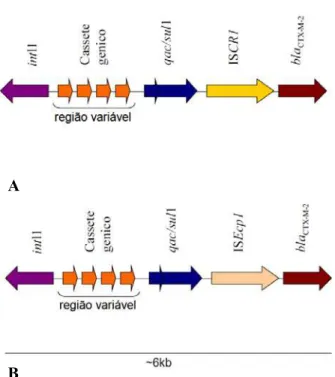
O ambiente genético dos genes de resistência encontrados foi pesquisado por PCR. Os genes blaSPM-1 estão localizados entre cópias do elemento de inserção ISCR4, uma upstrean e
outra downstrean ao gene,como esquematizado na Figura 1.
Figura 1. Ambiente genético do gene blaSPM-1 nos isolados estudados.
Todos os genes blaCTX-M-2 estavam associados a integron de classe 1, sendo que 3 isolados apresentaram o gene blaCTX-M-2 sendo mobilizado pelo elemento de inserção ISEcp1
Resultados | 23
Figura 2. Ambiente genético do gene blaCTX-M-2 nos isolados estudados. A. flanqueado pelo elemento de inserção ISCR1. B. flanqueado pelo elemento de inserção ISEcp1.
Os isolados produtores de KPC-2 não apresentaram amplicons para nenhum dos ambientes genéticos pesquisados para os genes blaKPC-2, como demonstrado na Tabela 3. Estratégias utilizando genewalking PCR foram utilizadas e demonstraram que o gene blaKPC-2
é flanqueado por duas proteínas hipotéticas, ainda sem função definida, de acordo com o banco de dados GenBank (http://www.ncbi.nlm.nih.gov/genbank/), sendo a proteína upstream
ao gene, aqui chamada Hyp1 (714pb) 76% idêntica à CP006982.1 (número de acesso no GenBank- posição 2734707-2735577) e a downstream ao gene, Hyp2, 87% idêntica à CP006982.1 (número de acesso no GenBank- posição 2736856-273600), sendo que ambas fazem parte do genoma de P. aeruginosa.
4.4 Clonalidade dos isolados de P. aeruginosa
4.4.1 Pulsotipos
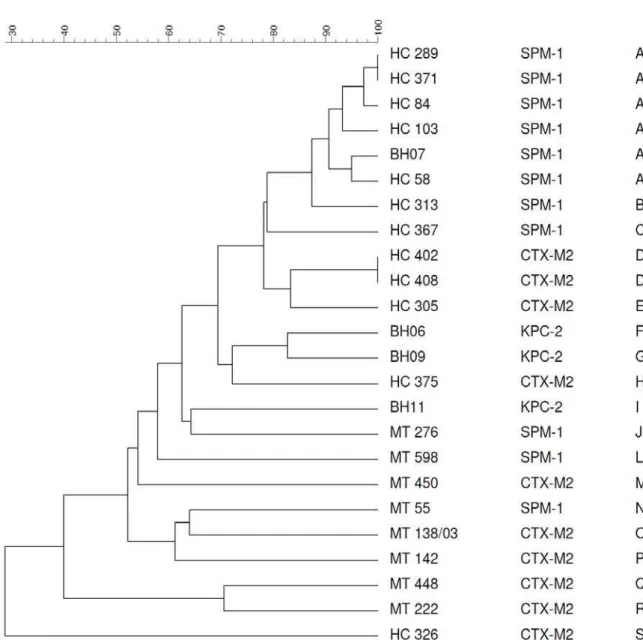
Vinte e quatro dos 31 isolados que apresentaram os genes bla foram tipados pela PFGE. Os perfis de macrorrestrição do DNA genômico destes isolados foram caracterizados após digestão utilizando a enzima de restrição SpeI e submetidos à PFGE.
De acordo com o dendrograma construído a partir do perfil de macrorrestrição dos isolados (Figura 3) e utilizando como critério similaridade superior a 90%, foram visualizados
A
18 perfis clonais nomeados de “A” a “S”. O grupo clonal A possui 6 isolados produtores de SPM-1, o grupo clonal B e C contém 1 isolado cada sendo os 2 produtores de SPM-1, o grupo clonal D possui dois isolados produtores de CTX-M-2 e os 14 perfis clonais restantes contém apenas 1 isolado cada, produtores de SPM-1, CTX-M-2 ou KPC-2 (Figura 3).
A maioria (6/11) dos isolados carreando o gene blaSPM-1 possuem o mesmo perfil clonal, sendo 5 deles provenientes do HCFMRP-USP e 1 de Belo Horizonte. É importante destacar que os isolados são de Estados diferentes e períodos diferentes, sendo os isolados provenientes do HCFMRP-USP em 2007 e, o proveniente de Belo Horizonte, em 2011. Os demais isolados produtores de SPM-1 provenientes do HCFMRP-USP possuem pulsotipos diferentes, porém com similaridade superior a 75% ao perfil clonal A. Os 3 isolados provenientes de Cuiabá possuem perfis clonais diferentes com menos de 60% de similaridade entre si.
Dois isolados carreando blaCTX-M-2 provenientes do HCFMRP-USP possuem o mesmo
perfil clonal e foram isolados no mesmo dia, na mesma clínica, porém em pacientes diferentes e de espécimes clínicos diferentes. Em geral, os isolados produtores de CTX-M-2 possuem grande diversidade clonal, principalmente aqueles isolados provenientes de Cuiabá, pois possuem menos de 60% de similaridade entre si.
Os isolados carreando gene blaKPC-2 também possuem perfis clonais diferentes sendo
que dois deles (BH6 e BH9) possuem aproximadamente 83% de similaridade entre si.
Não foi possível determinar o perfil clonal do isolado P. aeruginosa Pa128Rp produtor de SPM-1, pois o DNA apresentou-se degradado mesmo após várias tentativas de confecção de blocos e realização da eletroforese.
Resultados | 25
Figura 3. Dendrograma de similaridade genômica de P. aeruginosa carreando genes bla, dado pelo coeficiente de Dice gerado pelo método UPGMA utilizando o programa BioNumerics 5.01 a partir do perfil de macrorrestrição do DNA genômico clivado com enzima SpeI apresentado na PFGE. Os perfis estão representados por letras maiúsculas do alfabeto.
4.4.2 Tipo de Sequência gerada por MLST
Os isolados produtores de genes bla foram tipados por MLST e os alelos gerados estão apresentados no Anexo 2.
Além disso, os dados fornecidos pelo MLST demonstram que os três isolados produtores de KPC-2, do hospital de Belo Horizonte, pertencem a três STs diferentes (P. aeruginosa BH6-ST244, BH9-ST381 e BH11-ST261).
Tabela 3. Perfil de sensibilidade aos antibióticos e caracterização genética e molecular de P. aeruginosa que apresentaram genes bla.
Tipagem molecular
crDNA
Gene de resistência Determinação da CIM4 (µg/mL) Ambiente genético
P. aeruginosa
Pulsotipo1 ST2 bla Elemento
envolvido DNA
3 IMP MER CAZ AZT CPM POL
HC 58 A4 277 SPM-1 2 cópiasISCR4 Cr ≥32 ≥32 ≥256 6,0 ≥256 3,0 HC 84 A1 277 SPM-1 2 cópiasISCR4 Cr ≥32 ≥32 ≥256 4,0 ≥256 6,0
HC 103 A2 277 SPM-1 2 cópiasISCR4 Cr ≥32 ≥32 ≥256 6,0 ≥256 1,0
HC 289 A 277 SPM-1 2 cópiasISCR4 Cr ≥32 ≥32 ≥256 2,0 ≥256 2,0
HC 313 B 277 SPM-1 2 cópiasISCR4 Cr ≥32 ≥32 ≥256 4,0 ≥256 2,0 HC 367 C 277 SPM-1 2 cópiasISCR4 Cr ≥256 ≥256 ≥256 4,0 96 2,0
HC 371 A 277 SPM-1 2 cópiasISCR4 Cr ≥32 ≥32 ≥256 4,0 ≥256 2,0
BH 07 A3 277 SPM-1 2 cópiasISCR4 Cr ≥256 ≥256 ≥256 1,5 ≥256 1,5
MT 598* L 277 SPM-1 2 cópiasISCR4 Cr ≥16 ≥16 ≥64 16 ≥64 2,0
MT 55* J 277 SPM-1 2 cópiasISCR4 Cr ≥16 ≥16 ≥64 16 ≥64 2,0
MT 276* N 277 SPM-1 2 cópiasISCR4 Cr ≥16 ≥16 ≥64 16 ≥64 2,0
Pa128Rp* --- 277 SPM-1 2 cópiasISCR4 Cr ≥16 ≥16 ≥64 2,0 ≥64 2,0
BH 06 F 244 KPC-2 Hyp Pl ≥256 ≥256 ≥256 ≥256 ≥256 1,0
BH 09 G 381 KPC-2 Hyp Pl ≥256 ≥256 6,0 ≥256 48 1,5
BH 11 I 261 KPC-2 NS Cr ≥256 ≥256 ≥256 ≥256 ≥256 1,0
HC 305 E 244 CTX-M-2 ISEcp1-Int1 Cr ≥32 ≥32 16 96 ≥256 1,5
HC402 D 244 CTX-M-2 ISEcp1-Int1 Cr ≥256 ≥256 32 ≥256 ≥256 1,5
HC408 D 244 CYX-M-2 ISEcp1-Int1 Cr 6,0 ≥256 16 96 ≥256 1,5
HC 326 S 277 CTX-M-2 ISCR1-Int1 Cr ≥32 ≥32 4,0 24 64 1,0
MT 138-03* O 235 CTX-M-2 ISCR1-Int1 Cr ≥16 ≥16 ≥64 16 ≥64 2,0
MT 447* --- 532 CTX-M-2 ISCR1-Int1 Cr ≥16 ≥16 8,0 ≥64 ≥64 2,0
MT 448* Q 1081 CTX-M-2 ISCR1-Int1 Cr ≥16 ≥16 ≥64 16 ≥64 2,0
MT 450* M 244 CTX-M-2 ISCR1-Int1 Cr ≥16 ≥16 4,0 ≥64 ≥64 2,0
MT 142* P 244 CTX-M-2 ISCR1-Int1 Cr 4,0 ≥16 4,0 16 ≥64 ≤0,5
MT 222* R 235 CTX-M-2 ISCR1-Int1 Cr ≥16 ≥16 4,0 16 16 2,0
MT 279* --- 235 CTX-M-2 ISCR1-Int1 Cr 8,0 4,0 ≥64 16 ≥64 2,0
MT 140* --- 235 CTX-M-2 ISCR1-Int1 Cr ≥16 4,0 ≥64 16 ≥64 2,0
MT 137* --- 235 CTX-M-2 ISCR1-Int1 Cr 8,0 4,0 ≥64 ≥64 ≥64 2,0
MT 138-08* --- 244 CTX-M-2 ISCR1-Int1 Cr ≥16 ≥16 ≥64 ≥64 ≥64 ≤0,5
MT 141* --- 235 CTX-M-2 ISCR1-Int1 Cr 8,0 4,0 ≥64 16 ≥64 2,0
1
Resultado obtido por PFGE, 2 ST, tipo de sequencia por MLST, hyp: proteína hipotética,3Pl: Plasmídeo; Cr: cromossomo 4
Resultados | 27
4.5 Pesquisa e caracterização de plasmídeos
Utilizando a PFGE-S1 nuclease foi possível detectar plasmídeos em dois isolados produtores de KPC-2.
P. aeruginosa BH6 e BH11 apresentaram o mesmo padrão de 3 fragmentos de tamanho diferentes no gel de PFGE-S1 nuclease e houve hibridação com sonda específica para o gene blaKPC-2 nos três fragmentos (Figura 4A) demonstrando a localização plasmideal desse gene.. Após esse resultado, o pDNA dos isolados foi extraído e foram
realizados experimentos de restrição com enzima BAM-H1, que demonstraram a presença de apenas um plasmídeo de aproximadamente 3kb carreando o gene blaKPC-2 nos dois isolados. Além disso, o plasmídeo encontrado nesses isolados não pertence a nenhum grupo de incompatibilidade contemplado pelo esquema de tipagem por PBRT.
Para P. aeruginosa BH9 (produtor de KPC-2) não houve hibridação com sonda específica para blaKPC-2 nos géis de PFGE-S1 nuclease e sim nos gel de PFGE-I-Ceu-I
(Figura B1e B2) . Na figura B1 é possível observar que para este isolado houve hibridação com sonda específica para blaKPC-2 na banda referente ao DNA cromossômico. Além
disso, as figuras B1 e B2, confirmam que o gene blaKPC-2 nos isolados BH6 e BH11 está
inserido em plasmídeo pois é possível observar que na figura B2, que representa a
Figura 4. Filmes fotográficos demonstrando resultado de hibridação com sondas específicas para os isolados carreando blaKPC-2. A. hibridação com sonda específica para gene blaKPC-2 após PFGE-S1 nuclease. B1.
hibridação com sonda específica para gene blaKPC-2 após PFGE-I-Ceu-I. B2. hibridação com sonda específica para gene 16S rDNA após PFGE-I-Ceu-I.
Após PFGE-I-Ceu-I seguida de hibridação utilizando sondas específicas para o 16S rDNA e blaSPM-1 a localização cromossômica do gene blaSPM-1 foi confirmada para todos os
isolados produtores desse genes inseridos no estudo, assim como para os isolados produtores de CTX-M-2.
4.6 Pesquisa de genes codificadores de fatores de virulência
Os resultados da pesquisa dos genes relacionados com fatores de virulência investigados estão apresentados na Tabela 4: 100% dos isolados apresentaram gene algD,