L')
UNIVERSIDADEFEDERAL DE UBERLANDIA
CENTRO DE CIENCIAS
BIOMÉDICAS
CURSO
DE CIENCIAS
BIOLÓGICAS
Divergência Genética Interpopulacional em Tehagonisca
angus—alla
Latreille, 1811 (Hymenoptera,
:!
Apidae, Meliponinae)
Francis
de Morais
Franco Nunes
Monografia apresentada -à Coordenação
do Curso de Ciências Biológicas da Universidade Federal
de
Uberlândia pagaa obtenção do grau de Bacharel em CiênciasBiológicas
Uberlândia
-MG
tô
UNIVERSIDADE FEDERAL DE UBERLANDIA
CENTRO DE CIENCIAS
BIOMÉDICAS
CURSO
DE
CIENCIAS
BIOLÓGICAS
Divergência Genética Interpopulacional em Teh-agonia.?
angusmla
Latreille, 1811 (Hymenoptera, Apidae, Meliponinae)
Francis
de Morais
Franco Nunes
Orientador:
Prof. Dr. Warwick
Estevam
Kerr
»Monografia apresentada à Coordenação
de'
Cursº» de Ciências Biológicas daUniversidade Federal de Uberlândia para
a obtenção do grau -de Bacharel em Ciências Biológicas.
Uberlândia
-MG
UNIVERSIDADE FEDERAL DE UBERLANDIA
CENTRO DE CIENCIAS
BIOMÉDICASCURSO
DE CIENCIAS
BIOLOGICASDivergência Genética Interpopulacional
em
Tetragonz'scaangustula
Latreille,
1811(Hymenoptera, Apidae, Meliponinae)
Francis
de MoraisFranco Nunes
Aprovada
pela Banca Examinadora
emª/º/M
Nota
10039 .bm?/M
WProf.
Dr. Warwick Estevanln
ken
Orientador
”Qe/am &
Conab-a
dim
, x)“ ..,39 , . .
.
«_,; - aº S㺓 &“?» Profª. MSc.
Rosana
de Cassra OliveiraO
(ªx Wºº “ “ «ªº** .
ª
0 Gº““Orientadora
Dea'ícatázz'a
Ao
meu
[má/así
Antônia,-Aas
meu.;
z'mtâw;Frarzcíaâ
e
Femrtzfg—Aos meus avós,
joão
(írz mermm'arz),Tereza,
Assunção,
Maria
Aõacíia e Iníuí;
Aos mew tios
e
primas;
Peú
conf'atzça
újmz'fafp!
Especíaímente
à
7711571611mãe,
Rosa
Maria,
Pelo
amor
6
ii
AGRADECIMENTOS
Não queria nesse momento
ser
injusto, esquecendo nomes que colaboraramcomigo em minha formação profissional e da minha personalidade. No entanto, não posso deixar deagradeceraos nomes que agora vêm & minha mente! Desculpem-me ESQUECIDOS, porém
MUITO OBRIGADO!!!
Ao amigo Carlos Gustavo Nunes Siiva, por apresentar—me, em Santa Luziado Itanhy
— SE, 0 fascinante mundo das abelhas.
à amiga e orientadora Rosana de 'Câssiafºliveira, pelos
ensinamentos, apoio e constantes exemplos de boa conduta, determinação, responsabilidade, . Você foi uma dádiva de Deus que cruzou o meu caminho. Todas as palavras seriam poucas
para
agradecer—lhe tudoque
fez
por mimaté
hoje.Ao Prof. Dr. Warwick Estevam Kerr, Cientista e Educador do século, pela formação
político—científica, pelo modelode cidadão,—peloirradianteamorà Ciência e ao Brasii e pela
orientação desse trabalho. Obrigado pela paciência, por
ter
esperado tanto meu retorno, na qualidade de orientado.Ã Profa. Dra. Ana Maria Bonetti, com'quemconvivi
e aprendi'muito'em apenas *3
anos e 5 meses. A Senhora & realmente umexempio -a
ser
seguido.- Honestidade, Ética,Sinceridade, Objetivos, Responsabilidade,
(...)
foram valores importantes que me foramtransmitidos e que espero jamais esquecer. Por tudoisso,? hoje sou muito feliz! Ã Escola PÚBLICA que me ensinou desde a infância. Ã
todos os meus professores,
do pré—escolar & Universidade, por acreditarem na Educação
como ferramenta
transformadora da nossa (triste) realidade social. Persistam em seus ideais! Ã família Bernardeli (Sr. Mauro, Da. Lúcia,
Larissa, 'Mariinha' e Eduardo),“ por
completaremo vazio de um estudante que mora longe de casa. Obrigado pela receptividade,
simplicidade e horas de humor.
A amiga Kátia Bernardeli, meu braço-dimim'desde o início do
cum.
Fica uma!
iii
à amiga Ana Paula de Oliveira Ribeiro, pela convivência sadia, pela sinceridade
constante, pelas experiências de vida... Obrigado por todas as lições (desculpe-me, nem
sempre fui um bom alunol). Obrigadoã-Da. Eliana, pelos“ papos—cabeça'.
Ao amigo Rodrigo Lemes Martins... nossas diferenças nos completam, traduzindo-se
em crescimento pessoal.? Obrigado
por
- muitas vezester
contestado—me, o que mepossibilitou enxergar novos paradigmas. Se hoje sou uma pessoa um pouco melhor, devo
muitoo yoc'e'.
à CAPES, por
ter
inventado o"ProgramaEspecial de Treinamento; talvez-a maior
conquista de minha vida!
Ao PET/BIOLOGIA, minha PRIORIDADE NÚMERO 1. O que eu idealízei na Carta de
Intenção apresentada ao Grupo, antes da seleção,» foi
mito
pouco frente ao que estePrograma pôde oferecer—me e as possibilidades que juntos pudemos criar.
Aos amigos do PETIBIOLOGIA, por terem que escutar alguém tão prolixo. Quantas
histórias temos pra contar e relembrar! Obrigado: Bárbara, Fernando Biase, Elisângela, Luciana Paiva, Laiena, Braynner, Priscila, Cristiane, Kaila, Wilson, Raquel, Fernado Lourenço, Jaqueline, Carlos Roberto, Luciana Londe e Bruno. Vocês fazem parte de uma
passagemmuito especial'da minha vida.
Aos amigos da 42“ Turma de”Ciências Biológicas, especialmente& Tatyana, Andreia,
Francislenee Fabiane Sebaio, pela amizade, infindáveis horas de estudo e humor.
A todos os amigos do Laboratório
de
Genética, pelo conhecimento transmitida—ecompartilhado. Ressalvo aqui as boas risadas que demos juntos, que acabaram minimizando
meu stress. Aa Laboratório em si, pelo instrumental e aparelhagem necessários parana
execuçãodeste trabalho.
A todos os meus grandes amigos de ITUIUTABA—MG, especialmente André
e Aziz,
por entenderem minha ausênciae pelos bons momentos que 'curtímos' juntos!
A DEUS, pela vida
e
portudo isso-que relateiacima. Obrigadopeloaperto.
de
mão nas horas mais difíceis da minha caminhada!Somente as lágrimas que
morreram,
puderamtraduzir
silenciosamentea veracidade ' de minhas palavras.SUMÁRIO
ABREVIAÇÓES... v
RESUMO... I(í 1. INTRODUÇÃO... 01
1.1- Sobre-abelhas ... Q1 1.2 - Tetragonisca angustu/a ... 03
1.3- Biologia molecular... Q? 2. OBJETIVOS... 10
3. MATERIAL-EMÉTODOS ... 11
3.1 - Material biológico... 11
3.2- Extração
de
DNA.-.... 133.3 - Qualificação e quantificação do DNA e preparação de soluções de trabalho... 14
3.4- Amplificação doDNA porRAPD... 14
3.5- Análise de dados.... ... 1'7 4. RESULTADOSE DISCUSSÃO... 18
5.
CONCLUSÓES... 256.REFERÉNCIASBIBLIOGRÁFICAS... 26
% ºC uL 2n ABS -MgCÍz cm cM dATP dCTP dGTP dNTP dTTP DNA EDTA HCI KCl Mers mg min ABREVIAÇÓES
por cento (porcentagem) grau(s) Celsius
microlitro(s)
micromplar
diplóide
absorbância
cloreto de magnésio centímetro(s). centimorgan(s) 5ª—desoxiadenosina trifosfato 5'—desoxicitosina trifosfato 5'—desoxiguanosina trifosfato, 57-desoxinucleotideotrifosfato 5'-desoxitimidinatrifosfato ácido desoxirribonucléico
ácido etilenodiaminotetra acético sal dissódico
hora(s) '
ácido clorídrico »
cloreto de potássio molar
bases
em fitas simplesmiligramacs) minuto(s) mililitro(s) milimolar milímetro(=s) haplóide
cloreto
de
sódio nanograma(s)' nanômetro(s)pares de base
potencial hidrogeniônieo
pico-mol
ribonuclease-A
rotações
por minutosegundoás)
dodecilsulfato de sódio unidade(s)-de enzima
ultra—violeta
unweighted pair-group method using arithmetic
averages
voltsVi
RESUMO
Tetragonisca
angustula
(jataí)apresenta uma das mais amplas
distribuiçõesentre os
Meliponíneos,abrangendo
praticamentetoda a
America do Sul, Centrale
México.» A técnica de RAPD (Random'Amp/ifiedPo—lymorphic DNA)-tem sido utilizada
na
caracterização
de populações,detecção
de polimorfismos genéticos,análises
filogenéticas e, inclusive, confirmaçãode
taxonomiabaseada
— emdados
morfológicos.
Esse
trabalho objetivou-se estimara
diversidade genética entrepopulações
de T. angustulade três regiões
(TriânguloMineiro/MGz-Uberlândia 'l, 2, 3, 4e
5, Ituiutaba 1, 2e
3e
Uberaba 1e
2; Sul de Minas/MG:São
Miguel doAnta 1,2, 3,
4a
ti;-Argentina:Aristóbulo-delValle-e Posadas
1e
2),usando
Tetragomaclavipes como outgroup. Foram feitos bu/ks com 5 individuos por local de amostragem, utilizando-se »15
primers
(13curtos
e
2
longos) «comas
seguintes
condições
de
reação: 10ng de DNA, 10pmoles de primer, 2mM de MgClz, 1.5U de Taq DNA polimerase, vloopMde
cada
dNTP, 50mMde
KCle
iOmMde
Tris—HCl pH8.0 .Os produtos
das
amplificações foramseparados
em gel deagarose 12% e
fotografados. Os resultados foram
analisadospelo
método
de-porcentagem
dedesacordo e
análise de cluster por UPGMA. Ao nível de162%, as amostras
de jataí foram divididas emduas
clades: umacorrespondente à
região do Triângulo Mineiroe
outrapara as amostras
da Argentinae
Sul de Minas que, porsua
vez,se
divergiram em 2 grupos -a 14.2—%.-As
amostras
»do Triângulo Mineiroapresentaram
divergência máxima
de
6,5%, com 3 genótipos (doisde
Uberlândiae
umde
ituiutaba) que não apresentaram?dissimilaridade genética. As
amostras
do- Sul de Minasapresentaram
divergência genética máxima de 4.4%e as
da Argentina de55%.
Tetragona clavipes divergiu em69%das
amostras
de T. angustula. Os valoresencontrados
para T. angustula foram similaresaos encontrados
na literatura dentrode
uma mesma região - (Triangulo Mineiro); Noentanto,-para regiões
geograficamentedistantes
(Sul de Minas e Triângulo Mineiro), os valoresencontrados
foram superiores.isto
pode
ser
devidoao
uso de DNA proveniente debulks de cinco indivíduos, diferenças na Taq DNA polimerase
e
nos protocolos deextração. Novas-estimativas
de
divergênciapara
esta
espécie
usando—se bulks-odm maior número de indivíduos e maior número de primers forneceriamdados
conclusivossobre
o número mínimo “ amostral- porlocalidade e
taxa- de1JNTRODUÇÃO
1.1 -
Sobre
abelhas
A
grande preocupação
coma preservação
dos ambientes naturaisleva, hoje,
muitos
pesquisadores a
estudarem ossistemas
biológicos de váriasespécies,
sejamelas
animais,vegetais
ou microorganismos, na tentativa deconhece-las antes
de uma possível extinção ou ainda fazerdestes estudos ferramentas
úteispara
mantê— Iaspresentes
no planeta, assim comoentender
suas
relações na
natureza.Entre
as
inúmerasespécies
emquestão,
encontram—se os insetos que,apesar
daabundância de
populaçõese espécies,
chegarama
umestado
crítico de sobrevivência, colocando assim, outros organismos em risco, devidoàs
váriasinterações
dentro decomunidades
e
ecossistemas.
Pertencentes aos
insetos (ordem Hymenoptera),as abelhas,
cujosfósseis
mais velhos datam do Cretáceo,estão
intimamenterelacionadas ao
aparecimentodas
Angiospermas (CRUZ—LANDIM, 1960). Dentreessas,
muitasespécies estão
ameaçadas
pelaação
antrópica queaitera
os habitats naturais por meio dedesmatamentos,
uso indiscriminado de agrotóxicose ação
predatóriade
meleiros, alémda
concorrência com Apis mellifera(BALESTIERI, 1989; KERRet
al., 1996).As
abelhas
sem ferrão brasileiras, classificam-se na subfamília Meliponinae. WiLLE (1983) relatadescobertas recentes
de
fósseis europeus
por
KELNER-PILLAULT, do início doTerciário, sugerindo que os Meliponíneos surgiram na África,
portanto, não
se
restringindoàs
Américas. A hipótese que tem sido mais aceitaé
deKERR
e
MAULE (1964)que
consideram a América do Sul como o centro deorigem
África,
presença das espécies
mais primitivas (n=9)e
tambémas
maisespecializadas,
demonstrandoque
tiveram mais tempopara
diversificação.A situação
é preocupante
quandose
refereà
diminuição ou extinção depopulações e espécies
de Meliponíneos,responsáveis
por 40a
90% da polinização de regiões devegetação
nativa,dependendo do ecossistema.
Um exemploé a
destruição,
cada
vez maisacelerada, das
florestas nordestinas, em especiala
MataAtlântica e, na mesma medida, o fim
das
populações de
taisabelhas
(KERRet
al.,1996).
Nos meliponíneos, o comportamento classificado como eusocial, com relação
ao tamanho
de
colônias, diferença entre rainhae
operária, arquitetura do ninho,complexidade
de
códigos de comunicação,entre
outros,é
bem próximo ousemelhante
ao deApis mel/ifera (SAKAGAMI, 1971apud
BALESTIERI, 1989).Os
Meliponinae, há muitos anos, foramdomesticados
pelos povos pre-colombianos que lhes deram nomesainda
vigentes na cultura popular brasileira.Neste
grupode abelhas
conhece—se maisde
300espécies, das quais
pelo menos100 correm risco de extinção. A atual classificação zoológica
sugere
queesta
subfamília subdivide-se em
duas
tribos: Meliponinie
Trigonini (KERRet
al., 1996).A haplodiploidia característica
das abelhas
e uma ferramenta valiosapara
estudos de
genética,sobretudo
variabilidadee
manutenção
de polimorfismos, visto que asegregação
alélica dos machos (haplóides) reflete o genótipo da fêmea(diplóide) que os originou (FALCÃO,-1984). Dados apontam
para
uma diminuiçãoda
1.2 -
Tetragonisca
angustula
Tetragonisca
é
um—dos 53gêneros atuais da
tribo Trigoninique se destaca
pela ampla distribuição geográfica,
abrangendo
praticamente toda a Américado Sul, Centrale
México (BALESTlERl, 1989; FERREIRA,-1993; NOGUElRA-NETO, 1970).As
espécies
maisconhecidas são
Tetragonisca angustu/a Latreille,Tetragonisca buchwaldi
Friese
e
Tetragonisoa pfeifferíFriese (MICHENER, 1990). .Com relação
à
Tetragonisca angustu/a conhecem—seas subespécies
Tetragonisca angustufa angustufa-- Latreille -
e
» Tetragoniscaangustu/a
fiebrigi
Schwarz,
sendo
queesta
última restringe—seao Estadode
Santa Catarina, parte do Paraná, Argentina,Paraguaive
Vale do RioParaná
emSão
Paulo (NOGUElRA-NETO, 1970).A
espécie
Tetragoniscaangustuía possui
=comportamentos muitopeculiares quanto
à
reprodução, enxameagem, principalmente com relaçãoa
fisiologia comportamentalde
operáriase
rainhas virgens (FERREIRA, 1993).NOGUEIRA—NETO (1997) observou um
processo
deenxameação
emTetragonisca
angustularque
duroucerca
de 110 dias, tempobastante
longo quando comparado, por exemplo, a Plebeia droryana, em torno de 15 dias.MELLO (1981) estudou
a
bionomiade
»Tetragom'sca angustu/a,»descrevendo
relações
simples entreas castas.
A existência de cortedas operárias à
rainha foiobservada
e
esta,
porsua
vez,
não —possui,»na colméia, local preferencial depermanência. As operárias, mesmo em condições normais da colônia, também
podem ovipor. A autora
compara
as—características
vetológicas dev-Tetragoniscaangustu/a com outros Trigonini
estudados
e verifica umagrande semelhança
comTetragona
e
Cepha/otn'gona.GROSSO
e
BEGO' (1992) estudaram. divisãode
trabalho entre operárias deTetragonisca
angustu/a
angustula, considerandoa
relação idade do indivíduo eatividade por ele realizada. Verificou—se
que
o
»groomingé a
atividadede
maiorfrequência,
estendendo-se
dos primeiros dias de vidaaté cerca
do 55º dia. Outras atividadesapresentam duração
variável:- »visitasaos
potes de alimento — 50 dias,contato bucal — 49 dias, trabalho com cerume — 30 dias, trabalho com resina — 29
dias, trabalho com lixo — 29 dias-, participação no-
processo
de aprovisionamento.e
postura
das
células de cria (POP)— 15dias. As operárias de Tetragonisca angustu/aA longevidade média
alcançada pelas
operárias de Tetragoniscaangustula
angustu/a
foide
21901109?
dias.AlDAR(1999) em experimentos
deformação
de colônias filhas, observou queas
rainhas virgens de Tetragonisca angustu/a angustula levaram, em média, 20.9dias para acasalarem-se
e
iniciarema
postura.PIGNATA
e
STORT (1992) estudarama
quantidade relativa de coleta depolen em 4
espécies de
Me/ipona, Soaptot-n'gona post/cae
Tetragonisca angustula.Entre
as
meli'ponas,o transporte de polené
equivalente a 15% dopeso
da abelha. A proporçãonos
Trigonini é maior,sendo 193% para
8. posticae 337%
paraTetragonisca angustu/a.
CORTOPASSl—LAURINO' (1989) relata
a
produtividadede
Tetragonisçaangustula
durante todo o ano. A autora observoua
visitadessa
abelha a 326% das
190 plantas melíferas
estudadas
em uma região do Estado deSão
Paulo. A atividade de jataíé
tão intensa quefrequentemente
pilha ninhos fracos de Paratrigonasubnuda
e
Me/ipona quadrifasciata.As populações de Tetragonisca angustu/a
adaptam-se às
maisvariadas
condições
físicase
climáticas(BALESTIERI, 1989), com boa qualidadede
regulação de temperatura naárea
da criae
expressiva faixa de tolerância térmica (—1a
41ºC),tanto no inverno quanto no
verão
(PRONl, 1987).Segundo
PRONI (1987), Tetragonisca angustu/a comporta—se comopoiquilotérmicos típicos,
aumentando
a
taxa respiratória coma elevação
datemperatura. A termorregulação, no entanto,
é
diferente entreas duas subespécies:
para
Tetragoniscaangustu/a
frebrigia
faixa ótima no inverno varia entre 15a
25ºC,e
no verão, 25 a 30ºC, enquanto que, para Tetragonisca angustu/a angustu/a, omelhor ajustamento térmico
situa-se na
faixade
25a
30ºC no invernoe
40ºC no verão.Alguns
parâmetros
climáticosinfluenciama
atividade externadestas
abelhas,sendo a
temperatura,a
intensidade luminosa e umidade relativa os mais importantes na atividadede
vôo. Os indivíduosde
Tetragonisca angustula nãosaem das
colméias a baixas temperaturas, mesmo com boa intensidade luminosa (lWAMA,1977).
KNOLL (1985)
destaca que
condições metereológicas, principalmentetemperatura
e
pluviosidade, provocama
variação do número de indivíduos emcolônia.-Jataí, como
é
conhecida popularmente aespécie, é
umadas abelhas
indígenassem
ferrão maisconhecidas
e
mais facilmenteencontradas
no território brasileiro, inclusive em centros urbanos, onde nidifica com alta frequência porpossuir
hábitos de nidificação pouco exigentes,bastando
um lugaronde
haja uma cavidade suficiente para construção deseu
ninho (BALESTIERI, 1989; FERREIRA,1993).
A estrutura interna do ninho de Tetragonisca
angustula apresenta pequenos
potes
medindocerca
de 1.5 cm de altura, ondesão estocados
mele
pólen; favos de cria horizontais ou heiicoidaise
células reaisna
periferia. O ninhoapresenta
tambémum invólucro
bastante
desenvolvido e aentrada é
constituída de umtubo de cerume maleável,
amarelado
(BALESTIERI, 1989).Em
estudos
na região entre Piracicaba e Rio Claro /São
Paulo, LINDAUER
e
KERR (1960)descreveram que
as
colôniassão
geralmente populosas,
contendode
2000 a 5000 indivíduos.NOGUElRA—NETO (1997) incentiva
a
criação de
jataí, porser
uma
abelha
muito higiênica, resistente, fácil de manter
e
multiplicar, além de produzirum mel que
praticamente
dispensa
pausterização.O mel
desta abelha
é
muitoapreciado pelas populações
ruraise é
utilizado,empiricamente, no tratamento de
doenças
como glaucomae catarata
(IMPERATRIZ— FONSECAet
al., 1984a).BAZLEN (1997)
apud
AiDAR (1999) demonstrouação
bactericida domel de Tetragonisca angustu/a angustu/a quando colocados em meios de cultura para Escherichia coli
e
Staphyfococcusaureus.
Em Tetragonisca angustu/a, o pH médio do mel e
4.2, possuindo coloração muito
variada
entreas
colméias,de
praticamente incolora
castanho—escuro, ondeencontram-se presentes
glicose, frutose, galactose, maltose, lactose,sacarose,
melezitosee
refinose (lWAMA, 1977), alémde
água, proteínas, enzimase sais
minerais.
KNOLL (1985) constatou
que
a
proporção de indivíduosde
Tetragonísoa
angustu/a encontrados
comnéctar
foi maiorno verão, com pólen no outono,sendo a
coleta
de
resina, maior noinverno.Em
estudos
realizadosno
Campusda
Universidade deSão
Paulo (USP/SP);eabundante
no local, atrás,apenas,
de Trigona spinipese
Apis mellifera (IMPERATRIZ-FONSECAeta/.,
1984b; KNOLL, 1985, 1990).RABELO (1997)
estudando
interação abelha—planta naReserva
Ecológica doClube
Caça
ePesca
ltorcróde
Uberlândia-MG, considerou Tetragonisca angustula, entre outrasespécies
levantadas, como politróficas, por coletar néctar e/ou pólen emdiversos recursos
florais— Em-seus
dados,»esta
espécie aparece
como o terceiroapideo
mais frequente na Reserva (128%),atrás
de Apis mel/[fera (56%)e
Trigona spinipes (137%).É notável o
sucesso
dedispersão da
jataí, sobretudo emáreas
urbanas. Contando com um expressivo
númerode
alelossexuais,
os efeitosda
endogamiasão
menos prováveis de ocorrer. KERR (1994) relata que em Ribeirão Preto, 0 númerode
colméiasde jataí é
muito grande, facilitandoa
vida dos meliponicultoresque criam
essa
abelha,bastando
terapenas
uma colônia emsuas casas,
vistoque
a
área de
reprodução possui muito maisdo
que
as
44 colônias de meliponíneossugeridas
porKERR eVENCOVSKY (1982).KERR
e
SlLVElRA (1972)estudaram
() cariótipode
váriasespécies
demeliponíneos, encontrando 2n=34. POMPOLO (1992) refez
análise
citogenética emespécies de
Meliponinae, confirmando taisdados, e acrescentou
o predomínio decromossomos
submetacéntricose
metacêntricospara
essa
espécie.0
controle genético da glicerol-B—fosfatodesidrogenase efeito, na
maioria dosinsetos, por dois loci: um
gene que
se
expressa
durante todoo desenvolvimento(G-3-PDH1)
e
outro, típicoda fase
adulta (G—3—PDH2). CONTELet
al. (1992) estudaramessa
enzima em 21espécimes pertencentes a
12gêneros
de meliponíneosdetectando
polimorfismono
locus G-3—PDH1em
Tetragona clavipes-e
Tetragoniscaangustu/a angustu/a; polimorfismo doG-3—PDH2 foi detectado, também, em jatai.
FALCÃO (1984) encontrou homologia' genética em Tetragonísca angustu(a
1.3 - Biologia
molecular
BECKMANN (1988)
apud
'FERREiRAe
GRATTAPAGLIA (1998) enfocaa
mudança de paradigma genético,
da
GENÉTICA MENDELIANA (análise degenótipos
a
partir de fenótipos) paraa
atual GENÉTICA GENÓMICA (análise diretadas variações
do DNA).Com o advento
da
Biologia Molecular,— «astécnicas de
trabalho com DNAconstituem eximias
ferramentas
emestudos
aprofundados de genética, alémde
diversidadee
dinâmicade
populaçõese
estudos
de
evolução pelacapacidade
deinferir
dados
filogenéticos.KARY MULLIS, na
década de
80, desenvolveua
tecnologiada
Reação
emCadeia da Polimerase (PCR—Polymerase Chain Reaction),
que
lhe valeu o PrêmioNobel
de
Química em 1993. Tal técnica utiliza-sede
uma polimerase termoestável(Taq DNA polimerase) que
atua
na amplificação de fragmentos de DNA.Em 1990, em
estudos
-independentes,
WILLIANSet
ai.
e
'WELSHe
McCLELLAND, utilizaram primers de
sequências
arbitrárias, de poucospares
debase,
que
fianqueiam várias regiões complementares no DNA em estudo. A técnicafoi denominada por DNA Polimórfico Amplificado ao Acaso (RAPD
— Random
Amp/ified Polymorphic DNA) ou AP — PCR (Arbitrariily Primed—Polymerase Chain
Reaction).
A técnica
de
RAPD,baseada na
amplificação desegmentos
genômicos
múltiplos ao
acaso,
a partir de primers arbitrários, tem sido muito utilizadadevidoà
sua
rapideze
facilidade. Eia tem sidoempregada para
caracterizar populações,análises de
pedigree,estudos
filogenéticos, mapeamento genéticoe
melhoramentode plantas
e
animais domésticos (VALENTINiet
al.,1996).WILLIAMS
et
al. (1990) relatamque
diferençasde apenas
um par debases
são
suficientespara não
haver anelamentodos
primers.Diz-se que
a
técnica RAPDapresenta
dominância dos marcadores, ou seja,para
um indivíduo homozigoto dominante para um determinado aielo, amplificam—sedois fragmentos de mesmo
peso
molecular, quese
sobrepõem. Da mesma forma,para os
heterozigotos, há amplificaçãode
apenas
um alelo, com o mesmopeso
molecular, porém o fragmento gerado migrará como os do homozigoto. Neste caso,Na literatura, diversos trabalhos com
marcadores
RAPD têm sidoencontrados, sobretudo com relação
rãutilidade para
confirmaçãoda
taxonomiabaseada
emcaracteres
morfológicosde
muitasespécies.
KAZANet
al. (1993)estudaram
oparentesco
genético -e
variação emSty/osanthes
guiamensis
(LEGUMINOSAE), GONZÁLEZ
e
FERRER (1993) estudaram polimorfismos emHordeum sp
e
HOWELLet
al. (1994)utilizaram a técnica de RAPD para identificaçãoe
classificaçãode
Musa sp .HUNT
e
PAGE (1992) utilizaram-amplificaçãoao acaso de
fragmentos deDNA polimórficos (RAPD) a partir de cruzamentos artificiais de machos
e
rainhas de Apis mellifera. Eles definirampadrões de herança
parainsetos
haplodiplóides,classificados em 4 tipos de polimorfismos: tamanho de fragmentos, manifestação de
bandas
diplóide-especificas,presença/ausência de banda
e intensidade de bandas. Os dois últimos tipossão herdados
de maneira dominante, observandosegregação
mendeliana na progênie haplóidee
diplóide.Marcadores RAPD mostram—se muito eficientes no
mapeamento
genético. Noclássico de
HUNTe
PAGE (1995) forammapeados
3110 CM do genomade
Apismel/ifera, verificando grupos de
marcadores
ligados. Estimaram emaproximadamente 3450 CM o
tamanho
total do genomae
observaramumataxa
de recombinação muito alta
para esta
abelha.STILES
et
al. (1993),estudando
parentesco
entre cuitivares ,de mamão(Carica
papaya
L.) por RAPD, consideraram a técnica rápida, precisae
sensível paraanálises
genômicas.SCHlERWATER
e
ENDER (1993) utilizaram=13marcas
comerciais diferentesde
DNA polimerase termoestável, encontrando diferenças quantitativase
qualitativasnos padrões de
amplificação obtidosa
partirdas
várias combinaçõestestadas.
MEUNIER
e
GRlMONT (1993) testaram a reprodutibilidade da técnicautilizando dois termocicladores,
três marcas
de DNA polimerase termoestável, DNAde
trêsespécies
de bactériase
encontraram diferenças nospadrões
de amplificação. Sugeriramque
osdados resultantes de
RAPDsomente são
confiáveiscom
a
padronização da técnica, incluindo otimizaçãodas concentrações
dos
reagentes e
utilizaçãoconstante da
mesma aparelhagem.WEEDEN
et
al. (1992) analisarama
reprodutibilidade dos fenótipos RAPDConcluíram que tais modificações produzem diferenças nos
padrões
debandas,
masa
técnica foi útil na identificação devariedades e
construção demapas
genômicos.Na agricultura,
marcadores
RAPD têm sidousados
nacaracterização
de germoplasmas, facilitandoa
escolha
dos indivíduospara
cruzamento e»a
consequente expressão
de bons híbridos.O número de primers fa
serernusados
numa análise
de polimorfismo?deveestar
relacionadoaos
modos de reprodução,sistemas
de cruzamentoe
níveis de variação genéticadas espécies
estudadas
Isto- porque,para
espécies
muito
polimórficas talvez não hajanecessidade da
utilização de muitos primers, contrastando, porem, com ogrande
númerode
primers requeridospara analise
de indivíduosresultantes de propagação
vegetativa (LIUet
al., 1994). WlLKlEet
al.(1993)
ressalvam que
o perfilde bandas do
RAPD nãose
torna mais complexo como aumento de tamanho do genoma.
Em Meliponinae, encontram=se
poucos
-estudos sobre
variabilidade pormarcadores
RAPD. OLIVEIRA (1998) emestudo sobre
divergência genética pormarcadores
RAPD em Tetragonisca angustu/a, provenientesde
3países
(Brasil,Argentina
e
Panamá), estimoua
distância genética entre populações de diferenteslocais. A 13%
de
dissimilaridadegenética
houvea
formaçãode duas clades
(entituladas de grupos 1e
2). Encontrou-se uma distribuição geográfica maisabrangente
para o grupo 2,correspondente
a
Tetragoniscaangustu/a
fiebngi, do que o relatado pela literatura, alémde marcadores
moleculares que disoriminamestes
210
2. OBJETIVOS
Estimar & divergência genética, por-
marcadores
RAPD, -empopulações
de Tetragonisca angustu/ae
correlacionaras taxas encontradas
com outrosdados
de11
3. MATERIALE MÉTODOS
3.17
-Materiai biológiço
Coletou—seoperárias campeiras, na
entrada das
colônias, utiiizando-se puça. Asamostras
foram fixadas imediatamente em álcool absoluto, ou levadasdiretamente
ao
freezer
— 80ºC. No total, trabalhou-se com 18 colônias deTetragonisca
angustu/a e
uma de Tetragona clavipes,esta
última funcionando comooutgroup. As
amostras da
Argentina correspondemà
Tetragoniscaangustu/a
fiebrigi,enquanto as amostras
do Estado de Minas Gerais (Sul e Triângulo)são
)
))))))))))
))))))))
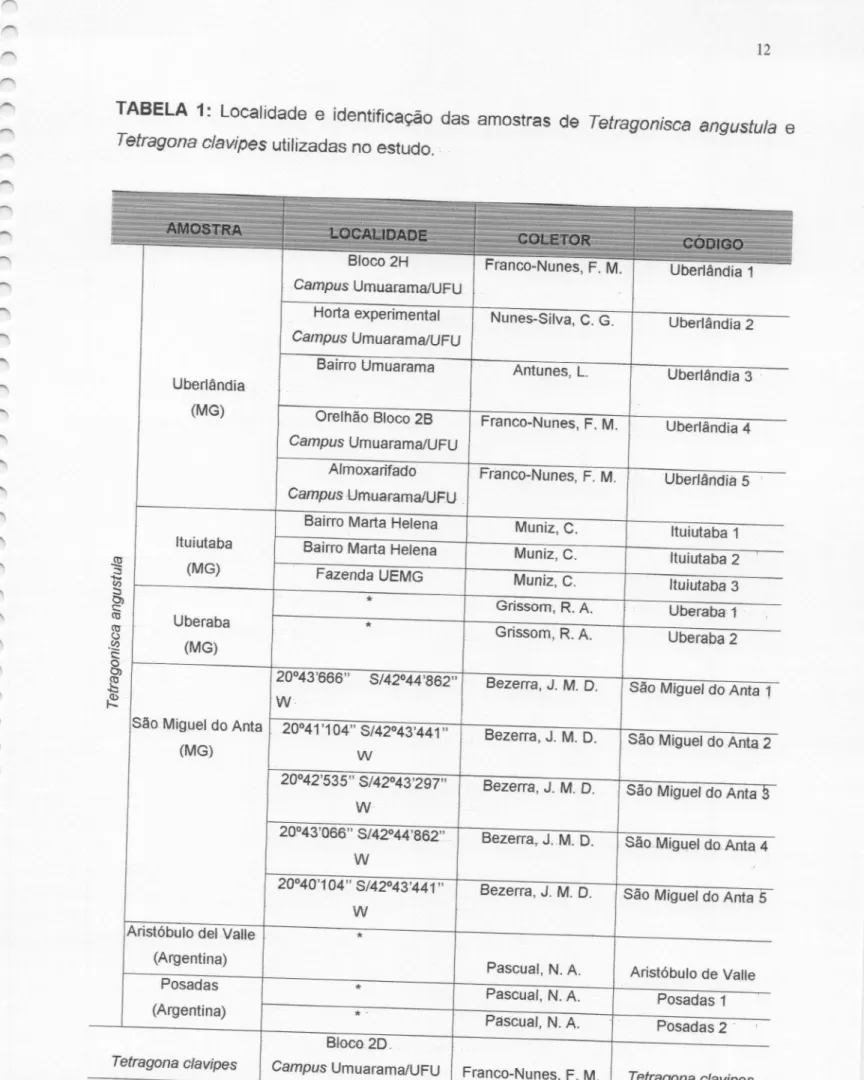
)))TABELA 1: Localidade
e
identificaçãodas amostras de
Tetragona clavipes utilizadas no estudo. =
Bloco 2H "
raco-Nune
F M.12
Tetragonisca angustu/a
e
"
Uberlanda 1
Campus Umuarama1UFU
Horta experimental Nunes-Silva,C. G.
Uberlândia2
Campus Umuarama/UFU
Bairro Umuarama Antunes,L.
Uberlândia3 '
Uberlândia
(MG) Orelhão Bloco25
Franco-Nunes, F.M. Uberlândia4 Campus Umuarama/UFU
Almoxarifado Franco-Nunes,F. M. Uberlândia
5 '
Campus 'UmuaramalUFU,
Bairro Marta Helena Muniz, C. Ituiutaba
1
Ituiutaba Bairro Mana Helena
Muniz,c. Ituiutaba 2 '
% (MG) Fazenda UEMG
Muniz, c. Ituiutaba 3
g, *
Grissom, R.A. Uberaba 1
É Uberaba *
Grissom,R.A. Uberaba2
"É, (MG)
É: 20º43'666" S/42º44'862“ Bezerra, J.
M. D. SãoMigueldoAnta 1
E ww
São MigueldoAnta 20º41'104" Sl42º43'441" Bezerra, J.
M. D. São Migueldo Anta2
(MG) W
20º42'535" S/42º43'297" Bezerra, J. M. D. São Migueldo
Antª
W'20º43º066" Sl42º44'862” Bezerra, J.,M. D. São,MigueldoAnta
4
W ]
20º40*104" S/42º43'441" Bezerra, J.M. D. São
Migueldo
Anta?
W
Aristóbulo del Valle *
(Argentina) Pascual, N. A.
An'stóbulo deValle
Posadas *
Pascual, N. A. Posadas1
(Argentina) *'
Pascual, N. A. Posadas2 '
Bloco2D,
Tetragona clavipes Campus Umuarama/UFU Franco-Nunes,F. M.
13
3.2 -
Extração de
DNAAntes
de
iniciara
extração,—utilizou—se lupa paraanálise das
patas- posteriores
de
cada
abelha, afimde
confirmar, pelamorfologia da unha, não-bifurcadas,
se os
indivíduos. eram operárias.Os
machos apresentam unhas
bifidas.
Cada
colôniafpirepresentada
por um bulk de 5abelhas
operárias. Os indivíduosconservados
em
álcool, foram reidratados em?
tampão
-(TrisCI-0.01M,—pH 8.0, NaCl 0.1M'
e
'—MgCI20.001M), para retiraro
excesso
de álcool.'
Para
extração
de
DNAgenômica
dos
iba/ks, utilizou-seo
protocolo. de
PAXTON
et
al. (1996), com modificações: 1 - Cincoabelhas
foramcolocadas
em
cadinhos de
porcelana, adicionando—se nitrogêniolíquidoe maceradas
em seguida;2 '- Adicionou—se 500pL
de
Tampão—-SET (NaCl 0.15M, Tris-HCl 0.02M, EDTA
1mMpH 8.0), transferindo o material para microtubo estéril de 1.5mL; 3 —Adicionou—se
em
cada
microtubo18pLde
proteinase K'(10mg/mL) e 22uLde
SDS 25%, homogenizandoa
solução em vórtex,e incubando—apor2h
a
55ºC;4- Adicionou—se 15pL de RNAse por microtubo
e
incubou-se por 1h
a
37ºC; ,5 — Adicionou-se 430uL de
NaCl 5M por microtubo, centrifugando o material
em seguida, por 10min
a
13000rpm; 6 — Osobrenadante
foitransferido
para
um novo microtubo de1.5mL,
adicionando-se 215pL de Tris-HCI (0.01M pH8.0) pormicrotubo; '
7 — Completou—se
o
volume
dos
»microtubos- com etanol995%
(—20ºÇ),
estocando
o material emfreezer
—20ºC por 2h ouovernight;8 - Centrifugou-se os- microtubos por» 15min
& 13000rpm, retirando-se
em
seguida
o etanol995% e acrescentando-se
500pL de Tris-HCl (0.01MpH 8.0) para
ressuspender
opel/ete
completandoo
volumedo
tubo comfenol,homogeneizando
() material em vórtex
e
centrifugando-se, em seguida, por5min
a
10000rpm; 9 — Osobrenadante
foitransferido para
um novo tuboe
o volume completadocom clorofil (clorofórmio/álcool isoamil, 24z1,
respectivamente),
homogeneizandoo
material emvórtexe
centrifugando-se,em
seguida,'por—-5mina10000rpm; 10 - Transferiu-se o
sobrenadante
para um novomicrotubo, repetindo-se o
14
11 — Retirou-se o etanol 99,5%
e
adicionou-se etanol 70%(20%),
invertendo-se
os
microtubospara
lavar bem-o pel/ete
centrifugando—se, em seguida, por15min
a
13000rpm;12 - Retirou-se
cuidadosamente
o— etanol70%
afimde não deslocar
-o pel/et, deixando o material
secar a
vácuo por, no mínimo, 3h ;13 - Redissolveu—se
opel/et
em 100 pLde
Tampão TE (Tris—HCl lomMiEDTA
1mM).
3.3 »—
Qualificação e
quantificação
-do DNA-e
preparação 'de soluções.
de
trabalho
Confirmou-se
a
qualidade ,das
'amostras
em= gelde agarose 08%.
A
quantificação do DNA foi feita em espectrofotômetro
para
leitura deabsorbância
&
260nm, utilizando-se 10 pL
de cada
amostra diluídos 100vezes
emágua
ultra—pura.Calculou-se a
concentração
de DNAa
partir da fórmula:[DNA]= ABS (260)=x50x
Fator
de
Diluição = .Em
seguida as amostras
de DNA foram diluídas para umaconcentração
de 5ng/pLe
constituiramas
soluções de
trabalho.3.4 -
Amplificação do
DNApor
RARDPor meio
de
umabateria
de-reações,
utilizando-seduas amostras
de localidades diferentes, selecionou-se pn'mers longos
e
curtos (10 Mers, da Operon Technologies). Assim, utilizou—se 15 primers informativos, ou seja,que
'denotaramum bom
padrão
debandas
para posterior análise. Os pn'mers utilizados)
)
)))))))))))))
)
)
))))))
))))))))))))))
)
)))))))
)
)
)
15
TABELA 2: Primers utilizados
e suas respectivas seqúências de
nucleotídeos.OPC12 '
Tarcrccc
OPC13 AAGCCTCGTC
OPC 14 TGCGTGCTTG
OPC18 ' TGAGTGGGTG »
OPF 09 . CCAAGCTTCC
.,, ? OPL 04 GACTGCACAC
É OPMos TCTG'ITCCCC *
3
opr
11 TTCCCCGCGAOPU01 - ACGGACGTCA
OPU 05 GGGTI'TGGCA
OPV oz AGTCACTCCC
opv
03 CTCCCTGCAA 'opz
13 , GACTAAGCCC8
MAU BZ GCCAGGCAGCAAG'ITCTCAGTAAT—' MAU 502 CAGCATI'CAGGGCTAACATC
Cada reação de
amplificação continha um volumefinal de 20pL, com:10.2er deágua ultrapura,
2pLde Tampão (Taq) 10X
2pLde dNTPs4mM.(dATP, dCTP, dGTP;dTTP)
1uLdeMgCIz 10mMIpL
2.5pL de primer4pmo|lpL
0.3pL de Taq DNA polimerase5U/uL
))
)
)
)
)
)
)))))
))
)
)
)
)
))
16
Acrescentou-se uma gota de óleo mineral (w20pL) para evitar
a evaporação
da
solução.Foram
feitas
amplificações- do material em termociciador M. J. Research—inc.,modelo PTC 100, por meio dos
seguintes passos:
"DESNATURAÇÃO: 94ºcnmin
ANELAMENTO DOSPRIMERS:35ºc11 min
EXTENSÃODASFITAS: 72ºC/2min
DESNATURAÇÃO: 94ºc1105ec
,ANELAMENTODOS-PRWERS:40ºCí20sec
EXTENSÃO DASFLTAS;72Bc/2min
EXTENSÃO FINAL DAS FITAS;72ºC/5min
Os
produtos ampiificados foramseparados
por eletroforese em gel-de
agarose 12%
comtampão
Tris-borato EDTA (TBE) 0.5X. O corante de DNA foibrometo
de
etídio, adicionando-se -1pL emcada
100mLde
gel. Os resultados foram visualizadosapós
2 horas de corridaa
150V, em transiluminador UVe
fotografadosemVDS Image System —Pharmacia.
As
reações
contiverem um controie negativo- com todos os produtosdas
reações,
exceto DNA,para
verificarse os reagentes
não estavam contaminadoscomDNA exógeno.
17
3.5 -
Análise de
dados
Apartir das bandas
obtidas foi«montada uma matriz binária (1 —presença de
banda e
O —ausência de
banda) para análise de distância genética pelo método dePorcentagem de Desacordo
e análise de clusterpelo
Método Não—ponderado qe Agrupamentoaos Pares
(UPGMA), com o auxílio do programa STATISTICA 4.5A. Estimou-sea
divergênciagenética entre
as
18— colônias de Tetragonisca18
4. RESULTADOSE DISCUSSÃO
O protocolo de
extração
de DNA seguidov_
PAXTONet
al.(1996)_
é
rápidoe
não utiliza produtos tóxicos. No entanto, a qualidade do DNAobservada
por meiodo pe/tet nos bulks não foi comum,
apresentando-se
“sujo”, quando deveriaser
transparente.
Observou-se dificuldade emressuspender
o pel/et,notando-se
muitos fragmentoscompactados.
-Aquantidade de
DNA extraída também foi—relativamenteinferior, comparada com
dados
obtidos com o uso de outros protocolos emetapas
preliminares. As modificações feitas
no
protocolo- foramas
inclusõesde
RNAse,fenol
e
clorofil(passos
4, 8e
9, respectivamente), comuns em outros protocolose
ausente
neste.Feitas
as
modificações,—opresente
protocolo tornou-se eficiente naextração extração de DNA
das amostras estudadas.
Vários
trabalhos
têm ressaltado a
- dificuldade naanálise das bandas
polimórficas
e
monomórficas produzidas por RAPD, visto que dois fragmentos demes-mo
peso
molecuiari comum-entreduas
ou-maisamostras,
podem—siginificar umalelo homólogo (herdado do ancestral) ou homoplásico (aparecimento
independente
na população). Assim como
em
outras
classes de marcadores
moleculares,encontra-se
dificuldade na análisee
interpretaçãodas bandas geradas.
Na análisede
RAPDdeve haver
flexibiiidade do pesquisador-emselecionaros marcadores
maisinformativos, ou seja, que
apresentam
melhor nitidez (FERREIRAe
GRATTAPAGLIA, 1998).
Dessa
forma,considerando apenas bandas intensas, pode-se
verificaruma alta taxa de polimorfismo em Tetragonisca angustula
e
Tetragona clavipes, paracada
primer analisado.Os
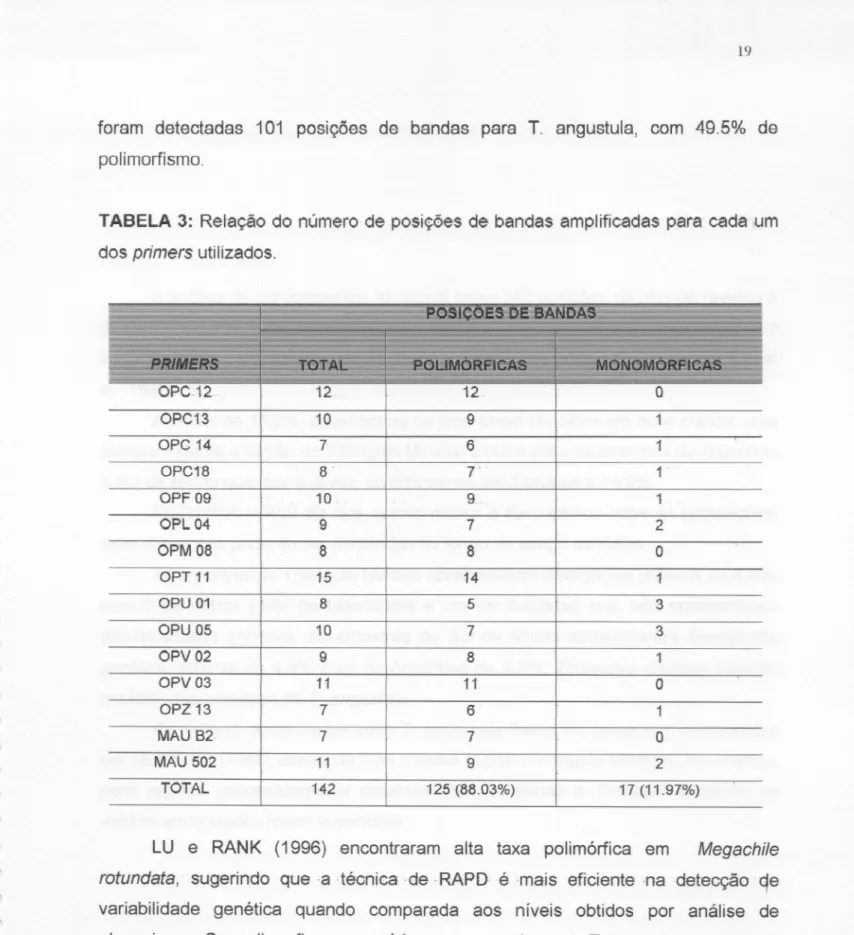
15 primers utitizados geraram 142posições de bandas
informativas (Tabela 3), uma média
de
9.46posições
debandas
por primer,das
)
)
)
)))
)')')ª)')')')))l)ª)')l)')')4)l)l)')l)º)l)l)l)!)lll)v)l)l)>)l)
)))))l)))')))))l)i)l)i)n
19
foram
detectadas
101posições
debandas
para T. angustuia, com495% de
polimorfismo.
TABELA 3: Relação do
númerode posições de bandas
ampliticadaspara cada
,umdos
primers utilizados.,12 ' '
12 " 12 o
OPC13 10 9 1
OPC 14 6 1
OPC1'8 8' 7"'
1 '
QPF 09 , 10, , 9 1 ,
OPL04 7 2
OPM 08 8 O
OPT11 15 14 1
CPU01 8 5 3
-OPU 05 10 7 3
OPV02 9 8 1
opv
03 11 11 oOPZ13 7 6 1
MAU 52, 7 7 o
MAU 502 11 9 2
TOTAL 142 125 (88.03%) 17 (11.97%)
LU
e
RANK (1996) encontraram alta taxa polimórfica em Megachilerotundata, sugerindo
que
«atécnica
de
RAPD é mais
eficientena detecçãode
variabilidade genética quandocomparada aos
níveis obtidos por análise de aloenzimas. Os polimorfismosgenéticos encontrados
em Tetragonisca angustui/a têm sido mais representativospara estudos
de variabilidade queos
descritos porFALCÃO(1984)
estudando
oitosistemas
enzimáticqs.C-HALMERS
et
al; (1992)aflrmamque RAPDé
uma ótimaferramenta para
22
Essa
diferenças de valores de divergênciaencontrados
podem
ser
decorrentes
de diferenças no número amostrale
também da metodologia empregada. Asseguintes observações
podemser
feitas:1 - Enquanto OLIVEIRA(1998)utilizou *Taq DNA
polimerase proveniente da Universidade Federal do Rio Grande do Sul (CENBIOT/RS),
neste estudo a
mesma enzima foi adquiridada empresa
P—ham1acia.-2 - As
amostras
utilizadas por OLIVEIRA (1998) foramrepresentadas
porapenas
um individuode cada
localidade.Aqui, utilizou—se bulkde
5 indivíduospara
representar cada
colônia amostrada, resultando numa maior diversidade genômica.No entanto, OLIVEIRA (1998)
estudou
umaextensão
geográfica mais
amplale
contínua, aopasso que neste
trabalhoanalisou-se apenas
3 localidades,separadas
geograficamentee
com númeroamostral
diferente entreelas
(dez do TriânguloMineiro, cinco do Sul de Minas
e três
da Argentina).3 - Os protocolos
de
extraçãode
DNA nos dois trabalhos foram distintos.4 - OLIVEIRA(1998) utilizou 18primers (11 curtos
e
7longos)
enquanto
que,para
este
estudo, foram utilizados 15 (13 curtose
2 longos).Apenas
dois dos primers utilizados emambos
os trabalhos foramcorrespondentes, sendo
um longo(MAU BZ)
e
um curto (OPL 04).O número amostral para
cada
localidade não foipadronizado devido-a pouca disponibilidade de exemplares fora do Triângulo Mineiro. Preferiu-se utilizar muitas
amostras
dessa
região,visando
estimara
divergência genéticapara-esta espécie
de forma precisa,a
fim de fornecer subsídios paraestudos
posteriores.De
qualquer
forma,ambos
trabalhos detectaram polimorfismos-genéticos significativos para Tetragonisca angustu/a, indicando diversidade biológicadessa
espécie, seja
ecológica, comportamental,-metabólica
e
ocorrência geográfica, queacabam
favorecendo a aclimataçãodas
populações emseus
nichos. Asvariações genotipicas
encontradas
dentrode
umaespécie,
expressa
pelo fenótipo,favorece,
a
melhoria naadaptação das
populações emseus
habitats.DOBZHANSKY
(1970)
apud- FALCÃO (1984) enfatiza queas
modificações ambientais poderiamser desastrosas
para
populações homozigóticas, por não possuírem uma variabilidade alélica suficiente paraadequar-se às
mudanças.Apesar
de pertencerema
mesma-tribo,osdados
obtidos mostraram pouca homologia entre Tetragonisca. angustulae
Tarragona clavipes: 69% de dissimilaridadegenetica.
Dados similares foram obtidos por OLIVEIRAl
))))l
)
)))))))
)
o
cluster
Tetragonisca. angustu/a com Apis mel/ifera (51%) e Tetragonisca buchwaldi (519%).A
técnica de
RAPD permite umaanálise
maisacurada
em investigações de
parentesco
entre populações de uma mesmaespécie,
ao invés deespécies
menos relacionadas (GONZALEZe
FERRER,-19953).Um exemplo
de
polimorfismogenético pode
serrcomprovado pelo
padrão-de
posições
debandas
na Figura 2, resultado da amplificaçãodas
18amostras
depopulações de
Tetragoniscaangustu/a
e
-1 amostrade
populaçãode
Tetragorra clavipes por meio do primer curto OPM 08.
Para este
primer foramanalisadas-8
posições de bandas de
forte intensidade. A técnica eletroforétioa possibilitaa
detecção
de variantes alélioas, possibilitando oestudo de
loci polimórficose
monomórficos (FALCÃO; 1984);HARTL-(1980)apud
FALCÃO-(1984) considera—a
eletroforese como
a
técnica mais útiljá idealizada para revelar variação genética. A variabilidade genéticaé
importanteà adaptação
e/ou
à processos
deespeciação
e,=dependendo
de
amplitude,-