UNIVERSIDADE DE SÃO PAULO
FACULDADE DE MEDICINA DE RIBEIRÃO PRETO
DEPARTAMENTO DE GENÉTICA
NATHALIA JOANNE BISPO CEZAR
Participação de genes relacionados ao processo inflamatório no
Diabetes Mellitus Gestacional
NATHALIA JOANNE BISPO CEZAR
Participação de genes relacionados ao processo inflamatório no
Diabetes Mellitus Gestacional
Dissertação apresentada à Faculdade de Medicina
de Ribeirão Preto da Universidade de São Paulo
para obtenção do título de Mestre em Ciências.
Versão corrigida. A versão original encontra-se na
secretaria da pós-graduação.
Área de Concentração: Genética
Orientador: Prof. Dr. Geraldo Aleixo da Silva
Passos Júnior
RIBEIRÃO PRETO
AUTORIZO A REPRODUÇÃO E DIVULGAÇÃO TOTAL OU PARCIAL DESTE TRABALHO, POR QUALQUER MEIO CONVENCIONAL OU ELETRÔNICO, PARA FINS DE ESTUDO E PESQUISA, DESDE QUE CITADA A FONTE.
FICHA CATALOGRÁFICA
Cezar, Nathalia Joanne Bispo
Participação de genes relacionados ao processo inflamatório no Diabetes Mellitus Gestacional. Ribeirão Preto, 2013.
113p.
Dissertação de Mestrado apresentada à Faculdade de Medicina de Ribeirão Preto da Universidade de São Paulo. Área de concentração: Genética.
Orientador: Passos, Geraldo Aleixo
FOLHA DE APROVAÇÃO
NATHALIA JOANNE BISPO CEZAR
Participação de genes relacionados ao processo inflamatório no Diabetes
Mellitus
Gestacional
Dissertação apresentada à Faculdade de Medicina
de Ribeirão Preto da Universidade de São Paulo
para obtenção do título de Mestre em Ciências.
Área de Concentração: Genética
Aprovado em:
__________________Banca Examinadora
Prof. Dr. ________________________________________________________
Instituição:___________________Assinatura: ___________________________
Prof. Dr. ________________________________________________________
Instituição:___________________Assinatura: ___________________________
Prof. Dr. ________________________________________________________
Dedico este trabalho
Aos meus pais, pelo apoio incondicinal e pelos valores que me trasmitiram desde os meus primeiros passos. O diferencial em minha formação foi ter tido vocês como exemplo de luta, coragem e perseverança.
AGRADECIMENTOS
A Deus, pela força e equilíbrio que me concedeu diante dos obstáculos enfrentados durante esta etapa.
Aos meus pais, Severiano Cezar e Rosa Bispo, por me ensinar a lutar, argumentar, perseverar e defender meus ideais dando exemplo de caráter e integridade. Obrigada por me convencer que sempre vale a pena arriscar para crescer, por mostrar desde cedo que lamentar não soluciona nossos problemas e o que precisamos é do respeito e não da piedade do mundo. Agradeço a participação diária, apesar da distância física, e pela alegria verdadeira diante das minhas conquistas. Ao meu irmão Augusto Bispo, por todo incentivo e pela torcida. Por nossa união tão pura e sincera nesses 25 anos da minha vida.
Ao meu Henrique Andrade, pela compreensão e apoio incondicional, por toda paciência durante esse período. Por sonhar e lutar junto comigo. Por estar ao meu lado independente de qualquer distância.
Ao professor Geraldo Passos, pela oportunidade, confiança, compreensão e suporte para o desenvolvimento deste trabalho. Agradeço a contribuição para minha formação científica.
Aos pesquisadores e participantes do projeto temático FAPESP, ao qual este trabalho está inserido: Profa. Dra. Elza Tiemi Sakamoto-Hojo e Prof. Dr. Eduardo Antônio Donadi e
aos pacientes, sem os quais esse trabalho não poderia ser realizado.
À Adriane Feijó, pela amizade e participação fundamental na concretização deste trabalho. Pela imensa ajuda, paciência e dedicação. Agradeço as orientações e o aprendizado.
À amiga Renata Almeida, pelo apoio e disponibilidade e por todas as discussões científicas que muito contribuíram para este trabalho.
Aos colegas do Laboratório de Mutagênese, Danilo Xavier, Paula Takahashi, Fernanda Caetano, Fernanda Paula e Leonardo Franchi pela receptividade. Em especial, a Danilo pela gentileza, disponibilidade, compromisso e pela contribuição fundamental.
Ao Programa de Pós-graduação em Genética da Faculdade de Medicina de Ribeirão Preto – USP, especialmente ao coordenador, Prof. Dr. Ademilson Spencer Egea Soares. A Secretaria do Departamento, em especial às secretárias Susie Nalon e Silvia Consiglieri pela atenção e dedicação.
Aos membros da banca examinadora pelas sugestões, contribuições e críticas.
À minha avó Matilde Bispo, pelo exemplo de força e luta, pelas palavras ditas na última vez que tivemos a oportunidade de conversar. Obrigada pela confiança.
Às amigas Fernanda Bueno e Lais Sacramento, pela união, parceria e compreensão nesses dois anos de convivência diária. Agradeço o apoio e a torcida sincera. Os momentos felizes que partilhamos estarão sempre em minha memória.
À amiga Ana Cláudia da Silveira, pelo carinho, incentivo, suporte e pelas infinitas orações. Às amigas Rebeka Maranhão, Carolina Vasconcelos e Danielly Cantarelli pela companhia, apesar da distância, e pelos muitos momentos felizes. Ao amigo Martone Souza, pelo estímulo.
“Tente uma, duas, três vezes e se possível tente a quarta, a quinta e quantas vezes forem necessárias. Só não desista nas primeiras tentativas, a persistência é amiga da conquista. Se você quer chegar aonde a maioria não chega, faça aquilo que a maioria não faz.”
RESUMO
CEZAR, NJB. Participação de genes relacionados ao processo inflamatório no diabetes
mellitus gestacional. 2013. 113p. Dissertação de Mestrado – Faculdade de Medicina de Ribeirão Preto, Universidade de São Paulo, Ribeirão Preto, Brasil, 2013.
O diabetes mellitus gestacional (DMG) é o distúrbio metabólico mais comum da gravidez. A definição padrão do DMG consiste no metabolismo anormal da glicose diagnosticado pela primeira vez durante a gestação. Mulheres que têm história de DMG geralmente apresentam diabetes pós-parto, resistência à insulina, síndrome metabólica, hipertensão e dislipidemia. A detecção precoce deste estado metabólico anormal é importante para eventual intervenção na tentativa de impedir ou mesmo retardar o aparecimento dos outros tipos de diabetes. Alguns estudos têm apontado, em mulheres com DMG, indução de genes envolvidos com resposta imune, particularmente aqueles associados com inflamação. A identificação de genes de inflamação induzidos em gestantes com DMG tem fornecido a base para elucidar a ligação entre vias inflamatórias e DMG. Para testar esta hipótese foi realizada a comparação do perfil transcricional de células mononucleares de sangue periférico (PBMCs) de pacientes com DMG e controles. As amostras de RNA total foram hibridadas utilizando oligo microarrays Agilent ® 4 x 44 K englobando o genoma funcional humano total. Os mRNAs diferencialmente expressos foram identificados aplicando-se a análise de Rank Products, e posteriormente submetidos ao agrupamento hierárquico de Pearson por meio do software Cluster. Utilizando o programa TreeView, foi realizada a construção dos dendrogramas com as representações espaciais dos mRNAs, classificados de acordo com suas funções moleculares e vias biológicas. A partir do banco de dados DAVID, foram identificados 130 processos biológicos significantes (P<0.05) incluindo os de resposta imune e defesa, resposta inflamatória, regulação de citocinas, apoptose, desenvolvimento de vasos sanguíneos e proliferação celular. Entre as vias de maior relevância destacamos a via de interação entre receptores de citocinas e a de sinalização do receptor NOD-like, além das vias de câncer, lúpus e asma. Adicionalmente, encontramos os transcritos dos genes IGFBP2, TCF3, OLR1, TCF7L2, previamente associados a alterações metabólicas, diferencialmente expressos nas gestantes com DMG. Também observamos que genes do complexo principal de histocompatibilidade (MHC), HLA-DRB6, HLA-DQA2, HLA-DQB2, HLA-DQB1, HLA-DOA, apresentaram mRNAs induzidos nas pacientes com DMG. A partir deste estudo, constatamos que vias relacionadas ao sistema imunológico e categorias funcionais associadas à inflamação participam da patogenia do DMG. Além disso, evidenciamos que transcritos de genes que pertencem ao MHC e aqueles envolvidos em processos metabólicos, estiveram diferencialmente expressos no DMG. Estes resultados confirmam nossa hipótese inicial e contribuem para o melhor entendimento das bases genéticas desta doença.
ABSTRACT
CEZAR, NJB. Participation of genes related to inflammatory process in gestational
diabetes mellitus. 2013. 113p. Master’s Dissertation – Faculty of Medicine of Ribeirão Preto, University of São Paulo, Ribeirão Preto, 2013.
Gestational Diabetes Mellitus (GDM) is the most common metabolic disorder found during pregnancy. The standard definition of GDM is the abnormal glucose metabolism first diagnosed during pregnancy. Women who have a history of GDM usually present postpartum diabetes, insulin resistance, metabolic syndrome, hypertension and dyslipidemia. Early detection of this abnormal metabolic status may permit early intervention to prevent or even delay the development of other types of diabetes. The induction of genes involved in immune response in women with GDM has been reported, particularly those associated with inflammatory pathways, providing basis proposing that inflammation genes might be associated to GDM. To test this hypothesis, we compared the transcriptome profiling of peripheral blood mononuclear cells (PBMCs) of GDM patients and controls. The total RNA samples were hybridized to Agilent ® 4 x 44 K oligo microarrays covering the whole human functional genome. Differentially expressed mRNAs were obtained by Rank Product analysis and then submitted to hierarchical clustering using the Cluster software . Dendrograms and spatial representations of mRNAs were constructed through the TreeView software . These mRNAs were classified according to their molecular functions and biological pathways using the DAVID database. We observed 130 significant biological processes (P<0.05), including immune and defense response, inflammatory response, regulation of cytokines, apoptosis, blood vessels development and cell proliferation. Among the most relevant pathways, we highlighted the interaction between cytokine receptors, NOD-like receptor signaling and cancer, lupus and asthma pathways. Additionally, we found transcripts of the genes IGFBP2, TCF3, OLR1, TCF7L2, which were previously associated with metabolic abnormalities, differentially expressed in pregnant women with GDM. Some major histocompatibility complex (MHC) genes (HLA-DRB6, HLA-DQA2, HLA-DQB2, HLA-DQB1, HLA-DOA) also presented mRNAs induced in patients with GDM. In conclusion, we found that immune-related pathways and functional categories associated with inflammation participate in the pathogenesis of DMG. Furthermore, we showed that transcripts of genes belonging to MHC and those involved in metabolic processes were differentially expressed in DMG. These results confirmed our initial hypothesis and contribute to a better understanding of the genetics basis of this disease.
LISTA DE FIGURAS
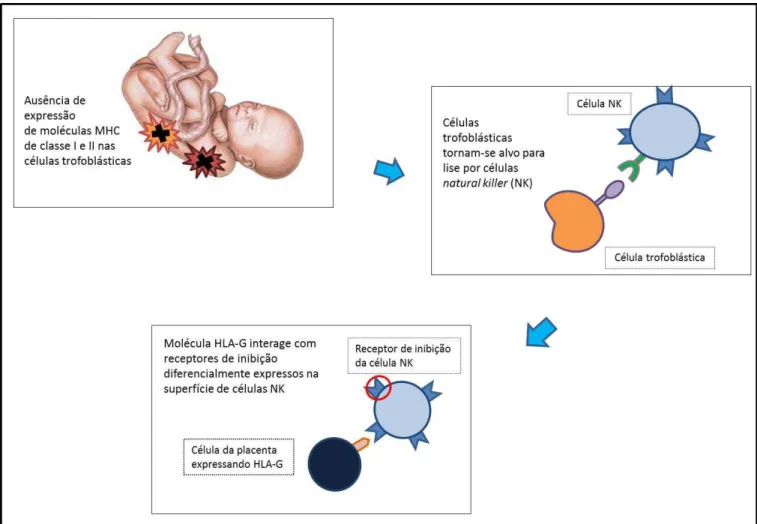
Figura 1 - Interação entre as moléculas HLA-G, expressas pelas células placentárias, e receptores inibitórios das células natural killer. A figura também ilustra a inibição das moléculas MHC classe I e II pelas células fetais ...
23
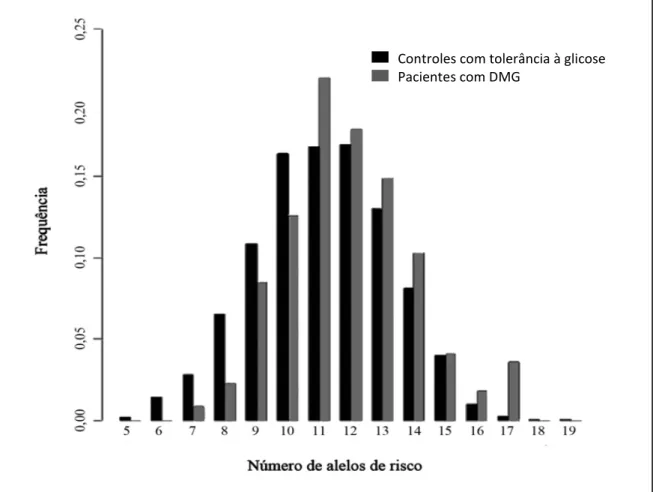
Figura 2 - Distribuição dos alelos de risco para DM2 em pacientes com DMG e em controles com tolerância a glicose (LAUENBORG, 2009)... 24
Figura 3 - Estapas gerais de experimentos de microarrays para análise de transcriptomas. Figura baseada nos sites (http://www.microarray.lu/en/ MICROARRAY_Overview.shtml) e (http://en.wikipedia.org/wiki/File: Heatmap.png) ... 27
Figura 4 - Densintometria da amostra de RNA de paciente com DMG isolada de PBMCs. RNA integrity number (RIN) = 9,0 ... 36
Figura 5 - Representação das reações de transcrição reversa, amplificação, purificação e hibridação de microarray da plataforma Agilent. Adaptado de (“G4140- 90040_GeneExpression_One-color_v6.5.pdf (objeto application/pdf)”... 38
Figura 6 - Delineamento experimental... 42
Figura 7 - Pipeline para análises de bioinformática dos dados de microarrays... 42
Figura 8 - Gráficos boxplot dos dados de microarrays utilizados neste trabalho após normalização a partir da metodologia quantile... 45
Figura 9 - Agrupamento hierárquico a partir da correlação de Pearson. Em destaque a separação entre os grupos de pacientes e controles... 46
Figura 10 - Heatmap ilustrativo das categorias identificadas a partir da ferramenta de análise funcional DAVID. Em destaque os processos mais significativos. A escala em cinza representa o valor de P em módulo logarítmico... 48
Figura 11 - Principais vias identificadas a partir da análise funcional usando DAVID. A escala em cinza representa o valor de P em módulo logarítmico... 49
Figura 12 - Representação do perfil de expressão dos mRNAs envolvidos com metabolismo entre pacientes com DMG e controles saudáveis. A figura ilustra os valores de fold change (FC) para cada transcrito e destaca a modulação positiva para IGFBP2 e TCF3 e negativa para OLR1 e TCF7L2... 53
Figura 14 - Gráficos com os valores de expressão gênica relativa (RQ) dos genes CXCL2 (A), NFKBIA (B) (Análise estatística teste t; P < 0.05). ... 56
LISTA DE TABELAS
Tabela 1 - Classificação etiológica do Diabetes Mellitus... 15
Tabela 2 - Características clínicas dos controles utilizados neste trabalho ... 33
Tabela 3 - Características clínicas das pacientes com DMG ... 34
Tabela 4 - Processos biológicos de maior relevância para este estudo. Em destaque os genes que fazem parte de cada processo, o valor do P-value e o código do Gene Ontology... 50
Tabela 5 - Vias biológicas de maior relevância para este estudo. Em destaque a categoria e os genes de cada via e o valor do P-value... 51
Tabela 6 - Oligonucleotídeos primers dos genes humanos utilizados nas reações de PCR... 55
SUMÁRIO
1 INTRODUÇÃO ...15
1.1 Definição e classificação do Diabetes Mellitus...15
1.2 Epidemiologia do Diabetes Mellitus...17
1.3 Diabetes Mellitus Gestacional ...18
1.4 Patogênese do Diabetes Mellitus Gestacional ...19
1.5 Imunogenética da gravidez...21
1.6 Genética do Diabetes Mellitus Gestacional ...23
1.7 Análise de transcriptomas pela tecnologia de microarrays...25
2 HIPÓTESE...29
3 OBJETIVOS ...31
3.1 Objetivo Geral ...31
3.2 Objetivos Específicos ...31
4 MATERIAL E MÉTODOS ...33
4.1 População de estudo ...33
4.2 Coleta de sangue e separação de células mononucleares de sangue periférico (PBMCs) de pacientes e controles ...34
4.3 Extração de RNA total e avaliação da integridade ...35
4.4 Reação de hibridação com oligo-microarrays...36
4.5 Análise dos dados de microarrays...39
4.5.1 Quantificação e normalização dos dados ...39
4.5.2 Análise estatística ...40
4.5.3 Análise funcional ...40
4.6 Confirmação dos dados de microarrays por PCR em tempo real ...41
4.7 Análise estatística dos dados de PCR em tempo real ...41
5 RESULTADOS ...44
5.1 Análise dos Dados de Microarrays...44
5.2 Análise funcional a partir da plataforma DAVID ...47
5.3 Perfil de expressão de genes envolvidos com metabolismo e componentes do MHC no DMG...52
6 DISCUSSÃO ...59
7 CONCLUSÕES...70
8 REFERÊNCIAS BIBLIOGRÁFICAS ...72
Introdução | 15
1
INTRODUÇÃO
1.1 Definição e classificação do Diabetes Mellitus
De acordo com a American Diabetes Association (ADA), o Diabetes Mellitus (DM) corresponde a um grupo de doenças metabólicas caracterizadas por hiperglicemia resultante de defeitos na secreção e/ou na ação da insulina. Vários processos patogênicos estão envolvidos no desenvolvimento do DM (ADA, 2010).
Os sintomas clínicos da resistência insulínica incluem reações de: poliúria, polidipsia, perda de peso, na maioria das vezes com polifagia ou ainda complicações agudas que podem levar a risco de vida. A hiperglicemia crônica está associada a dano, disfunção e falência de vários órgãos, especialmente olhos, rins, nervos, coração e vasos sanguíneos (GROSS et al., 2002).
A classificação do DM proposta pela Organização Mundial de Saúde (OMS) (ALBERTI; ZIMMET, 1998) inclui quatro classes clínicas de diabetes: Diabetes Mellitus tipo 1 (DM1), Diabetes Mellitus tipo 2 (DM2), outros tipos específicos de diabetes mellitus e Diabetes Mellitus Gestacional (DMG) (Tabela 1). Ainda existem duas categorias, a glicemia de jejum alterada e a tolerância à glicose diminuída. Essas categorias não são entidades clínicas, mas fatores de risco para o desenvolvimento do DM.
Introdução | 16
O DM1 tem início em indivíduos jovens sendo também denominado diabetes juvenil, embora possa se desenvolver em qualquer idade. É uma doença crônica autoimune que ocorre em indivíduos geneticamente susceptíveis (ATKINSON; EISENBARTH, 2001). O sistema imunológico do próprio corpo ataca as células betas das ilhotas de langerhans do pâncreas, destruindo-as reduzindo e eventualmente eliminando a produção de insulina (VAN BELLE; COPPIETERS; VON HERRATH, 2011).
Um locus crítico de susceptibilidade para doenças autoimunes em humanos, incluindo DM1, é a região HLA (antígeno leucocitário humano) no cromossomo 6p21 (NERUP et al., 1974). Inúmeros novos loci de susceptibilidade foram identificados desde então, mas nenhum deles coincide com a forte associação encontrada com a região HLA (CONCANNON; RICH; NEPOM, 2009).
O diabetes autoimune é raramente causado por defeitos mutacionais em um único gene. As formas monogênicas são geralmente acompanhadas de várias outras doenças autoimunes, devido ao rompimento de vias regulatórias comuns. Um exemplo são as mutações que ocorrem no fator de transcrição AIRE (regulador autoimune), que desencadeiam doenças autoimunes graves, nas quais em 20% dos casos há desenvolvimento de DM1 (VILLASEÑOR; BENOIST; MATHIS, 2005).
O DM2 representa 90-95% de todos os tipos de diabetes. A maioria dos pacientes com DM2 é obesa e a obesidade causa algum grau de resistência à insulina (ADA, 2010). Existem várias e diferentes causas para essa forma de diabetes, incluindo fatores genéticos e ambientais (GALINDO et al., 2009). Na maioria dos casos, o DM2 se comporta de acordo com o modelo de herança multifatorial ou poligênico (AGUIAR e SILVA, 2009), embora também sejam relatados casos de DM2 monogênico.
Na forma poligênica do DM2 vários genes atuam em fenótipos intermediários do diabetes que influenciam a homeostase glicêmica. Cada gene apresenta individualmente um efeito limitado, não podendo sozinho levar ao desenvolvimento da doença. Porém quando transmitidos juntos, e na presença de fatores ambientais desfavoráveis, podem ser expressos clinicamente. Os alelos de risco dos poligenes do diabetes podem ser raros ou apresentar alta prevalência na população. Sendo assim, grande parcela dos indivíduos pode ser susceptível ao diabetes se ocorrerem alterações nos hábitos de vida. Esta observação poderia justificar o aumento dos casos de obesidade e diabetes nas populações que tiveram seu estilo de vida alterado nas últimas décadas (REIS; VELHO, 2002).
Introdução | 17
em um só gene transmitida de forma autossômica-dominante é suficiente para promover a hiperglicemia (REIS; VELHO, 2002). A forma monogênica mais frequente do DM2 é Maturity Onset Diabetes of the Young (MODY). O fenótipo dos pacientes MODY é
caracterizado por hiperglicemia crônica de origem não autoimune. As formas mais graves acarretam desenvolvimento de complicações crônico-degenerativas semelhante ao DM2 clássico de início tardio (REIS; VELHO, 2002).
O DMG consiste na intolerância a glicose com início durante a gravidez (SHAAT et al., 2005). É associado tanto a resistência insulínica quanto a diminuição da função das células beta pancreáticas. O DMG será abordado em mais detalhes nas próximas sessões.
1.2 Epidemiologia do Diabetes Mellitus
No Brasil, um estudo multicêntrico realizado em nove capitais em 1988 demonstrou prevalência do diabetes em 7,6% dos indivíduos na faixa etária entre 30 e 69 anos. Neste mesmo estudo foi observado que 2,7% dos indivíduos na faixa etária de 30 a 59 anos apresentaram DM, contrastando com 17,4% para a de 60-69 anos, o que caracteriza um aumento de 6,4 vezes (MALERBI; FRANCO, 1992). Em outra pesquisa de rastreamento do diabetes, foi demonstrada a prevalência do diabetes em 11% para pessoas acima de 40 anos (Ministério da Saúde, 2001). Dados mais recentes apontam para taxas mais elevadas, como 12,1%, em um estudo desenvolvido em Ribeirão Preto, cidade de porte médio do estado de São Paulo (TORQUATO et al., 2003).
Entre os principais tipos de DM, o DMG é o distúrbio metabólico mais comum na gravidez (GUEUVOGHLANIAN-SILVA et al., 2012). Segundo a ADA (2010), 7% das gestantes apresentam DMG, o que representa mais de 200.000 casos por ano. A prevalência é de 1% a 14% do total de gestantes variando de acordo com a população avaliada e com o protocolo de diagnóstico aplicado. Algumas populações consideradas de alto risco mostram prevalências superiores a essa faixa, um exemplo é a prevalência no Qatar (16,3%), conhecida como uma das maiores do mundo, (BENER; SALEH; AL-HAMAQ, 2011).
Dados do Estudo Brasileiro de Diabetes Gestacional (EBDG) apontam prevalência de 7,6% de DMG no Brasil, sendo que 94% dos casos apresentam apenas tolerância diminuida à glicose e 6% hiperglicemia no nível do diabetes (Ministério da Saúde, 2001).
Introdução | 18
síndrome metabólica (GUEUVOGHLANIAN-SILVA et al., 2012), sendo considerado um problema de saúde pública.
1.3 Diabetes Mellitus Gestacional
A gravidez é um período progressivamente hiperglicêmico da vida, acompanhado pelo aumento da resistência à insulina a partir da metade da gestação. A hiperglicemia tem papel importante na nutrição e desenvolvimento do feto, fornecendo-lhe níveis adequados de glicose (VRACHNIS et al., 2012). O DMG é uma condição em que mulheres grávidas sem diagnóstico prévio de diabetes mellitus apresentam altos níveis de glicose no sangue (RAY et al., 2010).
No DMG, o quadro de hiperglicemia está associado com aumento da morbi-mortalidade fetal, caracterizada por alta frequência de malformações, macrossomia, hiperbilirrubinemia, policitemia, hipocalcemia, hipomagnesemia, cardiomiopatia hipertrófica e síndrome do desconforto respiratório do recém-nascido (CROWTHER et al., 2005). O estudo multicêntrico Hyperglycemia and Adverse Pregnancy Outcomes mostrou que a hiperglicêmia tem efeito significativo na gravidez, além de associação com peso aumentado do infante ao nascimento e complicações (MULLA; HENRY; HOMKO, 2010). O descontrole glicêmico durante a gestação está relacionado com doença hipertensiva específica da gravidez, indicação de cesáreas e hipertensão arterial.
Entre os fatores de risco sociodemográficos e ambientais que contribuem para o desenvolvimento do DMG, estão incluídos idade materna avançada, aumento do tecido adiposo materno, tabagismo e estilo de vida sedentário (GUEUVOGHLANIAN-SILVA et al., 2012). Sendo assim, as complicações para o feto e o risco aumentado da gestante permanecer diabética ou desenvolver a doença no futuro, tornam imperativa a necessidade de prevenção e controle eficaz (CROWTHER et al., 2005).
Introdução | 19
A maioria das mulheres com DMG é obesa, apresenta incapacidade funcional das células beta-pancreáticas e resistência à insulina (LAUENBORG et al., 2009). Algumas destas parecem ter uma predisposição genética para desenvolver intensa resposta inflamatória, o que pode aumentar o risco de desenvolver DMG (GUEUVOGHLANIAN-SILVA et al., 2012).
Evidências sugerem que duas vias principais podem desencadear o DMG: resistência à insulina e inflamação, além disso, a obesidade apresenta papel importante no desenvolvimento desta condição (VRACHNIS et al., 2012). Ambos DMG e obesidade são condições que têm em comum um estado de inflamação crônica subclínica, caracterizado pela produção anormal de citocinas e ativação de vias de sinalização relacionadas a inflamação (DAHER et al., 2011).
O aumento dos níveis de mediadores inflamatórios durante e após a gravidez é relatado em pacientes com DMG. Da mesma forma, o aumento da gordura corporal tem sido fortemente associado com inflamação, necrose dos adipócitos, hipóxia e liberação de quimiocinas (VRACHNIS et al., 2012). Um possível mecanismo patogênico partilhado tem sido atribuído aos macrófagos que secretam citocinas e ativam a produção de mediadores de inflamação. Como resultado, o tecido adiposo, torna-se infiltrado com macrofagos (VRACHNIS et al., 2012).
1.4 Patogênese do Diabetes Mellitus Gestacional
A necessidade de insulina é alta durante a gravidez normal e difere apenas ligeiramente entre mulheres saudáveis e diabéticas. No entanto, mulheres com DMG mostram consistentemente respostas reduzidas a este hormônio. A condição fisiológica do DMG é representada basicamente em função do aumento da resistência insulínica ou pela diminuição da sensibilidade a este hormônio em decorrência de um defeito na função pancreática das células beta, comumente encontrado em mulheres com DMG (OZTEKIN, 2007; KAAJA; RÖNNEMAA, 2008).
Introdução | 20
estão aumentadas durante a gravidez, a inflamação tem sido considerada fator determinante no desenvolvimento do DMG (DAHER et al., 2011) .
Os defeitos na função das células beta do pâncreas em mulheres com DMG podem ser decorrentes da destruição autoimune destas células, como ocorre no DM1. Esta destruição é caracterizada pela circulação de receptores dirigidos contra as ilhotas pancreáticas (anticorpos anti-células das ilhotas) ou antígenos de células beta (tais como descarboxilase do ácido glutâmico, GAD, ou autoanticorpos contra insulina, IAA). Essas pacientes parecem ter evolução para o DM1 (KAAJA; RÖNNEMAA, 2008). Os anticorpos anti-células das ilhotas ou anti-GAD estão presentes em menos de 10% das pacientes DMG. Essas mulheres podem desenvolver rapidamente diabetes após a gravidez. Outra causa para uma função beta-celular com defeito no DMG são mutações em autossomos, com subtipos genéticos denotados como MODY-1, MODY-2, entre outros. Mutações que causam subtipos MODY têm sido encontradas
em mulheres com DMG (BUCHANAN; XIANG, 2005)
Outra condição recentemente associada à patogênese do DMG aponta para defeitos pós-receptor presentes na via de sinalização da insulina na placenta de mulheres com gestações complicadas pelo diabetes e pela obesidade (COLOMIERE et al., 2009). Alterações na via de sinalização da insulina, localização anormal de transportadores GLUT4, diminuição da expressão de PPARy e transporte reduzido de glicose mediado por insulina já foram evidenciados no músculo esquelético e em células adiposas de mulheres com DMG (BUCHANAN; XIANG, 2005).
Embora os mecanismos patofisiológicos detalhados que levam ao DMG sejam ainda desconhecidos, alguns polimorfismos e a função alterada de determinados genes são os principais responsáveis pelo controle do metabolismo celular e da ação da insulina, podendo influenciar no desenvolvimento da doença (OZTEKIN, 2007). Tem sido proposto que os eventos que conduzem ao desenvolvimento do DMG são acionados por uma carga antigênica que é própria do feto. A expressão do antígeno leucocitário humano-G (HLA-G), que protege o feto do ataque imune através da baixa regulação das respostas de células T citotóxicas que estariam dirigidas a antígenos do trofoblasto fetal, é necessária para proteger as células das ilhotas pancreáticas. A interação entre HLA-G e o fator nuclear-kB (NF-kB), é sugerida como essencial nos eventos que levam ao desenvolvimento de DMG (KAAJA; RÖNNEMAA, 2008).
Introdução | 21
Adicionalmente, fatores humorais e neurais que incluem alterações de catecolaminas, cortisol e hormônios sexuais também são relevantes no desenvolvimento desta doença (OZTEKIN, 2007).
1.5 Imunogenética da gravidez
Na gravidez existe um contato próximo entre o sistema imune materno, células e tecidos fetais. O corpo feminino passa por diversas alterações em seu perfil imunológico para se adaptar à presença do feto. Sendo assim, mecanismos específicos devem atuar para modular o sistema imune materno, impedindo a rejeição do feto. Alterações nestes mecanismos podem gerar complicações durante a gestação (HVIID, 2006).
Alguns trabalhos têm relatado alterações imunológicas que ocorrem durante o período gestacional, incluindo aquelas envolvidas na tolerância ao feto. (ALUVIHARE; KALLIKOURDIS; BETZ, 2004; TROWSDALE; BETZ, 2006).
Os diversos fatores imunológicos que atuam na manutenção da homeostase durante a gestação incluem: o perfil de produção de citocinas do tipo Th2 (MARZI et al., 1996), a presença de células T regulatórias (SAITO; SASAKI; SAKAI, 2005) e a ação imunossupressora da molécula HLA-G (HVIID, 2006). Além disso, para que não haja rejeição, é essencial a ausência da expressão de moléculas MHC (Major Histocompatibility Complex) de classe I e II nas células trofoblásticas.
As duas principais populações efetoras de células T auxiliares regulam-se mutuamente e dividem-se em subpopulações: Th1 e Th2. As células Th1 produzem principalmente IFN- e IL-2 e desempenham papel nas respostas imunes celulares contra patógenos intracelulares. Já
as células Th2 secretam IL-4, IL-5 e IL-10 estando envolvidas na imunidade humoral (MOSMANN; SAD, 1996). Alguns estudos mostram que um padrão efetor Th2 está associado ao sucesso gestacional e que a manutenção de um padrão Th1 é prejudicial para a gravidez (MAKHSEED et al., 2001; CLARK; CROITORU, 2001).
Outro ponto importante consiste no fato das células T regulatórias atuarem em um papel crítico na tolerância materna ao feto (SAITO; SASAKI; SAKAI, 2005). O sistema imune fetal evita o ataque pelas células maternas que atravessam a placenta, através da ação de células T regulatórias capazes de reconhecer as células maternas (VIANNA, 2009).
Introdução | 22
As moléculas HLA são divididas nas classes I e II. As moléculas de classe I são expressas na maioria dos tipos celulares e apresentam antígenos endógenos para células T citotóxicas. Em humanos, a classe I de HLA é subdividida na classe Ia, denominada clássica e constituída pelas moléculas HLA-A, B e C e pela classe Ib, descrita como não-clássica e representada pelas moléculas HLA-E, F e G (NOLAN, 2008).
Introdução | 23
1.6Genética do Diabetes Mellitus Gestacional
Alguns loci associados a DM2 (KCNJ11, TCF7L2, CDKAL1, KCNQ1, CDKN2A/CDKN2B, HHEX / IDE, IGF2BP2, SLC30A8, TCF2, FTO) já foram relacionados
com o risco de desenvolver DMG, o que não é surpreendente, dado os precedentes comuns entre estes dois tipos de diabetes (LAUENBORG et al., 2009; SHAAT et al., 2005) (Figura 2). Porém, os achados relativos ao DMG são muitas vezes baseados em amostras pequenas em comparação a amostragem utilizada nos estudos para detectar novos marcadores de risco ao DM2 (WATANABE et al., 2007).
Introdução | 24
O efeito aditivo significativo de múltiplos alelos de risco ao DMG já foi relatado (LAUENBORG et al., 2009). Nesse estudo não foram identificados indivíduos com menos de cinco ou mais de 19 alelos de risco. Na maioria dos casos, semelhante aos outros tipos de diabetes, o risco genético para o DMG é estimado a partir de pequenas contribuições de diferentes genes em populações distintas, levando em consideração as prevalências dos alelos de risco na população (LAUENBORG et al., 2009). Embora, algumas mutações gênicas que causam a forma monogênica do diabetes também tenham sido encontradas em mulheres com DMG, é provável que nesta doença exista um fator de risco poligênico (BUCHANAN; XIANG, 2005; LAUENBORG et al., 2009).
O DMG mostra uma predisposição genética para o posterior desenvolvimento de DM1 e DM2, embora comumente evolua para o DM2, especialmente se outros fatores de risco, tais como a obesidade, estiverem presentes (MARTIN et al., 1985). No entanto, estudos mostram genes em comum envolvidos com o desenvolvimento de DM1 e DMG (RADAELLI et al., 2009; MCLELLAN et al., 1995; DÖRNER; PLAGEMANN; REINAGEL, 1987).
Um estudo global de expressão gênica, a partir de amostras de placenta, relatou a indução de genes envolvidos com resposta imune em mulheres com DMG
Controles com tolerância à glicose Pacientes com DMG
Introdução | 25
(ENQUOBAHRIE; WILLIAMS; QIU; MELLER; et al., 2009). Outro trabalho revelou genes de inflamação induzidos em gestantes com DMG, fornecendo a base para elucidar a relação entre vias inflamatórias e DMG, associadas com resistência à insulina (RADAELLI et al., 2003). Vias e categorias imunes significativas relacionadas ao DMG foram identificadas a partir da análise de amostras de sangue total e de placenta em mulheres com DMG (ZHAO et al., 2011). Assim, estes achados sugerem que o DMG pode ter fatores genéticos predisponentes comuns com DM1.
Uma evidência importante consiste no fato de que uma forma autoimune do diabetes é estimada em aproximadamente 10% das mulheres com DMG (LAPOLLA; DALFRÀ; FEDELE, 2009). Estudos sugerem que alguns antígenos HLA são mais frequentes no DMG (RUBINSTEIN et al., 1981).
Outro ponto importante é a relação entre alguns fatores genéticos relacionados ao feto associados com variações nas concentrações de glicose materna durante a gravidez. Uma pesquisa relatou a presença de maiores concentrações de glicose materna em mulheres grávidas de gêmeos do que naquelas que esperam uma criança (SCHWARTZ et al., 1999).
Um trabalho de estratificação de gestações de acordo com o sexo da prole identificou um aumento da incidência de DMG em mulheres que geram meninos, possivelmente devido a maior dieta energética na gestação de crianças do sexo masculino (TAMIMI et al., 2003).
Em resumo, o estudo da genética do DMG é dificultado por alguns fatores, como por exemplo as causas heterogêneas da doença e os critérios para o diagnóstico desta condição (LAPOLLA; DALFRÀ; FEDELE, 2009). Sendo assim, é necessária uma pesquisa mais abrangente empregando amostras significativas de pacientes diagnosticadas com DMG. Além disso, é importante agregar estudos de associação genômica em larga escala (GWAS) e de expressão gênica transcricional utilizando microarrays e RNA-Seq. Com isso, será possível estabelecer associações entre genes e DMG com maior poder estatístico.
1.7 Análise de transcriptomas pela tecnologia de microarrays
Introdução | 26
de genes e processos biológicos envolvidos na doença (PLANAS; PUJOL-BORRELL; VIVES-PI, 2010).
O transcriptoma compreende o conjunto de RNAs expressos em uma célula ou tecido em uma dada situação, incluindo transcritos alternativos e não-codificantes, cujas alterações refletem as condições fisiológicas ou patológicas em que o organismo se encontra (RUAN et al., 2004).
Alguns trabalhos têm mostrado que certos loci de susceptibilidade a doenças autoimunes, como o lúpus eritematoso sistêmico (TREVISAN et al., 2006), a artrite reumatóide (SILVA et al., 2007) e o DM1 (RASSI et al., 2008) são na realidade regiões cromossômicas que transcrevem mRNA.
A tecnologia dos microarrays permite a obtenção de um fingerprint molecular da expressão gênica (GROUSE; MUNSON; NELSON, 2001). Genes que são expressos de forma coordenada durante uma resposta biológica, compõem as chamadas “assinaturas de expressão gênica”(HASELTON; LLOYD; JOHNSON, 2000).
Os microarrays representam uma alternativa prática na análise da expressão gênica. Baseiam-se na ligação por complementaridade de bases de RNAs fita simples aos fragmentos de DNA, RNA ou oligonucleotídeos depositados em uma lâmina, (BRAZMA; VILO, 2001). Esta técnica tem sido largamente aplicada em biologia e medicina na aquisição de novos conhecimentos básicos e também na área diagnóstica. Com os microarrays é possível medir a expressão de milhares de genes em um único experimento por isso, este método passou a ser a escolha na exploração do transcriptoma em diversas situações normais e patológicas. A figura 3 ilustra o princípio geral da técnica.
Os experimentos com microarrays geram uma lista de genes diferencialmente expressos. A análise minuciosa desta lista proporciona grande quantidade de informações relevantes. A grande vantagem desta técnica consiste na possibilidade de atribuir novas funções a genes já descritos.
Além das assinaturas de expressão, muito úteis para o agrupamento de amostras e/ou de pacientes, as análises dos dados de microarrays também têm sido focadas na reconstrução de redes transcricionais regulatórias, ou seja, as interações entre moléculas de mRNAs. Tais redes visam encontrar e entender a interação gene-gene a partir de dados de expressão transcricional (WANG et al., 2006).
Introdução | 27
foram evidenciados diversos genes modulados relacionados à morfogênese, matriz extracelular, adesão celular, ligação de cálcio, ciclo celular, entre outros (DHAHBI et al., 2003; VAN LUNTEREN; MOYER, 2007).
Wang et al (2008) demonstraram que soros de pacientes diabéticos possuem fatores que induzem assinatura pró-inflamatória única em PBMCs refletindo auto-imunidade ativa. De fato, pesquisas conduzidas com PBMCs, sangue total e placenta empregando a tecnologia de microarrays, têm demonstrado padrões distintos de expressão gênica entre mulheres saudáveis e com DMG (ZHAO et al., 2011; RADAELLI et al., 2003; RADAELLI et al., 2009; ENQUOBAHRIE; WILLIAMS; QIU; MUHIE; et al., 2009).
Tais exemplos indicam a possibilidade de atribuir funções a regiões do genoma previamente associadas com DMG empregando a tecnologia dos microarrays e análise de transcriptomas.
Hipótese | 29
2 HIPÓTESE
Objetivos | 31
3 OBJETIVOS
3.1 Objetivo Geral
O objetivo geral deste trabalho foi descrever o perfil de expressão gênica transcricional (mRNA) de células mononucleares de sangue periférico (PBMCs) de pacientes com Diabetes Mellitus Gestacional focando em genes envolvidos com o processo inflamatório.
3.2 Objetivos Específicos
• Utilizar a tecnologia de oligo-microarrays para avaliar o perfil de expressão gênica transcricional das PBMCs de pacientes com Diabetes Mellitus Gestacional em comparação com gestantes saudáveis, focando em genes relacionados à inflamação;
• Fazer uso de programas de bioinformática dedicados à análise dos dados de microarrays. Utilizando o ambiente R, visamos estabelecer os perfis de expressão
gênica transcricional, a normalização dos dados e o teste estatístico; a partir dos programas Cluster e TreeView pretendemos realizar agrupamentos hierárquicos das pacientes, controles e mRNAs;
Material e Métodos | 33
4 MATERIAL E MÉTODOS
4.1 População de estudo
No presente estudo foram selecionadas 18 pacientes com DMG, com idade entre 19 e 40 anos (média = 31,5) diagnosticadas e acompanhadas na Divisão de Endocrinologia do Hospital das Clínicas da Faculdade de Medicina de Ribeirão Preto, Universidade de São Paulo (USP), segundo os critérios da Associação Americana de Diabetes (ADA). Pacientes que exibiram histórico de diabetes ants da gestação, doença autoimune, hipertensão e síndrom metabólica foram excluidas do estudo. Como grupo controle, foram selecionadas 10 gestantes sem antecedentes de DMG, exibindo níveis normais de glicose e pareadas com as pacientes em termos de idade, número de gestações e idade gestacional (Tabelas 2 e 3), a coleta foi realizada no Hospital Santa Casa de Misericórdia de Ribeirão Preto, com autorização concedida pela diretoria clínica do hospital (Anexo A). A cópia do termo de consentimento livre-esclarecido foi obtida de todas as pacientes e controles (Anexo B), e o protocolo de estudo foi aprovado pelo Comitê de Ética Local (Protocolo # 9153/2008) (Anexo C). Entre as grávidas não diabéticas que compõem o grupo controle, selecionamos aleatoriamente seis para avaliar o perfil transcricional do pool em duplicata. O presente trabalho é parte integrante do Projeto Temático FAPESP nº 08/56594-8 sob coordenação do Prof. Dr. Geraldo A. S. Passos Junior.
Tabela 2. Características clínicas dos controles utilizados neste trabalho.
Controle Idade (anos) Período gestacional (semanas) Número de gestações Glicemia
(mg/dL) HbA1c (%)
C_1 a 27 41 2 74 ---
C_2 a* 37 36 1 67 ---
C_3 a 21 38 1 72 ---
C_4 a 35 32 4 79 ---
C_5 a 26 37 3 82 ---
C_6 a 23 38 1 65 ---
C_7 20 39 1 82 ---
C_8 26 37 1 81 ---
C_9 19 38 1 78 ---
C_10 26 36 2 60 ---
C_11 19 36 2 66 ---
a
Controles que compõem o pool; a* Controle não marcado individualmente, usado
Material e Métodos | 34
Tabela 3. Características clínicas das pacientes com DMG.
Paciente Idade (anos)
Período gestacional
(semanas)
Número de gestações
Glicemia
(mg/dL) HbA1c (%)
DMG_1 35 30 1 88 5,6
DMG_2b 37 25 1 86 7,9
DMG_3 30 26 2 67 5,1
DMG_4 40 38 4 74 ---
DMG_5 35 27 1 94 8,8
DMG_6 29 34 1 65 5,7
DMG_7 24 29 1 74 5,4
DMG_8 23 34 2 86 5,4
DMG_9 33 35 2 80 5,0
DMG_10b 29 37 2 89 5,8
DMG_11 37 37 4 80 ---
DMG_12 39 32 4 72 5,7
DMG_13b 29 28 4 92 5,2
DMG_14 30 34 3 82 5,0
DMG_17 28 34 2 81 6,1
DMG_18 22 34 1 90 5,4
DMG_19 34 34 2 99 6,5
DMG_20 31 37 1 128 6,2
b
Pacientes que utilizam insulina.
4.2 Coleta de sangue e separação de células mononucleares de sangue periférico (PBMCs) de pacientes e controles
Foram coletados aproximadamente 20 mL de sangue venoso periférico de cada paciente e de cada controle em tubos contendo EDTA (1,8 mg/mL) (Sigma, St. Louis, MO, USA). O processamento do sangue foi realizado logo após a coleta sendo utilizado o protocolo descrito por Böyum (1968). O isolamento das células mononucleares foi realizado por centrifugação em gradiente de densidade utilizando Ficoll-Hypaque (GE Healthcare, Piscataway, NJ, USA).
Material e Métodos | 35
Ficoll-Hypaque na proporção (2:1). As amostras foram centrifugadas por 15 minutos a 1042 x
g – RCF (2300 rpm) à temperatura ambiente. Após a centrifugação, foi obtido um anel de células mononucleares, localizado na interface do Ficoll-Hypaque e do plasma sanguíneo. Estas células foram colhidas com auxílio de pipeta Pasteur e submetidas a duas lavagens com salina 0,9% estéril, sendo o pellet obtido ressuspenso em salina 0,9% tratado com dietilpirocarbonato (DEPC) (Sigma-Aldrich Brasil Ltda, São Paulo, Brasil). Em seguida, as células separadas de pacientes e controles foram submetidas à extração de RNA total.
4.3 Extração de RNA total e avaliação da integridade
As amostras de RNA total obtidas de PBMCs foram extraídas usando o reagente Trizol (Life Technologies, Carlsbad, CA, USA). Foi utilizado 1 mL do reagente para cada 5 mL de sangue. O RNA obtido foi suspenso em água tratada com DEPC (Sigma-Aldrich Brasil Ltda, São Paulo, Brasil) e armazenado em freezer a -80°C.
A partir da avaliação das absorbâncias e do cálculo das razões, usando espectrofotômetro NanoDrop ND-1000 (NanoDrop Products, Wilmington, DE, USA), foram obtidas as quantificações e o grau de pureza das extrações. Foram utilizadas apenas amostras de RNA livres de contaminação protéica (A260/A280 entre 1,8 – 2,2) e de fenol (A260/A220 entre 1,8 – 2,0).
Material e Métodos | 36
4.4 Reação de hibridação com oligo-microarrays
Para análise do transcriptoma das pacientes com DMG e controles foram utilizadas lâminas de microarrays Whole Human Gene Expression Microarrays Kit (4x44 K) com AMADID 014850 (Agilent Technologies, Santa Clara, CA, USA). Foi utilizada a metodologia monocolor Cy3 (cianina 3). Para a amplificação, marcação e hibridação dos mRNAs das amostras com os microarrays foi utilizado o Kit Agilent Quick Amp Labeling (Agilent Technologies, Santa Clara, CA, USA), seguindo as recomendações do fabricante. As etapas do procedimento de microarrays foram realizadas no Laboratório de Imunogenética Molecular no Departamento de
Genética da Faculdade de Medicina de Ribeirão Preto, Universidade de São Paulo (USP), Brasil. As etapas do processo estão esquematizadas na figura 5. As amostras de RNA controle (spike-in) foram misturadas a 500 ng de RNA total para transcrição reversa com utilização da
Material e Métodos | 37
enzima Moloney Murine Leukemia Virus – Reverse Transcriptase (MMLV-RT). Foram utilizados primers adaptados com timinas consecutivas acopladas a um promotor T7 para síntese de cDNAs dupla fita. Após essa reação, a enzima T7 RNA Polimerase foi adicionada com nucleotídeos marcados com o fluorocromo Cy3 para síntese de cRNAs e posterior amplificação.
Foi realizada a purificação das amostras de cRNAs, que em seguida foram submetidas à hibridação às lâminas de microarrays, utilizando-se câmaras de hibridação Agilent Microarray Hybridization Chambers (Agilent Technologies, Santa Clara, CA, USA). Posteriormente, as
lâminas foram incubadas em forno rotativo (Agilent Technologies) a 65°C por 17 horas. Após a hibridação, as lâminas foram lavadas com os tampões GE Wash Buffer 1 e GE Wash Buffer 2 para em seguida ser feita a aquisição das imagens (scanning).
As imagens de hibridação foram obtidas a partir do scanning dos microarrays (Scanner Agilent Bundle®) por meio do software Feature Extraction (Agilent Technologies,
Material e Métodos | 38
Cy3
Cy3 Cy3
Cy3 Cy3
Figura 5. Representação das reações de transcrição reversa, amplificação, purificação e
hibridação de microarrays da plataforma Agilent. Adaptado de (“G4140-
Material e Métodos | 39
4.5 Análise dos dados de microarrays
A correção e a normalização dos dados foram realizadas utilizando ambiente R. A linguagem de programação R (“The R Project for Statistical Computing”) consiste de ambiente estatístico-matemático com inúmeros pacotes de funções múltiplas, amplamente utilizados em análises de dados de microarrays ( endereço: http://www.r-project.org/).
4.5.1 Quantificação e normalização dos dados
A quantificação dos dados e o controle de qualidade foram obtidos por meio do software Feature Extraction. A tabela de dados quantificados foi utilizada para seleção das
colunas correspondentes as sondas, a mediana dos dados brutos (gMedianSignal) e a mediana do background (gBGMedianSignal). Foram gerados arquivos com extensão “.new” para todas as amostras e controles. Esta etapa foi realizada utilizando o sistema operacional Linux.
Em seguida, esses arquivos foram introduzidos em ambiente R versão 2.13.1 para ajuste do background a partir da subtração (gMedianSignal - gBGMedianSignal). Foi realizada a conversão dos valores para escala logarítmica (log2) para posteriormente ser gerado um único arquivo com todas as amostras e controles apresentando valor de background corrigido. Após esta etapa foi calculada mediana dos dados, a fim de eliminar
problemas de múltiplas sondas para o mesmo transcrito, mantendo-se apenas um valor de expressão para cada mRNA. Estes procedimentos compreendem a etapa de pré-processamento dos dados.
Os dados gerados no pré-processamento foram submetidos à normalização por quantile utilizando o pacote aroma.light. Tal método é considerado o mais robusto, corrigindo
as diferenças nas densidades de probabilidade de todas as amostras (BOLSTAD et al., 2003). Para a verificação dos dados após a normalização foram criados gráficos em formato boxplots.
Material e Métodos | 40
4.5.2 Análise estatística
Para detecção dos mRNAs diferencialmente expressos foi utilizada a análise estatística Rank Products (RP), o significado da detecção é avaliado por um teste de permutação
não-paramétrico, associado ao P-value e a taxa de falsa descoberta (False Discovery Rates - FDR) (HONG et al., 2006).
O RP proporciona uma maneira simples, mas estatisticamente rigorosa para determinar o nível de significância para cada transcrito, baseando-se em listas classificadas de acordo com os valores de fold change. Atualmente este teste é considerado um dos mais robustos para análise estatística de dados de microarrays. Além disso, a abordagem do RP é poderosa na identificação de alterações de expressão biologicamente relevantes (BREITLING et al., 2004). Essas evidências indicam que mesmo mRNAs com níveis de expressão relativamente baixos são considerados na análise, devido sua relevância biológica no estudo.
Neste trabalho, foram considerados mRNAs com valor de P < 0.01, sendo o teste aplicado de forma pareada, ou seja, DMG versus controle. Os transcritos significativos e diferencialmente expressos obtidos por RP foram submetidos ao agrupamento hierárquico de Pearson utilizando o software Cluster 3.0 (http://rana.lbl.gov/EisenSoftware .htm) (EISEN et
al., 1998). O arquivo gerado em extensão “.cdt”, foi usado como arquivo de entrada no programa TreeView (http://rana.lbl.gov/EisenSoftware .htm) (EISEN et al., 1998) que constrói dendrogramas com as representações espaciais dos genes.
4.5.3 Análise funcional
A lista dos mRNAs diferencialmente expressos obtida por RP foi submetida à análise de enriquecimento funcional a partir da ferramenta de anotação DAVID (Database for Annotation, Visualization, and Integrated Discovery) versão 6.7., que integra dados de
diversos bancos e está disponível no endereço: http://david.abcc.ncifcrf.gov.
Material e Métodos | 41
4.6 Confirmação dos dados de microarrays por PCR em tempo real
A PCR em tempo real foi realizada a partir da metodologia SYBR Green, de acordo com as instruções do fabricante (Applied Biosystems) utilizando-se o aparelho StepOne Real-Time PCR System (Applied Biosystems, USA).
A primeira etapa do processo consiste na avaliação da especificidade dos produtos de PCR a partir da análise de uma curva de dissociação do cDNA. É então realizada a quantificação absoluta de cada par de primer, incluindo um par que representa um gene constitutivo, neste trabalho utilizamos HPRT1. São feitas diluições seriadas 1:10 a 1:10000 de cDNA para calcular a eficiência da reação e, consequentemente, a qualidade dos primers. A reação consiste de amplificação seguida de dissociação.
Na segunda etapa foi realizada a quantificação relativa para avaliar a expressão dos transcritos estudados em cada uma das amostras e compara-las entre si. A reação final consiste em 20 µl, contendo 0,8 µl de cada primer, 1X solução SYBR Green e 1 µl de cDNA sintetizado a partir de uma quantidade padronizada de RNA total (2 µg de RNA). Cada primer foi desenhado a partir do software Primer3 (http://biotools.umassmed.edu/bioapps/ primer3_www.cgi).
4.7 Análise estatística dos dados de PCR em tempo real
A análise estatística dos dados de PCR foi realizada em ambiente R, utilizando o teste t não pareado (unpaired t test), os gráficos foram construídos por meio do software
Material e Métodos | 42
Plataforma R
Linux
DAVID
Figura 7. Pipeline para as análises de bioinformática dos dados de microarrays
Resultados | 44
5 RESULTADOS
5.1 Análise dos Dados de Microarrays
Após a obtenção dos dados de microarrays a partir do Software Feature Extraction, foi realizada a análise em ambiente R.
Para tornar as lâminas comparáveis e reduzir artefatos experimentais, foi realizada a normalização e a correção dos dados, com objetivo de obter valores de intensidade entre as lâminas em uma mesma escala. Na escolha do método de normalização, é fundamental que seja realizado um balanço dos níveis de intensidade ao longo dos experimentos, mantendo-se os efeitos biológicos investigados (DO; CHOI, 2006). A normalização foi realizada entre todos os pacientes e controles a partir do método quantile utilizando o pacote aroma.light, no qual os valores de expressão, em cada lâmina, são ordenados segundo a intensidade (ranked).
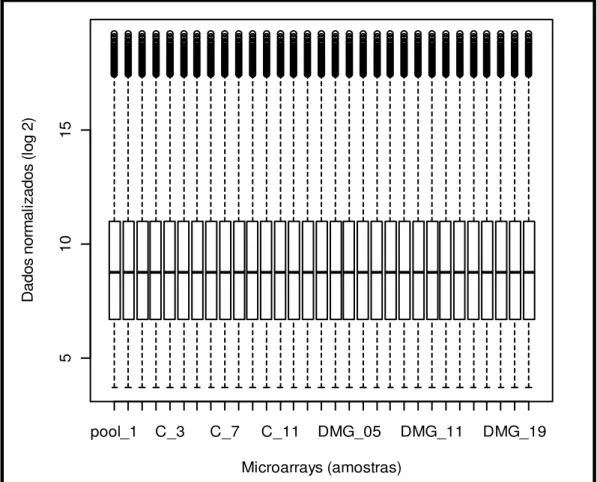
Os valores são substituídos pela média de intensidade obtida para determinada posição no ranking, obtendo-se um conjunto único de dados para todas as lâminas (BOLSTAD et al., 2003). Para essa normalização, é mais relevante a posição do gene no ranking do que a sua intensidade bruta. A figura 8 representa o gráfico boxplot que mostra as posições e comprimentos similares dos microarrays de todas as lâminas para todas as pacientes e controles avaliados.
Uma primeira análise foi realizada por agrupamento hierárquico onde foi evidenciada uma similaridade de acordo com o lote de hibridação em que cada amostra foi processada, e não com as variações biológicas. Reduzir o efeito “por lote de hibridação” foi um dos maiores desafios deste trabalho. Estes efeitos podem ser ocasionados por fatores diversos, incluindo os lotes de kits de amplificação utilizados, lotes das lâminas de marcação, hora que o experimento é realizado ou quando novas amostras são adicionadas a um conjunto de dados pré-existente, como aconteceu neste estudo.
Resultados | 45
Sendo removido o "efeito de lote" dos dados, foi possível observar o agrupamento das amostras de acordo com semelhanças biológicas. O heatmap final desta análise, proporcionando uma visão global dos dados, é ilustrado na figura 9.
Após a análise estatística por Rank Products, foram apontados 731 mRNAs considerados informativos (Anexo D). No agrupamento hierárquico, a partir da correlação de Pearson, esses mRNAs apresentaram-se bons marcadores na separação entre pacientes com
DMG e controles saudáveis, uma vez que houve a formação de dois grupos distintos.
pool_1 C_3 C_7 C_11 DMG_05 DMG_11 DMG_19
51
0
1
5
Microarrays (amostras)
Da
dos nor
m
a
li
z
ados (
log
2)
Figura 8. Gráficos boxplot dos dados de microarrays utilizados neste trabalho após normalização a
Resultados | 46
Resultados | 47
5.2 Análise funcional a partir da plataforma DAVID
Com a finalidade de encontrar categorias correspondentes aos mRNAs diferencialmente expressos, a lista dos 731 mRNAs modulados no DMG, foi submetida à análise no banco de dados DAVID, utilizando o Gene Ontology (GO) para identificação dos principais processos biológicos, e o banco de dados KEGG (Kyoto Encyclopedia of Genes and Genomes - http://www.genome.jp/kegg/) para a busca das vias biológicas relacionadas a estes
transcritos.
A figura 10 ilustra um total de 130 processos biológicos envolvidos com DMG. Em ordem decrescente de significância, os principais processos incluem: i) resposta de defesa (53 genes); ii) resposta a bactéria (25 genes); iii) resposta imune (51 genes). Os demais processos relevantes nesta análise foram: iv) resposta inflamatória (27 genes), v) regulação de citocinas (10 genes), vi) desenvolvimento de vasos sanguíneos (21 genes), vii) apoptose (47 genes), viii) regulação da proliferação celular (48 genes), entre outros (Anexo E).
De acordo com a análise funcional de vias de sinalização, um total de 9 vias biológicas foram identificadas (Figura 11). A via de interação entre receptores de citocinas (26 genes) foi a mais significativa, seguida da via de asma (7 genes) e câncer (25 genes). Outras vias incluindo a de lúpus eritematoso sistêmico (11 genes) e a via de sinalização de receptores NOD-like (8 genes) também foram identificadas nesta análise (Anexo F).
Resultados | 48
Figura 10. Heatmap ilustrativo das categorias identificadas a partir da ferramenta de análise funcional DAVID. Em destaque os
Resultados | 49
Figura 11. Principais vias de sinalização identificadas a partir da análise funcional usando DAVID. A escala em
cinza representa o valor de P em módulo logarítmico.
Resultados | 50
GO Processos biológicos Genes P-value
0006952 Resposta de defesa PGLYRP1, FOS, CCL3L3, HIST1H2BJ, IL1-Β,
LTF, CIITA, NFKBIZ, CRISP3, NCF2, CAMP, CD160, NLRP3, CD84, INHBA, SIGLEC1, CD83, PROK2, BPI, PPBP, LILRB5, DEFA4, DEFA3, CTSG, LALBA, CXCL1, TNF, CXCL3, C6, CLU, CXCL2, RSAD2, GPR68, DEFB127, CCL4, IL23A, CCL20, TFF3, PTX3, SCG2, PTPRC, IL5, CEBPB, RNASE3, IL8, OLR1, GABRA5, IL9, CYP4F11, COTL1, CD19, MPO, SELE
1.52E-07
0006955 Resposta imunológica PGLYRP1, SKAP1, CCL3L3, CEACAM8, IL1-Β,
ERAP1, LTF, CIITA, CRISP3, ICAM1, POU2AF1, NCF2, TNFRSF17, NLRP3, HLA-DQA2, OSM, CD83, BPI, LILRB5, PPBP, CTSG, CXCL1, HLA-DQB1, CPLX2, TNF, CXCL5, CXCL3, C6, CLU, CXCL2, RSAD2, DEFB127, PF4V1, CCL4, CHIT1, IL23A, CCL20, CD4, PTX3, HLA-DOA, PTPRC, IL5, CEBPB, IL8, OLR1, IGJ, IL9, GPI, ILF2, ETS1, CD300A
2.20E-05
0006954 Resposta inflamatória CXCL1, TNF, CXCL3, C6, CLU, CXCL2, GPR68,
CCL4, FOS, IL23A, CCL20, CCL3L3, IL1-Β, PTX3, SCG2, CIITA, NFKBIZ, IL5, CEBPB, IL8, OLR1, IL9, CYP4F11, NLRP3, SIGLEC1, PROK2, SELE
5.02E-04
0001817 Regulação da produção de
citocinas
CEBPB, TNF, IL6ST, IL9, ELANE, NLRP3, IL21, CD83, INHBA, BPI, REL, EREG, IL1-Β, RARA, CD4, AGPAT1
0.005334
0001568 Desenvolvimento de
vasos sanguíneos
TBX3, IL8, CDX4, PDGFA, FGF10, NCL, TCF7L2, JUNB, ZFP36L1, PROK2, GPI, EREG, ID1, BAX, PLXDC1, TDGF1, CASP8, ERAP1, IL1-Β, CEACAM1, SCG2
0.0016817932
0042981 Regulação da Apoptose LALBA, DPF2, IER3, TNF, PTGS2, MMP9, C6,
CLU, PPP3R1, NFKBIA, SOX4, TP63, HSPA1A, TCF7L2, PRUNE2, BLOC1S2, TDGF1, CASP8, IL1-Β, NGFRAP1, MYC, SCG2, ARHGEF4, PTPRC, CEBPB, TBX3, GFRAL, SOCS3, BCL2A1, PIM1, PIM3, PDE3A, NLRP3, PPIF, INHBA, PROK2, GCM2, CDKN1B, SSTR3, BTG2, DUSP1, ETS1, BBC3, BAX, MPO, ALOX12, TP53INP1
0.0069653625
0008284 Proliferação celular TNF, CXCL5, PTGS2, IL6ST, PDGFA, CLU,
NAP1L1, FGF10, SOX4, TP63, OSR2, BLOC1S2, TDGF1, IL1-Β, MYC, TCF3, THPO, SCG2, AGPAT1, PTPRC, IL5, TBX3, ELANE, IL9, IL21, CAPN1, OSM, VEGFB, CDKN1B, ATF3, EREG, ADM, MAB21L2, ALOX12
9.8E-5 Tabela 4. Processos biológicos de maior relevância para este estudo. Em destaque os genes que fazem parte de cada
Resultados | 51
Categoria Vias biológicas Genes P-value
KEGG Interação entre
receptores de citocinas
CXCL1, TNF, CXCL5, PDGFA, IL6ST, CXCL3,
CXCL2, CXCR3, PF4V1, CCL4, IL23A, CCL20,
CCL3L3, IL1-Β, AMHR2, IL5, IL8, IL9,
TNFRSF17, IL21, VEGFB, GH2, OSM, INHBA,
PPBP, IL3RA
4.50E-05
KEGG Lúpus eritematoso
sistêmico
HLA-DQB1, TNF, C6, HIST1H2AD,
HIST1H2AE, HIST1H2BJ, ELANE, HIST1H4E,
HLA-DOA, HLA-DQA2, CTSG
0.006392
KEGG Via de sinalização do
receptor NOD-like
CXCL1, TNF, IL8, CXCL2, CASP8, NFKBIA,
IL1-Β, NLRP3
0.011997
KEGG Via de câncer PTGS2, PDGFA, MMP9, FGF10, NFKBIA,
TCF7L2, MMP1, FOS, CDKN2B, CASP8, RARA,
MYC, WNT8B, CTBP1, IL8, LEF1, RALGDS,
WNT2B, VEGFB, JUP, CDKN1B, EP300,
LAMA3, ETS1, BAX
0.0031421885
KEGG Via de asma HLA-DQB1, IL5, TNF, RNASE3, IL9, HLA-DOA,
HLA-DQA2
8.9E-4 Tabela 5. Vias biológicas de maior relevância para este estudo. Em destaque a categoria e os genes de cada via e
Resultados | 52
5.3 Perfil de expressão de transcritos envolvidos com metabolismo e componentes do MHC no DMG
Entre os mRNAs diferencialmente expressos envolvidos com metabolismo do diabetes, destacamos de acordo com a análise estatística: IGFBP2 desempenhando papel importante na patogênese da obesidade, sendo associado à resistência insulínica, TCF3 como componente da via de sinalização Wnt, estando relacionado à patogênese do DM2, OLR1 participando de etapas chave do metabolismo de ácidos graxos e o TCF7L2 envolvido na regulação da homeostase da glicose sanguínea, também participando da via Wnt. A figura 12 ilustra a expressão destes mRNAs nas pacientes em relação aos controles, chamando atenção para modulação positiva dos transcritos IGFBP2 e TCF3 e negativa para OLR1 e TCF7L2, além disso, são mostrados os valores de fold change (FC) resultantes da análise estatística Rank Products.
Neste estudo também encontramos mRNAs que fazem parte do complexo principal de histocompatibilidade induzidos nas pacientes com DMG. Entre eles: HLA-DRB6 (major histocompatibility complex, class II, DR beta 6 - pseudogene), HLA-DQA2 (major
histocompatibility complex, class II, DQ alpha 2), HLA-DQB2 (major histocompatibility
complex, class II, DQ beta 2), HLA-DQB1 (major histocompatibility complex, class II, DQ
beta 1), HLA-DOA (major histocompatibility complex, class II, DO alpha). A figura 13
Resultados | 53
Figura 12. Representação do perfil de expressão dos mRNAs envolvidos com metabolismo entre pacientes com DMG e
controles saudáveis. A figura ilustra os valores de fold change (FC) para cada transcrito e destaca a modulação positiva para
IGFBP2 e TCF3 e negativa para OLR1 e TCF7L2.
Induzidos
TCF3 IGFBP2
Reprimidos
OLR1 TCF7L2
FC = 1.2 FC = 1.6
Resultados | 54
HLA-DOA
HLA-DQB1 HLA-DQB2
HLA-DQA2 HLA-DRB6
Figura 13. Representação do perfil de expressão dos transcritos dos genes MHC entre pacientes com DMG e controles saudáveis e seus
respectivos valores de fold change (FC).
FC = 1.6 FC = 1.1
FC = 1.4 FC = 1.4
Resultados | 55
5.4 Confirmação da expressão de genes relacionados ao processo inflamatório por PCR em tempo real
Foram utilizadas as mesmas amostras da análise por microarrays para a confirmação da expressão dos genes CXCL2, NFKBIA, TNF e IL1 . Estes genes estão associados à resposta inflamatória e seus transcritos apresentaram-se induzidos nas pacientes com DMG de acordo com a análise estatística Rank Products realizada neste trabalho. Foram feitas duplicatas experimentais de cada amostra. O gene HPRT1 foi utilizado como constitutivo para a normalização dos dados.
Os primers foram padronizados através da construção da curva padrão, para o cálculo de eficiência dos mesmos, e de uma curva de dissociação, para avaliar a ocorrência da amplificação de apenas um tipo de fragmento. Essas análises confirmaram a eficiência dos primers (99%), e a ausência de fragmentos inespecíficos. A tabela 6 mostra as sequências dos
primers dos genes selecionados para validação.
Os gráficos foram construídos de acordo com os valores de RQ (Relative Quantification), e os cálculos foram realizados utilizando como calibrador o menor dos
valores de ∆Ct. Em vista de o calibrador ser o menor valor de expressão, o perfil de repressão encontra-se próximo ao valor de RQ 0.00, enquanto o perfil de indução com valor próximo de
Gene Primers
CXCL2 Forward 5' CACACTCAAGAATGGGCAGA 3' Reverse 5' AGCTTCCTCCTTCCTTCTGG 3'
TNF Forward 5' AGCCCATGTTGTAGCAAACC 3' Reverse 5' TGAGGTACAGGCCCTCTGAT 3'
NFKBIA Forward 5' AGACCTGGCCTTCCTCAACT 3' Reverse 5' GTCTCGGAGCTCAGGATCAC 3'
IL1 Forward 5' CCACAGACCTTCCAGGAGAA 3' Reverse 5' GTGATCGTACAGGTGCATCG 3'
HPRT1 Forward 5' GACCAGTCAACAGGGGACAT 3' Reverse 5' CTGCATTGTTTTGCCAGTGT 3'
Resultados | 56
1.5. Os genes CXCL2 e NFKBIA apresentaram indução significativa nas pacientes com DMG em relação aos controles, aproximando-se dos perfis de expressão gênica gerados pela técnica de microarrays (Figura 14).
Os genes IL1-Β e TNF não apresentaram diferença estatística na comparação entre pacientes com DMG e controles (Figura 15).
.
Figura 14. Gráficos com os valores de expressão gênica relativa (RQ) dos genes CXCL2 (A), NFKBIA (B)
(Análise estatística teste t; P < 0.05).
A B
Figura 15. Gráficos com os valores de expressão gênica relativa (RQ) dos genes IL1B (C), TNF (D) (Análise
estatística teste t; P > 0.05).
Resultados | 57
Discussão | 59
6 DISCUSSÃO
Entre os principais tipos de diabetes, o DMG é o menos compreendido. A maior parte do conhecimento da manifestação desta doença vem de comparações com outros tipos de diabetes (LAUENBORG et al., 2009). Alguns estudos de transcriptoma têm comparado tecido placentário de grávidas acometidas por DM1 e DMG (RADAELLI et al., 2009), cuja maioria dos genes diferenciais também foram identificados em assinaturas de sangue total (ZHAO et al., 2011).
O presente trabalho refere-se ao estudo comparativo do perfil de expressão gênica transcricional em células mononucleares do sangue periférico (PBMCs) de mulheres com DMG em relação a gestantes saudáveis, focando em mRNAs relacionados à reposta inflamatória. O objetivo deste tipo de comparação consiste na caracterização dos perfis de expressão transcricionais das PBMCs no DMG. Estas células foram especialmente escolhidas como candidatas com base em estudos anteriores (ZHAO et al., 2011; KAIZER et al., 2007). Além disso, já foi relatado que as PBMCs expressam grande proporção dos mRNAs humanos, possibilitando o estudo de alterações clinicamente relevantes na expressão transcricional de maneira reprodutível (REYNIER et al., 2010; LIEW et al., 2006).
A análise aplicada neste estudo mostrou mRNAs envolvidos em processos biológicos significantes, incluindo resposta inflamatória, imunológica e de defesa. O perfil de expressão das PBMCs no DMG em relação aos controles revelou que mRNAs que regulam respostas inflamatórias representam o principal grupo funcional alterado no DMG. Além disso, transcritos relacionados ao metabolismo e componentes do MHC também estiveram diferencialmente expressos nas pacientes com DMG.