FLÁVIA LEONEL EMEDIATO
CARACTERIZAÇÃO DE GENES ANÁLOGOS DE RESISTÊNCIA (RGAS) EM CULTIVARES DE Musa acuminata CONTRASTANTES EM RESISTÊNCIA À
ESTRESSES BIÓTICOS
Dissertação apresentada ao Programa de Pós-Gradução Stricto Sensu em Ciências Genômicas e Biotecnologia, da Universidade Católica de Brasília, como requisito para a obtenção do Título de Mestre em Ciências Genômicas e Biotecnologia.
Orientador: Prof. Dr. Robert Neil Gerard Miller
Co-orientador: Prof. Dr. Georgios Joannis Pappas Jr
Brasília
Ficha elaborada pela Biblioteca Pós-Graduação da UCB E53c Emediato, Flávia Leonel
Caracterização de genes análogos de resistência (RGAS) em cultivares de musa acuminata contrastantes em resistência à estresses bióticos / Flávia Leonel Emediato. – 2009.
95f. : il. ; 30 cm
Dissertação (mestrado) – Universidade Católica de Brasília, 2009. Orientação: Robert Neil Gerard Miller
Co-orientador: Georgios Joannis Pappas Jr
1. Banana – Resistência à doenças e pragas. I. Miller, Robert Neil Gerard, orient. II. Pappas Jr, Georgios Joannis, co-orient. III. Título.
3 Dissertação de autoria de Flávia Leonel Emediato, intitulada “Caracterização de genes análogos de resistência (RGAs) em cultivares de Musa acuminata contrastantes em resistência à estresses bióticos”, apresentada como requisito parcial para a obtenção do grau de Mestre em Ciências Genômicas e Biotecnologia, defendida e aprovada em 18 de Dezembro de 2010, pela banca examinadora abaixo assinada:
Prof. Dr. Robert Neil Gerard Miller Orientador
Prof. Dra. Eliane Noronha Examinadora Interna
Prof. Dr. David Bertioli Examinador Externo
Brasília
5
AGRADECIMENTOS
Minha sincera gratidão ao meu orientador Professor Dr. Robert Miller pelos ensinamentos, apoio, confiança e por contribuir para meu crescimento pessoal e profissional.
Ao meu co-orientador Dr. Georgios Pappas pelo suporte em bioinformática.
A banca examinadora Dr. David Bertioli e Dr. Eliane Noronha, pela disponibilização de tempo e pelas correções que contribuíram para um melhor trabalho.
A equipe do laboratório de bioinformática da EMBRAPA Recursos Genéticos, Orzenil, Marcos Mota, Roberto Togawa e Natália Martins por todo apoio e dedicação oferecida nas análises de dados.
Aos amigos do laboratório da UCB Ciro, Aline e Cristiane por estarem sempre dispostos a ajudar.
Ao meu grande amigo Marco Aurélio pela sua amizade verdadeira, por estar sempre presente e pelo seu apoio em todos os momentos da minha vida.
Ao CNPq, FINEPE, IAEA, EMBRAPA E UCB pela concessão de bolsa, apoio técnico e financeiro.
Ao meu marido Eduardo, por todo o amor e paciência por ficar longe de mim e do Gabriel para que eu pudesse concluir meus estudos.
Aos meus irmãos Carol, Serginho e Paula e meus pais Sérgio e Regina por terem me dado toda a base para que pudesse estudar, pelo amor e pela ajuda com o Gabriel.
6
RESUMO
Referência: EMEDIATO, FL. Caracterização de genes análogos de
resistência (RGAs) em cultivares de Musa acuminata contrastantes em resistência
à estresses bióticos. 2009. Universidade Católica de Brasília, 2009.
Muitos cultivares de bananas (Musa spp.) são triplóides estéreis ou diplóides e sua evolução acontece somente por meio de mutação somática. Como conseqüência, nesta cultura pode faltar resistência a pragas e doenças. Vários genes de resistência (R genes) foram caracterizados em plantas, conferindo resistência a bactérias, fungos, nematóides e vírus. A caracterização de genes análogos de resistência (RGAs) foi realizada em cultivares de Musa acuminata contrastantes em resistência à doença Sigatoka negra causada pelo fungo Mycosphaerella fijiensis. O trabalho foi direcionado para as famílias de R genes NBS-LRR e família LRR extracelular com domínio transmembrana ligado a uma ser/thr quinase no citoplasma (RLK). Primers desenhados em trabalhos anteriores para NBS-LRR em banana geraram cerca de 200 sequências organizadas em 38 contigs com similaridade à RGAs desta classe. A diferenciação de contigs com base nos genótipos não foi observada, mostrando em um mesmo contig sequências provenientes tanto de cultivares resistentes à Sigatoka negra, como suscetíveis. As análises também foram realizadas para produtos amplificados por meio de primers direcionados à RLK, gerando 65 ORFs completas. Foi observada uma elevada porcentagem de clones seqüenciados que obtiveram similaridade à duas das famílias de R genes contendo quinase (RLK e STK) e mais uma vez a diferenciação dos contigs de proteínas Quinase não pôde ser feita com base em cultivares resistentes e suscetíveis a Sigatoka negra. A análise filogenética foi desenvolvida por máxima parcimônia com bootstrap 1000 réplicas, demonstrando alta divergência entre as sequências de RGAs tanto de NBS-LRR, quanto de RLK. A identificação e clonagem de R genes em Musa proporcionará novas oportunidades em potencial para o melhoramento genético através de seleção assistida por marcadores e modificação genética.
7
ABSTRACT
Many cultivars of bananas (Musa spp.) are triploid or sterile diploid and their evolution occurs only through somatic mutation. As a result, this culture may lack resistance to pests and diseases. Several resistance genes (R genes) have been characterized in plants, conferring resistance to bacteria, fungi, nematodes and viruses. The characterization of resistance gene analogues (RGAs) was held in Musa acuminata cultivars contrasting in resistance to Black Sigatoka disease caused by Mycosphaerella fijiensis. The work was directed to the families of R genes NBS-LRR and family of extracellular LRR transmembrane domain linked to a Ser / Thr quinase in the cytoplasm (RLK). Primers designed in previous work for NBS-LRR generated about 200 sequences organized into 38 contigs with similarity to RGAs in this class. The differentiation of contigs based on the genotypes was not observed, showing in the same contig sequences from both cultivars resistant to Black Sigatoka, as susceptible to it. The tests were also performed for products amplified by primers directed to RLK, generating 65 complete ORFs. There was a high percentage of clones sequenced that obtained similarity to two families from genes containing kinase (RLK and STK) and once again the differentiation of protein kinase contigs could not be made based on cultivars resistant and susceptible to Black Sigatoka . Phylogenetic analysis was carried out by maximum parsimony with bootstrap 1000 replicates, showing high divergence between the sequences of RGAs both NBS-LRR, RLK how much. The identification and cloning of R genes in Musa offer new potential for genetic improvement through marker-assisted selection and genetic modification.
8
LISTA DE ILUSTRAÇÕES
Figura 1 - Diversidade de banana à venda em uma loja no sul da Índia (Varkala, Kerala State) com diferentes composições de genoma.
Figura 2 - Sigatoka-amarela, mostrando coalescimento das lesões com necrose do tecido foliar.
Figura 3 - Ocorrência de Sigatoka negra no Brasil
Figura 4 - Sintomas causados pela Sigatoka negra. Alta freqüência de lesões foliares com estrias de coloração escura.
Figura 5 - Escurecimento dos vasos, observado por meio de corte realizado no pseudocaule de plantas afetadas pelo Mal do Panamá.
Figura 6 - Detalhe da exsudação de pus bacteriano, um dos principais sintomas da doença Moko.
Figura 7 - Maturação prematura dos frutos com murcha de Xanthomonas
Figura 8 - Tipos de combinações de genes e reações a doenças do sistema patógeno e hospedeiro no conceito “gene-a-gene”
Figura 9 - Famílias de genes de resistência identificados.
Figura 10 - Fluxograma resumindo a metodologia utilizada neste trabalho
Figura 11 - Fluxograma resumindo as etapas de Pré-assembly.
Figura 12 - Fluxograma resumindo as etapas de descoberta de genes.
Figura 13 - Regiões de anelamento de primers degenerados para a família NBS-LRR
Figura 14 - Etapas do processamento de seqüências por bioinformática.
Figura 15 - Alinhamento MUSCLE de seqüências de aminoácidos de 7 RGAs de banana, mostrando parte da região conservada de NBS.
Figura 16 - Análise filogenética de máxima parcimônia com Bootstrap 1.000 réplicas de seqüências de aminoácidos da família NBS-LRR de Musa.
Figura 17 - Alinhamento MUSCLE de seqüências de aminoácidos de 52 RGAs de banana, mostrando parte da região conservada do domínio pquinase de RLK e STK.
9
LISTADETABELAS
Tabela 1 - Grupos genômicos, subgrupos e nomes populares dos principais cultivares de bananeiras
Tabela 2 - Ranking dos países com maior produção de banana
Tabela 3 - Principais diferenças nos sintomas e epidemiologias entre Sigatoka negra e Sigatoka amarela
Tabela 4 - Lista de primers desenhados para a família RLK.
Tabela 5 - Lista dos conjuntos de primers desenhados para a família RLK com diferentes padrões como níveis de degeneração, tamanho do produto esperado e local de anelamento.
Tabela 6 - Lista de primers degenerados direcionados à família NBS_LRR com suas respectivas seqüências e motivos conservados.
Tabela 7 - Lista de primers específicos para espécie O. sativa com suas respectivas seqüências
Tabela 8 - Lista de primers degenerados testados e não utilizados nas análises e seus respectivos motivos de não uso.
Tabela 9 - Lista de NBS-LRR contigs com hits no genewise para domínios conservados e predição de pseudogenes, com respectivos cultivares e combinações de primers.
Tabela 10 - Análise das seqüências dos contigs por Blastx gerados com primers degenerados para a família de genes de resistência NBS-LRR
Tabela 11 - Acessos do Genbank e seus respectivos códigos utilizados na construção da árvore filogenética.
Tabela 12 - Lista de RLK contigs com respectivos hits no genewise para domínios conservados e predição de duas ORFs por contig, com respectivos cultivares e combinações de primers.
Tabela 13 - Eficiência dos combinações de primers na amplificação de RLK RGAs em Musa
Tabela 14 - Análise de Blastx para identificação das famílias gênicas.
10
LISTA DE GRÁFICOS
Gráfico 1 - Porcentagem de contigs composto de combinações específicos de primers para NBS-LRR RGAs.
11
LISTA DE ABREVIAÇÕES
Avr gene – Avirulence gene (gene de avirulência) AS – Ácido Salicílico
ATP – Adenosina tri-fosfato
BLSD – Black Leaf Streak Disease (Sigatoka Negra)
BAC – Bacterial Artificial Chromosome (cromossomo artificial de bactéria) Cc – Coiled coil (cauda espiralada)
COS – Sequência Ortóloga Conservada CDs – Região codificante
DNA – Deoxyribonucleic acid (Ácido desoxirribonucléico) ESTs – Expressed sequence Tags (Sequências expressas) FHIA – Fundación Hondureña de Investigacion Agrícola GLPs – Germin-like proteins
GTPs – Guanosina tri-fosfato HR – Reação de hipersensibilidade Il – Interleucina
JA – Ácido Jasmônico
OGMs – Organismos geneticamente modificados ORFs – Open Reading Frame
PRPs – Pathogenesis – Related Proteins (Proteínas relacionadas à patogenicidade) PCR – Polymerase Chain Reaction (Reação em Cadeia de Polimerase)
PROMUSA – Programa Internacional para melhoramento de Musa QTLs – Quantitative trait loci ( Locos de caracteres quantitativos) RGAs – Resistance Gene Analogs (Genes Análogos de Resistência)
ROIs – Reactive Oxygen Intermediates (Intermediários reativos de oxigênio) Rgene – Resistance genes (Genes de Resistência)
RLK – Receptor like Kinase RLP – Receptor Like Protein
SAR – Systemic Acquired Resistance (Resistência Sistêmica Adquirida) SD – Streat Disease (Sigatoka Amarela)
12 SNPs – Single Nucleotide Polymorphism (Polimorfismos nucleotídeos simples)
STK – Família de genes de resistência Serina/Treonina quinase SISGEN – Sistema Genoma
Thr - Treonina
TIR – Toll Interleucine receptor (Receptor Toll interleucina) TM – Domínio Transmembrana
13
SUMÁRIO
1. Introdução ... 14
1.1. Classificação Taxonômica ... 14
1.2. Importância sócio-econômica da banana ... 15
1.3. Estresses bióticos ... 16
1.4. Mecanismos de Resistência ... 26
1.5. Genes de Resistência ... 30
1.6. Melhoramento genético ... 34
1.7. Genômica de Musa ... 36
2. Objetivos ... 39
2.1. Objetivo Geral ... 39
2.2. objetivos específicos ... 39
3. Material e Métodos ... 40
3.1. Extração de DNA de M. acuminata ... 40
3.2. Desenho de primers degenerados ... 41
3.3. Amplificação de RGAs por PCR ... 44
3.5. Clonagem e sequenciamento de produtos de PCR ... 44
3.6. Análise bioinformática de seqüências ... 46
4. Resultados ... 49
4.1. Amplificação de produtos de PCR ... 49
4.3. processamento de sequências por bioinformática ... 50
4.4. Montagem das sequências em Contigs ... 51
5. Discussão ... 70
6. Conclusão ... 74
7. perspectivas futuras ... 75
8. Referências bibliográficas ... 76
14
1. INTRODUÇÃO
1.1. CLASSIFICAÇÃO TAXONÔMICA
Apesar de comumente ser confundida com espécie arbórea devido ao seu grande porte, a bananeira é uma herbácea que pode alcançar até três metros de altura, sem as lignificações ou espessamentos secundários das hastes que acontecem nas árvores.
Taxiconomicamente, a bananeira é classificada como membro da classe Liliopsida subclasse Zingiberidae, superordem Lilianae, ordem Zingiberales, família Musaceae (CRONQUIST, 1981, BELALCÁZAR & CARVAJAL, 1991). A família Musacea é dividida em três subfamílias: Heliconoideae, Strelitzoideae e Musoideae. Os gêneros Ensete e Musa pertencem à subfamília Musoideae, sendo que ao gênero Musa pertencem as bananeiras com frutos comestíveis. O gênero Musa está subdividido nas seções Australimusa, Callimusa, Rhodoclamys e Eumusa.
Descrita por STOVER AND SIMMONDS (1987), a classificação taxonômica da bananeira se deu com base na existência de dois cultivares selvagens denominados Musa acuminata (A) e Musa balbisiana (B). Posteriormente, os estudos de SIMMONDS & SHEPHERD (1955) levaram à constatação de grupos genômicos diplóides AA e AB; triplóides AAA, AAB e ABB; tetraplóides AAAA, AAAB, AABB e ABBB, sendo esta classificação adotada em todo o mundo. Além dos grupos genômicos, foi estabelecido o uso do termo subgrupo para denominar um complexo de cultivares, originários através de mutações de um único cultivar original, como nos casos dos grupos AAA, subgrupo Cavendish, e AAB, subgrupos Prata e Terra.
15 Tabela 1- Grupos genômicos, subgrupos e nomes populares dos principais cultivares de bananeiras
Grupo Genômico Sub grupo Cultivares
AA Ouro (Inajá, Bananinha, Imperador, Ouro
Paulista), Colatina Ouro.
AAA Cavendish Nanica (Caturra, Baé, Anã), Nanicão (Caturrão, N. Jangada, N. Eldorado), Grande Naine, Willians, Imperial, Valery, Lacatan.
AAB Prata Prata, Pacovan, Branca, Pranta Ponta Aparada, Prata-Zulu.
AAB Terra Terra (Comprida, Chifre-de-Boi), Pacova (Farta-Velhaco), Pacovaçu, D’Angola, Terrinha. AAAB Híbridos Pioneira, FHIA-01, FHIA-18.
1.2. IMPORTÂNCIA SÓCIO-ECONÔMICA DA BANANA
Dentro da fruticultura, a banana é responsável pela maior parcela da produção mundial, seguida pela laranja e maçã (FAOstat, 2009). Os seus atrativos comerciais, como seu alto teor nutritivo, preço acessível, gama de variedades e sabor, fazem com que esta cultura seja considerada a quarta mais importante em países em desenvolvimento, com uma produção mundial de cerca de 100 milhões de toneladas (FAOstat, 2007). De acordo com dados publicados pela FAOstat (2009), atualmente o Brasil está em quarto lugar no ranking de produção mundial de banana (Tabela 2).
No Brasil, a fruta tem grande importância econômica e social. Além de garantir ao país um lugar no ranking dos maiores produtores mundiais, juntamente com a Índia, China e as Filipinas, é também fundamental para a complementação da dieta alimentar das populações de baixa renda.
Tabela 2- Ranking dos países com maior produção de banana
País Produção (Milhões de Toneladas em 2007)
Índia 21.7 China 7.3 Filipinas 7.0 Brasil 6.9 Equador 6.1 Indonésia 5.0 Costa Rica 2.2
México 2.2 Tailândia 2.0 Colômbia 1.8 Burundi 1.6 Total Mundial 81,2
16 Amplamente distribuída por todo o globo, a banana tem como origem o sudeste Asiático, mais especificamente na Índia e Polinésia (SIMMONDS, 1962), sendo a Malásia e a Indonésia considerados os centros da diversidade (DANIELLS et al., 2001).
1.3. ESTRESSES BIÓTICOS
Ainda que existam incontáveis variedades de bananeiras (Figura 1), esses números se reduzem significativamente quando, entre as comercialmente viáveis, surgem necessidades de resistência a estresses bióticos. Isso porque, atualmente, a
cultura de banana é alvo de inúmeras pragas e doenças, que, encontrando condições favoráveis ao seu desenvolvimento, são capazes de causar prejuízos em lavouras. A quantidade e a qualidade das frutas são freqüentemente ameaçadas devido aos danos deixados na planta, que, dependendo da intensidade do ataque, pode torná-la improdutiva ou levar até a sua erradicação.
Figura 1- Diversidade de banana à venda em uma loja no sul da Índia (Varkala, Kerala State) com diferentes composições de genoma. © Copyright 2007: JS Heslop & Schwarzacher
Ocorrem atualmente aproximadamente 50 patógenos fúngicos em bananeiras, porém os mais importantes são Mycosphaerella fijiensis (Sigatoka negra), Mycosphaerella musicola (Sigatoka amarela) e Fusarium oxysporum f. sp. cubense (Mal do Panamá) (JONES, 2000).
17 tomate, tabaco e batata (NORMAN et al., 2009). A Podridão negra causada pela Xanthomonas campestris pv. campestris, ocorrente em diversas culturas como o brócolis e a couve, (VILLETH et al., 2009) também ocorre em cultivares de Musa devido a existência do patovar Xanthomonas campestris pv. musacearum, que provoca Murcha Bacteriana. Inicialmente descrita na Etiópia em 1968, o patógeno agora está se espalhando pela África Ocidental e Central (ARITUA, 2009).
O sub-grupo Cavendish, apesar da sua popularidade no mercado mundial, apresenta pouca diversidade genética (já que se reproduz assexuadamente), o que a torna particularmente vulnerável à doenças que ameaçam de igual forma, a agricultura de subsistência.
Nesse sentido, para que se possa atender tanto às exigências do mercado quanto às dos produtores, a busca por genes relacionados à resistência assume extrema importância para, justamente, conciliar sabor e produtividade, no desenvolvimento de bananas com mais qualidade e com reduzidos níveis de agrotóxicos, em benefício de consumidores e produtores.
1.3.1. A Sigatoka amarela
A Sigatoka amarela (SD), também conhecida como mal-de-sigatoka ou cercosporiose, é causada pelo fungo Mycosphaerella musicola, considerado um patógeno de elevada relevância, principalmente em altitudes maiores e também em áreas onde cultivares suscetíveis à Sigatoka amarela vem sendo cultivados.
Dois tipos de esporos estão envolvidos no aparecimento da doença: o esporo sexuado chamado, ascósporo, e o assexuado, denominado conidiósporo. As diferenças de comportamento entre eles podem refletir na epidemiologia da doença, que é fortemente influenciada pelas condições climáticas, onde temperaturas acima de 23°C, combinadas com umidade do ar maior que 80% e pluviosidade elevada, são as condições ideais para o desenvolvimento da doença (ISOKAZU et al., 2008).
18 amadurecer adequadamente; ataques menos graves resultam em diminuição do peso dos cachos, frutos menores, amadurecimento prematuro antes ou depois da colheita, descoloração da polpa e alteração no aroma e sabor dos frutos (CORDEIRO et al., 2005; MEREDITH, 1970).
Figura 2 - Sigatoka-amarela, mostrando coalescimento das lesões com necrose do tecido foliar. © Copyright 2003 Embrapa Mandioca e fruticultura
Dentre outros, alguns dos principais cultivares comercialmente viáveis são suscetíveis ao patógeno, como Nanica, Nanicão, Prata e Pacovan. Por outro lado, a resistência à doença é constatada em plátanos como Terra, D’Angola e Figo, assim como em bananas Mysore, Yangambi e Prata Zulu (TEIXEIRA et al., 1996). Enquanto as variedades de bananeiras suscetíveis a M. musicola apresentam menor período de incubação e maior número de manchas e esporulação nas folhas, em algumas variedades resistentes, a evolução dos sintomas é interrompida ainda nos primeiros estágios, pois com o aumento do nível de resistência aumenta-se também o tempo de transição entre os estágios de evolução da doença (STOVER, 1972; MEREDITH, 1970; FOURÉ, 1985; FOURÉ et al., 1990).
1.3.2. A Sigatoka negra
19 variando de 24 a 30ºC (GASPAROTTO, 2006), relativamente quente se comparado às condições ótimas para a Sigatoka amarela.
Além do Brasil, a doença também é bem disseminada em diversos outros pontos do globo. Surgiu pela primeira vez no sudeste da costa de Viti Levu, em Fiji, em 1963, sendo posteriormente relatada no restante da Ásia e África. Na América Latina foi identificada pela primeira vez em 1972 em Honduras, de onde se espalhou para o norte até Guatemala, Belize, sul do México, sul de El Salvador, Nicarágua, Costa Rica, Panamá, Colômbia e Equador. A doença foi depois reportada na Venezuela e Cuba (JONES et al. 1993).
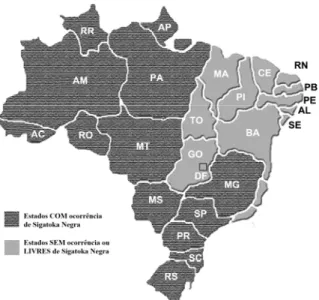
Atualmente, é considerada umas das doenças de bananeira de maior importância, principalmente em decorrência da sua rápida propagação. No Brasil, surgiu em 1998 na região do Amazonas, de onde se espalhou para quase todo o país (Figura 3). Estudos recentes mostram que a doença se encontra disseminada nos Estados do Amazonas, Pará, Roraima, Amapá, Acre, Rondônia, Mato Grosso, Mato Grosso do Sul, São Paulo, Paraná, Minas Gerais, Santa Catarina, Rio Grande do Sul (GASPAROTTO et al., 2006, MAPA 2007).
Figura 3- Ocorrência de Sigatoka negra no Brasil
20 Figura 4 - Sintomas causados pela Sigatoka negra. Alta freqüência de lesões foliares com estrias de coloração escura. © Copyright 2003 Embrapa Mandioca e fruticultura
Apesar da semelhança sintomatológica entre a Sigatoka negra e a Sigatoka amarela, há várias diferenças quem podem ser facilmente observadas. Enquanto nesta as folhas infectadas apresentam estrias de cor clara na face superior da folha, com lesões de freqüência bem mais baixas e bordos regulares de forma elíptica, naquela as estrias são marrons e visíveis na face inferior da folha, com freqüência de lesões bem mais alta e, nos estágios finais, estrias com bordos irregulares (FERRARI et al, 2008) (Tabela 3).
Tabela 3 - Comparação dos sintomas e epidemiologia da Sigatoka negra e Sigatoka amarela
Sigatoka negra Sigatoka amarela
Estrias iniciais marrom escuras. Estrias iniciais amarelo pálidas. Mais comum em ambientes mais quentes. Mais comum em ambientes mais frios. Ascósporos levados pelo vento são o inóculo mais
importante
Inóculo consiste de ambos, conídios (dispersos pela água) e ascósporos (dispersos pelo vento)
21
1.3.3. Mal do Panamá ou Murcha de Fusarium
O Mal do Panamá ou Murcha de Fusarium é causada por uma das cepas mais destrutivas do fungo Fusarium oxysporum f. sp. cubense (E.F. Smith) Sn e Hansen (FOURIE et al., 2009).
Durante a primeira metade do século passado foi a causa da destruição de mais de 50.000 ha de lavouras da variedade Gros Michel, gerando uma substituição por cultivares Cavendish (PÉREZ-VICENTE, 2004), inicialmente resistentes à doença.
Os sintomas mais comuns, que podem ser observados em bananeiras afetadas pela Murcha de Fusarium, é uma descoloração vascular pardo-avermelhada do pseudocaule de forma periférica, mantendo o centro claro (Figura 5). O nome Murcha de Fusarium se dá pela progressiva murcha das folhas mais velhas para as mais novas, precedido pelo amarelecimento das mesmas (CORDEIRO & KIMATI, 2005).
Figura 5 - Sintomatologia do Mal do Panamá. Escurecimento dos vasos, observado por meio de corte realizado no pseudocaule de plantas afetadas. © Copyright 2003 Embrapa Mandioca e fruticultura
São conhecidas quatro raças fisiológicas do patógeno. Entre elas, as raças 1, 2 e 4 possuem relevância justamente por atacarem bananeiras de interesse comercial. O termo raça fisiológica vem sendo utilizado para descrever os patógenos da mesma espécie, morfologicamente semelhantes e com mesma virulência. Patógenos de distintas raças fisiológicas apresentam diferentes níveis de virulência.
22 em torno da doença, por ser encontrada nos cultivares do subgrupo Cavendish, que inicialmente apresentavam resistência à doença (MOREIRA, 1999).
As principais formas de disseminação da doença são o contato dos sistemas radiculares de plantas sadias com esporos liberados por plantas doentes e, em muitas áreas, pelo uso de material de plantio contaminado. O fungo também é disseminado por água de irrigação, de drenagem, de inundação, assim como pelo homem, por animais e etc. (CORDEIRO, 2002).
1.3.4. Murcha Bacteriana Moko
Murcha bacteriana Moko, cujo nome foi derivado de um determinado cultivar de Musa severamente afetado pela doença no início do século XX (HAYWARD, 2006), é causada pela bactéria R. solanacearum, uma espécie complexa e tradicionalmente classificada em cinco “raças” baseadas na diferença de hospedeiros e propriedades químicas (EYRES et al, 2005).
A doença Moko é endêmica da América Central e do Sul, ocorrendo em Belize, Brasil, Colômbia, Costa Rica, Equador, El Salvador, Granada, Guatemala, Guiana, Honduras, México, Nicarágua, Panamá, Peru, Suriname, Trinidad e Venezuela. Em 2004, a doença foi confirmada em St James, Jamaica e Filipinas, na região de Mindanao (EYRES et al., 2005). No Brasil a bactéria é restrita aos estados do Amapá, Amazonas, Pará, Pernambuco, Rondônia, Roraima e Sergipe (MAPA, 2007).
O sintoma mais característico do Moko se manifesta por exsudação de pus bacteriano (Figura 6), descoloração vascular centralizada no pseudocaule e murcha das folhas em qualquer fase do ciclo vegetativo (VIEIRA et al., 2007).
O Moko pode ser disseminado por insetos, através de solos infectados ou pelo contato com a raiz. Estas características, associadas à indisponibilidade de cultivares resistentes e de baixa tecnologia de produção, tornam a doença um problema muito sério para as culturas de banana (BUDDENHAGEN 1961, STOVER 1972, TAKATSU 1986, MATOS et al. 1996).
23 variedades comerciais resistentes por meio de técnicas de reprodução convencionais. Então, o grande problema dessa doença é que nenhuma das bananas comestíveis conhecidas apresentam resistência a ela.
Figura 6 - Detalhe da exsudação de pus bacteriano, um dos principais sintomas da doença Moko. © Copyright 2009. Agro - NM
1.3.5. Murcha bacteriana de Xanthomonas
A doença provocada pela bactéria Xanthomonas vasicola pv. musacearum (sinônimo Xanthomonas campestris pv. musacearum) tem causado grande impacto na África, onde vem se espalhando rapidamente. Surgiu em 1960 na Etiópia (YIRGOU, 1968; BRADBURY 1974), em 2001 foi identificada em Uganda nos plantios de bananas “Kayinja” (ABB) e, logo em seguida, em países como República Democrática do Congo, Ruanda e Tanzânia (BIRUMA et al., 2007, NDUNGO et al.,2006, MGENZI et al., 2006).
24 Uma vez estabelecida em uma área, a doença se espalha rapidamente, podendo causar perda total da produção (EDEN-GREEN, 2004). As bananas afetadas desenvolvem sintomas como um progressivo amarelecimento e murcha das folhas (Figura 7), enrugamento da inflorescência masculina, amadurecimento precoce e desuniforme dos frutos, coloração amarronzada da polpa dos frutos e exsudação abundante de pus bacteriano amarelo (ARITUA et al., 2007). A bactéria causa infecção sistêmica na planta e se dissemina rapidamente para plantas sadias pelo transporte de pus bacteriano por meio de insetos vetores, de ferramentas ou equipamentos agrícolas contaminados.
Apesar dos sintomas serem muito parecidos com a doença Moko, causada por R. solanacearum, a murcha bacteriana de Xanthomonas de banana ainda não é encontrada no Brasil, e, por isso mesmo, o cuidado com a entrada de produtos importados deve ser rigorosamente exercido por medidas de inspeção do trânsito dos produtos, bem como a capacitação de pessoas que lidam diretamente com o cultivo para o reconhecimento dos sintomas causados por esta bactéria.
Agricultores da África Central e Oriental estão relutantes em empregar medidas de controle intensivo em suas plantações de banana, fazendo-se necessário o desenvolvimento de cultivares de banana resistentes à doença (TRIPATHI et al., 2009).
25
1.3.6. Mecanismos de Controle de doenças
Para ocorrer uma doença é necessário a interação de um patógeno, um hospedeiro suscetível e condições ambientais favoráveis para ocorrer os processos de infecção, colonização e reprodução. Após isto, visualizam-se os sintomas da doença nas plantas, identifica-se o agente causal e é adotada a estratégia mais adequada ao seu controle.
O controle da Sigatoka amarela e negra pode ser feito utilizando agrotóxicos e óleos (STOVER, 1972) via aplicação foliar, demandando elevados custos e aumentando o risco de contaminação ambiental. Pode-se, ainda, introduzir variedades resistentes (SILVA et al., 1998) ou, alternativamente, aplicar ambas as soluções, para se dificultar a proliferação do fungo.
O controle do Mal do Panamá ocorre de forma diferenciada, pois o fungo causador, ao contrário de algumas outras doenças, ataca pela raiz da planta. Destarte, para o controle do Mal do Panamá se adota, geralmente, a mudança geográfica da área de cultivo, já que o patógeno pode ficar no solo por até 40 anos. O F. oxysporum é um fungo de solo apresentando uma alta capacidade de sobrevivência na ausência do hospedeiro, decorrente, provavelmente, da formação de estruturas de resistência chamadas clamidósporos, que podem permanecer no solo em estado latente por longos anos (CORDEIRO et al., 2004). A medida de controle mais eficiente para essa doença é o cultivo de variedades resistentes (MOREIRA, 1987; MATOS et al., 2001; QUERINO et al., 2003; ROSALES et al., 2003).
O principal meio de controle da doença Moko é a exclusão de bananeiras das áreas onde ocorre a doença e a destruição das plantas infectadas. Isso porque, em decorrência do caráter sistemático da infecção, a destruição do agente acaba ensejando na morte da planta hospedeira. Em plantações comerciais, além dos prejuízos resultantes do extermínio das plantas infectadas, o produtor deverá, também, destruir as demais bananeiras da área, encarecendo ainda mais o procedimento para evitar a propagação da doença
26 1.4. MECANISMOS DE RESISTÊNCIA
Considerando que a maioria dos estresses bióticos que afeta as bananeiras vêm sendo tratados com a aplicação de produtos químicos, encarecendo a produção e fomentando à adaptação dos organismos patogênicos, além da própria degradação ambiental que o uso de agrotóxicos enseja, o desenvolvimento de plantas naturalmente resistentes às doenças talvez seja a medida de controle mais aceitável. Nada químico necessita ser aplicado, nem modificações no ambiente precisam ser efetuadas, pois é o próprio código genético da planta que oferece a resistência (BLANCHARD & TATTAR, 1997).
A resistência genética da planta está associada à reação de incompatibilidade entre a planta e o patógeno. Porém, um problema comumente encontrado diz respeito ao fato dos patógenos de plantas poderem evoluir para quebrar a resistência total ou para erodir a resistência parcial. A evolução das populações de patógenos depende de mutações, recombinações, alterações nas frequências alélicas, do fluxo gênico e da pressão de seleção exercida pelo hospedeiro (CARLIER, 2003).
1.4.1. Resistência raça-específica ou vertical
27 Figura 8 - Tipos de combinações de genes e reações a doenças do sistema patógeno e hospedeiro no conceito “gene-a-gene” (Agrios, 1997). Em “A” ocorre reação de incompatibilidade isto é, a planta é hospedeira do patógeno, porém a infecção não ocorre. Em “B” ocorre reação de compatibilidade. O patógeno e a planta não possuem os genes de Avirulência (Avr) e Resistência (R gene) respectivamente. Em “C” e “D” ocorre também reação de compatibilidade, onde em “C” o patógeno não possui o gene Avr e o hospedeiro possui o R gene, não havendo reconhecimento de ambas partes. Em “D” ocorre o inverso de “C”, o patógeno possui o gene Avr e o hospedeiro não possui o R gene, não havendo interação e conseqüentemente desenvolvendo a infecção.
Essa resposta pode ser local, havendo uma reação de hipersensibilidade (HR) no qual ocorre a morte programada de células no local da infecção. Nesta interação, o patógeno, ao penetrar o tecido da planta, dispara as respostas de defesa do hospedeiro, que, ativadas rápida e intensamente, impedem a multiplicação do patógeno evitando assim a doença (VANTINI et al., 2008). Um evento no início desta reação de hipersensibilidade é a geração de superóxido (O2-) e acúmulo de peróxido de hidrogênio (H2O2) em uma explosão oxidativa produzindo Reactive Oxygen Intermediates (ROIs), isto é, espécies ativas de oxigênio (FANG, 1997), que juntamente com a produção de oxido nítrico (NO), desempenham um papel chave na ativação dos mecanismos de
Patógeno Célula hospedeira Patógeno Célula hospedeira
Patógeno Célula hospedeira Patógeno Célula hospedeira
Fenótipo: Resistência
Fenótipo: Doença Fenótipo: Doença
Fenótipo: Doença B
C D
Elicitor
Elicitor Elicitor
Genes Avr presentes Genes de patogeneicidade
Genes de patogeneicidade
Genes de patogeneicidade Genes Avr presentes Genes de patogeneicidade Gene R
Gene R
28 resistência a doenças em animais e plantas (DELLEDONE et al., 2001). A ativação da explosão oxidativa na HR vegetal é parte de um sistema altamente amplificado e integrado de sinalização que envolve também o ácido salicílico (AS) e perturbações de Ca2+ citosólico para desencadear mecanismos de defesa (LAMB & DIXON, 1997). Ocorre também no processo a produção de fenóis, espessamento da parede celular da planta, acúmulo de fitohormôrnios, etileno e ácido jasmônico (JA), formação de fitoalexinas (proteínas antimicrobianas) e indução de expressão de PR (Pathogenesis related), proteínas relacionadas à defesa que limitam a colonização de tecidos vegetais e impedem a manifestação da doença (HAMMOND-KOSAK & JONES, 2003).
Esses eventos podem posteriormente desencadear uma resposta generalizada com longa duração, prolongando-a de forma sistêmica e atuando mesmo longe da infecção (SAR - Resistência Sistêmica Adquirida).
1.4.2. Resistência raça-não-específica ou horizontal
O que difere a resistência raça-específica da resistência raça-não-específica é que esta, provavelmente, é controlada por vários genes. Por ter vários genes envolvidos na produção de respostas de defesa, pode-se dizer que a resistência raça-não-específica é poligênica ou multigênica, sendo possível que ela seja mais durável pela menor probabilidade do patógeno quebrar a resistência (NELSON, 1971). Assim, esse mecanismo de resistência é considerado de fundamental importância para o melhorista.
Essa resistência se caracteriza por vários fenômenos: o patógeno apresenta baixa penetração e esporulação; os genes que condicionam a resistência são recessivos e interagem entre si; não há especificidade para uma determinada raça, ou seja, a resistência é efetiva para um amplo espectro de raças patogênicas (na maioria dos casos, atua uniformemente sobre todas as raças de um patógeno) (FEHR, 1987).
29 infecção, pequena produção de esporos e grande período de latência (VAN DER PLANK, 1963).
A desvantagem desse tipo de resistência está associada à dificuldade em se transferi-la de um genótipo para outro, pois a probabilidade de transferência de todos os alelos desejáveis a partir de um genótipo resistente para um suscetível é baixa, quando um grande número de alelos está envolvido. Considerando que os alelos favoráveis são gradualmente incorporados, se a reprodução da cultura for do tipo alógama, geralmente, se utiliza a seleção massal, onde as plantas mais resistentes têm suas sementes colhidas, misturadas e efetuando-se a semeadura de uma nova população. Já no caso de plantas autógamas, a fecundação cruzada tem que ser realizada manualmente, o que eleva o custo de produção. Atualmente tem-se utilizado a seleção por meio de marcadores moleculares, visando à identificação de genes de resistência, sendo mais fácil (GONÇALVES-VIDIGAL & POLETINE, 1999).
1.4.3. Resistência não hospedeiro
É, provavelmente, a resistência mais comum e durável de todas as citadas anteriormente. Isso decorre do fato de alguns patógenos possuírem afinidades por determinadas plantas, isto é, muitas vezes um patógeno que incide sobre um tomateiro, não ataca uma bananeira. Em comparação com a raça-específica, o entendimento da resistência não hospedeiro é menos completa. Este tipo de resistência, que pode ser exposta contra bactérias, fungos e agentes patogênicos, é conhecida por ser dependente de múltiplos componentes celulares que compreendem além de outros fatores, pré-barreiras (síntese de peptídios, proteínas e metabólitos secundários, que restringem a infecção por patógenos induzindo mecanismos de defesa) (HEATH, 2000).
30 Um certo número de moléculas de sinalização, como etileno, ácido salicílico, ácido jasmônico, induzidos por proteínas quinase (SIPK), estão também aplicados na resistência não hospedeiro. R-genes são também conhecidos por serem envolvidos neste tipo de resistência à doença. A glicerol quinase NHO1 de Arabidopsis por exemplo, confere resistência ao patógeno não hospedeiro Botrytis cinerea e Pseudomonas syringae (KANG et al., 2003). Até o momento, NHO1 é o único gene conhecido por ser envolvido em resistência não hospedeiro.
1.5. GENES DE RESISTÊNCIA
Foram identificadas até o momento cinco famílias de genes de resistência a estresses bióticos em plantas (MARTIN, 2003). Estes genes possuem como produto normalmente aminoácidos ou estruturas protéicas com motivos bem conservados (Figura 9), fatores que podem se tornar uma importante ferramenta para a biotecnologia auxiliando em estudos de doenças em plantas diferentes, porém provocadas pelo mesmo patógeno.
31
Classe 1 - Família NBS-LRR: Nucleotide binding site (NBS) and Leucine-rich repeat
(LRR) domains
É a maior e mais importante família de R genes, pois todos os integrantes desta família estão relacionados de alguma forma à resistência. A maioria dos genes clonados até hoje ligados à resistência estão incluídos nesta família, pois grande parte destes genes codificam proteínas NBS-LRR. A explicação se dá pelo fato de se acreditar que “...o domínio NBS seja importante na ligação de ATP e GTP e esteja atuando na transdução de sinais...” (ELLIS & JONES, 1998; van der BIEZEN & JONES 1998) e o LRR no reconhecimento do patógeno por meio da interação do produto do gene Avr com o produto do R gene. Então dessa forma, os dois agindo juntos, como um complexo bem coordenado traz grande eficácia na defesa das plantas contra as doenças mais importantes.
Muitos estudos têm sido elaborados em torno desta família de proteínas, pois a sua única função conhecida está relacionada à resistência (DANGL & JONES, 2001; MEYERS et al. 2005).
Foram identificados até hoje cerca de 150 genes da família NBS-LRR em Arabidopsis thaliana e mais de 400 em Oryza sativa (MEYERS, 2003). O domínio NBS compreende dois tipos de subclasse, conhecidos como TIR (Toll interleucine receptor) NBS ou Não-TIR NBS, baseados em aminoácidos localizados no N-terminal que mostram homologia com domínios de sinalização intracelular de Drosófila (Toll) e uma interleucina (IL) basal de mamíferos (PARKER et al., 1997). O receptor Toll controla a resposta imune a bactérias gram-positivas e fungos através da ativação de uma via de sinalização. O grupo TIR está presente em dicotiledôneas, enquanto o grupo não-TIR ocorre tanto em monocotiledôneas quanto em dicotiledôneas. O NBS também pode estar ligado a um CC (cauda espiralada) no N-terminal ou então pode estar ligado a um motivo TIR no N-terminal e um NLS e WRKY no C-terminal.
proteína-32 proteína (JONES, 1997). O domínio LRR está envolvido na determinação da especificidade de reconhecimento de várias proteínas R.
A estrutura de cristal de mais de 20 proteínas LRR tem revelado que este domínio possui como característica uma série de β-folhas que formam uma face côncava como uma ferradura (ENKHBAYAR, 2003), porém a sua estrutura quaternária ainda é pouco conhecida.
Estrutura 3D de determinadas proteínas LRR mostram que a superfície côncava do domínio é ideal para a interação com alfa-hélice, apoiando a hipótese de que sua estrutura proporciona um excelente enquadramento para alcançar diversas interações proteína-proteína (KOBE et al., 2001).
Classe 2 - Família LRR extracelular ancorado à membrana
Os R genes dessa família codificam proteínas que funcionam como receptores possuindo um domínio LRR extracelular ancorado à membrana com um pequeno domínio transmembrana. Neste caso não existem domínios sinalizadores intracelulares. Em tomate (Lycopersicon esculentum), os receptores LRR estão ancorados na membrana e são codificados por um complexo de genes cf que conferem resistência a Cladosporium fulvum (WULFF, 2009).
Classe 3 - Família LRR extracelular com domínio transmembrana ligado a
uma ser/thr kinase no citoplasma
O LRR além de ser encontrado no citoplasma, pode também ser encontrado fora da célula possuindo um domínio transmembrana, ligado a uma serina-treonina quinase no citoplasma. Essa organização é justificada com base na função do domínio LRR estar intimamente relacionada à recepção de estímulos externos e a ser/tre quinase às cadeias de fosforilações na transdução de sinais do citoplasma ao núcleo celular (SONG et al., 1995).
33 BLEECKER, 2003). Sabe-se também que o mesmo patógeno pode causar a murcha bacteriana em banana.
Esta classe parece indicar uma ligação evolutiva entre as diferentes classes de genes R em plantas, pois codifica um receptor tipo quinase caracterizado por um domínio LRR extracelular como os genes da classe 2 e uma região TM com uma ser/tre quinase intracelular como a classe 3 (XU et al., 2006; SONG 1997).
Classe 4 - Família ser/thr kinase citoplasmática
O gene Pto de L. esculentum foi o primeiro R gene a ser identificado nessa classe e confere resistência a Pseudomonas syringae, que expressa o gene Avr-Pto (MARTIN et al., 1993) e causa a mancha foliar do tomate (SONG et al., 1997, MELOTTO et al., 2004; ANDERSON et al., 2006). Esta classe caracteriza-se pela presença de uma quinase que fosforila resíduos de serina e treonina capazes de interagir com o produto do gene de Avr Pto (TANG et al., 1999). Este gene também já foi caracterizado em A. thaliana, Phaseolus vulgaris (MELOTTO et al., 2004) e M. acuminata (JAMES, 2008).
As proteínas quinases Pto, assim como Xa21 em arroz, são quinases ativas. Em experimentos in vitro, Pto se mostrou capaz de autofosforilação em vários sítios (Sessa et.al., 1998). A função do gene Pto requer mediação do gene Prf (FIELDS, et.al., 1994), membro da classe NBS-LRR (Jones et.al., 1997) que tem ligação próxima ao gene Pto (SALMERON et al., 2006).
34
Classe 5 - Família coiled coil ancorado à membrana
Domínio coiled coil ancorado à membrana (ex. gene RPW8)
O gene RPW8 de Arabidopsis codifica uma redutase localizada na membrana, com uma estrutura helicoidal, sem semelhança alguma com qualquer outro gene de resistência (KOBE e KAJAVA, 2001). O mesmo é induzido e dirigido especificamente para a membrana extra-haustorial, uma membrana interfacial derivada possivelmente da membrana plasmática da célula hospedeira (WANG et al., 2009).
O gene RWP8 ativa a produção de ácido salicílico (AS) como estratégia de defesa e promove o acúmulo local de H2O2, presumivelmente para restringir o crescimento do haustório reduzindo ao mesmo tempo o dano oxidativo às células do hospedeiro (WANG et al., 2009). A proteína confere um amplo espectro de resistência (XIAO et al., 2003).
Conhecendo bem a estrutura desses genes, bem como quais são os domínios conservados e os motivos de cada um, é possível desenhar primers degenerados para a busca desses genes em uma determinada espécie, através da amplificação e clonagem. Devido a alta similaridade com genes de resistência, estas seqüências são consideradas análogos de genes de resistência (RGAs).
1.6. MELHORAMENTO GENÉTICO
A preocupação com o consumo de alimentos que não contêm agrotóxicos vem aumentando a cada dia. A utilização de compostos químicos além de arriscar a vida de trabalhadores rurais (não são raros os casos de intoxicação por agrotóxicos), compromete a qualidade dos alimentos e, também, contamina o solo e a água (LEITE et al., 2009). Para evitar este tipo de problema, técnicas de melhoramento genético têm sido criadas.
35 Embora a banana seja classificada como a quarta mais importante mercadoria comercializada no mundo, depois do arroz, trigo e milho (HARPER et al., 2002), o melhoramento genético em Musa, em comparação com outras grandes culturas alimentares tem sido limitada. Cultivares têm evoluído desde diplóides, triplóides e tetraplóides selvagens asiáticos de espécies de M. acuminata (genoma A) e M. balbisiana (genoma B). Espécies selvagens são geralmente seminíferas, ao passo que hoje em dia a maioria dos cultivares comerciais são estéreis ou triplóides e diplóides, com frutas que se desenvolvem através de partenocarpia. Como esta cultura tem evoluído principalmente através de reprodução vegetativa assexuada, a variação genética é limitada, tornando-se uma cultura sem resistência a pragas e doenças. A solução para o problema se concentra em voltar a atenção e os esforços para busca de genes de importância em cultivares selvagens, fontes de resistência, como por exemplo Calcutta 4 para que posteriormente possam ser transferidos à cultivares comerciais (Cavendish).
As primeiras pesquisas com melhoramento genético da bananeira ocorreram em três diferentes locais: Honduras, em 1930 e 1959, pela “United Fruits Company”; Trinidad, em 1922, pelo “Imperial College of Tropical Agriculture”; e Jamaica, em 1924, pelo “Department of Agriculture”, motivadas pelo Mal do Panamá, que infestou a cultivar Gross Michel, plantado em grandes áreas da América Central e Jamaica. O objetivo era produzir um material com todas as características da Gross Michel, mas com resistência ao Mal do Panamá. Em 1930, devido à importância econômica da sigatoka, outro objetivo foi adicionado ao melhoramento de bananeira (SHEPHERD 1974, citado por SILVA et al., 2002).
36 Mais recentemente o centro de pesquisa Cirad/ Flhor iniciou pesquisa em genética e melhoramento da bananeira, em Guadalupe, Antilhas Francesas, Camarões, África, baseando-se em métodos tradicionais e biotecnológicos. Atualmente, além do melhoramento convencional, técnicas modernas, como hibridação somática e engenharia genética, vêm sendo utilizados com sucesso no melhoramento da bananeira (SHEPHERD 1992, MATSUMOTO e OKA 1997, citados por SILVA, 2002).
A Embrapa Mandioca e Fruticultura geralmente usa genótipos diplóide originários de M. acuminata em seu programa de melhoramento. Reprodução convencional em diplóides e triploides de Musa tem como resultado um baixo número ou ausência total de sementes, causado por uma falta de pólen viável, ou ineficiente polinização por parte dos insetos. Como conseqüência, cultivares comerciais Cavendish não produzem sementes quando polinizadas por diplóides resistentes. Um método usado para incorporar novas características em suscetíveis triplóides é o desenvolvimento de cultivares tetraplóides. Isso se dá através da fertilização entre pólen de machos haplóides com um gameta feminino triplóide, fazendo com que o pólen contribua com 25% para o novo genótipo (SILVA et al., 2002). O desenvolvimento de híbridos tetraplóides selecionados a partir de cruzamentos entre alguns cultivares triplóides semi-férteis e selvagens ou diplóides melhorados vem sendo a base de reprodução da banana, sendo esta a fonte da resistência a fatores bióticos e abióticos (CRESTE et al., 2006).
A transformação genética pode ser usada para obter plantas com melhor resistência a pragas, doenças e vírus, tolerância a herbicidas e características agronômicas. Existem duas categorias de sistemas de transformação. A primeira compreende a transformação indireta através da mediação por Agrobacterium tumefaciens e a outra, técnica direta de transferência do gene, em que se usam partícula de bombardeamento e eletroporação protoplástica. Esta tecnologia poderá produzir banana transgênica com resistência a viroses, pragas e doenças.
1.7. GENÔMICA DE MUSA
37
popularmente “transgênicos”. Esses avanços possibilitaram o desenvolvimento de bibliotecas de BAC para ambos os genomas A e B de banana.
O PROMUSA (Programa Internacional para o Melhoramento de Musa) foi
criado em 1997 para promover a cooperação internacional e facilitar a criação de sinergias entre os esforços de investigação em curso. O mesmo é dividido em grupos de trabalho com parcerias entre diversas instituições de pesquisa no mundo.
Os estudos de genômica em Musa têm conseguido diversos avanços através da contribuição de pesquisas com a citogenética molecular, identificação de marcadores moleculares, construção de mapas físico e genético, elucidação sobre os genes e sua expressão, bancos de dados de ESTs, análise de BACs e RGAs.
Pesquisas envolvendo a identificação de marcadores moleculares vêm sendo desenvolvida em Musa. Essa ferramenta molecular têm mostrado ser muito promissora. Por meio desta, é possível estabelecer mapas genéticos, bem como possibilitar uma seleção assistida por marcadores. Todavia, SNPs têm de ser acrescentados a esse repertório de marcadores, pois eles podem apresentar um impacto sobre a função das proteínas, especialmente quando aparecem em regiões codificantes. Além disso, marcadores moleculares podem ser usados para saturar mapas. O mapeamento visa identificar marcadores moleculares genética (mapas genéticos) ou fisicamente (mapas físicos) ligados a genes que contribuem para a manifestação de uma característica em particular ou continuamente variável de caracteres (por exemplo, um QTL). Assim, os marcadores podem ser explorados para isolar um gene de interesse, sendo estes, podendo ser utilizados para melhorar genótipos selecionados. Em Musa, pesquisas com mapeamento não estão muito avançadas, mas já existem vários grupos trabalhando nessa área.
38 Atualmente, 23.060 BAC ends de M. acuminata 'DH Pahang' estão em processo de seqüenciamento. Esse seqüenciamento completo do genoma da banana representará um enorme recurso para o estudo do genoma de Musa.
Numerosos RGAs pertencentes à família NBS-LRR foram recentemente caracterizados dos mais diversos cultivares dos genomas A e B de Musa (AZHAR & HESLOP-HARRISON, 2008). PEI et al. (2007), também ampliou essa busca na família non-TIR NBS em espécies selvagens de Musa, como Gongjiao, Xinyiyejiao, e espécies cultivadas como Zhongshandajiao, Fenjiao e Williams, utilizando primers previamente publicados para non-TIR NBS. ORTIZ-VÁSQUEZ et al., (2005) e ECHEVERRIA et al., (2007) também têm caracterizado o R gene Pto da família citoplasmática Ser / Thr quinase sem LRRs em M. acuminata.
MILLER et al (2008) tem utilizado seqüências ortólogas conservadas (COS), identificadas por meio de primers degenerados, como marcadores para a identificação de genes envolvidos na resistência à estresses bióticos. Este trabalho representa a primeira análise em grande escala de diversidade de RGAs em M. acuminata Calcutta 4 e oferece uma estratégia para o desenho de primers para RGAs/NBS-LRR.
A fim de contribuir para a descoberta de R-genes em Musa, um projeto colaborativo de genômica funcional está em curso entre a UCB, Embrapa, CIRAD e NIAS, analisando expressão gênica em M. acuminata Calcutta 4 (resistente) e M. acuminata Cavendish Grande Naine (suscetível) em folhas inoculadas in vitro com M . fijiensis. De 27648 clones preparados por biblioteca de cDNA, o seqüenciamento da extremidade 5' até agora tem sido realizado em mais de 8000 clones selecionados aleatoriamente por biblioteca (MILLER et al., 2008). Até o momento, alguns genes
39
2. OBJETIVOS
2.1. OBJETIVO GERAL
• Caracterizar análogos a genes de resistência (RGAs) em cultivares Lidi, Nyarma Yik, Pisang Berlin e Calcutta 4, de Musa acuminata contrastantes em resistência à estresses bióticos
2.2. OBJETIVOS ESPECÍFICOS
• Amplificar de forma especifica por PCR, RGAs em cultivares de M. acuminata contrastantes para resistência a Sigatoka negra utilizando primers degenerados desenhados a partir da família NBS-LRR;
• Desenhar primers degenerados para amplificação de RGAs da família gênica LRR extracelular com domínio transmembrana ligado com ser/thr quinases citoplasmáticas (RLK)
• Amplificar de forma especifica por PCR, RGAs (RLK) em cultivares de M. acuminata contrastantes para resistência a estresses bióticos
• Analisar através de técnicas de bioinformática seqüências de DNA: identificação de seqüências de DNA com similaridade a genes de resistência em bancos de dados de nucleotídeos e proteínas;
40
3. MATERIAL E MÉTODOS
O desenvolvimento metodológico guiou-se pelo esquema:
Figura 10 – Fluxograma resumindo a metodologia utilizada neste trabalho
3.1. EXTRAÇÃO DE DNA DE M. acuminata
41 com 400 µl de isopropanol a –20° C, adicionando em seguida a solução centrifugada a 12000 rpm por 15 minutos. O pelet resultante da centrifugação foi lavado em etanol 70% duas vezes e uma vez no etanol 100%. Logo após o pellet foi seco a temperatura ambiente e ressuspendido em 100 µl de tampão TE (Tris-HCl 10 mM, pH 8,0; EDTA 1 mM )
3.2. DESENHO DE PRIMERS DEGENERADOS
O desenho dos primers para PCR foi conduzido primeiramente por uma busca no Genbank (http://www.ncbi.nlm.nih.gov/Genbank/) de sequências contendo domínios conservados esperados para a família de genes de resistência de interesse RLK. Três seqüências principais de A. thaliana (gi:6522590), O. sativa (gi:1122442) e Triticum turgidum (gi:124378851) foram utilizadas e posteriormente analisadas por Blastx (ALTSCHUL et al., 1997 ), obtendo um total de 340 seqüências similares contendo os domínios de interesse. Estes domínios foram visualizados pelo programa Conserved Domain Database, v. 2.14 (Marchler-Bauer, 2007) e aquelas que não continham os domínios para resistência (RLK) foram eliminadas. Para a predição de região transmembrana foi utilizado o programa TMHMM v. 2.0 (KROGH et al., 2001 ), onde as seqüências verificadas sem esta região foram também excluídas.
Utilizando o programa CD HIT (Li et al., 2006) as redundâncias foram retiradas com um default igual a 90%. As seqüências foram alinhadas utilizando o programa MUSCLE (EDGARD, 2004) e para facilitar o processamento dos dados, as mesmas foram reorganizadas por nível de similaridade em subfamílias, utilizando o programa Clustering of Protein Sequences based on SMS a new similarity measure – CLUSS (ABDELLALI et al., 2007). Por fim, todas as seqüências contendo os domínios serviram como candidatas para o desenho de primers por meio do programa CODEHOP (ROSE et al, 1998).
42 Tabela 4 - Lista de primers desenhados para a família RLK. Em maiúsculo a região conservada e em minúsculo a região degenerada de cada par de primer.
Nome do Primer Seqüência do primer Subfamília CLUSS
RLK_S1_K_F 5’ CTGAGGAACATGCGGcaymgnaayyt 3’
1 RLK_S1_ID_R 5’ GCAGGAACCTGGCCadnccraartc 3’
1 RLK_S2_LRR_F 5’ CGTGATCTCCCCTCACrtnggnaaybt 3’
2 RLK_S2_LRR_R 5’ GAGAAGAAGTCCATGTTGswnadrttrta 3’
2 RLK_S3_K_F 5’ CTGAGGAGGATCCGGcaymgnaanyt 3’
3 RLK_S3_ID_R 5’ GGGGGCCACGTAGmcdatngtncc 3’
3 RLK_S4_K1_F 5’ CAGATCTCCCCCTCCbtnggnaayht 3
4 RLK_S4_ID_R 5’ GAAGATGCCCTTCGTGggnayntbncc 3’
4 RLK_S4_K2_F 5’ GCGGAACGTCCGGcaymgnaayht 3’
4 RLK_S4_ID2_R 5’ GAACATCTCCAGCAGCaynaynccraa 3’
4 RLK_S5_K_F 5’ GTAGCTGGACATGTCCtwyaayvrnyt 3’
5 RLK_S5_K_R 5’ GCTGCAGGTTGAACAcyttnayngcna 3’
5
Código degenerado de bases: I = inosine; R = A/G; Y = C/T; M = A/C; K = G/T; W = A/T; S = C/G; B = C/G/T; D = A/G/T; H = A/C/T; V = A/C/G; N = A/C/ G/T
Os primers foram desenhados utilizando diferentes padrões, como por exemplo, os níveis de degeneração ou até mesmo a localização de anelamento dos mesmos. A tabela 5 mostra os parâmetros utilizados no programa CODEHOP para o desenho dos primers.
Tabela 5 – Lista dos conjuntos de primers desenhados para a família RLK com diferentes padrões como níveis de degeneração, tamanho do produto esperado e local de anelamento.
Nome do Primer For/Rev Níveis de Degeneração Tamanho do produto esperado (pb) Localização Forward/Reverse
RLK_S1_K_F/RLK_S1_ID_R
128 297 Quinase/Inter domínio RLK_S2_LRR_F/RLK_S2_LRR_R
256 417 LRR/LRR
RLK_S3_K_F/RLK_S3_ID_R
128 405 Quinase/Inter domínio RLK_S4_K1_F/RLK_S4_ID_R
512 1512 Quinase/Inter domínio RLK_S4_K2_F/RLK_S4_ID2_R
128 504 Quinase/Inter domínio RLK_S5_K_F/RLK_S5_K_R
512 453 Quinase/Quinase
43 facilidade de anelar na região de interesse. Para contornar este problema e obter uma amplificação mais específica dos produtos esperados, foi adotado para os pares de primers das subfamílias 1, 3 e 4, sendo eles respectivamente, RLK_S1_K_F/ RLK_S1_ID_R, RLK_S3_K_F/RLK_S3_ID_R e RLK_S4_K2_F/RLK_S4_ID2_R, um parâmetro intermediário de degeneração (256) e por fim, um valor mínimo de degeneração (128) foi designado aos pares, RLK_S1_K_F/RLK_S1_ID_R e RLK_S4_K2_F/ RLK_S4_ID2_R.
A variação dos níveis de degeneração e a localização de anelamento possibilitam um maior espectro para os produtos de PCR, com maior ou menor especificidade, podendo obter diferentes domínios de interesse.
Outros primers degenerados direcionados às famílias NBS-LRR (Tabela 6) e primers específicos direcionados à família RLK em arroz (Tabela 7) também foram utilizados, porém os mesmos foram desenvolvidos em trabalhos anteriores.
Tabela 6 – Lista de primers degenerados direcionados à família NBS_LRR com suas respectivas seqüências e motivos conservados.
Nome do Primer
Seqüência do Primer Motivos
conservados
Autor
P1A (forward) GGIATGCCIGGIIIIGGIAARACIAC P-loop Bertioli et.al., 2003
P3A (reverse) AIITYIRIIRYIAGIGGYAAICC GLPL Bertioli et.al., 2003
P1B (forward) GGIATGGGIGGIIIIGGIAARACIAC P-loop Bertioli et.al., 2003 RNBSD-rev
(reverse)
GGRAAIARISHRCARTAIVIRAARC RNBS-D nao-TIR
Penuela et.al., 2002 1F (forward) GGCGGGGTGGGCaaracnacnht P-loop Miller et.al., 2008 P3B (reverse) AIITYIRIIRYIAGIGGIAGICC GLPL Miller et.al., 2008 3F2 (forward) GAGGTACTTCCTGGTGCTGgaygayrtbtgg Quinase 2 Miller et.al., 2008 13R1 (reverse) CGGCCAAGTCGTGCAyvakrtcrtgca LRR Miller et.al., 2008
Código degenerado de bases: I = inosine; R = A/G; Y = C/T; M = A/C; K = G/T; W = A/T; S = C/G; B = C/G/T; D = A/G/T; H = A/C/T; V = A/C/G; N = A/C/ G/T
Tabela 7 – Lista de primers específicos para espécie O. sativa com suas respectivas seqüências
Nome do Primer Seqüência do Primer Autor
RiceZ2 5’-ATTGAATAATTCACTGGGTATTGG-3’ Zai et. al., 2002 RiceZ3 5’-GTCTTGCCTTGCACTTCTGCACGA-3’ Zai et al., 2002
RiceKIN-1 5’ACTGGCCATCCTCTCATCACTCTAC-3’ Wang et.al.,2004 RiceKIN-2 5’TCAGATCGACTTCTGCAGTGGTAT-3’ Wang et.al., 2004
Rice3’Xa21R 5’GATCGGTATAACAGCAAAAC-3’ Wang et.al., 2004 RiceH3FragF 5’ATAGCAACTGATTGCTTGG-3’ Wang et.al.,2004
44 3.3. AMPLIFICAÇÃO DE RGAS POR PCR
Os conjuntos de primers sintetizados foram utilizados na amplificação por PCR, com o objetivo de obter produtos pertencentes às famílias NBS-LRR e RLK. O DNA genômico usado nas reações pertencem à espécie M. acuminata, com cultivares Calcutta 4 e Lidi, como fontes de resistência à Sigatoka negra, e Pisang Berlin e Nyama Yik sendo suscetíveis à Sigatoka negra.
As condições de PCR foram otimizados em termos de temperatura de anelamento, otimizada através de gradiente de PCR, e concentração final de reagentes BSA, DMSO, Taq e MgCl2. As reações foram realizadas com volume final de 12,5 µl, contendo 20 ng de DNA, 2.5 mM de MgCl2, 0.2 mM de dNTPs, 0.5µM do primer a ser utilizado, 1.25 unidades de DNA polimerase Taq Platinum (Phoneutria, Belo Horizonte, MG), e 1X de tampão da Taq Platinum (Phoneutria, Belo Horizonte, MG). As temperaturas dos ciclos de PCR foram de acordo com o seguinte programa: 94°C por 3 min; 35 ciclos de 94°C por 1 min, 45°C por 30s, 72°C por 1 min; e 10 min para extensão final a 72°C. Seguido da amplificação, os produtos de PCR foram visualizados sob luz U.V. em géis de agarose 1.5% corados com brometo de etídio a 0,15 µg/mL. Os marcadores de peso molecular 1Kb Plus DNA Ladder (Invitrogen, EUA ) e concentração de acido nucléico Low DNA Mass Ladder (Invitrogen, EUA) foram incluídos em todos os géis.
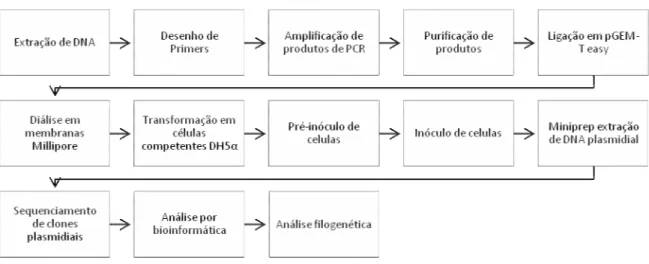
3.5. CLONAGEM E SEQUENCIAMENTO DE PRODUTOS DE PCR
45 transferida à uma cuveta e imediatamente levada ao eletroporador. Todo o procedimento foi feito com o material mantido no gelo.
Após a eletroporação, foi acrescentado 1mL de meio SOC na suspensão, sendo imediatamente incubada por 1 hora a 37°C. Em seguida, 100 e 200 µL de transformação foram plaqueados em meio LB (Luria-Bertani broth) Agar contendo o substrato Xgal (20mg/mL), o indutor do gene Lacz IPTG (0,1M) e ampicilina (100µg/mL). O material foi incubado a 37°C overnight.
Colônias brancas recombinantes foram selecionadas manualmente com até 96 clones preservados por combinação de primer e cultivar. Os transformantes foram então, inoculados em placa ELISA (Corning-costar 3799) com 128 µL de meio Circle Grow (CG) com ampicilina (100µg/mL) e incubados a 37°C por 15 horas (pré-inóculo). 50 ul de glicerol 20% foram adicionados a cada pré-inoculo, e as placas foram devidamente guardadas à temperatura de -80°C. O inóculo foi realizado em placas deep well contendo 1 mL de meio Circle Grow (CG) e ampicilina (100µg/mL) contendo 30 µL de pré-inóculo. O inóculo foi incubado a 37°C, 275 rpm, por cerca de 22 horas.
46 minutos e ressuspendido em 40 µL de água Milli-Q autoclavada. As placas foram armazenadas a -4ºC.
Os insertos de DNA plasmidial de 1.443 clones por combinação de primer foram seqüenciados unidirecionalmente em seqüenciadores ABI 377 e 3700 (Applied Biosystems, Foster City, CA, USA) utilizando respectivamente para cada sequenciador os kits DYEnamic ET Terminator Cycle Sequencing Kit (Amersham Biosciences, Piscataway, NJ, USA) e ABI BigDye Terminator Cycle Sequencing Kit (Applied Biosystems, Foster City, CA, USA).
3.6. ANÁLISE BIOINFORMÁTICA DE SEQÜÊNCIAS
Todos os procedimentos de processamento de seqüências abaixo citadas foram realizados na plataforma SISGEN (PAPPAS et al., 2008), por meio da compilação dos programas Cross-Match (http://www.phrap.org/phredphrap/general.html), SeqClean (CHEN et al., 2007), RepeatMasker (http://www.repeatmasker.org) e Lucy (CHOU & HOLMES, 2001).
Os dados gerados pelos seqüenciadores automáticos foram analisados, recebendo tratamentos especiais de mascaramento de vetor e regiões repetitivas, retirada de regiões de baixa complexidade, bem como o tratamento de qualidade com cortes de cauda PoliA/T. Os parâmetros utilizados para o controle de qualidade foram PHRED 15 e extensão mínima de 150 pb.
Posteriormente, as análises seguiram o fluxograma abaixo, Figura 11.
Figura 11 - Fluxograma resumindo as etapas de Pré-assembly.
47 procedimento foi realizado com o auxílio dos programas PHRAP v. 1.080812 e WCD (HAZELHURST et al., 2008). Posteriormente as seqüências foram submetidas ao programa AMOS (http://amos.sourceforge.net) onde sofreram marcações de regiões que possuíam mais de duas assinaturas de mis-assembly, isto é, regiões com baixa cobertura, alta cobertura ou alta incidência de polimorfismos. As seqüências marcadas foram excluídas.
Após todos os cuidados relacionados à qualidade das seqüências, foi realizado o clustering utilizando o montador genômico MIRA v. 3RC4 (CHEVREUX, 2004).
A descoberta de genes seguiu o fluxograma apresentado abaixo em Figura 12.
Figura 12 – Fluxograma resumindo as etapas de descoberta de genes.
49
4. RESULTADOS
4.1. AMPLIFICAÇÃO DE PRODUTOS DE PCR
4.2.1 Primers degenerados para a família de genes de resistência NBS-LRR
As combinações de primers P1A-P3A e 1F-P3B amplificaram produtos de aproximadamente 600 pb conforme esperado, por se localizarem em motivos conservados do domínio NBS que amplificam do motivo P-loop a GLPL. Os pares de primers P1B-RNBS-D localizados também em motivos conservados NBS geraram produtos de 500 pb amplificando os motivos P-loop e RNBS-D enquanto a combinação 3F2-13R1 amplificou a região que se inicia em um motivo quinase (domínio NBS) e termina em um motivo conservado do dominio LRR, obtendo produtos de cerca de 700 pb, como esperado (Figura 13).
Figura 13 - Regiões de anelamento de primers degenerados para a família NBS-LRR
4.2.2 Primers degenerados para a família de genes de resistência RLK
50 RLK_S5_R e RLK_S4_K2_F + RLK_S4_ID2_R, no qual o primeiro gerou bandas que variam de 700 pb a 2000 pb e o segundo gerou uma bandas de aproximadamente 400 pb e 450 pb. Nos dois casos, as duas bandas foram eluídas do gel e analisadas separaradamente.
Os primers específicos para Oryza (Zai et. al., 2002; Wang et.al., 2004 e Wang et.al., 2006) foram testados em Musa, porém não mostraram um resultado satisfatório, com a amplificação de não somente vários produtos de PCR, mas também amplificações não reproduzíveis. Os produtos conseqüentemente não foram seqüenciados e, portanto não utilizados nas análises posteriores (Tabela 8).
Tabela 8- Lista de primers degenerados testados e não utilizados nas análises e seus respectivos motivos de não uso.
Combinação de Primers Etapas Concluídas Motivo
P1A-P3A Amplificação por PCR, clonagem e seqüenciamento Não obteve similaridade a RGA RLK_S2_K_F / RLK_S2_ID_R Amplificação por PCR clonagem e seqüenciamento Não obteve similaridade a RGA RLK_S5_F / RLK_S5_R Amplificação por PCR, clonagem e seqüenciamento Não obteve similaridade a RGA RiceZ2/RiceZ3 Amplificação por PCR, clonagem e seqüenciamento Não obteve similaridade a RGA RiceKIN-1 / RiceKIN-2 Amplificação por PCR Produtos muito inespecíficos Rice3’Xa21R / RiceH3FragF Amplificação por PCR Produtos muito inespecíficos RiceXA21K_1 / RiceXA21K_2 Amplificação por PCR Produtos muito inespecíficos
4.3. PROCESSAMENTO DE SEQUÊNCIAS POR BIOINFORMÁTICA
Um total de 1443 sequências foram geradas, sendo 528 originadas a partir de primers degenerados direcionados à família de R genes NBS-LRR e 915 originadas a partir de primers degenerados direcionados à família de R genes RLK.
A submissão das seqüências ao Sistema Genoma (SISGEN) (PAPPAS, 2008) reduziu o número de seqüências para 699 aceitas, devido à exclusão das mesmas com qualidade inferior a PHRED 15 e seqüências com extensão inferior a 150 pb.
51 A figura 14 abaixo resume as etapas do processamento de seqüências por bioinformática.
A execução de pré-assembly iniciou com 417 seqüências totais para a família RLK, sendo 98 delas derivadas do cultivar Lidi, 82 de Pisang Berlin, 140 de Calcutta 4 e 97 de Nyarma Yik. Após retirada de possíveis erros de seqüenciamento, identificados pelos programas WCD e PHRAP, e também a remoção de seqüências com assinaturas de Misassembly (AMOS), restaram um total de 371 seqüências para a família RLK, sendo 67 derivadas do cultivar Lidi, 74 de Pisang Berlin, 93 de Nyarma Yik e 137 de Calcutta 4.
Figura 14 – Etapas do processamento de seqüências por bioinformática.
4.4. MONTAGEM DAS SEQUÊNCIAS EM CONTIGS
4.4.1. Contigs NBS-LRR