UNIVERSIDADE DE SÃO PAULO
INSTITUTO DE QUÍMICA
Programa de pós-graduação em Ciências Biológicas (Bioquímica)
JULIO CESAR LEVANO GARCIA
Caracterização bioquímica e imunológica das
enzimas recombinantes ATP-difosfohidrolases
1 e 2 do parasita
Schistosoma mansoni
São Paulo
JULIO CESAR LEVANO GARCIA
Caracterização bioquímica e imunológica das
enzimas recombinantes ATP-difosfohidrolases
1 e 2 do parasita
Schistosoma mansoni
Tese apresentada ao Instituto de
Química da Universidade de São Paulo
para obtenção do Título de Doutor em
Ciências (Bioquímica)
Orientador: Prof. Dr. Sergio Verjovski de Almeida
AGRADECIMENTOS
A Deus, que está sempre presente.
Agradeço ao Prof. Sergio Verjovski pela oportunidade de fazer doutorado e ter me apoiado e pela sua paciência. Agradeço muito por que sei que aprendi algumas coisas no seu laboratório e muitas outras que ainda preciso aprender, falando mais precisamente de virtudes.
Agradeço também a minha família que sempre me dá aquela forcinha. A meu pai Alejandro que sempre me aconselhou, a minha mãe pelo seu carinho e exemplo de pessoa, as minhas caras irmãs Miryam e Gladys, e ao meu grande irmão Ruben e os meus sobrinhos Bruno e Sebastian.
Aos Profs. Eduardo Moraes Reis e Ricardo DeMarco por seus conselhos e companheirismo. Aos meus colegas de laboratório. Ao Helder, Rodrigo e Thiago por serem bons amigos e os considero como irmãos. A Ana Claudia uma grande amiga e ótima colega de bancada, a Ana Paula por ter paciência comigo e ser muito gentil, a Katia e Giulliana por ser boas colegas, a Aninha Tahira, Ana Ayupe e Lauren. Um especial agradecimento a Camila que considero uma boa amiga, e sem esquecer da Ângela e Anna. Aos meninos Felipe, Carlos, Yuri, Renato e Jefferson. Um agradecimento muito especial a Erica Barroso, a minha namorada por ser ótima companheira e pela paciência comigo.
projeto de pesquisa, e sobre tudo por suas amizades. Agradeço também ao Prof. Paulo Lee Ho, Henrique, Leonardo e Silvia por sua hospitalidade, e por me deixar usar o espectropolarímetro dali.
Ao Prof. Renato Mortara da Escola Paulista de Medicina e Dra. Cybele Gargioni do Instituto Adolfo Lutz, por colaborarem com o meu projeto de pesquisa. Ao Prof. Alan Wilson e Simon Braschi da Universidade de York pela sua contribuição neste projeto.
Agradeço aos meus colegas do Instituto de Química, todas as pessoas que freqüentei, sobretudo aos funcionários da secretaria de pós-graduação da Bioquímica por serem bem atenciosos e pacientes comigo. Aos meus amigos peruanos Celso, Ricardo, Priscilla, Marcos, Miguel, César, Paola, Erick, Cesti, Juan, Fer, Mito e Diego. Aos meus amigos Brasileiros Diorge, Milton, Antero, Blenis, Paulo PCR, Paulinho, Marcelo e Miguelito.
A FAPESP, pelo seu apoio financeiro durante todo esse período. Com isto só me resta dizer MUITO OBRIGADO.
Posso ter defeitos, viver ansioso e ficar irritado algumas vezes, mas não esqueço que a minha vida é a maior empresa do mundo. E que posso evitar que ela vá à falência. Ser feliz é reconhecer que vale a pena viver, apesar de todos os desafios, incompreensões e períodos de crise. Ser feliz é deixar de ser vítima dos problemas e se tornar um autor da própria história. É atravessar desertos fora de si, mas ser capaz de encontrar um oásis no recôndito da sua alma. É agradecer a Deus a cada manhã pelo milagre da vida. Ser feliz é não ter medo dos próprios sentimentos. É saber falar de si mesmo. É ter coragem para ouvir um "não". É ter segurança para receber uma crítica, mesmo que injusta. Pedras no caminho? Guardo todas, um dia vou construir um castelo..."
RESUMO
Levano-Garcia, J. Caracterização bioquímica e imunológica das enzimas recombinantes ATP-difosfohidrolases 1 e 2 do parasita Schistosoma mansoni.
2008. 107p. Tese Doutoral. Programa de Pós-graduação em Bioquímica. Instituto de Química, Universidade de São Paulo, São Paulo.
ATPDases ou ATP-difosfohidrolases são enzimas que clivam o ATP e o ADP a AMP e Pi e estão envolvidos em inibição da agregação plaquetária. No parasita
Schistosoma mansoni nosso grupo identificou e clonou o gene da ATPDase1, e a proteína foi localizada na superfície do tegumento. Recentemente, clonamos o gene da ATPDase2 usando a informação do banco de dados de ESTs de S. mansoni e imunolocalizamos a sua proteína também no tegumento. ATPDase2 foi encontrada em ambas as membranas do tegumento basal e apical juntamente com a ATPDase1, entretanto ATPDase2 somente foi encontrada no espaço sincicial do tegumento. A presença de ambas as enzimas sobre a superfície externa do tegumento sugere um maior papel sobre a regulação de abundância de nucleotídeos. Análise da expressão de ambos os genes foram realizadas por RT-PCR em tempo real usando RNA de ovos, miracídeo, cercária, esquistossômulo e verme adulto. Os resultados mostraram que o gene da ATPDase1 foi mais expresso em ovos (7 vezes), adulto (6 vezes), cercária (3,5 vezes) e esquistossômulo (1,5 vezes) quando comparado ao miracídio, que foi tomado como referência. O gene da ATPDase2 foi mais expresso em ovos (16 vezes), cercária (11 vezes), miracídio (7 vezes) e verme adulto (2 vezes) quando comparado a esquistossômulo, mostrando que ambos os genes são modulados ao longo de seus estágios de ciclo de vida . Para maior caracterização destas enzimas, elas foram expressas heterologamente na levedura Pichia pastoris como proteínas de fusão com cauda de 6 histidinas e as proteínas recombinantes foram purificadas por cromatografia de afinidade com resina de Ni-NTA. As ATPDases recombinantes foram obtidas de forma ativa e medições de atividade enzimática foram realizadas. ATPDase1 mostrou atividades ATPásica e ADPásica em torno de 650 e 160 nmoles Pi.min
-1
UTPásica e UDPásica também foram encontradas nestas enzimas. Estudos de dicroísmo circular com estas duas enzimas elucidaram suas estruturas secundárias. Com isto, ATPDase1 (S66 to Q507) teve alfa-hélice (7 %), folha-beta (45 %) e estrutura randômica (48 %), e a ATPDase2 (N83 a K564) mostrou conter alfa-hélice (14 %), folha-beta (33 %) e estrutura randômica (53 %). Nós mostramos que a ATPDase2 é secretada pelo parasita no meio, de forma similar como descrito para as ATP-difosfohidrolases humanas CD39L2 e CD39L4. Adicionalmente, em ensaios de inibição de penetração (cercária em camundongo) usando anticorpo anti-ATPDase1 foi mostrada uma redução em 20 % da capacidade de penetração através da pele das cercárias previamente incubadas com o anti-soro. Devido a que a expressão do gene da ATPDase2 estava mais alta em miracídio e cercária, estágios que infectam caramujos e humanos, respectivamente, postulamos que ATPDase2 poderia ajudar no processo de invasão do parasita. No estágio ovo ambos os genes estão altamente expressados sugerindo um possível envolvimento das ATPDases na resposta de proteção contra o sistema imune humano. Ensaios de proteção contra S. mansoni em camundongos, usando as ATPDases 1 e 2 como antígenos, resultaram em uma baixa proteção obtendo-se não mais que 20% na redução da carga parasitária.
SUMMARY
Levano-Garcia, J. Biochemical and immunological characterization of ATP-diphosphohydrolases 1 and 2 from Schistosoma mansoni parasite. 2008. 107p. PhD Thesis - Graduate program in Biochemistry. Instituto de Química, Universidade de São Paulo, São Paulo.
ATPDases or ATP-diphosphohydrolases are enzymes that cleave ATP and ADP to AMP and Pi and are involved in inhibition of platelet aggregation. In the parasite Schistosoma mansoni our group had identified and cloned the ATPDase1 gene and localized the protein on the tegument surface. Recently, we cloned the
ATPDase2 gene using S. mansoni EST databank information and we
alpha helix (7%), beta sheet (45%) and random coil (48%), whereas ATPDase2 (N83 a K564) showed alpha helix (14%), beta sheet (33%) and random coil (53%). We found that ATPDase2 was secreted by the parasite to the medium, similar to what has been described for human CD39-L2 and CD39-L4 ATP-diphosphohydrolases. Additionally, a penetration assay (cercaria to mice) using antibody anti-ATPDase1 did show a decrease of 20% in the penetration capacity through mice skin of cercaria previously incubated with this antiserum. Because ATPDase2 gene expression was increased in miracidia and cercariae, the stages that infect snail and human, respectively, we postulate that ATPDase2 may help the parasite’s invasion process. In the egg stage both genes were highly expressed suggesting a possible involvement of the ATPDases in the protection response of eggs against the human immune system. Assays of protection against S. mansoni in mice, using recombinant ATPDase1 and 2 as antigens, resulted in low protection obtaining no more than 20% of parasite burden reduction.
Keywords: S. mansoni, ATP-diphosphohydrolases, P. pastoris, recombinant protein, antibody
ABREVIATURAS
AOX1 alcoolálcool oxidase gene 1
CFA adjuvante de Freund completo
Cy3 fluoróforo fluorescente vermelho da família das cyanines
dNTP deoxi-nucleostídeo trifosfatotrifosfatado
DTT dithiothreitol
EDTA ácido etilenodiamino tetra-acético
ELISA enzyme-linked immunosorbent assay
EST expression expressed sequence tag (etiqueta de sequência expressa)
F faraday
FITC Fluoresceine isothiocyanate, fluoróforo fluorescente verde
g gravidades
G418 antibiótico similar a canamicina
GFP green fluorescent protein
His+ células complementadas com o gene histidine dehidrogenase
HPR horsedarish peroxidase (peroxidase de rabanete)
IFA adjuvante de Freund incompleto
IgG imunoglobulina G
IPTG isopropyl-beta-D-thiogalactopyranoside
kDa quilodálton (s)
kV quilovolts
LB meio meio luria –broth
ml mililitro
mRNA RNA mensageiro
mU miliunidades
NDP nucleosídeo difosfatodifosfatado
ng nanogramas
N-terminal amino-terminal
NTP nucleosíideo trifosfatotrifosfatado
ºC graus Celsius
OD densidade óptica
OPD o-phenylenediamine
ORESTES Open Reading frame ESTs (ORESTES)
ORF open reading frame (fase aberta de leitura)
PAGE poliacrilamide polyacrylamide gel de electroforesiselectrophoresis (eletroforese em gel de poliacrilamida)
pb pares de bases
PBS solução salina tamponada com fosfato
PCR reação de polimerase em cadeia
pDNA DNA plasmidial
pH potencial de hidrogênio
Pi fosfato inorgânico
PMSF fluoreto de fenilmetilsulfonila
RACE rapid amplification cDNA ends
Real time RT-PCR PCR em tempo real
RNA ácido ribonucléico
Rpm rotações por minuto
RT-PCR transcrição reversa e PCR
s.c. subcutânea
SDS dodecil sulfato de sódio
SDS-PAGE eletroforese em gel de poliacrilamida com SDS
TAE tris, acetato, EDTA
TE tris, EDTA
TEMED N,N,N´,N´,-tetrametiletinelodiamida
Tris tris-hidroximetilaminometano
Triton X-100 polietilenglicol-terc-octilfenil éter
Trx tioredoxina
Tween 20 monolaurato de polioxietilenosorbitana
UTR região não traduzida do mRNA
V volt
X-Gal 5-bromo-4-cloro-3-indol-β-D-galactopiranosideo
YNB yeast nitrogen base (meio mínimo de suplemento de nitrogênio para levedura)
YPD yeast extract peptone dextrose (meio contendo extrato de levedura, peptona, dextrose)
β-ME mercaptoetanol
µg microgramas
LISTA DE TABELAS
Tabela 1.Nomenclatura da família E-NTPDase de membros mamíferos...8
Tabela 2.Metodologias de lavagens de corpos de inclusão...20
Tabela 3. Ensaio de proteção com SmATPDase 1: resposta humoral...74 Tabela 4. Desafio de camundongos C57BL/6 imunizados com a proteína
recombinante Apirase com diferentes adjuvantes...78
Tabela 5. Ensaio de proteção usando as SmATPDases 1 e 2 com o adjuvante
completo de Freund...81
LISTA DE FIGURAS
Figura 1. Schistosoma mansoni e áreas endêmicas com esquistossomose no
Brasil...1
Figura 2. Ciclo de vida do Parasita Schistosoma mansoni...2
Figura 3. Catabolismo de nucleotídeos extracelular localizados na superfície celular e ativação de receptores por nucleotídeos (P2 receptores) e adenosina (P1 receptores)...6
Figura 4. Árvore filogenética hipotética com 22 membros da família E-NTPDase (NTPDase1 a NTPDase8)...9
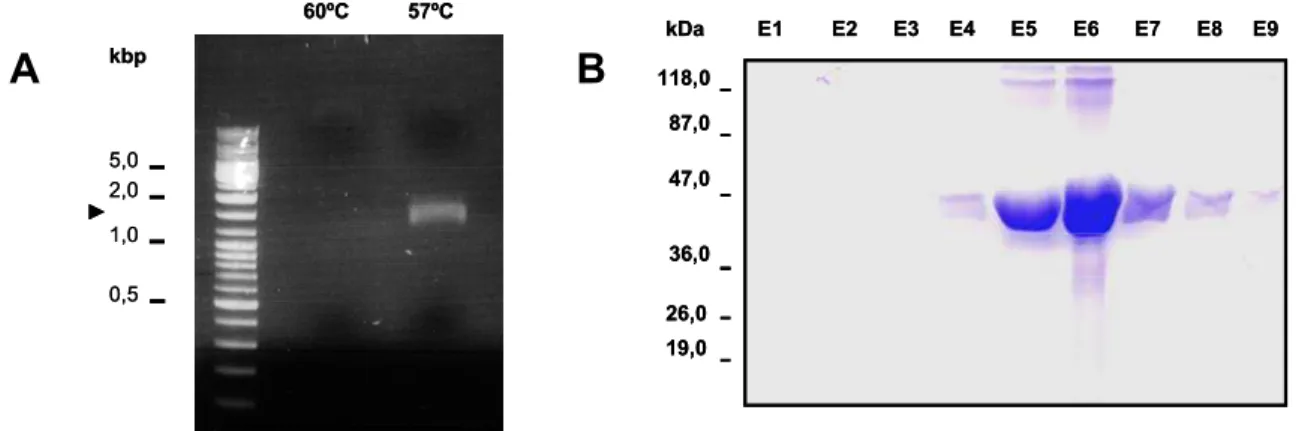
Figura 5. Expressão heteróloga de SmATPDase1 em E coli...39
Figura 6. Análise de expressão de APIRH em BL21(DE3)pLys sob diferentes temperaturas...39
Figura 7. Re-enovelamento e purificação de SmATPDase1...41
Figura 8. Análise de Expressão e purificação de APIRH-His8x em C41(DE3)...43
Figura 9. Construção da fusão GFP-SmATPDase1...44
Figura 10. Amplificação, reenovelamento e purificação de SmATPDase2. ...44
Figura 11. Amplificação da seqüência DNA que codifica Tioredoxina...45
Figura 12. Co-expressão de SmATPDase2 com tioredoxina usando baixa temperatura...46
Figura 13. Expressão de SmATPDase1 em P. pastoris GS115...47
Figura 14. Expressão e purificação de SmATPDase2 recombinante em P. pastoris ......49
Figura 15. Análise de glicosilação das enzimas recombinantes SmATPDase1 e 2 ...50
Figura 16. Efeito de compostos sobre a atividade ATPásica da enzima SmATPDase2...51
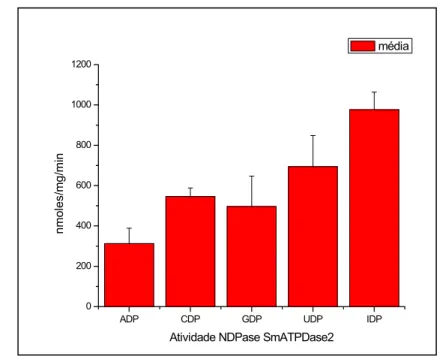
Figura 17. Atividade NTPásica das apirases recombinantes de S. mansoni......52
Figura 18A. Atividade NDPásica da apirase recombinante SmATPDase1...53
Figura 19 A. Efeito de cátions divalentes sobre a atividade ATPásica de
SmATPDase 1...55
Figura 19B. Efeito de cátions divalentes sobre a atividade ATPásica de
SmATPDase 2...56
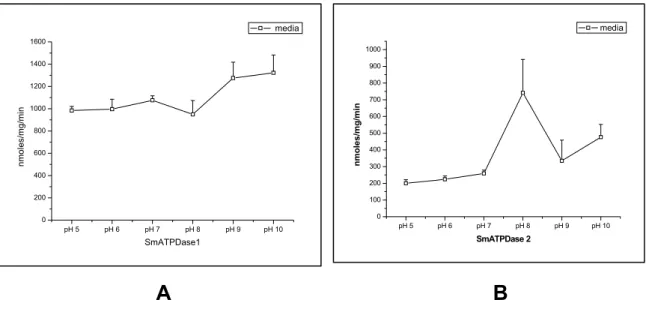
Figura 20. Efeito do pH na velocidade de hidrólise do substrato...57
Figura 21. Amplificação e coexpressão da seqüência de DNA que codifica para a chaperona PDI...58
Figura 22. Espectros CD de SmATPDase1 em 3 condições diferentes...60
Figura 23. Espectros CD de SmATPDase2 em 3 condições diferentes
...61
Figura 24. Formação de dímeros por tratamento com glutaraldeido... 62
Figura 25. Níveis de expressão de SmATPDase 1 e 2 em diferentes formas do ciclo de vida de S. mansoni determinado por Real-Time PCR...63
Figura 26A. Alinhamento de seqüências de aminoácidos de SmATPDase2 com outras apirases...65
Figura 26B. Árvore filogenética com várias ATP-difosfohidrolases de diferentes organismos... ...66
Figura 27. Amplificação e expressão dos fragmentos de DNA APIMEM e APISOL ...67
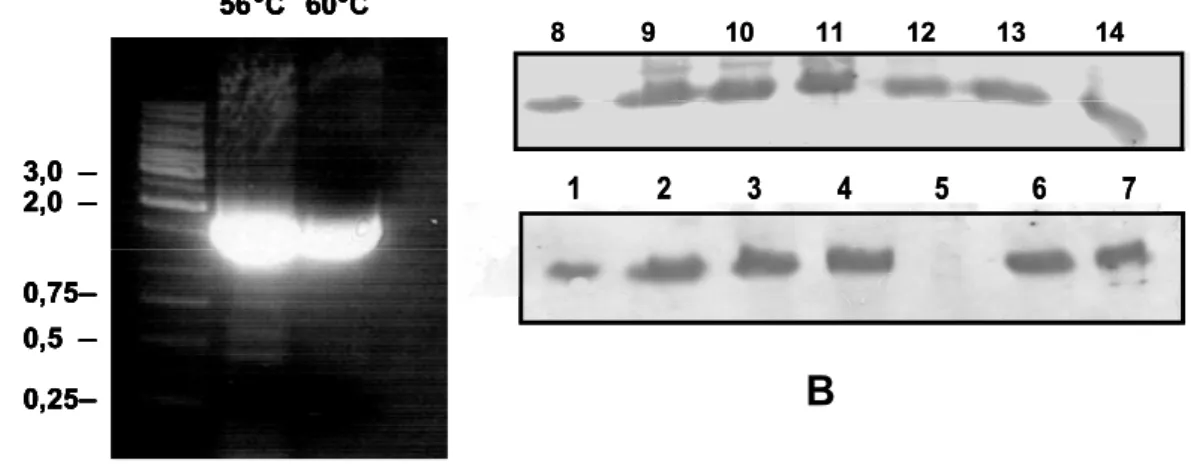
Figura 28. Purificação negativa de antisoros Anti-SmATPDase1 e 2...68
Figura 29. Detecção de SmATPDase 1 e 2 em frações solúvel e insolúvel do
tegumento do parasita...69
Figura 30A. Co-imunolocalização de SmATPDase1 e 2 em parasitas S. mansoni inteiros permeabilizados e cortes histológicos...71
Figura 30B. Desenho de co-imunolocalização das SmATPDase1 e 2 em parasitas
S. mansoni em corte histológico...72
Figura 31. Detecção de SmATPDase2 no sobrenadante de culturas in vitro de
vermes adultos S. mansoni...73
Figura 32. Ensaio de proteção com SmATPDase1: resposta humoral...74
Figura 33. Produção de anticorpos IgG anti-Apirase em camundongos imunizados com a proteína SmATPDase1 recombinante...75
Figura 35A. Isotipagem das IgG1 da sangria 4 de camundongos imunizados com
SmATPDase1 recombinante...77
Figura 35B. Isotipagem das IgG2a da sangria 4 de camundongos imunizados com SmATPDase1 recombinante. Titulação de IgG2a (resposta celular)...78
Figura 36. Ensaio de proteção 2...79
Figura 37. Titulação de IgG do ensaio de proteção 3... 80
Figura 38. Ensaio de proteção 3...81
Figura 39. Ensaio preliminar de proteção usando vacina de DNA...82
Figura 40. ELISPOT dos animais imunizados com a vacina de DNA...84
Figura 41. Efeito da concentração de apirase recombinante no ELISPOT dos animais imunizados com a vacina de DNA...84
Figura 42. Determinação do número de células produtoras de IFN- , a partir da cultura de esplenócitos de camundongos imunizados com a vacina de DNA...85
Figura 43. Gráfico de correlação entre a porcentagem de redução da carga parasitária em função da polarização da resposta imune obtida...86
Figura 44. Gráfico de correlação entre a porcentagem de redução da carga parasitária e o número de células secretoras das citocinas IL-4 e IFN-γ...87
ÍNDICE
1. INTRODUÇÃO...1
1.1 - A esquistossomose...1
1.2 - Nucleotídeos extracelulares como moléculas sinalizadoras...5
1.3 - As Ecto-nucleosídeo trifosfato difosfohidrolases (E-NTPDases)...7
1.4 - ATP-difosfohidrolases (ATPDases) de Schistosoma mansoni.....11
2. OBJETIVOS...14
3. MATERIAIS E METODOS...15
3. 1 - Manutenção de parasitas...15
3. 2 - Hospedeiros...15
3. 3 - Vetores...16
3. 4 - Amplificação e clonagem de SmATPDase1 ...16
3. 5 - Expressão de SmATPDase1 recombinante em E. coli......18
3. 6 - Lavagem e desnaturação de corpos de inclusão...19
3. 7 - Re-enovelamento de Proteínas desnaturadas...21
3.7.1 - Re-enovelamento por diálise de proteínas desnaturadas em Guanidina......21
3.7.2 - Re-enovelamento por diluição de proteínas desnaturadas com Guanidina...21
3.7.3 - Re-enovelamento ligado a uma matriz...21
3. 8 - Amplificação e clonagem da SmATPDase2...22
3. 9 - Clonagem e expressão da tioredoxin (Trx)...23
3. 11 - Purificação de proteínas recombinantes em E. coli......25
3. 11. 1 - Cromatografia de afinidade...25
3. 11. 2 - Cromatografia de troca iônica...25
3. 12 - Expressão de proteínas recombinantes em Pichia pastoris ...25
3. 13 - Purificação de SmATPDase1 e 2 do sobrenadante da cultura de P. pastoris...27
3. 14 - Clonagem e expressão da proteína dissulfeto isomerase (PDI) ...27
3. 15 - Caracterização enzimática de SmATPDases 1 e 2 recombinantes de S. mansoni...28
3. 16 - Dicroísmo circular...29
3. 17 - Western blot...29
3. 18 - Deglicosilação de apirase recombinante ...30
3. 19 - Imunização de camundongos e desafio com cercárias...31
3. 19. 1 - Imunização com Proteínas....31
3. 19. 2 - Imunização com plasmídeo (vacina de DNA)...32
3. 20 - ELISA...33
3. 21 - PCR em Tempo Real (Real Time PCR)...34
3. 22 - Imunofluorescência de parasitas inteiros e cortes histológicos ...34
3. 23 - Cultura in vitro de vermes adultos de S. mansoni...35
3. 24 - Ensaio de inibição de penetração pela pele...37
3. 25 - Cross-linking com glutaraldeido...37
4. RESULTADOS ...38
4.1 - Clonagem, expressão, purificação de SmATPDase1 ...38
4. 1. 1 - Construção I: Apirase recombinante com cauda de histidina...38
4. 1. 3 - Construção II : Proteína recombinante com cauda de histidina...42
4. 1. 4 - Construção III: Proteína recombinante de fusão com GFP...43
4. 2 - Amplificação e expressão de SmATPDase2 ...44
4. 3 - Construção do vetor de expressão com Tioredoxin (Trx) ...45
4. 4 - Co-expressão de SmATPDase2 com Tioredoxina em E. coli...46
4. 5 - Expressão e purificação de SmATPDase 1 e 2 recombinante no sistema P. pastoris...47
4. 6 - Deglicosilação de proteínas recombinantes...49
4. 7 - Atividade enzimática...50
4. 8 - Construção do vetor de expressão com Proteína dissulfeto isomerase e co-expressão com SmATPDase1 em P. pastoris ...58
4. 9 - Análise da estrutura secundaria das SmATPDases recombinantes por dicroísmo circular...59
4. 9. 1 - SmATPDase 1 e SmATPDase 2 formam dímeros...61
4. 10 - Análises dos níveis de expressão de SmATPDases 1 e 2...62
4. 11 - Análise filogenética de SmATPDase2...63
4. 12 - Expressão de fragmentos de SmATPDAse1 e 2 com baixa similaridade...66
4. 13 - Western Blot e imunolocalização de SmATPDases 1 e 2 de S. mansoni...68
4. 14 - SmATPDase2 é secretada pelo parasita no meio...72
4. 15 - Ensaios de Proteção contra Schistosoma usando SmATPDase1 e 2...73
4. 15. 1 - Ensaio de proteção usando SmATPDase recombinante: resposta humoral e ensaio funcional...73
4. 15. 1. 1 - Ensaio de proteção 1...74
4. 15. 1. 3 - Ensaio de Proteção 3...79
4. 15. 2 - Ensaio de proteção usando SmATPDase1 como Vacina de DNA...82
. 4. 15. 3 - Avaliação da resposta humoral em animais imunizados com a vacina de DNA SmATPDase1/pTARGET...83
4. 15. 4 - Avaliação da resposta celular em animais imunizados com SmATPDase1/ pTARGET (ELISPOT)...83
4. 16 - Ensaio de inibição de penetração pela pele...88
5. DISCUSSÃO...90
6. CONCLUSÃO...99
7. REFERÊNCIAS...100
LISTA DE ANEXOS
-Sumula curricular
1. INTRODUÇÃO
1. 1 - A esquistossomose
A esquistossomose é uma doença parasitária crônica, debilitante e, em cerca de 10% dos casos, fatal. Afeta principalmente indivíduos em áreas rurais, sendo endêmica em países tropicais e subtropicais. Ela constitui, na atualidade, um dos maiores problemas de saúde pública em termos globais, afetando em torno de 200 milhões de pessoas, e colocando em risco de infecção outras 650 milhões (WHO 2002). Esta doença é causada por um platelminto do gênero Schistosoma, sendo a espécie Schistosoma mansoni a única encontrada no Brasil (figura 1). Mesmo assim, estima-se que mais de 6 milhões de indivíduos estejam infectados, só neste país (http://www.fiocruz.br). Esta espécie também é encontrada na África e no Oriente médio.
Fig 1. Schistosoma mansoni e áreas endêmicas com esquistossomose no Brasil. (A) Distribuição
geográfica de áreas endêmicas com esquistossomose. Fonte: GT-Esquistossomose/CDTV/CGDT/SVS/MS. (Guia de vigilância epidemiológica) (Brasil 2005) (B) Microscopia eletrônica de varredura do parasita
Espécies deste gênero são encontradas em todo o mundo, como
Schistosoma haematobium na África e leste do Mediterrâneo e Schistosoma japonicum, no sudeste Asiático e no Pacifico Ocidental.
No ciclo de vida deste parasita, é necessária a passagem por dois hospedeiros, sendo o hospedeiro intermediário um gastrópode terrestre do gênero Biomphalaria e o definitivo, o ser humano. No caramujo, o parasita evolui da forma de miracídio à forma de cercária, passando por uma fase intermédia chamada esporocisto. Neste último estágio, o parasita consegue penetrar ativamente, através da pele, no hospedeiro humano, migrando até seus vasos sanguíneos, onde se desenvolverá até sua forma adulta, podendo viver ali por vários anos (Vermund et al. 1983).
A doença tem uma fase aguda e outra crônica. Na fase aguda, pode apresentar manifestações clínicas como coceiras e dermatites, febre, inapetência, tosse, diarréia, enjôos, vômitos e emagrecimento. Na fase crônica, geralmente assintomática, episódios de diarréia podem alternar-se com períodos de obstipação (prisão de ventre); e a doença pode evoluir para um quadro mais grave com aumento do fígado (hepatomegalia) e cirrose, aumento do baço (esplenomegalia), hemorragias provocadas por rompimento de veias do esôfago, e ascite ou barriga d’água, isto é, o abdômen fica dilatado e proeminente porque escapa plasma do sangue. As manifestações patológicas estão predominantemente relacionadas à deposição de numerosos ovos dos parasitas nos tecidos do hospedeiro, levando à resposta granulomatosa (Cheever et al. 2000).
estes mecanismos ainda não são bem conhecidos, sendo sua elucidação muito significante para o entendimento da relação parasita-hospedeiro.
Atualmente, existem medicamentos efetivos contra o parasita, como é o caso de Praziquantel cuja ação está associada à indução do influxo de cálcio através da membrana do tegumento. Este influxo causa uma rápida contração muscular e vascular do tegumento, matando desta forma o parasita (Kohler 2001). A esquistossomose tem progressão lenta e, na maioria dos casos, as pessoas infectadas procuram assistência médica apenas quando exibem a condição de hepatosplenomegalia, sendo, neste ponto, ineficaz o uso de medicamentos (Wilson e Coulson 1999). Outro aspecto fortemente negativo do tratamento por drogas é que o uso de medicamento evidentemente não impede a re-infecção dos indivíduos que vivem em áreas endêmicas. Além disso, em regiões onde foram realizados tratamentos em larga escala, já foram detectadas cepas resistentes ao Praziquantel (Ismail et al. 1996; Liang et al. 2001), embora o mecanismo de seleção de vermes resistentes em campo esteja sob discussão (Doenhoff et al. 2002).
Nas plaquetas foi observada uma ação contra esquistossômulo (Bout et al.
1986). A função do ADP como mediador primário de ativação de plaquetas já foi descrita, sendo que o ADP com efeito agregatório pode ser liberado na circulação sanguínea após danos nos tecidos (Marcus et al. 1997). Por outro lado, enzimas capazes de degradar o ADP exógeno têm sido encontradas na superfície externa de células do endotélio vascular e células sanguíneas como leucócitos, eritrócitos e plaquetas (Cote et al. 1991) tendo uma implicação na limitação da agregação plaquetária (Enjyoji et al. 1999).
1.2 - Nucleotídeos extracelulares como moléculas sinalizadoras
Nucleotídeos extracelulares possuem muita importância para o hospedeiro por representarem a substância de sinalização mais abundante entre células. Além disso, estas substâncias produzem respostas fisiológicas em cada tecido, sendo que estes exercem sua função através de receptores purinérgicos localizados na superfície celular (Chen et al. 1995). Isso demonstraria que no caso do ATP extracelular este tem diferentes efeitos em muitos processos biológicos, sendo os principais: contração do músculo liso, neurotransmissão, resposta imune, inflamação e dor (Ralevic e Burnstock 1998; Sitkovsky 1998; Sneddon et al. 1999; Ding et al.
Os nucleotídeos ATP e ADP podem ativar os neutrófilos e estimular a liberação de óxido nítrico (NO) de células endoteliais. No entanto, outros estudos mostraram que estes mesmos nucleotídeos secretados por plaquetas, glóbulos brancos e células endoteliais danificadas, mediam o processo de inflamação e trombose vascular observados durante rejeição a órgãos estranhos (Imai et al.
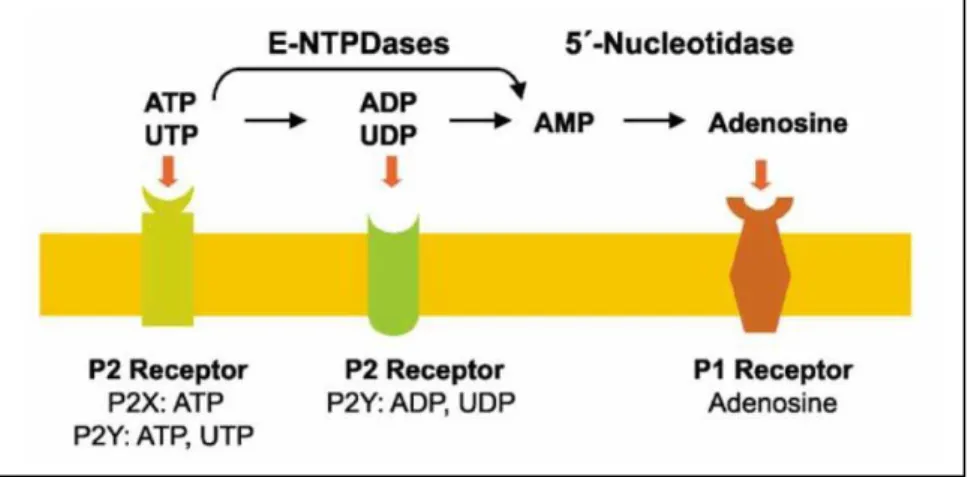
2000). Em muitos tecidos, concentrações extracelulares de ATP e ADP, assim como de outros nucleosídeos di e trifosfatados, parecem ser reguladas pela ação de um grupo de enzimas ligadas à membrana que hidrolisam nucleotídeos sendo uma destas a enzima ecto-ATP difosfohidrolase ou ecto-apirase (Zimmermann 1996). Essencialmente cada tipo celular em um organismo pode liberar estes mediadores, os quais vão sinalizar através de receptores para adenosina ou outros nucleotídeos localizados na superfície celular (figura 3).
Ao todo, duas famílias de receptores distintos estruturalmente têm sido identificadas e caracterizadas: os receptores acoplados à proteína G (P2Y) ou metabotrópicos, e os receptores com canais iônicos (P2X) ou ionotrópicos
Figura 3 .Catabolismo de nucleotídeos extracelular localizados na superficie celular e ativação
de receptores por nucleotídeos (P2 receptores) e adenosina (P1 receptores). A figura mostra as
(Fredholm e Altiok 1994; Buell et al. 1996; Atkinson et al. 2004). Os receptores P2X respondem a ATP, receptores P2Y podem ser ativados por ATP, ADP, UTP, UDP, ITP, e açúcares de nucleotídeos (Lazarowski et al. 2003) (figura 3).
1.3 – As Ecto-nucleosídeo trifosfato difosfohidrolases (E-NTPDases)
E-NTPDases (EC 3.6.1.5) ou também chamadas ATP-difosfohidrolases, ATPDases ou Apirases, são ecto-enzimas que hidrolisam nucleosídeos di e trifosfatados extracelulares e têm um definido perfil farmacológico. Elas são ativadas por elevadas concentrações de Ca2+ ou Mg2+ e não são inibidas pelos inibidores clássicos de ATPases intracelulares como as do tipo P, tipo F e tipo V ou fosfatase
alcalina (Vasconcelos et al. 1993; Plesner 1995; Zimmermann 1996).
Diferentemente das ATPases que hidrolisam apenas ATP, as apirases geralmente hidrolisam tanto ATP quanto ADP. Os produtos da hidrólise de ATP por apirase são AMP e dois ânions ortofosfato. A cadeia ou cascata ectonucleotidásica, iniciada por NTPDases, pode ser terminada pela ecto-5´-nucleotidase (CD73; EC 3.1.3.5) com hidrólise de nucleosídeos monofosfatados (Zimmermann 1992).
protótipo desta família de enzimas foi primeiro clonado e identificado como um antígeno de ativação celular linfóide (CD39) com função desconhecida (Maliszewski
et al. 1994).
Tabela 1. Nomenclatura da família E-NTPDase de mamíferos.
Subtipos de NTPDases tem diferenças em localização e propriedades funcionais. Com isso, quatro tipos foram localizados na superfície da célula (NTPDase1, 2, 3 e 8), os quais podem ser diferenciados de acordo à preferência pelo substrato, cátion divalente e formação de produto. Todas as NTPDases localizadas na superfície precisam dos íons Ca2+ ou Mg2+ na faixa milimolar para ter máxima atividade, e são inativas na ausência destes (Kukulski et al. 2004). Estas enzimas hidrolisam nucleosídeos trifosfatados incluindo os fisiologicamente ativos Nome da
proteína
Nome adicional Nome do gene
humano, e de camundongo
Número de acesso humano, e de camundongo NTPDase 1 CD39, ATPDase,
ectoapirase
ENTPD1, Entpd1 U87967, NM_009848 NTPDase 2 CD39L1, ecto-ATPase ENTPD2, Entpd2 AF144748,
AY376711
NTPDase 3 CD39L3, HB6 ENTPD3, Entpd3 AF034840,
AY376710
NTPDase 4 UDPase, LALP70 ENTPD4, Entpd4 AF016032,
NM_026174 NTPDase 5 CD39L4, ER-UDPase,
PCPH
ENTPD5, Entpd5 AF039918, AJ238636
NTPDase 6 CD39L2 ENTPD6, Entpd6 AY327581,
NM_17217
NTPDase 7 LALP1 ENTPD7, Entpd7 AF269255,
AF288221
NTPDase 8 hATPDase ENTPD8, Entpd8 AY430414,
AY364442
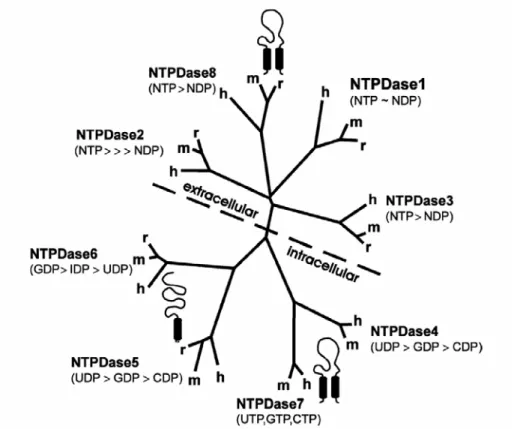
ATP e UTP. Notavelmente, a velocidade de hidrólise para nucleosídeos difosfatados varia consideravelmente entre subtipos (figura 4).
Adicionalmente, dados experimentais mostraram que enzimas do tipo NTPDase1 hidrolisam ATP e ADP em iguais proporções, e enzimas NTPDase3 e 8 revelaram ter maior hidrólise sobre ATP do que ADP (figura 4). Enzimas do tipo NTPDase2 tem uma elevada preferência pelos nucleosídeos trifosfatados, embora tenha sido classificada anteriormente como uma ecto-ATPase (Kirley et al. 2006). Com tudo, é muito possível que as diferenças entre suas seqüências e também entre suas estruturas secundária, terciária ou quaternária tenham influencia sobre as variações nas propriedades catalíticas dos subtipos de enzimas.
Figura 4. Árvore filogenética hipotética com 22 membros da família E-NTPDase
(NTPDase1 a NTPDase8). Esta árvore está baseada no alinhamento de seqüência de
As seqüências de identificação de todas as NTPDases são os cinco domínios altamente conservados conhecidos como “apyrase conserved regions” (ACR), os quais são chamados ACR1 até ACR5 (Handa e Guidotti 1996; Vasconcelos et al.
1996). Uma das primeiras enzimas desta família a ser caracterizada foi a apirase de batata, uma enzima de 49 kDa (Traverso-Cori et al. 1965). Enzimas ATP-difosfohidrolases ou apirases têm sido encontradas também na saliva de insetos hematófagos, onde estas enzimas possuem propriedades anti-hemostáticas (Reno e
Novak 2005). Estudos com a ATP-difosfohidrolase de placenta humana
(Christoforidis et al. 1995) mostraram que esta proteína era idêntica ao antígeno de ativação celular linfóide (CD39) (Maliszewski et al. 1994); esta mesma proteína CD39 foi encontrada inicialmente na superfície de células endoteliais e células derivadas de macrófagos (Kansas et al. 1991). A relação entre a proteína CD39 e a atividade ATP-difosfohidrolase foi demonstrada por estudos onde a expressão heteróloga de CD39 em células COS-7 levou a um incremento nas atividades ATPásica e ADPásica dependentes de Ca2+ ou Mg2+ (Kaczmarek et al. 1996; Wang e Guidotti 1996).
Entretanto, pouco é conhecido acerca da função das endo-apirases sendo sugerido envolvimento na glicosilação de proteínas (Gao et al. 1999; Uccelletti et al.
2007), e virulência do patógeno Legionella pneumophila (Sansom et al. 2007).
caracterizada como uma proteína tetramérica não covalente (Wang et al. 1998) que hidrolisa os dois substratos ATP e ADP com igual eficiência, enquanto a enzima ATPase somente hidrolisa o ATP (Atkinson et al. 2006).
1.4 - ATP-difosfohidrolases (ATPDases) de Schistosoma mansoni
No parasita Schistosoma mansoni foi possível purificar e caracterizar parcialmente uma proteína de superfície de aproximadamente 63 kDa no tegumento, que mostrava uma atividade ATP-difosfohidrolase (apirase, EC 3.6.1.5) sendo a primeira demonstração da presença deste tipo de enzima na superfície de um endoparasita (Vasconcelos et al. 1996; DeMarco et al. 2003). Esta enzima foi localizada na superfície externa do tegumento do parasita, e baseado na importância do ADP no processo de ativação das plaquetas foi proposto que a enzima ATP-difosfohidrolase poderia participar do mecanismo de escape do parasita à resposta do hospedeiro clivando ATP ou ADP, os quais poderiam ser liberados por plaquetas ativadas (Vasconcelos et al. 1993) ou linfócitos T citotóxicos. Interessantemente, outros estudos mostraram a importância da presença de enzimas que clivam nucleotídeos na superfície de patógenos na relação com o seu hospedeiro, mostrando a presença de uma enzima com atividade ecto-ATPase, dependente de magnésio, na superfície externa do parasita Leishmania tropica
(Meyer-Fernandes et al. 1997). Uma enzima similar a ATP-difosfohidrolase foi também demonstrada ser secretada no vacúolo celular de Toxoplasma gondii
ATP-difosfohidrolase 1 de S. mansoni compartilha várias características estruturais e funcionais com outras ATP-difosfohidrolases de diferentes origens, tendo especificidade por diferentes nucleotídeos (Vasconcelos et al. 1993; Vasconcelos et al. 1996).
Em nosso laboratório, foi possível clonar a seqüência de DNA que codifica a enzima ATP-difosfohidrolase 1 do parasita S. mansoni (DeMarco et al. 2003). Nesta enzima foi predita uma topologia com duas regiões transmembrânicas localizadas próximas às pontas amino e carboxilo terminal, mantendo similaridade com a topologia descrita para as proteínas CD39. Além disso, foi sugerido também que a região hidrofílica, localizada entre estas duas regiões transmembrana, estaria voltada para o lado externo da membrana.
Uma importante característica das ecto-apirases de vertebrados é a sensibilidade de sua atividade enzimática a muitos detergentes. As atividades enzimáticas de ATPases tipo E solúveis, incluindo a apirase de batata (S. tuberosum) (Kettlun et al. 2005) e as ecto-ATPases de parasitas (Smith et al. 1997) não são afetadas por detergentes, sendo a principal diferença entre as estruturas das ecto-apirases de mamíferos (sensíveis a detergente) e as apirases solúveis (resistentes a detergente) a presença de segmentos transmembrana, o que sugere que a interação dos domínios transmembrana com detergentes determina a sua sensibilidade.
Muitos trabalhos com apirases mostram que estas são proteínas glicosiladas (Plesner 1995; Mulero et al. 2000), sendo encontrados 7 e 6 potenciais sítios de N -glicosilação na ATP-difosfohidrolase de cérebro e ecto-apirase CD39 humana, respectivamente. Experiências com a apirase de cérebro humano mostraram que a deglicosilação da enzima tinha como conseqüência a perda de sua atividade (Smith
2. OBJETIVOS
Expressão heteróloga da região hidrofílica da SmATPDase1 no hospedeiro E.
coli.
Expressão heteróloga da região hidrofílica da SmATPDase1 no hospedeiro
Pichia pastoris
Clonagem de uma segunda isoforma de ATPDase do S. mansoni
Co-imunolocalização das duas SmATPDases no tegumento do parasita.
Análise de expressão dos genes SmATPDase1 e 2 nos diferentes estágios do
parasita.
Purificação das SmATPDases 1 e 2 recombinantes
Caracterização enzimática das proteínas purificadas
Caracterização imunológica das duas SmATPDases recombinantes usando o
3. MATERIAIS E MÉTODOS
3. 1 - Manutenção de parasitas
S. mansoni cepa BH foram mantidos por sucessivas infecções em camundongos com injeções subcutâneas de cercárias. Vermes adultos foram obtidos por perfusão de camundongos após 7-8 semanas de infecção. Os ovos foram obtidos a partir de fígados de camundongos infectados, e os miracídios foram obtidos a partir destes ovos purificados (Dalton et al. 1997). As formas cercárias foram obtidas após 30 dias de infecção de caramujos do gênero Biomphalaria. As formas esquistossômulo foram obtidas de culturas de cercárias in vitro por 7 dias (Basch 1981). Os parasitas em suas formas ovo, miracídio, cercária e esquistossômulo foram obtidos em nosso laboratório em colaboração com as doutorandas Kátia C. Oliveira e Giulliana Tessarini Almeida. Os parasitas adultos foram fornecidos pela Dra. Cybele Gargioni do Instituto Adolfo Lutz (Secção de enteroparasitoses).
3. 2 - Hospedeiros
A cepa E. coli DH10B (Gibco BRL) foi usada para transformação e propagação de plasmídeos recombinantes. As cepas E. coli BL21(DE3), Origami(DE3) (Novagen), C41(DE3) (Avidis) e ArcticExpress (DE3)RIL (Cpn10-Cpn60) foram usadas para estudos de expressão em hospedeiro procarionte.
3. 3 - Vetores
O vetor pGEM-T (Promega) foi usado para a clonagem dos produtos de PCR, e os vetores pET21b, pET36b (Novagen) e pAZ foram usados para a expressão intracelular em E. coli das seqüências que codificam para as regiões hidrofílicas e região com baixa similaridade entre ambas as SmATPDases 1 e 2. O vetor recombinante pAE-Trx codificando para Tioredoxina foi usado para co-expressão junto a SmATPDase2 em E. coli. O vetor pPIC9K (Invitrogen) foi usado para a expressão e secreção no meio de cultura das SmATPDases 1 e 2 recombinantes, e o vetor pPICHOLI-1 (Mobitech) foi usado para a expressão intracelular da seqüência que codifica para a proteína dissulfeto isomerase (PDI) em P. pastoris, e foi usado em testes de co-expressão com ambas as SmATPDases1 e 2. O vetor pTARGET (Promega) foi usado para a expressão intracelular das seqüências que codificam para as SmATPDases 1 e 2 inteiras (incluindo regiões transmembrana) em células de mamífero.
3. 4 - Amplificação e clonagem de SmATPDase1
As construções feitas contendo a seqüência de DNA que codifica para o domínio hidrofílico de SmATPDase1 (APIRH) foram realizadas usando como molde a construção pMIclone4/pET21b (apirase inteira), feita previamente no nosso laboratório (DeMarco et al. 2003). A seqüência de DNA que codifica a região
hidrofílica (S66 até Q507) foi amplificada usando os oligos
5´-TGAATTCTTATTGTGTACTGACAGA-3´ os quais continham os sítios de restrição (sublinhados) para Kpn I e EcoR I respectivamente, para a clonagem no vetor pAZ (em fusão com GFP). Adicionalmente, foram usados os oligos 5´-GCGTACGTAAGTAGTAACATTCC-3´ (“forward”), 5´-TAGAATTCTTAGTGGTGGT GGTGGTGGTGTTGTGTACTGACAGA-3´ (“reverse”) os quais continham os sítios
SnaB I e EcoR I para amplificação de APIRH e clonagem no vetor pPIC9K. As amplificações foram realizadas com a Advantage 2 polymerase (Clontech) usando,
20 pmol de cada oligo e 200 µM de dNTPs nas seguintes ciclagens: 95°C (1 min);
30 ciclos de 95°C (30 seg), 55°C (30 seg) e 68°C (3 min), seguido de uma extensão final de 68°C (3 min). A segunda amplificação de APIRH, para clonagem no vetor pAZ, foi com passo inicial de 95°C (1 min); 30 ciclos de 95°C (30 seg), 60°C (30 seg) e 68°C (3 min), seguido de uma extensão final de 68°C (3 min). A terceira e quarta amplificações foram realizadas usando as mesmas condições para a segunda amplificação, e os produtos clonados no vetor pPIC9K. Os produtos de PCR foram purificados de gel de agarose 1% usando o QIAquick Gel Extraction Kit (Qiagen). Os oligos usados nas amplificações foram sintetizados pelo laboratório de síntese de DNA do departamento de patologia da Universidade de Yale (USA).
Os produtos de PCR purificados foram ligados no vetor pGEM-T easy (Promega) a 4°C por 16 hrs. Células competentes E. coli DH10B foram transformadas por eletroporação (Dower et al. 1988) com as reações de ligação, e os clones foram selecionados no meio LB agar (10% Triptona, 5% extrato de
recombinantes foram selecionados por análise de restrição com as enzimas de restrição Nhe I/Kpn I, Kpn I/EcoR I e SnaB I/EcoR I (New England Biolabs), respectivamente. Os insertos dos plasmídeos foram seqüenciados automaticamente usando o Mega BACE 1000 DNA analysis system (Amersham Life Sciences) de acordo com o método de dideoxinucleotídeos (Sanger et al. 1977), utilizando ddNTPs modificados com uma molécula fluorescente diferente para cada base nitrogenada.
Os insertos dos plasmídeos recombinantes selecionados foram ligados em vetores de expressão, e as células E. coli DH10B transformadas com as novas construções. Os novos transformantes foram selecionadas no meio LB agar ampicilina (100 µg/ml) para construções no vetor pAZ, e LB agar canamicina (50 µg/ml) para construções nos vetores pET36b e pPIC9K. Em seguida, os plasmídeos recombinantes foram extraídos, e os clones foram selecionados por análise de restrição.
3. 5 - Expressão de SmATPDase1 recombinante em E. coli
Durante os ensaios de expressão da enzima recombinante, foram testadas as cepas BL21(DE3) e BL21(DE3)pLysS contendo a construção APIFRA6/pET21b a qual tem clonada a seqüência de DNA que codifica para a região hidrofílica (L65 até L515) da apirase SmATPDase1 (DeMarco et al. 2003). Neste sistema as bactérias
foram cultivadas no meio LB/ ampicilina (100 µg/ml) a 37oC com rotação em um
foi adicionado o IPTG (1 mM) para a indução da expressão da proteína; as bactérias foram coletadas após 6 horas de indução e lisadas com French Press (SIM-AMINCO) a 20 000 psi.
Em seguida foram testadas diferentes temperaturas de incubação da cultura
durante a expressão. Partiu-se de uma cultura de 50 ml com a cepa
BL21(DE3)pLys, a qual foi inoculada em um litro de meio de cultura, que foi incubado nas mesmas condições mencionadas anteriormente. A cultura foi dividida em 4 erlenmeyers (1L capacidade) com 250 ml de cultura cada, e incubado sob indução de IPTG (1mM) a 15oC, 20oC, 30oC e 37oC respectivamente por 6 horas. As células foram coletadas por centrifugação e normalizadas as concentrações de células de cada cultura com 20 mM Tris-HCl pH 7,5 de acordo com as OD600 obtidas
antes da coleta das bactérias. Os lisados celulares foram separados por centrifugação a 10 000 g por 30 min, e amostras das frações solúvel é insolúvel foram analisadas por SDS-PAGE 10%. Posteriormente foi usada a construção APIRH5-II/pET36b para expressar no hospedeiro E. coli C41(DE3), e a expressão de APIRH em fusão com a proteína GFP (N-terminal) foi realizada com células E.
coli Origami(DE3) e BL21(DE3) usando as mesmas condições descritas
anteriormente.
3. 6 - Lavagem e desnaturação de corpos de inclusão
com o processo de re-enovelamento e purificação, as proteínas recombinantes obtidas como corpos de inclusão foram submetidas a uma série de lavagens com diferentes tampões. Para aperfeiçoar este procedimento foram testadas algumas metodologias já descritas (I-VII; tabela 2).
Todas as centrifugações foram feitas a 10 000g por 20 min a 4oC, e em seguida as amostras dos corpos de inclusão foram analisadas em SDS-PAGE 10%. Em seguida, foi selecionado o melhor método de lavagem pela observação do perfil de bandas nos géis, e para a solubilização dos corpos de inclusão foram testados os seguintes desnaturantes: 6M Guanidina e 8M Uréia .
I Ressuspendido em 20 ml de 50 mM Tris-HCl pH 8,0, 2 mM EDTA, 100
µg/ml Lisozima, 0,1% Triton X-100. Incubado por 30 min a 37ºC (Gu et al. 2002), coletado por centrifugação e feitas 3 lavagens com 50 mM Tris-HCl pH 8,0.
II Ressuspendido em 20 ml de 50 mM Tris-HCl pH 8,0, 5 M Uréia, 2% Triton X-100. Incubado com rotação à temperatura ambiente por 30 min (Misawa e Kumagai 1999), coletado por centrifugação e feitas 3 lavagens com 50 mM Tris-HCl pH 8,0.
III Ressuspendido em 20 ml de 50 mM Tris-HCl pH 8,0, 100 µg/ml Lisozima; incubado por 30 min à temperatura ambiente, em seguida adicionados DNAse e MgCl2 com concentrações finais de 10 µg/ml e 3
mM respectivamente, incubado 30 min à temperatura ambiente. Em seguida foi adicionado Triton X-100, EDTA e NaCl para concentrações finais de 6%, 20 mM e 1,5 M respectivamente, e incubado por 30 min com rotação à temperatura ambiente (Richter et al. 2002), em seguida coletado por centrifugação e feitas 3 lavagens com 50 mM Tris-HCl pH 8,0.
IV Ressuspendido em 20 ml de 50 mM Tris-HCl pH 8,0, 2 M Uréia, incubado por 30 min com rotação à temperatura ambiente (Middelberg 2002). Em seguida coletado por centrifugação e feitas 3 lavagens com 50 mM Tris-HCl pH 8,0.
V Ressuspendido em 20 ml de 20 mM Tris-HCl pH 8,0, 2 M Uréia, 0,5 M NaCl. Incubado por 30 min com rotação à temperatura ambiente (Lemercier et al. 2003). Em seguida coletado por centrifugação e feitas 3 lavagens com 50 mM Tris-HCl pH 8,0.
VI Ressuspendido em 20 ml de 50 mM Tris-HCl pH 8,0 e feitas mais duas lavagens com o mesmo tampão.
VII Ressuspendido em 20 ml de 20 mM Tris-HCl pH 7,5 e feitas mais duas lavagens com o mesmo tampão.
3.7 – Re-enovelamento de Proteínas desnaturadas
Com a finalidade de obter a proteína recombinante APIRH desnaturada em forma solúvel, foram testados alguns métodos de re-enovelamento das proteínas, descritos a seguir:
3.7.1 - Re-enovelamento por diálise de proteínas desnaturadas em Guanidina
Proteína desnaturada em 6M Guanidina foi dialisada contra 50 volumes da solução 100 mM Tris-HCl pH 8,0, 0,5 M NaCl, 10% glicerol à temperatura ambiente (Sorensen et al. 2003).
3.7.2 – Re-enovelamento por diluição de proteínas desnaturadas com Guanidina
Proteína solubilizada (~23 mg/ml) em 2,5 ml de 6M Guanidina foi diluída em
500 ml de 100 mM Tris-HCl pH 8,5, 20% glicerol, Chaps 20µM. Foi adicionado ao
tampão de re-enovelamento CuSO4 (1µM) e mantido nas mesmas condições de
incubação como descrito anteriormente.
3.7.3 – Re-enovelamento ligado a uma matriz
Proteínas solubilizadas em 8M uréia, 20 mM Tris-HCl, pH 8,5 foram
3. 8 - Amplificação e clonagem da SmATPDase2
O mRNA de parasitas adultos foi obtido de vermes conservados em RNALater (Ambion) por extração do tecido com MAC mRNA isolation kit (Miltenyi Biotec). O cDNA completo foi obtido com o método RACE (“rapid amplification of cDNA ends”), usando 200 ng de mRNA e os kits 3´-RACE ou 5´-RACE (Gibco), com oligos específicos para o gene ATPDase2 de S. mansoni. Os oligos foram desenhados baseados na seqüência de um cluster derivado de ESTs obtidos no projeto de seqüenciamento em larga escala de transcritos de S. mansoni (Verjovski-Almeida et al. 2003) (SmAE608642.1, accessível em http://bioinfo.iq.usp.br/schisto/); as mini-bibliotecas de EST foram obtidas com oligos não-degenerados aleatórios e o método RT-PCR de baixa estringência (ORESTES) (Dias Neto et al. 1997). O passo de PCR nos experimentos RACE foi realizado com a Advantage II polymerase (Clontech), com o tampão fornecido pelo fabricante, 200 µM dNTPs e 200 nM de cada oligo usando a seguinte ciclagem: 95 oC (5min); 30 ciclos de 95 oC (30 seg), 60 oC (30 seg), e 68 oC (3 min); e extensão final de 68 oC (3 min).
3. 9 - Clonagem e expressão da tioredoxin (Trx)
A seqüência que codifica para a tioredoxina foi reamplificada por PCR a partir do vetor pET32a (Novagen) usando os oligos 5´-GAATTCTAATACGACTC ACTATAG-3´ (“forward”) e 5´-AAGCTTGGCCAGGTTAGCGTCG-3´ (“reverse”), clonada no vetor pGEM-T e seqüenciada. O fragmento de DNA Trx foi subclonado no vetor de expressão pET36b (resistência a canamicina) pelos sítios Eco RI e Hind
III. Após varredura de clones E. coli DH10B contendo o vetor de expressão recombinante, foi selecionado o clone contendo a construção Trx(1)/pET36b. Células competentes ArcticExpress(DE3) RIL (co-expressa as chaperonas Cpn10 e
Cpn60 da bactéria Oleispira antarctica) foram co-transformadas com as construções Trx(1)/pET36b e ATPDase2/pET21b por eletroporação e selecionadas no meio LB agar contendo ampicilina (100 µg/ml), canamicina (50 µg/ml) e gentamicina (20 µg/ml). Foi feito um pre-inóculo (5 ml) com os transformantes selecionados e adicionado a 1L de meio de LB contendo 1% glicose, com rotação de 210 rpm e incubado a 30ºC até atingir uma OD600 de 2.
3. 10 - Clonagem e expressão de fragmentos recombinantes da proteína
SmATPDase contendo baixa identidade entre SmATPDase1 e 2.
ATPDase2-sol/pET21b) permitiram a expressão dos fragmentos recombinantes SmATPDase1 e SmATPDase2 em fusão com uma cauda de histidinas no extremo carboxilo terminal.
3. 11 - Purificação de proteínas recombinantes em E. coli
3. 11. 1 - Cromatografia de afinidade
Fração solúvel de lisado bacteriano foi passada por uma coluna de Ni-NTA (1,5 x 8 cm) equilibrada com 20 mM Tris-HCl pH 8,5, 500 mM NaCl, 10% glicerol e lavada com dez volumes do tampão de equilíbrio contendo 20 mM de imidazol. Proteínas ligadas foram eluidas com o tampão contendo 500 mM de imidazol. As proteínas re-enoveladas na coluna de afinidade foram purificadas sob as mesmas condições.
3. 11. 2 - Cromatografia de troca iônica
As frações com as proteínas parcialmente purificadas foram dialisadas contra o tampão 20 mM Tris-HCl pH 9,0, 20 mM NaCl, 10% glicerol e 1 mM DTT, e passadas por uma coluna Q-sepharose high performance (Amersham Biosciences) equilibrada com o mesmo tampão usando uma bomba peristáltica Pump P-1. As proteínas foram eluídas com uma crescente concentração de NaCl até 1 M, usando um aparelho FPLC (Pharmacia Biotech).
3. 12 - Expressão de proteínas recombinantes em Pichia pastoris
selecionados SmATPDase1-(20)/pPIC9K, SmATPDase1-(16)/pPIC9K e SmATPDase2-(9)/pPIC9K foram linearizados com a enzima Sac I, e os produtos digeridos foram usados para transformar a levedura P. pastoris GS115 por eletroporação (Cregg et al. 1985) separadamente, usando os seguintes parâmetros: 25 F, 400 Ω, 1,5 kV. Os transformantes resultantes His+ selecionados no meio MD agar (1,34 % YNB, 4x10-5 % biotina, 1 % dextrose, 1,5 % agar) passaram por uma segunda triagem no meio YPD agar (1 % extrato de levedura, 2 % peptona, 2 % sucrose e 1,5 % agar) com 4 mg/ml de G418 sulfato (Invitrogen) e incubadas por 72
– 96 hrs a 30°C (Scorer et al. 1994). Os transformantes resistentes a esta alta concentração do antibiótico sugerem a presença de elevado número de cópias da construção inseridas no genoma da levedura.
3. 13 - Purificação de SmATPDase1 e 2 do sobrenadante da cultura de P.
pastoris
Após 5 dias de cultura, o meio foi coletado por centrifugação (4 000 g por 20 min) e o sobrenadante foi concentrado 30- 40 vezes por ultrafiltração, e depois dialisado contra 100 vezes o tampão de equilíbrio (20 mM Tris-HCl pH 8.5, 10 % glicerol, 500 mM NaCl) por 16 hrs a 4ºC, e depois foi carregada numa coluna de Ni-NTA (1,5 x 5 cm). A coluna foi lavada com o tampão de equilíbrio incluindo 10 mM imidazol e as proteínas foram eluídas com o mesmo tampão contendo 500 mM de imidazol. A proteína eluída foi analisada por SDS-PAGE 10%. As frações contendo apirase recombinante foram juntadas e concentradas por ultrafiltração usando filtros Amicon – ultra /10 000MWCO (Millipore). A concentração de proteína foi estimada pelo método de Bradford.
3. 14 - Clonagem e expressão da proteína dissulfeto isomerase (PDI)
O mRNA de uma cultura de Saccharomyces cerevisiae foi extraído usando o kit MAC RNA isolation (Miltenyi Biotec). O cDNA foi obtido por transcrição reversa usando oligo(dt)12-18 a partir de 100 ng de mRNA. A amplificação por PCR da
pICHOLI-1 (Mobitech) através dos sitios Not I e EcoR I; a expressão está sob controle do promotor do gene AOX1 que é induzido pelo metanol. O vetor resultante foi introduzido em E. coli DH10B por eletroporação e feita análise de restrição dos plasmídeos extraídos dos clones obtidos, para a seleção daqueles clones que continham o vetor de expressão recombinante.
Os clones SmATPDase1(16)/pPIC9K e SmATPDase2(9)/pPIC9K foram selecionadas pela resistência ao antibiótico G418 sulfato (4mg/ml) para a expressão das apirases recombinantes em fusão com uma cauda de histidina na região carboxilo terminal; células competentes foram transformadas por eletroporação com o vetor de expressão recombinante PDI/pICHOLI-1. Os novos transformantes foram selecionados no meio enriquecido YPD agar (1% extrato de levedura, 2% peptona, 2% glicose 1,5% agar) contendo G418 sulfato (4mg/ml) e Zeocina (100 µg/ml) e as placas incubadas a 30ºC por 4 dias. Foram selecionados aleatoreamente alguns clones para testes de expressão em pequena escala no meio mínimo BMG como descrito anteriormente. Os sobrenadantes das mini-culturas foram concentrados por ultrafiltração e as proteínas resolvidas em SDS-PAGE 10%.
3. 15 - Caracterização enzimática de SmATPDases 1 e 2 recombinantes de S.
mansoni.
Atividade apirásica foi determinada como descrito previamente (Schadeck et al. 1989); 134 µl de amostra contendo 0,2 µg de enzima foram incubados em tubos eppendorf 1,5ml com 166 µl de tampão de atividade 3x (150mM Tris-HCl pH 7.5, 12mM KCl, 15mM glicose, 15mM CaCl2,) e 200 ul de 7.5mM de nucleotídeo (NTP ou
NDP) a 37ºC por 20 min. O fosfato inorgânico liberado (Pi) foi quantificado
uma unidade de atividade enzimática foi definida como a quantidade de enzima necessária para liberar 1 µmol de Pi/min a 37ºC. A atividade da enzima purificada foi
ensaiada testando diferentes substratos: ATP, CTP, GTP, UTP, ADP, CDP, GDP, UDP e IDP; além disso foram avaliadas as atividades enzimáticas na presença dos cátions divalentes CaCl2, CoCl2, MgCl2, MnCl2, HgCl2, ZnCl2 e EDTA.
3. 16 - Dicroísmo circular
As proteínas recombinantes purificadas SmATPDase1 e 2 em concentrações de 0,33 mg/ml e 0,56 mg/ml respectivamente foram dialisadas “overnight” a 5 ºC, no tampão 10 mM NaH2PO4. Para cada enzima foram preparadas 3 amostras, uma em
tampão e as outras 2 em tampão com 5 e 10mM de MgSO4 respectivamente. Os
espectros de CD foram obtidos como a média de 5 leituras feitas pelo espectropolarímetro JASCO-810 (Instituto Butantã) a 20ºC entre os comprimentos de onda 190-260 nm, usando uma cubeta de 0,1cm. Os dados foram expressos como elipticidade média residual. Os valores de elipticidade molar residual [θ] (deg x cm2 x dmol-1) foram calculados utilizando a seguinte fórmula:
[elipticidade θ (mdeg) x massa molecular da proteína (Da)]
[10 x caminho da cubeta (cm) x concentração da prot. (mg/ml) x (nº de aminoácidos – 1)]
3. 17 - Western blot
Tris-HCl, pH 7,4, 8 % sacarose, 0,5 µg/ml leupeptin, 0.1 µg/ml pepstatin, 0,05 µg/ml inibidor de tripsina, e 8,7 µg/ml PMSF. Estas amostras foram estocadas em nitrogênio liquido até posterior uso. Amostras de tegumento foram previamente esquentadas antes do seu uso e solubilizadas com a solução contendo 40 mM Tris pH 7,4, 7 M ureia, 200 mM β-ME, 2 % Chaps e 1 % SDS, logo as frações solúvel e insolúvel foram separados por ultra-centrifugação a 100,000 g. Amostras foram resolvidas em SDS-PAGE 12 % e transferidas a uma membrana de nitrocelulose. A membrana foi bloqueada 12-16 hrs com a solução blotto (PBS, 5 % leite em pó desnatado, pH 7.4) e incubado separadamente por 4h com o soro anti-SmATPDase1 ou anti-SmATPDase2 (1:500 v:v), seguido de quatro lavagens de 5 min cada com PBS-Tween 20 e incubado posteriormente com o anticorpo secundário anti-mouse IgG ou anti-rabbit IgG (1: 2000 v:v) , HPR-linked (Amersham Biosciences) em solução blotto por 2 h. A IgG ligada foi detectada por quimioluminescência usando o kit ECL Western Blotting Detection reagents (Amersham Biosciences).
3. 18 - Deglicosilação de apirase recombinante
3. 19 - Imunização de camundongos e desafio com cercárias.
3. 19. 1 - Imunização com Proteínas
Inicialmente, para avaliar a resposta imunogênica da SmATPDase1, a proteína purificada foi obtida através de eletroforese seguida de eletroeluição de acordo com as recomendações do fornecedor Hoefer GE 200 Gel Eluter (Amersham Pharmacia Biotech). Posteriormente, proteínas purificadas das duas isoformas foram obtidas por re-enovelamento (E. coli) ou do sobrenadante da cultura de P. pastoris (solúvel e ativa). Foram testados neste ensaio os adjuvantes hidróxido de alumínio e de Freund.
Protocolo:
Camundongos fêmeas Balb/c de 4-6 semanas receberam 3 doses de 10µg de
proteína s.c (dorso) de 7 em 7 dias. Foi realizado o desafio com 110 cercárias (s.c. abdomen). A perfusão foi realizada após 7 semanas. As proteínas testadas foram apirase recombinante desnaturada obtida por eletroeluição, apirase recombinante re-enovelada por diluição e o controle positivo com Sm14 recombinante.
Camundongos fêmeas C57BL/6 de 4-6 semanas receberam 3 doses de 30
CpG, 3 reforços de CpG), grupo 3 (prime com alumínio, 3 reforços de CpG), grupo 4 (prime com CpG, 3 reforços com alumínio), grupos 5 e 6 são os controles negativos para Alumínio e CpG, respectivamente.
Camundongos fêmeas C57Bl/6 de 5-7 semanas de idade (10 animais por
grupo) receberam 3 doses. Um prime de 25 µg de proteína em PBS (1x) formulada no mesmo volume de adjuvante (CFA) por via subcutânea, e dois reforços com intervalo de 15 dias com 25 µg de proteína em PBS (1x), formulados no mesmo volume de adjuvante (IFA) (Sigma), via subcutânea. Os grupos testados foram SmATPDAse1 (grupo 1), SmATPDase2 (grupo 2) e SmATPDase1 + SmATPDase2 (grupo 3), grupo controle. Foi realizado o desafio com 110 cercárias (s.c. abdomen), e o sacrifício dos camundongos, seguido da perfusão do fígado para coleta de parasitas, foi realizado após 7 semanas.
3. 19. 2 - Imunização com plasmídeo (vacina de DNA)
Protocolo: Camundongos fêmeas Balb/c ou C57B/6 de 4-6 semanas,
receberam 50 µl em cada músculo quadríceps de uma solução 10 µM de
cardiotoxina (Latoxan). Decorrido 5 dias da injeção de cardiotoxina, o DNA
plasmidial (50 µg) foi injetado intramuscularmente com uma seringa de 1 ml e agulha Dia 70
Desafio 110 cercárias Sangria
Pré-imune
Dia 1 1° dose
Dia 14 3° dose Dia 7
2° dose
Dia 119 Perfusão Dia 6
Sangria 1 Dia 13 Sangria 2
com calibre de 29G. O volume total foi de 100 µl, com 50 µl injetado em cada músculo quadríceps. Os animais receberam um reforço 21 dias após a primeira dose.
3. 20 - ELISA
Microplacas de fundo chato foram revestidas com 0,5 µg/poço das proteínas recombinantes em tampão carbonato - bicarbonato 0,05 M (pH 9,6), durante 16 horas a 4°C. As placas foram lavadas em seguida 3 vezes com tampão fosfato salina (PBS) contendo 0,05% de Tween 20, e bloqueadas com 200 µl de uma solução 10% leite desnatado em PBS, por pelo menos 1 hora a 37°C. Depois de lavadas com PBS Tween 20 0,05%, 100 µl de uma diluição seriada do soro dos animais imunizados ou controles foram adicionados em cada poço, sendo a diluição inicial 1:20. As placas foram incubadas a 37°C durante 2 horas e depois lavadas três vezes com PBS Tween 20 0,05%. A seguir, 100 µl do anticorpo secundário conjugado a peroxidase foram adicionados em uma diluição 1:15 000 e incubados por 1 hora a 37°C. As placas foram então lavadas três vezes com PBS Tween 20
0,05%, e 100 µl do substrato OPD foi adicionado em cada poço. As placas foram lidas a 492 nm em um leitor de Elisa Labsystem.
Dia 50 Desafio 130 cercárias Sangria
Pré-imune
Dia 1 1° dose
Dia 21 2° dose
Dia 99 Perfusão Dia 20
Sangria 1
3. 21 - PCR em Tempo Real (Real Time PCR)
Os mRNAs das formas ovo, miracídio, cercária, esquistossômulo e verme adulto do parasita foram obtidas por extração de tecidos com MAC mRNA isolation kit (Miltenyi Biotec) usando amostras conservadas em RNALater (Ambion). Tres microgramas de mRNA foram tratadas com RQ1 RNAse-free DNAse (Promega), usando 1U em 10 µl de reação por 30 min a 37°C. Posteriormente, a DNase foi inativada a 65°C por 10min. Os produtos resultantes foram submetidos à transcrição reversa com “random hexamer primer” usando o protocolo do Superscript first strand system (Invitrogen) para RT-PCR. Foram usados como controle reações sem adição de transcriptase reversa as quais foram corridas em paralelo, e posteriormente usadas como molde para o controle negativo do PCR. Oligos para o Real-Time PCR foram desenhados usando a ferramenta Primer express program version 2.0.0 (Applied Biosystems). As reações do Real-Time PCR foram realizadas usando o reagente SYBR Green PCR master mix (Applied Biosystems) e os oligos específicos para SmATPDase2 ACAAAATCCTGTTGAAGGTGATGTAA-3´ (“Forward”),
5´-TTGTTGGGTGGATATCTGATAACATT-3´ (“Reverse”) e SmATPDase1
5´-GGAAAACAATTACATTTGATTCGAAA-3´ (“Forward”), 5´-TGCCCAGCTAACTGA TTTACCA-3´ (“Reverse”) usando o GeneAmp 5700 Sequence Detection System (Applied Biosystems). Os valores de significância (P-value) foram calculados usando o teste-t de Student. Todas as amostras foram usadas em triplicata e o gene alfa tubulina foi usado como um controle interno.
3. 22 - Imunofluorescência de parasitas inteiros e cortes histológicos
nas lâminas de vidro previamente tratadas poli-L-lisina e fixadas com acetona por 30 min a -20 °C. Após secagem, as secções foram bloqueadas por incubação em PBS contendo 1 % BSA e 0.1 % Tween 20 (solução de permeabilização) “overnight” a temperatura ambiente. Secções foram logo incubadas com os antisoros anti-SmATPDase1 e 2 (1:100 v:v) “overnight” a 5 °C. Após três lavagens com o tampão PBS, o anticorpo secundário anti-rabbit IgG conjugado com FITC (verde) e o anticorpo anti-mouse IgG conjugado com Cy3 (vermelho) (1:50 v:v) foram adicionados à solução de permeabilização com amostras por 1 h a temperatura ambiente. Secções foram lavadas, e montadas na solução glicerol 90 %, 50 mM Tris-HCl pH 9,0. Imagens foram obtidas usando o sistema confocal BioRad 1024UV conectado a um microscópio Zeiss Axiovert 100 equipado com uma lente objetiva de imersão 40x N.A 1.2 plan-apochromatic com contraste de interferência diferencial. O programa LaserSharp 1024 version 3.2T da Bio-Rad foi usado para a captação de imagens e o Adobe Photoshop 4.0 para o processamento destas. Os antisoros pre-immunes de coelho e camundongo foram usados como controle negativo. Formas inteiras de miracídio, cercária e esquistossômulo foram fixadas em metanol gelado por 10 min e incubadas “overnight” na solução de permeabilização PBS, 1 % BSA, 0,1% Tween 20; os passos seguintes foram realizados usando o mesmo procedimento descrito para cortes histológicos de vermes adultos.
3. 23 - Cultura in vitro de vermes adultos de S. mansoni
de cultura com o antibiótico antimicótico (Invitrogen) contendo 100 units/ml de Penicilina G, 100 µg/ml de Streptomicin sulfato, 0,25 µg/ml de anfotericin B e incubado por 48 hrs a 37ºC sob 5% CO2. Meio de cultura com antibióticos e sem
vermes foi usado em paralelo como controle negativo (30 ml). Experimentos foram realizados em duplicata (60ml) contendo um total de 1790 vermes. Após incubação de 48 hrs, o meio de cultura foi separado dos vermes por filtração usando papel filtro (Amersham Bioscience).
Os meios de cultura, das culturas de vermes adultos ou do controle, foram concentrados aproximadamente 5 vezes usando Amicon ultra filters 10000 MWCO (Millipore) e dialisados “overnight” contra 100 volumes do tampão de equilíbrio (50 mM Tris-HCl pH 7.5, 10% glicerol, 5 mM MgCl2, 20 mM NaCl) em baixa temperatura
3. 24 - Ensaio de inibição de penetração pela pele
O ensaio de inibição de penetração foi adaptado de uma metodologia já
descrita (Williamson et al. 2003). Resumidamente, 100-130 cercárias (em 100 µl)
foram incubadas com 50 µl de soro de camundongos imunizados com a proteína
apirase, ou 50 µl de soro de animais não imunizados. Após 1 h de incubação a
37°C, as cercárias juntamente com o anti-soro foram aplicadas em 850 µl de água corrente desclorada, para penetração pelo método do anel em camundongos anestesiados que tiveram seus abdomens previamente depilados. As cercárias foram deixadas penetrar durante 30 min à temperatura ambiente; após este período as larvas que não penetraram foram removidas da superfície dos animais e contadas. Para determinar se houve diferenças entre os grupos realizamos o teste-t de Student.
3. 25 - Cross-linking com glutaraldeido
As proteínas, 5 µM cada, foram incubadas à temperatura ambiente em 20 mM triethanolamine, pH 8,0, 100 mM NaCl e 0,2% glutaraldeido. Após 1 hora de incubação, a reação foi parada pela adição de Tris-HCl pH 7,5, na concentração final de 100 mM. As amostras foram resolvidas por SDS-PAGE 10%.