Diana Rosa Azevedo Marques
julho de 2015
Prevalência e susceptibilidade de isolados
clínicos de Pseudomonas aeruginosa numa
unidade hospitalar de Portugal
UMinho|20
15
Diana Rosa Aze
vedo Mar
ques
Pre
valência e suscep
tibilidade de isolados clínicos de
Pseudomonas aeruginosa
numa unidade hospit
alar de P
or
tugal
Universidade do Minho
Escola de Ciências
Diana Rosa Azevedo Marques
julho de 2015
Dissertação de Mestrado
Mestrado em Bioquímica Aplicada
Área de Especialização em Biomedicina
Prevalência e susceptibilidade de isolados
clínicos de Pseudomonas aeruginosa numa
unidade hospitalar de Portugal
Universidade do Minho
Escola de Ciências
Trabalho efectuado sob a orientação da
Professora Doutora Olga Cardoso
e co-orientação da
D
ECLARAÇÃO
Nome: Diana Rosa Azevedo Marques
Endereço electrónico: diana.r.marques@gmail.com Número do Cartão de Cidadão: 13599941
Título da dissertação: Prevalência e susceptibilidade de isolados clínicos de
Pseudomonas aeruginosa numa unidade hospitalar de Portugal
Orientadora: Professora Doutora Olga Cardoso Co-orientadora: Professora Doutora Paula Sampaio Ano de conclusão: 2015
Designação do mestrado: Bioquímica Aplicada, área de especialização em Biomedicina
É AUTORIZADA A REPRODUÇÃO INTEGRAL DESTA DISSERTAÇÃO APENAS PARA EFEITOS DE INVESTIGAÇÃO, MEDIANTE DECLARAÇÃO ESCRITA DO INTERESSADO, QUE A TAL SE COMPROMETE.
Universidade do Minho, 08 de Julho de 2015
iii
A
GRADECIMENTOS
A realização deste trabalho teve o apoio de várias pessoas, sem as quais nunca o teria levado a bom porto. Deixo-lhes o meu mais sincero agradecimento.
À minha orientadora, Doutora Olga Cardoso, que me possibilitou toda a logística para a realização deste projecto, que acompanhou a evolução do trabalho e a minha, e a quem devo muito do que aprendi ao longo deste tempo.
À minha co-orientadora, Doutora Paula Sampaio, que mesmo à distância se mostrou sempre disponível para me guiar e ajudar no passo seguinte.
Aos Hospitais da Universidade de Coimbra, na pessoa da Doutora Cristiana Canha, que me forneceu a “matéria-prima” para este projecto.
À Doutora Sónia Gonçalves Pereira, pelas aulas práticas, mas principalmente pelas conversas e novas perspectivas, às vezes à companhia de um café. Ao Diogo, por participar nas conversas e pagar o café.
À Sandra e à D. Celeste, pelo profissionalismo e disponibilidade, assim como pelos momentos de descontracção e pela boa disposição constante e contagiante.
À D. Conceição e à Joana, que me receberam em sua casa como se fosse mais um membro da família.
Ao Zé, que fez com que Coimbra se tornasse mais cheia e mais minha.
Aos membros do “Friday Procrastination” e do “Festão@Algures”, pelas gargalhadas, companheirismo e porto seguro.
E porque os últimos são os primeiros, obrigada aos meus pais, pelas pequenas e pelas grandes coisas, pelo que eu sei e pelo que eu nem sonho…
v
“Prevalência e susceptibilidade de isolados clínicos de Pseudomonas aeruginosa numa unidade hospitalar de Portugal”
R
ESUMO
A resistência bacteriana a antibióticos é hoje um grave problema de Saúde Pública de dimensão global, responsável por um aumento das taxas de morbilidade e mortalidade, por um acréscimo dos custos financeiros associados aos cuidados de saúde, e por um agravamento do risco inerente a uma panóplia de procedimentos médicos. De forma a combater estas tendências, a vigilância assume um papel fundamental, assim como um melhor entendimento dos mecanismos moleculares desta resistência e inclusive dos estímulos que levam à sua evolução e aos meios de transmissão.
O estudo de prevalência e susceptiblidade de isolados clínicos de Pseudomonas
aeruginosa, um dos principais patógenos humanos responsáveis por infecções
oportunistas em indivíduos imunocomprometidos, colhidos durante dois meses numa unidade hospitalar central portuguesa pretende ajudar a compreender a realidade deste problema.
A resistência de P. aeruginosa neste hospital é elevada, mostrando-se superior aos valores relativos a Portugal anunciados pelo ECDC em 2013, e sem indícios de melhorias significativas nos últimos anos. Testes fenotípicos indiciaram a produção de metalo-β-lactamases como o mecanismo de resistência aos antibióticos β-lactâmicos testados mais prevalente.
Com o aumento da resistência associada a esta espécie, e a sua comprovada tendência para evoluir para multirresistência, impõe-se atenção redobrada de forma a evitar um agravamento incomportável da situação. Nesse sentido, é recomendada a implementação ou aperfeiçoamento de medidas de prevenção e controlo de infecções e de resistência a antibióticos.
vii
“Prevalence and susceptibility of Pseudomonas aeruginosa clinical isolates in a hospital unit at Portugal”
A
BSTRACT
Antibiotic resistance is currently a major Public Health concern of global dimension, accountable for increasing morbidity and mortality rates, rising healthcare financial costs, and intensifying the risk associated with a variety of medical procedures. In order to fight these trends, surveillance is of the essence, as well as a better understanding of the underlying molecular mechanisms, stimuli of its evolution, and means of transmission.
The analysis of the prevalence and susceptibility of clinical isolates of Pseudomonas
aeruginosa, one of the main human pathogens responsible for opportunistic infections
of immunocompromised individuals, and one of the main bacterial species associated with nosocomial infections, collected over a period of two months in a Portuguese central hospital unit aims at helping to understand the reality of this issue.
P. aeruginosa resistance rates in this hospital are high, registering superior values than
those reported by ECDC when describing the Portuguese situation in 2013, and with no apparent improvements over the recent years. Phenotypic testing suggests that production of metallo-lactamases is the main resistance mechanism to the tested β-lactams.
Considering the increasing resistance rates associated with this species, and its established predisposition to develop multiple resistance, a renewed commitment is vital in order to avoid an unbearable aggravation of the circumstances. To achieve such goal, the implementation or adjustment of infections and antibiotic resistance prevention and control measures is highly recommended.
ix
Í
NDICE
Agradecimentos ... iii
Resumo ... v
Abstract ... vii
Lista de siglas e abreviaturas ... xi
Lista de figuras ... xiii
Lista de tabelas ... xv
1. Introdução ... 1
1.1. Pseudomonas aeruginosa ... 1
1.2. Resistência a antimicrobianos: situação global ... 4
1.2.1. Factores impulsionadores e vias de disseminação de resistência ... 7
1.2.2. Implicações sociais ... 11
1.2.3. Acções de controlo e prevenção de resistência ... 14
1.3. Mecanismos de resistência bacteriana ... 17
1.3.1. Resistência mediada por β-lactamases ... 19
1.3.2. Mecanismos de resistência em Pseudomonas aeruginosa ... 24
1.4. Objectivos do projecto ... 27
2. Materiais e métodos ... 28
2.1. Recolha de isolados de P. aeruginosa ... 28
2.2. Identificação dos isolados ... 28
2.3. Antibiogramas ... 29
3. Resultados ... 32
3.1. Epidemiologia ... 32
3.3. Perfil de susceptibilidade ... 34
3.4. Mecanismos de resistência a antibióticos ... 41
3.4.1. Pesquisa fenotípica de ESBLs ... 41
3.4.2. Pesquisa fenotípica de MBLs ... 41
3.4.3. Outros mecanismos de resistência ... 41
4. Discussão ... 43
4.1. Epidemiologia ... 43
4.2. Tendências dos perfis de susceptibilidade a antibióticos ... 44
4.2.1. Panorama português ... 44
x 4.3. Mecanismos de resistência... 50 4.4. Testes de susceptiblidade ... 52 5. Observações finais ... 54 Referências bibliográficas ... 56 Anexos ... 65
A redacção deste documento seguiu as normas de ortografia prévias à entrada em vigor do Acordo Ortográfico da Língua Portuguesa de 1990.
xi
L
ISTA DE SIGLAS E ABREVIATURAS
AK amicacina ATM aztreonamo
AUG amoxicilina e ácido clavulânico
CAZ ceftazidima
CDC Centro de Controlo e Prevenção de Doenças CIP ciprofloxacina
CLSI Clinical and Laboratory Standards Institute
CMI concentração mínima inibitória
DGS Direcção Geral de Saúde DNA ácido desoxirribonucleico EARS-NET Rede Europeia de Vigilância
de Resistência a Antimicrobianos
ECDC Centro Europeu de Controlo e Prevenção de Doenças EDTA ácido etilenodiamino
tetra-acético
EFSA Autoridade Europeia de Segurança Alimentar EMEA Agência Europeia de
Medicamentos
ESAC-NET Rede Europeia de Vigilância do Consumo de
Antimicrobianos ESBLs β-lactamases de largo
espectro
ETARs Estações de Tratamento de Águas Residuais
EUA Estados Unidos da América EUCAST Comité Europeu para o
Teste da Susceptibilidade Antimicrobiana
FDA Food and Drug
Administration
FEP cefepima
HUC Hospitais da Universidade de Coimbra I&D Investigação e Desenvolvimento IMI imipenemo MBLs metalo-β-lactamases MDR multirresistentes MRP meropenemo
MRSA Staphylococcus aureus
resistente a meticiclina
PBP penicillin-bindig proteins
PCR Polymerase chain reaction
PGA Programas de Gestão de Antibióticos
PRL piperacilina
QS quorum sensing
RNA ácido ribonucleico SDS dodecil sulfato de sódio UE União Europeia
WHO Organização Mundial de Saúde
xiii
L
ISTA DE FIGURAS
Figura 1: Representação temporal da introdução e detecção de resisitência a antimicrobianos no tratamento de infecções. Em cima, ano de introdução de novas classes de antimicrobianos e em baixo, ano de detecção de resistência às mesmas (Lewis 2013) ... 6 Figura 2: Principais fontes de pressão selectiva (vermelho), reservatórios ambientais (laranja) e vias de disseminação de antibióticos e resistência bacteriana (azul). Adaptado de Davies e Davies (2010). ... 8 Figura 3: Mecanismos de resistência a antibióticos β-lactâmicos mais comuns em bactérias de Gram positivo (à direita) e bactérias de Gram negativo (à esquerda) (adaptado de Tang et al. 2014) ... 18 Figura 4: Disseminação global de algumas famílias de Metalo-β-lactamases (Cornaglia
et al. 2011) ... 20
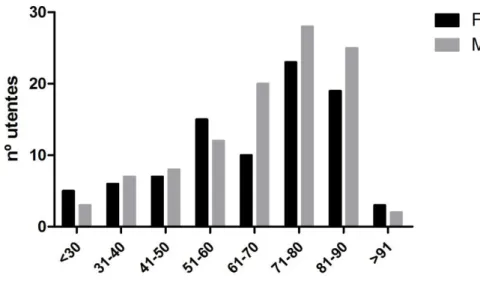
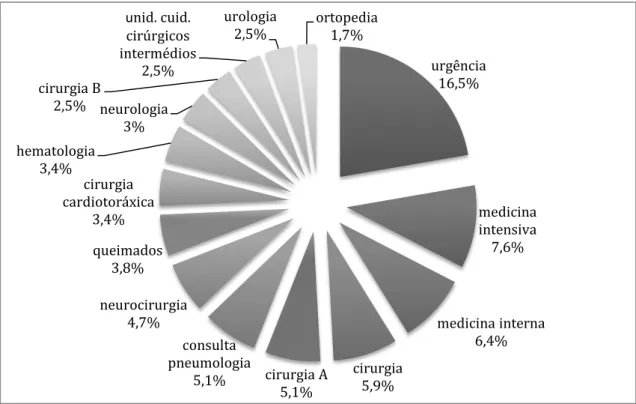
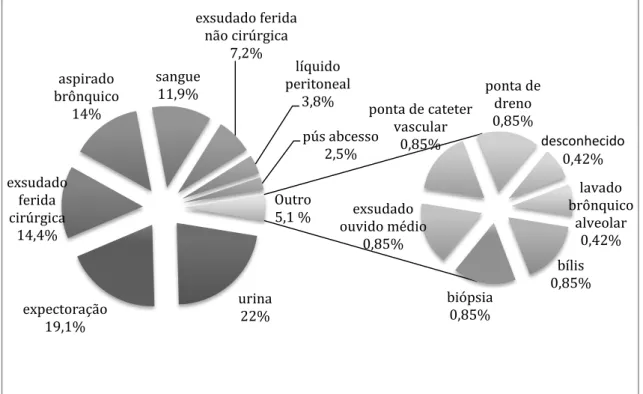
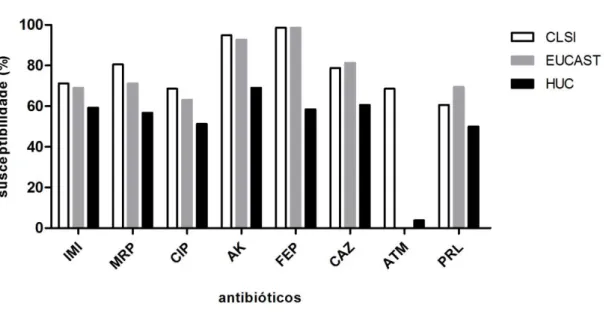
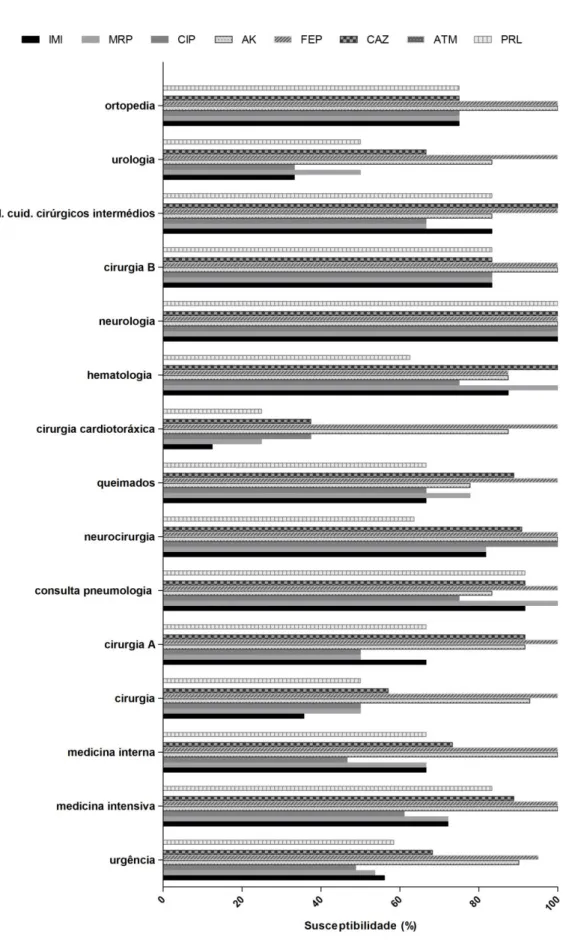
Figura 5: Distribuição dos utentes dos quais foram recolhidos os isolados por faixa etária e género (F – feminino; M – masculino). ... 32 Figura 6: Distribuição de 74,1% dos isolados pelos 15 serviços hospitalares mais contribuidores. ... 33 Figura 7: Distribuição dos isolados por produto biológico. ... 34 Figura 8: Susceptibilidade (%) dos isolados aos antibióticos utilizados. Comparação de resultados obtidos tendo em atenção os limites definidos pelos CLSI e EUCAST, e os obtidos pelo VITEK II. ... 35 Figura 9: Susceptibilidade (%) a todos os antibióticos testados, nos 15 serviços de origem de 74,1% dos isolados. ... 37 Figura 10: Isolados multirresistentes (%) distribuídos por serviço hospitalar (A) e produto biológico (B). ... 38 Figura 11: Susceptiblidade (%) aos antibióticos testados, por origem biológica... 39 Figura 12: Percentagem de isolados de P. aeruginosa resistentes a carbapenemos nos países europeus em 2013. Adaptado do Relatório Anual da EARS-NET de 2014. ... 47
xv
L
ISTA DE TABELAS
Tabela 1: Cronograma do projecto. ... 27 Tabela 2: Antibióticos utilizados nos antibiogramas de P. aeruginosa. Indicação dos limites (mm) definidos pelo CLSI e pelo EUCAST. ... 30 Tabela 3: Resistência (%) registada pelo ECDC para Portugal e média ponderada europeia no ano 2013 de P. aeruginosa em relação a carbapenemos, fluoroquinolonas, aminoglicosídeos, ceftazidima e piperacilina-tazobactam, e percentagem de multirresistência (ECDC 2014). Comparação com os valores equivalentes calculados para os HUC no presente estudo. ... 44
1
1.
I
NTRODUÇÃO
1.1.
P
SEUDOMONAS AERUGINOSAPseudomonas aeruginosa é um bacilo de Gram negativo mono-flagelado, pertencente
à família Pseudomonadaceae, incluída na classe Gammaproteobacteria. É um organismo quimio-heterotrófico, não-fermentativo e aeróbio facultativo, que na ausência de O2 usa NO3- como aceitador final de electrões. Tem necessidades
nutricionais básicas – tendo a capacidade de sobreviver em água destilada (Favero et
al. 1971) – e pode utilizar um vasto conjunto de compostos orgânicos como fonte de
carbono e energia. Possui elevada tolerância a vários factores de stress, tanto físicos como químicos; como exemplo, é capaz de se multiplicar a temperaturas entre os 4 e os 50 °C (Termine & Michel 2009; Pier & Ramphal 2010) e possui elevados níveis de tolerância a compostos de amónio quaternário (Langsrud et al. 2003).
A versatilidade metabólica deste organismo tem origem no seu genoma, um dos maiores do mundo bacteriano (aproximadamente 6 Mbp) (Stover et al. 2000), com uma incrível plasticidade genética (Mathee et al. 2008), e uma das maiores percentagens de genes reguladores (8,4%) (Kung et al. 2010). Por outro lado, cerca de 10% do seu material genético compõem o genoma acessório, um conjunto de ilhas genómicas adquiridas através de transferência horizontal de genes e diferente de estirpe para estirpe (Kung et al. 2010; Silby et al. 2011). Ou seja, além do vasto conjunto de mecanismos de adaptação inatos à espécie, cada isolado tem o seu próprio armamentarium para dar resposta a situações de stress.
Esta espécie possui assim uma ubiquidade inerente, sendo encontrada igualmente no solo e em ambientes aquáticos. Por outro lado, a mesma capacidade adaptativa que lhe permite prosperar em diferentes nichos ambientais confere-lhe as ferramentas necessárias para causar infecção em vários organismos, nomeadamente leveduras, plantas, nematodes, insectos e mamíferos (Battle et al. 2009). Quando em contexto de infecção, estes aspectos do seu comportamento denominam-se de factores de virulência.
2
A colecção de factores de virulência produzida por P. aeruginosa é vasta (Pereira et al. 2014). Este conjunto inclui várias enzimas que degradam moléculas efectoras do sistema imunitário e elementos essenciais à estrutura celular e tecidual do hospedeiro, como proteases e elastases. Inclui também as fenazinas e os seus derivados, como a piocianina, que estão associados à alteração de estados de oxidação celular, comunicação inter-celular, e regulação da expressão genética, assumindo um papel crucial na sobrevivência da célula, principalmente em contexto de infecção (Pierson & Pierson 2010). A pioverdina, um pigmento esverdeado, é um sideróforo que medeia a absorção de iões de ferro, um elemento essencial à sobrevivência e crescimento do organismo (Pier & Ramphal 2010). De referir também a presença de sistemas de transporte membranar, nomeadamente sistemas de secreção do tipo III, que, apesar de não serem eles próprios factores de virulência, medeiam o efluxo de proteínas responsáveis pela destruição de barreiras epiteliais e de células do hospedeiro, e interfere com a actividade de macrófagos e neutrófilos.
Esta bactéria tem a capacidade de alternar entre o estilo de vida planctónico e crescimento em biofilme, que a ajuda a sobreviver em nichos específicos, como cateteres e diversas superfícies hospitalares, conferindo-lhe uma maior tolerância a desinfectantes e antibióticos; está também associado a colonizações crónicas de dispositivos internos e material prostético, assim como de pulmões de pacientes com fibrose cística ou bronquiectasias (Kerr & Snelling 2009). O flagelo polar e pili do tipo IV estão envolvidos na formação de biofilme, e medeiam a capacidade de mobilidade e são relevantes na aderência a tecidos do hospedeiro (Pier & Ramphal 2010).
Todos estes determinantes, entre muitos outros, associam-se numa rede de regulação complexa, que sentencia a actuação da bactéria. Sistemas de quorum-sensing (QS) são essenciais neste processo, pois é neles que assentam sistemas de comunicação celular intra e inter-espécies. Através destes sistemas, é possível coordenar comportamentos entre todos os indivíduos da população ao estimar o número de células da mesma, assim como o estado de vulnerabilidade do hospedeiro, ajudando na regulação de todos os factores de virulência da bactéria (Balasubramanian et al. 2013). Estes traços são produzidos em diferentes fases do processo infeccioso, promovendo a alternância
3
entre infecções agudas e crónicas e, no geral, determinando a gravidade da doença (Strateva & Mitov 2011).
Considerando esta colectânea de factores de virulência, é natural que esta bactéria mereça especial atenção devido à sua potencialidade como patógeno humano. P.
aeruginosa é responsável por infecções oportunistas – tanto agudas como crónicas –
em indivíduos cujo sistema imunitário se encontre debilitado, tais como vítimas de queimaduras, seropositivos, transplantados, entre outros (Kerr & Snelling 2009). Em pacientes com fibrose cística, a colonização dos pulmões por esta bactéria é responsável por um agravamento significativo das taxas de morbilidade e mortalidade associadas à doença, devido à impossibilidade de erradicação da infecção, que provoca diminuição do desempenho e, eventualmente, falha total do órgão (Workentine et al. 2013). Apesar de ser maioritariamente isolada em infecções do trato respiratório, urinário e da corrente sanguínea, esta espécie tem a capacidade de colonizar virtualmente qualquer local e tecido (Xu et al. 2013).
Este patógeno inspira particular preocupação como causa de infecções associadas a cuidados de saúde, pois a sua resistência intrínseca a diversos detergentes e desinfectantes dificulta a sua erradicação de superfícies hospitalares, o que pode promover o contágio de pacientes (Kerr & Snelling 2009). Uma infecção nosocomial é definida como aquela que se manifesta 48 horas após a entrada no hospital, e até 10 dias após a alta, ou em pacientes residentes em lares de terceira idade e instituições semelhantes (de Abreu et al. 2014). Na União Europeia (UE), cerca de 6% dos pacientes internados em hospitais desenvolvem infecções nosocomiais, o que representa cerca de 3,2 milhões de pacientes por ano. P. aeruginosa está entre as quatro espécies mais prevalentes, sendo responsável por cerca de 9% destas ocorrências, a seguir a
Escherichia coli (16%), Staphylococcus aureus (12,3%) e Enterococcus spp. (9,6%). Por
outro lado, se forem consideradas apenas as pneumonias e infecções do tracto respiratório inferior, o tipo de infecção nosocomial mais comum (23,5%), esta percentagem sobe para cerca de 18%, sendo esta bactéria o agente infeccioso mais isolado neste tipo de situação (ECDC 2013).
4
Infecções causadas por P. aeruginosa representam um desafio complexo para os prestadores de cuidados de saúde, devido à capacidade da bactéria de colonização de qualquer tecido de um indivíduo imunocomprometido, e à sua incrível tolerância a factores de stress e adaptação a condições desfavoráveis. Com o aumento da resistência a antibióticos e a tendência para evoluir para múltipla resistência quando em contacto prolongado com antibióticos (Davies & Davies 2010) – um aspecto fulcral a ter em conta quando se trata de um organismo frequentemente isolado de ambientes hospitalares (de Abreu et al. 2014; Quick et al. 2014) – atingimos um patamar em que é exigido um cuidado redobrado de forma a evitar um agravamento da situação. Para que continue a ser possível combater as infecções desta bactéria, é necessário entender os mecanismos de resistência ao seu dispor, os meios de evolução e disseminação, e encontrar métodos para travar, ou pelo menos abrandar, este processo.
1.2.
R
ESISTÊNCIA A ANTIMICROBIANOS:
SITUAÇÃO GLOBALNo início do século passado, Paul Erhlich estudou pela primeira vez a aplicação de compostos químicos no tratamento de infecções, desenvolvendo um composto sintético que contém arsénio – o Salvarsan – no tratamento da sífilis, um grave problema de Saúde Pública da época (Kaufmann 2008). Mais tarde, em 1932 um outro bacteriologista alemão, Gerhard Domagk, demonstrou a actividade antibacteriana da sulfonamida. Quando Leonard Colebrook comprovou a eficácia deste composto no tratamento de infecções de Streptococcus na corrente sanguínea em ratos e humanos, teve início a era das sulfonamidas, com novos derivados mais eficientes e/ou menos tóxicos a surgirem num curto espaço de tempo. Por outro lado, em 1944 foi iniciada a produção em massa da penicilina, composto produzido pelo fungo Penicillum notatum, dando resposta à procura imensa causada pelo aumento de infecções associadas aos ferimentos e más condições de vida provocadas pela II Guerra Mundial. O efeito inibitório da penicilina na bactéria Staphylococcus aureus havia sido descoberto por
5
Alexander Fleming quase duas décadas antes, mas as limitações tecnológicas da altura impediram testes mais avançados (Britannica 2015).
Estes foram os três marcos do início da chamada Era Dourada dos antibióticos. Devido ao uso destes compostos, deu-se um manifesto aumento na esperança média e qualidade de vida, não só pelo seu efeito directo no tratamento de infecções bacterianas, mas também porque diminuiu o risco associado a uma panóplia de procedimentos médicos que comprometiam o sistema imunitário humano, convertendo a medicina numa disciplina terapêutica e não apenas de diagnóstico (Höjgård 2012).
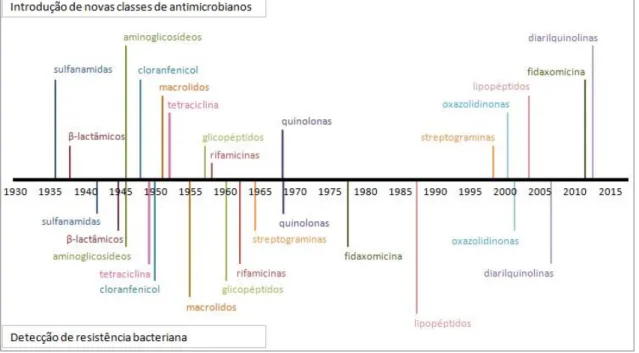
A Era Dourada prolongou-se por três décadas, durante a qual novas classes de antibióticos foram desenvolvidas (Figura 1). Entretanto, a partir do início da década de 1960, foram empregues esforços na compreensão do efeito da dosagem e vias de administração numa tentativa de melhorar o tratamento. Por outro lado, um crescente entendimento do mecanismo de acção dos antibióticos orientou modificações químicas dos compostos e, mais recentemente, o apoio de técnicas e estudos genéticos permitiu apostar na previsão de novos alvos (apesar de não ter tido os resultados esperados) (Davies & Davies 2010).
A partir da década de 1970 verificou-se uma queda abrupta na introdução de novas classes de antibióticos no tratamento de infecções bacterianas. Erradamente, a linha de pensamento da época defendia que as doenças infecciosas estariam na iminência da erradicação e seriam facilmente combatidas com os fármacos já desenvolvidos, o que levou à redução significativa do investimento na investigação e desenvolvimento (I&D) de novos antibióticos (Laxminarayan et al. 2013). Por outro lado, os regulamentos dos testes clínicos e, em última instância, da aprovação de novos fármacos estão mais exigentes. Actualmente, o processo de I&D é mais complexo, demorado, litigioso, e dispendioso, sem no entanto garantir taxas de sucesso superiores. Apesar de todas as classes de fármacos serem afectadas, os antibióticos parecem ter sofrido um maior impacto (Projan & Bradford 2007). Em 2012, um total de 28 compostos destinados ao tratamento de infecções bacterianas estavam em fase de
6
testes clínicos a nível mundial, sendo que apenas 8 eram dirigidos a organismos Gram-negativos (Theuretzbacher 2012).
A continuidade do sucesso de um agente terapêutico está restringida pela eventual manifestação de tolerância ou resistência a esse mesmo composto (Davies & Davies 2010). Em vários casos, a detecção de mecanismos de resistência a determinado antibiótico foi prévia à aplicação do composto em contexto clínico, como é o caso da tetraciclina, lipopéptidos ou diarilquinolonas (Figura 1).
Com o crescente uso de antibióticos, a pressão selectiva imposta sob a população bacteriana aumentou. Com os mais variados mecanismos a evoluírem, e diversas vias de transmissão e disseminação dos mesmos, a resistência a antimicrobianos é hoje um problema global (ECDC & EMEA 2009; Jindal et al. 2015).
Considerando apenas a espécie P. aeruginosa, em 2013, a maioria dos países participantes na Rede Europeia de Vigilância de Resistência a Antimicrobianos (EARS-Net) registou mais de 10% dos isolados resistentes a cada um dos antibióticos ou classes considerados (piperacilina-tazobactam, ceftazidima, carbapenemos, fluoroquinolonas e aminoglicosídeos). Portugal registou níveis de resistência quase
Figura 1: Representação temporal da introdução e detecção de resisitência a antimicrobianos no tratamento de infecções. Em cima, ano de introdução de novas classes de antimicrobianos e em baixo, ano de detecção de resistência às mesmas (Lewis 2013)
7
sempre acima da média europeia, entre 14,2% (aminoglicosídeos) e 24,1% (piperacilina + tazobactam) (ECDC 2014).
Com o aparecimento de estirpes multirresistentes com opções de tratamento extremamente limitadas, assistimos agora ao início da era pós-antibiótica, na qual os benefícios médicos, económicos e sociais trazidos por estes compostos serão atenuados, ou até mesmo anulados (ECDC & EMEA 2009; Jindal et al. 2015).
1.2.1.FACTORES IMPULSIONADORES E VIAS DE DISSEMINAÇÃO DE RESISTÊNCIA
A resistência bacteriana ocorre naturalmente graças a milhões de anos de evolução conjunta como mecanismo de defesa contra antibióticos segregados por outros microrganismos (D’Costa et al. 2011), e é transmitida em muitos casos através de transferência horizontal de genes (Forsberg et al. 2012). Todavia, a imensa pressão selectiva imposta ao longo de décadas de uso incorrecto e excessivo destes compostos estimulou a selecção de bactérias resistentes, num dos melhores exemplos da teoria da selecção natural de Charles Darwin (Davies & Davies 2010; Bell et al. 2014).
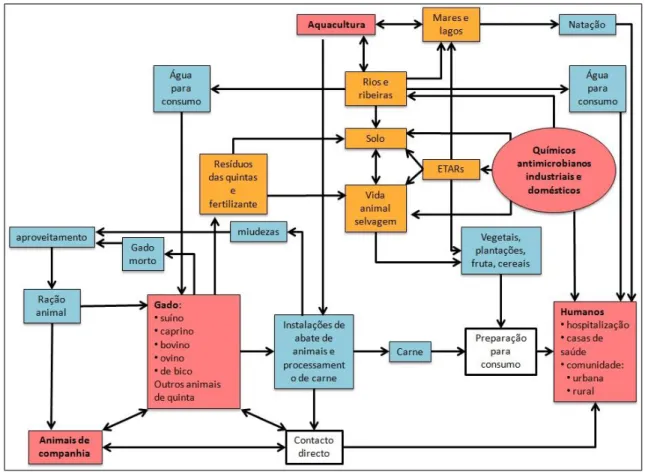
Desde meados do século XX, estima-se que milhares de toneladas de antibióticos tenham sido produzidas e usadas nas mais variadas aplicações, quase sempre sem ponderação dos seus efeitos nocivos a médio e longo prazo (Laxminarayan et al. 2013). Como é esquematizado na Figura 2, são várias e complexas as vias de disseminação de resistência a estes fármacos.
Uso clínico
No ano de 2012, estima-se que em 26 países da UE 3400 toneladas destes fármacos tenham sido aplicadas no tratamento de doenças infecciosas (68%) e em terapia profiláctica (ECDC et al. 2015), quer na comunidade, quer em pacientes internados. Porém, a Organização Mundial de Saúde (WHO) classifica a aplicação de metade dessa quantidade como desnecessária (Carlet et al. 2012).
8
A administração excessiva de antibióticos tem início na prescrição médica (Bell et al. 2014). No diagnóstico, geralmente não é feita a identificação no patógeno em causa nem confirmada a susceptibilidade a antimicrobianos, uma vez que a maioria dos testes disponíveis para o efeito demoram entre 36 a 48 horas, tornando-se obsoletos e impraticáveis. Na ausência de métodos auxiliares de diagnóstico, são administrados antibióticos com base em informações empíricas, que podem estar desactualizadas ou descontextualizadas. Em caso de dúvida, é normal os prestadores de cuidados de saúde tentarem cobrir todas as possibilidades, prescrevendo antibióticos de largo espectro, apesar de serem muitas vezes desnecessários. O facto de muitos destes prestadores não terem qualquer formação específica em doenças infecciosas agrava a situação (Carlet et al. 2012; Lee et al. 2013).
Por outro lado, em alguns países, os diferentes intervenientes na distribuição de antimicrobianos são incentivados no sentido de estimularem o seu consumo, geralmente por motivos monetários. Na China, uma parcela significativa da receita dos hospitais provém da venda destes compostos (Laxminarayan et al. 2013).
Figura 2: Principais fontes de pressão selectiva (vermelho), reservatórios ambientais (laranja) e vias de
9
Os pacientes representam também um papel importante neste problema. Segundo uma sondagem feita nos Estados Unidos da América (EUA), cerca de 48% dos inquiridos esperava receber um antibiótico quando se dirigia a um consultório médico (Vanden Eng et al. 2003), e pressionam os médicos nesse sentido. Porém, é comum os pacientes interromperem o tratamento assim que os sintomas atenuam, permitindo a evolução de novos mutantes resistentes ao agente terapêutico usado. A auto-medicação é também um factor importante, ainda que mais comum em países em desenvolvimento onde a compra sem prescrição médica é possível em farmácias ou noutros estabelecimentos de venda ao público. Actualmente, verifica-se ainda um aumento das vendas através da internet, que são muito difíceis de controlar. Além da questão óbvia da errada e desnecessária aplicação dos antimicrobianos, o comércio de fármacos contrafeitos com concentrações sub-óptimas constituem mais um factor promotor da resistência bacteriana (Carlet et al. 2012; Laxminarayan et al. 2013).
Uso veterinário
Apesar de os antibióticos serem primariamente necessários para a melhoria da saúde humana, o seu uso veterinário absorve a maior parte dos fármacos produzidos. Em 2012, cerca de 70% das mais de 11 000 toneladas consumidas em 26 países da UE foram empregues em quintas de criação de animais (ECDC et al. 2015).
A indicação destes compostos para uso veterinário ocorre para tratamento de doenças infecciosas e para a sua prevenção. Quando se verifica que um pequeno grupo de animais contraiu uma infecção, é administrado o fármaco não só aos animais doentes mas a toda a população, de modo a prevenir o contágio. Por outro lado, antibióticos são utilizados para compensar as más condições sanitárias (Laxminarayan et al. 2013). Simultaneamente, a mistura de doses sub-terapêuticas de antibióticos na alimentação dos animais como promotores do crescimento é comum. Embora tenha sido proibido na UE em 2006 (EFSA 2015), e a Food and Drug Administration (FDA) nos EUA tenha lançado orientações para o uso apropriado de antibióticos neste contexto (FDA 2014), esta prática ainda é bastante utilizada, favorecendo a selecção e disseminação de estirpes resistentes (E. Burow et al. 2014; E Burow et al. 2014).
10
Nos países da UE, a venda de todos os antimicrobianos para uso veterinário está sujeita a receita médica. No entanto, noutras regiões do planeta estes compostos são usados sem qualquer regulamentação ou controlo (Laxminarayan et al. 2013). Ademais, algumas das classes de antibióticos usadas em animais são as mesmas aplicadas em contexto clínico. Há indícios de que o uso veterinário tem contribuído para a evolução das estirpes multirresistentes que vêm a ameaçar a saúde humana, uma vez que as bactérias resistentes são transmitidas entre animais e humanos (Harrison et al. 2013).
A transmissão de microrganismos resistentes entre gado e humanos pode dar-se em dois momentos distintos: nos locais de criação e abate dos animais, ou nos locais de consumo. A transmissão para funcionários de quintas ou de matadouros é recorrente, ocorrendo através do contacto directo com os animais ou através do manuseamento de carne crua, ou ainda devido ao contacto com superfícies contaminadas (E. Burow et
al. 2014). Estas bactérias podem integrar a flora dos estabelecimentos referidos, onde
também têm oportunidade de transmitir os genes de resistência para outros organismos presentes nesse mesmo nicho. Por outro lado, a contaminação pode ocorrer em cozinhas. Apesar de a confecção destruir genes e bactérias, o manuseamento errado da carne crua permite a sua disseminação (Carlet et al. 2012).
Meio ambiente
Existem ainda vias de disseminação indirecta que conduzem à contaminação do meio ambiente e, mais tarde, de humanos. Produções agrícolas são adubadas com fertilizantes provenientes de quintas, ricos em bactérias comensais ou patogénicas potencialmente portadoras de genes de resistências. Mais tarde, os produtos consumidos em restaurantes e habitações podem ainda levar consigo vestígios desses organismos. Mais ainda, as águas de escorrência de locais de produção animal e agrícola, assim como águas provenientes de aquacultura (onde estes compostos também são aplicados) misturam-se com lençóis freáticos e cursos de água superficiais, infiltrando o sistema aquático a nível global. Através de sistemas de rega, consumo de alimentos de origem fluvial ou marinha, e contacto com a água, seja para consumo ou para banhos, estirpes bacterianas resistentes a antibióticos têm a
11
potencialidade para causar infecções adquiridas na comunidade (Heuer et al. 2011). Uma meta-análise efectuada por Bell et al. (2014) conclui que a pressão selectiva exercida a nível da população é um maior determinante do risco de albergar bactérias resistentes do que a pressão a nível individual.
As estações de tratamento de águas residuais (ETARs) são também uma grande fonte de genes de resistência. Afluentes abundantes em substância activa, com origem em fábricas de produção de antimicrobianos, hospitais e na comunidade em geral, desaguam nestas estações, exercendo uma pressão selectiva enorme na flora bacteriana local. Estas bactérias – ou os seus genes de resistência – são depois libertados para os solos e meio aquático, tal como os fármacos. O facto de a quantidade de genes de resistência e de antibióticos aumentar com a densidade populacional, ou com a proximidade a fábricas deste tipo de fármaco suporta este raciocínio (Graham et al. 2011; Khan et al. 2013).
1.2.2.IMPLICAÇÕES SOCIAIS
As crescentes taxas de resistência a antimicrobianos têm uma ampla influência na nossa sociedade, que pode ser observada em três sectores distintos, ainda que intimamente interligados: (i) maior morbilidade e mortalidade dos pacientes infectados; (ii) maiores custos financeiros associados aos cuidados de saúde e perda de productividade; e (iii) maior risco associado a uma panóplia de procedimentos médicos.
A resistência a antibióticos é hoje reconhecida como um móbil do aumento das taxas de morbilidade e mortalidade associadas a infecções bacterianas (Davies & Davies 2010). Em certos casos, a taxa de mortalidade pode subir para o dobro (Nathwani et al. 2014) ou até mesmo para o triplo (Zilberberg et al. 2014) quando comparadas às taxas associadas a infecções por microrganismos susceptíveis. Nos EUA, o Centro de Prevenção e Controlo de Doenças (CDC) estima que mais de dois milhões de pessoas sejam infectadas com bactérias resistentes anualmente, resultando em mais de 23 000 mortes (CDC 2013). Nos países da UE, o número de mortes anuais associados apenas a
12
bactérias multi-resistentes ronda as 25 000 (ECDC & EMEA 2009). Demora na aplicação de terapia apropriada, baixa eficácia e/ou toxicidade superior dos antibióticos, dosagem incorrecta, e maior probabilidade de necessitar de cirurgias ou procedimentos invasivos são alguns dos factores que explicam a incidência de taxas de mortalidade superiores associadas a bactérias resistentes (Maragakis et al. 2008). Taxas de morbilidade mais elevadas estão associadas a maiores tempos de internamento e a maiores custos associados aos cuidados de saúde. Por outro lado, há ainda a considerar o custo social das perdas de produtividade, que também acarretam encargos financeiros. Na Europa, os gastos motivados por infecções de apenas cinco organismos [S. aureus resistente a meticilina (MRSA), Enterococcus faecium resistente a vancomicina, Escherichia coli e Klebsiella pneumoniae resistentes a cefalosporinas de terceira geração, e P. aeruginosa resistente a carbapenemos] ascenderam a 1,5 mil milhões de euros, enquanto nos EUA foram gastos 2,7 mil milhões de dólares apenas com infecções de P. aeruginosa multi-resistente (ECDC & EMEA 2009). Um estudo do CDC estima que se gastem naquele país 20 mil milhões de dólares com cuidados médicos extraordinários, e ainda 35 mil milhões de forma a colmatar perdas de produtividade de pacientes infectados com bactérias resistentes (CDC 2013). Estes valores devem ser comparados com precaução, uma vez que os custos associados aos serviços de saúde estadunidenses são geralmente muito superiores aos europeus. A nível mundial, é difícil, se não impossível, estimar o custo extraordinário associado a estas infecções. Nos países subdesenvolvidos, a informação disponível é pouca e duvidosa. No entanto, maiores taxas de doenças infecciosas e acesso restrito a medicação sugerem uma sobrecarga superior à dos países desenvolvidos (Laxminarayan et al. 2013).
Por último, a resistência a antimicrobianos inflacciona o risco associado a vários procedimentos médicos que, de alguma forma, deixam o paciente mais propenso a contrair infecções bacterianas. Entre eles encontram-se a quimioterapia contra o cancro, tratamento de doenças auto-imunes, diálise, transplantes de órgãos e de medula, assim como toda e qualquer cirurgia (CDC 2013). Uma das grandes conquistas dos antibióticos foi permitir este tipo de procedimentos. A resistência bacteriana
13
ameaça o regresso à era pré-antibiótica ao aumentar a taxa de infecção pós-operatória para 50%, e a de mortalidade para 30% (Barriere 2015). Estes dados não são mais do que as melhores estimativas possíveis, dado os recursos e informação disponível. Por exemplo, no que diz respeito ao número de mortes associadas a esta problemática, a distinção entre mortes causadas por bactérias resistentes, mortes nas quais bactérias resistentes estiveram envolvidas mas não foram o único factor, ou mortes em que bactérias resistentes estavam presentes mas não estiveram associadas à causa da morte é feita de forma subjetiva. Por outro lado, em alguns casos são apenas contabilizadas as mortes que ocorreram em hospitais (CDC 2013). Estes valores, apesar de serem mal quantificados, representam provavelmente o maior fardo imposto pela resistência bacteriana à sociedade (Laxminarayan et al. 2013)
A estimativa dos custos financeiros associados a infecções por bactérias resistentes é ainda mais difícil de fazer. Avaliar o custo total da estadia e tratamento num hospital é mais complexo do que pode parecer, uma vez que é necessário incluir factores difíceis de quantificar monetariamente como, por exemplo, os recursos utilizados ou a baixa na produtividade. Por outro lado, há ainda quem defenda que nem todos os parâmetros estão a ser considerados neste cálculo, como é o caso da impossibilidade de um hospital receber um paciente no período de tempo a mais que outro é mantido internado devido a uma bactéria resistente (Maragakis et al. 2008).
Informação actualizada e completa acerca das reais implicações clínicas e económicas da resistência a antibióticos pode ajudar a orientar medidas para abordar esta questão, além de estimular os prestadores de cuidados de saúde a seguir as directrizes fornecidas. Admite-se ainda que possa sensibilizar o poder legislativo para a necessidade de aprovar políticas que ajudem a controlar a resistência antimicrobiana, e que estimulem o aumento de investimento na I&D de novos agentes antimicrobianos. Por último, estes dados podem também resultar em prognósticos de pacientes individuais mais precisos (Maragakis et al. 2008).
14
1.2.3.ACÇÕES DE CONTROLO E PREVENÇÃO DE RESISTÊNCIA
Uma vez que a produção de novos antibióticos estagnou, é urgente tomar medidas para prolongar a eficácia dos fármacos que já estão em uso. Nesse sentido, várias acções, sejam elas de nível local, nacional ou global, foram já postas em marcha para avaliar, disciplinar e aprimorar o uso de antibióticos.
Em 2001, a Organização Mundial de Saúde definiu uma estratégia global para contenção da resistência a antimicrobianos, e no Dia Mundial da Saúde de 2011 deu nova ênfase a esse conjunto de recomendações ao exortar os estados membros a adoptarem atitudes concretas para abordarem esta questão (WHO 2001; 2014). Estas recomendações foram feitas com consideração pelo facto de que recai sobre os países a principal responsabilidade do sucesso destas acções, já que depende dos governos a sua priorização, financiamento, execução e avaliação.
As atitudes a tomar devem seguir cinco domínios fundamentais: (i) vigilância do consumo de antibióticos e resistência; (ii) educação de prescritores, profissionais de saúde e grande público; (iii) regulamentação, nomeadamente da promoção dos antibióticos por parte da indústria farmacêutica; (iv) investigação dos mecanismos de resistência e vias de disseminação, assim como de novos agentes antimicrobianos com alvos diferentes; e (v) prevenção de infecções.
Na UE, vários programas foram criados para ajudar os estados-membro a cumprirem estas recomendações, tanto na área da vigilância como da educação. A EARS-Net fornece dados anuais relativos à prevalência e disseminação da resistência antimicrobiana na Europa. A Rede Europeia de Vigilância do Consumo de Antimicrobianos (ESAC-Net), por seu lado, compila informação respeitante ao consumo destes agentes farmacológicos, tanto na comunidade como em contexto hospitalar. Em ambos os casos, a informação compilada ou referente a países e/ou anos particulares está disponível para consulta na página online do Centro Europeu de Prevenção e Controlo de Doenças (ECDC) (www.ecdc.europa.eu/). A UE tem também promovido a 18 de Novembro de cada ano o Dia Europeu dos Antibióticos, uma iniciativa importante na educação e sensibilização do grande público (e, por vezes,
15
também dos profissionais de saúde), em que há um grande empenho no desenvolvimento de “infográficos” e restante material de campanha (ECDC 2015; Direcção Geral de Saúde 2014). A auto-medicação foi o tema abordado na edição de 2014.
A UE tem vindo também a aprovar legislação importante para o controlo da resistência, da qual são exemplos as proibições de compra de antibióticos sem prescrição médica e de uso destes fármacos na ração animal como factores de crescimento.
Na área da prevenção, práticas de higiene fundamentais têm sido enfatizadas, nomeadamente em locais de prestação de cuidados de saúde, desde centros ambulatórios até unidades de cuidados intensivos. A WHO iniciou uma campanha em 2009 para incentivar à higiene das mãos, considerando esta prática como o ponto de partida para reduzir infecções associadas a cuidados de saúde e aumentar a segurança do paciente (WHO 2015).
No entanto, estes esforços globais têm produzido efeitos limitados devido à falta de coordenação, principalmente com países em desenvolvimento, que têm maiores obstáculos em controlar o acesso e consumo de antibióticos, assim como em implementar medidas de higiene devido às más condições sanitárias (Carlet et al. 2012).
Em Portugal, além de serem seguidas as directrizes do ECDC e da WHO, a Direcção Geral de Saúde (DGS) instituiu o Programa de Prevenção e Controlo de Infecções e de Resistência a Antimicrobianos, ao abrigo do qual é publicado um relatório anual. Este documento comunica a situação nacional relativa às infecções associadas aos cuidados de saúde, consumo de antibióticos e resistência aos mesmos por parte de algumas espécies bacterianas mais alarmantes, e deixa algumas recomendações aos Conselhos de Administração e Conselhos Directivos das Unidades de Saúde portuguesas, assim como aos profissionais de saúde. No relatório de 2014, o foco dos conselhos da DGS centrava-se na redução da duração da terapêutica antibiótica, da profilaxia antibiótica cirúrgica com duração superior a 24 horas, e da percentagem de doentes com feridas crónicas a realizar terapêutica antibiótica sistémica. Era também referida a taxa de
16
infecção hospitalar em Portugal mais elevada do que a média europeia, e ficou registado o apelo à sua diminuição. Para a concretização destas directivas, é recomendado às Unidades de Saúde a adesão a todos os instrumentos de vigilância epidemiológica, a participação na Campanha Nacional de Precauções Básicas de Controlo de Infeção, e a implementação local de um programa de apoio à prescrição antibiótica. Finalmente, é relevada a necessidade de os profissionais de saúde terem formação da área da controlo e prevenção de infecções e de resistência a antimicrobianos. Contudo, este investimento na área é recente e os resultados positivos são ainda escassos (Paiva et al. 2014).
À parte destas iniciativas de grande escala, Programas de Gestão de Antibióticos (PGA) têm surtido algum efeito em centros hospitalares, embora ainda se encontrem em fase de optimização (Zhang & Singh 2015). Estes programas definem-se como um conjunto de actividades e políticas que visam melhorar o uso racional de antibióticos e optimizar o tratamento, por forma a diminuir a pressão selectiva e, consequentemente, reduzir a resistência bacteriana. Tais conjuntos de medidas podem diminuir o consumo de antibióticos (em 20 a 40%), a incidência de infecções nosocomiais, o tempo de internamento, e a prevalência de bactérias resistentes. Para atingir bons resultados, admite-se como relevante considerar as seguintes medidas: (i) lista restrita dos antibióticos em uso; (ii) directivas de tratamento gerais e padronizadas; (iii) auditoria de prescrições médicas; e (iv) educação/formação a todos os níveis (Laxminarayan et
al. 2013).
Contudo, todas estas etapas devem ser orientadas por um pleno conhecimento da situação de resistência bacteriana e consumo de antibióticos. A vigilância permanente é essencial para a promoção e orientação dos programas de gestão, assim como para a avaliação dos seus resultados (Laxminarayan et al. 2013). De facto, orientações baseadas em informações desactualizadas podem inclusivamente conduzir a um aumento da taxa de mortalidade (Lee et al. 2013).
Várias estratégias podem ser empregues no âmbito destes programas, tais como directrizes para a prescrição empírica, implementação de formulários e/ou sistemas de autorização prévia à prescrição, revisão do plano de tratamento após 48 a 72 horas do
17
seu início, alternância de antibióticos, ou uso de métodos auxiliares de diagnóstico (Lee et al. 2013). Até à data, a avaliação da eficácia de cada uma, ou da combinação de várias, das estratégias idealizadas e a comparação entre elas é um processo complexo, já que os vários estudos realizados utilizam diferentes critérios para medir o sucesso do programa em questão (Zhang & Singh 2015). Ainda assim, há indícios de que os PGA podem, de facto, ser úteis no controlo e diminuição da resistência a antibióticos, como se verificou no caso do MRSA na UE (ECDC 2014).
No entanto, os esforços empregues no controlo da resistência a antibióticos nunca irão além de adiar o inevitável, pois enquanto existirem antibióticos, existirá resistência microbiana, em permanente evolução (Lee et al. 2013). Esta noção eleva ainda mais a importância e a necessidade destas iniciativas e programas, já que concedem o tempo necessário ao desenvolvimento de antibióticos com alvos diferentes e que, nessa altura, prolongarão a sua eficácia até novos métodos de combate de infecções serem encontrados.
1.3.
M
ECANISMOS DE RESISTÊNCIA BACTERIANAAs células bacterianas encerram uma multitude de processos ou características que lhes são únicos e distintos dos encontrados em células eucariotas, nomeadamente nas humanas. Estas diferenças têm sido usadas como alvo de agentes antimicrobianos, assegurando assim que os fármacos são tóxicos para o patógeno, mas que os efeitos secundários para os hospedeiros são muito reduzidos. A inibição da síntese da parede celular por antibióticos β-lactâmicos, a inibição da síntese proteica por macrólidos, aminoglicosídeos e tetraciclinas, ou a interrupção de vias metabólicas necessárias à produção de nucleótidos por sulfonamidas são apenas alguns exemplos dos mecanismos de acção dos antibióticos (Dzidic et al. 2008). Os organismos bacterianos adaptaram-se à presença destes e de outros compostos prejudiciais à sua sobrevivência, e desenvolveram diversos mecanismos que anulam a sua actividade
18
Em termos genéticos, a resistência a antibióticos deve ser analisada em duas vertentes: o aparecimento de novos genes de resistência como resultado de mutações, e a disseminação destes através da transferência horizontal. As mutações são geralmente espontâneas e aleatórias, envolvem polimorfismos por substituição de um só nucleótido (SNP) e ocorrem a uma taxa de 10-9 a 10-10 por gene. A transferência destes genes é facilitada pelo facto de estarem usualmente inseridos em elementos genéticos móveis, como transposões, integrões e plasmídeos. Estes elementos actuam como vectores de transferência de informação genética durante processos de conjugação, transformação e transdução entre células da mesma espécie ou de espécies diferentes (Tang et al. 2014).
As estratégias empregues pelos microrganismos para resistirem a agentes antimicrobianos, apesar de numerosas e variadas, podem todas elas ser explicadas pelo conceito “bala-alvo”, ou seja, enquanto alguns mecanismos se baseiam na modificação do alvo molecular do agente farmacológico, outros impedem a actuação do antibiótico – a “bala” (Aminov 2010).
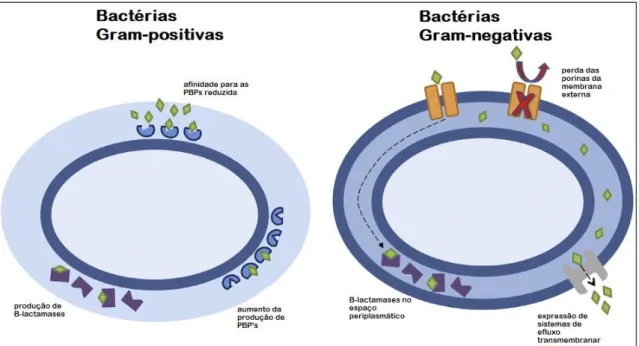
Um tipo de mecanismo relacionado com os alvos moleculares baseia-se na protecção destes através de modificações que impedem a ligação dos antibióticos. Estas modificações podem ter origem em alterações genéticas pontuais, como é o caso da
Figura 3: Mecanismos de resistência a antibióticos β-lactâmicos mais comuns em bactérias de Gram
19
mutação da RNA polimerase que confere resistência à rifampicina, ou em acção enzimática, sendo a metilação da sub-unidade 23S do rRNA responsável pela resistência a macrólidos, um possível exemplo (Aminov 2010). Por outro lado, os alvos moleculares podem ser totalmente substituídos devido à captação de DNA externo. Genes homólogos àqueles que codificam o alvo podem ser adquiridos durante o processo de transformação natural e expressarem proteínas insensíveis que substituem as afectadas por antibióticos, como acontece com o gene mecA, que codifica uma proteína homóloga ao alvo da meticilina e que torna o S. aureus resistente a ela (Blair et al. 2015).
Os alvos podem ainda ser protegidos indirectamente através do bloqueio do acesso do antibiótico ao espaço onde se encontra o alvo. A formação de biofilme é um método eficaz para dificultar a entrada do antibiótico no organismo, já que a secreção de grandes quantidades de exopolissacarídeos dificulta o contacto com o exterior da célula. Uma outra estratégia de resistência indirecta passa pela perda das porinas através das quais os antibióticos têm acesso ao interior da célula, ou pela alteração da permeabilidade das membranas celulares (Dzidic et al. 2008).
As estratégias de resistência que se focam na “bala” envolvem modificações enzimáticas do composto activo. Os aminoglicosídeos podem ser inactivados através da sua acetilação, ou da transferência de outros grupos químicos (Blair et al. 2015). Em alguns casos, a resistência pode ser conferida pela degradação enzimática do fármaco, como acontece com a hidrólise de β-lactâmicos por β-lactamases (Tang et al. 2014). A acção dos antibióticos pode ainda ser impedida devido à sua expulsão da célula através de sistemas de efluxo, um tipo de resistência intrínseca a muitas espécies de Gram negativo, e com grande relevância na tolerância a antibióticos usados contra infecções causadas por organismos de Gram positivo (Blair et al. 2015).
1.3.1.RESISTÊNCIA MEDIADA POR β-LACTAMASES
A resistência mediada por β-lactamases é um dos maiores desafios tanto para o tratamento de infecções, como para o desenvolvimento de novos e eficazes fármacos,
20
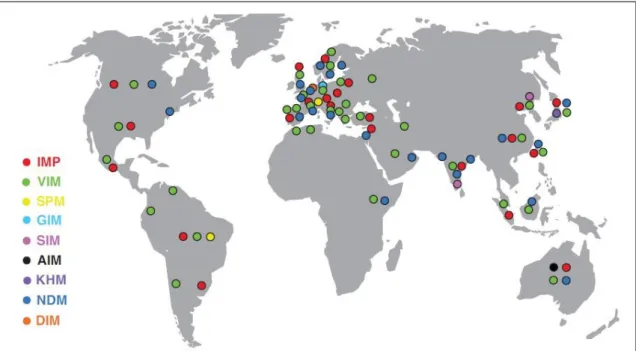
já que a sua evolução e disseminação é rápida e eficiente. Devido à facilidade que os genes de resistência aos β-lactâmicos têm em ser horizontalmente transferidos intra- e inter-espécies, e à pressão selectiva a que estão sujeitos, é possível que estes genes sejam os mais dispersos no planeta (Figura 4) (Davies & Davies 2010).
O alvo das β-lactamases: antibióticos β-lactâmicos
A parede celular bacteriana tem por base uma macromolécula elástica de peptidoglicano que protege a célula de stress osmóstico (dada a elevada pressão osmótica intracelular), define a forma da célula e medeia alterações morfológicas em resposta a condições adversas. A sua instabilidade conduz à lise e morte celular (Macheboeuf et al. 2006). Ao longo de uma só geração, mais de metade da parede celular é renovada, num processo complexo crucial em momentos de crescimento e divisão celular (Johnson et al. 2013).
As PBPs (Penicillin-Binding Proteins) possuem dois domínios distintos que catalisam reacções de transglicosilação e de transpeptidação responsáveis pelo processo de polimerização do peptidoglicano (Zapun et al. 2008; Johnson et al. 2013). Uma vez que o peptidoglicano é encontrado apenas em bactérias, as etapas da biossíntese desta molécula são alvo de vários antibióticos, nomeadamente dos β-lactâmicos.
21
As PBPs, tal como o nome indica, são o alvo dos β-lactâmicos. Estes antibióticos de baixa massa molecular imitam o substrato natural das PBPs e ligam-se ao centro activo do seu domínio transpeptídico. O complexo covalente que se forma entre o fármaco e as proteínas é hidrolisado de forma muito lenta – considerando o tempo do ciclo de vida bacteriano – impedindo que a enzima catalise as reacções de polimerização do peptidoglicano (Zapun et al. 2008; Worthington & Melander 2013).
Os antibióticos β-lactâmicos são os mais utilizados (Bell et al. 2014) e mais eficazes no tratamento de infeções provocadas por diversas espécies bacterianas (Worthington & Melander 2013). As penicilinas, cefalosporinas, carbapenemos, e monobactâmicos perfazem esta classe (Llarrull et al. 2010; Fair & Tor 2014). Para o tratamento de infecções adquiridas na comunidade, os fármacos mais frequentemente administrados são combinações de penicilinas com inibidores de β-lactamases, como é o caso de piperacilina e tazobactam, e amoxicilina com ácido clavulânico, ou cefalosporinas orais, como a cefixima e cefpodoxima. Ceftazidima, cefepima, e outras cefalosporinas de largo espectro são aplicadas no tratamento de infecções nosocomiais causadas por microrganismos não resistentes. Os carbapenemos são geralmente empregues em casos que envolvam patógenos multi-resistentes, já que não são inactivados pelos mecanismos de resistência mais comuns que afectam as sub-classes supracitadas (Bush 2010). Os monobactâmicos são igualmente utilizados em casos de resistência a outros antibióticos desta classe, e também de alergia (Trubiano & Phillips 2013).
A resistência a esta classe de antibióticos foi detectada pela primeira vez em 1944
(Figura 1). Desde então, foram identificadas várias estratégias de defesa contra estes
compostos. Em bactérias Gram-negativas, o mecanismo mais comum – e preocupante – é a produção de β-lactamases (Worthington & Melander 2013).
Caracterização de β-lactamases
O consumo de antibióticos conduz ao aumento da resistência bacteriana (Bell et al. 2014). No entanto, no caso dos antibióticos β-lactâmicos esta conexão é particularmente evidente (Davies & Davies 2010). Da mesma forma que estes antibióticos são os mais utlizados, os mecanismos de resistência bacteriana correspondentes são os mais difundidos. Até à data, já foram identificados mais de 2000 enzimas intervenientes neste tipo de resistência, incluindo novos genes e
22
derivados mutantes (Srivastava et al. 2014), e o espectro de resistência continua a alargar (Galán et al. 2013).
A classificação das β-lactamases não é consensual, existindo vários esquemas que utilizam diferentes aspectos como critério de classificação. Contudo, há duas que se destacam: a classificação de Ambler (Ambler 1980; Hall & Barlow 2005) e a de Bush-Jacoby (Bush & Bush-Jacoby 2010). Esta última tem em atenção o perfil hidrolítico e a sensibilidade a inibidores das enzimas e, portanto, fornece informação útil em contexto clínico, ao correlacionar as moléculas com os antibióticos a que conferem resistência. No entanto, tem uma componente subjectiva, e é pouco intuitiva. Já a classificação de Ambler utiliza a homologia das sequências proteicas e a estrutura molecular para agrupar as enzimas. É claramente a forma mais fácil, directa e objectiva de agrupar as β-lactamases, sendo a opção que menos controvérsia reúne.
A classificação de Ambler começa por separar as β-lactamases em dois grandes grupos de acordo com o seu centro activo. As metalo-β-lactamases (MBLs) dependem de iões de metal bivalentes (geralmente zinco) para hidrolisar o seu substrato, enquanto as restantes não são dependentes de um ião metálico, possuindo uma serina no seu lugar. Enquanto as MBLs compõem a classe B, as restantes estão divididas nas classes A, C e D, de acordo com a sua sequência amino-acídica.
Na classe A estão agrupadas as primeiras β-lactamases não dependentes de metal a serem identificadas. Os membros originais tinham um estreito espectro de actuação, no entanto desde a década de 1990 que são detectadas enzimas de largo espectro (ESBLs), capazes de hidrolisar penicilinas, cefalosporinas de primeira à quarta geração, e monobactâmicos (Meini et al. 2014). Famílias como a TEM e a SHV estão amplamente difundidas em Enterobacteriaceae e Pseudomonas spp.. ESBLs da família CTX-M, apesar de terem emergido mais tarde, são actualmente as mais prevalentes em centros hospitalares. Devido ao uso de carbapenemos contra estas ESBLs, as carbapenemases de classe A já são também uma realidade, sendo a família KPC (Klebsiella pneumoniae carbapenemase) a mais notável (Bush 2010; Meini et al. 2014). Quando foram identificadas β-lactamases com um centro activo de serina e uma sequência proteica demasiado diferente das enzimas da classe A, foi criada a classe C.
23
Esta classe é principalmente reconhecida pela β-lactamase cromossomal AmpC, embora também exista em plasmídeos (Pfeifer et al. 2010). As enzimas desta classe são das β-lactamases mais abundantes, a julgar pelo número de organismos que as produzem; a AmpC é produzida pela maioria das bactérias da família Enterobacteriaceae, e é também considerada um traço característico de P. aeruginosa (Bush 2010). Esta enzima cromossomal é geralmente expressa em níveis basais, podendo ser produzida em níveis superiores se induzida por alguma penicilina ou cefalosporina de estreito espectro, conferindo resistência contra esses mesmos antibióticos. Por outro lado, a desrepressão mutacional do gene ampC conduz à produção da enzima em quantidades muito elevadas, e à consequente resistência a antibióticos como cefalosporinas de terceira geração e monobactâmicos. Quando na presença de estratégias de resistência complementares (como a perda da porina OprD), esta mesma mutação conduz à resistência a carbapenemos (Poole 2011). A classe D de Ambler é também composta por enzimas não dependentes de metal com baixa homologia proteica com as restantes classes deste tipo. Esta família é bastante numerosa e possui uma grande variedade de moléculas, que podem ser codificadas por genes cromossomais ou localizados em plasmídeos, e está globalmente disseminada (Pfeifer et al. 2010). As β-lactamases OXA que compõem esta classe foram assim denominadas pela sua capacidade de degradação da oxacilina (da sub-classe das penicilinas), mas as cefalosporinas são também um substrato de algumas enzimas desta família, categorizadas como ESBLs. Por outro lado, outras OXA-β-lactamases alargaram o seu espectro noutro sentido, abrangendo agora os carbapenemos. Apesar da opinião corrente ser que estas carbapenemases ainda estão pouco disseminadas em P. aeruginosa (El Garch et al. 2011), uma outra hipótese é que o espectro de acção das enzimas da classe D presentes nesta espécie esteja mal avaliado (Antunes et al. 2014).
A classe B de β-lactamases pode ainda ser subdividida nas sub-classes B1, B2 e B3, de acordo com a homologia entre si. Estas enzimas são caracterizadas pela grande diversidade catalítica, podendo hidrolisar todos os antibióticos β-lactâmicos, à excepção dos monobactâmicos. Na sua maioria, estas enzimas são intrínsecas a diversas espécies bacterianas patogénicas e ambientais, codificadas por genes
24
inseridos no cromossoma. No entanto, algumas famílias de enzimas pertencentes à sub-classe B1 têm a sua informação genética inserida em plasmídeos, geralmente associada a integrões portadores de outros mecanismos de resistência, tais como sistemas de efluxo ou resistência a aminoglicosídeos e quinolonas (Fair & Tor 2014), assim como factores de virulência (Beceiro et al. 2013).
As MBLs são a principal causa dos elevados níveis de resistência a carbapenemos a nível mundial (Hawkey & Jones 2009). As famílias IMP, VIM e NDM – com 48, 43 e 14 variantes identificadas, respectivamente (McArthur et al. 2013) – são as mais relevantes (Figura 4). Por motivos ainda desconhecidos, o tipo VIM-2 tornou-se dominante e já foi identificado em pelo menos 32 países, incluindo Portugal (Hawkey & Jones 2009; Cardoso et al. 2002), sendo o mais prevalente da família, e dos mais disseminados entre todas as MBLs adquiridas, sendo que a larga maioria dos casos ocorre em P. aeruginosa (Walsh 2010).
1.3.2.MECANISMOS DE RESISTÊNCIA EM Pseudomonas aeruginosa
P. aeruginosa encerra uma grande variedade de estratégias de resistência a
antibióticos, tanto intrínsecas como adquiridas.
No primeiro grupo, é incluída a formação de biofilmes. Este tipo de organização celular é complexo, mas a baixa taxa metabólica e crescimento lento das células no interior da estrutura de biofilme podem explicar a maior tolerância a antibióticos, já que estes compostos têm maioritariamente processos metabólicos como alvo (Poole 2011; Balasubramanian et al. 2013).
A β-lactamase cromossomal AmpC é intrínseca a esta espécie e confere resistência a alguns β-lactâmicos, apesar de o seu espectro de acção ser alargado devido à acumulação com outras estratégias de resistência e/ou à desrepressão do gene ampC, o mecanismo de resistência a β-lactâmicos mais comum em P. aeruginosa (Poole 2011).
25
β-lactamases adquiridas são também encontradas nesta espécie. Pertencentes a diferentes famílias proteicas, representam um dos mediadores de resistência mais importantes não só nesta espécie, mas em todos os organismos de Gram negativo (Worthington & Melander 2013). Estas enzimas possuem diferentes espectros de acção e, cumulativamente, conferem resistência a todos os antibióticos β-lactâmicos utilizados (Bush 2010); β-lactamases pertencentes a todas as quatro classes de Ambler já foram identificadas em P. aeruginosa (Poole 2011).
Além de β-lactamases, este microrganismo recorre à acção de outras enzimas para sobreviver ao ataque de antibióticos. Transferases são importantes na resistência a aminoglicosídeos, já que catalisam a acetilação, adenilação ou fosforilação do antibiótico, inactivando-o. Metilases também são relevantes na resistência a estes agentes farmacológicos, pois transformam a sub-unidade ribossomal 30S e impedem a ligação de compostos clinicamente relevantes, como a gentamicina, tobramicina e amicacina (Poole 2011; Fair & Tor 2014).
Na resistência a aminoglicosídeos e a outras classes de antibióticos, sistemas de efluxo assumem também um papel importante. Até à data, foram descritas 5 famílias de sistemas de efluxo envolvidos em resistência bacteriana a antimicrobianos, no entanto a família RND parece ser a maior contribuidora para a resistência em P. aeruginosa, tendo sido identificados 12 sistemas dessa família neste organismo (Poole 2011). Embora alguns sejam específicos para um tipo de composto, muitos têm a capacidade de expulsar moléculas diferentes entre si, contribuindo para multirresistência (Dzidic
et al. 2008). Tal abrangência pode ser observada em três dos sistemas que conferem
resistência a fluorquinolonas, MexAB-OprM, MexCD-OprJ, e MexXY-OprM, que albergam igualmente β-lactâmicos, e o último listado está também associado a resistência a aminoglicosídeos (Poole 2011). Estes sistemas intrínsecos a várias espécies podem sofrer um incremento da sua expressão devido a mutações nas zonas de regulação, que conduzem a um aumento dos níveis de resistência (Dzidic et al. 2008).
A permeabilidade selectiva da membrana celular possui um papel central na resistência ou tolerância a compostos prejudiciais à célula, não só na sua expulsão,