Pró-reitora Acadêmica
Escola de Saúde
Programa de Pós-Graduação Stricto Sensu em Ciências
Genômicas e Biotecnologia
PAPEL IMUNITÁRIO DA AUTOFAGIA DE MACRÓFAGOS
CONTRA PARACOCCIDIOIDES SPP.
Brasília - DF
2015
KELLYANNE TEIXEIRA RANGEL
PAPEL IMUNITÁRIO DA AUTOFAGIA DE
MACRÓFAGOS CONTRA
PARACOCCIDIOIDES
SPP
.
Brasília/2015
Dissertação apresentada ao Programa de Pós-Graduação Stricto Sensu em Ciências Genômicas e Biotecnologia da Universidade Católica de Brasília, como requisito parcial para a obtenção do título de mestre em Ciências Genômicas e Biotecnologia.
Orientador: Prof. Dr. André Moraes Nicola
R 196p Rangel, Kellyanne Teixeira.
Papel imunitário da autofagia de macrófagos contra Paracoccidioides
spp. f Kellyanne Tei.'l:eira Rangel - 2015. 105 f.; i!.: 30 em
Dissertação (Mestrado) - Universidade Católica de Brasilia, 2015. Orientação: Prof. Dr. André Moraes Nico1a
Coorientaçào: Profa Dra. Maria Sueli Soares Felipe
1. Biotecnologia. 2. Paracoccidioidomicose. 3. P. brasiliensis. 4. Autofagia. 5. Lentivírus. I. Nico1a André l'vloraes, orient. 11. Felipe, l\•laria
Sueli Soares, coorient. In . Título.
CDU606
ll(
.· .•
セ@
Católica de Brasília
Dissettação de autoria de Kellyanne Teixeira Rangel, intitulada "Papel imunitário da autofagia de macrófagos contra Paracoccidioides spp", apresentada como requisito parcial para obtenção de grau de Mestre em Ciências Genômicas e Biotecnologia da Universidade Católica de Brasília, em 31 de Março de 2015, defendida e aprovada pela banca examinadora abaixo assinada:
Prof. Dr. André Moraes Nicola Orientador
(Programa de Pós Graduação Stricto Sensu em Ciências Genômicas e Biotecnologia-UCB)
Prof. Dr. Maria Sueli Soares Felipe Coorientadora
(Programa de Pós Graduação Stricto Sensu em Ciências Genômicas e Biotecnologia -UCB)
_ _.-/
-O 。カッセNM
cNNNvmoviセセ@
lb-
I?J<---tセ
M
Profa. Dra. Taia Maria Betto Rezende Membro Intema
(Programa de Pós Graduação Stricto Sensu em Ciências Genômicas e Biotecnologia-UCB)
Profa bra. Patrícia Albuquerque de Andrade Membro Extemo
(Universidade de Brasília- UnB)
'7 /}
\ . ,;,._l_fl
I ( V v-1 ·\A.L._ 1
Profa. 9 ra. Robe11 Edward Pogue Suplente
(Programa de Pós Graduação Stricto Sensu em Ciências Genômicas e Biotecnologia -UCB)
AGRADECIMENTO
Agradeço a Deus pela vida e pelo amadurecimento que Ele me permitiu vivenciar na minha vida nesse tempo de estudo e pesquisa.
Agradeço a minha família pelo apoio e compreensão.
Agradeço também por grandes amigos que me ajudaram a insistir em momentos difíceis, pelos conselhos e companhias. Amigos que se tornaram grandes irmãos, entre eles Felipe Daniel Horovits, que com grande paciência e sabedoria esteve ao meu lado.
RESUMO
RANGEL, Kellyanne Teixeira. Papel imunitário da autofagia de macrófagos
contra Paracoccidioides spp. 2015. Mestrado em Ciências Genômicas e
Biotecnologia – Universidade Católica de Brasília, 2015.
ABSTRACT
Paracoccidiodomycosis is a systemic disease caused by fungi’s two species of the genus Paracoccidioides, P. brasiliensis and P. lutzii. These fungi present thermal dimorphism, with characteristic mycelium to 22°C - 25°C temperature and 35°C - 37°C with yeast morphology. The human body responds to infection typically with phagocytosis mechanism and cellular immunity. A cell type that plays a key role in innate and acquired immunity is a monocyte enters tissue when it becomes macrophage. The main function of the macrophages is the phagocytosis of microorganisms. Another deterioration mechanism is autophagy, which originally was related to the recycling of damaged intracellular organelles also can act to combat pathogens. The aim of this work was to observe the cell efficiency to combat fungal infection by the formation of autofagossomo in macrophages infected with Paracoccidioides spp. The vector chosen in this work was a lentiviral vector to be able to insert the gene of interest into the host genome. The results demonstrate that in vitro infection experiments, fungi internalized by macrophages induce recruitment of LC3, an autophagosomes marker. Statistical analysis by ANOVA showed no statistically significant difference between the macrophages that received transduction and which on did not. In conclusion, it is observed that the immune action in autophagy has Paracoccidoides infection spp. Further, studies more detailed may reveal in more detail the importance of autophagy in PCM considering studies have proved the action of autophagy in bacterial infections and other fungal pathogens.
LISTA DE ILUSTRAÇÕES
Figura 01 – Fase saprobiótica e parasitária de P. brasiliensis (Bagagli, 2008)
... Página 18
Figura 02 – Morfologia do gênero Paracoccidioides (Franco, 1989)
... Página 19
Figura 03 – Distribuição geográfica de diferentes subespécies filogenéticas de
Paracoccidioides brasiliensis, de acordo com a informação atual disponível (Pereira, 2009) ... Página 20
Figura 04 – Formas clínicas da paracoccidioidomicose (PCM)
(Shikanai-Yassuda, 2006) ... Página 23
Figura 05– Exemplo do papel da autofagia na imunidade inata (Mintern, 2012)
... Página 28
Figura 06 – Papel da autofagia na imunidade adaptativa (Mintern, 2012)
... Página 29
Figura 07 – Formação do Autofagossomo - Proteínas Atg (Geng, 2008)
... Página 32
Figura 08 – Via de sinalização e regulação da autofagia (cellsignal, 2014)
... Página 33
Figura 09 – Resultado da purificação dos plasmídeos de empacotamento
lentiviral e shRNA... Página 44
Figura 10 – Imagem de sobreposição dos histogramas de distribuição de
tamanho... Página 46
Figura 11 – Controle negativo da quantificação lentiviral no qNano
... Página 47
Figura 12 – Resultado do knockdown da autofagia em células J774
Figura 13 – Resultado do qPCR. Expressão absoluta do gene em unidades arbitrárias (UA) ... Página 49
Figura 14 – Teste de atividade antifúngica de macrófagos infectados com P.
brasiliensis ... Página 50
Figura 15 - Imunolocalização de LC3 (verde) em células RAW264.7 infectadas
com Pb01 e Pb265 ... Página 51
Figura 16– Imunolocalização de LC3 (verde) em células RAW264.7 infectadas
LISTA DE TABELAS
Tabela 01 - Tabela 01 - Quantificação das partículas lentivirais para ATG5 de
LISTA DE ANEXOS
Anexo A: TAT ... Página 63
Anexo B: REV ... Página 64
Anexo C: GAG-POL ... Página 65
Anexo D: VSV-G ... Página 66
LISTA DE ABREVIATURAS
AMPK – Activated protein kinase α (proteína quinase α ativada)
ATG– AuTophaGy (autofagia – nomenclatura do gene) BHI – Brain Heart Infusion (infusão cérebro-coração)
CF488– Chromeo fluorescente 488 (cromóforo fluorescente)
cDNA– Acido desoxirribonucleico complementar DNA– Ácido Desoxirribonucleico
DMEM–Dulbecco’s Modified Eagle’s Medium
EGFP – Enhanced Green Fluorescent Protein (proteína verde fluorescente aprimorada)
HEK – Human Embryonic Kidney
HIV-1– Vírus da Imunodeficiência Humana
GFP-LC3– Proteína verde fluorescente conjugada com proteína cadeia leve 3.
Gp– glicoproteína
IFN-y– Interferon gama
IgG– Imunoglobulina G IL-2– Interleucina 2 LB– Luria-Bertani
LC3– Light Chain 3 (cadeia leve 3)
MHC – Major Histocompatibility Complex (complexo principal de histocompatibilidade)
NDP52– protein of nuclear domain 52 (proteína do domínio nuclear 52)
p62– Protein 62 (proteína 62)
PBS– Phosphate buffered Saline (tampão fosfato-salino) PCM - Paracoccidioidomicose
PE– phosphatidylethanolamine (fosfatidil etanolamina) RAW– Mouse Leukaemic Monocyte
RNA– Ribonucleic acid (ácido ribonucleico)
RNAi – Ácido Ribonucleico interferente
ROS – Reactive Oxygen Species (espécies reativas de oxigênio)
RT-PCR– Reação em Cadeia da Polimerase em Tempo Real
SBF– Fetal Bovine Serum (soro bovino fetal)
shRNA– pequeno grampo de ácido ribonucleico (small hairpin)
TAE– Tris-Acetato-EDTA
Th1 e Th2– T helper type I and II (T ajudador tipo I e II)
THP-1– linhagem celular de leucemia monocitica humana
TNF– fator de necrose tumora
TLR– Toll-Like Receptors (receptores Toll-like) TOR– Target o rapamycin (marcador de rapamicina)
Unidades de medidas
SUMÁRIO
1. INTRODUÇÃO ... 16
2. REVISÃO BIBLIOGRÁFICA ... 17
2.1. PARACOCCIDIOIDOMICOSE ... 17
2.2. DESENVOLVIMENTO DA DOENÇA ... 21
2.2.1. Aspectos clínicos e laboratoriais da PCM ... 22
2.2.2. Interação patógeno – hospedeiro ... 23
2.3. IMUNIDADE DOS MACRÓFAGOS ... 24
2.3.1. Papel imunológico da autofagia dos macrófagos ... 25
2.3.2. Autofagia e imunidade inata ... 26
2.4. AUTOFAGIA E IMUNIDADE ADAPTATIVA ... 28
2.5. FORMAÇÃO DO AUTOFAGOSSOMO ... 29
2.6. AUTOFAGIA E INFECÇÃO FÚNGICA ... 30
3. JUSTIFICATIVA ... 34
4. OBJETIVOS ... 35
4.1. OBJETIVO GERAL ... 35
4.2. OBJETIVOS ESPECÍFICOS ... 35
5. METODOLOGIA ... 36
5.1. CULTURA CELULAR ... 36
5.2. ENSAIO DE TRANSFECÇÃO COM A LINHAGEM CELULAR HEK 293T 37 5.2.1. Preparação plasmidial ... 37
5.2.2. Ensaio De Transfecção ... 38
5.3. CONTAGEM E CONCENTRAÇÃO DAS PARTICULAS LENTIVIRAIS UTILIZANDO RESISTENCIA DE PULSO ... 39
5.4. ENSAIO DE TRANSDUÇÃO DE CÉLULAS J774 COM LENTIVIRUS 39 5.5. KNOCKDOWN DA AUTOFAGIA EM CÉLULAS J774 – TRANSDUÇÃO, EXTRAÇÃO DE RNA E PCR ... 40
5.6. CULTURA FUNGICA ... 41
5.7. EXPERIMENTO DE CONTAGEM DE UNIDADES FORMADORAS DE COLONIAS –CFU’s ... 41
5.8. ENSAIO DE INFECÇÃO IN VITRO DE CÉLULAS RAW 264.7 COM Pb 42 5.9. IMUNOLOCALIZAÇÃO DE LC3 ... 42
6.1. FORMAÇÃO DA PARTICULA LENTIVIRAL ... 44
6.2. EXTRAÇÃO DE RNA ... 47
6.3. ENSAIO DE KNOCKDOWN ... 48
6.4. CONTAGEM DE CFU ... 49
6.5. ENSAIO DE IMUNOLOCALIZAÇÃO ... 50
7. DISCUSSÃO ... 52
8. CONCLUSÕES E PERSPECTIVAS... 57
9. REFERENCIAS ... 58
10. ANEXOS ... 63
INTRODUÇÃO
Paracoccidioidomicose – PCM – é a micose sistêmica de maior prevalência na américa latina (TRABULSI, 2008). O microrganismo patogênico causador da doença pertence ao gênero Paracoccidioides spp (P. brasiliensis e P. lutzii), caracterizado por ser dimórfico e saprobiótico. A infecção ocorre pela inalação dos conídios, caracterizando maior risco de aquisição da doença para profissionais com atividades relacionadas ao manuseio do solo, como trabalhadores agrícolas (SAN-BLAS, 2010).
O organismo humano geralmente responde à infecção com mecanismo de fagocitose e com a imunidade celular (YONA, 2009). A fagocitose é um mecanismo celular para degradar microrganismos que estão localizados externamente à célula. Autofagia, outro mecanismo de degradação celular também conhecido como macroautofagia, é uma via que realiza, entre outras ações, a reciclagem de material citoplasmático degradando agregados de proteínas e organelas defeituosas. Além dessa função, a autofagia apresenta papel importante na ação imunológica inata e adaptativa, eliminando microrganismos que invadem compartimentos intracelulares, como o Paracoccidioides brasliensis (KLIONSKY, 2005).
17
REVISÃO BIBLIOGRÁFICA
2.1. PARACOCCIDIOIDOMICOSE
Também conhecida como blastomicose sul-americana ou micose de Lutz-Splendore-Almeida, é uma doença sistêmica descrita pela primeira vez por Adolfo Lutz em 1908, que pode apresentar forma tegumentar, linfática, visceral ou mista (TRABULSI, 2008).
A incidência anual da doença no Brasil é de 10-30 infecções por milhão de habitantes, com taxa de mortalidade média de 1,4 por milhão de habitantes por ano, sendo assim caracterizada como maior causa de mortalidade entre as micoses sistêmicas (TEIXEIRA, 2014).
PCM pode ser considerada uma doença negligenciada, que segundo a organização mundial da saúde, são doenças concentradas em nações de baixa renda na África, Ásia e na América Latina, causadas por parasitas que são levados por insetos ou por água e solo contaminado. Porém, no Brasil não existem programas governamentais para PCM (GEGEMBAUER, 2014).
Não é uma doença de notificação compulsória e por isso existe uma dificuldade de se obter dados específicos sobre sua incidência na população. Possui característica endêmica de zona rural, com manifestações clínicas presentes principalmente em homens de 30 a 50 anos. Além de ser encontrado no solo, Paracoccidioides spp já foi isolado de morcegos, fezes de pinguim e comida de cachorro. Entretanto, esses achados foram casuais e sem reprodutibilidade. O achado mais confiável do fungo em animal selvagem foi em ninhos de tatu-galinha (Dasy novemcinctus), tanto em suas tocas quanto no próprio animal (PEREIRA, 2009).
Figura 01 – Fase saprobiótica e parasitária de P. brasiliensis. (A) Reprodução sexual e seleção natural exercida por microrganismos fagocíticos: aumento da frequência de variações nos traços de virulência. (B) aparecimento de novas variações de patógenos. (C) Morte do hospedeiro e retorno do P. brasiliensis para o meio saprobiótico.
Fonte: BAGAGLI, 2008
P. brasiliensis é um fungo que apresenta dimorfismo térmico, com característica micelar à temperatura 22ºC-25ºC, e à 35ºC-37ºC com morfologia leveduriforme. Outros fatores, como alterações nutricionais, podem também interferir na morfologia do fungo, levando-o a permanecer com forma de levedura à 25ºC (SANTOS ET AL, 2011). Os conídios são as partículas infectantes presentes no solo. Depois de inalados, os conídios passam para a forma de leveduras, desencadeando as manifestações clínicas características da doença
(SAN-BLAS, 2010).
19
informalmente chamados de “roda de leme de navio” e “Mickey Mouse” (PALMEIRO, 2005).
Figura 02– Morfologia do gênero Paracoccidioides. (A) Forma leveduriforme de
Paracoccidioides brasiliensis. Brotamento com característica de “roda de leme”. Fonte: Fortes, 2011. (B) Forma miceliana. Esporulação de um isolado de Pb18 com clamidósporo terminal em (C) e intercalado em (B).
Fonte: FRANCO, 1989.
O fungo, sob sua forma filamentosa e ao ser observado com auxílio de microscópio, apresenta hifas finas e septadas, com clamidósporos terminais e intercalados. Pode produzir artroconídios se cultivado em meio com pouca oferta de carboidrato. Entre as características macroscópicas da forma filamentosa está o aspecto algodonoso da colônia (SIDRIM, 2010).
formas. Quando há a mudança de forma micelar para leveduriforme e vice-versa, acontece, entre outros eventos, o envolvimento da enzima -glicana sintetase para que haja o predomínio da produção de α-glicana ou -glicana (MARQUES, 2007)
O gênero Paracoccidioides tem sido classificado na família Ajellomycetaceae. Nesse grupo também se encontram os gêneros Histoplasma e Blastomyces, dois outros fungos dimórfico causadores de infecções humanas. Por meio de diferentes estudos, o Paracocidioides brasiliensis revelou subespécies filogeneticamente diferentes também denominadas espécies ocultas. Elas são geograficamente diferenciadas (figura 03): S1 (espécie 1 do Brasil, Argentina, Paraguai, Peru e Venezuela), PS2 (espécie filogenética 2 do Brasil e Venezuela) e PS3 (espécie filogenética da Colômbia). Essas espécies são originadas de polimorfismo originada de eventos de especiação e recombinação genética (CARRERO, 2008)
Figura 03: Distribuição geográfica de diferentes subespécies filogenéticas de P.
brasiliensis, de acordo com a informação atual disponível. PS2 - Espécie filogenética 02 do Brasil e Venezuela; PS3 - Espécie filogenética da Colômbia; S1 – Espécie filogenética 01 do Brasil, Argentina, Paraguai, Peru e Venezuela.
21
2.2. DESENVOLVIMENTO DA DOENÇA
A infecção pelo fungo ocorre pela inspiração de conídios que estão presentes no solo. A ocorrência da infecção sem o desenvolvimento da doença é chamada de paracoccidioidomicose infecção. A identificação do estágio primário de infecção é rara por causa da frequente ausência de sintomas nessa etapa. Uma hipótese desenvolvida para justificar a ausência de sintomas com um resultado positivo em testes imunológicos é a formação de um complexo primário fúngico que retrocede, deixando uma hipersensibilidade retardada e anticorpos que indicam a ocorrência da infecção (KUROKAWA, 2005).
Esse complexo primário é formado geralmente no pulmão, começando com um processo inflamatório inespecífico decorrente da inalação de conídios que, depois de atingirem os alvéolos, se transformam em leveduras que são fagocitadas e transportadas para gânglios linfáticos do hilo pulmonar, criando o complexo primário. Esse complexo pode ser combatido com eficácia pelo sistema imunológico, destruindo o fungo. Caso o sistema imunológico esteja comprometido, o microrganismo se proliferará com maior facilidade pelo organismo a partir de linfonodos mediastinais para o restante do corpo, caracterizando a paracoccidioidomicose doença (SIDRIM, 2010).
O aparecimento de sintomas pode ser consequência ou da reativação de uma infecção que estava latente ou de uma infecção primária. O fungo pode se disseminar pelo organismo humano pela circulação sanguínea, pela circulação linfática ou pela via canalicular. Em pacientes imunossuprimidos, a disseminação sanguínea é mais frequente, levando ao surgimento de lesões no fígado, baço e medula óssea. Lesões na boca, orofaringe, mucosa nasal, intestino e na região anorretal são sugestivas de disseminação por via linfática. A via canalicular é representada por lesões pulmonares com secreções contaminadas conduzidas pelos brônquios (SIDRIM, 2010).
um diagnóstico indireto pode ser realizado pela titulação sorológica de anti-gp43 (GEGEMBAUER, 2014).
A glicoproteína gp43 é usada no diagnóstico sorológico da PCM, por representar o antígeno imunodominante de P. brasiliensis. O antígeno gp43 favorece a adesão, colonização e propagação do patógeno ao agir como um receptor de laminina. Pacientes com as formas severas da PCM apresentam altos níveis de anticorpos anti-gp43, sendo que essa alta titulação diminui com o curso do tratamento da doença, na maioria dos casos. A espécie P. lutzii apresenta baixa expressão de gp43, interferindo na realização do teste sorológico tradicional, podendo apresentar falsos negativos (GEGEMBAUER, 2014).
2.2.1. Aspectos clínicos e laboratoriais da PCM
A paracoccidioidomicose apresenta as formas clínicas: aguda, subaguda e crônica. As formas aguda e subaguda estão associadas a pacientes mais jovens, com história clínica mais curta, em média de dois meses, sendo responsável por 20 a 25% dos casos. A forma crônica com duração clínica de, em média, seis meses, representa 75% dos casos. As lesões pulmonares, de pele e mucosas caracterizam esta forma clínica, que afeta mais trabalhadores rurais homens e adultos, com idade média de 30 anos (SHIKANAI-YASUDA, 2006).
Figura 04: Formas clínicas da paracoccidioidomicose (PCM). Forma
23
borboleta”, opacidade nodulares e micronodulares difusa; (E) Acometimento peri-oral e mentoniano.
Fonte: SHIKANAI-YASSUDA, 2006
A forma aguda / subaguda é caracterizada por linfadenomegalia, manifestações digestivas, hepatoesplenomegalia, envolvimento ósteo-articular e lesões cutâneas. A forma crônica é caracterizada por fraqueza, emagrecimento, febre, tosse e dispneia (WANKE, 2009).
Os aspectos laboratoriais da doença incluem alterações nas funções hepáticas, nos níveis de uréia, creatinina, sódio e potássio. O padrão ouro para diagnóstico da PCM é encontrar o fungo nas amostras biológicas dos pacientes.
2.2.2. Interação patógeno – hospedeiro
intensidade da doença. Mulheres parecem ter a paracoccidioidomicose-infecção limitada pela ação de estrógenos. Pacientes mais jovens apresentam infecção mais generalizada, enquanto pacientes adulto possuem infecção mais restrita à porta de entrada do fungo, sendo a mais comum a entrada por via respiratória (SIDRIM, 2010).
O fungo aumenta sua capacidade de resistência à resposta imunológica quando a quantidade de microrganismo inalado é elevada, além da existência de fatores de virulência relacionada com a quantidade de α-glucana na parede da levedura, que dificulta a fagocitose, e com a interação do fungo com a matriz extracelular pela ligação de gp 43 e de gp 190 à laminina da membrana basal (FORTES, 2011).
Na fase inicial da doença, as células NK (natural killer) possuem um papel importante de inibir a ação das leveduras, porém sua atividade citotóxica diminue quando a PCM já está instalada (Peraçoli, 1995). Macrófagos também atuam contra a infecção, porém em alguns casos eles podem permitir a sobrevivência intracitoplasmática de leveduras fagocitadas, que também são destruídas na presença interferon-y (MONTEIRO, 2005).
Estudos indicam que a resposta celular Th-2 e Th-1 tem atividade na resistência e susceptibilidade fúngica, respectivamente. Pacientes que possuem a forma adulta da doença demonstram uma resposta imunológica mista, tanto com produção de citocinas da resposta Th-1 como IFN , TNF-α e IL-2, além de produção variável de citocinas relacionadas a resposta Th-2. Pacientes com a forma juvenil geralmente apresentam uma resposta Th-2 predominante, com produção de IL-4, IL-5 e IL-10. Novas populações de células T CD4 também estão envolvidas na resposta à infecção fúngica, como Th-7, Th-22 e Th-9, estimulando células inflamatórias a auxiliar no controle da infecção fúngica (CASTRO, 2013).
2.3. IMUNIDADE DOS MACRÓFAGOS
25
desenvolvimento de abundante citoplasma, sendo assim chamado de células epitelióides por se assemelharem às células epiteliais da pele é um exemplo. Outros podem se fundir e formar células multinucleadas gigantes. Macrófagos localizados no sistema nervoso central são chamados de micróglias. Células de Küpffer é o nome dado aos macrófagos localizados nos sinusoides vasculares do fígado, e osteoclastos são os macrófagos localizados no osso (ABBAS, 2008). A principal função dos macrófagos é a de fagocitose de microrganismos. Esse é um processo que engloba partículas maiores que 5µm de diâmetro para destruição em lisossomos que se fundirão com as vesículas (fagossomo) que contém o microrganismo fagocitado, resultando em um fagolisossomo. Existem receptores na membrana do macrófago que fazem com que essa célula fagocite apenas os microrganismos estranhos, e não outras moléculas ou células próprias do organismo. Um tipo de sinalização da fagocitose, chamada de opsonina, inclui anticorpos, proteínas do complemento e lectinas, e realiza a opsonização, um processo no qual o microrganismo é envolvido por essas proteínas para torná-lo alvo da fagocitose (SHALHOUB, 2011).
Outro mecanismo de degradação é a autofagia, que se diferencia da fagocitose por ser direcionado para degradação de moléculas e microrganismos localizados na parte interna da célula (KLIONSKY, 2005).
2.3.1. Papel imunológico da autofagia dos macrófagos
2.3.2. Autofagia e imunidade inata
Além da função de reciclagem de organelas intracelulares danificadas, a autofagia também é considerada um mecanismo eficiente para combater patógenos intracelulares, como Mycobacterium tuberculosis e Samonella spp. A xenofagia, nome que pode ser dado à autofagia quando realiza papel imunológico, é induzida por uma cascata de sinalização com receptores de “perigo” incluindo o TLR (Toll like receptors), que elimina bactérias que tem acesso ao citosol, como Streptococcus pyogenes, bactérias que estão presentes em vacúolos, como M. tuberculosis var bovis bacillus Calmette-Guerin (BCG), e patógenos virais, como o vírus Sindbis (LEVINE, 2005).
Um exemplo da ação imunológica da autofagia acontece quando o patógeno, por exemplo, uma bactéria, está dentro de um vacúolo e este é danificado, e os resíduos de açúcar da estrutura bacteriana são expostas, atraindo receptores de galectina 8 e recrutando a maquinaria autofágica (KLIONSKY, 2005).
Existem três proteínas que iniciam a autofagia seletiva para bactérias, sendo eles NDP52, p62 e optineurina. Elas vão detectar proteínas bacterianas poliubiquitinadas. Um desses sinais é a ligação da NDP52 à galectina-8, que funciona como um sinal de “coma-me” ubiquitina-independente que sinaliza vesículas danificadas por bactérias invasoras de citosol (S. LI, 2013).
27
liga à proteína NDP52 e limita a proliferação bacteriana por recrutar ao local a maquinaria autofágica (S. LI, 2013).
A autofagia pode prejudicar a replicação viral, “sequestrando” os produtos de replicação viral para compartimentos de endolisossomos onde eles são detectados por TLR e IFN tipo 1. Vários vírus desenvolveram mecanismos para desabilitar a autofagia, codificando proteínas que bloqueiam a iniciação da autofagia pela interferência da atividade de beclin 1 (MINTERN, 2012).
Outra ação da autofagia pode ser encontrada no controle de citocinas inflamatórias. Quando organelas, como a mitocôndria, ficam danificadas, citocinas inflamatórias agem na sua remoção. O acúmulo de mitocôndrias danificadas pode levar ao extravasamento de DNA mitocondrial para o citosol e aumentar as espécies reativas de oxigênio (ROS). Na figura 05 é possível observar uma das diferentes formas de controle que a autofagia exerce na cascata de citocina pró-inflamatórias. A influência da autofagia na secreção de IL-1 é a mais bem descritas. Alguns estudos realizados por NAKAHIRA 2011 e Crisan 2011 demonstraram que na ausência de autofagia, a produção de IL-1 e IL-18 são aumentadas. Em células humanas, essa resposta depende da fosforilação de proteínas quinase ativadas por mitógeno (MAPK).
Figura 05 – Exemplo do papel da autofagia na imunidade inata. (A)
Fonte: MINTERN, 2012.
2.4. AUTOFAGIA E IMUNIDADE ADAPTATIVA
A autofagia pode participar na formação de pool de peptídeos exibidos pelas moléculas de MHCII, durante seu papel no tráfico citosólico de proteínas para os lisossomos, o mesmo compartimento onde ocorre a biossíntese e carregamento de moléculas de MHCII.
29
Figura 06 – Papel da autofagia na imunidade adaptativa. (A) Antígenos
citosólicos são encaminhados para o compartimento de carregamento MHCII nas células apresentadoras de antígeno também pela autofagia, se caracterizando uma importante via de apresentação de antígeno para uma célula T. (B) As proteínas na maquinaria autofágica contribuem também para a fagocitose de antígeno extracelular. (C) A sobrevivência celular é melhorada com a reciclagem de organelas danificadas realizada pelo processo de autofagia.
Fonte: MINTERN, 2012.
2.5. FORMAÇÃO DO AUTOFAGOSSOMO
transdução de sinais (MINTERN, 2012). Os genes ATG (AuTophaGy) são os responsáveis pela tradução das proteínas envolvidas na formação do autofagossomo, sendo uma média de 18 ATG’s que compõe o núcleo da maquinaria autofágica.
Existem três estágios para a formação completa de um autofagossomo: a iniciação, a nucleação e a expansão. Todos os estágios são coordenados por proteínas ATG. Na iniciação ocorre a transmissão de sinais para direcionar a origem de formação da membrana por meio do recrutamento de complexos proteicos com funções essenciais. Para que ocorra a iniciação, vários complexos proteicos atuam juntos em uma cascata de sinalização como demonstrado na figura 08, regulando a magnitude da autofagia por vias de sinalização que “sentem” o estado nutricional da célula, como o complexo proteico ULK (LAMB, 2013).
A nucleação é caracterizada pela formação da membrana do autofagossomo, após a identificação do local de sua formação na etapa de iniciação. A expansão é o elongamento da membrana iniciada na nucleação até a formação completa do autofagossomo, com o auxílio do complexo ATG16L1 (ATG12-ATG5-ATG16L1) que é recrutado logo após a nucleação (SHPILKA, 2011). Antes do fechamento da membrana, criando um autofagossomo completo, as proteínas ATG’s que estão ligadas à membrana se dissociam, como pode ser visto na figura 07, mas a proteína LC3 e sua família continua ligada aonde vai se tornar a superfície interna do autofagossomo (LAMB, 2013).
2.6. AUTOFAGIA E INFECÇÃO FÚNGICA
Criptococcus neoformans (Cn) é um exemplo de patógeno fúngico que conhecidamente se replica dentro de um fagolisossomo, em um macrófago do hospedeiro infectado. Causador da meningoencefalite e criptococcose, o Cn é um fungo oportunista que pode apresentar em uma forma encapsulada, sendo o solo seu habitat natural (BLANCO, 2008).
31
autofágico começa seu envolvimento na infecção a partir do momento no qual proteínas responsáveis pela formação do autofagossomo, como Atg5, Atg9 e LC3, são recrutadas para o local onde o fungo se encontra intracitoplasmaticamente (BOTTS, 2010).
A influência da autofagia na infecção do Cn ainda é controversa. Segundo Quin, quando a atividade autofágica diminui em uma célula infectada por Cn, a infecção por Cn diminui. A ideia exposta por esse autor é que o Cn usa as proteínas do hospedeiro para facilitar sua replicação (QUIN, 2011).
Já o trabalho realizado por Nicola et al, 2012 mostra que a autofagia possui atividade imunológica contra a infecção por Criptococcus neoformans e Cândida albicans, no qual, no interior de autofagossomo foram encontrados peptídeos antifúngicos relacionados à digestão intracelular de Cn opsonizado por anticorpo. Este estudo mostrou que in vivo a ausência da autofagia fez com que a média vida de ratos infectados fosse menor que o controle, revelando a capacidade de contenção da infecção que a autofagia possui (NICOLA, 2012).
Apesar do estudo realizado in vivo por Nicola ter demostrado uma queda da sobrevida em ratos com knockdonw de autofagia com relação aos ratos controle, o estudo também mostrou que uma via alternativa de combate a infecção foi organizada pelo macrófago na ausência da autofagia. Isso comprova a complexidade do papel que autofagia desempenha na imunidade do macrófago.
Figura 07 – Formação do Autofagossomo (Proteínas Atg). Sistema de
cliva o Atg8 que está presente na superfície externa, retirando-o do autofagossomo formado.
Fonte: GENG, 2008.
Figura 08 – Via de sinalização e regulação da autofagia. Descrição da via: A
33
fusiona a um lisossomo, formando um autofagolisossomo, para proporcionar a degradação do conteúdo do autofagossomo.
JUSTIFICATIVA
35
OBJETIVOS
4.1. OBJETIVO GERAL
A pesquisa realizada nesse projeto teve como objetivo determinar o papel da atividade autofágica de macrófagos humanos e de camundongos na imunidade a Paracoccidioides spp.
4.2. OBJETIVOS ESPECÍFICOS
Observar se ocorre a formação de autofagossomos ao redor do fungo em macrófagos infectados com P. brasiliensis.
Alterar a expressão das proteínas conhecidamente envolvidas na formação do autofagossomo por meio de transdução de shRNAs em vetores lentivirais.
METODOLOGIA
5.1. CULTURA CELULAR
As células J774.16 e HEK 293T, cedidas pelo Doutor André Nicola, foram usadas para a os ensaios de transdução e transfecção. A célula J774.16 é uma linhagem de macrófago de camundongo que foi destinado nesse trabalho para uso no ensaio de transdução. A célula HEK 293t é uma linhagem de macrófago humano proveniente de rim embrionário, sendo destinado para o ensaio de transfecção.
As células foram mantidas, em placas de petri, com meio DMEM
(Dulbecocco’s Modified Eagle’s Medium – Sigma, com 4500 mg/L de glicose,
L-glutamina, sem bicarbonato de sódio, lote #SLBH2326V), meio NCTC-109 (Gibco, sem L-glutamina, na concentração de 1X, com volume total do recipiente de 500ml, Lote 1446526, importado e distribuído pela Life Technologies do Brasil Com. Prod. Biotec. LTDA), e ANE (Non-essential Amino Acid Solution na concentração de 100x,, filtrado esterilmente, testado para endotoxina, da empresa Sigma-Aldrich Life Science, lote RNBD0814). Uma terceira suplementação realizada com Soro Bovino Fetal (SBF) (Gibco, originado na América do Sul, distribuído pela empresa Life Technologies do Brasil Com. Prod. Biotec. LTDA). A cultura celular foi mantida em uma incubadora a 37 °C com fornecimento de 5 % de CO2.
37
5.2. ENSAIO DE TRANSFECÇÃO COM A LINHAGEM CELULAR HEK 293T
5.2.1. Preparação plasmidial
Para a realização do ensaio de transfecção, foram utilizados os plasmídeos de empacotamento: TAT, com 5706 pares de base; REV, com 4176 pares de base; GAG-POL, com tamanho desconhecido e VSV-G, com 6363 pares base; que foram originados de retrovírus (Vírus da Imunodeficiência humana 1 - HIV-1) e do vírus da estomatite vesicular humano (VSV). Todos de expressão em mamíferos e com marcador seletivo para ampicilina, no caso de multiplicação em bactérias. O plasmídeo pLK0.1, com 8901 pares de base, foi usado para determinar o tipo de RNA interferente que seria transcrito. O alvo foi o gene ATG5 de camundongo. O foco do experimento foi reduzido ao gene ATG5 de camundongo.
5.2.2. ENSAIO DE TRANSFECÇÃO
As células HEK 293T foram cultivadas até que atingissem a confluência de 90-95% da placa de petri. Após esse processo, a placa foi tripsinizada e contada de forma que se obtivesse 3,75x105 células.ml-1, ressuspendida em DMEM +
10% de SBF. Uma suspensão celular com 15 ml de DMEM + 10 % de SBF foi feita de forma que cada poço, de uma placa de seis poços, recebesse 2 ml da suspensão. Depois, 6 eppendorfs foram separados, sendo que em cada um foram adicionados 256 µl do mix de OptiMEM e lipofectamina, com o valor em µl correspondente a cada vetor pLK0.1 respectivo e 2 µl do meio de empacotamento, composto pelos plasmídeos TAT, REV, VSV-G e GAG-POL. O mix de meio de empacotamento foi feito para um volume final de 20 µl, para que cada poço recebesse 2 µl.
39
5.3. CONTAGEM E CONCENTRAÇÃO DAS PARTICULAS LENTIVIRAIS UTILIZANDO RESISTENCIA DE PULSO
Foi determinada a concentração das partículas lentivirais por mL e o diâmetro das partículas presentes no meio concentrado, por meio do equipamento qNano, da IZON, Christchurch, Nova Zelândia. As amostras foram diluídas em PBS 1X, e 40 µl da diluição de cada amostra foram lidas pelo equipamento com os seguintes parâmetros: poro de tamanho NP150, abertura do poro com 49mm, corrente de 0,28V e pressão a 1cm H2O. A corrente média entre todas as
amostras variou entre 126,83 nA e 129,30 nA, sendo que a calibração foi feita utilizando beads de diâmetros e concentrações conhecidas, que foram diluídos em PBS e medidos de forma idêntica às amostras.
A preparação do equipamento e da amostra para a leitura foi realizada da seguinte forma: a abertura dos dentes foi calibrada em 45 mm com o paquímetro; depois, 75µl de PBS foi colocado na coluna da célula inferior entre o poro, sendo posteriormente usado 40 µl de PBS para limpeza do poro. A voltagem foi ajustada para 0,80V, com corrente a 100mA e noise a aproximadamente a 10. Para a leitura da amostra, a cavidade onde é colocada a amostra foi lavada 3 vezes com 40 µl de PBS, e a pressão foi ajustada para 7 atm, sendo que deve ser feita a leitura de 500 partículas em 5 min. Após esse processo, a interpretação dos resultados obtidos da leitura foi realizada pelo software Izon Control Suite versão 2.2.
A validação do experimento de transfecção foi realizado por um controle negativo, onde as mesmas condições foram repetidas com a exceção da adição dos plasmídeos. As seis amostras que representam o controle negativo foram unidas em um pool para a leitura no qNano.
5.4. ENSAIO DE TRANSDUÇÃO DE CÉLULAS J774 COM LENTIVIRUS
a uma concentração de 105 células.ml-1, colocando 100µl de uma suspensão
celular de 3ml em 24 dos 96 poços. Depois sendo colocado na incubadora de cultura celular. No dia seguinte, o sobrenadante dos poços foi retirado e descartado. Ao meio usado para reposição adicionou-se 8 µl/ml de polybrene (brometo de hexadimetrina) e em cada poço foi adicionado 100 µl desse novo meio. Após, foi adicionado 10-100µl de cada tipo lentiviral produzido.
No dia seguinte o meio adicionado no dia anterior foi removido e, posteriormente 200µl de meio DMEM+10%SBF foi adicionado a cada poço usado no ensaio. No quarto dia de ensaio, as células transduzidas foram selecionadas com puromicina (Empresa Gibco, concentração 10mg/ml, lote 1416419), na concentração de 0,5 µg/ml. Depois de 48 horas em contato com a puromicina, células não transfectadas foram mortas pelo reagente. As células mortas foram removidas com o sobrenadante, e as células transduzidas, nesse ponto em uma placa de petri de seis poços, continuaram sendo cultivadas com meio DMEM+10%SBF.
5.5. KNOCKDOWN DA AUTOFAGIA EM CÉLULAS J774 – TRANSDUÇÃO, EXTRAÇÃO DE RNA E PCR
Células J774 sem transdução foram cultivadas em placas de 96 poços a 104 células por poço, sendo adicionada posteriormente a concentração de
lentivírus de 100µl, 50µl e 10µl. Depois de um tempo, foi transferido o volume da placa de 96 poços para uma placa de 6 poços, usando 5 µg/ml de puromicina. Após atingir a confluência necessária, o conteúdo da placa de seis poços foi duplicado, sendo separada uma das placas para a realização de qPCR. Isso para cada tipo de lentivírus produzido.
41
5.6. CULTURA FUNGICA
As linhagens Pb265 e Pb18 de P. brasiliensis e a linhagem Pb01 de P. lutzii foram mantidas em meio Fava-Netto, em estufa a 37ºC. A linhagem Pb18 é um tem origem de isolado clínico de grande virulência, enquanto Pb265 é um isolado menos virulento de P. brasiliensis. O isolado Pb01 é uma linhagem classificada como uma nova espécie, P. lutzi. O meio Fava-Netto usado é composto de Proteose peptona (3g/L), Peptona (10g/L), Extrato de carne (5g/L), Cloreto de sódio (5g/L), Dextrose (40g/L), e Ágar (18g/L) dissolvidos em água destilada. Os repiques foram realizados de 7 em 7 dias para manter a viabilidade dos fungos para realização dos experimentos. O quinto dia de crescimento fúngico foi o dia escolhido para a realização dos experimentos.
5.7. EXPERIMENTO DE CONTAGEM DE UNIDADES FORMADORAS DE COLONIAS –CFU’S
Para realizar a contagem das unidades formadoras de colônia, as células transfectadas foram cultivadas até atingir 80% de confluência e assim, 50µl de uma suspensão de 4x105 células.ml-1 dessas células foi plaqueada em uma
placa de 96 poços. Uma amostra celular de J774 que foi transduzida com 5ul do lentivírus 305, outra de J774 transduzida com 5ul do lentivírus 306, mais uma J774 transduzida com 50ul do lentivírus EGFP, além de uma amostra de J774 sem nocaute de autofagia foram colocadas na placa de 96 poços em cinco replicatas de cada amostra celular transduzida com três tipos lentivirais diferentes.
A linhagem Pb18 foi cultivada em caldo BHI durante 12 horas, e depois de crescido em BHI, o fungo foi sedimentado por centrifugação a 400g por 5 minutos e posteriormente ressuspendido em meio DMEM + 10% SBF. A contagem da densidade celular da cultura foi necessária para a realização da diluição de 5x105
O sobrenadante da placa de 96 poços foi descartado, e o meio reposto com a diluição de Pb, 100µl em cada poço.
Segundo a literatura, o macrófago deve ser ativado com IFN para que possa combater a infecção com Pb, por isso nesse experimento foram separadas placas onde o macrófago J774 com nocaute se manteve inativado, e outra situação onde o macrófago J774 foi ativado. Na situação onde o macrófago está inativado, foram posteriormente adicionados mais 50µl de DMEM + 10% SBF em cada poço. Ao local onde estava o macrófago que deveria ser ativado, foi adicionado em cada poço designado anteriormente, 50µl de DMEM + 200 unidades por ml de IFN de camundongo + 2mg/ml de LPS, com concentração final de 50%. Após 24 horas em câmara úmida, foi possível realizar a contagem.
5.8. ENSAIO DE INFECÇÃO IN VITRO DE CÉLULAS RAW 264.7 COM PB
Uma linhagem imortal de macrófagos de camundongo, RAW 264.7, foi cultivada nas mesmas condições que as células HEK293T e J774, em estufa a 37ºC com 5% de CO2 em meio DMEM + 10 % de SBF. As células foram
infectadas durante 24h a 37ºC com 5% de CO2, em placas de 24 poços com
lamínulas circulares. Após esse processo, uma amostra de Pb de cinco dias foi inoculada nestes poços na proporção 1:1, obtida após contagem das células leveduriformes.
5.9. IMUNOLOCALIZAÇÃO DE LC3
43
não coradas; um controle corado, no qual foi feito um controle negativo com células RAW264.7 não infectadas e coradas com anticorpos primário e secundário; e finalmente o teste em si com células RAW264.7 infectadas com Pb18, Pb265 e Pb01 e coradas com anticorpos primário e secundário. Outra condição experimental analisada foi a infecção de células RAW 264.7 com Pb18, que foram coradas apenas com o anticorpo secundário servindo como o controle negativo, e Cryptococcus neoformans, que foram coradas com ambos anticorpos primários e secundários sendo as células infectadas com esse fungo, o controle positivo do ensaio.
RESULTADOS
6.1. FORMAÇÃO DA PARTICULA LENTIVIRAL
Para se realizar o nocaute de autofagia e observar assim a real função da autofagia no combate ao Pb, foi usado RNA interferente para realizar o silenciamento gênico do gene ATG5. O vetor escolhido para transportar o RNAi para o interior da célula foi um vetor lentiviral de terceira geração, onde existe a separação dos genes de interesse em 4 plasmídeos diferentes.
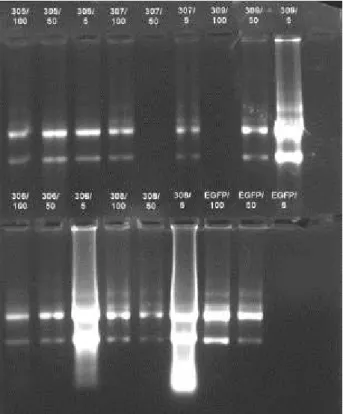
Foram multiplicados e purificados os plasmídeos necessários para a formação dos lentivírus, de acordo com a figura 09.
Figura 09 - Resultado da purificação dos plasmídeos de empacotamento
lentiviral e shRNA.
45
Tabela 01 - Quantificação das partículas lentivirais para ATG5 de macrófago de
camundongo no equipamento qNano. Amostras diluídas em PBS.
Amostra Diluição da amostra Concentração Bruta (partículas/mL) Diâmentro médio da partícula (nm) Taxa de partículas por minuto Partículas contadas
305 1:2 2,0e+10 124 1017,2 547
306 1:5 2,4e+10 117 483,6 523
307 1:5 1,3e+10 135 273,4 512
308 1:10 6,3e+10 121 63,7 517
309 1:10 2,4e+10 161 238,8 490
EGFP 1:5 6,5e+09 153 131,2 493
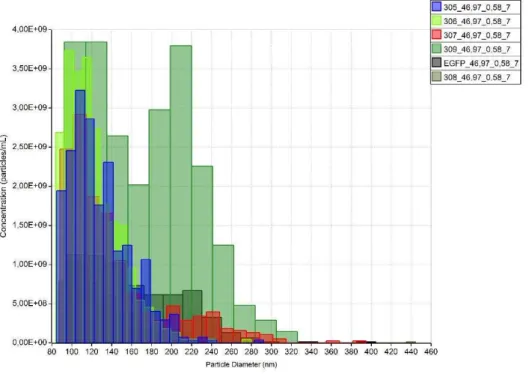
A figura seguinte mostra um histograma com a sobreposição da distribuição dos tamanhos encontrados nas cinco amostras diferentes. O tamanho de um lentivírus varia de 80 a 100nm, e nessa faixa onde se deseja encontrar a maior concentração de partículas observadas pelo equipamento.
Figura 10 – Imagem de sobreposição dos histogramas de distribuição de
tamanho. Amostra 305 (Diâmetro): Moda: 104 nm; Média: 124 nm; Máximo: 284 nm; Mínimo: 80 nm; Concentração Bruta (partículas/mL): 2,0e+10; Amostra
306 (Diâmetro): Moda: 93 nm; Média: 117 nm; Máximo: 274 nm; Mínimo: 79 nm;
EGFP (Diâmetro): Moda: 95 nm; Média: 153 nm; Máximo: 382 nm; Mínimo: 85 nm; Concentração Bruta (partículas/mL): 6,5e+09.
O controle negativo está representado na figura 11, onde é possível ver a presença de partículas de diâmetros variados, mas que se apresentam em maior quantidade na faixa de 100 a 300nm, diâmetro maior que o esperado para uma partícula lentiviral. Pode-se concluir que as partículas encontradas no controle negativo são restos de reagentes, agregados de partículas e restos celulares que conseguiram continuar nas amostras mesmo após o passar pela etapa de filtração antes da leitura no qNano.
Figura 11– Controle negativo da quantificação lentiviral no qNano. As mesmas
47
6.2. EXTRAÇÃO DE RNA
O próximo passo foi observar a capacidade de knockdown dos lentivírus no gene ATG5 em células J774. Para isso, o RNA total das células J774 foi extraído após as células serem transduzidas. Observa-se na figura 15 que o RNA total foi extraído com êxito.
Figura 12– Resultado do knockdown da autofagia em células J774. Referentes
6.3. ENSAIO DE KNOCKDOWN
Na figura 13 está o valor absoluto de expressão do gene ATG5 após o knockdown com todos os cinco tipos de lentivírus formados. Os que apresentam a menor expressão genética são os lentivírus escolhidos para prosseguir com o desenho experimental, que foi a infecção das células com knockdown com o Paracoccidioides.
Figura 13 – Resultado do qPCR. Expressão absoluta do gene em unidades
49
6.4. CONTAGEM DE CFU
Os lentivírus 305 e 305 na menor concentração foram usados para realizar novamente o experimento de transdução em células J774. Os macrófagos transduzidos foram infectados com Pb e ativados com IFN- e posteriormente lisados. A figura 14 mostra a contagem das unidades formadoras de colônias – UFC ou CFU, e a análise estatística realizada em cima do resultado obtido com a contagem.
Figura 14 - Teste de atividade antifúngica de macrófagos infectados com P.
brasiliensis. Macrófagos J774 transduzidos com dois diferentes shRNAs cujo
alvo é ATG5 ("305" e "306") e com um shRNA controle negativo ("EGFP") foram infectados com P. brasiliensis durante 24 h em meio de cultura contendo
IFN-gama. A amostra "J774" corresponde a macrófagos não transduzidos igualmente infectados e "Pb" corresponde a controle com poços contendo somente o fungo e meio de cultura, sem macrófagos. Após as 24 h de co-cultura, os macrófagos foram lisados e os fungos coletados e plaqueados para contagem de unidades formadoras de colônias. Análise estatística por ANOVA foi estatisticamente significativa (p<0,05), mas pós-testes mostraram diferença estatisticamente
0 0,5 1 1,5 2 2,5 3 3,5 U n id ad e s Arb itrá ri as Amostras
significativa somente da amostra "Pb" em relação à referência "EGFP". As barras correspondem a erro padrão da medida. Para cada grupo n = 4.
6.5. ENSAIO DE IMUNOLOCALIZAÇÃO
Nas figuras 15 e 16 pode-se observar células sem transdução infectadas com fungos RAW264.7, Pb01, Pb265 e Pb18. Com a imunolocalização de LC3, é possível observar a formação de um autofagossomo ao redor do fungo no citoplasma celular.
Figura 15 –Imunolocalização de LC3 (verde) em células RAW264.7 infectadas
51
Figura 16 –Imunolocalização de LC3 (verde) em células RAW264.7 infectadas
DISCUSSÃO
Os vetores lentivirais são capazes de transferir para a célula alvo insertos grandes. Possuem baixa imunogenicidade e podem infectar células quiescentes. Foram produzidos nesse trabalho em pequena escala com auxílio de compostos lipossomais, por meio da transfecção transiente de uma linhagem celular empacotadora, HEK 293T (TISCORNIA, 2006). Os lentivírus são vírus envelopados com pequenas projeções do envelope dispersas por sua superfície, pleomórficos, esferóides e com diâmetro de 80-100nm. O aumento de temperatura é capaz de inativar esse vírus, além da sensibilidade à ação de produtos químicos como fenóis, detergentes, hipoclorito, formalina e compostos quaternários de amônio (SILVA, 2007).
O objetivo de realizar o nocaute da autofagia, observando assim a real função desse mecanismo no combate ao Pb, foi possível pelo uso de RNA interferente – RNAi, que são originadas de moléculas de RNA de dupla fita gerados a partir da transdução de shRNA. O silenciamento gênico acontece pelo pareamento do siRNA de forma parcial ou total com o mRNA de interesse, impedindo sua tradução (HAMMOND, 2000). O alvo dos shRNA usados foi o RNAm transcritos do gene ATG5 de camundongo.
O vetor escolhido para transportar o RNAi para o interior da célula foi um vetor lentiviral por ser um vetor capaz de inserir o gene de interesse no genoma do hospedeiro, fazendo com que a replicação do gene seja feita pela própria célula hospedeira e diminui assim as chances de que a célula ataque a molécula formada. O lentivírus pertence ao gênero retrovírus e possui um período de incubação relativamente longo. O lentivírus produzido nesse trabalho é de terceira geração, onde existe a separação dos genes de interesse em 4 plasmídeos diferentes, para aumentar o título de produção lentiviral e a biossegurança, evitando uma possível restauração do fenótipo original
(TENÓRIO, 2008).
53
mesmo alvo e posteriormente testados com o objetivo de observar qual possui melhor eficiência no nocaute do gene.
Segundo as concentrações obtidas com o qNano, a amostra com maior concentração de partículas, fora o controle fluorescente EGFP, é a amostra 308, com 6,3e+10 partículas por mL. Porém isso não significa que todas as partículas contadas representam partículas lentivirais. Restos celulares, corpos multivesiculares e pequenas partes da membrana que formam micelas podem estar presentes na amostra mesmo depois da centrifugação realizada para sedimentar restos celulares após a coleta do sobrenadante, e antes da centrifugação para concentração dos lentivírus. Isso pode ser observado na figura 13, com distribuição de tamanho das partículas encontradas em cada amostra. A variação de tamanho encontrada revela a presença de outras partículas.
Para se ter uma noção se a contagem foi de partículas lentivirais ou de restos celulares, o mesmo experimento foi realizado sem a adição de plasmídeos de formação do vetor lentiviral nem do plasmídeo para transcrição do RNAi. Por comparação dos resultados obtidos com a leitura dessas amostras com as amostras do experimento, concluímos que as partículas lidas pelo qNano que tinham o diâmetro maior que 100nm eram, de fato, restos celulares, corpos multivesiculares e partículas oriundas dos reagentes lipossomais usados na metodologia do experimento.
O gene ATG5 codifica a proteína Atg5 que é uma E3 ubiquitina-ligase, necessária no estágio de elongação do autofagossomo, sendo ativada pela proteína Atg7 e, posteriormente, forma um complexo com Atg12 e Atg16L1. Este complexo desempenha função importante na conjugação do LC3-I ao PE, formando o LC3-II, marcadores que sinalizam que o processo autofágico foi completado (CODOGNO, 2006).
de menor concentração foram os escolhidas para o experimento de contagem das unidades formadoras de colônia, além da amostra com o lentivírus com EGFP, que vai demonstrar que a célula foi transduzida com sucesso e continua seu ciclo de vida (BAENS, 2006).
Macrófagos de camundongos foram usados nos experimentos. Uma das diferenças entre macrófago humano e de camundongo é falta de capacidade do macrófago humano de aumentar a expressão de arginase 1, um importante marcador da regulação de IL-4 de macrófago de camundongos (GEISSMANN,2010).
Os macrófagos são células fagocíticas encontradas em todos os tecidos do organismo humano. Têm diferentes origens como medula óssea e saco vitelino, além de possuir complexidade transcricional, são capazes de mudar seu fenótipo de acordo com a necessidade (WYNN, 2013). Várias tentativas foram feitas para classificar os macrófagos como o sistema fagocítico mononuclear (mononuclear phagocytic system – MPS), o qual considera duas classes, a de células final da linhagem fagocítica e os progenitores na medula óssea, levando em conta os macrófagos como células finais na linhagem fagocítica. Outra tentativa se refere a uma classificação binária que considera os estados inflamatórios para categorizar os macrófagos quando eles estão ativados e macrófagos ativados alternativamente (alternatively actvated macrofage - AAM), sendo estes estados definidos pela resposta a citocina interferon- (IFN- ), ativação de receptores “Toll-like” (TLRs) e interleucina 4 (IL-4) e IL-13, respectivamente (ARIEL, 2012).
55
As infecções fúngicas sistêmicas, como a PCM, são primeiramente combatidas pelo sistema imune inato. A interação patógeno-hospedeiro decorrente de uma infecção determina o desenvolvimento da doença. Um modelo de interação muito estudado é a que ocorre entre Candida albicans (C. albicans) e o macrófago (NETEA, 2008). Esse fagócito, como outros do sistema imune inato, reconhece patógenos fúngicos por meio de PAMPs – padrões moleculares associados a patógenos, cujos mais importantes são os carboidratos da parece celular fúngica: mananas, -glicanas e quitina (JIMÉNEZ-LÓPEZ, 2013).
A ativação clássica dos macrófagos ocorre pela combinação de dois sinais, IFN e o fator de necrose tumoral (TNF), que aprimora a capacidade do macrófago de combater infecções. INF é produzido pelas células NK como resposta a estímulos relacionados ao estresse e infecções, levando os macrófagos a secretarem citocinas pró-inflamatórias, aumentando a resistência do hospedeiro às infecções (MOSSER, 2008). Por isso, espera-se que os macrófagos que foram ativados com INF tenha uma maior capacidade de conter a infecção fúngica, reduzindo assim a quantidade de CFU’s.
A análise estatística por ANOVA, feita com base nos resultados encontrados da contagem de CFU’s, mostrou que não houve diferença estatisticamente significativa entre os macrófagos que receberam a transdução e os que não receberam. Isso pode ter ocorrido devido ao fato de apenas um dos genes que estão envolvidos na formação do autofagossomo ter sido alterado, possibilitando à célula lançar mão de outros mecanismos para compensar essa falta, pois a autofagia não é o único mecanismo imunológico para conter a infecção. Se mais de um gene responsável por formar o autofagossomo for alterado, existem maiores chances da diferença estatística ser mais relevante.
Com as outras condições experimentais pode-se observar a autofagia agindo na contenção da infecção, ao se observar o acúmulo de LC3 ao redor dos fungos usados na infecção. O anticorpo primário foi o IgG policlonal de coelho contra LC3 humana, e o secundário foi o IgG de cabra anti-IgG de coelho conjugado à CF88. Os vários pontos dispersos na célula representam o LC3 antes da lipidação, que naturalmente está presente no citoplasma. O acúmulo de LC3 ao redor do fungo demonstra a atividade imunológica que esse mecanismo também desempenha, ao conter seu crescimento intracelular, impedindo que o fungo tenha livre acesso às moléculas citoplasmáticas e levando o microrganismo para ser degradado por lisossomos.
A proteína de cadeia leve 3 (LC3) é normalmente usada como padrão ouro de identificação da formação do autofagossomo, indicando sua formação ou bloqueio de maturação (VIRGIN, 2009). Sua identificação geralmente é realizada por ensaios de imunolocalização com anticorpos conjugados, como o realizado nesse trabalho. Por ser previamente conhecido que o Cryptococcus neoformans se encontra em um autofagossomo durante sua infecção em macrófagos (NICOLA ET AL., 2012), este microrganismo foi escolhido como controle positivo, sendo possível validar o experimento.
Em outros trabalhos, já foi comprovada a ação de reconhecimento de fungos por macrófagos (ESTEBAN, 2011) e a participação da autofagia no combate a infecções bacterianas e virais tem sido estudada de forma extensiva, com a finalidade de encontrar meios alternativos de combate as doenças que mais afetam a população no geral (DERETIC, 2013).
57
CONCLUSÕES E PERSPECTIVAS
A produção lentiviral foi satisfatória, pois a menor concentração já foi o suficiente para alcançar o resultado desejado: produzir uma linhagem celular de camundongo geneticamente alterada. A partir desse momento, foi possível observar in vitro a infecção com o gênero Paracoccidioides.
O presente estudo mostrou que a atividade imunológica da autofagia existe, porém será estatisticamente significante se estudos posteriores envolvendo o knockdown de mais de um gene responsável pela formação do autofagossomo forem realizados. Essa etapa fica como sugestão para um futuro trabalho, no qual pode ser usada a célula THP-1 para passar pelo processo de nocaute dos genes ATG5 e ATG7. Essa linhagem celular, proveniente de uma leucemia monocítica aguda, é um valioso modelo no estudo dos mecanismos da homeostase e diferenciação de macrófagos.
REFERÊNCIAS
Ariel, A.; Maridonneau-Parini, I.; Rovere-Querini, P.; Levine, J. S.; Mühl, H.;
Macrophages in inflammation and its resolution; Frontiers in Immunology, v.
3, Article 324; November, 2012.
Auwerx, J.; The human leucemia cell line, THP-1: a multifacetted model for the study of monocyte-macrophage differentiation. Experientia, v. 15; n. 47; p. 22-31; Janeiro, 1991.
Bagagli, E. et al. Paracoccidioides brasiliensis: phylogenetic and
ecological aspects. Mycopathologia. v.165; p. 197–207, 2008.
Baens, M.; Noels, H.; Broeckx, V.; Hagens, S.; Fevery, S.; et al; The Dark Side
of EGFP: Defective Polyubiquitination. PLoS ONE, v. 1, n. 1, p. 54;
doi:10.1371/journal.pone.0000054, 2006.
Blanco J. L., Garcia M. E.; Immune response to fungal infections. Vet Immunol Immunopathol, v. 125; p. 47–70, 2008.
Botts M. R., Hull C. M.; Dueling in the lung: how Cryptococcus spores race
the host for survival. Curr Opin Microbiol, v. 13, p. 437–442; 2010.
Castro, L. F. et al; Characterization of the immune response in human
paracoccidioidomycosis; Journal of Infection, v. 67, p. 470 a 485; 2013.
Carrero L.L., et al; New Paracoccidioides brasiliensis isolate reveals
unexpected genomic variability in this human pathogen. Fung Genet Biol,
v. 45, p. 605-612, 2008.
Codogno, P.; Meijer, A. J.; Atg5: more than an autophagy factor; nature cell biology, v. 8, n. 10, October; 2006.
Deretic, V., et al; Autophagy in infection, inflammation and immunity;
59
Fortes, M.R.P., et al.; Imunologia da paracoccidioidomicose; An Bras
Dermatol; v. 86, n. 3, p. 516-25, 2011.
Franco, M.; et al; Chlamydospore formation by Paracoccidioides
brasiliensis mycelial form. Rev. Inst. Med. Trop. S. Paulo, v. 31, n. 3, p.
151-157, 1989.
Gegembauer G., et al.; Serology of Paracoccidioidomycosis Due to
Paracoccidioides lutzii. PLoS Negl Trop Dis; v. 8. Julho, 2014.
Geissmann, F. et al; Development of monocytes, macrophages and
dendritic cells; Science, NIH aublic access (author manuscript); v. 327; p.
656–661. February, 2010.
Hammond S. et al; An RNA-directed nuclease mediates post-transcriptional
gene silencing in Drosophila cells; Nature 404; v. 6775, p. 293–6, 2000.
Hippert, M.M. et al. Autophagy in cancer: good, bad, or both? Cancer Research. v. 66, n. 19, p. 9.349-51; 2006.
Jiménez-López, C.; Lorenz, M. C.; Fungal Immune Evasion in a Model Host–
Pathogen Interaction: Candida albicans Versus Macrophages. PLoS
Pathog v. 9, n. 11; November 21; 2013.
Klionsky, D. J.; The molecular machinery of autophagy: unanswered
questions. J. Cell Science; v. 118; p. 7-18; 2005.
Kurokawa, C.S. et al; Virulence profile of ten Paracoccidioides brasiliensis
isolates. Association with morphologic and genetic patterns. Rev. Inst. Med.
trop. S. Paulo, v. 47; n. 5; p. 257-262; 2005.
Lamb, C. A., et al. The autophagosome: origins unknown, biogenesis
complex; nature reviews molecular cell biology,v. 14, p. 759; December, 2013.
Marques S. A., et al; Paracoccidioidomicose: frequência, morfologia e
patogênese de lesões tegumentares; An Bras Dermatol; v. 82; p. 411-417;
2007.
Mintern J. D. and Villadangos J. A.; Autophagy and mechanisms of effective
immunity. Frontiers in Immunology; Antigen Presenting Cell Biology; v. 3,
Article 60; April 2012.
Monteiro da Silva, J.L. et al.; Interação de Paracoccidioides brasiliensis com
células endoteliais; Rev. Ciênc. Farm. Básica Apl., v. 26, n. 2, p. 149-156;
2005.
Mosser, D. M.; Edwards, J. P.; Exploring the full spectrum of macrophage
activation; Nat Rev Immunol; v. 8; n. 12; p. 958–969. December, 2008.
Netea, M.G. et al; An integrated model of the recognition of Candida
albicans by the innate immune system. Nat Rev Microbiol. v. 6, p. 67–78;
2008.
Nicola, A.M. et al; Macrophage autophagy in immunity to Cryptococcus
neoformans and Candida albicans. Infect Immun.; v. 80, n. 9, p. 3065-76.;
Sep 2012.
Palmeiro M, Cherubini K, Yurgel LS; Paracoccidioidomicose– revisão da literatura; Scientia Medica, Porto Alegre: PUCRS, v. 15, n.. 4, out./dez, 2005.
Pereira, V. B. R. et al; Molecular approaches for eco-epidemiological
studies of Paracoccidioides brasiliensis; Mem Inst Oswaldo Cruz, Rio de
Janeiro, v. 104; n. 4; p. 636-643; 2009.
Qin, Q. M. et al; Functional analysis of host factors that mediate the
intracellular lifestyle of Cryptococcus neoformans. PLoS Pathog.; v. 7; Jun,
2011.
S. Li, M. P. et al; Sterical hindrance promotes selectivity of the autophagy cargo receptor NDP52 for the danger receptor galectin-8 in antibacterial
61
Santos, R. S. et al.; O patógeno humano Paracoccidioides brasiliensis e a
paracoccidioidomicose: uma abordagem médica e molecular. Revista da
Universidade Vale do Rio Verde, Três Corações, v. 9, n. 2, p. 281-295; 2011.
San-Blas, G., Burger, E.; Experimental medical mycological research in
Latin America - a 2000-2009 overview; Revista Iberoamericana de Micología.
Published by Elsevier España, S.L. All rights reserved; 2010.
Shalhoub, J., et al; Innate immunity and monocyte-macrophage activation
in atherosclerosis. Journal of Inflammation; 2011.
Sidrim, J. J. C.; Micologia médica à luz de autores contemporâneos. Rio de Janeiro: Guanabara Koogan, 2010.
Silva, J. B. A., Lima, P. M.; Small ruminants Lentiviruses: etiological
characterization, infectivity, control, prevention and diagnosis; Acta Veterinaria Brasilica, v.1, n. 4, p.111-117, 2007.
Teixeira M. M., et al; Paracoccidioides Species Complex: Ecology,
Phylogeny, Sexual Reproduction, and Virulence.PLoS Pathog; v. 10; n. 10; 2014.
Tenório, L. Z., Silva, F. H., Han, S. W.; The Potentiality of Lentivectors in
Gene Therapy; Rev Bras Clin Med; v. 6; p. 260-267; 2008.
Tiscornia, G., Singer, O., Verma, I.M.; Production and purifiation of lentiviral
vectors. Nat Protoc; v. 1; p. 241-245; 2006.
Trabulsi, L. R.; Microbiologia. 5ª edição. São Paulo. Atheneu. 2008.
Vural, A.; Kehrl, J. H.; Autophagy in Macrophages: Impacting Inflammation
and Bacterial Infection; Article ID 825463, Hindawi Publishing Corporation
Scientifica. 2014.
Wynn, T. A.; Chawla, A.; Pollard, J. W.; Macrophage biology in development, homeostasis and disease; nature, v. 496; 2013.
63
ANEXOS
10.1. MAPA DOS PLASMÍDEOS
Anexo A: TAT - Transativador do ativador de transcrição (HIV-1)
A proteína derivada desse gene é rica em arginina (sequência que compreende aminoácidos 49 ao 57) e possui a habilidade de se movimentar através da membrana de plasma, característica que torna essa proteína interessante para entrega intracelular de moléculas e bioativos através da membrana plasmática (Saleh, 2010). É considerada uma proteína acessória relacionada com a regulação da expressão gênica, aumentando a velocidade de transcrição ao se ligar a uma “dobra”, conhecida como TAR – elemento de resposta ao transativador, localizado no terminal 5’ do RNA em formação. Sem essa proteína, as RNA polimerases não conseguem transcrever sequencias grandes, maiores que algumas centenas de nucleotídeos (Peçanha, 2002). Mapa do plasmídeo: