UNIVERSIDADE
CATÓLICA DE
BRASÍLIA
Programa de Pós-graduação Stricto
Sensu
em Ciências Genômicas e
Biotecnologia
.
Mestrado
CARACTERIZAÇÃO MOLECULAR DE ISOLADOS DE
PSEUDOMONAS AERUGINOSA
DO HOSPITAL DE BASE
DO DISTRITO FEDERAL
Autora: Patrícia Siqueira de Medeiros
Orientador: Prof. Dr. Ricardo Henrique Krüger
PATRÍCIA SIQUEIRA DE MEDEIROS
CARACTERIZAÇÃO MOLECULAR DE ISOLADOS
DE
PSEUDOMONAS AERUGINOSA
DO HOSPITAL DE
BASE DO DISTRITO FEDERAL
Dissertação apresentada ao Programa de Pós-Graduação “Stricto Sensu” em Ciências Genômicas e Biotecnologia da Universidade Católica de Brasília, como requisito para a obtenção do Título de Mestre em Ciências Genômicas e Biotecnologia.
Orientador: Prof. Dr. Ricardo Henrique Krüger
FICHA CATALOGRÁFICA
Medeiros, Patrícia Siqueira de.
Caracterização molecular de isolados de Pseudomonas Aeruginosa do Hospital de Base do Distrito Federal / Patrícia Siqueira de Medeiros. -- Brasília, 2008.
142f. : il.
Orientador: Ricardo Henrique Krüger
Dissertação (Mestrado em Ciências Genômicas e Biotecnologia) – Universidade Católica de Brasília, Brasília, 2008.
TERMO DE APROVAÇÃO
Dissertação defendida e aprovada como requisito parcial para a obtenção do Título de Mestre em Ciências Genômicas e Biotecnologia, defendida e aprovada, em 7 de maio de 2008, pela banca examinadora constituída por:
_________________________________________
Prof. Dr. Ricardo Henrique Krüger
_________________________________________
Prof. Dr. Julival Ribeiro
_________________________________________
Prof. Dr. Robert Neil Gerard Miller
Agradecimentos
A Deus, primeiramente, o meu reconhecimento pela sua intersecção, proporcionando-me
sabedoria sem a qual não teria sido possível a realização desta pesquisa.
Várias pessoas contribuíram direta ou indiretamente para o sucesso deste trabalho – meu
sincero agradecimento.
Ao professor Ricardo Henrique Krüger, por sua dedicação, paciência e empenho em
transmitir conhecimentos não só relacionados à biotecnologia, mas, principalmente ao
demonstrar a importância de reconhecer os limites humanos e que o sucesso da ciência não
está apenas em buscar a perfeição.
Agradeço o apoio à equipe do Hospital de Base do Distrito Federal e aos demais professores e
funcionários que integram o Curso de Pós-Graduação em Ciências Genômicas e
Biotecnologia da UCB.
Aos colegas de pesquisa, que ao longo do Curso, foram meus companheiros e dos quais
sentirei saudades. Meus sinceros agradecimentos à colega Adriane da Silva Kurokawa, pela
paciência, dedicação e empenho em transmitir conhecimentos relacionados à pesquisa.
Aos meus familiares, em especial à minha mãe Maria Zenilde Siqueira Medeiros e ao meu
esposo Rodrigo Araújo Damasceno, pelos momentos que se viram privados da minha
“Se o motivo da ciência é dominar a natureza, não é menos verdade que só podemos vencer a natureza obedecendo-lhe”.
SUMÁRIO
LISTA DE TABELAS ... 09
LISTA DE FIGURAS ... 10
ABREVIATURAS ... 11
RESUMO ... 14
ABSTRACT... 15
INTRODUÇÃO...16
1. MICROBIOLOGIA...18
1.1. Taxonomia...18
1.2. Considerações para a saúde humana...20
2. RESISTÊNCIA A ANTIBIÓTICOS... 25
2.1. Permeabilidade da membrana e sistemas de efluxo ... 29
2.2. Resistência a Antibióticos β-lactâmicos ... 31
2.3. Resistência aos Carbapenens ... 34
2.4. Resistência a Aminoglicosídios... 36
2.5. Resistência a Fluoroquinolonas ... 38
2.6. Resistência aos Macrolídios ... 39
3. VIRULÊNCIA... 40
4. GENOTIPAGEM ... 48
5. OBJETIVOS... 54
5.1. Objetivo geral ... 54
5.2. Objetivos específicos ... 54
6. MATERIAL E MÉTODOS... 55
6.1. Amostras de bactérias e cultura ... 55
6.2.Identificação Randômica do Polimorfismo do DNA genômico de P. aeruginosa... 57
6.2.1. Extração do DNA cromossomal ... 57
6.2.2. Análise RAPD ... 59
6.3. Macro-restrição do genoma de P. aeruginosa... 61
6.4. Análise dos dados ... 64
7. RESULTADOS ... 68
7.2. Perfil de Sensibilidade aos Antimicrobianos... 70
7.3. Análise RAPD ... 72
7.4. Macro-restrição... 77
8. DISCUSSÃO... 79
CONCLUSÃO... 93
PERSPECTIVAS ... 94
REFERÊNCIAS ... 95
APÊNDICES...106
LISTA DE TABELAS
Tabela 1 – Seqüências dos 10 primers selecionados que apresentam polimorfismos reproduzíveis para P. aeruginosa, segundo Mahenthiralgam et al (1996)... 55
Tabela 2 – Distribuição dos Pacientes por Idade e Sexo... 62
Tabela 3 – Distribuição das Amostras de P. aeruginosa, de acordo com o Setor de Atendimento ... 63
Tabela 4 – Distribuição das amostras de P. aeruginosa, conforme a fonte de isolamento ... 63
Tabela 5 - Lista dos Discos-Difusão (Kirby-Bauer) utilizados para o perfil das cepas ... 65
LISTA DE FIGURAS
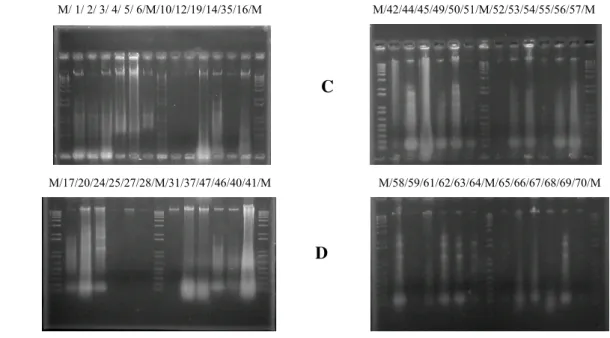
Figuras 1 – Padronização do DNA cromossomal extraído... 68
Figura 2 – Reação de PCR por RAPD (primer 208). ... 69
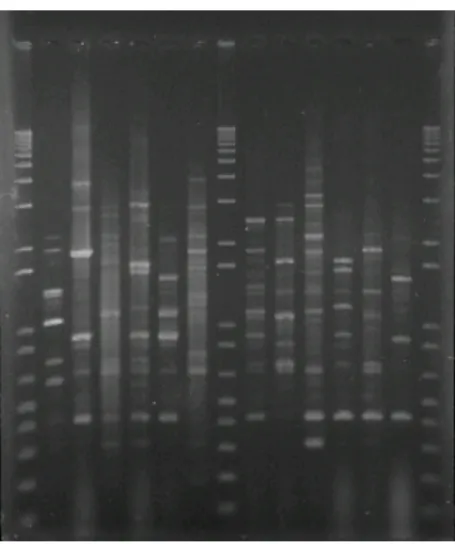
Figura 3 – Dendograma obtido pelo Programa BioNumerics para a técnica RAPD . ... 71
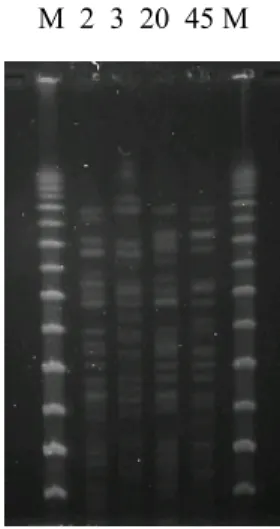
Figura 4 – Padrão de bandas obtidos por PFGE para as amostras 2 (2526/01/04), 3 (3328/01/04), 20 (3523/01/04) e 45 (3305/02/04)... 72
ABREVIATURAS
Aids – Acquired Immunodeficiency Syndrome AK – Amicacina
AmpC – β-lactamase induzível da classe C de Ambler AFLP - Amplified Fragment Length Polymorphism - AP-PCR - Arbitrarily Primed Polymerase Chain Reaction AT – Adenina-timina
ATM – Aztreonam CAZ - Ceftazidima CIP – Ciprofloxacino
CLSI - Clinical and Laboratory Standards Institute CN – Gentamicina
CTAB - Cetyl Trimethyl Ammonium Bromide
DNA – Deoxyribonucleic acid/ Ácido Desoxirribonucléico dNTP - Desoxirribonucleotídeos Fosfatados
EDTA - Ethylenediamine tetraacetic acid (Ácido Etilenodiamino Tetracético) ESBL – Extended-spectrum β-lactamase
EUA – Estados Unidos da América FEP - Cefepime
GC – Guanina-Cianina H - Horas
HBDF – Hospital de Base do Distrito Federal HIV – Human Immunodeficiency Virus IPM – Imipenem
IS – Insertion Sequence LB – Luria-Bertani Broth LMP – Low Melt Point LPS – Lipopolissacarídios M – Molar
mb - Megabase
MEP – Mucoid exopolysaccharide MgCl2 - Cloreto de Magnésio
μg – Micrograma
MIC – Concentração Inibitória Mínima Min - Minuto
mL – Mililitro
μL – Microlitro mm - Milímetro
MRSA – Multi-Resistent Staphylococcus aureus NaCl – Cloreto de Sódio
Na2CO3 – Carbonato de Sódio NaP - Fosfato de Sódio
NCCLS – National Commitee for Clinical Laboratories Standard – US 2002 nM - NanoMoles
OECD – Organization for Economic Co-operation and Development Pb – Pares de base
PB - Polimixina
PBP – Penicilin-binding protein
PCR – Reação de Polimerização em Cadeia PFGE - Pulse-field gel electrophoresis pH – potencial hidrogeniônico
pM - PicoMoles
PSE - Pseudomonas specific enzyme QS - Quorum-sensing system
RAPD – Random Amplified Polymorphic DNA RNA – Ribonucleic acid/ Ácido Ribonucléico RND – Resistance nodulation cell division rpm – Rotação por minuto
rRNA – RNA ribossomal SDS - Sodium Dodecyl Sulfate
SIDA – Síndrome da Imunodeficiência Adquirida SNP – single-nucleotide polymorphism
TE – Tris-EDTA
TSA – Tripticas Soy Agar; Caldo Tríptico de Soja TZP – Piperaciclina/Tarzobactam
U - unidades
UCB – Universidade Católica de Brasília UFC – Universidade Formadora de Colônia UTI – Unidade de Terapia Intensiva
UPGMA - Unweighted Pair Group Method Using Arithmetic Averages V/cm – Volts/centímetro
RESUMO
Pseudomonas aeruginosa constitui uma espécie bacteriana altamente versátil, de distribuição ubíqua, e um dos principais agentes causadores de infecções nosocomiais. O presente trabalho analisou 60 amostras de P. aeruginosa, obtidas de pacientes atendidos no Hospital de Base do Distrito Federal (HBDF), destacando-se como primeiro estudo de variabilidade genética desta bactéria realizado neste hospital. Os objetivos do trabalho foram identificar o perfil de sensibilidade microbiana dos isolados, pelo método Kirby-Bauer, conforme recomendações do National Committee for Clinical Laboratories (NCCLS); e realizar a genotipagem das amostras, utilizando a identificação randômica do polimorfismo de DNA amplificado (RAPD) e macro-restrição por eletroforese em gel de campo pulsado (PFGE). As amostras foram isoladas a partir de diferentes espécimes clínicos e ambientes hospitalares. Quanto à resistência bacteriana, 100% dos isolados foram sensíveis à Polimixina, 35% foram resistentes ao Ciprofloxacino, 30% apresentaram resistência concomitante ao Imipenem e à Gentamicina, 26% à Piperacilina/Tazobactam e 23% ao Meropenem. A análise dos resultados obtidos por RAPD e PFGE demonstrou grande diversidade genética, com formação de vários grupos entre as amostras estudadas. Embora não tenha havido relação direta entre o grau de resistência das amostras isoladas e os setores de atendimento onde foram coletadas, foi possível evidenciar grande variabilidade genética de amostras isoladas em um mesmo hospital terciário de Brasília. Os resultados demonstram a importância de realizar estudos que utilizam ferramentas de biologia molecular, para efetuar o monitoramento preciso dos pacientes infectados por P. aeruginosa, estabelecer uma conduta clínica mais adequada e evitar a seleção de cepas multi-resistentes.
ABSTRACT
This is the first study in the Distrito Federal to analyze the genetic diversity of 60 isolates of Pseudomonas aeruginosa, a ubiquitous and highly versatile bacterium, and one of the major nosocomial pathogens, obtained from patients from the Hospital de Base of the Distrito Federal (HBDF), Brazil. Samples were isolated from different clinical specimens and from different hospital sectors. The aims were: to identify the sensitivity profile of microbial isolates (Kirby-Bauer method recommended by the National Committee for Clinical Laboratories – NCCLS) and to perform genotyping of samples by random amplified polymorphic DNA (RAPD) and genome macrorestriction fingerprinting resolved by pulsed-field gel electrophoresis (PFGE). The results show that 100% of the isolates were sensitive to polymyxin, 35% were resistant to ciprofloxacin, 30% were resistant to imipenem as well gentamicin, 26% to piperacillin /tazobactam, 23% to meropenem, 15% to ceftazidime and 14% to amikacin. Analysis of RAPD and macro-restriction results with the BioNumerics program allowed the construction of cladograms which displayed a great number of clusters, suggesting high genetic diversity. However, no direct relationship between rate/degree of resistance collection origin was possible; the great genetic diversity in the same tertiary hospital was evident. The results demonstrate the importance of molecular biology tools to precise tracking of patients infected by P. aeruginosa, as a means to establish appropriate clinical practices to avoiding selection of multi resistant strains.
INTRODUÇÃO
Pseudomonas aeruginosa constitui uma espécie bacteriana bastante versátil do gênero
Pseudomonas, podendo apresentar crescimento tanto nos solos, áreas alagadas e ambientes marinhos, quanto em tecidos animais e vegetais (RHAME et al, 1997; RAHME et al, 2000;
VILA e MARCO, 2002; WOLFGANG et al, 2003; WALKER et al, 2004; WU et al, 2005);
possuindo inclusive capacidade de crescer em níveis extremamente baixos de nutrientes e em
condições de umidade e temperatura que favorecem sua colonização (SOKOL et al., 2000;
WU et al, 2005; GREEN et al, 1974 apud ORGANIZATION FOR ECONOMIC
CO-OPERATION AND DEVELOPMENT, 2006). Esta espécie apresenta, também, a capacidade
de formar biofilmes nas mais variadas superfícies (COSTERTON et al, 1999; DONLAN e
COSTERTON, 2002; WALKER et al, 2004; CHIANG e BURROWS, 2003). Dessa forma,
P. aeruginosa tem sido a espécie mais freqüentemente utilizada em pesquisas científicas, pois constitui um dos principais agentes causadores de infecções nosocomiais.
Durante as últimas décadas, o aparecimento desta bactéria como patógeno oportunista
tem se tornado cada vez mais evidente, possivelmente pela sua alta resistência aos
antibióticos. P. aeruginosa é atualmente uma fonte significativa de bacteremia em vítimas de queimaduras, infecções do trato urinário, em pacientes com cateteres ou ligados a respiradores
artificiais (COBBEN et al, 1996; DONLAN e COSTERTON, 2002). Esta espécie bacteriana
também é a causa predominante de morbidez e mortalidade em pacientes com fibrose cística,
cujo epitélio anormal dos pulmões permite a colonização pela bactéria, por longos períodos de
tempo (LYSACK et al, 2002; TÜMMLER e KIEWITZ, 1999). Com o intuito de promover
tratamentos hospitalares mais eficazes, amostras de P. aeruginosa têm sido identificadas, a partir de isolados clínicos, mediante amplificação por Reação em Cadeia da Polimerase
(PCR).
A utilização de técnicas moleculares é de relevante importância nos estudos de
amostras de P. aeruginosa envolvidas em infecções hospitalares, desde que permitam estabelecer o grau de similaridade entre as mesmas, a caracterização de surtos, a descoberta
Nesse sentido e considerando a escassez de dados relacionados à genotipagem de
amostras de P. aeruginosa na cidade de Brasília, a presente dissertação teve o objetivo de realizar a caracterização molecular de sessenta isolados clínicos de P. aeruginosa, coletados no ano de 2004, em diferentes unidades de atendimento do Hospital de Base do Distrito
Federal (HBDF), evidenciando sua diversidade genética e buscando correlacioná-la aos
1.
MICROBIOLOGIA
1.1. Taxonomia
As Pseudomonas são parte de um grupo de microrganismos heterogêneos e largamente distribuídos na natureza, com metabolismo altamente versátil. Apresentam-se
como bacilos gram-negativos, incapazes de fermentar diversos hidratos de carbono, entre eles
a glicose (VILA e MARCO, 2002). O gênero Pseudomonas possui mais de 140 espécies, sendo bastonetes ligeiramente curvados, em sua maioria, com flagelo polar. Estes
microrganismos são usualmente catalase e oxidase positivos. A taxonomia deste gênero tem
sido baseada na utilização de análises de seqüências 16S de RNA ribossomal (rRNA). Dentre
as espécies do gênero Pseudomonas, P. aeruginosa tem sido estudada mais detalhadamente, inclusive com a utilização de técnicas genéticas (TYLER et al, 1995; ORGANIZATION FOR
ECONOMIC CO-OPERATION AND DEVELOPMENT, 2006).
Até 1973, Pseudomonas eram vistas como um largo gênero heterogêneo com membros diferenciados por características fenotípicas. Palleroni et al. (1973), utilizando
homologia de seqüências de rRNA, concluíram que cinco grupos do gênero apresentavam
características filogenéticas distintas. Em 1989, Johnson e Palleroni confirmaram estas
observações, por meio de experimentos de hibridização de DNA, reafirmando unidades
taxonômicas baseadas em associações fenotípicas e genotípicas. Atualmente, novos gêneros
foram acrescentados aos cinco grupos originalmente identificados (ANEXO A): Bulkoderia, Stenotrophomonas, Comamonas, Shewanella, Ralstonia, Methylbacterium, Shingomonas, Acidovorax e Brevundimonas (SOARES, 2001; ORGANIZATION FOR ECONOMIC CO-OPERATION AND DEVELOPMENT, 2006). O gênero Pseudomonas, cuja espécie de maior representatividade é a espécie P. aeruginosa, está estritamente confinado aos membros do grupo I (TYLER et al., 1995; SOARES, 2001;ORGANIZATION FOR ECONOMIC
CO-OPERATION AND DEVELOPMENT, 2006).
A produção de pigmentos fluorescentes, por parte das espécies de P. aeruginosa, permite que estas sejam alocadas em um subgrupo dentro do grupo I, chamado de subgrupo
pigmento piocianina, de cor azul, seja fator de diagnóstico para P. aeruginosa, a situação inversa não pode ser sempre considerada como verdadeira (ORGANIZATION FOR
ECONOMIC CO-OPERATION AND DEVELOPMENT, 2006).
A taxonomia numérica tem sido um método tradicional de classificar membros do
gênero Pseudomonas, comparando múltiplas características do isolado com um banco de dados de características já descritas. A precisão desta abordagem depende principalmente da
quantidade e qualidade dos dados disponíveis, além da complexidade de relação entre as
características analisadas (ORGANIZATION FOR ECONOMIC CO-OPERATION AND
DEVELOPMENT, 2006).
Outra forma de identificação tem sido pelo uso de kits comerciais baseados no padrão
da utilização de fonte de carbono, e em características morfológicas e fisiológicas. São
exemplos destes Kits: os sistemas API20E (API, 200 Express Street, Plainview, New York
11803, USA; Biomerieux, F-69280 Marcy-L’Etoile, France) e BIOLOG (BIOLOG Inc.,
Hayward, Califórnia, USA). Entretanto, os dados para Pseudomonas utilizados nestes sistemas, são baseados principalmente em amostras de isolados clínicos e não em amostras
ambientais, restringindo inclusive, em alguns casos, a sua utilização. O uso destes Kits requer
habilidade, pois apresenta limitação significativa para a distinção de amostras de uma mesma
espécie (ORGANIZATION FOR ECONOMIC CO-OPERATION AND DEVELOPMENT,
2006).
A classificação atualmente aceita para o gênero Pseudomonas está baseada em homologias de rRNA, como alvo comum, pois todas as formas de vida possuem ribossomos.
Para isso, as regiões variáveis e conservadas da molécula de RNA são muito importantes,
sendo as variáveis utilizadas como alvo para sondas de hibridização e primers específicos; e,
as conservadas, para sítios de ligação de primers por PCR e para hibridização de sondas
universais (ORGANIZATION FOR ECONOMIC CO-OPERATION AND
DEVELOPMENT, 2006).
O perfil diagnóstico para microrganismos também pode ser obtido utilizando
eletroforese em gel de proteínas extraídas ou de DNA fingerprinting (impressão digital do DNA), inclusive a partir da digestão do DNA genômico por endonuclease, em razão do
enzimas como a SpeI, que corta o DNA em sítios específicos com alta composição de bases
adenina-timina (AT), vão digerir o DNA em alguns poucos sítios, formando grandes
fragmentos, podendo ser separados e analisados por meio de eletroforese em gel de campo
pulsado (PFGE – Pulse Field Gel Electrophoresis) (ORGANIZATION FOR ECONOMIC CO-OPERATION AND DEVELOPMENT, 2006), o qual corre utilizando múltiplos campos
elétricos, em que moléculas de DNA de alto peso molecular migram no referido gel, alterando
sua conformação e direção de deslocamento a cada pulso elétrico (BIRREN e LAI, 1993).
A taxonomia deve estar baseada no incremento de detalhamentos dos dados
fenotípicos e genotípicos, não limitada à simples homologia genômica DNA-DNA ou à
epidemiologia microbiológica (BELKUM et al., 2001). Embora sejam várias as técnicas que
permitem a identificação de Pseudomonas e de suas espécies, ressalta-se que, independentemente do método utilizado, a aplicabilidade dos resultados obtidos está
diretamente relacionada à qualidade dos dados disponíveis, para os quais as amostras estarão
referenciadas, bem como às condições para adequada terapêutica.
1.2. Considerações para a saúde humana
Apenas algumas espécies do gênero Pseudomonas têm sido associadas a doenças humanas, destacando-se a espécie P. aeruginosa que, embora ocorra em baixa freqüência na natureza, tem uma distribuição ubíqua. Cerca de 20 a 30% da população em geral apresentam
P. aeruginosa, inclusive indivíduos saudáveis. No entanto, a freqüência de indivíduos infectados aumenta expressivamente durante a hospitalização, em razão da maior ocorrência
deste microorganismo em ambiente hospitalar (ORGANIZATION FOR ECONOMIC
CO-OPERATION AND DEVELOPMENT, 2006). Assim, P. aeruginosa tem sido considerada importante causa de infecções nosocomiais, principalmente em pacientes queimados, de
unidades neonatais e em pacientes com leucemia ou outros tipos de câncer.
A transmissão nosocomial de P. aeruginosa pode ocorrer por contato direto ou indireto. Essa transmissão resulta do contato com fontes ambientais ou de paciente para
gastrointestinal do paciente (GOVAN, 2000; MATAR et al., 2005; ORGANIZATION FOR
ECONOMIC CO-OPERATION AND DEVELOPMENT, 2006).
Tümmler e Kiewitz (1999) sugeriram que a maioria de pacientes infectados em uma
mesma família ou que freqüentam a mesma escola tendem a apresentar o mesmo clone,
indicando uma provável disseminação via paciente-paciente ou uma fonte ambiental de
contaminação. Esta premissa foi confrontada por Govan (2000) ao considerar pacientes
fibrocísticos. O autor sugeriu que esta incidência poderia não ser tão elevada, na medida em
que evidências recentes têm mostrado que pacientes não-relacionados, com diagnóstico de
fibrose cística, tendem a ser cronicamente colonizados por tipos genômicos diferentes,
indicando assim, uma baixa disseminação entre pacientes ou em centros hospitalares. O autor
conclui que esta transmissibilidade relativamente baixa entre pacientes com fibrose cística
pode estar relacionada à transformação da P. aeruginosa típica não-mucóide, com fatores de virulência bem definidos (motilidade, adesinas, proteases, exotoxinas e LPS), para a forma
mucóide embebida em um biofilme, na qual a maioria dos fatores de virulência está
suprimida. Assim, a altamente transmissível bactéria saprófita de vida livre passa a ser um
parasita pobremente transmissível (GOVAN, 2000; NGUYEN e SINGH, 2006; SMITH et al.,
2006).
Outra fonte de infecção são objetos inanimados como equipamentos ou utensílios
hospitalares, incluindo produtos farmacêuticos, desinfetantes, jarros de água, cateteres
uretrais, equipamentos de anestesia, aparatos respiratórios, e ainda, utensílios para a
alimentação (COBBEN et al., 1996; DONLAN e COSTERTON, 2002; ORGANIZATION
FOR ECONOMIC CO-OPERATION AND DEVELOPMENT, 2006; REINHARDT et al.,
2007). Na endocardite por P. aeruginosa, dois fatores principais de predisposição têm sido associados: o uso de válvulas cardíacas protéticas e o uso de drogas ilícitas por via parenteral
(ORGANIZATION FOR ECONOMIC CO-OPERATION AND DEVELOPMENT, 2006).
Eventos de bacteremia são muitas vezes associados a P. aeruginosa e ocorrem principalmente em pacientes imunodeprimidos. Dentre os fatores associados à bacteremia
estão doenças malignas, transplantes, diabetes, cirrose hepática, insuficiência renal,
queimaduras, trauma, uso de drogas intravenosas, terapia com corticoesteróides, citotoxidade
quimioterápica, ressuscitação cardiopulmonar, deficiência de imunoglobulinas, Síndrome da
invasiva, etc (HILALI et al., 2000; ORGANIZATION FOR ECONOMIC CO-OPERATION
AND DEVELOPMENT, 2006).
O trato respiratório é uma das principais vias de colonização por P. aeruginosa. A pneumonia nosocomial é a segunda infecção mais comumente adquirida em hospitais nos
Estados Unidos, sendo que P. aeruginosa tem sido o agente etiológico mais prevalente, tanto em pneumonias mono quanto polimicrobianas (ZANETTI et al., 2003). No Brasil, infecções
por P. aeruginosa tem sido a primeira causa de pneumonia nosocomial (SADER et al., 2001; GALES et al., 2005). Porém, as pneumonias por P. aeruginosa, adquiridas em comunidades, têm uma incidência mais rara, entretanto, estão freqüentemente associadas a pacientes
portadores do vírus HIV (ORGANIZATION FOR ECONOMIC CO-OPERATION AND
DEVELOPMENT, 2006).
A colonização pulmonar crônica por P. aeruginosa em pacientes com fibrose cística resulta freqüentemente em episódios agudos de pneumonia e broncoestase crônica, com
obstrução das vias aéreas, dificuldade respiratória e, em alguns casos, óbito (GILLIGAN,
1991; OSTROFF et al, 1990; SOKOL et al, 1994; COEYNE et al, 2002; LYCZAK et al,
2002). Entretanto, alguns pacientes com fibrose cística possuem expressão genotípica da
doença de forma distinta, apresentando alteração pulmonar mais leve e/ou ausência de
sintomas digestivos, levando muitas vezes a um diagnóstico tardio ou equivocado, o qual
prejudica a adequada conduta terapêutica (LEMOS et al., 2004). Ademais, P. aeruginosa
permanece como o patógeno mais frequente em pacientes com fibrose cística, sendo
encontrado em 80-90% dos pacientes infectados (GOVAN, 2000; AGARWAL et al., 2005),
estando este microrganismo ligado à deterioração pulmonar, causando elevação das taxas de
morbidade e mortalidade (AGARWAL et al., 2005).
O diagnóstico da fibrose cística, uma doença genética autossômica recessiva, quando
efetuado na infância, antes dos 10 anos de idade, em geral demonstra uma expressão
fenotípica menos grave. A variabilidade desta expressão depende de fatores mutagênicos do
gene regulador da condutância transmembrana da fibrose cística (CFTR) e de fatores
ambientais (LEMOS et al., 2004). Em estudo realizado por Alvarez et al. (2004) foi
demonstrado que o diagnóstico de pacientes com fibrose cística no Brasil, em geral, é bem
mais tardio quando comparado a outros países como Estados Unidos e Canadá. O estudo
20 anos, conforme análise comparativa dos pacientes de outros países, por exemplo, dos
EUA, os quais apresentam uma sobrevida média de 31,6 anos. Entretanto, para Lemos et al.
(2004), o diagnóstico tardio não seria apenas pelo retardo nos procedimentos para a
confirmação da doença, mas também, em razão de alguns pacientes apresentarem uma
expressão genotípica diferente, resultando em doença pulmonar leve e ausência de sintomas
digestivos. Assim, estes autores ressaltam que devem ser investigados os pacientes com
infecção respiratória de repetição, sinusite e bronquiectasias, dentre outras infecções
pulmonares, em especial aquelas por P. aeruginosa.
Nas infecções por P. aeruginosa que afetam pulmões de pacientes com fibrose cística, uma transição de não-mucóide para mucóide é observada, indicando a natureza pleomórfica
destes organismos, e ocorrendo devido à produção aumentada de exo-polissacarídios
mucóides, também conhecidos como MEP – “mucoid exopolysaccharide” (OSTROFF et al,
1990; GILLIGAN, 1991; SOKOL et al, 1994; COEYNE et al, 2002; LYCZAK et al, 2002).
Além disso, a quantidade de produtos tóxicos gerados varia de acordo com o tipo de isolado,
sugerindo que os níveis de expressão de genes codificados em cromossomos estão sujeitos a
diferenças de amostras. Há evidências de que esta variação poderia ser atribuída à posição
variável dos genes no cromossomo (VASIL et al., 1990 apud ORGANIZATION FOR
ECONOMIC CO-OPERATION AND DEVELOPMENT, 2006).
O trato genitourinário também é uma das principais vias de contaminação por P. aeruginosa. Em raros casos, P. aeruginosa tem sido associada a meningite ou abscesso craniano. Estas condições são nosocomiais, ocorrendo em pacientes imunodeprimidos,
sofrendo de doenças malignas, submetidos a procedimentos invasivos ou com neutropenia
(ORGANIZATION FOR ECONOMIC CO-OPERATION AND DEVELOPMENT, 2006).
A umidade tem sido considerada como o principal fator determinante do crescimento
de P. aeruginosa, pois a pele normal e seca não favorece a proliferação deste microrganismo. Assim, infecções de pele por P. aeruginosa tendem a ser mais freqüentes em regiões tropicais e sub-tropicais ou estão associadas a piscinas, com tendência, entretanto, de serem infecções
localizadas, como foliculites ou otites (ORGANIZATION FOR ECONOMIC
Isolados clínicos obtidos a partir de ferimentos apresentam freqüentemente P. aeruginosa, em especial aqueles contaminados com solo, tecido vegetal ou água. Esta presença pode refletir colonização e não infecção, como uma conseqüência de sua larga
distribuição na natureza. Ferimentos pontuais, principalmente aqueles penetrantes até o tecido
ósseo, podem resultar em osteomielite ou osteocondrite, sendo comum em usuários de drogas
intravenosas e em ferimentos nos pés de crianças e de indivíduos diabéticos
(ORGANIZATION FOR ECONOMIC CO-OPERATION AND DEVELOPMENT, 2006).
Reinhardt et al. (2007), ao avaliarem pacientes intubados e com diagnóstico de
2.
RESISTÊNCIA A ANTIBIÓTICOS
P. aeruginosa é naturalmente resistente à maioria dos antibióticos utilizados em larga escala. Tal resistência dar-se em parte devido à impermeabilidade da membrana externa e à
produção de polissacarídios extracelulares. Este organismo, em geral, apresenta resistência
intrínseca ou natural a baixos níveis de canamicina, penicilina (com a exceção de penicilinas
anti-pseudomonas: carbenicilina, ticarcilina, piperacilina), maioria das cefalexinas de primeira
e segunda geração, clorafenicol, ácido nalidíxico, tetraciclinas, eritromicina, vancomicina,
sulfonamida, trimetropina e clindamicina (WASHINGTON et al, 1991; DARGIS et al, 1992;
GEORGOPAPADAKOU, 1993; GOVAN, 2000; GILBERT et al, 2001; LIVERMORE,
2002; DONAY et al, 2004;).
Considerando que amostras individuais podem ser resistentes a antibióticos para os
quais as espécies são geralmente suscetíveis, padrões de resistência não devem ser presumidos
de acordo com a verificação da espécie, mas acessados conforme a enfermidade de cada
paciente. Para Martinez et al. (1996), a variabilidade no tipo e na freqüência de agentes
causais e, particularmente, de sua interação com antimicrobianos, exige correlacionar,
periodicamente, dados clínicos e microbiológicos para atualizar e direcionar a
antibioticoterapia, segundo as necessidades locais. Os autores ressaltam, ainda, que é
fundamental preservar a vida útil dos antimicrobianos efetivos, restringindo seu uso a casos
com indicação clínica, microbiológica ou farmacológica especial, ou mesmo, quando não há
outra opção antiinfecciosa.
Assim, a antibioticoterapia para as infecções causadas por P. aeruginosa depende da topografia e da sensibilidade ao antibiótico testado, sendo que algumas espécies são
suscetíveis a uma restrita variedade de antibióticos. Ceftamizina, cefsulodina, imipenem,
ticarciclina – ácido clavulânico, azlocilina, piperacilina, aminoglicosídios, colistina e
ciprofloxacina são alguns dos antibióticos com uma alta percentagem de isolados suscetíveis
(ORGANIZATION FOR ECONOMIC CO-OPERATION AND DEVELOPMENT, 2006). A
terapia combinada, utilizando dois antibióticos efetivos, pode aumentar o índice de cura
aminoglicosídios e β- lactâmicos (que têm ação contra Pseudomonas) têm sido efetivas (BONE, 1993).
Segundo Reinhardt et al. (2007), os clínicos devem ser extremamente cautelosos ao
selecionarem a terapêutica e o período pelo qual será administrado o medicamento, pois P. aeruginosa tem uma excelente capacidade de se adaptar à pressão seletiva de diversas classes de agentes antimicrobianos e, após tratamentos que ultrapassam 6 dias, clones selecionados
podem reemergir rapidamente quando este mesmo tratamento for aplicado uma segunda vez.
Em menos de 10 dias, após o início do tratamento, todas as classes de antimicrobianos
selecionam cepas de P. aeruginosa resistentes. Quando do uso do imipenem e fluoroquinolonas essas cepas resistentes a esses antimicrobianos persistiam mesmo após a
retirada das drogas. Os autores também evidenciaram que os determinantes de resistência
apresentados pelas amostras estudadas não foram em decorrência da colonização cruzada com
novas amostras, mas em razão de mutações ocorridas nas amostras originalmente suscetíveis,
colonizando pacientes por longos períodos.
Um dos fatores que dificulta a erradicação de P. aeruginosa, principalmente em infecções crônicas, como em pacientes com fibrose cística, queimados ou imunodeprimidos,
tem sido a habilidade desta espécie bacteriana resistir a uma grande variedade de antibióticos.
Em um hospedeiro infectado, P. aeruginosa reside em biofilmes, os quais são altamente resistentes ao tratamento com antibióticos. Nos pacientes com fibrose cística, quando o
infiltrado de neutrófilos e as secreções mucóides aumentam, as células metabólicas da
bactéria e as células epiteliais do hospedeiro consomem o oxigênio disponível e o ambiente
torna-se gradativamente anaeróbico. Assim, os microrganismos passam a ser mucóides, pela
secreção do exopolissacarídeo alginato, o qual restringe ainda mais a difusão de oxigênio,
contribuindo para a formação de um ambiente microaeróbico ou anaeróbico (GILLIGAN,
1991; FILIATRAUT, 2006). Entretanto, nem sempre a produção de maiores quantidades de
alginato significa a formação de um biofilme mais robusto, o que demonstra que não há
correlação entre a produção de alginato e a formação inicial do biofilme (HEAD e YU, 2004).
A formação de biofilme pela P. aeruginosa pode ser um fator de resistência a agentes antimicrobianos, pois a produção de substâncias poliméricas que formam a matriz do biofilme
dificulta a penetração em profundidade do antibiótico utilizado, retardando sua difusão. Além
razão da limitação de nutrientes, sendo pouco suscetíveis aos antibióticos, quando comparadas
às células com metabolismo regular. Outra hipótese seria de que as células no biofilme
adquirem uma característica fenotípica resultante de uma resposta biológica ao crescimento
em uma superfície e não à limitação de nutrientes. É importante ressaltar que fragmentos de
biofilme podem se romper e colonizar novas superfícies, disseminando assim a infecção
(COSTERTON et al., 1998; KIRISITS et al., 2005).
Oliver et al. (2000), ao compararem a resistência aos antibióticos de isolados de P. aeruginosa de pacientes com fibrose cística e gene mutante, daqueles com fibrose cística sem gene mutante e de pacientes com outras patologias, observaram forte relação entre populações
mutantes de P. aeruginosa e elevada resistência a antibióticos. Os autores consideraram também que fatores como: uma alta densidade celular, uma multiplicidade de mudanças
ambientais, e uma mudança e compartimentalização do habitat brônquico, podem criar um
novo cenário para o aparecimento de espécies mutantes.
Embora a administração prolongada e frequente de antibióticos tenha sido útil no
tratamento de pacientes com fibrose cística, com complicações pulmonares, existe o risco de
desenvolvimento de amostras de P. aeruginosa multi-resistentes. A rotina de exames microbiológicos de amostras respiratórias obtidas a partir de pacientes com fibrose cística
pode contribuir para o estudo das características da infecção por P. aeruginosa (AGARWAL et al., 2005).
Donay et al. (2004) demonstraram que o sistema Phoenix, mas também o Vitek 2, que são sistemas automatizados para identificação bacteriana e detecção de resistência a
antibióticos em inóculos preparados manualmente, podem ser utilizados para a maioria dos
isolados de hospitais, com a possibilidade de serem associados a outros métodos utilizados
freqüentemente para a eventual comprovação dos dados obtidos, de forma a potencializar o
gerenciamento de infecções, principalmente aquelas causadas por cepas multi-resistentes.
Entretanto, estes sistemas podem falhar na realização do teste de sensibilidade às bactérias
gram-negativas não fermentadoras.
Ribeiro et al. (1999) avaliaram a resistência de amostras de Staphylococcus aureus
pacientes atendidos em Centro Médico da Califórnia - EUA, utilizando o equipamento Vitek
GPS-BS ou GPS-AS. Os autores concluíram que esta identificação pode apontar falsa
sensibilidade e resistência, sendo, portanto, necessário acrescentar um método de confirmação
para os resultados obtidos. Propuseram, então, o uso da reação de polimerase em cadeia
(PCR) para a identificação da presença ou não do gene mecA (confere resistência à oxacilina; ou apresenta uma linha de suscetibilidade), como técnica de padrão ouro para a identificação
de MRSA (RIBEIRO et al., 1999). Ressalta-se, porém, que o acesso à tecnologia baseada em
reações de PCR ainda tem sido restrito no Brasil, embora esteja reconhecida sua importância
para o diagnóstico preciso de infecções por patógenos multi-resistentes.
Com o objetivo de monitorar infecções nosocomiais e algumas infecções adquiridas
em comunidades, o Programa SENTRY (SENTRY Antimicrobial Resistance Surveillance Program), iniciado em 1997, promove levantamentos epidemiológicos em diversos países, para um monitoramento de hospitais igualmente distribuídos geograficamente, e ainda, com
as mesmas proporções de dimensão e atendimentos. No Brasil, em 1997 e 1998, foram
avaliados hospitais do Rio de Janeiro, São Paulo e Florianópolis (SADER et al., 2001). Até
2001, também faziam parte do Programa, hospitais de Porto Alegre e Brasília, sendo nesta
capital, o HBDF a entidade hospitalar selecionada. Os dados coletados no período de 1997 a
2001 demonstraram que a resistência para microrganismos gram-negativos na América Latina
foi bem mais elevada do que quando comparada a outras regiões do mundo, principalmente
América do Norte e Europa. Os maiores níveis de resistência estão associados a bacilos
gram-negativos não-fermentativos multi-resistentes, como Acinetobacter spp. e P. aeruginosa, tendo sido esta o terceiro patógeno isolado com maior frequência de infecções gerais e o
quinto de infecções sangüíneas (SADER et al., 2001; SADER et al., 2004).
Vários são os mecanismos de resistência aos antimicrobianos, como baixa
permeabilidade da membrana externa, sistemas de efluxo, alteração do alvo, produção de
enzimas (beta-lactamases, enzimas inativadoras de aminoglicosídios). Muitas cepas, além de
serem inerentemente resistentes a diferentes classes de drogas, têm a habilidade de adquirir
resistência via mutações e por meio de elementos genéticos móveis, como plasmídios,
seqüências de inserção, transposons e integrons (TSUDA et al, 1989; HILALI et al, 2000).
classificadas como AmpC, que podem ser induzíveis ou constitutivas. Mecanismos
regulatórios similares aos encontrados em enterobactérias, regulam a expressão da β -lactamase AmpC em P. aeruginosa (MOROSINI et al, 2000; LIVERMORE, 2002; RIBEIRO, 2004). A indução desta enzima é efetuada por antibióticos β-lactâmicos como cefalosporinas de primeira e segunda geração (cefoxitina), os quais são fortes indutores e
hidrolizados por esta β-lactamase. As cefalosporinas de terceira geração (ceftriaxone, ceftazidima) são fracas indutoras e, apesar de serem hidrolizadas, permanecem ativas porque
há pequena quantidade destas enzimas no espaço periplasmático para hidrolizar
completamente o antimicrobiano. A hiperprodução da enzima β-lactamase envolve a mutação do gene ampD (perda do mecanismo genético repressor), cujo fenômeno resulta em alto nível
de produção de β-lactamases, independentemente da concentração, sendo por isso considerada constitutiva. Apesar da alta produção desta enzima, essas cepas de P. aeruginosa
mantêm sensibilidade aos carbapenens, embora sejam estes fortes indutores desta enzima
(RIBEIRO, 2004).
Além disso, a presença de bombas de efluxo, que removem β-lactâmicos, cloranfenicol, fluoroquinolonas, novobiocina, sulfonamidas, tetraciclina, trimetroprim, além
de detergentes e corantes, aumenta expressivamente a resistência destas bactérias.
2.1. Permeabilidade da membrana e sistemas de efluxo
Segundo Vila e Marco (2002), os principais fatores que dificultam o êxito do
tratamento para infecções causadas por P. aeruginosa são: elevada resistência intrínseca a múltiplos antibióticos, reduzindo as possibilidades terapêuticas; e capacidade de adquirir
novos mecanismos de resistência, por meio de mutações. Embora a baixa permeabilidade da
membrana externa seja um mecanismo importante para a resistência intrínseca, o fator
relevante, talvez, seja a presença de bombas de efluxo, principalmente MexAB-OprM, com
capacidade para expulsar antibióticos: betalactâmicos, tetraciclina, clorafenicol, macrolídios,
fluoroquinonas, sulfonamidas e trimetoprim (VILA e MARCO, 2002; JO et al., 2003).
Os sistemas de efluxo apresentam importante papel na resistência intrínseca e na
componentes: uma membrana interna de regulação da divisão celular (RND), com a função
presumida de transportadora; um canal trimérico formando um poro na membrana externa; e
uma proteína periplasmática ligadora, a qual promove a interação entre o RND pump e o canal de efluxo, também chamada de porina (MASUDA et al., 2000b; JO et al., 2003).
A mais freqüente das diferentes porinas, que se encontram na membrana externa de P. aeruginosa, é a porina OprF, a qual é provavelmente utilizada pela maioria dos betalactâmicos para acessar o interior da bactéria (LIVERMORE, 2002; VILA e MARCO,
2002). As porinas OprC e OprE são canais inespecíficos, já que são empregados por vários
tipos de substratos, entre eles, alguns antibióticos. Uma quarta porina, a OprD, é utilizada
especificamente pelos carbapenens (VILA e MARCO, 2002; CRESPO et al., 2004). Uma
redução na expressão de OprF tem um escasso efeito sobre a Concentração Inibitória Mínima
(MIC) dos antibióticos β-lactâmicos, mas uma perda expressiva de OprD gera resistência a imipenem, em razão deste antibiótico utilizá-la para atravessar a membrana externa (TRIAS e
NIKAIDO, 1990; VILA e MARCO, 2002), e uma diminuição da sensibilidade ao
meropenem, sem afetar outros betalactâmicos (VILA e MARCO, 2002).
Segundo Sokol et al. (1994), proteínas de membrana externa são de particular interesse
em P. aeruginosa devido ao seu envolvimento no transporte de antibióticos, no transporte de fatores de virulência extracelulares e na ancoragem de estruturas que medeiam a adesão e
motilidade. Cerca de 150 genes codificam estas proteínas, um número desproporcional
quando comparados a outros genomas.
Têm sido descritos vários sistemas de bombas de efluxo em P. aeruginosa: o sistema MexAB-OprM, largamente distribuído nas células e associado tanto à resistência intrínseca,
quanto adquirida; o MexCD-OprJ e o MexEF-OprN, que são homólogos ao sistema
MexAB-OprM, mas não distribuídos como este, estando mais associados à resistência adquirida; e o
MexXY-OprM, o qual promove a extrusão de aminoglicosídios, tetraciclinas, eritromicina e
de alguns β-lactâmicos (AIRES et al., 1999; MASUDA et al., 2000b; VILA e MARCO,
2002). Em geral estes sistemas contribuem em maior ou menor escala no aumento da MIC de
carboxipenicilinas, ureidopenicilinas, cefalosporinas, monobactamas, fluoroquinolonas e, em
algumas ocasiões, aminoglicosídios. O sistema MexEF-OprN é regulado pelo gene nfxC, que por sua vez regula a OprD, causando uma redução em sua expressão, o que gera um aumento
Aendekerk et al. (2002) caracterizaram um novo sistema de efluxo, o qual
denominaram MexGHI-OpmD, em razão do mutante obtido apresentar três genes similares
aos sistemas de efluxo de P. aeruginosa, incluindo uma proteína de efluxo, uma proteína de fusão de membrana e uma porina de membrana externa. Os autores demonstraram que este
novo sistema confere resistência ao vanádio, substância com efeito bacteriostático sobre P. aeruginosa (SOARES, 2001), especialmente em condições de limitação de ferro (AENDEKERK et al, 2002).
2.2. Resistência a Antibióticos β-lactâmicos
Os antibióticos β-lactâmicos são agentes bactericidas de baixa toxicidade e largo espectro, que podem se apresentar como penicilinas bi-cíclicas (penams, penems,
carbapenems, oxapenams), cefalosporinas (cephems, cephamycins, oxacephems) ou
monobactâmicos mono-cíclicos. A suscetibilidade “natural” aos β-lactâmicos varia largamente entre as diferentes espécies bacterianas e está refletida na combinação de efeitos a
alvos ligantes, como proteínas ligadoras de penicilinas (penicilin-binding proteins - PBP), à estabilidade às β-lactamases, e em bactérias gram-negativas, à permeabilidade da membrana externa. De forma similar, a resistência a esta classe antibiótica reflete uma mudança em
qualquer destes três componentes. Nos últimos 50 anos, a resistência aos β-lactâmicos tem aumentado expressivamente, refletindo o extensivo uso desta classe de componentes
(GEORPAPADAKOU, 1993).
A espécie P. aeruginosa produz uma betalactamase cromossômica induzível tipo AmpC (β-lactamase induzível da classe C de Ambler) similar à encontrada em algumas enterobactérias (SOARES, 2001; LIVERMORE, 2002; VILA e MARCO, 2002; RIBEIRO,
2004). As ureidopenicilinas, cefalosporinas antipseudomônicas, monobactamas e
carbapenemas se mantêm ativas frente a cepas com produção basal de AmpC. A desrepressão
parcial ou total da enzima por mutação (ampD) leva a um aumento da MIC, havendo resistência a ticarcilina, piperaciclina, aztreonam, ceftazidima e cefepima.
(Pseudomonas specific enzyme) e, em menor freqüência, as betalactamases do tipo TEM e OXA. Caracterizam-se por hidrolizar ticarcilina e piperaciclina, esta última se vê menos
afetada e mantém atividade de ceftazidima, cefepime, aztreonam e carbapenens. Nos últimos
anos, têm aparecido novas betalactamases com um espectro muito mais amplo. A emergência
de metalo-β-lactamases (MβL) em patógenos clínicos, podendo conferir resistência aos antibióticos β-lactâmicos foi descrita no início dos anos 90 no Japão, sendo atualmente um problema de magnitude global. Giske et al. (2006) avaliaram amostras de P. aeruginosa
isoladas de pacientes da Grécia, Hungria, Itália e Suécia, com o propósito de obter um melhor
entendimento da epidemiologia internacional de amostras produtoras de MβL.
A enzima PER-1, de codificação plasmídica ou cromossômica, tem sido descrita na
Turquia (VAHABOGLU et al., 1990), França (MUGNIER et al, 1996), Itália (LUZZARO et
al., 2001; ENDIMIANI et al., 2006), etc. Esta enzima consiste em uma betalactamase de
classe A, que pode ser inibida pelo ácido clavulânico e pelas ceftazidima, cefepima,
aztreonam e ticarcilina; não conferindo resistência a piperaciclina, nem a carbapenens. As β -lactamases do tipo OXA, pertencentes à classe D de Ambler (AUBERT et al, 2001), conferem
resistência a ticarcilina, piperaciclina, ceftazidima, cefepima e aztreonam, mas não a
carbapenens (VILA e MARCO, 2002). São também encontradas na Turquia, e em menor
freqüência, na França, incluindo as OXA-11, 14, 16, 17, 19 e 28 (codificadas por integrons de localização plasmídica ou cromossômica), as quais são mutantes das OXA-10 e OXA-15,
enzimas plasmídicas, que por sua vez são derivadas da OXA–2.
Têm sido descritas carbapenases do tipo metalo-β-lactamases (MβL) que hidrolisam rapidamente carboxipenicilinas, ureidopenicilinas, ceftazidima, cefepima e carbapenens.
Entretanto, o aztreonam se mantém estável e conserva sua atividade. Estas enzimas são da
classe B, codificadas em integrons, os quais são inibidos pelo ácido etilenodiaminotetracético (EDTA). A enzima IMP-1 tem sido descrita principalmente no Japão, embora recentemente
tenha havido casos em outros países, a exemplo de Singapura e do Reino Unido (ARAKAWA
et al., 2000). Uma variante desta enzima, a IMP-7, foi descrita no Canadá (VILA e MARCO,
2002) e um outro tipo com estas características são as denominadas VIM, encontradas na
Itália (VIM-1) (GISKE et al., 2006; LAGATOLLA et al., 2006); França, Grécia e Koréia
O surgimento de espécies de bactérias gram-negativas resistentes a antibióticos β -lactâmicos de largo-espectro tem se tornado um problema clínico mundial. No Japão,
amostras resistentes aos carbepenens, sendo algumas produtoras da metalo-β-lactamase IMP-1, tendem a demonstrar resistência a cefalosporinas, cefamicinas e carbapenens. Arakawa et
al. (2000) propuseram, então, um método simples de difusão de disco, utilizando sais de
metais pesados e EDTA, para detectar este tipo de bactéria. Estes autores concluíram que o
método apresentou especificidade e sensibilidade comparável a análises de PCR, resultando
em uma ferramenta de rastreamento de menor custo, podendo ser disponibilizada para testes
clínicos de laboratórios (ARAKAWA et al., 2000).
A aquisição de β-lactamases, principalmente de PSE-1 e PSE-4, pode conferir resistência aos carbapenens, às cefalosporinas e aos monobactâmicos. A PER-1, uma β -lactamase de classe A, confere alta resistência a ceftazidime, e tem sido freqüentemente
relatada na Turquia (VAHABOGLU et al., 1990).
Zanetti et al. (2003) realizaram estudo comparativo entre monoterapia com cefepime e
a terapêutica com imipenem-cilastatina em pacientes acometidos por pneumonia,
provenientes de Unidades de Terapia Intensiva (UTI) de seis países europeus. Embora os
autores tenham evidenciado eficácia clínica similar para os dois tipos de terapêutica,
verificaram também que houve maior número de cepas de P. aeruginosa resistentes ao imipenem-cilastatina do que ao cefepime. Entretanto, os autores ressaltaram a ocorrência de
toxicidade renal como efeito colateral da monoterapia proposta.
Estas amostras altamente resistentes também estão emergindo na América, onde o
Programa de Vigilância SENTRY de dados da América Latina, em 2002, indicou a presença
de 18% de amostras de P. aeruginosa e Acinetobacter spp. resistentes aos carbapenens, sendo que 2% das P. aeruginosa eram produtoras de MβL (CRESPO et al., 2004). O Programa de 1999-2002 identificou estas amostras na Argentina, Chile, Brasil e Venezuela, exceto na
Colômbia (CRESPO et al, 2004). Recentemente, foi descrita uma nova subclasse de MβL, a SPM-1, a qual foi identificada em hospital terciário brasileiro, em São Paulo, a partir de
paciente da pediatria acometido por infecção do trato urinário (GALES et al., 2003; CRESPO
No período de janeiro de 2001 a dezembro de 2003, 1186 amostras de P. aeruginosa
foram coletadas pelo Programa SENTRY na América Latina, tendo sido 54 isolados
resistentes aos antibióticos β-lactâmicos, imipenem, meropenem e ceftazidime. Mais de 70% destes isolados foram provenientes de um mesmo centro médico da cidade de São Paulo. Este
programa de monitoramento tem detectado e caracterizado diversas novas enzimas MBL
nesta fase de surgimento de bacilos gram-negativos não-fermentadores multi-resistentes,
inferindo a possibilidade de que a disseminação de genes MBL pode representar o fator mais
importante no aumento da resistência aos carbapenems.
2.2. Resistência aos Carbapenens
Os Carbapenens agem pela inibição da síntese da parede celular e representam uma
modificação da estrutura do anel β-lactâmico. Devido ao seu largo espectro de atividade, à sua estabilidade e à hidrólise da maioria das β-lactamases, os carbapenens têm sido considerados medicamentos de escolha para o tratamento de infecções causadas por bactérias
gram-negativas resistentes às cefalosporinas (GOODMAN e GILMAN, 1991; TORTORA et
al., 2002).
Entretanto, estudo do SENTRY Antimicrobial Surveillance Program realizado em centros médicos da América Latina, no período entre 1997 e 2001, com a participação da
Argentina, Brasil, Chile, Colômbia, México, Uruguai e Venezuela, demonstrou que o
aumento da resistência aos antibióticos comumente utilizados é o maior problema terapêutico
desta região. Os carbapenens imipenem (IPM) e meropenem (MEM) são usualmente ativos
contra isolados de P. aeruginosa multi-resistentes. Todavia, a resistência a estes antimicrobianos tem sido enfatizada como um problema terapêutico (ANDRADE et al., 2003;
GALES et al., 2003; MENDES et al., 2004; SADER et al., 2005).
Os mecanismos responsáveis pela resistência de P. aeruginosa aos carbapenens incluem: redução da permeabilidade da membrana externa, em razão de mutação de porinas;
dependentes de zinco (MENDES et al., 2004; SADER et al., 2005a; SADER et al., 2005b;
REINHARDT et al., 2007).
Cardoso, Alves e Leitão (2008) relataram a presença de MBL VIM-2 em pacientes da
pediatria de hospital referência para monitoramento de fibrose cística, em Coimbra –
Portugal, confirmando a disseminação de clones multi-resistentes. Os autores evidenciaram
que das 15 amostras avaliadas, oito foram resistentes aos carbapenems, tendo sido também
positivas para a presença de MBL.
Carmeli et al. (1998) encontraram resistência aumentada, em duas vezes, do risco de
resistência ao imipenem (IPM), quando os resultados foram comparados com ciprofloxacim
(CIP), ceftazidime (CAZ) ou piperaciclina (TZP). No entanto, a combinação de terapias
poderia ser uma tentativa de prevenir a seleção de resistência mutacional, embora as
evidências ainda sejam escassas (LIVERMORE, 2002).
Gales et al. (2003), ao avaliarem 43 amostras de P. aeruginosa isoladas de pacientes de diversos hospitais brasileiros, dentre eles o HBDF, evidenciaram a ocorrência do clone SP
resistente a carbepenens. Os autores concluíram que, entre hospitais próximos, a transferência
do clone poderia ocorrer em razão do transporte de pacientes ou pelo próprio corpo clínico.
Entretanto, o mesmo clone foi também identificado em hospitais distantes, o que pode indicar
uma pressão seletiva em razão das terapias antimicrobianas utilizadas (GALES et al., 2003;
SADER et al., 2005).
Embora, tenha sido demonstrado o surgimento de resistência de P. aeruginosa ao imipenem (ANDRADE et al., 2003) e a outros antibióticos, medidas do controle de qualidade
são necessárias para prevenir resultados relacionados a falsa-resistência, devendo ser
observadas estritamente as condições laboratoriais de avaliação (CARMELI et al., 1998). A
perda da porina OprD, que forma canais na transmembrana, gera resistência ao imipenem e
reduz a suscetibilidade ao meropenem (LIVERMORE, 2002).
Sader et al. (2005) realizaram estudo com 183 amostras obtidas dos pacientes de
hospital terciário de São Paulo, para os quais foi inicialmente realizada a testagem para
resistência aos diversos antibióticos por meio da técnica de difusão de disco. As amostras
MβL. Os autores concluíram que os isolados de P. aeruginosa obtidos apresentaram alto grau de resistência à maioria dos agentes antimicrobianos utilizados, sendo que somente as
polimixinas (PB) demostraram consistente atividade in-vitro.
2.4. Resistência a Aminoglicosídios
Aminoglicosídios são antibióticos que, considerando sua estrutura química e
mecanismo de ação, estão entre aqueles que se ligam à subunidade ribossômica 30S e alteram
a síntese protéica, podendo causar a morte celular. Em razão de sua nefrotoxicidade e
ototoxicidade, os aminoglicosídios têm tido seu uso restrito. A neomicina e a gentamicina
(CN) são exemplos de antibióticos desta classe de inibidores da síntese protéica (GOODMAN
e GILMAN, 1991; TORTORA et al., 2002).
Os mecanismos mais importantes em P. aeruginosa relacionados à resistência aos aminoglicosídios são: inativação por enzimas modificantes, alterações na permeabilidade; e
eliminação por bombas de efluxo. As enzimas mais freqüentes em P. aeruginosa são: uma nucleotidiltransferase [ANT (2’’)-I], que confere resistência a gentamicina, tobramicina,
dibecacina e canamicina; e uma acetiltransferase [AAC (6’) – II], cujo substrato é a
gentamicina, a tobramicina e a netilmicina, ressaltando a freqüente combinação de diversas
enzimas. Alterações na permeabilidade comportam resistência a todos os aminoglicosídios e,
junto com as enzimas modificantes, constituem os mecanismos de resistência mais habituais.
Entretanto, as bases moleculares da resistência a aminoglicosídios são pouco conhecidas
(VILA e MARCO, 2002).
Três grandes famílias de proteínas da membrana externa foram relatadas a partir do
seqüenciamento genômico de P. aeruginosa realizado por Stover et al. (2000), os quais concluíram ter esta espécie apresentado o maior genoma bacteriano contido em um único
cromossomo circular (6,3Mbp), sequenciado até aquele momento. Os autores identificaram
também a presença de 14 homólogos da porina OprM, desconhecidos até aquele momento. A partir desse seqüenciamento genômico, Jo et al. (2003) demonstraram que três proteínas
(OpmG, OpmH e OpmI) influenciaram a suscetibilidade a aminoglicosídios, sendo que a
ruptura da OpmG afetou principalmente a suscetibilidade à gentamicina e a ruptura da OpmI,
apresentou um menor efeito na suscetibilidade aos antibióticos citados. Jo et al. (2003)
evidenciaram, ainda, que cada uma destas proteínas tem um efeito diferente e uma função
específica quanto aos aminoglicosídios, podendo ainda apresentar uma sobreposição em suas
ações.
Os aminoglicosídios são uma classe de antibióticos anfipáticos altamente catiônicos
que tem sido uma alternativa para microrganismos multi-resistentes. Dentre os sistemas de
efluxo descritos, somente o sistema MexX-MexY de P. aeruginosa tem sido associado à resistência aos aminoglicosídios (AIRES et al, 1999; MASUDA et al, 2000; LIVERMORE,
2002; JO et al., 2003; REINHARDT et al., 2007).
Aires et al. (1999), analisando sistemas de efluxo, concluíram que o sistema
MexAB-OprM confere, clinicamente, relevante resistência aos antibióticos β-lactâmicos, enquanto a super-expressão do sistema XY pode ser responsável pelos altos níveis de resistência aos
aminoglicosídios e às fluoroquinolonas, em investigações clínicas. Reinhardt et al. (2007)
ressaltaram a possibilidade de desenvolvimento de resistência aos aminiglicosídios por sua
inativação ou por sua extrusão pelos sistemas de efluxo MexXY. Entretanto, os autores
observaram um aumento de 20 vezes da expressão de 2 entre 11 isolados resistentes a este
antibiótico, em que não havia nenhuma correlação com a expressão desta bomba de efluxo,
sugerindo a existência de determinantes de resistência ainda desconhecidos.
A metilação de rRNA 16S tem surgido como mais um mecanismo de resistência aos
aminoglicosídios. Cinco metilases têm sido descritas: ArmA, RmtA, RmtB, RmtC, RmtD.
Doi et al. (2007) realizaram estudo em 45 pacientes de sete hospitais do Estado de São Paulo,
coletando 51 amostras resistentes ao imipenem, das quais 57% produziam MBL SPM-1 e
75% RmtD e 51% dos isolados produziram ambas enzimas, sugerindo que esta co-produção
pode ser freqüente na área geográfica estudada.
O aztreonam (ATM) foi o primeiro antibiótico monobactâmico liberado para uso nos
Estados Unidos, sendo ativo contra uma larga variedade de bactérias gram-negativas
aeróbicas e facultativas. Quando comparado aos aminoglicosídios, DeMaria et al. (1989)
demonstraram eficácia semelhante ao tratamento de pacientes com infecção por bacilos
gram-negativos, embora a concentração sérica do aztreonam tenha sido mais elevada, não houve
2.5. Resistência a Fluoroquinolonas
As quinolonas exercem efeito bactericida por inibição seletiva de uma enzima (DNA
girase) necessária para a replicação do DNA. Apesar de o ácido nalidíxico possuir um uso
limitado para infecções do trato urinário, na década de 80, foi desenvolvido um grupo de
antibióticos de largo espectro, as fluoroquinonas, as quais penetram muito bem nos tecidos,
sendo geralmente seguras para adultos, mas tendo seu uso limitado para crianças,
adolescentes e mulheres grávidas, em razão de possível alteração do desenvolvimento da
cartilagem (TORTORA et al., 2002).
As fluoroquinolonas são utilizadas freqüentemente em clínica médica para o
tratamento de infecções do trato respiratório e urinário (SOARES, 2001). Como ocorre com
as enterobactérias, a resistência de P. aeruginosa a fluoroquinolonas pode apresentar-se a partir de alterações protéicas (mutações na DNA girase e na topoisomerase IV), modificações
na permeabilidade ou sobre-expressão de bombas de efluxo. Têm sido descritas mutações do
gene gyrA, que codifica a subunidade A da DNA girase, alterando os aminoácidos situados na região NH2 terminal da proteína (SOARES, 2001; GILBERT et al., 2001; VILA e MARCO,
2002). A mudança em um único aminoácido já causaria uma resistência moderada às
fluoroquinolonas e uma dupla mutação do gene gyrA e do gene parC (subunidade A da topoisomerase IV) seria responsável por um elevado grau de resistência. As modificações das
porinas (quinolonas hidrófilas) ou em um lipopolissacarídio (quinolonas hidrófobas)
contribuem para reduzir a entrada das fluoroquinolonas no interior da célula, o que se traduz
em um aumento da resistência. A sobre-exposição das bombas de efluxo que ocorre com β -lactâmicos também provoca um aumento da resistência às fluoroquinolonas (VILA e
MARCO, 2002).
O uso do ciprofloxacino (CIP) por via oral, uma quinolona de ácido carboxílico pouco
nefrotóxica quando comparada aos aminoglicosídios, apresentou bons níveis séricos em
estudo realizado por Eron et al. (1985). Giamarellou et al. (2000) compararam a terapêutica
usualmente de escolha para pacientes com episódios de neutropenia febril, administração de
ceftazidime (CAZ) associada à amicacina (AK), à administração de ciprofloxacina (CIP)
menor dosagem. A justificativa para esta avaliação constituiu-se principalmente pela
nefrotoxicidade e ototoxicidade bem documentada dos aminiglicosídios e pela busca de
eficácia por meio da monoterapia.
Os referidos autores ainda concluíram que o procedimento utilizado foi pelo menos tão
efetivo quanto a terapia com ceftazidime associada a amicacina em pacientes com infecção
por P. aeruginosa. Entretanto, concluíram também que a seleção da antibioticoterapia adequada deveria estar associada à avaliação da presença de cepas multi-resistentes a
antibióticos específicos em determinados centros de atendimento, principalmente terciários
(GIAMARELLOU et al. 2000).
Gilbert et al. (2001) avaliaram a resistência fenotípica de sete espécies bacterianas,
dentre elas P. aeruginosa, para as fluoroquinonas ciprofloxacim (CIP), levofloxacim e trovafloxacim, demonstrando o aparecimento de resistência a estas drogas, embora tenha
havido diferenças entre as espécies estudadas. Para P. aeruginosa foram identificadas mais amostras mutantes para trovafloxacim do que para ciprofloxacina (CIP). Os autores
concluíram que embora a resistência fenotípica não denote uma referência específica aos
mecanismos genotípicos, estudos de características fenotípicas poderiam complementar
abordagens genotípicas, possibilitando melhor entendimento da dinâmica de populações
bacterianas.
2.6. Resistência aos Macrolídios
A antibioticoterapia em pacientes com fibrose cística acometidos por infecções
pulmonares por P. aeruginosa tem aumentado sua expectativa de vida. Nos últimos 10 anos, o uso da azitromicina para o controle da infecção e modulação da inflamação tem sido uma
nova abordagem. Entretanto, este antimicrobiano da classe dos macrolídios, os quais agem
pela inibição da síntese protéica, ainda tem seu mecanismo de ação pouco conhecido
3. VIRULÊNCIA
Os mecanismos envolvidos na virulência (capacidade de um microrganismo de causar
danos ao hospedeiro) de bactérias patogênicas e os que determinam a resistência de
determinada cepa bacteriana aos antibióticos são tópicos importantes e amplamente estudados
em microbiologia clínica (MOROSINI et al., 2000). Muito embora P. aeruginosa seja mais conhecida por sua alta taxa de resistência aos antibióticos do que por sua capacidade
virulenta, apresentando-se geralmente como patógeno oportunista, esta espécie bacteriana tem
causado infecções com freqüência cada vez maior. Assim, a baixa virulência da P. aeruginosa
é efetivamente compensada por uma alta taxa de resistência a antibióticos, combinação que
tem se mostrado mortal em muitos casos, sendo uma preocupação extrema em ambientes
hospitalares.
Dentre os fatores celulares que determinam a virulência de P. aeruginosa destacam-se: pilli; apêndices superficiais, que promovem a aderência do microrganismo a receptores de
gangliosídeo GM-1 presentes na superfície das células epiteliais do hospedeiro; flagelos, que
também participam da aderência (SOARES, 2005); e a endotoxina LPS (Lipolissacarídeo da
Parede Celular), responsável pela síndrome séptica, que ocorre em razão da ativação e
liberação de mediadores de vasodilatação, como interleucina 1 e fator de necrose tumoral, da
ativação do Complemento e da deflagração de coagulação intravascular disseminada, dentre
outros mecanismos (BONE, 1993).
Fatores extracelulares, em geral regulados por estímulos ambientais (RAHME et al.,
2000), também estão relacionados à virulência de P. aeruginosa, como: produção de alginato, polissacarídeo capsular que permite a aderência às superfícies epiteliais pulmonares com
formação de grandes conglomerados bacterianos, biofilme (HEAD e YU, 2004; FILITRAUT,
2006); produção de enzimas, como a elastase, que degrada imunoglobulinas e componentes
do Complemento, e como a exoenzima S, que inibe síntese protéica e tem função de adesina;
leucocidina, que inibe função de neutrófilos e linfócitos; piocianina, que impede o
crescimento de outras bactérias e inibe a atividade ciliar da mucosa respiratória; toxinas,
como a exotoxina A, a qual promove a destruição tecidual, inibindo a síntese protéica
É importante ressaltar que a expressão de exoprodutos obtidos em estudos in vitro
pode diferir significativamente da expressão a partir de infecções in vivo ou durante a co-cultivação em culturas celulares/tecidos (SAWA et al., 1998). Sawa et al. (1998) sugeriram
que a citotoxicidade induzida pela densidade pode ser resultante da expressão de
determinantes de virulência coordenadamente regulados pelos sistemas QS (quorum-sensing system). Esta comunicação inter-bacteriana, também chamada “quorum-sensing” (QS), é um mecanismo muito sofisticado pelo qual as moléculas agem como auto-indutoras de uma
variedade de funções biológicas, controlando não apenas a produção de fatores de virulência,
como também a formação de biofilme (NALCA et al., 2006). A presença ou ausência de um
simples gene ou de fatores que controlam os genes de virulência pode determinar se uma
amostra de P. aeruginosa será invasiva ou lítica para células epiteliais ou endoteliais in vitro
(TÜMMLER e KIEWITZ, 1999).
Em pacientes fibrocísticos, a produção do exopolissacarídio alginato, por meio de um
processo que consome energia pela conversão de fontes de carbono, evidenciando uma
necessidade de produção in vivo, tem sido apontada como a principal alteração fenotípica de
P. aeruginosa, permitindo inferir que o ambiente hostil dos pulmões destes pacientes induz não apenas mutações desta espécie bacteriana, mas também seleciona clones mutantes mais
aptos à sobrevivência naquele nicho específico (SILO-SUH et al., 2005). Embora esta
conversão fenotípica de não-mucóide para mucóide seja mais frequente em pacientes com
fibrose cística, ela também ocorre em indivíduos com infecções crônicas associadas a P. aeruginosa. O fator desencadeador desta mudança para mucóide pode ser a redução de nutrientes e/ou de energia (SOKOL et al., 1994). Sokol et al. (1994) ressaltaram também a
possibilidade de ocorrer um rearranjo do DNA cromossomal bacteriano, associado à
modificação fenotípica.
Os fatores de virulência são, por definição, requeridos para uma infecção aguda.
Entretanto, o sistema imune ao reconhecer os referidos fatores elimina células que os
possuem, selecionando dessa forma, células que apresentam mutações nestes fatores de
virulência e em seus reguladores (SMITH et al., 2006). Segundo Nguyen e Singh (2006), o
conceito de virulência é impreciso, pois “fatores de virulência” são necessários para a