Pró-Reitoria de Pós-Graduação e Pesquisa
Stricto Sensu em Ciências Genômicas e
Biotecnologia
Trabalho de Conclusão de Dissertação
EFEITO DE CANDIDATOS A SUPRESSORES DE
SILENCIAMENTO GÊNICO VIRAL EM EXPRESSÃO
DE PROTEÍNA RECOMBINANTE EM PLANTAS.
Autora: Raquel Medeiros Vasques Bonnet
Orientador: Prof. Dr.Tatsuya Nagata
Co-orientador: Dr. Cristiano Lacorte
RAQUEL MEDEIROS VASQUES BONNET
EFEITO DE CANDIDATOS A SUPRESSORES DE SILENCIAMENTO
GÊNICO VIRAL EM EXPRESSÃO DE PROTEÍNA RECOMBINANTE
EM PLANTAS.
Dissertação apresentada ao Programa de Pós-Graduação Stricto Sensu em Ciências Genômicas e Biotecnologia da Universidade Católica de Brasília, como requisito parcial para obtenção do Título de Mestre em Ciências Genômicas e Biotecnologia.
Orientador: Prof. Dr. Tatsuya Nagata Co-orientador: Prof. Dr. Cristiano Lacorte
Ficha elaborada pela Biblioteca Pós-Graduação da UCB
B717e Bonnet, Raquel Medeiros Vasques
Efeito de candidatos a supressores de silenciamento gênico viral em expressão de proteína recombinante em plantas. / Raquel Medeiros Vasques Bonnet – 2010.
60f.; il.: 30 cm
Dissertação (mestrado) – Universidade Católica de Brasília, 2010.
Orientação: Tatsuya Nagata Co-Orientação: Cristiano Lacorte
1. Virologia. 2. Biologia experimental. I. Nagata, Tatsuya, orient. II. Lacorte, Cristiano, co-orient. III. Título.
Dissertação da autoria de Raquel Medeiros Vasques Bonnet, intitulada “EFEITO DE CANDIDATOS A SUPRESSORES DE SILENCIAMENTO GÊNICO VIRAL EM EXPRESSÃO DE PROTEÍNA RECOMBINANTE EM PLANTAS”, apresentada como requisito parcial para obtenção do grau de Mestre em Ciências Genômicas e Biotecnologia da Universidade Católica de Brasília, em 13 de Dezembro de 2010, defendida e aprovada pela banca examinadora abaixo assinada:
Prof. Dr. Tatsuya Nagata Orientador
Biologia Molecular - UnB
Dr. Cristiano Lacorte Co-orientador Embrapa - Cenargen
Dr. Francisco José Lima Aragão Embrapa - Cenargen
Profª Drª Paula Andréia Silva
Ciências Genômica e Biotecnologia - UCB
AGRADECIMENTOS
Ao meu orientador Dr. Tatsuya Nagata pela oportunidade oferecida, pela confiança, atenção e paciência, pelo exemplo de profissionalismo e pela autonomia concedida, jamais esquecerei.
Ao meu co-orientador Dr. Cristiano Lacorte pelo conhecimento compartilhado, por todas as sugestões e por me fazer enxergar as “mini-preps” com outros olhos.
À minha colega, conterrânea, confidente e amiga Franciele, que foi a primeira pessoa a me ajudar, pelos medos e sonhos compartilhados, que sempre me deu força e tem sido uma grande parceira em aprendizados e trabalhos todos esses anos.
Às amigas que passaram mais tempo ao meu lado, Kelly, Shelly, Débora, Sônia Cris, Isabel, pelos cafés e risadas, choppinhos e gargalhadas, micheladas e guacamoles, vocês são inesquecíveis.
A todos os meus colegas e amigos dos laboratórios da UCB, CENARGEN e UnB pela credibilidade, pelas palavras de incentivo. Este tempo com vocês foi maravilhoso!
Ao meu marido querido Werner pelas palavras de apoio, por sempre acreditar que eu conseguiria e que muitas vezes precisou suportar meu mau-humor nos momentos “não tão bons”, as inúmeras horas na frente do computador ou os finais de semana no laboratório.
Aos professores, em especial à Profª Eliane Noronha, Prof. Dario Grattapaglia, Prof. Márcio Elias Ferreira, Profª Betânia Quirino, Profª Paula Andréia, pela contribuição na minha formação e no enriquecimento deste trabalho.
Agradeço aos meus pais, Suzana e Ricardo meus primeiros incentivadores, por me ensinarem que a realização dos sonhos mais distantes é possível, mesmo a distância sempre muito presentes, o amor e compreenção de vocês foram muito importantes para mim.
À coordenação e funcionários do Programa de Ciências Genômicas e Biotecnologia, por proporcionar as condições necessárias para a execução de todas as atividades desenvolvidas no mestrado.
À FAPDF por financiar parte desse sonho.
“Nas grandes batalhas da vida, o primeiro passo para a vitória é o desejo de vencer!”
RESUMO
REFERÊNCIA: VASQUES, Raquel. Efeito de candidatos a supressores de silenciamento gênico viral em expressão de proteína recombinante em plantas.
2010. 60 Fls. Mestrado em Ciências Genômicas e Biotecnologia. Universidade Católica de Brasília, Brasília, 2010.
O objetivo deste trabalho foi aumentar a eficiência do sistema de expressão transiente de proteínas heterólogas utilizando supressores de silenciamento gênico viral e Agrobacterium de maneira integrada. O uso de plantas como biorreator é uma
estratégia promissora para a produção em larga escala de proteínas heterólogas. Entretanto, o uso de plantas geneticamente modificadas pode levar a um baixo nível de expressão de proteína, um dos principais motivos é o forte sistema de degradação de mRNA do transgene, conhecido como o Silenciamento Gênico Pós-transcricional (PTGS). PTGS é um mecanismo de defesa contra vírus de RNA e retrotransposons em células eucarióticas, no entanto, este sistema é geralmente contra-atacado por supressores de silenciamento gênico, produzidos pelos vírus durante o processo de infecção. Para superar o baixo nível de expressão de proteínas recombinantes em plantas, a estratégia de coexpressão da proteína alvo com supressores de PTGS foi considerada. Para avaliar a utilidade desta estratégia, um gene repórter gfp, da Green Fluorescent Protein (GFP) foi clonado em vetor
binário para Agrobacterium tumefaciens transient expression (ATTE) e coinfiltrado
com três candidatos a supressores de PTGS separadamente, 126K de Pepper mild
mottle virus (Tobamovirus), HC-Pro de Brugmansia suaveolens mottle virus
(Potyvirus) e AC2 de Tomato severe rugose virus (Begomovirus). Os resultados
apontam para a viabilidade da estratégia, demonstrando que a coexpressão da proteína alvo com candidatos a supressores de silenciamento leva a um aumento na expressão da proteína heteróloga em questão.
ABSTRACT
The aim of this work was to increase the efficiency of transient heterolog protein expression system using viral gene-silencing suppressors and Agrobacterium
integrated. The use of plants as bioreactor is a promising strategy for large scale production of the heterolog protein. However, the use of genetically modified plants may lead to low protein expression due to the strong system of degradation of the transgene mRNA, known as the Post-Transcriptional Gene Silencing (PTGS). PTGS is a defense mechanism against RNA viruses and retrotransposons in eukaryotic cells, however, this system is often counter-attacked by gene virus silencing suppressors during infections. To overcome the low level of expression of recombinant proteins in plants, the strategy of co-expression of the target protein with PTGS suppressors was considered. To evaluate the usefulness of this strategy, a reporter gene gfp, coding for the Green Fluorescent Protein (GFP) was cloned into
binary vector for Agrobacterium tumefaciens transient expression (ATTE) and
co-infiltred separately, with three PTGS suppressor candidates, 126K protein of Pepper
mild mottle virus (Tobamovirus), Hc-Pro of Brugmansia suaveolans mottle virus
(Potyvirus) and AC2 of Tomato severe rugose virus (Begomovirus). Our results
indicate the feasibility of the strategy, demonstrating that co-expression of the target protein with a silencing supressor candidate leads to an increase on the heterologous protein expression in question.
LISTA DE ABREVIATURAS E SIGLAS
µg: Micrograma
µl: Microlitro
AC2: Geminivirus trans-activating F protein
A. tumefaciens: Agrobacterium tumefaciens
AIMV: Alfalfa mosaic virus
ATP: Adenosina trifosfato
ATTE: Agrobacterium tumefaciens transiente expression
BsMoV: Brugmansia suaveolens mottle virus
CMV: Cucumber mosaic virus
DCL: Dicer-like
Dil: Diluição
DNA: Ácido desoxirribonucléico dpi: Dias pós infiltração
dsRNA: RNA de fita dupla
E. coli: Escherichia coli
g: Força centrífuga
GFP: Proteína fluorescente verde HC-Pro: Helper component proteinase
HIV: Human immunodeficiency virus
LB: Luria-Bertani
mA: Miliampere
MES: Ácido 2-[N-morfolino] etanossulfônico
mg: Miligrama
ml: Mililitro
mRNA: RNA mensageiro
miRNA: Micro-RNA
N. benthamiana: Nicotiana benthamiana
N. tabacum: Nicotiana tabacum
ng: Nanograma
nm: Nanômetro
ORF: Open read frame
ORSV: Odontoglossum ringspot virus
p19: Proteína 19 kDa de tombusvirus pb: Pares de bases
pH: Potencial hidrogeniônico
PBS: Solução salina fosfatada tamponada PCR: Reação em cadeia de polimerase PMMoV: Pepper mild mottle virus
PTGS: Silenciamento gênico pós-transcricional PVDF: Polyvinylidene fluoride
PVX: Potato virus X
PVY: Potato virus Y
RISC: Complexo de silenciamento gênico induzido por RNA RNA: Ácido ribonucléico
RNAi: RNA de interferência RNase III: Ribonuclease III RNA pol II: RNA polimerase II RPM: Rotações por minuto
SARS: Síndrome respiratória aguda severa
SDS-PAGE: Sodium dodecyl sulfate- polyacrylamide gel electrophoresis siRNA: short-interfering RNA
ssDNA: DNA de fita dupla T-DNA: DNA de transferência
TEMED: N,N,N’,N’,tetrametilenodiamina TEV: Tobacco etch virus
TBSV: Tomato bushy stunt virus
Ti: Indutor de tumor
TVMV: Tobacco vein mottling virus
TMV: Tobacco mosaic virus
ToSRV: Tomato severe rugose virus
ToMV: Tomato mosaic virus
Tris: Tris (hidroximetil) amino metano UV: Ultravioleta
SUMÁRIO
1 INTRODUÇÃO ... 1
1.1 SISTEMAS DE EXPRESSÃO DE PROTEÍNA RECOMBINANTE ... 1
1.2 PLANTAS COMO BIORREATOR ... 2
1.3 SILENCIAMENTO GÊNICO MEDIADO POR RNA ... 6
1.4 SUPRESSORES VIRAIS DE SILENCIAMENTO GÊNICO ... 10
1.4.1 Candidatos a supressores virais utilizados no estudo ... 12
1.5 A PROTEÍNA VERDE FLUORESCENTE ... 15
2 JUSTIFICATIVA ... 18
3 OBJETIVOS ... 20
3.1 OBJETIVO GERAL... 20
3.2 OBJETIVOS ESPECÍFICOS ... 20
4 MATERIAIS E MÉTODOS ... 21
4.1 VETOR DE CLONAGEM ... 21
4.2 AMPLIFICAÇÃO DOS GENES CANDIDATOS À SUPRESSORES DE PTGS E DO GENE REPÓRTER GFP ... 22
4.3 DIGESTÃO DE VETOR DE CLONAGEM E INSERTOS ... 23
4.4 LIGAÇÃO DOS INSERTOS EM VETOR DE CLONAGEM ... 23
4.5 TRANSFORMAÇÃO DE BACTÉRIAS E.COLI ... 24
4.6 TRANSFORMAÇÃO EM A. TUMEFACIENS E AGROINOCULAÇÃO ... 24
4.7 OBSERVAÇÃO DA FLUORESCÊNCIA DE GFP ... 26
4.8 ANÁLISE POR WESTERN BLOT ... 26
4.9 AVALIAÇÃO DE INTENSIDADE DE SINAL DA REAÇÃO DE WESTERN BLOT ... 27
5 RESULTADOS E DISCUSSÃO ... 28
5.1 CLONAGEM DOS GENES EM VETOR BINÁRIO ... 28
5.2 AGROINFILTRAÇÃO E OBSERVAÇÃO DE FLUORESCÊNCIA DA GFP ... 28
5.3 DETECÇÃO DE GFP EM PLANTAS ... 31
6 CONCLUSÕES ... 34
7 PERSPECTIVAS ... 35
8 REFERÊNCIAS BIBLIOGRÁFICAS ... 36
9 APÊNDICE ... 46
9.1 SEQUÊNCIA DE HC-PRO DE BRUGMANSIA SUAVEOLENS MOTTLE VIRUS ISOLADO BR-CAMPINAS ... 46
9.2 SEQUÊNCIA DE 126 KDA DE PEPPER MILD MOTTLE VIRUS ISOLADO DF-BR01 ... 46
9.3 SEQUÊNCIA DE AC2 DE TOMATO SEVERE RUGOSE VIRUS ISOLADO DF-BR2 ... 48
9.4 SEQUÊNCIA DE P19 DE TOMATO BUSHY STUNT VIRUS ISOLADO TO4C ... 48
1 INTRODUÇÃO
1.1 SISTEMAS DE EXPRESSÃO DE PROTEÍNA RECOMBINANTE
Inúmeras técnicas de expressão são utilizadas para produção de proteínas heterólogas, ou seja, produzidas em organismos diferentes daqueles de sua origem, dentre os quais podemos citar os que utilizam células de bactérias, de leveduras, culturas de células de insetos, de mamíferos além de plantas e animais transgênicos (RYBICKI, 2009).
O sistema procariótico Escherichia coli é o mais utilizado por permitir que uma
grande quantidade de proteínas seja expressa de maneira rápida a um custo mais baixo que os sistemas eucarióticos e por possuir a facilidade de manutenção de culturas. No entanto o processamento das proteínas de origem eucarióticas em bactéria é incompleto e limitado a estruturas simples (peptídeos e proteínas pequenas) que não necessitem da maquinaria de processamento pós-traducional ou que estas sejam desnecessárias para a atividade biológica. Por exemplo, a maioria das proteínas humanas glicosiladas usadas como terapêuticos requerem glicosilação, processo o qual as bactérias não realizam (SHARMA & SHARMA, 2009).
Outra desvantagem do sistema bacteriano é que as proteínas podem formar agregados insolúveis, chamados corpúsculos de inclusão, que possuem a função de proteger as proteínas e para solubilizá-los utilizam-se agentes desnaturantes que podem causar danos estruturais e dificultar a recuperação das cadeias polipeptídicas, além de aumentar o custo do processo (MA et al., 2003). Há também a possibilidade dessas proteínas serem tóxicas para o organismo, o que poderia resultar em baixos níveis de produção (BENZ & SCHMIDT, 2002).
Entre os sistemas que utilizam células eucarióticas, a cultura de leveduras,
Saccharomyces cerevisiae, e mais recentemente Pichia pastoris, apresentam um
Outro sistema de expressão eucariótico é o que utiliza células de inseto infectadas por baculovírus. A principal vantagem consiste na presença dos fortes promotores de genes da poliedrina P10, o que resultará em alta expressão da proteína de interesse (O’REILLYet al., 1997).
O sistema que utiliza células de mamíferos já demonstrou ser um dos sistemas mais eficiente com relação às modificações pós-traducionais da proteína recombinante, principalmente se tratando de uma glicoproteína.
Essas duas últimas técnicas apresentam algumas desvantagens como, por exemplo, a manutenção das células com meios de cultura bastante complexos e uso de reatores sofisticados, o que vem a onerar muito a produção. Além disso, apresenta muitas vezes baixo rendimento, longo tempo para produção de proteína, alto custo de armazenamento e grande risco de contaminação das amostras (HAMILTON et al., 2006).
Uma das abordagens mais promissoras para produção de proteínas recombinantes tem sido a secreção de proteínas no leite de animais transgênicos. Bovinos, caprinos, suínos, ovinos, camundongos e coelhos, apresentam integrados aos seus genomas os genes de interesse controlados por promotores que restringem a expressão da proteína às glândulas mamárias. Uma desvantagem, porém é o tempo de espera razoavelmente grande para obtenção de proteínas recombinantes e, apesar de ter sido demonstrada a possibilidade de secreção de proteínas heterólogas na urina de animais transgênicos sob o controle de promotores tecido-específicos, o sistema continua com a grande desvantagem de ser um sistema muito caro, com o maior tempo de produção entre todos os sistemas vistos anteriormente e o que possui a menor capacidade de escalonamento de biossíntese de proteínas heterólogas, sem esquecer o risco de contaminação da amostra por vírus animais e príons (LARRICK & THOMAS, 2001; TWYMAN et al., 2003).
1.2 PLANTAS COMO BIORREATOR
de diversas doenças incluindo diarreia viral e bacteriana, raiva, carbúnculo hemático, SARS, sarampo, HIV, difteria, coqueluche, tétano, tuberculose, malária, astroenterites, vírus da peste bovina, citomegalovírus, parvovírus canino, gripe aviária e pneumonia bovina, entre outros, foram produzidos em plantas (revisado por SHARMAet al., 2009; revisado por TIWARI et al., 2009).
As plantas constituem, potencialmente, o sistema com custos mais baixos para produção em larga escala de proteínas recombinantes. Estima-se que essas proteínas possam ser produzidas em plantas por 2 - 10% do custo do sistema de fermentação microbiana, por cerca de 0,1% do custo da cultura de células de mamíferos e com uma redução de 10 - 50%, pelo menos, ao custo final do sistema bacteriano (TWYMAN et al., 2003; MA et al., 2003).
Outros fatores a favor dos sistemas vegetais incluem potencial para produção em larga escala, produção de biomassa mais econômica, eliminação de todos os custos relativos à purificação e processamento para o caso de produtos administrados oralmente (vacinas comestíveis), disponibilidade da tecnologia para colher e processar plantas e os seus produtos à escala industrial e são fáceis de modificar por engenharia genética.
Além disso, proteínas terapêuticas produzidas em plantas, purificadas ou não, têm menos probabilidade de contaminação com microorganismos patógenos humanos do que as derivadas de células animais, pois as plantas não são hospedeiras para agentes infecciosos humanos ou animais, endotoxinas e sequências de DNA oncogênicas (GIDDINGS et al., 2000).
Atualmente os biorreatores vegetais podem ser divididos conforme o tipo de expressão da proteína heteróloga, em dois grupos: os de expressão estável (plantas transgênicas) e os de expressão transiente (plantas infectadas por vetor viral vegetal e plantas agroinfiltradas).
Planta transgênica é aquela que contém um ou mais genes inseridos no seu genoma por meio da transformação genética (TORRES et al., 2000). Essas plantas representam um potencial estável e fonte de propagação barata, no entanto, o desenvolvimento e a seleção de uma linhagem transgênica apropriada podem demorar muitos meses e a produção com alto rendimento nem sempre é possível e estável (RYBICKI, 2009).
normas de boas práticas de fabricação (GMP) e o escalonamento podem dificultar o uso dessas plantas cultivadas no campo como fonte de antígenos para vacinas derivados de plantas (TIWARI et al. 2009). Além disso, a produtividade com alto rendimento nem sempre é atingível e estável devido ao fenômeno do mecanismo de silenciamento gênico (BAULCOMBE, 2005).
Outra estratégia é a de planta transplastômica que baseia na expressão de proteínas em cloroplastos transformados. Este sistema apresenta vantagens como, por exemplo, a integração do novo DNA dentro do genoma do cloroplasto via recombinação homóloga e o número elevado de cópias do transgene (o que leva este sistema a oferecer muitas vezes rendimentos elevados). Não somente o número da cópia do gene por célula, mas também a maquinaria de cloroplasto de produzir proteína é muito mais eficiente do que dos genes do núcleo. Este sistema também evita inúmeros problemas associados com a transformação nuclear devido a ausência de silenciamento gênico e a minimização de DNA cloroplasmático funcional em grãos de pólen, isto é, os genes de plastídeos são de herança uniparental, exclusivamente materna, fator que pode reduzir o risco de contaminação de transgenes para outras plantas (DANIELL, 2006).
A transformação cloroplasmática, no entanto, não é adequada para muitas proteínas complexas devido à incapacidade de realização de modificações pós-traducionais em cloroplastos, incluindo glicosilação. O sistema de transformação e seleção é difícil, ainda limitada a relativamente poucas espécies de plantas (DANIELL, 2006).
Outra tecnologia é a expressão transiente de proteínas heterólogas mediante a utilização de vírus de plantas como vetores de expressão. Ao ser introduzido na planta hospedeira, o vírus contendo o gene de interesse irá replicar e a proteína produzida em significantes quantidades. A utilização de vetores virais tem várias vantagens, entre elas a rapidez com que as proteínas heterólogas podem ser produzidas, o alto rendimento e o fato de que, como o gene não é incorporado no genoma da planta, não se tem uma característica hereditária e, assim é contido. Dentre os vários vetores virais de plantas, destacamos o Tobacco mosaic virus
(TMV), Alfalfa mosaic virus (AMV), Potato virus X (PVX) e Cucumber mosaic virus
(CMV) (YUSIBOV et al., 2006; GLEBA et al., 2007; MATSUO et al., 2007).
silenciamento gênico também pode ser inibido. Entretanto, o vetor viral facilmente exclui o transgene do seu genoma e ainda existe o risco de propagação do transgene para o meio ambiente que são as principais desvantagens do sistema.
Agrobacterium tumefaciens transient expression (ATTE) é uma das principais
ferramentas usadas para expressão transiente em plantas, o qual se aproveita da capacidade natural de Agrobacterium para transferir DNA exógeno para células
vegetais com parede celular intacta. A base molecular desta técnica é a transferência para a planta de uma região de um plasmídeo Ti (do inglês tumor
inducing). Essa região é denominada T-DNA (do inglês transferred DNA) e é
delimitada por duas sequências repetidas, borda esquerda e borda direita, contendo 25 pb cada, onde atuam endonucleases produzidas pela região de virulência (vir) do
plasmídeo Ti (GELVIN, 2003).
O processamento do T-DNA e sua transferência da bactéria para o genoma da célula da planta hospedeira resultam em grande parte da atividade dos produtos dos genes da região vir contidos no plasmídeo Ti, os quais têm expressão ótima sob
pH ácido e na presença de indutores fenólicos, como acetoseringona, que são liberados por ferimentos dos tecidos vegetais. O T-DNA é manipulado para conter os genes de seleção e os genes de interesse, os oncogenes são removidos e então o plasmídeo Ti é inserido em A. tumefaciens e utilizado para transformação em
plantas (BRASILEIRO & CARNEIRO, 1998).
Esse mecanismo de expressão transiente foi desenvolvido baseado na característica geral da Agrobacterium como explicado anteriormente, entretanto
infiltra-se uma suspensão de Agrobacterium contendo um vetor binário e o gene alvo
em folhas intactas com uma seringa ou a planta inteira sob pressão negativa. Desta forma um grande número de células é transformado atingindo altos níveis de expressão de proteínas por um curto período de tempo, com vantagens de baixo custo operacional e simplicidade dos protocolos de transformação e seleção. No entanto, geralmente, a técnica é adequada apenas para produção em pequena ou média escala (FISCHER et al., 2004).
permite avaliar como o gene exógeno é processado pela planta. Devido ao fato do silenciamento gênico pós-transcricional poder ser contornado com a coexpressão de proteínas supressoras de PTGS juntamente com o gene de interesse, esse fator pode ainda ser visto como uma vantagem desse sistema de expressão, pois devido à simplicidade da técnica, esse ensaio poderia funcionar como um antecessor de outros sistemas de produção heteróloga em plantas, permitindo prever o possível silenciamento gênico do transgene (CAZZONELLI & VELTEN, 2006).
A técnica de ATTE tem sido usada com sucesso para expressar genes repórter em tecidos de folhas de Nicotiana benthamiana, Nicotiana tabacum, Phaseolus vulgaris, Phaseolus acutifolius, Lactuca sativa, Arabidopsis thaliana,
Pisum sativum e Linum usitatissimum (VAN DER HOORN et al., 2000;
WROBLEWSKI et al., 2005).
Plantas de N. benthamiana são apropriadas a estes testes, pois além de sua
elevada eficiência de transferência de T-DNA (~100%) em células do mesófilo por infiltração com A. tumefaciens, possuem folhas de fácil infiltração que podem
produzir altas quantidades de proteínas em resposta a agroinfiltração (MARILLONNET et al., 2004).
Por todos esses motivos, a técnica de ATTE é bastante promissora comparando com outras estratégias, isso se deve também ao fato de ser rápida, sem necessidade de regeneração de plantas das células transformadas, e, portanto útil, para avaliar a expressão de construções múltiplas (HELLENS et al., 2005).
1.3 SILENCIAMENTO GÊNICO MEDIADO POR RNA
O silenciamento por RNA é um poderoso mecanismo de defesa que atua na regulação da expressão gênica, que está relacionado com o bloqueio na transcrição induzido por RNA antissenso derivado do próprio DNA que promoveria uma metilação na região promotora, e a homologia para a metilação dirigida ocorreria nas regiões transcritas e a nível pós-transcricional onde a degradação de RNAs homólogos no citoplasma levaria a não tradução (VAUCHERET et al., 2001).
e eucariotos inferiores (VAN DER KROL et al., 1990; ROMANO & MACINO, 1992;
FIRE, 1998; ROTH et al.; 2004).
O fenômeno de silenciamento gênico foi observado pela primeira vez em plantas transgênicas por dois grupos independentes de pesquisadores (NAPOLI et al., 1990; VAN DER KROL et al., 1990) onde o objetivo desses pesquisadores era criar petúnias cujas flores apresentassem uma coloração roxa muita escura, quase negra. A estratégia era superexpressar o gene que codifica a chalcone sintase (CHS), uma enzima chave na biossíntese de antocianinas. Para obter esse resultado, foram introduzidas cópias extras do gene da chalcone sintase sob controle do promotor 35S do Cauliflower mosaic cirus (CaMV), e, diferentemente do
que os pesquisadores esperavam, a introdução dessas inúmeras cópias do gene, produziram padrões distintos de tonalidades florais, incluindo linhagens que apresentavam flores totalmente brancas, ou seja, sem pigmento. Comprovou-se que a introdução do gene bloqueou efetivamente a biossíntese de antocianinas, inibindo simultaneamente, a expressão do gene endógeno pré-existente e da cópia introduzida. A inibição da pigmentação das flores foi diretamente correlacionada com uma redução específica no acúmulo de RNA mensageiro (mRNA) do gene Chs. O
fenômeno foi denominado cosupressão, pois a introdução de um transgene levou ao silenciamento simultâneo do próprio transgene e do gene endógeno homólogo.
Alguns anos mais tarde, Romano e Macino (1992) descreveram um fenômeno observado em fungos filamentosos da espécie Neurospora crassa, relacionado ao
de cosupressão, sendo este fenômeno conhecido como “Quelling”.
Em 1998, FIRE e colegas demonstraram que dsRNA era fundamental para induzir o silenciamento gênico em nematóides. Esse grupo ao injetar o mRNA responsável por comandar a síntese de uma proteína muscular, no nematóide
Caenorhabditis elegans, não notou qualquer efeito, assim como quando foi injetada
diversas outras proteínas do C. elegans e comprovaram que dsRNA silenciava o
gene que tinha a mesma sequência do RNA e, com isso, a proteína associada a esse gene deixava de ser produzida.
O termo silenciamento gênico refere-se a uma série de vias relacionadas encontradas em uma variedade de organismos eucarióticos. Estudos demonstraram a existência de três vias de silenciamento de RNA, e elas se sobrepõem em alguns pontos. A primeira via é a de silenciamento citoplasmático via siRNAs (em inglês
short-interfering RNAs), o qual o dsRNA pode ser originário da transcrição de um
gene endógeno, um transgene ou um intermediário da replicação de vírus com genoma de RNA e esta via está envolvida na degradação de RNA viral, interferindo ou até bloqueando a infecção. A segunda via refere-se ao silenciamento de mRNAs endógenos via miRNAs, onde estes regulam negativamente a expressão gênica por meio do pareamento de bases específica a mRNA alvo o que resulta na clivagem do mRNA ou na repressão de sua tradução. A terceira via provavelmente protege o indivíduo das desorganizações genômicas causadas por transposons e é nuclear e associada à metilação de DNA e à formação de heterocromatina (BAULCOMBE, 2005).
Pesquisas bioquímicas e genéticas estabeleceram um modelo geral do mecanismo para essas vias relacionadas e identificaram fatores que são necessários para o silenciamento gênico em diversos organismos. O processo normalmente é desencadeado pela formação de dsRNA, este dsRNA é clivado por um tipo de RNase III (endonuclease com afinidade por dsRNA) denominada Dicer em fragmentos de aproximadamente 21 – 24 nucleotídeos, os siRNAs, em um processo dependente de ATP. O modelo para a clivagem de dsRNA mediada por Dicer propõe que a enzima atue como um dímero antiparalelo, formando dois centros catalíticos que irão gerar os siRNA (BLASZCZYK at al., 2001).
Em Arabidopsis, a família gênica que codifica proteínas homólogas a Dicer
possui quatro membros homólogos (DCL), DCL-1 é necessária para o acúmulo de miRNAs; DCL-2 é necessária para a produção de siRNA derivados de vírus; DCL-3 produz siRNA endógenos e DCL-4 não tem sua função estabelecida, mas acredita-se que possa acredita-ser redundante com DCL-1 (HAMILTON et al., 2002; XIE et al., 2004). Os siRNAs são desnaturados e incorporados dentro de um complexo
ribonucleoprotéico denominado RISC (RNA- induced silencing complex) que possui
dependente de ATP e com atividade de uma RNA helicase. Uma vez incorporado ao complexo RISC, o dsRNA é desenrolado e somente uma das fitas de ~22 nt é mantida no complexo (ELBASHIR et al, 2001).
Cabe ressaltar que a porção 5’ da molécula antissenso de um siRNA funcional é termodinamicamente menos estável do que a porção 5’ da molécula senso, sendo esse o fator chave para seleção da molécula antissenso como guia pelo complexo RISC. A fita senso por sua vez é clivada por outras proteínas AGO (SHREY et al., 2009). A molécula guia de RNA antissenso é capaz de formar um duplex com a molécula complementar do mRNA alvo, e em seguida, pela ação da proteína AGO presente no RISC ativado, ocorre à clivagem do mRNA homólogo
(HAMMOND et al., 2000; ZAMORE et al., 2000; BERNSTEIN et al., 2001; ROTH et
al., 2004).
A especificidade do mecanismo é, portanto, determinada pelas moléculas de RNA antissenso presentes em RISC, e que são complementares à molécula de mRNA alvo. Se a complementaridade para tal for perfeita, como ocorre com os siRNAs, a fita de mRNA é clivada rapidamente por AGO que reconhece a região 3’ desprotegida (SHREY et al., 2009). Se a complementaridade não for perfeita, como acontece com os miRNAs, o complexo AGO/RISC bloqueia a tradução.
A repressão da tradução e a degradação do RNA ocorrem quando as moléculas de RNA guias têm somente complementaridade limitada à região alvo, com o pareamento de bases ocorrendo usualmente na região 3’ não traduzida (ZAMORE et al., 2000).
Uma característica marcante do silenciamento de RNA é seu caráter sistêmico, isto é, o processo não fica restrito às células iniciais em que foi disparado. Em plantas, acredita-se que o sinal sistêmico seja capaz de mover célula-a-célula via plasmodesmas e a longas distâncias via floema (MALLORY et al., 2002).
1.4 SUPRESSORES VIRAIS DE SILENCIAMENTO GÊNICO
Se o silenciamento gênico é um mecanismo de defesa contra vírus de plantas, não é de se surpreender que alguns vírus tenham vias envolvidas para contornar esse sistema de defesa. Estudos de sinergismos que ocorrem em infecções mistas foram fundamentais para o entendimento do mecanismo de supressão de silenciamento gênico utilizado pelos vírus.
Quando uma planta é infectada por duas ou mais espécies de vírus, a severidade dos sintomas é maior do que a adição dos efeitos dos vírus isolados. Muitos casos de sinergismo envolvem espécies do gênero Potyvirus, por exemplo,
Potato virus Y (PVY), Tobacco vein mottling virus (TVMV) e Tobacco etch virus
(TEV). Exemplos clássicos incluem a interação entre o potexvirus Potato virus X
(PVX) com os potyvírus citados, infectando espécies de Nicotiana. O resultado
desses estudos demonstra que o aumento da severidade dos sintomas é relacionado com um aumento do acúmulo de PVX, não havendo aumento ou decréscimo da concentração do potyvírus, concluindo que a replicação do genoma do potyvírus não é necessária para o sinergismo PVX/potyvírus e que a resposta é mediada pela expressão da proteína HC-Pro (VANCE et al., 1995).
Depois da descoberta de que uma proteína viral poderia ser capaz de suprimir o silenciamento, muitos pesquisadores sentiram-se incentivados a buscar novos exemplos em diversas espécies de vírus.
Os primeiros supressores virais relatados foram o HC-Pro e a proteína 2b codificada por potyvírus e cucumovírus, respectivamente (ANANDALAKSHMI et al., 1998; BRIGNET et al., 1998). Atualmente mais de 40 proteínas virais supressoras de silenciamento gênico foram identificadas em vírus de plantas, animais e insetos, incluindo a proteína NS1 do vírus da influenza (Influenza virus A, B e C), E3L do
Vaccinia virus (poxvírus) e B2 de Flock house virus (nodavírus) (LI & DING, 2006).
Dados disponíveis sugerem que todos os vírus de plantas codificam pelo menos um supressor viral de RNA. Embora a função dos supressores virais seja conservada entre as proteínas virais homólogas dentro de um gênero ou família como podemos citar 2b de cucumoviroses (BRIGNETI et al., 1998; LI et al., 1999), NS1 de três de quatro vírus da família Orthomyxoviridae (LI et al., 2004), B2 dos
gêneros da família Nodaviridae (LI et al., 2004; FENNER, et al., 2006), P19/P14 do
e AC2 e seus homólogos de dois dos quatro gêneros da família Geminiviridae
(BISARO, 2006); supressores virais de diferentes famílias muitas vezes não
compartilham homologia de sequência ou similaridade estrutural.
As proteínas supressoras podem possuir mais de uma função, e proteínas que são consideradas determinantes de patogenicidade poderão ser identificadas como supressores de silenciamento (BRIGNETI et al., 1998; ROTH et al., 2004). Essas proteínas podem inibir o silenciamento interferindo em diferentes etapas do mecanismo, incluindo a geração dos siRNAs, incorporação destes ao RISC, a atividade do RISC e a atividade do sinal sistêmico.
A proteína HC-Pro de potyvírus, por exemplo, exerce sua atividade supressora ao afetar negativamente o acúmulo dos siRNAs, reduzindo-os ou eliminando-os. Isso leva a crer que o silenciamento é bloqueado no passo de incorporação dos dsRNA pela Dicer. (JOHANSEN & CARRINGTON, 2001; MALLORY et al., 2002). Outros estudos mostraram que HC-Pro não só interfere com o acúmulo dos siRNA, como também no acúmulo dos miRNA endógenos, responsáveis pelo desenvolvimento de órgãos e tecidos, incluindo diversos fatores de transcrição (MALLORY et al., 2002; KASSCHAU et al., 2003).
Estudos envolvendo a proteína supressora p19 do tombusvírus Tomato bushy
stunt virus (TBSV) indicaram que o mecanismo de supressão de p19 é mediado pelo
sequestro dos siRNAs produzidos pela Dicer, impedindo que estes sejam incorporados ao RISC (SILHAVY et al.,2002).
A inibição do silenciamento sistêmico é outra estratégia dos supressores virais. A proteína 2b codificada pelo cucumovírus Cucumber mosaic virus (CMV) age
no silenciamento sistêmico bloqueando o movimento do sinal, mas possui um pequeno efeito no silenciamento local (BRIGNETI et al., 1998), e ainda interage diretamente com AGO1 (ZHANG at al., 2006).
suas proteínas supressoras afetam o silenciamento em pontos diferentes, ou no mesmo ponto, porém de forma distinta
1.4.1 Candidatos a supressores virais utilizados no estudo
1.4.1.1 Brugmansia suaveolens mottle virus – BsMoV
BsMoV foi o primeiro potyvírus isolado de Brugmansia suaveolens
(trombeta-de-anjo), uma solanácea arbustiva semi-lenhosa usada como planta ornamental, descrita no Brasil. O vírus (isolado Bs-Campinas) é transmitido por afídio e tem a capacidade de infectar algumas solanáceas e também duas espécies de
Chenopodium (LUCINDA et al., 2008).
A família Potyviridae constitui o maior e, do ponto de vista econômico, o mais
importante grupo de vírus de plantas onde cerca de 20% dos vírus de plantas pertencem a essa família, o que corresponde a 106 espécies de vírus nos seis gêneros (Potyvirus, Bymovirus, Rymovirus, Ipomovirus, Macluravirus e Tritimovirus)
que a compõe. Os potyvírus são cosmopolitas, sendo encontrados em todas as regiões do globo, infectando mais de 2.000 espécies de plantas em mais de 550 gêneros de 81 famílias. São vírus de RNA fita simples senso positivo transmitidos por pulgões de maneira não persistente (ADAMS et al., 2005).
O genoma de BsMoV consiste de 9.870 nt e sua proteína HC-Pro possui 456 aa e 64,16% de identidade com HC-Pro de Potato virus Y (PVY) (LUCINDA et
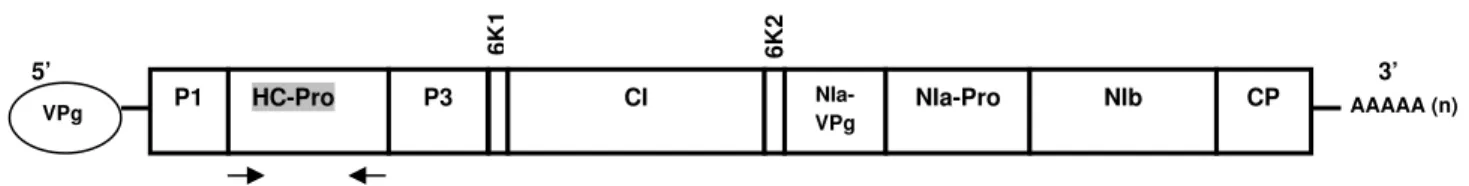
al., 2010) que é um conhecido supressor de PTGS (ANANDALAKSHMI et al., 1998). Utilizaremos no estudo o gene da proteína HC-Pro contendo 1.368 pb, conforme mapa genômico da figura 1.
Figura 1: Mapa genômico de Brugmansia suaveolens mottle virus (BsMoV). O gene supressor
de PTGS é HC-Pro. O mapa mostra o isolamento de gene de HC-Pro por RT-PCR. As setas indicam os primers para PCR.
P1 HC-Pro P3 CI
NIa-VPg NIa-Pro NIb CP
5’
VPg AAAAA (n)
3’
6K
1
6K
1.4.1.2 Pepper mild mottle virus – PMMoV
O gênero Tobamovirus, pertencente à família Virgaviridae, compreende 17
espécies de vírus de RNA fita simples senso positivo, distribuídas em três subgrupos, sendo o Pepper mild mottle virus pertencente ao subgrupo 1, assim
como Tobacco mosaic virus (TMV), o Tomato mosaic virus (ToMV), o Tobacco mild
green mosaic virus (TMGMV) e o Odontoglossum ringspot virus (ORSV) (LARTEY et
al., 1996).
PMMoV infecta pimenta (Capsicum baccatum L.) e pimentão (Capsicum
annuum L.) causando severos sintomas de mosaico nas folhas superiores de
desenvolvimento (KOBORI et al., 2001; EIRASet al., 2003).
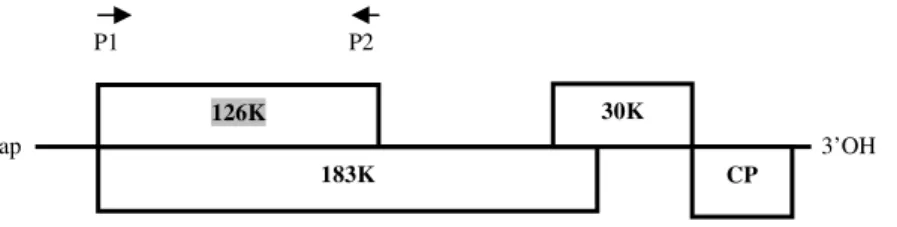
O genoma de PMMoV de 6.356 nucleotídeos possui quatro ORFs que codificam quatro proteínas: as proteínas 126-kDa e 183-kDa envolvidas no processo de replicação; a proteína de movimento 30-kDa e a proteína da capa protéica (ALONSO et al., 1991), conforme figura 2.
Estudos prévios sobre as funções da proteína 126K em tobamoviroses demonstraram que essa proteína possui várias funções, entre elas, replicação viral e supressão da maquinaria de silenciamento gênico agindo downstream à produção de siRNA, bloqueando o uso destes siRNA para incorporação ao RISC (KUBOTA et al., 2003; DING et al., 2004).
No estudo utilizaremos o gene da proteína 126K do isolado PMMoV BR-DF01 com 3.354 pb como candidato a supressor de silenciamento gênico.
P1 P2
cap 3’OH
Figura 2: Mapa genômico de Pepper mild mottle virus (PMMoV). O gene supressor de PTGS é a
126K. O mapa mostra o isolamento de gene da 126K por RT-PCR. P1 e P2 = primers para PCR. 126K
183K
30K
1.4.1.3 Tomato severe rugose virus – ToSRV
Os vírus da família Geminiviridae possuem a estrutura do capsídeo viral
formado por dois icosaédros incompletos geminados. Possuem genoma composto por DNA circular de fita simples (ssDNA) que se replica no núcleo de células infectadas via forma replicativa intermediária de DNA de fita dupla (dsDNA) (LAZAROWITZ & SHEPHERD, 1992). Os geminivirus estão divididos em quatro gêneros: Mastrevirus, Topocuvirus, Curtovirus e Begomovirus, gênero do qual
Tomato severe rugose virus está incluído (STANLEY et al., 2005).
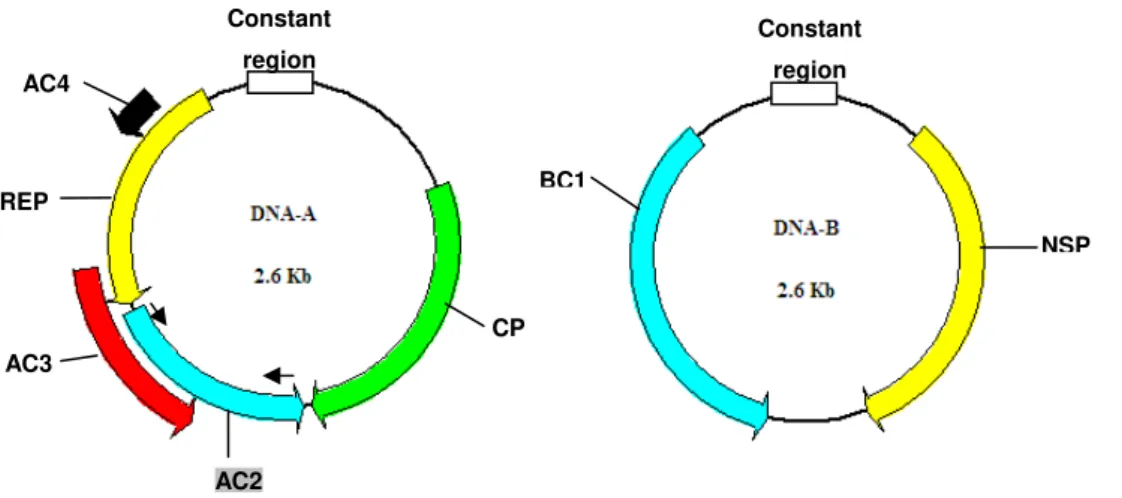
Os Begomovirus possuem espécies com um ou dois componentes
genômicos. As espécies que possuem apenas um componente, restritas ao “Velho Mundo” (Europa, Ásia e África), estão frequentemente associadas a um DNA satélite denominado DNAβ. Esse DNAβ codifica uma proteína supressora de silenciamento
gênico que induz sintomas mais severos quando presente (BRIDDON et al., 2003). No “Novo Mundo” (Américas) as espécies relatadas até o momento possuem dois componentes e normalmente não estão associadas a nenhum DNA satélite. O DNA A contém os genes relacionados com a replicação viral e a síntese da capa protéica, e o DNA B que possui os genes associados ao movimento e expressão de sintomas.
Os begomovírus transmitidos por mosca branca (Bemisia tabaci) infectam
muitas espécies cultivadas, como tomate, feijão, feijão caupi, pimentão, entre outras,
além de plantas invasoras de várias espécies; representam uma grande ameaça à agricultura em regiões tropicais e subtropicais e, por esse motivo, possuem grande importância econômica (MORALES & ANDERSON, 2001).
Tomato severe rugose virus já foi relatado nos estados de Goiás, Minas
Gerais, Pernambuco, Rio de Janeiro, Santa Catarina e São Paulo (FARIA et al., 2000; LIMA et al., 2001; RIBEIRO et al., 2002).
A proteína AL2 de 15 KDa encontrada em todos os begomovírus é também conhecida na literatura como TrAP (transcriptional activator protein), C2 ou AC2. As
metilação de DNA dirigida por siRNA. O mecanismo exato no qual ocorre a supressão ainda não está bem claro (VOINNET et al., 1999; BISARO, 2006).
Em nosso estudo, como candidato a supressor, utilizaremos o gene da proteína AC2 com 390 pb do isolado ToSRV DF, conforme figura 3.
Figura 3: Mapa genômico de Tomato severe rugose virus (ToSRV). O gene supressor de PTGS
é AC2. As setas indicam os primers para PCR.
1.5 A PROTEÍNA VERDE FLUORESCENTE
A proteína verde fluorescente ou GFP (do inglês green fluorescent protein)
produzida pelo cnidário Aequorea victoria foi descoberta em 1962 por Shimomura e
colaboradores,e tornou-se um gene repórter padrão em muitos sistemas biológicos. Essa proteína emite fluorescência na zona verde do espectro visível e é intrinsicamente fluorescente, isto é, não necessita de substratos ou co-fatores. Possui dois picos de excitação em UV e luz azul, um menor a 395 nm e um maior a 475 nm e seu pico de emissão é a 509 nm (CHALFIE et al., 1994; SIEMERING et al., 1996).
A estrutura primária de GFP e sua respectiva sequência de aminoácidos foram elucidadas em 1992 por Prasher e colaboradores, e foi determinado que a proteína possui 238 aminoácidos e massa molecular calculada em 26.888 Da.
CP
AC2 AC3
REP AC4
Constant region
Constant region
Sua fluorescência é explicada pela presença de um cromóforo denominado p
-hidroxibenzilideneimidazolidinona formado pela ciclização e oxidação dos resíduos de aminoácidos Ser65 - Tyr66 - Gly67(PRASHER et al.,1992; CODY et al., 1993).
O aminoácido Gly67 parece ser o único dos três aminoácidos indispensável para a correta formação do cromóforo, pois a substituição dos outros dois resíduos de aminoácidos não interfere na formação do cromóforo. Essa descoberta foi importante para a obtenção das primeiras variantes de GFP, BFP (blue fluorescent
protein) e CFP (cyan fluorescent protein) (TSIEN et al., 1998). Atualmente, diversas
outras proteínas que emitem fluorescência em diferentes cores são usadas em processos biotecnológicos.
Inúmeras razões tornam a GFP extremamente atrativa como alternativa aos genes repórteres atual, pois a GFP permite a visualização direta de expressão gênica e localização subcelular de proteínas de fusão em células vivas, sem necessidade de técnicas invasivas ou adição de co-fatores (CHALFIE, 1995). Além disso, facilita nossa compreensão dos processos biológicos fundamentais, interação entre microorganismos e plantas (GAGE et al., 1996), resposta à stress (FU et al., 2007) e mecanismos de silenciamento gênico (BAULCOMBE et al., 1995 e VOINNET et al., 1998).
O espectro de fluorescência é um ponto muito importante na aplicação de GFP como gene repórter. O pico de excitação de 395 nm é útil para o rastreio visual direto de organismos inteiros ou colônias de células através de lâmpadas de UV. O pico de excitação 475 nm é importante para aplicações tais como a microscopia de fluorescência ou confocal, onde conjuntos de filtros otimizados para a detecção de derivados de fluoresceína ou ainda excitação por laser azul são comumente usados (SIEMERING et al; 1996).
Apesar das muitas vantagens da GFP selvagem, sua utilização pode ser limitada pela formação lenta do cromóforo, o que normalmente ocorre aproximadamente duas horas após a síntese, o que limita os estudos que requerem monitoramento dentro deste período de tempo, como por exemplo, detecção de proteínas com meia vida curta. Além disso, GFP selvagem tende a formar corpos de inclusão em temperaturas acima de 37°C, e a intensi dade da fluorescência é relativamente baixa (HEIM et al., 1994).
base molecular da fluorescência, procedeu-se alterações mutagênicas da GFP produzindo uma série de variantes úteis, mais eficientes que a GFP nativa (CHALFIE et al., 1995).
A facilidade de detecção e a ausência de efeitos citotóxicos fazem da GFP uma poderosa ferramenta em estudos envolvendo biotecnologia de plantas, aplicada em três áreas distintas: visualização dinâmica de proteínas marcadas dentro das células; marcação seletiva e monitoramento de células em tecidos vegetais em crescimento e, identificação individual de plantas transgênicas que expressam a GFP (HASELOFF et al., 1997).
A utilização dessas proteínas para seleção de plantas geneticamente modificadas foi possível pela produção de mutantes de GFP que garantem a correta formação do cromóforo quando é expressa em plantas, além da seleção de moléculas com maior emissão de fluorescência como é o caso dos mutantes mGFP4, mGFP5 e sGFPS65T (SIEMERING et al., 1996; CHIU et al., 1996; HASELOFF et al., 1997). Após a manipulação dos espectros de fluorescência em um mutante de GFP (GFPA) com intensidade de fluorescência trinta e cinco vezes maiores que GFP selvagem quando expressa em E.coli a 37°C, foi produzido um
mutante termoestável (mgfp5) que pode ser facilmente excitado utilizando UV ou luz
azul e que resulta em níveis significantemente maiores de fluorescência em células de mamífero cultivadas a 37°C.
Assim como a GFP selvagem, GFP5 possui dois picos de excitação (λex: 395 nm ou 473 nm) e é, portanto, ideal como uma variante multi-expectral que pode ser usada para aplicações que requerem excitação por UV ou luz azul (SIEMERING et al., 1996).
2 JUSTIFICATIVA
Atualmente, o alvo de muitos estudos é a busca de sistemas eficientes para expressão de proteínas recombiantes, que possibilitem tanto o dobramento e a montagem corretos de proteínas heterólogas complexas, como o correto processamento pós-traducional dessas moléculas e que, além disso, possuam vantagens em termo de biossegurança a um baixo custo.
O uso de plantas para produzir proteínas recombinantes oferece inúmeras vantagens sobre os outros sistemas atualmente utilizados. Entre elas a ausência de risco de contaminação com patógenos animais incluindo vírus, pois nenhum vírus vegetal é considerado patógeno para humanos, além disso, o uso de plantas pode oferecer simples condições em manutenção e produção em larga escala.
Agrobacterium tumefaciens transiente expression (ATTE) surgiu como uma
metodologia alternativa para transferência direta de DNA. Este sistema tem a vantagem de aproveitar capacidade natural de A. tumefaciens de transferir
eficientemente contruções de DNA específicas contidas na região T-DNA do plasmídeo Ti para a célula da planta. Com esse sistema, um grande número de células vegetais são transformadas, atingindo altos níveis de expressão, ao contrário de outras tecnologias de transferência direta de DNA (JOHANSEN & CARRINGTON, 2001; CAZZONELLI & VELTEN., 2006).
Johansen & Carrington (2003), demonstraram que a redução dos níveis da expressão de transgenes no sistema de ATTE, uma das principais desvantagens dessa técnica, é decorrente do silenciamento gênico, porém na mesma época, Voinnet e colaboradores (2003) conseguiram aumentar os níveis de expressão de diversas proteínas, em pelo menos cinqüenta vezes, co-expressando a proteína p19 de TBSV, um conhecido inibidor de silenciamento gênico.
Para aumentar a quantidade de proteína heteróloga expressa em folhas de N.
benthamiana utilizando esta técnica, utilizaremos alguns candidatos a supressores
de PTGS como, por exemplo, a proteína viral HC-Pro de uma nova espécie de potyvírus relatada no Brasil (Brugmansia suaveolens mottle virus, BsMoV),
recentemente isolado e sequenciado (LUCINDA et al.,2008; LUCINDA et al., 2010),
a qual será utilizada devido à alta infectividade e agressividade em N. benthamiana.
do PTGS, relatados anteriormente, como proteína viral 126K do Pepper mild mottle
virus, PMMoV (Tobamovirus) e AC2 do Tomato severe rugose virus, ToSRV
(Begomovirus) serão testados separadamente monitorando o aumento de proteína
heteróloga, Green fluorescent protein (GFP).
GFP foi usada como um marcador sensível e não-invasivo para visualizar a expressão transiente em células transformadas.
3 OBJETIVOS
3.1 OBJETIVO GERAL
O presente estudo objetiva a otimização do sistema de expressão de proteínas recombiantes em plantas por meio da estratégia de Agrobacterium
tumefaciens transient expression (ATTE)através da coinfiltração das construções de
expressão via Agrobacterium tumefaciens junto com candidatos a supressores de
PTGS visando minimizar a supressão da produção de proteínas alvo pelo silenciamento gênico ou PTGS.
3.2 OBJETIVOS ESPECÍFICOS
1. Amplificação dos três genes candidatos a supressores de PTGS, de p19 de TBSV, do gene repórter mgfp5 e clonagem destes,
separadamente, em vetor binário pGreenII SK-62.
2. Coinfiltração dos genes candidatos a supressores e GFP em N. benthamiana.
3. Monitoramento da expressão da proteína GFP clonada no mesmo vetor, em N. benthamiana via ATTE para avaliação da qualidade do
sistema.
4. Testar os candidatos a supressores de silenciamento gênico. 5. Detecção da expressão das proteínas através de Western blot.
4 MATERIAIS E MÉTODOS
4.1 VETOR DE CLONAGEM
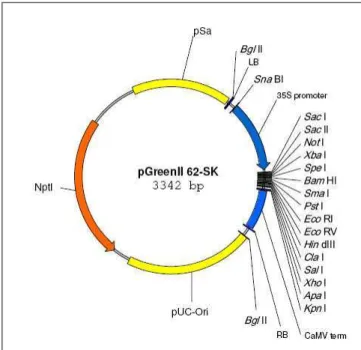
O vetor binário pGreenII 62-SK foi escolhido por seu tamanho reduzido, flexibilidade de transformação e seleção e um múltiplo sítio de clonagem. Além disso, a série II dos vetores pGreen, são ideais para o sistema de expressão transiente usando Agrobacterium, sendo gerados com a remoção de elementos
necessários para transformação estável, tais como marcadores de seleção, comuns nos vetores binários convencionais (HELLENS et al., 2000a; HELLENS et al.,
2000b).
Este, como observado abaixo na figura 4, contém sítiosde clonagens para as enzimas de restrição SacII e KpnI. Esse sítio foi eliminado pela inserção de nossos
genes de interesse com exceção do gene da proteína p19 de TBSV que foi inserido nos sítio de SacII e XhoI.
Figura 4: Mapa genético de pGreenII 62-SK. NptI = gene de resistência a canamicina, pSa = origem de replicação de Agrobacterium, pUC-Ori = origem de replicação de Escherichia coli.
4.2 AMPLIFICAÇÃO DOS GENES CANDIDATOS À SUPRESSORES DE PTGS E DO GENE REPÓRTER GFP
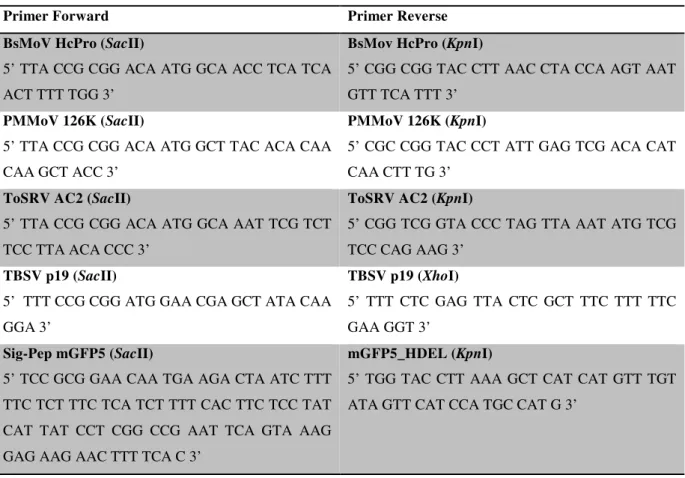
Os genes foram amplificados em Reação de Cadeia de Polimerase (PCR), utilizando os primers listados na tabela 1.
Tabela 1: Primers utilizados na amplificação dos genes.
Primer Forward Primer Reverse
BsMoV HcPro (SacII)
5’ TTA CCG CGG ACA ATG GCA ACC TCA TCA ACT TTT TGG 3’
BsMov HcPro (KpnI)
5’ CGG CGG TAC CTT AAC CTA CCA AGT AAT GTT TCA TTT 3’
PMMoV 126K (SacII)
5’ TTA CCG CGG ACA ATG GCT TAC ACA CAA CAA GCT ACC 3’
PMMoV 126K (KpnI)
5’ CGC CGG TAC CCT ATT GAG TCG ACA CAT CAA CTT TG 3’
ToSRV AC2 (SacII)
5’ TTA CCG CGG ACA ATG GCA AAT TCG TCT TCC TTA ACA CCC 3’
ToSRV AC2 (KpnI)
5’ CGG TCG GTA CCC TAG TTA AAT ATG TCG TCC CAG AAG 3’
TBSV p19 (SacII)
5’ TTT CCG CGG ATG GAA CGA GCT ATA CAA GGA 3’
TBSV p19 (XhoI)
5’ TTT CTC GAG TTA CTC GCT TTC TTT TTC GAA GGT 3’
Sig-Pep mGFP5 (SacII)
5’ TCC GCG GAA CAA TGA AGA CTA ATC TTT TTC TCT TTC TCA TCT TTT CAC TTC TCC TAT CAT TAT CCT CGG CCG AAT TCA GTA AAG GAG AAG AAC TTT TCA C 3’
mGFP5_HDEL (KpnI)
5’ TGG TAC CTT AAA GCT CAT CAT GTT TGT ATA GTT CAT CCA TGC CAT G 3’
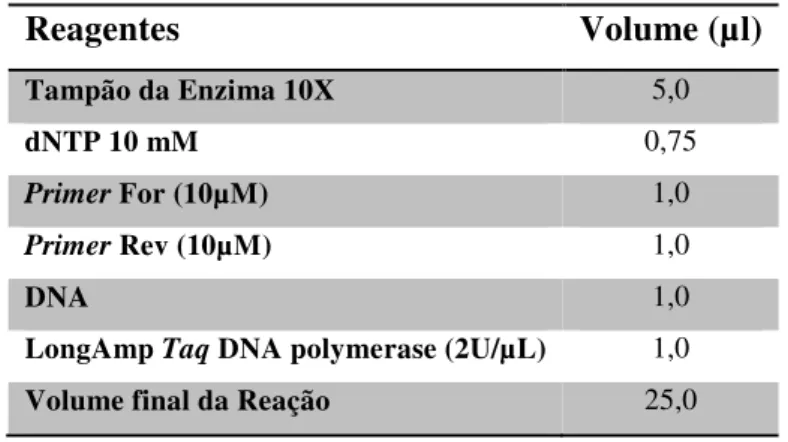
A amplificação dos insertos foi realizada utilizando a enzima “LongAmp Taq
DNA Polymerase” (New England BioLabs) realizada no termociclador (Verity,
Tabela 2: Protocolo da PCR utilizada para amplificação dos genes.
Reagentes Volume (µl)
Tampão da Enzima 10X 5,0
dNTP 10 mM 0,75
Primer For (10µM) 1,0
Primer Rev (10µM) 1,0
DNA 1,0
LongAmp Taq DNA polymerase (2U/µL) 1,0
Volume final da Reação 25,0
4.3 DIGESTÃO DE VETOR DE CLONAGEM E INSERTOS
O vetor de clonagem pGreenII 62-SK e os produtos de PCR foram submetidos a uma etapa de digestão, no qual cada fragmento amplificado foi digerido por um par de enzimas de restrição (SacII e KpnI ou SacII e XhoI), de
acordo com o sítio de restrição determinado pela sequência de primers utilizados na
etapa de amplificação. O protocolo foi utilizado conforme fornecido pelo laboratório
New England BioLabs (NEB). Foi realizada eletroforese em gel de agarose 1%
(SAMBROOK & RUSSEL, 2001) após a digestão para confirmar a eficiência do corte
das enzimas. Em seguida, foram corados em Violeta Cristal (40µg/ml) por 15 minutos e as bandas eluídas em kit de purificação (GE Healthcare) conforme protocolo do fabricante, com a finalidade de eliminar os resíduos da digestão.
4.4 LIGAÇÃO DOS INSERTOS EM VETOR DE CLONAGEM
cuja proporção de molaridade de inserto para vetor foi 3:1. Em cada ligação utilizou-se 100 ng do vetor pGreenII 62-SK.
4.5 TRANSFORMAÇÃO DE BACTÉRIAS E.COLI
Após a ligação dos insertos ao vetor pGreenII 62-SK, as construções foram, separadamente, transformadas em células de E. coli cepa DH5-α competentes para
eletroporação utilizando o equipamento Gene Pulser II (Bio Rad), conforme especificado por CHASSY & FLICKINGER, 1987.
Para selecionar os clones foram utilizados meio LB com canamicina (50µg/ml) conforme especificado em SAMBROOK & RUSSEL, 2001.
A extração dos plasmídeos foi feita com o Kit de purificação PureLinkTM Quick Plasmid Miniprep (Invitrogen), conforme recomendado pelo fabricante e as amostras foram enviadas para sequenciamento para confirmar a presença dos insertos.
4.6 TRANSFORMAÇÃO EM A. TUMEFACIENS E AGROINOCULAÇÃO
Após a confirmação da presença dos insertos nas construções, por sequenciamento, os vetores binários foram transformados por eletroporação em células de A. tumefaciens cepa GV3101 contendo o plasmídeo helper pSoup
(HELLENS et al., 2005) usando um eletroporador Gene Pulser II (Bio-Rad), conforme CHASSY & FLICKINGER, 1987. Os clones desejados foram selecionados utilizando canamicina (100 µg/ml), rifampicina (50 µg/ml) e tetraciclina (2 µg/ml).
Para inoculação na planta, 2 ml de cultura líquida crescida overnight foi
centrifugada a 1200 g por 10 min e ressuspendido em 5 ml de Meio MS (pH 5,5) suplementado com 100 µM de acetosiringona. O volume final foi ajustado para concentração bacteriana de 0,5 em A600.
Plantas de Nicotiana benthamiana com aproximandamente 5 semanas de
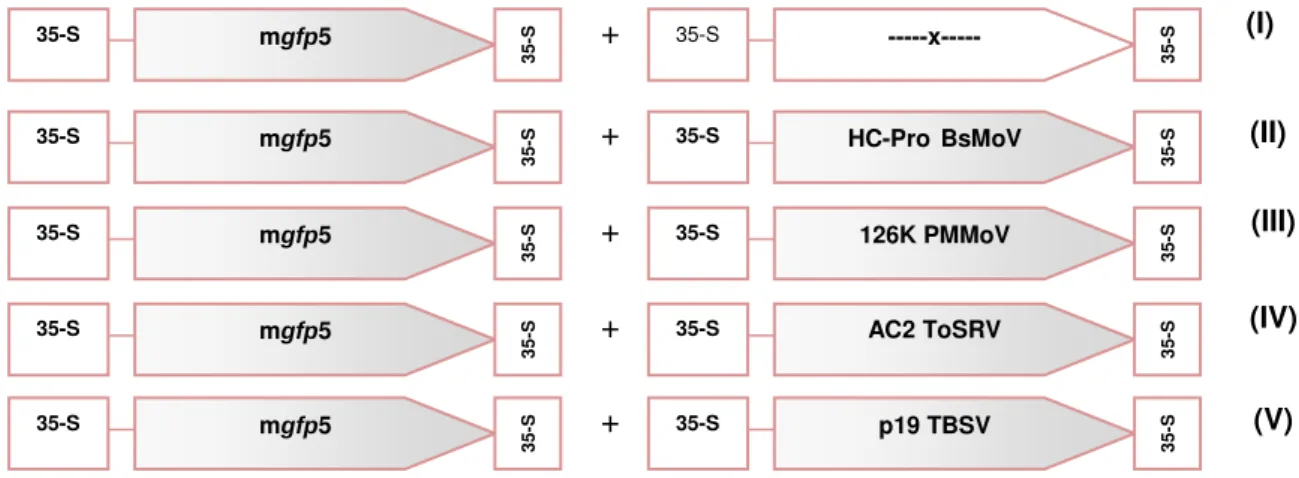
Quatro construções para agroinfiltração nas folhas de N. benthamiana foram
preparadas contendo os candidatos a supressores de silenciamento gênico (HC-Pro do BsMoV; 126K do PMMoV e AC2 do ToSRV) e a proteína p19, do TBSV, previamente caracterizada como supressora foi utilizada como controle positivo,abaixo na figura 5.
Usaremos como proteína-alvo em nosso estudo, a mGFP5 (SIEMERING et al., 1996) podendo futuramente ser substituída por outras proteínas de interesse e como controle positivo escolhemos a proteína viral p19 de Tomato brushy stunt
virus, TBSV (Tombusvirus) por ser um efetivo supressor de silenciamento utilizado
em vários trabalhos (VOINNET et al., 2003; BAULCOMBE & MOLNAR, 2004; LAKATOS et al., 2004).
Culturas de Agrobacterium contendo os clones selecionados foram infiltradas
na superfície abaxial de folhas de N. benthamiana. Em cada folha foram infiltradas
duas áreas, uma contendo a linhagem com os candidatos a supressores de silenciamento e a linhagem contendo pGreenII-mgfp5, na proporção 1:1. Na outra
área foi infiltrada apenas a linhagem contendo pGreenII-mgfp5.
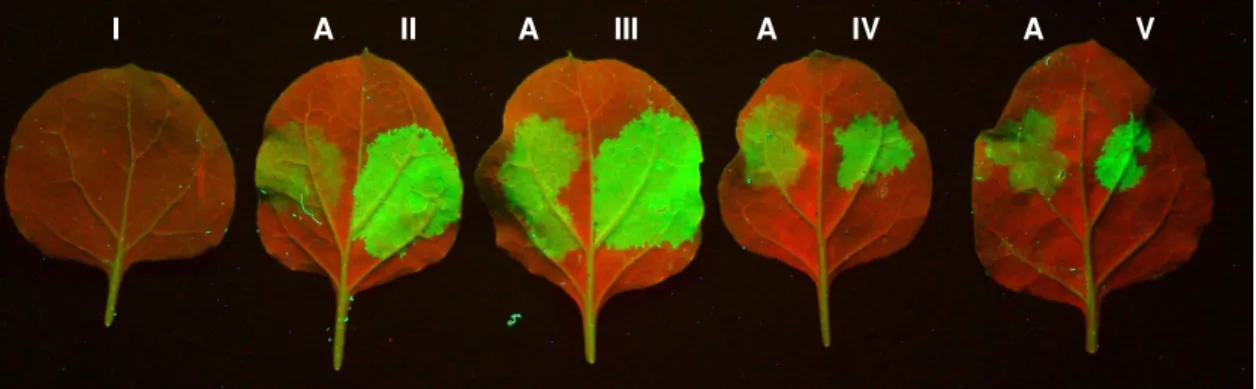
Figura 5: Construções utilizadas para agroinfiltrações em folhas de N. benthamiana. (I) gfp
coinfiltrada com o vetor pGreenII vazio; ou com os candidatos a supressores (II) HC-Pro BsMoV; (III) 126K PMMoV; (IV) AC2 ToSRV e (V) controle positivo p19 TBSV.
35-S mgfp5
35
-S
35-S mgfp5
35
-S
35-S mgfp5
35
-S
35-S mgfp5
35
-S
35-S mgfp5
35
-S
35-S ---x---
35
-S
35-S 126K PMMoV
35
-S
35-S AC2 ToSRV
35
-S
35-S p19 TBSV
35
-S
35-S HC-Pro BsMoV
4.7 OBSERVAÇÃO DA FLUORESCÊNCIA DE GFP
A fluorescência de GFP foi observada sob luz UV (365 nm, lâmpada modelo UVLM–28 EL, UVP) durante 10 dpi (dias pós infiltração) e fotografada usando uma máquina digital Nikon e filtro amarelo com 15 segundos de exposição.
4.8 ANÁLISE POR WESTERN BLOT
Aproximadamente 0,1g de folhas de N. bentahamiana agroinfiltradas foram
maceradas em tampão PBS 1X (NO2HPO4 10 mM; KH2PO4 1,7 mM; NaCl 140 mM e KCl 2,7 mM) baseado em SAMBROOK & RUSSELL 2001, contendo inibidor de proteases Protease Inhibitor Cocktail (Sigma-Aldrich). A solução extraída foi fervida
por 5 minutos e incubada no gelo por 3 minutos para inativar possíveis proteases que degradariam as proteínas de interesse. Os extratos protéicos foram submetidos à eletroforese em gel de poliacrilamida, corado com Comassie Blue (Comassie 0,25%, Metanol 50%, Ácido Acético 7%) e descorado com solução descorante (Ácido Acético 10%, Etanol 30%) a fim de se confirmar a integridade das amostras.
Após eletroforese em gel de poliacrilamida (SDS-PAGE, “stacking gel” 5% e “resolving gel” 15%), o gel foi transferido para membrana de PVDF (Hybond-P PVDF
Membrane) utilizando o equipamento Transblot SD Semi-Dry Electrophoretic
Transfer Cell(Bio-Rad) sob 100 mA e 10 V regulados.
4.9 AVALIAÇÃO DE INTENSIDADE DE SINAL DA REAÇÃO DE WESTERN BLOT
A membrana de PVDF resultante do western blot foi escaneada em
ImageScanner III, e utilizando o software ImageMaster 2D Platinum 7, foi feita a leitura da porcentagem de volume e intensidade de cada sinal da membrana.
5 RESULTADOS E DISCUSSÃO
5.1 CLONAGEM DOS GENES EM VETOR BINÁRIO
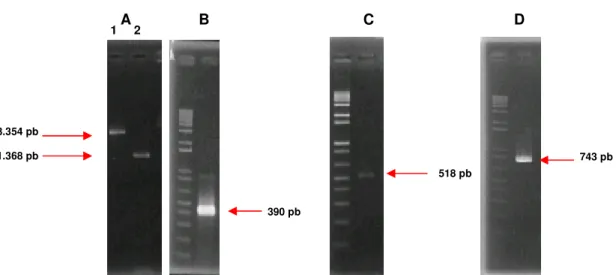
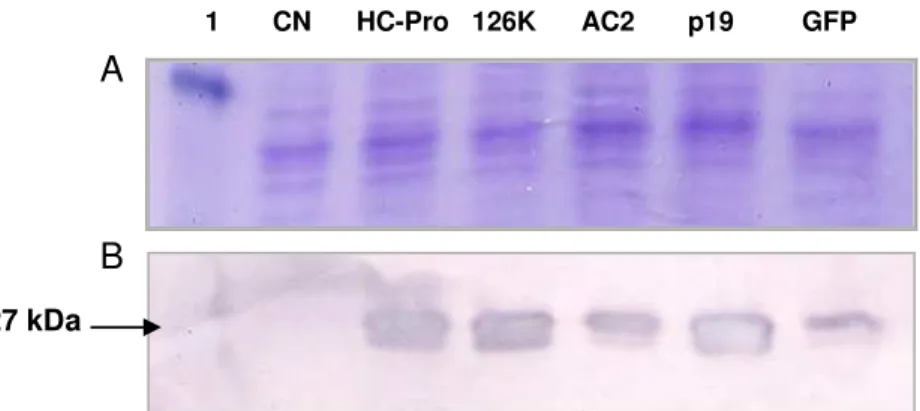
A PCR dos genes gerou fragmentos de DNA com tamanhos esperados: HC-Pro BsMoV (1.368 pb), 126K PMMoV (3.354 pb), AC2 ToSRV (390 pb), p19 TBSV (518 pb) e mgfp5 (743 pb), conforme a figura 6. Os fragmentos amplificados foram
clonados no plasmídeo pGreenII 62-SK nos sítios das enzimas de restrição específicas SacII e XhoI para o gene p19 e SacII e KpnI para os genes restantes. A
presença dos genes e suas sequências foram confirmadas por sequenciamento dos clones selecionados (Apêndice), que foram porteriormente transformados em A.
tumefaciens.
A B C D
Figura 6: Eletroforese de fragmentos de DNA gerado por PCR. (A.1): 126K PMMoV; (A.2): HC-Pro BsMoV; (B): AC2 ToSRV; (C): p19 TBSV e (D): mgfp5. Marcador utilizado:Kb ladder
(Invitrogen).
5.2 AGROINFILTRAÇÃO E OBSERVAÇÃO DE FLUORESCÊNCIA DA GFP
O gene repórter mGFP5 (gfp) foi utilizado como a proteína-alvo dos testes
para avaliar quais candidatos a supressores de silenciamento permitiram maior expressão de mGFP5 (GFP). As folhas de cada planta foram infiltradas do lado esquerdo com a construção de GFP e do lado direito com GFP coinfiltrado com
390 pb
518 pb
743 pb 3.354 pb
1.368 pb
supressores de silenciamento. A eficiência do sistema foi avaliada sob radiação ultravioleta a partir do segundo dia de infiltração, por um período de 10 dias.
Durante os dois primeiros dias não foi notada diferença de fluorescência de GFP entre os locais infiltrados com a GFP com ou sem supressores. Essa diferença foi percebida a partir de 3 dpi, onde as áreas coinfiltradas com supressores começaram a apresentar uma intensidade de fluorescência de GFP maior em relação a área infiltrada apenas com mGFP.
Após 4-5 dpi, uma queda nos níveis de intensidade de fluorescência de GFP era esperada, provavelmente resultante do silenciamento de RNA, porém, os locais infiltrados com GFP junto com os candidatos a supressores HC-Pro BsMoV, 126-kDa PMMov e AC2 ToSRV após o período de 5 dpi claramente exibiram um aumento do nível de fluorescência em relação aos controles, corroborando para os resultados encontrados em estudos com outros supressores virais (JOHANSEN & CARRINGTON, 2001; VOINNET et al., 2003).
A partir de 7 dpi o nível de intensidade de fluorescência nas áreas onde existia a coinfiltração com os candidatos a supressores começou a diminuir e com 10 dpi essa intensidade era baixa, quase imperceptível.
No experimento mostrado na figura 7, utilizou-se uma construção de pBIN19-ER-GFP. Este experimento permitiu testar os candidatos a supressores de PTGS induzidos pela super expressão simultânea do transgene agroinfiltrado expressando GFP através de vetor pBIN19 e as sequências codificantes selecionadas através de um sistema de vetor binário pGreenII (HELLENS et al., 2005) ambos sob o controle do promotor CaMV 35S. No experimento da figura 8 utilizou-se a construção de pGreenII-mgfp5 no vetor escolhido para esse estudo.
Figura 7: Sinais de GFP gerado por irradiação de UV onda longa nas folhas de N. benthamiana
agroinfiltradas, 5 dpi. (I) Controle negativo, planta sem agroinfiltração; GFP coinfiltrada com A136 (avirulenta) ou com os candidatos a supressores: (II) HC-Pro BsMoV; (III) 126K PMMoV; (IV) AC2 ToSRV e controle positivo (V) p19 TBSV.
Figura 8: Sinais de GFP gerado por irradiação de UV onda longa nas folhas de N. benthamiana
agroinfiltradas, 5 dpi. (A) GFP coinfiltrada com o vetor pGreenII vazio ou com os candidatos a supressores: (II) HC-Pro BsMoV; (III) 126K PMMoV; (IV) AC2 ToSRV e controle positivo (V) p19 TBSV.
Esses resultados fornecem indícios de que a expressão transiente do gene GFP foi reduzida pelo silenciamento gênico e que esse silenciamento foi alterado pelas proteínas virais HC-Pro de BsMoV, 126K de PMMoV, AC2 de ToSRV e por p19 de TBSV, utilizado como controle positivo, uma vez que é um supressor de silenciamento gênico bem caracterizado (VOINNET et al., 1999; VOINNET et al., 2003).
A diferença de intensidade de GFP, com ou sem supressores, ficou nítida 5 dias após a infiltração. Esses resultados também apóiam a hipótese lançada por JOHANSEN & CARRINGTON (2001) e VOINNET e colaboradores (2003), de que durante a expressão transiente, o silenciamento gênico é desencadeado, e em seguida, suprimido na presença de alguns supressores virais.
I A II A III A IV A V