Une étude des propriétés de l'ADN en relation avec le problème de l'auto-assemblage microscopique est également présentée. La section suivante propose une définition de l'auto-assemblage et de ses propriétés de base.
![Figure 1.1 - Micro-assemblage sériel. Transport, transfert et positionnement robotisés de micro- micro-objets [Golem 06].](https://thumb-eu.123doks.com/thumbv2/1bibliocom/464962.70326/15.892.190.624.753.989/figure-micro-assemblage-sériel-transport-transfert-positionnement-robotisés.webp)
Propriétés de l'auto-assemblage
Un état de l'art des travaux existants à l'échelle microscopique sur l'auto-assemblage inorganique et l'auto-assemblage par des processus biologiques est ensuite détaillé. Ce processus d’auto-assemblage biomoléculaire produit des complexes à partir de molécules dont la taille est mille fois plus petite que la taille des composants assemblés lors d’un auto-assemblage microscopique.
Etat de l'art
L'auto-assemblage microscopique inorganique
En effet, dans le domaine de la vie, l'auto-assemblage est un processus trivial et répétitif [Glade 04]. De plus, les processus d’auto-assemblage biomoléculaire sont connus pour être optimisés à un niveau d’énergie minimum.
L'auto-assemblage microscopique par processus biologiques
L'auto-assemblage par des processus biologiques est un processus dont les principales connexions doivent être étudiées et caractérisées. Pour l’auto-assemblage microscopique dans les processus biologiques, il est important de distinguer deux échelles concernant la force motrice.

Choix d'un processus biologique
D’une part, au niveau local, la force directrice augmente la probabilité de rencontres entre entités biomoléculaires collées à la surface des composants. En revanche, à l’échelle globale, la force dirigeante augmente la probabilité de rencontre entre les composants à assembler.
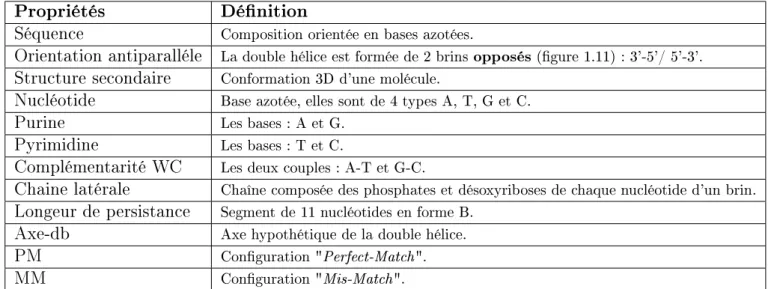
Structure de l'ADN
Pour la forme B, et une concentration de 1M en [N a+], le diamètre de la molécule est de 2n et la torsion d'une hélice est d'environ 0,34 nm. La notion de longueur de persistance dépend de la salinité du milieu et est déterminante pour l'élasticité de la molécule.
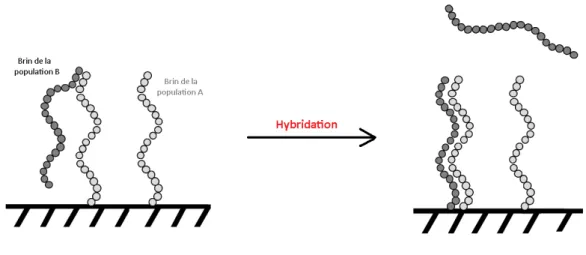
L'hybridation de l'ADN
La Spécicité
Problèmes d'auto-assemblage microscopique avec les processus biologiques La liaison H est une liaison chimique non covalente de type dipôle-dipôle (Figure 1.11). Lorsqu’une liaison hydrogène se forme, les deux hétéroatomes, les nucléotides, sont dans le même plan, distants d’environ 0,2 nm.
La Stabilité
Les paramètres de contrôle de l'hybridation
Paramètres extrinsèques
- La Température
- La Composition du milieu
La concentration en cations du milieu agit sur la configuration spatiale de la chaîne nucléique [Sae 83]. Ainsi, la composition du milieu aqueux joue un rôle très important dans l’équilibre et la stabilité de la double hélice et donc dans le processus d’hybridation.
Paramètres intrinsèques
Les bases azotées sont situées dans l'hélice et forment la partie hydrophobe de la molécule. À l'échelle nanoscopique, l'utilisation potentielle de la molécule dans l'auto-assemblage est largement exploitée et démontrée.
Caractérisation des forces d'interaction de l'ADN
Un résumé des exigences d’auto-assemblage microscopique est détaillé dans les parties 1.1 et 1.2 de ce chapitre. Les conditions environnementales de l'auto-assemblage microscopique peuvent être reproduites lors de la séparation de deux objets fonctionnalisés, telles que les forces de Van der Waals, l'électrostatique, l'attraction hydrodynamique, les vitesses d'approche-extraction et l'application d'une force externe.
Plan de la thése
L'étude de la stabilité nécessite donc l'expression et la quantification de cette propriété en fonction des paramètres de contrôle. La caractérisation de la stabilité à l'échelle individuelle en fonction de paramètres de contrôle est au cœur de cette partie.

Caractérisation de la stabilité
Le premier paragraphe présente les outils informatiques méthodologiques, issus de la modélisation moléculaire, utilisés pour quantifier la stabilité à cette échelle. Enfin, dans le troisième paragraphe, l’approche individuelle est analysée puis dans le contexte de la stabilité des AA microscopiques basées sur l’ADN.
Problématique de l'estimation de la force
La méthode utilisée pour estimer une force à partir de l'énergie consiste à approximer l'énergie d'interaction par une fonction analytique [Daunay 07] basée sur une série de points. Cette valeur est la première estimation de la force d'interaction entre deux brins d'ADN complémentaires rapportée à nos collaborateurs du projet Golem.
Discussion
L'élasticité de l'ADN
La structure remarquable de la molécule d’ADN est responsable de l’une de ses propriétés les plus importantes : l’élasticité. Cette structure remarquable est à l’origine de la propriété élastique, propriété essentielle au processus d’hybridation.
La densité de répartition des brins d'ADN
Pour chacun d’eux, l’emplacement du centre de masse de chaque segment change de manière aléatoire autour de leurs articulations. Appliquer cette solution à la méthode d’approximation énergétique consisterait à effectuer cette approximation pour chaque segment.
Conclusion
Dans la première section, l'objectif est d'identifier plus précisément le système étudié et les outils nécessaires pour quantifier la stabilité. Enfin, la méthode d'estimation de la force d'interaction basée sur l'énergie d'interaction caractéristique est proposée dans la dernière section.
Quantication de la stabilité
Enquête sur les paramètres de contrôle de stabilité Cependant, plusieurs inconvénients rendent l'approche individuelle inappropriée et difficile à mettre en œuvre dans le contexte du Velcro à base d'ADN. La caractérisation de ce processus est possible à partir de l'estimation de la différence d'énergie libre de liaison.
Modèle du plus proche voisin
Algorithme du modèle du plus proche voisin
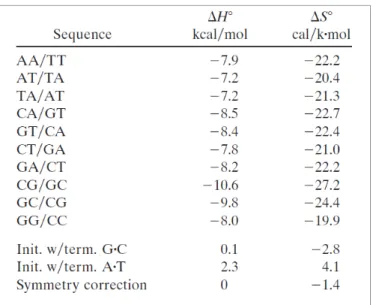
L'étude des paramètres de contrôle de stabilité comprend 5 couples de base : CG, GT, TT, TG, GA. Une fenêtre de paires de bases glisse le long de la représentation des deux séquences complémentaires hybridées.
Les paramètres intrinsèques
Par exemple, sur la figure 2.8.a, deux paires de bases sont illustrées : CA/GT et TG/AC appartenant respectivement à deux brins Velcro. La paire de paires de bases s’obtient en complétant la paire avec son complément.
Les paramètres environnementaux
La différence d'énergie libre ∆G37 pour la séquence dans les bases azotées peut être estimée pour une température fixe de 37 °C et une concentration de sel fixe [N a+] = 1M.
![Figure 2.10 - Evolution de ∆G pour une séquence de 120 bases en fonction de [N a + ] pour plusieurs valeurs xes de T.](https://thumb-eu.123doks.com/thumbv2/1bibliocom/464962.70326/49.892.68.789.192.512/figure-evolution-séquence-120-bases-fonction-valeurs-xes.webp)
Estimation de la force d'interaction à partir de ∆G
Les représentations graphiques des énergies d'interaction en fonction de la longueur de la séquence (en bases), pour ces deux séquences sont les droites DAT et DGC (figure 3.19). En raison de la linéarité de ces deux équations, leur dérivation donne deux valeurs constantes pour les forces d'interaction.
Récapitulatif
Ces deux valeurs sont respectivement la valeur moyenne minimale et la valeur moyenne maximale de la force d'interaction entre deux brins d'ADN pour les configurations de type PM. Cette conversion donne une valeur moyenne de la force d'interaction pour les configurations de type PM.
Principe général de l'AFM
Dans ce contexte expérimental, l'AFM s'écarte de sa fonction initiale de mesurer les forces d'interaction entre la pointe du faisceau et la surface du micro-objet. L’étalonnage du faisceau est donc une étape importante lors d’une expérience de mesure AFM.
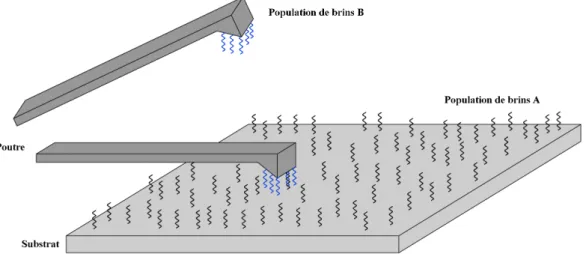
L'AFM pour les interactions biomoléculaires
Ceci est réalisé en mesurant et en représentant la déflexion de la poutre en fonction de sa position. La courbe est appelée courbe « d'approche/rétraction » (A/R) en raison du processus d'approche et de retrait du faisceau (ou du substrat) pendant la mesure.
Les variables de mesure et les paramétres expérimentaux
La force de rupture
Dans le cas particulier de la fonctionnalisation avec des brins d'ADN, plusieurs sauts correspondant à différents événements de cassure peuvent apparaître sur la courbe A/R de mesure des forces d'interaction dans l'axe de la double hélice. Sattin 04] pour mesurer les forces d'interaction entre différents brins d'ADN dans l'axe de la double hélice.
![Figure 3.5 - Protocole expérimental de Sattin et al. [Sattin 04] pour la mesure des forces d](https://thumb-eu.123doks.com/thumbv2/1bibliocom/464962.70326/59.892.107.736.475.825/figure-protocole-expérimental-sattin-sattin-teraction-diérents-hélice.webp)
Taux de chargement
Evans 97] par la théorie de la spectroscopie de force dynamique (DFS), et prouvé expérimentalement pour divers complexes protéiques [Janovjak 05, Odrowaz 06, Liu 08], ainsi que pour l'ADN [Strunz 99, Morll 07]. Ce temps varie en fonction de la force appliquée et du temps d'application de cette force.
Conlusion
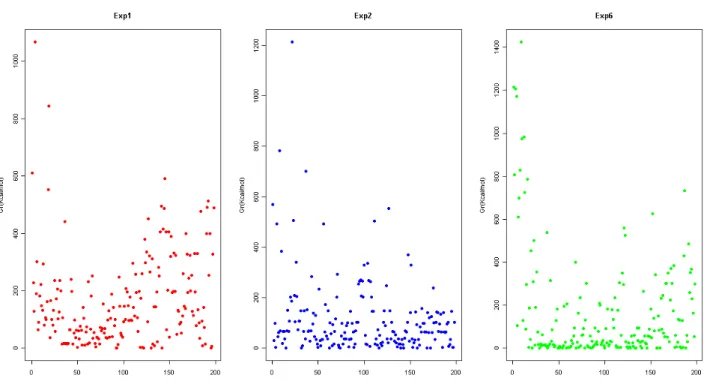
Caractérisation expérimentale de la stabilité A titre d'exemple, la courbe de la figure 3.10 illustre la mesure AFM obtenue pour la première itération de l'expérience Exp1 (v = 1µm/s, k = 0,1N/m, B). Il est important de faire la distinction entre les expériences dans lesquelles un bruit de mesure excessif empêche l'utilisation des données.
Méthode statistique pour l'identication des expériences signicatives
Pour l'estimation des forces de rupture, seules les expériences dont les mesures correspondent à des réponses moléculaires sont retenues grâce à l'analyse statistique présentée dans la section suivante. Dans l'hypothèse d'une dépendance due à des limitations inhérentes au protocole de mesure (manipulation de l'AFM, du matériel expérimental ou valeurs de paramètres expérimentaux inappropriées), la suppression de la partie d'approche des courbes A/R devrait permettre de distinguer les données significatives.
Identication des données exploitables
Moyennes et distributions des forces de ruptures
Conclusion
Variabilité de conguration et force de rupture
Le type de configuration adoptée par les deux brins dans cette phase dépend de la séquence de bases azotées des brins. Cette réponse dépend de la configuration adoptée lors de la phase d'approche, des paramètres environnementaux ainsi que des paramètres expérimentaux.
Etude des événements locaux de rupture
La superposition des valeurs de ∆Gnr sur ce diagramme est une méthode de classification des événements de rupture pour une évaluation de la stabilité d'un assemblage. Ainsi, pour les événements de catégorie 1, la variation d'énergie peut être plus importante que les prédictions théoriques en raison d'une sous-estimation de la distance de rupture prédite par l'hypothèse dr = n dbasis.
Discussion des résultats expérimentaux
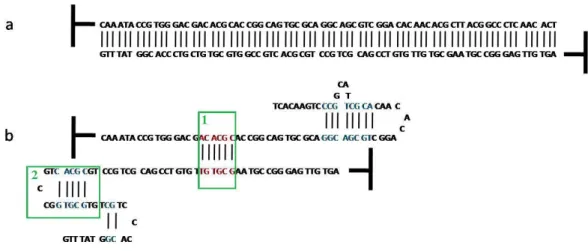
La caractérisation expérimentale de la stabilité du Velcro constitué de brins d'ADN de séquence S1 est réalisée au moyen d'expériences de mesure AFM. Il existe des recherches et une caractérisation expérimentale des spécificités clés de la programmation moléculaire de l'ADN.
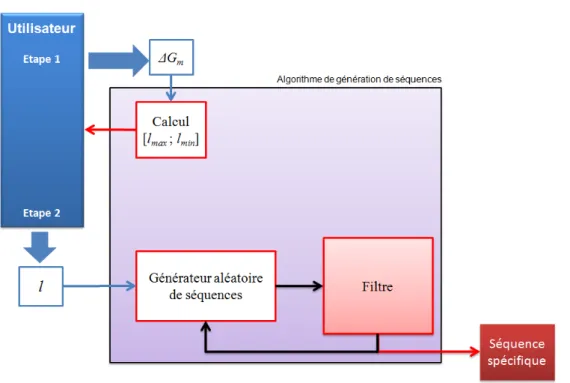
Principe de la génération de séquence
Pour éviter les cas B, C et D, chacun des deux segments xi etxj de la séquence produite doit être différent. Pour éviter le cas E, chacun des deux segmentsxi dhexRj de la séquence produite doit être différent.
Algorithme de génération de séquence
Analyse théorique d'une séquence générée 77 Le filtre ne fait que nier la spécificité de la séquence. La deuxième étape est la vérification de la spécificité de la séquence à travers le filtre annulé par les 3 règles.
Description des expériences
Analyse énergétique des événements de rupture locaux
Analyse expérimentale de la spécicité
Etude de la spécicité
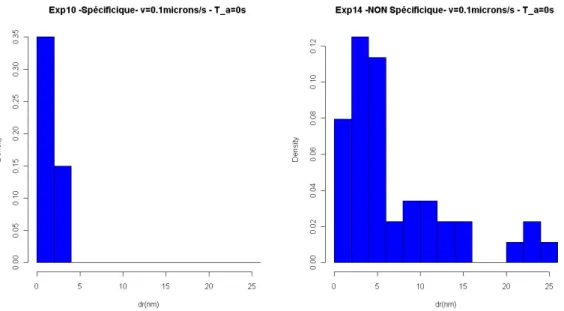
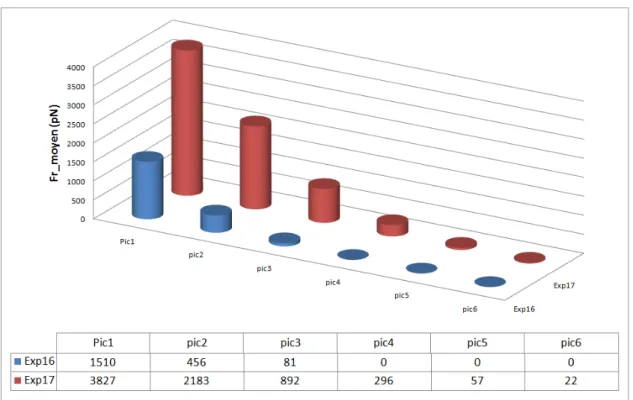
L'écart type évalué pour la force de rupture (tableau 4.4) et la distance de rupture (tableau 4.5) présente plusieurs caractéristiques. Comparaison de la répartition des forces de rupture mesurées pour les expériences Exp10 et Exp14.
Inuence de la vitesse sur la spécicité et la stabilité
Cependant, lorsque la distance de rupture est inférieure à cette valeur, il n’est pas possible de déduire le nombre de paires de brins en interaction. Comparaison de la répartition des distances de fracture mesurées pour les expériences Exp10 et Exp14.
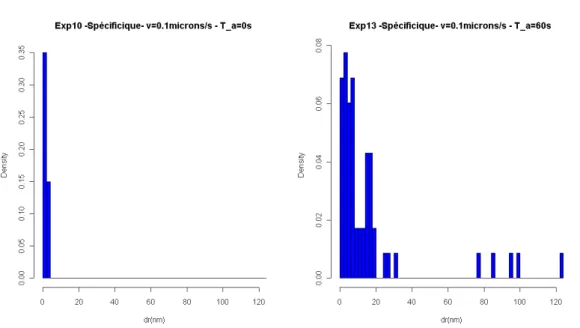
Inuence du temps d'attente
Comparaison de la répartition des forces de rupture mesurées pour les expériences Exp10(Ta=0s) et Exp13(Ta=60s). Comparaison de la répartition des forces de rupture mesurées pour les expériences Exp16(Ta=0s) et Exp17(Ta=60s).
Conclusion
Pour résoudre le problème de spécificité, un algorithme de génération de séquences est proposé dans ce chapitre. Ces expériences démontrent une meilleure stabilité et spécificité de la séquence conçue et valident l'approche proposée dans cette thèse.
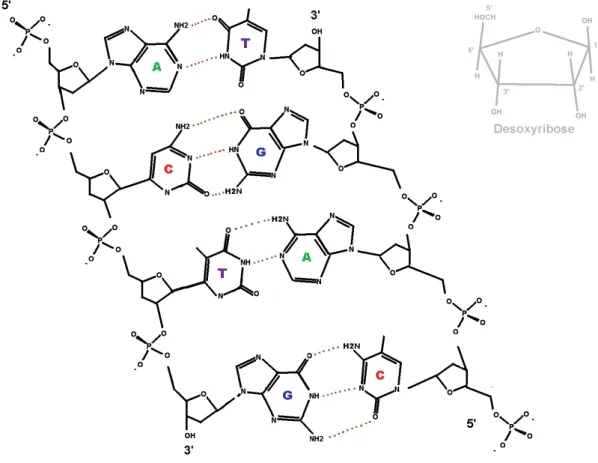
Structure 3D d'une double hélice d'ADN ; brin A (cgcganacgcc), brin B (ggcg-
Structures secondaires imparfaites : mismatch [Dimitrov 04]
Principe de l'A.A microscopique basé sur l'ADN
Axe hypothétique de la double hélice
Evolution de la force d'interaction estimée par extrapolation de l'énergie d'inter-
Modélisation par segmentation de l'hybridation dans les puces à ADN dans [Jaya-
Principe des puces à ADN. Hybridation entre deux populations de brins com-
Algorithme pour le calcul de ∆G . Une fenêtre de couple de paires de bases glisse
Tableau des paramètres standards du modèle du plus voisin pour l'ADN [Santa-
Les couples de paires signicatifs. a. les couples sont semblables, un des deux
Exemple d'une estimation de ∆G 37 pour une séquence CGTTGA [SantaLucia 98]. 36
Evolution de ∆G pour une séquence de 120 bases en fonction de T pour plusieurs
Energie d'interaction ∆G en fonction de la longueur de séquence pour les sé-
Principe général de l'AFM
Utilisation de l'AFM pour la caractérisation des surfaces. A gauche, une courbe
Principe d'une mesure des forces d'interaction biomoléculaires par l'AFM. a. For-
![Figure 1.3 - Structure 3D du complexe Cd1d-C6ph chez la souris [Schiefner 09]. En violet :Cd1d et en bleu : C6ph.](https://thumb-eu.123doks.com/thumbv2/1bibliocom/464962.70326/19.892.127.727.126.475/figure-structure-complexe-cd1d-c6ph-souris-schiefner-violet.webp)
![Figure 1.6 - Nanostructures polyhedrales [Yu He 08]. Images AFM et Cryo-EM. Les boites blanches indiquent les nanostructures.](https://thumb-eu.123doks.com/thumbv2/1bibliocom/464962.70326/23.892.174.684.746.1112/figure-nanostructures-polyhedrales-images-boites-blanches-indiquent-nanostructures.webp)
![Figure 1.7 - Moteur moléculaire protéique composé de myosine et de kinésine [Hess 04].](https://thumb-eu.123doks.com/thumbv2/1bibliocom/464962.70326/24.892.285.648.189.514/figure-moteur-moléculaire-protéique-composé-myosine-kinésine-hess.webp)
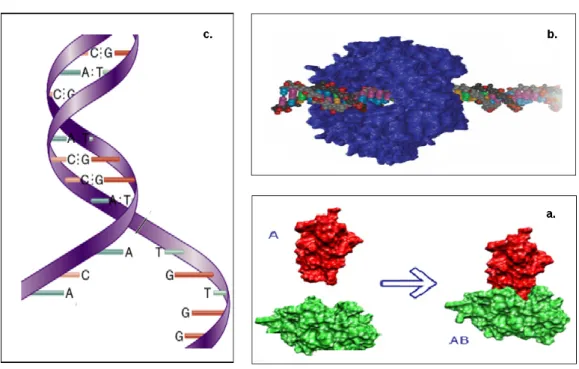
![Figure 1.12 - Structure 3D d'une double hélice d'ADN ; brin A (cgcganacgcc), brin B (ggcg- (ggcg-tatcgcg) [Beger 98].](https://thumb-eu.123doks.com/thumbv2/1bibliocom/464962.70326/29.892.170.684.290.577/figure-structure-double-hélice-adn-cgcganacgcc-tatcgcg-beger.webp)