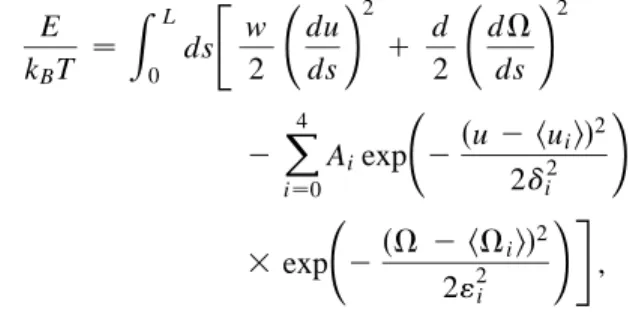Physique et Chimie de la Matière et des Matériaux Condensés, Possibilités de Surfaces et d'Interfaces, ULP, Strasbourg. Fin du soutien aux exercices de mécanique en 1ère année de DEUG, chimie, chimie biophysique. 1998 : Pierre Gelade, stage DEA de Physique des Liquides Paris VI, étude de la réponse électrique des microtubules.
2000 : Thomas Andlauer, stage de deuxième année de BTS Biotechnologie Strasbourg, stage de Biologie Moléculaire (6 semaines). De 1997 à 2003 j'ai été membre de la 28ème commission de spécialistes de la section locale à Strasbourg et membre du bureau de cette commission. Depuis juin 2007, je suis membre de la commission spécialisée section 28 de l'Université Paris VI.
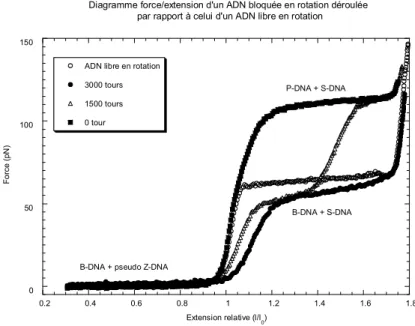
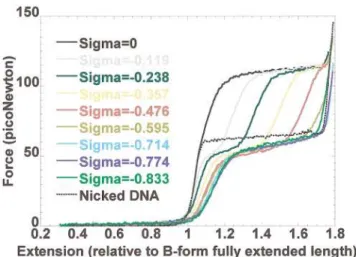
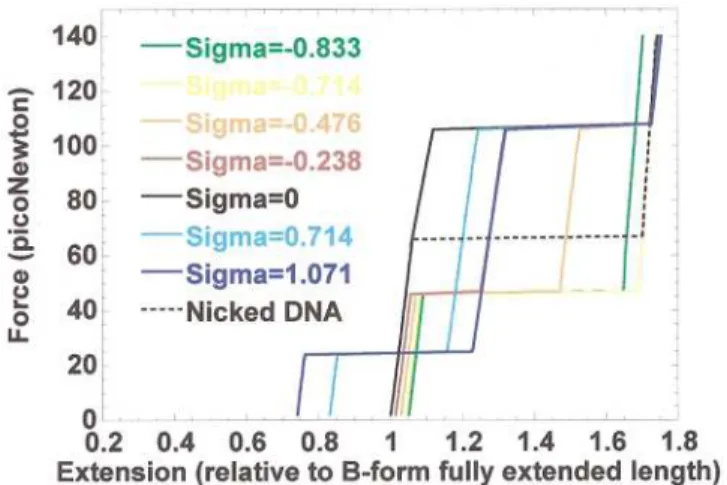
Au cours des cinq à six dernières années, le développement des techniques de mesure de force et de micromanipulation a permis l’étude de molécules biologiques à l’échelle d’une seule molécule. Cette transition est initialement observée avec des molécules chimiquement libres de tourner autour de l’axe de la double hélice. Des mesures de la cinétique de polymérisation de la protéine RecA autour de l'ADN ont été réalisées sur un seul ADN-A.
La protéine RecA interagit avec la partie thermiquement étirée de la molécule d'ADN et bloque la structure allongée de cette dernière.

RecA binding to a single double-stranded DNA molecule
A possible role of DNA conformational fluctuations
Etude du repliement secondaire post et en cours de transcription d'une molécule d'ARN
- Etude des structures secondaires d'un ARN par dénaturation mécanique
- Repliement de l'ARN en cours de transcription
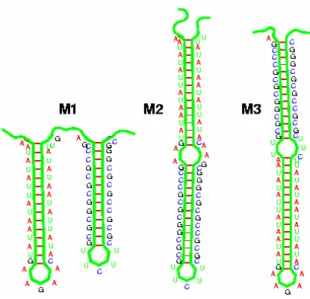
Local rearrangements, such as the formation of simple stem-loops, occur quite rapidly (< 1 ms) under low pulling force (or no force) and the number of possible hairpins (with a small loop) is proportional to the length of the stretched region of the RNA molecule. A histogram of the rupture force and a set of unfolding and refolding-extension force curves are shown for each motif in Figures 2A and B. For M1 and M2, these results can be simply attributed to the first opening of a weak (AU) helix followed by a stronger (GC) helix with greater applied force.
Stretching is induced by a slowly varying rigid constraint on the end-to-end distance of the RNA-dsDNA tweezers construct (rate ± 300 nm/s in ± 2 nm steps). In particular, they allow for the identification of likely intermediate structures involved in the refolding hysteresis, which mainly correspond to the formation of two non-native helices originating from each strand of the strong (GC) helix, Figure 3. As the piezo stage is displaced, the force starts to rise due to the elastic response of the DNA handles.
Colors correspond to consecutive stretching rounds of the same molecule (folding hysteresis not shown for clarity). Mechanical unfolding over the full stretch range of the molecule presents a characteristic unfolding plateau between 11 and 15 pN. Such a relative Spearman correlation coefficient is much more sensitive to specific unfolding signals, compared to the absolute Spearman correlation of the actual unfolding curves, which gives a higher but less discriminating correlation coefficient (see Fig 6 for details).
Spearman's correlation coefficients rs [24] are given between the deviations from the fit of the median line of the unfolded plateaus (see text and Figure 6). Therefore, the mechanical breaking of the natural structure does not occur with the successive opening of the entire native domains. A portion of the unpaired bases then typically rearranges into different helices that partially compensate for the sudden release of mechanical stress.
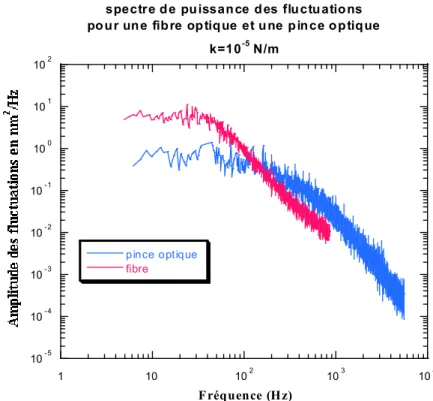
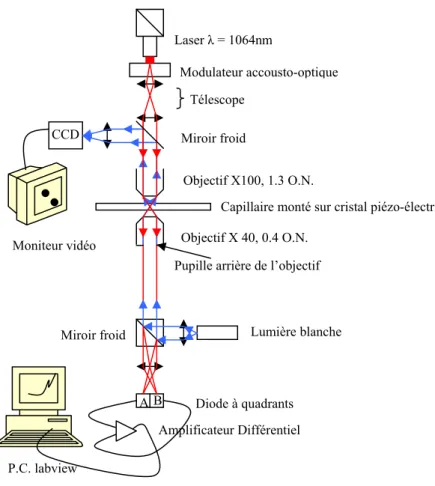
32] recently reported experimental results on the mechanical unfolding of the L-21 derivative of the Tetrahymena thermophila ribozyme, a 390-nucleotide catalytic RNA. Different hysteresis responses to applied force are presented for different parts of the molecule or in the presence of specific antisense oligos. The experiments described here were performed with a 50% reduction in laser intensity, which sets the stiffness of the optical trap around 7×10-5 N/m.
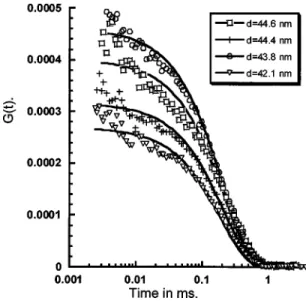
The voltage difference VA−VB which is proportional to the distance from the bead to the center of the trap is further amplified and filtered at 300 Hz by a low noise amplifier (SR-50, Stanford Research Instrument). The acquisition rate is 300 Hz, making the duration of the stretch/relaxation experiment approximately 10-20 seconds.
Subnanometric measurements of evanescent wave penetration depth using total internal reflection microscopy combined with fluorescent
- Motilité bactérienne et polymorphisme
- Projet de Recherche
- Introduction: généralités sur les plasmides
- Méthodes expérimentales
- Perspectives
- Publications
- Publication sans comité de lecture
- Participation à des congrès sous forme d'affiche; Ecoles
- Conférences orales
Ils sont attachés au corps de la bactérie et représentent un élément passif de motilité. D'un point de vue mécanique, le mouvement est assuré par trois éléments : un flagelle, un moteur et un crochet, identifiés par microscopie électronique. À l'intérieur de la membrane bactérienne, un moteur rotatif entraîne le mouvement du flagelle. Le flagelle est le résultat de la polymérisation d’une seule sous-unité protéique appelée flagelline.
A la fin de la période de commutation, la bactérie a une seconde direction aléatoire vers laquelle elle reprend son mouvement. La transition conformationnelle des hélices flagellaires peut être induite par des changements de pH ou de force ionique dans l'environnement bactérien. Nous avons développé un procédé de repolymérisation de la flagelline pour obtenir des flagelles d'env. 10 à 15 micromètres (environ 4 à 7 distances d'hélice).
L’un des principaux problèmes qu’il rencontra fut le greffage de la boule et de la lame de microscope aux extrémités du flagelle. Pour un même réplicon, c'est la combinaison de la régulation de la réplication de l'ADN et de la division des plasmides en cellules filles qui détermine le nombre moyen de copies plasmidiques dans une bactérie. En plus de l'étiquette antibiotique présente sur le plasmide, une séquence codant pour la protéine fluorescente mOrange est incorporée à l'aide de techniques standards de biologie moléculaire.
Trois plasmides ont été construits et mesurés en laboratoire : la séquence codant pour l'expression de la protéine fluorescente mOrange (excitation 548 nm, émission 562 nm) est identique pour tous les plasmides étudiés. L'excitation de fluorescence est obtenue via le port d'épifluorescence du microscope par un laser fixe émettant à 488 nm. Ce type de dispositif permet un éclairage homogène de la zone de passage bactérien sur une surface de côté égal à la.
La principale limitation de la durée de capture est due à l'augmentation du flux bactérien dans les canaux microfluidiques, qui provoque une saturation du signal de détection. La mesure de l'intensité de la fluorescence verte quantifie la variation de l'expression génétique bactérienne. En utilisant notre système bien caractérisé de mesure du nombre de copies de plasmides dans les bactéries, nous aborderons le problème de la compétition au sein d'une population bactérienne entre le coût métabolique de la maintenance des plasmides et la pression de sélection du milieu de culture.
Dans une première série d'expériences, on peut très simplement se demander quelle sera la réaction de la bactérie si elle est cultivée en l'absence d'antibiotique. Le basculement vers l'un ou l'autre des réservoirs se fera en fonction de l'intensité de fluorescence détectée lors du passage des bactéries en amont de la jonction.