CAROLINA TORRES ALEJO
Coronavírus em codornas: ocorrência, diversidade molecular e estudo do papel epidemiológico das codornas como reservatório para a bronquite infecciosa das
galinhas
CAROLINA TORRES ALEJO
Coronavírus em codornas: ocorrência, diversidade molecular e estudo do papel epidemiológico das codornas como reservatório para a bronquite infecciosa das
galinhas
Dissertação apresentada ao Programa de Pós-Graduação em Epidemiologia Experimental Aplicada às Zoonoses da Faculdade de Medicina Veterinária e Zootecnia da Universidade de São Paulo para obtenção do título de Mestre em Ciências
Departamento:
Medicina Veterinária Preventiva e Saúde Animal
Área de concentração:
Epidemiologia Experimental Aplicada às Zoonoses
Orientador:
Prof. Dr. Paulo Eduardo Brandão
Autorizo a reprodução parcial ou total desta obra, para fins acadêmicos, desde que citada a fonte.
DADOS INTERNACIONAIS DE CATALOGAÇÃO-NA-PUBLICAÇÃO
(Biblioteca Virginie Buff D’Ápice da Faculdade de Medicina Veterinária e Zootecnia da Universidade de São Paulo)
T.2553 Torres, Carolina Alejo
FMVZ Coronavírus em codornas: ocorrência, diversidade molecular e estudo do papel epidemiológico das codornas como reservatório para a bronquite infecciosa das galinhas / Carolina Torres Alejo. -- 2012.
76 p.
Dissertação (Mestrado) - Universidade de São Paulo. Faculdade de Medicina Veterinária e Zootecnia. Departamento de Medicina Veterinária Preventiva e Saúde Animal, São Paulo, 2012.
Programa de Pós-Graduação: Epidemiologia Experimental Aplicada às Zoonoses.
Área de concentração: Epidemiologia Experimental Aplicada às Zoonoses.
Orientador: Prof. Dr. Paulo Eduardo Brandão.
FOLHA DE AVALIAÇÃO
Nome: TORRES, Carolina Alejo
Título: Coronavírus em codornas: ocorrência, diversidade molecular e estudo do papel
epidemiológico das codornas como reservatório para a bronquite infecciosa das galinhas
Dissertação apresentada ao Programa de Pós-Graduação em Epidemiologia Experimental Aplicada às Zoonoses da Faculdade de Medicina Veterinária e Zootecnia da Universidade de São Paulo para obtenção do título de Mestre em Ciências
Data:___/___/___
Banca examinadora
Prof. Dr. _______________________________________________________ Instituição: ____________________ Julgamento: _________________
Prof. Dr. ______________________________________________________ Instituição: ____________________ Julgamento: _________________
A mis papás Luis y Vilma
y a mis hermanos Luisa y Daniel
por todo el amor, apoyo incondicional,
y por siempre incentivarme a correr atrás
ABSTRACT
TORRES, C. A. Coronavirus in quails: occurrence, molecular diversity and the role of quails as reservoir for avian infectious bronchitis virus [Coronavírus em codornas: ocorrência, diversidade molecular e estudo do papel epidemiológico das codornas como reservatório para a bronquite infecciosa das galinhas]. 2012. 76 f. Dissertação (Mestrado em Ciências) – Faculdade de Medicina Veterinária e Zootecnia, Universidade de São Paulo, São Paulo, 2012.
This study aimed to investigate the occurrence and molecular diversity of avian coronavirus in quails and laying hens, raised on the same farms and determine the role of quails in the epidemiology of avian infectious bronchitis (IB). To this end, pools of lungs, trachea, female reproductive tracts, kidneys and enteric contents were collected from quails and laying hens flocks with IB-like symptoms, co-housed in farms located in Sao Paulo and Espírito Santo states, Brazil, during 2009-2010. Chickens and quails samples were screened for IBV with an RT-PCR to the 3’UTR and positive samples were submitted to RT-PCRs to the dependent RNA-polymerase gene (RdRp) and two different RT-PCRs to the spike gene, including a typing-multiplex one. Amplicons of 3’UTR (from quails samples) were cloned and sequenced. Two other RT-PCRs were used to detect the avian metapneumovirus (aMPV) and Newcastle disease virus (NDV). Avian coronavirus was found in all types of samples analyzed in chickens and quails raised on the same farms, aMPV subtype B was found in chickens and the NDV was not observed in any samples. All avian coronavirus found were classified as variants by multiplex RT-PCR, however, DNA sequences for gene S were not obtained. Based on the DNA sequences for genes encoding the protein RdRp and the 3'UTR region can be show that avian coronavirus in quails are closely related to avian infectious bronchitis virus, with a molecular phylogeographic diversity for quails viruses; thus, quails might act as reservoirs for avian coronaviroses when in close contact with other avian species.
LISTA DE FIGURAS
Figura 1 - Sinais clínicos observados nas granjas do estudo. A: codorna jovem apresentando lacrimejamento; B: galinha jovem apresentado descarga nasal, lacrimejamento e tosse, decorrente da dificuldade respiratória; C e D alteração em casca (fina e porosa), no formato e na pigmentação em ovos de codorna e galinhas, respectivamente (D), São Paulo 2009- 2010... 30 Figura 2 - Distribuição das granjas estudadas no período de 2009-2010 no
município de Bastos/SP. A figura menor à direita mostra a localização do município de Bastos no estado de São Paulo e na área maior estão indicadas as distâncias entre as granjas paulistas do estudo. As setas indicam a localização dos galpões das codornas e das galinhas nas granjas estudadas e os pontos são as coordenadas obtidas a partir do
sistema de posicionamento global (Garmin GPSMAP®
60CS)... 31 Figura 3 - Representação esquemática das regiões amplificadas pela semi-nested
RT-PCR para a região 3’UTR e pela RT-PCR para a ORF 1B do gene
que codifica a RNA-polimerase RNA dependente... 35 Figura 4 - Representação esquemática da amplificação parcial pela semi-nested
RT-PCR para a região codificadora da subunidade S1 da proteína S de IBV... 37 Figura 5 - Representação esquemática da região flanqueadora da inserção do
amplicon de179pb no vetor plasmidial pTZ57R/T... 41 Figura 6 - Gel de agarose 1.5% corado com brometo de etídeo 0,5g/Ml do
produto (320pb) da PCR direcionada para a região flanqueadora da inserção do plasmídeo contendo o inserto de 179pb referente a
3’UTR... 53
Figura 7 - Árvore genealógica de distância com o algoritimo Neighbor-joining para
sorotipos/genótipos clássicos (com o numero de acesso do Genbank) e as amostras incluídas no presente estudo (o quadro representa as amostras das codornas, o triangulo as das galinhas e o rombo amostras de aves silvestres brasileiras). Os números acima de cada nó representam os valores de bootstrap para 1000 repetições (apenas os valores maiores que 50% estão demonstrados). A barra representa o número de substituições de nucleotídeos por sítio... 54 Figura 8 - Árvore genealógica de distância com o algoritimo Neighbor-joining para
LISTA DE GRÁFICOS
Gráfico 1 - Frequência de Gammacoronavirus nas granjas (galinhas e codornas) estudadas durante o período de 2009-2010... 46 Gráfico 2 - Ocorrência de Gammacoronavirus em codornas nos anos de 2009
e 2010 nas amostras estudadas dos municípios de Santa María de Jetibá/ES e Bastos/SP... 47 Gráfico 3 - Ocorrência de Gammacoronavirus em galinhas poedeiras nos
anos 2010 e nas amostras estudadas dos municípios de Santa Maria de Jetibá/ES e Bastos/SP... 47 Gráfico 4 - Ocorrência de Gammacoronavirus em codornas e galinhas
LISTA DE QUADROS
Quadro 1 - Descrição da localização, número de aves/lote e idade das aves nas granjas estudadas durante o período de 2009– 2010... 29 Quadro 2 - Resultados da genotipagem das amostras segundo o ano de
LISTA DE TABELAS
Tabela 1- Frequência de Gammacoronavirus nas granjas (galinhas e codornas) estudadas durante o período de 2009-2010... 46 Tabela 2 - Identidade média (em porcentagem) de nucleotídeos da região
LISTA DE ABREVIATURAS E SIGLAS
BLAST/n Basic Local Alignment Search Tool
cDNA DNA complementar
DEPC dietil-pirocarbonato
dNTP deoxinucleosídeo-trifosfato DNA ácido desoxirribonucleico DTT Dithiothreitol
et al. e colaboradores
EUA Estados Unidos da América
g aceleração da gravidade terrestre (9,8 m/s2)
g Gramas
kb Quilobase
kDa Quilo Dalton
M Molar
Min minuto de hora
mM Milimolar
M-MLV Moloney Murine Laeukemia Vírus
ng Nanogramas
mL Mililitro
µg Micrograma
µL Microlitro
ORF open reading frame
pb pares de bases
PCR reação em cadeia pela polimerase
pmol Picomoles
q.s.p Quantidade suficiente para RNA ácido ribonucleico
RT transcrição reversa
S Espícula
LISTA DE SÍMBOLOS
% Porcento
ºC graus Celsius
+ Positivo
SUMÁRIO
1 INTRODUÇÃO ... 21
2 OBJETIVOS ... 27
3 MATERIAL E MÉTODOS ... 28
3.1GRANJAS ... 28
3.2 AMOSTRAS ... 30
3.3 CONTROLES POSITIVOS E NEGATIVOS ... 32
3.4 EXTRAÇÃO DE RNA ... 32
3.5 REAÇÃO DE TRANSCRIÇÃO-REVERSA (RT) ... 32
3.6 DETECÇÃO DE CORONAVÍRUS AVIÁRIO MEDIANTE A REAÇÃO EM CADEIA PELA POLIMERASE SEMI-NESTED (SEMI-NESTED RT- PCR) DIRIGIDA A REGIÃO 3’UTR...33
3.7 AMPLIFICAÇÃO DE SEGMENTOS PARCIAIS DO GENE CODIFICADOR DA RNA-POLIMERASE RNA DEPENDENTE MEDIANTE UMA REAÇÃO EM CADEIA PELA POLIMERASE (PCR) ... 34
3.8 REAÇÃO DE TRANSCRIÇÃO-REVERSA SEGUIDA DE REAÇÃO EM CADEIA PELA POLIMERASE MULTIPLEX NESTED (MULTIPLEX NESTED RT-PCR) PARA GENOTIPAGEM DAS AMOSTRAS POSITIVAS PARA O CORONAVÍRUS AVIÁRIO...35
3.9 SEMI NESTED RT-PCR PARA AMPLIFICAÇÃO PARCIAL DA REGIÃO CODIFICADORA DA SUBUNIDADE S1 DA PROTEÍNA S DE IBV ... 36
3.10 DETECÇÃO DE METAPNEUMOVIRUS AVIÁRIO MEDIANTE A REAÇÃO EM CADEIA PELA POLIMERASE SEMI-NESTED (SEMI-NESTED RT- PCR) ... 38
3.11 DETECÇÃO DO VÍRUS DA DOENÇA DE NEWCASTLE MEDIANTE A REAÇÃO EM CADEIA PELA POLIMERASE (RT- PCR) ... 39
3.12.1 Purificação dos amplicons a partir do gel de agarose ... 40
3.12.2 Inserção no plasmídeo, transformação bacteriana e extração plasmidial ... 40
3.13 SEQÜENCIAMENTO DE DNA ... 41
3.14 EDIÇÃO DE SEQUENCIAS DE DNA ... 42
3.15 ANÁLISE FILOGENÉTICA ... 43
3.16 FORMA DE ANÁLISE DOS RESULTADOS ... 43
4 RESULTADOS ... 45
4.1 DETECÇÃO DE CORONAVÍRUS AVIÁRIOS MEDIANTE A REAÇÃO EM CADEIA PELA POLIMERASE SEMI-NESTED (SEMI-NESTED RT- PCR) DIRIGIDA A REGIÃO 3´UTR... ... 45
4.2 AMPLIFICAÇÃO DE SEGMENTOS PARCIAIS DO GENE CODIFICADOR DA RNA-POLIMERASE RNA DEPENDENTE MEDIANTE UMA REAÇÃO EM CADEIA PELA POLIMERASE (PCR) ... 48
4.3 REAÇÃO DE TRANSCRIÇÃO-REVERSA SEGUIDA DE REAÇÃO EM CADEIA PELA POLIMERASE MULTIPLEX NESTED (MULTIPLEX NESTED RT-PCR) PARA GENOTIPAGEM DAS AMOSTRAS POSITIVAS PARA O CORONAVÍRUS AVIÁRIO... ... 49
4.4 SEMI NESTED RT-PCR PARA AMPLIFICAÇÃO PARCIAL DA REGIÃO CODIFICADORA DA SUBUNIDADE S1 DA PROTEÍNA S DE IBV ... 51
4.5 DETECÇÃO DE METAPNEUMOVIRUS AVIÁRIO E VÍRUS DA DOENÇA DE NEWCASTLE MEDIANTE A REAÇÃO EM CADEIA PELA POLIMERASE SEMI-NESTED (SEMI-SEMI-NESTED RT- PCR) ... 52
4.6 CLONAGEM DO PRODUTO DA PCR DAS AMOSTRAS POSITIVAS DE CORDORNAS PARA 3’UTR ... 52
4.7 SEQÜENCIAMENTO DE DNA ... 53
4.8 ANÁLISE FILOGENÉTICA ... 53
5 DISCUSSÃO ... 57
6 CONCLUSÕES ... 67
21
1 INTRODUÇÃO
As codornas japonesas (Coturnix coturnix) são originárias do norte da África, da Europa e da Ásia, pertencendo à família Phasianidae e à sub-família Perdicinidae, sendo, portanto, da mesma família das galinhas e perdizes (PINTO et al., 2002).
São produzidas principalmente para carne na Europa e América, e para ovos na Ásia e no Brasil (MINVIELLE, 2004), onde a criação foi introduzida no início da década de 50, destacando-se no mercado agropecuário brasileiro pela excelente atividade produtiva, por requerer baixos custos com investimento inicial e mão-de-obra, utilizando pequenas áreas e proporcionando rápido retorno financeiro (PINTO et al., 2002; MINVIELLE, 2004).
De acordo com o levantamento sobre a Produção da Pecuária Municipal do Instituto Brasileiro de Geografia e Estatística (IBGE) (2010) a produção de ovos de codorna foi de 232 milhões de dúzias, sendo São Paulo (SP) o principal estado produtor e Espírito Santo (ES) o segundo. Os munícipios de Bastos (SP) e Santa Maria de Jetibá (ES) são uns dos mais importantes, sendo que o primeiro mantem 59,3% do total nacional da produção nacional.
O estado de São Paulo é também o maior produtor nacional de ovos de galinha, respondendo com uma participação de 28.7% (633 milhões de dúzias no segundo trimestre de 2011) (IBGE, 2011).
22
Dentre os vírus de elevada importância para a exploração avícola, tanto em função das perdas econômicas, quanto pela possibilidade de transmissão entre as espécies de aves, destacam-se o coronavírus aviário, o vírus da doença de Newcastle e o metapneumovírus aviário (COOK; CAVANAGH, 2002; CAVANAGH, 2007; MILLER, DECANINI; AFONSO, 2010)
Os coronavírus, nome proveniente da palavra coroa (do latim corona) sugerido em 1968 devido à morfologia observada no microscópio eletrônico, (WEISS; NAVAS-MARTIN, 2005), são classificados na ordem Nidovirales, família Coronaviridae, subfamília Coronavirinae, subdividida nos gêneros Alphacoronavirus, Betacoronavirus e Gammacoronavirus (ICTV, 2009).
Nos gêneros Alphacoronavirus e Betacoronavirus encontram-se os coronavírus dos mamíferos, enquanto o Gammacoronavirus contem as espécies Beluga wale coronavirus (BWCoV) e Coronavírus aviário (avian coronavirus), esta última incluindo o Vírus da Bronquite Infecciosa das Galinhas (IBV, Infectious Bronchitis Virus) e o Coronavírus dos Perus (TCoV), tendo sido descritos também coronavírus em faisões (PhCoV), patos (DCoV) e pombos (PCoV) (CAVANAGH, 2005; MCKINLEY; HILT; JACKWOOD, 2008). No presente trabalho, os termos Coronavírus aviário (no singular) e coronavírus aviários (no plural) serão utilizados indistintamente quando não se fizer referência específica ao IBV ou a outro tipo de Coronavírus aviário.
Doenças causadas por coronavírus já foram descritas há mais de 70 anos, mas esta família de vírus permaneceu relativamente obscura por décadas. No entanto, em 2003, quando se tornou claro que um novo coronavírus humano foi responsável por uma grave síndrome respiratória aguda (SARS), o coronavírus se tornou muito mais conhecido. A origem da SARS levanta questões interessantes sobre a evolução e a especificidade dos coronavírus pelas espécies (WEISS; NAVAS-MARTIN, 2005).
23
O envelope é composto por uma camada dupla de lipídeos, na qual estão localizadas três proteínas, no caso do Coronavírus aviário: a glicoproteína de espícula (S), a proteína de membrana (M) e a pequena proteína de membrana ou proteína de envelope (E) (LAI; CAVANAGH, 1997; CHU et al., 2006). Além disso, encontra-se a proteína de nucleocapsídio (N) associada com o genoma viral (CAVANAGH, 2007).
A proteína N é multifuncional, facilita a síntese de RNA, mas também possui um papel importante na imunidade mediada por células, na formação de partículas virais e provavelmente facilita o transporte do genoma viral desde o sítio de síntese até o local de formação das partículas virais (VERHEIJE et al., 2010).
A proteína S é uma glicoproteína do tipo I que forma os peplômeros das partículas virais (WEISS; NAVAS-MARTIN, 2005), oligomeriza-se na membrana do retículo endoplasmático e insere-se no viron através de interações não covalentes com as proteínas da membrana (GODEKE et al., 2000). Tem um peso molecular de 180 kDa e é clivada nas subunidades S1 e S2 (CAVANAGH, 2007). S1 é a responsável pela ligação aos receptores celulares (CASAIS et al., 2003), tropismo celular, especificidade pela espécie hospedeira, patogenicidade, indução de anticorpos neutralizantes e reposta imune celular e emergência de tipos variantes em no caso do coronavírus aviário (CASAIS et al., 2003; MCKINLEY; HILT; JACKWOOD, 2008; MONTASSIER et al., 2010); enquanto que S2 tem capacidade de fusão das membranas virais e celulares para a liberação do genoma no citoplasma (sítio de replicação do RNA) e formação de sincícios, sendo assim responsável pelo efeito citopático (CAVANAGH, 2005).
A glicoproteína de membrana M (de aproximadamente 230 aa) tem um papel importante na formação do envelope e do core do vírion, enquanto que a pequena proteína de envelope (E), composta de 84 a 109 aa, é necessária para a montagem das partículas virais (CAVANAGH, 2007).
24
descontínuo de transcrição que leva a geração de um conjunto de mRNAs co-terminais subgenômicos. (STEPHENSEN; CASEBOLT; GANGOPADHYAY, 1999; SAWICKI; SAWICKI; SIDDELL, 2007).
O RNA genômico dos coronavírus aviários tem um tamanho de 27kb, sendo de fita simples de sentido positivo (LAI; CAVANAGH, 1997; CAVANAGH, 2005). O número de genes varia entre os coronavírus, em coronavírus aviários o RNA genômico inclui os genes 3 e 5 que codificam as pequenas proteínas acessórias não estruturais 3a, 3b e 3c e 5a e 5b e podem ter um papel importante na patogênese (CASAIS et al., 2005). Nas terminações 5‘ e 3’ do genoma, estão localizadas regiões não-traduzidas (untranslated regions, UTRs) que atuam nos processo de replicação genômica e transcrição de mRNA (MASTERS, 2006).
As infecções por Coronavírus aviário não estão necessariamente associadas a sinais clínicos, visto que estes vírus foram também detectados no intestino de aves assintomáticas, incluindo pavões, marrecos (LIU et al., 2005), pombos selvagens (FELIPPE et al., 2010), gansos e pato-real, (JONASSEN et al., 2005) e diversas espécies de aves silvestres em cativeiro (CARDOSO et al., 2011).
Quanto ao IBV, patógeno epiteliotrópico de aves domésticas altamente contagioso em galinhas de todas as idades (MCKINLEY; HILT; JACKWOOD, 2008), a importância reside em perdas econômicas pelo retardo do crescimento, infertilidade, falsas poedeiras, declínio na produção de ovos, ovos com alterações internas e na casca, diarréia, aumento da susceptibilidade às infecções secundárias e moderada a grave mortalidade (CAVANAGH, 2001; CAVANAGH, 2003; SAIF, 2004; MENDONÇA PITANGUI et al., 2009; VILLAREAL, 2010a).
25
2005; OIE, 2011). As infecções com vírus virulento causam três formas clínicas bem definidas: (1) velogênica viscerotrópica, (2) velogênica neurotrópica e (3) mesogênica. O NDV pode se apresetar ainda como um vírus de baixa virulência (lentogênico), também classificado como entérico, de infecção assintomática sem sinais respiratórios (ALDOUS; ALEXANDER, 2001; MILLER; DECANINI; AFONSO, 2010).
O Metapneumovírus aviário, também denominado vírus da rinotraqueíte dos perus (TRT), pertence ao gênero Metapneumovirus, família Paramyxoviridae, sub-família Pneumovirinae e contém um RNA não-segmentado, de sentido negativo e um genoma de aproximadamente 13 kb, que codifica oito proteínas (YU et al., 1992; EASTON; DOMACHOWSKE; ROSENBERG, 2004). Causa infecção do trato respiratório superior em perus e em outras espécies aviárias (COOK; CAVANAGH, 2002), sendo associado à síndrome da cabeça inchada (SHS) em frangos e matrizes pesadas. Tanto em perus quanto em galinhas, foi descrita uma queda transitória na produção de ovos e/ou um aumento em anormalidades nos mesmos após infecção pelo aMPV (JONES, 1996; COOK et al., 2000).
Quatro subtipos (A, B, C e D) foram reconhecidos com base nas diferenças das sequências do gene que codifica a glicoproteína G (responsável pela ligação do vírus ao receptor celular) e em propriedades sorológicas (BAYON-AUBOYER et al., 2000; COOK; CAVANAGH, 2002), sendo que no Brasil já foram descritos os subtipos A e B em galinhas, perus e aves silvestres e sinatrópicas (D’ARCE et al., 2005; CHACÓN et al., 2007; CHACÓN et al., 2011a; FELIPPE et al., 2011).
26
O primeiro relato de coronavírus em codornas ocorreu em 2000 a partir de uma amostra isolada, proveniente do Estado de Rondonia/ Brasil de uma granja de codornas adultas sem histórico de doença e vacinação contra IBV (DI FABIO et al., 2000), para qual se demonstrou baixa identidade antigênica com os sorotipos de IBV Massachusetts, Connecticut, e D207.
Em estudos de transmissão do coronavírus da SARS a diversas aves, incluído codornas, demonstrou-se que este coronavírus não se transmitia para estas espécies (SWAYNE et al., 2004; WEINGARTL et al., 2004), evidenciando a ausência de susceptibilidade das aves a este vírus específico.
Em 2005, uma síndrome entérica foi observada em codornas criadas em uma fazenda no sul da Itália, sendo um coronavírus detectado no conteúdo intestinal destes animais, denominado de Quail coronavirus (QCoV). Com o estudo de fragmentos genômicos do gene codificador da RNA-polimerase RNA dependente, observou-se uma identidade de 16-18% com o IBV, sugerindo que especificamente este QCoV não é uma variante do IBV. Em contraste, a sequência de aminoácidos da porção S1 do QCoV teve 79-81% de identidade com as amostras de TCoV, o que pode significar que o QCoV seja um coronavírus similar ao dos perus (CIRCELLA et al., 2007).
Assim sendo, em relação ao coronavírus que infectam codornas, há ainda pouca clareza sobre sua caracterização e diversidade moleculares, bem como sobre o papel deste hospedeiro na epidemiologia das coronaviroses aviárias. Este conhecimento não é apenas essencial para se entenderem as relações genealógicas entre as diferentes espécies de coronavírus de aves, mas também para que se possam ter inferências sobre proximidade antigênica entre as mesmas e sobre sua transmissão a outras espécies de aves, para o desenvolvimento de medidas de profilaxia e controle adequadas.
27
2 OBJETIVOS
Este estudo teve por objetivos:
Pesquisar a ocorrência de coronavírus aviários em codornas e galinhas provenientes de granjas com relatos de manifestações clínicas compatíveis com bronquite infecciosa das galinhas a partir de amostras de órgãos e de conteúdo entérico.
Estudar a diversidade molecular dos coronavírus encontrados com base em sequencias de DNA para os genes codificadores das proteínas RNA-polimerase RNA-dependente e de espícula e da região não-traduzida 3’.
28
3 MATERIAL E MÉTODOS
3.1 GRANJAS
As aves do estudo eram procedentes de granjas de codornas e galinhas poedeiras criadas em mesmas propriedades. As granjas estavam localizadas no Estado de São Paulo e Espírito Santo, nos municípios de Bastos e Santa Maria de Jetibá, respectivamente (Quadro 1). Nestas granjas, havia histórico de galinhas e codornas com sinais respiratórios, dentre eles: descargas nasais, lacrimejamento, conjuntivite, tosse e sinais reprodutivos como queda na produção de ovos, alteração de casca (fina e porosa) ou alteração no formato dos ovos e falsas poedeiras (Figura 1).
Em todas as granjas estudadas, o esquema de vacinação das galinhas compreendia a primo vacinação de IBV com o sorotipo Massachusetts atenuado e reforços de vacina inativada com o mesmo sorotipo; vacinação com o subtipo A e B de aMPV e a vacina lentogênica de VDN.
29
Quadro 1 - Descrição da localização, número de aves/lote e idade das aves nas granjas estudadas
durante o período de 2009 – 2010
Ano Estado Município Granja Lote Idade
(meses) Espécie
Aves por lote Total de aves 2009 São
Paulo Bastos 1
R1 2
Codorna
2.000
50.000
R2 6 12.000
R3 9 12.000
R4A 11 12.000
R4B 13 12.000
Espírito Santo Santa Maria de Jetibá 2
1A 8.5
Codorna
37.798
181.000
1B 3 52.462
2 6.5 90.761
2010 Espírito Santo Santa Maria de Jetibá 3
1 11
Codorna 37.798 181.000
2 6 52.462
3 9 90.761
30 10
Galinhas 100.000 600.000
31 21 100.000
32 7 100.000
São
Paulo Bastos
4
4 2
Codorna 6.660 40.000
5 13 6.660
6 9 6.660
12 25
Galinhas
16.667
50.0000
13 20 16.667
14 10 16,667
15 12 25.000 50.0000
16 18 25.000
5 7 8 Codorna 5.820 5.820
20 15 Galinhas 3.898 3.898
6
8 6
Codorna 5.550 50.000
9 10 5.550
10 3 5.550
17 14
Galinhas
12,500 100.000
18 2.3 12.500 100.000
30
3.2 AMOSTRAS
Em cada granja foram coletadas cinco aves por galpão, numero suficiente para obter uma detecção de IBV bem sucedida (DE WIT, 2000). Na granja um (1) em 2009 foram amostrados cinco lotes de codornas, totalizando 25 aves. Em 2010 foram amostradas três granjas (uma delas coletada em 2009), totalizando sete lotes de codornas (35 aves ao total) e nove lotes de galinhas poedeiras (45 aves em total) (Figura 2). Foram coletadas aves (codornas e galinhas) com e sem sinais clínicos.
Também em 2009, na granja dois (2) foram amostradas cinco aves por lote em três galpões de codornas e em 2010 três lotes de codornas e três de galinhas poedeiras, totalizando 15 codornas e 15 galinhas amostradas em cada ano.
Figura 1 - Sinais clínicos observados nas granjas do estudo. A: codorna jovem apresentando
lacrimejamento; B: galinha jovem apresentado descarga nasal, lacrimejamento e tosse, decorrente da dificuldade respiratória; C e D alteração em casca (fina e porosa), no formato e na pigmentação em ovos de codorna e galinhas, respectivamente (D), São Paulo 2009- 2010
A.
C.
B.
31
Foi incluído no estudo um total de 90 amostras de codornas e 48 amostras de galinhas colhidas na forma de pools de órgãos de aparelho reprodutivo, pulmões, rins e traqueia de codornas e galinhas, e pools de amostras de conteúdo entérico de codornas, mantidas e transportadas sob refrigeração (4ºC) para o Laboratório de Biologia Molecular Aplicada e Sorologia (LABMAS) da Faculdade de Medicina Veterinária e Zootecnia da Universidade de São Paulo (FMVZ/USP), onde foram armazenadas a -20ºC até o momento do seu processamento.
Figura 2 - Distribuição das granjas estudadas no período de 2009-2010 no município de Bastos/SP.
32
3.3 CONTROLES POSITIVOS E NEGATIVOS
Como controle positivo das reações foi utilizada a amostra vacinal MA5 (sorotipo Massachusetts) de Bronquite infecciosa, amostra vacinal NOBILIS® RTV 8544 de metapneuovírus aviário e vírus da doença de Newcastle lentogênica (NOBILIS® ND LA SOTAL). Como controle negativo, utilizou-se água ultra-pura tratada com 0,1% de dietil-pirocarbonato (DEPC).
3.4 EXTRAÇÃO DE RNA
Cada pool de órgãos foi macerado com o uso placas de Petri esterilizadas descartáveis e tesouras e pinças esterilizadas, mantendo-se as amostras em gelo.
As amostras, suspendidas a 50% (volume/volume) em água DEPC, foram congeladas (-80°C por 15 min) e descongeladas (57°C por 15 min) por três ciclos, centrifugadas a 5.000 g a 4°C por 15 min. O sobrenadante resultante da centrifugação das amostras, bem como dos controles positivos e negativos foram utilizados para extração de RNA total com Trizol Reagent™ (Life Technologies Brasil), seguindo as instruções do fabricante.
3.5 REAÇÃO DE TRANSCRIÇÃO-REVERSA (RT)
33
DEPC para um volume final de 10µL. Em seguida a transcrição reversa foi realizada utilizando um ciclo de 42ºC/60 minutos e 72ºC/10 minutos.
3.6 DETECÇÃO DE CORONAVÍRUS AVIÁRIO MEDIANTE A REAÇÃO EM CADEIA
PELA POLIMERASE SEMI-NESTED (SEMI-NESTED RT- PCR) DIRIGIDA À REGIÃO 3’UTR
Utilizou-se a reação de semi-nested RT-PCR dirigida para a região 3’UTR do genoma dos Gammacoronavirus, de acordo com o protocolo proposto por Cavanagh et al. (2002).
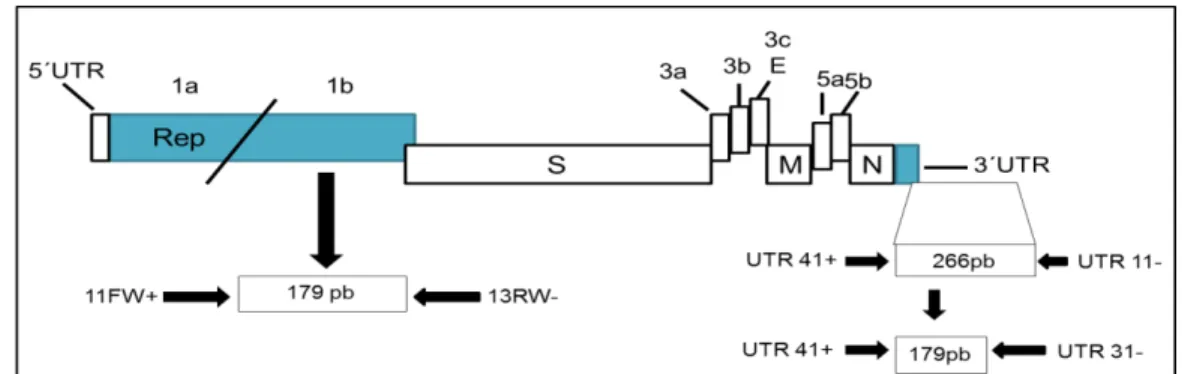
O par de primers externos (UTR41S 5’ ATGTCTATCGCCAGGGAAATGTC 3’ e
UTR11A 5’ GCTCTAACTCTATACTAGCCTA 3’) amplifica um fragmento 266pb (nt
27342 ao 27607 em relação à amostra Beaudette de IBV número de acesso GenBank M95169) e os internos (UTR41S 5’ ATGTCTATCGCCAGGGAAATGTC 3’ e UTR31A 5’ GGGCGTCCAAGTGCTGTACCC 3’) um fragmento de 179 pb (nt 27342 ao 27520 da
região 3’UTR, interno ao produto da primeira PCR) (Figura 3).
Para a primeira amplificação, 2.5µL de cada cDNA foram adicionados ao mix de PCR contendo 1 x PCR BufferTM (Life Technologies Brasil), 0,2mM de cada dNTP, 0,5 µM de cada primer (UTR41+ e UTR11-), 1,5mM MgCl2, 12,625µL água DEPC e 0,625U
de Platinum Taq DNA polimeraseTM (Life Technologies Brasil) para uma reação final de 25µL e submetidos a 35 ciclos a 94ºC/1 minuto, 48ºC/1,5 minutos e 72ºC/2 minutos, seguidos por 72ºC/10 minutos para extensão final.
A seguir, 5µL de cada DNA amplificado foram adicionados ao mix de nested (segunda amplificação) contendo1 x PCR BufferTM (Life Technologies Brasil), 0,2mM de cada dNTP, 0,5µM de cada primer (UTR31- e UTR41+), 1,5mM MgCl2, 20,25µL água
34
Foram consideradas positivas as amostras que resultaram na banda correspondente de 179pb em gel de agarose a 1,5% corado com brometo de etídeo 0,5
g/mL e observados sob luz ultravioleta.
3.7 AMPLIFICAÇÃO DE SEGMENTOS PARCIAIS DO GENE CODIFICADOR DA RNA-POLIMERASE RNA DEPENDENTE MEDIANTE UMA REAÇÃO EM CADEIA PELA POLIMERASE (PCR)
Nas amostras positivas para região 3‘UTR foi aplicada uma RT-PCR dirigida a ORF 1B contendo o gene codificador da RNA-polimerase RNA dependente (RdRp), de acordo com o proposto por Escutenaire et al. (2007), sendo altamente conservado para a subfamília Coronavirinae.
O par de primers 11-FW 5‘ TGATGATGSNGTTGTNTGYTAYAA 3‘ (senso) e 13-RV 5’GCATWGTRTGYTGNGARCARAATTC 3‘ (anti-senso), permitem a amplificação de um fragmento 179 pb da ORF 1B do gene codificador da RNA-Polimerase RNA dependente (nt 15647 ao 15801 em relação à amostra Tor2 do SARS-CoV (número de acesso no GenBank AY274119) (Figura 3).
Para a amplificação, 5µL de cada cDNA foram adicionados ao mix de PCR contendo 1 x PCR Buffer (Life Technologies Brasil), 0,2mM de cada dNTP, 0,5µM de cada primer (11FW+ e 13RV-), 1,5mM MgCl2, 20,25µL água DEPC e 1.25U de Platinum
Taq DNA polimerase (Life Technologies BrasilTM) para uma reação final de 50µL, e submetidos a 94ºC/5 minutos, 94°C/1 min, 40ºC/1 min e 72ºC/1 min, seguidos por 30 ciclos de 94ºC/1 min, 50ºC/1 min e 72ºC/1 min, seguidos por 72ºC/10 minutos para extensão final.
35
Figura 3 - Representação esquemática das regiões amplificadas pela semi-nested RT-PCR para a
região 3’ UTR e pela RT-PCR para a ORF 1B do gene que codifica a RNA-polimerase
RNA dependente
3.8 REAÇÃO DE TRANSCRIÇÃO-REVERSA SEGUIDA DE REAÇÃO EM CADEIA PELA POLIMERASE MULTIPLEX NESTED (MULTIPLEX NESTED RT-PCR) PARA GENOTIPAGEM DAS AMOSTRAS POSITIVAS PARA O CORONAVÍRUS AVIÁRIO
Para determinar se as amostras positivas para coronavírus aviário na triagem, utilizando a semi- nested RT-PCR (item 3.6) pertencem ou não a alguns sorotipos vacinais, as mesmas foram submetidas a uma multiplex nested RT-PCR descrita por Capua et al. (1999) para a amplificação de segmentos da região codificadora da subunidade S1 da proteína S dos sorotipos Massachusetts, D274 e 4/91 ou 793.
O par de primers externos (IBVS 5’ YACTGGYAATTTTTCAGATGG 3’ e IBVAS
5’ CWCTATAAACACCYTTRCA 3’) produz um fragmento de 464pb (nt 729 ao 1193 do
gene S1 em relação à amostra de IBV Beaudette), utilizado a seguir em uma multiplex nested PCR com o primer IBVNA (5’ CAGATTGCTTRCAACCACC 3’), desenhado para hibridizar em todos os três soro/genótipos, e os primers soro/genótipo-específicos: 793S
(5’AGTAGTTTTGTGTATAAAYCA 3’), D274S (5’ ATACAATTATATCAAACCAGC 3’) e
H120S (5’ AATACTACTTTTACGTTACAC 3’), gerando fragmentos de 153 pb (nt 958 a
1111), 217 pb (nt 895 a 1111) e 295 pb (nt 817 a 1111) respectivamente.
36
µM de cada primer (IBVS+ e IBVAS-), 1,5mM MgCl2, 12,625µL água DEPC e 0,625U
de Platinum Taq DNA polimerase™ (Life Technologies Brasil) para uma reação final de
25µL e submetidos a 35 ciclos a 94ºC/1 minuto, 48ºC/1,5 minutos e 72ºC/2 minutos, seguidos por 72ºC/10 minutos para extensão final.
Para a segunda amplificação, 2,5µL do produto da primeira amplificação foram utilizados no mix de nested, com1 x PCR Buffer™ (Life Technologies Brasil), 0,2mM de cada dNTP, 0,5µM de cada primer (IBVNA, 793S, D274S e H120S), 1,5mM MgCl2, 10,125µL água DEPC e 0,625U de Platinum Taq DNA polimerase™ (Life Technologies Brasil) para uma reação final de 25µL e submetidos a 35 ciclos a 94ºC/1 minuto, 48ºC/1,5 minutos e 72ºC/2 minutos, seguidos por 72ºC/ 10 minutos para extensão final. Um tubo contendo água DEPC foi adicionado a cada cinco amostras na reação de multiplex nested para o monitoramento de possíveis contaminações na manipulação do DNA amplificado.
A determinação do sorotipo/genótipo presente na amostra em estudo foi realizada pelo tamanho do fragmento obtido, pela eletroforese em gel de agarose a 1.5%, corados com brometo de etídeo a 0,5µg/mL e observados sob luz ultravioleta.
O amplicon de 153pb foi atribuído ao sorotipo 4/91, o de 217pb ao sorotipo D274 e o de 295pb ao sorotipo Massachusetts. Com a ausência de amplicons observada em gel de agarose submetido à eletroforese, a amostra foi considerada Variante.
3.9 SEMI NESTED RT-PCR PARA AMPLIFICAÇÃO PARCIAL DA REGIÃO
CODIFICADORA DA SUBUNIDADE S1 DA PROTEÍNA S DE IBV
37
TCCACCTCTATAAACACCYTT 3’) que produz um fragmento de 494 pb (nt 677 a 1170
do genoma do IBV) e o par de primers internos SX3S (5’
TAATACTGGYAATTTTTCAGA 3’) e SX4A (5’ AATACAGATTGCTTACAACCACC 3’) (Figura 4).
A seguir, 2.5µL de cada cDNA foram adicionados ao mix de PCR [1 x PCR Buffer™ (Life Technologies Brasil), 0,2mM de cada dNTP, 0,5µM de cada primer (SX1+ e SX2-), 1,5mM MgCl2, 12,625µL água DEPC e 0,625U de Platinum Taq DNA
polimerase™ (Life Technologies Brasil) para uma reação final de 25µL e submetidos a
35 ciclos a 94ºC/1 minuto, 48ºC/1,5 minutos e 72ºC/2 minutos, seguidos por 72ºC/10 minutos para extensão final.
Para segunda amplificação, 5µL do produto da PCR foram adicionados ao mix de nested PCR com 1 x PCR Buffer™ (Life Technologies Brasil), 0,2mM de cada dNTP, 0,5µM de cada primer (SX3+ e SX4-) 20,25 µL água DEPC e 1,25U de Platinum Taq DNA polymerase (Life Technologies BrasilTM) para uma reação final de 50µL e submetidos a 35 ciclos a 94ºC/1minuto, 48ºC/1,5minutos e 72ºC/2minutos, seguidos por 72ºC/10 minutos para extensão final. Para se avaliarem possíveis contaminações pela manipulação do DNA amplificado nesta etapa, foram adicionados tubos com água ultra-pura a cada cinco amostras.
As amostras que resultassem na banda correspondente de 390pb em gel de agarose a 1,5% submetido a eletroforese e corado com brometo de etídeo 0,5g/mL seriam consideradas positivas.
Figura 4 - Representação esquemática da amplificação parcial pela semi-nested RT-PCR para a região
38
3.10 DETECÇÃO DE METAPNEUMOVIRUS AVIÁRIO MEDIANTE A REAÇÃO EM CADEIA PELA POLIMERASE SEMI-NESTED (SEMI-NESTED RT- PCR)
Para investigar a presença de metapneumovírus aviário, utilizou-se uma semi-nested RT- PCR dirigida aos subtipos A e B do gene codificador da proteína G, descrita por Cavanagh et al. (1999).
O par de primers da primeira amplificação, G1 5’
GGGACAAGTATCT/CC/AT/GAT 3´(senso) com três bases degeneradas,
correspondente ao começo do gene (nt 1 ao 17) e o G6 5’
CTGACAAATTGGTCCTGATT 3’ (anti-senso) (nt 422 ao 441), são capazes de se hibridizar com o RNA dos dois subtipos. O produto de 444pb foi utilizado na nested com os primer G5 5’ CAAAGAA/GCCAATAAGCCCA 3’(anti-senso) (nt 401 ao 419), capaz de hibridizar com o RNA dos dois subtipos e os primers G8 5’
CACTCACTGTTAGCGTCATA 3’ senso) (nt 152 ao 171) e G9 5’
TAGTCCTCAAGCAAGTCCTC 3’ senso) (nt 68 ao 87) específicos para os subtipos A e B respetivamente.
A seguir, 2.5µL de cada DNA amplificado foram adicionados ao mix de nested (segunda amplificação) contendo 1 x PCR BufferTM (Life Technologies Brasil), 0,2mM de cada dNTP, 0,5 µM de cada primer (G5-, G8+A e G9+B), 1,5mM MgCl2, 12,625µL
água DEPC e 1,25U de Platinum Taq DNA polimeraseTM (Life Technologies Brasil) para uma reação final de 25µL e submetidos a 35 ciclos a 94ºC/1minuto, 48ºC/1,5minutos e 72ºC/2minutos, seguidos por 72ºC/10 minutos para extensão final. Nesta etapa, foram acrescentados tubos com água ultra-pura a cada cinco amostras para se avaliar possíveis contaminações pela manipulação do DNA amplificado.
39
3.11 DETECÇÃO DO VÍRUS DA DOENÇA DE NEWCASTLE MEDIANTE A REAÇÃO EM CADEIA PELA POLIMERASE (RT- PCR)
Com a finalidade de investigar a presença do vírus da doença de Newcastle, foi realizada uma RT- PCR dirigida ao uma região do gene que codifica a proteína F das linhagens virulentas e avirulentas descrita por Tiwari et al. (2004).
Para isso, 2,5µL de cada cDNA foram adicionados ao mix de PCR que contem 1 x PCR BufferTM (Life Technologies Brasil), 0,2mM de cada dNTP, 0,5µM de cada
primer NCF 5’ TTGATGGCAGGCCTCTTGC 3’ (senso) (nt 141 ao 159) e NCC 5’ (A/G)CG(A/T)CCCTGT(C/T)TCCC-30 (anti-senso) (nt 395 ao 380), ,5mM MgCl2,
12,625µL água DEPC e 0,625U de Platinum Taq DNA polimerase (Life Technologies BrasilTM) para uma reação final de 25µL e submetidos a 35 ciclos a 94ºC/1 minuto, 50ºC/1minuto e 30 segundos e 72ºC/2 minutos, seguidos por 72ºC/10 minutos para extensão final.
Dez microlitros do produto da PCR de cada amostra foram analisados em gel de agarose a 1,5%, submetido a eletroforese, corado com brometo de etídeo a 0,5µg/mL e observados sob luz ultravioleta. As amostras que resultaram numa banda correspondente a 255pb foram consideradas positivas.
3.12 CLONAGEM DO PRODUTO DA PCR DAS AMOSTRAS POSITIVAS DE CORDORNAS PARA 3’UTR
40
3.12.1 Purificação dos amplicons a partir do gel de agarose
Bandas de 179pb visualizadas em luz ultravioleta foram recortadas do gel de agarose e purificadas utilizando o kit GFX™ PCR DNA and Gel Band Purification kit (GE Healthcare, Fairfield, CT). A concentração total de dsDNA foi medida em
espectofotômetro Nanodrop™ ( Thermo Scientific), utilizando comprimento de onda de
280 nm.
3.12.2 Inserção no plasmídeo, transformação bacteriana e extração plasmidial
Os fragmentos purificados foram individualmente inseridos (na região lacZ) em um vetor plasmidial pTZ57R/T (na proporção plasmídeo-inserto de 1:3) utilizando-se o kit InsTaclone™ (Fermentas, Maryland, USA), de acordo com as recomendações do fabricante, e os plasmídeos foram usados separadamente para transformar células competentes da linhagem JM 109 de Escherichia coli.
Para tanto, 10µL de cada reação de ligação foram adicionados a100µL de E. coli JM 109 competente e incubados durante 30 min em gelo. A seguir, as amostras foram submetidas a um choque térmico aquecendo-se a 42º C por 2 min e imediatamente colocadas no gelo durante 2 min. As bactérias transformadas foram então cultivadas em 900µL de meio SOC por 1,5 horas à 37º C e 150 rpm. Após este período, 100µL das culturas foram plaqueadas em meio Luria–Bertani (LB) contendo 50µg/mL de ampicilina, IPTG (0,5mM) e X-Gal (40µg/ml) e incubadas a 37°C por 24 horas.
O sucesso da clonagem interrompe a sequência codificadora da β-galactosidase,
codificada na região do gene lacZ do plasmídeo, sendo que aqueles que receberam o inserto de cDNA formam colônias brancas por não metabolizarem o substrato X-gal e as que não o receberam, formam colônias azuis.
Finalmente, dois clones de JM109 para cada amplicon foram selecionados e submetidos à reação de PCR utilizando os primers M13/Puc (5’
41
antisenso direcionados para a região flanqueadora da inserção do plasmídeo seguindo as instruções do fabricante (Figura 5) para confirmação da presença do inserto. Em seguida os clones contendo os fragmentos desejados foram crescidos em meio LB
contendo ampicilina (50μg/mL) durante 12 horas (overnight). A extração plasmidial foi
realizada utilizando-se o kit NucleoSpin Plasmid™ (Macherey Nagel, Düren, Germany) como recomenda o protocolo do fabricante.
Após a corrida eletroforética em gel de agarose 1,5% corado com brometo de etídio 0,5µg/mL os produtos extraídos foram quantificados visualmente por meio de comparação das bandas obtidas com um padrão de tamanho molecular com Low Mass DNA Ladder (Life Technologies BrasilTM, EUA) segundo instruções do fabricante.
Figura 5 - Representação esquemática da região flanqueadora da inserção do amplicon de179pb
no vetor plasmidial pTZ57R/T
3.13 SEQÜENCIAMENTO DE DNA
A purificação dos amplicons de 179pb (RdRp) e 390pb (gene S) foi realizada utilizando-se GFX PCR DNA and gel band Purification Kit (GE Healthcare), sendo o DNA posteriormente quantificado visualmente com Low Mass DNA Ladder (Life Technologies BrasilTM) segundo instruções do fabricante. Os insertos de amplicons
referentes a 3’UTR foram purificados conforme descrito no item 3.10.1.A reação de
42
para a região 3’UTR consistiu em 0.5µL de BigDye Terminator v3.1 (Cycle
Sequencing Kit, Applied BiosystemsTM EUA, 2 µL de 1x Sequencing buffer (Applied
BiosystemsTM), 0,25M de cada primer (M13/pUC+ e M13/pUC-, 11FW+ e 13RV- ou SX3+ e SX4-) em reações separadas, 20ng do DNA alvo e água DEPC q.s.p. 10µL. As condições da reação de sequenciamento no termociclador Mastercycler Gradient (EppendorfTM) foram de 96ºC/1 minuto, 40 ciclos a 96ºC/15 minutos, 50ºC/15 minutos e 60ºC/4 minutos, com rampa de 1ºC por segundo entre cada temperatura.
A seguir, o produto desta reação foi precipitado à temperatura ambiente com 5
L de EDTA (125mM) e 60L de etanol absoluto, seguindo de homogeneização e incubação por 15 minutos em temperatura ambiente. Em seguida, a solução foi centrifugada a 16.000 x g/45 minutos a 4ºC, o sobrenadante removido, adicionado 65
L de etanol a 70% e centrifugado a 16.000 x g/15 minutos a 4ºC. Procedeu-se a secagem do precipitado a 95ºC/5 minutos. Após esta etapa, as sequências foram resolvidas em seqüenciador capilar Genetic Analyser 3500 (Applied Biosystems™).
3.14 EDIÇÃO DE SEQUENCIAS DE DNA
Os cromatogramas gerados para cada uma das sequências senso e antisenso de cada amostra e gene foram submetidos ao aplicativo Phred1 disponível online em:
http://asparagin.cenargen.embrapa.br/phph/ para avaliação da qualidade dos mesmos, utilizando-se apenas posições com escore superior a 20 (probabilidade de um erro a cada 100 nucleotídeos).
A seguir, os cromatogramas foram conferidos manualmente com o programa FinchTV 1.4 (© 2011 PerkinElmer) para a busca por erros de interpretação e discrepâncias entre cada uma das fitas seqüenciada. A sequência final de cada amostra foi obtida com o aplicativo Cap-Contig com o programa Bioedit v. 7.0.5.3
(HALL, 1999), sendo a mesma submetida ao BLASTn
43
(http://www.ncbi.nlm.nih.gov/BLAST/)2 para confirmação da especificidade do
seqüenciamento.
3.15 ANÁLISE FILOGENÉTICA
A árvore genealógica para as sequências de nucleotídeos de cada região genômica estudada foi gerada após o alinhamento das mesmas com sequências homólogas recuperadas do Genbank e correspondentes as diversas espécies de coronavírus aviário pelo método CLUSTAL/W com o programa Bioedit v. 7.0.5.3 (HALL, 1999).
O alinhamento assim obtido foi utilizado para a geração da árvore de distância, com algoritmo Neighbor-Joining e modelo evolutivo Maximum Composite Likelihood, com 1000 repetições de bootstrap, utilizando-se o programa MEGA 5.0 (TAMURA et al., 2011).
Para as sequencias referentes aos genes S e RdRp, árvores de aminoácidos seriam geradas a partir das traduções das sequências de nucleotídeos, com algoritmo Neighbor-Joining e modelo de Poisson, com 1000 repetições de bootstrap, utilizando-se o programa MEGA 5.0.
3.16 FORMA DE ANÁLISE DOS RESULTADOS
A frequência de ocorrência de coronavírus nas codornas e nas galinhas poedeiras foi determinada pela razão entre o número de amostras positivas pela semi-nested RT-PCR para a região 3’UTR para cada classe de amostras e o total de amostras testadas em cada classe. A associação entre a espécie de ave (codorna e
44
galinha) e a presença do vírus foi determinada com a aplicação do teste exato de Fisher com o programaMinitab® 15.1.0.0.(© 2006 Minitab Inc.).
As espécies de coronavírus encontradas nas codornas e nas galinhas foram
determinadas por meio do seqüenciamento e da filogenia da 3’UTR e o gene
codificador da proteína RNA- polimerase RNA dependente.
45
4 RESULTADOS
A ocorrência de coronavírus aviário em codornas e galinhas provenientes das mesmas granjas, a associação de co-infecção com o metapneumovírus aviário e vírus da doença de Newcastle, a classificação das amostras de coronavírus encontradas, assim com o sequenciamento parcial das regiões do genoma estudadas encontram-se descritos nos itens a seguir.
4.1 DETECÇÃO DE CORONAVÍRUS AVIÁRIOS MEDIANTE A REAÇÃO EM
CADEIA PELA POLIMERASE SEMI-NESTED (SEMI-NESTED RT- PCR) DIRIGIDA A REGIÃO 3´UTR
46
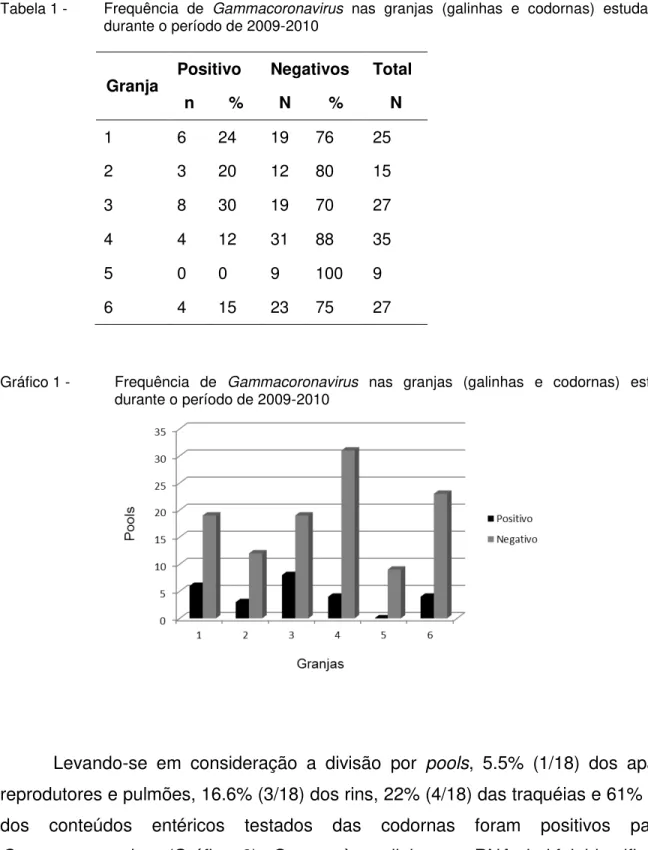
Tabela 1 - Frequência de Gammacoronavirus nas granjas (galinhas e codornas) estudadas
durante o período de 2009-2010
Granja Positivo Negativos Total
n % N % N
1 6 24 19 76 25
2 3 20 12 80 15
3 8 30 19 70 27
4 4 12 31 88 35
5 0 0 9 100 9
6 4 15 23 75 27
Levando-se em consideração a divisão por pools, 5.5% (1/18) dos aparelhos reprodutores e pulmões, 16.6% (3/18) dos rins, 22% (4/18) das traquéias e 61% (11/18) dos conteúdos entéricos testados das codornas foram positivos para os Gammacoronavirus (Gráfico 2). Quanto às galinhas, o RNA viral foi identificado em
pools dos pulmões (1/12 – 8.3%), dos rins (3/12 - 25%) e das traquéias (1/12- 8.3%) (Gráfico 3).
Gráfico 1 - Frequência de Gammacoronavirus nas granjas (galinhas e codornas) estudadas
47
Gráfico 3 - Ocorrência de Gammacoronavirus em galinhas poedeiras nos anos 2010 e nas
amostras estudadas dos municípios de Santa Maria de Jetibá/ES e Bastos/SP
A analise da presença de RNA dos Gammacoronavirus nos pools de órgãos das codornas e galinhas em 2010 evidenciou uma maior ocorrência no aparelho reprodutor (1/11 – 9%) e traquéia (2/11 – 18.2%) das amostras provenientes das codornas quando comprado com os resultados obtidos das galinhas, sendo que nenhum pool de aparelho reprodutor de galinhas foi positiva e em 9% (1/11) dos pools de traquéia foi confirmada a presença do RNA viral. No entanto, nos pools dos rins houve uma maior frequência
Gráfico 2 - Ocorrência de Gammacoronavirus em codornas nos anos de 2009 e 2010 nas
48
nas amostras provenientes das galinhas (3/11 – 27,2%) e no pool dos pulmões (1/11 – 9%) a frequência foi igual para as duas espécies. No total foram observados 54,5% (6/11) e 45,5% (5/11) de pools positivos para Gammacoronavirus nas codornas e galinhas, respectivamente (Gráfico 4).
Utilizando-se o programa Minitab® 15.1.0.0.(© 2006 Minitab Inc.), aplicou-se o teste exato de Fisher para se determinar a associação entre a espécie de ave e a presença de coronavírus aviário nos pools de órgãos avaliados em 2010, não tendo sido verificada associação estatisticamente significativa entre as mesmas (p=1).
4.2 AMPLIFICAÇÃO DE SEGMENTOS PARCIAIS DO GENE CODIFICADOR DA RNA-POLIMERASE RNA DEPENDENTE MEDIANTE UMA REAÇÃO EM CADEIA PELA POLIMERASE (PCR)
A reação de RT-PCR para o gene codificador da proteína RdRp foi positiva em pools de conteúdo entérico em seis (6) de vinte (20) amostras testadas das codornas, dos dois municípios em estudo, e seis (6) de dez (10) amostras das galinhas, sendo
Gráfico 4 - Ocorrência de Gammacoronavirus em codornas e galinhas poedeiras no ano 2010 nas
49
que o pool de traqueia pertencia ao município de Bastos e os cinco restantes a pools de conteúdo entérico de lotes de galinhas poedeiras de diversas regiões do Brasil, todas estas positivas para a RT-PCR tendo como alvo a 3’UTR.
As reações referentes ao controle negativo e aos controles incluídos na fase de nested (DPEC) não apresentaram quaisquer bandas.
4.3 REAÇÃO DE TRANSCRIÇÃO-REVERSA SEGUIDA DE REAÇÃO EM CADEIA PELA POLIMERASE MULTIPLEX NESTED (MULTIPLEX NESTED RT-PCR) PARA GENOTIPAGEM DAS AMOSTRAS POSITIVAS PARA O CORONAVÍRUS AVIÁRIO
Em todas as amostras positivas na triagem com a semi- nested-RT-PCR dirigida
à região 3’UTR foi aplicada o multiplex nested RT-PCR para a amplificação de
segmentos da região codificadora da subunidade S1 da proteína S dos sorotipos Massachusetts, D274 e 4/91 ou 793.
50
Quadro 2 - Resultados da genotipagem das amostras segundo o ano de
coleta, estado (ES= Espírito Santo; SC= Santa Catarina; SP= São Paulo; PR= Paraná; MG= Minas Gerais), lote, espécie e tipo de amostra.
Ano Estado Lote Espécie Amostra Resultado
2009 SP R1 Codorna Traquéia Variante
2009 SP R2 Codorna Conteúdo entérico Variante
2009 SP R2 Codorna Rim Variante
2009 SP R3 Codorna Conteúdo entérico Variante
2009 SP R4A Codorna Conteúdo entérico Variante
2009 SP R4B Codorna Traquéia Variante
2009 ES 1A Codorna Conteúdo entérico Variante
2009 ES 1B Codorna Conteúdo entérico Variante
2010 ES 1 Codorna reprodutor Aparelho Variante
2010 ES 1 Codorna Conteúdo entérico Variante
2010 ES 1 Codorna Rim Variante
2010 ES 1 Codorna Traquéia Variante
2010 ES 1 Codorna Traquéia Variante
2010 ES 2 Codorna Pulmão Variante
2010 ES 30 Galinha Rim Variante
2010 ES 31 Galinha Rim Variante
51
Quadro 2 - Resultados da genotipagem das amostras segundo o ano de coleta, estado
(ES= Espírito Santo; SC= Santa Catarina; SP= São Paulo; PR= Paraná; MG= Minas Gerais), lote, espécie e tipo de amostra (Continua)
Ano Estado Lote Espécie Amostra Resultado
2010 SP 5 Codorna Conteúdo
entérico Variante
2010 SP 5 Codorna Rim Variante
2010 SP 8 Codorna Conteúdo
entérico Variante
2010 SP 9 Codorna Conteúdo
entérico Variante
2010 SP 9 Codorna Traquéia Variante
2010 SP 10 Codorna Conteúdo
entérico Variante
2010 SP 13 Galinha Rim Variante
2010 SP 16 Galinha Traquéia Variante
2011 SC 09 Galinha Pulmão Variante
2011 PR 01 Galinha Conteúdo
entérico Variante
2011 RS 05 Galinha Conteúdo
entérico Variante
2011 MG 04 Galinha Rim Variante
2011 SP 16 Galinha Pulmão e
traquéia Variante
4.4 SEMI NESTED RT-PCR PARA AMPLIFICAÇÃO PARCIAL DA REGIÃO
CODIFICADORA DA SUBUNIDADE S1 DA PROTEÍNA S DE IBV
52
segmento de 390pb da região S1 do gene S que seriam utilizados no estudo da genealogia para este gene.
4.5 DETECÇÃO DE METAPNEUMOVIRUS AVIÁRIO E VÍRUS DA DOENÇA DE NEWCASTLE MEDIANTE A REAÇÃO EM CADEIA PELA POLIMERASE SEMI-NESTED (SEMI-SEMI-NESTED RT- PCR)
Nos pools de órgãos e conteúdo entérico das codornas o RNA viral de metapneumovírus aviário não foi detectado, no entanto em um dos pools de aparelho reprodutor e em dois pools de pulmão das galinhas provenientes dos dois municípios estudados, foi possível detectar o subtipo B de metapneumovírus.
Quanto ao vírus da doença de Newcastle, o RNA viral não foi detectado em nenhuma das amostras estudadas.
4.6 CLONAGEM DO PRODUTO DA PCR DAS AMOSTRAS POSITIVAS DE
CORDORNAS PARA 3’UTR
53
Figura 6 - Gel de agarose 1.5% corado com brometo de etídeo 0,5 g/Ml do produto (320pb) da
PCR direcionada para a região flanqueadora da inserção do plasmídeo contendo o
inserto de 179pb referente a 3’UTR
4.7 SEQÜENCIAMENTO DE DNA
Para os amplicons referentes à região 3’ UTR (produto da clonagem no caso das codornas) e da ORF1b do gene que codifica a proteína RdRp, 10 pools de amostras das galinhas (traquéia, rim e conteúdo entérico) e 20 pools de amostras das codornas (aparelho reprodutor, conteúdo entérico, pulmão, traquéia e rim), foram submetidos ao sequenciamento de DNA. Foram obtidas sequências de 97 nucleotídeos (3’ UTR) de 5 amostras das galinhas e 4 amostras de das codornas e de 116 nucleotídeos (RdRp) de 4 amostras das galinhas e 3 amostras das codornas.
4.8 ANÁLISE FILOGENÉTICA
54
das galinhas estudadas, que segregaram juntamente com duas sequencias correspondentes a linhagens vacinais (Massachusetts e Arkansas) e uma sequência originaria do TCoV e um outro grupamento próprio das codornas, subdivididos em dois clusters diferentes, um correspondente a granja 4 e o outro a granja 6, ambas localizadas na região de Bastos/SP.
Na árvore filogenética gerada para ao gene da RdRp (Figura 8), houve também a separação de um grupamento exclusivo para amostras de codornas, enquanto que as amostras de galinhas do presente estudo, segregaram com amostras de IBV do sorotipo Massachusetts.
GU323510.1 Porphryrio martinica GU323504.1Agamia agami
GU323508.1Penelope superciliaris GU323511.1Rupornis magnirostris GU323509.1 Porphryrio martinica DQ401286.1CECoVBR03USP-01 GU323505.1Aptenodytes patagonicus GU323506.1Pavo cristatus EU817497.1H52 FJ888351.1H120 Galinha/PT/2011/Granja16 Galinha/R/2011/Granja04 GQ504720.1ArkansasDPI Galinha/CE/2011/Granja01 Galinha/T/2011/Granja09 AY641576.1Peafowl DQ834384.1M41 Galinha/CE/2011/Granja05 GQ427176.1TCoV/TX-1038/98 EU095850.1TCoVMG10 Codorna/CE/2010/Granja6.2 Codorna/CE/2010/Granja4 Codorna/R/2010/Granja4 Codorna/CE/2010/Granja6.1
AJ619594.1 Pheasant coronavirus EU022526.1TCoV-ATCC EF165618.1| Ind/TN/92/03 EU526388.1 4/91 AY514485.1California 99 GU323507.Pavo cristatus 64 62 0.01
Figura 7 - Árvore genealógica de distância com o algoritimo Neighbor-joining para nucleotídeos da
região 3’ UTR (nt 27342 ao 27520), mostrando os sorotipos/genótipos clássicos (com o
55
Figura 8 - Árvore genealógica de distância com o algoritimo Neighbor-joining para nucleotídeos da
RdRp (nt 15647 ao 15801) mostrando os sorotipos/genótipos clássicos (com o numero de
acesso do Genbank) e as amostras incluídas no presente estudo (o quadro representa as amostras das codornas, o triangulo as das galinhas e o rombo a amostra CRG proveniente de um plantel avícola e isolada no laboratório em ovos SPF). Os números acima de cada nó representam os valores de bootstrap para 1000 repetições (apenas os valores maiores que 50% estão demonstrados). A barra representa o número de substituições de nucleotídeos por sítio MA5/2011 FJ888351.1H120 Galinha/CE/2011/Granja05 Galinha/CE/2011/Granja01 Codorna/CE/2010/Granja6 Codorna/CE/2010/Granja3 Codorna/CE/2009/Granja2 CRG/IBV EU022526.1 TCoV-ATCC EU526388.1 4/91 EU095850.1TCoV MG10 EU817497.1H52 DQ834384.1M41 NC001451.1 BEAUDETTE GQ427176.1TCoV/TX-1038/98 AY514485.1California99 GQ504720.1Ark GQ504720.1|ArkansasDPI 89 98 90 75 98 96 63 72 70 95 53 0.01
A identidade de nucleotídeos para região 3´UTR entre o do grupo das codornas foi de 98,9%, entre 97,5% e 97,6% em relação as galinhas e o genótipo Massachusetts (H120). Entre as amostras de galinhas não houve variação e entre o grupo das galinhas e o genótipo H120 a identidade foi de 98.9%.
56
Tabela 2 - Identidade média (em porcentagem) de nucleotídeos da região 3´UTR (nt 27342 ao 27520
em relação à amostra Beaudette de IBV e RdRp (nt 15647 ao 15801 em relação à amostra Tor2 do SARS-CoV) para os grupamentos de amostras estudadas no presente estudo (codornas e galinhas) e o sorotipos Massachusetts (H120 e Ma5). ND = Dados não disponíveis
Grupos 3´UTR RdRp
Média % Desvio padrão Média % Desvio padrão
Codornas vs
codornas 98 0,086 98,8 1,0392
Galinhas vs galinhas 100 0 90,5 0
Codornas vs galinhas 97,525 0,4887 92,13 2,4513
Codornas vs H120 97.68 2,442 93,1 0
Codornas vs Ma5 ND ND 93.1 0
Galinhas vs H120 98.9 0 95.3 4,041
57
5 DISCUSSÃO
Nesta investigação, o coronavírus aviário foi detectado em cinco de seis granjas de codornas e galinhas poedeiras estudadas. Algumas dessas granjas apresentaram o vírus em ambas às espécies de hospedeiro simultaneamente, sendo que tanto os coronavírus encontrados em codornas quanto em galinhas foram identificados como próximo àqueles relacionados ao vírus da bronquite infecciosa das galinhas.
Variáveis como o manejo, instalações, biossegurança, densidade populacional, região geográfica, número de animais amostrados e condições de coleta de amostras podem influenciar na detecção do coronavírus aviário e, portanto, nas diferenças encontradas nas frequências de ocorrência entre as granjas estudadas. Isso é devido às diferentes fases na dinâmica da infecção nas diferentes populações no momento da amostragem (JONASSEN et al., 2005).
A granja três, por exemplo, cumpria com a maioria das normas de biossegurança como: limpeza, desinfecção, controle de trânsito de pessoas, animais, veículos, aves de descarte, manutenção de registros sobre produção, morbidade, mortalidade, vacinações e testes de monitoramento; no entanto, a população era alta (181.000 codornas e 600.00 galinhas) e a distância entre os galpões de codornas e galinhas poedeiras era menor do que 100m, o que permite a transmissão de coronavírus aviários entre os lotes (DE WIT, 2000).
58
mínima de 2km entre os estabelecimentos avícolas com objetivos produtivos. Estes fatos significam que as populações aviárias nestas granjas se encontravam sob risco de ocorrência de diversos patógenos, entre eles os coronavírus aviários.
Quanto à granja quatro, os visitantes deviam tomar banho dentro das instalações da granja antes de ter contato com as aves, caso houvessem visitado outra propriedade no mesmo dia. A desinfecção do carro também era obrigatória. Além disso, árvores de eucalipto e seringueira isolavam a propriedade de outras granjas e uma cerca viva de eucalipto de 200m separava uma parte dos lotes de galinhas dos lotes das codornas. Nesses lotes de galinhas não foram encontradas amostras positivas para IBV.
A granja cinco possuía um menor número de animais na propriedade (5820 codornas e 3898 galinhas), quando comparada com as outras ganjas do estudo. Isso pode ter diminuído a probabilidade de detecção de amostras positivas para coronavírus aviário, pois nesta granja não foram encontradas amostras positivas.
Uma característica observada entre as granjas do estudo foi a curta distância entre lotes de idades diferentes e a proximidade entre os lotes de codornas e galinhas. Essa distância chegava ser de 50 m, como no caso da granja seis. Preconiza-se 500 m de distância entre lotes de diferentes idades e do lote em relação à estrada vicinal, rodovia estadual ou federal, e a distância entre galpões dos lotes deve ser o dobro da largura dos galpões (GONÇALVES, 2002). A alta proximidade entre os lotes pode elevar a transmissão de patógenos, explicando, em parte, a elevada frequência de coronavírus aviários detectados entre as granjas em estudo.
Em áreas onde existem granjas avícolas em contigüidade, é improvável manter as galinhas livres de IBV. Em função de baixa biossegurança, o vírus dissemina-se rapidamente, e, como medida de controle, utiliza-se a vacinação (CAVANAGH, 2007).
59
VILLARREAL et al., 2007a,b; MONTASSIER et al., 2008; MONTASSIER, 2010, FELIPPE et al., 2010; VILLARREAL, et al., 2010b; CHACÓN et al., 2011b).
Para as aves amostradas em 2010, a frequência de coronavírus aviário nos lotes das galinhas (4/12- 33.3%) foi baixa quando comparada aos de codornas (6/10 - 60%) nas 4 granjas estudadas. Uma vez que as galinhas receberam vacinação contra a bronquite infecciosa das galinhas (item 3.1), pode-se especular que uma das razões para uma menor detecção de coronavírus aviário nas galinhas seja algum grau de proteção conferida pelas vacinas, cuja ausência entre as codornas pode ter tido como consequência uma presença mais intensa deste vírus nestas aves.
É importante indicar que a semi-nested-RT-PCR utilizada no estudo para a detecção de coronavírus aviário não diferencia entre a presença de RNA genômico e RNA mensageiro. Dessa forma não é possível afirmar que nas amostras positivas o vírus estivesse se replicando. Após a replicação no trato respiratório superior e a fase de viremia, o vírus pode se disseminar para outros tecidos (LAI;CAVANAGH, 1997; COOK, 2008). Por outro lado, não se pode inferir uma infecção recente quando é detectado um vírus em um lote com suspeita de IBV previamente vacinado ou infectado (DE WIT, 2000).
Nas codornas, 5% dos pools de pulmões e 4% dos pools de traqueia foram positivos para coronavírus aviário, sendo que um dos pools estudados pertencia às codornas do lote R1, cuja idade era de 2 meses. Nesses animais foram observados sinais como lacrimejamento, tosse (Figura 2) e letargia. Estes sinais são compatíveis com os observados em galinhas de poucos dias ou semanas de idade, as quais apresentam corrimento nasal, ruídos secos (semelhante ao espirrar), olhos lacrimejantes e letargia (CAVANAGH; GELB, 2008; COOK, 2008).
60
Mycoplasma gallisepticum, Eschericha coli e Haemophilus paragallinarum que podem levar aos sinais observados (CAVANAGH; GELB 2008; MATTHIJS et al., 2009).
Nota-se que o coronavírus aviário foi encontrado no presente estudo em amostras de aparelhos reprodutivos de codornas e galinhas (item 4.1) que apresentavam queda na produção de ovos, cascas com menor espessura, maior porosidade e com alteração de formato (Figura 2), tendo sido observadas também falsas poedeiras.
Em aparelhos reprodutores de poedeiras, a manifestação mais comum de infecção por IBV é evidenciada na produção e qualidade de ovos. Isso se deve à replicação viral no oviduto, o que pode causar, em fêmeas jovens, dano permanente ao desenvolvimento do mesmo, observando-se atrofia parcial ou completa deste órgão, podendo as afetadas ovular normalmente mesmo sem produção de ovos (falsas poedeiras). A infecção das poedeiras durante o período de postura pode reduzir a produção em até 10% ou mais, dependendo da imunidade do lote (CAVANAGH; GELB 2008). A produção retorna ao normal de 4- a 6 semanas, mas pode ser observada uma deterioração na qualidade interna e externa do ovo (COOK, 2008), com hipoplasia glandular, que leva à redução da síntese de proteínas do albúmen, especialmente a ovomucina, lisozima e outras proteínas (DHINAKAR; JONES, 1997).
Assim, é provável que em codornas, a patogenia da infecção reprodutiva por coronavírus aviário seja similar àquela já descrita nas galinhas, levando estas a apresentarem sinais já relacionados à infecção por este vírus em aves com função de poedeiras e reprodutoras.