Aline de Sousa Marques
Uso da Espectroscopia do Infravermelho Próximo e técnicas multivariadas para
diferenciar
Escherichia coli
e Salmonella Enteritidis inoculadas em polpa
de fruta (abacaxi).
__________________________________
Dissertação de Mestrado
Natal/RN, julho de 2013
UNIVERSIDADE FEDERAL DO RIO GRANDE DO NORTE
CENTRO DE CIÊNCIAS EXATAS E DA TERRA
INSTITUTO DE QUÍMICA
UNIVERSIDADE FEDERAL DO RIO GRANDE DO NORTE
CENTRO DE CIÊNCIAS EXATAS E DA TERRA
PROGRAMA DE PÓS-GRADUAÇÃO EM QUÍMICA
Aline de Sousa Marques
Grupo de pesquisa em Quimiometria Aplicada – GPQA
Uso da Espectroscopia do Infravermelho Próximo e técnicas multivariadas para diferenciar Escherichia coli e Salmonella Enteritidis inoculadas em polpa de fruta
(abacaxi).
Natal/RN
2
Aline de Sousa Marques
Professor Orientador: Kássio Michell Gomes de Lima
Uso da Espectroscopia do Infravermelho Próximo e técnicas multivariadas para diferenciar Escherichia coli e Salmonella Enteritidis inoculadas em polpa de fruta
(abacaxi).
Dissertação de Mestrado apresentada ao Programa de Pós-Graduação em Química da Universidade Federal do Rio Grande do Norte, como parte dos requisitos necessários para obtenção do título de Mestre em Química.
NATAL/RN
UFRN / Biblioteca Central Zila Mamede. Catalogação da Publicação na Fonte.
Marques, Aline de Sousa.
Uso da espectroscopia do infravermelho próximo e técnicas multivariadas para diferenciar escherichia coli e salmonela enteritidis inoculadas em polpa de fruta (abacaxi). / Aline de SousaMarques. – Natal, RN, 2013.
67 f. : il.
Orientador: Prof. Dr. KássioMichell Gomes de Lima.
Dissertação (Mestrado) – Universidade Federal do Rio Grande do Norte. Centro de Ciências Exatas e da Terra. Instituto de Química. Programa de Pós-Graduação em Química.
1. Espectroscopia - Dissertação. 2. Transflectância NIR - Dissertação. 3. Escherichia Coli - Dissertação. 4. SalmonellaEnteritidis– Dissertação. 5. SIMCA – Dissertação. 6. PLS-DA – Dissertação. I. Lima, KássioMichell Gomes de. II. Universidade Federal do Rio Grande do Norte. III. Título.
4
Á Deus, por permitir e me dar a capacidade de tornar este trabalho possível, por me encorajar quando as minhas forças já não eram suficientes, por colocar no meu caminho o professor Kássio, que me motivou a caminhar por caminhos ainda não conhecidos.
5
AGRADECIMENTOS
Aos meus pais, João e Wilma, pelo amor, carinho, dedicação e educação que me deram ao longo da minha vida, permitindo meu crescimento pessoal para atingir mais essa conquista.
Às minhas irmãs Clarice, Valéria e Marília, pela relação de união que temos.
Ao meu noivo, Felipe, que esteve comigo por toda essa caminhada, acreditando, incentivando e muitas vezes me ajudando ‘cientificamente’ para que o trabalho fosse realizado, e por todo carinho e amor que fez a caminhada mais amena.
Às Helena’s do GPQA, Ana Carolina, Jábine, Fernanda e Raquel, por estarem sempre presentes dando sentido àquilo que não tinha, atribuindo conhecimento e tornando o trabalho mais divertido.
Ao professor Dr. Kássio Lima, pela oportunidade, orientação, apoio e confiança.
A professora Celeste por fornecer o seu conhecimento e laboratório para a aquisição das amostras e a Thiago seu orientando pelo apoio, dedicação e disposição.
A Jábine pelo companheirismo, pelas tardes e mais tardes de dedicação do seu tempo me ajudando em tudo o que eu precisava, o mérito também é dela.
Em especial, a Ana Carolina que esteve presente em toda caminhada, estudou, apoiou, acreditou, brigou, chorou, sempre ao meu lado, ela acreditou em mim até quando eu não acreditava, por tudo, obrigada.
6
RESUMO
Visando à segurança do consumidor, é de extrema importância identificar a presença de contaminantes patogênicos nos alimentos, pois os mesmos são responsáveis por surtos alimentares que dependendo do nível de contaminação pode chegar a causar a morte de quem os consome. Na industria há uma necessidade de que essa identificação de contaminantes seja rápida e rentável. Este estudo mostra a aplicação e utilidade de medidas espectrais de transflectância no infravermelho próximo (NIR) como um método alternativo para a identificação e classificação de Escherichia coli e Salmonella Enteritidis em polpa de fruta comercial (abacaxi). Análise de Componentes Principais (PCA), Modelagem Independente por Analogia Classe (SIMCA) e Análise Discriminante por Mínimos Quadrados Parciais (PLS-DA) foram utilizados na análise. Não foi possível obter uma separação total entre as amostras usando PCA e SIMCA. O PLS-DA apresentou bom desempenho na capacidade de predição alcançando 87,5% para E. coli e 88,3% para S. Enteritides, respectivamente. Os melhores modelos obtidos para o PLS-DA com os espectros tratados com segunda derivada apresentaram sensibilidade e especificidade de 0,87 e 0,83, repectivamente. Estes resultados sugerem que a espectroscopia NIR e PLS-DA podem ser usados para discriminar e detectar bactérias na polpa da fruta.
Palavras-chave: Transflectância NIR. Escherichia coli. Salmonella Enteritidis.
7
ABSTRACT
Aiming to consumer’s safety the presence of pathogenic contaminants in foods must be monitored because they are responsible for foodborne outbreaks that depending on the level of contamination can ultimately cause the death of those who consume them. In industry is necessary that this identification be fast and profitable. This study shows the utility and application of near-infrared (NIR) transflectance spectroscopy as an alternative method for the identification and classification of Escherichia coli and Salmonella Enteritidis in commercial fruit pulp (pineapple). Principal Component Analysis (PCA), Independent Modeling of Class Analogy (SIMCA) and Discriminant Analysis Partial Least Squares (PLS-DA) were used in the analysis. It was not possible to obtain total separation between samples using PCA and SIMCA. The PLS-DA showed good performance in prediction capacity reaching 87.5% for E. coli and 88.3% for S. Enteritides, respectively. The best models were obtained for the PLS-DA with second derivative spectra treated with a sensitivity and specificity of 0.87 and 0.83, respectively. These results suggest that the NIR spectroscopy and PLS-DA can be used to discriminate and detect bacteria in the fruit pulp.
Keywords: NIR Transflectance. Escherichia coli. Salmonella Enteritidis.
8
LISTA DE FIGURAS
Figura 1- Estrutura celular de organismos positivos e gram-negativos... 18
Figura 2- Imagem ampliada de Escherichia coli
O157:H7...
19
Figura 3- Imagem ampliada da Salmonella
entérica...
20
Figura 4- Crescimento de microrganismos fermentadores de lactose
(colônias rosadas) em ágar
VRBA...
22
Figura 5- Presença de gás e leve turbidez em tubos contendo caldo
Escherichia coli após 24h de
incubação...
23
Figura 6- Ágar SIM, (A) ágar sem contaminação, (B) teste de Indol positivo, (C) teste de Indol negativo e (D) produção de H2S com aparecimento de precipitado negro... 24
Figura 7- Esquema reativo de (A) produção do Indol e (B) reação do Indol
com o reativo de
Kovacs...
25
Figura 8- Procedimento de inoculação em ágar
TSI...
26
9
Figura 10- Reação de descarboxilação da Lisina através da enzima Lisina Descarboxilase (LDC), produzindo Cadaverina e dióxido de carbono... 28
Figura 11- Crescimento de Salmonella em ágar XLD com aparecimento de centros escuros devido a precipitação do H2S. ...
28
Figura 12- Crescimento de colônias rasadas em ágar Rambac, possível
contaminação por
Salmonella...
29
Figura 13- Ágar TSI, (1) contaminação por Salmonella, (2) não inoculado...
30
Figura 14- Regiões do espectro eletromagnético com ênfase na região do Infravermelho... 31
Figura 15- Tipos de vibrações
moleculares...
32
Figura 16- Transições eletrônicas entre níveis
vibracionais...
34
Figura 17- Diagrama de energia potencial de um oscilador: (A) harmônico e (B) anarmônico... 35
Figura 18- Representação de bandas de absorção na região do infravermelho próximo... 37
Figura 19- Esquema de um
espectrofotômetro...
10
Figura 20- Espectro obtido na região do infravermelho próximo...
38
Figura 21- Representação da simplificação de variárias por PCA...
42
Figura 22- Decomposição das matrizes X eY em matriz menores...
45
Figura 23- DFA dos espectros NIR de E.coli ATCC 25922 e E.coli K12 com
diferentes concentrações de
células...
46
Figura 24- Espectros do ‘Extato B’ do repolho picado, (a) espectro original
e (b) espectro com MSC + segunda
derivada...
47
Figura 25- Representação dos espectros de L.innocua ATCC 51742,
B.cereus ATCC 10876, E.coli O157:H7 ATCC 35150 e E.coli
O157:H7 ATCC 25522, diferenças na região de 2000-1000 cm-1 pode ser observado... 48
Figura 26- Representação de PC1xPC3 dos escores obtidos por regressão PLS1 de amostras com L.plantarum, L.mesenteroides e L.sakei
a níveis de concentração 3-9 log de ufc/mL...
48
Figura 27- Etapas de obtenção dos espectros. (a) fibra ótica na posição inicial, para a obtenção do Background, (b) posicionamento da sonda sobre a amostra para coleta dos espectros e (c) armazenamento de dados... 49
11
S. Enteritidis de 50 amostras, sendo 25 para E. coli e 25 para S. Enteritidis... 55
Figura 29- Espectros NIR das suspensões de bactérias E.coli e S. Enteritidis pré-processada com MSC e segunda derivada de Savitzky-Golay com janela de 15 pontos com atribuição das possíveis estruturas presentes na parece celular dos gram-negativos... 56
Figura 30- Escores de PCA dos espectros NIR das suspensões de E.coli e
S.
12
LISTA DE TABELAS
Tabela 1- Modelo SIMCA com segunda derivada na região de 1111-2000nm... 58
Tabela 2- Modelos PLS_DA com primeira e segunda derivada nas regiões
(A) espectro puro e (B)
1111-2000nm...
13
LISTA DE ABREVIATURAS
BHI- Ágar de infusão de cérebro e coração (do inglês, Brain Heart Infusion Agar)
EC- Eschecichia coli
FIR- infravermelho distante (do inglês, Far Infrared)
HUS- Síndrome Hemolítica-urêmica (do inglês, Hemolytic-uremic Sindrome)
ICMSF- Comissão Internacional de Especificações Microbiológicas em Alimentos (do inglês, International Comission on Microbiological Specifications for Food)
INCQS00258- Salmonella Enteritidis
INSCQ00171- Escherichia coli O157:H7
IR- infravermelho (do inglês, infrared)
LPS- Lipopolissacarídeo
MATLAB- software para cálculos numéricos (Matrix Laboratory)
MIR- infravermelho médio (do inglês, mid-infrared)
MSC- Correção do espalhamento multiplicativo (do inglês, Multiplicative Scatter Correction)
NIR- infravermelho próximo (do inglês, near infrared)
OMS- Organização Mundial de Saúde
PCA- análise por componentes principais (do inglês, principal component analysis)
PLS- Mínimos Quadrados Parciais (do inglês, Partial Least Square)
PLS-DA- Análise Discriminante por Mínimos Quadrados Parciais (do inglês,
Partial Least Square for Discriminant Analysis)
14
RMSEP- Raiz Quadrada do Erro Médio de Previsão (do inglês, Root Mean Square Error of Prediction)
SIMCA- Modelagem Independente por Analogia de Classes (do inglês, Soft Independent Modelling of Class Analogy)
STEC-EHEC- Escherichia coli Enteroemorrágica
TSB- Caldo soja tríptica (do inglês, Tryptic Soy Broth)
TSI- Ágar três açúcares ferro (do inglês, Triple Sugar Iron Ágar)
VRBA- Ágar vermelho violeta bile (do inglês, Violet Red Bile Agar)
15
SUMÁRIO
1 INTRODUÇÃO... 17
1.1 O ABACAXI... 17
1.2 MICROORGANISMOS PATOGÊNICOS E DOENÇAS TRANSMITIDAS POR ALIMENTOS ... 17
1.1.1 Gênero Escherichia... 18
1.1.2 Gênero Salmonella... 20
1.3 PROCEDIMENTO OPERACIONAL PADRÃO DE ANÁLISES MICROBIOLÓGICAS NA INDÚSTRIA DE ALIMENTOS... 21 1.3.1 Gênero Escherichia coli... 21
1.3.1.1 Prova presuntiva... 21
1.3.1.2 Prova confirmativa para coliformes termotolerantes... 22
1.3.1.3 Identificação bioquímica... 23
1.3.1.3.1 Motilidade e Indol... 23
1.3.1.3.2 Triagem para fermentação de glicose, lactose e sacarose... 25
1.3.2 Gênero Salmonella... 27
1.3.2.1 Pré-enriquecimento seletivo... 27
1.3.2.2 Isolamento e seleção... 27
1.3.2.3 Identificação bioquímica... 29
1.3.2.3.1 Motilidade, Indol e produção de H2S... 29
1.3.2.3.2 Triagem para a fermentação de glicose, lactose e sacarose... 29
1.4 ESPECTROSCOPIA... 30
1.4.1 Espectroscopia no infravermelho... 31
1.4.1.1 Fundamentação teórica... 32
1.4.1.2 Instrumentação... 37
1.5 QUIMIOMETRIA... 39
1.5.1 Pré-processamento de dados... 39
1.5.2 Técnicas quimiométricas... 39
41 1.5.2.1 Análise de Componentes Principais (PCA)... 41
16
1.6 REVISÃO BIBLIOGRÁFICA... 45
2 JUSTIFICATIVA E OBJETIVOS... 50
2.1 OBJETIVOS GERAIS... 50
2.2 OBJETIVOS ESPECÍFICOS... 50
3 PROCEDIMENTO EXPERIMENTAL... 51
3.1 PREPARAÇÃO DAS AMOSTRAS... 51
3.1.1 Polpa de fruta... 51
3.1.2 Cultura de bactérias e preparação do inóculo... 51 3.1.3 Preparação do inóculo... 51
3.2 INSTRUMENTAÇÃO... 51
3.3 QUIMIOMETRIA... 52
3.3.1 PCA e SIMCA... 52 3.3.2 PLS-DA... 53
3.4 AVALIAÇÃO DA PERFORMANCE DO MODELO... 53
3.5 SOFTWARE... 53
4 RESULTADOS E DISCUSSÃO... 55
4.1 ESPECTRO ORIGINAL... 55
4.2 PCA E SIMCA... 57
4.3 PLS-DA... 59
5 CONCLUSÕES... 61
REFERÊNCIAS……….……… 62
17
1 INTRODUÇÃO
1.1 O ABACAXI
O Abacaxi, de espécie Ananas comosus, é uma planta tropical com
múltiplas frutas comestíveis que pode ser consumido fresco, enlatado ou em forma de suco, também é encontrado em uma grande variedade de alimentos, tais como: sobremesas, salada de frutas, geléia, iogurte, sorvete, doces. Esta fruta possui baixa acidez, entre 0,6 a 1,62%, com uma média de pH acima de 4,51. A polpa apresenta cor branca, amarela ou laranja-avermelhada2.
Visando a segurança do consumidor, é de extrema importância compreender todas as respostas fisiológicas de microorganismos patogênicos a fatores de estresse sofridos durante o transporte, armazenamento e processamento da polpa dos frutos. E, diante destes fatores, aliar-se a uma combinação de diferentes barreiras para controlar a atividade microbiana, podendo, desta forma, aumentar a eficiência na preservação e diminuir o impacto sobre a qualidade do alimento.
1.2 MICRO-ORGANISMOS PATOGÊNICOS E DOENÇAS TRANSMITIDAS POR ALIMENTOS
Compreende-se micro-organismos patogênicos, aqueles que, dependendo das predisposições intrínsecas do hospedeiro, podem gerar ou não um estado patológico manifesto, denominado doença infecciosa, que também é transmissível3. De acordo com a ICMSF (Comissão Internacional de Especificações Microbiológicas em Alimentos), os organismos patogênicos são agrupados em três grupos: I – Micro-organismos que oferecem risco direto, moderado e com difusão limitada, II- Micro-organismos que oferecem risco direto, moderado e com difusão potencialmente extensa e III- Micro-organismos que oferecem um risco direto e grave4.
18
de toxinfecções alimentares estão a Escherichia coli e a Salmonella enterica5.
Ambas pertencem à família Enterobacteriaceae, que possui estrutura de bastonetes gram-negativos, anaeróbios facultativos, são móveis e com flagelos peritríquios e possuem como habitat natural o trato intestinal de humanos e animais. Os micro-organismos desta família são aeróbios e anaeróbios facultativos, fermentam a glicose e reduzem o nitrato ao nitrito. A principal característica que os tornam gram-negativos é a presença da parede celular, que é constituída de fosfolipídeos, proteínas e lipopolissacarídeos6.
Figura 1- Estrutura celular de organismos gram-negativos.
(Fonte:www.biomania.com.br, acessado em 5 de agosto de 2013.)
O lipopolissacarídeo (LPS) é o principal antígeno dessa família, ele é constituído por um oligossacarídeo em um lipídeo A. As enterobactérias podem ser classificadas sorologicamente de acordo com o componente do seu LPS: polissacarídeo somático (O). O antígeno somático (O) é um polissacarídeo termoestável e forma a parte do lipopolissacarídeo presente na membrana externa das bactérias Gram-negativas6,7.
1.2.1 Gênero Escherichia
19
A Escherichia coli (Figura 2) é uma das espécies de Escherichia, suas cepas estão presentes naturalmente no trato intestinal, em organismos debilitados/imunossuprimidos ou quando são introduzidas em outros tecidos após um trauma ou cirurgia, a sua forma não patogênica pode causar infecções. No entanto, a Escherichia coli geralmente é transmitida através da ingestão de alimentos ou água que entraram em contato com material fecal, sendo assim, um indicador de contaminação fecal.
Figura 2- Imagem ampliada de Escherichia coli O157:H7.
(Fonte: www.molecularstation.com)
A espécie E.coli patogênica é subdividida em duas subcategorias, a extraintestinal e a intraintestinal ou diarreiogênicas, sendo esta última subdividida em pelo menos cinco categorias. Dentre estas está a E.coli enterohemorrágica (STEC - EHEC) que é caracterizada pelas toxinas que produz sorotipos de E.coli
20 1.2.2 Gênero Salmonella
O gênero Salmonella também pertence à família Enterobacteriaceae.
Espécies desse gênero são móveis e gram-negativos. As Salmonellas são tipicamente definidas pela sua capacidade de utilização de citrato com única fonte de carbono e a lisina como única fonte de nitrogênio e por serem produtoras de H2S em ágar três açúcares, exceções a estas características são utilizadas para
definir seus sorotipos específicos.
O gênero Salmonella é composto de duas espécies, a Salmonella entérica
(Figura 3) e a Salmonella bongori, sendo a S. entérica subdividida em seis outras subespécies. Este gênero teve seu genoma analisado e foi encontrada uma alta variabilidade genética. Os membros deste gênero podem ser isolados a partir de alimentos ou água contaminados com fezes, eles não são organismos de vida livre no meio ambiente.
Figura 3- Imagem ampliada da Salmonella entérica.
(Fonte: Volker Brinkmann)
A sorotipagem é baseada na caracterização de três estruturas de superfícies: o antígeno S, que é a porção mais externa da camada que cobre a célula bacteriana, o antígeno H, que é a porção flagelar bacteriana e o antígeno Vi, que é um polissacarídeo capsular presente em sorotipos específicos. A
21
As estirpes de Salmonella são classificadas como tifoide ou não tifoide que corresponde à doença que está associada. Cepas de Salmonella não tifoide normalmente causam infecções intestinais, acompanhados por diarreia, febre e cólicas abdominais que normalmente duram uma semana ou mais, podendo também causar infecções extra intestinais como bacteremia ou osteomielite.
O contágio humano está comumente ligado aos alimentos, podendo ser transmitido pelo contato direto com animais, água contaminada e por contato humano. Cerca de 1,4 milhões de casos da doença e 600 mortes são causadas por salmonelose não tifoide nos Estados Unidos9.
1.3 PROCEDIMENTO OPERACIONAL PADRÃO DE ANÁLISES MICROBIOLÓGICAS NA INDÚSTRIA DE ALIMENTOS
1.3.1 Gênero Escherichia coli
1.3.1.1 Prova presuntiva
Para a identificação de Escherichia coli inicialmente realiza-se uma prova presuntiva por meio da inoculação de 1mL de diluições da amostra (10-1, 10-2,10
-3...) em placas de Petri cobertas com duas camadas de Ágar Cristal Violeta
22
Figura 4- Crescimento de microrganismos fermentadores de lactose (colônias rosadas) em ágar VRBA.
(Fonte:www.pvl.pt, acessado em 07 de Junho de 2013).
1.3.1.2 Prova confirmativa para coliformes termotolerantes
As colônias presentes na prova presuntiva passam, então, pela etapa de confirmação, nesta etapa as colônias suspeitas são inoculadas em tubos contendo Caldo Escherichia coli (EC) e incubadas a 45°±0,2°C por 24 a 48h. O caldo EC apresenta em sua composição uma mistura de fosfatos que lhe confere um poder tamponante impedindo a sua acidificação. A seletividade é dada através de sais biliares responsáveis pela inibição de micro-organismos Gram positivos10.
23
Figura 5- Presença de gás e leve turbidez em tubos contendo caldo Escherichia coli após 24h de incubação.
(Fonte: A autora).
1.3.1.3 Identificação bioquímica
As amostras que apresentarem-se positivas para a prova confirmativa, são submetidas à identificação bioquímica, que se baseia na evidenciação das propriedades fisiológicas e metabólicas das culturas suspeitas caracterizando o microrganismo11.
1.3.1.3.1 Motilidade e Indol
24
Figura 6- Ágar SIM, (A) ágar sem contaminação, (B) teste de Indol positivo, (C) teste de Indol negativo e (D) produção de H2S com aparecimento de precipitado
negro.
(Fonte : Adaptado de www.microbiology.scu.edu.tw, acessado em 27 de Junho de 2013).
O meio é composto por peptonas que são fontes de triptofano, o principal intermediário da degradação do triptofano é o ácido indolpirúvico que pode formar Indol (resultado positivo) por desaminação e Escatol (resultado negativo) por descarboxilação do ácido idolacético. A presença de Indol é detectada pela realização do teste colorimétrico (Figura 7), adicionando-se o reativo de Kovacs, que é composto de p-dimetilaminobenzaldeído, álcool amílico e ácido clorídrico. A
25
Figura 7- Esquema reativo de (A) produção do Indol e (B) reação do Indol com o reativo de Kovacs.
(Fonte: A autora).
1.3.1.3.2 Triagem para fermentação de glicose, lactose e sacarose
26
Figura 8- Procedimento de inoculação em ágar TSI.
(Fonte: A autora).
27
Figura 9- Ágar TSI, (1) sem inoculação e (2) inoculado com Escherichia coli.
(Fonte: A autora).
1.3.2 Gênero Salmonella
1.3.2.1 Pré-enriquecimento seletivo
Esta etapa tem o objetivo de minimizar os efeitos do processamento do alimento que podem promover stress nas células de Salmonella. É utilizada uma solução salina peptonada que mantém o pH, evitando que a microbiota acompanhante acidifique o meio, prejudicando a recuperação das células de
Salmonella. Adiciona-se 225 mL da solução a 25 g da amostra e incuba-se a 36±1 °C por 24 h11.
1.3.2.2 Isolamento e seleção
Após a etapa de pré-enriquecimento, realiza-se a seleção das colônias de
Salmonella em dois meios sólidos: o ágar XLD e o ágar Rambac pelo método de estriamento de superfície.
28
não fermenta a lactose e a sacarose, mas fermenta a xilose, mudando sua cor de vermelho para amarelo devido a presença do vermelho de fenol. Em seguida o pH aumenta devido a descarboxilação da Lisina (Figura 10), tornando o vermelho de fenol novamente vermelho. A produção de H2S produz colônias com um centro
negro (Figura 11)11.
Figura 10- Reação de descarboxilação da Lisina através da enzima Lisina Descarboxilase (LDC), produzindo Cadaverina e dióxido de carbono.
(Fonte: A autora).
Figura 11- Crescimento de Salmonella em ágar XLD com aparecimento de centros escuros devido a precipitação do H2S.
(Fonte: A autora).
29
Figura 12- Crescimento de colônias rosadas em ágar Rambac, possível contaminação por Salmonella.
(Fonte: http://www.chromagar.com/p-Chromogenic_agar_Technology, acessado em 15 de Junho de 2013)
1.3.2.3 Identificação bioquímica
1.3.2.3.1 Motilidade, Indol e produção de H2S
Para a Salmonella o ágar SIM se apresenta, quase sempre, escuro devido a reação do H2S com Citrato de Ferro e amônio (presentes do meio), formando
um precipitado negro, que dificulta a visualização da motilidade, que é positiva para a maior parte das Salmonellas. Enquanto que a presença de Indol é negativa (Figura 6-C)11.
1.3.2.3.2 Triagem para a fermentação de glicose, lactose e sacarose.
No ágar TSI ocorre a fermentação da glicose por aerobiose com produção de CO2, água e energia e fermentação da glicose por anaerobiose com produção
de ácidos orgânicos, aldeídos, álcoois, CO2, H2 e energia. A glicose está em
concentração dez vezes menor que os outros carboidratos e quando acaba o microrganismo começa a utilizar as peptonas e produzir compostos alcalinos. Como na superfície não há produção de ácidos o meio fica alcalino. Neste, também há o aparecimento de precipitado negro devido a produção de H2S pela
30
Figura 13- Ágar TSI, (1) contaminação por Salmonella, (2) não inoculado.
(Fonte: A autora).
1.4 ESPECTROSCOPIA
A espectroscopia mede interações da radiação com a matéria para obter informações sobre uma amostra. Inicialmente o analito encontra-se em seu estado de energia mais baixo (estado fundamental), depois de estimulado, algumas espécies no analito sofrem uma transição para um estado de energia maior (estado excitado).
Cada espécie molecular possui a capacidade de absorver frequências características. À medida que a luz atravessa o analito ela é absorvida e sua intensidade decresce a medida que o analito é excitado.
A transmitância, ou seja, a fração da radiação incidente transmitida pela solução é representada na equação 1.
(1)
Onde:
T = Transmitância;
P = Potência radiante transmitida;
Po = Potência radiante incidente.
31
A absorbância A de uma solução está relacionada com a transmitância de forma logarítmica, como mostrado na equação 2.
(2)
Quando excitadas, as moléculas são submetidas a três tipos de transições, as transições eletrônicas, as transições vibracionais e transições rotacionais. Não obstante, em fase condensada apenas as transições eletrônicas e vibracionais apresentam contribuições relevantes.
1.4.1 Espectroscopia no infravermelho
A espectroscopia na região do infravermelho possui uma ampla faixa espectral que é dividida em Infravermelho Próximo (NIR, 780 – 2500 nm), Infravermelho Médio (MIR, 2500 – 5x104) e Infravermelho Distante (FIR, 5x104 – 1x106). Como pode ser observada na Figura 14, essa radiação geralmente não é
suficiente para causar transições eletrônicas, podendo apenas induzir a transições vibracionais e rotacionais12.
Figura 14- Regiões do espectro eletromagnético com ênfase na região do Infravermelho.
32
1.4.1.1 Fundamentação teórica
Para que uma molécula diatômica absorva radiação na região do infravermelho ela precisa possuir um momento de dipolo permanente, podendo o campo elétrico alternado da radiação interagir com a molécula e causar variações na amplitude de seus movimentos.
Se a frequência da radiação coincidir com a frequência da molécula, ocorre uma transferência de energia e uma variação na amplitude da vibração é alcançada, consequentemente ocorre a absorção.
As vibrações se encaixam na categoria dos estiramentos e deformações angulares (Figura 15). A vibração de estiramento inclui uma variação constante na distância interatômica ao longo de um eixo da ligação entre dois átomos, enquanto que a vibração de deformação angular é caracterizada pela variação do ângulo entre duas ligações13.
Figura 15- Tipos de vibrações moleculares.
(Fonte: a autora)
33
(3)
√
Onde:
μ = massa reduzida m1xm2/m1+m2
A lei de Hooke aplicada a energia potencial (V) pode ser representada pela equação 4.
(4)
Onde:
k= constante de força;
x= coordenada de deslocamento.
A curva de energia potencial possui forma de parábola e é simétrica sob o comprimento da ligação, este modelo leva a frequência da ligação V0.
(5)
√
34
(6) –
Apesar de a mecânica quântica explicar as bandas de absorção observadas no Infravermelho originárias os modos fundamentais de vibração molecular, ela não consegue prever a presença de forças repulsivas entre átomos, a possibilidade de dissociação quando o comprimento da ligação excede a sua extensão e a presença de sobretons nos espectros NIR14. A região do NIR gera espectros que apresenta sobreposições e bandas de combinação das ligações CH, NH e OH de vários grupos funcionais15, outras ligações podem contribuir para o espectro NIR, sendo CC, CO, CN e PO, embora possuam sinal fraco quando comparados com as anteriormente citadas, atribuindo uma contribuição pouco significativa16.
O NIR baseia-se em absorção de energia por parte das ligações existentes nas moléculas de uma amostra que são causadas por três mecanismos diferentes: sobreposição de vibrações fundamentais, combinação de vibrações fundamentais e ainda absorções eletrônicas, gerando bandas de baixa seletividade15,16. Essas bandas são originadas de transições com ∆v=±2, ∆v=±3...,
não obedecendo a regra de seleção, como pode ser observado na Figura 16.
Figura 16- Transições entre níveis vibracionais.
35
Quando dois átomos de aproximam a repulsão coulombiana entre seus núcleos aumenta mais rapidamente que o previsto pelo modelo harmônico, e quando a distancia interatômica se aproxima daquela onde ocorre a dissociação dos átomos, um decréscimo de energia potencial ocorre, comportando-se anarmonicamente (Figura 17).
Figura 17- Diagrama de energia potencial de um oscilador: (A) harmônico e (B) anarmônico.
(Fonte: a autora)
Esse comportamento anarmônico é aproximado através da equação de Morse (7), que descreve a energia potencial da molécula diatômica, como:
(7)
Onde:
a = constante molecular; De = energia de dissociação;
re = distância interatômica de equilíbrio;
36
Desta forma, a equação resultante que descreve os níveis vibracionais é:
(8) Onde:
Xn = constante de anarmonicidade da vibração.
Assim, o modelo anarmônico explica a ocorrência de transições com Δ ± 2 ou maior (sobretons) e bandas de combinação entre vibrações, que são, ambos, os tipos de bandas com maior predominância na região espectral NIR. Outra contribuição importante é que o modelo prevê que a separação entre dois níveis vibracionais adjacentes diminui com o aumento do número quântico vibracional, υ, não sendo mais igualmente espaçadas, como previa o modelo harmônico17.
37
Figura 18- Representação de bandas de absorção na região do infravermelho próximo.
(Fonte: A autora)
1.4.1.2 Instrumentação
Um espectrofotômetro é um instrumento espectroscópico que utiliza um monocromador ou policromador que dispersa a radiação em seus comprimentos de onda constituintes, podendo variar o comprimento de onda sobre uma faixa espectral considerável, juntamente com um transdutor que converte as intensidades radiantes em sinais elétricos.
Os espectrofotômetros utilizam uma variedade de métodos para produzir espectros de radiação NIR, podendo ser instrumentos de filtro, instrumentos dispersivos ou interferômetros (transformada de Fourier), sendo este último o mais aplicado por atribuir precisão e exatidão no comprimento de onda, alta razão sinal/ruído e velocidade de varredura, podendo ser utilizado tanto para análises qualitativas quanto para quantitativas12.
38
Figura 19- Esquema de um espectrofotômetro.
(Fonte: a autora)
Para obter o espectro, primeiro obtém-se um espectro de fundo (Background), e em seguida é obtido o espectro da amostra, então a razão entre os dois espectros é calculada e a absorbância ou transmitância versus o comprimento de onda é registrado, como na Figura 20.
Figura 20- Espectro obtido na região do infravermelho próximo.
39
1.5 QUIMIOMETRIA
A quimiometria é uma área de estudo que aplica métodos matemáticos e estatísticos às ciências químicas, de modo a delinear procedimentos experimentais, obter a máxima informação química através dos dados obtidos, permitindo o desenvolvimento de modelos que estimam propriedades através da análise de outras propriedades19.
Graças à quimiometria foi possível empregar dados analíticos da região NIR, sendo a técnica considerada parte hardware (instrumento) e software (quimiometria).
1.5.1 Pré-processamento de dados
Inicialmente, os dados brutos gerados são tratados na etapa de pré-processamento. Isto deve-se ao fato de os dados analíticos de uma medida instrumental representarem uma propriedade físico-química em função de uma variável (pH, tempo, temperatura,...), e isto gera ruído, diferentes ordens de grandezas, variações sistemáticas de linha base e etc., o que dificulta a interpretação e modelagem destes sinais.
A apresentação de dados de natureza multivariada é feita, geralmente, em forma de tabelas, podendo ser representada por uma matriz de dimensões i x j sendo ‘i’ amostras e ‘j’ medidas experimentais.
Os pré-processamentos podem ser aplicados a variáveis ou as variações existentes entre as amostras. Quando aplicado a variáveis pode ser utilizado de três formas: centralização de dados na média, que consiste em subtrair o valor de cada elemento do vetor coluna (variável) pelo valor médio dos elementos dessa coluna; escalonamento, onde cada elemento de uma linha é dividida pelo desvio padrão e sua respectiva variável, ficando cada uma com a mesma influencia no modelo; e o auto escalonamento que consiste em centralizar os dados na média e em seguida efetuar o escalonamento, ficando a média igual a zero e o desvio padrão igual a 120. Quando aplicado às variações das amostras os métodos mais
40
Os métodos de derivações são aplicados quando deseja-se corrigir problemas relacionados com a variação da linha base, permitindo uma melhor visualização de picos existentes nos sinais originais. O cálculo da derivada pode ser representado por um modelo linear, como na Equação 11.
(11)
Na 1ª derivada o termo ‘b’ é removido do modelo linear, ela é utilizada para remover deslocamentos constantes da linha base.
(12)
Enquanto que na segunda derivada o termo ‘a’ é excluído da equação, nesta há uma eliminação de uma variação linear na linha base, normalmente devido a efeitos de espalhamento.
(13)
41
esse polinômio para estimar o ponto removido e desloca-se para o ponto seguinte repetindo o processo21.
1.5.2 Técnicas quimiométricas
1.5.2.1 Análise de Componentes Principais (PCA)
Uma vez que asrespostas instrumentais carregam informações químicas e físicas das amostras, a análise exploratória é usada para detecção de padrões de associação nos conjuntos de dados e, a partir destes padrões, é possível se estabelecer relações entre as amostras e variáveis, descobrir amostras anômalas (outliers) ou agrupa-las conforme determinadas características. Um dos métodos mais utilizados é a análise por componentes principais (PCA) que baseia-se na transformação ortogonal de um conjunto de observações em um conjunto de valores chamados componentes principais. Trata-se de um método que reduz a dimensionalidade através da representação do conjunto de dados em um novo sistema de eixos (componentes principais) preservando o máximo de informação útil à análise. O conjunto de dados resultante da PCA é muitas vezes utilizado como base para construção de diversos modelos multivariados e fornecem resultados mais satisfatórios quando comparados àqueles obtidos através do conjunto de dados originais22.
Uma trasfomação é realizada em que a matriz original é decomposta como o produto de duas outras matrizes, os escores e os loadings:
(9)
Onde:
X = Matriz de dados I x J;
T = Matriz de escores I x A;
P = Matriz de loadings J x A;
42
J = Número de variáveis;
A = Número de componentes calculados;
I = Número de objetos ou amostras.
Sabendo que os vetores escore ta são ortogonais (isto é, TtT = diag(a) e a
são autovalores da matriz XtX)23, Figura 21.
Figura 21- Representação da simplificação de variárias por PCA.
(Fonte: A autora)
43
dados estará contida na primeira PC; a segunda PC terá mais informação que a terceira, e assim por diante22.
O número ideal de componentes a ser utilizado deve ser encontrado no ponto médio entre os valores máximos do erro e estimativa de erro do modelo multivariado24. Para selecionar o número correto de componentes se utiliza uma
ferramenta chamada de validação cruzada (Cross Validation), ela calcula um erro estimado que um modelo multivariado apresenta frente a amostras desconhecidas na previsão de um parâmetro de interesse, para isso as mesmas amostras de calibração são utilizadas e suas respectivas respostas instrumentais que foram usadas para a construção do modelo.
O método de validação cruzada leave-one-out realiza esse cálculo removendo uma amostra por vez, e calculando o modelo na sua ausência. A habilidade de previsão deste modelo é, então, testada utilizando a amostra que foi mantida fora da construção do mesmo. Este procedimento é repetido até que todas as amostras de calibração disponíveis tenham sido excluídas uma vez e reincorporadas no modelo, individualmente. A estimativa de erro é dada pela raiz quadrada do erro médio de validação cruzada (RMSECV) que é definido como25:
(10) ∑ ⁄
Onde:
Ycv,i = Estimativa de Yi com a amostra i excluída
N = Número de amostras do conjunto de calibração
1.5.2.2 Modelagem Independente por Analogia de Classe (SIMCA)
44
diferenciação entre as classes é dada pela distancia e pelo resíduo entre elas. As amostras desconhecidas são comparadas com os modelos das classes e atribuídas as classes de acordo com a sua semelhança com as amostras de treinamento. Uma nova amostra será reconhecida como um membro de uma classe que seja suficientemente semelhante para os outros membros se não ela irá ser rejeitada. Cada classe é modelada usando modelos separados de PCA. O limite de distância do modelo Smax é utilizado para classificar as amostras novas e
Smax é calculado para o modelo da classe m como se segue:
(11)Smax(m)S0(m) Fc
Onde:
S0 = distância média dentro do modelo;
Fc = (Critério de Fisher) é o valor crítico fornecido pelas tabelas de Fisher-Snedecor. O valor Fc depende da percentagem de risco, geralmente fixada em 5%. A associação de classe é definida em um nível de significância de 2,5% do Smax15,19.
1.5.2.3 Análise Discriminante por Mínimos Quadrados Parciais (PLS-DA)
45
seus scores, T. Estes são obtidos pela decomposição de X e Y em matrizes menores, de acordo com a figura 2217.
Figura 22- Decomposição das matrizes X e Y em matrizes menores.
(Fonte: A autora)
A regressão PLS pode ser adaptada para o reconhecimento de padrões, dando origem ao método PLS-DA. O PLS-DA é realizado usando um código binário exclusivo. Durante o processo de calibração, o método PLS-DA é treinado para calcular os valores de "adesão", um para cada classe, quando o valor estiver acima de um limiar específico de predição a amostra é, então, atribuída a uma única classe, Este método, adaptado das regresões PLS1 ou PLS2, utiliza variáveis espectrais X como preditores e variáveis q (0 ou 1) como variáveis de resposta26.
1.6 REVISÃO BIBLIOGRÁFICA
46
identificar e quantificar duas cepas de E. coli ATCC 25922 e K12 crescido em meio líquido. Os autores empregaram a análise de componentes principais (PCA), análise de fator discriminante (DFA) e rede neural artificial (ANN) como ferramentas quimiométricas. Os modelos de PCA não conseguiram diferenciar as espécies. Para dados de classificação o DFA apresentou-se satisfatório, e mostrou diferenças entre grupos da mesma espécie e relacionou estas diferenças à concentração. Para relacionar dados qualitativos com quantitativos modelos de ANN foram construídos, estes apresentaram bons resultados de predição, com coeficiente de regressão de 0,9827.
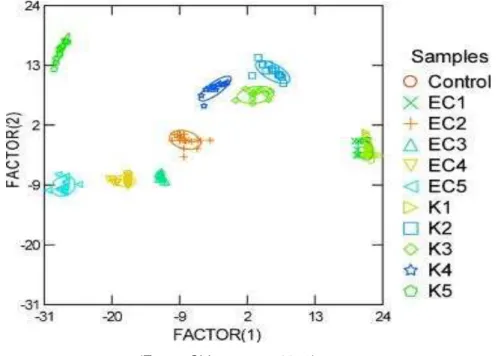
Figura 23- DFA dos espectros NIR de E.coli ATCC 25922 e E.coli K12 com diferentes concentrações de células.
(Fonte: Siripatrauan, 2010)
Suthiluk (2008) estudou o uso do NIR como um procedimento rápido e não destrutivo para medir a quantidade de contaminação por bactérias em repolho picado usando regressão por mínimos quadrados parciais (PLS). Resultados suficientemente precisos foram obtidos com o erro de polarização corrigida para o padrão de previsão (PTS) de 0,46 log ufc/g para a solução Stomacher e 0,44 log ufc/g para a solução de lavagem28.
47
Figura 24- Espectros do ‘extato b’ do repolho picado, (a) espectro original e (b) espectro com msc + segunda derivada.
(Fonte: Suthiluk, 2008).
Al-Holy, em seu trabalho,utilizou a espectroscopia NIR com transformada de Fourier para discriminar Escherichia coli O157:H7 ATCC 35150 de outras bactérias (E.coli ATCC 25522, Bacillus cereus ATCC 10876, Listeria innocua
ATCC 51742) em suco de maçã. Análises de Componentes principais (PCA) e Modelagem Independente por Analogia de Classe (SIMCA) foram utilizados. As amostras foram classificadas com 82% de limite de confiança para diferenciar
48
Figura 25. Representação dos espectros de L.innocua ATCC 51742, B.cereus ATCC 10876, E.coli
O157:H7 ATCC 35150 e E.coli O157:H7 ATCC 25522, diferenças na região de 2000-1000. cm-1 pode ser observado.
(Fonte: Al-Holy, 2006)
49
Figura 26- Representação de PC1xPC3 dos escores obtidos por regressão PLS1 de amostras com L.plantarum, L.mesenteroides e L.sakei a níveis de
concentração 3-9 log de ufc/mL.
50
2 JUSTIFICATIVA E OBJETIVOS
2.1 OBJETIVOS GERAIS
O objetivo do presente trabalho foi avaliar a eficiência da espectroscopia NIR na determinação dos micro-organismos (Escherichia coli e Salmonella
Enteritidis) inoculados em polpa de fruta (abacaxi), os quais apresentam um histórico de surtos associados a alimentos.
2.2 OBJETIVOS ESPECÍFICOS
Desenvolver uma metodologia de análise que atribua rapidez e precisão no controle de qualidade microbiológico de polpa de abacaxi industrial;
Utilizar a técnica da Espectroscopia da região do Infravermelho Próxima (NIR) como um método alternativo na determinação de Escherichia coli e Salmonella
Enteritidis em polpa de abacaxi;
Desenvolver modelos quimiométricos de classificação, utilizando aAnálises de Componentes Principais (PCA), Modelagem Independente por Analogia Classe (SIMCA) e Análise Discriminante por Mínimos Quadrados Parciais (PLS-DA); Contribuir para o desenvolvimento de métodos quimiométricos que possam ser
51
3 PROCEDIMENTO EXPERIMENTAL
3.1 PREPARAÇÃO DAS AMOSTRAS
3.1.1 Polpa de fruta
A polpa de fruta de abacaxi foi obtida de um supermercado em Natal, estado do Rio Grande do Norte, Brasil.
3.1.2 Cultura de bactérias e preparação do inóculo
As cepas de Escherichia coli – INCQS00171 e Salmonella Enteritidis – INCQS00258, utilizadas neste estudo, foram fornecidas pelo Laboratório de Microbiologia Médica, da Universidade Federal do Rio Grande do Norte, RN, Brasil. A cepas estavam sob congelamento em uma mistura de leite, BHI (Brain Heart Infusion; Oxoid Ltd. Basingstoke, England) e glicerol para manter o suprimento de nutrientes e a crioproteção.
3.1.3 Preparação do inóculo
As cepas foram inoculadas em 1 ml de Caldo TSB (Triptic Soy Broth, Becton, EUA), a 35 ° C durante 24 h. A suspensão bacteriana foi padronizada para 0,5 na escala de MacFarland (~ 108 ufc / mL) em solução salina estéril. Posteriormente, cada cultura foi centrifugada (4000 rpm, durante 5 min). O sobrenadante foi descartado e o precipitado resultante foi ressuspenso em 1 ml de solução salina a 0,9% e centrifugado novamente. Este processo foi repetido mais duas vezes para remover os componentes do meio. O precipitado obtido foi ressuspenso em 1 ml de polpa de abacaxi (~ 10 ° C).
3.2 INSTRUMENTAÇÃO
Como mostrado da Figura 27, os espectros foram medidos utilizando um Espectrofotômetro Antaris MX FT-NIR (Thermo Fisher Scientific Inc., EUA), equipado com sonda de fibra ótica de transflectância. Os espectros NIR foram obtidos ao longo de um gama de 10,000-4000 cm-1, ou 1000-2500 nm, e foram
52
de medição foi de 26 s (32 scans) por espectro. Os espectros de absorbância das amostras da suspensão bacteriana foram obtidos contra o espectro da sonda em repouso. Seringas descartáveis de 1 mL foram utilizadas para adicionar uma gota da amostra, aproximadamente 0,1 ml, em uma superfície de alumínio com aproximadamente 0,1 mm de espessura. A sonda de trasflectância foi posicionada sobre a superfície da amostra. Após cada amostra a sonda de transflectância foi lavada com etanol (70% v / v) e seca com papel. Cinqüenta espectros (N = 50) foram carregados seguindo a sequência de cada espécie à temperatura ambiente.
Figura 27- Etapas de obtenção dos espectros. (a) fibra ótica na posição inicial, para a obtenção do Background, (b) posicionamento da sonda sobre a
amostra para coleta dos espectros e (c) armazenamento de dados.
(Fonte: A autora).
3.3 QUIMIOMETRIA
3.3.1 PCA e SIMCA
Neste estudo, foi utilizado um total de 50 espectros NIR, divididos em
53
respectivamente, foram analisados. O desempenho do modelo SIMCA foi avaliado, comparando os valores de sensibilidade e especificidade.
3.3.2 PLS-DA
O método PLS-DA foi utilizado para diferenciar entre as duas classes diferentes de bactérias, e que foram divididas em dois conjuntos um de calibração e outro de validação. A análise PLS-DA foi realizada utilizando um conjunto de calibração com 15 e 21 amostras de E. coli e S. Enteritidis, respectivamente. Para a validação externa, 8 e 6 amostras de E.coli e S. Enteritidis, respectivamente. A validação cruzada foi realizada utilizando subconjuntos aleatórios, o que significa que cada conjunto de teste foi formado a partir de uma seleção de objetos, de forma que um único objeto é incluído em apenas um conjunto de testes. O número de variáveis latentes selecionados para cada modelo foi determinado utilizando o menor erro quadrático de previsão (RMSEP).
3.4 AVALIAÇÃO DA PERFORMANCE DO MODELO
A avaliação do desempenho do modelo foi efetuada através da análise de sensibilidade e especificidade. Sensibilidade é a estimativa percentual experimental de amostras classificadas corretamente em sua própria classe, e especificidade é a estimativa percentual experimental de amostras de outra classe que a classe modelo rejeita. Um modelo perfeito de classe tem 100% de sensibilidade e especificidade de 100%31.
3.5 SOFTWARE
54
55
4 RESULTADOS E DISCUSSÃO
4.1 ESPECTRO ORIGINAL
A Figura 28 mostra os espectros NIR das suspensões de bactérias de duas classes (E. coli e S. Enteritidis). Como pode ser observado há dificuldade, e até mesmo impossibilidade de encontrar diferenças significativas entre as duas classes apenas observando seus espectros puros, isto deve-se ao alto grau de sobreposição de bandas. Estas bandas são originadas do segundo sobretom do estiramento O–H (982 nm), do primeiro sobretom do estiramento O–H (1456 nm), e a combinação de bandas do estiramento assimétrico e deformação angular das vibrações do O–H (1940 nm). Também pode-se observar que houve um desvio da linha de base e bias consistente, que é muito comum para espectros NIR adquiridos por técnica de reflectância difusa devido ao alto espalhamento de luz32.
Figura 28- Espectros NIR originais das suspensões de bactérias E. coli e S. Enteritidis de 50 amostras, sendo 25 para E.coli e 25 para S. Enteritidis.
(Fonte: a Autora).
56
realizada para corrigir o efeito de dispersão da luz no espectro de absorção. Esta etapa foi essencial para a aplicação dos outros pré-processamentos. Foram construídos modelos com primeira e segunda derivada de Savitzky-Golay com janelas de 3 a 15 pontos (Apêndice A) nos espectros puros e em regiões específicas. Foi utilizada a derivada porque esta torna as características espectrais das diferentes cepas de bactérias mais proeminentes e por ser muito usada para processar dados espectrais, pois separa sobreposições de bandas de absorção, elimina desvios de linha de base e aumenta a resolução espectral aparente. Estes efeitos são mostrados na Figura 29.
Figura 29- Espectros NIR das suspensões de bactérias E.coli e S. Enteritidis pré-processada com MSC e segunda derivada de Savitzky-Golay com janela de
15 pontos com atribuição das possíveis estruturas presentes na parece celular dos gram-negativos.
57
4.2 PCA E SIMCA
Após os pré-processamentos, a faixa de 1000 a 1111 nm foi removida com a intenção de eliminar o ruído espectral e, em seguida, realizou-se a análise de PCA. Um modelo PCA foi construído a partir do conjunto de calibração usando 3 componentes principais, explicando juntos 98,62% do total de variância nos dados após a aplicação do pré-processamento (MSC e primeira derivada Savitzky-Golay com janela de 15 pontos). Como pode ser visto na Figura 30, não há separação clara entre as duas classes. Neste caso, a figura mostra que os escores de PCA resultantes da aplicação desta técnica, para os dados de espectroscopia NIR, não foram capazes de diferenciar as classes de bactérias.
Figura 30- Escores de PCA dos espectros NIR das suspensões de E.coli e S. Enteritidis.
(Fonte: a Autora).
58
concentração média de células bacterianas foi consistentemente 1 × 108 ufc / ml, que forneceu sinais intensos na região NIR. Por isso que o PCA, que é freqüentemente usado como um método de classificação não-supervisionado, não foi capaz de fornecer uma separação suficiente entre as classes, e também pelo fato do mesmo não ser eficaz quando a variação dentro do grupo é maior do que a diferença entre os grupos. Em tais situações, a utilização de métodos de classificação supervisionados deve ser considerada.
Para reduzir o efeito da água nos modelos supervisionados foi removido o pico principal da água (1900 nm), removendo, assim, a banda de estiramento O-H da água.
Antes da construção dos modelos, as amostras foram selecionadas utilizando o gráfico de PCA, pois mostrou-se mais satisfatório em relação à seleção de amostras utilizando o algorítmo Kennard-Stone (Apêndice B). O último não conseguiu diferenciar amostras por grupos, reconhecendo as amostras como sendo de um único grupo, este fato pode ser explicado pela alta variabilidade das amostras.
Foram costruídos modelos SIMCA para cada uma das duas classes usando um total de 50 amostras. Os modelos SIMCA apresentaram um baixo desempenho, como pode ser visto na Tabela 1 que mostra o melhor modelo obtido. Isto deve-se ao fato do modelo SIMCA ser baseado na Análise de Componentes Principais, não sendo capaz de identificar diferenças entre as classes e similaridades dentro de cada classe.
Tabela 1- Modelo SIMCA com segunda derivada na região de 1111-2000nm.
Bactéria
Calibração Previsão
Acertos %Variância Falsos Negativos
Acertos %Variância Falsos Negativos
E.coli 14 93,33 1 8 100 0
59
Atendo ao fato de que os modelos de PCA e SIMCA não foram capazes de diferenciar as duas classes de bactérias, modelos PLS-DA foram construídos. O PLS-DA foi escolhido por levar em consideração valores de X e Y, fornecendo mais informações ao modelo.
4.3 PLS-DA
O conjunto de dados com 256 variáveis (região selecionada) foi utilizado para o modelo de classificação PLS-DA. Durante a construção do modelo, algumas amostras foram removidas, tais como outliers com base na avaliação dos resíduos e T2 de Hotelling (Apêndice C). Modelos de classificação foram
construídos para todas as combinações de E. coli e S. Enteritidis. Os modelos foram validados por validação cruzada. Foi realizada a seleção dos dados de calibração para encontrar as variáveis ideais para a classificação. O modelo PLS-DA foi calculado e validado. O R2, e RMSEC RMSECV para os modelos PLS-DA de calibração foram 0,87, 0,46 e 0,69, respectivamente, para a E. coli. Para S. Enteritidis os valores R2, RMSEC e RMSECV foram 0,87, 0,60 e 0,68, respectivamente. O PLS-DA apresentou um bom desempenho, atingindo uma capacidade de predição de 87,5% para a E. coli e de 88,3% para S. Enteritidis, respectivamente. A Tabela 2 apresenta o resumo dos resultados obtidos para vários modelos de PLS-DA.
Tabela 2- Modelos PLS_DA com primeira e segunda derivada nas regiões (A) espectro puro e (B) 1111-2000nm.
Bacterium
Calibração Previsão
Acertos Variância Falsos Negativos
Acertos Variância Falsos Negativos
E.coli(A)¹ 12 77,70 3 6 75,00 2
S.enteritidis(A)¹ 18 85,70 3 3 58,30 3
E.coli(B)¹ 14 88,88 1 6 85,70 2
S.enteritidis(B)¹ 7 33,33 14 4 75,00 2
60
S.enteritidis(B)² 19 90,47 2 5 83,33 1
Os resultados dos diferentes modelos com os valores de sensibilidade e especificidade para a validação cruzada e os modelos testados foram calculados. Para os modelos PLS-DA os valores ideais obtidos para a sensibilidade e especificidade foram de 0,87 e 0,83, respectivamente, em modelos de diagnósticos um elevado valor de especificidade é muitas vezes preferido dado que tal reduz o número de falsos positivos.
Os resultados dos modelos PLS-DA sugerem que também é possível a utilização de técnicas quimiométricas e que estas são adequadas para a detecção de bactérias inoculadas em polpa de fruta.
Comparando os resultados obtidos em trabalhos anteriores29,33, pode ser observado que para construção de modelos com bactérias o SIMCA apresenta resultados altos na classificação, porém quando amostras de validação são testadas o modelo torna-se falho. Enquanto que, o PLS-DA, apresenta-se uma boa ferramenta para solucionar problemas de variabilidade dentro da classe, conseguindo bons modelos de validação.
O presente trabalho mostra-se promissor para solucionar deficiencias na indústria de polpa de fruta quanto à análises microbiológica das espécies
61
5 CONCLUSÕES
Tendo em vista os resultados obtidos, podemos concluir que é possível utilizar os métodos quimiométricos para diferenciar colônias de bactérias (Escherichia coli e Salmonella Enteritidis) a partir da espectroscopia NIR (1000– 2500 nm). Foi avaliado que o método PLS-DA apresentou melhores resultados.
Os resultados mostraram-se satisfatórios, porém apresentaram falhas na aquisição dos espectros que sofreu variações físicas (temperatura, tempo, etc) e baixa sensibilidade do equipamento. Para solucionar este problema técnicas quimiométricas tem sido desenvolvidas.
62
REFERÊNCIAS
1. KAEWTATHIP, T.; CHAROENREIN, S. Changes in volatile aroma compounds of pineapple (Ananas comosus) during freezing and thawing, International Journal of Food Science & Technology. v. 47, p. 985 – 990, 2012.
2. GIACORRELLI, E. J., PY, C. Abacaxi no Brasil. Campinas: Fundação Cargil, 1989.
3. CARROLL, C. K.; BUTEL, J. S. MORSE; S. A. Microbiologia Médica de Jawetz, Melnick e Adelberg. 25 ed. [S. l.]: Geo. F. Brooks, 2012.
4. INTERNATIONAL COMISSION OF MICROBIOLOGY SPECIFICATIONS IN FOOD. <http://www.icmsf.org/main/articles_papers.html>. Acesso em: fev. 2013.
5. MANNING, S.D. Escherichia coli infections, 1 ed., David Heymann,
United States of America: 1971.
5 MURRAY, P.R. Microbiologia Médica, 5 ed., Rio de Janeiro: Elsiever, 2006.
6 KAUFFMAN, F. The serology of the coli group, J. Immunol. v. 57, p. 71-11, 1947.
8. VERSALOVIC, J. Manual of Clinical Microbiologic, 10 ed., Texas Children’s Hospital: ASM press, 2011.
9. KONEMAN, E.W. Diagnóstico Microbiológico, 5 ed., Buenos Aires: Editora Médica Panamericana S.A., 1999.
63
Coordenação Geral de Laboratório Animal, 1992.
11. REGINA, J. H. Atlas da Microbiologia de Alimentos, 1 ed. [S.l]: Revista Indústria de Laticínios, 1998.
12. SKOOG, D.A. Princípios de Análise Instrumental, 5 ed. Porto Alegre: Bookman, 2002.
13. PASQUINI, C. Near Infrared Spectroscopy: fundamentals, practical aspects and analytical applications. J. Brasilian Chemical Society, v.14 , p.198-219, 2003.
14. BURNS, D.A.; CIURCZAK, E.W. Handbook of Near-infrared analysis, 3 ed. [S.l]: CRC Press, 2007.
15. NAES, T.; ISAKSSON, T.; FERN, T.; DAVIES, T. A user-friendly guide to multivariate calibration and classification, UK: NIR Publications, 2002.
16. BARKER, M.; RYENS, W. Partial leat squares for discrimintion, Journal of Chemometrics, v. 17, p. 166-173, 2003.
17. NEVES, A.C.O. Espectroscopia no infravermelho próximo e métodos de calibração multivariada aplicados à determinação simultânea de parâmetros bioquímicos em plasma sanguíneo, Natal-RN: Dissertação de Mestrado-UFRN, 2013.
18. MILLER, J.N.; MILLER, J.C., Statistics and chemometrics for analytical chemistry, 4 ed. Chinchester: Prentice Hall, 2000.
64
B., p. 385-413, 1998.
20. BRERETON, R. G. Introdution to multivariate calibration in analytical chemistry, [S.l]: Analyst, 2000.
21. SAVITZKY, A.; GOLAY, M.J.E. Smoothing and differentiation of data by simplified least squares procedures, Analytical chemistry, v. 36, p. 1627-1639, 1964.
22. NEVES, A.C.O.; ARAÚJO, B. L.; VALDERRAMA, P.; MARÇO, P.H.; LIMA, K.M.G. Near infrared spectroscopy and multivariate calibration for simultaneous determination of glucose, triglycerides and high-density lipoprotein in animal plasma, Journal o Pharmaceutical and Biomedical Analisys, v. 66, p. 252-257, 2012.
23. JOLIFFE, I. T. Principal Component Analysis, 2 ed. UK: Springer, 2002.
24. MATTHIAS, O. Chemometrics, Statistics and Computer Application in Analytical Chemistry, 2 ed. Weinheim: WILEY-VCH Verlag GmbH & Co. KGaA, 2007.
25. MARTEN, H.; NAES, T. Multivariate Calibration and Classification, 1 ed. Chichester-UK: NIR Publications, 2002.
26. BARKER, M.; RAYENS, W. Partil Least Squares for discrimination, Jornal of chemometrics, v. 17, p. 166-173, 2003.
27. SIRIPATRAWAN, U.; MAKINO, Y.; KAWAGOE, Y.; OSHITA, S. Near infrared spectroscopy integrated with chemometrics for rapid detection of
65
28. SUTHILUK, P.; SARANWONG, S.; KAWANO, S.; NUMTHUAM, S.; SATAKE, T. Possibility of using near infrared spectroscopy for evaluation of bacterial contamination in shredded cabbage, International Journal of Food Science and Technology, v. 43, p. 160-165, 2008.
29. AL-HOLY, M. A.; LIN, M.; CAVIINATO, A. C.; RASCO, B. The use of Fourier transform infrared spectroscopy to differentiate Escherichia coli
O157:H7 from others bacteria inoculated into apple juice, Food Microbiology, v. 23, p. 162-168, 2006.
30. MARTOS,C. F.; COSANO,Z. G.; RODRIGUEZ, P.F.; ROJOS, M. R.; Identification and quantification of lactic acid bacteria in a water-based matrix with near-infrared spectroscopy and multivariate regression modeling, Food Anal. Methods, v. 5, p. 19-28, 2012.
31. FORINA, M.; ARMANINO, C.; LEARD, R.; DRAVA, G. A class modeling technique based on potential functions, Journal of Chemometrics, v. 5, p. 435-453,1991.
32. WILLIAMS, P.; NORRIS, K. NIR Technology in the Agricultural and Food Industries, St. Paul: Am. Ass. Cereal Chemists, 1990.