UNIVERSIDADE FEDERAL DE OURO PRETO
NÚCLEO DE PESQUISAS EM CIÊNCIAS BIOLÓGICAS – NUPEB
PROGRAMA DE PÓS-GRADUAÇÃO EM CIÊNCIAS BIOLÓGICAS
Par ticipação de componentes do metabolismo do
fosfatidilinositol no contr ole de vias de sinalização induzidas
por glicose em Saccharomyces cerevisiae
AUTORA: FERNANDA MACHADO DE CARVALHO
ORIENTADOR: PROF. Dr. ROGELIO LOPES BRANDÃO CO-ORIENTADOR: PROF. Dr. LUCIANO GOMES FIETTO
Dissertação apresentada ao Programa de Pós-Graduação do Núcleo de Pesquisas em Ciências Biológicas da Universidade Federal de Ouro Preto, como parte integrante dos requisitos para a obtenção do Título de Mestre em Ciências Biológicas, área de concentração: Biologia Molecular
“E você aprende que realmente pode suportar... que realmente é forte, e que
pode ir muito mais longe depois de pensar que não se pode mais. E que
realmente a vida tem valor e que você tem valor diante da vida.”
(William Shakespear e)
D edico est e t rabalho a todos aqueles que com amizade, conf iança e
respeito estiveram ao meu lado durante a concretização do mesmo.
AGRADECIMENTOS
A Deus, meu refúgio e minha fortaleza.
Ao meu orientador professor Dr. Rogelio Lopes Brandão por ter confiado a mim o desenvolvimento desse projeto e pelo exemplo de profissionalismo e competência.
Ao meu co-orientador Dr. Luciano Gomes Fietto por todo o ensinamento científico, pela amizade, pela paciência, por ser tão solícito às dúvidas e questionamentos. Enfim, pela pessoa, pelo professor e pelo cientista que você é.
Ao professor Dr. Ieso de Miranda Castro pelo carinho e dedicação em me ajudar sempre que necessário.
À professora Dra. Juliana Fietto, a quem muito admiro pela competência profissional, o meu muito obrigado pela sua amizade e pelas sugestões e ensinamentos durante o desenvolvimento deste trabalho.
A todas as “gerações de alunos e funcionários do Laboratório de Biologia Celular e Molecular”, afinal ingressei nesse laboratório em 2002 tendo, assim, o privilégio de conviver com pessoas maravilhosas. A todos vocês o meu muito obrigado não somente pela amizade, convivência, companheirismo e diversão, mas pela troca de conhecimentos.
À querida Zezé, uma microbiologista de grande competência e capacidade, pelos ensinamentos, amizade e lições de vida.
À minha eterna amiga Anamaria, pelo real sentindo da amizade e confiança. E como doutoranda do LBCM, pelo compartilhamento da sua sabedoria científica. Você é mais que uma pessoa importante na minha vida, é parte dela. Ter sua companhia é o mesmo que estar vivendo com a certeza de que existem pessoas que realmente se importam conosco.
Aos colegas e amigos do mestrado Nilza, Cássio, Geórgia, Juliana e Marquinhos por todo apoio, divertimento e ensinamentos.
À secretária do NUPEB, Cida pela sua alegria contagiante e disposição a ajudar não somente a mim como a todos os alunos da pós-graduação.
À Juliana e ao Carlito, amigos da época da graduação e que também muito me apoiaram durante o mestrado.
Ás atuais moradoras e ex-alunas da República Girassol, pela amizade e apoio. Vocês realmente fazem parte desse “eu” que me tornei hoje.
RESUMO
Trabalhos realizados pelo nosso grupo de pesquisa demonstraram que a ativação, induzida por glicose, da H+-ATPase de membrana citoplasmática em S. cerevisiae é
dependente do cálcio extracelular e da ação da proteína Pkc1p, além disso, o metabolismo do fosfatidilinositol parece estar envolvido nesse processo: uma cepa de levedura (PJ69-2a) contendo a deleção do gene ARG82, que codifica uma quinase IP3
-IP4 específica, possui altos níveis de IP3, que controlam os níveis de cálcio citosólico
livre. Este estudo, também, mostrou que esse mutante não crescia na presença de galactose. Decidimos, então, analisar o crescimento celular em diferentes fontes de carbono de cepas que apresentavam deleção de genes que codificam proteínas envolvidas no metabolismo do fosfatidilinositol. Somente o mutante arg82∆ não
apresentou crescimento em fontes alternativas de carbono. Verificamos, ainda que o mesmo não apresenta a segunda fase de crescimento celular, possui problemas na desrepressão de genes controlados por glicose e confirmamos novamente a participação de Arg82p na ativação da H+-ATPase. Esses resultados são semelhantes àqueles já demonstrados por outros trabalhos de nosso grupo de pesquisa para o mutante pkc1Δ .
Recentemente, adquirimos a coleção de cepas mutantes EUROSCARF, apresentando deleções individuais em vários genes; decidimos então averiguar se o mutante arg82∆ dessa coleção apresentaria os mesmos resultados do mutante com o
qual vínhamos trabalhando. De forma intrigante, eles foram diferentes. Diante desse fato, uma vez que se tratava realmente dos mutantes arg82∆, o que foi confirmado por
reações de PCR, deletamos novamente o gene ARG82 na cepa selvagem PJ69-2a.
Além disso, construímos cepas de leveduras que apresentavam a deleção do gene ARG82 em associação com deleções de genes que codificam proteínas envolvidas
no metabolismo de IP3 e cálcio e analisamos seus fenótipos. Nossos resultados sugerem
que um fator específico do “background” PJ69-2a – uma possível superexpressão ou mutação do gene YVC1, codificante para um canal de cálcio vacuolar - em associação
ABSTRACT
Previous works from our research group demonstrated that the glucose-induced activation of plasma membrane H+-ATPase in S. cerevisiae is dependent of the
extracellular calcium and Pkc1 p action; furthermore, the phosphatidylinositol metabolism seems to be involved in this process: a yeast strain (PJ69-2a) presenting a deletion of gene ARG82, which encodes a dual-specificity IP3-IP4 protein kinase, shows
high levels of IP3, which controls the free cytosolic calcium levels. This study also
showed that this mutant does not grow in presence of galactose. In this context, we decided to analyze the cellular growth on different carbon sources of strains presenting deletion of genes that encodes proteins involved in the phosphatidylinositol metabolism. Only the arg82∆ mutant did not grow on media containing alternative carbon sources.
Subsequently, we verified that this mutant does not present a second phase of cellular growth; it shows problems in the derepression process of genes controlled by glucose and we confirmed again the involvement of Arg82 p in activation of H+-ATPase. These results are similar to pkc1Δ mutant phenotypes, which were demonstrated by others
works from our research group.
Recently, we acquired the EUROSCARF collection of yeast deleted strains and we decided to check if the EUROSCARF arg82∆ mutant would present the same
response of the mutant which has been used in our experiments. Intriguingly, they were different. In order to explain this, since both strains are real mutants that were confirmed by PCR, we deleted, again, the gene ARG82 in the wild type PJ69-2a.
Moreover, we constructed yeast strains presenting the deletion of gene ARG82 in
combination with deletions of genes that encode proteins involved in IP3 and calcium
metabolism and we analyzed their phenotypes. Our results suggest that, a specific factor of the background PJ69-2a – a possible overexpression or mutation of gene YVC1, that
ÍNDICE
RESUMO VI
ABSTRACT VII
LISTA DE ABREVIAÇÕES XI
LISTA DE TABELAS XVIII
LISTA DE FIGURAS XIX
1. INTRODUÇÃO... 1
1.1 Transdução de sinal em leveduras... 2
1.1.1 Vias de transdução de sinal reguladas por nutrientes... 3
1.1.2 Via de sinalização PKC MAP quinase... 5
1.2 Via de sinalização do fosfatidilinositol e de seus metabólitos... 8
1.2.1 Metabolismo do fosfatidilinositol... 10
1.2.2 O metabolismo do fosfatidilinositol e a regulação de processos celulares... 14
1.2.2.1 Regulação da expressão gênica... 15
1.2.2.2 Exportação nuclear do RNA mensageiro (RNAm)... 17
1.2.2.3 Elongação telomérica e mecanismo de reparo do DNA... 17
1.2.2.4 Tráfego vesicular e proliferação celular... 18
1.2.2.5 Secreção da enzima invertase... 19
1.2.2.6 Regulação de vias de sinalização dependentes de glicose... 19
2. OBJ ETIVOS... 24
2.1 Objetivo Geral... 25
2.2 Objetivos Específicos... 25
3.1 Cepas de Saccharomyces cerevisiae e construções utilizadas nos
experimentos... 27
3.2 Plasmídeo e vetor de expressão utilizados nos experimentos... 28
3.3 Meios de cultura... 29
3.3.1 Meio YP... 29
3.3.2 Meio mínimo sintético (SD)... 29
3.4 Teste de crescimento celular em meios de cultura contendo diferentes fontes de carbono... 29 3.5 Curva de crescimento... 30
3.5.1 Preparo do inóculo... 30
3.5.2 Construção da curva de crescimento... 30
3.6 Dosagem da taxa de consumo de glicose... 30
3.7 Dosagem da taxa de produção de etanol... 32
3.8 Dosagem da atividade enzimática específica da invertase... 33
3.8.1 Inóculo e condições de crescimento das células... 33
3.8.2 Preparação de extratos celulares para a determinação da atividade enzimática da invertase... 33
3.8.3 Dosagem de proteínas... 34
3.9 Dosagem da atividade enzimática específica da β-galactosidase... 34
3.9.1 Transformação de leveduras... 34
3.9.1.1 Preparo de células competentes... 34
3.9.1.2 Transformação de leveduras... 35
3.9.2 Inóculo e condições de crescimento das células de leveduras transformadas... 35 3.9.3 Ensaio da atividade enzimática específica da β -galactosidase... 36 3.10 Construção dos duplo mutantes arg82Δ plc1Δ e arg82Δ yvc1Δ ... 37
3.10.1 Inóculo e condições de crescimento das células... 37
3.10.3 Construção do duplo mutante arg82Δ plc1Δ ... 38
3.10.3.1 Amplificação da deleção do gene PLC1... 38
3.10.3.2 Confirmação da deleção dos genes ARG82 e PLC1... 39
3.10.4 Construção do duplo mutante arg82Δ yvc1Δ ... 40
3.11 Reações de polimerase em cadeia (PCR)... 41
3.12 Eletroforese em gel de agarose... 43
3.13 Acidificação extracelular... 43
3.13.1 Medida da acidificação extracelular induzida por glicose... 43
3.13.2 Determinação do peso seco... 44
3.13.3 Cálculo da taxa de acidificação extracelular... 44
3.14 Reprodutibilidade dos resultados e análise estatística... 47
4. RESULTADOS... 48
5. DISCUSSÃO... 75
6. CONCLUSÃO... 83
7. PERSPECTIVAS... 85
LISTA DE ABREVIAÇÕES
Acil-DHAP Acil-dihidroxicetona-fosfato
ADH Enzima álcool desidrogenase
ADP Adenosina 5`- bifosfato
Al-DH Enzima aldeído desidrogenase AMPc Adesosina monofosfato cíclico
Ar g80p Fator transcricional envolvido na regulação de genes controlados por arginina
ARG80 Gene que codifica para o fator transcricional Arg80p
Ar g81p Fator transcricional “zinc finger” envolvido na regulação de genes controlados por arginina
Ar g82p/Ipk2p Proteína inositol polifosfato multiquinase
ARG82/ARGIII/IPK2 Gene que codifica para a proteína Arg82p/Ipk2p.
Ar gR-Mcm1 Complexo transcricional envolvido no controle do metabolismo de arginina.
ATP Adenosina 5`-trifosfato
Aur 1p Proteína ceramida fosfoinositol transferase (IPC sintase), envolvida na síntese de esfingolipídeos
Ayr1p Proteína acil-dihidroxiacetona-fosfato redutase dependente de NADPH, envolvida na biossíntese do ácido fosfatídico
Bck1p Proteína quinase quinase quinase envolvida na síntese da parede celular
Ca++ Íon cálcio
ChIP “Chromatin immunoprecipitation” – imunoprecipitação de cromatina
Cdk1p Fator de transcrição repressor envolvido na inibição do crescimento celular durante a reprodução
Cds1p Proteína CDP-Diacilglicerol sintase C-terminal Carboxi terminal
DAG Diacilglicerol
Dak1p Proteína dihidroxiacetona quinase D-G-6-P Glicose-6-fosfato
DHA Dihidroxiacetona
DHAP Dihidroxiacetona fosfato
DNA Ácido desoxirribonucléico
D.O. Densidade ótica
Dpp1p/Lpp1p Proteínas fosfatases responsáveis pela defosforilação do DGPP a ácido fosfatídico (PA) e fosfato inorgânico; responsáveis também pela defosforilação de PA a DAG
Fab1p Proteína 1-fosfatidilinositol-3-fosfato 5 quinase
FKS2 Gene que codifica para uma proteína do complexo glucano sintase Fps1p Proteína-canal presente na membrana plasmática; envolvida no
efluxo de glicerol.
G6P-DH Enzima glicose-6-fosfato desidrogenase GAL Genes envolvidos no metabolismo de galactose GAL1 Gene que codifica para a galactoquinase GAL 2 Transportador de glicose
Gal4 p Fator transcricional envolvido no regulação de genes controlados por galactose
GDP Guanosina bifosfato
Glc7p Proteína fosfatase mediadora da repressão de SNF1
Gle1p Nucleoporina citoplasmática envolvida na exportação de RNAm e componente do complexo nuclear do poro Nup82.
Glk1p Proteína glicoquinase
Gat1p/Gat2p Proteínas glicerol 3- fosfato aciltransferases, envolvidas na acilação do glicerol 3-fosfato e dihidroacetona.
Gcy1p Proteína glicerol desidrogenase
Gpa2 p Proteína G de membrana
GPA2 Gene que codifica para a proteína G Gpa2p
GPCR “G-protein-coupled receptors”, receptor acoplado a proteína G Gpd1p/Gpd2p Proteínas glycerol 3-fosfato desidrogenases
Gpp1p/Gpp2p Proteínas DL-glicerol-3-fosfatases
Gpr 1 Receptor de membrana acoplado a proteína G Gr oPI Glicerofosfoinositóis
GTP Guanosina trifosfato
GTPases Família de enzimas responsáveis pela hidrólise de GTP Gup1p Proteína envolvida no transporte de glicerol
Gut1p Proteína glicerol quinase
GUT1 Gene que codifica para a proteína Gut1p Gut2p Proteína glicerol 3-fosfato desidrogenase
HK Proteína hexoquinase
Hos2 p Proteína histona deacetilase
Hxk1p Proteína Hexoquinase PI
Hxk2 p Proteína Hexoquinase P2
Hxtp Proteínas transportadoras de hexose
HXT Gene que codifica proteína transportadora de hexose Ino1p Proteína inositol 1- fosfato sintase
INO1 Gene que codifica para Ino1p
Inm1p/Inm2p Proteínas inositol monofosfatases, envolvidas na biossíntese do inositol
Inositol 1-P Inositol 1-fosfato
INV+ Gene que codifica para a enzima invertase de
Schizosaccharomyces pombe
IPs Inositóis polifosfatos
IP3/ I(1,4,5)P3 Inositol 1,4,5 -trifosfato
IP4 / I (1,3,4,5)P4 Inositol 1,3,4,5 - tetrafosfato
IP4 / I (1,4,5,6)P4 Inositol - 1,4,5,6 tetrafosfato
IP6/ I (1,2,3,4,5,6)P6 Inositol - 1,2,3,4,5,6 hexafosfato IP6k1p/ Ip6k2p Proteínas inositol quinase hexafosfato
IPC Ceramida fosfoinositol
IPCs Complexos esfingolipídeos
Ipk1p Proteína inositol 1,3,4,5,6-pentaquisfosfato 2-quinase IPK1 Gene que codifica para Ipk1p
Ipmkp Proteína inositol fosfato multiquinase IPS Atividade inositol difosfato sintase
Ips1p? Provável proteína que apresenta atividade IPS
Ipt1p Proteína inositol fosfotransferase 1, envolvida na síntese de M(IP)2C
Itr 1p/Itr2p Proteínas transportadoras de inositol
Kcs1p Proteína quinase C supressora do tipo 1 - Proteína inositol quinase hexafosfato
Lyso-PA Acil-glicerol-3-fosfato
MAL Genes envolvidos no metabolismo da maltose
MAL3R Gene que codifica para uma proteína indutora específica do metabolismo de maltose
MAP quinases Proteína quinase ativada por mitógeno MAPK Proteína quinase ativada por mitógeno
MAPKK Proteína quinase quinase ativada por mitógeno
MAPKKK Proteína quinase quinase quinase ativada por mitógeno Mcm1p Fator transcricional componente de ArgR-Mcm1 MCM1 Gene que codifica para o fator transcricional Mcm1p Mig1p Proteína repressora estimulada por glicose
MIPC Manosilinositol fosforilceramida M(IP)2C Manose -(inositol-P)2-ceramida
Mkk1p / Mkk2p Proteínas quinase quinase envolvidas na síntese da parede celular Mpk1p Proteína quinase envolvida na síntese da parede celular
NAD Nicotinamida adenine dincucleotídeo
NADH Nicotinamida adenine dincucleotídeo na sua forma reduzida NADP Nicotinamida adenina dinucleotídeo fosfato
NADPH Nicotinamida adenina dinucleotídeo fosfato na sua forma reduzida NHEJ Via de reparação do DNA - “non-homologous end joining”
NPCs Complexos nucleares do poro
Nup82p Subunidade do complexo nuclear do poro
ONP Orto-nitrofenol
ONPG Orto-nitrofenil β-D-galactopiranosídeo
p/v Peso por volume
PA Ácido fosfatídico
PC Fosfatidilcolina
PCR Reação de polimerase em cadeia
PGM1 Gene que codifica para a proteína fosfoglucomutase que é responsável pela conversão de glicose 6-fosfato a glicose 1-fosfato PGM2 Gene que codifica para a proteína fosfoglucomutase que é
responsável pela conversão de glicose 1-fosfato a glicose 6-fosfato PHO5 Gene que codifica a proteína fosfatase ácida que media hidrólise
do fosfato derivado de nucleotídeo extracelular PI /PtdIns Fosfatidilinositol
PI (3)P Fosfoinositol 3-fosfato PI (4)P Fosfoinositol 4-fosfato PI (3,5) P2 Fosfoinositol 3, 5 - bifosfato
PI (4,5)P2 Fosfoinositol 4, 5 - bifosfato
PI (3,4,5)P3 Fosfoinositol 3,4,5 - trifosfato
PKH2 Gene que codifica para uma proteína quinase Ser/Thr envolvida na via de sinalização mediada por esfingolipídeos.
Plc1p Proteína fosfolipase C inositol específica PLC1 Gene que codifica para Plc1p
Pmc1p Cálcio AT-Pase vacuolar; capta cálcio do citosol para o vacúolo PMC1 Gene que codifica para Pmc1p
PP-IP3 Difosfinositol trifosfato PP-IP4 Difosfoinositol tetrafosfato PP-IP5 Difosfoinositol pentafosfato (PP)2-IP2 Bis-difosfoinositol bifosfato (PP)2-IP3 Bis-difosfoinositol trifosfato (PP)2-IP4 Bis-difosfoinositol tetrafosfato
Plb1p/Plb2p/Plb3p Proteínas fosfolipases envolvidas no metabolismo de fosfolipídeos
Ras Proteína ativadora da enzima adenilato ciclase RE Retículo endoplasmático
Reg1p Subunidade regulatória (tipo 1) da proteína fosfatase Glc7p
Rgt2 p Proteína que atua como sensor de baixa afinidade para glicose e gera um sinal para indução da expressão do(s) gene(s) HXT RGT2 Gene que codifica para o sensor Rgt2 p
Rho1 Proteína pertencente a família das GTPases Rlm1 Regulador gênico para síntese da parede celular RNAm Ácido ribonucléico mensageiro
RT-PCR Reação de polimerase em cadeia e transcrição reversa – “reverse transcription- polimerase chain reaction”
Ser Serina
Slc1p Proteína aciltransferase responsável pela acilação de lyso PA a PA.
Snf1p Proteína quinase envolvida na via principal de repressão por glicose
Snf3 p Proteína que atua como sensor de alta afinidade para glicose e gera um sinal para indução da expressão do(s) gene(s) HXT
Snf4 p Subunidade ativada da proteína Snf1 Stt4p Proteína fosfatidilinositol- 4 quinase
SUC2 Gene que codifica para a invertase
Swi/Snfp Complexo remodelador de cromatina ATP-dependentes – “switch/sucrose nonfermentable”
Sur 1p/Csg2p Proteínas envolvidas na síntese de MIPC
Thr Treonina
URS Seqüência repressiva “’upstream” Vps34p PI 3-quinase
LISTA DE TABELAS
LISTA DE FIGURAS
Figura 1 Via principal de repressão por glicose. 6
Figura 2 Via de sinalização PKC MAP quinase. 9
Figura 3 Biossíntese de fosfolipídios em S. cerevisiae. 12
Figura 4 Via anabólica/catabólica do fosfatidilinositol em
S.cerevisiae.
13
Figura 5 Modelo hipotético da ativação, induzida por carboidrato, da H+- ATPase de membrana citoplasmática mediada por íons cálcio, via Pkc1 p.
22
Figura 6 Mapa do vetor de expressão pYES2/CT. 42
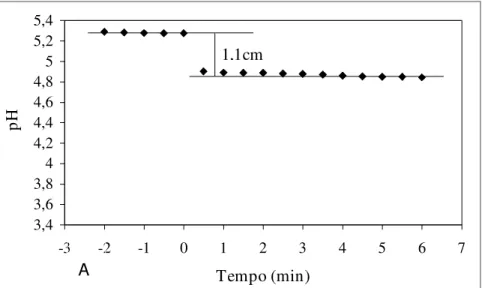
Figura 7 Representação gráfica de acidificação extracelular para o cálculo da taxa de extrusão de prótons.
46
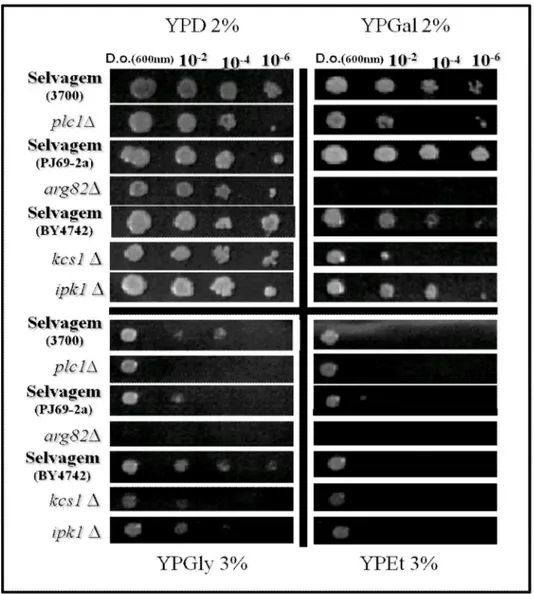
Figura 8 Teste de crescimento das cepas mutantes plc1Δ , arg82Δ , kcs1Δ ,
ipk1Δ e de suas respectivas selvagens 3700, PJ69-2a, BY4742, em
meios de cultura contendo diferentes fontes de carbono.
50
Figura 9 Curva de crescimento celular dos mutantes plc1Δ , arg82Δ , kcs1Δ ,
ipk1Δ e de suas respectivas cepas selvagens 3700, PJ69-2a,
BY4742.
Figura 10 Curva de crescimento celular, dosagem das taxas de consumo de glicose e produção de etanol da cepa selvagem PJ69-2a e do mutante arg82Δ , na presença e ausência de antimicina.
53
Figura 11 Atividade enzimática específica (em nmol de glicose. min-1 . mg proteína -1) da invertase de cepas mutantes
plc1Δ , arg82Δ , kcs1Δ ,
ipk1Δ e de suas respectivas selvagens 3700, PJ69-2a, BY4742,
crescidas na presença de glicose 4% – condição de repressão gênica – e de rafinose 2 % – condição de desrepressão gênica.
55
Figura 12 Atividade enzimática específica (nmol ONP. min-1.mg proteína -1) da β-galactosidase , em função do tempo, das cepas selvagem PJ69-2a e mutante arg82Δ , ambas contendo o plasmídeo
GAL1::lacZ .
57
Figura 13 Teste de crescimento das cepas mutantes arg82Δ e plc1Δ , de suas respectivas selvagens PJ69-2a e 3700, e do duplo mutante
arg82Δ plc1Δ em meios de cultura contendo diferentes fontes de
carbono.
59
Figura 14 Teste de crescimento das cepas mutantes arg82Δ , plc1Δ e de sua respectiva cepa selvagem, BY4742, em meios de cultura contendo diferentes fontes de carbono.
62
Figura 15 Atividade enzimática específica (em nmol de glicose. min-1. mg proteína -1) da invertase das cepas mutantes plc1Δ , arg82Δ , kcs1Δ ,
ipk1Δ e da selvagem BY4742, crescidas na presença de glicose
4% – condição de repressão gênica – e de rafinose 2 % – condição de desrepressão gênica.
Figura 16 Acidificação extracelular induzida por glicose (100mM) em cepas selvagens de S. cerevisiae, PJ69-2a e BY4741, e nos mutantes
arg82Δ provenientes desses “backgrounds”.
64
Figura 17 Análise da inserção do cassete KanMX4 na seqüência codificadora de Arg82p das cepas selvagens PJ69-2a e BY4742.
66
Figura 18 Curva de crescimento celular da cepa LBCM 512 na presença e ausência de antimicina .
68
Figura 19 Atividade enzimática específica (em nmol de glicose. min-1. mg proteína -1) da invertase da cepa LBCM 512 e de sua respectiva selvagem, PJ69-2a, crescidas na presença de glicose 4% – condição de repressão gênica – e de rafinose 2% – condição de desrepressão gênica.
69
Figura 20 Acidificação extracelular induzida por glicose (100 mM) nas cepas S. cerevisiae, selvagem PJ69-2a, no mutante arg82Δ e na cepa LBCM 512.
70
Figura 21 Acidificação extracelular induzida por glicose (100 mM) na cepa
S. cerevisiae, selvagem PJ69-2a e no duplo mutante
arg82Δ yvc1Δ .
73
Figura 22 Teste de crescimento da cepa mutante - arg82Δ yvc1Δ - e de sua respectiva cepa selvagem – PJ69-2a - em meios de cultura contendo diferentes fontes de carbono
1. Introdução
A habilidade dos organismos unicelulares ou multicelulares em detectar e reagir a sinais provenientes do meio externo é fundamental às suas sobrevivências. Entretanto, de acordo com registros fósseis, o surgimento dos organismos unicelulares data de cerca de 2,5 bilhões de anos anteriormente ao aparecimento dos primeiros organismos multicelulares (Alberts et al., 2002). Essa evidência está relacionada ao fato de que esse tipo de organismo encontrou dificuldades em desenvolver mecanismos elaborados de comunicação celular que permitissem a sua adaptação ao meio externo (Alberts et al., 2002), coordenando, assim, seu comportamento em benefício do organismo como um todo.
Os circuitos moleculares das células responsáveis pela detecção, amplificação, integração de diversos estímulos externos e subseqüente geração de respostas capazes de controlar fenômenos intracelulares caracterizam o processo de transdução de sinal (Hug & Sarre, 1993), que embora se diferencie quanto à sua complexidade e componentes celulares parece obedecer a certos princípios conservados entre a maioria dos organismos vivos (Hug & Sarre, 1993).
A detecção de sinais extracelulares ocorre através da difusão de compostos hidrofóbicos pela membrana plasmática ou pela ligação de moléculas sinalizadoras a receptores transmembranares, estimulando, diretamente ou em associação com transdutores, moléculas efetoras que por sua vez atuam no controle das quantidades e distribuição intracelular dos mensageiros secundários, responsáveis em modular a atividade de proteínas via modificações covalentes. Essas proteínas promovem, então, diretamente ou por uma cascata de fosforilações de alvos posteriores, a ativação e/ou inativação de enzimas, bem como a modulação de fatores transcricionais que alteram a expressão gênica da célula e, conseqüentemente, seu comportamento (Alberts et al., 2002).
1.1 – Tr ansdução de sinal em levedur as
encontrada em praticamente toda a superfície terrestre, desenvolvendo-se nos mais diversos ambientes, apresenta uma maquinaria de transdução celular típica de células eucarióticas e é empregada, biotecnologicamente, na síntese de compostos de largo interesse industrial como o etanol, glicerol, enzimas, bebidas alcoólicas, vitaminas e fermento (Winderickx et al., 2003). Além disso, possui o genoma completamente seqüenciado – 12 milhões de pares de bases presentes em pouco mais de 6000 genes e distribuídos em 16 cromossomos (Goffeau et al., 1997).
Uma série de vias de transdução de sinal presentes em leveduras encontra-se bem caracterizada entre elas a via Ras-adenilato ciclase e a via principal de repressão por glicose, que estão relacionadas à disponibilidade de nutrientes (Thevelein, 1994). Havendo ainda famílias de vias ativadas por mitógenos (MAP) como a via PKC MAP quinase (Levin et al. 1994), além da elucidação de vários componentes da via de sinalização do fosfatidilinositol e de seus metabólitos (Flanagan & Thorner, 1992; Patton & Lester, 1992; Flick & Thorner, 1993; Saiardi et al., 1999; Odom et al., 2000).
1.1.1 – Vias de transdução de sinal r eguladas por nutrientes
A disponibilidade de nutrientes no meio é um dos mais importantes fatores de controle do desenvolvimento e crescimento em microorganismos (Rolland et al. 2002), o que permite considerá-los como fontes primárias das vias de sinalização celular.
Em leveduras, a glicose, utilizada como fonte de carbono e energia, induz a biossíntese e/ou ativação das enzimas requeridas à sua ótima metabolização em associação com componentes requeridos para uma rápida taxa de crescimento celular (Cereghino & Scheffler, 1996; Muller et al., 1995, Wills, 1990), além da repressão de classes de genes que codificam para proteínas envolvidas nos processos de captação e metabolização de fontes alternativas de carbono (GAL, SUC2, MAL), respiração celular, gliconeogênese e ciclo do glioxilato (Gancedo, 1992; Johnston & Carlson, 1992; Thevelein, 1994; Trumbly, 1992).
O principal efeito, em nível transcricional, da adição de glicose a células de
S. cerevisiae, ocorre por meio da ativação da via principal de repressão por glicose, que
As concentrações de glicose disponíveis no meio afetam a expressão de genes que codificam para seus próprios carreadores, sendo o transporte intracelular da glicose um dos primeiros eventos essenciais à ativação das vias de sinalização dependentes dessa molécula sinalizadora.
A levedura S. cerevisiae possui em seu genoma 20 genes (HXT1-17, GAL2, SNF3 e RGT2) que codificam para proteínas envolvidas no sistema de transporte de glicose, entre os quais apenas sete (HXT1-7) são conhecidos por codificar transportadores funcionais (Carlson, 1998; Özcan & Johnston 1999), ao passo que, Snf3p e Rgt2p são consideradas proteínas homólogas aos transportadores de glicose, sendo incapazes de transportá-la, mas atuando como sensores dessa molécula e subseqüentemente, gerando um sinal intracelular que culmina com a indução da expressão dos genes HXT (Carlson, 1998; Özcan et al. 1996 e 1998).
A fosforilação da glicose necessária à ativação da via de sinalização desencadeada por essa hexose, é mediada pela ação de três hexoquinases: Hxk1p, Hxk2p e Glk1p (Hohmann et al., 1999), sendo que a hexoquinase 2 possui outros papéis no que se refere ao envolvimento na repressão de genes por glicose, podendo estar relacionada à regulação da função de Snf1p (mecanismo ainda não elucidado) ou ainda interagir diretamente com fatores de transcrição que controlam a expressão desses genes (Jiang et al., 2000).
A proteína quinase Snf1p tem como alvo principal inibir a repressão gênica induzida pelo fator transcricional denominado Mig1 p (Nehlin & Ronne, 1990). Evidências sugerem que a fosforilação de Mig1p, catalisada por Snf1p, e sua localização celular constituem-se em fatores determinantes à sua atividade. Em presença de glicose, essa proteína é rapidamente transportada para o núcleo, enquanto que sob a privação dessa fonte de carbono, Mig1p encontra-se fosforilado, o que promove sua interação com a exportina Msn5p, favorecendo, assim, sua exportação para o citoplasma e conseqüentemente a desrepressão de genes que estão sob o controle da glicose (De Vit & Johnston, 1997) (Figura 1).
Além da ativação da via principal de repressão por glicose, Eraso & Gancedo (1985) demonstraram que a adição dessa fonte de carbono fermentável às células de S.
cerevisiae elevam transitoriamente os níveis de AMPc, sendo esse feito associado à via
Ras-adenilato ciclase (Thevelein, 1991). Para que essa via de sinalização seja desencadeada é necessário que a molécula de glicose seja detectada pelo sistema receptor de membrana GPCR (Gpr1p – Gpa2p), internalizada e fosforilada. Esse sinal é então transmitido, via proteínas Ras1 e Ras2, à enzima adenilato ciclase, que uma vez ativada induz a síntese de AMPc a partir de ATP (Rolland et al.,2000).
O alvo intracelular do AMPc gerado é a proteína quinase A (Pka p), que amplifica o sinal extracelular através da ativação e/ou inativação de várias enzimas e fatores transcricionais por meio de uma cascata de fosforilações protéicas, resultando assim na regulação pós-transcricional de uma variedade de proteínas como as enzimas chaves da gliconeogênese e da glicólise, no controle transcricional de genes envolvidos na resposta geral ao estresse e codificadores para proteínas ribossomais (Neuman-Silberberg et al.,1995; Ruis & Schüller, 1995; Mager & De Kruijff, 1995).
1.1.2 – Via de sinalização PKC MAP quinase
Hxtp
Glicólise Glicose-6-P
núcleo Genes r epr imidos
por glicose Hxk2p Mig1p Snf4p
Snf1p
Glc7p Reg1p
Mig1p
P
Mig1p
citoplasma
Glicose
Hxk2p
Figura 1 – Via principal de repressão por glicose
A desrepressão de genes controlados por glicose ocorre pela inativação do repressor Mig1p através da fosforilação mediada pela proteína quinase Snf1p (associada à sua subunidade regulatória Snf4p) e do seu deslocamento do núcleo para o citoplasma. Sob a presença de glicose, a atividade de Snf1p é inibida pela proteína Glc7p e sua subunidade regulatória Reg1p, o que a impossibilita de exercer seu controle sobre Mig1p. Sugere-se, ainda, que Hxk2p forma um complexo com Snf1p, controlando sua atividade, ou, em associação com Mig1p, se liga diretamente a vários genes regulados por glicose impedindo sua transcrição.
A proteína quinase C (Pkc1p), identificada como integrante de uma cascata de MAP quinases (Hohmann & Mager, 1997), em S. cerevisiae está envolvida no controle da composição da parede celular dessa levedura, em resposta às modificações osmóticas do meio – estresse hipotônico ou choque hiposmótico (Levin et al., 1994 e Davenport et
al., 1994).
Em se tratando da via de sinalização PKC MAP quinase, sua ativação é desencadeada quando a proteína Rho1 (pertencente à família das GTPases), estimulada por altas concentrações de cálcio, limitação de nutrientes, altas temperaturas e/ou baixa osmolaridade promove a ativação de Pkc1p (Nonaka et al., 1995), que por sua vez, através de fosforilações proporciona a ativação em cascata dos demais componentes da via (Bck1p, Mkk1p/Mkk2p, Mpk1p; respectivamente, MAPKKK; MAPKKs; MAPK), desencadeando, subseqüentemente, a ativação de fatores de transcrição repressores, como Cdk1p (envolvido no processo de inibição do crescimento celular durante a reprodução) e/ou indutores, como Rlm1 (proteína que regula a expressão de genes envolvidos na síntese dos componentes da parede celular) (Banuett, 1998). O gene
FKS2 constitui um dos principais alvos dessa via (com indução de Rlm1) e codifica para uma proteína do complexo glucano sintase, envolvida na síntese da parede celular (Figura 2). Deleções em quaisquer genes que codificam para os componentes dessa via causam lise celular na ausência de estabilizadores osmóticos, como o sorbitol ou o cloreto de sódio. Contudo a deleção do gene PKC1 é mais severa quando comparada a dos demais genes componentes da via, sugerindo o possível envolvimento dessa proteína em outras vias de sinalização (Levin et al., 1994) como aquelas sensíveis à glicose (Souza et al., 2001; Brandão et al., 2002; Salgado et al., 2002; Gomes et al. 2005; Trópia et al., 2006).
desrepressão de genes que são reprimidos por glicose; contudo, os mecanismos envolvidos nesse processo ainda não estão completamente elucidados.
1.2. Via de sinalização do fosfatidilinositol e de seus metabólitos
O trabalho pioneiro de Hokin & Hokin (1953, 1955), constituiu-se em uma importante inovação no estudo do fosfatidilinositol e seus metabólitos, pois demonstraram que tanto eles como as enzimas responsáveis por suas sínteses possuem uma participação direta em vias de transdução celular.
Uma série de outras descobertas tem contribuído para que essas vias de sinalização sejam alvos primordiais das pesquisas biológicas. Sendo que os primeiros trabalhos desenvolvidos nessa área concentraram-se na descoberta e caracterização das enzimas responsáveis pelo metabolismo do fosfatidilinositol (fosfatidilinositol e inositol quinases, fosfatases e fosfolipases) (Cocco et al.,1998; York et al.,1998; Maraldi et al., 1999).
Embora resultados interessantes tenham sido obtidos com relação a essas enzimas, as pesquisas mais recentes têm direcionado seus estudos para os alvos finais dessas vias de sinalização, sendo isso viável devido: à síntese de análogos do fosfatidilinositol e de seus metabólitos para a realização de estudos in vitro, à clonagem dos genes que codificam as fosfatidilinositol e inositol quinases para estudos in vivo e à disponibilidade de inibidores potentes e específicos dessas enzimas. (Toker, 2002).
Complexo glucano sintase ↓Nutrientes ↑Temperatura ↓Osmolaridade
↑Ca++
núcleo Rho1p Pkc1p Bck1p Mkk1/2p Mpk1p
Outras vias de sinalização (?) Cdk1 FKS2
Fks 2p
Rlm1 Complexo glucano sintase ↓Nutrientes ↑Temperatura ↓Osmolaridade↑Ca++
núcleo Rho1p Pkc1p Bck1p Mkk1/2p Mpk1p
Outras vias de sinalização (?) Cdk1 FKS2
Fks 2p
Rlm1 Complexo glucano sintase ↓Nutrientes ↑Temperatura ↓Osmolaridade ↑Ca++ ↓Nutrientes↑Temperatura
↓Osmolaridade
↑Ca ↓Nutrientes
↑Temperatura
↓Osmolaridade
↑Ca++
núcleo Rho1p Pkc1p Pkc1p Bck1p Mkk1/2p Mpk1p
Outras vias de sinalização (?) Cdk1 FKS2
Fks 2p
Rlm1 Rlm1Figura 2 – Via de sinalização PKC MAP quinase
A via de PKC MAP quinase é desencadeada quando a proteína Rho1p é estimulada por altas temperaturas, altas concentrações de cálcio, limitação de nutrientes e/ou baixa osmolaridade e ativa a Pkc1p que através de fosforilações em cascata promove a ativação dos demais componentes da via: Bck1p (MAPKKK), Mkk1p/Mkk2p (MAPKKs) e Mpk1p (MAPK), resultando na ativação de alguns fatores de transcrição indutores e/ou repressores. Como exemplo, temos a regulação da transcrição do gene
FKS2 que codifica uma proteína do complexo glucano sintase. A Pkc1p pode, ainda, atuar em outras vias de sinalização.
1.2.1 – Metabolismo do fosfatidilinositol
O fosfatidilinositol (PI/PtdIns) é um fosfolipídio encontrado na membrana de células eucarióticas. Muitos de seus metabólitos, embora encontrados em concentrações menores, sofrem rápido “turnover” celular, sugerindo que pequenas alterações na concentração de PI ou de qualquer um dos seus metabólitos podem gerar profundos impactos em processos celulares importantes (Gardocki et al., 2005).
Em leveduras, os fosfolipídios são sintetizados no retículo endoplasmático (Kohlwein et al.1996), através de vias bioquímicas altamente conservadas em eucariotos. Sendo que PI, fosfatidilcolina (PC) e cardiolipina são sintetizados através de CDP-DAG enquanto que PI e PC podem ainda ser sintetizados, respectivamente, via inositol e CDP-colina (Greenberg et al., 1996; Henry et al., 1998; Carman et al., 1996 e 1999; McMillin et al., 2002; Schlame et al., 2000).
Para ativação da biossíntese do fosfatidilinositol via CDP-DAG é necessário que o glicerol seja transportado para o interior da célula através das proteínas Fps1p ou Slt1p, onde é fosforilado a glicerol 3-P pelo produto codificado pelo gene GUT1 (Athenstaedt et al., 1997 e 1999). A utilização do glicerol 3-P pode ser feita ao longo de duas vias bioquímicas que convergem para a síntese do ácido fosfatídico (PA), ou seja, as proteínas Gat1p/Gat2p e Slc1p através de uma série de acilações promovem, respectivamente, a conversão de glicerol 3-P a acil-glicerol 3-P (lyso-PA) e desse a PA, ao passo que Gut2p converge o glicerol 3-P a dihidroxiacetona (DHAP), enquanto que pela ação seqüencial de Gat1p/Gat2p e Ayr1p temos respectivamente, DHAP convertido a acil-dihidroxiacetona-fosfato (acil-DHAP) e a redução de acil-DHAP a lyso-PA (acil-glicerol 3-P). O PA gerado é, então, convertido a diacilglicerol (DAG) ou a CDP-DAG, respectivamente, pela ação das proteínas Dpp1p/Lpp1p e Cds1p (Athenstaedt et al., 2000) (Figura 3).
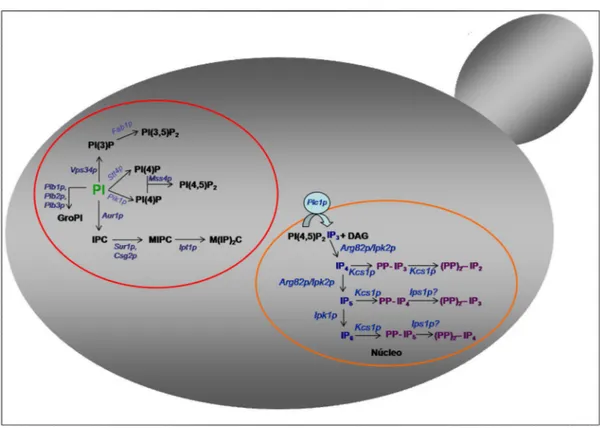
O fosfatidilinositol é usado direta ou indiretamente para sintetizar fosfoinositóis (PIPs), inositóis polifosfatos (IPs) e complexos esfingolipídios (IPCs), enquanto que seu catabolismo gera glicerofosfoinositóis (GroPI). Em leveduras, PI (4)P e PI (4,5)P2 são sintetizados a partir de PI pela ação das enzimas PI 4-quinases (Stt4p e Pik1p) e PI (4)P 5- quinase (Mss4p), respectivamente (Odorizzi et al., 2000; Wera et al., 2001) (Figura 4).
Diferentemente dos eucariotos superiores que possuem uma PI 3-quinase tipo 1 que converte PI(4,5)P2 a PI (3,4,5)P3, células de leveduras não possuem esse tipo de enzima, embora produzam PI(3)P e PI(3,5) P2 a partir de PI, mediado pela ação das enzimas PI 3-quinase (Vps34p) e PI(3)P 5-quinase (Fab1p) (Odorizzi et al., 2000; Wera
et al., 2001) (Figura 4).
Os IPs, inositóis polifosfatos solúveis em água, são considerados moléculas sinalizadoras constituintes da via de sinalização do inositol, que por sua vez, representa um dos maiores mecanismos geradores de respostas celulares às condições do meio externo (Berridge et al.,1984). Tais moléculas são geradas quando PI (4,5)P2 é clivada pela fosfolipase C PI – específica (codificada pelo gene PLC1), produzindo os mensageiros secundários, diacilglicerol (DAG) e IP3 (I(1,4,5)P3) (Shears et al., 2000; York et al., 2001). O IP3, entretanto, é fosforilado pelo produto do gene
ARG82/ARGIII/IPK2 gerando IP4 (I (1,3,4,5)P4 e I (1,4,5,6)P4) e IP5 (I (1,3,4,5,6)P5)
(Odom et al., 2000, Saiardi et al., 1999), que por sua vez, é fosforilado pela enzima Ipk1p a IP6 (I (1,2,3,4,5,6)P6) (Ives et.al., 2000) (Figura 4).
Figura 3 – Biossíntese de fosfolipídios em Saccharomyces cerevisiae
As enzimas relevantes à síntese de fosfolipídios estão indicadas em cor azul. DHA – dihidroxiacetona; DHAP – dihidroxiacetona fosfato; PA – ácido fosfatídico; DAG – diacilglicerol; PC – fosfatidilcolina; PI – fosfatidilinositol.
Figura 4 – Via anabólica/catabólica do fosfatidilinositol em S. cerevisiae
As enzimas relevantes a metabolização dos fosfolipídios estão indicadas em cor azul. PI(3)P, PI(4)P – fosfoinositóis monofosfato; PI (3,5)P2, PI(4,5)P2 – fosfoinositóis bifosfatos; GroPI – glicerofosfoinositóis; IPC, MIPC, M(IP)2C – complexos esfingolipídios; IP3, IP4, IP5, IP6 – inositóis polifosfatos; PP-IP4, PP-IP5, (PP)2-IP2, (PP)2-IP3, (PP)2-IP4 – inositóis pirofosfatos. A fosforilação de IP3, por Arg82p pode gerar tanto o IP4 I (1,4,5,6)P4) quanto o IP4 (I (1,3,4,5)P4), que por sua vez através da ação de Kcs1p, pode ser convertido ao inositol pirofosfato PP-IP3. Além disso, um novo componente que apresenta atividade IPS parece converter PP-IP4 e PP-IP5 a inositóis pirofosfatos mais complexos.
Recentemente, outros trabalhos têm sugerido a participação de novos componentes que contribuem para o aumento da complexidade do metabolismo dos inositóis polifosfatos e pirofosfatos, entre eles cita-se um segundo componente que possui uma atividade inositol difosfato sintase (IPS), assim como a apresentada por Kcs1p, sendo responsável pela geração de inositóis pirofosfatos mais complexos (Seeds
et al., 2005; York et al., 2005) (Figura 4).
1.2.2 – O metabolismo do fosfatidilinositol e a r egulação de processos celular es
Estudos genéticos e bioquímicos da via de sinalização do fosfatidilinositol e de seus metabólitos em leveduras têm evidenciado a sua participação em processos celulares bem distintos: regulação transcricional, remodelamento da cromatina, exportação nuclear do RNAm, manutenção da elongação telomérica, mecanismos de reparo do DNA, tráfego vesicular, proliferação celular, secreção da enzima invertase e regulação de vias de sinalização dependentes de glicose (Kaibuchi et al., 1986; Flick & Thorner, 1993; Brandão et al.,1994; Cocetti et al., 1998; Chi et al., 2004; York, 2006; Trópia et al., 2006).
Em contrapartida, os mecanismos e os receptores envolvidos na execução de tais papéis ainda não foram totalmente elucidados e caracterizados, tornando-se alvos de intensas pesquisas (Bennett et al., 2006). Nesse contexto, citamos o fato de que em células de mamíferos, em resposta a estímulos, a fosfolipase C hidrolisa o PI (3,5)P2 presente na membrana citoplasmática em dois componentes DAG e IP3. Sendo que o primeiro permanece na membrana e serve como mensageiro secundário para ativação da proteína quinase C enquanto que o IP3 também atua como mensageiro secundário para mobilização de Ca++ a partir de estoques intracelulares (Kaibuchi et al., 1986; Wera et
al., 2001). Embora todos esses componentes estejam presentes em células de leveduras, a via na qual eles interagem ainda permanece obscura. Já foi demonstrado que Plc1 p controla via DAG a proteína quinase C de leveduras, no entanto, a possível relação do IP3 no controle dos níveis de Ca++ não está bem estabelecida, já que o genoma de leveduras não apresenta nenhum receptor homólogo aos receptores para IP3 encontrados em mamíferos. Evidências que comprovam essa relação estão limitadas a estudos in
entanto, Silverman-Gavrila & Lew (2002) têm demonstrado a geração e manutenção de um gradiente de Ca++ intracelular em Neurospora crassa, através do seqüestro desse íon de vesículas vacuolares pela ligação de IP3 em um provável receptor na membrana dessa organela. Esse achado sustenta a hipótese de um possível receptor para IP3 em fungos, muito embora nenhum registro da presença do mesmo tenha sido indicado em células de S. cerevisiae.
1.2.2.1 – Regulação da expr essão gênica
A regulação da expressão gênica pelos inositóis é um tema que tem alcançado avanços importantes nas últimas duas décadas. Sendo que a primeira conexão entre a sinalização dependente do metabolismo do fosfatidilinositol e o controle transcricional foi demonstrada por Henry e colaboradores através do estudo do gene INO1 de leveduras, necessário à conversão de glicose 6-P a inositol 1-P, estando de tal forma intimamente relacionado à sobrevivência do microorganismo na ausência de suplementação exôgena de inositol (Hirsch & Henry, 1986; Lopes et al., 1991; Carman & Henry, 1999 ).
Além disso, a relação entre os inositóis polifosfatos e o controle da expressão gênica tem sido estabelecida por meio de estudos genéticos e bioquímicos da proteína Arg82p, primeiramente identificada como um fator de regulação transcricional de leveduras, e posteriormente como uma IP3 quinase ou inositol fosfato multiquinase (Ipmkp). Essas denominações se devem ao fato de que York e colaboradores ao clonarem o gene IPK2 de leveduras, responsável pela conversão de IP3 a IP4 e a IP5, demonstraram que se tratava exatamente do gene ARG82/ARGIII previamente caracterizado (Odom et al., 2000).
Steger et al. (2003) demonstraram que Arg82p e a produção de IP4 e IP5 participam , através do remodelamento da cromatina, do controle da expressão do gene
cromatina Swi/Snfp e Ino80p em induzir a transcrição do gene PHO5. Uma vez que o mutante arg82Δ apresenta altas concentrações de IP3 e plc1Δ dispõe de uma redução desse mesmo metabólito (Saiardi et al., 2000), desconsiderou-se a provável participação desse inositol na resposta fenotípica apresentada por ambos mutantes, sugerindo-se, então, que poderia estar relacionado à baixa produção de IP4 e IP5 (Steger et al., 2003).
Os inositóis pirofosfatos também têm sido relacionados ao controle da expressão gênica de PHO5, mas atuam diferentemente dos inositóis polifosfatos, sendo necessários à manutenção da repressão do gene PHO5 sob condições de altas concentrações de fosfato (El Alami et al., 2003).
O gene ARG82/ARGIII/IPK2 foi primeiramente identificado como um dos reguladores do metabolismo da arginina em S. cerevisiae (Béchet et al., 1970), podendo estar associada ao complexo transcricional ArgR-Mcm1 (Messenguy & Dubois, 2003; Yoon et al., 2004), que possui, além da Arg82p, outros três componentes: Arg80p, Mcm1p e Arg81p, sendo que os dois primeiros são membros da família de fatores transcricionais “MADS-box”, enquanto que a Arg81p, uma proteína “zinc finger”, é considerada como o sensor de arginina (Dubois et al., 2000). El Bakkoury et al. (2000) e Yoon et al. (2004) demonstraram através de ensaios de imunoprecipitação de cromatina (ChIP) que a proteína Arg82p parece estar associada a Mcm1p e é necessária à ativação de regiões transcricionais em resposta a arginina.
Segundo experimentos in vitro realizados por Dubois, Messenguy e colaboradores, a participação da proteína Arg82p no metabolismo de arginina estaria relacionada apenas a estabilização do complexo protéico ArgR-Mcm1p por meio de interações físicas diretas entre Arg82p e Mcm1p ou ainda entre Arg80p, sendo os níveis dessas proteínas significamente reduzidos em mutantes arg82Δ e a reversão fenotípica alcançada através da superexpressão gênica de MCM1 e ARG80 (Dubois & Messenguy, 1994; El Bakkory et al., 2000).
1.2.2.2 – Exportação nuclear do RNA mensageir o (RNAm)
York et al. (1999) com o intuito de investigar quais genes de levedura contribuíam para a exportação nuclear do RNAm, conduziram um rastreamento gênico através do qual observaram que mutações individuais nos genes PLC1, ARG82 ou IPK1 resultaram em uma produção deficiente de IP6 assim como uma ineficiente exportação nuclear de RNAm mediada por Gle1p. Além disso esse mesmo grupo, induzindo a produção de IP6 através da superexpressão de PLC1 em mutantes gle1Δ , detectaram a supressão de certos defeitos na exportação de RNAm característicos de tal mutante.
Em leveduras, Gle1p é uma nucleoporina citoplasmática, componente da subunidade Nup82p do complexo nuclear do poro (NPC), necessária ao transporte de macromoléculas como o RNAm, contribuindo assim para a regulação dos processos de transcrição do DNA no núcleo e síntese protéica citoplasmática (www.yeastgenome.org; Miller et al., 2004).
Com o intuito de averiguar se outras nucleoporinas ou fatores de transportes nucleocitoplasmáticos, assim como Gle1p, são influenciados pela atividade de Ipk1p e pela produção de IP6, Miller et al.(2004) identificaram uma série de genes cujas deleções, associadas ao mutante ipk1Δ , conferiram aos duplos mutantes fenótipos mais severos em relação à exportação nuclear do RNAm do que aqueles já descritos para o mutante ipklΔ . Os genes identificados codificam nucleoporinas localizadas na face citoplasmática dos complexos nucleares do poro (NPCs), sugerindo que o papel de IP6 na exportação do RNAm esteja restrito à face citoplasmática dos NPCs.
1.2.2.3 – Elongação telomér ica e mecanismo de reparo do DNA
Recentemente, alguns trabalhos têm demonstrado uma possível relação entre os inositóis polifosfatos e pirofosfatos e a regulação da elongação telomérica, morte celular e mecanismos de reparo do DNA. (York et al., 2005; Saiardi et al.,2005; Bennett et al., 2006; York, 2006).
da degradação do DNA e uma vez removida promove a ativação dos mecanismos de reparo, o comprometimento do ciclo celular, e a apoptose (Bennett et al., 2006).
Saiardi et al. (2005) demonstraram que em leveduras a proteína Kcs1p controla a atividade de proteínas que desempenham papéis importantes na manutenção dos telômeros. Hanakahi et al. (2000 e 2002) e Ma et al. (2002) por sua vez, sugerem que IP6 seja um regulador da via de reparo do DNA, denominada NHEJ (“non-homologous end joining”), entretanto, é necessário que outros estudos comprovem definitivamente tanto a participação de IP6 quanto dos inositóis pirofosfatos no metabolismo de DNA
1.2.2.4 - Tr áfego vesicular e prolifer ação celular
Os papéis biológicos desempenhados pelo metabolismo do fosfatidilinositol em relação aos eventos do tráfego vesicular incluem a secreção, endocitose e endereçamento protéico (Odorizzi et al., 2000).
Primeiramente, tem sido relatado o envolvimento dos inositóis polifosfatos e pirofosfatos no tráfego vesicular, seja pela ligação de IP6 ao complexo clatrina AP2 envolvido no processo de endocitose na membrana plasmática (Beck et al., 1991; Voglmaier et al., 1992), seja pela apresentação de uma morfologia vacuolar alterada (pequenos vacúolos fragmentados) por parte do mutante kcs1Δ (Saiardi et al., 2000), ou ainda pelo fato de que nesse mesmo mutante os endossomos ao invés de se ligarem ao vacúolo, formam intermediários multilamelares conferindo uma anormalidade no processo de endocitose celular ( Saiardi et al., 2002).
1.2.2.5 - Secr eção da enzima inver tase
A invertase é uma proteína naturalmente secretada em Saccharomyces sp., localizando-se no espaço periplasmático e parede celular dessas leveduras (Chi et al., 2004). Chi et al. (1997) demonstraram que a secreção da invertase em células de
Saccharomyces sp. W4 encontrava-se reprimida quando essas eram submetidas ao
crescimento em meio de cultura sintético contendo concentrações maiores que 0,2% (p/v) de glicose; entretanto, quando crescidas nesse mesmo meio suplementado com inositol, a repressão só era detectada quando as concentrações de glicose ou sacarose atingiam, respectivamente, valores maiores que 2% (p/v) ou de aproximadamente 4% (p/v), sugerindo que de alguma forma, o metabolismo do fosfatidilinositol estaria envolvido no processo de desrepressão da secreção da invertase.
Recentemente resultados obtidos por esse grupo de pesquisadores detectaram através da técnica de RT-PCR que células de leveduras reprimidas por glicose e crescidas em meio sintético suplementado com inositol, apresentavam uma concentração de RNAm codificante da invertase muito maior quando comparadas às células reprimidas por glicose crescidas em meio sintético e ausência de inositol, sugerindo que o metabolismo do fosfatidilinositol estaria envolvido não apenas no processo de secreção da invertase como também na regulação da expressão do gene codificante para essa proteína (Zhang & Chi et al., 2004). Entretanto, o mecanismo através do qual o inositol e o fosfatidilinositol desempenham esse papel precisa ser melhor elucidado.
1.2.2.6 – Regulação de vias de sinalização dependentes de glicose
constitui-se na proteína mais abundante na membrana citoplasmática de fungos e sua atividade é essencial à viabilidade desses organismos (Portillo, 2000; Ambesi et al., 2000), entretanto a via de sinalização responsável pela ativação dessa enzima ainda não foi totalmente elucidada, ainda que muitos componentes já tenham sido identificados assim como a necessidade da fosforilação direta da H+-ATPase (Chang & Slayman, 1991; De la Fuente & Portillo, 2000; Souza et al.,2001).
Inúmeros trabalhos têm sido desenvolvidos no intuito de descobrir a enzima responsável por fosforilar a H+-ATPase. Ulaszewski et al. (1989) sugeriram que a via de sinalização mediada por Pka p (RasAMPc) estaria envolvida no processo de ativação, por glicose, da H+-ATPase, sendo essa a enzima responsável por sua fosforilação. Entretanto, Becker dos Passos et al. (1992), ao trabalhar com mutantes da via RasAMPc, demonstraram que a ativação de H+-ATPase não era dependente dessa via.
Por sua vez, Brandão et al. (1994) ao estudarem o possível envolvimento da via de sinalização do fosfatidilinositol na regulação da H+-ATPase propuseram um mecanismo semelhante àquele da via do fosfatidilinositol de mamíferos, com a provável participação da Pkc1p na ativação induzida por glicose da H+-ATPase de levedura. Esses estudos tiveram como base o fato de que a adição de glicose a células de leveduras estimula também a metabolização do fosfatidilinositol (Kaibuch et al.,1986; Francescotti et al., 1990; Flick & Thorner, 1993). A hipótese de que a ativação da H+ -ATPase estaria ligada ao metabolismo do fosfatidilinositol foi reforçada pelos trabalhos de Coccetti et al. (1998) e Souza et al. (2001), sendo que o primeiro grupo de pesquisadores sugeriu o envolvimento da enzima fosfolipase C, tanto na indução por glicose no turnover do fosfatidilinositol quanto na ativação de H+-ATPase enquanto que o segundo demonstrou a participação das proteínas Gpa2p, do sensor de glicose Snf3p e de Pkc1p nessa via de sinalização.
glicose-6-P, que seria detectado pelo domínio C-terminal do sensor de glicose Snf3 p. Essa proteína, uma vez estimulada promoveria a inibição da Ca++-ATPase presente no vacúolo (Pmc1 p) e conseqüentemente alteraria os níveis de Ca++ citosólico. Em paralelo, e atuando em uma via independente daquela com o envolvimento de Snf3 p, a proteína Gpa2 p responderia a presença do açúcar fosforilado estimulando a ativação da enzima Plc1p. Essa enzima, uma vez ativada, hidrolisaria o PI (3,5)P2 gerando DAG e IP3. O inositol trifosfato por sua vez, atuaria sobre canais de cálcio presentes na membrana citoplasmática e/ou na membrana de organelas intracelulares, o que acarretaria uma elevação dos níveis de cálcio livre no citosol. Essa elevação seria resultado de dois mecanismos: um com o envolvimento de Snf3 p e outro com o envolvimento de Gpa2 p. A presença dos dois mensageiros secundários, DAG e cálcio, promoveria a ativação da proteína quinase C (Pkc1 p), culminando com a ativação da H+-ATPase de membrana citoplasmática por glicose (Trópia et al., 2006) (Figura 5).
A abordagem feita por esse modelo hipotético em relação ao possível envolvimento do inositol trifosfato (IP3) como mensageiro na resposta intracelular de cálcio e posterior ativação da H+-ATPase provém da evidência de que o mutante
arg82∆, que possui altos níveis de IP3, apresentou tanto a acidificação externa quanto a
ativação enzimática da H+-ATPase, ambas induzidas por glicose, mais intensas quando comparado à cepa selvagem correspondente (Trópia et al. 2006).
As cepas de S. cerevisiae envolvidas nos experimentos realizados por Trópia et
al. (2006) eram submetidas ao crescimento tanto em presença de glicose quanto de galactose, uma vez que Tokés-Fuzesi et al.(2002) demonstraram que células de leveduras crescidas em glicose ou galactose apresentam diferentes níveis na resposta da elevação transitória de cálcio citosólico, sendo tal situação associada a distintos níveis intracelulares de glicose 1-P e glicose 6-P gerados a partir dessas fontes de carbono (Aiello et al., 2002).
Figura 5 - Modelo hipotético da ativação, induzida por carboidrato, da H+- ATPase de membrana citoplasmática mediada por íons cálcio, via Pkc1 p.
deficiência em executar a desrepressão de sistemas enzimáticos relacionados ao metabolismo de carbono (Brandão et al., 2002; Salgado et al., 2002; Gomes et
al.,2005).
Devido ao fato dos mutantes pkc1∆ e arg82∆ compartilharem os fenótipos de deficiência de crescimento em fontes alternativas de carbono e a ausência dessas proteínas influenciarem a atividade da H+-ATPase de membrana citoplasmática (Souza
et al., 2001; Trópia et al. 2006) sugere-se que Pkc1p , o metabolismo do
fosfatidilinositol e possivelmente o metabolismo de cálcio estão de alguma maneira relacionados; embora em leveduras essa relação ainda não tenha sido diretamente demonstrada, em mamíferos elas estão intimamente ligadas (Kaibuchi et al., 1986; Wera et al., 2001).
2.Objetivos
2.1.Objetivo Geral:
Estudar a participação de componentes do metabolismo do fosfatidilinositol no controle de vias de sinalização induzidas por glicose em S. cerevisiae.
2.2.Objetivos Específicos:
a. Avaliar o metabolismo de carbono em cepas de S. cerevisiae derivadas de diferentes “backgrounds” genéticos , que contêm deleção em genes que codificam proteínas envolvidas na geração de inositóis fosfatos (PLC1, ARG82,
IPK1 e KCS1);
b. Caracterizar fenotipicamente os mutantes selecionados realizando ensaios de crescimento celular, dosagens do consumo de glicose e produção de etanol;
c. Verificar se a mutação presente nas cepas selecionadas afeta a atividade de sistemas enzimáticos que estão sob o controle de glicose;
3. Mater iais e métodos
3.1 – Cepas de Saccharomyces cerevisiae e construções utilizadas nos exper imentos
Nome Genótipo Or igem
PJ69- 2a
PJ69-4a 1
3700
3703
BY4742
YDR173C
YPL268W
YDR315C
YDR017C
Enzo Martegani, Dipartimento di Biotecnologie e Bioscienze, Università degli Studi di Milano-Bicocca, Italy.
MATa; trp-901; leu2-3; 112 ura3-52; his3-200; gal4Δ gal80Δ ; LYS::GAL1-HIS3;
GAL2-ADE2; met2::GAL7-lacZ
Enzo Martegani, Dipartimento di Biotecnologie e Bioscienze, Università degli Studi di Milano-Bicocca, Italy.
PJ69-2a; arg82::KanMX4
MATα; ura3-52; his3-11,15; trp1-Δ 901; ade2-101
Johan Thevelein, Laboratorium voor Moleculaire Celbiologie, Katholieke Universiteit Leuven, Belgium.
MATa; ura3-52; trp1-Δ 901; ade2-101; plc1 ::URA3
Johan Thevelein, Laboratorium voor Moleculaire Celbiologie, Katholieke Universiteit Leuven, Belgium.
Coleção de cepas Euroscarf.
Coleção de cepas Euroscarf.
BY4742; ipk1:: KanMX4
BY4742; arg82:: KanMX4 Coleção de cepas Euroscarf.
BY4742; plc1:: KanMX4 Coleção de cepas Euroscarf.
MATα ; his3Δ 1; leu2Δ 0; lys2Δ 0; ura3Δ 0
Nome Genótipo Or igem
BY4741
YDR173C
LBCM 465
LBCM 468
LBCM 506
LBCM 512
LBCM 513
3.2 - Plasmídeo e vetor de expr essão utilizados nos experimentos
pBM690: YCp50 GAL1::lacZ (inserido entre os sítios de restrição BamHI e SALI),
cedido por Mark Johnston, Washington University School of Medicine, United States.
pYES2/CT: vetor de expressão comercial (Invitrogen), que possui o gene URA3 como
marcador auxotrófico.
Coleção de cepas Euroscarf.
Coleção de cepas Euroscarf.
Cepa obtida durante o desenvolvimento da dissertação, Universidade Federal de Ouro Preto, Brasil.
Cepa obtida durante o desenvolvimento da dissertação, Universidade Federal de Ouro Preto, Brasil.
PJ69-2a; arg82::KanMX4 plc1::URA3
PJ69-2a + pBM690
PJ69-4a 1 + pBM690 Cepa obtida durante o desenvolvimento da dissertação, Universidade Federal de Ouro Preto, Brasil.
BY4741; arg82:: KanMX4
MATa; his3Δ 1; leu2Δ 0; met15Δ 0; ura3Δ 0
Cepa obtida durante o desenvolvimento da dissertação, Universidade Federal de Ouro Preto, Brasil.
Cepa obtida durante o desenvolvimento da dissertação, Universidade Federal de Ouro Preto, Brasil.
PJ69-2a; arg82::KanMX4 yvc1::URA3
3.3 - Meios de cultura 3.3.1 - Meio YP
O meio líquido YP é composto por extrato de leveduras 1% (p/v) e bactopeptona
2% (p/v). O preparo do meio sólido requer o acréscimo de ágar 1,5% (p/v).
As diferentes fontes de carbono (glicose 2%, galactose 2%, glicerol 3% e etanol 3%) utilizadas nos experimentos foram autoclavadas separadamente e acrescentadas, individualmente, ao meio YP estéril.
3.3.2 - Meio mínimo sintético (SD)
O meio líquido e sólido de SD são compostos por 0,67% (p/v) de base nitrogenada de leveduras sem aminoácidos, suplementado com os aminoácidos fenilalanina 50 mg/L, triptofano 100 mg/L, histidina 100 mg/L, ácido glutâmico 100
mg/L, ácido aspártico 100 mg/L, valina 100 mg/L, arginina 20 mg/L, metionina 20
mg/L, tirosina 30 mg/L, isoleucina 30 mg/L, lisina 30 mg/L, leucina 250 mg/L, treonina 200 mg/L e serina 375 mg/L e as bases adenina 50 mg/L e uracila 50 mg/L. Sendo o meio sólido acrescido de ágar 1,5% (p/v) e o pH acertado com KOH para o valor de 6.5. Em se tratando do preparo do meio SD líquido, o valor de pH foi acertado para 5.5. A glicose, como fonte de carbono, foi adicionada, separadamente, em uma concentração final de 2%.
3.4 – Teste de cr escimento celular em meios de cultura contendo difer entes fontes de carbono
utilizadas para inocular cada meio de cultura sólido acrescido de diferentes fontes de carbono. As placas foram incubadas, em estufa, a 30º C por um período de 48 horas.
3.5. - Cur va de cr escimento 3.5.1 - Prepar o do inóculo
As cepas de leveduras foram inoculadas em 5 mL de meio YP glicose 2% e submetidas ao crescimento a 30º C, por um período de 24 horas, sob agitação constante a 200 rpm (Incubador Rotatório New Brunswick Model G25). Mediu-se, em seguida, o valor da densidade ótica de tal suspensão celular a 600 nm (DU-68 Spectrophotometer-Beckman) e calculou-se qual o volume dessa cultura deveria ser acrescentado a 25 mL de meio de cultura (YP glicose 2%), contendo ou não antimicina na concentraçãode 1mg/mL, para que o valor final da densidade ótica fosse de 0.15. Tal inóculo foi incubado a 30º C., sob agitação constante de 200 rpm (Incubador Rotatório New Brunswick Model G25), por 24 horas.
3.5.2 – Construção da curva de crescimento
Durante um período de 60 horas, coletou-se, a cada 3 horas, alíquotas de 1 mL das amostras de células para a realização da leitura da densidade ótica a 600nm (DU-68 Spectrophotometer-Beckman) e subseqüente delineamento do perfil de crescimento das
cepas de S. cerevisiae selecionadas como alvo de nosso estudo.
A cada ponto correspondente à curva de crescimento foram retiradas alíquotas de 500 µL para o armazenamento em nitrogênio e posterior realização das dosagens do consumo de glicose e produção de etanol.
3.6 – Dosagem da taxa de consumo de glicose
Essa dosagem foi realizada em aparelho bioquímico COBAS-FARA (Roche) e
enzimáticos (Roche). A dosagem da glicose se processa por uma série de reações químicas descritas pelas seguintes equações:
D-Glicose + ATP D-G-6-P + ADP
Em pH 7.6, a enzima hexoquinase (HK) catalisa a fosforilação da D-glicose pelo ATP (adenosina 5`- trifosfato) obtendo-se simultaneamente a formação de ADP (adenosina 5’- bifosfato) e da D-glicose-6-fosfato (G-6-P).
A D-glicose-6-fosfato formada é oxidada pelo NADP (nicotinamida adenina dinucleotídeo fosfato) na presença de glicose - 6- fosfato desidrogenase (G6P-DH) a D-gluconato-6-fosfato com a simultânea redução do NADP a NADPH. O aumento de NADPH é medido pela absorvância a 334, 340 ou 365 nm.
D-G-6-P + NADP D-gluconato-6- fosfato + NADPH+ H+
Uma vez obtido os resultados em termos da absorvância do NADPH a concentração de glicose pode ser determinada a partir da seguinte equação:
c = (V x MV/ ε x d x v x 1000 x 2 ) x ∆A (g/L), onde:
• c – concentração de glicose (g/L);
• V – volume final em militro (mL);
• v - volume utilizado de amostra (mL);
• MW – peso molecular da substância que se deseja dosar (g/mol);
• d – caminho óptico (cm);
• ε – coeficiente de extinção molar segundo o comprimento de onda utilizado
para medida da absorvância do NADPH. HK