Autor (es): Carlos Emanoel Vieira Flores Soares Ano de Defesa: 2015
Curso/Programa: Pró - Reitoria Acadêmica Escola de Saúde Programa de Pós-Graduação Stricto Sensu em Ciências Genômicas e Biotecnologia.
CARLOS EMANOEL VIEIRA FLORES SOARES
CARACTERIZAÇÃO DE LEVEDURAS CONSUMIDORAS DE XILOSE POR ESPECTROMETRIA DE MASSA
MALDI – TOF
Dissertação apresentada ao curso de mestrado em Ciências Genômicas e Biotecnologia da Universidade Católica de Brasília, como requisito parcial para obtenção do Título de Mestre.
Orientadora: Profª. Dra. Beatriz Simas Magalhães.
Co-Orientador: Prof. Dr. João Ricardo Moreira de Almeida.
Ficha elaborada pela Biblioteca Pós-Graduação da UCB
Ficha elaborada pela Biblioteca Pós-Graduação da UCB S676c Soares, Carlos Emanoel Vieira Flores.
Caracterização de leveduras consumidoras de xilose por espectrometria de massa MALDI – TOF. / Carlos Emanoel Vieira Flores Soares – 2015.
84 f.; il.: 30 cm
Dissertação (Mestrado) – Universidade Católica de Brasília, 2015. Orientação: Profa. Dra. Beatriz Simas Magalhães
Coorientação: Prof. Dr. João Ricardo Moreira de Almeida
1. Biotecnologia. 2. Biotyper. 3. Leveduras. 4. Produção de etanol. 5. Xilose. 6. MALDI-TOF MS. I. Magalhães, Beatriz Simas, orient. II. Almeida, João Ricardo Moreira de, coorient. III. Título.
“Eu posso não ter ido para onde eu pretendia ir, mas eu acho que acabei terminando onde eu pretendia estar”.
SOARES, Carlos Emanoel Vieira Flores. Caracterização de Leveduras Consumidoras de Xilose por Espectrometria de Massa MALDI-TOF. 2015. 84 p. Mestrado em Ciências Genômicas e Biotecnologia pela Universidade Católica de Brasília, Brasília - DF, 2015.
As leveduras capazes de fermentar a xilose são importantes para a produção de etanol a partir de lignocelulose. Xilose é o segundo açúcar mais abundante na biomassa e pode explicar mais de 30% de açúcares em alguns materiais lignocelulósicos, como o bagaço de cana de açúcar. Assim, a identificação de novas espécies de leveduras que consomem xilose permite o aumento da produção de etanol sem aumentar a área cultivada. Várias espécies de levedura podem utilizar xilose para o crescimento, mas algumas espécies são capazes de fermentar esse açúcar. O objetivo deste estudo foi desenvolver uma metodologia para discriminar leveduras fermentadoras de xilose a partir de aqueles que consomem xilose usando espectrometria de massa. Para isso, um perfil de base de dados de proteínas de xilose-desgaste e leveduras que fermentam xilose foi construído utilizando uma metodologia de MALDI-TOF. Para criar o banco de dados de perfis de proteínas foram usadas cinco cepas de leveduras de referência, incluindo dois conhecidos que consome xilose Meyerozyma guilliermondii, Candida tenuis, e três que fermentam a
xilose Scheffersomyces stipitis, Spathaspora arborariae e Spathaspora passalidarum,
dez recém-isoladas (leveduras selvagens), nomeado A01 a A10. As proteínas foram extraídas com 70% de ácido fórmico e 100% de acetonitrila (1:1) a partir de leveduras cultivadas em meio sólido contendo xilose ou glicose como fonte de carbono. Os espectros foram adquiridos em equipamentos Autoflex III e adicionado à base de dados do programa Biotyper 3.0 (Bruker Daltonic). O espectrómetro de massa foi capaz de agrupar as leveduras selvagens em dois grupos, um conjunto com M. guiilliermondii e o
outro para mais perto das outras leveduras de referência. A metodologia desenvolvida é vantajosa porque permite que uma primeira seleção rápida de levedura que pode ter potencial biotecnológico para a produção de etanol a partir de xilose.
Yeasts capable of fermenting xylose are important for the production of ethanol from lignocellulose. Xylose is the second most abundant sugar in the biomass and can account to over 30% of sugars in some lignocellulosic materials, such as sugar cane bagasse. Thus, the identification of new species of xylose-consuming yeasts would allow increased ethanol production without increasing cultivated area. Several species of yeast can utilize xylose for growth but few species are able to ferment this sugar. The objective of this study was to develop a methodology to discriminate xylose-fermenting yeasts from xylose-consuming ones using mass spectrometry. For this, a protein database profile of xylose-consuming and xylose-fermenting yeast was constructed employing a MALDI-TOF methodology. To build the protein profiles database were used five reference yeast strains, including two known xylose-consuming Meyerozyma guilliermondii, Candida tenuis, and three xylose-fermenting Scheffersomyces stipitis, Spathaspora arborariae and Spathaspora passalidarum, ten newly isolated (wild) yeasts, named A01 through A10.
Proteins were extracted with 70% formic acid and 100% acetonitrile (1:1) from yeast grown in solid medium containing xylose or glucose as carbon source. Spectra were acquired in equipment Autoflex III and added to the program Biotyper system 3.0 database (Bruker Daltonic). The mass spectrometer was able to group the wild yeasts in two groups, one together with M. guiilliermondii and the other closer to the other
reference yeasts. The methodology developed is advantageous because it allows a fast first selection of yeast that may have biotechnological potential for the production of ethanol from xylose.
ACN: Acetonitrila
ACP: Análise de componente principal
AF: Ácido Fórmico
Da: Dalton
HPLC: High Perfomance Liquid Chromatography/ Cromatografia Líquida de Alta Performace
m/z: Razão massa dividida pela carga
MALDI: Matrix Assited Laser Dissociation Ionization / Ionização por Dissociação a Laser Assistida por Matriz
MS: Mass Spectrum/ Espectro de massa bruto
MSP: Espectro de massa Padrão
pb: Pares de base
PC: Componente Principal
PCR: Polymerase Chain Reaction/ Reação em Cadeia de Polimerase
pH: Potencial Hidrogeniônico.
TFA: Ácido trifluoracético
Figura 1: Fluxograma da transformação da cana de açúcar em etanol... 16 Figura 2: Esquema simplificado do metabolismo de levedura na produção de etanol a partir de xilose... 20 Figura 3: Árvore filogenética das sequencias aplicadas das regiões D1/D2 do DNAr 26S demonstrado a proximidade entre as leveduras S. passalidarum e S. arborariae, com
leveduras que foram isoladas de amostras cuja a fonte de carbono era baseado em xilose... 23 Figura 4: Regiões do RNA ribossômico utilizado na identificação de leveduras. Em destaque a região D1/D2 do gene 26S... 26 Figura 5: Exemplo de um perfil proteico gerado por meio de espectrometria de massa... 27 Figura 6: Exemplo de um perfil de microrganismo identificado pelo programa Biotyper, feita em setembro de 2014... 37 Figura 7: Espectro de massa da levedura S. cerevisiae CAT-1 obtido pelas A) análise direta;
B) após extração proteica da colônia; C) Tabela demonstrando a quantidade de vezes que os íons apareceram em todos os espectros de massa adquiridos do método direto; D) Tabela demonstrando a quantidade de vezes que os íons apareceram em todos os espectros de massa adquiridos no método extração proteica da colônia... 44 Figura 8: Dendrograma de classificação das leveduras controles agrupados separadamente por fonte de carbono. A) Meio sólido YPX; B) Meio sólido YPD... 45 Figura 9: Dendrograma de classificação das leveduras selvagens gerados a partir dos perfis proteicos inseridos no programa Biotyper. A) grupo A de leveduras selvagens; B) grupo B de leveduras selvagens... 48 Figura 10: Dendrograma de classificação das 15 leveduras em estudo, cultivadas em meio sólido YPX, realizados pelo programa Biotyper. A) grupo de leveduras selvagens próximas a levedura M. guilliermondii. B) levedura A06 se agrupa com a levedura S. passalidarum... 49
DNAr 26S das leveduras identificadas pelo Bioscan retiradas do banco de dados do CBS yeast 2011, demonstrando a real classifcação das leveduras selvagens quanto a sua proximidade com as leveduras controles... 54 Figura 14: Fermentação simples das leveduras em estudo crescidas em meio de cultura líquido YNB 1X + xilose 50 g/L durante 48h em estufa a 28°C. A) Consumo de xilose em 48h de crescimento; B) Produção de etanol e xilitol em 48h 56 Figura 15: Espectros de massa brutos adquiridos da levedura controle C. tenuis.
A) Meio sólido YPD; B) Meio sólido YPX... 70 Figura 16: Espectros de massa brutos adquiridos da levedura controle S. stiptis. A)
Meio sólido YPD; B) Meio sólido YPX... 70 Figura 17: Espectros de massa brutos adquiridos da levedura controle S. arborariae. A) Meio sólido YPD; B) Meio sólido YPX... 71
Figura 18: Espectros de massa brutos adquiridos da levedura controle S. passalidarum. A) Meio sólido YPD; B) Meio sólido YPX... 71
Figura 19: Espectros de massa brutos adquiridos da levedura controle M.
guilliermondii. A) Meio sólido YPD; B) Meio sólido YPX... 72
Tabela 1: Capacidade de fermentar xilose em etanol em concentrações mínimas da S. stiptis.... 22
1. INTRODUÇÃO... 15
2. REVISÃO BIBLIOGRÁFCA... 16
2.1ENERGIAS RENOVÁVEIS: PRODUÇÃO DE BIOETANOL... 16
2.2 FERMENTAÇÃO INDUSTRIAL E LEVEDURAS SACCHAROMYCES CEREVISIAE...... 17
2.3 XILOSE COMO SUBSTRATO PARA PRODUÇÃO DE ETANOL... 19
2.4LEVEDURAS COM POTENCIAL BIOTECNOLÓGICO: CONSUMIDORAS DE XILOSE VS. FERMENTADORAS DE XILOSE... 21
2.5 CARACTERIZAÇÃO E IDENTIFICAÇÃO DE LEVEDURAS... 24
2.5.1 Conceitos gerais: classificação e definição de leveduras... 24
2.5.2 Identificações por morfologia e fisiologia... 25
2.5.3 Identificações por sequenciamento de gene DNAr 26S... 25
2.5.4 Identificações por espectrometria de massa... 26
3. JUSTIFICATIVA... 30
4. HIPÓTESE... 31
5. OBJETIVOS... 32
5.1 OBJETIVOS GERAIS... 32
5.2 OBJETIVOS ESPECÍFICOS... 32
6. METODOLOGIA... 33
6.1 ISOLAMENTO DE LEVEDURAS SELVAGENS... 33
6.2 LEVEDURAS CONTROLES... 33
6.3 CULTIVO DE LEVEDURAS... 33
6.4 PREPARAO DA MATRIZ ÁCIDO-ALFA-CIANO-4-HIDROXICINÂMICO... 34
6.5 MÉTODOS DE DETECÇÃO DE PROTEÍNAS... 34
6.5.1 Método direto... 34
6.5.2 Método extração proteica da colônia... 34
6.6 AQUISIÇÕES DOS DADOS BRUTOS PELO PROGRAMA FLEX CONTROL... 35
6.7 ANÁLISES DOS DADOS DE ESPECTROMETRIA DE MASSA PELO PROGRAMA BIOTYPER... 35
6.8 ANÁLISE POR HPLC DA PRODUÇÃO DE PRODUTOS GERADOS PELA
FERMENTAÇÃO DE XILOSE... 38
6.9 SEQUENCIAMENTOS DAS REGIÕES D1/D2 DO DNAR 26S DAS LEVEDURAS SELVAGENS... 39
6.9.1 Reações de PCR... 39
6.9.2 Análises de DNA por eletroforese... 40
6.9.3 Purificações de DNA... 40
6.9.4 Sequenciamentos de DNA... 40
6.9.5 Análises filogenéticas... 41
6.10 IDENTIFICAÇÕES FISIOLÓGICAS... 44
7. RESULTADOS... 43
7.1 OTIMIZAÇÃO DO PROTOCOLO DE DETECÇÃO PROTEICA DAS CEPAS DE LEVEDURAS... 43
7.2 CRIAÇÃO DOS BANCOS DE DADOS DE PADRÃO... 45
7.3 CRIAÇÃO DOS BANCOS DE DADOS DE PERFIS PROTEICOS LEVEDURAS SELVANGES... 47
7.4 ANÁLISES DOS SEQUENCIAMENTOS DO GENE DNAR 26S... 50
7.5 CARACTERIZAÇÃO FISIOLÓGICAS... 52
7.6 LEVEDURAS SELVAGENS: CAPACIDADE DE FERMENTAÇÃO... 55
8. DISCUSSÃO... 57
9. CONLUSÃO... 61
10. REFERÊNCIAS... 62
APÊNDICE A – ESPECTROS DE MASSA BRUTO DAS LEVEDURAS CONTROLES... 70
APÊNDICE B – ESPECTROS DE MASSA BRUTO DAS LEVEDURAS SELVANGENS... 73
APÊNDICE C – DENDROGRMAS DE CLASSIFICAÇÃO INDIVIDUAL DAS LEVEDURAS SELVANGE... 76
1. INTRODUÇÃO
Leveduras são utilizadas para a produção de alimentos e bebidas alcoólicas há milhares de anos. Além de seu uso no setor alimentício estes organismos também tem o seu potencial aproveitado no setor energético, já que leveduras são utilizadas na produção industrial de álcool combustível durante a fermentação de açúcares.
Devido à crescente preocupação com a escassez do petróleo em âmbito mundial, há um aumento pela busca por fontes de energias renováveis que possam ser utilizadas de forma sustentável.
O etanol é uma das melhores opções quanto ao uso de novas energias renováveis, além de ser um ótimo substituto do petróleo por ser uma energia limpa e renovável, produzida a partir da biomassa vegetal. Seu uso ameniza os problemas ambientais em escala global como, por exemplo, diminuir a dependência pelo combustível fóssil e reduzir a poluição causada por ele e seus derivados (AZEVEDO; SANTOS; MAGALHÃES, 2012).
A produção de biocombustível proveniente da biomassa vegetal tem como princípio utilizar substratos ricos em celulose e não exercer competição com o setor alimentício, dessa forma abre uma perspectiva ambiental e econômica sustentável para o setor industrial responsável pela produção de biocombustíveis (WOHLBACH et al.,
2011).
No Brasil, por exemplo, cerca de 27 bilhões de litros de etanol foram produzidos em 2013, por meio da fermentação alcoólica utilizando a levedura Saccharomyces cerevisiae (BRASIL, 2014). Neste aspecto o setor industrial investe em estudos de
microrganismos que possuam potencial biotecnológico para a obtenção de energias a partir da biomassa vegetal (GOLDEMBERG, 2009).
O processo de identificação de espécies que possam ser utilizadas em larga escala na indústria é demorado além de requerer custo econômico alto. Sendo assim este trabalho visa propor o uso de uma nova metodologia que possa realizar uma seleção rápida de leveduras selvagens (leveduras de ambiente naturais que ainda não foram classificadas taxonomicamente) por meio da espectrometria de massas.
2. REVISÃO BIBLIOGRÁFICA
2.1 ENERGIAS RENOVÁVEIS: PRODUÇÃO DE BIOETANOL
O Brasil é o segundo maior produtor de etanol no mundo, produzindo 27, 2 bilhões de litros de etanol estando atrás dos EUA que produziu 50,4 bilhões de litros de etanol em 2013. O etanol do Brasil é produzido a partir da cana de açúcar enquanto que o etanol produzido nos EUA é proveniente do milho, uma matéria prima menos eficiente do que a cana de açúcar (CARVALHOet al., 2011; GOLDEMBERG; LUCON, 2007; BRASIL,
2014).
É possível afirmar que o Brasil possui vantagens para a produção de etanol, devido a fatores geográficos e climáticos que facilitam o cultivo da cana de açúcar, sendo um dos poucos países em que parte do combustível consumido é renovável (HOLFMANN, 2006; GOLDEMBERG; MOREIRA, 2005).
O etanol é produzido por uma série de etapas industriais sendo elas: a extração a partir da sacarose da moagem da cana de açúcar, preparo do caldo de cana tratado que recebe o nome de mosto e o processo de destilação, como mostra figura 1 (REIS, 2011; DAM et al., 2008).
Figura 1: Fluxograma da transformação da cana de açúcar em etanol.
Existem diferentes tipos de fermentações classificados em contínuos ou descontínuos que podem ou não reutilizar a mesma levedura em diferentes processos de fermentação. Atualmente, cerca de 70% das destilarias brasileiras utilizam o sistema de batelada alimentada (BRASIL, 2014; REIS, 2011; COSTA, 2014; FIGUEREIDO, 2008). O sistema em batelada Melle-Boinot (batelada alimentada) é o sistema mais comum de fermentação utilizado nas destilarias brasileiras, uma vez que este sistema consiste em realizar fermentações com altas concentrações de células e reciclagem de leveduras (FIGUEREIDO, 2008).
A fermentação em batelada alimentada ocorre com o mosto alimentado de forma contínua com uma vazão definida, de forma a diminuir o impacto da inibição das leveduras por concentrações altas de açúcares no substrato. Não há remoção de porções antes do final da fermentação, desta forma a fermentação do mosto ocorre durante a alimentação da dorna e continua mesmo após o seu enchimento (REIS, 2011).
O termino desta fermentação ocorre quando o teor de açúcar fermentável se torna desprezível. Após a fermentação o mosto fermentado, é separado do levedo por centrifugação e levado às colunas de destilação enquanto o leite de levedura (células em suspensão altamente concentradas) é tratado com pH ácido entre 2 a 3 em seguida é armazenado (REIS, 2011; COSTA, 2014; JÚNIOR, 2012).
Para que se possa produzir de forma eficiente e em larga escala o etanol a ser comercializado, é indispensável a participação de leveduras capazes de realizar o processo de fermentação alcoólica, sendo assim a fermentação industrial conta diretamente com o uso de leveduras da espécie Saccharomyces cerevisiae.
2.2FERMENTAÇÃO INDUSTRIAL E LEVEDURAS SACCHARMYCES CERVISIAE
O estudo de leveduras para o uso industrial tem como objetivo caracterizar novas espécies encontradas em ambientes naturais e é de importância tanto para a ciência básica como para aplicações industriais.
As principais leveduras estudadas são aquelas que possuem potencial biotecnológico a serem aplicadas em conjunto com a biomassa cultivada, uma vez que a mesma contém altas concentrações de carboidratos como a sacarose, glicose e a xilose que são a matéria prima para a produção de etanol.
pelas indústrias brasileiras por apresentarem um excelente potencial biotecnológico para a produção de etanol (HOWELL et al., 2004; JOHNSON, 2013).
A S. cerevisiae é a melhor opção para a produção de etanol em larga escala nas
destilarias brasileiras, devido à sua eficiência na prática da fermentação e produção do etanol proveniente do caldo da cana de açúcar tratado e também por ser altamente adaptada às condições do ambiente industrial (BASSO et al., 2008).
Esta espécie de levedura apresenta características importantes como, por exemplo, rápido crescimento em forma de pseudo-hifas, capacidade de produzir etanol, suportar variações de temperatura. É tolerante a altas concentrações de açúcar e sobrevive a níveis baixos de oxigênio, o que possibilita o seu uso no setor industrial de forma eficiente, maximizando a produção de etanol (REIS, 2011).
Em busca de aperfeiçoar o processo de fermentação e conseguir um aumento significativo na produção de etanol, foram isoladas diferentes cepas de S. cerevisiae,
adaptadas ao ambiente industrial como, por exemplo, as cepas CAT-1 (Fermentec-ESALQ) e PE-2 (Fermentc-(Fermentec-ESALQ). Estas foram isoladas das usinas de Catanduva e Usina da Pedra, por meio de técnicas de cariotipagem de processos fermentativos para a produção de etanol (ANTONANGELO, 2012; REIS, 2011; BASSO et al., 2008).
Um dos fatores que atrapalha a eficiência completa da S. cerevisiae no processo
de fermentação é a ocorrência de contaminação por bactérias e por espécies selvagens de leveduras Saccharomyces e não – Saccharomyces (SILVA-FILHO et al., 2005). Estes
contaminantes são capazes de competir com as cepas de S. cerevisiae e acabam por
prejudicar todo o processo de produção de etanol, provocando assim a formação de espuma e a diminuição da quantidade e qualidade de etanol desejada (HOWELLet al.,
2004).
A contaminação que ocorre durante o processo de fermentação se dá devido a dois principais fatores: 1º O processo de fermentação não é estéril o que facilita a contaminação por microrganismos que estejam presentes na água utilizada para a lavagem dos equipamentos (REIS, 2011; COSTA, 2014); 2º A reciclagem de leveduras utilizadas durante o processo de fermentação, provocando a reciclagem dos contaminantes (LUDWING; OLIVEIRTA-NETO; ANGELIS, 2001).
Outro fator que atrapalha a eficiência completa da S. cerevisiae é o fato de que ela
O etanol de primeira geração é produzido a partir de carboidratos e óleos de uma região específica, geralmente comestível, de plantas tradicionais como o milho e a cana de açúcar e que são acessíveis ao metabolismo de leveduras S. cerevisiae. O etanol de
segunda geração é produzido a partir de matérias primas celulósicas não alimentares como a xilose, encontrada em resíduos agroindustriais como o bagaço da cana de açúcar e não são metabolizados pelas S. cervisiae (MACHADO, 2013).
Neste aspecto, novas espécies de leveduras selvagens e espécies de leveduras recombinantes da S. cerevisiae, estão sendo estudadas para que se possa obter ter um
aproveitamento total da cana de açúcar na produção de etanol de segunda geração tendo a xilose como matéria prima (GRANGEet al., 2001).
2.3 XILOSE COMO SUBSTRATO PARA PRODUÇÃO DE ETANOL
A biomassa vegetal obtida da cana de açúcar dá origem ao caldo de cana tratado que é rico em carboidratos do tipo sacarose e glicose, utilizados no processo de produção de etanol (WOHLBACHet al., 2011).
O bagaço da cana de açúcar é altamente rico em celulose e hemicelulose onde é encontrada em grande quantidade a xilose considerada o segundo carboidrato mais abundante no bagaço. No entanto a xilose não é aproveitada na fermentação alcoólica já que as leveduras nativas de Saccharomyces não possuem a capacidade de metabolizáç-la
(KÖTTER; CIRIACY, 1993). A levedura Scheffersomyces stiptis é utilizada como
modelo para realizar experimentos de engenharia metabólica devido à eficiência das enzimas presentes em seu metabolismo capazes de fermentar a xilose e produzir etanol em maior quantidade (LEE et al., 2000; MACHADO, 2013).
O metabolismo de xilose em leveduras ocorre pela ação da enzima xilose redutase, que reduz a molécula de xilose em reação dependente de um cofator NADPH em xilitol, o qual é oxidado a xilulose pela enzima xilitol desidrogenase com auxílio do cofator NAD+. A xilulose por sua vez é metabolizada pela via das pentoses fosfato (PPP) e finalmente convertida a etanol, como demonstrado na figura 2 (BIER et al., 2007;
Figura 2: Esquema simplificado do metabolismo de levedura na produção de etanol a partir de xilose.
Fonte: Adaptado de WOHLBACH et al., 2011.
As leveduras Saccharomyces podem ser manipuladas geneticamente por meio de
engenharia genética, inserindo os genes responsáveis pela conversão do açúcar a xilulose. Para as etapas de engenharia, pode ser utilizada a via oxi/redutiva, composta pelas enzimas xilose redutase e xilitol desidrogenase, por exemplo, da levedura
Scheffersomyces stipitis, que as tornem aptas a consumir xilose (KÖTTER; CIRIACY,
1993; GRANGE et al., 2011). Entretanto é um método demorado e de custo alto o que
dificulta a utilização eficiente de leveduras recombinantes na produção de etanol de segunda geração.
2.4 LEVEDURAS COM POTENCIAL BIOLTECNOLÓGICO: CONSUMIDORAS DE XILOSE X FERMENTADORAS DE XILOSE
As leveduras têm como fonte de energia principal os carboidratos que podem ser metabolizados pela respiração celular quando na presença de oxigênio, onde o produto final do metabolismo é gás carbônico e água. Outra forma é pela fermentação, que ocorre na ausência de oxigênio e o produto final é gás carbônico e etanol. Durante o processo de fermentação industrial as leveduras S. cerevisiae metabolizam a glicose e a sacarose
presente no mosto diretamente a etanol (SILVA-FILHO, 2003).
No entanto apesar dessa levedura ser capaz de realizar o processo de fermentação alcoólica, esta levedura não é capaz de utilizar a xilose como fonte de carbono para realizar seus processos bioquímicos, incluindo a fermentação (FARWICK et al., 2014). Desta forma parte do etanol que poderia ser produzido a partir do bagaço da cana de açúcar que é rico em xilose acaba não sendo aproveitado.
Em busca de se obter o etanol de segunda geração tendo substratos ricos em xilose como matéria prima é necessária a identificação de espécies que sejam naturalmente capazes de realizar o processo de fermentação alcoólica a partir da xilose.
Sendo assim é preciso diferenciar dentre as leveduras consumidoras de xilose aquelas que são naturalmente capazes de fermentar xilose em etanol, já que toda levedura fermentadora de xilose também é uma consumidora, mas nem toda levedura consumidora de xilose é uma fermentadora (VALENTE et al., 2012).
Em geral leveduras consumidoras de xilose produzem mais xilitol do que etanol uma vez que requerem altas concentrações de oxigênio para o crescimento celular, produzido uma quantidade menor de etanol durante a assimilação da xilose (FARWICK et al., 2014; VALENTE et al., 2012; BIER et al., 2007). Já leveduras fermentadoras de xilose possuem em seu metabolismo a capacidade de reduzir a quantidade de xilitol produzido maximizando a produção de etanol tanto em condições aeróbicas quanto em condições anaeróbicas (LEE et al., 200; FARWICK et al., 2014; WHOLBACH et al., 2011; GARCIA, 2012).
2007, KURTZMAN; SUZUKIM 2010; LEE et al., 2000; GARCIA, 2012; REN et al., 2014; CADETE et al., 2009; NGUHEN et al., 2006).
A levedura S. stipitis é uma levedura aeróbica do filo Ascomiceta, antigamente
classificada como Pichia stipitis, hoje pertence ao gênero scheffersomyces, isolada de
amostras de madeira é capaz de crescer utilizando diferentes fontes de carbono dentre eles, a celobiose. Se destaca de outras leveduras fermentadoras de xilose como a
Pachysolen tannophilus por produzir menos xilitol embora ambas utilizem a enzima
xilose redutase (GARCIA, 2012).
É capaz de produzir etanol em concentrações mínimas de xilose como demonstrado na tabela 1. Entretanto ela requer condições micro aeróbicas bem controladas no processo de fermentação o que torna inviável o seu uso em larga escala na indústria (REN et al., 2014; MACHADO, 2013; LEE et al., 2000).
Tabela 1: Capacidade de fermentar xilose em etanol em concentrações mínimas da S. stiptis.
Fonte: Adaptado de LEE et al., 2000.
A levedura S. passalidarum é uma levedura do filo Ascomiceta do gênero Spathaspora. Foi isolada de uma amostra de madeira, esta levedura não só abrange uma
nova espécie como também um novo gênero. É uma levedura naturalmente consumidora e fermentadora de xilose que possui semelhanças tanto na fisiologia quanto no habitat e metabolismo com a S. stiptis. Esta levedura possui proximidade taxonômica com outras
leveduras consumidoras de xilose como a Candida Jeffriesii e Candida lyxospphila
conforme semelhanças encontradas em seus genes (NGUYEN et al., 2006).
A levedura S. arborariae é uma levedura do filo Ascomiceta, isolada de uma
amostra de madeira em decomposição coletada no Parque Nacional da Serra do Cipó, localizada em Minas Gerais – Brasil. Esta levedura pertence ao gênero Spathaspora,
assim como a S. passalidarum, no entanto ela está filogeneticamente mais próxima da
levedura C. jeffriessi (CADETE et al., 2009).
Concentração de xilose (g/L) 30 50 90 130 170
Etanol produzido (g/g) 0,37 0,36 0,33 0,29 0,26
Xilitol produzido (g/L) ND* 0,81 4,8 11,8 5,7
A S. arborariae é capaz de consumir e fermentar xilose em condições aeróbicas.
Esta espécie apresenta características semelhantes à espécie S. passalidarum, pois ambas
são sensíveis a inibidores que estão presentes nos hidrolisados de lignocelulose como o furfural (CADETEet al., 2009; NGUYEN et al., 2006).
Estudos recentes baseados no sequenciamento de DNAr 26S de leveduras encontradas em amostras de madeira, demonstrou que esta levedura juntamente com a S. passalidarum forma um grupo de leveduras naturalmente consumidoras de xilose que
esta geneticamente relacionadas com as espécies C. jeffriessi, C. lyxosopphila, C. materiae, C. insectamans, como demonstrado na figura 3 (NGUYEN et al., 2006;
CADETE et al., 2009).
Figura 3: Árvore filogenética das sequências aplicadas das regiões D1/D2 do DNAr 26S demonstrando a proximidade entre as leveduras S. passalidarum e S. arborariae, com
leveduras que foram isoladas de amostras cuja fonte de carbono era baseada em xilose.
Fonte: CADETE et al.,2009.
A levedura C. tenuis é uma levedura do filo Ascomiceta do gênero Candida que
faz parte do grupo de leveduras que possuem a capacidade de consumir e fermentar xilose, no entanto sua capacidade de fermentar a xilose não é comparável com a S. stiptis e a S. passalidarum (WOLBACH et al., 2011). Seu genoma é semelhante ao das leveduras do
gênero Spathaspora e Scheffersomyces, uma vez que possui a enzima xilose redutase que
A levedura M. guilliermondiii é uma do filo Ascomiceta, previamente era
classificada como C. guilliermondii por apresentar semelhanças quanto ao seu
crescimento e capacidade de metabolizar diversas fontes de carbono (BIER et al., 2007). O genoma da M. guilliermondii quando comparado com as demais espécies de Candida
apresenta diferenças significativas quanto ao número de pares de base (pb). O seu genoma apresentava mais proximidade com os genomas das espécies de Pichia o que mudou a
sua classificação para Pichia guilliermondii (KURTZMAN; SUZKI, 2010).
Recentemente foi proposta uma nova classificação para esta levedura e atualmente ela está incluída no gênero Meyerozyma recebendo a nomenclatura de M. guilliermondiii
(MAGUIREet al., 2013; WOLBACH et al., 2011; KURTZMAN; SUZUKI, 2010). A M. guilliermondii é capaz de consumir e fermentar xilose e pode ser encontrada em diversas
amostras ambientais (BIER et al., 2007).
As leveduras, S. stiptis, C. tenuis, M. guilliermondii, S. arborariae e S. passalidarum foram selecionadas para este trabalho devido ao fato de todas serem
leveduras que possuem capacidade de consumo de xilose. Dentre elas as leveduras S. stiptis e S. passalidarum são ótimas referências de leveduras naturalmente fermentadoras
de xilose.
2.5 CARACETERIZAÇÃO E IDENTIFICAÇÃO DE LEVEDURAS
2.5.1 Conceitos gerais: classificação e definição de leveduras
Leveduras são fungos unicelulares não filamentosos, seu aspecto morfológico é esférico ou oval e se reproduzem em geral por brotamento ou por cissiparidade, algumas espécies podem realizar o processo de fermentação alcoólica (KURTZMAN; FELL; BOEKHOUT, 2011).
As leveduras são classificadas como Ascomycota e Basidiomycota dentro da classificação geral do Reino Fungi uma vez que as espécies de leveduras apresentam a formação de ascósporo (ascomicetos) e basidiósporos (basidiomicetos), possuindo uma ampla diversidade genética tendo mais de 800 espécies descritas dentro da classe Saccharomycetes (KOUFOPANOU et al., 2013; JOHNSON, 2013).
Recentemente foi proposto por Kurtzman & Suzuki (2010) uma nova classificação para incluir novos gêneros de leveduras dentro dos ascomicetos com base em análises moleculares a partir da combinação de sequencias da região D1/D2 da subunidade maior do ribossomo 28S, dentre eles os gêneros Scheffersomyces, Babjeviella, Meyerozyma, Millerozyma e Priceomyces, que atualmente já são aceitos.
2.5.2 Identificações por morfologia e fisiologia
As leveduras são identificadas por um sistema clássico de identificação, constituído de características fisiológicas e morfológicas. No entanto muitas vezes não é possível diferenciar algumas espécies de leveduras tais como leveduras do gênero
Saccharomyces que são morfologicamente idênticas (STROPPA et al., 2003). Sendo
assim a identificação de leveduras ocorre principalmente por critérios genéticos e fisiológicos como metabolismo de açúcares (CARVALHO; BENTO; SILVA; 2006).
A identificação das leveduras por morfologia conta com o desenvolvimento de novos meios de culturas que atuam de maneira seletiva permitindo diferenciar e selecionar as leveduras que apresentam maior eficiência no consumo de uma fonte de carbono especifica (BASSOet al., 2008).
2.5.3 Identificações por sequenciamento de gene DNAr 26S
Entre as muitas técnicas de identificação a mais utilizada atualmente é a técnica do sequenciamento do gene de RNAr 16S para a identificação de bactérias e o sequenciamento do gene que codifica para o DNA ribossômico 28S para a identificação de leveduras (DWORKIN, 2006; ATIKINS; CLARK, 2004; MEYER et al., 2010).
O DNA ribossômico de fungos e leveduras é composto pelas seguintes estruturas: uma subunidade menor 18S, o espaço interno transcrito 1 (ITS1), a subunidade do gene 5.8S, o espaço interno transcrito 2 (ITS2) o gene 26S onde se encontra a região D1/D2 e uma subunidade maior 28S (ATIKINS; CLARK, 2004).
ROBNETT, 1998; COSTA, 2014; KURTZMAN; SUZIKI, 2010; COSTA 2014; FELL et al., 2000),
A identificação das leveduras por ferramentas moleculares como o método de SANGER requer uma amplificação do DNA por PCR (Polymerase chain reaction/ Reação em cadeia da polimerase) e comparar com banco de dados existentes para identificar uma nova espécie (AZEVEDO; SEVERINO; MAGALHÃES, 2004).
Uma alternativa recente para a classificação de microrganismos e que tem sido aplicada para o estudo de bactérias e leveduras desde a década de 90 é o uso da técnica de espectrometria de massa MALDI – TOF (CLAYDON et al., 1996).
Figura 4: Regiões do DNA ribossômico utilizado na identificação de leveduras. Em destaque a região D1/D2 do gene 26S.
Fonte: Imagem elaborada pelo autor.
2.5.4 Identificações por espectrometria de massa
Figura 5: Exemplo de um perfil proteico gerado por meio de espectrometria de massa.
Fonte: Imagem elaborada pelo autor.
Em um espectro de massa de qualidade pode ser observado um valor alto da razão sinal/ ruído dos íons, menor intensidade de laser no momento da aquisição dos espectros, pois diminui a possibilidade de saturação de sinal e uma maior quantidade de íons detectados de forma reprodutível, garantindo assim um espectro de qualidade (AGUSTINI et al., 20114; SOARES, 2013, BIZZINI et al., 2011).
Esta técnica foi aplicada inicialmente na identificação e caracterização de bactérias patogênica Gram negativas e positivas, (DEMIREV et al., 1999). A
identificação de leveduras pela espectrometria de massa tem a mesma eficiência e aplicabilidade para bactérias (MAIER; SCHAWARZ; KOSTRZEWA, 2008).
separados conforme a divisão taxonômica dos três domínios Bacteria, Eucaria e Archea
(DEMERIV; FELDMAN; LIN, 2004; MAIER; SCHAWARZ; KOSTRZEWA, 2008; RETTINGER et al., 2012, AGUSTINI, 2014).
A técnica de espectrometria de massa permite a análise das amostras de maneira rápida tanto da amostra sólida na forma de colônia intacta, quanto na forma líquida, ou seja, as proteínas extraídas da colônia em suspensão (NAGY et al., 2009). Atualmente os trabalhos realizados com aquisição de perfis proteicos por espectrometria de massa são realizados com protocolos pré-estabelecidos pelos fornecedores dos equipamentos.
Os bancos de dados responsáveis pelas identificações contam com um sistema estatístico que identifica o organismo em estudo por meio de comparação entre um perfil proteico bruto com os perfis proteicos existentes nos bancos de dados, gerando valores de semelhança (MAIER; SCHAWARZ; KOSTRZEWA, 2008; DEMERIV; FELDMAN; LIN; 2004; AUGUSTINIet al., 2014; SANTOSet al., 2011).
Os bancos de dados existentes para a identificação de microrganismos por espectrometria de massa são desenvolvidos basicamente pelas empresas que desenvolvem os equipamentos necessários. Os bancos de dados principais são a da SARAMISTM que pertence a empresa Shimadzu (SANTOS et al., 2011) e o do BIOTYPER que pertence a empresa Bruker Daltonics (SAUER et al., 2008; THEEL et
al., 2012).
Os bancos de dados de leveduras tiveram início devido ao interesse da área médica em se obter de forma rápida a identificação de leveduras patogênicas do gênero Candida
(SANTOS et al., 2011).
A partir deste início os bancos de dados de perfis proteicos vêm sendo constantemente atualizados com novos perfis de outras espécies de leveduras que não tenham só importância para a área médica, mas também apresentam importância para a área de pesquisa agropecuária, na identificação de diferentes cepas de S. cerevisiae
(BLÄTELL et al., 2013; QUIANet al., 2008).
Desta forma é possível obter a identificação de espécies de leveduras que são encontradas em plantas ou que vivem em simbiose com insetos como, por exemplo, a espécie de levedura S. stipitis, Pichia guilliermondii e leveduras do gênero Saccharomyces (AGUSTINI et al., 2014; SANTOSet al., 2011; THEEL et al., 2012).
Entretanto o seu banco de dados não é rico em informações de microrganismos ambientais, apesar disso o programa Biotyper permite aos usuários enriquecer o seu banco de dados com novos perfis proteicos, permitindo assim a construção de uma biblioteca de perfis proteicos de microrganismos ambientais dentro do banco de dados do Biotyper (AUGISTINI et al., 2014; SOARES, 2013).
Uma forma de utilizar a espectrometria de massa é no controle de qualidade de contaminação de diferentes amostras e no monitoramento de uma determinada levedura, como, por exemplo, em um trabalho realizado por Quian et al., (2008). Neste trabalho foi aplicado um monitoramento da levedura patogênica C. albicans durante quatro dias em
diferentes faixas de temperatura. Foram observados a mudança nos perfis proteicos da C. albicans, que indicava se havia ou não a contaminação de outra levedura, sendo possível
3. JUSTIFICATIVA
O interesse do presente projeto é de aplicar a espectrometria de massa MALDI – TOF para diferenciar leveduras que consomem xilose das que fermentam xilose de forma rápida e dessa forma fazer uma triagem de um banco de leveduras para as que apresentam assim potencial biotecnológico para a produção de biocombustíveis.
Além disso, a espectrometria de massa pode ser utilizada para a construção de uma biblioteca de espectros de massa para a rápida identificação de outros grupos de microrganismos que tenham algum tipo de importância industrial e como controle de qualidade, no cultivo de leveduras consumidoras de xilose.
4. HIPÓTESE
A técnica de espectrometria de massa MALDI – TOF permite diferenciar e caracterizar leveduras consumidoras e leveduras fermentadoras de xilose.
5. OBJETIVOS
5.1 OBJETIVO GERAL
Diferenciar e classificar leveduras consumidoras de xilose, por meio da utilização de espectrometria de massa MALDI – TOF e validar esta metodologia por outros métodos de classificação.
5.2 OBJETIVOS ESPECÍFICOS
1- Gerar perfis proteicos característicos das leveduras crescidas em meio contendo xilose.
2- Classificar as linhagens de leveduras selvagens consumidoras de xilose das espécies controles fermentadores de xilose.
3- Criar um banco de dados de espectros de massa característicos para as leveduras selvagens de forma a enriquecer o banco de dados do programa Biotyper.
4- Obter um perfil fisiológico das leveduras em estudo quanto a sua capacidade de produzir etanol a partir de xilose.
6. METODOLOGIA
6.1 ISOLAMENTOS DE LEVEDURAS SELVAGENS
As leveduras selvagens utilizadas neste estudo são oriundas da coleção de trabalho de microrganismos da EMBRAPA Agroenergia e foram isoladas de amostras de madeira em decomposição. As leveduras foram cultivadas em meio de cultura solido, preparado em laboratório, Yeast Peptone Dextose/ Levedura Peptonada Dextrose (YPD), utilizando os reagentes, extrato de levedura puríssima 2 %, peptona bacteriológico 2% e a fonte de carbono glicose 20 g/L e foram nomeadas A01, A02, A03, A04, A05, A06, A07, A08, A09 e A10.
6.2 LEVEDURAS CONTROLES
Foram utilizados os seguintes isolados como controle em todo o experimento, a espécie S. cervisiae CAT-1 Y-904(BASSO et al., 2008), para padronização do protocolo
de extração de proteínas e as espécies M. guilliermondii Y-324, C. tenuis Y-1498, S. stiptis Y-7124, S. passalidarum Y-27907 e S. arborariae Y-48658, (CADETE et al.,
2009; NYUGEN et al., 2006; BIER et al., 2007; WOLBACH et al., 2011; RAN et al., 2014; MACHADO et al., 2013).
As leveduras controles são oriundas da coleção de trabalho de microrganismos da EMBRAPA Agroenergia. Estes controles são sabidamente consumidores e fermentadores de xilose. Foram utilizados como referência para a criação do banco de dados de perfil proteicos no programa Biotyper e como referência para comparação com as leveduras selvagens.
6.3 CULTIVOS DE LEVEDURAS
O cultivo em meio sólido teve como material biológico inicial as leveduras em estoque -80° C, armazenados em glicerol 25%. Para os cultivos foi padronizado um intervalo de crescimento de 48h em estufa a 28º C, tempo ideal de crescimento para todas as leveduras em estudo.
peptona bacteriológico 2 %, as fontes de carbono glicose 20 g/L (YPD), xilose 20 g/L (YPX) e ágar bacteriológico 2%.
6.4 PPREPARO DE MATRIZ ÁCIDO ALFA-CIANO-4-HIDROXICINÂMICO
5 mg de matriz ácido alfa-ciano-4-hidroxicinâmico (10 mg/mL) foram pesados em uma balança analítica. Em seguida, foram adicionados 250 µL de Acetonitrila (ACN) 100%, 50 µL de TFA 3% e 200 µL de água ultrapura em um tubo polipropileno e levado ao vortéx até a matriz estivesse completamente solubilizada. A matriz foi preparada no momento do uso.
6.5 MÉTODOS DE DETECÇÃO DE PROTEÍNAS
6.5.1 Método direto
Com o auxílio de um palito de madeira estéril foi coletada uma colônia de cada levedura e transferida diretamente para a placa de inox de aquisição (Marca: Bruker Daltonics; usando diferentes tipos de placa). As colônias foram espalhadas em movimentos circulares até o preenchimento completo do poço de amostra. As cepas foram aplicadas em triplicatas. Após esperar a amostra secar no poço há temperatura ambiente foi adicionado sobre a superfície das colônias 1 µL de matriz ácido alfa-ciano-4-hidroxicinâmico.
6.5.2 Método extração proteica da colônia
Com o auxílio de um palito de madeira estéril foi coletada uma nova colônia de cada levedura e transferida para um tubo de polipropileno contendo 200 µL de etanol 70%. A solução foi centrifugada por 3 minutos a 20.000 x g. O etanol 70% foi removido e descartado. Em seguida foram adicionados ao pellet 10 µL de ácido fórmico (AF) 70% e 10 µL de Acetonitrila (ACN) 100% para a extração líquida das proteínas de cada levedura.
transferido para um tubo polipropileno contendo 3 µL de matriz ácido alfa-ciano-4-hidroxicinâmico, a amostra foi homogeneizada e aplicada 1 µL sobre à placa de inox de aquisição.
6.6 AQUSIÇÕES DOS DADOS BRUTOS PELO PROGRAMA FLEX CONTROL
Foram adquiridos vinte e quatro espectros de massa brutos para cada cepa de levedura, com o método aquisição MBT_FC.par de forma a garantir a reprodutibilidade dos perfis gerados com uma faixa de massa de 2000 a 20000 m/z utilizando o programa de aquisição Flex Control 3.0 (Bruker Daltonics). O banco de dados utilizado foi o Biotyper System 3.0 presente nos equipamentos da EMBRAPA – Recursos Genéticos e Biotecnologia. Os perfis proteicos gerados foram usados como base para as análises de componentes principais, análise de clusters e criação do banco de dados de perfis proteicos inseridos no programa Biotyper.
6.7 ANÁLISES DOS DADOS DE ESPECTROMETRIA DE MASSA PELO PROGRAMA BIOTYPER
6.7.1 Métodos de pré-processamento
6.7.2 Métodos de Criação do MSP, Dendrograma, ACP e Identificação de microrganismos
Para a confirmação dos dados obtidos por espectrometria de massa, análises estatísticas de componente principal (ACP) são realizadas por meio do programa Biotyper System 3.0 (MAIER; SCHAWARZ; KOSTRZEWA, 2008). O programa é capaz de gerar de maneira automática, a partir de uma média de 24 espectros de massa brutos, uma lista de massa semelhante no conjunto de espectros adquiridos de uma espécie (RETTINGER et al., 2012; THEEL et al., 2012; UHLIK et al., 2011). Assim um espectro de massa
padrão (MSP) é gerado contendo um total de 70 íons de relevância que possuem uma frequência mínima desejada de 25% nos espectros de massa brutos.
A identificação pode ser obtida de duas formas: utilizando o espectro de massa bruto individual do microrganismo ou pode ser obtida pela comparação dos MSP com o banco de dados que possui uma lista de massa dos espectros armazenados com informações de espécies e subespécies já conhecidas.
Durante a identificação as massas dos MS ou MSP são correlacionadas com a intensidade dos íons do banco de dados gerando um valor de semelhança na qual os valores de 0 a 1,6 registra dados não identificados, os valores de 1,7 a 1,9 são registrados como dados identificados como gênero e os valores acima de 2,0 representam um valor confiável possibilitando a identificação da amostra em estudo como espécie (NAGY et al., 2009; RETTINGER et al., 2012; VALENTINEet al., 2005), como demonstrado na
Figura 6: Exemplo de um perfil de microrganismo identificado pelo programa Biotyper, feita em setembro de 2014.
Fonte: Imagem elaborada pelo autor.
O programa Biotyper permite realizar análises estatísticas na forma de agrupamento e de criação de árvores filogenéticas, de duas maneiras principais: 1ª ao calcular a semelhança de todos os espectros brutos com os espectros do banco de dados, possibilitando a construção de um dendrograma de classificação. Este dendograma demonstra a posição de cada espécie em relação às outras conforme o grau de semelhança entre as espécies por meio de um método estatístico hierárquico (THEEL et al., 2011). 2ª
por meio de análises estatísticas multivariadas com base nas análises de componente principal para um determinado conjunto de espectros brutos (MAIER; SCHAWARZ; KOSTRZEWA, 2008).
A ACP utiliza os componentes principais (PC), a primeira a PC1 que é a combinação das variáveis de maneira linear, ou seja, agrupa o máximo de informação e a segunda à PC2 que também é a combinação das variáveis de maneira ortogonal a PC1; a terceira componente principal PC3 que também é a combinação das variáveis só que em ortogonal as duas primeiras, e assim por diante, de forma que as informações importantes fiquem concentradas nas primeiras PC devido ao cálculo do eixo ser de forma decrescente conforme a importância das varáveis contidas nos conjuntos de dados (NETO; SCARMINIO; BRUNS, 2006).
6.8 ANÁLISE POR HPLC DA PRODUÇÃO DE PRODUTOS GERADOS PELA FERMENTAÇÃO DE XILOSE
Em vista de obter os perfis fisiológicos das leveduras em estudo, as leveduras foram pré-inoculadas em 10 mL meio de cultura líquido YPD contendo xilose 20g/L crescidas em tubos tipo falcon de 50 mL em agitação durante 24h a 28ºC.
Após as 24h em agitação a cultura foi centrifugada durante 5 minutos a 4000 x g. Após a centrifugação o sobrenadante foi descartado e os pellets ressuspendidos em 3 mL de meio de cultura liquido com xilose 50 g/L e base de nitrogênio para levedura 100 g/L (YNB; Marca: Sigma – Aldrich). Em seguida as amostras foram inoculadas em triplicatas biológicas 1 mL em cada poço em uma microplaca de 96 poços de 1,1 mL com fundo em U e colocadas em estufas a 28ºC por 48h.
Após o período de crescimento de 48h as células de leveduras foram separadas do sobrenadante por centrifugação, repetida duas vezes por 5 minutos a 20.000 x g. 1 mL do sobrenadante foi transferido para um tubo voielt. As amostras foram analisadas por HPLC utilizando ácido sulfúrico (H2SO4-) a 5 mM como fase móvel (Biorad).
Tabela 2: Padrões estabelecidos para a curva padrão de HPLC.
Padrões HPLC
Padrão Xilose g/L Xilitol g/L Glicerol g/L Ácido Acético g/L Etanol g/L
p1 20 10,0 5,0 5,1 10,4
p2 10 5,0 2,5 2,5 5,1
p3 5 2,5 1,2 1,2 2,4
p4 2,5 1,3 0,6 0,6 1,2
p5 1,3 0,7 0,3 0,3 0,6
p6 0,7 0,3 0,2 0,2 0,3
p7 0,4 0,2 0,1 0,1 0,3
6.9 SEQUENCIAMENTOS DA REGIÃO D1/D2 DO DNAR 26S DAS LEVEDURAS SELVAGENS
6.9.1 Reações de PCR
Dez leveduras selvagens foram inoculadas em meio de cultura sólido YPD com glicose 20g/L, pelo método de esgotamento de placa e incubadas em um período de 48h nomeadas de A01 a A10.
Após o cultivo, uma Unidade formadora de colônia (UFC) foi diluída em micro tubo tipo eppendorf contendo 200 µL de água ultrapura esterilizada. As amostras foram homogeneizadas por 1 minuto, em seguida foram incubadas em termo bloco a 95ºC durante 5 minutos. Após a incubação 2 µL de amostra foram utilizados para a reação de PCR (MAUTONE, 2008).
O mix para a PCR foi preparado, utilizando de água ultrapura, tampão com KCl 1x, dNTP. 0,2 mM, MgCl2 2,5 mM, taq DNA polimerase 2 µL (Fermentas), 2 µL de DNA e os primers universais nas concentrações de 0,2 µM, descritos por Kurtzman & Robnett (1998), sendo eles o primer NL1 GCATATCAATAAGCGGAGGAAAAG e o primer NL4 GGTCCGTGTTTCAAGGACGG.
ocorre a extensão da cadeia de nucleotídeos pela Taq DNA polimerase a 72°C por 1 minuto. O terceiro estágio ocorre a extensão final a 72°C por 10 minutos e depois é armazenado a 4°C em ciclos infinitos (REIS, 2011; COSTA, 2014).
6.9.2 Análises de DNA por eletroforese
As amostras foram analisadas por técnica eletroforética em gel de agarose 1% após a reação de PCR. Foi utilizado o tampão SB 1X (NaoH 10 mM, pH 8,5, ajustado com ácido bórico) e utilizado 1 µL de Brometo de etídio 10 mg/mL para a criação da malha do gel. Para a corrida em gel as amostras foram homogeneizadas no tampão de amostra Loading Buffer (Tampão de carga). Foi utilizado como marcador de amostras o marcador Gene Ruler 1KB da Thermo (ANTONAGELO, 2012).
6.9.3 Purificações de DNA
Após a PCR as amostras amplificadas foram aplicadas em um gel de agarose a 1% para o corte das bandas. Após a eletroforese as bandas foram excisadas e purificadas pelo kit de purificação NucleoSpin® Gel and PCR Clean-up (750 preps) Macherey-Nagel, as bandas retiradas foram colocadas em tubos tipo eppendorf de 2 mL, e foi adicionado à solução NT1, os tubos foram aquecidos 15 minutos no termo agitador a 50º C até a banda estar completamente dissolvida.
Após o gel ter sido dissolvido na solução NT1, foi aplicado 700 µL em uma coluna para tubo de 2 mL e centrifugado por 30 segundo a 12.000 x g. Após a passagem na coluna foi adicionado 700 µL de solução NT3 e centrifugados por 30 segundos a 12. 000 x g. Após a centrifugação o NT3 foi descartado e foi adicionado a coluna 50 µL de solução NE e centrifugada por 30 segundos a 12.000 x g.
6.9.4 Sequenciamentos de DNA
de DNA, 3,2 pmol de primer NL1 e NL4 0,2 µM e água ultrapura, tendo um volume final de 7 µL. O sequenciamento foi realizado por método de SANGER.
6.9.5 Análise Filogenética
As sequencias foram analisadas utilizando os programas Bioedit para alinhamento das sequencias e o programa MEGA6 utilizado para a criação das árvores filogenéticas. As sequencias das leveduras controles foram retiradas do banco de dados CBS – KNAW Fungal Biodiverist Centre - Yeast 2011 (http://www.cbs.knaw.nl/). C. tenuis – CBS 615, M. guilliermondii – CBS 8417, S. stiptis– CBS 5773, S. passalidarum – DQ 109807; S. arborariae – CBS 11463. O método utilizado para a criação das árvores filogenéticas foi
o método estatístico Neighbor-Joining, realizado com um teste de bootstrap contendo 1000 repetições (CADETE, 2009).
6.10 IDENTIFICAÇÕES FISIOLÓGICAS
De forma a se obter dados taxonômicos baseados na fisiologia das leveduras a identificação foi realizada por meio de critérios metabólicos. Foi utilizado o equipamento Biolog MicrosStationTM, modelo Omnilog/Biolog. O banco de dados utilizado pelo Biolog é o GEN III que contém uma coleção com mais de 2.500 perfis metabólicos de bactérias, leveduras e fungos filamentosos, de interesses clínicos.
O Biolog realiza a identificação de leveduras estabelecendo um número de testes da capacidade de assimilação e oxidação de diferentes fontes de carbono e compara com os perfis metabólicos existentes no banco de dados do programa Management (Bioscan). Desta forma o programa estabelece um índice de semelhança entre o organismo estudado com o banco de dados de leveduras, obtendo as identificações para o organismo (PRAPHAILONG et al., 1997).
Em seguida foram transferidos 100 µL do líquido inoculante com as colônias inoculadas para uma microplaca de 96 poços (Biolog Yeast – YT plate). A microplaca contém dois poços com controles negativos, ou outros 94 poços contêm substratos para assimilação por oxidação.
Cada poço contém uma fonte de carbono específica e um corante violeta de tetrazólio que muda de incolor a roxo caso o substrato tenha sido oxidado (PRAPHAILNOG et al., 1997). Desta forma uma placa foi utilizada por levedura. Após todos os poços serem completamente preenchidos pela mesma levedura esta foi incubada em estufa a 28°C.
O índice de similaridade foi calculado com base nas reações do perfil metabólico da cepa em estudo comparado com as dez espécies do banco de dados que mais possuem similaridade com perfil metabólico da cepa em estudo. A leitura de cada levedura foi realizada com intervalos de 24h, durante 7 dias.
7. RESULTAODS
7.1 OTIMIZAÇÃO DO PROTOCOLO DE DETECÇÃO PROTEICA DAS CEPAS DE LEVEDURAS
Espectros de massa brutos foram obtidos utilizando a cepa de levedura S. cerevisiae CAT-1, como controle para a padronização do protocolo de detecção de
proteína, uma vez que o banco de dados do programa Biotyper é rico em perfis proteicos de diferentes linhagens de S. cerevisiae.
Foram propostos dois métodos de detecção de proteínas: método direto, onde o perfil proteico é adquirido da colônia intacta e o método extração proteica da colônia, onde o perfil proteico é adquirido a partir das proteínas extraídas em solução (ver item 6.5 p. 34).
Para estabelecer qual método é o mais eficiente na extração de proteínas, um total de seis espectros de massa bruto foi adquirido para cada método. O critério de seleção para o melhor método de detecção foi baseado no número de vezes que um íon de referência aparece nos seis espectros brutos (Figura 7).
Entre os métodos propostos é possível observar que o método direto não é tão eficaz para a detecção de íons de leveduras devido à baixa qualidade dos espectros adquiridos, essa qualidade se deve à quantidade de informação gerada pelo perfil bruto, tendo uma maior resolução e menos sensibilidade da quantidade de íons de referência adquiridos do perfil proteico.
Figura 7: Espectro de massa da levedura S. cerevisiae CAT-1 obtido pelas A) análise
7.2 CRIAÇÃO DO BANCO DE DADOS PADRÃO
Vinte e quatro espectros de massa brutos foram adquiridos de leveduras sabidamente fermentadoras de xilose (leveduras controles) cultivadas em dois meios de cultura sólido, um contendo glicose (YPD) e um contendo xilose (YPX). (Ver apêndice A p 70). Esses espectros foram utilizados para a criação de um espectro de massa padrão (MSP).
Os MSPs criados para cada levedura controle foram adicionados no programa de análise Biotyper 3.0, onde foi possível construir um dendrograma de classificação que demonstra o agrupamento das leveduras controles por fonte de carbono utilizada a uma semelhança quanto ao posicionamento das leveduras (Figura 8).
Foi possível observar que a levedura controle M. guilliermondii foi identificada a
nível de gênero pelo programa Biotyper como espécie Candida guilliermondii/Pichia guilliermondii, isso ocorre uma vez que o banco de dados do Biotyper contém a
classificação antiga desta levedura (AGUSTINI et al., 2014; KURTZMAN; SUZUKI,
2010), confirmando assim a qualidade da metodologia para a identificação de espécies de leveduras.
Nenhuma das outras leveduras controles que tiveram seus padrões adicionados no programa foram identificados, por não estarem de antemão no banco de dados do fabricante. Após a criação do banco de dados, novos espectros de massa brutos (MS) foram adquiridos para confirmar que as leveduras estavam sendo identificadas corretamente (Tabela3).
Tabela 3: Identificação de espectro brutos das leveduras controles após a criação do banco de dados.
Linhagem (MS-bruto) Identificação obtida Valor
identificação* Nível de identificação
C. tenuis C. tenuis SólidoYPD 2.8 Espécie
C. tenuis SólidoYPX 2.8 Espécie M. guilliermondii M. guilliermondii Sólido YPD 2.3 Espécie M. guilliermondii SólidoYPX 2.5 Espécie
C.guilliermondii/P.guilliermondii 1.7 Gênero S. stiptis S. stiptis SólidoYPX 2.1 Espécie S. stiptis SólidoYPD 2.0 Espécie S. arborariae S. arborariae SólidoYPD 2.9 Espécie S. arborariae SólidoYPX 2.7 Espécie S. passalidarum S. passalidarum SólidoYPD 2.9 Espécie S. passalidarum SólidoYPX 2.9 Espécie
* valor da identificação gerado pelo programa Biotyper: 0 a 1,6 não identificado; 1,7 a 1,9 identificação a nível de gênero; 2 a 3 identificação a nível de espécie.
7.3 CRIAÇÃO DO BANCO DE DADOS DE PERFIS PROTEICOS DE LEVEDURAS SELVAGENS SOMENTE EM XILOSE
Depois da criação do banco de dados para as leveduras controles, foram adquiridos os espectros de 10 leveduras selvagens, nomeadas de A01 a A10. Todas as 10 leveduras passaram pelo mesmo protocolo padrão de extração de proteínas e criação do espectro de massa padrão para adição ao banco de dados das leveduras controles.
É possível observar que elas são muito próximas entre si, uma vez que os espectros de massa bruto adquiridos das leveduras selvagens (ver apêndice B, p. 73) apresentam semelhanças quanto ao número íons e razão sinal/ ruído, além do valor de identificação acima de 2,0 gerado pelo programa para mais de uma levedura selvagem (Tabela 4).
Tabela 4: Identificação dos perfis proteicos brutos das leveduras selvagens.
Linhagem
(MS-bruto) Identificação obtida
Valor
identificação* Nível de identificação
A01 A01 Sólido YPX 2.4 Espécie
A03 Sólido YPX 2.2 Espécie
A02 A02 Sólido YPX 2.5 Espécie
M. guilliermondii Sólido YPX 2.1 Espécie
C. guilliermonii/P. guilliermondii 1.7 Gênero
A03 A03 Sólido YPX 2.7 Espécie
A01 Sólido YPX 2.2 Espécie
A05 Sólido YPX 1.7 Gênero
A04 A04 Sólido YPX 2.5 Espécie
A05 A05 Sólido YPX 2.6 Espécie
A03 Sólido YPX 1.7 Gênero
A06 A06 Sólido YPX 2.8 Espécie
A07 A07 Sólido YPX 2.7 Espécie
A08 A08 Sólido YPX 2.6 Espécie
A09 Sólido YPX 2.6 Espécie
A10 Sólido YPX 2.7 Espécie
A09 A09 Sólido YPX 2.7 Espécie
A08 Sólido YPX 2.6 Espécie
A10 Sólido YPX 2.5 Espécie
A10 A10 Sólido YPX 2.4 Espécie
A08 Sólido YPX 2.7 Espécie
A09 Sólido YPX 2.5 Espécie
A semelhança entre as leveduras pode ser tanto a nível de espécie quanto a nível de gênero, como é o caso da levedura A01 identificada como a mesma espécie da levedura A03 e a levedura A05 identificada como sendo do mesmo gênero com levedura A03.
As leveduras A08, A09 e A10 foram identificadas como a mesma espécie sendo elas mais próximas da levedura A02. Dentre as leveduras selvagens as mais diferentes são as leveduras A04, A06 e A07.
Foi possível observar que dentre as dez leveduras selvagens a levedura A02 foi identificada como M. guilliermondii. As demais leveduras selvagens não foram
identificadas com nenhuma das leveduras controles e nem com o banco de dados do fabricante.
Foi construído um dendrograma de classificação demonstrando o grau de semelhança entre as leveduras selvagens. No dendrograma há uma formação de dois grupos de leveduras, o grupo A contendo a leveduras A06, A01, A03, A04, A05 e A07 e o grupo B contendo as leveduras A02, A08, A09 e A10 (Figura 9).
Os MSP das leveduras selvagens foram comparados com os das leveduras controles de forma individual (ver apêndice C p. 76) e de forma coletiva onde é possível observar que existe uma formação de dois grupos de leveduras. O grupo A é composto pelas leveduras selvagens C. tenuis, M. guilliermondii, A02, A08, A09 e A10 estando a M. guilliermondii mais próxima da levedura A02. O grupo B composto pela levedura
selvagem A01, A03, A04, A05, A07 que não estão próximas a nenhuma levedura controle e a levedura selvagem A06 que se agrupa com a levedura controle S. passalidarum (figura
10).
7.4 ANÁLISES DPS SEQUENCIAMENTOS DO GENE DNAR 26S
A identificação da análise da sequência de DNAr 26S foi realizada para se obter uma possível identificação a nível de espécie para as leveduras selvagens. Além disso o sequenciamento foi utilizado como comparação para validar dos resultados obtidos pela espectrometria de massa.
Assim como na espectrometria de massa, há uma formação de dois grupos de leveduras selvagens, o grupo A contendo as leveduras A01, A03, A04, A05, A06, A07 e A09. O grupo B é composto pelas leveduras A02, A08 e A10 (Figura 11).
Figura 11: Árvore filogenética construída a partir das sequencias de DNAr 26S no programa MEGA 6. A) grupo A de leveduras selvagens; B) grupo B de leveduras selvagens.
Uma segunda árvore filogenética foi realizada entre as leveduras selvagens e as leveduras controles, neste caso há uma formação de dois grupos de leveduras, sendo o grupo A as leveduras selvagens que são mais próximas das leveduras controles S. passalidarum e S. arborariae e o grupo B das leveduras selvagens que é mais próximo
Figura 12: Árvore filogenética gerada pelo programa MEGA 6 por Neighbor-joining, a partir das sequencias de DNAr 26S das leveduras selvagens e controles.
Ao comparar os resultados obtidos de ambas as metodologias há uma semelhança quanto à formação dos grupos de leveduras selvagens e suas relações de proximidade entre si. Quando elas leveduras selvagens são comparadas com as leveduras controles é possível observar que há diferenças de classificação, como por exemplo a relação de proximidade da levedura selvagem A09 com os controles que muda conforme a metodologia utilizada.
Na identificação por espectrometria de massa a levedura A09 tem se agrupado com as leveduras A08 e A10 que são mais próximas da levedura controle M. guilliermondii enquanto que pelo sequenciamento do DNAr 26S ela tem sido identificada
7.5 CARACTERIZAÇÃO FISIOLÓGICA
De forma a se obter dados taxonômicos quanto à fisiologia das leveduras selvagens foi realizada a identificação das mesmas utilizando o banco de dados do Bioscan. O banco de dados do Bioscan é limitado a leveduras que são conhecidas apenas para uso clínico e algumas leveduras de amostras ambientais, assim a maioria das leveduras selvagens não foram corretamente identificadas.
Apesar da limitação foi possível obter uma identificação que fosse mais próxima da espécie em estudo. Com base no valor de referência estabelecido pelo fabricante para a identificação de leveduras, onde com 24h de crescimento o índice de similaridade (SIM) deve ser 0,9 para ser considerado aceitável. Com 48h de crescimento o SIM deve de 0,7; com 72h de crescimento o SIM deve ser de 0,65; com 96h ou mais horas de crescimento o SIM deve ser de 0,6 para ser considerável aceitável (ver item 6.10 p. 40).
As espécies obtidas abaixo do valor de referencias do Bioscan não tiveram suas sequencias inseridas na árvore filogenetica. As espécies obtidas na identificação do Bioscan (Tabela 5) foram utilizadas para enriquecer a árvore filogenética das leveduras e assim obter uma árvore curada que demonstrasse maior semelhança entre as leveduras selvagens e controles (Figura 13).
É possivel observar que a levedura A09 não teve uma identificação precisa nesta metodologia, sendo assim nenhuma das três técnicas de identificação utilizadas neste trabalho foram suficientes para determinar a real espécie desta levedura.
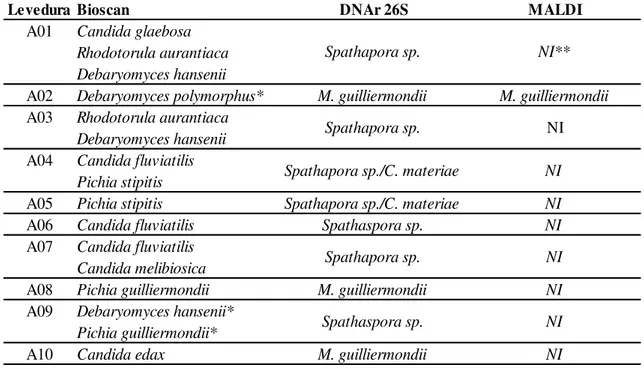
Tabela 5: Comparação pelos três metodos de identificação das leveduras selvagens.
Levedura Bioscan DNAr 26S MALDI
A01 Candida glaebosa Rhodotorula aurantiaca Debaryomyces hansenii
A02 Debaryomyces polymorphus* M. guilliermondii M. guilliermondii
A03 Rhodotorula aurantiaca Debaryomyces hansenii
A04 Candida fluviatilis Pichia stipitis
A05 Pichia stipitis Spathapora sp./C. materiae NI
A06 Candida fluviatilis Spathaspora sp. NI
A07 Candida fluviatilis Candida melibiosica
A08 Pichia guilliermondii M. guilliermondii NI
A09 Debaryomyces hansenii* Pichia guilliermondii*
A10 Candida edax M. guilliermondii NI
* Identificação obtida abaixo do valor de referencia do Bioscan. ** Não Identificado pelo banco de dados do biotyper
Spathapora sp. NI
Spathaspora sp. NI Spathapora sp. NI**
Spathapora sp. NI
7.6 LEVEDURAS SELVANGES: CAPACIDADE DE FERMENTAÇÃO
A fim de comprovar o perfil fisiológico das leveduras selvagens quanto a sua capacidade de produção de etanol e demais produtos derivados do consumo de xilose, foram realizadas análises de HPLC para quantificar a produção de etanol, xilitol, ácido acético e glicerol, após a fermentação de xilose.
Desta forma foi possível confirmar que as leveduras selvagens possuem um perfil fisiológico muito semelhante quanto a sua capacidade de consumir e fermentar xilose. Todas as leveduras selvagens são consumidoras de xilose uma vez que seu perfil fisiológico é bastante semelhante ao da levedura controle M. guilliermondii e S. arborariae quanto a quantidade de xilitol produzido (Figura 14A).
Assim como as leveduras S. arborirae e M. guilliermondii, as leveduras selvagens
utilizadas neste estudo possuem uma pequena capacidade de fermentar etanol chegando a menos de 2 g/L, tendo o seu metabolismo a preferência pela produção do xilitol, bem como a não produção de glicerol e ácido acético, o que é característico de leveduras consumidoras de xilose (Figura 14B). O perfil fisiológico foi obtido com um período de 48h uma vez que este é o tempo de crescimento para estas leveduras.
Apesar de não terem um perfil proteico semelhante as leveduras fermentadoras de xilose S. passalidarum e S. stiptis, as leveduras selvagens A01, A05 e A09, se destacam