D
INORAM
ARIA DAS
ILVAL
OPESF
ERREIRAR
R
E
E
S
S
I
I
S
S
T
T
Ê
Ê
N
N
C
C
I
I
A
A
A
A
A
A
N
N
T
T
I
I
M
M
A
A
L
L
Á
Á
R
R
I
I
C
C
O
O
S
S
E
E
M
M
P
P
l
l
a
a
s
s
m
m
o
o
d
d
i
i
u
u
m
m
f
f
a
a
l
l
c
c
i
i
p
p
a
a
r
r
u
u
m
m
:
:
E
E
N
N
V
V
O
O
L
L
V
V
I
I
M
M
E
E
N
N
T
T
O
O
D
D
O
O
S
S
G
G
E
E
N
N
E
E
S
S
p
p
f
f
c
c
r
r
t
t
E
E
p
p
f
f
m
m
d
d
r
r
1
1
I
I
NSNSTTIITTUUTTO O DDEEH
H
IGIGIIEENNEE EEM
M
EEDDIICCIINNAAT
T
ROROPPIICCALALU
U
NINIVVEERRSSIIDDAADDEEN
N
OOVVAA DDEEL
L
IISSBBOOAA LISBOA 2004DINORA MARIA DA SILVA LOPES FERREIRA
BOLSEIRA DA FUNDAÇÃO PARA A CIÊNCIA E TECNOLOGIA / PRAXIS XXI NO CENTRO DE MALÁRIA E OUTRAS DOENÇAS TROPICAIS / INSTITUTO DE HIGIENE E MEDICINA TROPICAL / UNIVERSIDADE NOVA DE
LISBOA (BOLSA BD/21573/99)
Resistência a Antimaláricos em Plasmodium
falciparum: Envolvimento dos Genes pfcrt e pfmdr1
Dissertação para obtenção do grau de Doutor No ramo das ciências biomédicas
Especialidade Biologia Celular e Molecular
Centro de Malária e Outras Doenças Tropicais/ Unidade de Investigação e Ensino de Malária/
Instituto de Higiene e Medicina Tropical
UNIVERSIDADE NOVA DE LISBOA LISBOA
A
A
GGRRAADDEECCIIMMEENNTTOOSSEste trabalho foi realizado no Centro de Malária e outras Doenças Tropicais (IHMT) e foi financiado pelo Programa PRAXIS XXI (BD/21573/99) e pelos projectos ”Busca de novos marcadores de resistência a antimaláricos” (PRAXIS/P/SAU/14070/98) e “RESMALSHIP – Development of a malaria resistance DNA chip as a public health tool for managment of Plasmodium falciparum malaria drug resistance” (QLK2-CT-2002-01503). Para que a sua realização fosse possível, além das referidas entidades, foi fundamental poder contar com o apoio e colaboração de diversas pessoas, às quais quero aqui expressar os meus sinceros agradecimentos:
Ao Prof. Doutor Virgílio do Rosário, Coordenador Cientifico do Centro de Malária e Outras Doenças Tropicais e Professor Catedrático da Unidade de Investigação e Ensino de Malária do Instituto de Higiene e Medicina Tropical, pela orientação deste trabalho bem como pelo apoio e disponibilidade que sempre demonstrou no estabelecimento de colaborações com outras instituições de forma a tornar possível a realização de algumas fases deste trabalho.
Aos Doutores José Pedro Gil (CMDT/IHMT e Instituto Karolinska, Suécia) e Pedro Lemos Cravo (CMDT e IHMT) pela componente de supervisão técnica e científica, a qual foi fundamental para a realização do trabalho desta tese de doutoramento.
À Prof. Doutora Isabel Sá-Correia, do Instituto Superior Técnico, ter-me recebido no seu laboratório, onde grande desenvolvimento na área de transfecção heteróloga teve lugar.
À Doutora Sandra Tenreiro pelo apoio dado durante a realização dos estudos no Instituto Superior Técnico e pela sua regular atenção à parte técnica da investigação. À Fátima Nogueira, pelo apoio na parte técnica e pelo excelente trabalho de equipa realizado na República Democrática de São Tomé e Príncipe e República de Angola. Ao Dr. Luís Bernardino do Hospital Pediátrico de Luanda, ao Doutor Luís Varandas do UEI Clínica de Doenças Tropicais/IHMT e a toda a equipa do serviço de urgências daquele hospital, pelo apoio e disponibilidade demonstrados para o desenvolvimento das actividades na República de Angola.
Ao Dr. Vílfrido Gil e aos técnicos do Centro Nacional de Endemias da República Democrática de São Tomé e Príncipe, todo o apoio dado durante o período de colheitas. À Prof. Doutora Sosdri Thaithong e Dra. Kanchana Rungsihirunrat, Universidade de Chulalongkorn, Bangkok, Tailândia, à Dra. Sílvia di Santi de Sucen, São Paulo, Brasil, à Dra. Salma Oliveira e Dra. Marinete Povoa do Instituto Evandro Chagas, Belém, Brasil, pelo envio de isolados de P. falciparum utilizados neste estudo.
Ao Prof. Doutor David Walliker, Universidade de Edimburgo, pela disponibilidade do seu centro de investigação, onde parte do trabalho de formação ocorreu.
À Prof. Doutora Aida Esteves e Prof. Doutora Lenea Campino, do Instituto de Higiene e Medicina Tropical, pela disponibilidade enquanto elementos da comissão tutorial e pelas sugestões tão importantes para o desenvolvimento deste estudo.
E a toda a equipa do CMDT, em especial às colegas Catarina Alves, Celeste Figueiredo, Ana Afonso e à D. Encarnação Horta pelo apoio e amizade sempre demonstrado.
Aos meus pais a quem quero expressar um obrigado muito especial por tudo o que me ensinaram e por terem tornado possível a realização de mais este objectivo. A eles e à Ju, à Adélia, ao Luís, ao Anselmo e aos “pequenitos”, quero agradecer a amizade, o apoio e o encorajamento nos bons e maus momentos.
Ao Tiago, pela companhia nas colheitas nocturnas, pelos fins-de-semana passados no CMDT e pela amizade e apoio constante e incondicional, que me foi dando forças para continuar, mesmo quando o Plasmodium parecia estar quase a ganhar a batalha.
P
P
UUBBLLIICCAAÇÇÕÕEESSA partir do conjunto de resultados, apresentados nesta dissertação, foram efectuadas apresentações em congressos e foram publicados os seguintes artigos científicos:
Lopes, D., Nogueira, F., Gil, J.P., Ferreira, C., do Rosário, V.E., Cravo, P. 2002. pfcrt and pfmdr1 mutations and chloroquine resistance in Plasmodium falciparum from Sao Tome and Principe, West Africa. Annals of Tropical Medicine and Parasitology, Dec; 96 (8): 831-4.
Lopes, D., Rungsihirunrat, K., Nogueira, F., Seugorn, A., Gil, J.P., do Rosário, V.E., Cravo, P. 2002. Molecular characterisation of drug-resistant Plasmodium falciparum from Thailand. Malaria Journal, Oct 14; 1 (1): 12.
S
S
UUMMÁÁRRIIOOA malária causada por Plasmodium falciparum é, em conjunto com a tuberculose e o HIV/SIDA, a maior causa de mortalidade mundial entre as doenças infecciosas e parasitárias. Considera-se que um diagnóstico atempado e um tratamento correcto são os elementos básicos para um programa de controlo da malária bem sucedido. No entanto, nas últimas décadas, o controlo e tratamento têm sido bastante dificultados pelo aparecimento e disseminação da resistência parasitária aos antimaláricos mais utilizados, nomeadamente à cloroquina Os mecanismos que conferem ao parasita a capacidade de resistir à maioria dos antimaláricos disponíveis, principalmente da classe das quinoleínas, não se encontram completamente elucidados. Consequentemente, o objectivo principal deste trabalho era o de avaliar o envolvimento dos genes pfcrt e pfmdr1 na resistência a esta classe de antimaláricos. Assim, recorrendo a culturas in vitro e micro-testes de susceptibilidade, foram primariamente caracterizados os perfis fenotipicos em isolados de P. falciparum colectados na RD de STP, R de Angola, Tailândia e Brasil, relativamente à sua fármaco - susceptibilidade a quatro antimaláricos da classe das quinoleínas: cloroquina, mefloquina, quinino e amodiaquina. Em paralelo, através da técnica de PCR-RFLP, determinou-se o genótipo de cada uma das amostras relativamente aos polimorfismos pfcrt K76T e pfmdr1 N86Y, N1042D e N1246Y. Os dados fenotipicos e genotipicos foram subsequentemente integrados e analisados estatisticamente, no intuito de detectar associações significativas entre um determinado marcador e a resposta do parasita a cada fármaco. Foram também avaliados os níveis de expressão basal destes dois genes em parasitas com diferentes susceptibilidades à cloroquina e à mefloquina, bem como a possibilidade de esta expressão poder ser induzida pelos referidos fármacos. Por último foram efectuados estudos de expressão heteróloga destes genes de Plasmodium em Saccharomyces cerevisiae de forma a avaliar o seu envolvimento no fenótipo de resistência a estes antimaláricos. Os resultados deste estudo demonstram a gravidade deste problema de resistência a antimaláricos, evidenciado pelas elevadas prevalências de resistência relativas a cada um dos fármacos em estudo, tendo sido também observado, que em algumas destas áreas endémicas, o fenótipo de multi-resistência se encontra completamente instalado,
níveis. Relativamente aos genótipos estes resultados demonstram que o gene pfcrt apresenta um envolvimento importante na resistência à cloroquina, quer pelo polimorfismo K76T, em especial na Tailândia, quer pelo seu aumento de expressão basal verificado em parasitas resistentes. Por outro lado, a mefloquina parece induzir o aumento de expressão deste gene, indiciando um possível envolvimento do mesmo no fenótipo de resistência a esta quinoleína. O polimorfismo N86 do gene pfmdr1 demonstrou uma associação com a resistência do parasita à mefloquina na Tailândia e quando é estudada a totalidade da amostra, tendo também sido encontrada uma associação entre este polimorfismo e o fenótipo de resistência ao quinino nos isolados provenientes do continente Africano e Sudeste Asiático. Os resultados dos estudos de expressão heteróloga demonstraram alguma inconclusividade, tendo no entanto permitido a obtenção de conhecimentos importantes para estudos que possam ser realizados no futuro.
Ficando assim evidente que os referidos fenótipos não são determinados exclusivamente pelos genes em estudo, dependendo provavelmente de factores adicionais, hipoteticamente outros polimorfismos e/ou acção de genes ainda não identificados.
A
A
BBSSTTRRAACCTTOf all infectious diseases, Plasmodium falciparum malaria is one of the major causes of morbidity and mortality worldwide, along with TB and HIV/AIDS. It is generally accepted that timely diagnosis and adequate treatment are the key elements for a well succeeded malaria control programme. However, over the past decades, the success of malaria control programmes based on drug treatment has been seriously hampered by the emergence and spread of parasites resistance to the most widely used antimalarials, such as chloroquine.
The mechanisms by which drug-resistant malaria parasites are able to by-pass the lethal effect of most drugs, especially of quinolines, have not been fully elucidated. Consequently, the main aim of this study was to evaluate the involvement of the pfmdr1 and pfcrt genes in resistance to quinoline-containing drugs. As such, making use of in vitro drug susceptibility micro-tests, the phenotypic profiles of several P. falciparum isolates collected in the RD of STP, R Angola, Thailand and Brazil, were determined in relation to their responses to four quinoline drugs: chloroquine, mefloquine, quinine e amodiaquine. In parallel to this, the genotype of each sample was determined, relative to the polymorphisms pfcrt K76T, pfmdr1 N86Y, N1042D and N1246Y. Subsequently, phenotypic and genotypic data were combined in order to search for significant associations between each genetic marker and the parasite´s response to a given drug. In addition, the basal expression levels of these genes were compared between parasites with different susceptibilities to the drugs chloroquine and mefloquine, and the possibility that this expression could be induced by drug challenge, was studied. Lastly, the pfcrt and pfmdr1 genes were expressed in the heterologous model system of Saccharomyces cerevisiae, as an additional tool to investigate their potential involvement in resistance to the drugs under study.
The results of this study demonstrated that antimalarial drug resistance of was a well established feature in natural parasite populations, as reflected by high prevalences of P. falciparum isolates resistant to individual drugs in all geographical areas studied. In addition, in some places, multi-drug resistance was also a common event, affecting most of the presently available compounds. The genetic and molecular analysis of P. falciparum field-collected isolates and laboratory-adapted strains, revealed that the pfcrt gene plays a major role in chloroquine resistance through its K76T polymorphism
(especially in Thailand) and/or by the increase in gene expression observed in resistant parasites. On the other hand, mefloquine appeared to induce an increase in the expression of this gene in cultured parasites, possibly indicating that higher levels of pfcrt transcripts may increase the parasite’s tolerance to this quinoline-containing compound. Overall, the allele 86N of the pfmdr1 gene, demonstrated to be statistically associated with mefloquine resistance in all areas studied, and in addition, an association was also verified to occur between this genotype and quinine resistance in isolates from Africa and South-east Asia.
The results regarding heterologous expression of the pfcrt and pfmdr1 genes in S. cerevisiae were limiting in their interpretation, but nevertheless, contributed to the gathering of important information that may be used in future studies.
In conclusion, the observations arising from this work demonstrated that antimalarial resistance of P. falciparum to quinoline drugs is widespread, and that the pfcrt and pfmdr1 genes appear to play an important, but not exclusive role, in the modulation of these phenotypes.
SOMMAIRE
La malaria causée par Plasmodium falciparum est, avec la tuberculose et le HIV/SIDA, la plus grande cause de mortalité mondiale dans les maladies infectieuses et parasitaires. On considère qu’un diagnostique précoce et un traitement correct sont les éléments basiques pour un programme du contrôle de la malaria bien réussi. Néanmoins, dans les dernières décennies, le contrôle et le traitement ont été difficulté par l’apparition et la dessiccation de la résistance parasitaire aux antipaludiques plus utilisés, notamment à la Chloroquine. Les mécanismes qui confèrent au parasite la capacité de résister à la plupart des antipaludiques disponibles, principalement ceux de la classe des quinoléines, ne sont pas totalement éclaircis. Par conséquent, l’objectif principal de ce travail à était celui d’évaluer l`engagement des gènes pfcrt et pfmdr 1 dans la résistance à cette classe de antipaludiques. Donc, en utilisant la culture in vitro et les micro-testes de susceptibilité, on a caractérisé, en premier, les profils phénotypiques des isolant de P. falciparum collectés en RD de STP, en R Angola, en Thaïlande et au Brésil, en ce qui concerne leur susceptibilité à quatre antipaludiques de la classe des quinoléines : chloroquine, méfloquine, quinine et amodiaquine. En parallèle, à travers la technique de PCR-RFLP, on a déterminé le genotype de chacun des échantillons relatif aux polymorphisme pfcrt K76T et pfmdr1 N86Y, N1042D et N1246Y. Les données phénotypiques et genotypiques ont été ensuite intégrées et analysées statistiquement, dans le but de détecter des associations significatives entre un marqueur déterminé et la réponse du parasite à chaque médicament. Les niveaux d`expression basale de ces deux génes dans des parasites avec de différentes susceptibilités à la chloroquine et à la méfloquine ont aussi été évalués, ainsi que la possibilité de cette expression pouvoir être poussée par les médicament déjà cité. Finalement, on a effectué des études d’expression hétérologue de ces gènes de Plasmodium en Saccharomyces cerevisiae de façon à évaluer son engagement dans le phénotype de résistance à ces antipaludiques .Les résultats de cette étude montrent la gravité du problème de la résistance aux antipaludiques, mis en évidence par ses hautes résistances a chacun de ces médicaments étudiés, l’observation ayant aussi démontré que dans certaines des surfaces endémiques, le phénotype de multi-résistance se trouve
actuellement disponible. En ce qui concerne les génotypes, ces résultats démontrent que le gène pfcrt présente un engagement important dans la résistance à la chloroquine, tant par le polymorphisme K76T, spécialement en Thailande, tant par son augmentation d’expression basale vérifiée dans les parasites résistants. D’un autre coté, la méfloquine paraît induire une augmentation de l’expression de ce gène, indiquant un possible investissement du même dans le phénotype de résistance à cette quinoléine. Le polymorphisme N86 du gène pfmdr1 a démontré une association avec la résistance du parasite à la méfloquine en Thaïlande et, quand on étudie la totalité de l’échantillon, on a aussi trouvé une association entre ce polymorphisme et le phénotype de résistance à la quinine dans les isolés provenant des continents africain et sud-est asiatique. Les résultats des études de l’expression hétérologue n`ont pas été conclusifs, ayant néanmoins permis l’obtention de connaissances importantes pour les études qui peuvent être réalisées dans le futur. Sachant ainsi avec évidence que les phénotypes cités ne sont pas déterminés exclusivement par les gènes en cours d’étude, ceux-ci dépendants probablement de facteurs adipocires, on peut penser, par hypothèse, que d’autres polymorphismes et/ou action de gène n’ont pas encore été identifiés.
L
L
IISSTTAADDEEA
A
BBRREEVVIIAATTUURRAASS∆∆∆∆ - Variação ou diferença
aa – Aminoácido
ABC – Região de ligação ao ATP (ATP binding cassette) ACR – Adequate Clinical Response
Amp – Ampicilina AMQ – Amodiaquina
ATP – Trifosfato de adenosina bp – Pares de bases
BSA – Albumina de soro bovino
cDNA – Ácido desoxirribonucleico complementar CMDT – Centro de Malária e Outras Doenças Tropicais CQ – Cloroquina
Ct – Treshold cycle (ciclo onde ocorre um aumento significativo da fluorescência emitida)
CYP – Citocromos P450 DEPC – Dietilpirocarbonato DHFR – Dihidrofolato reductase DHPS – Dihidropteroato sintetase DNA – Ácido desoxirribonucleico DNAse – Deoxiribonuclease I
dNTP – 3’-deoxinucleósido-5’-trifosfato dp – Desvio padrão
dTMP – Timidina monofosfato
DO600nm – Densidade óptica determinada a um comprimento de onda de 600 nm EDTA – Ácido etileno diamino tetra acético
ETF – Early treatment failure FPXI – Ferriprotoporfirina IX
GFP – Gene que codifica a proteína verde fluorescente de Aequorea Victoria (Green Fluorescent Protein)
GPD – Glico fosfato desidrogenase
γγγγGCS – γ glutamilcisteinil sintetase GPx – Glutatião peroxidase GR – Glutatião reductase GSH – Glutatião GSSH – Dissulfito de glutatião GST – Glutationa S-transferase HRPII - Histidine rich protein II IC – Concentração inibitoria
IHMT – Instituto de Higiene e Medicina Tropical Kb – Kilo bases (1000 bp)
kDa – Kilo dalton
LacZ – Gene de Escherichia coli que codifica a enzima β galactosidase LB – Meio Luria-Bertani
LDH – Lactato desidrogenase Log – Logaritmo base 10 LTF – Late treatment failure MDR – Multi drug resistance
min – Minutos MQ – Mefloquina N – Número
OMS – Organização Mundial de Saúde PABA – Ácido ρ-aminobenzoíco pBC – pBluescript
PBS – Phosphate buffered saline
PCR – Reacção de polimerização em cadeia (Polimerase Chain Reaction) PFCRT – Proteína Plasmodium falciparum Chloroquine resistance tansporter pfcrt – Gene Plasmodium falciparum chloroquine resistance transporter pfββββ-actinaI – Gene Plasmodium falciparum beta actina I
pfmdr1 – Gene Plasmodium falciparum multidrug resistance 1 Pgh1 – P glicoproteína 1
6PGD – Enzima 6 fosfato glicodesidrogenase p/v – Peso/ volume
QUIN – Quinino R – Resistente
R Angola – República de Angola
RDSTP – República Democrática de São Tomé e Príncipe RFLP – Restriction fragment length polymorphism
Rn – Sinal de fluorescência do produto amplificado em qualquer momento da reacção Rpm – Rotações por minuto
RPMIc – Meio de cultura completo RPMIinc – Meio de cultura incompleto
RT-PCR – Reverse transcriptase – polymerase chain reaction rRNA – Ácido ribonucleico ribossomal
S – Sensível
SDS – Dodecil sulfato de sódio SEAsiático – Sudoeste Asiático
SERCA – Retículo sarco/endoplasmático Ca2+ ATPase TBE – Tampão constituído por Tris, ácido bórico e EDTA TCTP – Translationally controlled tumor protein
TE – Tampão de eluição constituído por Tris EDTA TEMED – N,N,N’,N’ tetramethylethylenediamine TRIS – Tris (hidroximetil) aminometano
tRNA – Ácido ribonucleico de transferência TS – Timidilato sintetase
U – Unidades
UEI – Unidade de Investigação e Ensino UNL – Universidade Nova de Lisboa UV – Ultra violeta
V – Volt
v/v – Volume/ volume
Í
ÍNNDDIICCEEGGEERRAALL
Agradecimentos i
Artigos científicos iii
Sumário iv
Abstract vi
Sommaire viii Lista de abreviaturas x Índice geral xii
Índice de figuras xxv
Índice de tabelas xxxi
I I..IINNTTRROODDUUÇÇÃÃOO 1 I.1. Ciclo de vida de Plasmodium 2
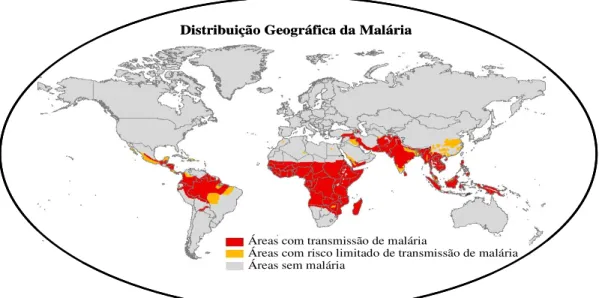
I.2. Distribuição geográfica da malária 4
I.3. O material genético de Plasmodium 5
I.4. Fármacos disponíveis para o tratamento da malária 7
I.5. A resistência aos antimaláricos – dados gerais 9
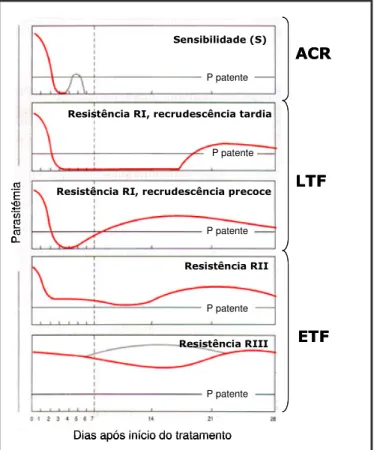
I.5.1. Classificação de resistência in vivo 9
I.5.2. Factores que influenciam o aparecimento de resistência 10
I.5.2.1. Características associadas ao hospedeiro humano 11
I.5.2.2. Características associadas ao parasita 11
I.5.2.3. Características associadas ao agente antimalárico 12
I.5.3. Espécies de Plasmodium resistentes a antimaláricos 12
I.5.4. Multi-resistência 13
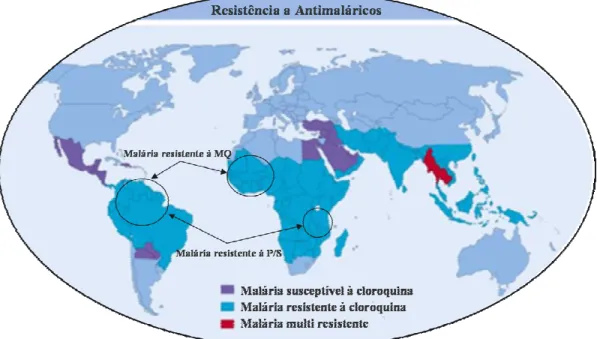
I.5.5. Focos de origem de resistência a antimaláricos e propagação 14 I I..66..MMaarrccaaddoorreessmmoolleeccuullaarreessddeerreessiissttêênncciiaa 16 I I..77..AAssppeeccttoossmmeettaabbóólliiccoossddooppaarraassiittaarreellaacciioonnaaddoossccoommaarreessiissttêênncciiaaaa a annttiimmaalláárriiccooss 17
20 I.8. Modos de acção dos antimaláricos e mecanismos de resistência
associados
I
I..88..11..AAnnttiimmaalláárriiccoossddooggrruuppooddaassqquuiinnoolleeíínnaass 20
I
I..88..11..11..CClloorrooqquuiinnaa 21 I.8.1.1.1. Modo de acção da cloroquina
21
I.8.1.1.2. Mecanismo de resistência à cloroquina
22
I.8.1.2. AAmmooddiiaaqquuiinnaa
28
I.8.1.2.1. Modo de acção da amodiaquina
28
I.8.1.2.2. Mecanismo de resistência à amodiaquina
29
I
I..88..11..33..QQuuiinniinnoo 29
I.8.1.3.1. Modo de acção do quinino
30
I.8.1.3.2. Mecanismo de resistência ao quinino
30
I
I..88..11..44..MMeeffllooqquuiinnaa 31 I.8.1.4.1. Modo de acção da mefloquina
31
I.8.1.4.2. Mecanismo de resistência à Mefloquina
31
I.8.2. Grupo dos antifolatos
33
I.8.2.1. Pirimetamina/ sulfadoxina
33
I.8.2.1.1. Modo de acção da pirimetamina/ sulfadoxina
33
I.8.2.1.2. Mecanismo de resistência da pirimetamina/ sulfadoxina
34
I.8.3. Derivados de Qinghaosu
36
I.8.3.1. AArrtteemmiissiinniinnaaeesseeuussddeerriivvaaddooss
36
I.8.3.1.1. Modo de acção da artemisinina e seus derivados
37
I.8.3.1.2. Mecanismo de resistência aos derivados de Qinghaosu
I I..99..OOBBJJEECCTTIIVVOOSS 39 I III..MMAATTEERRIIAALLEEMMÉÉTTOODDOOSS 42 II.1. MATERIAL 43 II.1.1. P. falciparum 43
II.1.1.1. Clones de referência
43
II.1.1.2. Isolados de P. falciparum
44
II.1.2. Escherichia coli
44
II.1.3. Saccharomyces cerevisae
44
II.1.4. Vectores de clonagem
44 II.1.4.1. pCR2.1 TOPO 44 II.1.4.2. pBluescript SK+/-(pBC) 45 II.1.4.3. p416GPD 46 II.1.4.4. p426GPD 46 II.1.4.5. pMET25_GFP 47 II.2. MÉTODOS 48 II.2.1. ESTUDO EPIDEMIOLÓGICO DA RESISTÊNCIA
49
II.2.1.1. Selecção de isolados de P. falciparum
50
II.2.1.2. Colheita e armazenamento de isolados de P. falciparum
51
II.2.1.3. Determinação de parasitémias
51
II.2.1.3.1. Parasitas por µl de sangue
51
II.2.1.3.2. Parasitas por 100 eritrocítos
52
II.2.1.4. Culturas in vitro de P. falciparum
52
II.2.1.4.1. Crio-preservação de amostras de P. falciparum em
II.2.1.4.2. Descongelação de amostras crio-preservadas de P.
falciparum 53
II.2.1.4.3. Sincronização de culturas in vitro de P. falciparum com
sorbitol 53
II.2.1.5. Ensaios de susceptibilidade in vitro a antimaláricos
54
II.2.1.5.1. Preparação das soluções de fármacos
54
II.2.1.5.2. Testes in vitro de susceptibilidade a antimaláricos segundo a
metodologia de MARK III, OMS 55 II.2.1.5.3. Leitura dos resultados dos ensaios de susceptibilidade in
vitro 56
II.2.1.6. Extracção de DNA de P. falciparum
57
II.2.1.6.1. Método de extracção com fenol-clorofórmio
57
II.2.1.6.2. Método de extracção com Chelex
58
II.2.1.7. PCR “Polimerase Chain Reaction”
58
II.2.1.7.1. Desenho de primers
59
II.2.1.7.2. Reacções de PCR
60
II.2.1.8. Identificação dos polimorfismos por RFLP
61
II.2.1.9. Electroforese de DNA
62
II.2.1.9.1. Electroforese em gel de agarose
63
II.2.1.9.2. Electroforese em gel de acrilamida
63
II.2.1.10. Tratamento estatístico
63
II.2.1.11. Sequenciação de DNA
64
II.2.1.11.1. Amplificação por PCR da sequência dos blocos B1, B2, B3 64 II.2.1.11.2. Clonagem dos fragmentos do gene pfcrt
65
II.2.1.11.3. Purificação de DNA
66
II.2.1.11.4. PCR para sequenciação
67
II.2.1.11.5. Análise de sequências
II.2.2. ESTUDOS DOS NÍVEIS DE EXPRESSÃO DO pfcrt E pfmdr1
69
II.2.2.1. Micro-cultura de P. falciparum para ensaios de expressão dos
genes pfcrt e pfmdr1 70
II.2.2.1.1. Durante o ciclo intra-eritrocitário na ausência de fármaco
70
II.2.2.1.2. Na fase de trofozoíto maduro na ausência de fármaco
71
II.2.2.1.3. Na fase de trofozoíto maduro na presença de fármaco
71
II.2.2.2. Extracção de RNA total
72
II.2.2.3. Síntese de cDNA
73
II.2.2.3.1. Tratamento com DNAseI
73 II.2.2.3.2. Transcrição reversa de cDNA
73
II.2.2.4. PCR em tempo real
74
II.2.2.4.1. Condições de PCR
74
II.2.2.4.2. Optimização da concentração de primers
76
II.2.2.4.3. Validação dos resultados
76
II.2.2.4.4. Avaliação da especificidade das reacções
77
II.2.2.4.5. Análise de resultados
78
II.2.3. ESTUDOS DE EXPRESSÃO HETERÓLOGA DOS GENES PFCRT E PFMDR1
EM S. CEREVISIAE 81
II.2.3.1. Avaliação do fenótipo a antimaláricos dos clones de Levedura 82 II.2.3.1.1. Amplificação da sequência codificante total dos genes pfcrt
e pfmdr1 82
II.2.3.1.1.1. Desenho de primers
82
II.2.3.1.1.2. Reacção de PCR
83
II.2.3.1.2. Preparação de DNA e vectores para clonagem 85
II.2.3.1.3.1. Preparação de células competentes de E. coli
86 II.2.3.1.4. Clonagem dos produtos de PCR no vector intermédio pBC 87
II.2.3.1.5. Transformação de células de E. coli 87
II.2.3.1.6. Confirmação das construções genéticas, com o vector pBC 88
II.2.3.1.7. Re-clonagem nos vectores p416 GPD e p426 GPD 89
II.2.3.1.8. Confirmação das construções genéticas entre os genes pfcrt /
pfmdr1 e os vectores p416GPD e 426 GPD 90
II.2.3.1.9. Cultura in vitro de Saccharomyces cerevisiae 91
II.2.3.1.9.1. Preparação de células competentes de S. cerevisiae 92
II.2.3.1.10. Transformação de S. cerevisiae 92
II.2.3.1.11.Testes de susceptibilidade a antimaláricos em leveduras
transformadas 93
II.2.3.2. Localização sub-celular das proteínas PFCRT e Pgh1 expressas
em levedura 94
II.2.3.2.1. Amplificação da sequência codificante total dos genes pfcrt
e pfmdr1 94
II.2.3.2.1.1. Desenho de primers 95
II.2.3.2.1.2. Reacção de PCR 96
II.2.3.2.2. Preparação de DNA e vector para clonagem 96
II.2.3.2.3. Construção das fusões genéticas GFP_ pfcrt e GFP_ pfmdr1 97 II.2.3.2.4. Localização sub-celular das proteínas por Microscopia de
Fluorescência 98
II.2.3.2.5. Confirmação da construção das fusões genética GFP_ pfcrt
e GFP_ pfmdr1 98
I
IIIII..RREESSUULLTTAADDOOSS 100
III.1. Estudo epidemiológico da resistência aos antimaláricos
a) Testes in vitro de susceptibilidade a antimaláricos – áreas de estudo e
fármacos testados 106
b) PCR-RFLP para estudo de frequências alélicas nos genes pfcrt e pfmdr1
107
I
IIIII..11..11..RReeppúúbblliiccaaDDeemmooccrrááttiiccaaddeeSSããooTToommééeePPrríínncciippee 110 III.1.1.1. Amostragem
110
III.1.1.2. Resposta in vitro aos antimaláricos
110
III.1.1.2.1. Cloroquina
110
III.1.1.3. Frequência alélica
111
III.1.1.4. Associação entre os marcadores moleculares e a resposta à
cloroquina 113
III.1.1.5 Resumo
114
III.1.2. República de Angola
115
III.1.2.1. Amostragem
115
III.1.2.2. Resposta in vitro aos antimaláricos
115 III.1.2.2.1. Cloroquina 115 III.1.2.2.2. Mefloquina 116 III.1.2.2.3. Quinino 116 III.1.2.2.4. Multi-resistência 116
III.1.2.3. Frequência alélica
117
III.1.2.4. Associação entre os marcadores moleculares e a resposta
aos fármacos 119 III.1.2.4.1. Cloroquina 120 III.1.2.4.2. Mefloquina 121 III.1.2.4.3. Quinino 121 III.1.2.5. Resumo 122 III.1.3. Tailândia 123
III.1.3.1. Amostragem
123
III.1.3.2. Resposta in vitro aos antimaláricos
124 III.1.3.2.1. Cloroquina 124 III.1.3.2.2. Mefloquina 124 III.1.3.2.3. Quinino 124 III.1.3.2.4. Amodiaquina 124 III.1.3.2.5 Multi-resistência 125
III.1.3.3. Frequência alélica
126
III.1.3.4. Associação entre os marcadores moleculares e a resposta
aos fármacos 127 III.1.3.4.1. Cloroquina 128 III.1.3.4.2. Mefloquina 129 III.1.3.4.3. Quinino 130 III.1.3.4.4. Amodiaquina 131 III.1.3.5. Resumo 132 III.1.4. Brasil 133 III.1.4.1. Amostragem 133
III.1.4.2. Resposta in vitro a antimaláricos
134 III.1.4.2.1. Cloroquina 134 III.1.4.2.2. Mefloquina 134 III.1.4.2.3. Quinino 134 III.1.4.2.4. Amodiaquina 135 III.1.4.2.5. Multi-resistência 135
III.1.4.3. Frequência alélica
III.1.4.4. Associação entre os marcadores moleculares e a resposta aos fármacos 138 III.1.4.4.1. Cloroquina 139 III.1.4.4.2. Mefloquina 140 III.1.4.4.3. Quinino 140 III.1.4.4.4. Amodiaquina 141 III.1.4.5. Resumo 142
III.1.5. Estudo global
143
III.1.5.1. Amostragem
143
III.1.5.2. Resposta in vitro a antimaláricos
143 III.1.5.2.1. Cloroquina 144 III.1.5.2.2. Mefloquina 145 III.1.5.2.3. Quinino 145 III.1.5.2.4. Amodiaquina 145 III.1.5.2.5. Multi-resistência 145
III.1.5.3. Frequência alélica
147
III.1.5.4. Associação entre os marcadores moleculares e a resposta
aos fármacos 149 III.1.5.4.1. Cloroquina 150 III.1.5.4.2. Mefloquina 150 III.1.5.4.3. Quinino 150 III.1.5.4.4. Amodiaquina 150 III.1.5.5. Resumo 151
III.1.6. Caracterização dos genótipos dos isolados multi-resistentes
152
III.1.7. Haplótipos do gene pfcrt
III.2. ESTUDOS DOS NÍVEIS DE EXPRESSÃO DOS GENES PFCRT E PFMDR1
155
a) Selecção dos isolados de P. falciparum
155
a.1) Determinação dos IC’s 50 de cloroquina e mefloquina
155
b) Optimização das condições de PCR em tempo real
159
III.2.1. Avaliação dos níveis de expressão basal dos genes pfcrt e pfmdr1
ao longo do ciclo intra-eritrocitário de P. falciparum 162
III.2.1.1. Caracterização do material por microscopia óptica
162
III.2.1.2. Quantificação relativa dos genes ao longo do ciclo de vida do Plasmodium
163 III.2.1.3. Resumo
165 III.2.2. Estudo comparativo do nível de expressão basal dos genes pfcrt
e pfmdr1 em parasitas com diferentes fenótipos 165
III.2.2.1. Caracterização do material por microscopia óptica
165
III.2.2.2. Expressão basal do gene pfcrt
166 III.2.2.3. Expressão basal do gene pfmdr1
167
III.2.2.4. Resumo
168
III.2.3. Estudos da expressão dos genes pfcrt e pfmdr1, na presença dos
fármacos cloroquina e mefloquina 169
III.2.3.1. Caracterização do material das colheitas por Microscopia
Óptica 169
III.2.3.2.1. Estudos de expressão dos genes pfcrt e pfmdr1 em
parasitas submetidos à cloroquina 169
III.2.3.2.1.1. Estudos de expressão dos genes no clone Dd2
170
III.2.3.2.1.2. Estudos de expressão dos genes clone 3D7
171
III.2.3.2.1.3. Estudos de expressão dos genes no Isolado STP 611
173
III.2.3.2.1.4. Estudos de expressão dos genes no Isolado STP 595
174
III.2.3.2.2. Estudos de expressão dos genes pfcrt e pfmdr1 em parasitas
III.2.3.2.2.1. Estudos de expressão dos genes no clone Dd2
176
III.2.3.2.2.2. Estudos de expressão dos genes no clone 3D7
178
III.2.3.2.2.3. Estudos de expressão dos genes no Isolado STP 611
180
III.2.3.2.2.4. Estudos de expressão dos genes no Isolado STP 595
181
III.2.3.2.3. Resumo
183
III.3. Estudos de expressão heteróloga dos genes pfcrt e pfmdr1 em Saccharomyces cerevisiae
185 a) Selecção da estirpe de levedura
185
b) Amplificação da sequência codificante total dos genes pfcrt e pfmdr1
187
c) Clonagem dos genes pfcrt e pfmdr1
189
III.3.1. Avaliação da susceptibilidade a antimaláricos da levedura transformada com os genes pfcrt e pfmdr1 de P. falciparum
192
III.3.2. Localização sub celular das proteínas, codificadas pelos referidos genes, no sistema heterólogo, S. cerevisiae
195 III.3.3. Resumo 198 I IVV..DDIISSCCUUSSSSÃÃOO 199 I IVV..11..EESSTTUUDDOOEEPPIIDDEEMMIIOOLLÓÓGGIICCOODDAARREESSIISSTTÊÊNNCCIIAA 200 IV.1.1. RD STP 205 IV.1.2. R Angola 208 IV.1.3. Tailândia 210 IV.1.4. Brasil 213 IV.1.5. Analise global
215 I
IVV..22EESSTTUUDDOOSSDDOOSSNNÍÍVVEEIISSDDEEEEXXPPRREESSSSÃÃOODDOOppffccrrttEEppffmmddrr11 222
IV.2.1. Avaliação dos níveis de expressão basal dos genes pfcrt e pfmdr1
ao longo do ciclo intra-eritrocitário de P. falciparum 225
IV.2.2. Estudo comparativo do nível de expressão basal dos genes pfcrt
IV.2.3. Avaliação da resposta do parasita à presença de cloroquina e
mefloquina relativamente à expressão dos genes pfcrt e pfmdr1 228
I IVV..33..EESSTTUUDDOOSSDDEEEEXXPPRREESSSSÃÃOOHHEETTEERROOLLOOGGAADDOOSSGGEENNEESSppffccrrttEEppffmmddrr11 E EMMSS..CCEERREEVVIISSIIAAEE 231 V V..PPRRIINNCCIIPPAAIISSCCOONNCCLLUUSSÕÕEESSDDEESSTTEEEESSTTUUDDOO 237 V VII..PPEERRSSPPEECCTTIIVVAASSDDEETTRRAABBAALLHHOOFFUUTTUURROO 240 V VIIII..RREEFFEERRÊÊNNCCIIAASSBBIIBBLLIIOOGGRRÁÁFFIICCAASS 242 V VIIIIII..AANNEEXXOOSS 283 A
ANNEEXXOO11-- Principais soluções utilizadas neste trabalho 284
A
ANNEEXXOO22-- Preparação de meios e aminoácidos 285
AANNEEXXOO33 - Preparação de eritrócitos não parasitados
287
AANNEEXXOO44-- Preparação do gel de poliacrilamida 8%
288 AAnneexxoo55-- Primers utilizados para PCR RFLP
289
AANNEEXXOO66-- Primers para amplificação dos “blocos” do gene pfcrt para
sequenciação
290 AANNEEXXOO77-- Primers para amplificação por PCR em tempo real
291
A
ANNEEXXOO88-- Primers para amplificação da sequência codificante total dos
genes para expressão em S. cerevisiae 292
A
ANNEEXXOO99-- Mapas de restrição dos genes pfcrt e pfmdr1 293
A
ANNEEXXOO1100 - Primers utilizados na amplificação das sequências
codificantes dos genes para construção de fusões génicas com a GFP 294
A
ANNEEXXOO1111-- Resultados da caracterização dos isolados de P. falciparum
da RDSTP 295
A
ANNEEXXOO1122-- Resultados da caracterização dos isolados de P. falciparum
da R Angola 296
A
ANNEEXXOO1133 - Resultados da caracterização dos isolados de P. falciparum
da Tailândia 297
A
ANNEEXXOO1144-- Resultados da caracterização dos isolados de P. falciparum
A
ANNEEXXOO1155-- Avaliação dos haplótipos para associação aos fenótipos 299
A
ANNEEXXOO1166-- Resultados da sequenciação do gene pfcrt 300
I
Í
ÍNNDDIICCEE DDEEFFIIGGUURRAASS
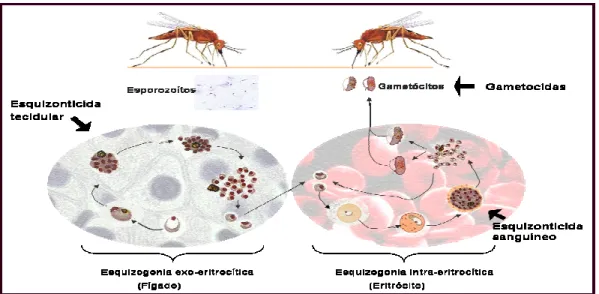
Figura I.1 – Ciclo de vida do parasita Plasmodium falciparum. 3
FIGURA I.2 – Mapa da distribuição geográfica da malária em 2002. 5
Figura I.3 – Fases do ciclo de vida onde actuam os antimaláricos. 9
Figura I.4 – Resposta clínica do indivíduo ao tratamento, segundo as duas
classificações propostas pela OMS 10
FIGURA I.5 – Mapa das áreas geográficas onde o P. falciparum apresenta
resistência aos fármacos mais utilizados. 16
Figura I.6 – Esquema resumido dos principais aspectos metabólicos do ciclo
de vida do parasita, relacionados com o modo de acção dos antimaláricos. 20
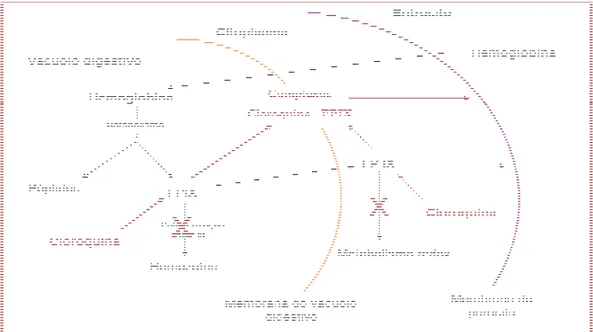
Figura I.7 – Esquema representativo do modo de acção da cloroquina. 22
Figura I.8 – Resumo da via biosintética do ácido fólico alvo da acção do
Fansidar®. 34
Figura I.9 – Esquema resumido do modo de acção como antimalárico da
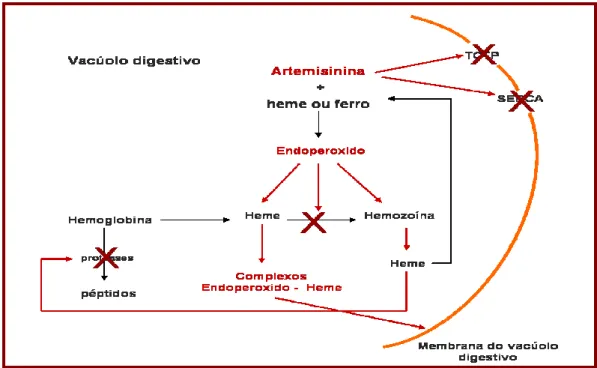
artemisinina e seus derivados. 38
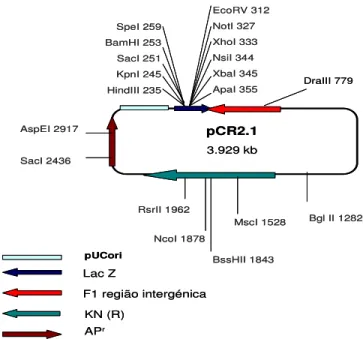
Figura II.1 – Esquema representativo do mapa físico do vector pCR2.1. 45
Figura II.2 – Esquema representativo do mapa físico do vector pBC. 45
Figura II.3 – Esquema representativo do mapa físico do vector p416GPD. 46
Figura II.4 – Esquema representativo do mapa físico do vector p426GPD. 47
Figura II.5 – Esquema representativo do mapa físico do vector
pMET25_GFP. 47
Figura II.6 – Esquema da metodologia utilizada na linha directriz 1 – Estudo
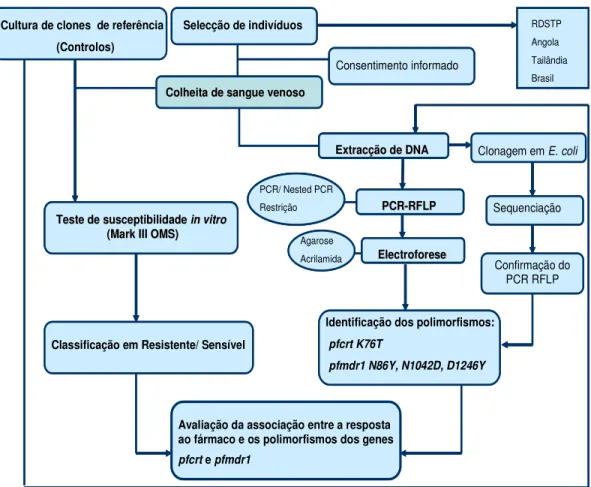
epidemiológico da resistência. 49
Figura II.7 – Esquema das placas em que são efectuados os testes de
susceptibilidade 56
Figura II.8 – Esquema representativo da metodologia adoptada para sequenciação dos fragmentos (B1, B2, B3), do gene pfcrt para clonagem no
vector pBC2.1 e transformação de E. coli. 68
Figura II.9 – Esquema da metodologia utilizada na linha directriz 2 – Estudo
Figura II.10 – Representação da emissão de fluorescência pelo Sybr green
durante a reacção de PCR. 75
Figura II.11 – Curvas de dissociação 78
Figura II.12 – Fotografia do gel de agarose 2% correspondente à eletroforese
dos produtos dos 3 genes amplificados por PCR em tempo real. 78
Figura II.13 – Representação do sinal de fluorescência do produto amplificado (Rn) em função do número de ciclos, onde as curvas de
amplificação quantificam o produto amplificado ao longo da reacção de PCR. 79
Figura II.14 – Esquema da metodologia utilizada na linha directriz 3 –
Estudos de expressão heteróloga dos genes pfcrt e pfmdr1 em S. cerevisiae. 81
Figura II.15 – Esquema representativo da reacção de recombinação
homóloga entre os genes de Plasmodium e o vector pMET25. 95
Figura III.1 – Géis correspondentes às electroforeses realizadas com os
produtos de PCR e produtos das digestões com endonucleases. 108
Figura III.2 – Mapa do arquipélago da RDSTP com as regiões onde foram
efectuadas as colheitas assinaladas com um círculo azul. 110
Figura III.3 – Prevalência da resistência à cloroquina na RD STP. 111
Figura III.4 – Frequências alélicas encontrados nos isolados estudados da
RDSTP. 112
Figura III.5 – Representação gráfica dos alelos dos genes pfcrt e pfmdr1
agrupados de acordo com a sua resposta in vitro à cloroquina. 114
Figura III.6 – Mapa da R Angola com a região onde foram efectuadas as
colheitas assinalada com um círculo azul. 115
Figura III.7 – Representação das prevalências de resistência aos
antimaláricos encontradas nos isolados de R Angola. 117
Figura III.8 – Representação gráfica das frequências alélicas encontrados
nos isolados da R Angola. 118
Figura III.9 – Representação gráfica do número de alelos dos genes pfcrt e
pfmdr1, agrupados de acordo com a sua resposta in vitro à cloroquina. 120
Figura III.10 – Representação gráfica do número de alelos dos genes pfcrt e
Figura III.11 – Representação gráfica do número de alelos dos genes pfcrt e
pfmdr1, agrupados de acordo com a sua resposta in vitro ao quinino. 122
Figura III.12 – Mapa da Tailândia com as regiões onde foram efectuadas as
colheitas assinaladas com um círculo azul. 123
Figura III.13 – Representação das prevalências de resistência in vitro aos
antimaláricos verificadas nos isolados provenientes da Tailândia. 125
Figura III.14 – Representação gráfica das frequências de cada alelo
encontradas nos isolados da Tailândia. 127
Figura III.15 – Representação gráfica do número de alelos dos genes pfcrt e
pfmdr1, agrupados de acordo com a sua resposta in vitro à cloroquina. 129
Figura III.16 – Representação gráfica do número de alelos dos genes pfcrt e
pfmdr1, agrupados de acordo com a sua resposta in vitro à mefloquina. 130
Figura III.17 – Representação gráfica do número de alelos dos genes pfcrt e
pfmdr1, agrupados de acordo com a sua resposta in vitro ao quinino. 131
Figura III.18 – Representação gráfica do número de alelos dos genes pfcrt e
pfmdr1, agrupados de acordo com a sua resposta in vitro à amodiaquina. 132
Figura III.19 – Mapa do Brasil com as regiões onde foram efectuadas as
colheitas assinaladas com um círculo azul. 133
Figura III.20 – Representação das percentagens de resistência in vitro aos
antimaláricos verificadas nos isolados do Brasil. 136
Figura III.21 – Representação gráfica das frequências de cada alelo nos
isolados do Brasil. 137
Figura III.22 – Representação gráfica do número de alelos dos genes pfcrt e
pfmdr1, agrupados de acordo com a sua resposta in vitro à cloroquina. 139
Figura III.23 – Representação gráfica do número de alelos dos genes pfcrt e
pfmdr1, agrupados de acordo com a sua resposta in vitro à mefloquina. 140
Figura III.24 – Representação gráfica do número de alelos dos genes pfcrt e
pfmdr1, agrupados de acordo com a sua resposta in vitro ao quinino. 141
Figura III.25 – Representação gráfica do número de alelos dos genes pfcrt e
pfmdr1, agrupados de acordo com a sua resposta in vitro à amodiaquina. 142
Figura III.26 – Distribuição geográfica dos isolados incluídos no estudo
Figura III.27 – Representação das percentagens de resistência aos antimaláricos verificadas em cada uma das áreas endémicas incluídas no
estudo. 147
Figura III.28 – Representação gráfica, em percentagem, das frequências de
cada alelo, encontrados na totalidade dos isolados estudados. 148
Figura III.29 – Gráfico dos resultados dos testes de susceptibilidade à
cloroquina do isolado STP 595. 156
Figura III.30 – Gráfico dos resultados dos testes de susceptibilidade à
cloroquina do isolado STP 611. 156
Figura III.31 – Gráfico dos resultados dos testes de susceptibilidade à
mefloquina do isolado do isolado STP 595. 157
Figura III.32 – Gráfico dos resultados dos testes de susceptibilidade à
mefloquina do isolado STP 611. 157
Figura III.33 – Gráfico dos resultados dos testes de susceptibilidade à
mefloquina do clone Dd2. 157
Figura III.34 – Representação gráfica dos valores Ct em função do log da concentração de DNA obtidos por PCR em tempo real, bem como a
representação da regressão linear que lhes está associada. 160
Figura III.35 – Esquema representativo do estadio de desenvolvimento dos
parasitas em cada colheita. 163
Figura III.36 – Perfil de expressão dos genes pfcrt e pfmdr1 ao longo do
ciclo intra eritrocitário do clone de P. falciparum Dd2. 164
Figura III.37 – Esquema representativo do estadio de desenvolvimento dos
parasitas no início do ensaio e no momento da colheita de material celular. 165
Figura III.38 – Representação do nível de expressão basal dos genes pfcrt e pfmdr1 em culturas sincronizadas na fase de trofozoíto maduro, em cada um dos parasitas estudados, relativamente ao clone sensível de P. falciparum
3D7, utilizado como controlo (Nfold). 168
Figura III.39 – Representação esquemática das fases de desenvolvimento do parasita, identificadas por microscopia óptica, no momento das colheitas de
material. 169
Figura III.40 – Expressão dos genes pfcrt e pfmdr1 observada no clone Dd2 submetido a cloroquina, relativamente à expressão basal no mesmo clone
Figura III.41 – Expressão dos genes pfcrt e pfmdr1 observada no clone 3D7 submetido a cloroquina, relativamente à expressão no mesmo clone sem
fármaco (Nfold), na fase de trofozoíto maduro. 172
Figura III.42 – Expressão dos genes pfcrt e pfmdr1 observada no isolado 611 submetido a cloroquina, relativamente à expressão no mesmo isolado
sem fármaco (Nfold), na fase de trofozoíto maduro. 174
Figura III.43 – Expressão dos genes pfcrt e pfmdr1 observada no isolado STP 595 submetido a cloroquina, relativamente à expressão no mesmo
isolado sem fármaco (Nfold), na fase de trofozoíto maduro. 175
Figura III.44 – Expressão dos genes pfcrt e pfmdr1 observada nos parasitas estudados submetidos a cloroquina, cada um deles relativamente à sua
expressão basal (Nfold), na fase de trofozoíto maduro. 176
Figura III.45 – Expressão dos genes pfcrt e pfmdr1 observada no clone Dd2 submetido a mefloquina, relativamente à sua expressão basal (Nfold), na fase
de trofozoíto maduro. 178
Figura III.46 – Expressão dos genes pfcrt e pfmdr1 observada no clone 3D7 submetido a mefloquina, relativamente à expressão no mesmo clone sem
fármaco (Nfold), na fase de trofozoíto maduro. 179
Figura III.47 – Expressão dos genes pfcrt e pfmdr1 observada no isolado STP 611 submetido a mefloquina, relativamente ao seu nível de expressão
basal (Nfold), na fase de trofozoíto maduro. 181
Figura III.48 – Expressão dos genes pfcrt e pfmdr1 observada no isolado STP 595 submetido a mefloquina, relativamente à expressão no mesmo clone
sem fármaco (Nfold), na fase de trofozoíto maduro. 182
Figura III.49 – Expressão dos genes pfcrt e pfmdr1 observada nos parasitas estudados submetidos à mefloquina, cada um deles relativamente à sua
expressão basal (Nfold), na fase de trofozoíto maduro. 183
Figura III.50 – Fotografias dos spots de crescimento das duas estirpes de levedura By4146, By4146∆erg6, resultantes dos testes de susceptibilidade
aos antimaláricos. 186
Figura III.51 – Fotografias de géis de agarose, resultantes da eletroforese
realizada aos produtos amplificados do gene pfcrt (A) e pfmdr1 (B). 188
Figura III.52 – Fotografia do gel de agarose 0,8% da eletroforese do DNA
da sequência codificante dos genes pfcrt e pfmdr1. 188
Figura III.53 – Fotografia dos géis dos produtos de restrição para
Figura III.54 – Géis de confirmação das construções génicas do gene pfcrt
com os vectores 416 e 426 GPD. 190
Figura III.55 – Fotografia do gel de agarose da electroforese dos produtos da digestão para confirmação da construção genética do gene pfmdr1 com o
vector 416 GPD. 191
Figura III.56 – Crescimento apresentado pelos clones By4146∆erg6 – pfcrt nos testes de susceptibilidade a antimaláricos, comparação entre vários
fármacos. 193
Figura III.57 – Crescimento apresentado pelos clones By4146∆erg6 – pfmdr1 nos testes de susceptibilidade a antimaláricos, comparação entre
vários fármacos. 194
Figura III.58 – Fluorescência apresentada pelas células de S. cerevisiae
albergando o plasmídeo pMET25 com a fusão proteica pfcrt_GFP. 196
Figura III.59 – Fluorescência apresentada pelas células de S. cerevisiae
Í
ÍNNDDIICCEE DDEETTAABBEELLAASS
Tabela I.1 – Classes de antimaláricos de acordo com a fase do ciclo do P.
falciparum em que actuam. 8
Tabela II.1 – Caracterização dos clones de P. falciparum de referência. 43
Tabela II.2 – Períodos de segurança relativamente à toma de antimaláricos. 50
Tabela II.3 – Critérios de inclusão em testes de susceptibilidade segundo
MARK III da OMS. 50
Tabela II.4 – Concentrações (nM) de antimalárico testadas nos testes in vitro. 55
Tabela II.5 – Classificação dos parasitas (MARK III - OMS), relativamente à
concentração de antimalárico (IC99). 57
Tabela II.6 – Sequências dos primers e condições de PCR, para amplificação
dos fragmentos dos genes pfcrt e pfmdr1. 60
Tabela II.7 – Enzimas e condições de restrição, para identificação dos
polimorfismos. 61
Tabela II.8 – Sequência dos primers e condições de PCR para amplificação
dos blocos do gene pfcrt. 65
Tabela II.9 – Polimorfismos associados a cada um dos blocos amplificados
por PCR, B1, B2 e B3. 65
Tabela II.10 – Mistura e condições da reacção de PCR de sequenciação. 67
Tabela II.11 – Sequência dos primers e condições de reacção de PCR em
tempo real. 75
Tabela II.12 – Mistura de reacção de PCR em tempo real. 76
Tabela II.13 – Misturas de reacção utilizadas no protocolo de Long PCR. 83
Tabela II.14 – Sequência dos primers e condições de PCR, para amplificação
dos genes pfcrt e pfmdr1 Nested long PCR. 84
Tabela II.15 – Enzimas e condições de restrição para criação de extremidades
Tabela II.16 – Restrições para confirmação das construções genéticas entre os
genes pfcrt/ pfmdr1 com pBC. 89
Tabela II.17 – Restrições para confirmação das construções genéticas com
p416GPD e 426GPD. 91
Tabela II.18 – Concentrações de fármacos testadas nos testes de
susceptibilidade realizados pela técnica de spots. 94
Tabela II.19. – Sequência dos primes e condições de PCR utilizados para
amplificação dos genes para construção de fusões génicas com GFP. 96
Tabela II.20 – Reacções de restrição para confirmação das fusões pfcrt_GFP e
pfmdr1 _GFP. 99
Tabela III.1 – Antimaláricos testados em cada uma das áreas endémicas. 107
Tabela III.2 – Genótipos, dos clones de referência utilizados como controlos,
relativamente aos genes pfcrt e pfmdr1. 109
Tabela III.3 – Frequências alélicas nos isolados da RDSTP. 112
Tabela III.4 – Isolados agrupados de acordo com a susceptibilidade à
cloroquina e os alelos dos genes pfcrt e pfmdr1. 113
Tabela III.5 – Resultados dos testes de susceptibilidade in vitro aos diferentes
antimaláricos. 116
Tabela III.6 – Resultados da análise dos polimorfismos, relativos aos codões em estudo, bem como as frequências alélicas, nos isolados da República de
Angola. 117
Tabela III.7 – Isolados agrupados de acordo com a susceptibilidade a cada
fármaco e os alelos dos genes pfcrt e pfmdr1. 119
Tabela III.8 – Resultados dos testes de susceptibilidade in vitro aos diferentes
antimaláricos. 125
Tabela III.9 – Sumário dos resultados da análise dos polimorfismos, relativos aos codões em estudo, bem como as frequências alélicas, nos isolados da
Tailândia. 126
Tabela III.10 – Isolados agrupados de acordo com a susceptibilidade a cada
fármaco e os alelos dos genes pfcrt e pfmdr1. 128
Tabela III.11 – Resultados dos testes de susceptibilidade in vitro aos
Tabela III.12 – Sumário dos resultados da análise dos polimorfismos, relativos aos codões em estudo, bem como as frequências alélicas, nos isolados do
Brasil. 136
Tabela III.13 – Isolados agrupados de acordo com a susceptibilidade in vitro a
cada fármaco e os alelos dos genes pfcrt e pfmdr1. 138
Tabela III.14 – Percentagem de resistência verificada nas áreas endémicas
estudadas, bem como na totalidade das zonas estudadas. 146
Tabela III.15 – Sumário dos resultados da análise dos polimorfismos, relativos aos códões em estudo, bem como as frequências alélicas, em todos os isolados
incluídos neste estudo. 148
Tabela III.16 – Isolados agrupados de acordo com a susceptibilidade a cada
fármaco e os alelos dos genes pfcrt e pfmdr1. 149
Tabela III.17 – Resumo global da dos resultados obtidos no estudo
epidemiológico da resistência a antimaláricos. 151
Tabela III.18 – Resultados da sequenciação efectuada a algumas amostras com
diferentes proveniências geográficas. 153
Tabela III.19 – Número de isolados agrupados por alelos e por
susceptibilidade à cloroquina. 154
Tabela III.20 – Caracterização fenotipica e genotipica dos parasitas utilizados
nos estudos de expressão. 158
Tabela III.21 – Média dos Cts (triplicados) e respectivos desvios padrão (DP)
nas reacções de amplificação dos genes pfcrt, pfmdr1 e pfβactinaI. 159
Tabela III.22 – Eficiências das reacções de amplificação dos genes alvo (pfcrt
e pfmdr1) e do gene de referência (pfβ actinaI). 160
Tabela III.23 – Média dos Cts (triplicados) e respectivos desvios padrão (DP)
nas reacções de amplificação dos genes pfcrt, pfmdr1 e pfβ actinaI. 160
Tabela III.24 – Declives das regressões lineares associadas às reacções de
amplificação dos genes alvo relativamente ao gene de referência. 161
Tabela III.25 – Resultados obtidos por PCR em tempo real, relativamente ao
gene pfcrt no clone Dd2, ao longo do ciclo eritrocitário. 163
Tabela III.26 – Resultados obtidos por PCR em tempo real, relativamente ao
Tabela III.27 – Resultados obtidos por PCR em tempo real, relativamente ao gene pfcrt, em parasitas com diferentes susceptibilidades aos fármacos na fase
de trofozoíto maduro, no clone Dd2, ao longo do ciclo eritrocitário. 166
Tabela III.28 – Resultados obtidos por PCR em tempo real, relativamente ao gene pfmdr1, em parasitas com diferentes susceptibilidades aos fármacos na de
trofozoíto maduro. 167
Tabela III.29 – Resumo dos resultados da expressão basal dos genes, bem
como os seus IC’s 50 para a cloroquina e mefloquina. 168
Tabela III.30 – Tratamento dos resultados de PCR em tempo real para a
expressão do gene pfcrt em Dd2. 170
Tabela III.31 – Tratamento dos resultados de PCR em tempo real para a
expressão do gene pfmdr1 em Dd2. 170
Tabela III.32 – Tratamento dos resultados de PCR em tempo real para a
expressão do gene pfcrt em 3D7. 171
Tabela III.33 – Tratamento dos resultados de PCR em tempo real para a
expressão do gene pfmdr1 em 3D7. 172
Tabela III.34 – Tratamento dos resultados de PCR em tempo real para a
expressão do gene pfcrt no isolado 611. 173
Tabela III.35 – Tratamento dos resultados de PCR em tempo real para a
expressão do gene pfmdr1 no isolado STP 611. 173
Tabela III.36 – Tratamento dos resultados de PCR em tempo real para a
expressão do gene pfcrt no isolado STP 595. 174
Tabela III.37 – Tratamento dos resultados de PCR em tempo real para a
expressão do gene pfmdr1 no isolado STP 595. 175
Tabela III.38 – Tratamento dos resultados de PCR em tempo real para a
expressão do gene pfcrt no clone Dd2. 177
Tabela III.39 – Tratamento dos resultados de PCR em tempo real para a
expressão do gene pfmdr1 no clone Dd2. 177
Tabela III.40 – Tratamento dos resultados de PCR em tempo real para a
expressão do gene pfcrt no clone 3D7. 178
Tabela III.41 – Tratamento dos resultados de PCR em tempo real para a